Method Article
לחקור m6A ו- m5C Epitranscriptomes על זיהום ויראלי: דוגמה עם HIV
In This Article
Summary
התפקיד של שינויים RNA בזיהומים ויראליים רק מתחיל להיחקר והוא יכול להדגיש מנגנוני אינטראקציה ויראלי מארח חדשים. בעבודה זו, אנו מספקים צינור לחקור m6A ו m5C RNA שינויים בהקשר של זיהומים ויראליים.
Abstract
תפקידם של שינויים ברנ"א בתהליכים ביולוגיים היה במוקד של מספר גדל והולך של מחקרים בשנים האחרונות וידוע כיום כאפיטרנכתובת. בין היתר, N6-מתילדנוסין (m6A) ו 5-מתילציטוסין (m5C) שינויים RNA תוארו על מולקולות mRNA, עשוי להיות תפקיד באפנון תהליכים תאיים. Epitranscriptomics הוא אפוא שכבה חדשה של רגולציה שיש לשקול בנוסף ניתוחים transcriptomic, כפי שהוא יכול גם להיות שונה או מווסת על ידי חשיפה לכל חומר כימי או ביולוגי, כולל זיהומים ויראליים.
כאן, אנו מציגים זרימת עבודה המאפשרת ניתוח של הנוף האפיטרנולוגי התאי והוויראלי המשותף של סימני m6A ו- m5C בו זמנית, בתאים נגועים או לא בנגיף הכשל החיסוני האנושי (HIV). לאחר בידוד mRNA ופיצול מתאים נגועים ב- HIV ולא נגועים, השתמשנו בשני הליכים שונים: MeRIP-Seq, טכניקה מבוססת אימונופרציפיטציה של RNA, כדי להעשיר עבור שברי RNA המכילים את סימן m6A ו- BS-Seq, טכניקה מבוססת המרה ביסולפיט, כדי לזהות את סימן m5C ברזולוציה נוקלאוטיד אחת. עם לכידת מתילציה ספציפית, ספריות RNA מוכנות לרצף בעל תפוקה גבוהה. פיתחנו גם צינור ביואינפורמטיקה ייעודי לזיהוי תמלילים מתילאטים (DM) באופן דיפרנציאלי ללא תלות מפרופיל הביטוי הבזלי שלהם.
בסך הכל, המתודולוגיה מאפשרת לחקור סימנים אפיטרנראציים מרובים בו זמנית ומספקת אטלס של תמלילי DM על זיהום ויראלי או כל שיבוש תאים אחר. גישה זו מציעה הזדמנויות חדשות לזהות שחקנים חדשים ומנגנונים חדשניים של תגובת תאים, כגון גורמים תאיים המקדמים או מגבילים שכפול ויראלי.
Introduction
זה זמן רב ידוע כי מולקולות RNA ניתן לשנות, ויותר מ 150 שינויים לאחר שעתוק תוארו עד כה1. הם מורכבים בנוסף של קבוצות כימיות, בעיקר קבוצות מתיל, כמעט כל מיקום של טבעות pyrimidine ו purine של מולקולות RNA2. שינויים כאלה לאחר התמלול כבר הוכחו להיות מועשר מאוד בRNA העברה (tRNA) ו RNA ריבוזומלי (rRNA) ולאחרונה תוארו על מולקולות mRNA גם כן.
עלייתן של טכנולוגיות חדשות, כגון רצף הדור הבא (NGS), וייצור נוגדנים ספציפיים המזהים שינויים כימיים מוגדרים אפשרו, לראשונה, את חקירת המיקום ואת תדירות השינויים הכימיים הספציפיים ברמה של תמלול. התפתחויות אלה הובילו להבנה טובה יותר של שינויי RNA ומיפוי של מספר שינויים במולקולות mRNA3,4.
בעוד אפיגנטיקה חוקרת את התפקיד של שינויי DNA והיסטון בוויסות התמלול, epitranscriptomics באופן דומה מתמקד שינויים RNA ואת תפקידם. חקירת השינויים האפיטרנכתוביים מספקת הזדמנויות חדשות להדגיש מנגנוני רגולציה חדשניים שעשויים לכוונן מגוון תהליכים תאיים (כלומר, חבית RNA, ייצוא, יציבות ותרגום)5. לכן לא הייתה זו הפתעה גדולה כי מחקרים אחרונים חשפו שינויים אפיטרנכרומיים רבים על זיהום ויראלי הן ב- RNAs הסלולר והן ב- RNAs6 הנגיפי. וירוסים שנחקרו עד כה כוללים הן DNA והן וירוסי RNA; ביניהם, HIV יכול להיחשב כדוגמה חלוצית. בסך הכל, גילוי מתילציה של RNA בהקשר של זיהומים ויראליים עשוי לאפשר חקירה של מנגנונים שטרם נרשמו של ביטוי ויראלי או שכפול, ובכך לספק כלים ויעדים חדשים לשלוט בהם7.
בתחום האפיטרנכתובת של HIV, שינויים של תמלילים ויראליים נחקרו בהרחבה והראו כי נוכחותו של שינוי זה מועילה לשכפול נגיפי8,9,10,11,12,13. עד כה ניתן להשתמש בטכניקות שונות כדי לזהות סימנים אפיטרנראתומים ברמה של תמלול. הטכניקות הנפוצות ביותר לזיהוי m6A מסתמכות על טכניקות משקעים חיסוניות כגון MeRIP-Seq ו- miCLIP. בעוד MeRIP-Seq מסתמך על פיצול RNA כדי ללכוד שברים המכילים שאריות מתילציה, miCLIP מבוסס על הדור של α-m6A נוגדנים ספציפיים מוטציות חתימה על RNA-נוגדן UV crosslinking, ובכך מאפשר מיפוי מדויק יותר.
זיהוי של שינוי m5C יכול להיות מושגת על ידי טכנולוגיות מבוססות נוגדנים הדומות לזיהוי m6A (m5C RIP), או על ידי המרת ביסולפיט או על ידי AZA-IP או על ידי miCLIP. הן Aza-IP והן m5C miCLIP משתמשים במתיל-טרנספראז ספציפי כפיתיון ל-RNA ממוקד תוך כדי עבירת מתילציה של RNA. ב Aza-IP, תאי היעד נחשפים 5-azacytidine, וכתוצאה מכך המבוא אקראי של אתרי 5-אזציטידין אנלוגי ציטדין לתוך RNA המתהווה. ב- miCLIP, מתילטרנספראז NSun2 מהונדס גנטית כדי לספק מוטציה C271A14,15.
בעבודה זו, אנו מתמקדים באפיון הכפול של m6A ו- m5C שינויים בתאים נגועים, באמצעות HIV כמודל. לאחר אופטימיזציה מתודולוגית, פיתחנו זרימת עבודה המשלבת אימונופרציפיטציה של RNA מתילציה (MeRIP) והמרת RNA ביסולפיט (BS), המאפשרת חקירה סימולטנית של סימנים אפיטרנרתיים m6A ו- m5C ברמה רחבת התמלול, הן בהקשרים תאיים והן בהקשרים ויראליים. זרימת עבודה זו יכולה להיות מיושמת על תמציות RNA סלולרי, כמו גם על RNA מבודד מחלקיקים ויראליים.
הגישה של RNA ImmunoPrecipitation (MeRIP)16 המאפשרת חקירה של m6A ברמה הרחבה של התמלול מבוססת היטב ומערך של נוגדנים ספציפיים ל- m6A זמינים מסחרית עד כה17. שיטה זו מורכבת מלכידה סלקטיבית של חלקי RNA המכילים m6A באמצעות נוגדן ספציפי m6A. שני החסרונות העיקריים של טכניקה זו הם (i) הרזולוציה המוגבלת, התלויה מאוד בגודל של שברי RNA ובכך מספקת מיקום ואזור משוערים המכילים את שאריות המתילציה, ו- (ii) כמות החומר הגדולה הדרושה לביצוע הניתוח. בפרוטוקול הממוטב הבא, התקנו את גודל הקטע לכ-150 nt והפחתנו את כמות החומר ההתחלתי מ-10 מיקרוגרם של RNA שנבחר בפולי-A, שהוא כיום הכמות המומלצת של חומר התחלתי, ל-1 מיקרוגרם בלבד של RNA שנבחר בפולי-A. כמו כן, הגדלנו את יעילות ההתאוששות של שברי m6A RNA הקשורים לנוגדנים ספציפיים באמצעות אלוטציה על ידי גישת תחרות עם פפטיד m6A במקום שיטות אלוטציה קונבנציונליות יותר ופחות ספציפיות באמצעות טכניקות מבוססות פנול או פרוטאינאז K. המגבלה העיקרית של עשייה מבוססת RIP זו, עם זאת, נשארת הרזולוציה התת-אופטימלית שאינה מאפשרת זיהוי של נוקלאוטיד A שונה מדויק.
ניתוח של סימן m5C יכול להתבצע כעת באמצעות שתי גישות שונות: שיטה מבוססת RIP עם נוגדנים ספציפיים m5C והמרת RNA ביסולפיט. כמו RIP מציע רק רזולוציה מוגבלת על זיהוי של שאריות מתילציה, השתמשנו המרה bisulfite שיכול להציע רזולוציה נוקלאוטיד יחיד. חשיפה ל-RNA לביסולפיט (BS) מובילה לדיהון ציטוסין, ובכך ממירה את שאריות הציטוסין לאורסיל. לכן, במהלך תגובת ההמרה RNA bisulfite, כל ציטוסין שאינו מתילציה הוא deaminated וומר uracil, בעוד נוכחות של קבוצת מתיל בעמדה 5 של ציטוסין יש השפעה מגנה, מניעת deamination הנגרמת על ידי BS ושמירה על שאריות ציטוסין. הגישה המבוססת על BS מאפשרת זיהוי של נוקלאוטיד שונה m5C ברזולוציית בסיס יחיד ולהערכת תדירות המתילציה של כל תעתיק, ומספקת תובנות על דינמיקת שינוי m5C18. המגבלה העיקרית של טכניקה זו עם זאת מסתמכת על שיעור חיובי שווא של שאריות מתיל. ואכן, המרת BS יעילה ברנ"א חד-גדילי עם שאריות C נגישות. עם זאת, נוכחות של מבנה משני RNA הדוק יכול להסוות את המיקום N5C ולעכב המרת BS, וכתוצאה מכך שאריות C שאינן מתילות שאינן מומרות לשאריות U, ובכך חיוביות שגויות. כדי לעקוף בעיה זו ולמזער את שיעור החיובי השגוי, החלנו 3 סיבובים של דנטורציה ומחזורי המרה bisulfite19. הצגנו גם 2 פקדים בדגימות כדי לאפשר הערכה של יעילות ההמרה של ביסולפיט: אנו ספייק-בבקרות רצף ERCC (רצפים מתוקננים ללא מתילציה וזמינים מסחרית)20, כמו גם RNAs פולי-A-מרוקן כדי להעריך את שיעור ההמרה bisulfite מצד אחד, וכדי לוודא על ידי RT-PCR נוכחות של אתר מתיל ידוע ושמורה היטב, C4447, על RNA ריבוזומלי 28S מצד שני21.
בתחום הוירולוגיה, צימוד שתי שיטות חקירה אפיטרנראולוגיות אלה עם הדור הבא רצף וניתוח ביואינפורמטי מדויק מאפשר מחקר מעמיק של דינמיקה m6A ו- m5C (כלומר, שינוי RNA שינויים זמניים שעלולים להתרחש עם זיהום ויראלי ויכול לחשוף מערך של מטרות חדשות רלוונטיות מבחינה טיפולית לשימוש קליני).
Protocol
1. הכנת תאים
הערה: בהתאם לסוג התא ותוכן ה-RNA שלו, מספר התאים ההתחלתי עשוי להשתנות.
- יש מספיק תאים כדי להשיג בין 200-500 מיקרוגרם של RNA הכולל או 5-7 מיקרוגרם של פולי-A-נבחר RNA. לדוגמה, 50 x 106 תאי SupT1 צריך להניב סביב 500 מיקרוגרם של RNA הכולל עם החילוץ עם ריאגנטים מבוססי פנול, ולכן נדרש עבור כל מצב בודד נבדק.
- הכן את מספר התאים הנדרש על פי העיצוב הניסיוני, וכך על פי מספר התנאים שנבדקו (זיהום, נקודות זמן, טיפול). אם הניסוי נועד להשיג תאים שאינם נגועים ותאים נגועים ב- HIV בשעה 24 שעות לאחר ההדבקה, יש צורך בסך הכל 100 x 106 תאים, מחציתם למצב שאינו נגוע ומחצית למצב נגוע.
2. מיצוי RNA
- מתאים: מיצוי RNA עם פנול-כלורופורם
- עבור כל תנאי, לאסוף תאים (למשל,50 x 106) על ידי צנטריפוגה ולהשליך את supernatant.
- הוסף 5 מ"ל של ריאגנט מבוסס פנול לכל גלולה של 50 x 106 תאים וערבבו על ידי צנרת למעלה ולמטה מספר פעמים.
- דגירה במשך 5 דקות בטמפרטורת החדר כדי לאפשר תמוזה מלאה. ניתן לאחסן תאים lysed ב -80 °C (80 °F) או מעובד ישירות.
הערה: במידת הצורך, תאים ניתן לחלק גם aliquots של 10 x 106 תאים לכל צינור בצינורות 1.5 מ"ל ו lysed ב 1 מ"ל של ריאגנט מבוסס פנול לאחסון נוח יותר. - מוסיפים 1 מ"ל של כלורופורם ומערבבים לפי היפוך.
- דגירה במשך 3 דקות בטמפרטורת החדר.
- צנטריפוגה למשך 15 דקות ב 2,000 x g ו 4 °C (70 °F).
- פיפטה את השלב מימי (שלב עליון) ולעבור לצינור חדש. לסיים את העברת השלב מימי על ידי angling את הצינור ב 45° בזהירות pipetting את הפתרון החוצה.
הערה: כמות השלב מימי עשוי להשתנות בין דגימות אבל צריך להיות קרוב לכמות כלורופורם הוסיף המדגם (כלומר, 1 מ"ל). אין להעביר שכבה בין-פאזית או אורגנית! השימוש בצינורות נעילת פאזה או יוצר פאזה יכול להקל על תהליך זה. - הוסף 0.5 מ"ל של איזופרופנול מולקולרי 100% מולקולרי לשלב מימי.
- דגירה במשך 1 שעות ב -80 °C (80 °F) כדי לאפשר משקעים RNA.
- צנטריפוגה למשך 10 דקות ב 12,000 x g ו 4 °C (5 °F) כדי גלולה RNA זירוז.
- השלך את supernatant ו resuspend כדורי RNA ב 1 מ"ל של 75% ביולוגיה מולקולרית כיתה אתנול. מערבולת בקצרה.
- צנטריפוגה למשך 5 דקות ב 7,500 x g ו 4 °C (7 °F) להשליך את supernatant.
- ייבשו את הכדור במשך 15 דקות.
- resuspend הכדור ב 20 μL של מים ללא RNase ולהעביר צינור חדש.
- לשטוף את הצינור הריק עם 20 μL נוסף של מים כדי למקסם את התאוששות RNA, ובריכה עם נפח 20 μL הראשון.
- לכמת את הרנ"א הכולל עם ספקטרופוטומטר ולהעריך את איכות ה- RNA באמצעות מנתח שברים.
- מחלקיקים ויראליים: מיצוי RNA עם ערכת חילוץ RNA ויראלית מבוססת טור
הערה: מיצוי RNA מחלקיקים ויראליים עם תוצאות ריאגנט מבוסס פנול ברנ"א ויראלי באיכות נמוכה ובספריות באיכות נמוכה יותר. לכן יש להעדיף חילוץ RNA מבוסס טורים. ערכות חילוץ RNA באמצעות RNA נושא עבור elution RNA והתאוששות אינם מתאימים להליך זה ויש להימנע. מאז HIV RNA הוא poly-Adenylated, מיצוי RNA ישיר ללא בידוד mRNA נוסף מספיק כדי להיכנס צינורות MeRIP-Seq ו- BS-Seq. בדרך כלל 1-2 מ"ל של supernatant ויראלי מתאים נגועים אוניברסלית צריך לספק מספיק RNA כדי לבצע את זרימת העבודה כולה.- הכן את המאגר על ידי הוספת 150 μL של בטא-mercaptoethanol ל 30 מ"ל של מאגר תמוגה. ליישב מחדש את מאגר הכביסה הנגיפי על ידי הוספת 96 מ"ל של 100% אתנול.
- לאסוף וירוסים המכילים supernatants וצנטריפוגה כדי גלולה פסולת תא כדי למזער זיהום RNA הסלולר.
- העבר 1 מ"ל של supernatant ויראלי לצינור 15 מ"ל.
- הוסף 3 מ"ל של מאגר RNA ויראלי ל 1 מ"ל של מדגם ויראלי ולערבב על ידי מערבולת.
- העבר 700 μL של מדגם בעמודה, מוכנס בצינור אוסף.
- צנטריפוגה למשך 2 דקות ב-13,000 על גרם בטמפרטורת החדר.
- תזרוק את הזרימה.
- חזור על 3 השלבים הקודמים עד שכל המדגם עובד, ולכן כל ה- RNA נלכד בעמודת המטריצה המבוססת על סיליקה.
- הוסף 500 μL של מאגר כביסה ויראלי לעמודה.
- צנטריפוגה למשך דקה אחת בטמפרטורת החדר. תזרוק את הזרימה.
- הוסף 200 μL של מאגר כביסה ויראלי לעמודה.
- צנטריפוגה למשך דקה אחת בטמפרטורת החדר. תזרוק את הזרימה.
- מקם את העמודה בצינור איסוף ריק.
- צנטריפוגה למשך דקה אחת בטמפרטורת החדר היא להשליך עוד יותר את כל מזהם מאגר הכביסה שנותר.
- בזהירות להעביר את העמודה לתוך צינור 1.5 מ"ל.
- הוסף 20 μL של מים ללא DNase / RNase ישירות למרכז מטריצת העמוד וצנטריפוגה ב 10,000 x g עבור 30 s בטמפרטורת החדר.
- הוסף 10 μL נוספים של מים ללא DNase / RNase ישירות למרכז מטריצת העמודה וצנטריפוגה שוב במשך 30 s.
- לכמת את הרנ"א הכולל עם ספקטרופוטומטר ולהעריך את איכות ה- RNA באמצעות מנתח שברים.
הערה: מיצוי RNA יכול להתבצע בכל שיטה, אם האיכות של RNA שאוחזר גבוהה, עם תקינות RNA / מספר איכות > 9. ניתן לאחסן RNA כולל ב-80 °C (80 °F) עד לעיבוד נוסף.
3. בידוד mRNA על ידי בחירת פולי-A עם אוליגו(dT)25
הערה: בשל נוכחות של RNA ריבוזומלי מתילציה מאוד בתמציות הסלולר, מומלץ מאוד לבודד פולי-A RNA או על ידי דלדול rRNA או מועדף על ידי בחירה חיובית poly-A. שלב זה הוא אופציונלי ויש לבצעו עבור דגימות RNA סלולריות בלבד, כדי להשיג תוצאות רצף ברזולוציה גבוהה יותר. אם מנתחים מתילציה של RNAs ויראליים שאינם פולי-אדנילציה, העדיפו דלדול rRNA במקום בחירת פולי-A או שבסופו של דבר לבצע את הניתוח על RNA הכולל.
-
הכנת חרוזים ללכידת פולי-A
- Resuspend אוליגו(dT)25 מעין חרוזים מגנטיים מלאי על ידי מערבולת עבור >30 s.
- העבר 200 μL של חרוזים מגנטיים לצינור 1.5 מ"ל. הכן את מספר הצינורות עם חרוזים מגנטיים על פי הכמות הכוללת של דגימות RNA לעיבוד.
הערה: צינור אחד עם 200 μL של פתרון מלאי Dynabead מתאים 1 מ ג של חרוזים והוא יכול להכיל מדגם של 75 מיקרוגרם של RNA הכולל. - מניחים את הצינורות על מגנט במשך 1 דקות ולהשליך את supernatant. הסר את הצינורות מהמגנט.
- הוסף 1 מ"ל של חוצץ כריכה (20 מ"מ טריס-HCl, pH 7.5, 1.0 M LiCl, 2 מ"מ EDTA) ו resuspend על ידי מערבולת. מניחים את הצינורות על המגנט במשך 1 דקות ולהשליך את supernatant. הסר את הצינורות מהמגנט. חוזר.
- יש לתתכוד מחדש את החרוזים המגנטיים שנשטפו ב-100 מיקרו-אל של מאגר כריכה.
-
הכנה כוללת לרנ"א
- לדלל את הרנ"א הכולל בריכוז סופי של 0.75 מיקרוגרם / μL עם מים ללא RNase, אשר מתאים 75 מיקרוגרם / 100 μL.
הערה: אם RNA נמצא בריכוז נמוך יותר, המשך כמתואר להלן מבלי לשנות את אמצעי האחסון. - Aliquot הרנ"א הכולל בצינורות מרובים על ידי חלוקת 100 μL של דגימת RNA לכל צינור.
- הוסף 100 μL של מאגר איגוד לכל דגימת RNA.
- מחממים את הרנ"א הכולל ל-65 מעלות צלזיוס למשך 2 דקות כדי לשבש מבנים משניים.
- מניחים מיד על הקרח עד שהם מוכנים להמשיך לשלב הבא.
הערה: זמן הדגירה עשוי להשתנות בהתאם למספר הדגימות שיש לעבד אך לא יעלה על 1 שעות כדי למנוע כל השפלה RNA.
- לדלל את הרנ"א הכולל בריכוז סופי של 0.75 מיקרוגרם / μL עם מים ללא RNase, אשר מתאים 75 מיקרוגרם / 100 μL.
-
בחירת פולי-איי
- לכל צינור RNA (החל מהשלב 3.2), הוסיפו 100 מיקרו-אל של חרוזים מגנטיים שטופים (החל מהשלב 3.1).
- מערבבים היטב על ידי צנרת למעלה ולמטה ומאפשרים כריכה על גלגל מסתובב בטמפרטורת החדר במשך 15 דקות.
- פתחו את כל הצינורות, הניחו אותם על המגנט למשך דקה אחת, והוציאו בזהירות את כל הסופר-טבעי.
- לשחזר את supernatant בצינור חדש ולשמור בצד לסיבוב שני של לכידת RNA (שלב 3.3.14), על מנת לשפר את ההתאוששות הסופית poly-A.
- הסר את הצינור מן המגנט ולהוסיף 200 μL של חוצץ כביסה (10 mM Tris-HCl, pH 7.5, 0.15 M LiCl, 1 mM EDTA). מערבבים על ידי צנרת בזהירות 4 עד 5 פעמים.
- מניחים את הצינור על המגנט למשך דקה אחת ומשליכים את הסופר-נט.
- חזור על שלב הכביסה פעם אחת (חזור על שלבים 3.3.5 ו- 3.3.6).
- הוסף 20 μL של קר כקרח 10 mM Tris-HCl כדי לחמוק פולי-A RNA מן החרוזים.
- דגירה ב 80 °C (70 °F) במשך 2 דקות.
- הנח את הצינור על המגנט והעבר במהירות את ה- supernatant המכיל את ה- poly-A RNA לצינור חדש ללא RNase. הנח את הצינור על קרח.
- חזור על שלב ההעמקה (שלבים 3.3.8 עד 3.3.10) כדי להגדיל את התשואה.
- לשטוף את אותם חרוזים פעם אחת עם 200 μL של חוצץ כביסה. מערבבים על ידי צנרת בזהירות 4 עד 5 פעמים.
- מניחים על המגנט במשך דקה אחת ומשליכים את חיץ הכביסה.
- הוסף את הזרימה מהשלב 3.3.4 לחרוזים וחזר על ההליך מחייב לאלוטיון (שלבים 3.3.2 עד 3.3.10). שמור את eluates RNA בצינורות נפרדים לעת עתה.
הערה: באופן אופציונלי, שמור שוב את המקבילה supernatant לשלב 3.3.4 בצינור חדש כפי שהוא יכול לשמש כפקד. בסוף ההליך, לטהר ולרכז את הרנ"א על ידי משקעים אתנול או עם שיטת בחירה מבוססת טור (כלומר, RNA נקי ורכז). מדגם זה מתאים לדגימת RNA מרוקנת מפולי-A וניתן להשתמש בה כפקד להמרת ביסולפיט (שלב 8.2.2). - לכמת את הרנ"א הנמהר עם ספקטרופוטומטר ולשמור על aliquot 2 μL כדי להעריך עוד יותר את איכות ה- RNA עם מנתח שברים.
הערה: פולי-A RNA ניתן לאחסן ב -80 °C (80 °F) עד הצורך.
4. זרימת עבודה של RNA
- חלקו את דגימות ה-פולי-A RNA התאיות (mRNA) ודגימות הרנ"א הנגיפיות ל-2 עליקוטים, המוקדשים לצינור הניתוח האפיטרנכתובי המתאים:
(i) 5 מיקרוגרם של mRNA סלולרי או 1 מיקרוגרם של RNA ויראלי עבור MeRIP-Seq ובקרות קלט (עבור לשלבים 5 עד 7, ושלב 9).
(ii) 1 מיקרוגרם של mRNA סלולרי או 500 ננוגרם של RNA ויראלי עבור BS-Seq (עבור לשלבים 8 ו -9).
5. פיצול RNA
הערה: פיצול RNA מתבצע עם ריאגנט פיצול RNA ומיועד לדגימות MeRIP-Seq ולשלוט ב- RNA. זהו צעד חשוב מאוד הדורש אופטימיזציה זהירה על מנת להשיג שברים הנעים בין 100-200 nt.
- חלק את הנפח הכולל של mRNA לצינורות PCR 0.2 מ"ל עם 18 μL של mRNA / צינור.
הערה: עבוד במהירות. אין לעבוד עם יותר מ-8 דגימות בכל פעם כדי לקבל תוצאות ניתנות לשחזור. הגדלת עוצמת הקול לא תבטיח פיצול אחיד ושחזורי. - לחמם תרמוציקלר ב 70 °C (70 °F).
- הוסף 2 μL של ריאגנט פיצול בקצה של כל צינור PCR.
- סגור את הצינור ולהסתובב למטה (כך ריאגנט מקבל במגע עם RNA באותו זמן עבור 8 צינורות).
- לדגור על הדגימות 15 דקות ב 70 °C (70 °F) תרמוציקלר מחומם מראש.
- ברגע הדגירה נגמרת, להוסיף במהירות 2 μL של פתרון עצור בכל צינור.
- תסתובבו ותנו לשבת על הקרח עד שיהיו מוכנים להמשיך לשלב הבא.
הערה: זמן הדגירה עשוי להשתנות בהתאם למספר הדגימות שיש לעבד אך לא יעלה על 1 שעות כדי למנוע כל השפלה RNA. - חזור על ההליך עבור כל הדגימות (אם יש יותר מ 8 aliquots).
- איחדו את הצינורות והמשיכו לטיהור RNA עם ערכת RNA נקייה ורכזת (שלב 6) או כל ערכה מותאמת אישית המבוססת על עמודות כדי להיפטר מהמאגרים ולשחזר RNA מקוטע נקי במים.
6. טיהור RNA
הערה: שלב זה יכול להתבצע על ידי משקעים אתנול או עם כל סוג של שיטת טיהור וריכוז RNA מבוססת עמודה (כלומר, RNA נקי ורכז).
- יש לפנות או לתתכון מחדש את הרנ"א המטוהר בנפח כולל של 50-75 מיקרול של מים נטולי DNase/RNase.
הערה: אם נעשה שימוש בשיטה מבוססת עמודה, מומלץ מאוד לשני סבבי עילה כדי להבטיח שחזור מרבי. - לכמת את mRNA מקוטע מטוהר עם ספקטרופוטומטר ולהעריך את איכות ה- RNA עם מנתח שברים.
- שמור 100 ננוגרם של mRNA מקוטע כבקרת קלט להכנת הספרייה ורצף (עבור לשלב 9). ניתן להשתמש ב- mRNA המפוצל הנותר (מינימום 2.5 מיקרוגרם) עבור MeRIP (עבור לשלב 7.2).
7. מריפ
הערה: נדרש מינימום של 2.5 מיקרוגרם של mRNA מקוטע עבור כל אימונופרציפיטציה (IP), בין אם באמצעות נוגדן אנטי-m6A ספציפי (מצב בדיקה) או באמצעות נוגדן אנטי-IgG (שליטה שלילית).
- הכנת חרוז מגנטי לאימונופרציפיטציה
- עבור כל מדגם, הכן 4 מ"ל של 1x מאגר IP בצינור חרוטי חדש על ידי דילול 800 μL של מאגר MRNA IP 5x (50 mM Tris-HCl pH 7.4, 750 mM NaCl, 0.5% CA-630 Igepal ומים ללא נוקלאז) עם 3.2 מ"ל של מים ללא נוקלאז.
הערה: דרושות לפחות 2 תגובות (בדיקה אחת ובקרת IgG אחת). - הנח את הצינור על קרח.
- סמן את המספר המתאים של צינורות מיקרוצנטריפוגה 1.5 מ"ל עבור מספר תגובות ה- IP הרצויות:
n צינורות (בדיקה) לנוגדן נגד m6A.
n צינורות (שליטה שלילית) עבור עכבר רגיל IgG. - resuspend החרוזים המגנטיים (למשל, חלבון ChIP מגנה A/G ) על ידי היפוך מערבולת. אין לראות גושי חרוזים.
- עבור כל תגובה מתוכננת, להעביר 25 μL של חרוזים מגנטיים לצינור microcentrifuge.
- הוסף פי עשרה יותר 1x מאגר IP (מהשלב 7.1.1) ביחס לנפח המקורי של חרוזים בשימוש (כלומר, 250 μL של 1x מאגר IP לכל 25 μL של חרוזים מגנטיים).
- מערבבים את החרוזים על ידי צנרת בעדינות למעלה ולמטה מספר פעמים לחידוש מלא.
- מניחים את הצינור על המפריד המגנטי למשך דקה אחת.
- הסר והשלך את supernatant, הקפדה לא לשאוף כל חרוזים מגנטיים. הסר את הצינור מהמגנט.
- חזור על שלב הכביסה (שלבים 7.1.6 עד 7.1.9).
- resuspend החרוזים ב 100 μL של 1x מאגר IP לכל 25 μL של נפח מקורי של חרוזים מגנטיים.
- הוסף 5 μL של נוגדן (1 מיקרוגרם / μL) לכל 25 μL של נפח מקורי של חרוזים מגנטיים.
n צינורות (בדיקה) עם נוגדן נגד m6A (שיבוט 17-3-4-1) [1 מיקרוגרם/μL].
n צינורות (שליטה שלילית) עם IgG עכבר רגיל (1 מיקרוגרם / μL). - דגירה על הגלגל המסתובב במשך 30 דקות בטמפרטורת החדר כדי לאפשר הטיות של הנוגדנים עם החרוזים המגנטיים.
- מניחים את הצינור על המפריד המגנטי למשך דקה אחת. זרוק את סופר-טבעי. הסר את הצינור מן המגנט ו resuspend תערובת נוגדנים-חרוזים ב 100 μL של 1x IP Buffer.
- עבור כל מדגם, הכן 4 מ"ל של 1x מאגר IP בצינור חרוטי חדש על ידי דילול 800 μL של מאגר MRNA IP 5x (50 mM Tris-HCl pH 7.4, 750 mM NaCl, 0.5% CA-630 Igepal ומים ללא נוקלאז) עם 3.2 מ"ל של מים ללא נוקלאז.
- RNA Immunoprecipitation (RIP)
- הכן 500 μL של תערובת תגובת RIP עבור כל מדגם mRNA 2.5 מיקרוגרם כדלקמן: 2.5 מיקרוגרם ב 100 μL של RNA מקוטע (בשלב 6.12); 295 μL של מים ללא נוקלאז; 5 μL של 40 מעכבי RNase U/μL; ו- 100 μL של מאגר IP 5x.
- הוסף 500 μL של תערובת תגובת RIP לכל תערובת נוגדנים-חרוזים (~ 100 μL מ שלב 7.1.14). מערבבים על ידי צנרת עדינה מספר פעמים כדי להצמיד מחדש את החרוזים לחלוטין. מניחים על קרח.
- לדגור את כל צינורות RIP על גלגל מסתובב במשך 2 שעות ב 4 °C (70 °F).
- צנטריפוגות תגובות MeRIP בקצרה כדי לסובב את טיפות נוזלים מן המכסה ואת צדי הצינור. מניחים את הצינורות על מפריד מגנטי במשך 1 דקות.
- העבר את supernatant בצינור צנטריפוגה חדש, נזהר לא להפריע חרוזים מגנטיים .
הערה: ניתן לשמור על Flowthrough כפקד כדי לאמת את יעילות RIP (עבור לשלב 7.3.9). - הסר צינורות מהמגנט. לשטוף את החרוזים על ידי הוספת 500 μL של חיץ IP 1x קר. מערבבים את החרוזים על ידי צנרת עדינה מספר פעמים כדי resuspend החרוזים לחלוטין.
- מניחים את הצינורות על מפריד מגנטי במשך 1 דקות ולהשליך supernatant.
- חזור על הליך הכביסה (שלבים 7.2.6-7.2.7) פעמיים עבור סך של 3 כביסות.
- מניחים את הצינורות על קרח ומיד ממשיכים לתהלייה.
- אלוטיון
- הכן פתרון 20 mM m6A על ידי המסת 10 מ"ג של N6-Methyladenosine, 5′-מונופוספט מלח נתרן (m6A) ב 1.3 מ"ל של מים ללא נוקלאז. הכן 150 עליקוטים μL ולאחסן ב -20 °C (50 °F).
- עבור כל מדגם (בדיקה ובקרות): הכן 225 μL של מאגר אלוטיון על ידי ערבוב הרכיבים הבאים: 45 μL של 5x IP Buffer, 75 μL של 20 mM m6A, 3.5 μL של מעכב RNase 40U / μL, ו 101.5 μL של מים ללא נוקלאז.
- הוסף 100 μL של מאגר אלוטיון (מהשלב 7.3.2) אל החרוזים (מהשלב 7.2.9). מערבבים על ידי צנרת עדינה מספר פעמים כדי resuspend חרוזים לחלוטין.
- לדגור את כל הצינורות במשך 1 שעות עם רעד מתמשך על רוקר ב 4 °C (50 °F).
- צנטריפוגות תגובות RIP בקצרה כדי לסובב טיפות נוזליות מן המכסה ואת צדי הצינור. מניחים את הצינורות על מפריד מגנטי במשך 1 דקות.
- העבר את supernatant המכיל שברי RNA אלים לצינור microcentrifuge 1.5 מ"ל חדש. היזהר לא לשאוף את החרוזים, כפי שהוא יגביר את רעשי הרקע.
- חזור על שלבי ההעפרה (7.3.3 עד 7.3.6) על-ידי הוספת 100 מיקרו-אל של חוצץ אילוטיון, דגירה של שעה אחת ב-4 °C (4 °F) ואיסוף האלאט לאחר ההפרדה המגנטית.
- שלב את כל eluates מאותה מדגם (נפח elution הכולל צריך להיות 200 μL).
- טהרו את ה-RNA הנמהר ואת הזרימה (אופציונלי, החל מהשלב 7.2.5) על ידי משקעים אתנול או בשיטת בחירה מבוססת טורים (כלומר, RNA נקי ורכז).
- להעריך את כמות ה- RNA ואת איכות הזרימה ודגימות eluted עם מנתח שברים באמצעות ערכת זיהוי רגישות גבוהה. אם איכות ה- RNA משביעת רצון, המשך להכנת הספרייה ולרצף תפוקה גבוהה (שלב 9).
הערה: כמות הרנ"א שאוחזרה ב- MeRIP נמוכה מאוד, ובהכרח דורשת ערכות לזיהוי רגישות גבוהה כדי להבטיח כימות. אם אין ביואנלייזר זמין, ניתן להמשיך בעיוורון להכנת הספרייה.
8. המרת RNA ביסולפיט
- הכנה לבקרה וריאגנט
- בקרת ספייק-אין לערבב ERCC: הוסף תערובת ERCC בהתאם להוראות היצרן, הממליצות על תוספת של 0.5 μL של תערובת ERCC לא מדוללת ל- 500 ננוגרם של mRNA. פקד זה יכול לעזור להעריך את היעילות של המרת ביסולפיט.
- ספייק פולי-A-מרוקן RNA (מ שלב 3.3.14) ביחס 1/1000 (כלומר, 500 pg של פולי-A-מרוקן RNA עבור 500 ננוגרם של mRNA). מדגם זה מועשר ברנ"א ריבוזומלי ולכן צריך להכיל את 28S rRNA, שליטה חיובית להמרת ביסולפיט.
הערה: סה"כ RNA יכול לשמש גם כשליטה חיובית במקום RNA מרוקן פולי-A. - בצע המרת ביסולפיט עם ערכת מתילציה RNA (למשל, זימו EZ).
- חוצץ שטיפת RNA: יש להוסיף 48 מ"ל של 100% אתנול (או 52 מ"ל של 95% אתנול) ל-12 מ"ל של תרכיז מאגר שטיפת RNA לפני השימוש.
- המרת ביסולפיט
הערה: המרת Bisulfite בוצעה עם ערכת המרה RNA bisulfite זמין מסחרית בעקבות הליך היצרן כאמור להלן.- בצינורות PCR של 0.2 מ"ל, הוסף 1000 ננוגרם של mRNA (או בין 300 ל-1000 ננוגרם). הוסף פקדי ספייק-אין: 1 μL של תערובת ERCC (שלב 8.1.1) ו- 1000 pg של RNA מרוקן פולי-A (שלב 8.1.2). נפח מלא עד 20 μL עם מים ללא DNase /RNase.
- הוסף 130 μL של ריאגנט המרת RNA לכל דגימת RNA של 20 μL.
- מערבבים את הדגימה על ידי צנרת למעלה ולמטה.
- ספין למטה בקצרה כדי להבטיח שאין טיפות במכסה או בצדדים של הצינור.
- מניחים את צינורות PCR באופניים תרמיים ולבצע את השלבים הבאים: denaturation ב 70 °C (5 דקות); המרה ב 54 °C (54 °F) במשך 45 דקות; חזור על שלבי דנטורציה והמרה עבור סך של 3 מחזורים; ולאחר מכן להחזיק ב 4 °C ללא הגבלת זמן.
הערה: שלושה מחזורים של denaturation והמרת bisulfite להבטיח המרה ביסולפיט מלא של המדגם. ניתן לאחסן דגימות ב- -80 °C (80 °F) או לעבד אותן ישירות. - המשך עם ביטול ההסתה בעמודה. מקם עמודה בצינור אוסף ריק והוסף 250 μL של מאגר איגוד RNA לעמודה.
- טען את המדגם (~ 150 μL משלב 8.2.5) לעמודה המכילה את מאגר איגוד ה- RNA וערבוב על-ידי צנרת למעלה ולמטה.
- הוסף 400 μL של 95-100% אתנול לתערובת אגף כריכת RNA מדגם בעמודה. סגור את המכסה ומיד מערבבים על-ידי היפוך העמודה מספר פעמים.
- צנטריפוגה במלוא המהירות (≥ 10,000 x g) עבור 30 s. תזרוק את הזרימה.
- הוסף 200 μL של מאגר שטיפת RNA לעמודה וצנטריפוגה במהירות מלאה עבור 30 s.
- הוסף 200 μL של מאגר Desulphonation RNA לעמודה ודגר בטמפרטורת החדר במשך 30 דקות. לאחר הדגירה, צנטריפוגה במלוא המהירות במשך 30 s. תזרוק את הזרימה.
- הוסף 400 μL של מאגר שטיפת RNA לעמודה וצנטריפוגה במהירות מלאה עבור 30 s. חזור על שלב הכביסה עם 400 μL נוסף של מאגר שטיפת RNA. תזרוק את הזרימה.
- צנטריפוגה העמוד בצינור האוסף המרוקן במלוא המהירות במשך 2 דקות. העבר את העמודה לצינור ללא RNase.
- הוסיפו ≥ 10 מיקרו-אל של מים נטולי DNase/RNase ישירות למטריצת העמודות, ודגרו במשך דקה אחת בטמפרטורת החדר. צנטריפוגה במלוא המהירות ל-30 שנה.
הערה: אנחנו בדרך כלל לברוח בנפח של 20 μL. RNA eluted ניתן להשתמש באופן מיידי או מאוחסן ב -20 °C (50 °F) עד 3 חודשים. לאחסון לטווח ארוך, יש לשמור על -80 °C (80 °F). - להוציא 2.5 μL להערכת מנתח קטעים של איכות RNA וכמות ולהמשיך להכנת הספרייה ורצף תפוקה גבוהה (שלב 9).
- קח 4 μL של RNA מומר לשליטת המרה bisulfite של יעילות (שלב 8.3).
- בקרת המרה ביסולפיט על ידי RT-PCR
הערה: שלב זה מבטיח כי המרת bisulfite הצליחה לפני שתמשיך לרצף . RNA ריבוזומלי 28S מהומו ספיינס ישמש כבקרה חיובית לניתוח מתילציה של RNA, שכן שאריות C בעמדה 4447 (גישה של GenBank # NR_003287) תוארה כ- 100% מתילציה.
רצפי פריימר:
H 28SF primer: 5'-GGGGTTTTAYGATTTTTGATTTTTTGG-3'
H 28SR פריימר: 5'-CCAACTCACRTTCTATTAAATAAAAAC-3'- הכן תמלול הפוך (RT) תערובת תגובה באמצעות ערכת תמלול הפוך cDNA בקיבולת גבוהה. להפשיר את רכיבי הערכה על קרח ולהכין את תערובת מאסטר RT על קרח כדלקמן:
4 μL של RNA מומר ביסולפיט (החל בשלב 8.2.14):
2 מיקרו-אל של מאגר 10xRT
0.8 μL של 25x dNTP מיקס [100 מ"מ]
2 מיקרו-אל של פריימרים אקראיים RT 10x
1 μL של תמלול הפוך של ריבוי סופרים
1 μL של מעכב RNase
9.2 μL של H2O ללא נוקלאז
הערה: כל תגובת RT צריכה להכיל נפח סופי של 20 μL בצינורות PCR של 0.2 מ"ל. - שים את הצינורות ברכיב האופניים התרמי עם תוכנית RT הבאה: 25 °C (70 °F) למשך 10 דקות; 37 °C (77 °F) למשך 120 דקות; 85 °C (5 דקות; ואז ב 4 °C ללא הגבלת זמן.
- הכן את תגובת ה-PCR כדי להגביר במיוחד את rRNA 28S עם אנזים הגהה PCR. מפשירים את רכיבי הערכה על קרח, מערבולת בעדינות וצנטריפוגה קצרה. הכן את תערובת מאסטר PCR על קרח או על מחזיק צלחת מתכת קר כקרח כדלקמן:
0.6 μL של 10 מיקרומטר H 28SF פריימר
0.6 μL של 10 מיקרומטר H 28SF פריימר
6.5 μL של תבנית cDNA
22.5 μL של תמהיל מאסטר פולימראז DNA
הערה: כל תגובת PCR צריכה להכיל נפח סופי של 20 μL בצינורות PCR של 0.2 מ"ל. - שים את הצינורות ברכיב האופניים התרמי עם תוכנית PCR הבאה: denaturation הראשוני ב 95 °C במשך 5 דקות; 45 מחזורים של denaturation (95 °C (15 °C) עבור 15 s), חישול (57 °C (30 °C) והארכות (72 °C (15 °C), התארכות סופית ב 72 °C במשך 10 דקות, ולאחר מכן מחזיק ב 4 °C ללא הגבלת זמן.
- הפעל 10 μL של התגובה על ג'ל אגרוז 2%. גודל הרצועה הצפוי הוא 130 - 200 bp.
- הכן תמלול הפוך (RT) תערובת תגובה באמצעות ערכת תמלול הפוך cDNA בקיבולת גבוהה. להפשיר את רכיבי הערכה על קרח ולהכין את תערובת מאסטר RT על קרח כדלקמן:
- רצף של מוצרי PCR
- לטהר את מוצרי PCR עם שיטה מבוססת עמודה של בחירה כדי להסיר אנזימים ושאריות dNTP ולחמוק ה- DNA המוגבר לפחות 20 μL של מים ללא DNase / RNase.
- לכמת את הדנ"א המטוהר עם ספקטרופוטומטר.
- תגובת רצף
- השתמש ב- 40 ננוגרם של תגובת מוצר/רצף PCR.
- רצף בשני הכיוונים עם פריימרים H 28SF ו- H 28SR.
- ישר את הרצפים עם הרצף הלא-מומר הידוע (28S ribosomal N5 (RNA28SN5). בדוק את נוכחותם של שאריות C במיקום C4447, ואת שאריות T במקום C במקום במקום אחר.
9. הכנת הספרייה ורצף תפוקה גבוהה
- הכן ספריות לרצף באמצעות ערכות mRNA (לדוגמה, Illumina TruSeq Stranded), הפעלת הפרוטוקול בשלב Elute-Prime-Fragment והוראות היצרן.
- עם זאת, עבור קלט RNA-Seq ו MeRIP-Seq דגימות, לדגור את הדגימות ב 80 °C (80 °F) במשך 2 דקות רק פריים אבל לא עוד לפצל אותם.
- בצע רצף באמצעות פלטפורמות Illumina. תגובות רצף יכולות להתבצע על פי העדפות ועיצוב ניסיוני, קצוות בודדים או מצמידים, עם אורך מינימלי של 100 nt.
10. ניתוחים ביואינפורמטיים
- עיבוד נתונים m6A
- הפעל FASTQC24 כדי להעריך את איכות הקריאה ב- m6A והקלט קבצי FASTQ מרצף.
- הפעל את Atropos25 כדי לקצץ רצפי קצה ומתאם באיכות נמוכה מהקריאות. הגדר את הפרמטרים הבאים בהפעלת Atropos.
- הסר את רצפי המתאם הבאים: AGATCGGAAGAG, CTCTCCGATCT, AACACTCTTTCCCT, AGATCGGAAGAGCG, AGGGAAAGAGTGTT, CGCTTCCGATCT.
- השתמש בקיצוץ האיכותי הבא של Phred: 5, לגיזום קצוות באיכות נמוכה כפי שצוין על-ידי היצרן (https://support.illumina.com/downloads/illumina-adapter-sequences-document-1000000002694.html).
- השתמש באורך הקריאה המינימלי הבא לאחר החיתוך: 25 זוגות בסיס.
- מזג את הגנום האנושי GRh38 ו- HIV [pNL4-3Env-GFP משולב] בפורמט FASTA.
- אינדקס ההפניה הממוזגת עם HISAT226.
- הפעל את HISAT2 בקריאות חתוכות כדי ליישר את ההפניה הכלולה באינדקס. השתמש בפרמטרים של HISAT המוגדרים כברירת מחדל.
- מיין וציין אינדקס של הקריאות המיושרות באמצעות SAMtools27.
- הפעל את הסטטיסטיקה של SAMtools ואת Qualimap 228, לבדיקת איכות לאחר היישור של הספריות המרוצפות.
- לחלופין, לאסוף ולסכם מדדי איכות מהשלב הקודם עם multiQC29.
- לגנום HIV יש רצפי 634 bp הומולוגיים ב- LTR 5 ' ו- LTR 3 ': יישר מחדש קריאות מרובות מיפויים מ- 5 ' LTR לאזור LTR המתאים 3 ' עם SAMtools.
- על מנת לזהות את פסגות m6A, הפעל את תוכנת הקריאה שיא MACS230 (v 2.1.2). בחר בקפידה פרמטרים פועלים MACS2, על מנת להבטיח תפקוד נכון על נתוני RNA-Seq כמו שיא שיחות יכול להיות מושפע על ידי רמת ביטוי גנים, ו exons קצר עשוי להיות misיקרא פסגות. לפיכך, יש להחסיר אות קלט מאות m6A, ללא החלקה המופעלת באופן שגרתי על-ידי MACS2 לנתונים מבוססי DNA. החל את הפרמטרים הבאים על פקודת המשנה 'callpeak' מ- MACS2:
-keep-dup auto (שולט בהתנהגות MACS2 כלפי קריאות כפולות, 'אוטומטי' מאפשר ל- MACS לחשב את המספר המרבי של קריאות באותו מיקום בדיוק בהתבסס על התפלגות בינומית באמצעות 1e-5 כניתוק ערך p)
-g 2.7e9 (גודל הגנום האנושי ב- bp)
-q 0.01 (ניתוק מינימלי של FDR כדי לקרוא לשיאים משמעותיים)
-נודל (לעקוף את בניית מודל ההזזה, המותאם לניסויי ChIP-Seq)
-slocal 0
-llocal 0 (הגדרת פרמטר זה ואת הפרמטר הקודם ל- 0 מאפשרת ל- MACS2 להחסיר ישירות, ללא החלקה, את קריאות הקלט מה- m6A קורא)
-extsize 100 (אורך ממוצע של שברים ב- bp)
-B - הפעל את שיא דיפרנציאל קורא תת הפקודה של MACS2, 'bdgdiff' כדי להשוות דגימות נגועות לעומת לא נגועות. 'bdgdiff' לוקח כמו קלט קבצי bedGraph שנוצרו על ידי 'callpeak' בשלב הקודם. עבור כל נקודת זמן, להפעיל את ההשוואה של דגימות נגועות לעומת לא נגוע עם 'bdgdiff', חיסור אות הקלט המתאים מן האות m6A ומספק את הפרמטרים הנוספים: -g 60 -l 120.
- עיבוד נתונים m5C
- הפעל את Cutadapt31 כדי לחתוך רצפי מתאם מהקריאות הגולמיות, עם הפרמטרים הבאים:
מתאם "AGATCGGAAGAGCACGTCTGAAC"
-אורך מינימלי=25. - המשלים לאחור את הקריאות הקצוצות באמצעות seqkit32, כאשר פרוטוקול הרצף מפיק קריאות מהגדיל ההפוך.
- הפעל FastQC כדי לבחון את איכות הקריאה.
- מיזוג GRh38 גנום אנושי ו- HIV [pNL4-3Env-GFP משולב] הפניה בפורמט FASTA.
- אינדקס ההפניה הממוזגת עם meRanGh היישום מחבילת meRanTK33.
- ישר עם meRanGh עם הפרמטרים הבאים:
-UN מאפשר לכתוב קריאות לא ממופות לקבצי פלט
-MM המאפשר כתיבת קריאות מרובות ממופות לקובץ פלט
- bg לפלט במיטהגרף
-mbgc 10 מסנן דיווח אזור לפי כיסוי (לפחות 10 קריאות של כיסוי) - לגנום HIV יש רצפי 634 bp הומולוגיים ב- LTR 5 ' ו- LTR 3 ': יישר מחדש קריאות מרובות מיפויים מ- 5 ' LTR לאזור LTR המתאים 3 ' עם SAMtools.
- הפעל שיחות מתילציה באמצעות הכלי meRanCall, המסופק על ידי meRanTK, עם הפרמטרים הבאים:
-rl = 126, אורך קריאה
-ei = 0.1, מרווח זמן לשגיאות עבור חישוב ערך p-value של קצב מתילציה
-cr = 0.99, המרה צפויה - הפעל את estimateSizeFactors.pl השירות של MeRanTK להערכת גורמי הגודל של כל מדגם. גורמי הגודל ישמשו כפרמטרים בשלב הבא.
- הפעל MeRanCompare לניתוח מתילציה דיפרנציאלית של לא נגוע לעומת נגוע על פני נקודות זמן 12, 24, ו 36h. הפרמטרים הבאים מוחלים: ערך משמעות של .01 כסף מינימלי לדיווח וגורמי גודל מהשלב הקודם.
- הפעל את Cutadapt31 כדי לחתוך רצפי מתאם מהקריאות הגולמיות, עם הפרמטרים הבאים:
תוצאות
זרימת עבודה זו הוכיחה שימושי לחקור את התפקיד של m6A ו m5C מתילציה בהקשר של זיהום HIV. בשביל זה, השתמשנו במודל קו תאים CD4+ T (SupT1) שאנחנו גם להדביק עם HIV או לא מטופל. התחלנו את זרימת העבודה עם 50 מיליון תאים לכל תנאי וקיבלנו בממוצע 500 מיקרוגרם של RNA כולל עם מספר איכות RNA של 10 (איור 1A-B). בבחירת פולי-A חזרנו בין 10 ל-12 מיקרוגרם של mRNA לכל תנאי (המייצגים כ-2% מסך הרנ"א) (איור 1B). בשלב זה, השתמשנו 5 מיקרוגרם של פולי-A-נבחר RNA עבור צינור MeRIP-Seq ו 1 מיקרוגרם עבור צינור BS-Seq. מאז HIV RNA הוא poly-adenylated, אין צורך בפעולה נוספת וניתן ליישם ישירות הליכי MeRIP-Seq ו- BS-Seq.
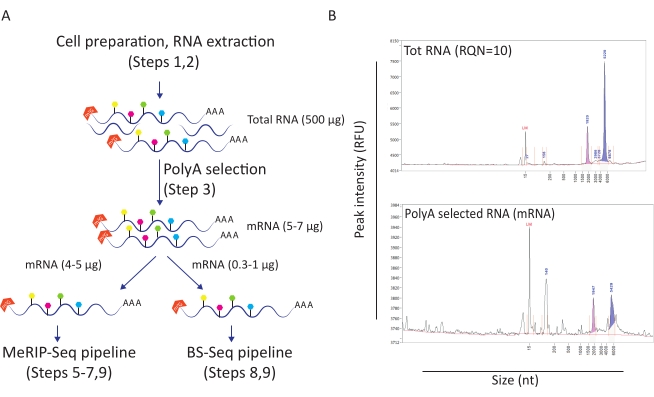
איור 1: הכנת RNA ליישומים במורד הזרם. A) זרימת עבודה המתארת הכנה והפצה של RNA עבור צינורות MeRIP-Seq ו- BS-Seq בו-זמניים. כל צורת משושה ממולאת מייצגת סוג שינוי RNA, כגון m6A (ירוק) או m5C (ורוד). כמויות של חומר RNA הדרושים לביצוע הניסוי מסומנים. B) תוצאות מייצגות המתארות פרופילי הפצת RNA צפויים (גודל וכמות) על חילוץ RNA כולל (פאנל עליון) ובחירת פולי-A (החלונית התחתונה). דגימות נטענו על מנתח הקטעים עם ערכת רגישות סטנדרטית על מנת להעריך את איכות ה- RNA לפני הזנת הליכי MeRIP-Seq ו- BS-Seq ספציפיים. RQN: מספר איכות RNA; nt: נוקלאוטידים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
צינור MeRIP-Seq הוא טכניקה מבוססת אימונופרציפיטציה של RNA המאפשרת חקירה של שינוי m6A לאורך מולקולות RNA. בשביל זה, RNA הוא תחילה מקוטע ולאחר מכן דגירה עם נוגדנים ספציפיים m6A בשילוב חרוזים מגנטיים עבור immunoprecipitation ולכידה. שברי RNA מועשרים ב-MeRIP והשבר שלא נגעו בהם (קלט) רצפים ומושווים לזיהוי אזורי RNA שעברו שינוי m6A וכך תעתיקים מתילציה m6A(איור 2A). הרזולוציה של הטכניקה מסתמכת על היעילות של פיצול RNA. ואכן, שברים קצרים יותר מאפשרים לוקליזציה מדויקת יותר של שאריות m6A. כאן, RNAs הסלולר שנבחר על-ידי פולי-A ו- RNAs ויראליים היו נתונים לפיצול מבוסס יונים עם מאגר פיצול RNA במהלך 15 דקות בנפח סופי של 20 μL כדי להשיג שברי RNA של 100-150 nt. החל מ-5 מיקרוגרם של mRNA, התאוששנו מ-4.5 מיקרוגרם של רנ"א מקוטע, המקביל לשיעור התאוששות של 90% (איור 2B). השתמשנו ב-100 ננוגרם של רנ"א מקוטע ומטוהר כבקרת קלט, הכפופה ישירות להכנת הספרייה ולרצף. הרנ"א הנותר (~4.4 מיקרוגרם) עובד על פי צינור MeRIP-Seq, שמתחיל בדגורה של RNA מקוטע עם חרוזים הקשורים לנוגדנים ספציפיים נגד m6A או לנוגדנים אנטי-IgG כשליטה. RIP (MeRIP) ספציפי ל-m6A של 2.5 מיקרוגרם של רנ"א מקוטע אפשר לאחזר כ-15 ננוגרם של חומר מועשר ב-m6A, שעבר הכנה ורצף בספרייה (איור 2B). RIP עם בקרת אנטי-IgG, כצפוי, לא הניב מספיק RNA כדי לאפשר ניתוח נוסף (איור 2B).
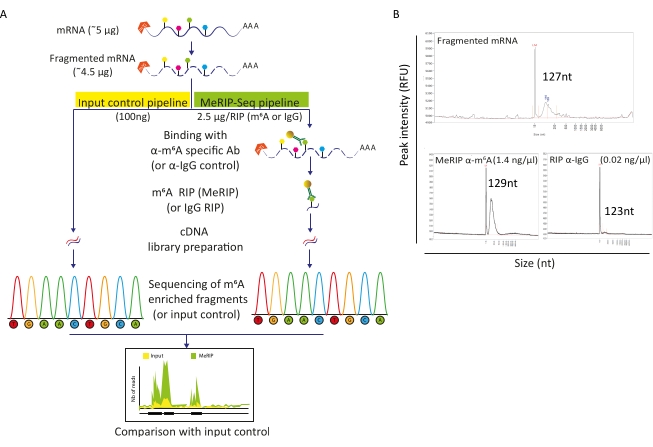
איור 2: צינור MeRIP-Seq. א) ייצוג סכמטי של זרימת עבודה של MeRIP-Seq ובקרת קלט. עם בחירת פולי-A, דגימות היו מקוטעות לחתיכות 120-150 nt, או ישירות נתון רצף (100 ננוגרם, בקרת קלט), או משמש עבור אימונופרציפיטציה RNA (2.5 מיקרוגרם, RIP) עם נוגדן ספציפי נגד m6A או נוגדן IgG כשליטה שלילית לפני הרצף. B) תוצאות מייצגות המציגות פרופילי הפצת RNA צפויים (גודל וכמות) בעת פיצול (החלונית העליונה) ו- RIP (חלוניות תחתונות, MeRIP: שמאל, פקד IgG: מימין). דגימות נטענו על מנתח קטעים כדי להעריך את איכות RNA וריכוז לפני עיבוד נוסף להכנת הספרייה ורצף. ניתוח RNA מקוטע בוצע באמצעות ערכת הרגישות הסטנדרטית של RNA בעוד ש-RNA ללא חיסון השתמש בערכת הרגישות הגבוהה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
צינור BS-Seq מאפשר חקר של שינוי RNA m5C ברזולוציית נוקלאוטיד ומוביל לזיהוי של תמלילים m5C-מתילציה. עם המרת ביסולפיט, ציטוצינים שאינם מתילאטים מומרים לאורסיל, בעוד ציטוצינים מתילאט נשארים ללא שינוי (איור 3A). בשל התנאים הקשים של הליך המרת ביסולפיט (כלומר, טמפרטורה גבוהה ו- pH נמוך), mRNAs שהומרו מושפלים מאוד (איור 3B), אולם זה לא מפריע להכנת הספרייה ורצף. המרת ביסולפיט יעילה רק ברנ"א חד-גדילי, ולכן עלולה להיות מעוכבת על ידי מבני RNA דו-גדיליים משניים. כדי להעריך את היעילות של המרת C-U הצגנו שני פקדים. כשליטה חיובית, ניצלנו את הנוכחות שתוארה בעבר של ציטוסין מתילציה מאוד בעמדה C4447 של 28S rRNA23. עם הגברה RT-PCR ורצף של שבר 200 bp המקיף את האתר מתילציה אנו יכולים לראות כי כל ציטוזינים הוסבו בהצלחה uracils, ובכך הופיע כמו thymidines ברצף ה- DNA, למעט ציטוסין במיקום 4447 שנותר ללא שינוי. כפקד על קצב ההמרה של ביסולפיט, השתמשנו ברצפי RNA סינתטיים זמינים מסחרית של ERCC. תערובת זו מורכבת ממאגר של רצפי RNA ידועים, לא מתילציה ופולי-אדנילציה, עם מגוון מבנים ואורכי משניים. עם הכנת הספרייה ורצף, התמקדנו אלה רצפי ERCC כדי לחשב את שיעור ההמרה, אשר ניתן לבצע על ידי ספירת מספר C המרה בין שאריות C הכולל בכל רצפי ERCC ובכל מדגם. השגנו שיעור המרה של 99.5%, המאשר את היעילות וההצלחה של תגובת ההמרה של ביסולפיט (איור 3D).
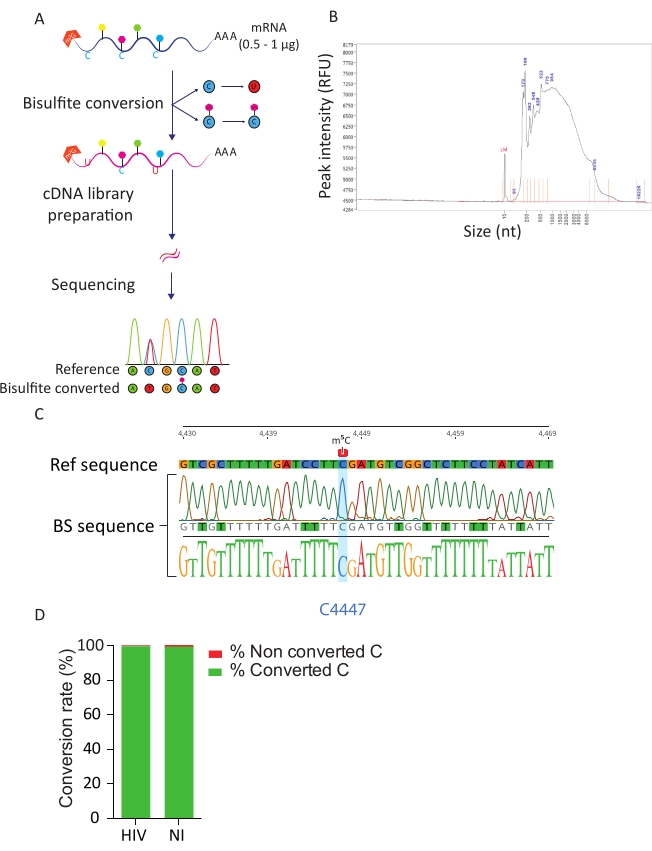
איור 3: צינור ב.ס.ס.ס.ק. א) ייצוג סכמטי של זרימת עבודה של BS-Seq. עם בחירת פולי-A, דגימות חשופות ביסולפיט, וכתוצאה מכך המרה C ל- U (עקב deamination) עבור שאריות C שאינן מתיל. לעומת זאת, שאריות C מתילאט (m5C) אינן מושפעות מטיפול ביסולפיט ונשארות ללא שינוי. ב) תוצאה מייצגת של פרופיל הפצת RNA (גודל וכמות) שעבר ביסולפיט בעת ניתוח מנתח קטעים עם ערכת רגישות סטנדרטית. C) אלקטרופרוגרמה המציגה תוצאת רצף מייצגת של אמפליסון RT-PCR של האזור המקיף את 100% מתילציה C במיקום 4447 ב 28S rRNA (מודגש בכחול). לעומת זאת, שאריות C של רצף הייחוס זוהו כשאריות T ברצף האמפליקון עקב הצלחת המרת ביסולפיט. ד) הערכה של שיעור ההמרה C-U על ידי ניתוח של רצפי ספייק-אין ERCC בתאים נגועים ב- HIV ולא נגועים. שיעור ההמרה הממוצע הוא 99.5%. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
דגימות מועשרות M6A, דגימות מומרים של ביסולפיט ובקרות קלט מעובדות עוד יותר להכנת הספרייה, רצף וניתוח ביואינפורמטי (איור 4). על פי התכנון הניסיוני ושאלות ביולוגיות שנדונו, ניתן ליישם ניתוחים ביואינפורמטיים מרובים. כהוכחה עקרונית כאן, אנו מציגים תוצאות מייצגות מיישום פוטנציאלי אחד (כלומר, ניתוח מתילציה דיפרנציאלי), המתמקד בזיהוי של תמלילים מתילציה דיפרנציאלית המושרה על הידבקות ב- HIV. בקצרה, חקרנו את רמת המתילציה m6A או m5C של תמלילים, ללא תלות ברמת ביטוי הגנים שלהם, הן בתאים שאינם נגועים והן בתאים נגועים ב- HIV, על מנת להבין עוד יותר את תפקידם של מתילציות RNA במהלך מחזור החיים הנגיפי. עם נורמליזציה של ביטוי הגנים, זיהינו כי התמליל ZNF469 היה דיפרנציאלי m6A-מתילציה בהתאם למצב הזיהום, אכן תעתיק זה לא היה מתילציה בתאים שאינם נגועים בזמן שהוא הציג כמה פסגות מתילציה על זיהום HIV (איור 5A). ניתוח מתילציה דיפרנציאלי דומה ב-m5C גילה כי התמליל PHLPP1 הכיל מספר שאריות מתילציה, אשר נוטות להיות מתילציה בתדירות גבוהה יותר במצב HIV (איור 5B). בהקשר זה, שני הניתוחים מראים כי זיהום HIV משפיע על epitranscriptome הסלולר.
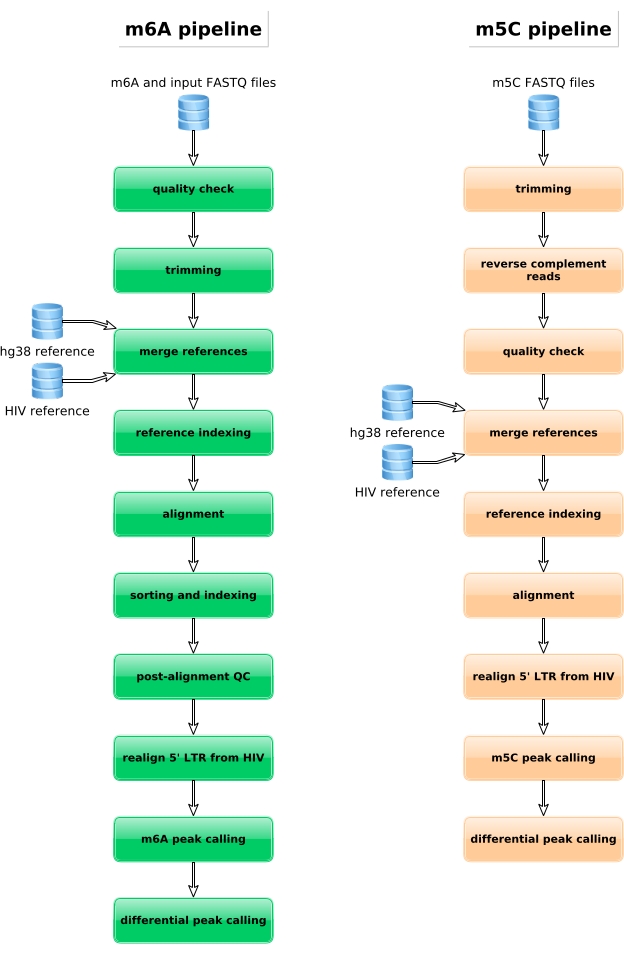
איור 4: ייצוג סכמטי של זרימת העבודה הביו-אינפורמטית לניתוח נתוני m6A ו- m5C. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
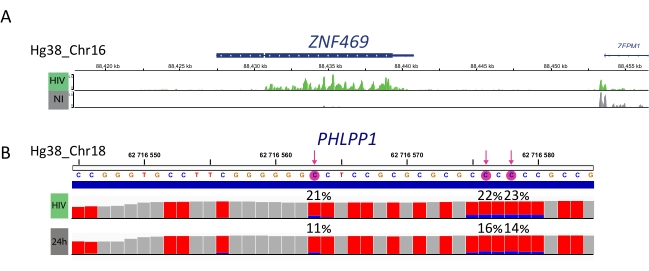
איור 5: דוגמה לתעתיקים מתילים דיפרנציאליים עם זיהום. A) תוצאה מייצגת המציגה m6A מתילציה של ZNF459 תעתיק בתאים נגועים ב- HIV (ירוק) ולא נגועים (אפור). עוצמת שיא (עם חיסור ביטוי קלט) מוצגת בציר ה- y ובמיקום בכרומוזום לאורך ציר ה- x. ניתוח מתילציה דיפרנציאלי מגלה כי ZFN469 תעתיק הוא hypermethylated על זיהום HIV. B) תוצאה מייצגת של גן מתילציה m5C בתאים נגועים ב- HIV (נתיב עליון) ותאים שאינם נגועים (נתיב תחתון). הגובה של כל סרגל מייצג את מספר הקריאות לכל נוקלאוטיד ומאפשר הערכת כיסוי. כל שאריות C ב מיוצג באדום, ואת החלק של C מתילציה מיוצג בכחול. שיעור המתילציה המדויק (%) מדווח מעל כל שאריות C. חצים מדגישים באופן סטטיסטי משמעותי מבחינה דיפרנציאלית C. דוגמאות היו דמיינו באמצעות מציג IGV. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
Discussion
תפקידם של שינויים ב- RNA בזיהום נגיפי עדיין אינו ידוע ברובו. הבנה טובה יותר של תפקידם של שינויים אפיטרנראציים בהקשר של זיהום ויראלי יכולה לתרום לחיפוש אחר מטרות טיפול אנטי ויראליות חדשות.
בעבודה זו, אנו מספקים זרימת עבודה מלאה המאפשרת חקירה של m6A ו- m5C epitranscriptomes של תאים נגועים. בהתאם לשאלה הביולוגית, אנו ממליצים להשתמש ב- RNA שנבחר בפולי-A כחומר התחלתי. למרות אופציונלי, כמו הצינור יכול לשמש עם RNA הכולל, חשוב לזכור כי rRNAs, כמו גם RNAs קטנים הם מאוד שונה ומכילים מספר חשוב של שאריות מתילציה. הדבר עלול לגרום לירידה באיכות ובכמות של נתוני רצף משמעותיים.
עם זאת, אם המוקד של המחקר הוא RNA שאינו פולי-adenylated, שלב החילוץ RNA צריך להיות מותאם על מנת למנוע השלכת RNA קטן (במקרה של חילוץ RNA מבוסס עמודה) וכדי הרשאות ריבוזום-דלדול טכניקות ולא בחירת poly-A להיכנס לצנרת.
על מנת להבטיח RNA באיכות גבוהה, פיצול נכון ואיכות RNA מועשרת m6A ו- BS מתאים להכנת הספרייה אנו ממליצים בחום להשתמש מנתח שברים או ביואנלייזר. עם זאת, ציוד זה אינו זמין תמיד. כחלופה, איכות הרנ"א, ה-mRNA והגודל של RNA מקוטע ניתן גם להעריך על ידי הדמיה על ג'ל אגרוז. לחלופין, ניתן לבצע הכנת ספריה ללא הערכה קודמת של כמות RNA.
השתמשנו בטכניקת MeRIP-Seq16 המבוססת על נוגדנים כדי לחקור את הנוף האפיטרנכתובי m6A. טכניקה זו מבוססת על אימונופרציפיטציה של RNA והיא מוצלחת; עם זאת, שלבים מסוימים זקוקים למיטוב זהיר ויכולים להיות קריטיים. למרות מתילציה m6A תואר להתרחש בעיקר בתוך רצף הקונצנזוס RRA * CH, מוטיב זה הוא שכיח מאוד לאורך מולקולות mRNA ואינו מאפשר זיהוי מדויק של האתר מתילציה. לכן חשוב להשיג פיצול RNA לשחזור ועקבי, יצירת שברי RNA קטנים, כדי לשפר את הרזולוציה מבוססת RIP. בפרוטוקול זה, אנו ממליצים על הליך ממוטב, המספק תוצאות ניתנות לשחזור ועקביות בסביבה הניסיונית שלנו; עם זאת, שלב פיצול זה עשוי להזדקק למיטוב נוסף בהתאם לתכונות לדוגמה ספציפיות.
לאחרונה תוארה טכניקה חדשה המאפשרת רצף ישיר של m6A. הוא מבוסס על השימוש בגרסאות תמלול הפוכות ספציפיות המציגות חתימות RT ייחודיות כתגובה למפגש עם m6A RNA modification24. טכנולוגיה זו, לאחר אופטימיזציה זהירה, יכולה לעקוף את המגבלה העיקרית העומדת בפני MeRIP-Seq (הפחתת כמות החומר הראשוני ומאפשרת רזולוציה גבוהה יותר). כדי לחקור את שינוי m5C החלטנו להשתמש בטכניקת ההמרה bisulfite על מנת לזהות ברזולוציית נוקלאוטיד שאריות C שונה. על מנת להפחית את שיעור החיובי השגוי בשל נוכחותם של מבנים משניים RNA, ביצענו 3 מחזורים של המרת denaturation / bisulfite ולשלוט נוסף בביצועי שיעור ההמרה bisulfite הודות לשימוש של בקרת ספייק-אין ERCC. אחת המגבלות הקשורות לטכניקה זו היא כי המרת bisulfite הוא קשה מאוד ושלושה מחזורים של denaturation / ביסולפיט המרה יכול לבזות כמה RNA ולכן להפחית את הרזולוציה. עם זאת, בהגדרה שלנו, בחרנו להסתפק ברזולוציה מעט נמוכה יותר על מנת להגדיל את איכות ערכת הנתונים.
הודות לאופטימיזציות ובקרות אלה, הצלחנו לספק זרימת עבודה אמינה וקול שניתן לנצל כדי לחקור את הנוף האפיטרנכתובי ואת השינוי שלו בהקשר של זיהומים ויראליים, אינטראקציות מארח-פתוגן, או כל חשיפה לטיפולים ספציפיים.
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
עבודה זו נתמכה על ידי הקרן הלאומית למדע בשווייץ (מענקים 31003A_166412 ו 314730_188877).
Materials
| Name | Company | Catalog Number | Comments |
| AccuPrime Pfx SuperMix | Invitrogen | 12344-040 | |
| anti-m6A antibody _Clone 17-3-4-1 | Millipore | MABE1006 | |
| Chloroform | Merck | 67-66-3 | |
| ERCC | Invitrogen | 4456740 | |
| EZ RNA Methylation Kit | Zymo Research | EZR5001 | |
| Fragment analyzer RNA Kit - HS RNA Kit | Agilent | DNF-472-0500 | |
| Fragment analyzer RNA Kit - RNA Kit | Agilent | DNF-471-0500 | |
| High-Capacity cDNA Reverse Transcription Kit | Applied Biosystem | 4368814 | |
| Illumina TruSeq Stranded mRNA | Illumina | 20020594 | |
| Magnetic Beads A/G Blend | Merck | 16-663 | |
| N6-Methyladenosine, 5′-monophosphate sodium salt (m6A) | Sigma Aldrich | M2780-10MG | |
| Normal Mouse IgG | Merk | 12371 | |
| Oligo(dT)25 | Life Technologies | 61005, | |
| PCRapace | Stratec | 1020220300 | |
| Quick RNA Viral Kit | Zymo Research | 1034 | |
| RNA Clean & Concentrator | Zymo Research | R1015 | |
| RNA Fragmentation Reagent | Ambion | AM8740 | |
| RNase Inhibitor | Ambion | AM2684 | |
| Trizol | TRIzol Reagent | 15596026 |
References
- Machnicka, M. A., et al. MODOMICS: a database of RNA modification pathways--2013 update. Nucleic Acids Research. 41, 262-267 (2013).
- Zaccara, S., Ries, R. J., Jaffrey, S. R. Reading, writing and erasing mRNA methylation. Nature Reviews Molecular Cell Biology. 20 (10), 608-624 (2019).
- Davalos, V., Blanco, S., Esteller, M. SnapShot: Messenger RNA Modifications. Cell. 174 (2), 498 (2018).
- Saletore, Y., et al. The birth of the Epitranscriptome: deciphering the function of RNA modifications. Genome Biology. 13 (10), 175 (2012).
- Zhao, B. S., Roundtree, I. A., He, C. Post-transcriptional gene regulation by mRNA modifications. Nature Reviews Molecular Cell Biology. 18 (1), 31-42 (2017).
- Netzband, R., Pager, C. T. Epitranscriptomic marks: Emerging modulators of RNA virus gene expression. Wiley Interdisciplinary Reviews: RNA. 11 (3), 1576 (2020).
- Pereira-Montecinos, C., Valiente-Echeverria, F., Soto-Rifo, R. Epitranscriptomic regulation of viral replication. Biochimica et Biophysica Acta. 1860 (4), 460-471 (2017).
- Lichinchi, G., et al. Dynamics of the human and viral m(6)A RNA methylomes during HIV-1 infection of T cells. Nature Microbiology. 1, 16011 (2016).
- Courtney, D. G., et al. Epitranscriptomic Addition of m(5)C to HIV-1 Transcripts Regulates Viral Gene Expression. Cell Host & Microbe. 26 (2), 217-227 (2019).
- Kennedy, E. M., et al. Posttranscriptional m(6)A Editing of HIV-1 mRNAs Enhances Viral Gene Expression. Cell Host & Microbe. 19 (5), 675-685 (2016).
- Tirumuru, N., Wu, L. HIV-1 envelope proteins up-regulate N (6)-methyladenosine levels of cellular RNA independently of viral replication. Journal of Biological Chemistry. 294 (9), 3249-3260 (2019).
- Tirumuru, N., et al. N(6)-methyladenosine of HIV-1 RNA regulates viral infection and HIV-1 Gag protein expression. Elife. 5, (2016).
- Cristinelli, S., Angelino, P., Janowczyk, A., Delorenzi, M., Ciuffi, A. HIV Modifies the m6A and m5C Epitranscriptomic Landscape of the Host Cell. Frontiers in Virology. 1 (11), (2021).
- Khoddami, V., Cairns, B. R. Transcriptome-wide target profiling of RNA cytosine methyltransferases using the mechanism-based enrichment procedure Aza-IP. Nature Protocols. 9 (2), 337-361 (2014).
- Hussain, S., Aleksic, J., Blanco, S., Dietmann, S., Frye, M. Characterizing 5-methylcytosine in the mammalian epitranscriptome. Genome Biology. 14 (11), 215 (2013).
- Dominissini, D., Moshitch-Moshkovitz, S., Salmon-Divon, M., Amariglio, N., Rechavi, G. Transcriptome-wide mapping of N6-methyladenosine by m6A-seq based on immunocapturing and massively parallel sequencing. Nature Protocols. 8 (1), 176-189 (2013).
- Dominissini, D., et al. Topology of the human and mouse m6A RNA methylomes revealed by m6A-seq. Nature. 485 (7397), 201-206 (2012).
- Shobbir Hussain, J. A., Blanco, S., Dietmann, S., Frye, M. Characterizing 5-methylcytosine in the mammalian epitranscriptome. Genome Biology. 14 (215), (2013).
- Amort, T., et al. Distinct 5-methylcytosine profiles in poly(A) RNA from mouse embryonic stem cells and brain. Genome Biology. 18 (1), 1 (2017).
- Endrullat, C., Glökler, J., Franke, P., Frohme, M. Standardization and quality management in next-generation sequencing. Applied & Translational Genomics. 10, 2-9 (2016).
- Schaefer, M., Pollex, T., Hanna, K., Lyko, F. RNA cytosine methylation analysis by bisulfite sequencing. Nucleic Acids Research. 37 (2), 12 (2009).
- Cristinelli, S., Angelino, P., Janowczyk, A., Delorenzi, M., Ciuffi, A. HIV Modifies the m6A and m5C Epitranscriptomic Landscape of the Host Cell. biorxiv. 1 (11), (2021).
- Squires, J. E., et al. Widespread occurrence of 5-methylcytosine in human coding and noncoding RNA. Nucleic Acids Research. 40 (11), 5023-5033 (2012).
- Aschenbrenner, J., et al. Engineering of a DNA Polymerase for Direct m(6) A Sequencing. Angewandte Chemie (International ed. in English). 57 (2), 417-421 (2018).
- Didion, J. P., Martin, M., Collins, F. S. Atropos: specific, sensitive, and speedy trimming of sequencing reads. PeerJ. 5, 3720 (2017).
- Kim, D., Langmead, B., Salzberg, S. L. HISAT: a fast spliced aligner with low memory requirements. Nature Methods. 12 (4), 357-360 (2015).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Okonechnikov, K., Conesa, A., García-Alcalde, F. Qualimap 2: advanced multi-sample quality control for high-throughput sequencing data. Bioinformatics. 32 (2), 292-294 (2016).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Zhang, Y., et al. Model-based Analysis of ChIP-Seq (MACS). Genome Biology. 9 (9), 137 (2008).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet Journal. 17 (1), (2011).
- Shen, W., Le, S., Li, Y., Hu, F. SeqKit: A Cross-Platform and Ultrafast Toolkit for FASTA/Q File Manipulation. PLOS ONE. 11 (10), 0163962 (2016).
- Rieder, D., Amort, T., Kugler, E., Lusser, A., Trajanoski, Z. meRanTK: methylated RNA analysis ToolKit. Bioinformatics. 32 (5), 782-785 (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved