Method Article
Erforschung von m6A- und m5C-Epitranskriptomen bei Virusinfektion: ein Beispiel mit HIV
In diesem Artikel
Zusammenfassung
Die Rolle von RNA-Modifikationen bei Virusinfektionen beginnt gerade erst erforscht zu werden und könnte neue Viral-Wirt-Interaktionsmechanismen aufzeigen. In dieser Arbeit stellen wir eine Pipeline zur Verfügung, um m6A- und m5C-RNA-Modifikationen im Zusammenhang mit Virusinfektionen zu untersuchen.
Zusammenfassung
Die Rolle von RNA-Modifikationen in biologischen Prozessen stand in den letzten Jahren immer mehr Studien im Fokus und wird heute als Epitranskriptomik bezeichnet. Unter anderem wurden N6-Methyladenosin (m6A) und 5-Methylcytosin (m5C) RNA-Modifikationen an mRNA-Molekülen beschrieben und können eine Rolle bei der Modulation zellulärer Prozesse spielen. Epitranscriptomics ist somit eine neue Regulierungsebene, die zusätzlich zu transkriptomischen Analysen berücksichtigt werden muss, da sie auch durch die Exposition gegenüber chemischen oder biologischen Agenzien, einschließlich Virusinfektionen, verändert oder moduliert werden kann.
Hier stellen wir einen Workflow vor, der die Analyse der gemeinsamen zellulären und viralen epitranskriptomischen Landschaft der m6A- und m5C-Markierungen gleichzeitig in Zellen ermöglicht, die mit dem humanen Immunschwächevirus (HIV) infiziert sind oder nicht. Nach der Isolierung und Fragmentierung von mRNA aus HIV-infizierten und nicht infizierten Zellen verwendeten wir zwei verschiedene Verfahren: MeRIP-Seq, eine auf RNA-Immunpräzipitation basierende Technik, um RNA-Fragmente mit der m6A-Markierung anzureichern, und BS-Seq, eine auf Bisulfit-Umwandlung basierende Technik, um die m5C-Markierung mit einer einzigen Nukleotidauflösung zu identifizieren. Nach der methylierungsspezifischen Erfassung werden RNA-Bibliotheken für die Hochdurchsatzsequenzierung vorbereitet. Wir haben auch eine dedizierte Bioinformatik-Pipeline entwickelt, um differentiell methylierte (DM) Transkripte unabhängig von ihrem basalen Expressionsprofil zu identifizieren.
Insgesamt ermöglicht die Methodik die gleichzeitige Untersuchung mehrerer epitranskriptomischer Markierungen und liefert einen Atlas von DM-Transkripten bei Virusinfektion oder anderen Zellstörungen. Dieser Ansatz bietet neue Möglichkeiten, neue Akteure und neuartige Mechanismen der Zellantwort zu identifizieren, wie z.B. zelluläre Faktoren, die die virale Replikation fördern oder einschränken.
Einleitung
Es ist seit langem bekannt, dass RNA-Moleküle modifiziert werden können, und bis heute wurden mehr als 150 posttranskriptionelle Modifikationen beschrieben1. Sie bestehen in der Addition von chemischen Gruppen, hauptsächlich Methylgruppen, zu praktisch jeder Position der Pyrimidin- und Purinringe von RNA-Molekülen2. Solche posttranskriptionellen Modifikationen haben sich bereits als hochangereichert in Transfer-RNA (tRNA) und ribosomaler RNA (rRNA) erwiesen und wurden kürzlich auch an mRNA-Molekülen beschrieben.
Der Aufstieg neuer Technologien wie Next Generation Sequencing (NGS) und die Produktion spezifischer Antikörper, die bestimmte chemische Modifikationen erkennen, ermöglichten erstmals die Untersuchung des Ortes und der Häufigkeit spezifischer chemischer Modifikationen auf transkriptomweiter Ebene. Diese Fortschritte haben zu einem besseren Verständnis von RNA-Modifikationen und zur Kartierung mehrerer Modifikationen an mRNA-Molekülen geführt3,4.
Während die Epigenetik die Rolle von DNA- und Histonmodifikationen bei der Transkriptomregulation untersucht, konzentriert sich die Epitranskriptomik in ähnlicher Weise auf RNA-Modifikationen und ihre Rolle. Die Untersuchung epitranskriptomischer Modifikationen bietet neue Möglichkeiten, neuartige Regulationsmechanismen hervorzuheben, die eine Vielzahl zellulärer Prozesse (d.h. RNA-Spleißen, Export, Stabilität und Translation) abstimmen können5. Es war daher keine große Überraschung, dass neuere Studien viele epitranskriptomische Modifikationen bei einer Virusinfektion sowohl in zellulären als auch in viralen RNAs aufdeckten6. Zu den bisher untersuchten Viren gehören sowohl DNA- als auch RNA-Viren; unter ihnen kann HIV als wegweisendes Beispiel angesehen werden. Insgesamt könnte die Entdeckung der RNA-Methylierung im Zusammenhang mit Virusinfektionen die Untersuchung noch unbeschriebener Mechanismen der viralen Expression oder Replikation ermöglichen und so neue Werkzeuge und Ziele zu deren Kontrolle liefern7.
Auf dem Gebiet der HIV-Epitranskriptomik wurden Modifikationen viraler Transkripte umfassend untersucht und haben gezeigt, dass das Vorhandensein dieser Modifikation für die Virusreplikation von Vorteil war8,9,10,11,12,13. Bis heute können verschiedene Techniken verwendet werden, um epitranskriptomische Markierungen auf transkriptomweiter Ebene zu erkennen. Die am häufigsten verwendeten Techniken zur m6A-Identifizierung basieren auf Immunfällungstechniken wie MeRIP-Seq und miCLIP. Während MeRIP-Seq auf RNA-Fragmentierung setzt, um Fragmente zu erfassen, die methylierte Reste enthalten, basiert miCLIP auf der Erzeugung von α-m6A-Antikörper-spezifischen Signaturmutationen bei RNA-Antikörper-UV-Vernetzung, was eine genauere Kartierung ermöglicht.
Der Nachweis der m5C-Modifikation kann entweder durch Antikörper-basierte Technologien ähnlich dem m6A-Nachweis (m5C RIP) oder durch Bisulfit-Umwandlung oder durch AZA-IP oder miCLIP erreicht werden. Sowohl Aza-IP als auch m5C miCLIP verwenden eine spezifische Methyltransferase als Köder, um RNA anzugreifen, während sie die RNA-Methylierung durchlaufen. In Aza-IP werden Zielzellen mit 5-Azacytidin exponiert, was zur zufälligen Einführung von Cytidin-analogen 5-Azacytidin-Stellen in die entstehende RNA führt. In miCLIP ist die NSun2-Methyltransferase genetisch so verändert, dass sie die C271A-Mutation enthält14,15.
In dieser Arbeit konzentrieren wir uns auf die doppelte Charakterisierung von m6A- und m5C-Modifikationen in infizierten Zellen am Beispiel von HIV. Nach methodischer Optimierung haben wir einen Workflow entwickelt, der methylierte RNA-Immunpräzipitation (MeRIP) und RNA-Bisulfit-Umwandlung (BS) kombiniert und so die gleichzeitige Erforschung von m6A- und m5C-epitranskriptomischen Markierungen auf transkriptomweiter Ebene sowohl in zellulären als auch in viralen Kontexten ermöglicht. Dieser Workflow kann sowohl auf zellulären RNA-Extrakten als auch auf RNA, die aus viralen Partikeln isoliert wurde, implementiert werden.
Der Methylated RNA ImmunoPrecipitation (MeRIP)16-Ansatz, der die Untersuchung von m6A auf transkriptomweiter Ebene ermöglicht, ist gut etabliert und eine Reihe von m6A-spezifischen Antikörpern ist bis heute kommerziell erhältlich17. Diese Methode besteht in der selektiven Erfassung von m6A-haltigen RNA-Stücken mit einem m6A-spezifischen Antikörper. Die beiden Hauptnachteile dieser Technik sind (i) die begrenzte Auflösung, die stark von der Größe der RNA-Fragmente abhängt und somit einen ungefähren Ort und eine Region liefert, die den methylierten Rückstand enthält, und (ii) die große Menge an Material, die zur Durchführung der Analyse benötigt wird. Im folgenden optimierten Protokoll haben wir die Fragmentgröße auf etwa 150 nt standardisiert und die Menge an Ausgangsmaterial von 10 μg Poly-A-selektierter RNA, was derzeit die empfohlene Menge an Ausgangsmaterial ist, auf nur 1 μg Poly-A-selektierte RNA reduziert. Wir maximierten auch die Rückgewinnungseffizienz von m6A-RNA-Fragmenten, die an spezifische Antikörper gebunden sind, indem wir eine Elution durch einen Wettbewerbsansatz mit einem m6A-Peptid anstelle von konventionelleren und weniger spezifischen Elutionsmethoden mit phenolbasierten Techniken oder Proteinase K verwendeten. Die Haupteinschränkung dieses RIP-basierten Assays bleibt jedoch die suboptimale Auflösung, die die Identifizierung des präzisen modifizierten A-Nukleotids nicht zulässt.
Die Analyse der m5C-Markierung kann derzeit mit zwei verschiedenen Ansätzen durchgeführt werden: einer RIP-basierten Methode mit m5C-spezifischen Antikörpern und einer RNA-Bisulfit-Umwandlung. Da RIP nur eine begrenzte Auflösung für die Identifizierung des methylierten Rückstands bietet, haben wir die Bisulfitkonversion verwendet, die eine Einzelnukleotidauflösung bieten kann. Die RNA-Exposition gegenüber Bisulfit (BS) führt zu einer Cytosin-Desaminierung, wodurch der Cytosinrest in Uracil umgewandelt wird. So wird während der RNA-Bisulfit-Umwandlungsreaktion jedes nicht methylierte Cytosin deaminiert und in Uracil umgewandelt, während das Vorhandensein einer Methylgruppe in Position 5 des Cytosins eine schützende Wirkung hat, die BS-induzierte Desaminierung verhindert und den Cytosinrückstand konserviert. Der BS-basierte Ansatz ermöglicht den Nachweis eines m5C-modifizierten Nukleotids mit einzelner Basenauflösung und die Bewertung der Methylierungsfrequenz jedes Transkripts, wodurch Einblicke in die m5C-Modifikationsdynamik18 gegeben werden. Die Haupteinschränkung dieser Technik beruht jedoch auf der Falsch-Positiv-Rate methylierter Rückstände. Tatsächlich ist die BS-Umwandlung auf einzelsträngige RNA mit zugänglichen C-Resten wirksam. Das Vorhandensein einer engen RNA-Sekundärstruktur könnte jedoch die N5C-Position maskieren und die BS-Umwandlung behindern, was zu nicht methylierten C-Resten führt, die nicht in U-Reste umgewandelt werden, und somit zu falsch positiven Ergebnissen. Um dieses Problem zu umgehen und die Falsch-Positiv-Rate zu minimieren, haben wir 3 Runden Denaturierungs- und Bisulfit-Umwandlungszyklen angewendet19. Wir haben auch 2 Kontrollen in die Proben eingeführt, um die Abschätzung der Bisulfit-Umwandlungseffizienz zu ermöglichen: Wir spike-in ERCC-Sequenzierungskontrollen (nicht methylierte standardisierte und kommerziell erhältliche Sequenzen)20 sowie Poly-A-depletierte RNAs, um einerseits die Bisulfit-Umwandlungsrate zu bewerten und durch RT-PCR das Vorhandensein einer bekannten und gut konservierten methylierten Stelle, C4447, zu überprüfen. auf 28S ribosomaler RNA hingegen21.
Im Bereich der Virologie ermöglicht die Kopplung dieser beiden epitranskriptomischen Untersuchungsmethoden mit next generation sequencing und genauer bioinformatischer Analyse eine eingehende Untersuchung der m6A- und m5C-Dynamik (d.h. zeitliche Veränderungen der RNA-Modifikation, die bei einer Virusinfektion auftreten könnten und eine Reihe neuer therapeutisch relevanter Ziele für den klinischen Einsatz aufdecken könnten).
Protokoll
1. Zellpräparation
HINWEIS: Je nach Zelltyp und RNA-Gehalt kann die Ausgangsanzahl der Zellen variieren.
- Haben Sie genug Zellen, um zwischen 200-500 μg Gesamt-RNA oder 5-7 μg Poly-A-selektierte RNA zu erhalten. Beispielsweise sollten 50 x 106 SupT1-Zellen bei extraktion mit phenolbasierten Reagenzien rund 500 μg Gesamt-RNA liefern und werden daher für jede einzelne getestete Bedingung benötigt.
- Bereiten Sie die erforderliche Anzahl von Zellen entsprechend dem experimentellen Design und damit entsprechend der Anzahl der getesteten Bedingungen (Infektion, Zeitpunkte, Behandlung) vor. Wenn das Experiment darauf abzielt, 24 Stunden nach der Infektion nicht infizierte Zellen und HIV-infizierte Zellen zu erhalten, werden insgesamt 100 x 106 Zellen benötigt, die Hälfte für den nicht 0-infizierten Zustand und die Hälfte für den infizierten Zustand.
2. RNA-Extraktion
- Aus Zellen: RNA-Extraktion mit Phenol-Chloroform
- Sammeln Sie für jede Bedingung Zellen (z. B. 50 x 106) durch Zentrifugation und verwerfen Sie den Überstand.
- Fügen Sie 5 ml Reagenz auf Phenolbasis zu jedem 50 x 106 Zellpellet hinzu und mischen Sie es durch mehrmaliges Pipettieren auf und ab.
- Inkubieren Sie für 5 min bei Raumtemperatur, um eine vollständige Lyse zu ermöglichen. Lysierte Zellen können bei -80 °C gelagert oder direkt verarbeitet werden.
HINWEIS: Bei Bedarf können die Zellen auch in Aliquots von 10 x 106 Zellen pro Röhrchen in 1,5 ml Röhrchen aufgeteilt und zur bequemeren Lagerung in 1 ml Reagenz auf Phenolbasis lysiert werden. - 1 ml Chloroform zugeben und durch Inversion mischen.
- 3 min bei Raumtemperatur inkubieren.
- Zentrifuge für 15 min bei 2.000 x g und 4 °C.
- Die wässrige Phase (obere Phase) pipettieren und in ein neues Röhrchen überführen. Beenden Sie die Übertragung der wässrigen Phase, indem Sie das Rohr bei 45 ° anwinkeln und die Lösung vorsichtig pipettieren.
HINWEIS: Die Menge der wässrigen Phase kann zwischen den Proben variieren, sollte jedoch in der Nähe der Menge an Chloroform liegen, die der Probe zugesetzt wird (d. h. 1 ml). Übertragen Sie keine Interphasen- oder organischen Schichten! Die Verwendung von Phasensperr- oder Phasenmacherrohren kann diesen Prozess erleichtern. - Fügen Sie 0,5 ml 100% molekulares Isopropanol zur wässrigen Phase hinzu.
- 1 h bei -80 °C inkubieren, um eine RNA-Ausfällung zu ermöglichen.
- Zentrifugiere für 10 min bei 12.000 x g und 4 °C, um die ausgefällte RNA zu pelletieren.
- Verwerfen Sie den Überstand und resuspenieren Sie das RNA-Pellet in 1 ml Ethanol in 75% molekularbiologischer Qualität. Wirbel kurz.
- 5 min bei 7.500 x g und 4 °C zentrifugieren und den Überstand entsorgen.
- Trocknen Sie das Pellet 15 Minuten lang an der Luft.
- Resuspendieren Sie das Pellet in 20 μL RNase-freiem Wasser und geben Sie es in ein neues Röhrchen um.
- Waschen Sie das leere Röhrchen mit zusätzlichen 20 μL Wasser, um die RNA-Rückgewinnung zu maximieren, und poolen Sie es mit dem ersten 20 μL-Volumen.
- Quantifizieren Sie die Gesamt-RNA mit einem Spektralphotometer und bewerten Sie die RNA-Qualität mit einem Fragmentanalysator.
- Aus viralen Partikeln: RNA-Extraktion mit säulenbasiertem viralem RNA-Extraktionskit
HINWEIS: Die RNA-Extraktion aus viralen Partikeln mit phenolbasiertem Reagenz führt zu viraler RNA von geringer Qualität und zu Bibliotheken mit geringerer Qualität. Eine säulenbasierte RNA-Extraktion sollte daher bevorzugt werden. RNA-Extraktionskits, die Träger-RNA zur RNA-Elution und -Rückgewinnung verwenden, sind für dieses Verfahren nicht geeignet und sollten vermieden werden. Da HIV-RNA poly-adenyliert ist, reicht eine direkte RNA-Extraktion ohne weitere mRNA-Isolierung aus, um in die MeRIP-Seq- und BS-Seq-Pipelines zu gelangen. Normalerweise sollten 1-2 ml viraler Überstand aus universell infizierten Zellen genügend RNA liefern, um den gesamten Workflow durchzuführen.- Bereiten Sie den Puffer vor, indem Sie 150 μL Beta-Mercaptoethanol zu 30 ml Lysepuffer hinzufügen. Rekonstituieren Sie den Viral Wash Buffer durch Zugabe von 96 ml 100% Ethanol.
- Sammeln Sie virushaltige Überstände und zentrifugieren Sie zu Pelletzelltrümmern, um die zelluläre RNA-Kontamination zu minimieren.
- Übertragen Sie 1 ml viralen Überstand in eine 15 ml Röhre.
- Fügen Sie 3 ml viralen RNA-Puffer zu 1 ml viraler Probe hinzu und mischen Sie durch Vortexing.
- Übertragen Sie 700 μL Probe in eine Säule, die in ein Sammelröhrchen eingeführt wird.
- Zentrifuge für 2 min bei 13.000 x g bei Raumtemperatur.
- Verwerfen Sie den Durchfluss.
- Wiederholen Sie die 3 vorherigen Schritte, bis die gesamte Probe verarbeitet wurde und somit die gesamte RNA auf der Siliziumdioxid-basierten Matrixsäule eingefangen wurde.
- Geben Sie 500 μL Viral Wash Buffer in die Spalte.
- Zentrifuge für 1 min bei 10.000 x g bei Raumtemperatur. Verwerfen Sie den Durchfluss.
- Fügen Sie 200 μL Viral Wash Buffer in die Spalte hinzu.
- Zentrifuge für 1 min bei 10.000 x g bei Raumtemperatur. Verwerfen Sie den Durchfluss.
- Legen Sie die Säule in ein leeres Auffangrohr.
- Zentrifugieren Sie für 1 min bei 10.000 x g bei Raumtemperatur, um verbleibende Waschpufferverunreinigungen weiter zu entsorgen.
- Die Säule vorsichtig in ein 1,5-ml-Röhrchen geben.
- 20 μL DNase/RNase-freies Wasser direkt in die Mitte der Säulenmatrix geben und bei 10.000 x g für 30 s bei Raumtemperatur zentrifugieren.
- Zusätzliche 10 μL DNase/RNase-freies Wasser direkt in die Mitte der Säulenmatrix geben und erneut für 30 s zentrifugieren.
- Quantifizieren Sie die Gesamt-RNA mit einem Spektralphotometer und bewerten Sie die RNA-Qualität mit einem Fragmentanalysator.
HINWEIS: Die RNA-Extraktion kann mit jeder Methode durchgeführt werden, wenn die Qualität der abgerufenen RNA hoch ist, mit einer RNA-Integritäts-/Qualitätszahl > 9. Die Gesamt-RNA kann bis zur Weiterverarbeitung bei -80 °C gespeichert werden.
3. mRNA-Isolierung durch Poly-A-Selektion mit Oligo(dT)25
HINWEIS: Aufgrund des Vorhandenseins von hochmethylierter ribosomaler RNA in zellulären Extrakten wird dringend empfohlen, Poly-A-RNA entweder durch rRNA-Depletion oder vorzugsweise durch Poly-A-positive Selektion zu isolieren. Dieser Schritt ist optional und sollte nur für zelluläre RNA-Proben durchgeführt werden, um Sequenzierungsergebnisse mit höherer Auflösung zu erhalten. Wenn Sie die Methylierung von nicht poly-adenylierten viralen RNAs analysieren, bevorzugen Sie die rRNA-Depletion anstelle der Poly-A-Selektion oder führen Sie schließlich die Analyse der Gesamt-RNA durch.
-
Perlenvorbereitung für Poly-A-Capture
- Resuspendieren Sie die Oligo(dT)25 Magnetperlen durch Vortexing für >30 s.
- Übertragen Sie 200 μL Magnetperlen in ein 1,5 ml Rohr. Bereiten Sie die Anzahl der Röhrchen mit magnetischen Kügelchen entsprechend der Gesamtmenge der zu verarbeitenden RNA-Proben vor.
HINWEIS: Ein Röhrchen mit 200 μL Dynabead-Stammlösung entspricht 1 mg Perlen und kann eine Probe von 75 μg Gesamt-RNA aufnehmen. - Legen Sie die Rohre für 1 min auf einen Magneten und entsorgen Sie den Überstand. Entfernen Sie die Rohre vom Magneten.
- Fügen Sie 1 ml Bindungspuffer (20 mM Tris-HCl, pH 7,5, 1,0 m LiCl, 2 mM EDTA) hinzu und resuspendieren Sie durch Vortexing. Legen Sie die Rohre für 1 min auf den Magneten und entsorgen Sie den Überstand. Entfernen Sie die Rohre vom Magneten. Wiederholen.
- Resuspendieren Sie die gewaschenen Magnetperlen in 100 μL Bindungspuffer.
-
Gesamt-RNA-Präparation
- Verdünnen Sie die Gesamt-RNA in einer Endkonzentration von 0,75 μg/μL mit RNase-freiem Wasser, was 75 μg/100 μL entspricht.
HINWEIS: Wenn die RNA eine niedrigere Konzentration aufweist, gehen Sie wie folgt vor, ohne die Volumina zu verändern. - Aliquotieren Sie die Gesamt-RNA in mehreren Röhrchen, indem Sie 100 μL RNA-Probe pro Röhrchen abgeben.
- Jeder RNA-Probe werden 100 μL Bindungspuffer zugesetzt.
- Erhitzen Sie die Gesamt-RNA für 2 min auf 65 °C, um sekundäre Strukturen zu stören.
- Sofort auf Eis legen, bis sie bereit sind, mit dem nächsten Schritt fortzufahren.
HINWEIS: Die Inkubationszeit kann je nach Anzahl der zu verarbeitenden Proben variieren, sollte jedoch 1 h nicht überschreiten, um einen RNA-Abbau zu vermeiden.
- Verdünnen Sie die Gesamt-RNA in einer Endkonzentration von 0,75 μg/μL mit RNase-freiem Wasser, was 75 μg/100 μL entspricht.
-
Poly-A Auswahl
- Zu jedem RNA-Röhrchen (ab Schritt 3.2) werden 100 μL gewaschene Magnetperlen (ab Schritt 3.1) gegeben.
- Durch Pipettieren auf und ab gründlich mischen und das Binden auf einem rotierenden Rad bei Raumtemperatur für 15 min zulassen.
- Öffnen Sie alle Rohre, legen Sie sie für 1 Minute auf den Magneten und entfernen Sie vorsichtig den gesamten Überstand.
- Gewinnen Sie den Überstand in einem neuen Röhrchen zurück und bewahren Sie ihn für eine zweite Runde der RNA-Erfassung (Schritt 3.3.14) auf, um die endgültige Poly-A-Erholung zu verbessern.
- Entfernen Sie das Röhrchen vom Magneten und fügen Sie 200 μL Waschpuffer hinzu (10 mM Tris-HCl, pH 7,5, 0,15 M LiCl, 1 mM EDTA). 4 bis 5 Mal durch Pipettieren vorsichtig mischen.
- Legen Sie das Rohr für 1 min auf den Magneten und entsorgen Sie den Überstand.
- Wiederholen Sie den Waschschritt einmal (wiederholen Sie die Schritte 3.3.5 und 3.3.6).
- Fügen Sie 20 μL eiskaltes 10 mM Tris-HCl hinzu, um Poly-A-RNA aus den Kügelchen zu eluieren.
- Bei 80 °C für 2 min inkubieren.
- Legen Sie das Rohr auf den Magneten und übertragen Sie den Überstand, der die Poly-A-RNA enthält, schnell in ein neues RNase-freies Röhrchen. Stellen Sie die Röhre auf Eis.
- Wiederholen Sie den Elutionsschritt (Schritte 3.3.8 bis 3.3.10), um die Ausbeute zu erhöhen.
- Waschen Sie die gleichen Perlen einmal mit 200 μL Waschpuffer. 4 bis 5 Mal durch Pipettieren vorsichtig mischen.
- Für 1 min auf den Magneten legen und waschpuffer entsorgen.
- Fügen Sie den Flowthrough aus Schritt 3.3.4 zu den Perlen hinzu und wiederholen Sie den Vorgang von der Bindung bis zur Elution (Schritte 3.3.2 bis 3.3.10). Bewahren Sie die RNA-Eluate vorerst in separaten Röhrchen auf.
HINWEIS: Optional sollte der Überstand, der Schritt 3.3.4 entspricht, in einem neuen Rohr aufbewahrt werden, da er als Steuerung verwendet werden kann. Am Ende des Verfahrens reinigen und konzentrieren Sie die RNA durch Ethanolfällung oder mit einer säulenbasierten Methode ihrer Wahl (d. H. RNA-Reinigung und Konzentrator). Diese Probe entspricht einer Poly-A-depletierten RNA-Probe und kann als Kontrolle für die Bisulfit-Umwandlung verwendet werden (Schritt 8.2.2). - Quantifizieren Sie die eluierte RNA mit einem Spektralphotometer und halten Sie ein 2 μL Aliquot, um die RNA-Qualität mit einem Fragmentanalysator weiter zu beurteilen.
HINWEIS: Poly-A-RNA kann bei -80 °C gelagert werden, bis sie benötigt wird.
4. RNA-Workflow
- Teilen Sie die zelluläre Poly-A-RNA (mRNA) und die viralen RNA-Proben in 2 Aliquots auf, die der jeweiligen epitranskriptomischen Analysepipeline gewidmet sind:
(i) 5 μg zelluläre mRNA oder 1 μg virale RNA für MeRIP-Seq und Eingabekontrollen (siehe Schritte 5 bis 7 und Schritt 9).
(ii) 1 μg zelluläre mRNA oder 500 ng virale RNA für BS-Seq (siehe Schritte 8 und 9).
5. RNA-Fragmentierung
HINWEIS: Die RNA-Fragmentierung erfolgt mit dem RNA-Fragmentierungsreagenz und ist für MeRIP-Seq- und Kontroll-RNA-Proben bestimmt. Dies ist ein sehr wichtiger Schritt, der eine sorgfältige Optimierung erfordert, um Fragmente im Bereich zwischen 100 und 200 nt zu erhalten.
- Teilen Sie das Gesamtvolumen der mRNA in 0,2 ml PCR-Röhrchen mit 18 μL mRNA/Röhrchen auf.
HINWEIS: Arbeiten Sie schnell. Arbeiten Sie nicht mit mehr als 8 Proben gleichzeitig, um reproduzierbare Ergebnisse zu erzielen. Eine Skalierung des Volumens garantiert keine reproduzierbare und gleichmäßige Fragmentierung. - Wärmen Sie einen Thermocycler bei 70 °C auf.
- Fügen Sie 2 μL Fragmentierungsreagenz am Rand jedes PCR-Röhrchens hinzu.
- Schließen Sie das Röhrchen und drehen Sie es nach unten (so dass das Reagenz gleichzeitig für die 8 Röhrchen mit RNA in Kontakt kommt).
- Inkubieren Sie die Proben 15 min bei 70 °C im vorgewärmten Thermocycler.
- Sobald die Inkubation vorbei ist, schnell 2 μL Stop-Lösung in jedes Röhrchen geben.
- Drehen Sie sich nach unten und lassen Sie es auf dem Eis sitzen, bis Sie bereit sind, mit dem nächsten Schritt fortzufahren.
HINWEIS: Die Inkubationszeit kann je nach Anzahl der zu verarbeitenden Proben variieren, sollte jedoch 1 h nicht überschreiten, um einen RNA-Abbau zu vermeiden. - Wiederholen Sie den Vorgang für alle Proben (wenn mehr als 8 Aliquots vorhanden sind).
- Poolen Sie die Röhrchen zusammen und fahren Sie mit der RNA-Reinigung mit einem RNA-Reinigungs- und Konzentrator-Kit (Schritt 6) oder einem kundenspezifischen säulenbasierten Kit fort, um die Puffer loszuwerden und saubere fragmentierte RNA in Wasser wiederherzustellen.
6. RNA-Aufreinigung
HINWEIS: Dieser Schritt kann durch Ethanolfällung oder mit jeder Art von säulenbasierter RNA-Aufreinigungs- und Konzentrationsmethode (d. H. RNA Clean und Concentrator) durchgeführt werden.
- Eluieren oder resuspenieren Sie die gereinigte RNA in einem Gesamtvolumen von 50-75 μL DNase/RNase-freiem Wasser.
HINWEIS: Wenn eine spaltenbasierte Methode verwendet wird, werden zwei Elutionsrunden dringend empfohlen, um eine maximale Wiederherstellung zu gewährleisten. - Quantifizieren Sie die gereinigte fragmentierte mRNA mit einem Spektralphotometer und beurteilen Sie die RNA-Qualität mit einem Fragmentanalysator.
- Bewahren Sie 100 ng fragmentierte mRNA als Eingabekontrolle für die Bibliotheksvorbereitung und -sequenzierung auf (weiter zu Schritt 9). Die verbleibende fragmentierte mRNA (mindestens 2,5 μg) kann für MeRIP verwendet werden (siehe Schritt 7.2).
7. MeRIP
HINWEIS: Für jede Immunpräzipitation (IP) sind mindestens 2,5 μg fragmentierte mRNA erforderlich, entweder unter Verwendung eines spezifischen Anti-m6A-Antikörpers (Testbedingung) oder unter Verwendung eines Anti-IgG-Antikörpers (Negativkontrolle).
- Magnetisches Perlenpräparat zur Immunpräzipitation
- Für jede Probe werden 4 ml 1x IP-Puffer in einem neuen konischen Röhrchen hergestellt, indem 800 μL mRNA-IP-Puffer 5x (50 mM Tris-HCl pH 7,4, 750 mM NaCl, 0,5% Igepal CA-630 und nukleasefreies Wasser) mit 3,2 ml nukleasefreiem Wasser verdünnt werden.
HINWEIS: Es sind mindestens 2 Reaktionen erforderlich (ein Test und eine IgG-Kontrolle). - Stellen Sie die Röhre auf Eis.
- Markieren Sie die entsprechende Anzahl von 1,5 ml Mikrozentrifugenröhrchen für die Anzahl der gewünschten IP-Reaktionen:
n Röhrchen (Test) für Anti-m6A-Antikörper.
n Röhren (Negativkontrolle) für Normale Maus IgG. - Resuspendieren Sie die magnetischen Kügelchen (z. B. Magna ChIP Protein A / G ) durch Invertieren und Wirbeln. Es sollten keine Perlenklumpen sichtbar sein.
- Für jede geplante Reaktion werden 25 μL Magnetperlen in ein Mikrozentrifugenröhrchen überführt.
- Fügen Sie zehnmal mehr 1x IP-Puffer (aus Schritt 7.1.1) in Bezug auf das ursprüngliche Volumen der verwendeten Perlen hinzu (d. H. 250 μL 1x IP-Puffer pro 25 μL Magnetperlen).
- Mischen Sie die Perlen, indem Sie mehrmals vorsichtig auf und ab pipettieren, um eine vollständige Wiederverwendung zu erreichen.
- Legen Sie das Rohr für 1 min auf den Magnetabscheider.
- Entfernen und entsorgen Sie den Überstand und achten Sie darauf, keine magnetischen Perlen abzusaugen. Entfernen Sie das Rohr vom Magneten.
- Wiederholen Sie den Waschschritt (Schritte 7.1.6 bis 7.1.9).
- Resuspendieren Sie die Perlen in 100 μL 1x IP-Puffer pro 25 μL Originalvolumen magnetischer Perlen.
- Fügen Sie 5 μL Antikörper (1 μg/μL) pro 25 μL ursprüngliches Volumen magnetischer Kügelchen hinzu.
n Röhrchen (Test) mit Anti-m6A-Antikörper (Klon 17-3-4-1) [1 μg/μL].
n Röhrchen (Negativkontrolle) mit Normal Mouse IgG (1 μg/μL). - Auf dem rotierenden Rad für 30 min bei Raumtemperatur inkubieren, um die Konjugation der Antikörper mit den magnetischen Kügelchen zu ermöglichen.
- Legen Sie das Rohr für 1 min auf den Magnetabscheider. Verwerfen Sie den Überstand. Entfernen Sie das Röhrchen vom Magneten und resuspendieren Sie das Antikörper-Perlen-Gemisch in 100 μL 1x IP-Puffer.
- Für jede Probe werden 4 ml 1x IP-Puffer in einem neuen konischen Röhrchen hergestellt, indem 800 μL mRNA-IP-Puffer 5x (50 mM Tris-HCl pH 7,4, 750 mM NaCl, 0,5% Igepal CA-630 und nukleasefreies Wasser) mit 3,2 ml nukleasefreiem Wasser verdünnt werden.
- RNA-Immunpräzipitation (RIP)
- Bereiten Sie 500 μL RIP-Reaktionsmischung für jede 2,5 μg mRNA-Probe wie folgt vor: 2,5 μg in 100 μL fragmentierter RNA (ab Schritt 6.12); 295 μL nukleasefreies Wasser; 5 μL 40 U/μL RNase-Inhibitor; und 100 μL 5x IP-Puffer.
- Zu jedem Antikörper-Perlen-Gemisch werden 500 μL RIP-Reaktionsgemisch (~100 μL aus Schritt 7.1.14) gegeben. Mischen Sie durch mehrmaliges vorsichtiges Pipettieren, um die Perlen vollständig neu zu versenken. Auf Eis legen.
- Inkubieren Sie alle RIP-Rohre auf einem rotierenden Rad für 2 Stunden bei 4 °C.
- Zentrifugieren Sie die MeRIP-Reaktionen kurz, um Flüssigkeitströpfchen von der Kappen- und Röhrchenseite herunterzudrehen. Legen Sie die Rohre für 1 min auf einen Magnetabscheider.
- Übertragen Sie den Überstand in ein neues Zentrifugenrohr, wobei darauf zu achten ist, dass die magnetischen Kügelchen nicht gestört werden.
HINWEIS: Flowthrough kann als Kontrolle beibehalten werden, um die RIP-Effizienz zu überprüfen (siehe Schritt 7.3.9). - Entfernen Sie die Schläuche vom Magneten. Waschen Sie die Perlen, indem Sie 500 μL kalten 1x IP-Puffer hinzufügen. Mischen Sie die Perlen, indem Sie mehrmals vorsichtig pipettieren, um die Perlen vollständig wieder aufzunehmen.
- Legen Sie die Rohre für 1 min auf einen Magnetabscheider und entsorgen Sie den Überstand.
- Wiederholen Sie den Waschvorgang (Schritte 7.2.6-7.2.7) zweimal für insgesamt 3 Wäschen.
- Legen Sie die Röhrchen auf Eis und fahren Sie sofort mit der Elution fort.
- Elution
- Herstellen einer 20 mM m6A-Lösung durch Lösen von 10 mg N6-Methyladenosin, 5′-Monophosphat-Natriumsalz (m6A) in 1,3 ml nukleasefreiem Wasser. 150 μL Aliquots zubereiten und bei -20 °C lagern.
- Für jede Probe (Test und Kontrollen): Bereiten Sie 225 μL Elutionspuffer vor, indem Sie die folgenden Komponenten mischen: 45 μL 5x IP-Puffer, 75 μL 20 mM m6A, 3,5 μL 40U/μL RNase-Inhibitor und 101,5 μL nukleasefreies Wasser.
- 100 μL Elutionspuffer (ab Schritt 7.3.2) zu den Kügelchen (ab Schritt 7.2.9) geben. Mischen Sie durch mehrmaliges vorsichtiges Pipettieren, um die Perlen vollständig zu resuspendieren.
- Alle Röhrchen für 1 h mit kontinuierlichem Schütteln auf einer Wippe bei 4 °C inkubieren.
- Zentrifugieren Sie die RIP-Reaktionen kurz, um Flüssigkeitströpfchen von der Kappen- und Röhrchenseite herunterzuschleudern. Legen Sie die Rohre für 1 min auf einen Magnetabscheider.
- Übertragen Sie den Überstand, der eluierte RNA-Fragmente enthält, in ein neues 1,5-ml-Mikrozentrifugenröhrchen. Achten Sie darauf, die Perlen nicht abzusaugen, da dies die Hintergrundgeräusche erhöht.
- Wiederholen Sie die Elutionsschritte (7.3.3 bis 7.3.6), indem Sie erneut 100 μL Elutionspuffer hinzufügen, 1 h bei 4 °C inkubieren und das Eluat nach magnetischer Trennung sammeln.
- Kombinieren Sie alle Eluate aus derselben Probe (das Gesamtelutionsvolumen sollte 200 μL betragen).
- Reinigen Sie die eluierte RNA und den Durchfluss (optional, ab Schritt 7.2.5) durch Ethanolfällung oder durch eine säulenbasierte Methode ihrer Wahl (d. h. RNA Clean und Concentrator).
- Bewerten Sie die RNA-Quantität und -Qualität von Durchfluss- und eluierten Proben mit einem Fragmentanalysator unter Verwendung eines hochempfindlichen Nachweiskits. Wenn die Qualität der RNA zufriedenstellend ist, fahren Sie mit der Bibliotheksvorbereitung und der Hochdurchsatzsequenzierung fort (Schritt 9).
HINWEIS: Die Menge an RNA, die bei MeRIP abgerufen wird, ist sehr gering und erfordert zwingend hochempfindliche Nachweiskits, um die Quantifizierung sicherzustellen. Wenn kein Bioanalytiker verfügbar ist, ist es möglich, blind mit der Bibliotheksvorbereitung fortzufahren.
8. RNA-Bisulfit-Umwandlung
- Kontrolle und Reagenzienaufbereitung
- ERCC-Mix-Spike-in-Kontrolle: Fügen Sie ERCC-Mix gemäß den Anweisungen des Herstellers hinzu, die die Zugabe von 0,5 μL unverdünnter ERCC-Mischung zu 500 ng mRNA empfehlen. Diese Kontrolle kann helfen, die Effizienz der Bisulfitumwandlung zu beurteilen.
- Spike Poly-A-depletierte RNA (ab Schritt 3.3.14) im Verhältnis 1/1000 (d.h. 500 pg Poly-A-depletierte RNA für 500 ng mRNA). Diese Probe ist mit ribosomaler RNA angereichert und sollte daher die 28S rRNA enthalten, eine Positivkontrolle für die Bisulfit-Umwandlung.
HINWEIS: Gesamt-RNA kann auch als Positivkontrolle anstelle von Poly-A-depletierter RNA verwendet werden. - Führen Sie die Bisulfit-Umwandlung mit einem RNA-Methylierungskit (z. B. Zymo EZ) durch.
- RNA Wash Buffer: Vor Gebrauch 48 ml 100% Ethanol (oder 52 ml 95% Ethanol) zu 12 ml RNA Wash Buffer Konzentrat geben.
- Bisulfit-Umwandlung
HINWEIS: Die Bisulfit-Umwandlung wurde mit einem kommerziell erhältlichen RNA-Bisulfit-Umwandlungskit nach dem unten angegebenen Verfahren des Herstellers durchgeführt.- In 0,2 mL PCR-Röhrchen 1000 ng mRNA (oder zwischen 300 und 1000 ng) hinzufügen. Spike-in-Kontrollen hinzufügen: 1 μL ERCC-Mix (Schritt 8.1.1) und 1000 pg Poly-A-depletierte RNA (Schritt 8.1.2). Komplettes Volumen bis 20 μL mit DNase/RNase-freiem Wasser.
- Fügen Sie 130 μL RNA-Umwandlungsreagenz zu jeder 20 μL RNA-Probe hinzu.
- Mischen Sie die Probe durch Pipettieren auf und ab.
- Drehen Sie kurz nach unten, um sicherzustellen, dass sich keine Tröpfchen in der Kappe oder an den Seiten des Rohrs befinden.
- Legen Sie die PCR-Röhrchen in einen Thermocycler und führen Sie die folgenden Schritte durch: Denaturierung bei 70 °C für 5 min; Umwandlung bei 54 °C für 45 min; Wiederholung der Denaturierungs- und Umwandlungsschritte für insgesamt 3 Zyklen; und dann auf unbestimmte Zeit bei 4 °C halten.
ANMERKUNG: Drei Zyklen der Denaturierung und Bisulfitumwandlung gewährleisten eine vollständige Bisulfitumwandlung der Probe. Proben können bei -80 °C gelagert oder direkt aufbereitet werden. - Fahren Sie mit der Spaltenentschwingung fort. Legen Sie eine Säule in ein leeres Sammelröhrchen und geben Sie 250 μL RNA-Bindungspuffer in die Säule.
- Laden Sie die Probe (~150 μL aus Schritt 8.2.5) in die Säule, die den RNA-Bindungspuffer enthält, und mischen Sie sie durch Pipettieren auf und ab.
- 400 μL 95-100% Ethanol in die Proben-RNA-Bindungspuffermischung in der Säule geben. Schließen Sie die Kappe und mischen Sie sofort, indem Sie die Spalte mehrmals umkehren.
- Zentrifuge mit voller Geschwindigkeit (≥ 10.000 x g) für 30 s. Verwerfen Sie den Durchfluss.
- 200 μL RNA Wash Buffer in die Säule geben und mit voller Geschwindigkeit für 30 s zentrifugieren.
- 200 μL RNA-Desulfonierungspuffer in die Säule geben und 30 min bei Raumtemperatur inkubieren. Nach der Inkubation 30 s mit voller Geschwindigkeit zentrifugieren. Verwerfen Sie den Durchfluss.
- 400 μL RNA Wash Buffer in die Säule geben und mit voller Geschwindigkeit für 30 s zentrifugieren. Wiederholen Sie den Waschschritt mit einem zusätzlichen 400 μL RNA Wash Buffer. Verwerfen Sie den Durchfluss.
- Zentrifugieren Sie die Säule im entleerten Sammelrohr mit voller Geschwindigkeit für 2 min. Übertragen Sie die Säule in ein RNase-freies Rohr.
- Geben Sie ≥ 10 μL DNase/RNase-freies Wasser direkt in die Säulenmatrix und inkubieren Sie für 1 min bei Raumtemperatur. Zentrifuge mit voller Geschwindigkeit für 30 s.
HINWEIS: Wir eluieren normalerweise in einem Volumen von 20 μL. Die eluierte RNA kann sofort verwendet oder bei -20 °C bis zu 3 Monate gelagert werden. Für die Langzeitlagerung bei -80 °C aufbewahren. - Nehmen Sie 2,5 μL für die Bewertung der RNA-Qualität und -Quantität durch den Fragmentanalysator heraus und fahren Sie mit der Bibliotheksvorbereitung und der Hochdurchsatzsequenzierung fort (Schritt 9).
- Nehmen Sie 4 μL konvertierte RNA zur Bisulfit-Umwandlungskontrolle der Effizienz (Schritt 8.3).
- Bisulfit-Konversionskontrolle durch RT-PCR
HINWEIS: Dieser Schritt stellt sicher, dass die Bisulfitkonvertierung erfolgreich war, bevor mit der Sequenzierung fortgefahren wird. 28S ribosomale RNA von Homo sapiens wird als Positivkontrolle für die RNA-Methylierungsanalyse verwendet, da der C-Rest an Position 4447 (GenBank-Beitritt # NR_003287) als 100% methyliert beschrieben wurde.
Primer-Sequenzen:
H 28SF Grundierung: 5'-GGGGTTTTAYGATTTTTTTTTGATTTTTTTGGG-3'
H 28SR Primer: 5'-CCAACTCACRTTCCCTATTAATAAATAAAC-3'- Bereiten Sie den Reverse Transcription (RT) Reaktionsmix mit einem High-Capacity cDNA Reverse Transcription Kit vor. Tauen Sie die Kit-Komponenten auf Eis auf und bereiten Sie den RT-Master-Mix auf Eis wie folgt vor:
4 μL Bisulfit-umgewandelte RNA (ab Schritt 8.2.14):
2 μL 10xRT Puffer
0,8 μL 25x dNTP Mix [100 mM]
2 μL 10x RT Random Primer
1 μL MultiScribe Reverse Transkriptase
1 μL RNase-Inhibitor
9,2 μL nukleasefreies H2O
HINWEIS: Jede RT-Reaktion sollte ein Endvolumen von 20 μL in 0,2 ml PCR-Röhrchen enthalten. - Legen Sie die Rohre mit folgendem RT-Programm in den Thermocycler: 25 °C für 10 min; 37 °C für 120 min; 85 °C für 5 min; dann bei 4 °C auf unbestimmte Zeit.
- Bereiten Sie die PCR-Reaktion vor, um die 28S rRNA mit einem PCR-Korrekturlesezym spezifisch zu amplifizieren. Die Kit-Komponenten auf Eis auftauen, vorsichtig wirbeln und kurz zentrifugieren. Bereiten Sie die PCR-Mastermischung auf Eis oder auf einem eiskalten Metallplattenhalter wie folgt vor:
0,6 μL 10 μM H 28SF Primer
0,6 μL 10 μM H 28SF Primer
6,5 μL der Vorlage cDNA
22,5 μL DNA Polymerase Master Mix
HINWEIS: Jede PCR-Reaktion sollte ein Endvolumen von 20 μL in 0,2 ml PCR-Röhrchen enthalten. - Legen Sie die Röhrchen mit folgendem PCR-Programm in den Thermocycler: Initialdenaturierung bei 95 °C für 5 min; 45 Zyklen der Denaturierung (95 °C für 15 s), des Glühens (57 °C für 30 s) und der Dehnung (72 °C für 15 s), der endgültigen Dehnung bei 72 °C für 10 min und dann des Haltens bei 4 °C auf unbestimmte Zeit.
- 10 μL der Reaktion auf einem 2% igen Agarosegel laufen lassen. Die erwartete Bandgröße beträgt 130 - 200 bp.
- Bereiten Sie den Reverse Transcription (RT) Reaktionsmix mit einem High-Capacity cDNA Reverse Transcription Kit vor. Tauen Sie die Kit-Komponenten auf Eis auf und bereiten Sie den RT-Master-Mix auf Eis wie folgt vor:
- Sequenzierung von PCR-Produkten
- Reinigen Sie die PCR-Produkte mit einer säulenbasierten Methode Ihrer Wahl, um Enzyme und dNTP-Rückstände zu entfernen und die amplifizierte DNA in mindestens 20 μL DNase/RNase-freiem Wasser zu eluieren.
- Quantifizieren Sie die gereinigte DNA mit einem Spektralphotometer.
- Sequenzierungsreaktion
- Verwenden Sie 40 ng PCR-Produkt/Sequenzierungsreaktion.
- Sequenz in beide Richtungen mit den Primern H 28SF und H 28SR.
- Richten Sie die Sequenzen mit der bekannten nicht konvertierten Sequenz (28S ribosomales N5 (RNA28SN5) aus. Prüfen Sie, ob an der Position C4447 ein C-Rückstand vorhanden ist, und an anderer Stelle auf T-Rückstände anstelle von C.
9. Bibliotheksvorbereitung und Hochdurchsatzsequenzierung
- Bereiten Sie Bibliotheken für die Sequenzierung mit mRNA-Kits (z. B. Illumina TruSeq Stranded) vor, starten Sie das Protokoll im Elute-Prime-Fragment-Schritt und befolgen Sie die Anweisungen des Herstellers.
- Für die Eingangsproben RNA-Seq und MeRIP-Seq werden die Proben jedoch 2 min lang bei 80 °C inkubiert, um sie nur zu primen, aber nicht weiter zu fragmentieren.
- Führen Sie die Sequenzierung mit Illumina-Plattformen durch. Sequenzierungsreaktionen können nach Präferenzen und experimentellem Design durchgeführt werden, entweder einzelne oder gepaarte Enden, mit einer Länge von mindestens 100 nt.
10. Bioinformatische Analysen
- m6A Datenverarbeitung
- Führen Sie FASTQC24 aus, um die Lesequalität in m6A zu bewerten und FASTQ-Dateien aus der Sequenzierung einzugeben.
- Führen Sie Atropos25 aus, um minderwertige End- und Adaptersequenzen aus den Lesevorgängen zu entfernen. Legen Sie die folgenden Parameter beim Ausführen von Atropos fest.
- Entfernen Sie die folgenden Adaptersequenzen: AGATCGGAAGAG, CTCTTCCGATCT, AACACTCTTTCCCT, AGATCGGAAGAGCG, AGGGAAAGAGTGTT, CGCTCTTCCGATCT.
- Verwenden Sie den folgenden Phred-Qualitätsgrenzwert: 5, um minderwertige Enden gemäß den Angaben des Herstellers zu trimmen (https://support.illumina.com/downloads/illumina-adapter-sequences-document-1000000002694.html).
- Verwenden Sie nach dem Trimmen die folgende Mindestlesedauer: 25 Basispaare.
- Verschmelzen Sie das GRh38-Humangenom und die HIV-Referenz [Integrated linear pNL4-3Env-GFP] im FASTA-Format.
- Indizieren Sie die zusammengeführte Referenz mit HISAT226.
- Führen Sie HISAT2 für zugeschnittene Lesevorgänge aus, um sie an der indizierten Referenz auszurichten. Verwenden Sie die standardmäßigen HISAT-Parameter.
- Sortieren und indizieren Sie die ausgerichteten Lesevorgänge mit SAMtools27.
- Führen Sie SAMtools stat und Qualimap 228 aus, um die Qualität der sequenzierten Bibliotheken nach der Ausrichtung zu überprüfen.
- Sammeln und fassen Sie optional Qualitätsmaße aus dem vorherigen Schritt mit multiQC29 zusammen.
- Das HIV-Genom hat homologe 634 bp-Sequenzen in der 5' LTR und 3' LTR: Reign multimapping liest von 5' LTR auf die entsprechende 3' LTR-Region mit SAMtools.
- Um die m6A-Peaks zu identifizieren, führen Sie die Peak-Calling-Software MACS230 (v 2.1.2) aus. Wählen Sie die MACS2-Laufparameter sorgfältig aus, um eine korrekte Funktion der RNA-Seq-Daten sicherzustellen, da der Peak-Calling durch das Genexpressionsniveau beeinflusst werden kann und kurze Exons fälschlicherweise als Peaks bezeichnet werden können. Daher muss das Eingangssignal vom m6A-Signal subtrahiert werden, ohne dass MACS2 routinemäßig dnabasierte Daten glättet. Wenden Sie die folgenden Parameter auf den Unterbefehl 'callpeak' von MACS2 an:
-keep-dup auto (steuert das MACS2-Verhalten gegenüber doppelten Lesevorgängen, 'auto' ermöglicht es MACS, die maximale Anzahl von Lesevorgängen an genau derselben Stelle basierend auf der Binomialverteilung zu berechnen, wobei 1e-5 als p-Wert-Cutoff verwendet wird)
-g 2,7e9 (Größe des menschlichen Genoms in bp)
-q 0,01 (minimaler FDR-Grenzwert, um signifikante Spitzen auszurufen)
-nomodel (um den Aufbau des Shifting-Modells zu umgehen, das auf ChIP-Seq-Experimente zugeschnitten ist)
-slokal 0
-llocal 0 (wenn Sie diesen und den vorherigen Parameter auf 0 setzen, kann MACS2 die Eingangslesevorgänge von den m6A-Lesevorgängen direkt subtrahieren, ohne zu glätten)
-extsize 100 (durchschnittliche Länge der Fragmente in bp)
-B - Führen Sie den Differentiellen Peak Calling-Unterbefehl von MACS2, 'bdgdiff', aus, um infizierte und nicht infizierte Samples zu vergleichen. 'bdgdiff' nimmt als Eingaben die bedGraph-Dateien, die von 'callpeak' im vorherigen Schritt generiert wurden. Führen Sie für jeden Zeitpunkt den Vergleich von infizierten und nicht infizierten Proben mit "bdgdiff" durch, subtrahieren Sie das jeweilige Eingangssignal vom m6A-Signal und geben Sie die zusätzlichen Parameter an: -g 60 -l 120.
- m5C Datenverarbeitung
- Führen Sie Cutadapt31 aus, um Adaptersequenzen aus den unformatierten Lesevorgängen mit den folgenden Parametern zu trimmen:
Adapter "AGATCGGAAGAGCACACGTCTGAAC"
-minimum-length=25. - Komplementieren Sie die getrimmten Lesevorgänge mit seqkit32, da das Sequenzierungsprotokoll Lesevorgänge aus dem umgekehrten Strang erzeugt.
- Führen Sie FastQC aus, um die Lesequalität zu überprüfen.
- GRh38 Humangenom und HIV [Integrated linear pNL4-3Env-GFP] Referenz im FASTA-Format zusammenführen.
- Indizieren Sie die zusammengeführte Referenz mit der Anwendung meRanGh aus dem meRanTK-Paket33.
- Richten Sie sich mit den folgenden Parametern an meRanGh aus:
-UN ermöglicht das Schreiben nicht zugeordneter Lesevorgänge in Ausgabedateien
-MM ermöglicht das Schreiben von Multi-Mapped-Lesevorgängen in die Ausgabedatei
-bg für Ausgabe in bedGraph
-mbgc 10 Filter gemeldete Region nach Abdeckung (mindestens 10 Messwerte der Abdeckung) - Das HIV-Genom hat homologe 634 bp-Sequenzen in der 5' LTR und 3' LTR: Richten Sie Multimapping-Messwerte von 5' LTR auf die entsprechende 3' LTR-Region mit SAMtools aus.
- Führen Sie den Methylierungsaufruf über das von meRanTK bereitgestellte MeRanCall-Tool mit den folgenden Parametern aus:
-rl = 126, Leselänge
-ei = 0,1, Fehlerintervall für die Berechnung der Methylierungsrate p-Wert
-cr = 0,99, erwartete Konvertierung - Führen Sie das Dienstprogramm meRanTK estimateSizeFactors.pl aus, um die Größenfaktoren jeder Stichprobe zu schätzen. Die Größenfaktoren werden im nächsten Schritt als Parameter verwendet.
- Führen Sie MeRanCompare für die differentielle Methylierungsanalyse von nicht infizierten vs. infizierten Über die Zeiträume 12, 24 und 36h aus. Die folgenden Parameter werden angewendet: ein Signifikanzwert von .01 als minimaler Schwellenwert für Berichts- und Größenfaktoren aus dem vorherigen Schritt.
- Führen Sie Cutadapt31 aus, um Adaptersequenzen aus den unformatierten Lesevorgängen mit den folgenden Parametern zu trimmen:
Ergebnisse
Dieser Workflow hat sich als nützlich erwiesen, um die Rolle der m6A- und m5C-Methylierung im Zusammenhang mit einer HIV-Infektion zu untersuchen. Dazu haben wir ein CD4+ T-Zelllinienmodell (SupT1) verwendet, das wir entweder mit HIV infizieren oder unbehandelt lassen. Wir begannen den Workflow mit 50 Millionen Zellen pro Zustand und erhielten durchschnittlich 500 μg Gesamt-RNA mit einer RNA-Qualitätszahl von 10 (Abbildung 1A-B). Bei der Poly-A-Selektion haben wir zwischen 10 und 12 μg mRNA pro Zustand (was etwa 2% der gesamten RNA entspricht) gewonnen (Abbildung 1B). Zu diesem Zeitpunkt verwendeten wir 5 μg Poly-A-selektierte RNA für die MeRIP-Seq-Pipeline und 1 μg für die BS-Seq-Pipeline. Da HIV-RNA poly-adenyliert ist, ist keine weitere Aktion erforderlich und MeRIP-Seq- und BS-Seq-Verfahren können direkt angewendet werden.
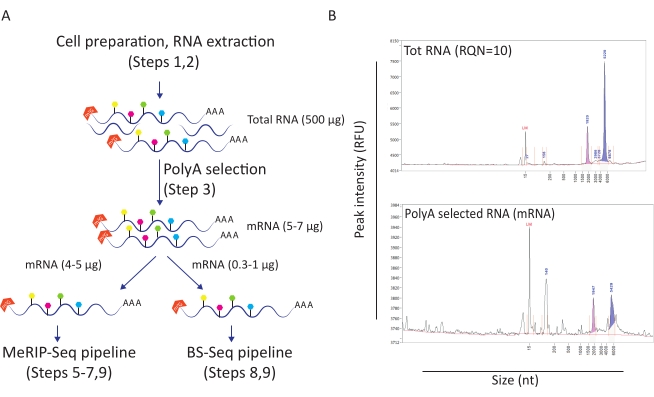
Abbildung 1: RNA-Vorbereitung für nachgeschaltete Anwendungen. A) Workflow zur Darstellung der RNA-Vorbereitung und -Verteilung für gleichzeitige MeRIP-Seq- und BS-Seq-Pipelines. Jede gefüllte sechseckige Form stellt einen RNA-Modifikationstyp dar, z. B. m6A (grün) oder m5C (rosa). Mengen an RNA-Material, die für die Durchführung des Experiments benötigt werden, sind angegeben. B) Repräsentative Ergebnisse, die die erwarteten RNA-Verteilungsprofile (Größe und Menge) bei der Gesamt-RNA-Extraktion (oberes Bild) und der Poly-A-Selektion (unteres Panel) darstellen. Die Proben wurden mit einem Standard-Sensitivitätskit auf den Fragmentanalysator geladen, um die RNA-Qualität zu beurteilen, bevor spezifische MeRIP-Seq- und BS-Seq-Verfahren eingegeben wurden. RQN: RNA-Qualitätszahl; nt: Nukleotide. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Die MeRIP-Seq-Pipeline ist eine auf RNA-Immunpräzipitation basierende Technik, die die Untersuchung der m6A-Modifikation entlang von RNA-Molekülen ermöglicht. Dazu wird RNA zunächst fragmentiert und dann mit m6A-spezifischen Antikörpern inkubiert, die zur Immunpräzipitation und -erfassung an magnetische Kügelchen gekoppelt sind. MeRIP-angereicherte RNA-Fragmente und die unberührte (Input-)Fraktion werden dann sequenziert und verglichen, um m6A-modifizierte RNA-Regionen und damit m6A-methylierte Transkripte zu identifizieren (Abbildung 2A). Die Auflösung der Technik beruht auf der Effizienz der RNA-Fragmentierung. Tatsächlich ermöglichen kürzere Fragmente eine genauere Lokalisierung des m6A-Rückstands. Hier wurden zelluläre Poly-A-selektierte RNAs und virale RNAs während 15 min in einem 20 μL-Endvolumen einer ionenbasierten Fragmentierung mit RNA-Fragmentierungspuffer unterzogen, um RNA-Fragmente von 100-150 nt zu erhalten. Ausgehend von 5 μg mRNA gewannen wir 4,5 μg fragmentierte RNA zurück, was einer Rückgewinnungsrate von 90% entspricht (Abbildung 2B). Wir verwendeten 100 ng fragmentierte, gereinigte RNA als Inputkontrolle, die direkt der Bibliotheksvorbereitung und -sequenzierung unterzogen wurde. Die verbleibende RNA (~4,4 μg) wurde gemäß der MeRIP-Seq-Pipeline verarbeitet, die mit der Inkubation von fragmentierter RNA mit Kügelchen beginnt, die entweder an Anti-m6A-spezifische Antikörper oder an Anti-IgG-Antikörper als Kontrolle gebunden sind. m6A-spezifische RIP (MeRIP) von 2,5 μg fragmentierter RNA ermöglichte die Rückholung von etwa 15 ng m6A-angereichertem Material, das einer Bibliotheksvorbereitung und -sequenzierung unterzogen wurde (Abbildung 2B). RIP mit Anti-IgG-Kontrolle lieferte erwartungsgemäß nicht genügend RNA, um eine weitere Analyse zu ermöglichen (Abbildung 2B).
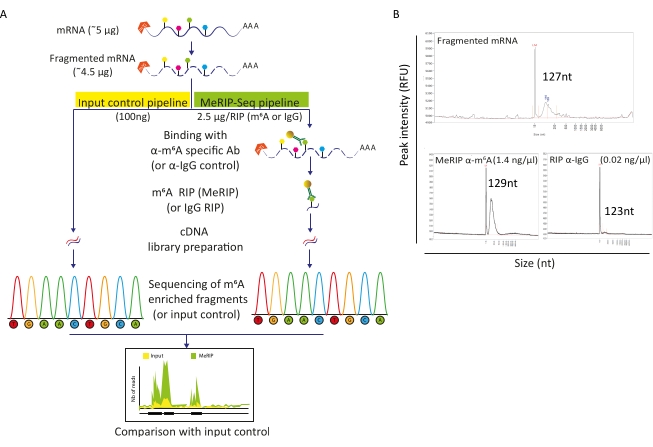
Abbildung 2: MeRIP-Seq-Pipeline. A) Schematische Darstellung des MeRIP-Seq-Workflows und der Eingabesteuerung. Bei der Poly-A-Selektion wurden die Proben in 120-150 nt Stücke fragmentiert und entweder direkt einer Sequenzierung (100 ng, Inputkontrolle) unterzogen oder für die RNA-Immunpräzipitation (2,5 μg, RIP) mit Anti-m6A-spezifischen Antikörpern oder Anti-IgG-Antikörpern als Negativkontrolle vor der Sequenzierung verwendet. B) Repräsentative Ergebnisse, die die erwarteten RNA-Verteilungsprofile (Größe und Menge) bei Fragmentierung (oberes Bild) und RIP (untere Panels, MeRIP: links, IgG-Kontrolle: rechts) zeigen. Die Proben wurden auf den Fragmentanalysator geladen, um die RNA-Qualität und -Konzentration vor der weiteren Verarbeitung zur Bibliotheksvorbereitung und -sequenzierung zu bewerten. Die fragmentierte RNA-Analyse wurde mit dem RNA-Standard-Sensitivitätskit durchgeführt, während immunpräzipitierte RNA das Hochempfindlichkeitskit verwendete. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Die BS-Seq-Pipeline ermöglicht die Erforschung der m5C-RNA-Modifikation bei Nukleotidauflösung und führt zur Identifizierung von m5C-methylierten Transkripten. Bei der Umwandlung von Bisulfit werden nicht methylierte Cytosine in Uracil umgewandelt, während methylierte Cytosine unverändert bleiben (Abbildung 3A). Aufgrund der rauen Bedingungen des Bisulfit-Umwandlungsverfahrens (d. h. hohe Temperatur und niedriger pH-Wert) sind konvertierte mRNAs stark degradiert (Abbildung 3B), was jedoch die Bibliotheksvorbereitung und -sequenzierung nicht beeinträchtigt. Die Bisulfit-Umwandlung ist nur auf einzelsträngiger RNA effizient und kann daher potenziell durch sekundäre doppelsträngige RNA-Strukturen behindert werden. Um die Effizienz der C-U-Konvertierung zu bewerten, haben wir zwei Steuerelemente eingeführt. Als Positivkontrolle nutzten wir das zuvor beschriebene Vorhandensein eines hochmethylierten Cytosins in Position C4447 der 28S rRNA23. Bei der RT-PCR-Amplifikation und Sequenzierung eines 200 bp-Fragments, das die methylierte Stelle umgibt, konnten wir beobachten, dass alle Cytosine erfolgreich in Uracile umgewandelt wurden und dadurch als Thymidine in der DNA-Sequenz auftraten, mit Ausnahme des Cytosins in Position 4447, das unverändert blieb. Als Kontrolle für die Bisulfit-Umwandlungsrate verwendeten wir kommerziell erhältliche synthetische ERCC-RNA-Sequenzen. Diese Mischung besteht aus einem Pool bekannter, nicht methylierter und poly-adenylierter RNA-Sequenzen mit einer Vielzahl von Sekundärstrukturen und -längen. Bei der Bibliotheksvorbereitung und -sequenzierung konzentrierten wir uns auf diese ERCC-Sequenzen, um die Umwandlungsrate zu berechnen, die durchgeführt werden kann, indem die Anzahl der konvertierten C-Rückstände unter den gesamten C-Resten in allen ERCC-Sequenzen und in jeder Probe gezählt wird. Wir erhielten eine Umwandlungsrate von 99,5%, was die Effizienz und den Erfolg der Bisulfit-Umwandlungsreaktion bestätigt (Abbildung 3D).
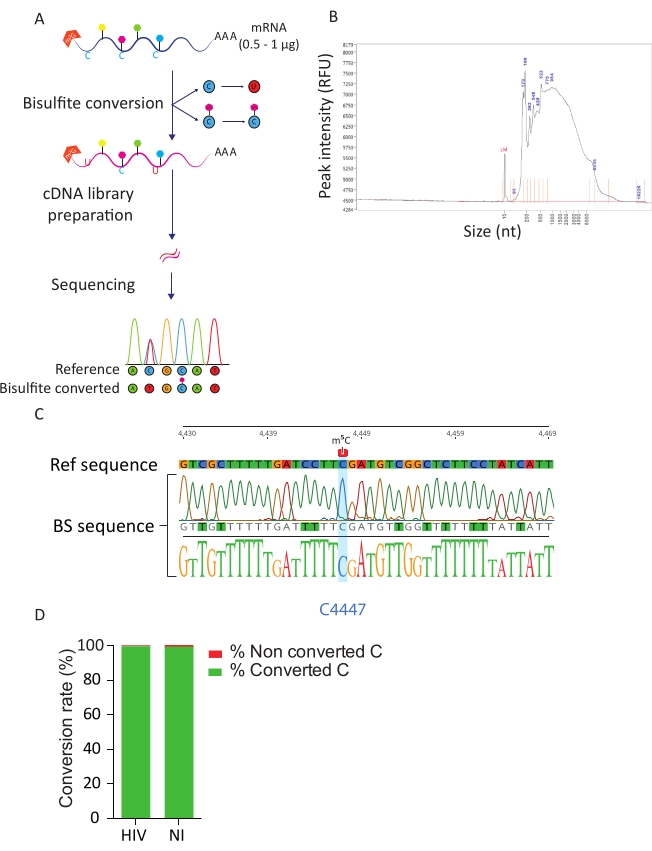
Abbildung 3: BS-Seq-Pipeline. A) Schematische Darstellung des BS-Seq-Workflows. Bei der Poly-A-Selektion werden die Proben Bisulfit ausgesetzt, was zu einer C-zu-U-Umwandlung (aufgrund von Deaminierung) für nicht methylierte C-Reste führt. Im Gegensatz dazu werden methylierte C-Reste (m5C) durch die Bisulfitbehandlung nicht beeinflusst und bleiben unverändert. B) Repräsentatives Ergebnis des in Bisulfit konvertierten RNA-Verteilungsprofils (Größe und Menge) bei der Analyse auf einem Fragmentanalysator mit einem Standard-Sensitivitätskit. C) Elektropherogramm mit repräsentativem Sequenzierungsergebnis des RT-PCR-Amplikons der Region um das 100% methylierte C an Position 4447 in 28S rRNA (blau hervorgehoben). Im Gegensatz dazu wurden C-Reste der Referenzsequenz aufgrund von Bisulfit-Umwandlungserfolgen als T-Reste in der Amplikonsequenz identifiziert. D) Bewertung der C-U-Umwandlungsrate durch Analyse von ERCC-Spike-in-Sequenzen in HIV-infizierten und nicht infizierten Zellen. Die durchschnittliche Conversion Rate liegt bei 99,5%. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
M6A-angereicherte Proben, Bisulfit-konvertierte Proben und Eingangskontrollen werden für die Bibliotheksvorbereitung, Sequenzierung und bioinformatische Analyse weiterverarbeitet (Abbildung 4). Je nach experimentellem Design und der behandelten biologischen Frage(n) können mehrere bioinformatische Analysen angewendet werden. Als Prinzipnachweis zeigen wir hier repräsentative Ergebnisse aus einer möglichen Anwendung (d.h. der differentiellen Methylierungsanalyse), die sich auf die Identifizierung von differentiell methylierten Transkripten konzentriert, die bei einer HIV-Infektion induziert werden. Kurz gesagt, wir untersuchten den m6A- oder m5C-Methylierungsgrad von Transkripten, unabhängig von ihrem Genexpressionsniveau, sowohl in nicht infizierten als auch in HIV-infizierten Zellen, um die Rolle von RNA-Methylierungen während des viralen Lebenszyklus besser zu verstehen. Bei der Normalisierung der Genexpression stellten wir fest, dass das ZNF469-Transkript je nach Infektionsstatus unterschiedlich m6A-methyliert war, tatsächlich war dieses Transkript in nicht infizierten Zellen nicht methyliert, während es bei einer HIV-Infektion mehrere methylierte Peaks aufwies (Abbildung 5A). Eine ähnliche differentielle Methylierungsanalyse an m5C ergab, dass das PHLPP1-Transkript mehrere methylierte Rückstände enthielt, die im HIV-Zustand tendenziell häufiger methyliert werden (Abbildung 5B). In diesem Zusammenhang deuten beide Analysen darauf hin, dass eine HIV-Infektion das zelluläre Epitranskriptom beeinflusst.
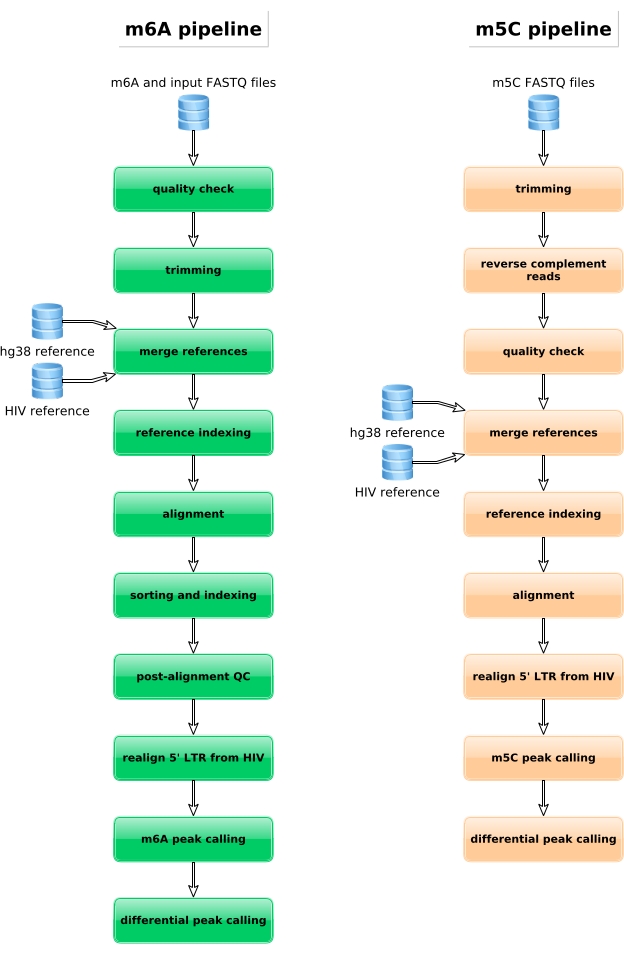
Abbildung 4: Schematische Darstellung des bioinformatischen Workflows für die Analyse von m6A- und m5C-Daten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
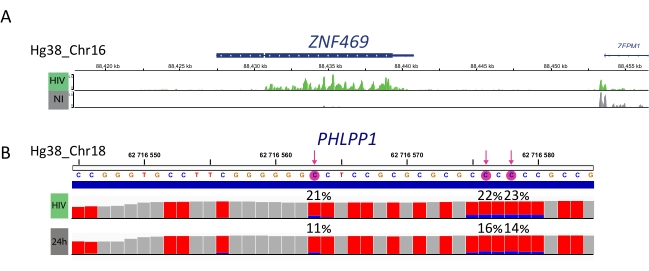
Abbildung 5: Beispiel für differentiell methylierte Transkripte bei der Infektion. A) Repräsentatives Ergebnis, das die m6A-Methylierung des ZNF459-Transkripts in HIV-infizierten (grün) und nicht infizierten (grauen) Zellen zeigt. Die Spitzenintensität (bei Subtraktion der Eingabeexpression) wird auf der y-Achse und der Position im Chromosom entlang der x-Achse angezeigt. Die analyse der differentiellen Methylierung zeigt, dass das ZFN469-Transkript bei einer HIV-Infektion hypermethyliert ist. B) Repräsentatives Ergebnis des m5C-methylierten Gens in HIV-infizierten (obere Spur) und nicht infizierten (untere Spur) Zellen. Die Höhe jedes Balkens stellt die Anzahl der Lesevorgänge pro Nukleotid dar und ermöglicht die Bewertung der Abdeckung. Jeder C-Rest ist rot dargestellt, und der Anteil an methyliertem C ist blau dargestellt. Die genaue Methylierungsrate (%) wird über jedem C-Rückstand angegeben. Pfeile heben statistisch signifikante differentiell methylierte C hervor. Proben wurden mit dem IGV-Viewer visualisiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Die Rolle von RNA-Modifikationen bei der Virusinfektion ist noch weitgehend unbekannt. Ein besseres Verständnis der Rolle epitranskriptomischer Modifikationen im Zusammenhang mit Virusinfektionen könnte zur Suche nach neuen antiviralen Behandlungszielen beitragen.
In dieser Arbeit stellen wir einen kompletten Workflow zur Verfügung, der die Untersuchung der m6A- und m5C-Epitranskriptome infizierter Zellen ermöglicht. Je nach biologischer Fragestellung empfehlen wir, Poly-A-selektierte RNA als Ausgangsmaterial zu verwenden. Obwohl optional, da die Pipeline mit Gesamt-RNA verwendet werden könnte, ist es wichtig zu bedenken, dass sowohl rRNAs als auch kleine RNAs stark modifiziert sind und eine wichtige Anzahl methylierter Rückstände enthalten. Dies könnte zu einer verminderten Qualität und Quantität aussagekräftiger Sequenzierungsdaten führen.
Wenn der Schwerpunkt der Studie jedoch auf nicht-poly-adenylierter RNA liegt, sollte der RNA-Extraktionsschritt angepasst werden, um zu vermeiden, dass kleine RNA verworfen wird (im Falle einer säulenbasierten RNA-Extraktion) und Ribosomen-Depletionstechniken anstelle der Poly-A-Selektion bevorzugt werden, um in die Pipeline zu gelangen.
Um eine hohe Qualität der RNA, eine korrekte Fragmentierung und eine geeignete m6A-angereicherte und BS-konvertierte RNA-Qualität für die Bibliotheksvorbereitung zu gewährleisten, empfehlen wir dringend, einen Fragmentanalysator oder einen Bioanalytiker zu verwenden. Diese Ausrüstung ist jedoch nicht immer verfügbar. Alternativ könnten die Qualität von RNA, mRNA und die Größe der fragmentierten RNA auch durch Visualisierung auf Agarosegel beurteilt werden. Alternativ kann die Bibliotheksvorbereitung ohne vorherige Beurteilung der RNA-Menge durchgeführt werden.
Wir verwendeten die Antikörper-basierte MeRIP-Seq16-Technik, um die epitranskriptomische m6A-Landschaft zu erforschen. Diese Technik basiert auf RNA-Immunpräzipitation und ist erfolgreich; Einige Schritte müssen jedoch sorgfältig optimiert werden und können kritisch sein. Obwohl beschrieben wurde, dass die m6A-Methylierung hauptsächlich innerhalb der Konsensussequenz RRA*CH auftritt, ist dieses Motiv entlang von mRNA-Molekülen sehr häufig und erlaubt keine genaue Identifizierung der methylierten Stelle. Es ist daher entscheidend, eine reproduzierbare und konsistente RNA-Fragmentierung zu erreichen, die kleine RNA-Fragmente erzeugt, um die RIP-basierte Auflösung zu verbessern. In diesem Protokoll empfehlen wir ein optimiertes Verfahren, das reproduzierbare und konsistente Ergebnisse in unserem experimentellen Umfeld liefert; Dieser Fragmentierungsschritt muss jedoch möglicherweise entsprechend bestimmten Beispielfunktionen weiter optimiert werden.
Kürzlich wurde eine neue Technik beschrieben, die eine direkte m6A-Sequenzierung ermöglicht. Es basiert auf der Verwendung spezifischer Reverse-Transkriptase-Varianten, die einzigartige RT-Signaturen als Reaktion auf die Begegnung mit der m6A-RNA-Modifikation aufweisen24. Diese Technologie könnte bei sorgfältiger Optimierung die Haupteinschränkung von MeRIP-Seq umgehen (Verringerung der Menge an Ausgangsmaterial und Ermöglichung einer höheren Auflösung). Um die m5C-Modifikation zu untersuchen, haben wir uns für die Bisulfit-Umwandlungstechnik entschieden, um die modifizierten C-Reste mit Nukleotidauflösung nachzuweisen. Um die Falsch-Positiv-Rate aufgrund des Vorhandenseins von RNA-Sekundärstrukturen zu reduzieren, haben wir 3 Zyklen der Denaturierung / Bisulfit-Umwandlung durchgeführt und die Bisulfit-Umwandlungsrate dank der Verwendung von ERCC-Spike-in-Kontrollen weiter kontrolliert. Eine der Einschränkungen, die mit dieser Technik verbunden sind, besteht darin, dass die Bisulfitkonversion sehr hart ist und drei Zyklen der Denaturierung / Bisulfit-Umwandlung etwas RNA abbauen und somit die Auflösung verringern könnten. In unserer Umgebung haben wir uns jedoch für eine möglicherweise etwas niedrigere Auflösung entschieden, um die Qualität des Datensatzes zu erhöhen.
Dank dieser Optimierungen und Kontrollen konnten wir einen zuverlässigen und soliden Workflow bereitstellen, der genutzt werden kann, um die epitranskriptomische Landschaft und ihre Veränderung im Zusammenhang mit Virusinfektionen, Wirt-Pathogen-Interaktionen oder einer Exposition gegenüber bestimmten Behandlungen zu untersuchen.
Offenlegungen
Die Autoren haben nichts preiszugeben.
Danksagungen
Diese Arbeit wurde vom Schweizerischen Nationalfonds unterstützt (Grants 31003A_166412 und 314730_188877).
Materialien
| Name | Company | Catalog Number | Comments |
| AccuPrime Pfx SuperMix | Invitrogen | 12344-040 | |
| anti-m6A antibody _Clone 17-3-4-1 | Millipore | MABE1006 | |
| Chloroform | Merck | 67-66-3 | |
| ERCC | Invitrogen | 4456740 | |
| EZ RNA Methylation Kit | Zymo Research | EZR5001 | |
| Fragment analyzer RNA Kit - HS RNA Kit | Agilent | DNF-472-0500 | |
| Fragment analyzer RNA Kit - RNA Kit | Agilent | DNF-471-0500 | |
| High-Capacity cDNA Reverse Transcription Kit | Applied Biosystem | 4368814 | |
| Illumina TruSeq Stranded mRNA | Illumina | 20020594 | |
| Magnetic Beads A/G Blend | Merck | 16-663 | |
| N6-Methyladenosine, 5′-monophosphate sodium salt (m6A) | Sigma Aldrich | M2780-10MG | |
| Normal Mouse IgG | Merk | 12371 | |
| Oligo(dT)25 | Life Technologies | 61005, | |
| PCRapace | Stratec | 1020220300 | |
| Quick RNA Viral Kit | Zymo Research | 1034 | |
| RNA Clean & Concentrator | Zymo Research | R1015 | |
| RNA Fragmentation Reagent | Ambion | AM8740 | |
| RNase Inhibitor | Ambion | AM2684 | |
| Trizol | TRIzol Reagent | 15596026 |
Referenzen
- Machnicka, M. A., et al. MODOMICS: a database of RNA modification pathways--2013 update. Nucleic Acids Research. 41, Database issue 262-267 (2013).
- Zaccara, S., Ries, R. J., Jaffrey, S. R. Reading, writing and erasing mRNA methylation. Nature Reviews Molecular Cell Biology. 20 (10), 608-624 (2019).
- Davalos, V., Blanco, S., Esteller, M. SnapShot: Messenger RNA Modifications. Cell. 174 (2), 498(2018).
- Saletore, Y., et al. The birth of the Epitranscriptome: deciphering the function of RNA modifications. Genome Biology. 13 (10), 175(2012).
- Zhao, B. S., Roundtree, I. A., He, C. Post-transcriptional gene regulation by mRNA modifications. Nature Reviews Molecular Cell Biology. 18 (1), 31-42 (2017).
- Netzband, R., Pager, C. T. Epitranscriptomic marks: Emerging modulators of RNA virus gene expression. Wiley Interdisciplinary Reviews: RNA. 11 (3), 1576(2020).
- Pereira-Montecinos, C., Valiente-Echeverria, F., Soto-Rifo, R. Epitranscriptomic regulation of viral replication. Biochimica et Biophysica Acta. 1860 (4), 460-471 (2017).
- Lichinchi, G., et al. Dynamics of the human and viral m(6)A RNA methylomes during HIV-1 infection of T cells. Nature Microbiology. 1, 16011(2016).
- Courtney, D. G., et al. Epitranscriptomic Addition of m(5)C to HIV-1 Transcripts Regulates Viral Gene Expression. Cell Host & Microbe. 26 (2), 217-227 (2019).
- Kennedy, E. M., et al. Posttranscriptional m(6)A Editing of HIV-1 mRNAs Enhances Viral Gene Expression. Cell Host & Microbe. 19 (5), 675-685 (2016).
- Tirumuru, N., Wu, L. HIV-1 envelope proteins up-regulate N (6)-methyladenosine levels of cellular RNA independently of viral replication. Journal of Biological Chemistry. 294 (9), 3249-3260 (2019).
- Tirumuru, N., et al. N(6)-methyladenosine of HIV-1 RNA regulates viral infection and HIV-1 Gag protein expression. Elife. 5, (2016).
- Cristinelli, S., Angelino, P., Janowczyk, A., Delorenzi, M., Ciuffi, A. HIV Modifies the m6A and m5C Epitranscriptomic Landscape of the Host Cell. Frontiers in Virology. 1 (11), (2021).
- Khoddami, V., Cairns, B. R. Transcriptome-wide target profiling of RNA cytosine methyltransferases using the mechanism-based enrichment procedure Aza-IP. Nature Protocols. 9 (2), 337-361 (2014).
- Hussain, S., Aleksic, J., Blanco, S., Dietmann, S., Frye, M. Characterizing 5-methylcytosine in the mammalian epitranscriptome. Genome Biology. 14 (11), 215(2013).
- Dominissini, D., Moshitch-Moshkovitz, S., Salmon-Divon, M., Amariglio, N., Rechavi, G. Transcriptome-wide mapping of N6-methyladenosine by m6A-seq based on immunocapturing and massively parallel sequencing. Nature Protocols. 8 (1), 176-189 (2013).
- Dominissini, D., et al. Topology of the human and mouse m6A RNA methylomes revealed by m6A-seq. Nature. 485 (7397), 201-206 (2012).
- Shobbir Hussain, J. A., Blanco, S., Dietmann, S., Frye, M. Characterizing 5-methylcytosine in the mammalian epitranscriptome. Genome Biology. 14 (215), (2013).
- Amort, T., et al. Distinct 5-methylcytosine profiles in poly(A) RNA from mouse embryonic stem cells and brain. Genome Biology. 18 (1), 1(2017).
- Endrullat, C., Glökler, J., Franke, P., Frohme, M. Standardization and quality management in next-generation sequencing. Applied & Translational Genomics. 10, 2-9 (2016).
- Schaefer, M., Pollex, T., Hanna, K., Lyko, F. RNA cytosine methylation analysis by bisulfite sequencing. Nucleic Acids Research. 37 (2), 12(2009).
- Cristinelli, S., Angelino, P., Janowczyk, A., Delorenzi, M., Ciuffi, A. HIV Modifies the m6A and m5C Epitranscriptomic Landscape of the Host Cell. biorxiv. 1 (11), (2021).
- Squires, J. E., et al. Widespread occurrence of 5-methylcytosine in human coding and noncoding RNA. Nucleic Acids Research. 40 (11), 5023-5033 (2012).
- Aschenbrenner, J., et al. Engineering of a DNA Polymerase for Direct m(6) A Sequencing. Angewandte Chemie (International ed. in English). 57 (2), 417-421 (2018).
- Bioinformatics. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc (2021).
- Didion, J. P., Martin, M., Collins, F. S. Atropos: specific, sensitive, and speedy trimming of sequencing reads. PeerJ. 5, 3720(2017).
- Kim, D., Langmead, B., Salzberg, S. L. HISAT: a fast spliced aligner with low memory requirements. Nature Methods. 12 (4), 357-360 (2015).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Okonechnikov, K., Conesa, A., García-Alcalde, F. Qualimap 2: advanced multi-sample quality control for high-throughput sequencing data. Bioinformatics. 32 (2), Oxford, England. 292-294 (2016).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), Oxford, England. 3047-3048 (2016).
- Zhang, Y., et al. Model-based Analysis of ChIP-Seq (MACS). Genome Biology. 9 (9), 137(2008).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet Journal. 17 (1), (2011).
- Shen, W., Le, S., Li, Y., Hu, F. SeqKit: A Cross-Platform and Ultrafast Toolkit for FASTA/Q File Manipulation. PLOS ONE. 11 (10), 0163962(2016).
- Rieder, D., Amort, T., Kugler, E., Lusser, A., Trajanoski, Z. meRanTK: methylated RNA analysis ToolKit. Bioinformatics. 32 (5), 782-785 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten