Method Article
Esplorare gli epitrascrittomi m6A e m5C sull'infezione virale: un esempio di HIV
In questo articolo
Riepilogo
Il ruolo delle modificazioni dell'RNA nelle infezioni virali sta appena iniziando ad essere esplorato e potrebbe evidenziare nuovi meccanismi di interazione virale-ospite. In questo lavoro, forniamo una pipeline per studiare le modifiche dell'RNA m6A e m5C nel contesto delle infezioni virali.
Abstract
Il ruolo delle modificazioni dell'RNA nei processi biologici è stato al centro di un numero crescente di studi negli ultimi anni ed è oggi noto come epitrascrittomica. Tra gli altri, le modificazioni dell'RNA N6-metiladenosina (m6A) e 5-metilcitosina (m5C) sono state descritte su molecole di mRNA e possono avere un ruolo nella modulazione dei processi cellulari. L'epitrascrittomica è quindi un nuovo livello di regolazione che deve essere considerato in aggiunta alle analisi trascrittomiche, in quanto può anche essere alterato o modulato dall'esposizione a qualsiasi agente chimico o biologico, comprese le infezioni virali.
Qui, presentiamo un flusso di lavoro che consente l'analisi del paesaggio epitrascrittomico cellulare e virale congiunto dei segni m6A e m5C contemporaneamente, in cellule infette o meno con il virus dell'immunodeficienza umana (HIV). Dopo l'isolamento e la frammentazione dell'mRNA da cellule infette da HIV e non infette, abbiamo utilizzato due diverse procedure: MeRIP-Seq, una tecnica basata sull'immunoprecipitazione dell'RNA, per arricchire i frammenti di RNA contenenti il marchio m6A e BS-Seq, una tecnica basata sulla conversione del bisolfito, per identificare il marchio m5C a una singola risoluzione nucleotidica. Dopo l'acquisizione specifica per metilazione, le librerie di RNA vengono preparate per il sequenziamento ad alto rendimento. Abbiamo anche sviluppato una pipeline bioinformatica dedicata per identificare i trascritti differenzialmente metilati (DM) indipendentemente dal loro profilo di espressione basale.
Nel complesso, la metodologia consente l'esplorazione di più segni epitrascrittomici contemporaneamente e fornisce un atlante dei trascritti DM su infezione virale o qualsiasi altra perturbazione cellulare. Questo approccio offre nuove opportunità per identificare nuovi attori e nuovi meccanismi di risposta cellulare, come i fattori cellulari che promuovono o limitano la replicazione virale.
Introduzione
È noto da tempo che le molecole di RNA possono essere modificate e fino ad oggi sono state descritte più di 150 modifiche post-trascrizionali1. Consistono nell'aggiunta di gruppi chimici, principalmente gruppi metilici, praticamente in qualsiasi posizione degli anelli pirimidina e purina delle molecole di RNA2. Tali modificazioni post-trascrizionali hanno già dimostrato di essere altamente arricchite nell'RNA di trasferimento (tRNA) e nell'RNA ribosomiale (rRNA) e sono state recentemente descritte anche su molecole di mRNA.
L'ascesa di nuove tecnologie, come il Next Generation Sequencing (NGS), e la produzione di anticorpi specifici che riconoscono determinate modificazioni chimiche hanno permesso, per la prima volta, di indagare la posizione e la frequenza di specifiche modificazioni chimiche a livello di trascrittoma. Questi progressi hanno portato ad una migliore comprensione delle modificazioni dell'RNA e alla mappatura di diverse modifiche sulle molecole di mRNA3,4.
Mentre l'epigenetica indaga il ruolo delle modifiche del DNA e degli istoni nella regolazione del trascrittoma, l'epitrascrittomica in modo simile si concentra sulle modifiche dell'RNA e sul loro ruolo. Lo studio delle modificazioni epitrascrittomiche offre nuove opportunità per evidenziare nuovi meccanismi di regolazione che possono sintonizzare una varietà di processi cellulari (ad esempio, splicing dell'RNA, esportazione, stabilità e traduzione)5. Non è stata quindi una grande sorpresa che studi recenti abbiano scoperto molte modifiche epitrascrittomiche sull'infezione virale negli RNA sia cellulari che virali6. I virus studiati finora includono sia virus a DNA che a RNA; tra questi, l'HIV può essere considerato un esempio pionieristico. Complessivamente, la scoperta della metilazione dell'RNA nel contesto delle infezioni virali può consentire lo studio di meccanismi ancora non descritti di espressione o replicazione virale, fornendo così nuovi strumenti e bersagli per controllarli7.
Nel campo dell'epitrascrittomica dell'HIV, le modifiche dei trascritti virali sono state ampiamente studiate e hanno dimostrato che la presenza di questa modifica era benefica per la replicazione virale8,9,10,11,12,13. Ad oggi varie tecniche possono essere utilizzate per rilevare segni epitrascrittomici a livello di trascrittoma. Le tecniche più utilizzate per l'identificazione di m6A si basano su tecniche di precipitazione immunitaria come MeRIP-Seq e miCLIP. Mentre MeRIP-Seq si basa sulla frammentazione dell'RNA per catturare frammenti contenenti residui metilati, miCLIP si basa sulla generazione di mutazioni specifiche della firma anticorpale α-m6A sulla reticolazione UV RNA-anticorpo, consentendo così una mappatura più precisa.
Il rilevamento della modifica di m5C può essere ottenuto sia con tecnologie basate su anticorpi simili al rilevamento di m6A (m5C RIP), sia con la conversione del bisolfito o con AZA-IP o con miCLIP. Sia Aza-IP che m5C miCLIP utilizzano una metiltransferasi specifica come esca per colpire l'RNA durante la metilazione dell'RNA. In Aza-IP, le cellule bersaglio sono esposte alla 5-azacitidina, con conseguente introduzione casuale di siti di 5-azacitidina analoga alla citidina nell'RNA nascente. In miCLIP, la NSun2 metiltransferasi è geneticamente modificata per ospitare la mutazione C271A14,15.
In questo lavoro, ci concentriamo sulla doppia caratterizzazione delle modifiche m6A e m5C nelle cellule infette, utilizzando l'HIV come modello. Dopo l'ottimizzazione metodologica, abbiamo sviluppato un flusso di lavoro che combina l'immunoprecipitazione dell'RNA metilato (MeRIP) e la conversione del bisolfito dell'RNA (BS), consentendo l'esplorazione simultanea dei segni epitrascrittomici m6A e m5C a livello di trascrittoma, sia in contesti cellulari che virali. Questo flusso di lavoro può essere implementato su estratti di RNA cellulare e su RNA isolato da particelle virali.
L'approccio MeRIP)16 dell'RNA metilato immunoprecipitazione (MeRIP)16 che consente di studiare m6A a livello di trascrittoma è ben consolidato e una serie di anticorpi specifici per m6A sono disponibili in commercio fino ad oggi17. Questo metodo consiste nella cattura selettiva di pezzi di RNA contenenti m6A utilizzando un anticorpo specifico di m6A. I due principali svantaggi di questa tecnica sono (i) la risoluzione limitata, che dipende fortemente dalle dimensioni dei frammenti di RNA e quindi fornisce una posizione e una regione approssimate contenenti il residuo metilato, e (ii) la grande quantità di materiale necessaria per eseguire l'analisi. Nel seguente protocollo ottimizzato, abbiamo standardizzato la dimensione del frammento a circa 150 nt e ridotto la quantità di materiale di partenza da 10 μg di RNA poli-A-selezionato, che è attualmente la quantità consigliata di materiale di partenza, a solo 1 μg di RNA poli-A-selezionato. Abbiamo anche massimizzato l'efficienza di recupero dei frammenti di RNA m6A legati a anticorpi specifici utilizzando un approccio di eluizione mediante competizione con un peptide m6A invece di metodi di eluizione più convenzionali e meno specifici utilizzando tecniche a base di fenolo o proteinasi K. Il principale limite di questo test basato su RIP, tuttavia, rimane la risoluzione non ottimale che non consente l'identificazione del nucleotide A modificato preciso.
L'analisi del marchio m5C può essere attualmente eseguita utilizzando due diversi approcci: un metodo basato su RIP con anticorpi specifici per m5C e la conversione del bisolfito di RNA. Poiché il RIP offre solo una risoluzione limitata sull'identificazione del residuo metilato, abbiamo utilizzato la conversione del bisolfito che può offrire una risoluzione a singolo nucleotide. L'esposizione dell'RNA al bisolfito (BS) porta alla deaminazione della citosina, convertendo così il residuo di citosina in uracile. Così, durante la reazione di conversione del bisolfito dell'RNA, ogni citosina non metilata viene deaminata e convertita in uracile, mentre la presenza di un gruppo metilico in posizione 5 della citosina ha un effetto protettivo, prevenendo la deaminazione indotta da BS e preservando il residuo di citosina. L'approccio basato su BS consente il rilevamento di un nucleotide modificato m5C a risoluzione a base singola e la valutazione della frequenza di metilazione di ciascun trascritto, fornendo approfondimenti sulla dinamica di modifica di m5C18. Il limite principale di questa tecnica si basa tuttavia sul tasso di falsi positivi dei residui metilati. Infatti, la conversione BS è efficace sull'RNA a singolo filamento con residui di C accessibili. Tuttavia, la presenza di una struttura secondaria di RNA stretta potrebbe mascherare la posizione N5C e ostacolare la conversione BS, con conseguente residuo C non metilato che non viene convertito in residui U, e quindi falsi positivi. Per aggirare questo problema e ridurre al minimo il tasso di falsi positivi, abbiamo applicato 3 cicli di denaturazione e conversione del bisolfito19. Abbiamo inoltre introdotto 2 controlli nei campioni per consentire la stima dell'efficienza di conversione del bisolfito: abbiamo inserito controlli di sequenziamento ERCC (sequenze standardizzate non metilate e disponibili in commercio)20 e RNA impoveriti di poli-A per valutare il tasso di conversione del bisolfito da un lato e per verificare mediante RT-PCR la presenza di un sito metilato noto e ben conservato, C4447, sull'RNA ribosomiale 28S invece21.
Nel campo della virologia, l'accoppiamento di questi due metodi di indagine epitrascrittomica con il sequenziamento di nuova generazione e un'accurata analisi bioinformatica consente lo studio approfondito delle dinamiche m6A e m5C (cioè i cambiamenti temporali di modifica dell'RNA che potrebbero verificarsi in seguito all'infezione virale e potrebbero scoprire una serie di nuovi bersagli terapeuticamente rilevanti per uso clinico).
Protocollo
1. Preparazione delle cellule
NOTA: a seconda del tipo di cellula e del suo contenuto di RNA, il numero iniziale di cellule può variare.
- Avere abbastanza cellule per ottenere tra 200-500 μg di RNA totale o 5-7 μg di RNA poli-A-selezionato. Ad esempio, 50 x 106 cellule SupT1 dovrebbero produrre circa 500 μg di RNA totale dopo l'estrazione con reagenti a base di fenolo, ed è quindi necessario per ogni singola condizione testata.
- Preparare il numero richiesto di cellule secondo il disegno sperimentale, e quindi in base al numero di condizioni testate (infezione, timepoint, trattamento). Se l'esperimento mira a ottenere cellule non infette e cellule infette da HIV a 24 ore dopo l'infezione, è necessario un totale di 100 x 106 cellule, metà per la condizione non infetta e metà per la condizione infetta.
2. Estrazione dell'RNA
- Dalle cellule: estrazione dell'RNA con fenolo-cloroformio
- Per ogni condizione, raccogliere le cellule (ad esempio, 50 x 106) mediante centrifugazione ed eliminare il surnatante.
- Aggiungere 5 ml di reagente a base di fenolo a ogni pellet di celle 50 x 106 e mescolare tubando su e giù più volte.
- Incubare per 5 minuti a temperatura ambiente per consentire la completa lisi. Le celle lisate possono essere conservate a -80 °C o lavorate direttamente.
NOTA: Se necessario, le cellule possono anche essere divise in aliquote di 10 x 106 celle per tubo in tubi da 1,5 ml e lisate in 1 mL di reagente a base di fenolo per una conservazione più comoda. - Aggiungere 1 mL di cloroformio e mescolare per inversione.
- Incubare per 3 minuti a temperatura ambiente.
- Centrifuga per 15 min a 2.000 x g e 4 °C.
- Pipettare la fase acquosa (fase superiore) e trasferirla in un nuovo tubo. Terminare il trasferimento della fase acquosa angolando il tubo a 45° e pipettando accuratamente la soluzione.
NOTA: La quantità di fase acquosa può variare tra i campioni, ma deve essere vicina alla quantità di cloroformio aggiunta al campione (cioè 1 mL). Non trasferire alcun interfase o strato organico! L'uso di tubi phase-lock o phase-maker può facilitare questo processo. - Aggiungere 0,5 ml di isopropanolo di grado molecolare al 100% alla fase acquosa.
- Incubare per 1 ora a -80 °C per consentire la precipitazione dell'RNA.
- Centrifugare per 10 minuti a 12.000 x g e 4 °C per pellettizzare l'RNA precipitato.
- Scartare il surnatante e risospesare il pellet di RNA in 1 mL di etanolo di grado di biologia molecolare al 75%. Vortice brevemente.
- Centrifugare per 5 minuti a 7.500 x g e 4 °C ed eliminare il surnatante.
- Asciugare all'aria il pellet per 15 min.
- Risospesciare il pellet in 20 μL di acqua priva di RNasi e trasferirlo in un nuovo tubo.
- Lavare il tubo vuoto con ulteriori 20 μL di acqua per massimizzare il recupero dell'RNA e raggruppare con il primo volume di 20 μL.
- Quantificare l'RNA totale con uno spettrofotometro e valutare la qualità dell'RNA con un analizzatore di frammenti.
- Da particelle virali: estrazione di RNA con kit di estrazione dell'RNA virale basato su colonna
NOTA: l'estrazione di RNA da particelle virali con reagente a base di fenolo si traduce in RNA virale di bassa qualità e in librerie di qualità inferiore. Un'estrazione di RNA basata su colonne dovrebbe quindi essere favorita. I kit di estrazione dell'RNA che utilizzano l'RNA vettore per l'eluizione e il recupero dell'RNA non sono appropriati per questa procedura e devono essere evitati. Poiché l'HIV RNA è poli-adenilato, l'estrazione diretta dell'RNA senza ulteriore isolamento dell'mRNA è sufficiente per entrare nelle pipeline MeRIP-Seq e BS-Seq. Normalmente 1-2 ml di surnatante virale da cellule universalmente infette dovrebbero fornire abbastanza RNA per eseguire l'intero flusso di lavoro.- Preparare il tampone aggiungendo 150 μL di beta-mercaptoetanolo a 30 ml di tampone di lisi. Ricostituire il tampone di lavaggio virale aggiungendo 96 ml di etanolo al 100%.
- Raccogliere supernatanti contenenti virus e centrifugare per pellet detriti cellulari per ridurre al minimo la contaminazione da RNA cellulare.
- Trasferire 1 mL di surnatante virale in un tubo da 15 mL.
- Aggiungere 3 mL di Viral RNA Buffer a 1 mL di campione virale e mescolare mediante vortice.
- Trasferire 700 μL di campione in una colonna, inserito in un tubo di raccolta.
- Centrifugare per 2 min a 13.000 x g a temperatura ambiente.
- Eliminare il flusso.
- Ripetere i 3 passaggi precedenti fino a quando l'intero campione è stato elaborato, e quindi tutto l'RNA è stato catturato sulla colonna della matrice basata sulla silice.
- Aggiungere 500 μL di viral wash buffer alla colonna.
- Centrifuga per 1 min a 10.000 x g a temperatura ambiente. Eliminare il flusso.
- Aggiungere 200 μL di viral wash buffer alla colonna.
- Centrifuga per 1 min a 10.000 x g a temperatura ambiente. Eliminare il flusso.
- Posizionare la colonna in un tubo di raccolta vuoto.
- Centrifugare per 1 minuto a 10.000 x g a temperatura ambiente per scartare ulteriormente qualsiasi residuo contaminante tampone di lavaggio.
- Trasferire con attenzione la colonna in un tubo da 1,5 ml.
- Aggiungere 20 μL di acqua priva di DNasi/RNasi direttamente al centro della matrice della colonna e centrifugare a 10.000 x g per 30 s a temperatura ambiente.
- Aggiungere altri 10 μL di acqua priva di DNasi/RNasi direttamente al centro della matrice della colonna e centrifugare nuovamente per 30 s.
- Quantificare l'RNA totale con uno spettrofotometro e valutare la qualità dell'RNA con un analizzatore di frammenti.
NOTA: l'estrazione dell'RNA può essere effettuata con qualsiasi metodo, se la qualità dell'RNA recuperato è elevata, con un numero di integrità/qualità dell'RNA > 9. L'RNA totale può essere immagazzinato a -80 °C fino a un'ulteriore elaborazione.
3. Isolamento dell'mRNA mediante selezione poly-A con Oligo(dT)25
NOTA: A causa della presenza di RNA ribosomiale altamente metilato negli estratti cellulari, si raccomanda vivamente di isolare l'RNA poli-A per deplezione di rRNA o preferenzialmente per selezione poli-A positiva. Questo passaggio è facoltativo e deve essere eseguito solo per campioni di RNA cellulare, per ottenere risultati di sequenziamento a risoluzione più elevata. Se si analizza la metilazione di RNA virali non poli-adenilati, favorire l'esaurimento dell'rRNA piuttosto che la selezione poli-A o eventualmente eseguire l'analisi sull'RNA totale.
-
Preparazione del tallone per la cattura poly-A
- Risospesso il flaconcino di perline magnetiche Oligo(dT)25 mediante vortice per >30 s.
- Trasferire 200 μL di perline magnetiche in un tubo da 1,5 mL. Preparare il numero di tubi con perline magnetiche in base alla quantità totale di campioni di RNA da elaborare.
NOTA: Un tubo con 200 μL di soluzione madre Dynabead corrisponde a 1 mg di perline e può ospitare un campione di 75 μg di RNA totale. - Posizionare i tubi su un magnete per 1 minuto ed eliminare il surnatante. Rimuovere i tubi dal magnete.
- Aggiungere 1 mL di binding buffer (20 mM Tris-HCl, pH 7,5, 1,0 M LiCl, 2 mM EDTA) e risospesso mediante vortice. Posizionare i tubi sul magnete per 1 minuto ed eliminare il surnatante. Rimuovere i tubi dal magnete. Ripetere.
- Risospendare le perline magnetiche lavate in 100 μL di Binding Buffer.
-
Preparazione totale dell'RNA
- Diluire l'RNA totale ad una concentrazione finale di 0,75 μg/μL con acqua priva di RNasi, che corrisponde a 75 μg/100 μL.
NOTA: Se l'RNA è ad una concentrazione inferiore, procedere come descritto di seguito senza modificare i volumi. - Aliquotare l'RNA totale in più provette erogando 100 μL di campione di RNA per tubo.
- Aggiungere 100 μL di binding buffer a ciascun campione di RNA.
- Riscaldare l'RNA totale a 65 °C per 2 minuti per interrompere le strutture secondarie.
- Mettere immediatamente sul ghiaccio fino a quando non è pronto per procedere al passaggio successivo.
NOTA: Il tempo di incubazione può variare in base al numero di campioni da elaborare, ma non deve superare 1 ora per evitare qualsiasi degradazione dell'RNA.
- Diluire l'RNA totale ad una concentrazione finale di 0,75 μg/μL con acqua priva di RNasi, che corrisponde a 75 μg/100 μL.
-
Selezione Poly-A
- A ciascun tubo di RNA (dal punto 3.2), aggiungere 100 μL di perline magnetiche lavate (dal passaggio 3.1).
- Mescolare accuratamente con il pipettaggio su e giù e lasciare legare su una ruota rotante a temperatura ambiente per 15 minuti.
- Aprire tutti i tubi, posizionarli sul magnete per 1 minuto e rimuovere con attenzione tutto il surnatante.
- Recuperare il surnatante in un nuovo tubo e tenerlo da parte per un secondo ciclo di cattura dell'RNA (passo 3.3.14), al fine di migliorare il recupero finale poli-A.
- Rimuovere il tubo dal magnete e aggiungere 200 μL di tampone di lavaggio (10 mM Tris-HCl, pH 7,5, 0,15 M LiCl, 1 mM EDTA). Mescolare con cura 4 o 5 volte.
- Posizionare il tubo sul magnete per 1 minuto ed eliminare il surnatante.
- Ripetere la fase di lavaggio una volta (ripetere i passaggi 3.3.5 e 3.3.6).
- Aggiungere 20 μL di Tris-HCl 10 mM ghiacciato per eluire l'RNA poli-A dalle perline.
- Incubare a 80 °C per 2 min.
- Posizionare il tubo sul magnete e trasferire rapidamente il surnatante contenente l'RNA poli-A in un nuovo tubo privo di RNasi. Posizionare il tubo sul ghiaccio.
- Ripetere la fase di eluizione (passaggi da 3.3.8 a 3.3.10) per aumentare la resa.
- Lavare le stesse perline una volta con 200 μL di tampone di lavaggio. Mescolare con cura 4 o 5 volte.
- Posizionare sul magnete per 1 minuto e scartare il tampone di lavaggio.
- Aggiungere il flusso dal passaggio 3.3.4 alle perline e ripetere la procedura dall'associazione all'eluizione (passaggi da 3.3.2 a 3.3.10). Mantenere l'RNA eluato in tubi separati per ora.
NOTA: Facoltativamente, tenere nuovamente il surnatante equivalente al punto 3.3.4 in un nuovo tubo in quanto può essere utilizzato come controllo. Alla fine della procedura, purificare e concentrare l'RNA mediante precipitazione di etanolo o con un metodo di scelta basato su colonna (ad esempio, RNA pulito e concentratore). Questo campione corrisponde a un campione di RNA impoverito di poli-A e può essere utilizzato come controllo per la conversione del bisolfito (fase 8.2.2). - Quantificare l'RNA eluito con uno spettrofotometro e mantenere un'aliquota di 2 μL per valutare ulteriormente la qualità dell'RNA con un analizzatore di frammenti.
NOTA: l'RNA Poly-A può essere conservato a -80 °C fino a quando necessario.
4. Flusso di lavoro dell'RNA
- Dividere i campioni di RNA poli-A cellulare (mRNA) e RNA virale in 2 aliquote, dedicate alla rispettiva pipeline di analisi epitrascrittomica:
(i) 5 μg di mRNA cellulare o 1 μg di RNA virale per MeRIP-Seq e controlli di input (andare ai passaggi da 5 a 7 e al passaggio 9).
(ii) 1 μg di mRNA cellulare o 500 ng di RNA virale per BS-Seq (andare ai passaggi 8 e 9).
5. Frammentazione dell'RNA
NOTA: la frammentazione dell'RNA viene effettuata con il reagente di frammentazione dell'RNA ed è destinata a MeRIP-Seq e campioni di RNA di controllo. Si tratta di un passaggio molto importante che richiede un'attenta ottimizzazione al fine di ottenere frammenti che vanno tra 100-200 nt.
- Dividere il volume totale di mRNA in tubi PCR da 0,2 mL con 18 μL di mRNA/tubo.
NOTA: lavora rapidamente. Non lavorare con più di 8 campioni alla volta per avere risultati riproducibili. L'aumento del volume non garantirà una frammentazione riproducibile e uniforme. - Riscaldare un termociclatore a 70 °C.
- Aggiungere 2 μL di reagente di frammentazione sul bordo di ciascun tubo PCR.
- Chiudere il tubo e ruotare verso il basso (in modo che il reagente entri in contatto con l'RNA contemporaneamente per gli 8 tubi).
- Incubare i campioni 15 min a 70 °C nel termociclatore preriscaldato.
- Non appena l'incubazione è terminata, aggiungere rapidamente 2 μL di soluzione Stop in ciascun tubo.
- Girare verso il basso e lasciare riposare sul ghiaccio fino a quando non si è pronti a procedere al passaggio successivo.
NOTA: Il tempo di incubazione può variare in base al numero di campioni da elaborare, ma non deve superare 1 ora per evitare qualsiasi degradazione dell'RNA. - Ripetere la procedura per tutti i campioni (se ci sono più di 8 aliquote).
- Raggruppare i tubi insieme e procedere alla purificazione dell'RNA con un kit di pulizia e concentratore di RNA (fase 6) o qualsiasi kit personalizzato basato su colonne per eliminare i buffer e recuperare l'RNA frammentato pulito in acqua.
6. Purificazione dell'RNA
NOTA: Questa fase può essere eseguita mediante precipitazione di etanolo o con qualsiasi tipo di metodo di purificazione e concentrazione dell'RNA basato su colonna (ad esempio, RNA Clean e Concentrator).
- Eluire o risospendare l'RNA purificato in un volume totale di 50-75 μL di acqua priva di DNasi/RNasi.
NOTA: se si utilizza un metodo basato su colonne, si consiglia vivamente di eseguire due cicli di eluizione per garantire il massimo recupero. - Quantificare l'mRNA frammentato purificato con uno spettrofotometro e valutare la qualità dell'RNA con un analizzatore di frammenti.
- Mantenere 100 ng di mRNA frammentato come controllo di input per la preparazione e il sequenziamento della libreria (andare al passaggio 9). L'mRNA frammentato rimanente (minimo 2,5 μg) può essere utilizzato per MeRIP (andare al passaggio 7.2).
7. MeRIP
NOTA: Per ogni immunoprecipitazione (IP) è richiesto un minimo di 2,5 μg di mRNA frammentato, utilizzando uno specifico anticorpo anti-m6A (condizione di prova) o un anticorpo anti-IgG (controllo negativo).
- Preparazione del tallone magnetico per l'immunoprecipitazione
- Per ogni campione, preparare 4 mL di 1x tampone IP in un nuovo tubo conico diluendo 800 μL di tampone IP mRNA 5x (50 mM Tris-HCl pH 7,4, 750 mM NaCl, 0,5% Igepal CA-630 e acqua priva di nucleasi) con 3,2 mL di acqua priva di nucleasi.
NOTA: Sono necessarie almeno 2 reazioni (un test e un controllo IgG). - Posizionare il tubo sul ghiaccio.
- Etichettare il numero appropriato di tubi microcentrifuga da 1,5 mL per il numero di reazioni IP desiderate:
n provette (test) per l'anticorpo anti-m6A.
n tubi (controllo negativo) per Normal Mouse IgG. - Risospendare le perle magnetiche (ad esempio, Magna ChIP Protein A/G ) invertendo e vorticando. Nessun ciuffo di perline dovrebbe essere visibile.
- Per ogni reazione pianificata, trasferire 25 μL di perline magnetiche a un tubo microcentrifuga.
- Aggiungere dieci volte più buffer IP 1x (dal passaggio 7.1.1) rispetto al volume originale di perline utilizzate (ad esempio, 250 μL di 1x buffer IP per 25 μL di perline magnetiche).
- Mescolare le perline pipettando delicatamente su e giù più volte per una completa risospensione.
- Posizionare il tubo sul separatore magnetico per 1 min.
- Rimuovere ed eliminare il surnatante, assicurandosi di non aspirare perline magnetiche. Rimuovere il tubo dal magnete.
- Ripetere la fase di lavaggio (passaggi da 7.1.6 a 7.1.9).
- Sospendere le perline in 100 μL di 1x IP Buffer per 25 μL di volume originale di perline magnetiche.
- Aggiungere 5 μL di anticorpi (1 μg/μL) per 25 μL di volume originale di perline magnetiche.
n provette (test) con anticorpo anti-m6A (clone 17-3-4-1) [1 μg/μL].
n tubi (controllo negativo) con Normal Mouse IgG (1 μg/μL). - Incubare sulla ruota rotante per 30 minuti a temperatura ambiente per consentire la coniugazione degli anticorpi con le perle magnetiche.
- Posizionare il tubo sul separatore magnetico per 1 min. Scartare il surnatante. Rimuovere il tubo dal magnete e risospesciare la miscela anticorpo-perline in 100 μL di 1x IP Buffer.
- Per ogni campione, preparare 4 mL di 1x tampone IP in un nuovo tubo conico diluendo 800 μL di tampone IP mRNA 5x (50 mM Tris-HCl pH 7,4, 750 mM NaCl, 0,5% Igepal CA-630 e acqua priva di nucleasi) con 3,2 mL di acqua priva di nucleasi.
- Immunoprecipitazione dell'RNA (RIP)
- Preparare 500 μL di miscela di reazione RIP per ogni campione di mRNA da 2,5 μg come segue: 2,5 μg in 100 μL di RNA frammentato (dallo stadio 6.12); 295 μL di acqua priva di nucleasi; 5 μL di 40 U/μL di inibitore della RNasi; e 100 μL di 5x buffer IP.
- Aggiungere 500 μL di miscela di reazione RIP a ciascuna miscela anticorpo-perline (~100 μL dal passaggio 7.1.14). Mescolare delicatamente pipettando più volte per risospescere completamente le perline. Mettere sul ghiaccio.
- Incubare tutti i tubi RIP su una ruota rotante per 2 ore a 4 °C.
- Centrifugare brevemente le reazioni MeRIP per far cadere le goccioline liquide dai lati del cappuccio e del tubo. Posizionare i tubi su un separatore magnetico per 1 min.
- Trasferire il surnatante in un nuovo tubo centrifugo, facendo attenzione a non disturbare le perle magnetiche.
NOTA: Flowthrough può essere mantenuto come controllo per verificare l'efficienza rip (andare al passaggio 7.3.9). - Rimuovere i tubi dal magnete. Lavare le perline aggiungendo 500 μL di buffer IP freddo 1x. Mescolare le perline pipettando delicatamente più volte per risospescere completamente le perline.
- Posizionare i tubi su un separatore magnetico per 1 minuto e scartare il surnatante.
- Ripetere la procedura di lavaggio (passaggi 7.2.6-7.2.7) due volte per un totale di 3 lavaggi.
- Posizionare i tubi sul ghiaccio e procedere immediatamente all'eluizione.
- Eluizione
- Preparare una soluzione di mM m6A sciogliendo 10 mg di N6-metildedenosina, sale sodico 5′-monofosfato (m6A) in 1,3 ml di acqua priva di nucleasi. Preparare aliquote da 150 μL e conservare a -20 °C.
- Per ogni campione (test e controlli): Preparare 225 μL di tampone di eluizione mescolando i seguenti componenti: 45 μL di 5x IP Buffer, 75 μL di 20 mM m6A, 3,5 μL di 40U/μL di inibitore della RNasi 40U/μL e 101,5 μL di acqua priva di nucleasi.
- Aggiungere 100 μL di tampone di eluizione (dal punto 7.3.2) alle perline (dal passaggio 7.2.9). Mescolare delicatamente pipettando più volte per risospendare completamente le perline.
- Incubare tutti i tubi per 1 ora con agitazione continua su un bilanciere a 4 °C.
- Centrifugare brevemente le reazioni RIP per far scorrere le goccioline liquide dai lati del cappuccio e del tubo. Posizionare i tubi su un separatore magnetico per 1 min.
- Trasferire il surnatante contenente frammenti di RNA eluiti in un nuovo tubo microcentrifuga da 1,5 ml. Fare attenzione a non aspirare le perline, in quanto aumenterà il rumore di fondo.
- Ripetere le fasi di eluizione (da 7.3.3 a 7.3.6) aggiungendo nuovamente 100 μL di tampone di eluizione, incubando 1 ora a 4 °C e raccogliendo l'eluato dopo la separazione magnetica.
- Combinare tutti gli eluati dello stesso campione (il volume totale di eluizione deve essere di 200 μL).
- Purificare l'RNA eluito e il flusso attraverso (facoltativo, dal punto 7.2.5) mediante precipitazione di etanolo o con un metodo di scelta basato su colonne (ad esempio, RNA Clean e Concentrator).
- Valuta la quantità e la qualità dell'RNA dei campioni flowthrough ed eluiti con un analizzatore di frammenti utilizzando un kit di rilevamento ad alta sensibilità. Se la qualità dell'RNA è soddisfacente, procedere alla preparazione della libreria e al sequenziamento ad alto rendimento (fase 9).
NOTA: La quantità di RNA recuperata su MeRIP è molto bassa e richiede imperativamente kit di rilevamento ad alta sensibilità per garantire la quantificazione. Se non è disponibile alcun bioanalizzatore, è possibile procedere ciecamente alla preparazione della libreria.
8. Conversione del bisolfito dell'RNA
- Controllo e preparazione dei reagenti
- Controllo spike-in della miscela ERCC: aggiungere la miscela ERCC seguendo le istruzioni del produttore, che raccomandano l'aggiunta di 0,5 μL di miscela ERCC non diluita a 500 ng di mRNA. Questo controllo può aiutare a valutare l'efficienza della conversione del bisolfito.
- Spike Poly-A-depleted RNA (dallo step 3.3.14) con un rapporto 1/1000 (cioè 500 pg di RNA poli-A-impoverito per 500 ng di mRNA). Questo campione è arricchito in RNA ribosomiale e dovrebbe quindi contenere l'rRNA 28S, un controllo positivo per la conversione del bisolfito.
NOTA: L'RNA totale può anche essere usato come controllo positivo invece dell'RNA impoverito di poli-A. - Eseguire la conversione del bisolfito con un kit di metilazione dell'RNA (ad esempio, Zymo EZ).
- Tampone di lavaggio RNA: aggiungere 48 mL di etanolo al 100% (o 52 ml di etanolo al 95%) a 12 mL di concentrato di tampone di lavaggio RNA prima dell'uso.
- Conversione del bisolfito
NOTA: La conversione del bisolfito è stata effettuata con un kit di conversione del bisolfito di RNA disponibile in commercio seguendo la procedura del produttore come indicato di seguito.- Nei tubi PCR da 0,2 ml, aggiungere 1000 ng di mRNA (o tra 300 e 1000 ng). Aggiungere controlli spike-in: 1 μL di miscela ERCC (fase 8.1.1) e 1000 pg di RNA impoverito di poli-A (fase 8.1.2). Volume completo fino a 20 μL con acqua priva di DNasi/RNasi.
- Aggiungere 130 μL di reagente di conversione dell'RNA a ciascun campione di RNA da 20 μL.
- Mescolare il campione tubando su e giù.
- Ruotare brevemente verso il basso per assicurarsi che non ci siano goccioline nel cappuccio o nei lati del tubo.
- Posizionare i tubi PCR in un termociclatore ed eseguire i seguenti passaggi: denaturazione a 70 °C per 5 min; conversione a 54 °C per 45 min; ripetere le fasi di denaturazione e conversione per un totale di 3 cicli; e quindi tenere a 4 °C a tempo indeterminato.
NOTA: Tre cicli di denaturazione e conversione del bisolfito assicurano la completa conversione del bisolfito del campione. I campioni possono essere conservati a -80 °C o lavorati direttamente. - Procedere con la desolfonia in colonna. Posizionare una colonna in un tubo di raccolta vuoto e aggiungere 250 μL di RNA Binding Buffer alla colonna.
- Caricare il campione (~150 μL dal passo 8.2.5) nella colonna contenente il buffer di legame dell'RNA e mescolare mediante pipettaggio su e giù.
- Aggiungere 400 μL di etanolo al 95-100% alla miscela sample-RNA Binding Buffer nella colonna. Chiudere il tappo e mescolare immediatamente invertendo la colonna più volte.
- Centrifuga a piena velocità (≥ 10.000 x g) per 30 s. Eliminare il flusso.
- Aggiungere 200 μL di tampone di lavaggio RNA alla colonna e centrifugare a piena velocità per 30 s.
- Aggiungere 200 μL di RNA Desulphonation Buffer alla colonna e incubare a temperatura ambiente per 30 min. Dopo l'incubazione, centrifugare a piena velocità per 30 s. Eliminare il flusso.
- Aggiungere 400 μL di tampone di lavaggio RNA alla colonna e centrifugare a piena velocità per 30 s. Ripetere la fase di lavaggio con altri 400 μL di tampone di lavaggio dell'RNA. Eliminare il flusso.
- Centrifugare la colonna nel tubo di raccolta svuotato a piena velocità per 2 min. Trasferire la colonna in un tubo privo di RNasi.
- Aggiungere ≥ 10 μL di acqua priva di DNasi/RNasi direttamente alla matrice della colonna e incubare per 1 minuto a temperatura ambiente. Centrifuga a tutta velocità per 30 s.
NOTA: Di solito eluiamo in un volume di 20 μL. L'RNA eluito può essere utilizzato immediatamente o conservato a -20 °C per un massimo di 3 mesi. Per la conservazione a lungo termine, conservare a -80 °C. - Estrarre 2,5 μL per la valutazione dell'analizzatore di frammenti della qualità e della quantità dell'RNA e procedere alla preparazione della libreria e al sequenziamento ad alto rendimento (fase 9).
- Prendi 4 μL di RNA convertito per il controllo dell'efficienza della conversione del bisolfito (fase 8.3).
- Controllo della conversione del bisolfito mediante RT-PCR
NOTA: questo passaggio garantisce che la conversione del bisolfito abbia avuto esito positivo prima di procedere al sequenziamento. L'RNA ribosomiale 28S di Homo sapiens sarà utilizzato come controllo positivo per l'analisi di metilazione dell'RNA, poiché il residuo C in posizione 4447 (adesione GenBank # NR_003287) è stato descritto come metilato al 100%.
Sequenze di primer:
Primer H 28SF: 5'-GGGGTTTTAYGATTTTTTTGATTTTTTGGG-3'
H 28SR primer: 5'-CCAACTCACRTTCCCTATTAATAAATAAAC-3'- Preparare la miscela di reazione a trascrizione inversa (RT) utilizzando un kit di trascrizione inversa cDNA ad alta capacità. Scongelare i componenti del kit sul ghiaccio e preparare la miscela master RT sul ghiaccio come segue:
4 μL di RNA convertito bisolfito (dal passo 8.2.14):
2 μL di buffer 10xRT
0,8 μL di 25x dNTP Mix [100 mM]
2 μL di 10x RT Random Primers
1 μL di trascrittasi inversa MultiScribe
1 μL di inibitore della RNasi
9,2 μL di H2O senza nucleasi
NOTA: Ogni reazione RT deve contenere un volume finale di 20 μL in tubi PCR da 0,2 mL. - Mettere i tubi nel termociclatore con il seguente programma RT: 25 °C per 10 min; 37 °C per 120 min; 85 °C per 5 min; poi a 4 °C a tempo indeterminato.
- Preparare la reazione PCR per amplificare specificamente l'rRNA 28S con un enzima di correzione di bozze PCR. Scongelare i componenti del kit sul ghiaccio, vorticare delicatamente e centrifugare brevemente. Preparare la miscela master PCR su ghiaccio o su un supporto per piastre metalliche ghiacciate come segue:
0,6 μL di primer H 28SF da 10 μM
0,6 μL di primer H 28SF da 10 μM
6,5 μL di Template cDNA
22,5 μL di miscela master di DNA polimerasi
NOTA: Ogni reazione PCR deve contenere un volume finale di 20 μL in tubi PCR da 0,2 mL. - Mettere i tubi nel termociclatore con il seguente programma PCR: denaturazione iniziale a 95 °C per 5 min; 45 cicli di denaturazione (95 °C per 15 s), ricottura (57 °C per 30 s) e allungamento (72 °C per 15 s), allungamento finale a 72°C per 10 min, e poi mantenimento a 4 °C a tempo indeterminato.
- Eseguire 10 μL della reazione su un gel di agarosio al 2%. La dimensione della banda prevista è 130 - 200 bp.
- Preparare la miscela di reazione a trascrizione inversa (RT) utilizzando un kit di trascrizione inversa cDNA ad alta capacità. Scongelare i componenti del kit sul ghiaccio e preparare la miscela master RT sul ghiaccio come segue:
- Sequenziamento dei prodotti PCR
- Purificare i prodotti PCR con un metodo di scelta basato su colonne per rimuovere enzimi e residui di dNTP ed eluire il DNA amplificato in almeno 20 μL di acqua priva di DNasi/RNasi.
- Quantificare il DNA purificato con lo spettrofotometro.
- Reazione di sequenziamento
- Utilizzare 40 ng di prodotto PCR/reazione di sequenziamento.
- Sequenza in entrambe le direzioni con i primer H 28SF e H 28SR.
- Allineare le sequenze con la sequenza nota non convertita (28S ribosomiale N5 (RNA28SN5). Verificare la presenza di un residuo C in posizione C4447 e residui T anziché C altrove.
9. Preparazione della libreria e sequenziamento ad alto throughput
- Preparare le librerie per il sequenziamento utilizzando kit di mRNA (ad esempio, Illumina TruSeq Stranded), avviando il protocollo nella fase Elute-Prime-Fragment e seguendo le istruzioni del produttore.
- Tuttavia, per i campioni di input RNA-Seq e MeRIP-Seq, incubare i campioni a 80 °C per 2 minuti per innescarli solo ma non frammentarli ulteriormente.
- Eseguire il sequenziamento utilizzando le piattaforme Illumina. Le reazioni di sequenziamento possono essere eseguite in base alle preferenze e al design sperimentale, sia singole che accoppiate, con una lunghezza minima di 100 nt.
10. Analisi bioinformatiche
- m6A Trattamento dei dati
- Eseguire FASTQC24 per valutare la qualità di lettura in m6A e inserire i file FASTQ dalla sequenziazione.
- Eseguire Atropos25 per tagliare le sequenze di estremità e adattatori di bassa qualità dalle letture. Impostare i seguenti parametri nell'esecuzione di Atropos.
- Rimuovere le seguenti sequenze di adattatori: AGATCGGAAGAG, CTCTTCCGATCT, AACACTCTTTCCCT, AGATCGGAAGAGCG, AGGGAAAGAGTGTT, CGCTCTTCCGATCT.
- Utilizzare il seguente limite di qualità Phred: 5, per tagliare estremità di bassa qualità come specificato dal produttore (https://support.illumina.com/downloads/illumina-adapter-sequences-document-1000000002694.html).
- Utilizzare la seguente lunghezza minima di lettura dopo il taglio: 25 coppie di basi.
- Unire il genoma umano GRh38 e il riferimento HIV [Integrated linear pNL4-3Env-GFP] in formato FASTA.
- Indicizzare il riferimento unito con HISAT226.
- Eseguite HISAT2 su letture tagliate per allinearle al riferimento indicizzato. Utilizzare i parametri HISAT predefiniti.
- Ordinare e indicizzare le letture allineate con SAMtools27.
- Esegui SAMtools stat e Qualimap 228, per il controllo della qualità post-allineamento delle librerie sequenziate.
- Facoltativamente, raccogliere e riepilogare le misure di qualità del passaggio precedente con multiQC29.
- Il genoma dell'HIV ha sequenze omologhe a 634 bp nel 5' LTR e 3' LTR: il riallineamento multimapping legge da 5' LTR alla corrispondente regione 3' LTR con SAMtools.
- Per identificare i picchi m6A, eseguire il software di chiamata di picco MACS230 (v 2.1.2). Selezionare attentamente i parametri di esecuzione macs2, al fine di garantire il corretto funzionamento dei dati RNA-Seq poiché le chiamate di picco possono essere influenzate dal livello di espressione genica e gli esoni brevi possono essere erroneamente chiamati picchi. Quindi, il segnale di ingresso deve essere sottratto dal segnale m6A, senza la levigatura applicata di routine da MACS2 ai dati basati sul DNA. Applicare i seguenti parametri al sottocomizio 'callpeak' da MACS2:
-keep-dup auto (controlla il comportamento macs2 verso le letture duplicate, 'auto' consente a MACS di calcolare il numero massimo di letture nella stessa identica posizione in base alla distribuzione binomiale utilizzando 1e-5 come cutoff p-value)
-g 2.7e9 (dimensione del genoma umano in bp)
-q 0,01 (cutoff FDR minimo per chiamare picchi significativi)
-nomodel (per bypassare la costruzione del modello di spostamento, che è su misura per gli esperimenti ChIP-Seq)
-slocal 0
-llocal 0 (impostando questo e il parametro precedente su 0 permette a MACS2 di sottrarre direttamente, senza smussare, l'input legge dalle letture m6A)
-extsize 100 (lunghezza media dei frammenti in bp)
-B - Esegui il sottocomando di chiamata di picco differenziale di MACS2, 'bdgdiff' per confrontare campioni infetti e non infetti. 'bdgdiff' prende come input i file bedGraph generati da 'callpeak' nel passaggio precedente. Per ogni punto temporale, eseguire il confronto tra campioni infetti e non infetti con "bdgdiff", sottraendo il rispettivo segnale di ingresso dal segnale m6A e fornendo i parametri aggiuntivi: -g 60 -l 120.
- Elaborazione dati m5C
- Eseguire Cutadapt31 per tagliare le sequenze dell'adattatore dalle letture non elaborate, con i seguenti parametri:
adattatore "AGATCGGAAGAGCACACACGTCTGAAC"
-lunghezza minima=25. - Completare inversamente le letture tagliate usando seqkit32, poiché il protocollo di sequenziamento produce letture dal filo inverso.
- Eseguire FastQC per esaminare la qualità di lettura.
- Unire il genoma umano GRh38 e il riferimento HIV [Integrated linear pNL4-3Env-GFP] in formato FASTA.
- Indicizzare il riferimento unito con l'applicazione meRanGh dal pacchetto meRanTK33.
- Allinearsi con meRanGh con i seguenti parametri:
-UN abilita le letture non mappate da scrivere nei file di output
-MM che consente di scrivere letture multi-mappate nel file di output
-bg per l'uscita in bedGraph
-mbgc 10 filtra la regione segnalata per copertura (almeno 10 letture di copertura) - Il genoma dell'HIV ha sequenze omologhe di 634 bp nel 5' LTR e 3' LTR: riallinea le letture multimapping da 5' LTR alla corrispondente regione LTR 3' con SAMtools.
- Esegui chiamate di metilazione tramite lo strumento meRanCall, fornito da meRanTK, con i seguenti parametri:
-rl = 126, lunghezza di lettura
-ei = 0,1, intervallo di errore per il calcolo del valore p del tasso di metilazione
-cr = 0,99, conversione prevista - Eseguire l'utilità meRanTK estimateSizeFactors.pl per stimare i fattori dimensionali di ciascun campione. I fattori di dimensione verranno utilizzati come parametri nel passaggio successivo.
- Esegui MeRanCompare per l'analisi della metilazione differenziale di non infetti vs infetti nei punti temporali 12, 24 e 36h. Vengono applicati i seguenti parametri: un valore di significatività di .01 come soglia minima per i fattori di reporting e dimensione del passaggio precedente.
- Eseguire Cutadapt31 per tagliare le sequenze dell'adattatore dalle letture non elaborate, con i seguenti parametri:
Risultati
Questo flusso di lavoro si è dimostrato utile per studiare il ruolo della metilazione di m6A e m5C nel contesto dell'infezione da HIV. Per questo, abbiamo usato un modello di linea cellulare T CD4 + (SupT1) che abbiamo infettato con l'HIV o lasciato non trattato. Abbiamo iniziato il flusso di lavoro con 50 milioni di cellule per condizione e ottenuto una media di 500 μg di RNA totale con un numero di qualità dell'RNA di 10 (Figura 1A-B). Dopo la selezione poly-A abbiamo recuperato tra 10 e 12 μg di mRNA per condizione (che rappresenta circa il 2% dell'RNA totale) (Figura 1B). A questo punto, abbiamo utilizzato 5 μg di RNA poli-A-selezionato per la pipeline MeRIP-Seq e 1 μg per la pipeline BS-Seq. Poiché l'HIV RNA è poli-adenilato, non sono necessarie ulteriori azioni e le procedure MeRIP-Seq e BS-Seq possono essere applicate direttamente.
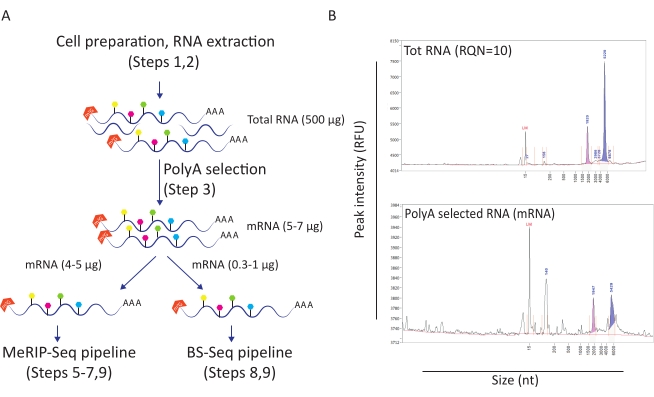
Figura 1: Preparazione dell'RNA per applicazioni a valle. A) Flusso di lavoro raffigurante la preparazione e la distribuzione dell'RNA per pipeline simultanee MeRIP-Seq e BS-Seq. Ogni forma esagonale riempita rappresenta un tipo di modifica dell'RNA, come m6A (verde) o m5C (rosa). Sono indicate le quantità di materiale RNA necessarie per effettuare l'esperimento. B) Risultati rappresentativi che descrivono i profili di distribuzione dell'RNA attesi (dimensioni e quantità) all'estrazione totale dell'RNA (pannello superiore) e la selezione poly-A (pannello inferiore). I campioni sono stati caricati sull'analizzatore di frammenti con kit di sensibilità standard al fine di valutare la qualità dell'RNA prima di entrare in specifiche procedure MeRIP-Seq e BS-Seq. RQN: numero di qualità dell'RNA; nt: nucleotidi. Fare clic qui per visualizzare una versione più grande di questa figura.
La pipeline MeRIP-Seq è una tecnica basata sull'immunoprecipitazione dell'RNA che consente di studiare la modifica di m6A lungo le molecole di RNA. Per questo, l'RNA viene prima frammentato e poi incubato con anticorpi specifici di m6A accoppiati a perline magnetiche per immunoprecipitazione e cattura. I frammenti di RNA arricchiti con MeRIP e la frazione non toccata (input) vengono quindi sequenziati e confrontati per identificare le regioni di RNA modificate da m6A e quindi i trascritti metilati m6A (Figura 2A). La risoluzione della tecnica si basa sull'efficienza della frammentazione dell'RNA. Infatti, frammenti più corti consentono una localizzazione più precisa del residuo m6A. Qui, gli RNA poli-A-selezionati cellulari e gli RNA virali sono stati sottoposti a frammentazione basata su ioni con tampone di frammentazione dell'RNA durante 15 minuti in un volume finale di 20 μL per ottenere frammenti di RNA di 100-150 nt. Partendo da 5 μg di mRNA, abbiamo recuperato 4,5 μg di RNA frammentato, corrispondenti a un tasso di recupero del 90% (Figura 2B). Abbiamo usato 100 ng di RNA frammentato e purificato come controllo di input, sottoposto direttamente alla preparazione e al sequenziamento della libreria. L'RNA rimanente (~4,4 μg) è stato elaborato secondo la pipeline MeRIP-Seq, che inizia con l'incubazione di RNA frammentato con perline legate ad anticorpi specifici anti-m6A o ad anticorpi anti-IgG come controllo. Il RIP specifico per m6A (MeRIP) di 2,5 μg di RNA frammentato ha permesso di recuperare circa 15 ng di materiale arricchito con m6A sottoposto a preparazione e sequenziamento della libreria (Figura 2B). Il RIP con controllo anti-IgG, come previsto, non ha prodotto abbastanza RNA per consentire ulteriori analisi (Figura 2B).
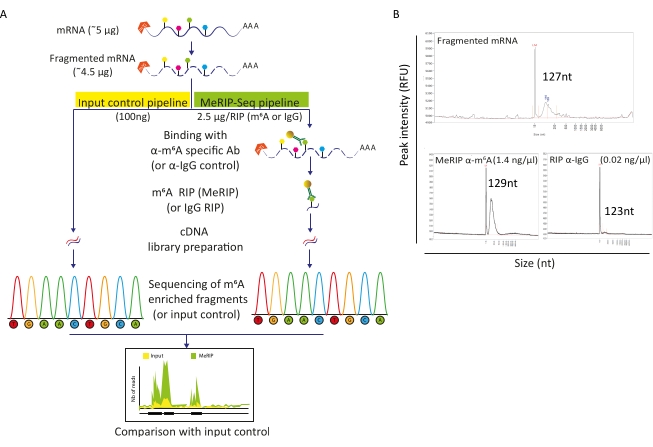
Figura 2: Pipeline MeRIP-Seq. A) Rappresentazione schematica del flusso di lavoro MeRIP-Seq e del controllo dell'input. Dopo la selezione poly-A, i campioni sono stati frammentati in pezzi da 120-150 nt e, direttamente sottoposti a sequenziamento (100 ng, controllo di ingresso), o utilizzati per l'immunoprecipitazione dell'RNA (2,5 μg, RIP) con anticorpo specifico anti-m6A o anticorpo anti-IgG come controllo negativo prima del sequenziamento. B) Risultati rappresentativi che mostrano i profili di distribuzione dell'RNA attesi (dimensioni e quantità) alla frammentazione (pannello superiore) e RIP (pannelli inferiori, MeRIP: sinistra, controllo IgG: destra). I campioni sono stati caricati sull'analizzatore di frammenti per valutare la qualità e la concentrazione dell'RNA prima di un'ulteriore elaborazione per la preparazione e il sequenziamento della libreria. L'analisi dell'RNA frammentato è stata eseguita utilizzando il kit di sensibilità standard dell'RNA mentre l'RNA immunoprecipitato ha utilizzato il kit ad alta sensibilità. Fare clic qui per visualizzare una versione più grande di questa figura.
La pipeline BS-Seq consente l'esplorazione della modifica dell'RNA m5C alla risoluzione nucleotidica e porta all'identificazione di trascritti m5C-metilati. Dopo la conversione del bisolfito, le citosine non metilate vengono convertite in uracile, mentre le citosine metilate rimangono invariate (Figura 3A). A causa delle dure condizioni della procedura di conversione del bisolfito (cioè alta temperatura e basso pH), gli mRNA convertiti sono altamente degradati (Figura 3B), tuttavia ciò non interferisce con la preparazione e il sequenziamento della libreria. La conversione del bisolfito è efficiente solo sull'RNA a singolo filamento e può quindi essere potenzialmente ostacolata da strutture secondarie di RNA a doppio filamento. Per valutare l'efficienza della conversione C-U abbiamo introdotto due controlli. Come controllo positivo, abbiamo sfruttato la presenza precedentemente descritta di una citosina altamente metilata in posizione C4447 dell'rRNA 28S23. Dopo l'amplificazione RT-PCR e il sequenziamento di un frammento di 200 bp che circonda il sito metilato abbiamo potuto osservare che tutte le citosine sono state convertite con successo in uracili, apparendo così come timidine nella sequenza del DNA, ad eccezione della citosina in posizione 4447 che è rimasta invariata. Come controllo per il tasso di conversione del bisolfito, abbiamo utilizzato sequenze di RNA ERCC sintetico disponibili in commercio. Questa miscela consiste in un pool di sequenze di RNA note, non metilate e poli-adenilate, con una varietà di strutture e lunghezze secondarie. Dopo la preparazione e il sequenziamento della libreria, ci siamo concentrati su queste sequenze ERCC per calcolare il tasso di conversione, che può essere eseguito contando il numero di C convertiti tra i residui C totali in tutte le sequenze ERCC e in ciascun campione. Abbiamo ottenuto un tasso di conversione del 99,5%, confermando l'efficienza e il successo della reazione di conversione del bisolfito (Figura 3D).
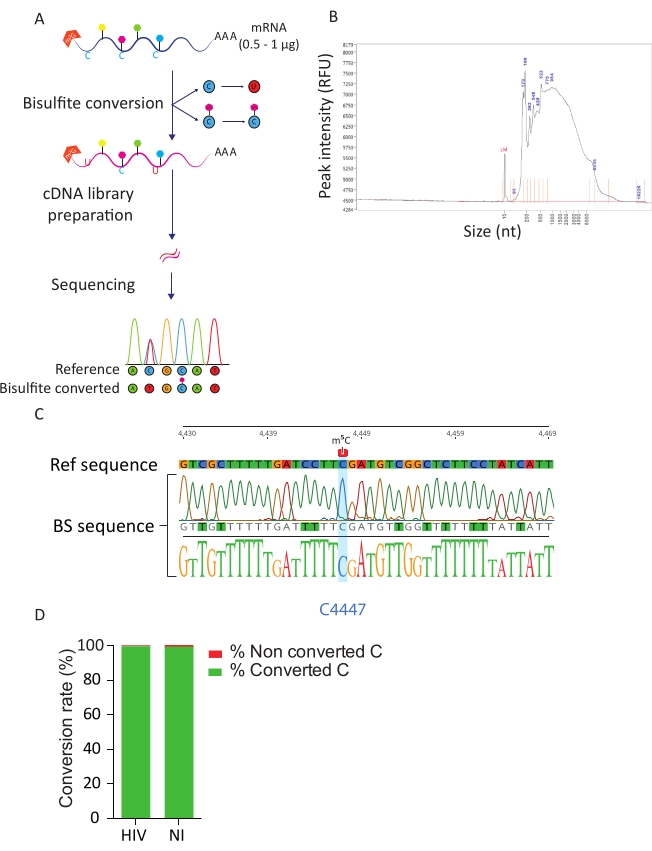
Figura 3: Pipeline BS-Seq. A) Rappresentazione schematica del flusso di lavoro BS-Seq. Dopo la selezione del poli-A, i campioni vengono esposti al bisolfito, con conseguente conversione da C a U (a causa della deaminazione) per i residui C non metilati. Al contrario, i residui C metilati (m5C) non sono influenzati dal trattamento con bisolfito e rimangono invariati. B) Risultato rappresentativo del profilo di distribuzione dell'RNA convertito in bisolfito (dimensioni e quantità) dopo l'analisi su analizzatore di frammenti con un kit di sensibilità standard. C) Elettroferogramma che mostra il risultato rappresentativo del sequenziamento dell'amplicone RT-PCR della regione che circonda il C metilato al 100% in posizione 4447 in rRNA 28S (evidenziato in blu). Al contrario, i residui C della sequenza di riferimento sono stati identificati come residui T nella sequenza amplicon a causa del successo della conversione del bisolfito. D) Valutazione del tasso di conversione C-U mediante analisi di sequenze di spike-in ERCC in cellule infette da HIV e non infette. Il tasso di conversione medio è del 99,5%. Fare clic qui per visualizzare una versione più grande di questa figura.
I campioni arricchiti con M6A, i campioni convertiti in bisolfito e i controlli di input vengono ulteriormente elaborati per la preparazione della libreria, il sequenziamento e l'analisi bioinformatica (Figura 4). In base al disegno sperimentale e alle domande biologiche affrontate, possono essere applicate più analisi bioinformatiche. Come prova di principio qui, mostriamo risultati rappresentativi da una potenziale applicazione (cioè l'analisi di metilazione differenziale), che si concentra sull'identificazione di trascrizioni differenzialmente metilate indotte dall'infezione da HIV. In breve, abbiamo studiato il livello di metilazione m6A o m5C dei trascritti, indipendentemente dal loro livello di espressione genica, sia in cellule non infette che infette da HIV, al fine di comprendere ulteriormente il ruolo delle metilazioni dell'RNA durante il ciclo di vita virale. Dopo la normalizzazione dell'espressione genica, abbiamo identificato che il trascritto ZNF469 era differenzialmente m6A-metilato in base allo stato dell'infezione, infatti questo trascritto non era metilato nelle cellule non infette mentre mostrava diversi picchi metilati sull'infezione da HIV (Figura 5A). Un'analisi di metilazione differenziale simile su m5C ha rivelato che il trascritto PHLPP1 conteneva diversi residui metilati, che tendono ad essere più frequentemente metilati nella condizione di HIV (Figura 5B). In questo contesto, entrambe le analisi suggeriscono che l'infezione da HIV influisce sull'epitrastoma cellulare.
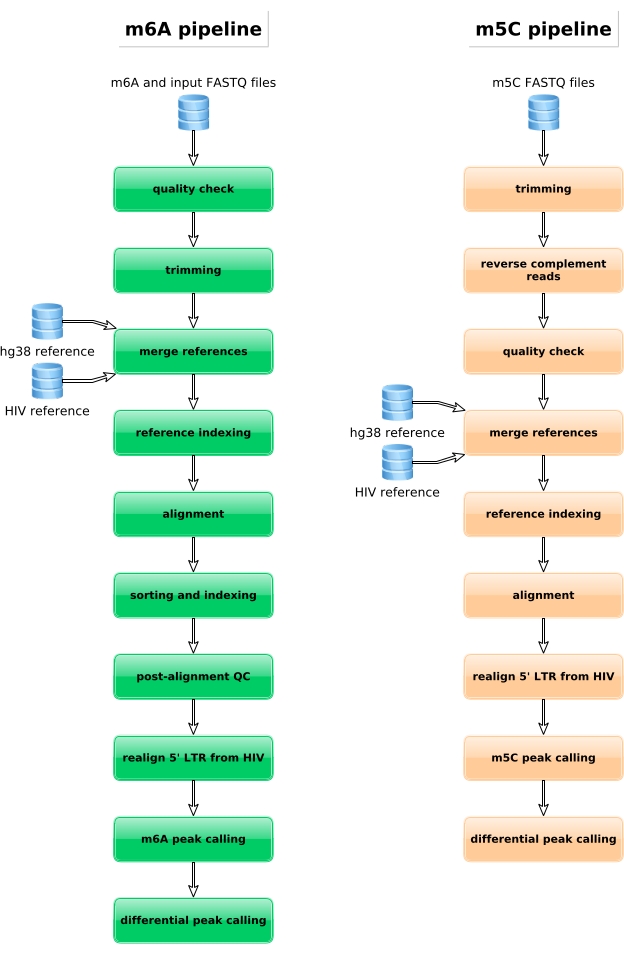
Figura 4: Rappresentazione schematica del flusso di lavoro bioinformatico per l'analisi dei dati m6A e m5C. Fare clic qui per visualizzare una versione più grande di questa figura.
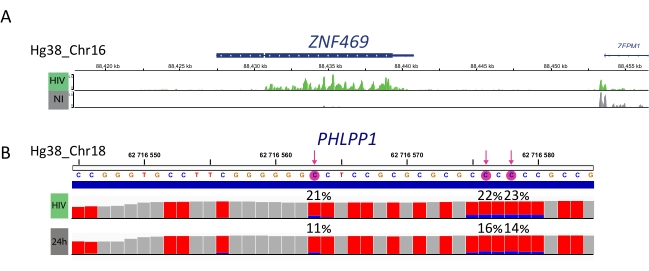
Figura 5: Esempio di trascrizioni differenzialmente metilate in caso di infezione. A) Risultato rappresentativo che mostra la metilazione m6A del trascritto ZNF459 in cellule infette da HIV (verdi) e non infette (grigie). L'intensità di picco (alla sottrazione dell'espressione di input) è mostrata sull'asse y e sulla posizione nel cromosoma lungo l'asse x. L'analisi di metilazione differenziale rivela che il trascritto ZFN469 è ipermetilato dopo l'infezione da HIV. B) Risultato rappresentativo del gene metilato m5C in cellule infette da HIV (corsia superiore) e non infette (corsia inferiore). L'altezza di ogni barra rappresenta il numero di letture per nucleotide e consente la valutazione della copertura. Ogni residuo C è rappresentato in rosso e la proporzione di C metilato è rappresentata in blu. L'esatto tasso di metilazione (%) è riportato al di sopra di ciascun residuo C. Le frecce evidenziano C. I campioni sono stati visualizzati utilizzando il visualizzatore IGV statisticamente significativo. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Il ruolo delle modificazioni dell'RNA nell'infezione virale è ancora in gran parte sconosciuto. Una migliore comprensione del ruolo delle modificazioni epitrascrittomiche nel contesto dell'infezione virale potrebbe contribuire alla ricerca di nuovi bersagli terapeutici antivirali.
In questo lavoro, forniamo un flusso di lavoro completo che consente di indagare gli epitrascrittomi m6A e m5C delle cellule infette. A seconda della domanda biologica, consigliamo di utilizzare l'RNA selezionato poli-A come materiale di partenza. Sebbene facoltativo, poiché la pipeline potrebbe essere utilizzata con l'RNA totale, è importante tenere presente che gli rRNA e i piccoli RNA sono altamente modificati e contengono un numero importante di residui metilati. Ciò potrebbe comportare una diminuzione della qualità e della quantità di dati di sequenziamento significativi.
Tuttavia, se il focus dello studio è l'RNA non poli-adenilato, la fase di estrazione dell'RNA dovrebbe essere adattata per evitare di scartare l'RNA piccolo (in caso di estrazione dell'RNA basata su colonna) e per privilegiare le tecniche di deplezione dei ribosomi piuttosto che la selezione poli-A per entrare nella pipeline.
Al fine di garantire RNA di alta qualità, corretta frammentazione e un'adeguata qualità di RNA arricchito con m6A e BS convertito per la preparazione della libreria, consigliamo vivamente di utilizzare un analizzatore di frammenti o un bioanalizzatore. Tuttavia, questa apparecchiatura non è sempre disponibile. In alternativa, la qualità dell'RNA, dell'mRNA e la dimensione dell'RNA frammentato potrebbero anche essere valutate mediante la visualizzazione su gel di agarosio. In alternativa, la preparazione della libreria può essere eseguita senza previa valutazione della quantità di RNA.
Abbiamo usato la tecnica MeRIP-Seq16 basata su anticorpi per esplorare il panorama epitrascrittomico m6A. Questa tecnica si basa sull'immunoprecipitazione dell'RNA e ha successo; tuttavia, alcuni passaggi richiedono un'attenta ottimizzazione e possono essere critici. Sebbene sia stato descritto che la metilazione m6A avviene principalmente all'interno della sequenza di consenso RRA*CH, questo motivo è molto frequente lungo le molecole di mRNA e non consente l'identificazione precisa del sito metilato. È quindi fondamentale ottenere una frammentazione dell'RNA riproducibile e coerente, generando piccoli frammenti di RNA, per migliorare la risoluzione basata su RIP. In questo protocollo, raccomandiamo una procedura ottimizzata, fornendo risultati riproducibili e coerenti nel nostro ambiente sperimentale; tuttavia, questo passaggio di frammentazione potrebbe richiedere un'ulteriore ottimizzazione in base a specifiche funzionalità di esempio.
Recentemente è stata descritta una nuova tecnica che consente il sequenziamento diretto m6A. Si basa sull'uso di specifiche varianti della trascrittasi inversa che presentano firme RT uniche come risposta all'incontro con la modifica dell'RNA m6A24. Questa tecnologia, dopo un'attenta ottimizzazione, potrebbe aggirare la principale limitazione affrontata da MeRIP-Seq (diminuendo la quantità di materiale iniziale e consentendo una risoluzione più elevata). Per esplorare la modifica m5C abbiamo deciso di utilizzare la tecnica di conversione del bisolfito al fine di rilevare a risoluzione nucleotidica i residui C modificati. Al fine di ridurre il tasso di falsi positivi dovuto alla presenza di strutture secondarie di RNA, abbiamo eseguito 3 cicli di conversione di denaturazione / bisolfito e ulteriormente controllato le prestazioni del tasso di conversione del bisolfito grazie all'uso di controlli spike-in ERCC. Una delle limitazioni legate a questa tecnica è che la conversione del bisolfito è molto dura e tre cicli di conversione di denaturazione/bisolfito potrebbero degradare parte dell'RNA e quindi ridurre la risoluzione. Tuttavia, nella nostra impostazione, abbiamo scelto di accontentarci di una risoluzione potenzialmente leggermente inferiore al fine di aumentare la qualità del set di dati.
Grazie a queste ottimizzazioni e controlli, siamo stati in grado di fornire un flusso di lavoro affidabile e solido che può essere sfruttato per indagare il panorama epitrascrittomico e la sua alterazione nel contesto di infezioni virali, interazioni ospite-patogeno o qualsiasi esposizione a trattamenti specifici.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto dal Fondo nazionale svizzero (sovvenzioni 31003A_166412 e 314730_188877).
Materiali
| Name | Company | Catalog Number | Comments |
| AccuPrime Pfx SuperMix | Invitrogen | 12344-040 | |
| anti-m6A antibody _Clone 17-3-4-1 | Millipore | MABE1006 | |
| Chloroform | Merck | 67-66-3 | |
| ERCC | Invitrogen | 4456740 | |
| EZ RNA Methylation Kit | Zymo Research | EZR5001 | |
| Fragment analyzer RNA Kit - HS RNA Kit | Agilent | DNF-472-0500 | |
| Fragment analyzer RNA Kit - RNA Kit | Agilent | DNF-471-0500 | |
| High-Capacity cDNA Reverse Transcription Kit | Applied Biosystem | 4368814 | |
| Illumina TruSeq Stranded mRNA | Illumina | 20020594 | |
| Magnetic Beads A/G Blend | Merck | 16-663 | |
| N6-Methyladenosine, 5′-monophosphate sodium salt (m6A) | Sigma Aldrich | M2780-10MG | |
| Normal Mouse IgG | Merk | 12371 | |
| Oligo(dT)25 | Life Technologies | 61005, | |
| PCRapace | Stratec | 1020220300 | |
| Quick RNA Viral Kit | Zymo Research | 1034 | |
| RNA Clean & Concentrator | Zymo Research | R1015 | |
| RNA Fragmentation Reagent | Ambion | AM8740 | |
| RNase Inhibitor | Ambion | AM2684 | |
| Trizol | TRIzol Reagent | 15596026 |
Riferimenti
- Machnicka, M. A., et al. MODOMICS: a database of RNA modification pathways--2013 update. Nucleic Acids Research. 41, 262-267 (2013).
- Zaccara, S., Ries, R. J., Jaffrey, S. R. Reading, writing and erasing mRNA methylation. Nature Reviews Molecular Cell Biology. 20 (10), 608-624 (2019).
- Davalos, V., Blanco, S., Esteller, M. SnapShot: Messenger RNA Modifications. Cell. 174 (2), 498 (2018).
- Saletore, Y., et al. The birth of the Epitranscriptome: deciphering the function of RNA modifications. Genome Biology. 13 (10), 175 (2012).
- Zhao, B. S., Roundtree, I. A., He, C. Post-transcriptional gene regulation by mRNA modifications. Nature Reviews Molecular Cell Biology. 18 (1), 31-42 (2017).
- Netzband, R., Pager, C. T. Epitranscriptomic marks: Emerging modulators of RNA virus gene expression. Wiley Interdisciplinary Reviews: RNA. 11 (3), 1576 (2020).
- Pereira-Montecinos, C., Valiente-Echeverria, F., Soto-Rifo, R. Epitranscriptomic regulation of viral replication. Biochimica et Biophysica Acta. 1860 (4), 460-471 (2017).
- Lichinchi, G., et al. Dynamics of the human and viral m(6)A RNA methylomes during HIV-1 infection of T cells. Nature Microbiology. 1, 16011 (2016).
- Courtney, D. G., et al. Epitranscriptomic Addition of m(5)C to HIV-1 Transcripts Regulates Viral Gene Expression. Cell Host & Microbe. 26 (2), 217-227 (2019).
- Kennedy, E. M., et al. Posttranscriptional m(6)A Editing of HIV-1 mRNAs Enhances Viral Gene Expression. Cell Host & Microbe. 19 (5), 675-685 (2016).
- Tirumuru, N., Wu, L. HIV-1 envelope proteins up-regulate N (6)-methyladenosine levels of cellular RNA independently of viral replication. Journal of Biological Chemistry. 294 (9), 3249-3260 (2019).
- Tirumuru, N., et al. N(6)-methyladenosine of HIV-1 RNA regulates viral infection and HIV-1 Gag protein expression. Elife. 5, (2016).
- Cristinelli, S., Angelino, P., Janowczyk, A., Delorenzi, M., Ciuffi, A. HIV Modifies the m6A and m5C Epitranscriptomic Landscape of the Host Cell. Frontiers in Virology. 1 (11), (2021).
- Khoddami, V., Cairns, B. R. Transcriptome-wide target profiling of RNA cytosine methyltransferases using the mechanism-based enrichment procedure Aza-IP. Nature Protocols. 9 (2), 337-361 (2014).
- Hussain, S., Aleksic, J., Blanco, S., Dietmann, S., Frye, M. Characterizing 5-methylcytosine in the mammalian epitranscriptome. Genome Biology. 14 (11), 215 (2013).
- Dominissini, D., Moshitch-Moshkovitz, S., Salmon-Divon, M., Amariglio, N., Rechavi, G. Transcriptome-wide mapping of N6-methyladenosine by m6A-seq based on immunocapturing and massively parallel sequencing. Nature Protocols. 8 (1), 176-189 (2013).
- Dominissini, D., et al. Topology of the human and mouse m6A RNA methylomes revealed by m6A-seq. Nature. 485 (7397), 201-206 (2012).
- Shobbir Hussain, J. A., Blanco, S., Dietmann, S., Frye, M. Characterizing 5-methylcytosine in the mammalian epitranscriptome. Genome Biology. 14 (215), (2013).
- Amort, T., et al. Distinct 5-methylcytosine profiles in poly(A) RNA from mouse embryonic stem cells and brain. Genome Biology. 18 (1), 1 (2017).
- Endrullat, C., Glökler, J., Franke, P., Frohme, M. Standardization and quality management in next-generation sequencing. Applied & Translational Genomics. 10, 2-9 (2016).
- Schaefer, M., Pollex, T., Hanna, K., Lyko, F. RNA cytosine methylation analysis by bisulfite sequencing. Nucleic Acids Research. 37 (2), 12 (2009).
- Cristinelli, S., Angelino, P., Janowczyk, A., Delorenzi, M., Ciuffi, A. HIV Modifies the m6A and m5C Epitranscriptomic Landscape of the Host Cell. biorxiv. 1 (11), (2021).
- Squires, J. E., et al. Widespread occurrence of 5-methylcytosine in human coding and noncoding RNA. Nucleic Acids Research. 40 (11), 5023-5033 (2012).
- Aschenbrenner, J., et al. Engineering of a DNA Polymerase for Direct m(6) A Sequencing. Angewandte Chemie (International ed. in English). 57 (2), 417-421 (2018).
- Didion, J. P., Martin, M., Collins, F. S. Atropos: specific, sensitive, and speedy trimming of sequencing reads. PeerJ. 5, 3720 (2017).
- Kim, D., Langmead, B., Salzberg, S. L. HISAT: a fast spliced aligner with low memory requirements. Nature Methods. 12 (4), 357-360 (2015).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Okonechnikov, K., Conesa, A., García-Alcalde, F. Qualimap 2: advanced multi-sample quality control for high-throughput sequencing data. Bioinformatics. 32 (2), 292-294 (2016).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Zhang, Y., et al. Model-based Analysis of ChIP-Seq (MACS). Genome Biology. 9 (9), 137 (2008).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet Journal. 17 (1), (2011).
- Shen, W., Le, S., Li, Y., Hu, F. SeqKit: A Cross-Platform and Ultrafast Toolkit for FASTA/Q File Manipulation. PLOS ONE. 11 (10), 0163962 (2016).
- Rieder, D., Amort, T., Kugler, E., Lusser, A., Trajanoski, Z. meRanTK: methylated RNA analysis ToolKit. Bioinformatics. 32 (5), 782-785 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon