Method Article
Изучение эпитранскриптомов m6A и m5C при вирусной инфекции: пример с ВИЧ
В этой статье
Резюме
Роль модификаций РНК в вирусных инфекциях только начинает изучаться и может выявить новые механизмы взаимодействия вируса и хозяина. В этой работе мы предоставляем конвейер для исследования модификаций РНК m6A и m5C в контексте вирусных инфекций.
Аннотация
Роль модификаций РНК в биологических процессах была в центре внимания все большего числа исследований в последние несколько лет и в настоящее время известна как эпитранскриптомика. Среди прочего, модификации РНК N6-метиладенозина (m6A) и 5-метилцитозина (m5C) были описаны на молекулах мРНК и могут играть роль в модуляции клеточных процессов. Таким образом, эпитранскриптомика является новым слоем регуляции, который необходимо рассматривать в дополнение к транскриптомному анализу, поскольку он также может быть изменен или модулирован воздействием любого химического или биологического агента, включая вирусные инфекции.
Здесь мы представляем рабочий процесс, который позволяет анализировать совместный клеточный и вирусный эпитранскриптомный ландшафт меток m6A и m5C одновременно, в клетках, инфицированных или не инфицированных вирусом иммунодефицита человека (ВИЧ). При выделении и фрагментации мРНК из ВИЧ-инфицированных и неинфицированных клеток мы использовали две различные процедуры: MeRIP-Seq, метод на основе иммунопреципитации РНК, для обогащения фрагментов РНК, содержащих метку m6A, и BS-Seq, метод на основе конверсии бисульфита, для идентификации метки m5C с разрешением одного нуклеотида. После метилирования-специфического захвата библиотеки РНК подготавливаются для высокопроизводительного секвенирования. Мы также разработали специальный конвейер биоинформатики для идентификации дифференциально метилированных (DM) транскриптов независимо от их базального профиля экспрессии.
В целом, методология позволяет исследовать несколько эпитранскриптомных меток одновременно и предоставляет атлас транскриптов СД при вирусной инфекции или любом другом клеточном возмущении. Этот подход предлагает новые возможности для выявления новых игроков и новых механизмов клеточного ответа, таких как клеточные факторы, способствующие или ограничивающие репликацию вируса.
Введение
Давно известно, что молекулы РНК могут быть модифицированы, и на сегодняшний день описано более 150 посттранскрипционных модификаций1. Они заключаются в добавлении химических групп, главным образом метильных групп, практически к любому положению пиримидиновых и пуриновых колец молекул РНК2. Уже было показано, что такие посттранскрипционные модификации сильно обогащены трансферной РНК (тРНК) и рибосомной РНК (рРНК) и недавно были описаны на молекулах мРНК.
Появление новых технологий, таких как секвенирование следующего поколения (NGS), и производство специфических антител, распознающих определенные химические модификации, позволили впервые исследовать местоположение и частоту конкретных химических модификаций на уровне транскриптома. Эти достижения привели к лучшему пониманию модификаций РНК и картированию нескольких модификаций на молекулах мРНК3,4.
В то время как эпигенетика исследует роль модификаций ДНК и гистонов в регуляции транскриптома, эпитранскриптомика аналогичным образом фокусируется на модификациях РНК и их роли. Исследование эпитранскриптомных модификаций предоставляет новые возможности для выявления новых механизмов регуляции, которые могут настраивать различные клеточные процессы (т.е. сплайсинг, экспорт, стабильность и трансляцию РНК)5. Поэтому неудивительно, что недавние исследования обнаружили множество эпитранскриптомных модификаций при вирусной инфекции как в клеточных, так и в вирусных РНК6. Вирусы, исследованные до сих пор, включают как ДНК, так и РНК-вирусы; среди них ВИЧ можно рассматривать как новаторский пример. В целом, открытие метилирования РНК в контексте вирусных инфекций может позволить исследовать еще не описанные механизмы вирусной экспрессии или репликации, тем самым предоставляя новые инструменты и цели для их контроля7.
В области эпитранскриптомики ВИЧ модификации вирусных транскриптов были широко исследованы и показали, что наличие этой модификации было полезно для репликации вируса8,9,10,11,12,13. На сегодняшний день различные методы могут быть использованы для обнаружения эпитранскриптомных меток на уровне транскриптома. Наиболее часто используемые методы идентификации m6A основаны на методах иммунного осаждения, таких как MeRIP-Seq и miCLIP. В то время как MeRIP-Seq полагается на фрагментацию РНК для захвата фрагментов, содержащих метилированные остатки, miCLIP основан на генерации специфических сигнатурных мутаций антител α-m6A при Сшивании УФ-излучения РНК-антитела, что позволяет более точное картирование.
Обнаружение модификации m5C может быть достигнуто либо с помощью технологий на основе антител, аналогичных обнаружению m6A (m5C RIP), либо путем конверсии бисульфита, либо с помощью AZA-IP, либо с помощью miCLIP. Как Aza-IP, так и m5C miCLIP используют специфическую метилтрансферазу в качестве приманки для нацеливания на РНК при прохождении метилирования РНК. В Aza-IP клетки-мишени подвергаются воздействию 5-азацитидина, что приводит к случайному введению цитидиновых аналоговых 5-азацитидиновых сайтов в зарождающуюся РНК. В miCLIP метилтрансфераза NSun2 генетически модифицирована для содержания мутации C271A14,15.
В этой работе мы фокусируемся на двойной характеристике модификаций m6A и m5C в инфицированных клетках, используя ВИЧ в качестве модели. После методологической оптимизации мы разработали рабочий процесс, который сочетает в себе иммунопреципитацию метилированной РНК (MeRIP) и конверсию бисульфита РНК (BS), что позволяет одновременно исследовать эпитранскриптомные метки m6A и m5C на уровне транскриптома как в клеточном, так и в вирусном контекстах. Этот рабочий процесс может быть реализован на клеточных экстрактах РНК, а также на РНК, выделенной из вирусных частиц.
Подход метилированной РНК ImmunoPrecipitation (MeRIP)16 , позволяющий исследовать m6A на уровне транскриптома, хорошо известен, и на сегодняшний день коммерчески доступен массив m6A-специфических антител17. Этот метод заключается в селективном захвате м6А-содержащих фрагментов РНК с использованием м6А-специфического антитела. Двумя основными недостатками этого метода являются (i) ограниченное разрешение, которое сильно зависит от размера фрагментов РНК и, таким образом, обеспечивает приблизительное местоположение и область, содержащую метилированный остаток, и (ii) большое количество материала, необходимого для выполнения анализа. В следующем оптимизированном протоколе мы стандартизировали размер фрагмента примерно до 150 нт и уменьшили количество исходного материала с 10 мкг поли-А-выбранной РНК, которая в настоящее время является рекомендуемым количеством исходного материала, до всего лишь 1 мкг поли-А-выбранной РНК. Мы также максимизировали эффективность восстановления фрагментов РНК m6A, связанных со специфическими антителами, используя элюирование с помощью конкурентного подхода с пептидом m6A вместо более традиционных и менее специфических методов элюирования с использованием методов на основе фенола или протеиназы K. Однако основным ограничением этого анализа на основе RIP остается неоптимальное разрешение, которое не позволяет идентифицировать точный модифицированный нуклеотид А.
Анализ марки m5C в настоящее время может быть выполнен с использованием двух различных подходов: метода на основе RIP с m5C-специфическими антителами и конверсией РНК-бисульфита. Поскольку RIP предлагает только ограниченное разрешение на идентификацию метилированного остатка, мы использовали бисульфитовую конверсию, которая может предложить разрешение одного нуклеотида. Воздействие РНК бисульфита (BS) приводит к дезаминированию цитозина, тем самым превращая остаток цитозина в урацил. Таким образом, во время реакции превращения бисульфита РНК каждый неметилированный цитозин дезаминируется и превращается в урацил, при этом присутствие метильной группы в положении 5 цитозина оказывает защитное действие, предотвращая дезаминирование, индуцированное BS, и сохраняя остаток цитозина. Подход, основанный на BS, позволяет обнаруживать модифицированный нуклеотид m5C с разрешением одного основания и оценивать частоту метилирования каждого транскрипта, обеспечивая понимание динамики модификации m5C18. Однако основное ограничение этого метода зависит от ложноположительной скорости метилированных остатков. Действительно, конверсия BS эффективна на одноцепочечной РНК с доступными остатками C. Однако наличие плотной вторичной структуры РНК может маскировать положение N5C и препятствовать конверсии BS, что приводит к неметилированным остаткам C, которые не превращаются в остатки U, и, следовательно, ложноположительным результатам. Чтобы обойти эту проблему и свести к минимуму частоту ложных срабатываний, мы применили 3 цикла денатурации и конверсии бисульфитов19. Мы также ввели 2 контрольных элемента в образцах, чтобы можно было оценить эффективность конверсии бисульфита: мы увеличили контроль секвенирования ERCC (неметилированные стандартизированные и коммерчески доступные последовательности)20, а также поли-А-обедненные РНК для оценки скорости конверсии бисульфита, с одной стороны, и для проверки с помощью ОТ-ПЦР наличия известного и хорошо законсервированного метилированного сайта C4447, на 28S рибосомной РНК с другой стороны21.
В области вирусологии соединение этих двух эпитранскриптомных методов исследования с секвенированием следующего поколения и точным биоинформационным анализом позволяет проводить углубленное изучение динамики m6A и m5C (т.е. временных изменений модификации РНК, которые могут произойти при вирусной инфекции и могут выявить множество новых терапевтически значимых целей для клинического использования).
протокол
1. Клеточная подготовка
ПРИМЕЧАНИЕ: В зависимости от типа клетки и содержания в ней РНК, начальное число клеток может варьироваться.
- Иметь достаточно клеток, чтобы получить от 200 до 500 мкг общей РНК или 5-7 мкг поли-А-выбранной РНК. Например, 50 x 106 клеток SupT1 должны давать около 500 мкг общей РНК при экстракции реагентами на основе фенола и, таким образом, необходимы для каждого отдельного тестируемого состояния.
- Подготовьте необходимое количество клеток согласно экспериментальному проекту, и, таким образом, по количеству тестируемых состояний (инфекция, временные точки, лечение). Если эксперимент направлен на получение неинфицированных клеток и ВИЧ-инфицированных клеток через 24 часа после заражения, необходимо в общей сложности 100 x 106 клеток, половина для не0-инфицированного состояния и половина для инфицированного состояния.
2. Экстракция РНК
- Из клеток: экстракция РНК фенол-хлороформом
- Для каждого состояния соберите клетки (например, 50 х 106) путем центрифугирования и выбросьте супернатант.
- Добавьте 5 мл реагента на основе фенола к каждой грануле 50 x 106 клеток и перемешайте путем пипетирования вверх и вниз несколько раз.
- Инкубировать в течение 5 мин при комнатной температуре, чтобы обеспечить полный лизис. Лизированные ячейки могут храниться при -80 °C или обрабатываться напрямую.
ПРИМЕЧАНИЕ: При необходимости клетки также могут быть разделены на аликвоты по 10 х 106 клеток на трубку в пробирках по 1,5 мл и лизированы в 1 мл реагента на основе фенола для более удобного хранения. - Добавить 1 мл хлороформа и перемешать путем инверсии.
- Инкубировать в течение 3 мин при комнатной температуре.
- Центрифуга в течение 15 мин при 2 000 х г и 4 °C.
- Пипетку выводят из водной фазы (верхнюю фазу) и переносят в новую трубку. Закончите перенос водной фазы, зацепив трубку под углом 45° и тщательно выпустив раствор.
ПРИМЕЧАНИЕ: Количество водной фазы может варьироваться между образцами, но должно быть близко к количеству хлороформа, добавленного в образец (т.е. 1 мл). Не переносите никаких межфазных или органических слоев! Использование фазоблокировочных или фазогенераторных трубок может облегчить этот процесс. - Добавьте 0,5 мл 100% молекулярного изопропанола в водную фазу.
- Инкубировать в течение 1 ч при -80 °C, чтобы обеспечить осаждение РНК.
- Центрифугу в течение 10 мин при 12 000 х г и 4 °C до гранулированной осажденной РНК.
- Откажитесь от супернатанта и повторно суспендируйте гранулу РНК в 1 мл этанола 75% класса молекулярной биологии. Вихрь кратко.
- Центрифугу в течение 5 мин при 7,500 х г и 4 °C и выбросьте супернатант.
- Высушите гранулы на воздухе в течение 15 мин.
- Повторно суспендировать гранулы в 20 мкл воды, не содержащей РНКазы, и перенести в новую трубку.
- Промыть пустую трубку дополнительными 20 мкл воды, чтобы максимизировать восстановление РНК, и объединить с первым объемом 20 мкл.
- Количественно оцените общую РНК с помощью спектрофотометра и оцените качество РНК с помощью анализатора фрагментов.
- Из вирусных частиц: экстракция РНК с помощью набора для экстракции вирусной РНК на основе колонки
ПРИМЕЧАНИЕ: Экстракция РНК из вирусных частиц с помощью реагента на основе фенола приводит к низкому качеству вирусной РНК и к библиотекам более низкого качества. Таким образом, следует отдавать предпочтение извлечению РНК на основе столбцов. Наборы для экстракции РНК, использующие несущую РНК для элюирования и восстановления РНК, не подходят для этой процедуры и их следует избегать. Поскольку РНК ВИЧ является полиаденилированной, прямой экстракции РНК без дальнейшего выделения мРНК достаточно для попадания в трубопроводы MeRIP-Seq и BS-Seq. Обычно 1-2 мл вирусного супернатанта из универсально инфицированных клеток должны обеспечить достаточное количество РНК для выполнения всего рабочего процесса.- Готовят буфер, добавляя 150 мкл бета-меркаптоэтанола к 30 мл буфера лизиса. Восстановите буфер вирусной промывки, добавив 96 мл 100% этанола.
- Собирайте вируссодержащие супернатанты и центрифуги в обломки клеток гранул, чтобы свести к минимуму загрязнение клеточной РНК.
- Перенесите 1 мл вирусного супернатанта в пробирку объемом 15 мл.
- Добавьте 3 мл буфера вирусной РНК к 1 мл вирусного образца и перемешайте путем вихря.
- Переложите 700 мкл образца в колонку, вставленную в сборную трубку.
- Центрифуга в течение 2 мин при 13 000 х г при комнатной температуре.
- Отбросьте сквозной поток.
- Повторяйте 3 предыдущих шага до тех пор, пока весь образец не будет обработан, и, таким образом, вся РНК не будет захвачена на матричной колонке на основе кремнезема.
- Добавьте 500 мкл вирусного буфера стирки в колонку.
- Центрифуга в течение 1 мин при 10 000 х г при комнатной температуре. Отбросьте сквозной поток.
- Добавьте 200 мкл вирусного буфера стирки в колонку.
- Центрифуга в течение 1 мин при 10 000 х г при комнатной температуре. Отбросьте сквозной поток.
- Поместите столбец в пустую коллекционную трубку.
- Центрифуга в течение 1 мин при 10 000 х г при комнатной температуре для дальнейшего удаления любого оставшегося загрязняющего вещества в буфере промывки.
- Аккуратно переложите колонну в трубку объемом 1,5 мл.
- Добавьте 20 мкл воды, не содержащей ДНКазы/РНКазы, непосредственно в центр матрицы колонны и центрифугу при 10 000 х г в течение 30 с при комнатной температуре.
- Добавьте еще 10 мкл воды, не содержащей ДНКазы/РНКазы, непосредственно в центр матрицы колонны и снова в центрифугу в течение 30 с.
- Количественно оцените общую РНК с помощью спектрофотометра и оцените качество РНК с помощью анализатора фрагментов.
ПРИМЕЧАНИЕ: Экстракция РНК может быть проведена любым методом, если качество извлеченной РНК высокое, с числом целостности/качества РНК > 9. Общая РНК может храниться при -80 °C до дальнейшей обработки.
3. Выделение мРНК методом поли-А селекции с помощью Oligo(dT)25
ПРИМЕЧАНИЕ: Из-за наличия высокометилированной рибосомной РНК в клеточных экстрактах настоятельно рекомендуется выделять поли-А РНК либо путем истощения рРНК, либо преимущественно путем поли-А положительного отбора. Этот шаг является необязательным и должен выполняться только для образцов клеточной РНК для получения результатов секвенирования с более высоким разрешением. При анализе метилирования неполиаденилированных вирусных РНК отдайте предпочтение истощению рРНК, а не поли-А отбору или в конечном итоге выполните анализ общей РНК.
-
Подготовка бусин к поли-А захвату
- Повторное суспендирование флакона с магнитными шариками Oligo(dT)25 путем вихря в течение >30 с.
- Перенесите 200 мкл магнитных шариков в трубку объемом 1,5 мл. Подготовьте количество пробирок с магнитными шариками в соответствии с общим количеством образцов РНК, подлежащих обработке.
ПРИМЕЧАНИЕ: Одна пробирка с 200 мкл раствора Dynabead соответствует 1 мг шариков и может вместить образец из 75 мкг общей РНК. - Поместите трубки на магнит на 1 мин и выбросьте супернатант. Снимите трубки с магнита.
- Добавьте 1 мл связывающего буфера (20 мМ Tris-HCl, pH 7,5, 1,0 М LiCl, 2 мМ ЭДТА) и повторно суспендируйте путем вихря. Поместите трубки на магнит на 1 мин и выбросьте супернатант. Снимите трубки с магнита. Повторять.
- Повторно суспендируйте промытые магнитные шарики в 100 мкл связывающего буфера.
-
Общий препарат РНК
- Разбавляют общую РНК в конечной концентрации 0,75 мкг/мкл водой без РНКазы, что соответствует 75 мкг/100 мкл.
ПРИМЕЧАНИЕ: Если РНК находится в более низкой концентрации, действуйте так, как описано ниже, не изменяя объемы. - Аликвотирование общей РНК в нескольких пробирках путем дозирования 100 мкл образца РНК на пробирку.
- Добавьте 100 мкл связывающего буфера к каждому образцу РНК.
- Нагревайте общую РНК до 65 °C в течение 2 мин, чтобы разрушить вторичные структуры.
- Поместите сразу на лед, пока не будете готовы перейти к следующему шагу.
ПРИМЕЧАНИЕ: Время инкубации может варьироваться в зависимости от количества образцов, подлежащих обработке, но не должно превышать 1 ч, чтобы избежать деградации РНК.
- Разбавляют общую РНК в конечной концентрации 0,75 мкг/мкл водой без РНКазы, что соответствует 75 мкг/100 мкл.
-
Выбор Poly-A
- К каждой трубке РНК (из стадии 3.2) добавляют 100 мкл промытых магнитных шариков (из шага 3.1).
- Тщательно перемешайте путем пипетки вверх и вниз и оставьте связывание на вращающемся колесе при комнатной температуре в течение 15 мин.
- Откройте все трубки, поместите их на магнит на 1 мин и аккуратно удалите все надотворные вещества.
- Восстановите супернатант в новой трубке и отложите в сторону для второго раунда захвата РНК (шаг 3.3.14), чтобы улучшить окончательное восстановление поли-А.
- Извлеките трубку из магнита и добавьте 200 мкл моющего буфера (10 мМ Tris-HCl, рН 7,5, 0,15 М LiCl, 1 мМ ЭДТА). Перемешайте путем тщательной пипетки 4-5 раз.
- Поместите трубку на магнит на 1 мин и выбросьте супернатант.
- Повторите этап стирки один раз (повторите шаги 3.3.5 и 3.3.6).
- Добавьте 20 мкл ледяного 10 мМ Tris-HCl к элюированию поли-А РНК из шариков.
- Инкубировать при 80 °C в течение 2 мин.
- Поместите трубку на магнит и быстро перенесите супернатант, содержащий поли-А РНК, в новую трубку без РНКазы. Поместите трубку на лед.
- Повторите этап элюирования (шаги 3.3.8-3.3.10), чтобы увеличить выход.
- Вымойте те же бусины один раз с 200 мкл промывочного буфера. Перемешайте путем тщательной пипетки 4-5 раз.
- Поместите на магнит на 1 мин и выбросьте буфер для промывки.
- Добавьте прохождение из шага 3.3.4 к шарикам и повторите процедуру от связывания до элюирования (этапы 3.3.2-3.3.10). Пока держите элюаты РНК в отдельных пробирках.
ПРИМЕЧАНИЕ: При желании снова сохраните супернатант, эквивалентный шагу 3.3.4, в новой трубке, так как его можно использовать в качестве элемента управления. В конце процедуры очищают и концентрируют РНК путем осаждения этанола или с помощью колоночного метода выбора (т.е. РНК чистой и концентраторной). Этот образец соответствует образцу РНК, обедненному поли-А, и может быть использован в качестве контроля для конверсии бисульфита (этап 8.2.2). - Количественно оцените элюированную РНК с помощью спектрофотометра и сохраните аликвоту 2 мкл для дальнейшей оценки качества РНК с помощью анализатора фрагментов.
ПРИМЕЧАНИЕ: Поли-А РНК можно хранить при -80 °C до тех пор, пока это не понадобится.
4. Рабочий процесс РНК
- Разделите образцы клеточной поли-А РНК (мРНК) и вирусной РНК на 2 аликвоты, предназначенные для соответствующего конвейера эпитранскриптомного анализа:
(i) 5 мкг клеточной мРНК или 1 мкг вирусной РНК для MeRIP-Seq и входного контроля (перейдите к шагам 5-7 и шагу 9).
(ii) 1 мкг клеточной мРНК или 500 нг вирусной РНК для BS-Seq (перейдите к шагам 8 и 9).
5. Фрагментация РНК
ПРИМЕЧАНИЕ: Фрагментация РНК осуществляется с помощью реагента фрагментации РНК и предназначена для Образцов MeRIP-Seq и контрольной РНК. Это очень важный шаг, который требует тщательной оптимизации для получения фрагментов в диапазоне 100-200 нт.
- Разделите общий объем мРНК на 0,2 мл ПЦР-пробирок с 18 мРНК/трубкой.
ПРИМЕЧАНИЕ: Работайте быстро. Не работайте с более чем 8 образцами одновременно, чтобы получить воспроизводимые результаты. Увеличение объема не гарантирует воспроизводимой и равномерной фрагментации. - Разогрейте термоциклер при 70 °C.
- Добавьте 2 мкл фрагментационного реагента на край каждой трубки ПЦР.
- Закройте трубку и раскрутите вниз (чтобы реагент одновременно соприкасался с РНК для 8 пробирок).
- Инкубируйте образцы 15 мин при 70 °C в предварительно нагретом термоциклере.
- Как только инкубация закончится, быстро добавьте в каждую пробирку 2 мкл стоп-раствора.
- Повернитесь вниз и дайте посидеть на льду, пока не будете готовы перейти к следующему шагу.
ПРИМЕЧАНИЕ: Время инкубации может варьироваться в зависимости от количества образцов, подлежащих обработке, но не должно превышать 1 ч, чтобы избежать деградации РНК. - Повторите процедуру для всех образцов (если имеется более 8 аликвот).
- Объедините трубки вместе и приступайте к очистке РНК с помощью комплекта чистых и концентраторов РНК (шаг 6) или любого настраиваемого набора на основе колонки, чтобы избавиться от буферов и восстановить чистую фрагментированную РНК в воде.
6. Очистка РНК
ПРИМЕЧАНИЕ: Эта стадия может быть осуществлена путем осаждения этанола или с помощью любого вида метода очистки и концентрации РНК на основе колонки (т.е. РНК Clean and Concentrator).
- Элюируют или повторно суспендируют очищенную РНК в общем объеме 50-75 мкл воды, свободной от ДНКазы/РНКазы.
ПРИМЕЧАНИЕ: Если используется метод на основе столбцов, настоятельно рекомендуется два раунда элюирования для обеспечения максимального восстановления. - Количественно оценить очищенную фрагментированную мРНК с помощью спектрофотометра и оценить качество РНК с помощью анализатора фрагментов.
- Сохраните 100 нг фрагментированной мРНК в качестве входного контроля для подготовки библиотеки и секвенирования (перейдите к шагу 9). Оставшаяся фрагментированная мРНК (минимум 2,5 мкг) может быть использована для MeRIP (перейдите к шагу 7.2).
7. MeRIP
ПРИМЕЧАНИЕ: Для каждой иммунопреципитации (IP) требуется минимум 2,5 мкг фрагментированной мРНК, либо с использованием специфического антитела против m6A (тестовое состояние), либо с использованием антитела против IgG (отрицательный контроль).
- Магнитный шарик Препарат для иммунопреципитации
- Для каждого образца подготовьте 4 мл 1x IP-буфера в новой конической трубке, разбавляя 800 мкл IP-буфера мРНК 5x (50 мМ Tris-HCl pH 7,4, 750 мМ NaCl, 0,5% Igepal CA-630 и безнуклеазной воды) 3,2 мл воды без нуклеазы.
ПРИМЕЧАНИЕ: Требуется не менее 2 реакций (один тест и один контроль IgG). - Поместите трубку на лед.
- Обозначьте соответствующее количество микроцентрифужных пробирок объемом 1,5 мл для количества желаемых IP-реакций:
n пробирок (тест) на антитела против m6A.
n трубок (отрицательный контроль) для нормальной мыши IgG. - Повторное суспендирование магнитных шариков (например, Magna ChIP Protein A/G) путем инвертирования и вихря. Никаких комочков бусин быть не должно быть видно.
- Для каждой запланированной реакции переложите 25 мкл магнитных шариков в микроцентрифужную трубку.
- Добавьте в десять раз больше 1x IP-буфера (из шага 7.1.1) по отношению к исходному объему используемых бусин (т.е. 250 мкл 1x IP-буфера на 25 мкл магнитных шариков).
- Смешайте бусины, аккуратно испешивая вверх и вниз несколько раз для полного повторного суспендирования.
- Поместите трубку на магнитный сепаратор на 1 мин.
- Удалите и выбросьте супернатант, убедившись, что вы не аспирируете какие-либо магнитные шарики. Извлеките трубку из магнита.
- Повторите этап стирки (шаги 7.1.6-7.1.9).
- Повторное суспендирование бусин в 100 мкл 1x IP Buffer на 25 мкл исходного объема магнитных шариков.
- Добавьте 5 мкл антитела (1 мкг/мкл) на 25 мкл исходного объема магнитных шариков.
n пробирок (тест) с антителом против m6A (клон 17-3-4-1) [1 мкг/мкл].
n пробирок (отрицательный контроль) с нормальным Mouse IgG (1 мкг/мкл). - Инкубируют на вращающемся колесе в течение 30 мин при комнатной температуре, чтобы обеспечить сопряжение антител с магнитными шариками.
- Поместите трубку на магнитный сепаратор на 1 мин. Выбросьте супернатант. Извлеките трубку из магнита и повторно суспендируйте смесь антитело-шарик в 100 мкл 1x IP-буфера.
- Для каждого образца подготовьте 4 мл 1x IP-буфера в новой конической трубке, разбавляя 800 мкл IP-буфера мРНК 5x (50 мМ Tris-HCl pH 7,4, 750 мМ NaCl, 0,5% Igepal CA-630 и безнуклеазной воды) 3,2 мл воды без нуклеазы.
- Иммунопреципитация РНК (RIP)
- Готовят 500 мкл реакционной смеси RIP на каждый образец мРНК весом 2,5 мкг следующим образом: 2,5 мкг в 100 мкл фрагментированной РНК (из стадии 6.12); 295 мкл воды без нуклеазы; 5 мкл ингибитора РНКАЗЫ 40 Ед/мкл; и 100 мкл 5x IP-буфера.
- Добавьте 500 мкл реакционной смеси RIP к каждой смеси антитело и шарика (~100 мкл со стадии 7.1.14). Перемешайте, аккуратно дозировав несколько раз, чтобы полностью повторно суспендировать бусины. Место на льду.
- Инкубируйте все трубки RIP на вращающемся колесе в течение 2 часов при 4 °C.
- Центрифугируйте реакции MeRIP на короткое время, чтобы раскрутить капли жидкости со стороны колпачка и трубки. Поместите трубки на магнитный сепаратор на 1 мин.
- Перенесите супернатант в новую центрифужную трубку, стараясь не потревожить магнитные шарики.
ПРИМЕЧАНИЕ: Проточное прохождение может быть сохранено в качестве контроля для проверки эффективности RIP (перейдите к шагу 7.3.9). - Снимите трубки с магнита. Вымойте бусины, добавив 500 мкл холодного 1x IP-буфера. Смешайте бусины, аккуратно испешивая несколько раз, чтобы полностью повторно суспендировать бусины.
- Поместите трубки на магнитный сепаратор на 1 мин и выбросьте супернатант.
- Повторите процедуру стирки (этапы 7.2.6-7.2.7) дважды, в общей сложности 3 промывки.
- Поместите трубки на лед и сразу приступайте к элюированию.
- Элюирование
- Готовят раствор 20 мМ м6А, растворяя 10 мг N6-метиладенозина, 5'-монофосфатной натриевой соли (м6А) в 1,3 мл воды без нуклеазы. Приготовить 150 мкл аликвот и хранить при -20 °C.
- Для каждого образца (тест и контроль): Подготовьте 225 мкл элюционного буфера путем смешивания следующих компонентов: 45 мкл 5x IP-буфера, 75 мкл 20 мМ м6А, 3,5 мкл ингибитора РНКазы 40U/μL и 101,5 мкл воды без нуклеазы.
- Добавьте 100 мкл буфера элюирования (из шага 7.3.2) к шарикам (из шага 7.2.9). Перемешайте, аккуратно дозировав несколько раз, чтобы полностью повторно суспендировать бусины.
- Инкубировать все трубки в течение 1 ч с непрерывным встряхиванием на коромысле при 4 °C.
- Центрифугируйте реакции RIP ненадолго, чтобы раскрутить капли жидкости со стороны колпачка и трубки. Поместите трубки на магнитный сепаратор на 1 мин.
- Перенесите супернатант, содержащий элюированные фрагменты РНК, в новую микроцентрифужную трубку объемом 1,5 мл. Будьте осторожны, чтобы не аспирировать бусины, так как это увеличит фоновый шум.
- Повторите этапы элюирования (от 7.3.3 до 7.3.6), снова добавив 100 мкл буфера элюирования, инкубируя 1 ч при 4 °C и собирая элюат после магнитного разделения.
- Объединить все элюаты из одного образца (общий объем элюирования должен составлять 200 мкл).
- Очищают элюированную РНК и прохождение (необязательно, начиная с этапа 7.2.5) осаждением этанола или методом выбора на основе столбцов (т.е. РНК Clean and Concentrator).
- Оцените количество РНК и качество проточных и элюированных образцов с помощью анализатора фрагментов с использованием высокочувствительного комплекта для детектирования. Если качество РНК удовлетворительное, приступайте к подготовке библиотеки и высокопроизводительному секвенированию (шаг 9).
ПРИМЕЧАНИЕ: Количество РНК, извлекаемой при MeRIP, очень низкое и в обязательном порядке требует наборов для обнаружения высокой чувствительности для обеспечения количественной оценки. Если биоанализатор отсутствует, можно вслепую приступить к подготовке библиотеки.
8. Конверсия РНК-бисульфита
- Контроль и подготовка реагентов
- Контроль спайка смеси ERCC: Добавьте смесь ERCC в соответствии с инструкциями производителя, которые рекомендуют добавлять 0,5 мкл неразбавленной смеси ERCC к 500 нг мРНК. Этот контроль может помочь оценить эффективность конверсии бисульфита.
- Спайк Поли-А-обедненная РНК (со стадии 3.3.14) в соотношении 1/1000 (т.е. 500 пг поли-А-истощенной РНК на 500 нг мРНК). Этот образец обогащен рибосомной РНК и, таким образом, должен содержать 28S рРНК, положительный контроль для превращения бисульфитов.
ПРИМЕЧАНИЕ: Общая РНК также может быть использована в качестве положительного контроля вместо поли-А-истощенной РНК. - Выполняют превращение бисульфита с помощью набора метилирования РНК (например, Zymo EZ).
- Буфер промывки РНК: Добавьте 48 мл 100% этанола (или 52 мл 95% этанола) к 12 мл концентрата РНК Wash Buffer перед использованием.
- Конверсия бисульфита
ПРИМЕЧАНИЕ: Конверсию бисульфита проводили с помощью коммерчески доступного набора для конверсии РНК-бисульфита в соответствии с процедурой производителя, как указано ниже.- В пробирки ПЦР 0,2 мл добавляют 1000 нг мРНК (или от 300 до 1000 нг). Добавьте контрольные элементы управления: 1 мкл смеси ERCC (этап 8.1.1) и 1000 пг поли-А-истощенной РНК (этап 8.1.2). Полный объем до 20 мкл с водой без ДНКазы/РНКазы.
- Добавьте 130 мкл реагента преобразования РНК к каждому образцу РНК объемом 20 мкл.
- Перемешайте образец, пипетируя вверх и вниз.
- Открутите ненадолго, чтобы убедиться, что в колпачке или боковых сторонах трубки нет капель.
- Поместите трубки ПЦР в термоциклер и выполните следующие этапы: денатурацию при 70 °C в течение 5 мин; конверсия при 54 °C в течение 45 мин; повторять этапы денатурации и преобразования в общей сложности 3 цикла; а затем удерживать при 4 °C неопределенно долго.
ПРИМЕЧАНИЕ: Три цикла денатурации и превращения бисульфита обеспечивают полную конверсию бисульфита образца. Образцы могут храниться при -80 °C или непосредственно обрабатываться. - Приступайте к десульфонированию в колонке. Поместите столбец в пустую коллекторную трубку и добавьте в колонку 250 мкл буфера связывания РНК.
- Загрузите образец (~150 мкл из шага 8.2.5) в столбец, содержащий буфер связывания РНК, и перемешайте путем пипетирования вверх и вниз.
- Добавьте 400 мкл 95-100% этанола в образец-связывающую буферную смесь РНК в колонке. Закройте колпачок и сразу же перемешайте, перевернув колонку несколько раз.
- Центрифуга на полной скорости (≥ 10 000 х г) в течение 30 с. Отбросьте сквозной поток.
- Добавьте 200 мкл РНК Wash Buffer в колонку и центрифугу на полной скорости в течение 30 с.
- Добавьте в колонку 200 мкл буфера десульфирования РНК и инкубируйте при комнатной температуре в течение 30 мин. После инкубации центрифугируют на полной скорости в течение 30 с. Отбросьте сквозной поток.
- Добавьте 400 мкл РНК Wash Buffer в колонку и центрифугу на полной скорости в течение 30 с. Повторите этап промывки с дополнительными 400 мкл РНК Wash Buffer. Отбросьте сквозной поток.
- Центрифугируйте колонну в опорожненной коллекторной трубке на полной скорости в течение 2 мин. Перенесите колонку в трубку, свободную от РНКазы.
- Добавьте ≥ 10 мкл воды, не содержащей ДНКазы/РНКазы, непосредственно в матрицу колонки и инкубируйте в течение 1 мин при комнатной температуре. Центрифуга на полной скорости в течение 30 с.
ПРИМЕЧАНИЕ: Мы обычно элюируем в объеме 20 мкл. Элюированная РНК может быть использована немедленно или храниться при -20 °C до 3 месяцев. Для длительного хранения хранить при температуре -80 °C. - Вынимаем 2,5 мкл для оценки качества и количества РНК анализатором фрагментов и переходим к подготовке библиотеки и высокопроизводительному секвенированию (шаг 9).
- Взять 4 мкл преобразованной РНК для контроля эффективности конверсии бисульфитов (этап 8.3).
- Контроль конверсии бисульфитов методом ОТ-ПЦР
ПРИМЕЧАНИЕ: Этот шаг гарантирует, что конверсия бисульфита была успешной, прежде чем приступить к секвенированию. Рибосомная РНК 28S от Homo sapiens будет использоваться в качестве положительного контроля для анализа метилирования РНК, поскольку остаток C в позиции 4447 (присоединение GenBank # NR_003287) был описан как 100% метилированный.
Последовательности праймеров:
Грунтовка H 28SF: 5'-GGGGTTTTAYGATTTTTTTTTTTTTTTTTGGG-3'
Грунтовка H 28SR: 5'-CCAACTCACRTTCCCTATTAATAATAAATAAAC-3'- Подготовьте реакционную смесь обратной транскрипции (RT) с помощью набора обратной транскрипции кДНК с высокой емкостью. Разморозьте компоненты комплекта на льду и приготовьте мастер-микс RT на льду следующим образом:
4 мкл бисульфит-преобразованной РНК (из стадии 8.2.14):
2 мкл буфера 10xRT
0,8 мкл 25x dNTP Mix [100 мМ]
2 мкл 10 случайных праймеров RT
1 мкл мультикрибаритной обратной транскриптазы
1 мкл ингибитора РНКАЗЫ
9,2 мкл H2O без нуклеазы
ПРИМЕЧАНИЕ: Каждая реакция РТ должна содержать конечный объем 20 мкл в пробирках ПЦР объемом 0,2 мл. - Поместите трубки в термоциклер со следующей программой RT: 25 °C в течение 10 мин; 37 °C в течение 120 мин; 85 °C в течение 5 мин; затем при 4 °C бессрочно.
- Подготовьте реакцию ПЦР для амплиации специфической рРНК 28S с помощью фермента корректуры ПЦР. Разморозьте компоненты комплекта на льду, осторожно вихрь и ненадолго центрифугируйте. Приготовьте мастер-микс ПЦР на льду или на держателе металлической пластины со льдом следующим образом:
0,6 мкл грунтовки 10 мкМ H 28SF
0,6 мкл грунтовки 10 мкМ H 28SF
6,5 мкл шаблонной кДНК
22,5 мкл мастер-микса ДНК-полимеразы
ПРИМЕЧАНИЕ: Каждая реакция ПЦР должна содержать конечный объем 20 мкл в пробирках ПЦР объемом 0,2 мл. - Поместите трубки в термоциклер со следующей программой ПЦР: начальная денатурация при 95 °C в течение 5 мин; 45 циклов денатурации (95 °C в течение 15 с), отжига (57 °C в течение 30 с) и относительного удлинения (72 °C в течение 15 с), окончательного удлинения при 72 °C в течение 10 мин, а затем удержания при 4 °C неопределенное время.
- Запустите 10 мкл реакции на 2% агарозном геле. Ожидаемый размер полосы составляет 130 - 200 bp.
- Подготовьте реакционную смесь обратной транскрипции (RT) с помощью набора обратной транскрипции кДНК с высокой емкостью. Разморозьте компоненты комплекта на льду и приготовьте мастер-микс RT на льду следующим образом:
- Секвенирование продуктов ПЦР
- Очищают продукты ПЦР с помощью выбранного на основе колонного метода для удаления ферментов и остатков dNTP и элюляции амплифицированной ДНК по меньшей мере в 20 мкл воды, свободной от ДНКазы/РНКазы.
- Количественно оцените очищенную ДНК с помощью спектрофотометра.
- Реакция секвенирования
- Используйте 40 нг препарата ПЦР/реакции секвенирования.
- Последовательность в обоих направлениях с помощью праймеров H 28SF и H 28SR.
- Выровняйте последовательности с известной непреобразованной последовательностью (28S рибосомная N5 (RNA28SN5). Проверьте наличие остатка C в положении C4447 и остатков T вместо C в другом месте.
9. Подготовка библиотеки и секвенирование высокой пропускной способности
- Подготовьте библиотеки для секвенирования с помощью наборов мРНК (например, Illumina TruSeq Stranded), запустив протокол на этапе Elute-Prime-Fragment и следуя инструкциям производителя.
- Однако для входных образцов RNA-Seq и MeRIP-Seq инкубируйте образцы при 80 °C в течение 2 мин, чтобы получить только прайм, но не далее фрагментировать их.
- Проводите секвенирование с помощью платформ Illumina. Реакции секвенирования могут проводиться в соответствии с предпочтениями и экспериментальным дизайном, как одиночными, так и парными концами, с минимальной длиной 100 нт.
10. Биоинформатические анализы
- m6A Обработка данных
- Запустите FASTQC24 для оценки качества чтения в m6A и ввода файлов FASTQ из последовательности.
- Запустите Atropos25 , чтобы обрезать некачественные конечные и переходные последовательности из чтения. Задайте следующие параметры при запуске Atropos.
- Удалите следующие последовательности адаптеров: AGATCGGAAGAG, CTCTTCCGATCT, AACACTCTTTCCCT, AGATCGGAAGAGCG, AGGGAAAGAGTGTT, CGCTCTTCCGATCT.
- Используйте следующую отсечку качества Phred: 5, для обрезки некачественных концов, как указано производителем (https://support.illumina.com/downloads/illumina-adapter-sequences-document-1000000002694.html).
- Используйте следующую минимальную длину чтения после обрезки: 25 пар оснований.
- Объединить геном человека GRh38 и ссылку на ВИЧ [Интегрированный линейный pNL4-3Env-GFP] в формате FASTA.
- Проиндексируйте объединенную ссылку с помощью HISAT226.
- Запустите HISAT2 на обрезанных чтениях, чтобы выровнять их по индексированной ссылке. Используйте параметры HISAT по умолчанию.
- Сортируйте и индексируйте выровненные чтения с помощью SAMtools27.
- Запустите SAMtools stat и Qualimap 228 для проверки качества секвенированных библиотек после выравнивания.
- При необходимости соберите и обобщите показатели качества предыдущего шага с помощью multiQC29.
- Геном ВИЧ имеет гомологичные 634 последовательности bp в 5' LTR и 3' LTR: Перестройка мультикармирования считывает из 5' LTR в соответствующую область 3' LTR с SAMtools.
- Чтобы определить пики m6A, запустите программное обеспечение пиковых вызовов MACS230 (версия 2.1.2). Тщательно выбирайте параметры работы MACS2, чтобы обеспечить правильное функционирование данных RNA-Seq, поскольку пиковый вызов может зависеть от уровня экспрессии генов, а короткие экзоны могут быть неправильно названы пиками. Следовательно, входной сигнал должен быть вычтен из сигнала m6A без сглаживания, обычно применяемого MACS2 к данным на основе ДНК. Примените следующие параметры к подкоманде 'callpeak' из MACS2:
-keep-dup auto (управляет поведением MACS2 по отношению к дублированию считывания, 'auto' позволяет MACS вычислять максимальное количество считываний в том же месте на основе биномиального распределения с использованием 1e-5 в качестве отсечки p-значения)
-g 2.7e9 (размер генома человека в bp)
-q 0.01 (минимальная отсечка FDR для вызова значительных пиков)
-nomodel (в обход построения модели смещения, которая предназначена для экспериментов ChIP-Seq)
-слокальный 0
-llocal 0 (установка этого и предыдущего параметров на 0 позволяет MACS2 напрямую вычитать, без сглаживания, вход считывает с чтения m6A)
-extsize 100 (средняя длина фрагментов в bp)
-В - Выполните подкоманду дифференциального пикового вызова MACS2, 'bdgdiff', чтобы сравнить зараженные и незараженные образцы. 'bdgdiff' принимает в качестве входных данных файлы bedGraph, сгенерированные 'callpeak' на предыдущем шаге. Для каждой точки времени проводите сравнение инфицированных и незараженных образцов с помощью 'bdgdiff', вычитая соответствующий входной сигнал из сигнала m6A и предоставляя дополнительные параметры: -g 60 -l 120.
- Обработка данных m5C
- Запустите Cutadapt31 , чтобы обрезать последовательности адаптеров из необработанных чтений со следующими параметрами:
адаптер "АГАТЧГААГАГКАКАККТКТГААК"
-минимальная длина=25. - Обратное дополнение обрезанных считываний с помощью seqkit32, так как протокол секвенирования производит считывание с обратной нити.
- Запустите FastQC, чтобы проверить качество чтения.
- Слияние генома человека GRh38 и ВИЧ [Интегрированная линейная ссылка pNL4-3Env-GFP] в формате FASTA.
- Проиндексируйте объединенную ссылку с приложением meRanGh из пакета meRanTK33.
- Выровнять с meRanGh со следующими параметрами:
-UN, позволяющий записывать несопоставленные чтения в выходные файлы
-MM, позволяющий записывать многосопоставленные чтения в выходной файл
-bg для вывода в bedGraph
-mbgc 10 фильтр сообщается по регионам покрытия (не менее 10 считываний покрытия) - Геном ВИЧ имеет гомологичные 634 последовательности bp в 5' LTR и 3' LTR: выравнивание мультикармирования считывает из 5' LTR в соответствующую область 3' LTR с помощью SAMtools.
- Запустите вызов метилирования с помощью инструмента meRanCall, предоставляемого meRanTK, со следующими параметрами:
-rl = 126, длина чтения
-ei = 0,1, интервал погрешности для расчета p-значения скорости метилирования
-cr = 0,99, ожидаемое преобразование - Запустите служебную estimateSizeFactors.pl MeRanTK для оценки коэффициентов размера каждого образца. Коэффициенты размера будут использоваться в качестве параметров на следующем шаге.
- Запустите MeRanCompare для дифференциального анализа метилирования неинфицированных и инфицированных в точки времени 12, 24 и 36 часов. Применяются следующие параметры: значение значимости 0,01 в качестве минимального порога для отчетности и коэффициенты размера по сравнению с предыдущим шагом.
- Запустите Cutadapt31 , чтобы обрезать последовательности адаптеров из необработанных чтений со следующими параметрами:
Результаты
Этот рабочий процесс оказался полезным для изучения роли метилирования m6A и m5C в контексте ВИЧ-инфекции. Для этого мы использовали модель клеточной линии CD4 + T (SupT1), которую мы либо заражаем ВИЧ, либо оставляем без лечения. Мы начали рабочий процесс с 50 миллионами клеток на состояние и получили в среднем 500 мкг общей РНК с числом качества РНК 10 (рисунок 1A-B). При поли-А отборе мы извлекли от 10 до 12 мкг мРНК на условие (что составляет около 2% от общей РНК) (рисунок 1B). На данный момент мы использовали 5 мкг поли-A-выбранной РНК для трубопровода MeRIP-Seq и 1 мкг для трубопровода BS-Seq. Поскольку РНК ВИЧ полиаденилирована, никаких дальнейших действий не требуется, и процедуры MeRIP-Seq и BS-Seq могут применяться напрямую.
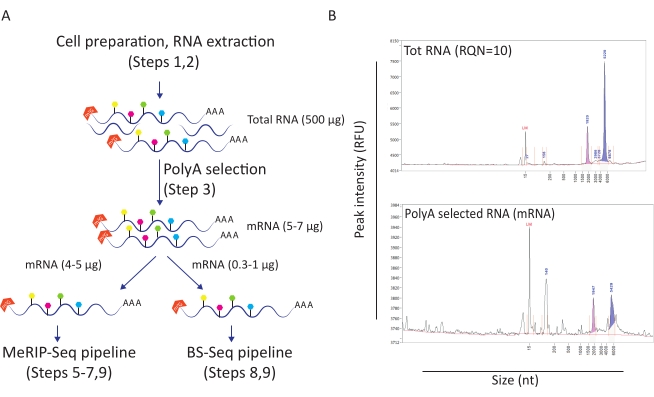
Рисунок 1: Подготовка РНК для последующих применений. A) Рабочий процесс, отображающий подготовку и распределение РНК для одновременных конвейеров MeRIP-Seq и BS-Seq. Каждая заполненная гексагональная форма представляет собой тип модификации РНК, такой как m6A (зеленый) или m5C (розовый). Указаны количества материала РНК, необходимые для проведения эксперимента. B) Репрезентативные результаты, показывающие ожидаемые профили распределения РНК (размер и количество) при общей экстракции РНК (верхняя панель) и выборе поли-А (нижняя панель). Образцы были загружены на анализатор фрагментов со стандартным набором чувствительности для оценки качества РНК перед входом в конкретные процедуры MeRIP-Seq и BS-Seq. RQN: число качества РНК; nt: нуклеотиды. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Трубопровод MeRIP-Seq представляет собой метод на основе иммунопреципитации РНК, который позволяет исследовать модификацию m6A вдоль молекул РНК. Для этого РНК сначала фрагментируют, а затем инкубируют с m6A-специфическими антителами, соединенными с магнитными шариками для иммунопреципитации и захвата. Обогащенные MeRIP фрагменты РНК и нетронутая (входная) фракция затем секвенируются и сравниваются для идентификации m6A-модифицированных областей РНК и, таким образом, m6A-метилированных транскриптов (рисунок 2A). Разрешение методики зависит от эффективности фрагментации РНК. Действительно, более короткие фрагменты позволяют более точно локализовать остаток m6A. Здесь клеточные поли-А-выбранные РНК и вирусные РНК подвергали фрагментации на основе ионов с буфером фрагментации РНК в течение 15 мин в конечном объеме 20 мкл с получением фрагментов РНК 100-150 нт. Начиная с 5 мкг мРНК, мы восстановили 4,5 мкг фрагментированной РНК, что соответствует скорости восстановления 90% (рисунок 2B). Мы использовали 100 нг фрагментированной, очищенной РНК в качестве входного контроля, подвергая непосредственно подготовке библиотеки и секвенированию. Оставшуюся РНК (~4,4 мкг) обрабатывали по конвейеру MeRIP-Seq, который начинается с инкубации фрагментированной РНК с шариками, связанными либо со специфическими антителами против m6A, либо с антителами против IgG в качестве контроля. m6A-специфический RIP (MeRIP) из 2,5 мкг фрагментированной РНК позволил извлечь около 15 нг обогащенного m6A материала, который подвергся библиотечной подготовке и секвенированию (рисунок 2B). RIP с контролем анти-IgG, как и ожидалось, не дал достаточного количества РНК, чтобы позволить дальнейший анализ (рисунок 2B).
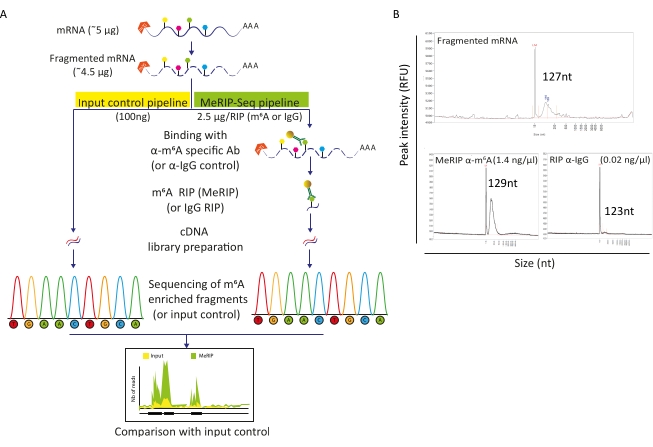
Рисунок 2: Конвейер MeRIP-Seq. A) Схематическое представление рабочего процесса MeRIP-Seq и управления входом. При поли-А отборе образцы фрагментировали на 120-150 нт кусочков и либо непосредственно подвергали секвенированию (100 нг, входной контроль), либо использовали для иммунопреципитации РНК (2,5 мкг, RIP) со специфическим антителом анти-m6A или анти-IgG антителом в качестве отрицательного контроля перед секвенированием. B) Репрезентативные результаты, показывающие ожидаемые профили распределения РНК (размер и количество) при фрагментации (верхняя панель) и RIP (нижние панели, MeRIP: левый, контроль IgG: правый). Образцы были загружены на анализатор фрагментов для оценки качества и концентрации РНК перед дальнейшей обработкой до подготовки библиотеки и секвенирования. Фрагментированный анализ РНК проводили с использованием стандартного набора чувствительности РНК, в то время как иммунопреципитированную РНК использовали набор с высокой чувствительностью. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Трубопровод BS-Seq позволяет исследовать модификацию рНК m5C с разрешением нуклеотидов и приводит к идентификации m5C-метилированных транскриптов. После превращения бисульфита неметилированные цитозины превращаются в урацил, в то время как метилированные цитозины остаются неизменными (рисунок 3А). Из-за суровых условий процедуры конверсии бисульфита (т.е. высокой температуры и низкого рН) преобразованные мРНК сильно деградируют (рисунок 3B), однако это не мешает подготовке библиотеки и секвенированию. Превращение бисульфита эффективно только на одноцепочечной РНК и, таким образом, потенциально может быть затруднено вторичными двухцепочечными структурами РНК. Для оценки эффективности преобразования C-U мы ввели два элемента управления. В качестве положительного контроля мы воспользовались ранее описанным присутствием высокометилированного цитозина в положении C4447 28S rRNA23. При амплификации и секвенировании ОТ-ПЦР фрагмента 200 bp, окружающего метилированный участок, мы могли наблюдать, что все цитозины были успешно преобразованы в урацилы, тем самым появляясь в виде тимидинов в последовательности ДНК, за исключением цитозина в позиции 4447, который остался неизменным. В качестве контроля скорости конверсии бисульфитов мы использовали коммерчески доступные синтетические последовательности РНК ERCC. Эта смесь состоит из пула известных, неметилированных и полиаденилированных последовательностей РНК с различными вторичными структурами и длинами. После подготовки библиотеки и секвенирования мы сосредоточились на этих последовательностях ERCC для расчета коэффициента преобразования, который может быть выполнен путем подсчета количества преобразованного C среди общих остатков C во всех последовательностях ERCC и в каждом образце. Мы получили коэффициент конверсии 99,5%, подтверждающий эффективность и успешность реакции превращения бисульфита (рисунок 3D).
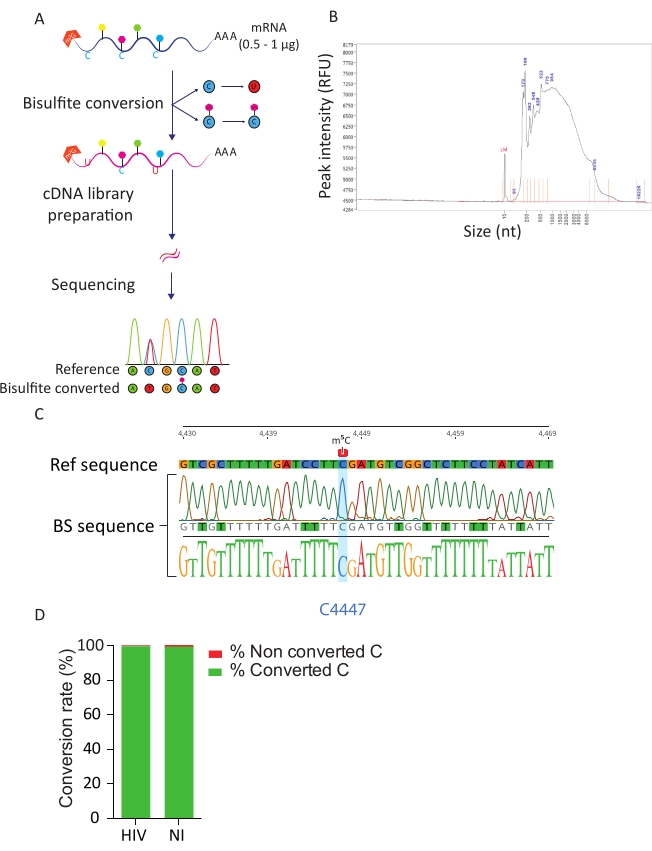
Рисунок 3: Конвейер BS-Seq. A) Схематическое представление рабочего процесса BS-Seq. При поли-А отборе образцы подвергают воздействию бисульфита, что приводит к конверсии С в U (из-за дезаминирования) для неметилированных остатков С. Напротив, метилированные остатки С (m5C) не подвергаются обработке бисульфитом и остаются неизменными. Б) Репрезентативный результат профиля распределения (размер и количество) преобразованной в бисульфит РНК при анализе на анализаторе фрагментов со стандартным комплектом чувствительности. C) Электроферограмма, показывающая репрезентативный результат секвенирования ампликона ОТ-ПЦР области, окружающей 100% метилированный С в положении 4447 в 28S рРНК (выделена синим цветом). Напротив, остатки С эталонной последовательности были идентифицированы как Т-остатки в последовательности ампликонов из-за успеха конверсии бисульфита. D) Оценка коэффициента конверсии C-U путем анализа последовательностей всплеска ERCC в ВИЧ-инфицированных и неинфицированных клетках. Средний коэффициент конверсии составляет 99,5%. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Образцы, обогащенные M6A, образцы, преобразованные в бисульфит, и входные элементы управления дополнительно обрабатываются для подготовки библиотеки, секвенирования и биоинформационного анализа (рисунок 4). В соответствии с экспериментальным дизайном и рассмотренным биологическим вопросом (вопросами), могут быть применены множественные биоинформационные анализы. В качестве принципиального доказательства здесь мы показываем репрезентативные результаты одного потенциального применения (т.е. дифференциального анализа метилирования), который фокусируется на идентификации дифференциально метилированных транскриптов, индуцированных при ВИЧ-инфекции. Вкратце, мы исследовали уровень метилирования транскриптов m6A или m5C, независимо от уровня экспрессии их генов, как в неинфицированных, так и в ВИЧ-инфицированных клетках, чтобы лучше понять роль метилирования РНК в течение жизненного цикла вируса. После нормализации экспрессии генов мы определили, что транскрипт ZNF469 был дифференциально m6A-метилирован в соответствии со статусом инфекции, на самом деле этот транскрипт не был метилирован в неинфицированных клетках, в то время как он демонстрировал несколько метилированных пиков при ВИЧ-инфекции (рисунок 5A). Аналогичный анализ дифференциального метилирования m5C показал, что транскрипт PHLPP1 содержит несколько метилированных остатков, которые, как правило, чаще метилируются в состоянии ВИЧ (рисунок 5B). В этом контексте оба анализа показывают, что ВИЧ-инфекция влияет на клеточный эпитранскриптом.
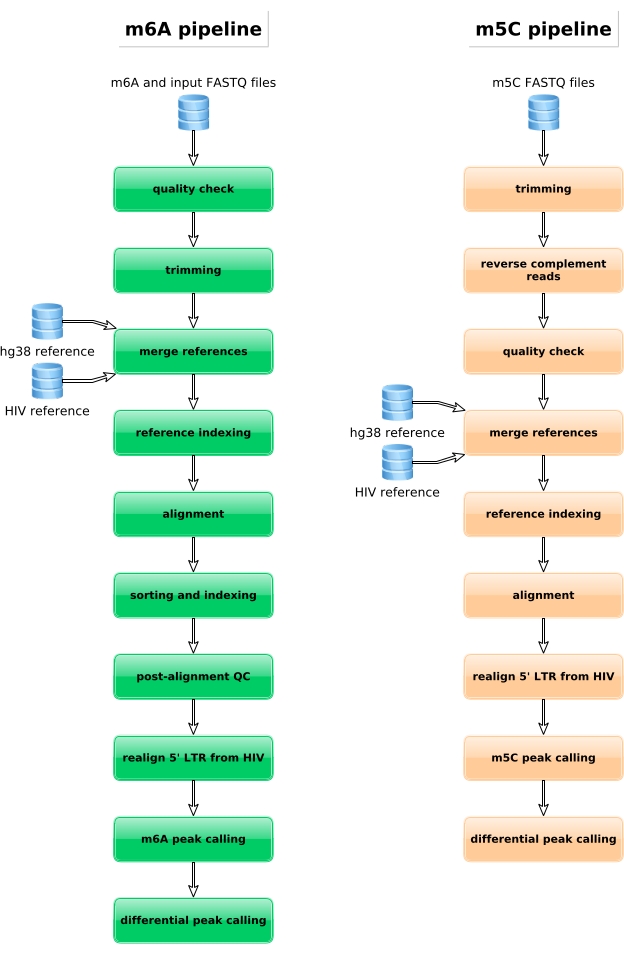
Рисунок 4: Схематическое представление биоинформационного рабочего процесса для анализа данных m6A и m5C. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
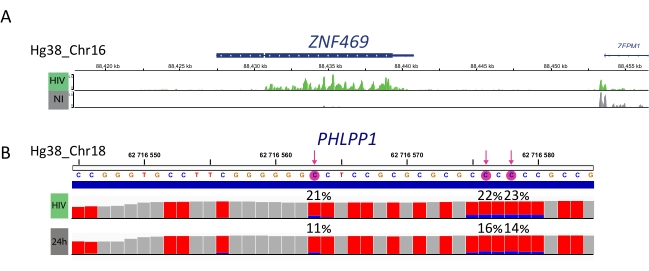
Рисунок 5: Пример дифференциально метилированных транскриптов при инфекции. A) Репрезентативный результат, показывающий метилирование m6A транскрипта ZNF459 в ВИЧ-инфицированных (зеленых) и неинфицированных (серых) клетках. Пиковая интенсивность (при вычитании входной экспрессии) показана на оси y и положении в хромосоме вдоль оси x. Дифференциальный анализ метилирования показывает, что транскрипт ZFN469 гиперметилируется при ВИЧ-инфекции. B) Репрезентативный результат метилированного гена m5C в ВИЧ-инфицированных (верхняя полоса) и неинфицированных (нижняя полоса) клетках. Высота каждого стержня представляет собой количество считываний на нуклеотид и позволяет оценить охват. Каждый остаток С представлен красным цветом, а доля метилированного С представлена синим цветом. Точная скорость метилирования (%) указана выше каждого остатка С. Стрелки выделяют статистически значимый дифференциально метилированный C. Образцы были визуализированы с помощью IGV viewer. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Обсуждение
Роль модификаций РНК в вирусной инфекции до сих пор в значительной степени неизвестна. Лучшее понимание роли эпитранскриптомных модификаций в контексте вирусной инфекции может способствовать поиску новых целей противовирусного лечения.
В этой работе мы обеспечиваем полный рабочий процесс, который позволяет исследовать эпитранскриптомы m6A и m5C инфицированных клеток. В зависимости от биологического вопроса мы советуем использовать поли-А-выбранную РНК в качестве исходного материала. Хотя это необязательно, поскольку конвейер может использоваться с общей РНК, важно иметь в виду, что рРНК, а также малые РНК сильно модифицированы и содержат значительное количество метилированных остатков. Это может привести к снижению качества и количества значимых данных о последовательности.
Однако, если основное внимание в исследовании уделяется неполиаденилированной РНК, этап экстракции РНК должен быть адаптирован, чтобы избежать отбрасывания малой РНК (в случае экстракции РНК на основе колонки) и дать предпочтение методам истощения рибосом, а не отбора поли-А для входа в конвейер.
Для обеспечения высокого качества РНК, правильной фрагментации и подходящего качества РНК, обогащенной m6A и преобразованной BS, для подготовки библиотеки мы настоятельно рекомендуем использовать анализатор фрагментов или биоанализатор. Однако это оборудование не всегда доступно. В качестве альтернативы качество РНК, мРНК и размер фрагментированной РНК также могут быть оценены путем визуализации на агарозном геле. Альтернативно, подготовка библиотеки может быть выполнена без предварительной оценки количества РНК.
Мы использовали технику MeRIP-Seq16 на основе антител для изучения эпитранскриптомного ландшафта m6A. Данная методика основана на иммунопреципитации РНК и является успешной; однако некоторые шаги требуют тщательной оптимизации и могут быть критическими. Хотя было описано, что метилирование m6A происходит в основном в рамках консенсусной последовательности RRA*CH, этот мотив очень часто встречается вдоль молекул мРНК и не позволяет точно идентифицировать метилированный участок. Таким образом, крайне важно достичь воспроизводимой и последовательной фрагментации РНК, генерируя небольшие фрагменты РНК, для улучшения разрешения на основе RIP. В этом протоколе мы рекомендуем оптимизированную процедуру, обеспечивающую воспроизводимые и последовательные результаты в наших экспериментальных условиях; однако этот этап фрагментации может потребовать дальнейшей оптимизации в соответствии с конкретными характеристиками выборки.
Недавно была описана новая методика, позволяющая прямое секвенирование m6A. Он основан на использовании специфических вариантов обратной транскриптазы, которые демонстрируют уникальные RT-сигнатуры в ответ на столкновение с модификацией РНК m6A24. Эта технология, при тщательной оптимизации, может обойти основное ограничение, с которым сталкивается MeRIP-Seq (уменьшение количества исходного материала и обеспечение более высокого разрешения). Для изучения модификации m5C мы решили использовать метод конверсии бисульфитов для обнаружения с разрешением нуклеотидов модифицированных остатков C. Чтобы снизить частоту ложных срабатываний из-за наличия вторичных структур РНК, мы выполнили 3 цикла денатурации / превращения бисульфита и дополнительно контролировали скорость превращения бисульфита благодаря использованию элементов управления всплесками ERCC. Одним из ограничений, связанных с этим методом, является то, что превращение бисульфита очень жесткое, и три цикла денатурации / превращения бисульфита могут разрушать некоторые РНК и, следовательно, уменьшать разрешение. Однако в наших настройках мы решили довольствоваться потенциально немного более низким разрешением, чтобы повысить качество набора данных.
Благодаря этим оптимизациям и контролю мы смогли обеспечить надежный и надежный рабочий процесс, который можно использовать для исследования эпитранскриптомного ландшафта и его изменения в контексте вирусных инфекций, взаимодействий хозяин-патоген или любого воздействия конкретных методов лечения.
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Эта работа была поддержана Швейцарским национальным научным фондом (гранты 31003A_166412 и 314730_188877).
Материалы
| Name | Company | Catalog Number | Comments |
| AccuPrime Pfx SuperMix | Invitrogen | 12344-040 | |
| anti-m6A antibody _Clone 17-3-4-1 | Millipore | MABE1006 | |
| Chloroform | Merck | 67-66-3 | |
| ERCC | Invitrogen | 4456740 | |
| EZ RNA Methylation Kit | Zymo Research | EZR5001 | |
| Fragment analyzer RNA Kit - HS RNA Kit | Agilent | DNF-472-0500 | |
| Fragment analyzer RNA Kit - RNA Kit | Agilent | DNF-471-0500 | |
| High-Capacity cDNA Reverse Transcription Kit | Applied Biosystem | 4368814 | |
| Illumina TruSeq Stranded mRNA | Illumina | 20020594 | |
| Magnetic Beads A/G Blend | Merck | 16-663 | |
| N6-Methyladenosine, 5′-monophosphate sodium salt (m6A) | Sigma Aldrich | M2780-10MG | |
| Normal Mouse IgG | Merk | 12371 | |
| Oligo(dT)25 | Life Technologies | 61005, | |
| PCRapace | Stratec | 1020220300 | |
| Quick RNA Viral Kit | Zymo Research | 1034 | |
| RNA Clean & Concentrator | Zymo Research | R1015 | |
| RNA Fragmentation Reagent | Ambion | AM8740 | |
| RNase Inhibitor | Ambion | AM2684 | |
| Trizol | TRIzol Reagent | 15596026 |
Ссылки
- Machnicka, M. A., et al. MODOMICS: a database of RNA modification pathways--2013 update. Nucleic Acids Research. 41, 262-267 (2013).
- Zaccara, S., Ries, R. J., Jaffrey, S. R. Reading, writing and erasing mRNA methylation. Nature Reviews Molecular Cell Biology. 20 (10), 608-624 (2019).
- Davalos, V., Blanco, S., Esteller, M. SnapShot: Messenger RNA Modifications. Cell. 174 (2), 498 (2018).
- Saletore, Y., et al. The birth of the Epitranscriptome: deciphering the function of RNA modifications. Genome Biology. 13 (10), 175 (2012).
- Zhao, B. S., Roundtree, I. A., He, C. Post-transcriptional gene regulation by mRNA modifications. Nature Reviews Molecular Cell Biology. 18 (1), 31-42 (2017).
- Netzband, R., Pager, C. T. Epitranscriptomic marks: Emerging modulators of RNA virus gene expression. Wiley Interdisciplinary Reviews: RNA. 11 (3), 1576 (2020).
- Pereira-Montecinos, C., Valiente-Echeverria, F., Soto-Rifo, R. Epitranscriptomic regulation of viral replication. Biochimica et Biophysica Acta. 1860 (4), 460-471 (2017).
- Lichinchi, G., et al. Dynamics of the human and viral m(6)A RNA methylomes during HIV-1 infection of T cells. Nature Microbiology. 1, 16011 (2016).
- Courtney, D. G., et al. Epitranscriptomic Addition of m(5)C to HIV-1 Transcripts Regulates Viral Gene Expression. Cell Host & Microbe. 26 (2), 217-227 (2019).
- Kennedy, E. M., et al. Posttranscriptional m(6)A Editing of HIV-1 mRNAs Enhances Viral Gene Expression. Cell Host & Microbe. 19 (5), 675-685 (2016).
- Tirumuru, N., Wu, L. HIV-1 envelope proteins up-regulate N (6)-methyladenosine levels of cellular RNA independently of viral replication. Journal of Biological Chemistry. 294 (9), 3249-3260 (2019).
- Tirumuru, N., et al. N(6)-methyladenosine of HIV-1 RNA regulates viral infection and HIV-1 Gag protein expression. Elife. 5, (2016).
- Cristinelli, S., Angelino, P., Janowczyk, A., Delorenzi, M., Ciuffi, A. HIV Modifies the m6A and m5C Epitranscriptomic Landscape of the Host Cell. Frontiers in Virology. 1 (11), (2021).
- Khoddami, V., Cairns, B. R. Transcriptome-wide target profiling of RNA cytosine methyltransferases using the mechanism-based enrichment procedure Aza-IP. Nature Protocols. 9 (2), 337-361 (2014).
- Hussain, S., Aleksic, J., Blanco, S., Dietmann, S., Frye, M. Characterizing 5-methylcytosine in the mammalian epitranscriptome. Genome Biology. 14 (11), 215 (2013).
- Dominissini, D., Moshitch-Moshkovitz, S., Salmon-Divon, M., Amariglio, N., Rechavi, G. Transcriptome-wide mapping of N6-methyladenosine by m6A-seq based on immunocapturing and massively parallel sequencing. Nature Protocols. 8 (1), 176-189 (2013).
- Dominissini, D., et al. Topology of the human and mouse m6A RNA methylomes revealed by m6A-seq. Nature. 485 (7397), 201-206 (2012).
- Shobbir Hussain, J. A., Blanco, S., Dietmann, S., Frye, M. Characterizing 5-methylcytosine in the mammalian epitranscriptome. Genome Biology. 14 (215), (2013).
- Amort, T., et al. Distinct 5-methylcytosine profiles in poly(A) RNA from mouse embryonic stem cells and brain. Genome Biology. 18 (1), 1 (2017).
- Endrullat, C., Glökler, J., Franke, P., Frohme, M. Standardization and quality management in next-generation sequencing. Applied & Translational Genomics. 10, 2-9 (2016).
- Schaefer, M., Pollex, T., Hanna, K., Lyko, F. RNA cytosine methylation analysis by bisulfite sequencing. Nucleic Acids Research. 37 (2), 12 (2009).
- Cristinelli, S., Angelino, P., Janowczyk, A., Delorenzi, M., Ciuffi, A. HIV Modifies the m6A and m5C Epitranscriptomic Landscape of the Host Cell. biorxiv. 1 (11), (2021).
- Squires, J. E., et al. Widespread occurrence of 5-methylcytosine in human coding and noncoding RNA. Nucleic Acids Research. 40 (11), 5023-5033 (2012).
- Aschenbrenner, J., et al. Engineering of a DNA Polymerase for Direct m(6) A Sequencing. Angewandte Chemie (International ed. in English). 57 (2), 417-421 (2018).
- Didion, J. P., Martin, M., Collins, F. S. Atropos: specific, sensitive, and speedy trimming of sequencing reads. PeerJ. 5, 3720 (2017).
- Kim, D., Langmead, B., Salzberg, S. L. HISAT: a fast spliced aligner with low memory requirements. Nature Methods. 12 (4), 357-360 (2015).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Okonechnikov, K., Conesa, A., García-Alcalde, F. Qualimap 2: advanced multi-sample quality control for high-throughput sequencing data. Bioinformatics. 32 (2), 292-294 (2016).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Zhang, Y., et al. Model-based Analysis of ChIP-Seq (MACS). Genome Biology. 9 (9), 137 (2008).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet Journal. 17 (1), (2011).
- Shen, W., Le, S., Li, Y., Hu, F. SeqKit: A Cross-Platform and Ultrafast Toolkit for FASTA/Q File Manipulation. PLOS ONE. 11 (10), 0163962 (2016).
- Rieder, D., Amort, T., Kugler, E., Lusser, A., Trajanoski, Z. meRanTK: methylated RNA analysis ToolKit. Bioinformatics. 32 (5), 782-785 (2015).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены