Method Article
바이러스 감염시 m6A 및 m5C 에피레미테롬 탐험: HIV를 가진 예
요약
바이러스 감염에 있는 RNA 수정의 역할은 탐구하기 시작하고 새로운 바이러스성 호스트 상호 작용 기계장치를 강조할 수 있었습니다. 이 작업에서는 바이러스 감염의 맥락에서 m6A 및 m5C RNA 수정을 조사하는 파이프 라인을 제공합니다.
초록
생물학적 과정에 있는 RNA 수정의 역할은 지난 몇 년 동안 연구 결과의 증가 수의 초점이 되고 고 epitranscriptomics로 요즘 알려져 있습니다. 그 중에서도, N6-메틸라데로신(m6A) 및 5-메틸시토신(m5C) RNA 수정은 mRNA 분자에 기술되어 있고 세포 프로세스를 조절하는 역할을 할 수 있다. Epitranscriptomics는 따라서 바이러스 감염을 포함하여 어떤 화학 또는 생물학 에이전트에 노출에 의해 변경되거나 변조될 수 있기 때문에, 전사 분석 이외에 고려되어야 하는 규정의 새로운 층입니다.
여기서, 우리는 인간 면역 결핍 바이러스 (HIV)로 감염되었거나 하지 않는 세포에서 m6A 및 m5C 마크의 관절 세포 및 바이러스 상피 적 경관의 분석을 동시에 할 수있는 워크플로우를 제시한다. HIV-감염 및 비감염 세포로부터의 mRNA 격리 및 단편화시, 우리는 RNA 면역 침전 기반 기술인 MeRIP-Seq, rna 면역 침전 기반 기술, m6A 마크및 BS-Seq, 양성환전환 기반 기술을 포함하는 RNA 단편을 농축하여 단일 핵티드 분해능에서 m5C 마크를 식별하는 두 가지 다른 절차를 사용했습니다. 메틸화 별 포획시, RNA 라이브러리는 고처리량 시퀀싱을 위해 준비됩니다. 또한 기초 발현 프로파일과 는 별개로 차별화된 메틸화(DM) 성적증명서를 식별하기 위해 전용 생물정보학 파이프라인을 개발했습니다.
전반적으로, 방법론은 동시에 다중 epitranscriptomic 마크의 탐구를 허용하고 바이러스 감염 또는 그밖 세포 동요에 DM 전사의 아틀라스를 제공합니다. 이 접근은 바이러스성 복제를 승진하거나 제한하는 세포 요인과 같은 새로운 플레이어 및 세포 반응의 새로운 기계장치를 식별할 수 있는 새로운 기회를 제공합니다.
서문
RNA 분자가 변형될 수 있고, 150개 이상의 전사 후 수정이 현재까지 기술되었다는 것은 오래 알려져 있다1. 그(것)들은 RNA 분자2의 pyrimidine 및 purine 고리의 거의 모든 위치에 화학 단, 주로 메틸 단의 추가로 이루어져 있습니다. 이러한 전사 후 수정은 이미 전사 RNA (tRNA) 및 리보소말 RNA (rRNA)에서 고도로 농축되는 것으로 나타났으며 최근에는 mRNA 분자에도 설명되어 있다.
차세대 시퀀싱(NGS)과 같은 신기술의 부상과 명확한 화학적 수정을 인식하는 특정 항체의 생산은 처음으로 전사 적 수준에서 특정 화학 적 수정의 위치 및 주파수에 대한 조사가 허용되었다. 이러한 발전은 RNA 수정의 더 나은 이해와 mRNA 분자3,4에 대한 여러 수정의 매핑으로 이어졌습니다.
후성 유전학은 전사 성 규제에서 DNA와 히스톤 변형의 역할을 조사하는 동안, 유사한 방식으로 epitranscriptomics는 RNA 수정및 그들의 역할에 초점을 맞추고 있습니다. epitranscriptomic 수정의 조사는 다양한 세포 프로세스 (즉, RNA 접합, 수출, 안정성 및 번역)5를 조정할 수있는 새로운 규칙 메커니즘을 강조 할 수있는 새로운 기회를 제공합니다. 최근 연구에서 세포와 바이러스 성 RNAs6 모두에서 바이러스 감염에 많은 상피 수정을 발견 했다 따라서 큰 놀라운 일이 아니었다. 지금까지 조사된 바이러스에는 DNA와 RNA 바이러스가 모두 포함됩니다. 그 중에서도 HIV는 선구적인 예로 간주될 수 있습니다. 전부, 바이러스 감염의 맥락에서 RNA 메틸화의 발견은 바이러스성 발현 또는 복제의 아직 설명되지 않은 기계장치의 조사를 허용할 수 있습니다, 따라서 그(것)들을 통제하는 새로운 공구 및 표적을 제공7.
HIV 에피레미토믹스 분야에서 바이러스 성 성적 증명서의 수정은 널리 조사되었으며이 수정의 존재가 바이러스 복제8,9,10,11,12,13에 도움이되었음을 보여주었습니다. 현재까지 다양한 기술을 사용하여 전사 적 수준에서 에피레미토믹 마크를 검출할 수 있다. m6A 식별에 가장 많이 사용되는 기술은 MeRIP-Seq 및 miCLIP과 같은 면역 강수 기술에 의존합니다. MeRIP-Seq는 RNA 단편화에 의존하여 메틸화 잔류물을 포함하는 단편을 포착하는 반면, miCLIP은 RNA-항체 UV 교차 링크시 α-m6A 항체 특이적 시그니처 돌연변이의 생성을 기반으로 하므로 보다 정밀한 매핑을 허용합니다.
m5C 변형의 검출은 m6A 검출(m5C RIP)과 유사한 항체 기반 기술, 또는 비설피트 변환 또는 AZA-IP 또는 miCLIP에 의해 달성될 수 있다. Aza-IP와 m5C miCLIP 은 RNA 메틸화를 거치면서 RNA를 표적으로 하는 미끼로서 특정 메틸 트랜스퍼라아제를 사용한다. Aza-IP에서, 표적 세포는 5-azacytidine에 노출되어, 초기 RNA로 cytidine 아날로그 5-azacytidine 사이트의 임의로 소개의 결과로. miCLIP에서, NSun2 메틸 트랜스퍼라제는 C271A 돌연변이14,15를 항구하기 위하여 유전자 변형됩니다.
이 작품에서는 HIV를 모델로 사용하여 감염된 세포의 m6A 및 m5C 변형의 이중 특성화에 중점을 둡니다. 방법론적 최적화시, 우리는 세포 및 바이러스 성 맥락에서 전사적 수준에서 m6A 및 m5C 상피 마크의 동시 탐사를 허용하는 메틸화 RNA 면역 침전(MeRIP)과 RNA 편황피화 변환(BS)을 결합한 워크플로우를 개발했습니다. 이 워크플로우는 바이러스 성 입자로부터 분리된 RNA뿐만 아니라 세포 RNA 추출물에 구현될 수 있다.
메틸화 RNA 면역전수량(MeRIP)16 접근법은 전사-전체 수준에서 m6A의 조사를 허용하는 것이 잘 확립되고 m6A 특이적 항체의 배열이 현재까지 상용화되어 있다17. 이러한 방법은 m6A 특이적 항체를 이용한 m6A 함유 RNA 조각을 선택적으로 포획하는 것으로 구성된다. 이 기술의 2개의 주요 단점은 (i) RNA 단편의 크기에 크게 의존하고 따라서 메틸화 잔류물을 포함하는 대략적인 위치 및 영역을 제공하고, (ii) 분석을 수행하는 데 필요한 다량의 물질을 제공한다. 다음 최적화 된 프로토콜에서, 우리는 약 150 nt로 단편 크기를 표준화하고 폴리 A 선택 RNA의 10 μg에서 시작 물질의 양을 감소, 이는 현재 시작 물질의 권고 된 양, 폴리 A 선택 RNA의 단지 1 μg. 또한 페놀계 기술 또는 단백질아제 K를 이용한 보다 종래의 덜 특이적 용용 방법 대신 m6A 펩타이드와의 경쟁 접근법에 의한 경쟁 접근법에 의한 용출을 이용하여 특정 항체에 결합된 m6A RNA 단편의 회수 효율을 극대화했다. 그러나 이 RIP 기반 분석의 주요 제한은 정밀하게 변형된 A 뉴클레오티드의 식별을 허용하지 않는 최적이 아닌 해상도로 남아 있다.
m5C 마크의 분석은 현재 두 가지 다른 접근법을 사용하여 수행될 수 있다: m5C 특이적 항체 및 RNA bisulfite 변환을 이용한 RIP 기반 방법. RIP는 메틸화 잔류물의 식별에 대한 제한된 해상도만 제공하므로 단일 뉴클레오티드 분해능을 제공할 수 있는 양성환 변환을 사용했습니다. 비설핏(BS)에 대한 RNA 노출은 시토신 탈약으로 이어져 사이토신 잔류물을 우라실로 변환한다. 따라서, RNA bisulfite 변환 반응 도중, 모든 비메틸화 된 사이토신은 탈액및 우라실로 변환되고, 사이토신의 위치 5에 메틸 군의 존재는 BS 유도 된 탈충을 방지하고 사이토신 잔류물을 보존하는 보호 효과가 있다. BS 기반 접근법은 단일 염기 해상도에서 m5C 변형 뉴클레오티드를 검출하고 각 성적증명서의 메틸화 주파수를 평가하여 m5C 수정 역학18에 대한 통찰력을 제공합니다. 그러나이 기술의 주요 제한은 메틸화 잔류물의 거짓 긍정 비율에 의존한다. 실제로, BS 변환은 접근 가능한 C 잔류물을 가진 단 하나 좌초 RNA에 효과적입니다. 그러나, 단단한 RNA 이차 구조의 존재는 N5C 위치를 가리고 BS 변환을 방해할 수 있고, U 잔류물으로 변환되지 않는 비메틸화 C 잔류물의 결과로, 따라서 거짓 긍정. 이 문제를 회피하고 거짓 긍정 률을 최소화하기 위해 3 차례의 디마튜션 및 비황피전환 주기19를 적용했습니다. 또한 비설피테 변환 효율의 추정을 가능하게 하는 2개의 컨트롤을 시료에 도입했습니다: 우리는 ERCC 시퀀싱 제어(비메틸화 표준화 및 시판 시퀀스)20뿐만 아니라 폴리 A-A 고갈된 RNA를 한 손에 는 양황피전환율을 평가하고 RT-PCR에 의해 잘 알려진 것으로 알려진 C4의 존재를 검증하고, 잘 알려진 C4의 존재를 검증하기 위해 에 28S 리보소말 RNA 다른 한편으로는21.
바이러스학 분야에서, 차세대 시퀀싱과 정확한 생물 정보 분석과 이 두 가지 epitranscriptomic 조사 방법을 결합하면 m6A 및 m5C 역학의 심층 연구를 가능하게 합니다 (즉, 바이러스 감염 시 발생할 수 있는 RNA 수정 측두경 변경및 임상 사용에 대한 새로운 치료 관련 표적의 배열을 발견할 수 있음).
프로토콜
1. 세포 준비
참고: 세포 유형 및 RNA 함량에 따라 세포의 시작 수는 다를 수 있습니다.
- 총 RNA의 200-500 μg 또는 다중 A 선택 RNA의 5-7 μg 사이에서 얻을 수 있는 충분한 세포가 있다. 예를 들어, 50 x 106 SupT1 세포는 페놀 기반 시약을 추출할 때 총 RNA의 약 500 μg를 산출해야 하며, 따라서 테스트된 각 개별 조건에 대해 요구된다.
- 실험 설계에 따라 필요한 수의 세포를 준비하여, 따라서 시험된 조건의 수에 따라서 (감염, 정점, 처리). 실험이 감염되지 않은 세포와 HIV 감염 세포를 24시간 감염 후 에서 얻는 것을 목표로 하는 경우 총 100 x 106 세포가 필요하며, 절반은 감염되지 않은 상태의 경우 절반, 감염된 상태의 절반은 필요합니다.
2. RNA 추출
- 세포에서: 페놀 클로로폼을 가진 RNA 추출
- 각 조건에 대해, 원심 분리에 의해 세포 (예를 들어,50 x 106)를 수집하고 상체를 폐기합니다.
- 각 50 x 106 셀 펠릿에 페놀 기반 시약 5mL을 추가하고 여러 번 위아래로 파이프를 사용하여 혼합합니다.
- 실온에서 5분 동안 배양하여 완전한 용해를 허용합니다. Lysed 세포는 -80 °C에 저장하거나 직접 처리 할 수 있습니다.
참고: 필요한 경우, 세포는 1.5mL 튜브에서 튜브 당 10 x 106 세포의 알리쿼트로 분할되고 보다 편리한 저장을 위해 페놀 기반 시약 1mL로 용액될 수 있습니다. - 클로로폼 1mL을 넣고 반전으로 섞습니다.
- 실온에서 3분 동안 배양하십시오.
- 2,000 x g 및 4 °C에서 15 분 동안 원심 분리기.
- 수성 상(상층)을 피펫하고 새로운 튜브로 이송합니다. 튜브를 45°로 기울여 수성 위상을 옮기고 조심스럽게 용액을 피펫으로 마무리하십시오.
참고: 수성 상의 양은 샘플마다 다를 수 있지만 시료에 첨가된 클로로폼의 양 (즉, 1mL)에 가깝어야 합니다. 어떤 단계 또는 유기 층을 전송하지 마십시오! 위상 잠금 또는 위상 메이커 튜브를 사용하면 이 프로세스를 용이하게 할 수 있습니다. - 수성 상에 100% 분자 급 이소프로판올의 0.5 mL를 추가합니다.
- RNA 강수량을 허용하기 위해 -80°C에서 1시간 동안 배양한다.
- 12,000 x g 및 4°C에서 10분 동안 원심분리기를 사용하여 침전된 RNA를 펠릿합니다.
- 상체를 버리고 RNA 펠릿을 75% 분자 생물학 등급 에탄올의 1 mL에서 재중단한다. 소용돌이가 짧게.
- 7,500 x g 및 4 °C에서 5 분 동안 원심 분리기를 하고 상체를 폐기하십시오.
- 펠릿을 15분 동안 공기 건조시합니다.
- RNase가 없는 물의 20 μL에서 펠릿을 다시 중단하고 새로운 튜브로 옮습니다.
- 빈 튜브를 추가로 20 μL의 물로 씻어 RNA 복구를 최대화하고 처음 20 μL 부피를 사용하여 풀을 하십시오.
- 분광계로 총 RNA를 정량화하고 단편 분석기로 RNA 품질을 평가합니다.
- 바이러스 성 입자에서: 컬럼 기반 바이러스 RNA 추출 키트와 RNA 추출
참고: 페놀 기반 시약을 가진 바이러스 성 입자에서 RNA 추출은 낮은 품질의 바이러스 RNA와 낮은 품질의 라이브러리에서 발생합니다. 따라서 컬럼 기반 RNA 추출을 선호해야 한다. RNA 용출 및 회수를 위한 담체 RNA를 이용한 RNA 추출 키트는 이 절차에 적합하지 않으며 피해야 합니다. HIV RNA는 폴리-아데닐화되기 때문에, 추가 mRNA 절연 없이 직접 RNA 추출은 MeRIP-Seq 및 BS-Seq 파이프라인에 들어가기에 충분합니다. 보편적으로 감염된 세포로부터의 바이러스 성 상피물의 일반적으로 1-2 mL은 전체 워크플로우를 수행하기에 충분한 RNA를 제공해야한다.- 30mL의 용해 버퍼에 베타-메르카토에탄올 150 μL을 추가하여 버퍼를 준비합니다. 100% 에탄올의 96mL를 추가하여 바이러스 성 세척 버퍼를 재구성합니다.
- 바이러스가 함유된 수퍼나티와 원심분리기를 수집하여 세포 이물질을 펠릿하여 세포 RNA 오염을 최소화합니다.
- 15 mL 튜브에 바이러스 성 상류체의 1 mL을 전송합니다.
- 바이러스 RNA 버퍼 3mL을 바이러스 성 샘플 1 mL에 넣고 소용돌이에 섞습니다.
- 수집 튜브에 삽입된 컬럼에 700 μL의 샘플을 전송합니다.
- 상온에서 13,000 x g에서 2 분 동안 원심 분리기.
- 흐름을 삭제합니다.
- 전체 샘플이 처리될 때까지 3개의 이전 단계를 반복하여 모든 RNA가 실리카 계 매트릭스 컬럼상에 포획되었다.
- 열에 바이러스 세척 버퍼 500 μL을 추가합니다.
- 상온에서 10,000 x g에서 1 분 동안 원심 분리기. 흐름을 삭제합니다.
- 열에 바이러스 세척 버퍼 200 μL을 추가합니다.
- 상온에서 10,000 x g에서 1 분 동안 원심 분리기. 흐름을 삭제합니다.
- 열을 빈 수집 튜브에 넣습니다.
- 원심분리기는 실온에서 10,000 x g에서 1분 동안 원심분리기를 사용하여 남은 세척 완충물질 오염 물질을 더 폐기합니다.
- 조심스럽게 1.5 mL 튜브로 컬럼을 전송합니다.
- 실온에서 30s에 대한 열 매트릭스와 원심 분리기의 중심에 직접 DNase / RNase가없는 물의 20 μL을 추가합니다.
- 열 매트릭스의 중앙에 직접 DNase/RNase가 없는 물의 10μL을 추가하고 원심분리기를 다시 30초 동안 추가로 추가합니다.
- 분광계로 총 RNA를 정량화하고 단편 분석기로 RNA 품질을 평가합니다.
참고: RNA 추출은 검색된 RNA의 품질이 높은 경우, RNA 무결성/품질 번호 > 9를 사용하여 임의의 방법으로 수행될 수 있다. 총 RNA는 추가 처리될 때까지 -80°C에서 저장할 수 있습니다.
3. oligo (dT)25와 폴리 A 선택에 의해 mRNA 절연
참고: 세포 추출물에서 고도로 메틸화된 리보소말 RNA의 존재로 인해, rRNA 고갈또는 폴리-A 양성 선택에 의해 폴리-A RNA를 우선적으로 분리하는 것이 좋습니다. 이 단계는 선택 사항이며 세포 RNA 샘플만 수행하여 더 높은 해상도로 시퀀싱 결과를 얻어야 합니다. 비 다중 아데닐라기 바이러스 RNA의 메틸화를 분석하는 경우, 폴리-A 선택보다는 rRNA 고갈을 선호하거나 결국 총 RNA에 대한 분석을 수행한다.
-
폴리 A 캡처를위한 비드 준비
- 올리고 (dT)25 마그네틱 비즈 주식 바이알을 >30 s에 대한 소용돌이를 다시 중단합니다.
- 자기 구슬 200 μL을 1.5mL 튜브로 옮기습니다. 처리될 RNA 샘플의 총 수량에 따라 자기 구슬이 있는 튜브수를 준비한다.
참고: 다이나비드 스톡 용액200μL을 가진 튜브 1개는 구슬 1mg에 해당하며 총 RNA 75μg 샘플을 수용할 수 있습니다. - 튜브를 자석에 1분 동안 놓고 상체를 폐기합니다. 자석에서 튜브를 제거합니다.
- 바인딩 버퍼 1mL(20m Tris-HCl, pH 7.5, 1.0 M LiCl, 2mM EDTA)를 추가하고 소용돌이에 의해 재중단됩니다. 자석에 튜브를 1분 동안 놓고 상체를 폐기합니다. 자석에서 튜브를 제거합니다. 반복하다.
- 바인딩 버퍼의 100 μL에서 세척 된 자기 구슬을 다시 일시 중지합니다.
-
총 RNA 제제
- 75 μg/100 μL에 해당하는 RNase 가 없는 물로 총 RNA를 0.75 μg/μL의 최종 농도로 희석시 희석시.
참고: RNA가 농도가 낮은 경우 볼륨을 수정하지 않고 아래에 설명된 대로 진행합니다. - Aliquot는 튜브 당 RNA 샘플 의 100 μL을 분배하여 다중 튜브에서 총 RNA를 인용합니다.
- 각 RNA 샘플에 결합 버퍼 100 μL을 추가합니다.
- 총 RNA를 65°C로 가열하여 2분 동안 가열하여 이차 구조를 방해합니다.
- 다음 단계로 나아갈 준비가 될 때까지 얼음 위에 즉시 놓습니다.
참고: 인큐베이션 시간은 처리되는 시료의 수에 따라 달라질 수 있지만 RNA 분해를 피하기 위해 1h를 초과해서는 안 됩니다.
- 75 μg/100 μL에 해당하는 RNase 가 없는 물로 총 RNA를 0.75 μg/μL의 최종 농도로 희석시 희석시.
-
폴리-A 선택
- 각 RNA 튜브(3.2단계에서)에 세척된 자기 구슬 100μL를 추가합니다(3.1단계에서).
- 위아래로 파이프를 돌리면 철저히 섞어서 실온에서 회전하는 휠에 15분 동안 바인딩을 허용합니다.
- 모든 튜브를 열고 자석에 1 분 동안 놓고 모든 상퍼를 조심스럽게 제거하십시오.
- 새로운 튜브에서 상체를 회수하고 폴리-A 최종 회복을 개선하기 위해 두 번째 RNA 포획(Step 3.3.14)을 위해 제쳐두십시오.
- 자석에서 튜브를 제거하고 세척 버퍼의 200 μL을 추가 (10m Tris-HCl, pH 7.5, 0.15 M LiCl, 1 mM EDTA). 조심스럽게 4 ~ 5 번 파이펫으로 섞는다.
- 튜브를 자석에 1분 동안 놓고 상체를 폐기합니다.
- 세척 단계를 한 번 반복합니다(반복 단계 3.3.5 및 3.3.6).
- 비드에서 폴리-A RNA를 엘루트하기 위해 얼음 감기 10m Tris-HCl 20 μL을 추가합니다.
- 80 °C에서 2 분 동안 인큐베이션하십시오.
- 자석에 튜브를 놓고 폴리-A RNA를 함유한 상체를 새로운 RNase 가 없는 튜브로 신속하게 이송합니다. 튜브를 얼음 위에 놓습니다.
- 용출 단계(단계 3.3.8 내지 3.3.10)를 반복하여 수율을 늘립니다.
- 세탁 버퍼 200 μL로 동일한 구슬을 한 번 씻으시면 됩니다. 조심스럽게 4 ~ 5 번 파이펫으로 섞는다.
- 자석에 1분 동안 놓고 세척 버퍼를 버리십시오.
- 3.3.4 단계에서 유량을 구슬에 추가하고 구속력에서 용출까지의 절차를 반복합니다(3.3.2 ~ 3.10 단계). RNA용용유는 당분간 별도의 튜브에 보관하십시오.
참고: 선택적으로, 컨트롤로 사용할 수 있으므로 새 튜브에서 3.3.4 단계와 동등한 상체를 다시 유지합니다. 절차의 끝에서, 에탄올 강수량 또는 선택의 컬럼 기반 방법 (즉, RNA 청소 및 농축기)에 의해 RNA를 정화하고 농축한다. 이 샘플은 다중 A-고갈 된 RNA 샘플에 대응하고 bisulfite 변환을위한 제어로 사용될 수있다 (단계 8.2.2). - 용출된 RNA를 분광광계로 정량화하고 2 μL 알리쿼트를 유지하여 단편 분석기로 RNA 품질을 더욱 평가합니다.
참고: 폴리-A RNA는 필요할 때까지 -80°C에 보관할 수 있다.
4. RNA 워크플로우
- 세포 폴리-A RNA(mRNA) 및 바이러스 RNA 샘플을 각각의 상피 분석 파이프라인에 전념하는 2개의 알리쿼트로 나눕니다.
(i) 세포 mRNA 5 μg 또는 MeRIP-Seq 및 입력 제어를 위한 바이러스 RNA 의 1 μg (단계 5 ~ 7, 및 단계 9로 이동).
(ii) BS-Seq에 대한 바이러스 RNA의 세포 mRNA 또는 500 ng의 1 μg (8 단계 및 9단계로 이동).
5. RNA 단편화
참고: RNA 단편화는 RNA 단편화 시약으로 수행되며 MeRIP-Seq 및 제어 RNA 샘플을 위한 것입니다. 이것은 100-200 nt 사이의 조각을 얻기 위해 신중한 최적화가 필요한 매우 중요한 단계입니다.
- mRNA의 총 부피를 mRNA/튜브의 18 μL로 0.2 mL PCR 튜브로 나눕니다.
참고: 신속하게 작업할 수 있습니다. 재현 가능한 결과를 얻으려면 한 번에 8개 이상의 샘플로 작업하지 마십시오. 볼륨을 확장해도 재현 가능하고 균일한 조각화를 보장하지는 않습니다. - 70°C에서 열순환기를 데우겠습니다.
- 각 PCR 튜브의 가장자리에 조각화 시약 2 μL을 추가합니다.
- 튜브를 닫고 스핀 다운하십시오 (시약이 8 개의 튜브에 대해 동시에 RNA와 접촉하게됩니다).
- 예열된 열순환기에서 70°C에서 15분 동안 시료를 배양한다.
- 인큐베이션이 끝난 즉시 각 튜브에 2 μL의 정지 용액을 빠르게 추가합니다.
- 스핀 다운하고 다음 단계로 진행할 준비가 될 때까지 얼음에 앉아 보자.
참고: 인큐베이션 시간은 처리되는 시료의 수에 따라 달라질 수 있지만 RNA 분해를 피하기 위해 1h를 초과해서는 안 됩니다. - 모든 샘플에 대한 절차를 반복합니다(8개 이상의 알리쿼트가 있는 경우).
- 튜브를 함께 풀링하고 RNA 청정 및 농축기 키트(6단계) 또는 맞춤형 컬럼 기반 키트로 RNA 정화를 진행하여 버퍼를 제거하고 물에 있는 깨끗한 단편화된 RNA를 회수합니다.
6. RNA 정화
참고: 이 단계는 에탄올 침전 또는 임의의 종류의 컬럼 기반 RNA 정제 및 농도 방법(즉, RNA 클린 및 농축기)에 의해 수행될 수 있다.
- 엘ute 또는 DNase /RNase가없는 물의 총 부피 50-75 μL의 정제 된 RNA를 다시 중단합니다.
참고: 열 기반 방법을 사용하는 경우 최대 복구를 위해 두 라운드의 용출을 강력하게 권장합니다. - 분광계로 정제된 단편화된 mRNA를 정량화하고 단편 분석기로 RNA 품질을 평가한다.
- 라이브러리 준비 및 시퀀싱을 위한 입력 컨트롤로 조각화된 mRNA 100 ng를 유지합니다(9단계로 이동). 나머지 단편화된 mRNA(최소 2.5 μg)는 MeRIP(7.2단계로 이동)에 사용할 수 있습니다.
7. 메립
참고: 단편화된 mRNA의 최소 2.5μg은 각 면역침전수(IP)에 필요하며, 특정 항m6A 항체(test condition)를 사용하거나 안티-IgG 항체(음성 제어)를 사용한다.
- 면역 침전을위한 자기 비드 준비
- 각 시료에 대해, mRNA IP 완충제 5배(50m Tris-HCl pH 7.4, 750mMM NaCl, 0.5% Igepal CA-630, 뉴클레아스 프리 워터)의 800 μL을 3.2mL의 뉴클레아스 워터로 희석하여 새로운 원내 튜브에 1x IP 버퍼 4mL를 준비한다.
참고: 적어도 2개의 반응이 필요합니다(테스트 1회 및 IgG 제어 1개). - 튜브를 얼음 위에 놓습니다.
- 원하는 IP 반응 수에 대해 1.5mL 마이크로원심분리기 튜브의 적절한 수에 라벨을 부착하십시오.
항m6A 항체용 n관(test).
일반 마우스 IgG용 n 튜브(음수 제어) - 반전 및 소용돌이에 의해 자기 구슬 (예를 들어, Magna ChIP 단백질 A/G)을 다시 중단합니다. 구슬 덩어리를 볼 수 없습니다.
- 계획된 각 반응에 대해, 자기 구슬의 25 μL을 마이크로 센심분리기 튜브로 옮기다.
- 사용된 구슬의 원래 부피(즉, 자기 비드의 25 μL 당 1배 IP 버퍼의 250 μL)에 대하여 10배 더 많은 1배 IP 버퍼(단계 7.1.1)를 추가합니다.
- 구슬을 여러 번 부드럽게 배관하여 혼합하여 완전한 재서스펜션을 합니다.
- 1 분 동안 자기 분리기에 튜브를 배치합니다.
- 체퍼를 제거하고 폐기하여 자기 구슬을 흡인하지 않도록 하십시오. 자석에서 튜브를 제거합니다.
- 세척 단계를 반복합니다(7.1.6~ 7.1.9단계).
- 구슬의 원래 부피의 25 μL 당 1배 IP 버퍼의 100 μL로 구슬을 재연한다.
- 자기 구슬의 원래 부피의 25 μL 당 항체의 5 μL (1 μg/μL)를 추가합니다.
항m6A 항체(클론 17-3-4-1) [1 μg/μL]을 가진 n 관(test).
일반 마우스 IgG (1 μg /μL)와 n 튜브 (음수 제어). - 자기 구슬과 항체의 연상을 허용하기 위해 실온에서 30 분 동안 회전 휠에 배양.
- 1 분 동안 자기 분리기에 튜브를 배치합니다. 상부체를 폐기합니다. 자석에서 튜브를 제거하고 1x IP 버퍼의 100 μL에서 항체 비드 혼합물을 다시 분리합니다.
- 각 시료에 대해, mRNA IP 완충제 5배(50m Tris-HCl pH 7.4, 750mMM NaCl, 0.5% Igepal CA-630, 뉴클레아스 프리 워터)의 800 μL을 3.2mL의 뉴클레아스 워터로 희석하여 새로운 원내 튜브에 1x IP 버퍼 4mL를 준비한다.
- RNA 면역 침전 (RIP)
- 다음과 같이 각 2.5 μg mRNA 샘플에 대해 RIP 반응 혼합물 500 μL을 준비하십시오: 단편화된 RNA의 100μL에서 2.5 μg (단계 6.12에서); 뉴클레아제 없는 물의 295 μL; 40 U/μL RNase 억제제의 5 μL; 및 5배 IP 버퍼의 100 μL.
- 각 항체 비드 혼합물에 RIP 반응 혼합물 500 μL을 추가합니다(7.1.14 단계에서~100 μL). 구슬을 완전히 재보페수 있도록 여러 번 부드럽게 파이펫을 섞는다. 얼음 위에 놓습니다.
- 4°C에서 2시간 동안 회전 휠에 모든 RIP 튜브를 배양합니다.
- 원심 분리는 MeRIP 반응을 간략하게 반응하여 캡과 튜브 측면에서 액체 방울을 회전시합니다. 튜브를 자기 분리기위에 1분 동안 놓습니다.
- 새로운 원심분리기 튜브에서 상체를 옮기고 자기 구슬을 방해하지 않도록 주의한다.
참고: 플로우루는 RIP 효율을 확인하기 위한 제어로 보관할 수 있습니다(7.3.9단계로 이동). - 자석에서 튜브를 제거합니다. 차가운 1x IP 버퍼의 500 μL을 추가하여 구슬을 씻으세요. 구슬을 여러 번 부드럽게 배관하여 구슬을 완전히 재보페팅합니다.
- 튜브를 자기 분리기위에 1분 동안 놓고 상체를 폐기합니다.
- 총 3개의 세척에 대해 세척 절차(단계 7.2.6-7.7.7)를 두 번 반복합니다.
- 튜브를 얼음 위에 놓고 즉시 용해로 진행합니다.
- 용출
- 20mMm6A 용액을 10 mg의 N6-메틸라데로신, 5′모노포세산염 나트륨(m6A)을 1.3mL의 뉴클레아제 없는 물로 용해시게 한다. 150 μL 알리코를 준비하고 -20 °C에 보관하십시오.
- 각 시료(테스트 및 제어):5배 IP 버퍼 45μL, 20mM m6A 75μL, 40U/μL RNase 억제제의 3.5 μL, 뉴클레아자유수의 101.5 μL 등 다음 성분을 혼합하여 225μL의 용출 버퍼를 준비한다.
- 용출 버퍼 100 μL(7.3.2 단계)을 구슬에 추가합니다(7.2.9 단계부터). 구슬을 완전히 재보페수 있도록 여러 번 부드럽게 파이펫을 섞는다.
- 4 °C에서 로커에 연속 흔들림으로 모든 튜브를 1 시간 동안 배양하십시오.
- RIP 반응을 원심분리하여 캡과 튜브 측면에서 액체 방울을 회전시합니다. 튜브를 자기 분리기위에 1분 동안 놓습니다.
- 용출된 RNA 단편을 포함하는 상체를 새로운 1.5 mL 마이크로센심분리기 튜브로 이송한다. 비드를 흡인하지 않도록 주의하십시오.
- 용출 단계(7.3.3 ~ 7.3.6)를 다시 100 μL의 용출 버퍼를 추가하고, 4°C에서 1h를 배양하고, 자기 분리 후 용출을 수집한다.
- 동일한 샘플에서 모든 용출을 결합합니다(총 용출 부피는 200 μL이어야 합니다).
- 에탄올 강수량 또는 열 기반 선택 방법(즉, RNA 클린 및 농축기)에 의해 용출된 RNA 및 유량(7.2.5단계으로부터 선택 사항)을 정화합니다.
- 고감도 검출 키트를 사용하여 단편 분석기를 사용하여 RNA 수량 및 유량 및 용출 된 샘플을 평가합니다. RNA의 품질이 만족하는 경우, 라이브러리 준비 및 높은 처리량 시퀀싱(단계 9)을 진행한다.
참고: MeRIP에서 검색된 RNA의 양은 매우 낮으며, 정량화를 보장하기 위해 반드시 고감도 검출 키트가 필요합니다. 생체 분석기를 사용할 수 없는 경우, 도서관 준비에 맹목적으로 진행할 수 있다.
8. RNA 비설피테 변환
- 제어 및 시약 준비
- ERCC 믹스 스파이크 인 제어: mRNA의 500 ng에 희석되지 않은 ERCC 믹스의 0.5 μL을 추가하는 것이 좋습니다 제조업체의 지시에 따라 ERCC 믹스를 추가합니다. 이 제어는 비설피트 변환의 효율성을 평가하는 데 도움이 될 수 있습니다.
- 스파이크 폴리-A 고갈 RNA (단계로부터 3.3.14) 비율 1/1000 (즉, mRNA의 500 ng에 대한 폴리 A 고갈 RNA의 500 pg). 이 샘플은 리보소말 RNA에서 농축되어 따라서 28S rRNA, 양성 조절을 포함해야 한다.
참고: 총 RNA는 또한 다중 A 고갈 RNA 대신 양성 대조군으로 사용될 수 있습니다. - RNA 메틸화 키트(예: Zymo EZ)로 비설피트 변환을 수행합니다.
- RNA 세척 버퍼: 사용하기 전에 RNA 세척 버퍼 농축물의 12mL에 100% 에탄올(또는 95% 에탄올의 52mL)의 48mL을 추가합니다.
- 비설피트 변환
참고: 비설피테 변환은 아래에 명시된 대로 제조업체의 절차에 따라 상업적으로 이용 가능한 RNA bisulfite 변환 키트로 수행되었다.- 0.2 mL PCR 튜브에서 1000 ng의 mRNA (또는 300 에서 1000 ng 사이)를 추가하십시오. 스파이크 인 컨트롤 추가 : ERCC 믹스의 1 μL (단계 8.1.1) 및 폴리 A 고갈 RNA의 1000 pg (단계 8.1.2). DNase/RNase가 없는 물로 최대 20μL의 완전한 부피를 완성합니다.
- RNA 변환 시약 130μL을 각 20 μL RNA 샘플에 추가합니다.
- 위아래로 파이프를 사용하여 샘플을 혼합합니다.
- 튜브의 캡이나 측면에 물방울이 없는지 확인하기 위해 잠시 회전하십시오.
- PCR 튜브를 열 사이클러에 놓고 다음 단계를 수행하십시오: 5분 동안 70°C에서 의 분해; 45 분 동안 54 °C에서 변환; 총 3사이클에 대한 반복적인 거부 및 변환 단계; 그런 다음 4 °C에서 무기한 으로 유지하십시오.
참고: 3주기의 데마산및 비황피트 변환은 시료의 완전한 비황피트 변환을 보장합니다. 샘플은 -80°C에 저장하거나 직접 처리할 수 있습니다. - 열 내 탈황을 진행합니다. 컬럼을 빈 수집 튜브에 넣고 컬럼에 RNA 바인딩 버퍼 250 μL을 추가합니다.
- RNA 바인딩 버퍼를 포함하는 컬럼에 샘플(~150 μL)을 로드하고 위아래로 배관하여 혼합합니다.
- 매컬의 샘플 RNA 결합 버퍼 혼합물에 95-100% 에탄올의 400 μL을 추가합니다. 캡을 닫고 열을 여러 번 반전하여 즉시 섞습니다.
- 30 초용 최고 속도 (≥ 10,000 x g)의 원심 분리기. 흐름을 삭제합니다.
- RNA 워시 버퍼 200μL을 컬럼에 넣고 원심분리기를 최고 속도로 30초간 추가합니다.
- RNA 황전 버퍼 200μL을 컬럼에 넣고 실온에서 30분 동안 배양합니다. 인큐베이션 후, 30 초 전속력의 원심분리기. 흐름을 삭제합니다.
- RNA 워시 버퍼 400μL을 컬럼에 넣고 원심분리기를 최고 속도로 30초간 추가합니다. RNA 세척 버퍼의 추가 400 μL로 세척 단계를 반복합니다. 흐름을 삭제합니다.
- 2 분 동안 최고 속도로 비워진 컬렉션 튜브의 컬럼을 원심 분리합니다. 컬럼을 RNase 가 없는 튜브로 옮기십시오.
- 단열 매트릭스에 직접 DNase/RNase가 없는 물의 ≥ 10μL을 추가하고 실온에서 1분 동안 배양합니다. 30 초전속의 원심분리기.
참고: 우리는 보통 20 μL의 부피로 엘ute. 용출 된 RNA는 즉시 사용하거나 최대 3 개월 동안 -20 °C에서 저장할 수 있습니다. 장기 보관을 위해 -80°C로 유지하십시오. - RNA 품질 및 수량의 단편 분석기 평가를 위해 2.5 μL을 꺼내 서고 준비 및 고처리량 시퀀싱(단계 9)을 진행한다.
- 효율의 편유피 변환 제어를 위해 변환 된 RNA의 4 μL을 가져 가라 (단계 8.3).
- RT-PCR에 의한 비설피트 변환 제어
참고: 이 단계는 시퀀싱을 진행하기 전에 비설피트 변환이 성공했습니다. 호모 사피엔스의 28S 리보소말 RNA는 RNA 메틸화 분석에 대한 양성 대조군으로 사용될 것이며, C 잔류물은 4447(GenBank 가입 #NR_003287)로 100% 메틸화된 것으로 설명되고 있다.
프라이머 시퀀스:
H 28SF 프라이머: 5'-GGGGTTAYGATTTTTTTTTTGGG-3'
H 28SR 프라이머: 5'-CCAACTCACRTTCTATTAATAATAATAAC-3'- 대용량 cDNA 역전사 키트를 사용하여 역전사(RT) 반응 믹스를 준비한다. 얼음에 키트 구성 요소를 해동하고 다음과 같이 얼음에 RT 마스터 믹스를 준비 :
비술피테의 4 μL 변환 RNA (단계 8.2.14에서):
10xRT 버퍼의 2 μL
0.8 μL 의 25배 dNTP 믹스 [100mMM]
10x RT 랜덤 프라이머 의 2 μL
멀티스크리브 역전사 1μL
RNase 억제제 의 1 μL
뉴클레아제 프리 H2O의 9.2 μL
참고: 각 RT 반응에는 0.2mL PCR 튜브의 20 μL 최종 부피가 포함되어야 합니다. - 다음 RT 프로그램과 함께 열 사이클러에 튜브를 넣어 : 10 분 동안 25 °C; 120 분 동안 37 °C; 5 분 동안 85 °C; 그런 다음 4 °C에서 무기한.
- PCR 교정 효소를 사용하여 28S rRNA를 증폭시키기 위해 PCR 반응을 준비합니다. 키트 구성 요소를 얼음, 부드럽게 소용돌이 및 간략하게 원심분리기에 적어 보냅니다. 다음과 같이 얼음이나 얼음 냉철 판 홀더에 PCR 마스터 믹스를 준비하십시오.
10 μM H 28SF 프라이머의 0.6 μL
10 μM H 28SF 프라이머의 0.6 μL
템플릿 cDNA의 6.5 μL
DNA 폴리머라제 마스터 믹스의 22.5 μL
참고: 각 PCR 반응에는 0.2mL PCR 튜브의 20 μL 최종 부피가 포함되어야 합니다. - 다음 PCR 프로그램과 함께 열 사이클러에 튜브를 넣어 : 5 분 동안 95 °C에서 초기 분해; 45사이클의 데나포화(15s용 95°C), 아닐링(30초동안 57°C), 신장(15초동안 72°C), 72°C에서 10분 동안 최종 신장, 그리고 4°C에서 무기한 으로 유지한다.
- 2% 아가로즈 젤에 반응의 10 μL을 실행합니다. 예상 대역 크기는 130 - 200 bp입니다.
- 대용량 cDNA 역전사 키트를 사용하여 역전사(RT) 반응 믹스를 준비한다. 얼음에 키트 구성 요소를 해동하고 다음과 같이 얼음에 RT 마스터 믹스를 준비 :
- PCR 제품의 시퀀싱
- 효소및 dNTP 잔류물을 제거하고 증폭된 DNA를 DNase/RNase 가 없는 물의 최소 20μL에서 염화한 DNA를 예테하는 것이 선택되는 컬럼 기반 방법으로 PCR 제품을 정화합니다.
- 정제된 DNA를 분광광계로 정량화합니다.
- 시퀀싱 반응
- PCR 제품/염기서열 분석 반응의 40 ng를 사용합니다.
- H 28SF 및 H 28SR 프라이머를 사용 하 여 양방향으로 서열.
- 알려진 비변환 시퀀스와 서열을 정렬 (28S 리보소말 N5 (RNA28SN5). 위치 C4447에서 C 잔류물의 존재 여부를 확인하고 다른 곳에서 C 대신 T 잔류물의 경우.
9. 라이브러리 준비 및 고처리량 시퀀싱
- elute-Prime-Fragment 단계에서 프로토콜을 시작하고 제조업체 지침을 따르기 위해 mRNA 키트(예: 일루미나 TruSeq Stranded)를 사용하여 시퀀싱을 위한 라이브러리를 준비합니다.
- 그러나, 입력 RNA-Seq 및 MeRIP-Seq 샘플의 경우, 샘플을 80°C에서 2분 동안 배양하여 전성기에만 만주하지만 더 이상 단편화하지는 않는다.
- 일루미나 플랫폼을 사용하여 시퀀싱을 수행합니다. 시퀀싱 반응은 최소 100nt 길이로 단일 또는 페어링 된 끝 중 하나 또는 쌍의 종결환경 및 실험 설계에 따라 수행 될 수 있습니다.
10. 생물정보학 분석
- m6A 데이터 처리
- FASTQC24를 실행하여 m6A의 판독 품질을 평가하고 시퀀싱에서 FASTQ 파일을 입력합니다.
- Atropos25를 실행하여 판독에서 저품질 엔드 및 어댑터 시퀀스를 다듬습니다. Atropos를 실행에서 다음 매개 변수를 설정합니다.
- AGATCGGAAGAG, CTCTTCCGATCT, AACACTCTTTCCCT, AGATCGGAAGAGCG, AGGGAAAGAGTGTT, CGCTCTTCCGATCT : 다음과 같은 어댑터 시퀀스를 제거합니다.
- 제조업체(https://support.illumina.com/downloads/illumina-adapter-sequences-document-1000000002694.html)가 지정한 저품질 단자를 트리밍하기 위해 5.
- 트리밍 후 다음 최소 읽기 길이를 사용합니다: 25 개의 기본 쌍.
- GRh38 인간 게놈 및 HIV [통합 선형 pNL4-3Env-GFP] 참조를 FASTA 형식으로 병합합니다.
- 합병된 참조를 HISAT226으로 인덱싱합니다.
- 트리밍된 읽기에서 HISAT2를 실행하여 인덱싱된 참조에 정렬합니다. 기본 HISAT 매개 변수를 사용합니다.
- 정렬된 읽기를 SAMtools27로 정렬하고 인덱싱합니다.
- SAMtools 통계 및 Qualimap 228을 실행하여 시퀀스 라이브러리의 정렬 후 품질 검사를 위해 실행합니다.
- 선택적으로 multiQC29를 사용하면 이전 단계의 품질 측정값을 수집하고 요약합니다.
- HIV 게놈은 5'LTR 및 3'LTR에서 동종 634 bp 서열을 가지고 있습니다: SAMtools를 가진 해당 3' LTR 지구에 5'LTR에서 멀티 매핑 판독을 재조정합니다.
- m6A 피크를 식별하려면 피크 호출 소프트웨어 MACS230(v 2.1.2)을 실행합니다. MACS2 실행 매개변수를 신중하게 선택하여, 최고 호출이 유전자 발현 수준에 의해 영향을 받을 수 있고, 짧은 엑소온이 피크로 잘못 호출될 수 있기 때문에 RNA-Seq 데이터에 대한 올바른 기능을 보장하기 위해. 따라서 MACS2에서 DNA 기반 데이터에 일상적으로 적용하지 않고 입력 신호를 m6A 신호에서 빼야 합니다. MACS2의 '콜피크' 하위 명령에 다음 매개 변수를 적용합니다.
- 유지 DUP 자동 (중복 읽기로 MACS2 동작을 제어, '자동'MACS는 p 값 컷오프로 1e-5를 사용하여 이소미 분포를 기반으로 정확히 동일한 위치에서 읽기의 최대 수를 계산 할 수 있습니다)
-g 2.7e9(bp의 인간 게놈 크기)
-q 0.01 (최소 FDR 컷오프가 중요한 피크를 호출)
- 노모델 (ChIP-Seq 실험에 맞게 조정된 변속 모델 구축 우회)
-slocal 0
-llocal 0 (이 및 이전 매개 변수를 0으로 설정하면 MACS2가 매끄럽지 않고 직접 빼고 m6A 읽기에서 입력이 읽습니다).
-내전 100(bp의 조각의 평균 길이)
-B - MACS2의 하위 명령을 호출하는 차동 피크를 실행, 'bdgdiff' 감염된 대 비 감염 된 샘플을 비교합니다. 'bdgdiff'는 이전 단계에서 'callpeak'에 의해 생성된 bedGraph 파일을 입력하는 것으로 수행됩니다. 각 시간 지점에 대해, 감염된 대 비감염 시료를 'bdgdiff'로 비교하여 m6A 신호로부터 각각의 입력 신호를 빼고 추가 매개변수를 제공합니다: -g 60-l 120.
- m5C 데이터 처리
- Cutadapt31을 실행하여 다음 매개 변수를 사용하여 원시 읽기에서 어댑터 시퀀스를 다듬습니다.
어댑터 "아가트CG가가카카크GTGTCTGAAC"
- 최소 길이 =25. - 시퀀싱 프로토콜이 역방향 가닥에서 읽기를 생성하므로 seqkit32를 사용하여 트리밍된 읽기를 역으로 보완합니다.
- FastQC를 실행하여 판독 품질을 검사합니다.
- FASTA 형식으로 GRh38 인간 게놈 및 HIV[통합 선형 pNL4-3Env-GFP] 참조를 병합합니다.
- 메란TK 패키지33에서 응용 프로그램 meRanGh와 병합 된 참조를 색인.
- 다음과 같은 매개 변수와 meRanGh와 정렬 :
- UN을 가능하게 하는 매핑되지 않은 읽기는 출력 파일에 기록될 수 있습니다.
-MM 을 사용하면 멀티 매핑된 읽기를 출력 파일에 기록할 수 있습니다.
-베드그래프출력용 bg
-mbgc 10 필터 커버리지에 의해 보고 된 지역 (적어도 10 판독 범위) - HIV 게놈은 5'LTR 및 3'LTR에서 동종 634 bp 서열을 가지고 있습니다: 멀티 매핑 판독을 5' LTR에서 SAMtools를 사용한 해당 3' LTR 영역으로 재정렬됩니다.
- meRanTK에서 제공하는 meRanCall 도구를 통해 메틸화 호출을 실행합니다.
-rl = 126, 읽기 길이
-ei = 0.1, 메틸화 율 p-값 계산에 대한 오차 간격
-cr = 0.99, 예상 전환 - 각 샘플의 크기 요인을 추정하기 위해 MeRanTK의 유틸리티 estimateSizeFactors.pl 실행합니다. 크기 요소는 다음 단계에서 매개 변수로 사용됩니다.
- 실행 MeRanCompare 비감염 대 시간 점 12, 24 및 36h에 걸쳐 감염의 차동 메틸화 분석을 위해 비교합니다. 다음 매개 변수가 적용됩니다: 이전 단계의 보고 및 크기 요인에 대한 최소 임계값으로 .01의 중요도 값이 적용됩니다.
- Cutadapt31을 실행하여 다음 매개 변수를 사용하여 원시 읽기에서 어댑터 시퀀스를 다듬습니다.
결과
이 워크플로우는 HIV 감염의 맥락에서 m6A 및 m5C 메틸화의 역할을 조사하는 데 유용합니다. 이를 위해, 우리는 우리가 HIV에 감염하거나 치료되지 않은 떠나는 CD4+ T 세포주 모형 (SupT1)를 사용했습니다. 우리는 조건당 5천만 개의 세포로 워크플로우를 시작하고 RNA 질 수가 10 (그림 1A-B)로 총 RNA의 평균 500 μg를 얻었습니다. 폴리-A 선택에 따라 조건당 mRNA의 10 에서 12 μg 사이를 검색합니다 (총 RNA의 약 2 %를 나타냅니다)(그림 1B). 이 시점에서, 우리는 MeRIP-Seq 파이프라인에 대한 폴리 A 선택 RNA 5 μg와 BS-Seq 파이프라인에 대한 1 μg를 사용했습니다. HIV RNA는 폴리-아덴레이트되기 때문에 추가 조치가 필요하지 않으며 MeRIP-Seq 및 BS-Seq 절차를 직접 적용할 수 있습니다.
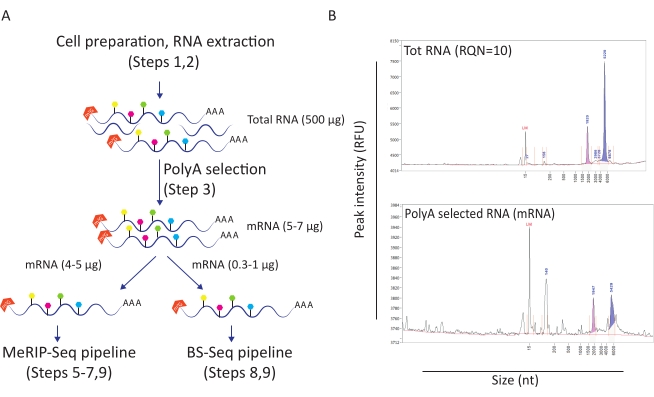
그림 1: 다운스트림 응용 을 위한 RNA 준비. A) 동시 MeRIP-Seq 및 BS-Seq 파이프라인에 대한 RNA 준비 및 분포를 묘사하는 워크플로우. 채워진 모든 육각형 형상은 m6A(녹색) 또는 m5C(분홍색)와 같은 RNA 변형 유형을 나타낸다. 실험을 수행하는 데 필요한 RNA 물질의 양이 표시됩니다. B) 총 RNA 추출(상부 패널) 및 다중A 선택(하부 패널)에 대한 예상 RNA 분포 프로파일(크기 및 양)을 기재하는 대표적인 결과. 샘플은 특정 MeRIP-Seq 및 BS-Seq 절차를 입력하기 전에 RNA 품질을 평가하기 위해 표준 감도 키트를 사용하여 단편 분석기에 로드되었습니다. RQN: RNA 품질 번호; nt: 뉴클레오티드. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
MeRIP-Seq 파이프라인은 RNA 분자를 따라 m6A 변형을 조사할 수 있는 RNA 면역 침전 기반 기술입니다. 이를 위해, RNA는 면역 침전 및 포획을 위해 자기 구슬에 결합된 m6A 특이적 항체로 먼저 단편화되고 그 후 배양된다. MeRIP 농축 RNA 단편및 손길이 닿지 않은 (입력) 분획은 m6A 변형 RNA 영역과 따라서 m6A 메틸화 된 전사체를 식별하기 위해 시퀀스되고 비교된다 (도 2A). 기술의 해상도는 RNA 단편화의 효율성에 의존한다. 실제로, 짧은 조각은 m6A 잔류물의 보다 정확한 국소화를 가능하게 합니다. 여기서, 세포 다중-A 선택 된 RNA 및 바이러스 RNA는 100-150 nt의 RNA 단편을 얻기 위해 20 μL 최종 부피에서 15 분 동안 RNA 단편화 버퍼로 이온 기반 단편화를 실시하였다. mRNA의 5 μg로 시작하여, 우리는 90 %의 회복 속도에 해당하는 단편화된 RNA의 4.5 μg를 회수했습니다 (그림 2B). 우리는 라이브러리 준비 및 시퀀싱을 직접 실시입력 제어로 단편화, 정제 된 RNA의 100 ng를 사용했다. 나머지 RNA(~4.4 μg)는 MeRIP-Seq 파이프라인에 따라 처리되었으며, 이는 항체가 있는 단편화된 RNA의 배양으로 시작하여 항m6A 특이적 항체또는 반대로 IgG 항체를 대조군으로 결합하였다. m6A 특이적 RIP(MeRIP)의 단편화된 RNA의 2.5 μg는 라이브러리 준비 및 시퀀싱(도 2B)을 받은 m6A 농축 물질의 약 15ng을 회수할 수 있었습니다. 예상대로 안티-IgG 제어를 가진 RIP는 추가 분석을 허용할 충분한 RNA를 산출하지 못했습니다(도 2B).
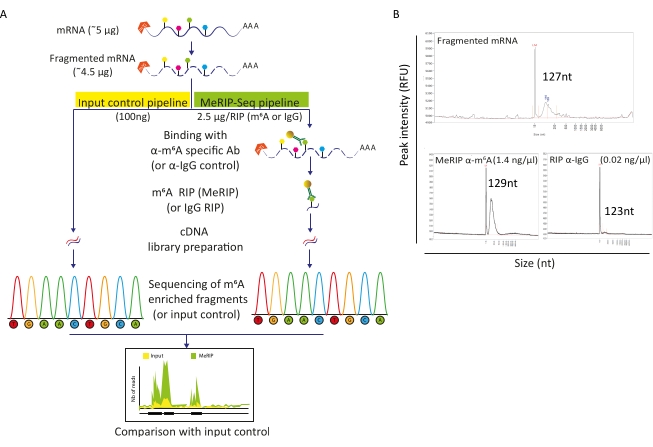
그림 2: MeRIP-Seq 파이프라인. A) MeRIP-Seq 워크플로우 및 입력 제어의 회로도 표현. 폴리-A 선택에 따라 시료는 120-150nt 조각으로 단편화되었고, 시퀀싱(100 ng, 입력 제어)을 직접 실시하거나, 항체 를 이용한 RNA 면역침전(2.5 μg, RIP)에 사용되는 경우, 시퀀싱 전에 음의 대조군으로 사용하였다. B) 단편화(상부 패널) 및 RIP(하부 패널, MeRIP: 왼쪽, IgG 제어: 오른쪽)에 기대되는 RNA 분포 프로파일(크기 및 양)을 나타내는 대표적인 결과. 샘플은 라이브러리 준비 및 시퀀싱에 추가 처리하기 전에 RNA 품질과 농도를 평가하기 위해 단편 분석기에 로드되었습니다. 단편화된 RNA 분석은 RNA 표준 감도 키트를 사용하여 수행되었으며 면역 침전 RNA는 고감도 키트를 사용했다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
BS-Seq 파이프라인은 뉴클레오티드 해상도에서 m5C RNA 변형을 탐색할 수 있으며 m5C-메틸화 된 성적 증명서의 식별으로 이어집니다. 비황피티 전환시 메틸화된 사이토신은 우라실로 변환되고 메틸화된 사이토신은 변하지 않습니다(그림 3A). 비설피테 변환 절차 (예: 고온 및 낮은 pH)의 열악한 조건으로 인해 변환된 mRNA는 고도로 저하(그림 3B)이지만, 이는 도서관 준비 및 시퀀싱을 방해하지 않습니다. Bisulfite 변환은 단 하나 좌초 RNA에 만 효율적이므로 잠재적으로 이차 이중 좌초 RNA 구조물에 의해 방해될 수 있습니다. C-U 변환의 효율성을 평가하기 위해 두 개의 컨트롤을 도입했습니다. 긍정적인 대조군으로서, 우리는 28S rRNA23의 위치 C4447에서 고도로 메틸화 된 사이토신의 이전에 설명 된 존재를 이용했다. RT-PCR 증폭 및 메틸화 부위를 둘러싼 200 bp 단편의 시퀀싱에 따라 우리는 모든 사이토신이 우라실로 성공적으로 변환되어 DNA 서열에서 티미딘으로 나타나는 것을 관찰할 수 있었으며, 그 결과, 위치 4447의 시토신을 제외하고는 DNA 서열에서 티미딘으로 나타났다. 비설피테 전환율에 대한 대조군으로서, 우리는 시판되는 합성 ERCC RNA 서열을 사용했습니다. 이 혼합물은 알려진, 비 메틸화 및 다중 아데니레이션 RNA 서열의 풀로 구성되며, 다양한 이차 구조 및 길이를 가진다. 라이브러리 준비 및 시퀀싱시, 우리는 모든 ERCC 서열 및 각 샘플에서 총 C 잔류물 중 변환 된 C의 수를 계산하여 수행 할 수있는 전환율을 계산하기 위해 이러한 ERCC 서열에 초점을 맞추었다. 전환율은 99.5%로 비설피트 전환 반응의 효율과 성공을 확인하였다(그림 3D).
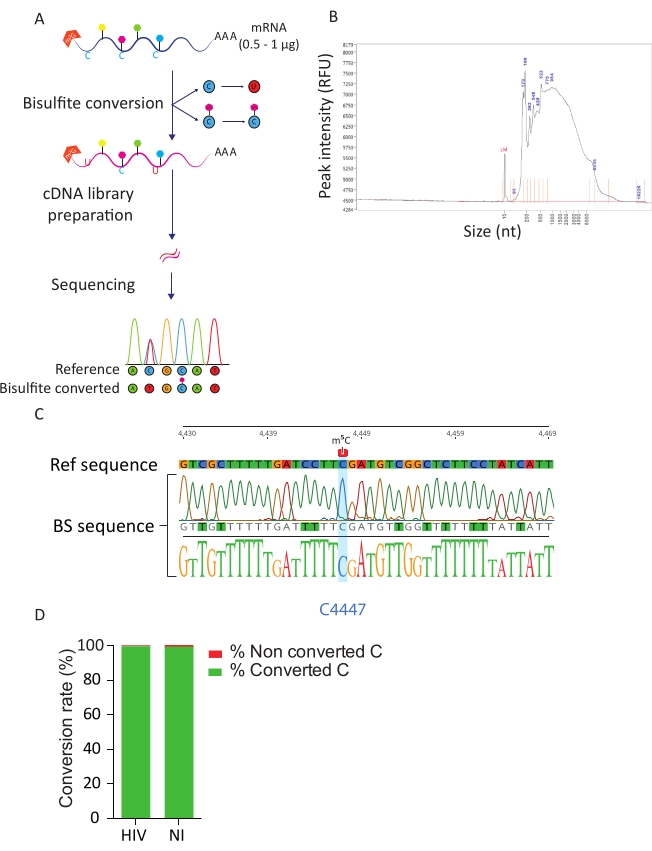
그림 3: BS-Seq 파이프라인. A) BS-Seq 워크플로우의 회로도 표현. 폴리A 선택에 따라 샘플은 비황핏에 노출되어, 비메틸화 C 잔류물의 C변환(탈약으로 인한)이 발생합니다. 대조적으로, 메틸화 된 C 잔류물 (m5C)는 양성환 처리에 의해 영향을 받지 않으며 변경되지 않습니다. B) 표준 감도 키트를 가진 단편 분석기에 대한 분석 시 비설피테 변환 RNA 분포 프로파일(크기 및 양)의 대표적인 결과. C) 28S rRNA에서 위치 4447에서 100% 메틸화 C를 둘러싼 지역의 RT-PCR 앰플리콘의 대표적인 시퀀싱 결과를 나타내는 일렉트로페로그램(파란색으로 강조). 대조적으로, 기준 서열의 C 잔류물은 양성환 변환 성공으로 인한 앰플리콘 서열에서 T 잔류물으로 확인되었다. D) HIV 감염 및 비감염 된 세포에서 ERCC 스파이크 인 서열의 분석에 의한 C-U 전환율의 평가. 평균 전환율은 99.5%입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
M6A 농축 시료, 비설피테 변환된 샘플 및 입력 제어는 라이브러리 준비, 시퀀싱 및 생물정보 분석(그림 4)을 위해 추가로 처리됩니다. 실험 설계 및 생물학적 질문에 따르면 다중 생체 정보 분석을 적용할 수 있습니다. 원칙의 증거로, 우리는 HIV 감염에 유도 된 차별화 메틸화 성적 증명서의 식별에 초점을 맞춘 하나의 잠재적 인 응용 프로그램 (즉, 차동 메틸화 분석)에서 대표적인 결과를 보여줍니다. 간단히, 우리는 바이러스 성 수명 주기 동안 RNA 메틸화의 역할을 더 이해하기 위해 유전자 발현 수준과 독립적으로 유전자 발현 수준과 독립적으로 m6A 또는 m5C 메틸화 수준을 조사했습니다. 유전자 발현 정상화시, 우리는 ZNF469 전사체가 감염 상태에 따라 m6A-메틸화되었다는 것을 확인했으며, 실제로 이 성적증명서는 HIV 감염 시 여러 메틸화 피크를 표시하는 동안 비감염된 세포에서 메틸화되지 않았다는 것을 확인했습니다(그림 5A). m5C에 유사한 차동 메틸화 분석은 PHLPP1 성적 증명서가 HIV 상태에서 더 자주 메틸화되는 경향이 있는 몇몇 메틸화 된 잔류물을 포함하는 것으로 나타났습니다 (도 5B). 이 맥락에서, 두 분석은 HIV 감염이 세포 에피레마에 영향을 미친다는 것을 건의합니다.
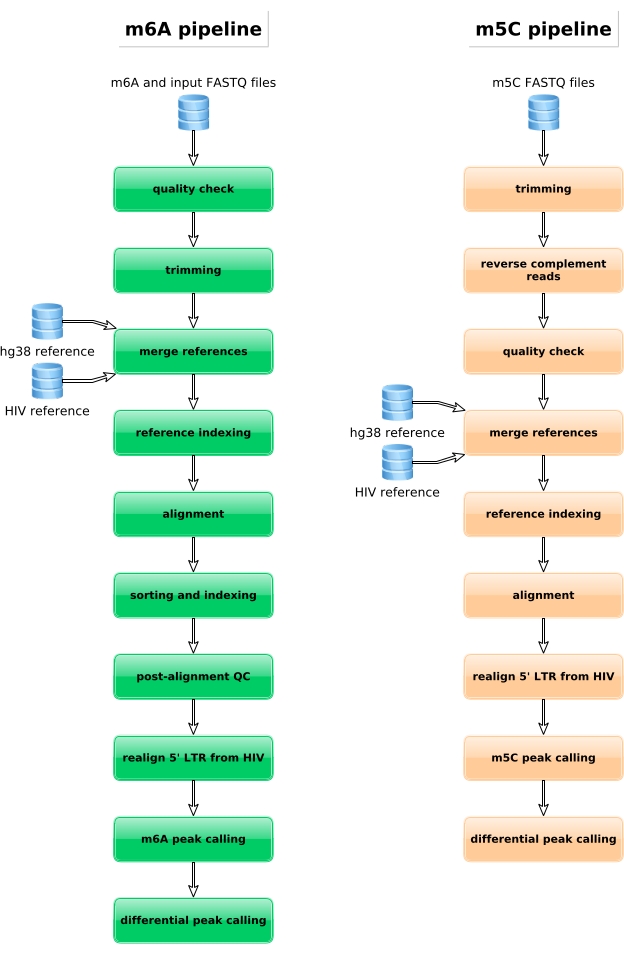
그림 4: m6A 및 m5C 데이터 분석을 위한 생물학적 워크플로우의 회로도 표현.
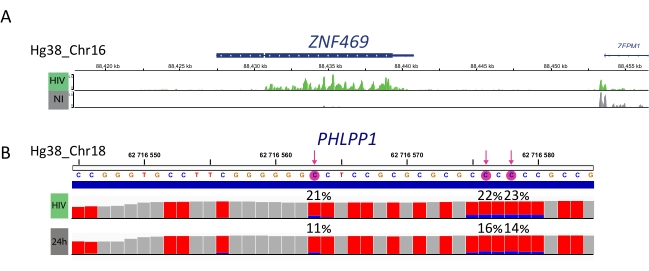
그림 5: 감염 시 차분하게 메틸화된 성적증명서의 예. A) HIV 감염(green) 및 비감염(grey) 세포에서 ZNF459 전사체의 m6A 메틸화를 보여주는 대표적인 결과. 피크 강도(입력 식 빼기 시)는 x축을 따라 염색체의 y축 및 위치에 표시됩니다. 차동 메틸화 분석은 ZFN469 성적 증명서가 HIV 감염시 과구화된다는 것을 보여줍니다. B) HIV 감염(상부 차선) 및 비감염(하부 차선) 세포에서 m5C 메틸화 유전자의 대표적인 결과. 각 막대의 높이는 뉴클레오티드당 읽기 수를 나타내며 커버리지 평가를 허용합니다. 각 C 잔류물은 빨간색으로 표시되고 메틸화 된 C의 비율이 파란색으로 표시됩니다. 정확한 메틸화 율(%)은 각 C 잔류물 위에 보고됩니다. 화살표는 IGV 뷰어를 사용하여 통계적으로 유의한 분화 된 C. 샘플을 강조합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
바이러스 감염에 있는 RNA 수정의 역할은 아직도 크게 알려지지 않습니다. 바이러스 감염의 맥락에서 epitranscriptomic 수정의 역할의 더 나은 이해는 새로운 항 바이러스 치료 목표에 대 한 탐구에 기여할 수 있습니다.
이 작품에서는 감염된 세포의 m6A 및 m5C 에피레모니오크를 조사할 수 있는 완벽한 워크플로우를 제공합니다. 생물학적 질문에 따라 폴리A 선택 RNA를 시동 재료로 사용하는 것이 좋습니다. 비록 선택적이지만, 파이프라인은 총 RNA와 함께 사용될 수 있기 때문에 rRNA뿐만 아니라 작은 RNA가 고도로 수정되고 메틸화 잔류물의 중요한 수를 포함하는 것을 명심하는 것이 중요합니다. 이로 인해 의미 있는 시퀀싱 데이터의 품질과 수량이 감소할 수 있습니다.
그러나, 연구의 초점이 비다성 RNA인 경우, RNA 추출 단계는 작은 RNA(컬럼 계 RNA 추출의 경우)를 폐기하지 않도록 하고, 파이프라인에 진입하기 위해 다중A 선택보다는 리보솜 고갈 기술을 특권하기 위해 적응되어야 한다.
고품질 RNA, 올바른 단편화 및 적절한 m6A 농축 및 BS 변환 RNA 품질을 보장하기 위해 라이브러리 준비를 위해 RNA 품질을 변환하여 단편 분석기 또는 생체 분석기를 사용하는 것이 좋습니다. 그러나 이 장비가 항상 사용할 수 있는 것은 아닙니다. 대안으로, RNA의 품질, mRNA 및 단편화된 RNA의 크기는 또한 아가로즈 젤에 시각화에 의해 평가될 수 있었습니다. 대안적으로, 라이브러리 제제는 RNA 수량에 대한 이전의 평가 없이 수행될 수 있다.
우리는 m6A 에피레마레토믹 풍경을 탐구하기 위해 항체 기반의 MeRIP-Seq16 기술을 사용했습니다. 이 기술은 RNA 면역 침전을 기반으로 하고 성공적입니다; 그러나 일부 단계는 신중한 최적화가 필요하며 중요할 수 있습니다. m6A 메틸화는 주로 합의 서열 RRA*CH 내에서 발생하도록 기술되었지만, 이 모티프는 mRNA 분자를 따라 매우 빈번하며 메틸화 부위의 정확한 식별을 허용하지 않는다. 따라서 RIP 기반 해상도를 개선하기 위해, 작은 RNA 단편을 생성, 재현 가능하고 일관된 RNA 단편화를 달성하는 것이 중요합니다. 이 프로토콜에서는 최적화된 절차를 권장하며 실험 환경에서 재현 가능하고 일관된 결과를 제공하는 것이 좋습니다. 그러나 이러한 조각화 단계는 특정 샘플 피쳐에 따라 추가 최적화가 필요할 수 있습니다.
최근에는 m6A 직접 시퀀싱을 허용하는 새로운 기술이 설명되었다. 그것은 m6A RNA 수정을 접는 것에 대한 응답으로 독특한 RT 서명을 나타내는 특정 역 전사 변이체의 사용을 기반으로합니다24. 이 기술은 신중한 최적화시 MeRIP-Seq(초기 재료의 양을 줄이고 더 높은 해상도를 허용)에 직면한 주요 한계를 우회할 수 있습니다. m5C 수정을 탐구하기 위해 우리는 뉴클레오티드 해상도에서 수정된 C 잔류물을 검출하기 위해 비설피테 변환 기술을 사용하기로 결정했습니다. RNA 이차 구조의 존재로 인한 거짓 양성률을 줄이기 위해 ERCC 스파이크인 컨트롤의 사용으로 인해 3주기의 내성/양성환 변환을 수행하고 양성환 전환율 성능을 더욱 조절했습니다. 이 기술에 연결된 한계 의 한개는 bisulfite 변환이 아주 가혹하고 데니션/bisulfite 변환의 3 주기는 몇몇 RNA를 저하시키고 그러므로 해결책을 감소시킬 수 있었다는 것입니다. 그러나 설정에서 데이터 집합의 품질을 높이기 위해 잠재적으로 약간 낮은 해상도에 정착하기로 결정했습니다.
이러한 최적화 및 제어 덕분에 바이러스 감염, 숙주 병원체 상호 작용 또는 특정 치료에 대한 노출의 맥락에서 상피 지형 및 변경 사항을 조사하기 위해 악용 할 수있는 신뢰할 수있는 사운드 워크플로우를 제공 할 수있었습니다.
공개
저자는 공개 할 것이 없습니다.
감사의 말
이 작품은 스위스 국립 과학 재단 (31003A_166412 보조금 및 314730_188877)에 의해 지원되었다.
자료
| Name | Company | Catalog Number | Comments |
| AccuPrime Pfx SuperMix | Invitrogen | 12344-040 | |
| anti-m6A antibody _Clone 17-3-4-1 | Millipore | MABE1006 | |
| Chloroform | Merck | 67-66-3 | |
| ERCC | Invitrogen | 4456740 | |
| EZ RNA Methylation Kit | Zymo Research | EZR5001 | |
| Fragment analyzer RNA Kit - HS RNA Kit | Agilent | DNF-472-0500 | |
| Fragment analyzer RNA Kit - RNA Kit | Agilent | DNF-471-0500 | |
| High-Capacity cDNA Reverse Transcription Kit | Applied Biosystem | 4368814 | |
| Illumina TruSeq Stranded mRNA | Illumina | 20020594 | |
| Magnetic Beads A/G Blend | Merck | 16-663 | |
| N6-Methyladenosine, 5′-monophosphate sodium salt (m6A) | Sigma Aldrich | M2780-10MG | |
| Normal Mouse IgG | Merk | 12371 | |
| Oligo(dT)25 | Life Technologies | 61005, | |
| PCRapace | Stratec | 1020220300 | |
| Quick RNA Viral Kit | Zymo Research | 1034 | |
| RNA Clean & Concentrator | Zymo Research | R1015 | |
| RNA Fragmentation Reagent | Ambion | AM8740 | |
| RNase Inhibitor | Ambion | AM2684 | |
| Trizol | TRIzol Reagent | 15596026 |
참고문헌
- Machnicka, M. A., et al. MODOMICS: a database of RNA modification pathways--2013 update. Nucleic Acids Research. 41, Database issue 262-267 (2013).
- Zaccara, S., Ries, R. J., Jaffrey, S. R. Reading, writing and erasing mRNA methylation. Nature Reviews Molecular Cell Biology. 20 (10), 608-624 (2019).
- Davalos, V., Blanco, S., Esteller, M. SnapShot: Messenger RNA Modifications. Cell. 174 (2), 498(2018).
- Saletore, Y., et al. The birth of the Epitranscriptome: deciphering the function of RNA modifications. Genome Biology. 13 (10), 175(2012).
- Zhao, B. S., Roundtree, I. A., He, C. Post-transcriptional gene regulation by mRNA modifications. Nature Reviews Molecular Cell Biology. 18 (1), 31-42 (2017).
- Netzband, R., Pager, C. T. Epitranscriptomic marks: Emerging modulators of RNA virus gene expression. Wiley Interdisciplinary Reviews: RNA. 11 (3), 1576(2020).
- Pereira-Montecinos, C., Valiente-Echeverria, F., Soto-Rifo, R. Epitranscriptomic regulation of viral replication. Biochimica et Biophysica Acta. 1860 (4), 460-471 (2017).
- Lichinchi, G., et al. Dynamics of the human and viral m(6)A RNA methylomes during HIV-1 infection of T cells. Nature Microbiology. 1, 16011(2016).
- Courtney, D. G., et al. Epitranscriptomic Addition of m(5)C to HIV-1 Transcripts Regulates Viral Gene Expression. Cell Host & Microbe. 26 (2), 217-227 (2019).
- Kennedy, E. M., et al. Posttranscriptional m(6)A Editing of HIV-1 mRNAs Enhances Viral Gene Expression. Cell Host & Microbe. 19 (5), 675-685 (2016).
- Tirumuru, N., Wu, L. HIV-1 envelope proteins up-regulate N (6)-methyladenosine levels of cellular RNA independently of viral replication. Journal of Biological Chemistry. 294 (9), 3249-3260 (2019).
- Tirumuru, N., et al. N(6)-methyladenosine of HIV-1 RNA regulates viral infection and HIV-1 Gag protein expression. Elife. 5, (2016).
- Cristinelli, S., Angelino, P., Janowczyk, A., Delorenzi, M., Ciuffi, A. HIV Modifies the m6A and m5C Epitranscriptomic Landscape of the Host Cell. Frontiers in Virology. 1 (11), (2021).
- Khoddami, V., Cairns, B. R. Transcriptome-wide target profiling of RNA cytosine methyltransferases using the mechanism-based enrichment procedure Aza-IP. Nature Protocols. 9 (2), 337-361 (2014).
- Hussain, S., Aleksic, J., Blanco, S., Dietmann, S., Frye, M. Characterizing 5-methylcytosine in the mammalian epitranscriptome. Genome Biology. 14 (11), 215(2013).
- Dominissini, D., Moshitch-Moshkovitz, S., Salmon-Divon, M., Amariglio, N., Rechavi, G. Transcriptome-wide mapping of N6-methyladenosine by m6A-seq based on immunocapturing and massively parallel sequencing. Nature Protocols. 8 (1), 176-189 (2013).
- Dominissini, D., et al. Topology of the human and mouse m6A RNA methylomes revealed by m6A-seq. Nature. 485 (7397), 201-206 (2012).
- Shobbir Hussain, J. A., Blanco, S., Dietmann, S., Frye, M. Characterizing 5-methylcytosine in the mammalian epitranscriptome. Genome Biology. 14 (215), (2013).
- Amort, T., et al. Distinct 5-methylcytosine profiles in poly(A) RNA from mouse embryonic stem cells and brain. Genome Biology. 18 (1), 1(2017).
- Endrullat, C., Glökler, J., Franke, P., Frohme, M. Standardization and quality management in next-generation sequencing. Applied & Translational Genomics. 10, 2-9 (2016).
- Schaefer, M., Pollex, T., Hanna, K., Lyko, F. RNA cytosine methylation analysis by bisulfite sequencing. Nucleic Acids Research. 37 (2), 12(2009).
- Cristinelli, S., Angelino, P., Janowczyk, A., Delorenzi, M., Ciuffi, A. HIV Modifies the m6A and m5C Epitranscriptomic Landscape of the Host Cell. biorxiv. 1 (11), (2021).
- Squires, J. E., et al. Widespread occurrence of 5-methylcytosine in human coding and noncoding RNA. Nucleic Acids Research. 40 (11), 5023-5033 (2012).
- Aschenbrenner, J., et al. Engineering of a DNA Polymerase for Direct m(6) A Sequencing. Angewandte Chemie (International ed. in English). 57 (2), 417-421 (2018).
- Bioinformatics. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc (2021).
- Didion, J. P., Martin, M., Collins, F. S. Atropos: specific, sensitive, and speedy trimming of sequencing reads. PeerJ. 5, 3720(2017).
- Kim, D., Langmead, B., Salzberg, S. L. HISAT: a fast spliced aligner with low memory requirements. Nature Methods. 12 (4), 357-360 (2015).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Okonechnikov, K., Conesa, A., García-Alcalde, F. Qualimap 2: advanced multi-sample quality control for high-throughput sequencing data. Bioinformatics. 32 (2), Oxford, England. 292-294 (2016).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), Oxford, England. 3047-3048 (2016).
- Zhang, Y., et al. Model-based Analysis of ChIP-Seq (MACS). Genome Biology. 9 (9), 137(2008).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet Journal. 17 (1), (2011).
- Shen, W., Le, S., Li, Y., Hu, F. SeqKit: A Cross-Platform and Ultrafast Toolkit for FASTA/Q File Manipulation. PLOS ONE. 11 (10), 0163962(2016).
- Rieder, D., Amort, T., Kugler, E., Lusser, A., Trajanoski, Z. meRanTK: methylated RNA analysis ToolKit. Bioinformatics. 32 (5), 782-785 (2015).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유