Method Article
Viral Enfeksiyon Üzerine m6A ve m5C Epitranscriptomes'i Keşfetmek: HIV ile Bir Örnek
Bu Makalede
Özet
Viral enfeksiyonlarda RNA modifikasyonlarının rolü yeni yeni araştırılmaya başlanmıştır ve yeni viral konak etkileşim mekanizmalarını vurgulayabilir. Bu çalışmada, viral enfeksiyonlar bağlamında m6A ve m5C RNA modifikasyonlarını araştırmak için bir boru hattı sağlıyoruz.
Özet
RNA modifikasyonlarının biyolojik süreçlerdeki rolü, son birkaç yılda artan sayıda çalışmanın odak noktası olmuştur ve günümüzde epitranscriptomics olarak bilinmektedir. Diğerleri arasında, N6-metildenozin (m6A) ve 5-metilsitozin (m5C) RNA modifikasyonları mRNA molekülleri üzerinde tanımlanmıştır ve hücresel süreçlerin modülasyonunda rol alabilir. Epitranscriptomics, viral enfeksiyonlar da dahil olmak üzere herhangi bir kimyasal veya biyolojik maddeye maruz kalarak da değiştirilebileceği veya modüle edilebildiği için transkriptomik analizlere ek olarak dikkate alınması gereken yeni bir düzenleme katmanıdır.
Burada, insan immün yetmezlik virüsü (HIV) ile enfekte olmuş veya enfekte olmayan hücrelerde, m6A ve m5C izlerinin eklem hücresel ve viral epitranscriptomik manzarasının aynı anda analizini sağlayan bir iş akışı sunuyoruz. HIV ile enfekte ve enfekte olmayan hücrelerden mRNA izolasyonu ve parçalanması üzerine, iki farklı prosedür kullandık: M6A işareti içeren RNA parçaları için zenginleştirmek için RNA immün önkoşullama tabanlı bir teknik olan MeRIP-Seq ve iki çekirdekli dönüşüm tabanlı bir teknik olan BS-Seq, m5C işaretini tek bir nükleotid çözünürlüğünde tanımlamak için. Metilasyona özgü yakalama üzerine, RNA kitaplıkları yüksek verimli sıralama için hazırlanır. Ayrıca, bazal ifade profillerinden bağımsız olarak farklı metillenmiş (DM) transkriptleri tanımlamak için özel bir biyoinformatik boru hattı geliştirdik.
Genel olarak, metodoloji aynı anda birden fazla epitranscriptomic işaretin araştırılmasına izin verir ve viral enfeksiyon veya başka bir hücre pertürbasyonu üzerine bir DM transkript atlası sağlar. Bu yaklaşım, viral replikasyonu teşvik eden veya kısıtlayan hücresel faktörler gibi yeni oyuncuları ve hücre tepkisinin yeni mekanizmalarını tanımlamak için yeni fırsatlar sunar.
Giriş
RNA moleküllerinin değiştirilebildiği uzun zamandır bilinmektedir ve bugüne kadar 150'den fazla transkripsiyon sonrası modifikasyon tanımlanmıştır1. RNA moleküllerinin pirimidin ve pürin halkalarının hemen hemen her pozisyonuna başta metil grupları olmak üzere kimyasal grupların eklenmesinden oluşurlar2. Bu tür transkripsiyon sonrası değişikliklerin transfer RNA (tRNA) ve ribozomal RNA'da (rRNA) oldukça zenginleştirilmiş olduğu ve son zamanlarda mRNA molekülleri üzerinde de tanımlandığı gösterilmiştir.
Yeni Nesil Dizileme (NGS) gibi yeni teknolojilerin yükselişi ve kesin kimyasal modifikasyonları tanıyan spesifik antikorların üretilmesi, ilk kez transcriptome çapında belirli kimyasal modifikasyonların yerinin ve sıklığının araştırılmasına izin verildi. Bu gelişmeler, RNA modifikasyonlarının daha iyi anlaşılmasına ve mRNA molekülleri üzerinde çeşitli değişikliklerin haritalanmasına yol açmıştır3,4.
Epigenetik, transkriptomi regülasyonunda DNA ve histon modifikasyonlarının rolünü araştırırken, benzer bir şekilde epitranscriptomics RNA modifikasyonlarına ve rollerine odaklanmaktadır. Epitranscriptomic modifikasyonların araştırılması, çeşitli hücresel süreçleri (örneğin, RNA birleştirme, dışa aktarma, kararlılık ve çeviri) ayarlayabilecek yeni düzenleme mekanizmalarını vurgulamak için yeni fırsatlar sağlar 5. Bu nedenle, son çalışmaların hem hücresel hem de viral RNA'larda viral enfeksiyon üzerine birçok epitranscriptomik modifikasyonu ortaya çıkarması büyük bir sürpriz değildi. Şimdiye kadar araştırılan virüsler hem DNA hem de RNA virüslerini içerir; bunlar arasında HIV öncü bir örnek olarak kabul edilebilir. Tamamen, viral enfeksiyonlar bağlamında RNA metilasyonunun keşfi, viral ifade veya replikasyonun henüz atfedilmemiş mekanizmalarının araştırılmasına izin verebilir, böylece onları kontrol etmek için yeni araçlar ve hedefler sağlayabilir7.
HIV epitranscriptomics alanında, viral transkriptlerin modifikasyonları yaygın olarak araştırılmış ve bu değişikliğin varlığının viral replikasyon için yararlı olduğunu göstermiştir8,9,10,11,12,13. Bugüne kadar transkriptom düzeyinde epitranscriptomic izleri tespit etmek için çeşitli teknikler kullanılabilir. m6A tanımlama için en çok kullanılan teknikler MeRIP-Seq ve miCLIP gibi immün çökeltme tekniklerine dayanır. MeRIP-Seq metillenmiş kalıntılar içeren parçaları yakalamak için RNA parçalanmasına güvenirken, miCLIP RNA-antikor UV çapraz bağlama üzerine α-m6A antikor spesifik imza mutasyonlarının üretimine dayanır, böylece daha kesin bir haritalama sağlar.
m5C modifikasyonunun tespiti, m6A algılamasına (m5C RIP) benzer antikor bazlı teknolojilerle veya bisülfit dönüşümüyle veya AZA-IP veya miCLIP ile elde edilebilir. Hem Aza-IP hem de m5C miCLIP, RNA metilasyonundan geçerken RNA'yı hedeflemek için yem olarak belirli bir metiltransfenaz kullanır. Aza-IP'de, hedef hücreler 5-azacytidin'e maruz kalır ve bu da sitidin analog 5-azasitidin bölgelerinin nascent RNA'ya rastgele girmesiyle sonuçlanır. MiCLIP'te, NSun2 metiltransfez C271A mutasyonu14,15'i barındıracak şekilde genetik olarak değiştirilmiştir.
Bu çalışmada, HIV'i bir model olarak kullanarak, enfekte hücrelerde m6A ve m5C modifikasyonlarının ikili karakterizasyonuna odaklanıyoruz. Metodolojik optimizasyon üzerine, metillenmiş RNA immünödeksiyasyon (MeRIP) ve RNA bisülfit dönüşümünü (BS) birleştiren, m6A ve m5C epitranscriptomic izlerinin hem hücresel hem de viral bağlamlarda transkriptom çapında eşzamanlı olarak araştırılmasını sağlayan bir iş akışı geliştirdik. Bu iş akışı hücresel RNA özlerine ve viral parçacıklardan izole edilmiş RNA'ya uygulanabilir.
Metillenmiş RNA İmmün Önkseçasyon (MeRIP)16 yaklaşımı, m6A'nın transkriptome-wide düzeyinde araştırılmasına izin verir ve bugüne kadar bir dizi m6A spesifik antikor ticari olarak mevcuttur17. Bu yöntem, m6A içeren RNA parçalarının m6A'ya özgü bir antikor kullanılarak seçici olarak yakalanmasından oluşur. Bu tekniğin iki önemli dezavantajı(i) RNA parçalarının boyutuna oldukça bağlı olan ve böylece metillenmiş kalıntıyı içeren yaklaşık bir konum ve bölge sağlayan sınırlı çözünürlük ve (ii) analizi gerçekleştirmek için gereken büyük miktarda malzemedir. Aşağıdaki optimize edilmiş protokolde, parça boyutunu yaklaşık 150 nt'ye standartlaştırdık ve başlangıç malzemesi miktarını şu anda tavsiye edilen başlangıç malzemesi miktarı olan 10 μg poli-A seçilen RNA'dan sadece 1 μg poli-A seçilen RNA'ya düşürdük. Ayrıca fenol bazlı teknikler veya proteinaz K kullanarak daha geleneksel ve daha az spesifik elution yöntemleri yerine m6A peptitli bir rekabet yaklaşımı ile bir elution kullanarak spesifik antikorlara bağlı m6A RNA parçalarının geri kazanım verimliliğini en üst düzeye çıkardık. Bununla birlikte, bu RIP tabanlı tahlilin ana sınırlaması, kesin olarak değiştirilmiş A nükleotidinin tanımlanmasına izin vermeyen yetersiz çözünürlük olmaya devam etmektedir.
M5C işaretinin analizi şu anda iki farklı yaklaşım kullanılarak yapılabilir: m5C'ye özgü antikorlara sahip RIP tabanlı bir yöntem ve RNA bisülfit dönüşümü. RIP metillenmiş kalıntının tanımlanmasında sadece sınırlı çözünürlük sunduğundan, tek nükleotid çözünürlüğü sunabilen bisülfit dönüşümünü kullandık. Bisülfit (BS) RNA maruziyeti sitozin deaminasyonuna yol açar, böylece sitozin kalıntısını urasiye dönüştürür. Bu nedenle, RNA bisülfit dönüşüm reaksiyonu sırasında, metil olmayan her sitozin deaminasyona edilir ve urasil haline dönüştürülürken, sitozinin 5 pozisyonunda bir metil grubunun varlığı koruyucu bir etkiye sahiptir, BS kaynaklı deaminasyonu önler ve sitozin kalıntılarını korur. BS tabanlı yaklaşım, m5C modifiye nükleotitin tek baz çözünürlükte algılanmasına ve her transkriptin metilasyon sıklığının değerlendirilmesine olanak tanır ve m5C modifikasyon dinamikleri hakkında içgörü sağlar18. Bununla birlikte, bu tekniğin ana sınırlaması metillenmiş kalıntıların yanlış pozitif oranına dayanır. Gerçekten de, BS dönüşümü erişilebilir C kalıntılarına sahip tek iplikli RNA üzerinde etkilidir. Bununla birlikte, sıkı bir RNA ikincil yapısının varlığı N5C konumunu maskeleyebilir ve BS dönüşümünü engelleyebilir, bu da U kalıntılarına dönüştürülmeyen metillenmiş olmayan C kalıntılarına ve dolayısıyla yanlış pozitiflere neden olabilir. Bu sorunu atlatmak ve yanlış pozitif oranı en aza indirmek için 3 tur denatürasyon ve bisülfit dönüşüm döngüsü uyguladık19. Ayrıca, bisülfit dönüşüm verimliliğinin tahmin edilmesini sağlamak için numunelerde 2 kontrol sunduk: ERCC sıralama kontrollerinde (metillenmemiş standartlaştırılmış ve ticari olarak kullanılabilen diziler)20 ve bir yandan bisülfit dönüşüm oranını değerlendirmek için poli-A tükenmiş RNA'lar, ve RT-PCR tarafından bilinen ve iyi korunmuş bir metillenmiş site olan C4447'nin varlığını doğrulamak için, diğer yandan 28S ribozomal RNA üzerinde21.
Viroloji alanında, bu iki epitranscriptomik araştırma yönteminin yeni nesil dizileme ve doğru biyoinformatik analiz ile birlenmesi, m6A ve m5C dinamiklerinin derinlemesine incelenmesine izin verir (yani, viral enfeksiyon üzerine ortaya çıkabilecek ve klinik kullanım için terapötik olarak ilgili bir dizi yeni hedefi ortaya çıkarabilecek RNA modifikasyon zamansal değişiklikleri).
Protokol
1. Hücre Hazırlama
NOT: Hücre türüne ve RNA içeriğine bağlı olarak, başlangıç hücre sayısı değişebilir.
- Toplam RNA'nın 200-500 μg'si veya 5-7 μg poli-A-seçilmiş RNA'sını elde etmek için yeterli hücreye sahip olmak. Örneğin, 50 x 106 SupT1 hücresi fenol bazlı reaktiflerle ekstraksiyon sırasında yaklaşık 500 μg toplam RNA vermelidir ve bu nedenle test edilen her bir durum için gereklidir.
- Deneysel tasarıma göre gerekli hücre sayısını ve böylece test edilen durum sayısına (enfeksiyon, zaman noktaları, tedavi) göre hazırlayın. Deney, enfekte olmayan hücreleri ve HIV ile enfekte olmuş hücreleri enfeksiyon sonrası 24 saat içinde elde etmeyi amaçlıyorsa, yarısı enfekte olmayan durum için, yarısı enfekte durum için olmak üzere toplam 100 x 106 hücreye ihtiyaç vardır.
2. RNA Ekstraksiyonu
- Hücrelerden: Fenol-Kloroform ile RNA Ekstraksiyonu
- Her koşul için, hücreleri (örneğin, 50 x 106) santrifüjleme ile toplayın ve üstnatantı atın.
- Her 50 x 106 hücre peleti için 5 mL fenol bazlı reaktif ekleyin ve birkaç kez yukarı ve aşağı pipetleme yaparak karıştırın.
- Tam lizise izin vermek için oda sıcaklığında 5 dakika kuluçkaya yatır. Lislenmiş hücreler -80 °C'de saklanabilir veya doğrudan işlenebilir.
NOT: Gerekirse, hücreler 1,5 mL tüplerde tüp başına 10 x 106 hücreli aliquotlara bölünebilir ve daha uygun depolama için 1 mL fenol bazlı reaktifte lysed edilebilir. - 1 mL kloroform ekleyin ve ters çevrilerek karıştırın.
- Oda sıcaklığında 3 dakika kuluçkaya yatır.
- 2.000 x g ve 4 °C'de 15 dakika santrifüj.
- Sulu fazı (üst faz) pipetle dışarı atın ve yeni bir tüpe aktarın. Tüpü 45° 'de angling ederek ve çözeltiyi dikkatlice pipetle çıkararak sulu fazı aktarmayı bitirin.
NOT: Sulu faz miktarı örnekler arasında değişebilir, ancak numuneye eklenen kloroform miktarına yakın olmalıdır (yani, 1 mL). Herhangi bir ara faz veya organik tabaka aktarmayın! Faz kilidi veya faz yapıcı tüplerin kullanımı bu işlemi kolaylaştırabilir. - Sulu faza %100 moleküler sınıf izopropanol 0,5 mL ekleyin.
- RNA yağışına izin vermek için -80 °C'de 1 saat kuluçkaya yaslanın.
- Çökemiş RNA'yı peletmek için 12.000 x g ve 4 °C'de 10 dakika santrifüj.
- Süpernatant atın ve RNA peletini% 75 moleküler biyoloji sınıfı etanol 1 mL'de yeniden dirildi. Kısaca Girdap.
- 7.500 x g ve 4 °C'de 5 dakika santrifüj ve süpernatantı atın.
- Peletin havayla 15 dakika kurutun.
- Peleti 20 μL RNase içermeyen suda yeniden boşaltın ve yeni bir tüpe aktarın.
- RNA geri kazanımını en üst düzeye çıkarmak için boş tüpü ek 20 μL suyla yıkayın ve ilk 20 μL hacme sahip havuzalayın.
- Toplam RNA'yı bir spektrofotometre ile ölçün ve RNA kalitesini bir parça analizörü ile değerlendirin.
- Viral Parçacıklardan: Sütun Bazlı Viral RNA Ekstraksiyon Kiti ile RNA Ekstraksiyonu
NOT: Fenol bazlı reaktifli viral parçacıklardan RNA ekstraksiyonu, düşük kaliteli viral RNA ve daha düşük kaliteli kütüphanelerde sonuçlanır. Sütun tabanlı bir RNA ayıklaması bu nedenle tercih edilmelidir. RNA elüasyonu ve geri kazanımı için taşıyıcı RNA kullanan RNA ekstraksiyon kitleri bu prosedür için uygun değildir ve kaçınılmalıdır. HIV RNA poli-Adenilasyonlu olduğundan, Daha fazla mRNA izolasyonu olmadan doğrudan RNA ekstraksiyonu MeRIP-Seq ve BS-Seq boru hatlarına girmek için yeterlidir. Normalde evrensel olarak enfekte olmuş hücrelerden 1-2 mL viral süpernatant, tüm iş akışını gerçekleştirmek için yeterli RNA sağlamalıdır.- 30 mL liziz tampona 150 μL beta-mercaptoethanol ekleyerek tamponu hazırlayın. %100 etanol 96 mL ekleyerek Viral Yıkama Tamponu yeniden inşa edin.
- Hücresel RNA kontaminasyonini en aza indirmek için virüs içeren süpernatantları ve santrifüjleri pelet hücresi kalıntılarına toplayın.
- 1 mL viral süpernatant 15 mL tüpe aktarın.
- 1 mL viral örneğe 3 mL Viral RNA Tampon ekleyin ve girdapla karıştırın.
- Bir Koleksiyon Tüpüne yerleştirilmiş bir sütuna 700 μL numune aktarın.
- Oda sıcaklığında 13.000 x g'da 2 dakika santrifüj.
- Akışa göre atın.
- Tüm örnek işlenene ve böylece tüm RNA silika tabanlı matris sütununa yakalanana kadar önceki 3 adımı tekrarlayın.
- Sütuna 500 μL Viral Yıkama Tamponu ekleyin.
- Oda sıcaklığında 10.000 x g'da 1 dakika santrifüj. Akışa göre atın.
- Sütuna 200 μL Viral Yıkama Tamponu ekleyin.
- Oda sıcaklığında 10.000 x g'da 1 dakika santrifüj. Akışa göre atın.
- Sütunu boş bir toplama tüpüne yerleştirin.
- Kalan yıkama tampon kirleticilerini daha fazla atmak için oda sıcaklığında 10.000 x g'da 1 dakika santrifüj.
- Sütunu dikkatlice 1,5 mL'lik bir tüpe aktarın.
- Sütun matrisinin ortasına doğrudan 20 μL DNase/RNase içermeyen su ekleyin ve oda sıcaklığında 30 s için 10.000 x g'da santrifüj ekleyin.
- Sütun matrisinin ortasına doğrudan 10 μL DNase/RNase içermeyen su ekleyin ve 30 sn boyunca tekrar santrifüjleyin.
- Toplam RNA'yı bir spektrofotometre ile ölçün ve RNA kalitesini bir parça analizörü ile değerlendirin.
NOT: Alınan RNA'nın kalitesi yüksekse, RNA bütünlüğü/kalite numarası 9'> herhangi bir yöntemle RNA ekstraksiyonu yapılabilir. Toplam RNA, bir sonraki işleme kadar -80 °C'de saklanabilir.
3. oligo(dT)25 ile poli-A Seçimi ile mRNA İzolasyonu
NOT: Hücresel özlerde yüksek metillenmiş ribozomal RNA bulunması nedeniyle, poli-A RNA'sının rRNA tükenmesi veya tercihen poli-A pozitif seçimi ile izole etmesi şiddetle tavsiye edilir. Bu adım isteğe bağlıdır ve daha yüksek çözünürlükte sıralama sonuçları elde etmek için yalnızca hücresel RNA örnekleri için gerçekleştirilmelidir. Poli-adenilasyonlu olmayan viral RNA'ların metilasyonunun analiz edilirse, poli-A seçimi yerine rRNA tükenmesini tercih edin veya sonunda analizi toplam RNA üzerinde gerçekleştirin.
-
Poli-A Yakalama için Boncuk Hazırlama
- Oligo(dT)25 manyetik boncuk şişesini >30 sn girdapla yeniden depolayarak yeniden depolar.
- 200 μL manyetik boncukları 1,5 mL'lik bir tüpe aktarın. İşlenecek RNA numunelerinin toplam miktarına göre manyetik boncuklu tüp sayısını hazırlayın.
NOT: 200 μL Dynabead stok çözeltisine sahip bir tüp 1 mg boncuklara karşılık gelir ve toplam RNA'nın 75 μg'lik bir örneğini barındırabilir. - Tüpleri 1 dakika boyunca bir mıknatısa yerleştirin ve üstnatant atın. Tüpleri mıknatıstan çıkarın.
- 1 mL Bağlama Tamponu (20 mM Tris-HCl, pH 7.5, 1.0 M LiCl, 2 mM EDTA) ekleyin ve vorteks ile yeniden depolanın. Tüpleri mıknatısın üzerine 1 dakika yerleştirin ve üstnatant atın. Tüpleri mıknatıstan çıkarın. Yinelemek.
- Yıkanmış manyetik boncukları 100 μL Bağlama Tamponunda yeniden diriltin.
-
Toplam RNA hazırlığı
- Toplam RNA'yı 75 μg/100 μL'ye karşılık gelen RNase içermeyen su ile 0,75 μg/μL'lik son konsantrasyonda seyreltin.
NOT: RNA daha düşük konsantrasyondaysa, birimleri değiştirmeden aşağıda açıklandığı gibi devam edin. - Tüp başına 100 μL RNA örneği dağıtarak birden fazla tüpte toplam RNA'yı aliquot.
- Her RNA örneğine 100 μL Bağlama arabelleği ekleyin.
- İkincil yapıları bozmak için toplam RNA'yı 2 dakika boyunca 65 °C'ye ısıtın.
- Bir sonraki adıma geçme hazır olana kadar hemen buza yerleştirin.
NOT: Kuluçka süresi, işlenecek numune sayısına göre değişebilir, ancak herhangi bir RNA bozulmasını önlemek için 1 saati geçmemelidir.
- Toplam RNA'yı 75 μg/100 μL'ye karşılık gelen RNase içermeyen su ile 0,75 μg/μL'lik son konsantrasyonda seyreltin.
-
Çoklu A Seçimi
- Her RNA tüpüne (adım 3.2'den itibaren), 100 μL yıkanmış manyetik boncuk ekleyin (adım 3.1'den itibaren).
- Yukarı ve aşağı pipetleme ile iyice karıştırın ve 15 dakika boyunca oda sıcaklığında dönen bir tekerlek üzerinde bağlamaya izin verin.
- Tüm tüpleri açın, 1 dakika boyunca mıknatısın üzerine yerleştirin ve tüm süpernatantları dikkatlice çıkarın.
- Yeni bir tüpte süpernatant kurtarın ve poli-A son iyileşmeyi iyileştirmek için ikinci tur RNA yakalama (adım 3.3.14) için bir kenara bırakın.
- Tüpü mıknatıstan çıkarın ve 200 μL Yıkama Tamponu ekleyin (10 mM Tris-HCl, pH 7.5, 0.15 M LiCl, 1 mM EDTA). 4 ila 5 kez dikkatlice pipetleyarak karıştırın.
- Tüpü mıknatısın üzerine 1 dakika yerleştirin ve üstnatant atın.
- Yıkama adımını bir kez tekrarlayın (3.3.5 ve 3.3.6 adımlarını tekrarlayın).
- Boncuklardan poli-A RNA'sı elute etmek için 20 μL buz gibi 10 mM Tris-HCl ekleyin.
- 80 °C'de 2 dakika kuluçkaya yatır.
- Tüpü mıknatısın üzerine yerleştirin ve poli-A RNA'yı içeren süpernatant'ı yeni bir RNase içermeyen tüpe hızlı bir şekilde aktarın. Tüpü buza yerleştirin.
- Verimi artırmak için elution adımını (adım 3.3.8 ila 3.3.10) tekrarlayın.
- Aynı boncukları 200 μL yıkama tamponu ile bir kez yıkayın. 4 ila 5 kez dikkatlice pipetleyarak karıştırın.
- Mıknatısa 1 dakika yerleştirin ve yıkama tamponu atın.
- Akışa adım 3.3.4'ten boncuklara ekleyin ve bağlamadan elution'a (adım 3.3.2 ile 3.3.10) yordamı tekrarlayın. RNA eluatlarını şimdilik ayrı tüplerde tutun.
NOT: İsteğe bağlı olarak, kontrol olarak kullanılabileceği için 3.3.4 adımına eşdeğer olan süpernatant'ı yine yeni bir tüpte tutun. İşlemin sonunda, RNA'yı etanol çökeltme veya sütun tabanlı bir seçim yöntemi (örneğin, RNA temiz ve yoğunlaştırıcı) ile arındırın ve konsantre edin. Bu örnek poli-A-tükenmiş bir RNA örneğine karşılık gelir ve bisülfit dönüşümü için bir kontrol olarak kullanılabilir (adım 8.2.2). - Bir spektrofotometre ile eluted RNA'yı ölçün ve RNA kalitesini bir parça analizörü ile daha fazla değerlendirmek için 2 μL aliquot tutun.
NOT: Poly-A RNA ihtiyaç duyulana kadar -80 °C'de saklanabilir.
4. RNA iş akışı
- Hücresel poli-A RNA (mRNA) ve viral RNA örneklerini ilgili epitranscriptomic analiz boru hattına adanmış 2 aliquot'a bölün:
(i) MeRIP-Seq ve giriş kontrolleri için 5 μg hücresel mRNA veya 1 μg viral RNA (5 ila 7 ve adım 9'a gidin).
(ii) BS-Seq için 1 μg hücresel mRNA veya 500 ng viral RNA (8 ve 9. adımlara gidin).
5. RNA Parçalanması
NOT: RNA parçalanması RNA parçalanma reaktifi ile gerçekleştirilir ve MeRIP-Seq ve kontrol RNA örnekleri için tasarlanmıştır. Bu, 100-200 nt arasında değişen parçalar elde etmek için dikkatli iyileştirme gerektiren çok önemli bir adımdır.
- Toplam mRNA hacmini 18 μL mRNA/tüp ile 0,2 mL PCR tüplere bölün.
NOT: Hızlı çalışın. Tekrarlanabilir sonuçlar elde etmek için aynı anda 8'den fazla örnekle çalışmayın. Birimin ölçeklendirmesi, tekrarlanabilir ve düzgün bir parçalanmayı garanti etmez. - 70 °C'de bir termosikleyici ısıtın.
- Her PCR tüpünün kenarına 2 μL parçalanma reaktifi ekleyin.
- Tüpü kapatın ve aşağı doğru döndürün (böylece reaktif 8 tüp için aynı anda RNA ile temas eder).
- Önceden ısıtılmış termositte numuneleri 70 °C'de 15 dk kuluçkaya yatırın.
- Kuluçka biter bitmez, her tüpe hızlı bir şekilde 2 μL Stop çözeltisi ekleyin.
- Aşağı dön ve bir sonraki adıma geçme hazır olana kadar buzun üzerine otur.
NOT: Kuluçka süresi, işlenecek numune sayısına göre değişebilir, ancak herhangi bir RNA bozulmasını önlemek için 1 saati geçmemelidir. - Tüm örnekler için prosedürü tekrarlayın (8'den fazla aliquot varsa).
- Tüpleri bir araya toplayın ve tamponlardan kurtulmak ve sudaki temiz parçalanmış RNA'yı kurtarmak için bir RNA temiz ve yoğunlaştırıcı Kit (adım 6) veya herhangi bir özelleştirilmiş sütun tabanlı kit ile RNA saflaştırmaya geçin.
6. RNA Saflaştırma
NOT: Bu adım etanol çökeltme veya sütun bazlı RNA saflaştırma ve konsantrasyon yöntemi (örneğin, RNA Clean ve Concentrateor) ile gerçekleştirilebilir.
- Arıtılmış RNA'yı toplam 50-75 μL DNase/RNase içermeyen su hacminde boşaltın veya yeniden hissedin.
NOT: Sütun tabanlı bir yöntem kullanılıyorsa, maksimum kurtarma sağlamak için iki tur elution önerilir. - Saflaştırılmış parçalanmış mRNA'yı spektrofotometre ile ölçün ve RNA kalitesini bir parça analizörü ile değerlendirin.
- Kütüphane hazırlama ve sıralama için giriş kontrolü olarak 100 ng parçalı mRNA tutun (adım 9'a gidin). Kalan parçalanmış mRNA (minimum 2,5 μg) MeRIP için kullanılabilir (adım 7,2'ye gidin).
7. MeRIP
NOT: Her immün özürlü solunum (IP) için, belirli bir anti-m6A antikoru (test durumu) veya bir anti-IgG antikoru (negatif kontrol) kullanılarak en az 2,5 μg parçalı mRNA gereklidir.
- İmmün Önseçme için Manyetik Boncuk Hazırlama
- Her numune için, 800 μL mRNA IP tamponu 5x (50 mM Tris-HCl pH 7.4) seyrelterek yeni bir konik tüpte 4 mL 1x IP tampon hazırlayın, 750 mM NaCl, %0,5 Igepal CA-630 ve nükleaz içermeyen su) 3,2 mL çekirdeksiz su ile.
NOT: En az 2 reaksiyon gereklidir (bir test ve bir IgG kontrolü). - Tüpü buza yerleştirin.
- İstenilen IP reaksiyonlarının sayısı için uygun sayıda 1,5 mL mikrosantrifüj tüpü etiketle:
anti-m6A antikor için n tüpler (test).
Normal Fare IgG için n tüpler (negatif kontrol). - Manyetik boncukları (örneğin, Magna ChIP Protein A/G) ters çevirerek ve girdaplayarak yeniden depolar. Boncuk yığınları görünmemelidir.
- Planlanan her reaksiyon için, 25 μL manyetik boncukları bir mikrosantrifüj tüpüne aktarın.
- Kullanılan boncukların orijinal hacmine göre on kat daha fazla 1x IP arabelleği (adım 7.1.1'den itibaren) ekleyin (yani, 25 μL manyetik boncuk başına 250 μL 1x IP arabellek).
- Tam resüspensyon için birkaç kez hafifçe yukarı ve aşağı pipetleme yaparak boncukları karıştırın.
- Tüpü manyetik ayırıcıya 1 dakika yerleştirin.
- Herhangi bir manyetik boncuk aspire olmadığından emin olarak süpernatantı çıkarın ve atın. Tüpü mıknatıstan çıkarın.
- Yıkama adımını tekrarlayın (adım 7.1.6 ila 7.1.9).
- Boncukları, orijinal manyetik boncuk hacmi başına 100 μL 1x IP Tamponda yeniden diriltin.
- 25 μL orijinal manyetik boncuk hacmi başına 5 μL antikor (1 μg/μL) ekleyin.
n tüpler (test) anti-m6A antikorlu (klon 17-3-4-1) [1 μg/μL].
Normal Fare IgG (1 μg/μL) ile n tüpler (negatif kontrol). - Manyetik boncuklarla antikorların konjugasyonuna izin vermek için oda sıcaklığında 30 dakika boyunca dönen tekerlek üzerinde kuluçkaya yatırın.
- Tüpü manyetik ayırıcıya 1 dakika yerleştirin. Üstnatant atın. Tüpü mıknatıstan çıkarın ve antikor-boncuk karışımını 100 μL 1x IP Tamponda yeniden atın.
- Her numune için, 800 μL mRNA IP tamponu 5x (50 mM Tris-HCl pH 7.4) seyrelterek yeni bir konik tüpte 4 mL 1x IP tampon hazırlayın, 750 mM NaCl, %0,5 Igepal CA-630 ve nükleaz içermeyen su) 3,2 mL çekirdeksiz su ile.
- RNA İmmün Önseçme (RIP)
- Her 2,5 μg mRNA numunesi için aşağıdaki gibi 500 μL RIP reaksiyon karışımı hazırlayın: 100 μL Parçalı RNA'da 2,5 μg (adım 6,12'den itibaren); 295 μL çekirdeksiz su; 5 μL 40 U/μL RNaz inhibitörü; ve 100 μL 5x IP tampon.
- Her antikor boncuk karışımına 500 μL RIP reaksiyon karışımı ekleyin (adım 7.1.14'ten ~100 μL). Boncukları tamamen yeniden çıkarmak için birkaç kez hafifçe pipetleme ile karıştırın. Buzun üzerine koy.
- Tüm RIP tüplerini dönen bir tekerlek üzerinde 4 °C'de 2 saat kuluçkaya yatırın.
- Kapak ve tüp yanlarından sıvı damlacıkları döndürmek için MeRIP reaksiyonlarını kısaca santrifüj edin. Tüpleri 1 dakika boyunca manyetik bir ayırıcıya yerleştirin.
- Süpernatantı yeni bir santrifüj tüpüne aktarın, manyetik boncukları rahatsız etmemeye dikkat edin.
NOT: Akış, RIP verimliliğini doğrulamak için kontrol olarak tutulabilir (adım 7.3.9'a gidin). - Tüpleri mıknatıstan çıkarın. Boncukları 500 μL soğuk 1x IP tampon ekleyerek yıkayın. Boncukları tamamen yeniden çıkarmak için birkaç kez hafifçe pipetleyarak boncukları karıştırın.
- Tüpleri 1 dakika boyunca manyetik bir ayırıcıya yerleştirin ve süpernatant atın.
- Toplam 3 yıkama için yıkama prosedürünü (adım 7.2.6-7.2.7) iki kez tekrarlayın.
- Tüpleri buza yerleştirin ve hemen yağ vermeye devam edin.
- İfade
- 10 mg N6-Metildenozin, 5′-monofosfat sodyum tuzu (m6A) 1,3 mL çekirdeksiz suda eriterek 20 mM m6A çözeltisi hazırlayın. 150 μL aliquot hazırlayın ve -20 °C'de saklayın.
- Her numune için (test ve kontroller): Aşağıdaki bileşenleri karıştırarak 225 μL elüasyon tamponu hazırlayın: 45 μL 5x IP Tampon, 75 μL 20 mM m6A, 3,5 μL 40U/μL RNase Inhibitörü ve 101,5 μL çekirdeksiz su.
- Boncuklara (adım 7.2.9'dan itibaren) 100 μL elution tamponu ekleyin ( adım 7.3.2'den). Boncukları tamamen yeniden sulandırmak için birkaç kez hafifçe pipetleme yaparak karıştırın.
- Tüm tüpleri 4 °C'de bir rocker üzerinde sürekli sallanarak 1 saat boyunca kuluçkaya yatırın.
- Kapak ve tüp yanlarından sıvı damlacıkları aşağı döndürmek için RIP reaksiyonlarını kısa bir süre santrifüj edin. Tüpleri 1 dakika boyunca manyetik bir ayırıcıya yerleştirin.
- Eluted RNA parçaları içeren süpernatantı yeni bir 1,5 mL mikrosantrifüj tüpüne aktarın. Arka plan gürültüsünü artıracağından boncukları aspire etmemeye dikkat edin.
- Tekrar 100 μL elüsyon tamponu ekleyerek, 4 °C'de 1 saat inkübe ederek ve manyetik ayırmadan sonra eluat toplayarak elution adımlarını (7.3.3 ila 7.3.6) tekrarlayın.
- Aynı numuneden tüm eluatları birleştirin (toplam elution hacmi 200 μL olmalıdır).
- Elde edilen RNA'yı ve akış yüzeyini (isteğe bağlı olarak, adım 7.2.5'ten) etanol çökeltme veya sütun tabanlı bir seçim yöntemi (örneğin, RNA Clean and Concentrateor) ile arındırın.
- Yüksek hassasiyet algılama kiti kullanarak RNA miktarını ve akış kalitesini ve eluted numuneleri bir parça analizörü ile değerlendirin. RNA'nın kalitesi tatmin ediciyse, kitaplık hazırlama ve yüksek verimli sıralamaya geçin (adım 9).
NOT: MeRIP'te alınan RNA miktarı çok düşüktür ve nicelik sağlamak için zorunlu olarak yüksek hassasiyet algılama kitleri gerektirir. Biyoanalyzer yoksa, kütüphane hazırlığına körü körüne devam etmek mümkündür.
8. RNA Bisulfite Dönüşümü
- Kontrol ve Reaktif Hazırlama
- ERCC mix spike-in kontrolü: 500 ng mRNA'ya 0,5 μL sulandırılmamış ERCC karışımı eklenmesini öneren üreticinin talimatlarını takiben ERCC karışımını ekleyin. Bu kontrol, bisulfite dönüşümünün verimliliğini değerlendirmeye yardımcı olabilir.
- Spike Poly-A-tükenmiş RNA (adım 3.3.14'ten itibaren) 1/1000 oranında (yani, 500 ng mRNA için 500 pg poli-A-tükenmiş RNA). Bu örnek ribozomal RNA ile zenginleştirilmiştir ve bu nedenle bisülfit dönüşümü için pozitif bir kontrol olan 28S rRNA içermelidir.
NOT: Toplam RNA, poli-A-tükenmiş RNA yerine pozitif kontrol olarak da kullanılabilir. - RNA metilasyon kiti (örneğin, Zymo EZ) ile bisülfit dönüştürme gerçekleştirin.
- RNA Yıkama Tamponu: Kullanmadan önce 12 mL RNA Yıkama Tampon konsantresine 48 mL% 100 etanol (veya % 95 etanol 52 mL) ekleyin.
- Bisulfite Dönüştürme
NOT: Bisulfite dönüştürme, aşağıda belirtildiği gibi üreticinin prosedürünü takiben ticari olarak kullanılabilen bir RNA bisulfite dönüştürme kiti ile gerçekleştirilmiştir.- 0,2 mL PCR tüplerinde 1000 ng mRNA (veya 300 ila 1000 ng arasında) ekleyin. Çivili kontroller ekleyin: 1 μL ERCC karışımı (adım 8.1.1) ve 1000 pg poli-A tükenmiş RNA (adım 8.1.2). DNase/RNase içermeyen su ile 20 μL'ye kadar komple hacim.
- Her 20 μL RNA örneğine 130 μL RNA Dönüştürme Reaktifi ekleyin.
- Numuneyi yukarı ve aşağı pipetleme ile karıştırın.
- Tüpün kapağında veya yanlarında damlacık olmadığından emin olmak için kısa bir süre aşağı çevirin.
- PCR tüplerini bir termal çevrime yerleştirin ve aşağıdaki adımları uygulayın: 5 dakika boyunca 70 ° C'de denatürasyon; 45 dakika boyunca 54 °C'de dönüştürme; toplam 3 döngü boyunca denatürasyon ve dönüştürme adımlarını tekrarlayın; ve ardından süresiz olarak 4 °C'de tutun.
NOT: Üç denatürasyon ve bisulfite dönüştürme döngüsü, numunenin tam bisülfit dönüşümünü sağlar. Numuneler -80 °C'de saklanabilir veya doğrudan işlenebilir. - Sütun içi desulphonation ile devam edin. Boş bir toplama tüpüne bir sütun yerleştirin ve sütuna 250 μL RNA Bağlama Arabelleği ekleyin.
- Örneği (~150 μL adım 8.2.5'ten) RNA Bağlama Tamponu'nun bulunduğu sütuna yükleyin ve yukarı ve aşağı pipetleme yaparak karıştırın.
- Sütundaki örnek RNA Bağlama Tampon karışımına 400 μL% 95-100 etanol ekleyin. Kapağı kapatın ve sütunu birkaç kez ters çevirerek hemen karıştırın.
- 30 sn için tam hızda (≥ 10.000 x g) santrifüj. Akışa göre atın.
- Kolona 200 μL RNA Yıkama Tamponu ekleyin ve 30 sn boyunca tam hızda santrifüj ekleyin.
- Kolona 200 μL RNA Desülfonasyon Tamponu ekleyin ve oda sıcaklığında 30 dakika kuluçkaya yatırın. Kuluçkadan sonra, 30 s için tam hızda santrifüj. Akışa göre atın.
- Kolona 400 μL RNA Yıkama Tamponu ekleyin ve 30 sn boyunca tam hızda santrifüj ekleyin. Yıkama adımını ek 400 μL RNA Yıkama Tamponu ile tekrarlayın. Akışa göre atın.
- Boşaltılan Toplama Tüpündeki sütunu 2 dakika boyunca tam hızda santrifüjlayın. Sütunu RNase içermeyen bir tüpe aktarın.
- ≥ 10 μL DNase/RNase içermeyen suyu doğrudan sütun matrisine ekleyin ve oda sıcaklığında 1 dakika kuluçkaya yatırın. 30 sn boyunca tam hızda santrifüj.
NOT: Genellikle 20 μL'lik bir hacimde elute ediyoruz. Eluted RNA hemen kullanılabilir veya 3 aya kadar -20 °C'de saklanabilir. Uzun süreli depolama için -80 °C'de tutun. - RNA kalitesi ve miktarının parça analizörü değerlendirmesi için 2,5 μL'yi çıkarın ve kütüphane hazırlama ve yüksek verimli sıralamaya geçin (adım 9).
- Verimliliğin bisülfit dönüşüm kontrolü için 4 μL dönüştürülmüş RNA alın (adım 8.3).
- RT-PCR tarafından bisulfite Dönüştürme Kontrolü
NOT: Bu adım, sıralamaya geçmeden önce bisülfit dönüştürmenin başarılı olmasını sağlar. Homo sapiens'ten 28S ribozomal RNA, RNA metilasyon analizi için pozitif kontrol olarak kullanılacaktır, çünkü 4447 pozisyonundaki C kalıntısı (GenBank katılım # NR_003287) % 100 metillenmiş olarak tanımlanmıştır.
Astar Dizileri:
H 28SF astar: 5'-GGGGTTTTAYGATTTTTTTTTGATTTTTTGGG-3'
H 28SR astar: 5'-CCAACTCACRTTCCCTATTAATAAATAAAC-3'- Yüksek Kapasiteli cDNA Ters Transkripsiyon Kiti kullanarak Ters Transkripsiyon (RT) reaksiyon karışımını hazırlayın. Kit bileşenlerini buz üzerinde çözün ve RT master karışımını buz üzerinde aşağıdaki gibi hazırlayın:
4 μL Bisulfite dönüştürülmüş RNA (adım 8.2.14'ten itibaren):
2 μL 10xRT Tampon
0,8 μL 25x dNTP Karışımı [100 mM]
2 μL 10x RT Rastgele Astar
1 μL MultiScribe Ters Transkriptaz
1 μL RNaz Inhibitörü
9.2 μL çekirdeksiz H2O
NOT: Her RT reaksiyonı 0,2 mL PCR tüplerinde 20 μL son hacim içermelidir. - Tüpleri aşağıdaki RT programıyla termal çevrim içine koyun: 10 dakika boyunca 25 °C; 120 dakika için 37 °C; 5 dakika için 85 °C; daha sonra süresiz olarak 4 °C'de.
- PCR reaksiyonlarını, özellikle 28S rRNA'yı bir PCR düzeltme enzimi ile yükseltmek için hazırlayın. Kit bileşenlerini buz üzerinde çözün, hafifçe girdaplayın ve kısaca santrifüj edin. PCR master karışımını buz üzerinde veya buz gibi metal bir plaka tutucusuna aşağıdaki gibi hazırlayın:
10 μM H 28SF astarın 0,6 μL'si
10 μM H 28SF astarın 0,6 μL'si
6,5 μL Şablon cDNA
22,5 μL DNA Polimeraz ana karışımı
NOT: Her PCR reaksiyon, 0,2 mL PCR tüplerinde 20 μL son hacim içermelidir. - Tüpleri aşağıdaki PCR programıyla termal çevrime koyun: 5 dakika boyunca 95 °C'de ilk denatürasyon; 45 denatürasyon döngüsü (15 s için 95 °C), tavlama (30 s için 57 °C) ve uzama (15 s için 72 °C), 10 dakika boyunca 72 °C'de son uzama ve ardından süresiz olarak 4 °C'de tutma.
- Reaksiyonun 10 μL'sini% 2 agarose jel üzerinde çalıştırın. Beklenen bant boyutu 130 - 200 bp'dir.
- Yüksek Kapasiteli cDNA Ters Transkripsiyon Kiti kullanarak Ters Transkripsiyon (RT) reaksiyon karışımını hazırlayın. Kit bileşenlerini buz üzerinde çözün ve RT master karışımını buz üzerinde aşağıdaki gibi hazırlayın:
- PCR ürünlerinin dizilimi
- Enzimleri ve dNTP kalıntılarını gidermek ve güçlendirilmiş DNA'yı en az 20 μL DNaz/RNase içermeyen suda elute etmek için tercih edilen sütun bazlı bir yöntemle PCR ürünlerini saflaştırın.
- Saflaştırılmış DNA'yı spektrofotometre ile ölçün.
- Sıralama reaksiyon
- 40 ng PCR ürün/dizileme reaksiyoni kullanın.
- H 28SF ve H 28SR astarlarla her iki yönde sıralayın.
- Dizileri bilinen dönüştürülmemiş sırayla hizalayın (28S ribozomal N5 (RNA28SN5). C4447 konumunda C kalıntısı olup olmadığını ve başka bir yerde C yerine T kalıntısı olup olmadığını kontrol edin.
9. Kütüphane Hazırlama ve Yüksek Verimli Sıralama
- Elute-Prime-Fragment adımında protokolü başlatarak ve üretici talimatlarını izleyerek mRNA kitlerini (örneğin, Illumina TruSeq Stranded) kullanarak sıralama için kitaplıkları hazırlayın.
- Bununla birlikte, giriş RNA-Seq ve MeRIP-Seq örnekleri için, numuneleri 80 °C'de sadece asal olarak 2 dakika kuluçkaya yatırın, ancak daha fazla parçalamayın.
- Illumina platformlarını kullanarak sıralama gerçekleştirin. Sıralama reaksiyonları, tercihlere ve deneysel tasarıma göre, tek veya eşleştirilmiş uçlar, en az 100 nt uzunluğunda gerçekleştirilebilir.
10. Biyoinformatik Analizleri
- m6A Veri İşleme
- m6A'daki okuma kalitesini değerlendirmek ve fastq dosyalarını sıralamadan girmek için FASTQC24'i çalıştırın.
- Okumalardan düşük kaliteli uç ve bağdaştırıcı dizilerini kırpmak için Atropos25'i çalıştırın. Atropos çalışırken aşağıdaki parametreleri ayarlayın.
- Aşağıdaki bağdaştırıcı sıralarını kaldırın: AGATCGGAAGAG, CTCTTCCGATCT, AACACTCTTTCCCT, AGATCGGAAGAGCG, AGGGAAAGAGTGTT, CGCTCTTCCGATCT.
- Aşağıdaki Phred kalite kesmesini kullanın: 5, üretici tarafından belirtilen düşük kaliteli uçları kırpmak için (https://support.illumina.com/downloads/illumina-adapter-sequences-document-1000000002694.html).
- Kırpmadan sonra aşağıdaki minimum okuma uzunluğunu kullanın: 25 baz çift.
- GRh38 insan genom ve HIV [Entegre lineer pNL4-3Env-GFP] referansını FASTA formatında birleştirin.
- Birleştirilmiş başvuruyu HISAT226 ile dizine dizine dizin.
- Dizinlenmiş başvuruya hizalamak için kırpılmış okumalarda HISAT2'yi çalıştırın. Varsayılan HISAT parametrelerini kullanın.
- Hizalanmış okumaları SAMtools27 ile sıralayın ve dizine dizine ayırın.
- Sıralı kitaplıkların hizalama sonrası kalite denetimi için SAMtools stat ve Qualimap 228'i çalıştırın.
- İsteğe bağlı olarak, multiQC29 ile önceki adımdaki kalite ölçülerini toplayın ve özetleyin.
- HIV genomu 5' LTR ve 3' LTR'de homolog 634 bp diziye sahiptir: 5' LTR'den SAMtools ile ilgili 3' LTR bölgesine realign çoklumaing okumaları.
- m6A tepelerini tanımlamak için, macs230 (v 2.1.2) çağırma yazılımını çalıştırın. Tepe çağırma gen ifade düzeyinden etkilenebileceği ve kısa eksonlar tepe noktası olarak yanlış adlandırılabildiği için RNA-Seq verilerinde doğru çalışmayı sağlamak için MACS2 çalışma parametrelerini dikkatlice seçin. Bu nedenle, giriş sinyali, MACS2 tarafından DNA tabanlı verilere rutin olarak uygulanmadan m6A sinyalinden çıkarılmalıdır. MACS2'nin 'callpeak' alt komutuna aşağıdaki parametreleri uygulayın:
-keep-dup auto (MACS2 davranışını yinelenen okumalara doğru kontrol eder, 'otomatik', MACS'in 1e-5'i p-değer kesmesi olarak kullanarak binom dağılımına göre aynı konumdaki maksimum okuma sayısını hesaplamasına izin verir)
-g 2.7e9 (bp'de insan genomunun boyutu)
-q 0.01 (önemli zirveleri çağırmak için minimum FDR kesmesi)
-nomodel (ChIP-Seq deneyleri için özel olarak tasarlanmış değişen modelin yapımını atlamak için)
-slocal 0
-llocal 0 (bunu ve önceki parametreyi 0 olarak ayarlamak, MACS2'nin yumuşatma olmadan doğrudan çıkarmasını sağlar, giriş m6A okur)
-extsize 100 (bp'deki parçaların ortalama uzunluğu)
-B - Virüslü ve enfekte olmayan örnekleri karşılaştırmak için MACS2, 'bdgdiff' alt komutunu çağıran diferansiyel tepeyi çalıştırın. 'bdgdiff', önceki adımda 'callpeak' tarafından oluşturulan bedGraph dosyalarını girdi olarak alır. Her zaman noktası için, enfekte olmayan örneklerle enfekte olmayan örneklerin 'bdgdiff' ile karşılaştırılması, ilgili giriş sinyalinin m6A sinyalinden çıkarılması ve ek parametrelerin sağlanmasını çalıştırın: -g 60 -l 120.
- m5C Veri İşleme
- Bağdaştırıcı sıralarını ham okumalardan kırpmak için Cutadapt31'i aşağıdaki parametrelerle çalıştırın:
bağdaştırıcı "AGATCGGAAGAGCACACGTCTGAAC"
-minimum uzunluk=25. - Sıralama protokolü ters iplikçikten okumalar ürettiğinden, seqkit32 kullanarak kırpılan okumaları ters tamamla.
- Okuma kalitesini incelemek için FastQC'yi çalıştırın.
- GRh38 insan genom ve HIV [Entegre lineer pNL4-3Env-GFP] referansını FASTA formatında birleştirin.
- MeRanTK package33'ten meRanGh uygulamasıyla birleştirilmiş başvuruyu dizine dizine katın.
- meRanGh ile aşağıdaki parametrelerle hizalayın:
-UN, eşlenmemiş okumaların çıktı dosyalarına yazılmasını sağlar
-MM, çok eşlenmiş okumaların çıktı dosyasına yazılmasını sağlar
BedGraph'ta çıktı için -bg
-mbgc 10 filtre kapsamına göre bildirilen bölge (en az 10 kapsama alanı okuması) - HIV genomu 5' LTR ve 3' LTR'de homolog 634 bp diziye sahiptir: 5' LTR'den SAMtools ile ilgili 3' LTR bölgesine yeniden hizalanmış çokluma okumaları.
- meRanTK tarafından sağlanan meRanCall aracı aracılığıyla metilasyon çağırmayı aşağıdaki parametrelerle çalıştırın:
-rl = 126, okuma uzunluğu
-ei = 0.1, metilasyon oranı p-değer hesaplaması için hata aralığı
-cr = 0,99, beklenen dönüştürme - Her örneğin boyut faktörlerini tahmin etmek için MeRanTK'nin yardımcı program estimateSizeFactors.pl çalıştırın. Boyut faktörleri bir sonraki adımda parametre olarak kullanılacaktır.
- MeRanCompare'i, 12, 24 ve 36h zaman noktalarında enfekte olmayan vs enfekte olanların diferansiyel metilasyon analizi için çalıştırın. Aşağıdaki parametreler uygulanır: önceki adımdan raporlama ve boyut faktörleri için en az eşik olarak .01 önem değeri.
- Bağdaştırıcı sıralarını ham okumalardan kırpmak için Cutadapt31'i aşağıdaki parametrelerle çalıştırın:
Sonuçlar
Bu iş akışı, HIV enfeksiyonu bağlamında m6A ve m5C metilasyonunun rolünü araştırmak için yararlı olmuştur. Bunun için HIV ile enfekte ettiğimiz veya tedavi edilmeyen bir CD4+ T hücre hattı modeli (SupT1) kullandık. İş akışını durum başına 50 milyon hücre ile başlattık ve RNA kalite sayısı 10 olan ortalama 500 μg toplam RNA elde ettik (Şekil 1A-B). Poli-A seçimi üzerine durum başına 10 ila 12 μg mRNA (toplam RNA'nın yaklaşık% 2'sini temsil eden) aldık (Şekil 1B). Bu noktada, MeRIP-Seq boru hattı için 5 μg poli-A seçilen RNA ve BS-Seq boru hattı için 1 μg kullandık. HIV RNA'ları poli-adenilasyonlu olduğundan, başka bir eyleme gerek yoktur ve MeRIP-Seq ve BS-Seq prosedürleri doğrudan uygulanabilir.
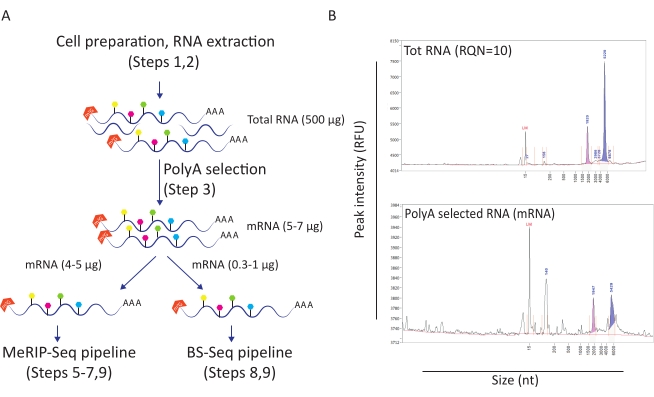
Şekil 1: Aşağı akış uygulamaları için RNA hazırlığı. A) Eşzamanlı MeRIP-Seq ve BS-Seq işlem hatları için RNA hazırlama ve dağıtımını gösteren iş akışı. Her dolgulu altıgen şekil, m6A (yeşil) veya m5C (pembe) gibi bir RNA değişiklik türünü temsil eder. Deneyi gerçekleştirmek için gereken RNA malzemesi miktarları belirtilir. B) Toplam RNA ekstraksiyonu (üst panel) ve poli-A seçimi (alt panel) üzerine beklenen RNA dağıtım profillerini (boyut ve miktar) gösteren temsili sonuçlar. Numuneler, belirli MeRIP-Seq ve BS-Seq prosedürlerine girmeden önce RNA kalitesini değerlendirmek için standart hassasiyet kiti ile parça analizörüne yüklendi. RQN: RNA kalite numarası; nt: nükleotidler. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
MeRIP-Seq boru hattı, RNA molekülleri boyunca m6A modifikasyonunun araştırılmasına izin veren RNA immün özürlüğe dayalı bir tekniktir. Bunun için RNA önce parçalanır ve daha sonra immün özürlülüğü ve yakalanması için manyetik boncuklara bağlı m6A spesifik antikorlarla inkübe edilir. MeRIP ile zenginleştirilmiş RNA parçaları ve el değmemiş (giriş) fraksiyonu daha sonra sıralanır ve m6A modifiye RNA bölgelerini ve dolayısıyla m6A metillenmiş transkriptleri tanımlamakla karşılaştırılır (Şekil 2A). Tekniğin çözünürlüğü RNA parçalanmasının verimliliğine dayanır. Aslında, daha kısa parçalar m6A kalıntısının daha hassas bir şekilde lokalizasyonunu sağlar. Burada hücresel poli-A seçilen RNA'lar ve viral RNA'lar 100-150 nt RNA parçaları elde etmek için 20 μL son hacimde 15 dakika boyunca RNA parçalanma tamponu ile iyon bazlı parçalanmaya maruz kaldı. 5 μg mRNA ile başlayarak, %90'lık bir geri kazanım oranına karşılık gelen 4,5 μg parçalı RNA'yı geri kazandık (Şekil 2B). Giriş kontrolü olarak doğrudan kütüphane hazırlığı ve dizilimine tabi tutulan 100 ng parçalı, saflaştırılmış RNA kullandık. Kalan RNA (~4.4 μg), parçalanmış RNA'nın anti-m6A spesifik antikorlara veya kontrol olarak anti-IgG antikorlarına bağlı boncuklarla inkübasyonu ile başlayan MeRIP-Seq boru hattına göre işlendi. 2,5 μg parçalı RNA'nın m6A'ya özgü RIP 'i (MeRIP), kütüphane hazırlığı ve dizilimine tabi olan yaklaşık 15 ng m6A ile zenginleştirilmiş malzemenin geri alınmasına izin verdi (Şekil 2B). Anti-IgG kontrollü RIP, beklendiği gibi, daha fazla analize izin verecek kadar RNA vermedi (Şekil 2B).
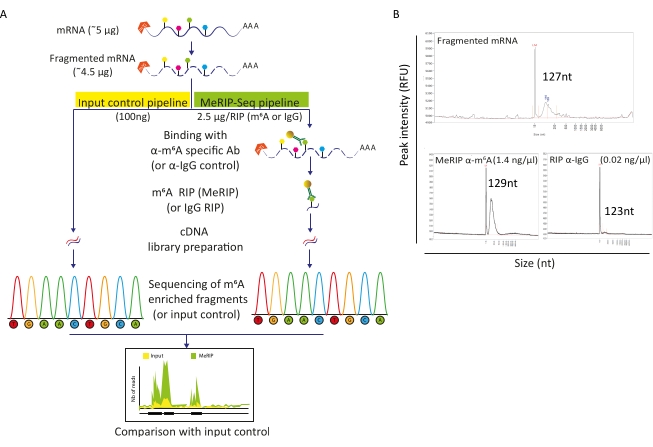
Şekil 2: MeRIP-Seq ardışık düzeni. A) MeRIP-Seq iş akışının ve giriş denetiminin şematik gösterimi. Poli-A seçimi üzerine, numuneler 120-150 nt parçalara ayrıldı ve doğrudan sıralamaya (100 ng, giriş kontrolü) tabi tutuldu veya sıralamadan önce negatif kontrol olarak anti-m6A spesifik antikor veya anti-IgG antikoru ile RNA immün önkekipitasyon (2.5 μg, RIP) için kullanıldı. B) Parçalanma (üst panel) ve RIP (alt paneller, MeRIP: sol, IgG kontrolü: sağ) üzerine beklenen RNA dağıtım profillerini (boyut ve miktar) gösteren temsili sonuçlar. Numuneler, kütüphane hazırlama ve sıralamaya daha fazla işlemeden önce RNA kalitesini ve konsantrasyonu değerlendirmek için parça analizörüne yüklendi. Parçalanmış RNA analizi RNA standart duyarlılık kiti kullanılarak yapılırken, immün özürlü RNA yüksek hassasiyet kiti kullanılmıştır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
BS-Seq boru hattı, nükleotid çözünürlükte m5C RNA modifikasyonunun araştırılmasına izin verir ve m5C metillenmiş transkriptlerin tanımlanmasına yol açar. Bisülfit dönüşümünde metilasyonsuz sitozinler urasil haline dönüştürülürken metillenmiş sitozinler değişmeden kalır (Şekil 3A). Bisülfit dönüştürme prosedürünün zorlu koşulları (yani, yüksek sıcaklık ve düşük pH) nedeniyle, dönüştürülmüş mRNA'lar yüksek oranda bozulur (Şekil 3B), ancak bu kütüphane hazırlama ve sıralamayı engellemez. Bisulfite dönüşümü sadece tek iplikli RNA'da etkilidir ve bu nedenle ikincil çift iplikli RNA yapıları tarafından engellenebilir. C-U dönüşümünün verimliliğini değerlendirmek için iki kontrol sunduk. Pozitif bir kontrol olarak, 28S rRNA23'ün C4447 pozisyonunda yüksek metillenmiş bir sitozin varlığından yararlandık. Metillenmiş bölgeyi çevreleyen 200 bp'lik bir parçanın RT-PCR amplifikasyonu ve dizilimi üzerine, tüm sitozinlerin başarıyla urasillere dönüştürüldüğünü ve böylece 4447 pozisyonundaki sitozin hariç, DNA dizisinde timistinler olarak göründüğünü gözlemleyebiliriz. Bisülfit dönüşüm oranı için bir kontrol olarak, piyasada bulunan sentetik ERCC RNA dizilerini kullandık. Bu karışım, çeşitli ikincil yapılara ve uzunluklara sahip bilinen, metillenmemiş ve poli-adenilasyonlu RNA dizilerinden oluşan bir havuzdan oluşur. Kütüphane hazırlığı ve dizileme üzerine, tüm ERCC dizilerinde ve her örnekte toplam C kalıntıları arasında dönüştürülen C sayısı sayılarak gerçekleştirilebilecek dönüşüm oranını hesaplamak için bu ERCC dizilerine odaklandık. Bisülfit dönüşüm reaksiyonunun verimliliğini ve başarısını doğrulayarak% 99,5'lik bir dönüşüm oranı elde ettik (Şekil 3D).
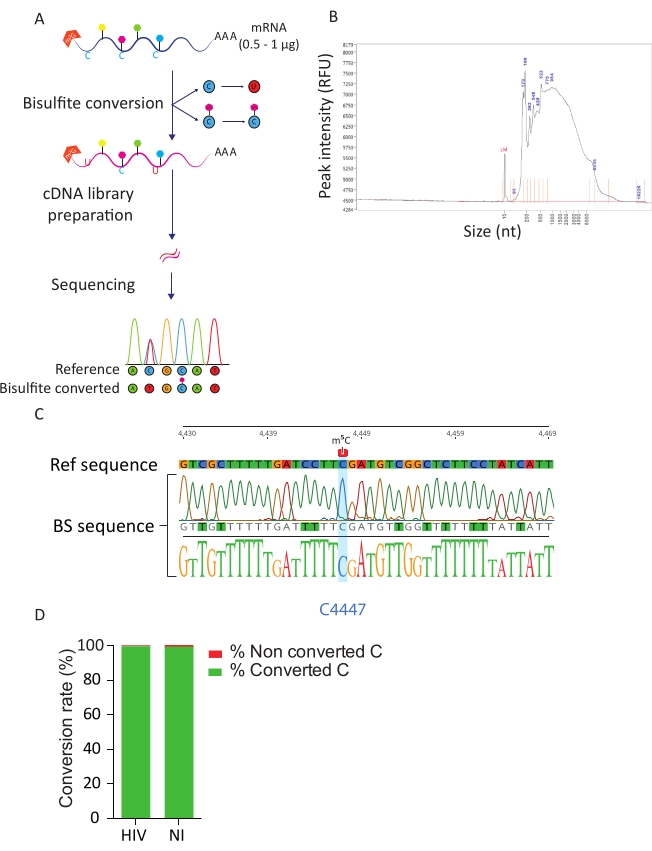
Şekil 3: BS-Seq ardışık düzeni. A) BS-Seq iş akışının şematik gösterimi. Poli-A seçimi üzerine, numuneler bisülfitlere maruz kalır ve metillenmiş olmayan C kalıntıları için C'den U'ya dönüşüme (deaminasyon nedeniyle) neden olur. Buna karşılık metillenmiş C kalıntıları (m5C) bisülfit tedavisinden etkilenmez ve değişmeden kalır. B) Standart hassasiyet kiti ile parça analizörü üzerinde yapılan analizler sonucunda bisülfit dönüştürülmüş RNA dağılım profilinin (boyut ve miktar) temsili sonucu. C) 28S rRNA'da 4447 pozisyonunda %100 metillenmiş C'yi çevreleyen bölgenin RT-PCR ampliconunun temsili dizileme sonucunu gösteren elektroferogram (mavi ile vurgulanır). Buna karşılık, referans dizisinin C kalıntıları bisülfit dönüşüm başarısı nedeniyle amplicon dizisinde T kalıntısı olarak tanımlanmıştır. D) HIV ile enfekte ve enfekte olmayan hücrelerde ERCC spike-in dizilerinin analizi ile C-U dönüşüm oranının değerlendirilmesi. Ortalama dönüşüm oranı %99,5'tir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
M6A ile zenginleştirilmiş numuneler, bisülfit dönüştürülmüş numuneler ve giriş kontrolleri kütüphane hazırlama, sıralama ve biyoinformatik analiz için daha fazla işlenir (Şekil 4). Ele alınan deneysel tasarım ve biyolojik soru(lar)a göre birden fazla biyoinformatik analiz uygulanabilir. Burada ilke kanıtı olarak, HIV enfeksiyonuna bağlı farklı metillenmiş transkriptlerin tanımlanmasına odaklanan bir potansiyel uygulamadan (yani diferansiyel metilasyon analizi) temsili sonuçlar gösteriyoruz. Kısaca, viral yaşam döngüsü sırasında RNA metilasyonlarının rolünü daha iyi anlamak için, hem enfekte olmayan hem de HIV ile enfekte olmuş hücrelerde gen ekspresyon seviyelerinden bağımsız olarak transkriptlerin m6A veya m5C metilasyon seviyesini araştırdık. Gen ekspresyonu normalleşmesi üzerine, ZNF469 transkriptinin enfeksiyon durumuna göre farklı m6A-metillendiğini tespit ettik, gerçekten de bu transkript HIV enfeksiyonu üzerinde birkaç metillenmiş zirve gösterirken enfekte olmayan hücrelerde metillenmedi (Şekil 5A). m5C'deki benzer bir diferansiyel metilasyon analizi, PHLPP1 transkriptinin HIV durumunda daha sık metillenme eğiliminde olan birkaç metillenmiş kalıntı içerdiğini ortaya koydu (Şekil 5B). Bu bağlamda, her iki analiz de HIV enfeksiyonlarının hücresel epitranscriptomu etkilediğini göstermektedir.
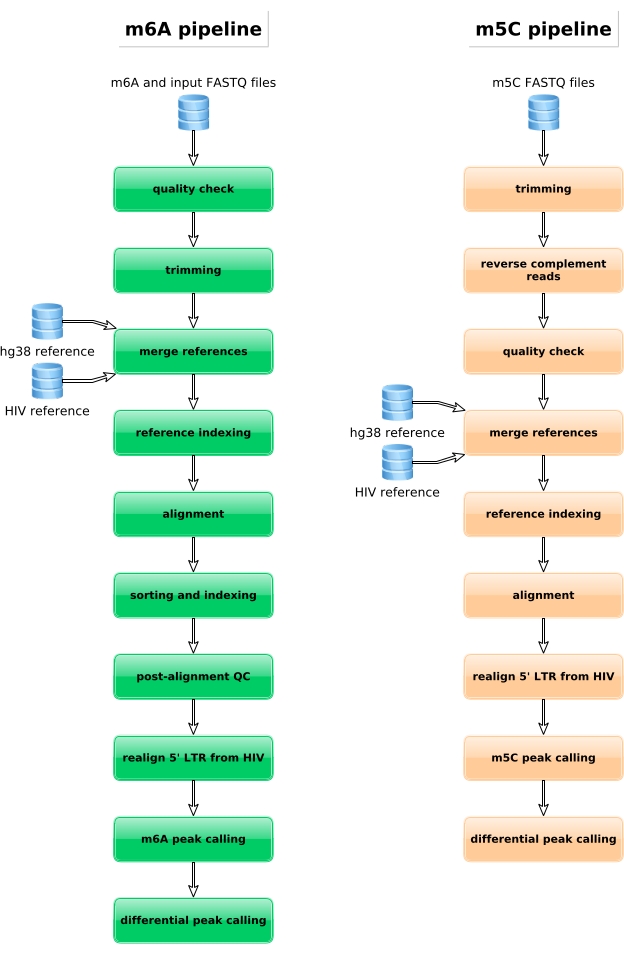
Şekil 4: m6A ve m5C verilerinin analizi için biyoinformatik iş akışının şematik gösterimi. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
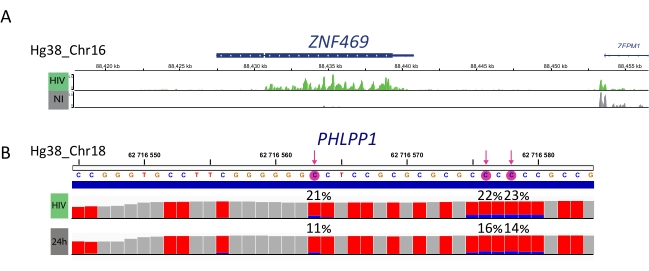
Şekil 5: Enfeksiyon üzerine farklı metillenmiş transkript örneği. A) HIV ile enfekte (yeşil) ve enfekte olmayan (gri) hücrelerde ZNF459 transkriptinin m6A metilasyonunun temsili sonucu. Tepe yoğunluğu (giriş ifadesi çıkarma üzerine) x ekseni boyunca kromozomdaki y ekseninde ve konumunda gösterilir. Diferansiyel metilasyon analizi, ZFN469 transkriptinin HIV enfeksiyonu üzerine hipermetilasyon olduğunu ortaya koymaktadır. B) HIV ile enfekte (üst şerit) ve enfekte olmayan (alt şerit) hücrelerde m5C metillenmiş genin temsili sonucu. Her çubuğun yüksekliği nükleotid başına okuma sayısını temsil eder ve kapsama değerlendirmesine izin verir. Her C kalıntısı kırmızı ile temsil edilir ve metillenmiş C oranı mavi ile temsil edilir. Tam metilasyon oranı (%) her C kalıntısının üzerinde bildirilmektedir. Oklar istatistiksel olarak anlamlı olarak metillenmiş C'yi vurgular. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Tartışmalar
Viral enfeksiyonda RNA modifikasyonlarının rolü hala büyük ölçüde bilinmemektedir. Epitranscriptomic modifikasyonların viral enfeksiyon bağlamındaki rolünün daha iyi anlaşılması, yeni antiviral tedavi hedefleri arayışına katkıda bulunabilir.
Bu çalışmada, enfekte hücrelerin m6A ve m5C epitranscriptomelerinin araştırılmasına izin veren eksiksiz bir iş akışı sağlıyoruz. Biyolojik soruya bağlı olarak, başlangıç malzemesi olarak poli-A-seçilmiş RNA'yı kullanmanızı tavsiye ederiz. İsteğe bağlı olsa da, işlem hattı toplam RNA ile kullanılabileceğinden, rRNA'ların yanı sıra küçük RNA'ların da yüksek oranda değiştirildiğini ve önemli sayıda metillenmiş kalıntı içerdiğini akılda tutmak önemlidir. Bu, anlamlı sıralama verilerinin kalitesinin ve miktarının azalmasına neden olabilir.
Bununla birlikte, çalışmanın odak noktası poli-adenilasyonlu olmayan RNA ise, RNA ekstraksiyon adımı, küçük RNA'nın atılmasını önlemek (sütun bazlı RNA ekstraksiyonu durumunda) ve boru hattına girmek için poli-A seçimi yerine ribozom tükenmesi tekniklerini imtiyaz etmek için uyarlanmalıdır.
Kütüphane hazırlığı için yüksek kaliteli RNA, doğru parçalanma ve uygun m6A ile zenginleştirilmiş ve BS dönüştürülmüş RNA kalitesi sağlamak için bir parça analizörü veya biyoanalizör kullanmanızı şiddetle tavsiye ederiz. Ancak, bu ekipman her zaman mevcut değildir. Alternatif olarak, RNA, mRNA ve parçalanmış RNA'nın büyüklüğü de agarose jel üzerinde görselleştirme ile değerlendirilebilir. Alternatif olarak, kütüphane hazırlığı RNA miktarı önceden değerlendirilmeden gerçekleştirilebilir.
M6A epitranscriptomic manzarayı keşfetmek için antikor bazlı MeRIP-Seq16 tekniğini kullandık. Bu teknik RNA immün önksepitasyona dayanır ve başarılıdır; ancak, bazı adımların dikkatli bir optimizasyona ihtiyacı vardır ve kritik olabilir. M6A metilasyonunun esas olarak konsensüs dizisi RRA*CH içinde gerçekleştiği açıklansa da, bu motif mRNA molekülleri boyunca oldukça sık görülür ve metilasyon bölgesinin kesin olarak tanımlanmasına izin vermez. Bu nedenle, RIP tabanlı çözünürlüğü iyileştirmek için küçük RNA parçaları üreten tekrarlanabilir ve tutarlı bir RNA parçalanması elde etmek önemlidir. Bu protokolde, deneysel ortamımızda tekrarlanabilir ve tutarlı sonuçlar sağlayan optimize edilmiş bir prosedür öneriyoruz; ancak, bu parçalanma adımının belirli örnek özelliklere göre daha fazla iyileştirmeye ihtiyacı olabilir.
Son zamanlarda m6A doğrudan dizileme izin yeni bir teknik tanımlanmıştır. M6A RNA modifikasyonu24 ile karşılaşmaya yanıt olarak benzersiz RT imzaları sergileyen belirli ters transkriptaz varyantlarının kullanımına dayanmaktadır. Bu teknoloji, dikkatli optimizasyon üzerine, MeRIP-Seq ile karşı karşıya kalınmış büyük sınırlamayı aşabilir (ilk malzeme miktarını azaltır ve daha yüksek bir çözünürlüğe izin verir). M5C modifikasyonu keşfetmek için, modifiye C kalıntılarını nükleotid çözünürlüğünde tespit etmek için bisülfit dönüştürme tekniğini kullanmaya karar verdik. RNA ikincil yapıların varlığı nedeniyle yanlış pozitif oranı azaltmak için, ERCC çivili kontrollerin kullanımı sayesinde 3 döngü denatürasyon / bisülfit dönüştürme ve bisülfit dönüşüm oranı performansını daha fazla kontrol ettik. Bu tekniğe bağlı sınırlamalardan biri, bisülfit dönüşümünün çok sert olması ve üç denatürasyon / bisulfite dönüştürme döngüsünün bazı RNA'ları bozabileceği ve bu nedenle çözünürlüğü azaltabileceğidir. Ancak, bizim ayarımızda, veri kümesinin kalitesini artırmak için potansiyel olarak biraz daha düşük bir çözünürlükle yetinmeyi seçtik.
Bu optimizasyonlar ve kontroller sayesinde, epitranscriptomik manzarayı ve viral enfeksiyonlar, konak-patojen etkileşimleri veya belirli tedavilere maruz kalma bağlamındaki değişimini araştırmak için yararlanılabilen güvenilir ve sağlam bir iş akışı sağlayabildik.
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Bu çalışma İsviçre Ulusal Bilim Vakfı (31003A_166412 ve 314730_188877 hibeleri) tarafından desteklendi.
Malzemeler
| Name | Company | Catalog Number | Comments |
| AccuPrime Pfx SuperMix | Invitrogen | 12344-040 | |
| anti-m6A antibody _Clone 17-3-4-1 | Millipore | MABE1006 | |
| Chloroform | Merck | 67-66-3 | |
| ERCC | Invitrogen | 4456740 | |
| EZ RNA Methylation Kit | Zymo Research | EZR5001 | |
| Fragment analyzer RNA Kit - HS RNA Kit | Agilent | DNF-472-0500 | |
| Fragment analyzer RNA Kit - RNA Kit | Agilent | DNF-471-0500 | |
| High-Capacity cDNA Reverse Transcription Kit | Applied Biosystem | 4368814 | |
| Illumina TruSeq Stranded mRNA | Illumina | 20020594 | |
| Magnetic Beads A/G Blend | Merck | 16-663 | |
| N6-Methyladenosine, 5′-monophosphate sodium salt (m6A) | Sigma Aldrich | M2780-10MG | |
| Normal Mouse IgG | Merk | 12371 | |
| Oligo(dT)25 | Life Technologies | 61005, | |
| PCRapace | Stratec | 1020220300 | |
| Quick RNA Viral Kit | Zymo Research | 1034 | |
| RNA Clean & Concentrator | Zymo Research | R1015 | |
| RNA Fragmentation Reagent | Ambion | AM8740 | |
| RNase Inhibitor | Ambion | AM2684 | |
| Trizol | TRIzol Reagent | 15596026 |
Referanslar
- Machnicka, M. A., et al. MODOMICS: a database of RNA modification pathways--2013 update. Nucleic Acids Research. 41, Database issue 262-267 (2013).
- Zaccara, S., Ries, R. J., Jaffrey, S. R. Reading, writing and erasing mRNA methylation. Nature Reviews Molecular Cell Biology. 20 (10), 608-624 (2019).
- Davalos, V., Blanco, S., Esteller, M. SnapShot: Messenger RNA Modifications. Cell. 174 (2), 498(2018).
- Saletore, Y., et al. The birth of the Epitranscriptome: deciphering the function of RNA modifications. Genome Biology. 13 (10), 175(2012).
- Zhao, B. S., Roundtree, I. A., He, C. Post-transcriptional gene regulation by mRNA modifications. Nature Reviews Molecular Cell Biology. 18 (1), 31-42 (2017).
- Netzband, R., Pager, C. T. Epitranscriptomic marks: Emerging modulators of RNA virus gene expression. Wiley Interdisciplinary Reviews: RNA. 11 (3), 1576(2020).
- Pereira-Montecinos, C., Valiente-Echeverria, F., Soto-Rifo, R. Epitranscriptomic regulation of viral replication. Biochimica et Biophysica Acta. 1860 (4), 460-471 (2017).
- Lichinchi, G., et al. Dynamics of the human and viral m(6)A RNA methylomes during HIV-1 infection of T cells. Nature Microbiology. 1, 16011(2016).
- Courtney, D. G., et al. Epitranscriptomic Addition of m(5)C to HIV-1 Transcripts Regulates Viral Gene Expression. Cell Host & Microbe. 26 (2), 217-227 (2019).
- Kennedy, E. M., et al. Posttranscriptional m(6)A Editing of HIV-1 mRNAs Enhances Viral Gene Expression. Cell Host & Microbe. 19 (5), 675-685 (2016).
- Tirumuru, N., Wu, L. HIV-1 envelope proteins up-regulate N (6)-methyladenosine levels of cellular RNA independently of viral replication. Journal of Biological Chemistry. 294 (9), 3249-3260 (2019).
- Tirumuru, N., et al. N(6)-methyladenosine of HIV-1 RNA regulates viral infection and HIV-1 Gag protein expression. Elife. 5, (2016).
- Cristinelli, S., Angelino, P., Janowczyk, A., Delorenzi, M., Ciuffi, A. HIV Modifies the m6A and m5C Epitranscriptomic Landscape of the Host Cell. Frontiers in Virology. 1 (11), (2021).
- Khoddami, V., Cairns, B. R. Transcriptome-wide target profiling of RNA cytosine methyltransferases using the mechanism-based enrichment procedure Aza-IP. Nature Protocols. 9 (2), 337-361 (2014).
- Hussain, S., Aleksic, J., Blanco, S., Dietmann, S., Frye, M. Characterizing 5-methylcytosine in the mammalian epitranscriptome. Genome Biology. 14 (11), 215(2013).
- Dominissini, D., Moshitch-Moshkovitz, S., Salmon-Divon, M., Amariglio, N., Rechavi, G. Transcriptome-wide mapping of N6-methyladenosine by m6A-seq based on immunocapturing and massively parallel sequencing. Nature Protocols. 8 (1), 176-189 (2013).
- Dominissini, D., et al. Topology of the human and mouse m6A RNA methylomes revealed by m6A-seq. Nature. 485 (7397), 201-206 (2012).
- Shobbir Hussain, J. A., Blanco, S., Dietmann, S., Frye, M. Characterizing 5-methylcytosine in the mammalian epitranscriptome. Genome Biology. 14 (215), (2013).
- Amort, T., et al. Distinct 5-methylcytosine profiles in poly(A) RNA from mouse embryonic stem cells and brain. Genome Biology. 18 (1), 1(2017).
- Endrullat, C., Glökler, J., Franke, P., Frohme, M. Standardization and quality management in next-generation sequencing. Applied & Translational Genomics. 10, 2-9 (2016).
- Schaefer, M., Pollex, T., Hanna, K., Lyko, F. RNA cytosine methylation analysis by bisulfite sequencing. Nucleic Acids Research. 37 (2), 12(2009).
- Cristinelli, S., Angelino, P., Janowczyk, A., Delorenzi, M., Ciuffi, A. HIV Modifies the m6A and m5C Epitranscriptomic Landscape of the Host Cell. biorxiv. 1 (11), (2021).
- Squires, J. E., et al. Widespread occurrence of 5-methylcytosine in human coding and noncoding RNA. Nucleic Acids Research. 40 (11), 5023-5033 (2012).
- Aschenbrenner, J., et al. Engineering of a DNA Polymerase for Direct m(6) A Sequencing. Angewandte Chemie (International ed. in English). 57 (2), 417-421 (2018).
- Bioinformatics. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc (2021).
- Didion, J. P., Martin, M., Collins, F. S. Atropos: specific, sensitive, and speedy trimming of sequencing reads. PeerJ. 5, 3720(2017).
- Kim, D., Langmead, B., Salzberg, S. L. HISAT: a fast spliced aligner with low memory requirements. Nature Methods. 12 (4), 357-360 (2015).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Okonechnikov, K., Conesa, A., García-Alcalde, F. Qualimap 2: advanced multi-sample quality control for high-throughput sequencing data. Bioinformatics. 32 (2), Oxford, England. 292-294 (2016).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), Oxford, England. 3047-3048 (2016).
- Zhang, Y., et al. Model-based Analysis of ChIP-Seq (MACS). Genome Biology. 9 (9), 137(2008).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet Journal. 17 (1), (2011).
- Shen, W., Le, S., Li, Y., Hu, F. SeqKit: A Cross-Platform and Ultrafast Toolkit for FASTA/Q File Manipulation. PLOS ONE. 11 (10), 0163962(2016).
- Rieder, D., Amort, T., Kugler, E., Lusser, A., Trajanoski, Z. meRanTK: methylated RNA analysis ToolKit. Bioinformatics. 32 (5), 782-785 (2015).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır