Method Article
שיטה מבוססת ליפידים-למידת מכונה לאפיון וכימות משקעי שומנים באמצעות אפיתל פיגמנט רשתית שמקורו ב-iPSC
In This Article
Summary
מחלות עיניים ניווניות המשפיעות על שכבת אפיתל פיגמנט הרשתית של העין יש מקורות מונוגניים ופוליגניים. מספר מודלים של מחלות ויישום תוכנה, LipidUNet, פותחו כדי לחקור מנגנונים של מחלות, כמו גם התערבויות טיפוליות פוטנציאליות.
Abstract
אפיתל פיגמנט הרשתית (RPE) הוא חד-שכבתי של תאים משושים הממוקמים בחלק האחורי של העין. הוא מספק הזנה ותמיכה לקולטני אור ונימים כורואידיים, מבצע פגוציטוזה של מקטעים חיצוניים של קולטני אור (POS), ומפריש ציטוקינים באופן מקוטב לשמירה על הומאוסטזיס של הרשתית החיצונית. RPE לא מתפקד, הנגרם על ידי מוטציות, הזדקנות וגורמים סביבתיים, גורם לניוון של שכבות רשתית אחרות וגורם לאובדן ראייה. מאפיין פנוטיפי מובהק של RPE מנוון הוא מרבצי שומנים עשירים בשומנים תוך ות-תאיים. משקעים אלה הם פנוטיפ נפוץ במחלות ניווניות שונות ברשתית. כדי לשחזר את פנוטיפ משקעי השומנים של ניוון רשתית מונוגני במבחנה, נוצר RPE המושרה פלוריפוטנטית שמקורה בתאי גזע (iRPE) מפיברובלסטים של חולים. קווי תאים שנוצרו מחולים עם Stargardt ומחלת ניוון רשתית מאוחרת (L-ORD) הוזנו עם קופה במשך 7 ימים כדי לשכפל את הפונקציה הפיזיולוגית RPE, מה שגרם לפתולוגיה הנגרמת על ידי קופה phagocytosis במחלות אלה. כדי ליצור מודל לניוון מקולרי תלוי גיל (AMD), מחלה פוליגנית הקשורה להפעלה משלימה חלופית, iRPE אותגר עם אנפילטוקסינים משלימים חלופיים. משקעי השומנים התוך-תאיים והתת-תאיים אופיינו באמצעות אדום הנילוס, בורון-דיפירומתין (BODIPY) ואפוליפופרוטאין E (APOE). כדי לכמת את צפיפות משקעי השומנים, פותחה תוכנה מבוססת למידת מכונה, LipidUNet. התוכנה אומנה על תמונות הקרנה בעוצמה מרבית של iRPE על משטחי תרבית. בעתיד, הוא יאומן לנתח תמונות תלת מימדיות (3D) ולכמת את נפח טיפות השומנים. תוכנת LipidUNet תהיה משאב רב ערך לגילוי תרופות המפחיתות את הצטברות השומנים במודלים של מחלות.
Introduction
אפיתל פיגמנט הרשתית (RPE) הוא חד-שכבה של תאים הממוקמת בחלק האחורי של העין בסמוך לקולטני אור ברשתית. RPE ממלא תפקיד חיוני בשמירה על ראייה תקינה על ידי מתן תמיכה מטבולית ומבנית לקולטני האור. תאי RPE בריאים מאופיינים במורפולוגיה משושה מובהקת. הם מחוברים על ידי צמתים הדוקים, המאפשרים RPE לפעול כמחסום בין choriocapillaris הממוקם בצד הבסיס שלה photoreceptors הממוקם apically. כדי לשמור על המערכת האקולוגית של הרשתית, RPE מעביר מטבוליטים מרכזיים, למשל גלוקוז, לקולטני אור באופן שממזער את צריכת הגלוקוז ב-RPE1. בשל מגבלה זו, RPE תלוי במטבוליטים אחרים כדי לשמור על הצרכים המטבוליים שלהם, כולל חומצות שומן, אשר RPE ממיר לקטונים באמצעות חמצון β2. בהתחשב בנטייה של RPE להשתמש בחומצות שומן, אשר ככל הנראה ממוחזרות מעיכול מקטע חיצוני של קולטני אור (POS), כמקור אנרגיה, שינויים מזיקים במסלולי עיבוד השומנים ב- RPE מובילים לעתים קרובות, או מעורבים, הן במחלות רשתית ניווניות מונוגניות והן פוליגניות3.
ניוון מקולרי תלוי גיל (AMD), מחלת עיניים ניוונית פוליגנית הגורמת לניוון RPE, נקשרה גם לאוטופגיה חריגה ולמטבוליזם שומנים בחד-שכבה RPE. הכישלון של חד-שכבה RPE לא מתפקדת לעבד קופה ולבצע פונקציות קריטיות אחרות מוביל למשקעים חוץ-תאיים (תת-RPE) הנקראים משקעים ליניאריים בסיסיים (BLinD) הממוקמים בין RPE לממברנה של ברוך - סימן היכר של פתולוגיות AMD. המרכיבים העיקריים של BLinD כוללים ליפופרוטאינים, שהנפוץ שבהם הוא אפוליפופרוטאין E (APOE)4. הצטברות של שכבות דקות של BLinD יכולה להוביל לדרוסן רך, המוכר כסימפטום קליני של AMD 5,6.
מספר קבוצות הראו כי מודלים של תאי גזע שמקורם במבחנה הגורמים לתפקוד לקוי של RPE כוללים הצטברות שומנים תת-RPE 7,8,9. Hallam et al. (2017) יצרו RPE מושרה פלוריפוטנטית נגזר תאי גזע (iRPE) מחולים עם סיכון גבוה ל- AMD עקב פולימורפיזם של הגן CFH. ה- iRPE הראה הצטברות דרוסן, כפי שמסומן על ידי APOE, וה- RPE בסיכון גבוה צבר משקעים גדולים יותר מאשר iRPE שנוצר מחולים בסיכון נמוך10.
כדי ליצור מודל במבחנה המשחזר סימני היכר תאיים של AMD, כגון טיפות שומנים ושקיעת דרוסן, קווי iRPE שנוצרו מדגימות דם של מטופלים נקבעו באמצעות פרוטוקול מונחה התפתחותית11 שפורסם בעבר. ה- iRPE היו נתונים לסרום אנושי משלים (CC-HS), תמיסה המכילה אנפילטוקסינים המחקים גורם אפשרי אחד ל- AMD: איתות משלים חלופי מוגבר8. השקיעה התאית והתת-תאית של משקעי שומנים נמדדה באמצעות סמני שומנים וליפופרוטאינים נפוצים, APOE, אדום הנילוס ו-BODIPY. באמצעות סמנים אלה, הוכח כי איתות משלים פעיל באמצעות CC-HS החמיר הצטברות שומנים בתאי iRPE8.
כדי לפתח מודל מחלה למחלה ניוונית של רשתית מונוגנית, פותחו קווי iRPE מחולים עם מחלת סטארגארדט, מחלה הנגרמת על ידי מוטציות בגן ABCA4 ב- RPE. בעבר הוכח כי כאשר ABCA4 הוא דפק החוצה, A2E lipofuscin, משקעים תוך תאיים הידוע להכיל רמות גבוהות של פוספוליפידים ותוצרי חמצון שומנים תלויי אור, מצטבר בתוך RPE12. קווי נוקאאוט ABCA4 פותחו לצד קווי המטופלים, ושניהם היו נתונים להזנת קופה. ה- Stargardt iRPE הדגים פתולוגיה הנגרמת על ידי קופה phagocytosis, והציג הצטברות שומנים מוגברת שכומתה על ידי צביעת BODIPY. RPE שמקורו ב- ABCA4 KO iPSCs היו נתונים לטיפול CC-HS; כימות אות BODIPY הראה פגם בטיפול בשומנים גם במודל מחלת סטארגרדט9.
לאור שכיחותן של מחלות אלו והצורך בטיפולים יעילים, לצד מודלי המחלה הרלוונטיים שתוארו לעיל, יש צורך לבסס שיטות איתנות לכימות יעילותם של טיפולים פוטנציאליים. כדי לכמת משקעי שומנים באופן אובייקטיבי, אוטומטי וסטנדרטי, נוצרה תוכנה מבוססת למידת מכונה, LipidUNet, כך שבשילוב עם כלי ניתוח מסכות, ניתן לזהות במהירות וביעילות שקיעת שומנים באמצעות הסמנים הנפוצים Nile Red, BODIPY ו-APOE. לאחר מכן ניתן לנתח ולהציג באופן גרפי את הנתונים הסטטיסטיים המסכמים המתקבלים באמצעות צינור ניתוח זה, מה שמאפשר השוואה קלה של תנאי הטיפול. הסכימה של הפרוטוקול מוצגת באיור 1.
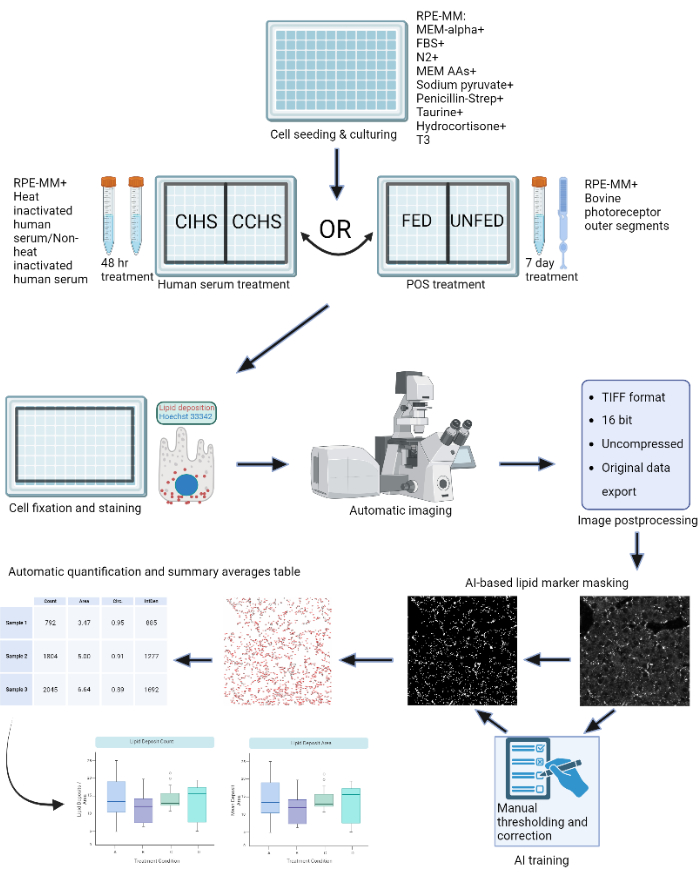
איור 1: סכמה של הפרוטוקול: תאי RPE גדלים על צלחת של 96 בארות ומאותגרים עם סרום אנושי פעיל או מקטעים חיצוניים מטוהרים של בקר כדי למדל סוגים שונים של ניוון רשתית במבחנה. תאי RPE קבועים ומוכתמים עבור משקעי ליפופרוטאין עם אדום הנילוס, BODIPY ו- APOE. מיקרוסקופ קונפוקלי משמש לרכישת ערימות Z של חלקיקי שומנים בעלי תווית פלואורסצנטית, אשר מעובדים לאחר מכן להקרנות דו-ממדיות בעוצמה מרבית. אלגוריתם למידת מכונה אומן לזהות ולפלח נכון חלקיקי ליפופרוטאין. טבלאות סיכום המכילות ספירת חלקיקים ומדדי צורה שונים נוצרות וניתן להשתמש בהן לצורך התוויית נתונים וניתוח סטטיסטי עוקבים. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
Protocol
כל שלבי הפרוטוקול מצייתים להנחיות שנקבעו על ידי ועדת האתיקה של המחקר האנושי של ה-NIH. עבודת תאי גזע ואיסוף דגימות מטופלים אושרו על ידי המועצה המשולבת לסקירה מוסדית של מדעי המוח (CNS IRB) תחת המשרד להגנה על מחקר אנושי (OHRP), NIH, בהתאם להנחיות 45 CFR 46 של ממשלת ארה"ב. דגימות המטופלים נאספו באמצעות טופס הסכמה שאושר על ידי CNS IRB בהתאם לקריטריונים שנקבעו על ידי הצהרת הלסינקי תחת מספר הפרוטוקול NCT01432847 (https://clinicaltrials.gov/ct2/show/NCT01432847?cond=NCT01432847&draw=2&rank=1).
1. דור iRPE
- צור iRPE מ- iPSC שמקורו בדם המטופל בעקבות הפרוטוקול שפורסם על-ידי Sharma et al., 202211 (איור 2 ואיור 3).
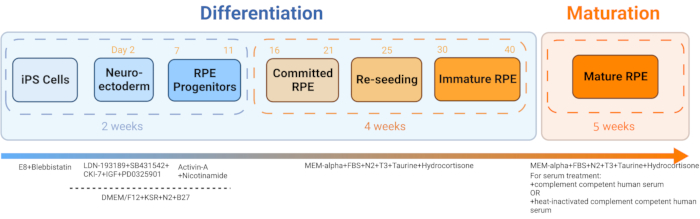
איור 2: סכמה של התמיינות והתבגרות iRPE. כדי ליצור iRPE, פרוטוקול התמיינות מבוסס היה אחריו, ואת התאים הורשו להבשיל במשך 5 שבועות. תרבית התאים המתקבלת פועלת כמודל במבחנה שניתן לתפעל עם טיפולים שונים כדי לחקות תפקוד לקוי של RPE במחלות כמו AMD ומחלת סטארגארדט. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
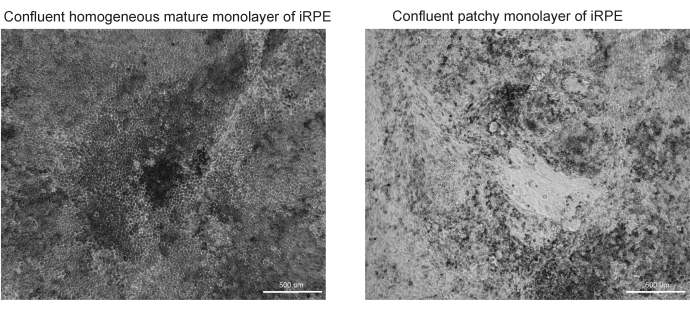
איור 3: תמונות מייצגות של בידול והתבגרות מוצלחים ולא מוצלחים של RPE. שתי תמונות שדה בהיר בהגדלה של פי 10 של TJP1 RPE מוצגות ביום ה-42 של פרוטוקול iRPE. (A) התמיינות והבשלה מוצלחות יראו RPE משולב עם פיגמנטציה ומורפולוגיה מצולעת. (B) התמיינות והבשלה לא מוצלחות יראו צבירים של תאים גוססים, כפי שמוצג כאן. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
2. הכנת מדיית תחזוקת RPE (RPE-MM)
- הפשיר את תוסף N2 ב 4 ° C במשך הלילה. הפשירו את כל הריאגנטים האחרים בטמפרטורת החדר (RT).
- בתנאים סטריליים, הוסף את הריאגנטים המפורטים בטבלה 1 בגורמי הדילול המפורטים, בהתאם לפרוטוקול שנקבע על ידי שארמה ואחרים, 202211.
- ערבבו היטב את המדיה וסננו אותה באמצעות יחידת סינון של 0.22 מיקרומטר.
הערה: חומרי הדפסה מתאימים לשימוש תוך שבועיים אם הם מאוחסנים בטמפרטורה של 4°C.
3. זריעת צלחת 96 בארות
- הפשירו אליקוט של ויטרונקטין ב-RT למשך 3-5 דקות או עד שהקרח נמס לחלוטין.
- דללו את הוויטרונקטין במי מלח חוצצי פוספט (DPBS) 1x Dulbecco כדי להשיג את פתרון העבודה הרצוי באמצעות דילול 1:200 (ויטרונקטין: DPBS). לקבלת צלחת 96 בארות, מצפים כל באר עם 200 μL של פתרון העבודה.
- שלב מעכב ROCK מופשר (Y-27632 dihydrochloride) עם RPE-MM בדילול 1:1000 כדי להשיג ריכוז סופי של 10 μM. זוהי מדיית הציפוי עבור תאי RPE.
- הפשירו את בקבוקון ה-iRPE באמצעות מערכת הפשרת תאים אוטומטית והעבירו את מתלה תאי ה-iRPE לצינור של 50 מ"ל.
- מדללים את מתלה התא עם מדיית הציפוי בדילול של 1:10. צנטריפוגה את הצינור ב 400 x גרם במשך 5 דקות.
- בזהירות לשאוף את supernatant ו resuspend את התאים ב 10 מ"ל של אמצעי ציפוי.
- ערבבו 400 μL של מדיית הציפוי עם 100 μL של תמיסת התא המרחפת מחדש לספירת תאים. השתמש aliquot זה כדי לקבוע את ריכוז התא קיימא של תרחיף התא באמצעות מונה כדאיות התא.
- לדלל את תרחיף התא עם מצע הציפוי לריכוז סופי של 60,000 תאים/מ"ל.
- שאפו לחלוטין את תמיסת ציפוי הוויטרונקטין מהצלחת בת 96 הבארות ושחררו 200 מיקרוליטר של תרחיף התא לכל באר. יהיו בערך 12,000 תאים / באר או ~ 200 תאים / מ"מ2.
- לדגור על לוחות תא זרע במשך 48 שעות ב 37 ° C ו 5% CO2. לאחר 48 שעות, שנה את המדיה ל- RPE-MM ללא תוספת מעכבי ROCK. שנה את המדיה כל 2-3 ימים במהלך תקופת ההבשלה של 5 שבועות.
4. מודלים של מחלות חוץ גופיות
- טיפול משלים בסרום אנושי מוסמך (CC-HS)
- יש להפשיר סרום משלים אנושי בטמפרטורה של 4°C למשך הלילה.
- הכינו את CC-HS והשלימו מדיה של סרום אנושי לא כשיר (CI-HS).
- כדי להכין מדיית CC-HS 5%, ערבבו את הסרום האנושי המשלים המופשר עם RPE-MM בדילול של 1:20. סנן את התמיסה דרך מסנן מדיה בגודל 0.22 מיקרומטר לפני השימוש.
- כדי להכין מדיית סרום אנושי לא כשיר (CI-HS) 5% משלימה, יש לחמם תחילה את ה-CC-HS באמבט מים בטמפרטורה של 57°C למשך 30 דקות ולאחר מכן לערבב עם אמצעי התרבית בדילול של 1:20. סנן את התמיסה דרך מסנן מדיה בגודל 0.22 מיקרומטר לפני השימוש.
- סרום לטפל בתאים עם 200 μL של 5% CC-HS או 5% CI-HS מדיה במשך זמן הדגירה הכולל של 48 שעות, רענון המדיה לאחר 24 שעות.
- שטפו את התאים עם 1x DPBS וקבעו אותם עם 4% פרפורמלדהיד למשך 20 דקות ב-RT. שטפו פעם נוספת עם DPBS 1x ואחסנו את הדגימות ב-4°C, שקועות ב-200 מיקרוליטר של DPBS.
- אופציונלי: אם תרצה, ליז את התאים מהצלחת כדי להציג רק שקיעת שומנים תת-RPE.
- כדי lyse את התאים ולהשאיר רק משקעי שומנים, להסיר את התקשורת ולהוסיף 200 μL של מים deionized לכל באר.
- דוגרים במשך 10-15 דקות, פיפטה למעלה ולמטה עד להסרת תאים. לשטוף שוב עם 200 μL של מים deionized ומיד לתקן את התאים עם 4% paraformaldehyde.
- אשר את יעילות הסרת התאים עם צביעה גרעינית באמצעות Hoechst. הוסף את Hoechst בדילול 1:2000 לתמיסת DPBS 1x המכילה אלבומין בסרום בקר (BSA), 0.5% Tween 20 ו-0.5% Triton-X 100. דגרו ב-RT במשך שעה אחת בחושך. לאחר מכן, לשטוף עם 1x DPBS.
- טיפול במקטע חיצוני של קולטני אור (POS) ב- iRPE
- הכנת קופה
- הסר את צינור גלולות הקופה מאחסון -80 ° C והפשיר לילה ב -4 ° C בדלי קרח מכוסה.
- הכן חיץ שטיפה קופה על ידי ערבוב 10 גרם סוכרוז ב 40 מ"ל של H 2 O כפול deionized (ddH2O).
- מחממים את התערובת ב-40-50°C תוך ערבוב עדין במשך 15 דקות. מוסיפים 840 מ"ג סודיום ביקרבונט לתערובת ומערבבים תוך כדי חימום במשך 10 דקות.
- התאם את הנפח הכולל של מאגר השטיפה של הקופה ל -100 מ"ל עם ddH2O והתאם את ה- pH של התמיסה ל- 8.3 עם 1 N HCl או 1 N NaOH, לפי הצורך. סנן את תמיסת הכביסה באמצעות מסנן 0.22 מיקרומטר.
הערה: ניתן להשהות את הפרוטוקול כאן; ניתן לאחסן את מאגר הכביסה של הקופה ב -4 ° C למשך הלילה. - לאחר ההפשרה, להשהות את הגלולה ב 15 מ"ל של חיץ שטיפה קופה. היו עדינים במהלך מתלי גלולות כדי להבטיח את שלמות הקופה. צנטריפוגו את מתלי הקופה ב 600 x גרם ב 4 ° C למשך 20 דקות ולאחר מכן לשאוף את supernatant.
- השהה מחדש את גלולת הקופה ב -10 מ"ל של מאגר השטיפה של הקופה.
- הסר aliquot 100 μL של קופה + קופה לשטוף חיץ (תמיסת POS) ולדלל ב 400 μL של 1x DPBS. מורחים 50 μL של תמיסת הקופה המדוללת על צלחת אגר דם וצלחת אגרוז כדי לבדוק מזהמים חיידקיים ופטרייתיים. הכינו בקרות חיוביות לכל אחד מהם ודגרו על כל הצלחות במשך 48 שעות בטמפרטורה של 37°C.
- בצע בדיקת qPCR על ידי הוספת 1 μL של פתרון קופה לבאר הזיהוי כדי לבדוק מיקופלסמה. כדי להגביר מקטעי DNA, בצע 40 מחזורים של דנטורציה (95°C, 15 שניות), וחישול והתארכות (60°C, דקה אחת). הפריימרים קדימה ואחורה לאיתור מיקופלסמה בדגימת הקופה הם כדלקמן:
פריימר קדמי: GGA TTA GAT ACC CTG GTA GTC CAC G
פריימר הפוך: CGT CAA TTC CTT TAA GTT TCA CTC TTG GC - למדוד ריכוז קופה באמצעות מנתח תאים aliquot על פי הצורך. עבור באר אחת של 96 צלחות באר עם תאי RPE, 3 x 106 קופה מספיק. היחס הרצוי הוא תא 10 POS/RPE. אחסן את aliquots ב 80 ° C לשימוש עתידי.
- הוספת קופה לתאים
- הפשירו בקבוקונים של קופה באמבט קרח.
- מערבבים את הכמות המחושבת של קופה מוכנה עם RPE-MM ומטפלים בתאים עם קופה פעם ביום למשך 7 ימים.
הערה: הכן פתרון קופה טרי מדי יום. - שטפו את התאים עם DPBS 1x ולאחר מכן תקנו אותם עם 4% פרפורמלדהיד למשך 20 דקות ב-RT. שטפו עם DPBS פעם נוספת ואחסנו דגימות ב-4°C, שקועות ב-200 מיקרוליטר של DPBS.
- הכנת קופה
5. צביעה על פיקדונות תת-RPE
- פרוטוקול צביעה אדומה של הנילוס
- לאחר קיבוע PFA, לשטוף את הדגימות 3 פעמים עם 1x DPBS.
הערה: אם לא נעשה שימוש מיידי, ניתן להשהות את הפרוטוקול כאן, אך יש לאחסן את הדגימות בתמיסת DPBS 1x + 0.02% נתרן אזיד ב- 4 °C. - להכנת תמיסת הנילוס אדום, יש להמיס את אבקת הנילוס האדום באצטון בריכוז של 3 מ"ג/מ"ל. דוגרים במשך 15 דקות ב-RT עם ערבוב תקופתי. סנן את התמיסות עם מסנן 0.22 מיקרומטר פעם או פעמיים בהתאם לרמת המשקע שנותר בתמיסה.
הערה: הגן על פתרון המלאי מפני אור. - כדי להכין את פתרון העבודה, דלל את פתרון המניות ביחס של 1:500 ב- DPBS 1x. הוסף 200 μL של פתרון העבודה לדגימה למשך 30 דקות ב- RT על שייקר והגן עליו מפני אור.
- יש לשטוף 3 פעמים עם 1x PBS ולאחסן את הדגימות בטמפרטורה של 4°C, שקועות ב-200 μL של DPBS.
הערה: אם עורכים ניסוי על טרנסוולים במקום צלחת של 96 בארות, ניתן להרכיב דגימות על שקופית עם אמצעי הרכבה, לכסות בכיסוי זכוכית ולאטום בלק שקוף. יש להקפיד להרכיב את הדגימה כשהתאים פונים כלפי מעלה.
- לאחר קיבוע PFA, לשטוף את הדגימות 3 פעמים עם 1x DPBS.
- פרוטוקול צביעת BODIPY
- עבור תמיסת מלאי, להמיס BODIPY ב dimethyl sulfoxide נטול מים (DMSO) כדי להשיג ריכוז מלאי של 3.8 mM.
- עבור דגימות קבועות של PFA, דלל מלאי BODIPY בשעה 1:300 ב- DPBS אחד. הוסיפו 200 μL לתאים ודגרו לילה על נדנדה ב-RT.
- יש לשטוף 3 פעמים עם DPBS 1x ולאחסן את הדגימות בטמפרטורה של 4°C, שקועות ב-200 μL של DPBS.
הערה: אם עורכים ניסוי על טרנסוולים במקום צלחת של 96 בארות, ניתן להרכיב דגימות על שקופית עם אמצעי הרכבה, לכסות בכיסוי זכוכית ולאטום בלק שקוף. יש להקפיד להרכיב את הדגימה כשהתאים פונים כלפי מעלה.
- פרוטוקול APOE immunostaining
- שלבו DPBS אחד עם אלבומין 1% בסרום בקר (BSA), 0.5% Tween 20 ו-0.5% Triton-X 100 ליצירת תמיסת חיץ.
- עבור דגימות קבועות של PFA, חסום וחדר את הדגימה ב- 200 μL של תמיסת החיץ למשך שעה אחת ב- RT.
- הוסף נוגדן ראשוני APOE מדולל בשעה 1:100 בתמיסת החיץ ודגור למשך הלילה ב- RT.
- למחרת, לשטוף את הדגימות 3 פעמים עם 1x DPBS.
- הוסף נוגדן משני בדילול 1:1000 בתמיסת החיץ והוסף 200 μL של התמיסה לתאים למשך שעה אחת ב- RT.
- יש לשטוף 3 פעמים עם DPBS אחד ולאחסן דגימות בטמפרטורה של 4°C, שקועות ב-200 מיקרוליטר של DPBS.
הערה: אם עורכים ניסוי על טרנסוולים במקום צלחת של 96 בארות, ניתן להרכיב דגימות על שקופית עם אמצעי הרכבה (Fluoromount), לכסות בכיסוי זכוכית ולאטום בלק שקוף. יש להקפיד להרכיב את הדגימה כשהתאים פונים כלפי מעלה.
6. אוטומציה ועיבוד תמונה
- סריקת תמונה אוטומטית
הערה: במחקר זה נעשה שימוש במיקרוסקופ סריקה קונפוקלי הפוך Zeiss LSM 800 ובתוכנת ZEN 3.2 (מהדורה כחולה). ודא שהצלחת בעלת 96 הבארות מחוממת ל-RT למשך 60 דקות לפחות לפני ההדמיה כדי למנוע סחף של מישור המוקד במהלך הסריקה עקב שינוי במקדם השבירה של התווך עם שינוי טמפרטורה.- באמצעות מיקרוסקופ קונפוקלי ומטרה של 40x, ליצור פרופיל סריקה עם תעלות פלואורסצנטיות מתאימות עבור סמן השומנים בשימוש וכל נוגדנים נוספים.
- השתמש בתיבת הסימון אריחים כדי להגדיר אוטומציית תמונות. כדי לכייל את לוח 96 הקידוחים, ודא שמדידות נשא הדגימה הנכונות הוזנו ונבחרו. לאחר מכן, לחץ על כיול כפתור כדי לכייל את הלוח בהתאם להוראות, מה שדורש שימוש במטרה 10x.
- בחר בתצוגת Advanced Setup כדי לבחור בארות מתאימות ולהוסיף 3 נקודות הדמיה שונות ליד מרכז הבאר באמצעות הפונקציה Position. ניתן לעשות זאת באופן ידני תחת לשונית המשנה מיקום או באופן אקראי באמצעות הכרטיסיה הגדרת מיקום ובחירה באפשרות הגדרה לפי ספק. חזור על כל הבארות של אותו צביעה.
- למיקוד מיטבי ולמיקום מחסנית Z במהלך אוטומציה, עבור אל הכרטיסיה אסטרטגיית מיקוד כדי לבחור השתמש בערכי Focus/Z Defined by Tiles Setup. שיטות חלופיות יכולות להשתמש באסטרטגיות מיקוד אחרות, אך מומלץ להשתמש בהגדרה זו לקבלת התוצאות העקביות ביותר.
- תחת הכרטיסיה אריחים, לחץ על אמת מיקומים והגדר ידנית את מישור Z המרכזי עבור כל מיקום. ההגדרות בכרטיסיית המשנה אפשרויות יסדרו את רכישת התמונות, לכן בדוק זאת לפני שתתחיל לצלם. לקבלת תמונות לפי סדר בחירת המיקומים, בטל את הבחירה בתיבות הסימון אזורים/מיקומים ובארות מוביל/מכיל. בחרו 'פצל סצינות לקבצים נפרדים' כדי להקל על עיבוד התמונה.
- ודאו שהכרטיסייה Z-Stack מוגדרת למרכז, טווח מוזן להעדפות המשתמש והלחצן 'אופטימלי ' נבחר לקביעת מרווח הזמן לפרוסה.
- לאחר מיטוב הכרטיסיות מצב רכישה, ערוצים, אסטרטגיית מיקוד, Z-Stack ואריחים , התחל את הניסוי.
- עיבוד תמונה
- בעזרת שיטת עיבוד תמונה באצווה, צרו הקרנות מרביות של כל ערימת Z בשיטה 'עומק מיקוד מורחב '.
- בשיטת עיבוד תמונות אצווה, יצאו את קובצי ההקרנה המרביים כתמונות TIFF של 16 סיביות. הגדר את הדחיסה ללא וודא שהנתונים המקוריים מסומנים. התמונה המתקבלת צריכה להיות TIFF בגווני אפור הקרנה מרבי של ערוץ הפלואורסצנטיות שבו מבוטא סמן השומנים.
7. פילוח וכימות
הערה: תוכנית LipidUNet אומנה על תמונות 40x מצלחת 96 בארות. מומלץ מאוד להשתמש בתמונות שהתקבלו באמצעות מטרה 40x.
- התקן את תוכנת LipidUNet. ניתן להוריד את LipidUNet ממאגר GitHub הבא: https://github.com/RPEGoogleMap/LipidUNet
- זהה את תמונות TIFF המייצגות את Nile Red, Bodipy או APOE והעבר אותן לתיקייה בשם imgs בספרייה בשם Nile_Red, Bodipy או APOE, בהתאם לשיטה שבה נעשה שימוש.
הערה: יש להשתמש במוסכמות שמות מדויקות כדי שתוכנית LipidUNet תזהה את הספריות. - פתחו את תוכנת LipidUNet (איור 4).
- בכרטיסייה חיזוי של התוכנה, בחר את הספרייה הרלוונטית (Nile_Red, Bodipy או APOE) על ידי לחיצה על שלוש הנקודות וניווט לספרייה בעלת השם. ודא שהתוכנית LipidUNet זיהתה כראוי את התמונות על-ידי בדיקת הערך Class.
- בחר סף הסתברות עבור האלגוריתם בין 0.01 ל- 0.99. ערך גבוה יותר יבטל יותר תוצאות חיוביות שגויות אך עלול לגרום ליותר תוצאות שליליות שגויות, וערכים נמוכים יותר עשויים להציג יותר תוצאות חיוביות שגויות תוך ביטול יותר תוצאות שליליות שגויות. ערך של 0.65 הוא ברירת המחדל והוא מומלץ.
- לחץ על חזוי.
הערה: התוכנה תחזור על כל התמונות באופן אוטומטי ותיצור תיקייה חדשה בשם predicted_masks בספרייה שנבחרה. - השתמשו בכלי ניתוח מסיכות כדי לעבור על המסיכות שנוצרו ולספק ספירה כמותית של משקעי השומנים הסף מתמונות המסיכה.
- נתח את נתוני הספירה שנוצרו כדי להשוות בין תנאי הטיפול.
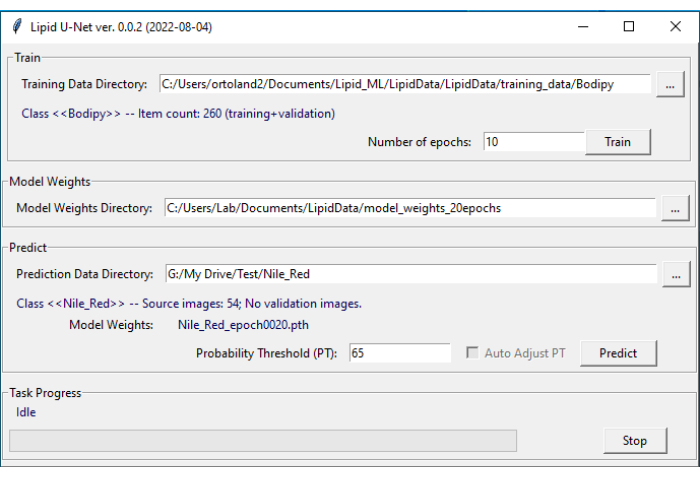
איור 4: ממשק המשתמש של LipidUNet. תוכנת LipidUNet כוללת מקטעים שונים לבחירה עבור ספריית נתוני האימון, שבהם תמונות של משקעי שומנים זוהו כראוי; מדריך משקולות המודל, המופק מנתוני האימונים; וספריית נתוני החיזוי שבה המשתמש יזין את התמונות שלו לצורך סגמנטציה. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
תוצאות
פרוטוקול זה מספק זרימת עבודה לזיהוי משקעי שומנים המוכתמים על ידי Nile Red, BODIPY ו- APOE. התוכנה שפותחה יכולה לזהות ולכמת באופן אוטומטי משקעי שומנים והביצועים הטובים ביותר כאשר הפרוטוקול המתואר ממוטב. כלולות דוגמאות של RPE ממוין בהצלחה (איור 3A) ו-RPE ממוין גרוע (איור 3B), מאחר שאיכות מודל התא משפיעה מאוד על איכות פילוח התמונה הנכון.
שניים מתוך שלושת הסמנים המתוארים בפרוטוקול, אדום הנילוס ו-BODIPY, מזוהים כנקודות מעגליות קטנות ובהירות באופן מובהק בתמונות פלואורסצנטיות (איור 5 ואיור 6). תמונה "חיובית" מהפרוטוקול תהיה זיהוי הולם של המרבצים השונים האלה (איור 5A-D ואיור 5E-H). תוצאה "שלילית" תציג פילוח שגוי של התמונה על-ידי חישוב שגוי של פלואורסצנטיות הרקע כפיקדון, בין אם בשל צביעה חלשה (איור 6A-C ואיור 6D-F) או בשל עוצמת רקע גבוהה (איור 6G-I).
למרבצי APOE יש מגוון גדלים וצורות, והם נראים יותר אליפטיים או לא סדירים מאשר המרבצים המעגליים של אדום הנילוס והבודיפי. פיקדונות אלה הם גם פחות מנוקבים, ועוצמת האות יכולה להיות שונה בין פיקדונות עקב שינויים בחדירות המדגם. זיהוי נכון יזהה כל מרבץ, כולל מרבצים פחות רוויים (איור 5I-L), בעוד שסגמנטציה שגויה לא תאסוף את המרבצים הללו (איור 6J-L). לכן, חשוב לייעל את שיטות הצביעה וההדמיה כדי למנוע שונות דרסטית. אחת הדרכים לעשות זאת היא על ידי מתן תשומת לב קפדנית לשלבי חדירת הדגימה תוך כדי חיסון. כדי למטב את האות הפלואורסצנטי, ניתן לבצע ליזה של תאים לפני קיבוע וצביעת מערכת החיסון עבור APOE, מה שמביא לרוויה אחידה ופילוח טוב יותר של מרבצי ה-APOE.
מסופקות גם תמונות מקוטעות של תאים שהתבגרו על פלטפורמת תרבית שאינה צלחת באר 96. תוכנת LipidUNet הורצה על תמונות של תאים שגודלו בתרבית על גבי טרנסוול, ובעוד משקעי השומנים הם סף, כך גם הנקבוביות בקרום הטרנסוול (איור 6M-O). בגלל הדמיון בצורה ובגודל, תוכנת LipidUNet בצורתה הנוכחית תסווה הן את משקעי השומנים והן את נקבוביות השקיפות ללא הבחנה.
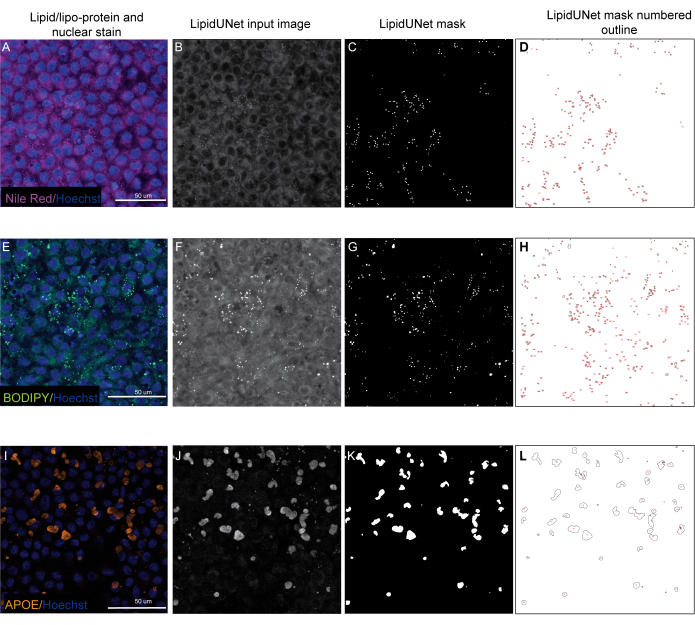
איור 5: תוצאות מייצגות. (A,E,I) RPE מצופה 96 היטב מוכתמים בצביעה גרעינית Hoechst (כחול) ואדום נילוס (מגנטה), BODIPY (ירוק) או APOE (כתום) והם הקרנות העוצמה המרבית של ערימת Z. (ב,ו,י) תמונות הקלט בגווני אפור עבור תוכנת LipidUNet לאחר עיבוד תמונה. (ג,ג,יא) מסכות שנוצרו על ידי LipidUNet, שבו כל הפיקדונות מזוהים כראוי. (ד,ח,ל) קווי המתאר של כל חלקיק מוסווה ממוספרים. תוויות אלה מאפשרות לחבר כל חלקיק בתמונה לערך ב- spreadheet עם הנתונים הגולמיים. (A-D) מראה כתמים בצבע אדום הנילוס, והתוכנה מסוגלת לזהות את המרבצים על רקע במדויק למרות אות חלש יותר. (E-H) מראה ניגוד חזק בין אות BODIPY לבין הרקע, שהוא אידיאלי. LipidUNet מזהה נכון כל הפקדה בתמונה. (I-L) מראה אות APOE חזק ומייצג את השונות של רוויית האות שנראית לעתים קרובות עם כתם זה. עם זאת, פילוח תמונות מסוגל לזהות את הגבולות של כל מרבץ APOE. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
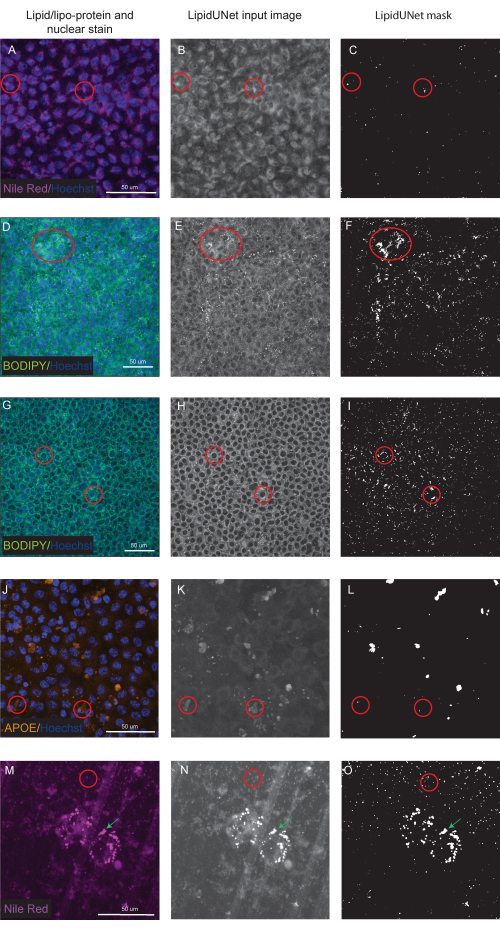
איור 6: תוצאות תת-אופטימליות. (A,D,G,J,M) RPE מצופה היטב 96 מוכתמים בצביעה גרעינית Hoechst (כחול) ואדום נילוס (מגנטה), BODIPY (ירוק) או APOE (כתום) והם הקרנות העוצמה המרבית של ערימת Z. (ב,ה,ח,ק,נ) תמונות הקלט בגווני אפור עבור תוכנת LipidUNet לאחר עיבוד תמונה. (ג,ו,י,ל,א) המסכות השגויות שנוצרו על ידי LipidUNet. עיגולים אדומים מציינים היכן התוכנה זיהתה באופן שגוי פיקדון שומנים. (א-ג) עיבוד Nile Red שגוי מכיוון שהתוכנה זיהתה את צביעת הרקע כפיקדון. זה יכול לקרות לעתים קרובות יותר כאשר יש רקע גבוה אבל מעט משקעי שומנים בתמונה. שתי דוגמאות לצביעת BODIPY מוצגות: תמונה באיכות ירודה עקב (D-F) צביעת BODIPY חלשה ו- (G - I) אות BODIPY חזק עם רקע גבוה. בשני המקרים, התוכנה אינה מסוגלת להבחין בין משקעי שומנים קטנים ועגולים לבין הטבעת המעגלית המקיפה את הגרעין. בעוד צביעה והדמיה צריך להיות אופטימיזציה כדי למנוע שגיאות אלה, הגרסה העדכנית ביותר של LipidUNet משופרת במידה רבה עבור תמונות אלה. (י-ל) פילוח APOE שגוי. מכיוון שהפיקדונות משתנים יותר בגודל וברוויה של האות, התוכנה מתקשה לזהות כמה פיקדונות. (מ-ו) RPE זרע על טרנסוול ומוכתם באדום הנילוס. פרוסה מערימת Z מוצגת כאן עם משקעי שומנים אדומים של הנילוס ונקבוביות טרנסוול. התוכנה אינה מסוגלת להבחין בין השניים, כפי שניתן לראות על ידי העיגול האדום המכיל נקבוביות טרנסוול והחץ הירוק המצביע על מרבצי הנילוס האדומים. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.

איור 7: השוואת כלי מסיכה. (A,B,C) RPE מצופה 96 היטב עם כמויות משתנות של שקיעת שומנים מזוהים עם אדום הנילוס (אדום). התמונות מוסוות באמצעות שלוש שיטות מסיכה נפוצות שונות, Find Maxima, Max Entropy ו-Renyi Entropy, ומושווים למסיכה שנוצרה על ידי LipidUNet. התמונה המקורית מלווה בספירה ידנית של משקעי השומנים, ואילו המסכות מציגות את הספירה החזויה בכל שיטת סגמנטציה. שיעור השגיאה הממוצע חושב עבור כל שיטת פילוח באמצעות הנוסחה הבאה: mean[(|ספירה חזויה - ספירה ידנית|/ספירה ידנית) x 100]. המסכה שנוצרה על-ידי LipidUNet מזהה באופן מדויק יותר משקעי שומנים בתמונות עם שיקוע משתנה בהשוואה לשיטות מיסוך אחרות (שיעורי שגיאה ממוצעים: 23% LipidUnet, 1164% Find Maxima, 851% Max Entropy, 203% Renyi Entropy). אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
| רכיב | מספר חתול | מניית קונק. | קונק סופי. | מ"ל |
| MEM אלפא | 12571-063 | נה | 500 | |
| תוספת N2 | 17502-048 | נה | 1% | 5 |
| FBS מנוטרל בחום | SH30071.03 | נה | 5% | 25 |
| NMEM NEAA | 11140-050 | 10 מ"מ | 0.01 מ"מ | 5 |
| נתרן פירובט | 11360-070 | 100mM | 1 מ"מ | 5 |
| פניצילין-סטרפטומיצין | 15140-122 | 10000u/מ"ל | 100U/מ"ל | 5 |
| טאורין | T4571 | 50 מ"ג/מ"ל | 250 מ"ג/מ"ל | 2.5 |
| הידרוקורטיזון | H6909 | 18.1 מ"ג/ליטר | 20 מ"ג/ליטר | 0.553 |
| T3 | T5516 | 20 מ"ג/ליטר | 0.013ug/L | 0.33 |
| נפח כולל, מ"ל | 548.383 | |||
טבלה 1: הרכב מגיב RPE-MM. רשימה של ריאגנטים וריכוזים אופטימליים עבור RPE-MM.
Discussion
פרוטוקול זה מספק שיטה לתיוג, תמונה וכימות יעילים של משקעי שומנים במודלים מונוגניים ופוליגניים של מחלות מבחנה עבור מחלות עיניים ניווניות. התוכנה מבוססת הבינה המלאכותית, LipidUNet, ניתנת ליישום על שלושה סמני שומנים נפוצים, APOE, Nile Red ו-BODIPY, ומספקת שיטה מהירה ואוטומטית לניתוח המאפשרת לכמת להיות סטנדרטית ולא מוטה.
המגבלה העיקרית של LipidUNet היא העובדה שמערך נתוני האימון עבור הבינה המלאכותית הוגבל לתמונות הגדלה פי 40 של תאים בתרבית בלוח של 96 בארות. כתוצאה מסט תמונות האימון, LipidUNet, בצורתו הנוכחית, מוגבל לניתוח תמונות הגדלה פי 40. התוכנה יכולה לשמש לניתוח תמונות 40x של תאים בתרבית על משטחי תרבית אחרים מלבד צלחת 96 באר, אך יש להקפיד לבחון את מסכות הפלט שנוצרו כדי לאמת סף מדויק על ידי התוכנה. יהיה צורך בערכות תמונות נוספות (בהגדלות שונות) כדי להרחיב את טווח הדגימות/תמונות שהוא יכול לנתח.
לפרוטוקול מספר שלבים קריטיים. בשלב סמן השומנים, על המשתמש לאשר כי תרכובת הסימון שנבחרה (BODIPY, APOE, Nile Red) תייגה את הדגימה שלו ביעילות. תאי RPE בוגרים הם לעתים קרובות פיגמנטים כבדים, אשר יכולים לפגוע באות הפלואורסצנטי של נוגדנים immunostaining. כאשר האות הפלואורסצנטי חלש או כאשר יש יותר מדי כתמי רקע, LipidUNet אינו יכול להבחין בטיפות שומנים במדויק. מסיבה דומה, יש להשתמש בהגדרות רכישה שנבחרו כראוי לשלב ההדמיה האוטומטית של הפרוטוקול. אם התמונות המתקבלות הן באיכות ירודה, LipidUNet יתקשה להסוות כראוי תמונות ולכן הכימות יהיה לא מדויק (איור 6A-L). לבסוף, לאחר עיבוד של התמונות הוא צעד חשוב, כמו LipidUNet יש דרישות ספציפיות עבור התוכנה לעבוד.
בהשוואה לתהליכי עבודה לניתוח שומנים המשתמשים בסף ידני, או טכניקות הכוללות סף אוטומטי בתוכנה כמו פיג'י, LipidUNet מציעה פילוח לא מוטה ואמין על פני תמונות עם שקיעת שומנים משתנה, כפי שמשתקף בשיעור שגיאות קטן בזיהוי חלקיקי שומנים (איור 7). התוכנה מאפשרת כניסת משתמש לתמונות הדרכה נוספות, ומאפשרת ניתוח של מערכי תמונות מעבר לאלו המשתמשות ביעד הגדלה של פי 40 או אפילו כאלה המשתמשות בטוש שומנים אחר, כמפורט בפרוטוקול. בעתיד, התוכנה תהיה מאומנת לנתח תמונות 3D כך כימות של נפח הפקדת שומנים אפשרי. מחלות עיניים ניווניות הכרוכות בשקיעת שומנים כתורמת עיקרית לפתולוגיה נפוצות, והמקרים צפויים לעלות ככל שאוכלוסיית הקשישים מתרחבת13. מודלים מדויקים של מחלות וכלי ניתוח יעילים, כפי שתיארנו בפרוטוקול זה, יאפשרו פיתוח התערבויות טיפוליות חדשניות.
Disclosures
אין גילויים.
Acknowledgements
אנו מודים לליבת ההיסטולוגיה של המכון הלאומי לעיניים (NEI) על השימוש במערכת הקונפוקלית Zeiss. עבודה זו נתמכה על ידי קרנות NEI IRP (מספר מענק ZIA EY000533-04).
Materials
| Name | Company | Catalog Number | Comments |
| 0.22 µm Steriflip filter system | EMD Millipore | SCGP00525 | |
| 1x Dulbecco's Phosphate Buffered Saline | Gibco | 14190-144 | |
| 3,3',5-Triiodo-L-thyronine | Sigma | T5516 | |
| Albumin Bovine, Fraction V | MP Biomedical | 160069 | |
| Alexa Fluor 555 rabbit anti-goat IgG (H+L) | Invitrogen | A21431 | APOE secondary antibody |
| APOE primary antibody | Millipore Sigma | AB947 | |
| BODIPY 493/503 | Invitrogen | D3922 | Protect from light |
| Complement competent human serum | Millipore Sigma | S1-LITER | |
| CTS N2 Supplement | Life Technologies | A13707-01 | |
| Fetal Bovine Serum | Hyclone | SH30071.03 | |
| Fluoromount-G | SouthernBiotech | 0100-01 | Slide mounting media |
| Glass Cover Slips #1 1/2 22 mm x 22 mm | Electron Microscopy Sciences | 72204-01 | |
| Glass Microscope Slide 25 mm x 75 mm- 1.2 mm Thick | Electron Microscopy Sciences | 71870-01 | |
| Hydrocortisone | Sigma | H0396 | |
| MEM Alpha | Life Technologies | 12571-063 | |
| MEM non-essential Amino Acids | Life Technologies | 11140 | |
| Nile Red | Sigma | 72485-100MG | Protect from light |
| Paraformaldehyde 16% Solution, EM Grade | Electron Microscopy Sciences | 15710 | |
| Penicillin-Strep | Life Technologies | 15140-148 | |
| Phosphate Buffered Saline 10x | Gibco | 70011-044 | |
| Rod Outer Segments (OS) | InVision Bioresources | 98740 | |
| Sodium bicarbonate | Sigma Aldrich | S5761 | |
| Sodium Pyruvate | Life Technologies | 11360-070 | |
| Sucrose | Sigma Aldrich | S1888 | |
| SYBR Green Master Mix | Bio-Rad | 1725274 | |
| Taurine | Sigma | T0625 | |
| Triton X-100 | Sigma | 9002-93-1 | |
| Tween 20 Ultrapure | Affymetrix | 9005-64-5 | |
| Vitronectin | Life Technologies | A14701SA | |
| Y-27632 dihydrochloride | R&D Systems | 1254 |
References
- Kanow, M. A., et al. Biochemical adaptations of the retina and retinal pigment epithelium support a metabolic ecosystem in the vertebrate eye. eLife. 6, e28899(2017).
- Adijanto, J., et al. The retinal pigment epithelium utilizes fatty acids for ketogenesis. The Journal of Biological Chemistry. 289 (30), 20570-20582 (2014).
- Farnoodian, M., et al. Cell-autonomous lipid-handling defects in Stargardt iPSC-derived retinal pigment epithelium cells. Stem Cell Reports. 17 (11), 2438-2450 (2022).
- Curcio, C. A. Soft drusen in age-related macular degeneration: Biology and targeting via the oil spill strategies. Investigative Ophthalmology & Visual Science. 59 (4), AMD160-AMD181 (2018).
- Cankova, Z., Huang, J. D., Kruth, H. S., Johnson, M. Passage of low-density lipoproteins through Bruch's membrane and choroid. Experimental Eye Research. 93 (6), 947-955 (2011).
- Curcio, C. A., et al. Esterified and unesterified cholesterol in drusen and basal deposits of eyes with age-related maculopathy. Experimental Eye Research. 81 (6), 731-741 (2005).
- Miyagishima, K. J., et al. AMPK modulation ameliorates dominant disease phenotypes of CTRP5 variant in retinal degeneration. Communications Biology. 4 (1), 1360(2021).
- Sharma, R., et al. Epithelial phenotype restoring drugs suppress macular degeneration phenotypes in an iPSC model. Nature Communications. 12 (1), 7293(2021).
- Farnoodian, M., et al. Cell-autonomous lipid-handling defects in Stargardt iPSC-derived retinal pigment epithelium cells. Stem Cell Reports. 17 (11), 2438-2450 (2022).
- Hallam, D., et al. An induced pluripotent stem cell patient specific model of complement factor H (Y402H) polymorphism displays characteristic features of age-related macular degeneration and indicates a beneficial role for UV light exposure. Stem Cells (Dayton, Ohio). 35 (11), 2305-2320 (2017).
- Sharma, R., Bose, D., Montford, J., Ortolan, D., Bharti, K. Triphasic developmentally guided protocol to generate retinal pigment epithelium from induced pluripotent stem cells. STAR Protocols. 3 (3), 101582(2022).
- Issa, P. C., Barnard, A. R., Herrmann, P., Washington, I., MacLaren, R. E. Rescue of the Stargardt phenotype in Abca4 knockout mice through inhibition of vitamin A dimerization. Proceedings of the National Academy of Sciences of the United States of America. 112 (27), 8415-8420 (2015).
- GBD 2019 Blindness and Vision Impairment Collaborators. Blindness and Vision Impairment Collaborators. Causes of blindness and vision impairment in 2020 and trends over 30 years, and prevalence of avoidable blindness in relation to VISION 2020: the Right to Sight: an analysis for the Global Burden of Disease Study. Lancet Global Health. 9 (2), e144-e160 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved