Method Article
LipidUNet-Machine-Learning-basierte Methode zur Charakterisierung und Quantifizierung von Lipidablagerungen unter Verwendung von iPSC-abgeleitetem retinalem Pigmentepithel
In diesem Artikel
Zusammenfassung
Degenerative Augenerkrankungen, die die retinale Pigmentepithelschicht des Auges betreffen, sind monogenen und polygenen Ursprungs. Mehrere Krankheitsmodelle und eine Softwareanwendung, LipidUNet, wurden entwickelt, um Krankheitsmechanismen sowie mögliche therapeutische Interventionen zu untersuchen.
Zusammenfassung
Das retinale Pigmentepithel (RPE) ist eine Monoschicht aus hexagonalen Zellen, die sich im hinteren Teil des Auges befindet. Es nährt und unterstützt Photorezeptoren und Aderhautkapillaren, führt eine Phagozytose der äußeren Photorezeptorsegmente (POS) durch und sezerniert Zytokine auf polarisierte Weise, um die Homöostase der äußeren Netzhaut aufrechtzuerhalten. Dysfunktionales RPE, das durch Mutationen, Alterung und Umweltfaktoren verursacht wird, führt zur Degeneration anderer Netzhautschichten und zum Verlust des Sehvermögens. Ein charakteristisches phänotypisches Merkmal degenerierender RPE sind intra- und subzelluläre lipidreiche Ablagerungen. Diese Ablagerungen sind ein häufiger Phänotyp bei verschiedenen degenerativen Erkrankungen der Netzhaut. Um den Lipiddepot-Phänotyp monogener Netzhautdegenerationen in vitro zu reproduzieren, wurde induziertes pluripotentes Stammzell-abgeleitetes RPE (iRPE) aus den Fibroblasten der Patienten erzeugt. Zelllinien, die von Patienten mit Stargardt und spät einsetzender Netzhautdegeneration (L-ORD) generiert wurden, wurden 7 Tage lang mit POS gefüttert, um die physiologische Funktion von RPE zu replizieren, die bei diesen Erkrankungen eine durch POS-Phagozytose induzierte Pathologie verursachte. Um ein Modell für die altersbedingte Makuladegeneration (AMD) zu entwickeln, eine polygene Erkrankung, die mit alternativer Komplementaktivierung assoziiert ist, wurde iRPE mit alternativen Komplement-Anaphylatoxinen konfrontiert. Die intra- und subzellulären Lipiddepots wurden mit Nilrot, Bor-Dipyrromethen (BODIPY) und Apolipoprotein E (APOE) charakterisiert. Um die Dichte von Lipidablagerungen zu quantifizieren, wurde eine auf maschinellem Lernen basierende Software, LipidUNet, entwickelt. Die Software wurde mit Projektionsbildern von iRPE auf Kulturoberflächen mit maximaler Intensität trainiert. In Zukunft soll es darauf trainiert werden, dreidimensionale (3D) Bilder zu analysieren und das Volumen von Lipidtröpfchen zu quantifizieren. Die LipidUNet-Software wird eine wertvolle Ressource für die Entdeckung von Medikamenten sein, die die Lipidakkumulation in Krankheitsmodellen verringern.
Einleitung
Das retinale Pigmentepithel (RPE) ist eine Monoschicht von Zellen, die sich im hinteren Teil des Auges neben retinalen Photorezeptoren befindet. RPE spielt eine wichtige Rolle bei der Aufrechterhaltung des richtigen Sehvermögens, indem es die Photorezeptoren metabolisch und strukturell unterstützt. Gesunde RPE-Zellen zeichnen sich durch eine ausgeprägte hexagonale Morphologie aus. Sie sind durch Tight Junctions verbunden, die es dem RPE ermöglichen, als Barriere zwischen der auf der Basalseite befindlichen Choriokapillaris und den apikal gelegenen Photorezeptoren zu fungieren. Um das Ökosystem der Netzhaut zu erhalten, transportiert RPE wichtige Metaboliten, z. B. Glukose, so zu den Photorezeptoren, dass der Glukoseverbrauch im RPEminimiert wird 1. Aufgrund dieser Einschränkung ist RPE auf andere Metaboliten angewiesen, um seinen Stoffwechselbedarf aufrechtzuerhalten, einschließlich Fettsäuren, die RPE durch β-Oxidation in Ketone umwandelt2. Angesichts der Neigung von RPE, Fettsäuren, die wahrscheinlich aus der Verdauung des äußeren Photorezeptorsegments (POS) recycelt werden, als Energiequelle zu nutzen, führen nachteilige Veränderungen der Lipidverarbeitungswege bei RPE häufig zu monogenen und polygenen degenerativen Netzhauterkrankungen oder sind daran beteiligt3.
Die altersbedingte Makuladegeneration (AMD), eine polygene degenerative Augenerkrankung, die eine RPE-Degeneration verursacht, wurde ebenfalls mit einer fehlerhaften Autophagie und einem abweichenden Fettstoffwechsel in der RPE-Monoschicht in Verbindung gebracht. Das Versagen einer dysfunktionalen RPE-Monoschicht, POS zu verarbeiten und andere kritische Funktionen auszuführen, führt zu extrazellulären (sub-RPE) Ablagerungen, die als basale lineare Ablagerungen (BLinD) bezeichnet werden und sich zwischen dem RPE und der Bruchschen Membran befinden - ein Kennzeichen von AMD-Pathologien. Zu den Hauptbestandteilen von BLinD gehören Lipoproteine, von denen das Apolipoprotein E (APOE)4 am häufigsten vorkommt. Die Anhäufung dünner BLinD-Schichten kann zu weichen Drusen führen, die als klinisches Symptom der AMD 5,6 anerkannt sind.
Mehrere Gruppen haben gezeigt, dass aus Stammzellen gewonnene In-vitro-Krankheitsmodelle, die eine RPE-Dysfunktion verursachen, eine Sub-RPE-Lipidakkumulation aufweisen 7,8,9. Hallam et al. (2017) generierten induziertes pluripotentes Stammzell-abgeleitetes RPE (iRPE) von Patienten mit einem hohen Risiko für AMD aufgrund eines Polymorphismus des CFH-Gens. Das iRPE zeigte eine Drusenakkumulation, wie sie durch APOE gekennzeichnet war, und das Hochrisiko-RPE akkumulierte größere Ablagerungen als das iRPE, das von Patienten mit niedrigem Risiko generiert wurde10.
Um ein In-vitro-Modell zu erstellen, das zelluläre Merkmale der AMD, wie z. B. Lipidtröpfchen und Drusenablagerungen, rekapituliert, wurden iRPE-Linien, die aus Blutproben von Patienten generiert wurden, unter Verwendung eines zuvor veröffentlichten entwicklungsgesteuerten Protokolls etabliert11. Die iRPE wurden komplementkompetentem humanem Serum (CC-HS) unterzogen, einer Lösung, die Anaphylatoxine enthält, die eine mögliche Ursache der AMD nachahmen: eine erhöhte Signalübertragung durch alternative Komplemente8. Die daraus resultierende zelluläre und subzelluläre Ablagerung von Lipidablagerungen wurde mit den gängigen Lipid- und Lipoproteinmarkern APOE, Nile Red und BODIPY gemessen. Anhand dieser Marker konnte gezeigt werden, dass die aktivierte Komplement-Signaltransduktion über CC-HS die Lipidakkumulation in iRPE-Zellen verstärkte8.
Um ein Krankheitsmodell für eine monogene degenerative Erkrankung der Netzhaut zu entwickeln, wurden iRPE-Linien von Patienten mit Morbus Stargardt entwickelt, einer Erkrankung, die durch Mutationen des ABCA4-Gens in RPE verursacht wird. Es wurde bereits gezeigt, dass sich A2E-Lipofuscin, eine intrazelluläre Ablagerung, von der bekannt ist, dass sie hohe Mengen an Phospholipiden und lichtabhängigen Lipidperoxidationsprodukten enthält, im RPE12 ansammelt, wenn ABCA4 ausgeschaltet wird. ABCA4-Knockout-Linien wurden parallel zu den Patientenlinien entwickelt, und beide wurden einer POS-Fütterung unterzogen. Das Stargardt iRPE zeigte eine POS-Phagozytose-induzierte Pathologie mit erhöhter Lipidakkumulation, die durch BODIPY-Färbung quantifiziert wurde. RPE, die aus ABCA4 KO iPSCs gewonnen wurden, wurden einer CC-HS-Behandlung unterzogen; Die Quantifizierung des BODIPY-Signals zeigte auch im Stargardt-Modell einen Defekt im Lipidhandling9.
Angesichts der Prävalenz dieser Krankheiten und des Bedarfs an wirksamen Therapeutika sowie den oben beschriebenen relevanten Krankheitsmodellen besteht die Notwendigkeit, robuste Methoden zur Quantifizierung der Wirksamkeit potenzieller Behandlungen zu etablieren. Um Lipidablagerungen objektiv, automatisiert und standardisiert zu quantifizieren, wurde eine auf maschinellem Lernen basierende Software, LipidUNet, entwickelt, so dass in Kombination mit Maskenanalysewerkzeugen die Lipidablagerung mithilfe der gängigen Marker Nile Red, BODIPY und APOE schnell und effektiv identifiziert werden kann. Die mit dieser Analysepipeline erhaltenen zusammenfassenden Statistiken können dann analysiert und grafisch dargestellt werden, was einen einfachen Vergleich der Behandlungsbedingungen ermöglicht. Der Schaltplan des Protokolls ist in Abbildung 1 dargestellt.
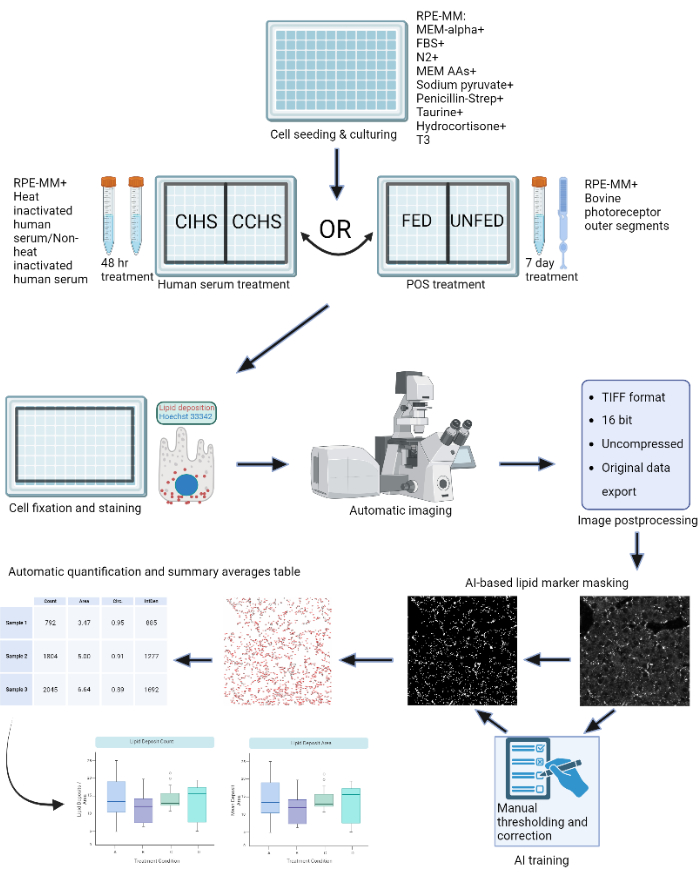
Abbildung 1: Schematische Darstellung des Protokolls: RPE-Zellen werden auf einer 96-Well-Platte gezüchtet und mit aktivem humanem Serum oder gereinigten Rinderaußensegmenten herausgefordert, um verschiedene Arten von Netzhautdegenerationen in vitro zu modellieren. RPE-Zellen werden fixiert und für Lipoproteinablagerungen mit Nilrot, BODIPY und APOE gefärbt. Ein konfokales Mikroskop wird verwendet, um Z-Stapel von fluoreszenzmarkierten Lipidpartikeln zu erfassen, die anschließend zu 2D-Projektionen mit maximaler Intensität verarbeitet werden. Ein Machine-Learning-Algorithmus wurde darauf trainiert, Lipoproteinpartikel zu erkennen und korrekt zu segmentieren. Zusammenfassende Tabellen mit Partikelanzahl und verschiedenen Formmetriken werden generiert und können für nachfolgende Diagramme und statistische Analysen verwendet werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Protokoll
Alle Protokollschritte entsprechen den Richtlinien der Ethikkommission für Humanforschung der NIH. Die Arbeit mit Stammzellen und die Entnahme von Patientenproben wurden vom Combined NeuroScience Institutional Review Board (CNS IRB) unter dem Office of Human Research Protection (OHRP), NIH, gemäß den Richtlinien der US-Regierung gemäß 45 CFR 46 genehmigt. Die Patientenproben wurden unter Verwendung einer von der CNS IRB genehmigten Einverständniserklärung gemäß den Kriterien der Deklaration von Helsinki unter der Protokollnummer NCT01432847 (https://clinicaltrials.gov/ct2/show/NCT01432847?cond=NCT01432847&draw=2&rank=1) entnommen.
1. iRPE-Generierung
- Generieren Sie iRPE aus aus dem Blut von Patienten stammenden iPS-Zellen gemäß dem veröffentlichten Protokoll von Sharma et al., 202211 (Abbildung 2 und Abbildung 3).
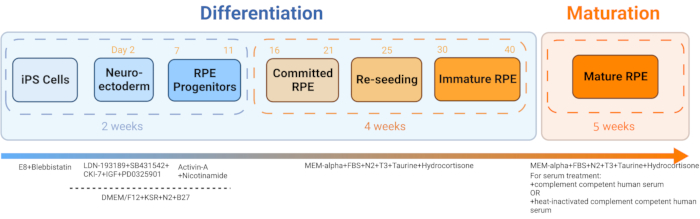
Abbildung 2: Schematische Darstellung der Differenzierung und Reifung von iRPE. Um iRPE zu erzeugen, wurde ein etabliertes Differenzierungsprotokoll befolgt, und die Zellen wurden 5 Wochen lang reifen gelassen. Die resultierende Zellkultur fungiert als In-vitro-Modell , das mit verschiedenen Behandlungen manipuliert werden kann, um eine RPE-Dysfunktion bei Krankheiten wie AMD und Morbus Stargardt nachzuahmen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
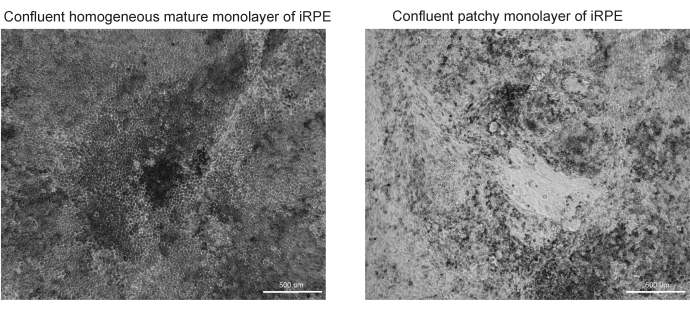
Abbildung 3: Repräsentative Bilder erfolgreicher und erfolgloser RPE-Differenzierung und -Reifung. Zwei Hellfeldbilder mit 10-facher Vergrößerung von TJP1 RPE sind an Tag 42 des iRPE-Protokolls zu sehen. (A) Erfolgreiche Differenzierung und Reifung zeigen konfluierendes RPE mit Pigmentierung und polygonaler Morphologie. (B) Eine erfolglose Differenzierung und Reifung zeigt Cluster von absterbenden Zellen, wie hier gezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
2. Vorbereitung der RPE-Wartungsmedien (RPE-MM)
- Tauen Sie das N2-Präparat über Nacht bei 4 °C auf. Tauen Sie alle anderen Reagenzien bei Raumtemperatur (RT) auf.
- Unter sterilen Bedingungen werden die in Tabelle 1 aufgeführten Reagenzien mit den aufgeführten Verdünnungsfaktoren gemäß dem von Sharma et al., 202211, festgelegten Protokoll hinzugefügt.
- Mischen Sie das Medium gut und filtrieren Sie es mit einer 0,22-μm-Filtereinheit.
HINWEIS: Das Medium ist für den Gebrauch innerhalb von 2 Wochen geeignet, wenn es bei 4 °C gelagert wird.
3. Aussaat von 96-Well-Platten
- Tauen Sie einen aliquoten Teil des Vitronektins bei RT für 3-5 Minuten auf oder bis das Eis vollständig geschmolzen ist.
- Verdünnen Sie das Vitronektin mit 1x phosphatgepufferter Kochsalzlösung (DPBS) von Dulbecco, um die gewünschte Arbeitslösung in einer Verdünnung von 1:200 (Vitronektin: DPBS) zu erhalten. Bei einer 96-Well-Platte wird jede Well mit 200 μl der Arbeitslösung beschichtet.
- Den aufgetauten ROCK-Inhibitor (Y-27632-Dihydrochlorid) mit dem RPE-MM in einer Verdünnung von 1:1000 kombinieren, um eine Endkonzentration von 10 μM zu erreichen. Dies ist das Beschichtungsmedium für RPE-Zellen.
- Tauen Sie das iRPE-Fläschchen mit einem automatisierten Zellauftausystem auf und übertragen Sie die iRPE-Zellsuspension in ein 50-ml-Röhrchen.
- Verdünnen Sie die Zellsuspension mit dem Beschichtungsmedium in einer Verdünnung von 1:10. Zentrifugieren Sie das Röhrchen bei 400 x g für 5 Minuten.
- Saugen Sie den Überstand vorsichtig ab und resuspendieren Sie die Zellen in 10 ml des Beschichtungsmediums.
- Mischen Sie 400 μl des Beschichtungsmediums mit 100 μl der resuspendierten Zelllösung für die Zellzählung. Verwenden Sie dieses Aliquot, um die lebensfähige Zellkonzentration der Zellsuspension mit einem Zellviabilitätszähler zu bestimmen.
- Verdünnen Sie die Zellsuspension mit dem Beschichtungsmedium auf eine Endkonzentration von 60.000 Zellen/ml.
- Saugen Sie die Vitronektin-Beschichtungslösung vollständig von der 96-Well-Platte ab und geben Sie 200 μl der Zellsuspension in jede Well. Es wird ungefähr 12.000 Zellen/Well oder ~200 Zellen/mm2 geben.
- Die ausgesäten Zellplatten werden 48 h bei 37 °C und 5 % CO2 inkubiert. Nach 48 h ist das Medium auf RPE-MM ohne ROCK-Inhibitor-Supplementierung umzustellen. Wechseln Sie das Medium während der 5-wöchigen Reifezeit alle 2-3 Tage.
4. In-vitro-Krankheitsmodelle
- Ergänzen Sie die kompetente Behandlung mit humanem Serum (CC-HS)
- Tauen Sie das humane Komplement-kompetente Serum über Nacht bei 4 °C auf.
- Bereiten Sie CC-HS vor und ergänzen Sie inkompetente Humanserummedien (CI-HS).
- Um 5%ige CC-HS-Medien herzustellen, mischen Sie das aufgetaute komplementkompetente Humanserum mit RPE-MM in einer Verdünnung von 1:20. Filtern Sie die Lösung vor Gebrauch durch einen 0,22-μm-Medienfilter.
- Zur Herstellung von 5%igen komplementinkompetenten Humanserummedien (CI-HS) wird das CC-HS zunächst 30 min lang in einem 57 °C warmen Wasserbad erhitzt und dann mit dem Nährmedium in einer Verdünnung von 1:20 vermischt. Filtern Sie die Lösung vor Gebrauch durch einen 0,22-μm-Medienfilter.
- Das Serum behandelt die Zellen mit 200 μl entweder 5 % CC-HS oder 5 % CI-HS für eine Gesamtinkubationszeit von 48 h und frischt das Medium nach 24 h auf.
- Waschen Sie die Zellen mit 1x DPBS und fixieren Sie sie mit 4% Paraformaldehyd für 20 min bei RT. Waschen Sie sie noch einmal mit 1x DPBS und lagern Sie die Proben bei 4 °C, eingetaucht in 200 μL DPBS.
- OPTIONAL: Falls gewünscht, lysieren Sie die Zellen von der Platte, um nur eine Sub-RPE-Lipidablagerung zu zeigen.
- Um die Zellen zu lysieren und nur Lipidablagerungen zu hinterlassen, entfernen Sie das Medium und geben Sie 200 μl deionisiertes Wasser in jede Vertiefung.
- 10-15 Minuten inkubieren, auf und ab pipettieren, bis die Zellen entfernt sind. Waschen Sie noch einmal mit 200 μl deionisiertem Wasser und fixieren Sie die Zellen sofort mit 4% Paraformaldehyd.
- Bestätigen Sie die Wirksamkeit der Zellentfernung mit einer Kernfärbung mit Hoechst. Hoechst wird in einer Verdünnung von 1:2000 zu einer 1x DPBS-Lösung hinzugefügt, die 1 % Rinderserumalbumin (BSA), 0,5 % Tween 20 und 0,5 % Triton-X 100 enthält. Bei RT 1 h im Dunkeln inkubieren. Anschließend mit 1x DPBS waschen.
- Behandlung des äußeren Segments des Photorezeptors (POS) auf iRPE
- POS-Vorbereitung
- Nehmen Sie das POS-Pelletrohr aus dem -80 °C-Lager und tauen Sie es über Nacht bei 4 °C in einem abgedeckten Eiskübel auf.
- Bereiten Sie den POS-Waschpuffer vor, indem Sie 10 g Saccharose in 40 ml doppelt deionisiertemH2O(ddH2O) mischen.
- Mischung bei 40-50 °C unter leichtem Rühren 15 min erhitzen. Fügen Sie der Mischung 840 mg Natriumbicarbonat hinzu und rühren Sie unter Rühren 10 Minuten lang.
- Stellen Sie das Gesamtvolumen des POS-Waschpuffers mit ddH2O auf 100 mL ein und stellen Sie den pH-Wert der Lösung mit 1 N HCl oder 1 N NaOH je nach Bedarf auf 8,3 ein. Filtern Sie die Waschlösung mit einem 0,22 μm Filter.
HINWEIS: Das Protokoll kann hier pausiert werden. der POS-Waschpuffer kann über Nacht bei 4 °C gelagert werden. - Nach dem Auftauen das Pellet in 15 ml POS-Waschpuffer suspendieren. Seien Sie bei der Pelletsuspension sanft, um die Integrität des POS zu gewährleisten. Die POS-Suspension bei 600 x g bei 4 °C für 20 min zentrifugieren und anschließend den Überstand absaugen.
- Resuspendieren Sie das POS-Pellet in 10 ml des POS-Waschpuffers.
- Entfernen Sie ein 100 μL Aliquot des POS + POS Waschpuffers (POS-Lösung) und verdünnen Sie es in 400 μL 1x DPBS. Verteilen Sie 50 μl der verdünnten POS-Lösung auf einer Blutagarplatte und einer Agaroseplatte, um sie auf bakterielle und pilzliche Verunreinigungen zu überprüfen. Bereiten Sie jeweils Positivkontrollen vor und inkubieren Sie alle Platten 48 h lang bei 37 °C.
- Führen Sie einen qPCR-Assay durch, indem Sie 1 μl POS-Lösung in die Nachweisvertiefung geben, um auf Mykoplasmen zu testen. Um DNA-Fragmente zu vervielfältigen, führen Sie 40 Zyklen Denaturierung (95 °C, 15 s) sowie Glühen und Elongieren (60 °C, 1 min) durch. Die Vorwärts- und Rückwärtsprimer für den Nachweis von Mykoplasmen in der POS-Probe lauten wie folgt:
Vorwärts-Grundierung: GGA TTA GAT ACC CTG GTA GTC CAC G
Reverse primer: CGT CAA TTC CTT TAA GTT TCA CTC TTG GC - Messen Sie die POS-Konzentration mit einem Zellanalysator und aliquotieren Sie sie nach Bedarf. Für ein Well mit 96-Well-Platten mit RPE-Zellen sind 3 x 106 POS ausreichend. Das gewünschte Verhältnis beträgt 10 POS/RPE-Zellen. Lagern Sie die Aliquots für die spätere Verwendung bei 80 °C.
- Hinzufügen von POS zu Zellen
- Fläschchen mit POS in einem Eisbad auftauen.
- Mischen Sie die errechnete Menge des vorbereiteten POS mit RPE-MM und behandeln Sie die Zellen 7 Tage lang einmal täglich mit POS.
Anmerkungen: Bereiten Sie die POS-Lösung täglich frisch zu. - Waschen Sie die Zellen mit 1x DPBS und fixieren Sie sie dann mit 4% Paraformaldehyd für 20 min bei RT. Waschen Sie sie erneut mit DPBS und lagern Sie die Proben bei 4 °C, eingetaucht in 200 μL DPBS.
- POS-Vorbereitung
5. Färbung für Sub-RPE-Ablagerungen
- Nilrot-Färbeprotokoll
- Nach der PFA-Fixierung waschen Sie die Proben 3 Mal mit 1x DPBS.
HINWEIS: Wenn das Protokoll nicht sofort verwendet wird, kann es hier pausiert werden, aber die Proben müssen in einer 1x DPBS + 0,02%igen Natriumazidlösung bei 4 °C gelagert werden. - Um die Nilrot-Stammlösung herzustellen, lösen Sie Nilrot-Pulver in Aceton mit einer Konzentration von 3 mg/ml auf. 15 min bei RT unter regelmäßigem Mischen inkubieren. Filtern Sie die Lösungen mit einem 0,22-μm-Filter ein- oder zweimal, je nachdem, wie viel Niederschlag in der Lösung verbleibt.
Anmerkungen: Schützen Sie die Stammlösung vor Licht. - Um die Arbeitslösung herzustellen, verdünnen Sie die Stammlösung im Verhältnis 1:500 in 1x DPBS. 200 μl der Arbeitslösung werden der Probe für 30 min bei RT auf einem Schüttler zugegeben und vor Licht geschützt.
- 3 mal mit 1x PBS waschen und die Proben bei 4 °C lagern, eingetaucht in 200 μL DPBS.
HINWEIS: Wenn Sie ein Experiment mit Transwells anstelle einer 96-Well-Platte durchführen, können die Proben auf einen Objektträger mit Eindeckmedium montiert, mit einem Glasdeckglas abgedeckt und mit transparentem Nagellack versiegelt werden. Es sollte darauf geachtet werden, dass die Probe mit den Zellen nach oben montiert wird.
- Nach der PFA-Fixierung waschen Sie die Proben 3 Mal mit 1x DPBS.
- BODIPY-Färbeprotokoll
- Für die Stammlösung wird BODIPY in wasserfreiem Dimethylsulfoxid (DMSO) gelöst, um eine Stammkonzentration von 3,8 mM zu erreichen.
- Bei fixierten PFA-Proben BODIPY-Material im Verhältnis 1:300 in 1x DPBS verdünnen. Fügen Sie den Zellen 200 μL hinzu und inkubieren Sie sie über Nacht auf einer Wippe bei RT.
- 3 mal mit 1x DPBS waschen und die Proben bei 4 °C lagern, eingetaucht in 200 μL DPBS.
HINWEIS: Wenn Sie ein Experiment mit Transwells anstelle einer 96-Well-Platte durchführen, können die Proben auf einen Objektträger mit Eindeckmedium montiert, mit einem Glasdeckglas abgedeckt und mit transparentem Nagellack versiegelt werden. Es sollte darauf geachtet werden, dass die Probe mit den Zellen nach oben montiert wird.
- APOE-Immunfärbungsprotokoll
- Kombinieren Sie 1x DPBS mit 1 % Rinderserumalbumin (BSA), 0,5 % Tween 20 und 0,5 % Triton-X 100, um eine Pufferlösung zu erhalten.
- Bei PFA-fixierten Proben wird die Probe blockiert und in 200 μl der Pufferlösung für 1 h bei RT permeabilisiert.
- APOE-Primärantikörper im Verhältnis 1:100 verdünnt in die Pufferlösung geben und über Nacht bei RT inkubieren.
- Waschen Sie die Proben am nächsten Tag 3 Mal mit 1x DPBS.
- Geben Sie einen sekundären Antikörper in 1:1000 Verdünnung in die Pufferlösung und geben Sie 200 μL der Lösung für 1 h bei RT in die Zellen.
- 3 Mal mit 1x DPBS waschen und die Proben bei 4 °C lagern, eingetaucht in 200 μl DPBS.
HINWEIS: Wenn Sie ein Experiment an Transwells anstelle einer 96-Well-Platte durchführen, können die Proben auf einen Objektträger mit Eindeckmedium (Fluoromount) montiert, mit einem Glasdeckglas abgedeckt und mit transparentem Nagellack versiegelt werden. Es sollte darauf geachtet werden, dass die Probe mit den Zellen nach oben montiert wird.
6. Bildautomatisierung und -verarbeitung
- Automatisierter Bildscan
HINWEIS: In dieser Studie wurden das inverse konfokale Rastermikroskop Zeiss LSM 800 und die Software ZEN 3.2 (blue edition) verwendet. Stellen Sie sicher, dass die 96-Well-Platte vor der Bildgebung mindestens 60 Minuten lang auf RT erwärmt wird, um eine Drift der Brennebene während des Scans aufgrund einer Änderung des Brechungsindex des Mediums bei Temperaturänderung zu vermeiden.- Erstellen Sie mit einem konfokalen Mikroskop und einem 40x-Objektiv ein Scanprofil mit den entsprechenden Fluoreszenzkanälen für den verwendeten Lipidmarker und eventuelle zusätzliche Antikörper.
- Verwenden Sie das Kontrollkästchen Kacheln , um die Bildautomatisierung einzurichten. Um die 96-Well-Platte zu kalibrieren, stellen Sie sicher, dass die richtigen Probenträgermessungen eingegeben und ausgewählt wurden. Klicken Sie dann auf die Schaltfläche Kalibrieren , um die Platte gemäß den Anweisungen zu kalibrieren, was die Verwendung des 10x-Objektivs erfordert.
- Wählen Sie die Ansicht "Erweiterte Einstellungen", um geeignete Vertiefungspunkte auszuwählen, und fügen Sie mit der Funktion "Positionen" 3 verschiedene Abbildungspunkte in der Nähe der Mitte des Vertiefungsbereichs hinzu. Dies kann manuell auf der Unterregisterkarte "Position" oder nach dem Zufallsprinzip auf der Registerkarte "Position Setup" und der Auswahl von "Setup by Carrier" erfolgen. Wiederholen Sie dies für alle Vertiefungen mit derselben Färbung.
- Um eine optimale Fokussierung und Z-Stapel-Positionierung während der Automatisierung zu erzielen, wechseln Sie zur Registerkarte Fokusstrategie , um Fokusoberfläche/Z-Werte verwenden auszuwählen, die durch Kachel-Setup definiert sind. Alternative Methoden können andere Fokusstrategien verwenden, es wird jedoch empfohlen, diese Einstellung zu verwenden, um die konsistentesten Ergebnisse zu erzielen.
- Klicken Sie auf der Registerkarte Kacheln auf Positionen überprüfen , und legen Sie die zentrale Z-Ebene für jede Position manuell fest. Die Einstellungen auf der Unterregisterkarte Optionen ordnen die Aufnahme von Bildern, also überprüfen Sie dies, bevor Sie mit der Aufnahme beginnen. Um Bilder in der Reihenfolge aufzunehmen, in der die Positionen ausgewählt wurden, deaktivieren Sie die Kontrollkästchen " Kachelbereiche/Positionen " und "Trägermulden/Container ". Wählen Sie Szenen in separate Dateien aufteilen , um die Bildverarbeitung zu vereinfachen.
- Stellen Sie sicher, dass die Registerkarte " Z-Stack " auf " Mitte" eingestellt ist, ein Bereich in die Einstellungen des Benutzers eingegeben wird und die Schaltfläche "Optimal " ausgewählt ist, um das Slice-Intervall festzulegen.
- Nachdem Sie die Registerkarten "Erfassungsmodus", "Kanäle", "Fokusstrategie", "Z-Stapel" und " Kacheln" optimiert haben, starten Sie das Experiment.
- Bildverarbeitung
- Erstellen Sie mithilfe einer Batch-Bildverarbeitungsmethode maximale Projektionen jedes Z-Stapels mit der Methode " Erweiterte Tiefenschärfe ".
- Exportieren Sie mithilfe einer Batch-Bildverarbeitungsmethode die maximalen Projektionsdateien als 16-Bit-TIFF-Bilder. Legen Sie die Komprimierung auf " Keine " fest, und stellen Sie sicher, dass "Originaldaten " aktiviert ist. Das resultierende Bild sollte ein Maximum-Projektions-Graustufen-TIFF sein, das nur den Fluoreszenzkanal zeigt, auf dem der Lipidmarker exprimiert wird.
7. Segmentierung und Quantifizierung
HINWEIS: Das LipidUNet-Programm wurde mit 40-fachen Bildern von einer 96-Well-Platte trainiert. Es wird dringend empfohlen, Bilder zu verwenden, die mit einem 40x-Objektiv aufgenommen wurden.
- Installieren Sie die LipidUNet-Software. LipidUNet kann aus dem folgenden GitHub-Repository heruntergeladen werden: https://github.com/RPEGoogleMap/LipidUNet
- Identifizieren Sie die TIFF-Bilder, die entweder Nile Red, Bodipy oder APOE darstellen, und verschieben Sie sie in einen Ordner mit dem Namen imgs in einem Verzeichnis mit dem Namen Nile_Red, Bodipy oder APOE, je nach verwendeter Methode.
HINWEIS: Für das LipidUNet-Programm müssen exakte Namenskonventionen verwendet werden, um die Verzeichnisse zu erkennen. - Öffnen Sie die LipidUNet-Software (Abbildung 4).
- Wählen Sie auf der Registerkarte Predict der Software das entsprechende Verzeichnis (Nile_Red, Bodipy oder APOE) aus, indem Sie auf die Auslassungspunkte klicken und zum benannten Verzeichnis navigieren. Vergewissern Sie sich, dass das LipidUNet-Programm die Bilder korrekt identifiziert hat, indem Sie den Klasseneintrag überprüfen.
- Wählen Sie einen Wahrscheinlichkeitsschwellenwert für den Algorithmus zwischen 0,01 und 0,99 aus. Ein höherer Wert eliminiert mehr falsch positive Ergebnisse, kann aber zu mehr falsch negativen Ergebnissen führen, und niedrigere Werte können zu mehr falsch positiven Ergebnissen führen, während mehr falsch negative Ergebnisse eliminiert werden. Ein Wert von 0,65 ist der Standardwert und wird empfohlen.
- Klicken Sie auf Vorhersage.
HINWEIS: Die Software durchläuft automatisch alle Bilder und erstellt einen neuen Ordner mit dem Namen predicted_masks im ausgewählten Verzeichnis. - Verwenden Sie ein Maskenanalysewerkzeug, um die generierten Masken zu durchlaufen und eine quantitative Anzahl der Lipidablagerungen mit Schwellenwert aus den Maskenbildern bereitzustellen.
- Analysieren Sie die generierten Zähldaten, um die Behandlungsbedingungen zu vergleichen.
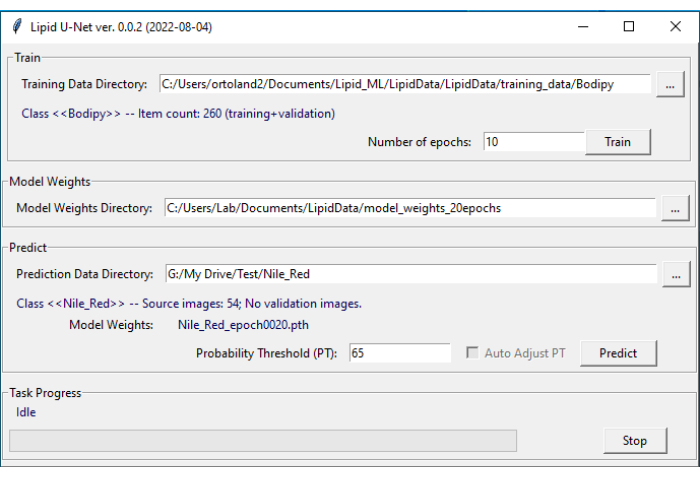
Abbildung 4: LipidUNet-Benutzeroberfläche. Die LipidUNet-Software verfügt über verschiedene Abschnitte, die für das Trainingsdatenverzeichnis ausgewählt werden können, in denen Bilder von Lipidablagerungen korrekt identifiziert wurden. das Modellgewichtungsverzeichnis, das aus den Trainingsdaten erstellt wird. und das Vorhersagedatenverzeichnis, in das der Benutzer seine Bilder zur Segmentierung eingibt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Ergebnisse
Dieses Protokoll bietet einen Arbeitsablauf zur Identifizierung von Lipidablagerungen, die mit Nilrot, BODIPY und APOE gefärbt wurden. Die entwickelte Software kann Lipidablagerungen automatisch identifizieren und quantifizieren und funktioniert am besten, wenn das skizzierte Protokoll optimiert ist. Enthalten sind Beispiele für erfolgreich differenzierte RPE (Abbildung 3A) und schlecht differenzierte RPE (Abbildung 3B), da die Qualität des Zellmodells einen großen Einfluss auf die Qualität der korrekten Bildsegmentierung hat.
Zwei der drei im Protokoll beschriebenen Marker, Nile Red und BODIPY, werden als kleine kreisförmige Punkte identifiziert, die in fluoreszierenden Bildern deutlich hell sind (Abbildung 5 und Abbildung 6). Ein "positives" Bild aus dem Protokoll wäre eine geeignete Identifizierung dieser unterschiedlichen Lagerstätten (Abbildung 5A-D und Abbildung 5E-H). Ein "negatives" Ergebnis würde eine falsche Segmentierung des Bildes zeigen, indem die Hintergrundfluoreszenz als Ablagerung verwechselt wird, entweder aufgrund einer schwachen Färbung (Abbildung 6A-C und Abbildung 6D-F) oder aufgrund einer hohen Hintergrundintensität (Abbildung 6G-I).
APOE-Ablagerungen haben eine Vielzahl von Größen und Formen und erscheinen eher oval oder unregelmäßig als die kreisförmigen Ablagerungen von Nilrot und BODIPY. Diese Ablagerungen sind auch weniger punktförmig, und die Signalintensität kann zwischen den Ablagerungen aufgrund von Schwankungen in der Permeabilisierung der Probe variieren. Eine korrekte Identifizierung identifiziert jede Lagerstätte, einschließlich derjenigen, die weniger gesättigt sind (Abbildung 5I-L), während eine falsche Segmentierung diese Lagerstätten nicht erfasst (Abbildung 6J-L). Daher ist es wichtig, die Färbe- und Bildgebungsmethoden zu optimieren, um drastische Abweichungen zu vermeiden. Eine Möglichkeit, dies zu tun, besteht darin, bei der Immunfärbung sorgfältig auf die Schritte der Probenpermeabilisierung zu achten. Um das Fluoreszenzsignal zu optimieren, können die Zellen vor der Fixierung und Immunfärbung für APOE lysiert werden, was zu einer gleichmäßigen Sättigung und besseren Segmentierung der APOE-Ablagerungen führt.
Bereitgestellt werden auch segmentierte Bilder von Zellen, die auf einer anderen Kulturplattform als einer 96-Well-Platte gereift sind. Die LipidUNet-Software wurde mit Bildern von Zellen ausgeführt, die auf einem Transwell kultiviert wurden, und während die Lipidablagerungen einen Schwellenwert aufweisen, sind dies auch die Poren in der Transwell-Membran (Abbildung 6M-O). Aufgrund der Ähnlichkeit in Form und Größe maskiert die LipidUNet-Software in ihrer jetzigen Form wahllos sowohl die Lipidablagerungen als auch die Transwellporen.
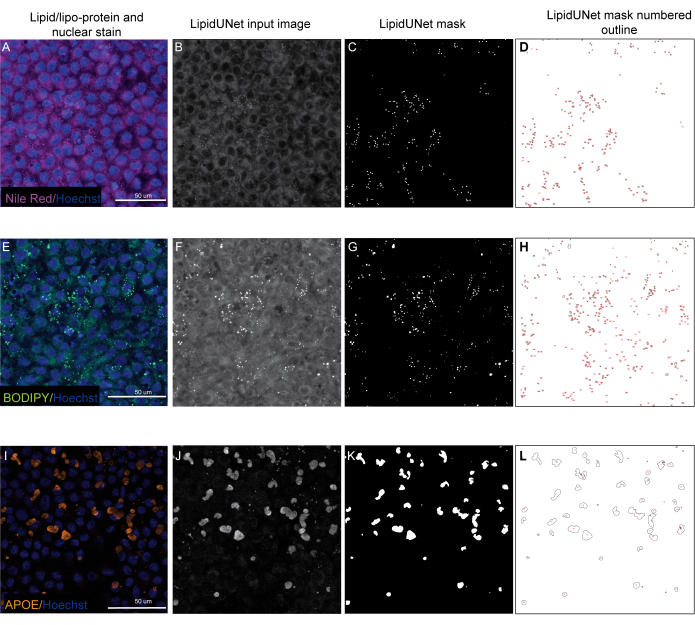
Abbildung 5: Repräsentative Ergebnisse. (A,E,I) 96-Well-plattierte RPE werden mit Hoechst-Kernfärbung (blau) und entweder Nilrot (Magenta), BODIPY (grün) oder APOE (orange) gefärbt und sind die Projektionen der maximalen Intensität eines Z-Stapels. (B,F,J) Die Graustufen-Eingabebilder für die LipidUNet-Software nach der Bildverarbeitung. (C,G,K) Von LipidUNet generierte Masken, bei denen alle Ablagerungen korrekt identifiziert werden. (D,H,L) Die Umrisse der einzelnen maskierten Partikel sind nummeriert. Diese Beschriftungen ermöglichen es, jedes Partikel im Bild mit einem Eintrag im Spreadheet mit den Rohdaten zu verbinden. (A-D) zeigt eine Nilrot-Färbung, und die Software ist in der Lage, die Ablagerungen vor dem Hintergrund trotz eines schwächeren Signals genau zu erkennen. (E-H) zeigt einen starken Kontrast zwischen dem BODIPY-Signal und dem Hintergrund, was ideal ist. LipidUNet identifiziert jede Ablagerung im Bild korrekt. (I-L) zeigt ein starkes APOE-Signal und repräsentiert die Variabilität der Signalsättigung, die bei dieser Färbung häufig zu beobachten ist. Nichtsdestotrotz ist die Bildsegmentierung in der Lage, die Grenzen jeder APOE-Lagerstätte zu identifizieren. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
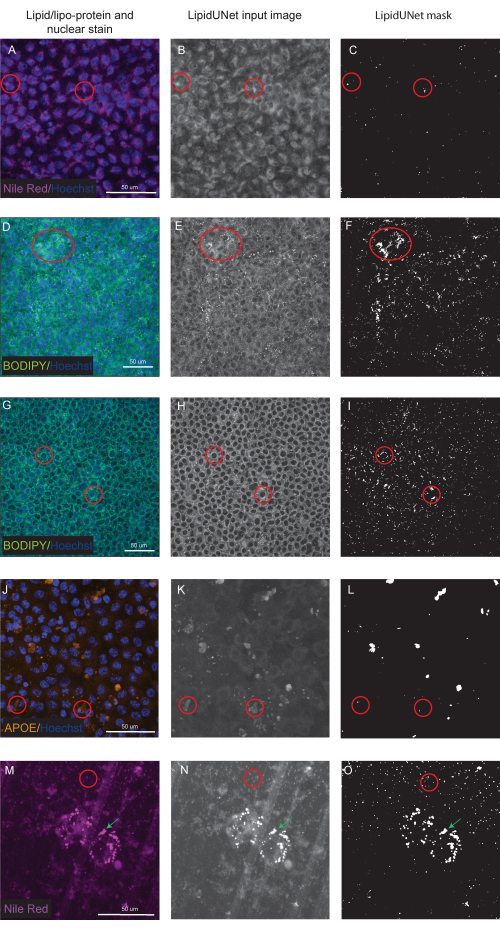
Abbildung 6: Suboptimale Ergebnisse. (A,D,G,J,M) 96-gut plattierte RPE werden mit Hoechst-Kernfärbung (blau) und entweder Nilrot (Magenta), BODIPY (grün) oder APOE (orange) gefärbt und sind die maximalen Intensitätsprojektionen eines Z-Stapels. (B,E,H,K,N) Die Graustufen-Eingabebilder für die LipidUNet-Software nach der Bildverarbeitung. (C,F,I,L,O) Die falschen Masken, die von LipidUNet generiert wurden. Rote Kreise zeigen an, wo die Software eine Lipidablagerung falsch erkannt hat. (A-C) Die Verarbeitung von Nile Red ist fehlerhaft, da die Software die Hintergrundfärbung als Ablagerung identifiziert hat. Dies kann häufiger passieren, wenn ein hoher Hintergrund, aber nur wenige Lipidablagerungen im Bild vorhanden sind. Es werden zwei Beispiele für BODIPY-Färbung gezeigt: eine schlechte Bildqualität aufgrund einer (D-F) schwachen BODIPY-Färbung und (G - I) ein starkes BODIPY-Signal mit hohem Hintergrund. In beiden Fällen ist die Software nicht in der Lage, kleine, kreisförmige Lipidablagerungen von dem kreisförmigen Ring im Hintergrund zu unterscheiden, der den Zellkern umgibt. Während die Färbung und Bildgebung optimiert werden sollten, um diese Fehler zu vermeiden, ist die neueste Version von LipidUNet für diese Bilder weitgehend verbessert. (J-L) Falsche APOE-Segmentierung. Da die Ablagerungen in Größe und Signalsättigung variabler sind, hat die Software Schwierigkeiten, einige Ablagerungen zu erkennen. (M-O) RPE auf ein Transwell gesät und mit Nilrot gefärbt. Hier ist ein Ausschnitt des Z-Stapels mit Nilrot-Lipidablagerungen und Transwell-Poren dargestellt. Die Software ist nicht in der Lage, zwischen den beiden zu unterscheiden, wie der rote Kreis mit den Transwell-Poren und der grüne Pfeil, der auf die Ablagerungen von Nil Red zeigt, zeigen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 7: Vergleich des Maskenwerkzeugs. (A,B,C) 96-Well-plattierte RPE mit unterschiedlichen Mengen an Lipidablagerungen werden mit Nilrot (rot) identifiziert. Die Bilder werden mit drei verschiedenen gängigen Maskierungsmethoden maskiert: Find Maxima, Max Entropy und Renyi Entropy, und mit der von LipidUNet generierten Maske verglichen. Das Originalbild wird von einer manuellen Zählung der Lipidablagerungen begleitet, während die Masken die vorhergesagten Zahlen für jede Segmentierungsmethode anzeigen. Die durchschnittliche Fehlerquote wurde für jede Segmentierungsmethode mit folgender Formel berechnet: Mittelwert[(|Prognostizierte Anzahl - Manuelle Zählung|/Manuelle Zählung) x 100]. Die von LipidUNet generierte Maske identifiziert im Vergleich zu anderen Maskierungsmethoden Lipidablagerungen in Bildern mit variabler Ablagerung genauer (durchschnittliche Fehlerraten: 23 % LipidUnet, 1164 % Find Maxima, 851 % Max Entropy, 203 % Renyi Entropy). Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
| Bestandteil | Kat-Nummer | Aktien-Konz. | Finale Konz. | Ml |
| MEM alpha | 12571-063 | NA | 500 | |
| N2-Ergänzung | 17502-048 | NA | 1% | 5 |
| Hitzeinaktiviertes FBS | SH30071.03 | NA | 5% | 25 |
| NMEM NEAA | 11140-050 | 10mM | 0,01 Mio. | 5 |
| Natriumpyruvat | 11360-070 | 100mM | 1mM | 5 |
| Penicillin-Streptomycin | 15140-122 | 10000u/ml | 100U/ml | 5 |
| Taurin | T4571 | 50mg/ml | 250ug/ml | 2.5 |
| Hydrocortison | H6909 | 18,1 mg/l | 20ug/L | 0.553 |
| T3 | T5516 | 20ug/L | 0,013 ug/L | 0.33 |
| Gesamtvolumen, ml | 548.383 | |||
Tabelle 1: Zusammensetzung der RPE-MM-Reagenzien. Eine Liste der Reagenzien und optimalen Konzentrationen für RPE-MM.
Diskussion
Dieses Protokoll bietet eine Methode zur effizienten Markierung, Abbildung und Quantifizierung von Lipidablagerungen in monogenen und polygenen In-vitro-Krankheitsmodellen für degenerative Augenerkrankungen. Die KI-basierte Software LipidUNet kann auf drei gängige Lipidmarker, APOE, Nile Red und BODIPY, angewendet werden und bietet eine schnelle, automatische Analysemethode, die eine standardisierte und unvoreingenommene Quantifizierung ermöglicht.
Die Haupteinschränkung von LipidUNet ist die Tatsache, dass der Trainingsdatensatz für die KI auf Bilder mit 40-facher Vergrößerung von Zellen beschränkt war, die in einer 96-Well-Platte kultiviert wurden. Aufgrund des Trainingsbildsatzes ist LipidUNet in seiner jetzigen Form auf die Analyse von Bildern mit 40-facher Vergrößerung beschränkt. Die Software kann verwendet werden, um 40-fache Bilder von Zellen zu analysieren, die auf anderen Kulturoberflächen als einer 96-Well-Platte kultiviert wurden, aber es sollte darauf geachtet werden, die generierten Ausgabemasken zu untersuchen, um die genaue Schwellenwertbildung durch die Software zu überprüfen. Es werden mehr Bildsätze (bei unterschiedlichen Vergrößerungen) benötigt, um den Umfang der zu analysierenden Proben/Bilder zu erweitern.
Das Protokoll besteht aus mehreren kritischen Schritten. Im Schritt des Lipidmarkers sollte der Benutzer bestätigen, dass die von ihm gewählte Markierungsverbindung (BODIPY, APOE, Nilrot) seine Probe effektiv markiert hat. Reife RPE-Zellen sind oft stark pigmentiert, was das Fluoreszenzsignal der Antikörper-Immunfärbung beeinträchtigen kann. Wenn das Fluoreszenzsignal schwach ist oder wenn die Hintergrundfärbung zu stark ist, kann LipidUNet Lipidtröpfchen nicht genau erkennen. Aus einem ähnlichen Grund müssen ordnungsgemäß ausgewählte Erfassungseinstellungen für den automatischen Bildgebungsschritt des Protokolls verwendet werden. Wenn die aufgenommenen Bilder von schlechter Qualität sind, hat LipidUNet Schwierigkeiten, die Bilder richtig zu maskieren, und daher ist die Quantifizierung ungenau (Abbildung 6A-L). Schließlich ist die Nachbearbeitung der Bilder ein wichtiger Schritt, da LipidUNet spezifische Anforderungen an die Software stellt, um zu funktionieren.
Im Vergleich zu Workflows für die Lipidanalyse, die manuelle Schwellenwerte verwenden, oder Techniken, die eine automatische Schwellenwertbildung in Software wie Fiji beinhalten, bietet LipidUNet eine unverzerrte und zuverlässige Segmentierung über Bilder mit variabler Lipidablagerung, was sich in einer geringen Fehlerrate bei der Identifizierung von Lipidpartikeln widerspiegelt (Abbildung 7). Die Software ermöglicht dem Benutzer die Eingabe zusätzlicher Trainingsbilder, was die Analyse von Bildsätzen ermöglicht, die über diejenigen hinausgehen, die ein 40-faches Vergrößerungsobjektiv verwenden, oder sogar solche, die einen anderen Lipidmarker verwenden, wie im Protokoll beschrieben. In Zukunft soll die Software darauf trainiert werden, 3D-Bilder zu analysieren, so dass die Quantifizierung des Lipidablagerungsvolumens möglich ist. Degenerative Augenerkrankungen, die die Ablagerung von Lipiden als Hauptursache für die Pathologie implizieren, sind weit verbreitet, und es wird prognostiziert, dass die Fälle mit zunehmender älterer Bevölkerung zunehmenwerden 13. Genaue Krankheitsmodelle und effiziente Analyseinstrumente, wie wir sie in diesem Protokoll skizziert haben, werden die Entwicklung neuartiger therapeutischer Interventionen ermöglichen.
Offenlegungen
Keine Offenlegungen.
Danksagungen
Wir danken dem Histologie-Kern des National Eye Institute (NEI) für den Einsatz des konfokalen Zeiss-Systems. Diese Arbeit wurde aus Mitteln des NEI IRP (Förderkennzeichen ZIA EY000533-04) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.22 µm Steriflip filter system | EMD Millipore | SCGP00525 | |
| 1x Dulbecco's Phosphate Buffered Saline | Gibco | 14190-144 | |
| 3,3',5-Triiodo-L-thyronine | Sigma | T5516 | |
| Albumin Bovine, Fraction V | MP Biomedical | 160069 | |
| Alexa Fluor 555 rabbit anti-goat IgG (H+L) | Invitrogen | A21431 | APOE secondary antibody |
| APOE primary antibody | Millipore Sigma | AB947 | |
| BODIPY 493/503 | Invitrogen | D3922 | Protect from light |
| Complement competent human serum | Millipore Sigma | S1-LITER | |
| CTS N2 Supplement | Life Technologies | A13707-01 | |
| Fetal Bovine Serum | Hyclone | SH30071.03 | |
| Fluoromount-G | SouthernBiotech | 0100-01 | Slide mounting media |
| Glass Cover Slips #1 1/2 22 mm x 22 mm | Electron Microscopy Sciences | 72204-01 | |
| Glass Microscope Slide 25 mm x 75 mm- 1.2 mm Thick | Electron Microscopy Sciences | 71870-01 | |
| Hydrocortisone | Sigma | H0396 | |
| MEM Alpha | Life Technologies | 12571-063 | |
| MEM non-essential Amino Acids | Life Technologies | 11140 | |
| Nile Red | Sigma | 72485-100MG | Protect from light |
| Paraformaldehyde 16% Solution, EM Grade | Electron Microscopy Sciences | 15710 | |
| Penicillin-Strep | Life Technologies | 15140-148 | |
| Phosphate Buffered Saline 10x | Gibco | 70011-044 | |
| Rod Outer Segments (OS) | InVision Bioresources | 98740 | |
| Sodium bicarbonate | Sigma Aldrich | S5761 | |
| Sodium Pyruvate | Life Technologies | 11360-070 | |
| Sucrose | Sigma Aldrich | S1888 | |
| SYBR Green Master Mix | Bio-Rad | 1725274 | |
| Taurine | Sigma | T0625 | |
| Triton X-100 | Sigma | 9002-93-1 | |
| Tween 20 Ultrapure | Affymetrix | 9005-64-5 | |
| Vitronectin | Life Technologies | A14701SA | |
| Y-27632 dihydrochloride | R&D Systems | 1254 |
Referenzen
- Kanow, M. A., et al. Biochemical adaptations of the retina and retinal pigment epithelium support a metabolic ecosystem in the vertebrate eye. eLife. 6, e28899(2017).
- Adijanto, J., et al. The retinal pigment epithelium utilizes fatty acids for ketogenesis. The Journal of Biological Chemistry. 289 (30), 20570-20582 (2014).
- Farnoodian, M., et al. Cell-autonomous lipid-handling defects in Stargardt iPSC-derived retinal pigment epithelium cells. Stem Cell Reports. 17 (11), 2438-2450 (2022).
- Curcio, C. A. Soft drusen in age-related macular degeneration: Biology and targeting via the oil spill strategies. Investigative Ophthalmology & Visual Science. 59 (4), AMD160-AMD181 (2018).
- Cankova, Z., Huang, J. D., Kruth, H. S., Johnson, M. Passage of low-density lipoproteins through Bruch's membrane and choroid. Experimental Eye Research. 93 (6), 947-955 (2011).
- Curcio, C. A., et al. Esterified and unesterified cholesterol in drusen and basal deposits of eyes with age-related maculopathy. Experimental Eye Research. 81 (6), 731-741 (2005).
- Miyagishima, K. J., et al. AMPK modulation ameliorates dominant disease phenotypes of CTRP5 variant in retinal degeneration. Communications Biology. 4 (1), 1360(2021).
- Sharma, R., et al. Epithelial phenotype restoring drugs suppress macular degeneration phenotypes in an iPSC model. Nature Communications. 12 (1), 7293(2021).
- Farnoodian, M., et al. Cell-autonomous lipid-handling defects in Stargardt iPSC-derived retinal pigment epithelium cells. Stem Cell Reports. 17 (11), 2438-2450 (2022).
- Hallam, D., et al. An induced pluripotent stem cell patient specific model of complement factor H (Y402H) polymorphism displays characteristic features of age-related macular degeneration and indicates a beneficial role for UV light exposure. Stem Cells (Dayton, Ohio). 35 (11), 2305-2320 (2017).
- Sharma, R., Bose, D., Montford, J., Ortolan, D., Bharti, K. Triphasic developmentally guided protocol to generate retinal pigment epithelium from induced pluripotent stem cells. STAR Protocols. 3 (3), 101582(2022).
- Issa, P. C., Barnard, A. R., Herrmann, P., Washington, I., MacLaren, R. E. Rescue of the Stargardt phenotype in Abca4 knockout mice through inhibition of vitamin A dimerization. Proceedings of the National Academy of Sciences of the United States of America. 112 (27), 8415-8420 (2015).
- GBD 2019 Blindness and Vision Impairment Collaborators. Blindness and Vision Impairment Collaborators. Causes of blindness and vision impairment in 2020 and trends over 30 years, and prevalence of avoidable blindness in relation to VISION 2020: the Right to Sight: an analysis for the Global Burden of Disease Study. Lancet Global Health. 9 (2), e144-e160 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten