Method Article
LipidUNet-機械学習に基づくiPS細胞由来網膜色素上皮を用いた脂質沈着物のキャラクタリゼーション・定量法
要約
眼の網膜色素上皮層に影響を及ぼす変性眼疾患は、単一遺伝子および多遺伝子起源を有する。いくつかの疾患モデルとソフトウェアアプリケーションであるLipidUNetは、疾患のメカニズムと潜在的な治療介入を研究するために開発されました。
要約
網膜色素上皮(RPE)は、目の後ろにある六角形の細胞の単層です。光受容体と脈絡膜毛細血管に栄養とサポートを提供し、光受容体外側セグメント(POS)の食作用を行い、外網膜の恒常性を維持するために二極的にサイトカインを分泌します。突然変異、加齢、環境要因によって引き起こされる機能不全のRPEは、他の網膜層の変性をもたらし、視力喪失を引き起こします。変性RPEの特徴的な表現型の特徴は、細胞内および細胞内の脂質リッチ沈着物です。これらの沈着物は、さまざまな網膜変性疾患に共通の表現型です。単遺伝子網膜変性の脂質沈着表現型を in vitroで再現するために、患者の線維芽細胞から人工多能性幹細胞由来RPE(iRPE)を作製した。Stargardtおよび遅発性網膜変性症(L-ORD)の患者から作製した細胞株にPOSを7日間与えて、RPE生理機能を再現し、これらの疾患でPOS食作用誘発病理を引き起こしました。代替補体活性化に関連する多遺伝子性疾患である加齢黄斑変性症(AMD)のモデルを生成するために、iRPEは代替補体アナフィラトキシンで挑戦されました。細胞内および細胞内脂質沈着物は、ナイルレッド、ホウ素ジピロメテン(BODIPY)、およびアポリポタンパク質E(APOE)を使用して特徴付けられました。脂質沈着物の密度を定量化するために、機械学習ベースのソフトウェアであるLipidUNetが開発されました。このソフトウェアは、培養表面上のiRPEの最大強度投影画像でトレーニングされました。将来的には、3次元(3D)画像を解析し、脂肪滴の体積を定量化するように訓練される予定です。LipidUNetソフトウェアは、疾患モデルにおける脂質蓄積を減少させる薬剤を発見するための貴重なリソースとなるでしょう。
概要
網膜色素上皮(RPE)は、網膜光受容体に隣接する眼の後ろに位置する細胞の単層です。RPEは、光受容体に代謝的および構造的サポートを提供することにより、適切な視力を維持する上で重要な役割を果たします。健康なRPE細胞は、明確な六方晶形態によって特徴付けられる。それらはタイトジャンクションによって接続されており、RPEはその基底側に位置する絨毛毛細血管と頂端に位置する光受容体との間の障壁として作用することを可能にする。網膜生態系を維持するために、RPEは、RPE1におけるグルコース消費を最小限に抑える方法で、主要な代謝産物、例えばグルコースを光受容体にシャトルする。この制限により、RPEは、β酸化によってケトンに変換する脂肪酸を含む、代謝ニーズを維持するために他の代謝物に依存しています2。視細胞外帯(POS)消化からリサイクルされる可能性が高い脂肪酸をエネルギー源として利用するRPEの傾向を考えると、RPEの脂質処理経路の有害な変化は、単遺伝子性および多遺伝子性変性網膜疾患の両方につながるか、関与することがよくあります3。
RPE変性を引き起こす多遺伝子性変性眼疾患である加齢黄斑変性症(AMD)も、RPE単層における異常なオートファジーおよび脂質代謝に関連しています。機能不全のRPE単層がPOSを処理し、他の重要な機能を実行できないと、RPEとブルッフ膜の間に位置する基底線形沈着物(BLinD)と呼ばれる細胞外(サブRPE)沈着物が発生します-AMD病理の特徴です。BLinDの主成分にはリポタンパク質が含まれ、その中で最も豊富なのはアポリポタンパク質E(APOE)4です。BLinDの薄層の蓄積は、AMD 5,6の臨床症状として認識されている柔らかいドルーセンにつながる可能性があります。
いくつかのグループは、RPE機能障害を引き起こす幹細胞由来のin vitro疾患モデルがサブRPE脂質蓄積を特徴とすることを示しています7、8、9。Hallam et al. (2017)は、CFH遺伝子の多型によりAMDのリスクが高い患者から人工多能性幹細胞由来RPE(iRPE)を生成しました。iRPEはAPOEでマークされるようにドルーゼン蓄積を示し、高リスクRPEは低リスク患者から生成されたiRPEよりも大きな沈着物を蓄積しました10。
脂肪滴やドルーゼン沈着などのAMDの細胞の特徴を再現する in vitro モデルを作成するために、患者の血液サンプルから生成されたiRPEラインを、以前に公開された発達ガイドプロトコル11を使用して確立しました。iRPEは、AMDの考えられる原因の1つである代替補体シグナル伝達の増加を模倣するアナフィラトキシンを含む溶液である補体コンピテントヒト血清(CC-HS)に供されました8。得られた脂質沈着物の細胞内沈着を、一般的に使用される脂質およびリポタンパク質マーカー、APOE、ナイルレッド、およびBODIPYを用いて測定した。これらのマーカーを通じて、CC-HSを介した活性化補体シグナル伝達がiRPE細胞8における脂質蓄積を悪化させることが示された。
単遺伝子網膜変性疾患の疾患モデルを開発するために、RPEの ABCA4 遺伝子の変異によって引き起こされる疾患であるスターガルト病の患者からiRPE株を開発しました。 ABCA4 がノックアウトされると、高レベルのリン脂質と光依存性脂質過酸化生成物を含むことが知られている細胞内沈着物であるA2EリポフスチンがRPE12の内部に蓄積することが以前に示されています。 ABCA4 ノックアウトラインは患者ラインと一緒に開発され、両方ともPOS給餌を受けました。Stargardt iRPEは、POS食作用誘発病理を示し、BODIPY染色によって定量化された脂質蓄積の増加を示しました。 ABCA4 KO由来のRPEをCC-HS処理した。BODIPYシグナルの定量化は、同様にスターガルト病モデルにおける脂質取り扱いの欠陥を示した9。
これらの疾患の有病率と効果的な治療法の必要性を考えると、上記の関連する疾患モデルとともに、潜在的な治療法の有効性を定量化するための堅牢な方法を確立する必要があります。客観的、自動化、標準化された方法で脂質沈着を定量するために、機械学習ベースのソフトウェアであるLipidUNetが作成され、マスク分析ツールと組み合わせると、一般的なマーカーであるNile Red、BODIPY、およびAPOEを使用して脂質沈着を迅速かつ効果的に識別できます。この分析パイプラインを使用して得られた要約統計量は、分析してグラフィカルに表示することができ、治療条件の比較が容易になります。プロトコルの回路図を 図1に示します。
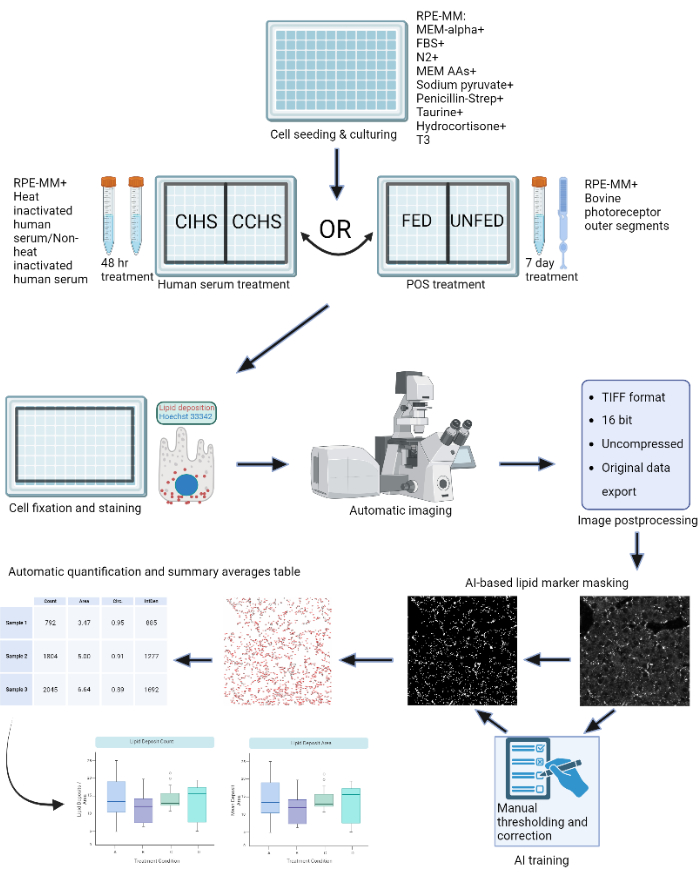
図1:プロトコルの概略図:RPE細胞を96ウェルプレート上で増殖させ、活性ヒト血清または精製ウシ外側セグメントでチャレンジして、in vitroでさまざまな種類の網膜変性をモデル化します。RPE細胞は、ナイルレッド、ボディピー、およびAPOEでリポタンパク質沈着物について固定および染色されます。共焦点顕微鏡を使用して、蛍光標識された脂質粒子のZスタックを取得し、その後、2D最大強度投影に加工します。機械学習アルゴリズムは、リポタンパク質粒子を認識して正しくセグメント化するようにトレーニングされました。粒子数とさまざまな形状メトリックを含むサマリーテーブルが生成され、その後のプロットと統計分析に使用できます。この図の拡大版を表示するには、ここをクリックしてください。
プロトコル
すべてのプロトコルステップは、NIHの人間研究倫理委員会によって定められたガイドラインに準拠しています。幹細胞の研究と患者のサンプル収集は、米国政府の45 CFR 46ガイドラインに従って、NIHの人間研究保護局(OHRP)の下の複合神経科学施設内審査委員会(CNS IRB)によって承認されました。患者サンプルは、プロトコル番号NCT01432847(https://clinicaltrials.gov/ct2/show/NCT01432847?cond=NCT01432847&draw=2&rank=1)の下でヘルシンキ宣言によって設定された基準に従って、CNS IRB承認の同意書を使用して収集されました。
1. iRPE の生成
- Sharmaらによって公開されたプロトコルに従って、患者の血液由来iPS細胞からiRPEを生成します, 202211 (図2 および 図3)。
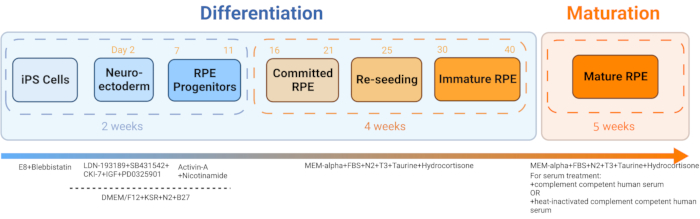
図2:iRPEの分化と成熟の概略図。iRPEを生成するために、確立された分化プロトコルに従い、細胞を5週間成熟させました。得られた細胞培養は、AMDやスターガルト病などの疾患におけるRPE機能障害を模倣するために様々な処理で操作することができるin vitroモデルとして機能します。この図の拡大版を表示するには、ここをクリックしてください。
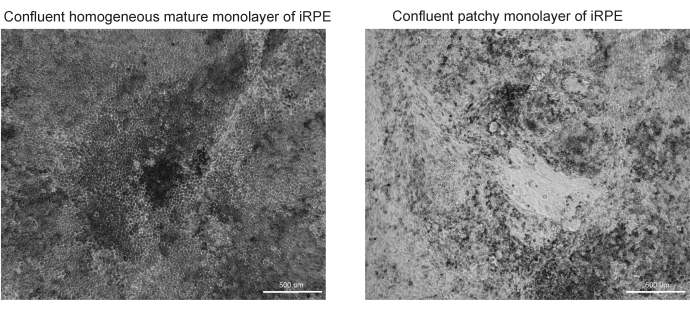
図3:RPEの分化と成熟の成功と失敗の代表的な画像。 TJP1 RPEの倍率10倍の2つの明視野画像が、iRPEプロトコルの42日目に表示されます。(A)分化と成熟が成功すると、色素沈着と多角形の形態を伴うコンフルエントなRPEが示されます。(B)分化と成熟の失敗は、ここに示すように、死にかけている細胞のクラスターを示します。 この図の拡大版を表示するには、ここをクリックしてください。
2. RPE メンテナンス メディア (RPE-MM) の準備
- N2サプリメントを4°Cで一晩解凍します。他のすべての試薬を室温(RT)で解凍します。
- 無菌条件下で、Sharmaら、202211によって確立されたプロトコルに従って、表1にリストされている試薬をリストされた希釈係数で追加します。
- 培地をよく混合し、0.22 μmのろ過ユニットを使用してろ過します。
注意: メディアは、2°Cで保管した場合、4週間以内の使用に適しています。
3. 96ウェルプレート播種
- ビトロネクチンのアリコートをRTで3〜5分間、または氷が完全に溶けるまで解凍します。
- ビトロネクチンを1xダルベッコリン酸緩衝生理食塩水(DPBS)で希釈し、1:200希釈液(ビトロネクチン:DPBS)を使用して目的の作業溶液を取得します。96ウェルプレートの場合、各ウェルに200 μLの作業溶液をコーティングします。
- 解凍したROCK阻害剤(Y-27632二塩酸塩)とRPE-MMを1:1000の希釈で組み合わせて、最終濃度10 μMにします。RPEセルのめっき媒体です。
- 自動細胞融解システムを使用してiRPEバイアルを解凍し、iRPE細胞懸濁液を50 mLチューブに移します。
- 細胞懸濁液を1:10希釈でめっき培地で希釈します。チューブを400 x g で5分間遠心分離します。
- 上清を注意深く吸引し、細胞を10 mLのプレーティング培地に再懸濁します。
- 細胞計数のために、400 μLのめっき培地を再懸濁した細胞溶液100 μLと混合します。このアリコートを使用して、細胞生存率カウンターを使用して細胞懸濁液の生細胞濃度を決定します。
- 細胞懸濁液をプレーティング培地で最終濃度60,000細胞/mLに希釈します。
- 96ウェルプレートからビトロネクチンコーティング溶液を完全に吸引し、200 μLの細胞懸濁液を各ウェルに分注します。約12,000セル/ウェルまたは~200セル/mm2になります。
- 播種した細胞プレートを37°Cおよび5%CO2で48時間インキュベートします。48時間後、培地をROCK阻害剤サプリメントなしのRPE-MMに変更します。.5週間の成熟期間中、2〜3日ごとに培地を交換してください。
4. 体外 疾患モデル
- コンピテントヒト血清(CC-HS)治療を補完する
- ヒト補体コンピテントセラムを4°Cで一晩解凍します。
- CC-HSを調製し、無能ヒト血清(CI-HS)培地を補完します。
- 5%CC-HS培地を調製するには、解凍した補体コンピテントヒト血清をRPE-MMと1:20希釈で混合します。使用前に0.22 μmのメディアフィルターで溶液をろ過してください。
- 5%補体不適格ヒト血清(CI-HS)培地を調製するには、まずCC-HSを57°Cの水浴中で30分間熱不活化し、次に1:20希釈で培地と混合します。使用前に0.22 μmのメディアフィルターで溶液をろ過してください。
- 血清は、細胞を200 μLの5%CC-HS培地または5%CI-HS培地で合計48時間インキュベーション時間で処理し、24時間後に培地をリフレッシュします。
- 細胞を1x DPBSで洗浄し、4%パラホルムアルデヒドでRTで20分間固定します。 1x DPBSでもう一度洗浄し、サンプルを4°Cで保存し、200 μLのDPBSに浸します。
- オプション:必要に応じて、プレートから細胞を溶解して、サブRPE脂質沈着のみを表示します。
- 細胞を溶解し、脂質沈着物のみを残すには、培地を除去し、各ウェルに200 μLの脱イオン水を加えます。
- 10〜15分間インキュベートし、細胞が除去されるまでピペットで上下に動かします。200 μLの脱イオン水でもう一度洗浄し、すぐに4%パラホルムアルデヒドで細胞を固定します。
- ヘキストを用いた核染色で細胞除去効果を確認。1%ウシ血清アルブミン(BSA)、0.5%トゥイーン20、および0.5%Triton-X 100を含む1x DPBS溶液に、1:2000希釈でヘキストを加えます。暗所で1時間RTでインキュベートします。その後、1x DPBSで洗浄します。
- iRPEにおける視細胞アウターセグメント(POS)治療
- POS の準備
- POSペレットチューブを-80°Cの保管場所から取り出し、蓋付きのアイスバケットで4°Cで一晩解凍します。
- 40mLの二重脱イオンH2O(ddH2O)中に10gのスクロースを混合することにより、POS洗浄バッファーを調製する。
- 穏やかに15分間攪拌しながら、混合物を40〜50°Cで加熱する。混合物に重炭酸ナトリウム840mgを加え、10分間加熱しながら攪拌する。
- 必要に応じて、POS洗浄バッファーの総容量をddH2Oで100 mLに調整し、必要に応じて1 N HClまたは1 N NaOHで溶液のpHを8.3に調整します。0.22 μmフィルターを使用して洗浄液をろ過します。
注:プロトコルはここで一時停止できます。POS洗浄バッファーは4°Cで一晩保存できます。 - 解凍したら、ペレットを15 mLのPOS洗浄バッファーに懸濁します。ペレット懸濁液中は、POSの完全性を確保するために穏やかにしてください。POS懸濁液を600 x g で4°Cで20分間遠心分離し、上清を吸引します。
- POSペレットを10 mLのPOS洗浄バッファーに再懸濁します。
- 100 μLのPOS + POS洗浄バッファー(POS溶液)を除去し、400 μLの1x DPBSで希釈します。希釈したPOS溶液50 μLを血液寒天プレートとアガロースプレートに広げ、細菌および真菌の汚染物質をチェックします。それぞれについてポジティブコントロールを準備し、すべてのプレートを37°Cで48時間インキュベートします。
- 1 μLのPOS溶液を検出ウェルに追加してqPCRアッセイを実行し、マイコプラズマをテストします。DNA断片を増幅するには、変性(95°C、15秒)、アニーリングと伸長(60°C、1分)を40サイクル行います。POSサンプル中のマイコプラズマを検出するためのフォワードプライマーとリバースプライマーは次のとおりです。
フォワードプライマー:GGA TTA GAT ACC CTG GTA GTC CAC G
リバースプライマー:CGT CAA TTC CTT TAA GTT TCC TTG GC - 細胞分析装置を使用してPOS濃度を測定し、必要に応じてアリコートします。RPE細胞を有する96ウェルプレートの1ウェルの場合、3 x 106 POSで十分です。望ましい比率は10 POS / RPEセルです。アリコートは将来の使用のために80°Cで保管してください。
- セルへのPOSの追加
- POSのバイアルを氷浴で解凍します。
- 調製したPOSの計算量をRPE-MMと混合し、細胞を1日1回POSで7日間処理します。
注: 毎日新鮮な POS ソリューションを準備します。 - 細胞を1x DPBSで洗浄し、4%パラホルムアルデヒドでRTで20分間固定します。 もう一度DPBSで洗浄し、サンプルを4°Cで保存し、200 μLのDPBSに浸します。
- POS の準備
5. サブRPE堆積物の染色
- ナイルレッド染色プロトコル
- PFA固定後、サンプルを1x DPBSで3回洗浄します。
注:すぐに使用しない場合は、ここでプロトコルを一時停止できますが、サンプルは4°Cで1x DPBS + 0.02%アジ化ナトリウム溶液に保存する必要があります。 - ナイルレッドストック溶液を調製するには、ナイルレッド粉末をアセトンに3 mg / mL濃度で溶解します。定期的に混合しながらRTで15分間インキュベートします。溶液中に残っている沈殿物のレベルに応じて、0.22μmフィルターで溶液を1〜2回ろ過します。
注意: ストック溶液を光から保護してください。 - 作業溶液を調製するには、ストック溶液を1x DPBSで1:500の比率で希釈します。200 μLの作業溶液をシェーカーのRTで30分間サンプルに加え、光から保護します。
- 1x PBSで3回洗浄し、サンプルを4°Cで保存し、200 μLのDPBSに浸します。
注:96ウェルプレートの代わりにトランスウェルで実験を行う場合は、サンプルを封入剤付きのスライドにマウントし、ガラスカバースリップで覆い、透明なマニキュアで密封することができます。細胞を上に向けてサンプルを取り付けるように注意する必要があります。
- PFA固定後、サンプルを1x DPBSで3回洗浄します。
- ボディ染色プロトコル
- 原液は、BODIPYを無水ジメチルスルホキシド(DMSO)に溶解し、原液濃度3.8mMにします。
- PFA固定サンプルの場合は、ボディディピーストックを1xDPBSで1:300に希釈します。細胞に200 μLを加え、RTのロッカーで一晩インキュベートします。
- 1x DPBSで3回洗浄し、サンプルを4°Cで保存し、200 μLのDPBSに浸します。
注:96ウェルプレートの代わりにトランスウェルで実験を行う場合は、サンプルを封入剤付きのスライドにマウントし、ガラスカバースリップで覆い、透明なマニキュアで密封することができます。細胞を上に向けてサンプルを取り付けるように注意する必要があります。
- APOE免疫染色プロトコル
- 1x DPBSを1%ウシ血清アルブミン(BSA)、0.5%トゥイーン20、および0.5%Triton-X 100と組み合わせて、緩衝液を作成します。
- PFA固定サンプルの場合は、サンプルをブロックし、200 μLの緩衝液でRTで1時間透過処理します。
- 1:100に希釈したAPOE一次抗体を緩衝液に加え、RTで一晩インキュベートします。
- 翌日、サンプルを1x DPBSで3回洗浄します。
- バッファー溶液に1:1000希釈で二次抗体を加え、200 μLの溶液をRTで1時間細胞に加えます。
- 1x DPBSで3回洗浄し、サンプルを4°Cで保存し、200 μLのDPBSに浸します。
注:96ウェルプレートの代わりにトランスウェルで実験を行う場合は、サンプルを封入剤(フルオロマウント)でスライドにマウントし、ガラスカバースリップで覆い、透明なマニキュアで密封することができます。細胞を上に向けてサンプルを取り付けるように注意する必要があります。
6.画像の自動化と処理
- 自動画像スキャン
注:この研究では、Zeiss LSM 800倒立共焦点走査型顕微鏡とZEN 3.2(青版)ソフトウェアを使用しました。温度変化に伴う媒体の屈折率の変化によるスキャン中の焦点面のドリフトを回避するために、イメージングの前に96ウェルプレートを最低60分間RTに温めてください。- 共焦点顕微鏡と40倍対物レンズを使用して、使用する脂質マーカーと追加の抗体に適した蛍光チャネルを含むスキャンプロファイルを作成します。
- [ タイル] チェックボックスを使用して、画像の自動化を設定します。96ウェルプレートを校正するには、正しいサンプルキャリア測定値が入力され、選択されていることを確認します。次に、[ キャリブレーション ]ボタンをクリックして、指示に従ってプレートをキャリブレーションしますが、これには10倍の対物レンズを使用する必要があります。
- 詳細設定ビューを選択して適切なウェルを選択し、位置機能を使用してウェルの中心近くに3つの異なるイメージングポイントを追加します。これは、[位置]サブタブで手動で行うことも、[位置設定]タブを使用して[運送業者による設定]を選択してランダムに行うこともできます。同じ染色のすべてのウェルについて繰り返します。
- オートメーション中にフォーカスと Z スタックの配置を最適化するには、[フォーカス 戦略 ] タブに移動して、[ タイル設定で定義されたフォーカス サーフェス/Z 値を使用] を選択します。別の方法で他のフォーカス戦略を使用することもできますが、最も一貫性のある結果を得るには、この設定を使用することをお勧めします。
- [タイル]タブで、[位置を確認]をクリックし、各位置の中央Z平面を手動で設定します。[オプション]サブタブの設定で画像の取得順序が決まるので、画像撮影を開始する前に確認してください。位置が選択された順序で画像を取り込むには、「タイル領域/位置」および「キャリアウェル/コンテナ」チェックボックスの選択を解除します。画像処理を容易にするために、シーンを別々のファイルに分割を選択します。
- [Z スタック] タブが [中央] に設定され、ユーザーの好みに合わせて範囲が入力され、[最適] ボタンが選択されてスライス間隔が設定されていることを確認します。
- [取得モード]、[チャンネル]、[フォーカス戦略]、[Z スタック]、および [タイル] タブを最適化したら、実験を開始します。
- 画像処理
- バッチ画像処理方法を使用して、 拡張焦点深度 法を使用して各Zスタックの最大投影を作成します。
- バッチ画像処理方法を使用して、最大の投影ファイルを16ビットTIFF画像としてエクスポートします。圧縮を [なし ] に設定し、[ 元のデータ ] がオンになっていることを確認します。結果として得られる画像は、脂質マーカーが発現される蛍光チャネルのみの最大投影グレースケールTIFFである必要があります。
7. セグメンテーションと定量化
注:LipidUNetプログラムは、96ウェルプレートからの40倍の画像でトレーニングされました。40倍の対物レンズを使用して取得した画像を使用することを強くお勧めします。
- LipidUNet ソフトウェアをインストールします。LipidUNet は、次の GitHub リポジトリからダウンロードできます。 https://github.com/RPEGoogleMap/LipidUNet
- ナイルレッド、ボディピー、または APOE のいずれかを表す TIFF イメージを識別し、使用する方法に応じて、Nile_Red、ボディピー、または APOE のいずれかという名前のディレクトリ内の imgs という名前のフォルダーに移動します。
注: LipidUNet プログラムがディレクトリを認識するには、正確な命名規則を使用する必要があります。 - LipidUNetソフトウェアを開きます(図4)。
- ソフトウェアの [予測 ]タブで、省略記号をクリックして名前付きディレクトリに移動して、関連するディレクトリ(Nile_Red、Bodipy、 または APOE)を選択します。クラスエントリをチェックして、LipidUNetプログラムが画像を正しく識別したことを確認します。
- アルゴリズムの確率しきい値を 0.01 から 0.99 の間で選択します。値を大きくすると、より多くの偽陽性が排除されますが、より多くの偽陰性が発生する可能性があり、値を小さくすると、より多くの偽陰性が排除される一方で、より多くの偽陽性が発生する可能性があります。既定値は 0.65 であり、推奨されます。
- [ 予測] をクリックします。
注:ソフトウェアはすべての画像を自動的に反復処理し、選択したディレクトリにpredicted_masksという新しいフォルダを作成します。 - マスク分析ツールを使用して、生成されたマスクを反復処理し、 マスク 画像から閾値脂質沈着物の定量的カウントを提供します。
- 生成されたカウントデータを分析して、処理条件を比較します。
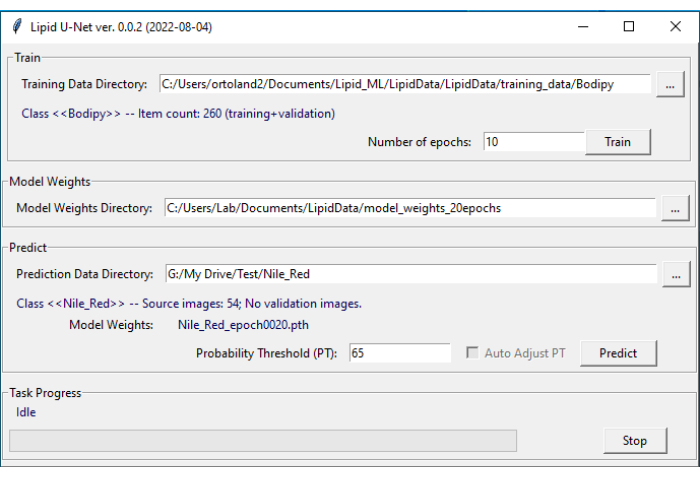
図4:脂質UNetユーザーインターフェイス。 LipidUNetソフトウェアには、脂質沈着物の画像が正しく識別されているトレーニングデータディレクトリ用に選択するさまざまなセクションがあります。トレーニング データから生成されるモデルの重み付けディレクトリ。ユーザーがセグメンテーションのために画像を入力する予測データディレクトリ。 この図の拡大版を表示するには、ここをクリックしてください。
結果
このプロトコルは、ナイルレッド、ボディピー、およびAPOEによって染色された脂質沈着物を特定するためのワークフローを提供します。開発されたソフトウェアは、脂質沈着物を自動的に識別して定量することができ、概説されたプロトコルが最適化されたときに最高のパフォーマンスを発揮します。細胞モデルの品質は適切な画像セグメンテーションの品質に大きく影響するため、分化に成功したRPE(図3A)と低分化したRPE(図3B)の例が含まれています。
プロトコルに記載されている3つのマーカーのうち2つ、ナイルレッドとボディピーは、蛍光画像ではっきりと明るい小さな円形のポイントとして識別されます(図5および図6)。プロトコルからの「ポジティブ」画像は、これらの異なる堆積物の適切な識別です(図5A-Dおよび図5E-H)。「ネガティブ」の結果は、弱い染色(図6A-Cおよび図6D-F)または高いバックグラウンド強度(図6G-I)のいずれかのために、バックグラウンド蛍光を堆積物と間違えることにより、画像の誤ったセグメンテーションを示します。
APOE鉱床にはさまざまなサイズと形状があり、ナイルレッドやボディピーの円形の堆積物ではなく、より楕円形または不規則に見えます。これらの堆積物はまた、点状が少なく、サンプルの透過性の変動により、信号強度が堆積物間で異なる可能性があります。正しい識別は、飽和度の低いものを含む各鉱床を識別しますが(図5I-L)、誤ったセグメンテーションはこれらの鉱床を拾いません(図6J-L)。したがって、急激なばらつきを避けるために、染色およびイメージング方法を最適化することが重要です。これを行う1つの方法は、免疫染色中にサンプルの透過処理ステップに注意を払うことです。蛍光シグナルを最適化するために、APOEの固定および免疫染色の前に細胞を溶解することができ、その結果、APOE沈着物の均一な飽和とより良いセグメンテーションが得られます。
96ウェルプレート以外の培養プラットフォームで成熟した細胞のセグメント化された画像も提供される。LipidUNetソフトウェアは、トランスウェルで培養された細胞の画像で実行され、脂質沈着物が閾値化される一方で、トランスウェル膜の細孔も閾値化されます(図6M-O)。形状とサイズが類似しているため、現在の形のLipidUNetソフトウェアは、脂質沈着物とトランスウェル孔の両方を無差別にマスクします。
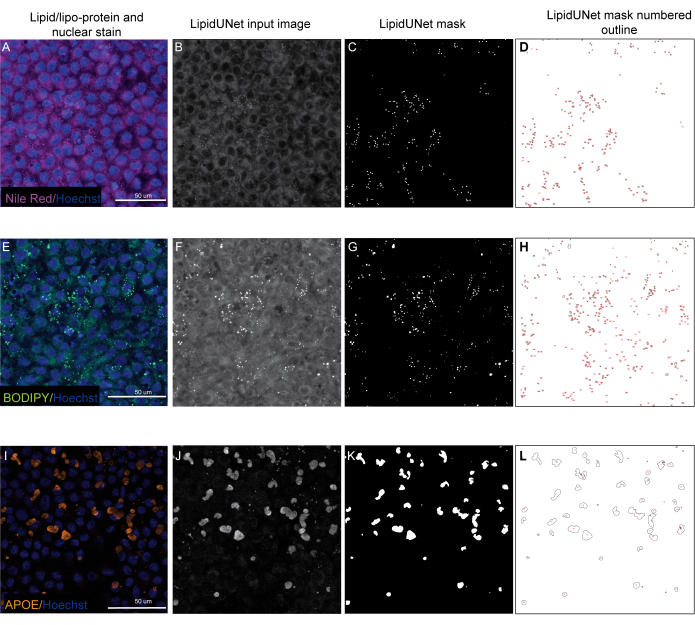
図5:代表的な結果。 (A、E、I)96ウェルメッキRPEは、ヘキスト核染色(青)およびナイルレッド(マゼンタ)、ボディピー(緑)、またはAPOE(オレンジ)のいずれかで染色され、Zスタックの最大強度投影です。(B,F,J)画像処理後のLipidUNetソフトウェアのグレースケール入力画像。(C,G,K)すべての堆積物が正しく識別されるLipidUNetによって生成されたマスク。(D,H,L)マスクされた各パーティクルのアウトラインには番号が付けられます。これらのラベルを使用すると、画像内の各粒子を生データを含むスプレッドのエントリに接続できます。(A-D)はナイルレッド染色を示しており、ソフトウェアは弱い信号にもかかわらずバックグラウンドに対する堆積物を正確に認識することができます。(E-H)は、BODIPY信号とバックグラウンドの間に強いコントラストを示し、これは理想的です。LipidUNetは、画像内のすべての堆積物を正しく識別します。(I-L)は強いAPOEシグナルを示し、この染色でよく見られるシグナル飽和の変動性を表します。それにもかかわらず、画像セグメンテーションは各APOEデポジットの境界を識別することができます。この図の拡大版を表示するには、ここをクリックしてください。
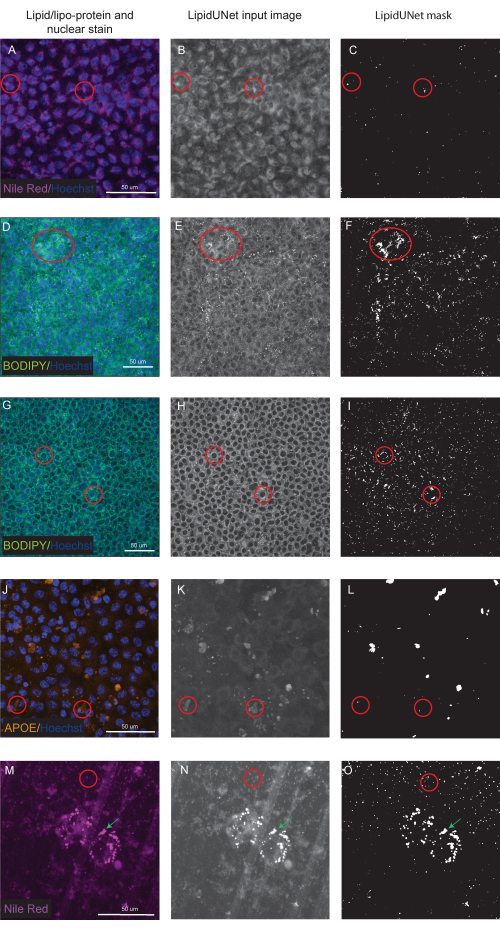
図6:最適ではない結果。 (A、D、G、J、M)96ウェルメッキRPEは、ヘキスト核染色(青)およびナイルレッド(マゼンタ)、ボディピー(緑)、またはAPOE(オレンジ)のいずれかで染色され、Zスタックの最大強度投影です。(B,E,H,K,N)画像処理後のLipidUNetソフトウェアのグレースケール入力画像。(C,F,I,L,O)LipidUNetによって生成されたマスクが正しくありません。赤い円は、ソフトウェアが脂質沈着物を誤って識別した場所を示します。(A-C) ソフトウェアがバックグラウンド染色を堆積物として識別したため、Nile Redの処理が正しくありません。これは、画像にバックグラウンドは高いが脂質沈着物がほとんどない場合に、より頻繁に発生する可能性があります。BODIPY染色の2つの例が示されています:(D-F)弱いBODIPY染色による低品質の画像と(G-I)高いバックグラウンドの強いBODIPYシグナル。どちらの場合も、ソフトウェアは、核を囲む背景の円形リングから小さな円形の脂質沈着物を区別することができません。染色とイメージングはこれらのエラーを回避するために最適化する必要がありますが、最新バージョンのLipidUNetはこれらの画像に対して大幅に改善されています。(J-L)APOEセグメンテーションが正しくありません。堆積物は信号のサイズと飽和度がより可変であるため、ソフトウェアはいくつかの堆積物を認識するのが困難です。(M-O)RPEをトランスウェルに播種し、ナイルレッドで染色した。Zスタックのスライスは、ナイルレッド脂質沈着物とトランスウェル孔の両方とともにここに示されています。ソフトウェアは、トランスウェル孔を含む赤い円とナイルレッド鉱床を指す緑色の矢印で示されているように、2つを区別できません。この図の拡大版を表示するには、ここをクリックしてください。

図7:マスクツールの比較 。 (A、B、C)脂質沈着量が変動する96ウェルメッキRPEは、ナイルレッド(赤)で識別されます。画像は、最大検索、最大エントロピー、およびRenyiエントロピーの3つの異なる一般的なマスク方法を使用してマスクされ、LipidUNetで生成されたマスクと比較されます。元の画像には脂質沈着物の手動カウントが添付され、マスクには各セグメンテーション方法による予測カウントが表示されます。平均エラー率は、次の式を使用してセグメンテーションの各方法について計算されました:mean[(|予測カウント - 手動カウント|/手動カウント) x 100]。LipidUNetで生成されたマスクは、他のマスキング方法と比較して、沈着が変動する画像全体で脂質沈着をより正確に識別します(平均エラー率:23%LipidUnet、1164%Find Maxima、851%最大エントロピー、203%Renyiエントロピー)。 この図の拡大版を表示するには、ここをクリックしてください。
| コンポーネント | 猫番号 | ストックコンク | 最終コンク | ミリリットル |
| MEM アルファ | 12571-063 | 該当なし | 500 | |
| N2サプリメント | 17502-048 | 該当なし | 1% | 5 |
| 熱不活性化FBS | SH30071.03 | 該当なし | 5% | 25 |
| NMEM NEAA | 11140-050 | 10ミリメートル | 0.01ミリメートル | 5 |
| ピルビン酸ナトリウム | 11360-070 | 100ミリメートル | 1ミリメートル | 5 |
| ペニシリン-ストレプトマイシン | 15140-122 | 10000u/mL | 100U/mL | 5 |
| タウリン | T4571 | 50ミリグラム/ミリリットル | 250グラム/ミリリットル | 2.5 |
| ヒドロコルチゾン | H6909 | 18.1ミリグラム/L | 20ug / L | 0.553 |
| T3 | T5516 | 20ug / L | 0.013ug/L | 0.33 |
| 総容量、mL | 548.383 | |||
表1:RPE-MM試薬組成。 RPE-MMの試薬と最適濃度のリスト。
ディスカッション
このプロトコルは、変性眼疾患の単一遺伝子および多遺伝子 のin vitro 疾患モデルにおいて脂質沈着を効率的に標識、画像化、および定量する方法を提供します。AIベースのソフトウェアであるLipidUNetは、APOE、ナイルレッド、およびBODIPYの3つの一般的な脂質マーカーに適用でき、定量を標準的かつ偏りのない高速自動分析方法を提供します。
LipidUNetの主な制限は、AIのトレーニングデータセットが96ウェルプレートで培養された細胞の40倍の倍率画像に制限されていたという事実です。トレーニング画像セットの結果として、LipidUNetは、現在の形式では、40倍の倍率画像の分析に限定されます。このソフトウェアは、96ウェルプレート以外の他の培養表面で培養された細胞の40倍の画像を分析するために使用できますが、ソフトウェアによる正確な閾値を検証するために、生成された出力マスクを調べるように注意する必要があります。分析できるサンプル/画像の範囲を拡大するには、より多くの画像セット(異なる倍率)が必要になります。
プロトコルにはいくつかの重要なステップがあります。脂質マーカーステップでは、ユーザーは選択した標識化合物(BODIPY、APOE、ナイルレッド)がサンプルを効果的に標識したことを確認する必要があります。成熟RPE細胞は色素沈着が強いことが多く、抗体免疫染色の蛍光シグナルを損なう可能性があります。蛍光シグナルが弱い場合やバックグラウンド染色が多すぎる場合、LipidUNetは脂肪滴を正確に識別できません。同様の理由から、プロトコルの自動イメージングステップに対して適切に選択された取得設定を使用する必要があります。取得した画像の品質が低い場合、LipidUNetは画像を適切にマスクするのに苦労するため、定量化は不正確になります(図6A-L)。最後に、LipidUNetにはソフトウェアが機能するための特定の要件があるため、画像の後処理は重要なステップです。
手動閾値を使用する脂質分析のワークフロー、またはフィジーのようなソフトウェアで自動閾値化を含む技術と比較すると、LipidUNetは、脂質粒子の同定における小さなエラー率に反映されるように、可変脂質沈着を伴う画像全体で非バイアスで信頼性の高いセグメンテーションを提供します(図7)。このソフトウェアでは、追加のトレーニング画像をユーザーが入力できるため、プロトコルで概説されているように、40倍の倍率の対物レンズを利用する画像セットや、異なる脂質マーカーを利用する画像セットの解析も可能です。将来的には、脂質沈着量の定量化が可能になるように、3D画像を解析するようにソフトウェアをトレーニングする予定です。脂質沈着を病態の主な原因とする変性眼疾患が蔓延しており、高齢者人口の拡大に伴い症例が増加すると予測されています13。このプロトコルで概説したように、正確な疾患モデルと効率的な分析ツールは、新しい治療介入の開発を可能にします。
開示事項
開示なし。
謝辞
ツァイス共焦点システムの使用について、国立眼科研究所(NEI)の組織学コアに感謝します。この作業は、NEI IRP基金(助成金番号ZIA EY000533-04)によって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| 0.22 µm Steriflip filter system | EMD Millipore | SCGP00525 | |
| 1x Dulbecco's Phosphate Buffered Saline | Gibco | 14190-144 | |
| 3,3',5-Triiodo-L-thyronine | Sigma | T5516 | |
| Albumin Bovine, Fraction V | MP Biomedical | 160069 | |
| Alexa Fluor 555 rabbit anti-goat IgG (H+L) | Invitrogen | A21431 | APOE secondary antibody |
| APOE primary antibody | Millipore Sigma | AB947 | |
| BODIPY 493/503 | Invitrogen | D3922 | Protect from light |
| Complement competent human serum | Millipore Sigma | S1-LITER | |
| CTS N2 Supplement | Life Technologies | A13707-01 | |
| Fetal Bovine Serum | Hyclone | SH30071.03 | |
| Fluoromount-G | SouthernBiotech | 0100-01 | Slide mounting media |
| Glass Cover Slips #1 1/2 22 mm x 22 mm | Electron Microscopy Sciences | 72204-01 | |
| Glass Microscope Slide 25 mm x 75 mm- 1.2 mm Thick | Electron Microscopy Sciences | 71870-01 | |
| Hydrocortisone | Sigma | H0396 | |
| MEM Alpha | Life Technologies | 12571-063 | |
| MEM non-essential Amino Acids | Life Technologies | 11140 | |
| Nile Red | Sigma | 72485-100MG | Protect from light |
| Paraformaldehyde 16% Solution, EM Grade | Electron Microscopy Sciences | 15710 | |
| Penicillin-Strep | Life Technologies | 15140-148 | |
| Phosphate Buffered Saline 10x | Gibco | 70011-044 | |
| Rod Outer Segments (OS) | InVision Bioresources | 98740 | |
| Sodium bicarbonate | Sigma Aldrich | S5761 | |
| Sodium Pyruvate | Life Technologies | 11360-070 | |
| Sucrose | Sigma Aldrich | S1888 | |
| SYBR Green Master Mix | Bio-Rad | 1725274 | |
| Taurine | Sigma | T0625 | |
| Triton X-100 | Sigma | 9002-93-1 | |
| Tween 20 Ultrapure | Affymetrix | 9005-64-5 | |
| Vitronectin | Life Technologies | A14701SA | |
| Y-27632 dihydrochloride | R&D Systems | 1254 |
参考文献
- Kanow, M. A., et al. Biochemical adaptations of the retina and retinal pigment epithelium support a metabolic ecosystem in the vertebrate eye. eLife. 6, e28899(2017).
- Adijanto, J., et al. The retinal pigment epithelium utilizes fatty acids for ketogenesis. The Journal of Biological Chemistry. 289 (30), 20570-20582 (2014).
- Farnoodian, M., et al. Cell-autonomous lipid-handling defects in Stargardt iPSC-derived retinal pigment epithelium cells. Stem Cell Reports. 17 (11), 2438-2450 (2022).
- Curcio, C. A. Soft drusen in age-related macular degeneration: Biology and targeting via the oil spill strategies. Investigative Ophthalmology & Visual Science. 59 (4), AMD160-AMD181 (2018).
- Cankova, Z., Huang, J. D., Kruth, H. S., Johnson, M. Passage of low-density lipoproteins through Bruch's membrane and choroid. Experimental Eye Research. 93 (6), 947-955 (2011).
- Curcio, C. A., et al. Esterified and unesterified cholesterol in drusen and basal deposits of eyes with age-related maculopathy. Experimental Eye Research. 81 (6), 731-741 (2005).
- Miyagishima, K. J., et al. AMPK modulation ameliorates dominant disease phenotypes of CTRP5 variant in retinal degeneration. Communications Biology. 4 (1), 1360(2021).
- Sharma, R., et al. Epithelial phenotype restoring drugs suppress macular degeneration phenotypes in an iPSC model. Nature Communications. 12 (1), 7293(2021).
- Farnoodian, M., et al. Cell-autonomous lipid-handling defects in Stargardt iPSC-derived retinal pigment epithelium cells. Stem Cell Reports. 17 (11), 2438-2450 (2022).
- Hallam, D., et al. An induced pluripotent stem cell patient specific model of complement factor H (Y402H) polymorphism displays characteristic features of age-related macular degeneration and indicates a beneficial role for UV light exposure. Stem Cells (Dayton, Ohio). 35 (11), 2305-2320 (2017).
- Sharma, R., Bose, D., Montford, J., Ortolan, D., Bharti, K. Triphasic developmentally guided protocol to generate retinal pigment epithelium from induced pluripotent stem cells. STAR Protocols. 3 (3), 101582(2022).
- Issa, P. C., Barnard, A. R., Herrmann, P., Washington, I., MacLaren, R. E. Rescue of the Stargardt phenotype in Abca4 knockout mice through inhibition of vitamin A dimerization. Proceedings of the National Academy of Sciences of the United States of America. 112 (27), 8415-8420 (2015).
- GBD 2019 Blindness and Vision Impairment Collaborators. Blindness and Vision Impairment Collaborators. Causes of blindness and vision impairment in 2020 and trends over 30 years, and prevalence of avoidable blindness in relation to VISION 2020: the Right to Sight: an analysis for the Global Burden of Disease Study. Lancet Global Health. 9 (2), e144-e160 (2021).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved