Method Article
Metodo basato sull'apprendimento automatico di LipidUNet per la caratterizzazione e la quantificazione dei depositi lipidici utilizzando l'epitelio pigmentato retinico derivato da iPSC
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Le malattie degenerative dell'occhio che colpiscono lo strato di epitelio pigmentato retinico dell'occhio hanno origini monogeniche e poligeniche. Diversi modelli di malattia e un'applicazione software, LipidUNet, sono stati sviluppati per studiare i meccanismi della malattia, nonché potenziali interventi terapeutici.
Abstract
L'epitelio pigmentato retinico (RPE) è un monostrato di cellule esagonali situato nella parte posteriore dell'occhio. Fornisce nutrimento e supporto ai fotorecettori e ai capillari coroideali, esegue la fagocitosi dei segmenti esterni dei fotorecettori (POS) e secerne citochine in modo polarizzato per mantenere l'omeostasi della retina esterna. L'RPE disfunzionale, causata da mutazioni, invecchiamento e fattori ambientali, provoca la degenerazione di altri strati retinici e causa la perdita della vista. Una caratteristica fenotipica della degenerazione dell'RPE sono i depositi ricchi di lipidi intra e subcellulari. Questi depositi sono un fenotipo comune in diverse malattie degenerative della retina. Per riprodurre il fenotipo del deposito lipidico delle degenerazioni retiniche monogeniche in vitro, è stata generata RPE derivata da cellule staminali pluripotenti (iRPE) indotta dai fibroblasti dei pazienti. Le linee cellulari generate da pazienti con malattia da degenerazione retinica di Stargardt e ad esordio tardivo (L-ORD) sono state alimentate con POS per 7 giorni per replicare la funzione fisiologica RPE, che ha causato la patologia indotta dalla fagocitosi POS in queste malattie. Per generare un modello per la degenerazione maculare legata all'età (AMD), una malattia poligenica associata all'attivazione del complemento alternativo, l'iRPE è stato sfidato con anafilatossine del complemento alternative. I depositi lipidici intra e subcellulari sono stati caratterizzati utilizzando rosso del Nilo, boro-dipirrometene (BODIPY) e apolipoproteina E (APOE). Per quantificare la densità dei depositi lipidici, è stato sviluppato un software basato sull'apprendimento automatico, LipidUNet. Il software è stato addestrato su immagini di proiezione a massima intensità di iRPE su superfici di coltura. In futuro, sarà addestrato ad analizzare immagini tridimensionali (3D) e quantificare il volume delle goccioline lipidiche. Il software LipidUNet sarà una risorsa preziosa per scoprire farmaci che riducono l'accumulo di lipidi nei modelli di malattia.
Introduzione
L'epitelio pigmentato retinico (RPE) è un monostrato di cellule situato nella parte posteriore dell'occhio adiacente ai fotorecettori retinici. RPE svolge un ruolo vitale nel mantenimento di una corretta visione fornendo supporto metabolico e strutturale ai fotorecettori. Le cellule RPE sane sono caratterizzate da una morfologia esagonale distinta. Sono collegati da giunzioni strette, che consentono all'RPE di fungere da barriera tra il coriocapillare situato sul suo lato basale e i fotorecettori situati apicalmente. Per mantenere l'ecosistema retinico, RPE trasporta metaboliti chiave, ad esempio glucosio, ai fotorecettori in modo da ridurre al minimo il consumo di glucosio nell'RPE1. A causa di questa limitazione, RPE dipende da altri metaboliti per mantenere le loro esigenze metaboliche, compresi gli acidi grassi, che RPE converte in chetoni attraverso β-ossidazione2. Data la propensione dell'RPE a utilizzare acidi grassi, che sono probabilmente riciclati dalla digestione del segmento esterno dei fotorecettori (POS), come fonte di energia, cambiamenti dannosi nelle vie di elaborazione dei lipidi nell'RPE spesso portano a, o sono implicati in, malattie degenerative della retina sia monogeniche che poligeniche3.
La degenerazione maculare legata all'età (AMD), una malattia degenerativa poligenica dell'occhio che causa la degenerazione RPE, è stata anche collegata all'autofagia aberrante e al metabolismo lipidico nel monostrato RPE. L'incapacità di un monostrato RPE disfunzionale di elaborare POS e svolgere altre funzioni critiche porta a depositi extracellulari (sub-RPE) chiamati depositi lineari basali (BLinD) situati tra l'RPE e la membrana di Bruch - un segno distintivo delle patologie AMD. I componenti principali di BLinD includono le lipoproteine, la più abbondante delle quali è l'apolipoproteina E (APOE)4. L'accumulo di strati sottili di BLinD può portare a drusen morbide, che è riconosciuto come un sintomo clinico di AMD 5,6.
Diversi gruppi hanno dimostrato che i modelli di malattia in vitro derivati da cellule staminali che causano disfunzione RPE presentano un accumulo lipidico sub-RPE 7,8,9. Hallam et al. (2017) hanno generato RPE derivata da cellule staminali pluripotenti indotte (iRPE) da pazienti ad alto rischio di AMD a causa di un polimorfismo del gene CFH. L'iRPE ha mostrato l'accumulo di drusen, come contrassegnato da APOE, e l'RPE ad alto rischio ha accumulato depositi più grandi rispetto all'iRPE generato da pazienti a basso rischio10.
Per creare un modello in vitro che ricapitola i segni distintivi cellulari dell'AMD, come le goccioline lipidiche e la deposizione di drusen, sono state stabilite linee iRPE generate da campioni di sangue dei pazienti utilizzando un protocollo11 guidato dallo sviluppo precedentemente pubblicato. Gli iRPE sono stati sottoposti a siero umano competente per il complemento (CC-HS), una soluzione contenente anafilautotossine che imitano una possibile causa di AMD: aumento della segnalazione del complemento alternativo8. La deposizione cellulare e subcellulare risultante di depositi lipidici è stata misurata utilizzando marcatori lipidici e lipoproteici comunemente usati, APOE, Nile Red e BODIPY. Attraverso questi marcatori, è stato dimostrato che la segnalazione attivata del complemento tramite CC-HS ha esacerbato l'accumulo di lipidi nelle cellule iRPE8.
Per sviluppare un modello di malattia per una malattia degenerativa retinica monogenica, sono state sviluppate linee iRPE da pazienti con malattia di Stargardt, una malattia causata da mutazioni del gene ABCA4 nella RPE. È stato precedentemente dimostrato che quando ABCA4 viene eliminato, la lipofuscina A2E, un deposito intracellulare noto per contenere alti livelli di fosfolipidi e prodotti di perossidazione lipidica dipendenti dalla luce, si accumula all'interno dell'RPE12. Le linee knockout ABCA4 sono state sviluppate insieme alle linee paziente ed entrambe sono state sottoposte ad alimentazione POS. L'iRPE Stargardt ha dimostrato una patologia indotta da fagocitosi POS, che mostra un aumento dell'accumulo lipidico quantificato dalla colorazione BODIPY. RPE derivato da ABCA4 KO iPSC sono stati sottoposti a trattamento CC-HS; la quantificazione del segnale BODIPY ha mostrato un difetto nella gestione dei lipidi anche nel modello di malattia di Stargardt9.
Data la prevalenza di queste malattie e la necessità di terapie efficaci, insieme ai modelli di malattia pertinenti sopra descritti, è necessario stabilire metodi solidi per quantificare l'efficacia dei potenziali trattamenti. Per quantificare i depositi lipidici in modo oggettivo, automatizzato e standardizzato, è stato creato un software basato sull'apprendimento automatico, LipidUNet, in modo che, se abbinato a strumenti di analisi della maschera, la deposizione lipidica possa essere identificata rapidamente ed efficacemente utilizzando i marcatori comuni Nile Red, BODIPY e APOE. Le statistiche riassuntive ottenute utilizzando questa pipeline di analisi possono quindi essere analizzate e visualizzate graficamente, consentendo un facile confronto delle condizioni di trattamento. Lo schema del protocollo è illustrato nella Figura 1.
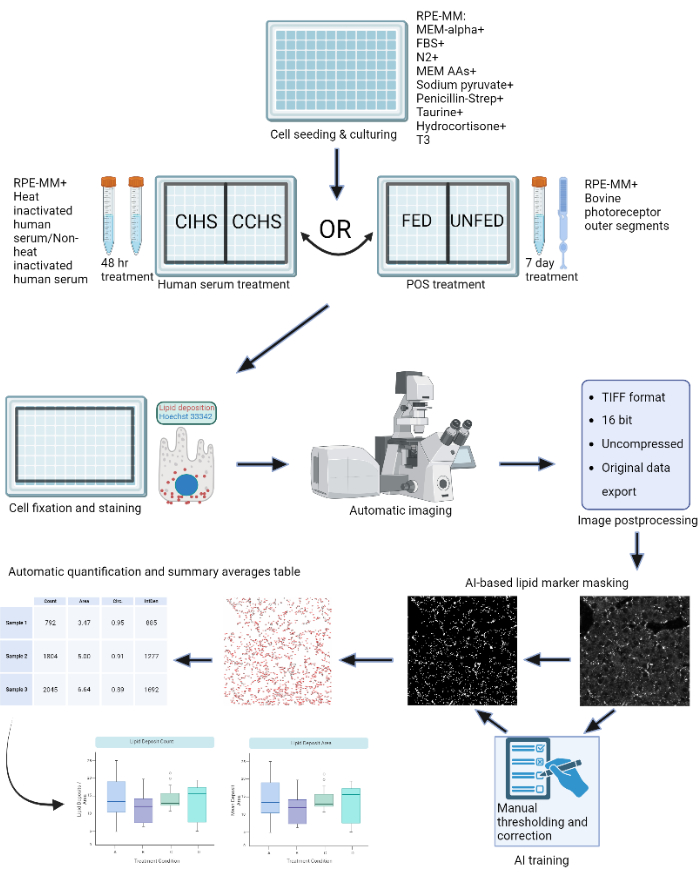
Figura 1: Schema del protocollo: le cellule RPE vengono coltivate su una piastra a 96 pozzetti e sfidate con siero umano attivo o segmenti esterni bovini purificati per modellare diversi tipi di degenerazioni retiniche in vitro. Le cellule RPE sono fissate e colorate per i depositi di lipoproteine con Nilo Red, BODIPY e APOE. Un microscopio confocale viene utilizzato per acquisire Z-stack di particelle lipidiche marcate con fluorescenza, che vengono successivamente elaborate in proiezioni di massima intensità 2D. Un algoritmo di apprendimento automatico è stato addestrato per riconoscere e segmentare correttamente le particelle lipoproteiche. Vengono generate tabelle riassuntive contenenti il conteggio delle particelle e varie metriche di forma che possono essere utilizzate per la successiva rappresentazione grafica e analisi statistica. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Protocollo
Tutte le fasi del protocollo aderiscono alle linee guida stabilite dal comitato etico per la ricerca umana del NIH. Il lavoro sulle cellule staminali e la raccolta di campioni dei pazienti sono stati approvati dal Combined NeuroScience Institutional Review Board (CNS IRB) sotto l'Office of Human Research Protection (OHRP), NIH, secondo le linee guida 45 CFR 46 del governo degli Stati Uniti. I campioni dei pazienti sono stati raccolti utilizzando il modulo di consenso approvato dall'IRB del CNS in conformità con i criteri stabiliti dalla Dichiarazione di Helsinki con il numero di protocollo NCT01432847 (https://clinicaltrials.gov/ct2/show/NCT01432847?cond=NCT01432847&draw=2&rank=1).
1. Generazione iRPE
- Generare iRPE da iPSC derivate dal sangue del paziente seguendo il protocollo pubblicato da Sharma et al., 202211 (Figura 2 e Figura 3).
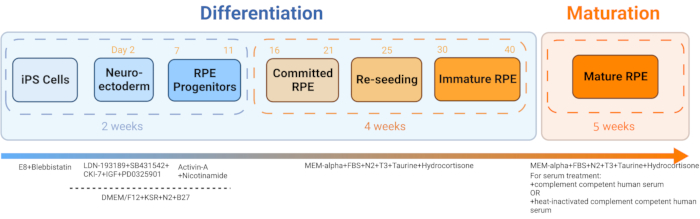
Figura 2: Schema della differenziazione e della maturazione delle iRPE. Per generare iRPE, è stato seguito un protocollo di differenziazione stabilito e le cellule sono state lasciate maturare per 5 settimane. La coltura cellulare risultante agisce come un modello in vitro che può essere manipolato con vari trattamenti per imitare la disfunzione RPE in malattie come AMD e malattia di Stargardt. Fare clic qui per visualizzare una versione ingrandita di questa figura.
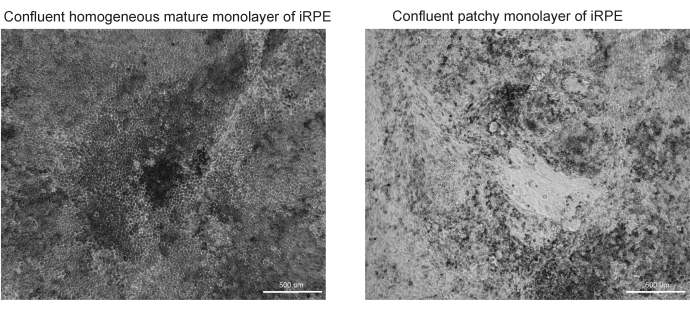
Figura 3: Immagini rappresentative della differenziazione e della maturazione RPE di successo e di quelle non riuscite. Due immagini in campo chiaro con ingrandimento 10x di TJP1 RPE sono mostrate al giorno 42 del protocollo iRPE. (A) Il successo della differenziazione e della maturazione mostrerà RPE confluente con pigmentazione e morfologia poligonale. (B) La differenziazione e la maturazione infruttuose mostreranno gruppi di cellule morenti, come mostrato qui. Fare clic qui per visualizzare una versione ingrandita di questa figura.
2. Preparazione dei supporti di manutenzione RPE (RPE-MM)
- Scongelare il supplemento N2 a 4 °C durante la notte. Scongelare tutti gli altri reagenti a temperatura ambiente (RT).
- In condizioni sterili, aggiungere i reagenti elencati nella Tabella 1 ai fattori di diluizione elencati, secondo il protocollo stabilito da Sharma et al., 202211.
- Mescolare bene il fluido e filtrarlo utilizzando un'unità di filtrazione da 0,22 μm.
NOTA: Il supporto è adatto per l'uso entro 2 settimane se conservato a 4 °C.
3. Semina della piastra a 96 pozzetti
- Scongelare un'aliquota di vitronectina a RT per 3-5 minuti o fino a quando il ghiaccio non è completamente sciolto.
- Diluire la vitronectina con soluzione salina tamponata fosfato 1x Dulbecco (DPBS) per ottenere la soluzione di lavoro desiderata utilizzando una diluizione 1:200 (vitronectina: DPBS). Per una piastra da 96 pozzetti, rivestire ciascun pozzetto con 200 μL della soluzione di lavoro.
- Combinare l'inibitore ROCK scongelato (Y-27632 dicloridrato) con RPE-MM ad una diluizione 1:1000 per ottenere una concentrazione finale di 10 μM. Questo è il supporto di placcatura per le celle RPE.
- Scongelare il flaconcino iRPE utilizzando un sistema automatico di scongelamento cellulare e trasferire la sospensione della cella iRPE in un tubo da 50 ml.
- Diluire la sospensione cellulare con il mezzo di placcatura ad una diluizione 1:10. Centrifugare il tubo a 400 x g per 5 min.
- Aspirare con cautela il surnatante e risospendere le cellule in 10 ml del mezzo di placcatura.
- Miscelare 400 μL del mezzo di placcatura con 100 μL della soluzione cellulare risospesa per il conteggio delle cellule. Utilizzare questa aliquota per determinare la concentrazione cellulare vitale della sospensione cellulare utilizzando un contatore di vitalità cellulare.
- Diluire la sospensione cellulare con il mezzo di placcatura ad una concentrazione finale di 60.000 cellule/ml.
- Aspirare completamente la soluzione di rivestimento di vitronectina dalla piastra a 96 pozzetti ed erogare 200 μL della sospensione cellulare in ciascun pozzetto. Ci saranno circa 12.000 celle / pozzetto o ~ 200 celle / mm2.
- Incubare le piastre cellulari seminate per 48 ore a 37 °C e 5% di CO2. Dopo 48 ore, cambiare il supporto a RPE-MM senza supplemento inibitore ROCK. Cambiare il terreno ogni 2-3 giorni durante il periodo di maturazione di 5 settimane.
4. Modelli di malattia in vitro
- Trattamento complementare con siero umano competente (CC-HS)
- Scongelare il siero umano complementare competente a 4°C durante la notte.
- Preparare CC-HS e integrare i mezzi di siero umano incompetente (CI-HS).
- Per preparare il supporto CC-HS al 5%, mescolare il siero umano competente per il complemento scongelato con RPE-MM ad una diluizione 1:20. Filtrare la soluzione attraverso un filtro da 0,22 μm prima dell'uso.
- Per preparare il 5% di siero umano incompetente (CI-HS), prima inattivare a caldo il CC-HS a bagnomaria a 57 °C per 30 minuti e quindi miscelare con i terreni di coltura ad una diluizione 1:20. Filtrare la soluzione attraverso un filtro da 0,22 μm prima dell'uso.
- Il siero tratta le cellule con 200 μL di mezzi CC-HS al 5% o CI-HS al 5% per un tempo di incubazione totale di 48 ore, rinfrescando il terreno dopo 24 ore.
- Lavare le cellule con 1x DPBS e fissarle con paraformaldeide al 4% per 20 minuti a RT. Lavare ancora una volta con 1x DPBS e conservare i campioni a 4 °C, immersi in 200 μL di DPBS.
- FACOLTATIVO: Se lo si desidera, lisare le cellule dalla piastra per mostrare solo la deposizione lipidica sub-RPE.
- Per lisare le cellule e lasciare solo depositi lipidici, rimuovere il mezzo e aggiungere 200 μL di acqua deionizzata a ciascun pozzetto.
- Incubare per 10-15 minuti, pipettare su e giù fino a quando le cellule non vengono rimosse. Lavare ancora una volta con 200 μL di acqua deionizzata e fissare immediatamente le cellule con paraformaldeide al 4%.
- Confermare l'efficacia della rimozione cellulare con la colorazione nucleare utilizzando Hoechst. Aggiungere Hoechst alla diluizione 1:2000 in una soluzione 1x DPBS contenente albumina sierica bovina (BSA) all'1%, allo 0,5% di Tween 20 e allo 0,5% di Triton-X 100. Incubare a RT per 1 ora al buio. Successivamente, lavare con 1x DPBS.
- Trattamento del segmento esterno (POS) dei fotorecettori su iRPE
- Preparazione POS
- Rimuovere il tubo del pellet POS dallo stoccaggio a -80 °C e scongelarlo per una notte a 4 °C in un secchiello per il ghiaccio coperto.
- Preparare il tampone di lavaggio POS mescolando 10 g di saccarosio in 40 ml di H 2 O (ddH2O) a doppia deionizzazione.
- Riscaldare la miscela a 40-50 °C mescolando delicatamente per 15 min. Aggiungere 840 mg di bicarbonato di sodio alla miscela e mescolare riscaldando per 10 minuti.
- Regolare il volume totale del tampone di lavaggio POS a 100 ml con ddH2O e regolare il pH della soluzione a 8,3 con 1 N HCl o 1 N NaOH, se necessario. Filtrare la soluzione di lavaggio utilizzando un filtro da 0,22 μm.
NOTA: il protocollo può essere messo in pausa qui; il tampone di lavaggio POS può essere conservato a 4 °C durante la notte. - Una volta scongelato, sospendere il pellet in 15 ml di tampone di lavaggio POS. Sii delicato durante le sospensioni del pellet per garantire l'integrità del POS. Centrifugare la sospensione POS a 600 x g a 4 °C per 20 minuti, quindi aspirare il surnatante.
- Risospendere il pellet POS in 10 ml del tampone di lavaggio POS.
- Rimuovere un'aliquota da 100 μL del tampone di lavaggio POS + POS (soluzione POS) e diluire in 400 μL di 1x DPBS. Distribuire 50 μL della soluzione POS diluita su una piastra di agar sanguigno e una piastra di agarosio per verificare la presenza di contaminanti batterici e fungini. Preparare controlli positivi per ciascuno e incubare tutte le piastre per 48 ore a 37 °C.
- Eseguire un test qPCR aggiungendo 1 μL di soluzione POS al pozzetto di rilevamento per testare il micoplasma. Per amplificare i frammenti di DNA, eseguire 40 cicli di denaturazione (95°C, 15 s), ricottura e allungamento (60°C, 1 min). I primer avanti e indietro per rilevare il micoplasma nel campione POS sono i seguenti:
Primer anteriore: GGA TTA GAT ACC CTG GTA GTC CAC G
Primer inverso: CGT CAA TTC CTT TAA GTT TCA CTC TTG GC - Misurare la concentrazione POS utilizzando un analizzatore cellulare e aliquote in base alle necessità. Per un pozzetto di 96 piastre con celle RPE, 3 x 106 POS sono sufficienti. Il rapporto desiderato è 10 celle POS/RPE. Conservare le aliquote a 80 °C per un uso futuro.
- Aggiunta di POS alle celle
- Scongelare le fiale di POS in un bagno di ghiaccio.
- Mescolare la quantità calcolata di POS preparato con RPE-MM e trattare le celle con POS una volta al giorno per 7 giorni.
NOTA: Preparare la soluzione POS fresca ogni giorno. - Lavare le cellule con 1x DPBS e quindi fissarle con paraformaldeide al 4% per 20 minuti a RT. Lavare nuovamente con DPBS e conservare i campioni a 4 °C, immersi in 200 μL di DPBS.
- Preparazione POS
5. Colorazione per depositi sub-RPE
- Protocollo di colorazione rosso Nilo
- Dopo la fissazione del PFA, lavare i campioni 3 volte con 1x DPBS.
NOTA: se non utilizzato immediatamente, il protocollo può essere messo in pausa qui, ma i campioni devono essere conservati in una soluzione di azoturo di sodio 1x DPBS + 0,02% a 4 °C. - Per preparare la soluzione madre di rosso Nilo, sciogliere la polvere di rosso del Nilo in acetone a una concentrazione di 3 mg/ml. Incubare per 15 minuti a RT con miscelazione periodica. Filtrare le soluzioni con un filtro da 0,22 μm una o due volte a seconda del livello di precipitato rimanente nella soluzione.
NOTA: proteggere la soluzione madre dalla luce. - Per preparare la soluzione di lavoro, diluire la soluzione madre in un rapporto di 1:500 in 1x DPBS. Aggiungere 200 μL della soluzione di lavoro al campione per 30 minuti a RT su uno shaker e proteggerlo dalla luce.
- Lavare 3 volte con 1x PBS e conservare i campioni a 4 °C, immersi in 200 μL di DPBS.
NOTA: se si esegue un esperimento su transwell invece di una piastra da 96 pozzetti, i campioni possono essere montati su un vetrino con supporti di montaggio, coperti con un vetrino di vetro e sigillati con smalto trasparente. Prestare attenzione a montare il campione con le cellule rivolte verso l'alto.
- Dopo la fissazione del PFA, lavare i campioni 3 volte con 1x DPBS.
- Protocollo di colorazione BODIPY
- Per la soluzione madre, sciogliere BODIPY in dimetilsolfossido anidro (DMSO) per ottenere una concentrazione di 3,8 mM.
- Per i campioni fissi PFA, diluire il brodo BODIPY a 1:300 in 1x DPBS. Aggiungere 200 μL alle cellule e incubare durante la notte su un bilanciere a RT.
- Lavare 3 volte con 1x DPBS e conservare i campioni a 4 °C, immersi in 200 μL di DPBS.
NOTA: se si esegue un esperimento su transwell invece di una piastra da 96 pozzetti, i campioni possono essere montati su un vetrino con supporti di montaggio, coperti con un vetrino di vetro e sigillati con smalto trasparente. Prestare attenzione a montare il campione con le cellule rivolte verso l'alto.
- Protocollo immunocolorante APOE
- Combinare 1x DPBS con albumina sierica bovina (BSA) all'1%, 0,5% Tween 20 e Triton-X 100 allo 0,5% per creare una soluzione tampone.
- Per i campioni fissi PFA, bloccare e permeabilizzare il campione in 200 μL della soluzione tampone per 1 ora a RT.
- Aggiungere l'anticorpo primario APOE diluito a 1:100 nella soluzione tampone e incubare durante la notte a RT.
- Il giorno seguente, lavare i campioni 3 volte con 1x DPBS.
- Aggiungere un anticorpo secondario alla diluizione 1:1000 nella soluzione tampone e aggiungere 200 μL della soluzione alle cellule per 1 ora a RT.
- Lavare 3 volte con 1x DPBS e conservare i campioni a 4 °C, immersi in 200 μL di DPBS.
NOTA: Se si esegue un esperimento su transwell invece di una piastra a 96 pozzetti, i campioni possono essere montati su un vetrino con supporto di montaggio (Fluoromount), coperti con un vetrino di copertura e sigillati con smalto trasparente. Prestare attenzione a montare il campione con le cellule rivolte verso l'alto.
6. Automazione ed elaborazione delle immagini
- Scansione automatica delle immagini
NOTA: in questo studio sono stati utilizzati il microscopio a scansione confocale invertita Zeiss LSM 800 e il software ZEN 3.2 (blue edition). Assicurarsi che la piastra a 96 pozzetti sia riscaldata a RT per un minimo di 60 minuti prima dell'imaging per evitare la deriva del piano focale durante la scansione a causa di un cambiamento nell'indice di rifrazione del mezzo con il cambiamento di temperatura.- Utilizzando un microscopio confocale e un obiettivo 40x, creare un profilo di scansione con i canali fluorescenti appropriati per il marcatore lipidico utilizzato e gli eventuali anticorpi aggiuntivi.
- Usa la casella di controllo Riquadri per configurare l'automazione delle immagini. Per calibrare la piastra a 96 pozzetti, assicurarsi che vengano inserite e selezionate le misure corrette del supporto del campione. Quindi, fai clic sul pulsante Calibra per calibrare la piastra in base alle istruzioni, che richiede l'utilizzo dell'obiettivo 10x.
- Scegliere la vista Configurazione avanzata per selezionare i pozzi appropriati e aggiungere 3 diversi punti di imaging vicino al centro del pozzo utilizzando la funzione Posizioni . Questa operazione può essere eseguita manualmente nella sottoscheda Posizione o in modo casuale utilizzando la scheda Impostazione posizione e selezionando Imposta per operatore. Ripeti per tutti i pozzetti della stessa colorazione.
- Per una messa a fuoco e un posizionamento Z-stack ottimali durante l'automazione, vai alla scheda Strategia di messa a fuoco per selezionare Usa superficie di messa a fuoco/valori Z definiti da Impostazione riquadri. I metodi alternativi possono utilizzare altre strategie di messa a fuoco, ma si consiglia di utilizzare questa impostazione per ottenere risultati più coerenti.
- Nella scheda Riquadri fare clic su Verifica posizioni e impostare manualmente il piano Z centrale per ogni posizione. Le impostazioni nella sottoscheda Opzioni ordineranno l'acquisizione delle immagini, quindi controlla questo prima di iniziare a creare immagini. Per acquisire immagini nell'ordine in cui sono state selezionate le posizioni, deseleziona le caselle di controllo Regioni affiancamento/Posizioni e Pozzi portatori/Contenitore . Selezionare Dividi scene in file separati per facilitare l'elaborazione delle immagini.
- Assicurarsi che la scheda Z-Stack sia impostata su Centro, che venga inserito un intervallo in base alle preferenze dell'utente e che sia selezionato il pulsante Ottimale per impostare l'intervallo di sezione.
- Dopo aver ottimizzato le schede Modalità acquisizione, Canali, Strategia di messa a fuoco, Z-Stack e Riquadri , avviare l'esperimento.
- Elaborazione di immagini
- Utilizzando un metodo di elaborazione delle immagini batch, create proiezioni massime di ogni Z-stack con il metodo Extended Depth of Focus .
- Utilizzando un metodo di elaborazione delle immagini batch, esportare i file di proiezione massimi come immagini TIFF a 16 bit. Impostare la compressione su Nessuno e assicurarsi che i dati originali siano selezionati. L'immagine risultante dovrebbe essere un TIFF in scala di grigi di proiezione massima del solo canale di fluorescenza su cui è espresso il marcatore lipidico.
7. Segmentazione e quantificazione
NOTA: Il programma LipidUNet è stato addestrato su immagini 40x da una piastra da 96 pozzetti. Si consiglia vivamente di utilizzare immagini ottenute utilizzando un obiettivo 40x.
- Installare il software LipidUNet. LipidUNet può essere scaricato dal seguente repository GitHub: https://github.com/RPEGoogleMap/LipidUNet
- Identificare le immagini TIFF che rappresentano Nile Red, Bodipy o APOE e spostarle in una cartella denominata imgs all'interno di una directory denominata Nile_Red, Bodipy o APOE, a seconda del metodo utilizzato.
Nota : le convenzioni di denominazione esatte devono essere utilizzate affinché il programma LipidUNet riconosca le directory. - Aprire il software LipidUNet (Figura 4).
- Nella scheda Predict del software, selezionare la directory pertinente (Nile_Red, Bodipy o APOE) facendo clic sui puntini di sospensione e passando alla directory denominata. Verificare che il programma LipidUNet abbia identificato correttamente le immagini controllando la voce Classe.
- Selezionare una soglia di probabilità per l'algoritmo compresa tra 0,01 e 0,99. Un valore più alto eliminerà più falsi positivi, ma potrebbe causare più falsi negativi, mentre valori più bassi potrebbero introdurre più falsi positivi eliminando più falsi negativi. Il valore predefinito è 0,65 ed è consigliato.
- Fai clic su Prevedi.
NOTA: il software scorrerà automaticamente tutte le immagini e creerà una nuova cartella chiamata predicted_masks nella directory selezionata. - Utilizzare uno strumento di analisi delle maschere per scorrere le maschere generate e fornire un conteggio quantitativo dei depositi lipidici sospesi dalle immagini della maschera.
- Analizzare i dati generati per confrontare le condizioni di trattamento.
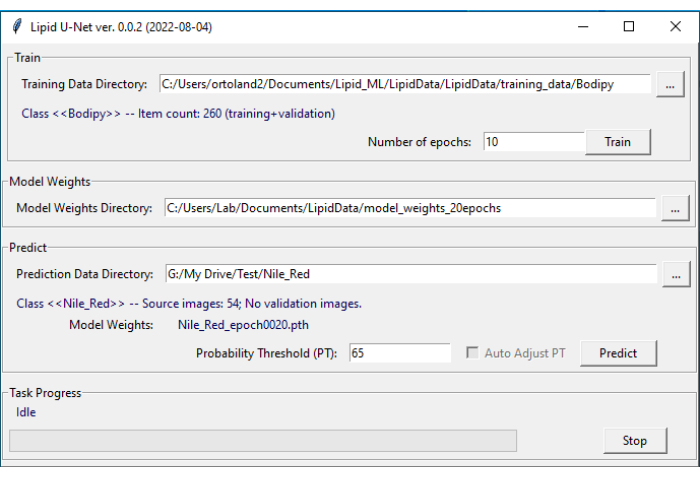
Figura 4: Interfaccia utente di LipidUNet. Il software LipidUNet ha diverse sezioni da selezionare per la directory dei dati di allenamento, dove le immagini dei depositi lipidici sono state identificate correttamente; la directory dei pesi del modello, prodotta dai dati di addestramento; e la directory dei dati di previsione in cui l'utente inserirà le proprie immagini per la segmentazione. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Risultati
Questo protocollo fornisce un flusso di lavoro per identificare i depositi lipidici colorati da Nilo Red, BODIPY e APOE. Il software sviluppato è in grado di identificare e quantificare automaticamente i depositi lipidici e funziona meglio quando il protocollo delineato è ottimizzato. Sono inclusi esempi di RPE differenziato con successo (Figura 3A) e RPE scarsamente differenziato (Figura 3B), poiché la qualità del modello cellulare influisce notevolmente sulla qualità della corretta segmentazione dell'immagine.
Due dei tre marcatori descritti nel protocollo, Nile Red e BODIPY, sono identificati come piccoli punti circolari che sono distintamente luminosi nelle immagini fluorescenti (Figura 5 e Figura 6). Un'immagine "positiva" del protocollo sarebbe un'identificazione appropriata di questi depositi distinti (Figura 5A-D e Figura 5E-H). Un risultato "negativo" mostrerebbe una segmentazione errata dell'immagine scambiando la fluorescenza di fondo come un deposito, a causa di una debole colorazione (Figura 6A-C e Figura 6D-F) o a causa dell'elevata intensità dello sfondo (Figura 6G-I).
I depositi di APOE hanno una varietà di dimensioni e forme, apparendo più ovali o irregolari piuttosto che i depositi circolari di Nilo Red e BODIPY. Questi depositi sono anche meno puntati e l'intensità del segnale può differire tra i depositi a causa delle variazioni nella permeabilizzazione del campione. Una corretta identificazione identificherà ogni deposito, compresi quelli meno saturi (Figura 5I-L), mentre una segmentazione errata non raccoglierà questi depositi (Figura 6J-L). Pertanto, è importante ottimizzare i metodi di colorazione e imaging per evitare variazioni drastiche. Un modo per farlo è prestare particolare attenzione alle fasi di permeabilizzazione del campione durante l'immunocolorazione. Per ottimizzare il segnale fluorescente, le cellule possono essere lisate prima della fissazione e dell'immunocolorazione per l'APOE, il che si traduce in una saturazione uniforme e una migliore segmentazione dei depositi di APOE.
Vengono fornite anche immagini segmentate di cellule maturate su una piattaforma di coltura diversa da una piastra a 96 pozzetti. Il software LipidUNet è stato eseguito su immagini di cellule coltivate su un transwell, e mentre i depositi lipidici sono sogliati, lo sono anche i pori nella membrana transwell (Figura 6M-O). A causa della somiglianza nella forma e nelle dimensioni, il software LipidUNet nella sua forma attuale maschererà indiscriminatamente sia i depositi lipidici che i pori transwell.
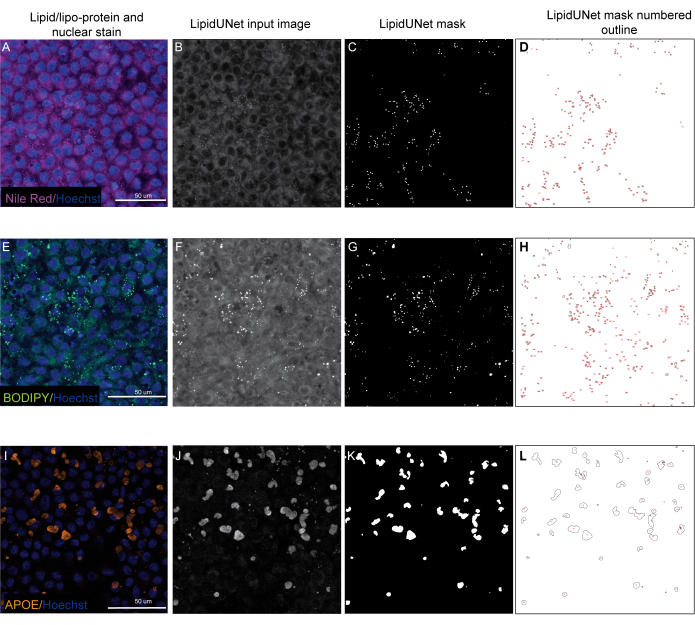
Figura 5: Risultati rappresentativi. (A,E,I) Gli RPE placcati a 96 pozzetti sono colorati con colorazione nucleare di Hoechst (blu) e rosso Nilo (magenta), BODIPY (verde) o APOE (arancione) e sono le proiezioni di massima intensità di una pila Z. (B,F,J) Le immagini di input in scala di grigi per il software LipidUNet dopo l'elaborazione delle immagini. (C,G,K) Maschere generate da LipidUNet, dove tutti i depositi sono identificati correttamente. (D,H,L) I contorni di ogni particella mascherata sono numerati. Queste etichette consentono di collegare ogni particella nell'immagine a una voce nello spreadheet con i dati grezzi. (A-D) mostra la colorazione rosso del Nilo e il software è in grado di riconoscere accuratamente i depositi sullo sfondo nonostante un segnale più debole. (E-H) mostra un forte contrasto tra il segnale BODIPY e lo sfondo, che è l'ideale. LipidUNet identifica correttamente ogni deposito nell'immagine. (I-L) mostra un forte segnale APOE e rappresenta la variabilità della saturazione del segnale che si vede spesso con questa macchia. Tuttavia, la segmentazione delle immagini è in grado di identificare i confini di ciascun deposito APOE. Fare clic qui per visualizzare una versione ingrandita di questa figura.
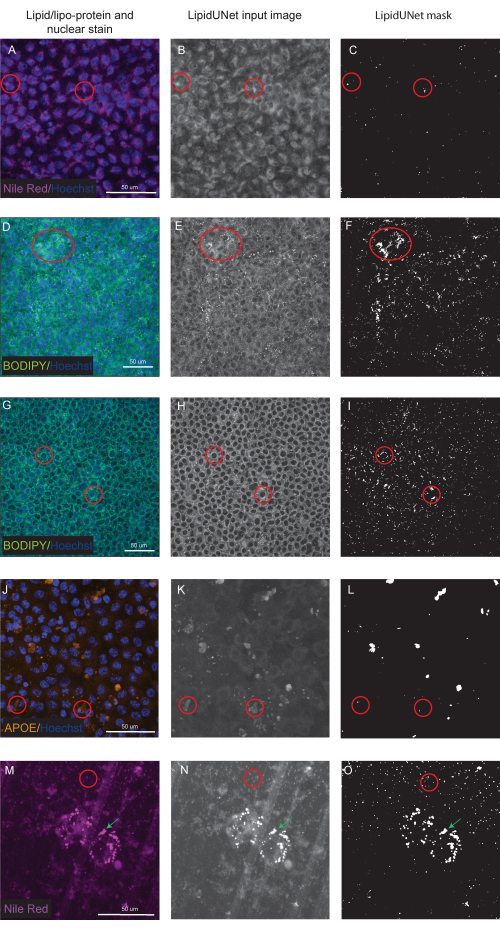
Figura 6: Risultati non ottimali. (A,D,G,J,M) Gli RPE ben placcati a 96 sono colorati con colorazione nucleare Hoechst (blu) e rosso Nilo (magenta), BODIPY (verde) o APOE (arancione) e sono le proiezioni di massima intensità di uno Z-stack. (B,E,H,K,N) Le immagini di input in scala di grigi per il software LipidUNet dopo l'elaborazione delle immagini. (C,F,I,L,O) Le maschere errate generate da LipidUNet. I cerchi rossi indicano dove il software ha identificato erroneamente un deposito lipidico. (A-C) L'elaborazione del rosso Nilo non è corretta perché il software ha identificato la colorazione dello sfondo come un deposito. Questo può accadere più spesso quando c'è uno sfondo alto ma pochi depositi lipidici nell'immagine. Vengono mostrati due esempi di colorazione BODIPY: un'immagine di scarsa qualità dovuta alla debole colorazione BODIPY (D-F) e (G - I) un forte segnale BODIPY con sfondo elevato. In entrambi i casi, il software non è in grado di distinguere piccoli depositi lipidici circolari dall'anello circolare di fondo che circonda il nucleo. Mentre la colorazione e l'imaging dovrebbero essere ottimizzati per evitare questi errori, la versione più recente di LipidUNet è ampiamente migliorata per queste immagini. (J-L) Segmentazione APOE errata. Poiché i depositi sono più variabili per dimensioni e saturazione del segnale, il software ha difficoltà a riconoscere alcuni depositi. (M-O) RPE seminato su un transwell e colorato di rosso Nilo. Una fetta dello Z-stack è mostrata qui con entrambi i depositi lipidici rosso Nilo e pori transwell. Il software non è in grado di distinguere tra i due, come mostrato dal cerchio rosso contenente i pori transwell e dalla freccia verde che punta ai depositi rosso del Nilo. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 7: Confronto tra gli strumenti maschera. (A,B,C) Gli RPE placcati a 96 pozzetti con quantità variabili di deposizione lipidica sono identificati con il rosso del Nilo (rosso). Le immagini sono mascherate utilizzando tre diversi metodi di mascheramento comuni, Find Maxima, Max Entropy e Renyi Entropy, e confrontate con la maschera generata da LipidUNet. L'immagine originale è accompagnata da un conteggio manuale dei depositi lipidici, mentre le maschere mostrano i conteggi previsti da ciascun metodo di segmentazione. Il tasso di errore medio è stato calcolato per ciascun metodo di segmentazione utilizzando la seguente formula: media[(|Conteggio previsto - Conteggio manuale|/Conteggio manuale) x 100]. La maschera generata da LipidUNet identifica più accuratamente i depositi lipidici attraverso le immagini con deposizione variabile rispetto ad altri metodi di mascheramento (tassi di errore medi: 23% LipidUnet, 1164% Trova massimi, 851% Entropia massima, 203% entropia Renyi). Fare clic qui per visualizzare una versione ingrandita di questa figura.
| Componente | Numero di gatto | Stock Conc. | Conc. finale | Ml |
| MEM alfa | 12571-063 | NA | 500 | |
| Supplemento N2 | 17502-048 | NA | 1% | 5 |
| FBS inattivato dal calore | SH30071.03 | NA | 5% | 25 |
| NMEM NEAA | 11140-050 | 10mM | 0,01 mM | 5 |
| Piruvato di sodio | 11360-070 | 100mM | 1mM | 5 |
| Penicillina-streptomicina | 15140-122 | 10000u/mL | 100U/mL | 5 |
| Taurina | T4571 | 50mg/mL | 250ug/mL | 2.5 |
| Idrocortisone | H6909 | 18,1 mg / L | 20ug/L | 0.553 |
| T3 | T5516 | 20ug/L | 0,013ug/L | 0.33 |
| Volume totale, mL | 548.383 | |||
Tabella 1: Composizione del reagente RPE-MM. Un elenco di reagenti e concentrazioni ottimali per RPE-MM.
Discussione
Questo protocollo fornisce un metodo per etichettare, visualizzare e quantificare in modo efficiente i depositi lipidici in modelli di malattia monogenica e poligenica in vitro per le malattie degenerative dell'occhio. Il software basato sull'intelligenza artificiale, LipidUNet, può essere applicato a tre marcatori lipidici comuni, APOE, Nile Red e BODIPY, e fornisce un metodo rapido e automatico per l'analisi che consente alla quantificazione di essere standard e imparziale.
La principale limitazione di LipidUNet è il fatto che il set di dati di addestramento per l'IA era limitato a immagini di ingrandimento 40x di cellule coltivate in una piastra a 96 pozzetti. Come risultato del set di immagini di addestramento, LipidUNet, nella sua forma attuale, è limitato all'analisi di immagini di ingrandimento 40x. Il software può essere utilizzato per analizzare immagini 40x di cellule coltivate su altre superfici di coltura oltre a una piastra da 96 pozzetti, ma è necessario prestare attenzione a esaminare le maschere di output generate per verificare la soglia accurata da parte del software. Saranno necessari più set di immagini (a diversi ingrandimenti) per espandere la portata di quali campioni / immagini può analizzare.
Il protocollo ha diversi passaggi critici. Nella fase del marcatore lipidico, l'utente deve confermare che il composto di etichettatura scelto (BODIPY, APOE, Nilo Red) ha etichettato il proprio campione in modo efficace. Le cellule RPE mature sono spesso fortemente pigmentate, il che può compromettere il segnale fluorescente dell'immunocolorazione anticorpale. Quando il segnale di fluorescenza è debole o quando c'è troppa colorazione di fondo, LipidUNet non è in grado di discernere con precisione le goccioline lipidiche. Per un motivo simile, è necessario utilizzare le impostazioni di acquisizione correttamente selezionate per la fase di imaging automatico del protocollo. Se le immagini acquisite sono di scarsa qualità, LipidUNet farà fatica a mascherare correttamente le immagini e, pertanto, la quantificazione sarà imprecisa (Figura 6A-L). Infine, la post-elaborazione delle immagini è un passo importante, in quanto LipidUNet ha requisiti specifici per il funzionamento del software.
Rispetto ai flussi di lavoro per l'analisi dei lipidi che utilizzano la soglia manuale o alle tecniche che comportano la soglia automatica in software come Fiji, LipidUNet offre una segmentazione imparziale e affidabile tra le immagini con deposizione lipidica variabile, come riflesso da un piccolo tasso di errore nell'identificazione delle particelle lipidiche (Figura 7). Il software consente all'utente di inserire ulteriori immagini di allenamento, consentendo l'analisi di set di immagini oltre a quelli che utilizzano un obiettivo di ingrandimento 40x o anche quelli che utilizzano un marcatore lipidico diverso, come indicato nel protocollo. In futuro, il software sarà addestrato ad analizzare immagini 3D in modo che sia possibile la quantificazione del volume dei depositi lipidici. Le malattie degenerative degli occhi che implicano la deposizione di lipidi come un importante contributo alla patologia sono prevalenti e si prevede che i casi aumenteranno man mano che la popolazione anziana si espande13. Modelli di malattia accurati e strumenti di analisi efficienti, come abbiamo delineato in questo protocollo, consentiranno lo sviluppo di nuovi interventi terapeutici.
Divulgazioni
Nessuna divulgazione.
Riconoscimenti
Si ringrazia il nucleo istologico del National Eye Institute (NEI) per l'utilizzo del sistema confocale Zeiss. Questo lavoro è stato sostenuto dai fondi IRP NEI (numero di sovvenzione ZIA EY000533-04).
Materiali
| Name | Company | Catalog Number | Comments |
| 0.22 µm Steriflip filter system | EMD Millipore | SCGP00525 | |
| 1x Dulbecco's Phosphate Buffered Saline | Gibco | 14190-144 | |
| 3,3',5-Triiodo-L-thyronine | Sigma | T5516 | |
| Albumin Bovine, Fraction V | MP Biomedical | 160069 | |
| Alexa Fluor 555 rabbit anti-goat IgG (H+L) | Invitrogen | A21431 | APOE secondary antibody |
| APOE primary antibody | Millipore Sigma | AB947 | |
| BODIPY 493/503 | Invitrogen | D3922 | Protect from light |
| Complement competent human serum | Millipore Sigma | S1-LITER | |
| CTS N2 Supplement | Life Technologies | A13707-01 | |
| Fetal Bovine Serum | Hyclone | SH30071.03 | |
| Fluoromount-G | SouthernBiotech | 0100-01 | Slide mounting media |
| Glass Cover Slips #1 1/2 22 mm x 22 mm | Electron Microscopy Sciences | 72204-01 | |
| Glass Microscope Slide 25 mm x 75 mm- 1.2 mm Thick | Electron Microscopy Sciences | 71870-01 | |
| Hydrocortisone | Sigma | H0396 | |
| MEM Alpha | Life Technologies | 12571-063 | |
| MEM non-essential Amino Acids | Life Technologies | 11140 | |
| Nile Red | Sigma | 72485-100MG | Protect from light |
| Paraformaldehyde 16% Solution, EM Grade | Electron Microscopy Sciences | 15710 | |
| Penicillin-Strep | Life Technologies | 15140-148 | |
| Phosphate Buffered Saline 10x | Gibco | 70011-044 | |
| Rod Outer Segments (OS) | InVision Bioresources | 98740 | |
| Sodium bicarbonate | Sigma Aldrich | S5761 | |
| Sodium Pyruvate | Life Technologies | 11360-070 | |
| Sucrose | Sigma Aldrich | S1888 | |
| SYBR Green Master Mix | Bio-Rad | 1725274 | |
| Taurine | Sigma | T0625 | |
| Triton X-100 | Sigma | 9002-93-1 | |
| Tween 20 Ultrapure | Affymetrix | 9005-64-5 | |
| Vitronectin | Life Technologies | A14701SA | |
| Y-27632 dihydrochloride | R&D Systems | 1254 |
Riferimenti
- Kanow, M. A., et al. Biochemical adaptations of the retina and retinal pigment epithelium support a metabolic ecosystem in the vertebrate eye. eLife. 6, e28899(2017).
- Adijanto, J., et al. The retinal pigment epithelium utilizes fatty acids for ketogenesis. The Journal of Biological Chemistry. 289 (30), 20570-20582 (2014).
- Farnoodian, M., et al. Cell-autonomous lipid-handling defects in Stargardt iPSC-derived retinal pigment epithelium cells. Stem Cell Reports. 17 (11), 2438-2450 (2022).
- Curcio, C. A. Soft drusen in age-related macular degeneration: Biology and targeting via the oil spill strategies. Investigative Ophthalmology & Visual Science. 59 (4), AMD160-AMD181 (2018).
- Cankova, Z., Huang, J. D., Kruth, H. S., Johnson, M. Passage of low-density lipoproteins through Bruch's membrane and choroid. Experimental Eye Research. 93 (6), 947-955 (2011).
- Curcio, C. A., et al. Esterified and unesterified cholesterol in drusen and basal deposits of eyes with age-related maculopathy. Experimental Eye Research. 81 (6), 731-741 (2005).
- Miyagishima, K. J., et al. AMPK modulation ameliorates dominant disease phenotypes of CTRP5 variant in retinal degeneration. Communications Biology. 4 (1), 1360(2021).
- Sharma, R., et al. Epithelial phenotype restoring drugs suppress macular degeneration phenotypes in an iPSC model. Nature Communications. 12 (1), 7293(2021).
- Farnoodian, M., et al. Cell-autonomous lipid-handling defects in Stargardt iPSC-derived retinal pigment epithelium cells. Stem Cell Reports. 17 (11), 2438-2450 (2022).
- Hallam, D., et al. An induced pluripotent stem cell patient specific model of complement factor H (Y402H) polymorphism displays characteristic features of age-related macular degeneration and indicates a beneficial role for UV light exposure. Stem Cells (Dayton, Ohio). 35 (11), 2305-2320 (2017).
- Sharma, R., Bose, D., Montford, J., Ortolan, D., Bharti, K. Triphasic developmentally guided protocol to generate retinal pigment epithelium from induced pluripotent stem cells. STAR Protocols. 3 (3), 101582(2022).
- Issa, P. C., Barnard, A. R., Herrmann, P., Washington, I., MacLaren, R. E. Rescue of the Stargardt phenotype in Abca4 knockout mice through inhibition of vitamin A dimerization. Proceedings of the National Academy of Sciences of the United States of America. 112 (27), 8415-8420 (2015).
- GBD 2019 Blindness and Vision Impairment Collaborators. Blindness and Vision Impairment Collaborators. Causes of blindness and vision impairment in 2020 and trends over 30 years, and prevalence of avoidable blindness in relation to VISION 2020: the Right to Sight: an analysis for the Global Burden of Disease Study. Lancet Global Health. 9 (2), e144-e160 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon