Method Article
Méthode de caractérisation et de quantification de dépôts lipidiques basée sur l’apprentissage automatique à l’aide d’un épithélium pigmentaire rétinien dérivé d’iPSC
Dans cet article
Résumé
Les maladies oculaires dégénératives qui affectent la couche d’épithélium pigmentaire rétinien de l’œil ont des origines monogéniques et polygéniques. Plusieurs modèles de maladies et une application logicielle, LipidUNet, ont été développés pour étudier les mécanismes de la maladie, ainsi que les interventions thérapeutiques potentielles.
Résumé
L’épithélium pigmentaire rétinien (EPR) est une monocouche de cellules hexagonales située à l’arrière de l’œil. Il nourrit et soutient les photorécepteurs et les capillaires choroïdiens, effectue une phagocytose des segments externes des photorécepteurs (POS) et sécrète des cytokines de manière polarisée pour maintenir l’homéostasie de la rétine externe. L’EPR dysfonctionnelle, causée par des mutations, le vieillissement et des facteurs environnementaux, entraîne la dégénérescence d’autres couches rétiniennes et entraîne une perte de vision. Une caractéristique phénotypique caractéristique de la dégénérescence de l’EPR est les dépôts intra et subcellulaires riches en lipides. Ces dépôts sont un phénotype commun à différentes maladies dégénératives de la rétine. Pour reproduire le phénotype de dépôt lipidique des dégénérescences rétiniennes monogéniques in vitro, un EPR induit dérivé de cellules souches pluripotentes (iRPE) a été généré à partir des fibroblastes des patients. Les lignées cellulaires générées par des patients atteints de Stargardt et de dégénérescence rétinienne tardive (L-ORD) ont été nourries avec POS pendant 7 jours pour reproduire la fonction physiologique RPE, ce qui a provoqué une pathologie induite par la phagocytose POS dans ces maladies. Pour générer un modèle de dégénérescence maculaire liée à l’âge (DMLA), une maladie polygénique associée à une activation alternative du complément, l’iRPE a été mis au défi avec d’autres anaphylatoxines du complément. Les dépôts lipidiques intra et subcellulaires ont été caractérisés à l’aide de rouge de Nil, de bore-dipyrrométène (BODIPY) et d’apolipoprotéine E (APOE). Pour quantifier la densité des dépôts lipidiques, un logiciel basé sur l’apprentissage automatique, LipidUNet, a été développé. Le logiciel a été formé sur des images de projection d’intensité maximale d’iRPE sur des surfaces de culture. À l’avenir, il sera formé pour analyser des images tridimensionnelles (3D) et quantifier le volume de gouttelettes lipidiques. Le logiciel LipidUNet sera une ressource précieuse pour découvrir des médicaments qui diminuent l’accumulation de lipides dans les modèles de maladies.
Introduction
L’épithélium pigmentaire rétinien (EPR) est une monocouche de cellules située à l’arrière de l’œil adjacente aux photorécepteurs rétiniens. L’EPR joue un rôle essentiel dans le maintien d’une bonne vision en fournissant un soutien métabolique et structurel aux photorécepteurs. Les cellules RPE saines sont caractérisées par une morphologie hexagonale distincte. Ils sont reliés par des jonctions serrées, ce qui permet à l’EPR d’agir comme une barrière entre le choriocapillaris situé sur sa face basale et les photorécepteurs situés apicalement. Pour maintenir l’écosystème rétinien, l’EPR transporte des métabolites clés, par exemple le glucose, vers les photorécepteurs de manière à minimiser la consommation de glucose dans l’EPR1. En raison de cette limitation, l’EPR dépend d’autres métabolites pour maintenir leurs besoins métaboliques, y compris les acides gras, que l’EPR convertit en cétones par β-oxydation2. Compte tenu de la propension de l’EPR à utiliser les acides gras, qui sont probablement recyclés à partir de la digestion du segment externe des photorécepteurs (POS), comme source d’énergie, les changements préjudiciables aux voies de traitement des lipides dans l’EPR conduisent souvent à des maladies dégénératives rétiniennes monogéniques et polygéniques3.
La dégénérescence maculaire liée à l’âge (DMLA), une maladie oculaire dégénérative polygénique qui provoque la dégénérescence de l’EPR, a également été liée à une autophagie aberrante et au métabolisme des lipides dans la monocouche de l’EPR. L’échec d’une monocouche RPE dysfonctionnelle à traiter le POS et à remplir d’autres fonctions critiques conduit à des dépôts extracellulaires (sous-RPE) appelés dépôts linéaires basaux (BLinD) situés entre l’EPR et la membrane de Bruch - une caractéristique des pathologies de la DMLA. Les principaux composants de BLinD comprennent les lipoprotéines, dont la plus abondante est l’apolipoprotéine E (APOE)4. L’accumulation de couches minces de BLinD peut conduire à des drusen mous, qui sont reconnus comme un symptôme clinique de la DMLA 5,6.
Plusieurs groupes ont montré que les modèles de maladies in vitro dérivés de cellules souches qui causent un dysfonctionnement de l’EPR présentent une accumulation lipidique inférieureà l’EPR 7,8,9. Hallam et coll. (2017) ont généré une EPR induite dérivée de cellules souches pluripotentes (iRPE) chez des patients présentant un risque élevé de DMLA en raison d’un polymorphisme du gène ZH. L’iRPE a montré une accumulation de drusen, marquée par l’APOE, et l’EPR à haut risque a accumulé des dépôts plus importants que l’iRPE générée par les patients à faible risque10.
Pour créer un modèle in vitro qui récapitule les caractéristiques cellulaires de la DMLA, telles que les gouttelettes lipidiques et le dépôt de drusen, des lignées iRPE générées à partir d’échantillons de sang de patients ont été établies à l’aide d’un protocole guidé par le développement11 publié précédemment. Les iRPE ont été soumis au sérum humain à complément compétent (CC-HS), une solution contenant des anaphylatoxines qui imitent une cause possible de DMLA : augmentation de la signalisation alternativedu complément 8. Le dépôt cellulaire et sous-cellulaire résultant des dépôts lipidiques a été mesuré à l’aide de marqueurs lipidiques et lipoprotéiques couramment utilisés, APOE, Nile Red et BODIPY. Grâce à ces marqueurs, il a été démontré que la signalisation activée du complément via CC-HS exacerbait l’accumulation de lipides dans les cellules iRPE8.
Pour développer un modèle de maladie pour une maladie dégénérative rétinienne monogénique, des lignées iRPE ont été développées à partir de patients atteints de la maladie de Stargardt, une maladie causée par des mutations du gène ABCA4 dans l’EPR. Il a déjà été démontré que lorsque ABCA4 est éliminé, la lipofuscine A2E, un dépôt intracellulaire connu pour contenir des niveaux élevés de phospholipides et de produits de peroxydation lipidiques dépendants de la lumière, s’accumule à l’intérieur de l’EPR12. Les lignes knockout ABCA4 ont été développées parallèlement aux lignes de patients, et les deux ont été soumises à une alimentation POS. L’iRPE de Stargardt a démontré une pathologie induite par la phagocytose POS, présentant une accumulation accrue de lipides quantifiée par la coloration BODIPY. L’EPR dérivé des CSPi ABCA4 KO a été soumis à un traitement CC-HS; la quantification du signal BODIPY a également montré un défaut dans la manipulation des lipides dans le modèle de la maladie de Stargardt9.
Compte tenu de la prévalence de ces maladies et de la nécessité de mettre en place des traitements efficaces, ainsi que des modèles de maladies pertinents décrits ci-dessus, il est nécessaire d’établir des méthodes robustes pour quantifier l’efficacité des traitements potentiels. Pour quantifier les dépôts lipidiques de manière objective, automatisée et standardisée, un logiciel basé sur l’apprentissage automatique, LipidUNet, a été créé afin que, lorsqu’il est associé à des outils d’analyse de masque, les dépôts lipidiques puissent être identifiés rapidement et efficacement à l’aide des marqueurs communs Nile Red, BODIPY et APOE. Les statistiques sommaires obtenues à l’aide de ce pipeline d’analyse peuvent ensuite être analysées et affichées graphiquement, ce qui permet de comparer facilement les conditions de traitement. Le schéma du protocole est illustré à la figure 1.
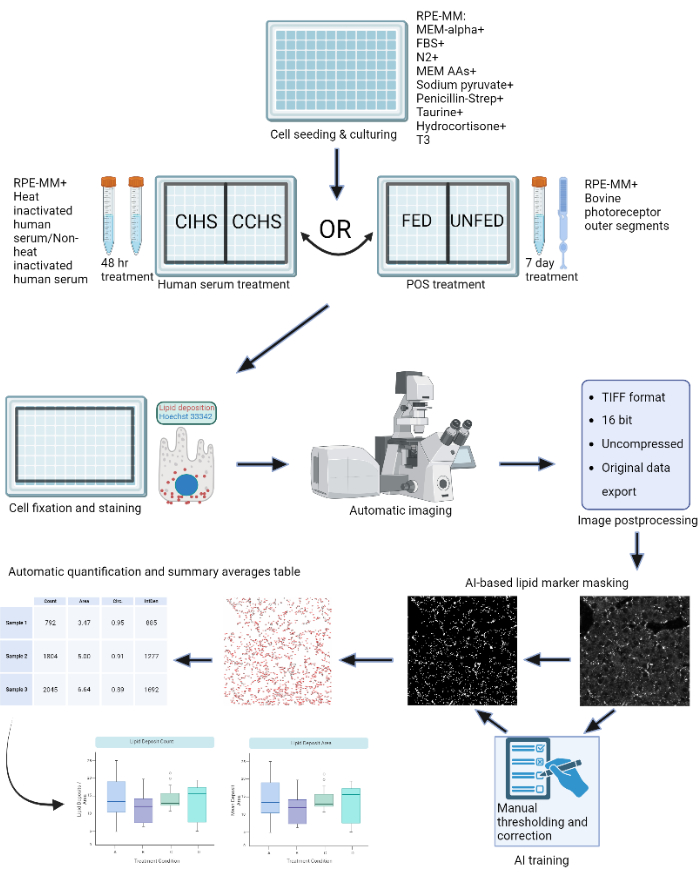
Figure 1 : Schéma du protocole : Les cellules EPR sont cultivées sur une plaque de 96 puits et mises au défi avec du sérum humain actif ou des segments externes bovins purifiés pour modéliser différents types de dégénérescence rétinienne in vitro. Les cellules EPR sont fixées et colorées pour les dépôts de lipoprotéines avec Nile Red, BODIPY et APOE. Un microscope confocal est utilisé pour acquérir des piles Z de particules lipidiques marquées par fluorescence, qui sont ensuite transformées en projections d’intensité maximale 2D. Un algorithme d’apprentissage automatique a été formé pour reconnaître et segmenter correctement les particules de lipoprotéines. Des tableaux récapitulatifs contenant le nombre de particules et diverses mesures de forme sont générés et peuvent être utilisés pour le traçage ultérieur et l’analyse statistique. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
Toutes les étapes du protocole respectent les lignes directrices établies par le comité d’éthique de la recherche humaine des NIH. Le travail sur les cellules souches et la collecte d’échantillons de patients ont été approuvés par le Combined NeuroScience Institutional Review Board (CNS IRB) sous l’égide de l’Office of Human Research Protection (OHRP), NIH, conformément aux directives 45 CFR 46 du gouvernement américain. Les échantillons de patients ont été prélevés à l’aide du formulaire de consentement approuvé par la CISR conformément aux critères fixés par la Déclaration d’Helsinki sous le numéro de protocole NCT01432847 (https://clinicaltrials.gov/ct2/show/NCT01432847?cond=NCT01432847&draw=2&rank=1).
1. Génération iRPE
- Générer de l’iRPE à partir de CSPi dérivée du sang des patients en suivant le protocole publié par Sharma et coll., 202211 (Figure 2 et Figure 3).
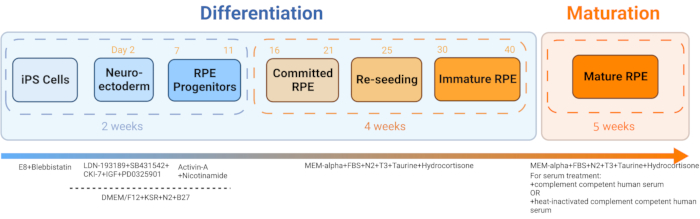
Figure 2 : Schéma de la différenciation et de la maturation de l’iRPE. Pour générer l’iRPE, un protocole de différenciation établi a été suivi et les cellules ont été autorisées à mûrir pendant 5 semaines. La culture cellulaire résultante agit comme un modèle in vitro qui peut être manipulé avec divers traitements pour imiter le dysfonctionnement de l’EPR dans des maladies comme la DMLA et la maladie de Stargardt. Veuillez cliquer ici pour voir une version agrandie de cette figure.
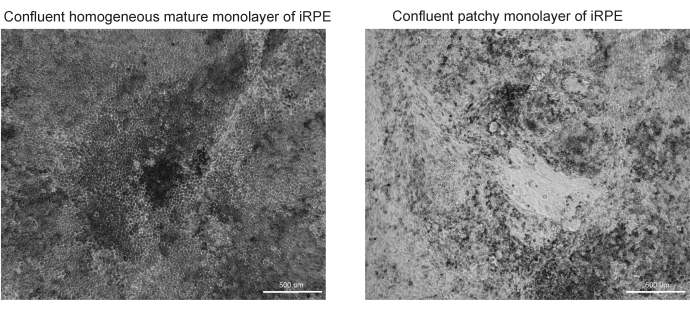
Figure 3 : Images représentatives de la différenciation et de la maturation réussies et infructueuses de l’EPR. Deux images en fond clair à un grossissement de 10x de TJP1 RPE sont affichées au jour 42 du protocole iRPE. (A) Une différenciation et une maturation réussies montreront une EPR confluente avec pigmentation et morphologie polygonale. (B) Une différenciation et une maturation infructueuses montreront des amas de cellules mourantes, comme indiqué ici. Veuillez cliquer ici pour voir une version agrandie de cette figure.
2. Préparation des supports de maintenance RPE (RPE-MM)
- Décongeler le supplément de N2 à 4 °C pendant la nuit. Décongeler tous les autres réactifs à température ambiante (RT).
- Dans des conditions stériles, ajouter les réactifs énumérés au tableau 1 aux facteurs de dilution indiqués, conformément au protocole établi par Sharma et coll., 202211.
- Bien mélanger le média et le filtrer à l’aide d’une unité de filtration de 0,22 μm.
REMARQUE: Le support peut être utilisé dans les 2 semaines s’il est conservé à 4 ° C.
3. Ensemencement de plaques à 96 puits
- Décongeler une partie aliquote de vitronectine à TA pendant 3-5 minutes ou jusqu’à ce que la glace soit complètement fondue.
- Diluer la vitronectine avec 1x la solution saline tamponnée au phosphate (DPBS) de Dulbecco pour obtenir la solution de travail souhaitée en utilisant une dilution 1:200 (vitronectine: DPBS). Pour une plaque de 96 puits, enduisez chaque puits de 200 μL de la solution de travail.
- Combiner l’inhibiteur de ROCK décongelé (chlorhydrate Y-27632) avec le RPE-MM à une dilution de 1:1000 pour obtenir une concentration finale de 10 μM. C’est le milieu de placage pour les cellules EPR.
- Décongeler le flacon d’iRPE à l’aide d’un système automatisé de décongélation cellulaire et transférer la suspension cellulaire iRPE dans un tube de 50 mL.
- Diluer la suspension cellulaire avec le média de placage à une dilution de 1:10. Centrifuger le tube à 400 x g pendant 5 min.
- Aspirer soigneusement le surnageant et remettre les cellules en suspension dans 10 ml du média de placage.
- Mélanger 400 μL du média de placage avec 100 μL de la solution cellulaire remise en suspension pour le comptage cellulaire. Utilisez cette partie aliquote pour déterminer la concentration cellulaire viable de la suspension cellulaire à l’aide d’un compteur de viabilité cellulaire.
- Diluer la suspension cellulaire avec le média de placage jusqu’à une concentration finale de 60 000 cellules/mL.
- Aspirer complètement la solution de revêtement de vitronectine de la plaque de 96 puits et distribuer 200 μL de la suspension cellulaire dans chaque puits. Il y aura environ 12 000 cellules/puits ou ~200 cellules/mm2.
- Incuber les plaques cellulaires ensemencées pendant 48 h à 37 °C et 5% de CO2. Après 48 h, changer le support en RPE-MM sans supplément d’inhibiteur de ROCK. Changez le support tous les 2-3 jours pendant la période de maturation de 5 semaines.
4. Modèles de maladies in vitro
- Complément de traitement au sérum humain compétent (CC-HS)
- Décongeler le sérum compétent du complément humain à 4 °C pendant la nuit.
- Préparer le CC-HS et compléter les milieux de sérum humain incompétent (CI-HS).
- Pour préparer des milieux CC-HS à 5 %, mélanger le sérum humain compétent du complément décongelé avec le RPE-MM à une dilution de 1:20. Filtrer la solution à travers un filtre à média de 0,22 μm avant utilisation.
- Pour préparer un complément à 5 % de milieu de sérum humain incompétent (CI-HS), inactiver d’abord le CC-HS dans un bain-marie à 57 °C pendant 30 minutes, puis mélanger avec le milieu de culture à une dilution de 1:20. Filtrer la solution à travers un filtre à média de 0,22 μm avant utilisation.
- Sérum traite les cellules avec 200 μL de milieux CC-HS à 5% ou CI-HS à 5% pour un temps d’incubation total de 48 heures, rafraîchissant le milieu après 24 heures.
- Lavez les cellules avec 1x DPBS et fixez-les avec 4% de paraformaldéhyde pendant 20 min à TA. Lavez à nouveau avec 1x DPBS et conservez les échantillons à 4 °C, immergés dans 200 μL de DPBS.
- FACULTATIF: Si vous le souhaitez, lyser les cellules de la plaque pour ne montrer que le dépôt lipidique sous-RPE.
- Pour lyser les cellules et ne laisser que des dépôts lipidiques, retirez le média et ajoutez 200 μL d’eau désionisée à chaque puits.
- Incuber pendant 10-15 min, pipeter de haut en bas jusqu’à ce que les cellules soient enlevées. Laver à nouveau avec 200 μL d’eau désionisée et fixer immédiatement les cellules avec 4% de paraformaldéhyde.
- Confirmer l’efficacité de l’élimination cellulaire avec la coloration nucléaire à l’aide de Hoechst. Ajouter Hoechst à dilution 1:2000 dans une solution 1x de DPBS contenant 1 % d’albumine sérique bovine (BSA), 0,5 % de Tween 20 et 0,5 % de Triton-X 100. Incuber à TA pendant 1 h dans l’obscurité. Ensuite, laver avec 1x DPBS.
- Traitement du segment externe du photorécepteur (POS) sur iRPE
- Préparation POS
- Retirer le tube de granulés du PDV de l’entreposage à -80 °C et décongeler pendant la nuit à 4 °C dans un seau à glace couvert.
- Préparer le tampon de lavage POS en mélangeant 10 g de saccharose dans 40 ml deH2Odouble désionisé (ddH2O).
- Chauffer le mélange à 40-50 °C en agitant doucement pendant 15 min. Ajouter 840 mg de bicarbonate de sodium au mélange et remuer en chauffant pendant 10 min.
- Ajuster le volume total du tampon de lavage POS à 100 mL avec ddH2O et ajuster le pH de la solution à 8,3 avec 1 N HCl ou 1 N NaOH, au besoin. Filtrer la solution de lavage à l’aide d’un filtre de 0,22 μm.
REMARQUE: Le protocole peut être mis en pause ici; le tampon de lavage POS peut être stocké à 4 °C pendant la nuit. - Une fois décongelé, suspendre la pastille dans 15 ml de tampon de lavage POS. Soyez doux pendant les suspensions de granulés pour assurer l’intégrité du point de vente. Centrifuger la suspension POS à 600 x g à 4 °C pendant 20 min puis aspirer le surnageant.
- Remettez en suspension la pastille POS dans 10 ml du tampon de lavage POS.
- Retirer une aliquote de 100 μL du tampon de lavage POS + POS (solution POS) et diluer dans 400 μL de 1x DPBS. Étaler 50 μL de la solution PDV diluée sur une plaque de gélose au sang et une plaque d’agarose pour vérifier la présence de contaminants bactériens et fongiques. Préparer des témoins positifs pour chacun et incuber toutes les plaques pendant 48 h à 37 °C.
- Effectuer un test qPCR en ajoutant 1 μL de solution POS au puits de détection pour tester les mycoplasmes. Pour amplifier les fragments d’ADN, effectuer 40 cycles de dénaturation (95 °C, 15 s), de recuit et d’allongement (60 °C, 1 min). Les amorces avant et arrière pour la détection des mycoplasmes dans l’échantillon POS sont les suivantes:
Introduction avant: GGA TTA GAT ACC CTG GTA GTC CAC G
Introduction inversée: CGT CAA TTC CTT TAA GTT TCA CTC TTG GC - Mesurer la concentration de PDV à l’aide d’un analyseur de cellules et d’une partie aliquote selon les besoins. Pour un puits de 96 plaques de puits avec cellules EPR, 3 x 106 POS suffisent. Le ratio souhaité est de 10 cellules POS/RPE. Conserver les aliquotes à 80 °C en vue d’une utilisation ultérieure.
- Ajout de POS aux cellules
- Décongeler des flacons de POS dans un bain de glace.
- Mélanger la quantité calculée de POS préparé avec RPE-MM et traiter les cellules avec POS une fois par jour pendant 7 jours.
REMARQUE: Préparez la solution POS fraîche tous les jours. - Lavez les cellules avec 1x DPBS, puis fixez-les avec 4% de paraformaldéhyde pendant 20 min à TA. Laver à nouveau avec DPBS et conserver les échantillons à 4 °C, immergés dans 200 μL de DPBS.
- Préparation POS
5. Coloration pour les dépôts sous-RPE
- Protocole de coloration rouge du Nil
- Après fixation PFA, laver les échantillons 3 fois avec 1x DPBS.
REMARQUE: S’il n’est pas utilisé immédiatement, le protocole peut être interrompu ici, mais les échantillons doivent être conservés dans une solution 1x DPBS + 0,02% d’azoture de sodium à 4 ° C. - Pour préparer la solution mère de rouge du Nil, dissoudre la poudre de rouge du Nil dans de l’acétone à une concentration de 3 mg/mL. Incuber pendant 15 min à TA avec mélange périodique. Filtrer les solutions avec un filtre de 0,22 μm une ou deux fois selon le niveau de précipité restant dans la solution.
REMARQUE: Protéger la solution mère de la lumière. - Pour préparer la solution de travail, diluer la solution mère dans un rapport de 1:500 dans 1x DPBS. Ajouter 200 μL de la solution de travail à l’échantillon pendant 30 min à TA sur un agitateur et le protéger de la lumière.
- Laver 3 fois avec 1x PBS et conserver les échantillons à 4 °C, immergés dans 200 μL de DPBS.
REMARQUE: Si vous menez une expérience sur des transpuits au lieu d’une plaque de 96 puits, les échantillons peuvent être montés sur une lame avec un support de montage, recouverte d’une lamelle de verre et scellée avec du vernis à ongles transparent. Il faut prendre soin de monter l’échantillon avec les cellules tournées vers le haut.
- Après fixation PFA, laver les échantillons 3 fois avec 1x DPBS.
- Protocole de coloration BODIPY
- Pour la solution mère, dissoudre BODIPY dans du diméthylsulfoxyde anhydre (DMSO) pour obtenir une concentration mère de 3,8 mM.
- Pour les échantillons fixes PFA, diluer le stock BODIPY à 1:300 dans 1x DPBS. Ajouter 200 μL aux cellules et incuber pendant une nuit sur une bascule à TA.
- Laver 3 fois avec 1x DPBS et conserver les échantillons à 4 °C, immergés dans 200 μL de DPBS.
REMARQUE: Si vous menez une expérience sur des transpuits au lieu d’une plaque de 96 puits, les échantillons peuvent être montés sur une lame avec un support de montage, recouverte d’une lamelle de verre et scellée avec du vernis à ongles transparent. Il faut prendre soin de monter l’échantillon avec les cellules tournées vers le haut.
- Protocole d’immunomarquage APOE
- Combinez 1x DPBS avec 1% d’albumine sérique bovine (BSA), 0,5% Tween 20 et 0,5% Triton-X 100 pour créer une solution tampon.
- Pour les échantillons fixés de PFA, bloquer et perméabiliser l’échantillon dans 200 μL de la solution tampon pendant 1 h à TA.
- Ajouter l’anticorps primaire APOE dilué à 1:100 dans la solution tampon et incuber pendant une nuit à TA.
- Le lendemain, lavez les échantillons 3 fois avec 1x DPBS.
- Ajouter un anticorps secondaire à 1:1000 de dilution dans la solution tampon et ajouter 200 μL de la solution aux cellules pendant 1 h à TA.
- Laver 3 fois avec 1x DPBS et conserver les échantillons à 4 °C, immergés dans 200 μL de DPBS.
REMARQUE: Si vous effectuez une expérience sur des puits au lieu d’une plaque de 96 puits, les échantillons peuvent être montés sur une lame avec support de montage (Fluoromount), recouverte d’un couvercle en verre et scellée avec du vernis à ongles transparent. Il faut prendre soin de monter l’échantillon avec les cellules tournées vers le haut.
6. Automatisation et traitement des images
- Numérisation automatisée d’images
REMARQUE: Le microscope confocale inversé Zeiss LSM 800 et le logiciel ZEN 3.2 (édition bleue) ont été utilisés dans cette étude. Assurez-vous que la plaque de 96 puits est chauffée à RT pendant au moins 60 minutes avant l’imagerie afin d’éviter la dérive du plan focal pendant le balayage due à un changement de l’indice de réfraction du milieu avec changement de température.- À l’aide d’un microscope confocal et d’un objectif 40x, créer un profil de balayage avec les canaux fluorescents appropriés pour le marqueur lipidique utilisé et tout anticorps supplémentaire.
- Utilisez la case à cocher Vignettes pour configurer l’automatisation des images. Pour calibrer la plaque à 96 puits, assurez-vous que les mesures correctes du support d’échantillon sont saisies et sélectionnées. Cliquez ensuite sur le bouton Calibrer pour calibrer la plaque conformément aux instructions, ce qui nécessite l’utilisation de l’objectif 10x.
- Choisissez la vue Configuration avancée pour sélectionner les puits appropriés et ajouter 3 points d’imagerie différents près du centre du puits à l’aide de la fonction Positions . Cela peut être fait manuellement sous le sous-onglet Position ou de manière aléatoire en utilisant l’onglet Configuration de la position et en sélectionnant Configuration par transporteur. Répétez pour tous les puits de même coloration.
- Pour une mise au point optimale et un positionnement de la pile Z pendant l’automatisation, accédez à l’onglet Stratégie de mise au point pour sélectionner Utiliser les valeurs de surface de mise au point/Z définies par la configuration des vignettes. Les méthodes alternatives peuvent utiliser d’autres stratégies de concentration, mais il est recommandé d’utiliser ce paramètre pour obtenir les résultats les plus cohérents.
- Sous l’onglet Vignettes , cliquez sur Vérifier les positions et définissez manuellement le plan Z central pour chaque position. Les paramètres du sous-onglet Options ordonneront l’acquisition des images, alors vérifiez-le avant de commencer à imager. Pour acquérir des images dans l’ordre dans lequel les positions ont été sélectionnées, décochez les cases Régions/Positions de vignette et Puits porteurs/Conteneur . Sélectionnez Diviser les scènes en fichiers séparés pour faciliter le traitement de l’image.
- Assurez-vous que l’onglet Z-Stack est défini sur Centrer, qu’une plage est saisie dans les préférences de l’utilisateur et que le bouton Optimal est sélectionné pour définir l’intervalle de tranche.
- Après avoir optimisé les onglets Mode d’acquisition, Canaux, Stratégie de mise au point, Z-Stack et Tuiles , démarrez l’expérience.
- Traitement d'images
- À l’aide d’une méthode de traitement d’image par lots, créez des projections maximales de chaque pile Z avec la méthode de profondeur de champ étendue .
- À l’aide d’une méthode de traitement d’image par lots, exportez les fichiers de projection maximale sous forme d’images TIFF 16 bits. Définissez la compression sur Aucun et assurez-vous que Données d’origine est cochée. L’image résultante doit être un TIFF de projection maximale en niveaux de gris du seul canal de fluorescence sur lequel le marqueur lipidique est exprimé.
7. Segmentation et quantification
REMARQUE: Le programme LipidUNet a été formé sur des images 40x provenant d’une plaque de 96 puits. Il est fortement recommandé d’utiliser des images obtenues à l’aide d’un objectif 40x.
- Installez le logiciel LipidUNet. LipidUNet peut être téléchargé à partir du référentiel GitHub suivant : https://github.com/RPEGoogleMap/LipidUNet
- Identifiez les images TIFF représentant Nile Red, Bodipy ou APOE et déplacez-les dans un dossier nommé imgs dans un répertoire nommé Nile_Red, Bodipy ou APOE, selon la méthode utilisée.
Remarque : Les conventions d’affectation de noms exactes doivent être utilisées pour que le programme LipidUNet reconnaisse les répertoires. - Ouvrez le logiciel LipidUNet (Figure 4).
- Dans l’onglet Prédiction du logiciel, sélectionnez le répertoire approprié (Nile_Red, Bodipy ou APOE) en cliquant sur les points de suspension et en naviguant vers le répertoire nommé. Vérifiez que le programme LipidUNet a correctement identifié les images en vérifiant l’entrée Classe.
- Sélectionnez un seuil de probabilité pour l’algorithme compris entre 0,01 et 0,99. Une valeur plus élevée éliminera plus de faux positifs, mais pourrait causer plus de faux négatifs, et des valeurs plus faibles pourraient introduire plus de faux positifs tout en éliminant plus de faux négatifs. Une valeur par défaut de 0,65 est recommandée et est recommandée.
- Cliquez sur Prédire.
REMARQUE: Le logiciel parcourra automatiquement toutes les images et créera un nouveau dossier appelé predicted_masks dans le répertoire sélectionné. - Utilisez un outil d’analyse de masque pour parcourir les masques générés et fournir un décompte quantitatif des dépôts lipidiques seuillés à partir des images de masque.
- Analyser les données de comptage générées pour comparer les conditions de traitement.
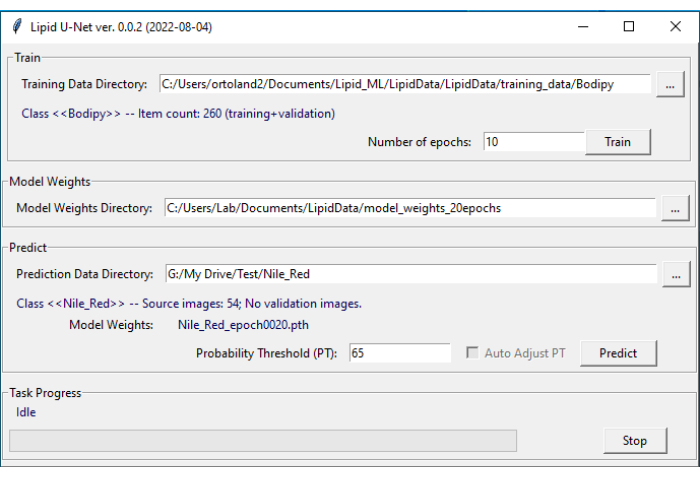
Figure 4 : Interface utilisateur de LipidUNet. Le logiciel LipidUNet a différentes sections à sélectionner pour le répertoire de données d’entraînement, où les images des dépôts lipidiques ont été identifiées correctement; le répertoire des poids des modèles, qui est produit à partir des données d’entraînement; et le répertoire de données de prédiction dans lequel l’utilisateur entrera ses images pour la segmentation. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Résultats
Ce protocole fournit un flux de travail pour identifier les dépôts lipidiques colorés par Nile Red, BODIPY et APOE. Le logiciel développé peut identifier et quantifier automatiquement les dépôts lipidiques et fonctionne mieux lorsque le protocole décrit est optimisé. On y trouve des exemples de RPE différencié avec succès (Figure 3A) et d’EPR peu différencié (Figure 3B), car la qualité du modèle cellulaire a un impact considérable sur la qualité de la segmentation correcte de l’image.
Deux des trois marqueurs décrits dans le protocole, Nile Red et BODIPY, sont identifiés comme de petits points circulaires qui sont nettement lumineux dans les images fluorescentes (Figure 5 et Figure 6). Une image « positive » du protocole serait une identification appropriée de ces dépôts distincts (figure 5A-D et figure 5E-H). Un résultat « négatif » montrerait une segmentation incorrecte de l’image en confondant la fluorescence de fond avec un dépôt, soit en raison d’une faible coloration (Figure 6A-C et Figure 6D-F), soit en raison d’une intensité de fond élevée (Figure 6G-I).
Les dépôts d’APOE ont une variété de tailles et de formes, apparaissant plus ovales ou irrégulières que les dépôts circulaires de Nile Red et BODIPY. Ces dépôts sont également moins ponctués, et l’intensité du signal peut varier d’un dépôt à l’autre en raison des variations de perméabilisation de l’échantillon. Une identification correcte permettra d’identifier chaque gisement, y compris ceux qui sont moins saturés (Figure 5I-L), tandis qu’une segmentation incorrecte ne détectera pas ces dépôts (Figure 6J-L). Par conséquent, il est important d’optimiser les méthodes de coloration et d’imagerie pour éviter les variations drastiques. Une façon de le faire est de porter une attention particulière aux étapes de perméabilisation de l’échantillon pendant l’immunomarquage. Pour optimiser le signal fluorescent, les cellules peuvent être lysées avant la fixation et l’immunomarquage pour l’APOE, ce qui entraîne une saturation uniforme et une meilleure segmentation des dépôts APOE.
Des images segmentées de cellules mûries sur une plate-forme de culture autre qu’une plaque de 96 puits sont également fournies. Le logiciel LipidUNet a été exécuté sur des images de cellules cultivées sur un transwell, et bien que les dépôts lipidiques soient seuillés, il en va de même pour les pores de la membrane transwell (Figure 6M-O). En raison de la similitude de forme et de taille, le logiciel LipidUNet dans sa forme actuelle masquera à la fois les dépôts lipidiques et les pores transwell sans discernement.
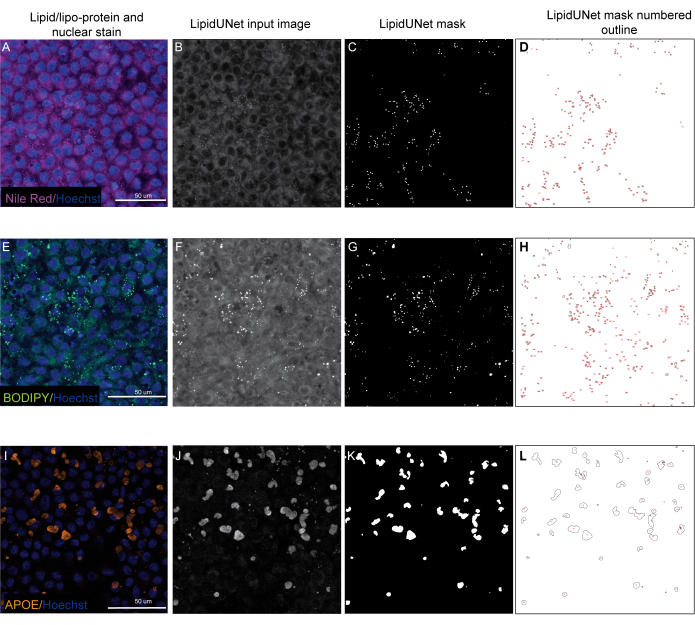
Figure 5 : Résultats représentatifs. (A, E, I) Les EPR plaqués 96 puits sont colorés avec une coloration nucléaire Hoechst (bleu) et soit rouge du Nil (magenta), BODIPY (vert) ou APOE (orange) et sont les projections d’intensité maximale d’une pile Z. (B, F, J) Les images d’entrée en niveaux de gris pour le logiciel LipidUNet après traitement d’image. (C,G,K) Masques générés par LipidUNet, où tous les dépôts sont identifiés correctement. (D,H,L) Les contours de chaque particule masquée sont numérotés. Ces étiquettes permettent de connecter chaque particule de l’image à une entrée dans le spread heet avec les données brutes. (A-D) montre la coloration rouge du Nil, et le logiciel est capable de reconnaître les dépôts sur l’arrière-plan avec précision malgré un signal plus faible. (E-H) montre un fort contraste entre le signal BODIPY et l’arrière-plan, ce qui est idéal. LipidUNet identifie correctement chaque dépôt dans l’image. (I-L) montre un signal APOE fort et représente la variabilité de la saturation du signal qui est souvent observée avec cette coloration. Néanmoins, la segmentation des images est capable d’identifier les limites de chaque gisement APOE. Veuillez cliquer ici pour voir une version agrandie de cette figure.
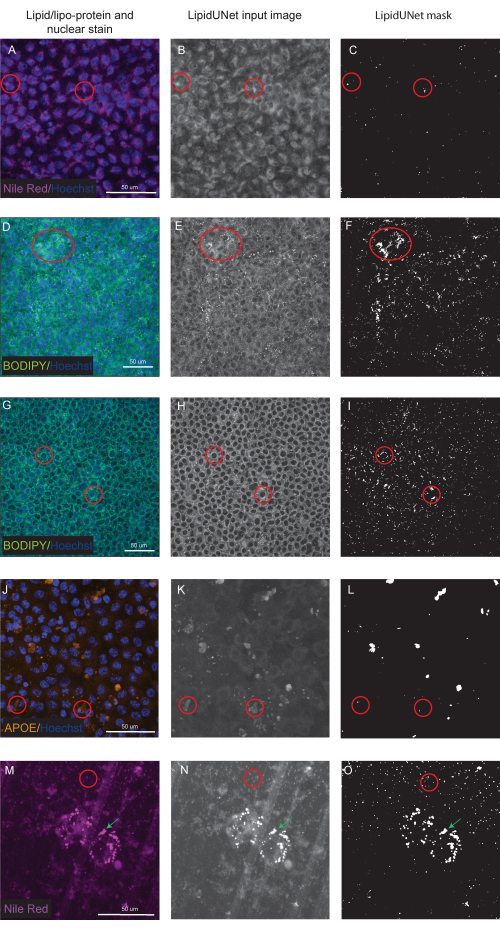
Figure 6 : Résultats sous-optimaux. (A, D, G, J, M) Les EPR à 96 puits sont colorés avec une coloration nucléaire Hoechst (bleu) et un rouge du Nil (magenta), BODIPY (vert) ou APOE (orange) et sont les projections d’intensité maximale d’une pile Z. (B,E,H,K,N) Les images d’entrée en niveaux de gris pour le logiciel LipidUNet après traitement d’image. (C,F,I,L,O) Les masques incorrects générés par LipidUNet. Les cercles rouges indiquent où le logiciel a mal identifié un dépôt lipidique. (A-C) Le traitement du rouge du Nil est incorrect car le logiciel a identifié la coloration de fond comme un dépôt. Cela peut se produire plus souvent lorsqu’il y a un arrière-plan élevé mais peu de dépôts lipidiques dans l’image. Deux exemples de coloration BODIPY sont montrés : une image de mauvaise qualité due à (D-F) une faible coloration BODIPY et (G - I) un signal BODIPY fort avec un arrière-plan élevé. Dans les deux cas, le logiciel est incapable de distinguer les petits dépôts lipidiques circulaires de l’anneau circulaire de fond entourant le noyau. Bien que la coloration et l’imagerie doivent être optimisées pour éviter ces erreurs, la version la plus récente de LipidUNet est largement améliorée pour ces images. (J-L) Segmentation APOE incorrecte. Comme les dépôts sont plus variables en taille et en saturation du signal, le logiciel a du mal à reconnaître certains dépôts. (M-O) RPE ensemencé sur un transwell et taché de rouge du Nil. Une tranche de la pile Z est montrée ici avec à la fois des dépôts lipidiques rouge du Nil et des pores transwell. Le logiciel est incapable de faire la distinction entre les deux, comme le montre le cercle rouge contenant les pores transwell et la flèche verte pointant vers les dépôts rouge du Nil. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 7 : Comparaison des outils de masque. (A, B, C) L’EPR plaqué 96 puits avec des quantités variables de dépôts lipidiques est identifié avec le rouge du Nil (rouge). Les images sont masquées à l’aide de trois méthodes de masquage courantes différentes, Find Maxima, Max Entropy et Renyi Entropy, et comparées au masque généré par LipidUNet. L’image originale est accompagnée d’un comptage manuel des dépôts lipidiques, tandis que les masques affichent les comptages prédits par chaque méthode de segmentation. Le taux d’erreur moyen a été calculé pour chaque méthode de segmentation à l’aide de la formule suivante : moyenne[(|Nombre prévu - Dénombrement manuel|/Dénombrement manuel) x 100]. Le masque généré par LipidUNet identifie plus précisément les dépôts lipidiques à travers les images avec un dépôt variable par rapport aux autres méthodes de masquage (taux d’erreur moyens: 23% LipidUnet, 1164% Find Maxima, 851% Entropie Max, 203% Entropie Renyi). Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Composant | Numéro de chat | Stock Conc. | Conc. finale | Ml |
| MEM alpha | 12571-063 | NA | 500 | |
| Supplément N2 | 17502-048 | NA | 1% | 5 |
| FBS inactivé par la chaleur | SH30071.03 | NA | 5% | 25 |
| NMEM NEAA | 11140-050 | 10mM | 0.01mM | 5 |
| Sodium Pyruvate | 11360-070 | 100mM | 1mM | 5 |
| Pénicilline-streptomycine | 15140-122 | 10000u/ml | 100U/ml | 5 |
| Taurine | T4571 | 50 mg/mL | 250ug/ml | 2.5 |
| Hydrocortisone | N° H6909 | 18,1 mg/L | 20ug/L | 0.553 |
| T3 | T5516 | 20ug/L | 0,013 ug/L | 0.33 |
| Volume total, mL | 548.383 | |||
Tableau 1 : Composition du réactif RPE-MM. Une liste des réactifs et des concentrations optimales pour l’EPR-MM.
Discussion
Ce protocole fournit une méthode pour étiqueter, imager et quantifier efficacement les dépôts lipidiques dans des modèles de maladies in vitro monogéniques et polygéniques pour les maladies oculaires dégénératives. Le logiciel basé sur l’IA, LipidUNet, peut être appliqué à trois marqueurs lipidiques courants, APOE, Nile Red et BODIPY, et fournit une méthode d’analyse rapide et automatique qui permet à la quantification d’être standard et impartiale.
La principale limitation de LipidUNet est le fait que l’ensemble de données d’apprentissage pour l’IA était limité à des images de grossissement 40x de cellules cultivées dans une plaque de 96 puits. À la suite de l’ensemble d’images d’apprentissage, LipidUNet, dans sa forme actuelle, est limité à l’analyse d’images d’agrandissement 40x. Le logiciel peut être utilisé pour analyser des images 40x de cellules cultivées sur d’autres surfaces de culture en plus d’une plaque de 96 puits, mais il faut prendre soin d’examiner les masques de sortie générés pour vérifier le seuillage précis par le logiciel. Plus d’ensembles d’images (à différents grossissements) seront nécessaires pour élargir la portée des échantillons/images qu’il peut analyser.
Le protocole comporte plusieurs étapes critiques. À l’étape du marqueur lipidique, l’utilisateur doit confirmer que le composé de marquage choisi (BODIPY, APOE, Nile Red) a marqué efficacement son échantillon. Les cellules EPR matures sont souvent fortement pigmentées, ce qui peut altérer le signal fluorescent de l’immunomarquage des anticorps. Lorsque le signal de fluorescence est faible ou lorsqu’il y a trop de coloration de fond, LipidUNet ne peut pas discerner les gouttelettes lipidiques avec précision. Pour une raison similaire, des paramètres d’acquisition correctement sélectionnés pour l’étape d’imagerie automatique du protocole doivent être utilisés. Si les images acquises sont de mauvaise qualité, LipidUNet aura du mal à masquer correctement les images et, par conséquent, la quantification sera inexacte (Figure 6A-L). Enfin, le post-traitement des images est une étape importante, car LipidUNet a des exigences spécifiques pour que le logiciel fonctionne.
Par rapport aux flux de travail pour l’analyse des lipides qui utilisent le seuillage manuel, ou aux techniques qui impliquent un seuillage automatique dans des logiciels comme Fiji, LipidUNet offre une segmentation non biaisée et fiable entre les images avec un dépôt lipidique variable, comme en témoigne un faible taux d’erreur dans l’identification des particules lipidiques (Figure 7). Le logiciel permet à l’utilisateur d’entrer des images d’entraînement supplémentaires, permettant l’analyse d’ensembles d’images au-delà de celles qui utilisent un objectif de grossissement 40x ou même celles qui utilisent un marqueur lipidique différent, comme indiqué dans le protocole. À l’avenir, le logiciel sera formé pour analyser des images 3D afin de permettre la quantification du volume de dépôt lipidique. Les maladies oculaires dégénératives qui impliquent les dépôts de lipides comme un contributeur majeur à la pathologie sont répandues, et les cas devraient augmenter à mesure que la population âgée augmente13. Des modèles de maladie précis et des outils d’analyse efficaces, comme nous l’avons souligné dans ce protocole, permettront le développement de nouvelles interventions thérapeutiques.
Déclarations de divulgation
Aucune divulgation.
Remerciements
Nous remercions le noyau d’histologie du National Eye Institute (NEI) pour l’utilisation du système confocal Zeiss. Ce travail a été financé par les fonds du PRI NEI (numéro de subvention ZIA EY000533-04).
matériels
| Name | Company | Catalog Number | Comments |
| 0.22 µm Steriflip filter system | EMD Millipore | SCGP00525 | |
| 1x Dulbecco's Phosphate Buffered Saline | Gibco | 14190-144 | |
| 3,3',5-Triiodo-L-thyronine | Sigma | T5516 | |
| Albumin Bovine, Fraction V | MP Biomedical | 160069 | |
| Alexa Fluor 555 rabbit anti-goat IgG (H+L) | Invitrogen | A21431 | APOE secondary antibody |
| APOE primary antibody | Millipore Sigma | AB947 | |
| BODIPY 493/503 | Invitrogen | D3922 | Protect from light |
| Complement competent human serum | Millipore Sigma | S1-LITER | |
| CTS N2 Supplement | Life Technologies | A13707-01 | |
| Fetal Bovine Serum | Hyclone | SH30071.03 | |
| Fluoromount-G | SouthernBiotech | 0100-01 | Slide mounting media |
| Glass Cover Slips #1 1/2 22 mm x 22 mm | Electron Microscopy Sciences | 72204-01 | |
| Glass Microscope Slide 25 mm x 75 mm- 1.2 mm Thick | Electron Microscopy Sciences | 71870-01 | |
| Hydrocortisone | Sigma | H0396 | |
| MEM Alpha | Life Technologies | 12571-063 | |
| MEM non-essential Amino Acids | Life Technologies | 11140 | |
| Nile Red | Sigma | 72485-100MG | Protect from light |
| Paraformaldehyde 16% Solution, EM Grade | Electron Microscopy Sciences | 15710 | |
| Penicillin-Strep | Life Technologies | 15140-148 | |
| Phosphate Buffered Saline 10x | Gibco | 70011-044 | |
| Rod Outer Segments (OS) | InVision Bioresources | 98740 | |
| Sodium bicarbonate | Sigma Aldrich | S5761 | |
| Sodium Pyruvate | Life Technologies | 11360-070 | |
| Sucrose | Sigma Aldrich | S1888 | |
| SYBR Green Master Mix | Bio-Rad | 1725274 | |
| Taurine | Sigma | T0625 | |
| Triton X-100 | Sigma | 9002-93-1 | |
| Tween 20 Ultrapure | Affymetrix | 9005-64-5 | |
| Vitronectin | Life Technologies | A14701SA | |
| Y-27632 dihydrochloride | R&D Systems | 1254 |
Références
- Kanow, M. A., et al. Biochemical adaptations of the retina and retinal pigment epithelium support a metabolic ecosystem in the vertebrate eye. eLife. 6, e28899 (2017).
- Adijanto, J., et al. The retinal pigment epithelium utilizes fatty acids for ketogenesis. The Journal of Biological Chemistry. 289 (30), 20570-20582 (2014).
- Farnoodian, M., et al. Cell-autonomous lipid-handling defects in Stargardt iPSC-derived retinal pigment epithelium cells. Stem Cell Reports. 17 (11), 2438-2450 (2022).
- Curcio, C. A. Soft drusen in age-related macular degeneration: Biology and targeting via the oil spill strategies. Investigative Ophthalmology & Visual Science. 59 (4), AMD160-AMD181 (2018).
- Cankova, Z., Huang, J. D., Kruth, H. S., Johnson, M. Passage of low-density lipoproteins through Bruch's membrane and choroid. Experimental Eye Research. 93 (6), 947-955 (2011).
- Curcio, C. A., et al. Esterified and unesterified cholesterol in drusen and basal deposits of eyes with age-related maculopathy. Experimental Eye Research. 81 (6), 731-741 (2005).
- Miyagishima, K. J., et al. AMPK modulation ameliorates dominant disease phenotypes of CTRP5 variant in retinal degeneration. Communications Biology. 4 (1), 1360 (2021).
- Sharma, R., et al. Epithelial phenotype restoring drugs suppress macular degeneration phenotypes in an iPSC model. Nature Communications. 12 (1), 7293 (2021).
- Farnoodian, M., et al. Cell-autonomous lipid-handling defects in Stargardt iPSC-derived retinal pigment epithelium cells. Stem Cell Reports. 17 (11), 2438-2450 (2022).
- Hallam, D., et al. An induced pluripotent stem cell patient specific model of complement factor H (Y402H) polymorphism displays characteristic features of age-related macular degeneration and indicates a beneficial role for UV light exposure. Stem Cells (Dayton, Ohio). 35 (11), 2305-2320 (2017).
- Sharma, R., Bose, D., Montford, J., Ortolan, D., Bharti, K. Triphasic developmentally guided protocol to generate retinal pigment epithelium from induced pluripotent stem cells. STAR Protocols. 3 (3), 101582 (2022).
- Issa, P. C., Barnard, A. R., Herrmann, P., Washington, I., MacLaren, R. E. Rescue of the Stargardt phenotype in Abca4 knockout mice through inhibition of vitamin A dimerization. Proceedings of the National Academy of Sciences of the United States of America. 112 (27), 8415-8420 (2015).
- GBD 2019 Blindness and Vision Impairment Collaborators. Blindness and Vision Impairment Collaborators. Causes of blindness and vision impairment in 2020 and trends over 30 years, and prevalence of avoidable blindness in relation to VISION 2020: the Right to Sight: an analysis for the Global Burden of Disease Study. Lancet Global Health. 9 (2), e144-e160 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon