Method Article
LipidUNet-Machine Learning Based Method of Characterization and Quantification of Lipid Deposits Using iPSC-Derived Retinal Pigment Epithelium
En este artículo
Resumen
Las enfermedades oculares degenerativas que afectan la capa del epitelio pigmentario de la retina del ojo tienen orígenes monogénicos y poligénicos. Se han desarrollado varios modelos de enfermedades y una aplicación de software, LipidUNet, para estudiar los mecanismos de la enfermedad, así como posibles intervenciones terapéuticas.
Resumen
El epitelio pigmentario de la retina (EPR) es una monocapa de células hexagonales ubicadas en la parte posterior del ojo. Proporciona alimento y apoyo a los fotorreceptores y capilares coroideos, realiza fagocitosis de segmentos externos fotorreceptores (POS) y secreta citoquinas de manera polarizada para mantener la homeostasis de la retina externa. El EPR disfuncional, causado por mutaciones, envejecimiento y factores ambientales, resulta en la degeneración de otras capas de la retina y causa pérdida de la visión. Una característica fenotípica distintiva de la degeneración del EPR son los depósitos ricos en lípidos intra y subcelulares. Estos depósitos son un fenotipo común en diferentes enfermedades degenerativas de la retina. Para reproducir el fenotipo de depósito lipídico de las degeneraciones retinianas monogénicas in vitro, se generó RPE derivado de células madre pluripotentes inducidas (iRPE) a partir de los fibroblastos de los pacientes. Las líneas celulares generadas a partir de pacientes con Stargardt y enfermedad de degeneración retiniana de inicio tardío (L-ORD) se alimentaron con POS durante 7 días para replicar la función fisiológica del EPR, lo que causó la patología inducida por fagocitosis POS en estas enfermedades. Para generar un modelo para la degeneración macular relacionada con la edad (DMAE), una enfermedad poligénica asociada con la activación alternativa del complemento, el iRPE fue desafiado con anafilatoxinas alternativas del complemento. Los depósitos lipídicos intra y subcelulares se caracterizaron utilizando rojo del Nilo, boro-diprometeno (BODIPY) y apolipoproteína E (APOE). Para cuantificar la densidad de los depósitos lipídicos, se desarrolló un software basado en el aprendizaje automático, LipidUNet. El software fue entrenado en imágenes de proyección de máxima intensidad de iRPE en superficies de cultivo. En el futuro, será entrenado para analizar imágenes tridimensionales (3D) y cuantificar el volumen de gotas de lípidos. El software LipidUNet será un recurso valioso para descubrir fármacos que disminuyan la acumulación de lípidos en modelos de enfermedades.
Introducción
El epitelio pigmentario de la retina (EPR) es una monocapa de células ubicadas en la parte posterior del ojo adyacentes a los fotorreceptores de la retina. RPE juega un papel vital en el mantenimiento de una visión adecuada al proporcionar apoyo metabólico y estructural a los fotorreceptores. Las células sanas del EPR se caracterizan por una morfología hexagonal distinta. Están conectados por uniones estrechas, que permiten que el EPR actúe como una barrera entre el coriocapilar ubicado en su lado basal y los fotorreceptores ubicados apicalmente. Para mantener el ecosistema retiniano, el EPR transporta metabolitos clave, por ejemplo, glucosa, a los fotorreceptores de una manera que minimiza el consumo de glucosa en el EPR1. Debido a esta limitación, el EPR depende de otros metabolitos para mantener sus necesidades metabólicas, incluidos los ácidos grasos, que el EPR convierte en cetonas a través de la β-oxidación2. Dada la propensión del EPR a utilizar ácidos grasos, que probablemente se reciclan de la digestión del segmento externo de los fotorreceptores (POS), como fuente de energía, los cambios perjudiciales en las vías de procesamiento de lípidos en el EPR a menudo conducen a, o están implicados en, enfermedades degenerativas de la retina monogénicas y poligénicas3.
La degeneración macular relacionada con la edad (DMAE), una enfermedad ocular degenerativa poligénica que causa la degeneración del EPR, también se ha relacionado con la autofagia aberrante y el metabolismo de los lípidos en la monocapa del EPR. La falla de una monocapa de EPR disfuncional para procesar POS y realizar otras funciones críticas conduce a depósitos extracelulares (sub-RPE) llamados depósitos lineales basales (BLinD) ubicados entre el EPR y la membrana de Bruch, un sello distintivo de las patologías de DMAE. Los principales componentes de BLinD incluyen lipoproteínas, la más abundante de las cuales es la apolipoproteína E (APOE)4. La acumulación de capas delgadas de BLinD puede conducir a drusas blandas, que se reconoce como un síntoma clínico de AMD 5,6.
Varios grupos han demostrado que los modelos de enfermedad in vitro derivados de células madre que causan disfunción del EPR presentan acumulación de lípidos sub-EPR 7,8,9. Hallam et al. (2017) generaron RPE derivado de células madre pluripotentes inducidas (iRPE) de pacientes con un alto riesgo de DMAE debido a un polimorfismo del gen CFH. El iRPE mostró acumulación de drusas, marcada por APOE, y el RPE de alto riesgo acumuló depósitos mayores que el iRPE generado a partir de pacientes de bajo riesgo10.
Para crear un modelo in vitro que recapitule las características celulares de la DMAE, como las gotas de lípidos y la deposición de drusas, se establecieron líneas de iRPE generadas a partir de muestras de sangre de pacientes utilizando un protocolo guiado por el desarrollopreviamente publicado 11. Los iRPE fueron sometidos a suero humano competente para el complemento (CC-HS), una solución que contiene anafilatoxinas que imitan una posible causa de DMAE: aumento de la señalización alternativa del complemento8. La deposición celular y subcelular resultante de depósitos de lípidos se midió utilizando marcadores lipídicos y lipoproteicos de uso común, APOE, Nile Red y BODIPY. A través de estos marcadores, se demostró que la señalización activada del complemento a través de CC-HS exacerbó la acumulación de lípidos en las células iRPE8.
Para desarrollar un modelo de enfermedad para una enfermedad degenerativa retiniana monogénica, se desarrollaron líneas iRPE a partir de pacientes con enfermedad de Stargardt, una enfermedad causada por mutaciones en el gen ABCA4 en RPE. Se ha demostrado previamente que cuando ABCA4 es eliminado, la lipofuscina A2E, un depósito intracelular conocido por contener altos niveles de fosfolípidos y productos de peroxidación lipídica dependientes de la luz, se acumula dentro del RPE12. Las líneas knockout ABCA4 se desarrollaron junto con las líneas de pacientes, y ambas fueron sometidas a alimentación POS. El Stargardt iRPE demostró patología inducida por fagocitosis POS, exhibiendo una mayor acumulación de lípidos cuantificada por la tinción BODIPY. Los EPR derivados de las iPSC ABCA4 KO se sometieron a tratamiento CC-HS; la cuantificación de la señal de BODIPY también mostró un defecto en el manejo de lípidos en el modelo de enfermedad de Stargardt9.
Dada la prevalencia de estas enfermedades y la necesidad de terapias eficaces, junto con los modelos de enfermedad relevantes descritos anteriormente, es necesario establecer métodos sólidos para cuantificar la eficacia de los tratamientos potenciales. Para cuantificar los depósitos de lípidos de una manera objetiva, automatizada y estandarizada, se creó un software basado en el aprendizaje automático, LipidUNet, para que, cuando se combina con herramientas de análisis de máscaras, la deposición de lípidos se pueda identificar de manera rápida y efectiva utilizando los marcadores comunes Nile Red, BODIPY y APOE. Las estadísticas resumidas obtenidas utilizando esta tubería de análisis se pueden analizar y mostrar gráficamente, lo que permite una fácil comparación de las condiciones de tratamiento. El esquema del protocolo se muestra en la Figura 1.
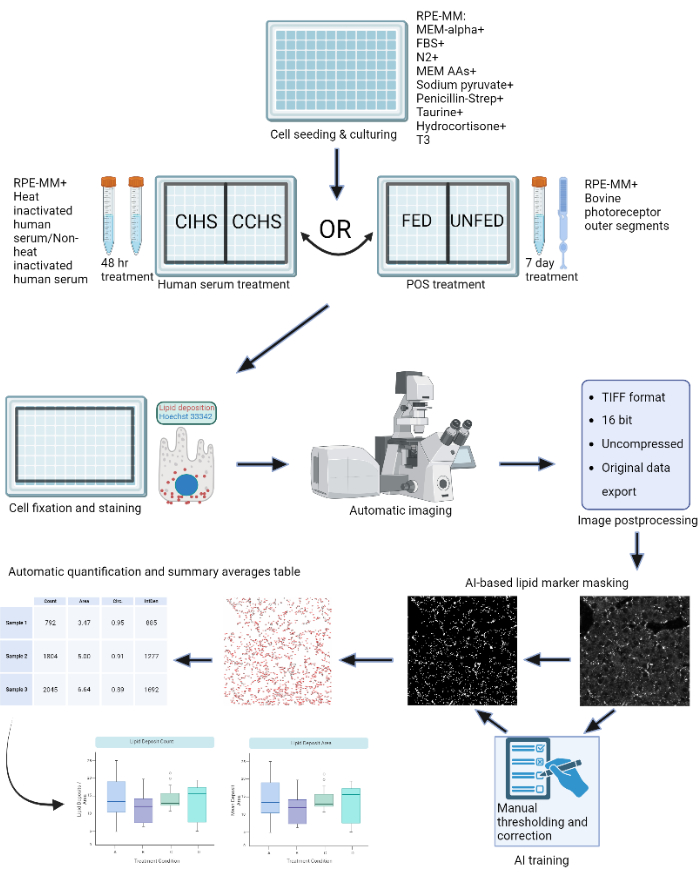
Figura 1: Esquema del protocolo: las células del EPR se cultivan en una placa de 96 pocillos y se desafían con suero humano activo o segmentos externos bovinos purificados para modelar diferentes tipos de degeneraciones retinianas in vitro. Las células del EPR se fijan y tiñen para depósitos de lipoproteínas con Nile Red, BODIPY y APOE. Se utiliza un microscopio confocal para adquirir pilas Z de partículas lipídicas marcadas con fluorescencia, que posteriormente se procesan en proyecciones de intensidad máxima 2D. Se entrenó un algoritmo de aprendizaje automático para reconocer y segmentar correctamente las partículas de lipoproteínas. Se generan tablas de resumen que contienen el recuento de partículas y varias métricas de forma que se pueden utilizar para el trazado posterior y el análisis estadístico. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
Todos los pasos del protocolo se adhieren a las pautas establecidas por el comité de ética de investigación humana de los NIH. El trabajo con células madre y la recolección de muestras de pacientes fueron aprobados por la Junta de Revisión Institucional de Neurociencia Combinada (CNS IRB) bajo la Oficina de Protección de la Investigación Humana (OHRP), NIH, según las pautas 45 CFR 46 del Gobierno de los Estados Unidos. Las muestras de pacientes se recogieron utilizando el formulario de consentimiento aprobado por el IRB del SNC de acuerdo con los criterios establecidos por la Declaración de Helsinki bajo el número de protocolo NCT01432847 (https://clinicaltrials.gov/ct2/show/NCT01432847?cond=NCT01432847&draw=2&rank=1).
1. Generación de iRPE
- Generar iRPE a partir de iPSC derivadas de la sangre del paciente siguiendo el protocolo publicado por Sharma et al., 202211 (Figura 2 y Figura 3).
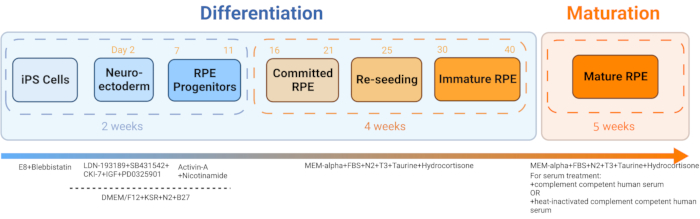
Figura 2: Esquema de diferenciación y maduración de iRPE. Para generar iRPE, se siguió un protocolo de diferenciación establecido y se dejó madurar a las células durante 5 semanas. El cultivo celular resultante actúa como un modelo in vitro que puede manipularse con diversos tratamientos para imitar la disfunción del EPR en enfermedades como la DMAE y la enfermedad de Stargardt. Haga clic aquí para ver una versión más grande de esta figura.
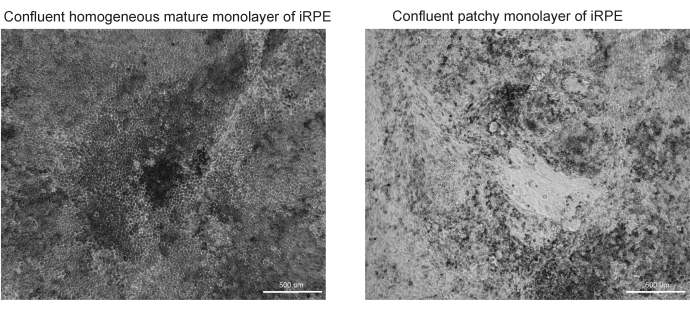
Figura 3: Imágenes representativas de la diferenciación y maduración exitosa y no exitosa del EPR. Dos imágenes de campo claro con un aumento de 10x de TJP1 RPE se muestran en el día 42 del protocolo iRPE. (A) La diferenciación y maduración exitosas mostrarán EPR confluente con pigmentación y morfología poligonal. (B) La diferenciación y maduración fallidas mostrarán grupos de células moribundas, como se muestra aquí. Haga clic aquí para ver una versión más grande de esta figura.
2. Preparación de los medios de mantenimiento ERP (RPE-MM)
- Descongele el suplemento de N2 a 4 °C durante la noche. Descongele todos los demás reactivos a temperatura ambiente (RT).
- En condiciones estériles, agregue los reactivos enumerados en la Tabla 1 a los factores de dilución enumerados, según el protocolo establecido por Sharma et al., 202211.
- Mezcle bien el medio y fílelo con una unidad de filtración de 0,22 μm.
NOTA: El medio es adecuado para su uso dentro de las 2 semanas si se almacena a 4 ° C.
3. Siembra de placas de 96 pocillos
- Descongele una alícuota de vitronectina en RT durante 3-5 minutos o hasta que el hielo esté completamente derretido.
- Diluir la vitronectina con 1x solución salina tamponada con fosfato de Dulbecco (DPBS) para obtener la solución de trabajo deseada utilizando una dilución 1:200 (vitronectina: DPBS). Para una placa de 96 pocillos, cubra cada pocillo con 200 μL de la solución de trabajo.
- Combine el inhibidor ROCK descongelado (diclorhidrato Y-27632) con el RPE-MM en una dilución 1:1000 para alcanzar una concentración final de 10 μM. Este es el medio de recubrimiento para las células RPE.
- Descongele el vial de iRPE utilizando un sistema automatizado de descongelación celular y transfiera la suspensión de células iRPE a un tubo de 50 ml.
- Diluir la suspensión celular con el medio de recubrimiento en una dilución de 1:10. Centrifugar el tubo a 400 x g durante 5 min.
- Aspirar cuidadosamente el sobrenadante y resuspender las células en 10 ml del medio de recubrimiento.
- Mezclar 400 μL del medio de recubrimiento con 100 μL de la solución celular resuspendida para el recuento celular. Utilice esta alícuota para determinar la concentración celular viable de la suspensión celular utilizando un contador de viabilidad celular.
- Diluir la suspensión celular con el medio de recubrimiento hasta una concentración final de 60.000 células/ml.
- Aspirar completamente la solución de recubrimiento de vitronectina de la placa de 96 pocillos y dispensar 200 μL de la suspensión celular en cada pocillo. Habrá aproximadamente 12,000 células / pocillo o ~ 200 células / mm2.
- Incubar las placas celulares sembradas durante 48 h a 37 °C y 5% deCO2. Después de 48 h, cambie el medio a RPE-MM sin suplemento inhibidor de ROCK. Cambie los medios cada 2-3 días durante el período de maduración de 5 semanas.
4. Modelos de enfermedad in vitro
- Tratamiento con suero humano competente para el complemento (CC-HS)
- Descongelar el suero competente del complemento humano a 4 °C durante la noche.
- Preparar CC-HS y complementar los medios de suero humano incompetente (CI-HS).
- Para preparar medios CC-HS al 5%, mezcle el suero humano competente del complemento descongelado con RPE-MM en una dilución de 1:20. Filtrar la solución a través de un filtro de medios de 0,22 μm antes de su uso.
- Para preparar un medio de suero humano incompetente (CI-HS) del complemento al 5%, primero inactive el CC-HS en un baño de agua a 57 °C durante 30 min y luego mezcle con los medios de cultivo a una dilución de 1:20. Filtrar la solución a través de un filtro de medios de 0,22 μm antes de su uso.
- El suero trata las células con 200 μL de medios CC-HS al 5% o CI-HS al 5% durante un tiempo total de incubación de 48 h, refrescando los medios después de 24 h.
- Lave las células con 1x DPBS y fíjelas con paraformaldehído al 4% durante 20 min en RT. Lavar una vez más con 1x DPBS y almacenar las muestras a 4 °C, sumergidas en 200 μL de DPBS.
- OPCIONAL: Si lo desea, lisar las células de la placa para mostrar solo la deposición de lípidos por debajo del EPR.
- Para lisar las células y dejar solo depósitos de lípidos, retire los medios y agregue 200 μL de agua desionizada a cada pozo.
- Incubar durante 10-15 min, pipetear hacia arriba y hacia abajo hasta que se eliminen las células. Lavar una vez más con 200 μL de agua desionizada e inmediatamente fijar las células con paraformaldehído al 4%.
- Confirme la eficacia de eliminación celular con tinción nuclear usando Hoechst. Agregue Hoechst a una dilución de 1:2000 a una solución 1x DPBS que contenga 1% de albúmina sérica bovina (BSA), 0,5% de Tween 20 y 0,5% de Triton-X 100. Incubar a RT durante 1 h en la oscuridad. Posteriormente, lavar con 1x DPBS.
- Tratamiento del segmento externo de los fotorreceptores (POS) en el iRPE
- Preparación de TPV
- Retire el tubo de pellets POS del almacenamiento de -80 °C y descongele durante la noche a 4 °C en una cubitera cubierta.
- Prepare el tampón de lavado POS mezclando 10 g de sacarosa en 40 ml deH2O(ddH2O) doblemente desionizado.
- Calentar la mezcla a 40-50 °C mientras se agita suavemente durante 15 min. Agregue 840 mg de bicarbonato de sodio a la mezcla y revuelva mientras calienta durante 10 minutos.
- Ajuste el volumen total del tampón de lavado POS a 100 ml conddH2Oy ajuste el pH de la solución a 8.3 con 1 N HCl o 1 N NaOH, según sea necesario. Filtrar la solución de lavado con un filtro de 0,22 μm.
NOTA: El protocolo se puede pausar aquí; el tampón de lavado POS se puede almacenar a 4 °C durante la noche. - Una vez descongelado, suspender el pellet en 15 mL de tampón de lavado POS. Sea suave durante las suspensiones de pellets para garantizar la integridad del punto de venta. Centrifugar la suspensión POS a 600 x g a 4 °C durante 20 min y luego aspirar el sobrenadante.
- Vuelva a suspender el pellet POS en 10 ml del tampón de lavado POS.
- Retire una alícuota de 100 μL del tampón de lavado POS + POS (solución POS) y diluya en 400 μL de 1x DPBS. Extienda 50 μL de la solución POS diluida en una placa de agar sangre y una placa de agarosa para verificar si hay contaminantes bacterianos y fúngicos. Preparar controles positivos para cada uno e incubar todas las placas durante 48 h a 37 °C.
- Realice un ensayo de qPCR agregando 1 μL de solución POS al pocillo de detección para detectar micoplasma. Para amplificar fragmentos de ADN, realice 40 ciclos de desnaturalización (95°C, 15 s), y recocido y alargamiento (60°C, 1 min). Los cebadores directos e inversos para detectar micoplasma en la muestra POS son los siguientes:
Imprimación delantera: GGA TTA GAT ACC CTG GTA GTC CAC G
Cebador inverso: CGT CAA TTC CTT TAA GTT TCA CTC TTG GC - Mida la concentración de POS utilizando un analizador de células y alícuota según la necesidad. Para un pocillo de 96 placas de pocillos con celdas RPE, 3 x 106 POS es suficiente. La relación deseada es 10 celdas POS/RPE. Conservar las alícuotas a 80 °C para utilizarlas en el futuro.
- Adición de POS a las celdas
- Descongele los viales de POS en un baño de hielo.
- Mezcle la cantidad calculada de POS preparado con RPE-MM y trate las células con POS una vez al día durante 7 días.
NOTA: Prepare la solución POS fresca diariamente. - Lave las células con 1x DPBS y luego fíjelas con paraformaldehído al 4% durante 20 minutos en RT. Lavar con DPBS una vez más y almacenar muestras a 4 °C, sumergidas en 200 μL de DPBS.
- Preparación de TPV
5. Tinción para depósitos de sub-EPR
- Protocolo de tinción de rojo del Nilo
- Después de la fijación de PFA, lave las muestras 3 veces con 1x DPBS.
NOTA: Si no se usa inmediatamente, el protocolo se puede pausar aquí, pero las muestras deben almacenarse en una solución de azida de sodio 1x DPBS + 0.02% a 4 °C. - Para preparar la solución madre de Nile Red, disolver el polvo de Nile Red en acetona a una concentración de 3 mg/ml. Incubar durante 15 min a RT con mezcla periódica. Filtrar las soluciones con un filtro de 0,22 μm una o dos veces, dependiendo del nivel de precipitado que quede en la solución.
NOTA: Proteja la solución madre de la luz. - Para preparar la solución de trabajo, diluya la solución madre en una proporción de 1:500 en 1x DPBS. Añadir 200 μL de la solución de trabajo a la muestra durante 30 min a RT en un agitador y protegerla de la luz.
- Lavar 3 veces con 1x PBS y almacenar las muestras a 4 °C, sumergidas en 200 μL de DPBS.
NOTA: Si se realiza un experimento en transpozos en lugar de una placa de 96 pocillos, las muestras se pueden montar en un portaobjetos con medios de montaje, cubrirse con un cubreobjetos de vidrio y sellarse con esmalte de uñas transparente. Se debe tener cuidado de montar la muestra con las células hacia arriba.
- Después de la fijación de PFA, lave las muestras 3 veces con 1x DPBS.
- Protocolo de tinción BODIPY
- Para la solución madre, disolver BODIPY en dimetilsulfóxido anhidro (DMSO) para alcanzar una concentración madre de 3,8 mM.
- Para muestras fijas de PFA, diluir el stock de BODIPY a 1:300 en 1x DPBS. Agregue 200 μL a las células e incube durante la noche en un balancín en RT.
- Lavar 3 veces con 1x DPBS y almacenar las muestras a 4 °C, sumergidas en 200 μL de DPBS.
NOTA: Si se realiza un experimento en transpozos en lugar de una placa de 96 pocillos, las muestras se pueden montar en un portaobjetos con medios de montaje, cubrirse con un cubreobjetos de vidrio y sellarse con esmalte de uñas transparente. Se debe tener cuidado de montar la muestra con las células hacia arriba.
- Protocolo de inmunotinción APOE
- Combine 1x DPBS con albúmina sérica bovina (BSA) al 1%, Tween 20 al 0,5% y Triton-X 100 al 0,5% para crear una solución tampón.
- Para muestras fijas de PFA, bloquear y permeabilizar la muestra en 200 μL de la solución tampón durante 1 h a RT.
- Añadir el anticuerpo primario APOE diluido a 1:100 en la solución tampón e incubar durante la noche en RT.
- Al día siguiente, lave las muestras 3 veces con 1x DPBS.
- Añadir un anticuerpo secundario a una dilución 1:1000 en la solución tampón y añadir 200 μL de la solución a las células durante 1 h en RT.
- Lavar 3 veces con 1x DPBS y almacenar las muestras a 4 °C, sumergidas en 200 μL de DPBS.
NOTA: Si se realiza un experimento en pozos de transwell en lugar de una placa de 96 pocillos, las muestras se pueden montar en un portaobjetos con medios de montaje (Fluoromount), cubrirse con un cubreobjetos de vidrio y sellarse con esmalte de uñas transparente. Se debe tener cuidado de montar la muestra con las células hacia arriba.
6. Automatización y procesamiento de imágenes
- Escaneo automatizado de imágenes
NOTA: En este estudio se utilizó el microscopio de barrido confocal invertido Zeiss LSM 800 y el software ZEN 3.2 (edición azul). Asegúrese de que la placa de 96 pocillos se caliente a RT durante un mínimo de 60 minutos antes de obtener imágenes para evitar la desviación del plano focal durante el escaneo debido a un cambio en el índice de refracción del medio con el cambio de temperatura.- Usando un microscopio confocal y un objetivo 40x, cree un perfil de escaneo con los canales fluorescentes apropiados para el marcador lipídico utilizado y cualquier anticuerpo adicional.
- Utilice la casilla de verificación Mosaicos para configurar la automatización de imágenes. Para calibrar la placa de 96 pocillos, asegúrese de que se introduzcan y seleccionen las mediciones correctas del portador de muestras. Luego, haga clic en el botón Calibrar para calibrar la placa de acuerdo con las instrucciones, lo que requiere usar el objetivo 10x.
- Elija la vista Configuración avanzada para seleccionar los pozos apropiados y agregue 3 puntos de imagen diferentes cerca del centro del pozo utilizando la función Posiciones . Esto se puede hacer manualmente en la subpestaña Posición o aleatoriamente usando la pestaña Configuración de posición y seleccionando Configuración por operador. Repita para todos los pocillos de la misma tinción.
- Para un enfoque óptimo y un posicionamiento de la pila Z durante la automatización, vaya a la pestaña Estrategia de enfoque para seleccionar Usar valores de superficie de enfoque/Z definidos por la configuración de mosaicos. Los métodos alternativos pueden usar otras estrategias de enfoque, pero se recomienda usar esta configuración para obtener los resultados más consistentes.
- En la pestaña Mosaicos , haga clic en Verificar posiciones y establezca manualmente el plano Z central para cada posición. La configuración en la subpestaña Opciones ordenará la adquisición de imágenes, así que verifique esto antes de comenzar a la imagen. Para adquirir imágenes en el orden en que se seleccionaron las posiciones, anule la selección de las casillas de verificación Regiones/Posiciones de mosaico y Pozos portadores/Contenedores . Seleccione Dividir escenas en archivos separados para facilitar el procesamiento de imágenes.
- Asegúrese de que la pestaña Z-Stack esté establecida en Centro, que se introduzca un rango en las preferencias del usuario y que se seleccione el botón Óptimo para establecer el intervalo de segmento.
- Después de optimizar las pestañas Modo de adquisición, Canales, Estrategia de enfoque, Z-Stack y Mosaicos , inicie el experimento.
- Tratamiento de imágenes
- Mediante un método de procesamiento de imágenes por lotes, cree proyecciones máximas de cada pila Z con el método de profundidad de enfoque extendida .
- Mediante un método de procesamiento de imágenes por lotes, exporte los archivos de proyección máxima como imágenes TIFF de 16 bits. Establezca la compresión en Ninguno y asegúrese de que los datos originales estén marcados. La imagen resultante debe ser un TIFF en escala de grises de proyección máxima de solo el canal de fluorescencia en el que se expresa el marcador lipídico.
7. Segmentación y cuantificación
NOTA: El programa LipidUNet fue entrenado en imágenes 40x de una placa de 96 pocillos. Es muy recomendable utilizar imágenes obtenidas utilizando un objetivo de 40x.
- Instale el software LipidUNet. LipidUNet se puede descargar desde el siguiente repositorio de GitHub: https://github.com/RPEGoogleMap/LipidUNet
- Identifique las imágenes TIFF que representan Nile Red, Bodipy o APOE y muévalas a una carpeta denominada imgs dentro de un directorio denominado Nile_Red, Bodipy o APOE, según el método utilizado.
NOTA: Se deben utilizar convenciones de nomenclatura exactas para que el programa LipidUNet reconozca los directorios. - Abra el software LipidUNet (Figura 4).
- En la pestaña Predecir del software, seleccione el directorio correspondiente (Nile_Red, Bodipy o APOE) haciendo clic en los puntos suspensivos y navegando hasta el directorio nombrado. Confirme que el programa LipidUNet ha identificado correctamente las imágenes comprobando la entrada Clase.
- Seleccione un umbral de probabilidad para el algoritmo entre 0,01 y 0,99. Un valor más alto eliminará más falsos positivos, pero podría causar más falsos negativos, y los valores más bajos podrían introducir más falsos positivos mientras eliminan más falsos negativos. Un valor de 0,65 es el valor predeterminado y se recomienda.
- Haga clic en Predict.
NOTA: El software iterará a través de todas las imágenes automáticamente y creará una nueva carpeta llamada predicted_masks en el directorio seleccionado. - Utilice una herramienta de análisis de máscara para iterar a través de las máscaras generadas y proporcionar un recuento cuantitativo de los depósitos de lípidos umbrales de las imágenes de máscara.
- Analice los datos de recuentos generados para comparar las condiciones de tratamiento.
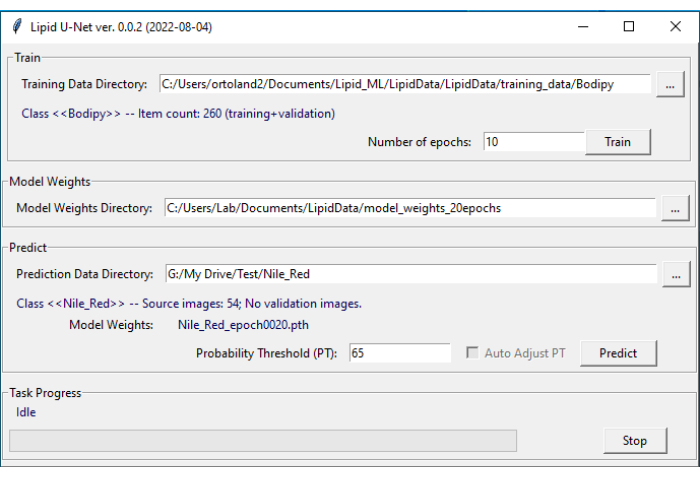
Figura 4: Interfaz de usuario de LipidUNet. El software LipidUNet tiene diferentes secciones para seleccionar para el directorio de datos de entrenamiento, donde las imágenes de los depósitos de lípidos se han identificado correctamente; el directorio de ponderaciones del modelo, que se produce a partir de los datos de entrenamiento; y el directorio de datos de predicción en el que el usuario ingresará sus imágenes para la segmentación. Haga clic aquí para ver una versión más grande de esta figura.
Resultados
Este protocolo proporciona un flujo de trabajo para identificar depósitos de lípidos teñidos por Nile Red, BODIPY y APOE. El software desarrollado puede identificar y cuantificar automáticamente los depósitos de lípidos y funciona mejor cuando se optimiza el protocolo descrito. Se incluyen ejemplos de EPR diferenciado con éxito (Figura 3A) y EPR pobremente diferenciado (Figura 3B), ya que la calidad del modelo celular afecta en gran medida la calidad de la segmentación adecuada de la imagen.
Dos de los tres marcadores descritos en el protocolo, Nile Red y BODIPY, se identifican como pequeños puntos circulares que son claramente brillantes en imágenes fluorescentes (Figura 5 y Figura 6). Una imagen "positiva" del protocolo sería una identificación apropiada de estos depósitos distintos (Figura 5A-D y Figura 5E-H). Un resultado "negativo" mostraría una segmentación incorrecta de la imagen al confundir la fluorescencia de fondo con un depósito, ya sea debido a una tinción débil (Figura 6A-C y Figura 6D-F) o debido a una alta intensidad de fondo (Figura 6G-I).
Los depósitos de APOE tienen una variedad de tamaños y formas, apareciendo más ovalados o irregulares en lugar de los depósitos circulares de Nile Red y BODIPY. Estos depósitos también son menos punteados, y la intensidad de la señal puede diferir entre depósitos debido a variaciones en la permeabilización de la muestra. La identificación correcta identificará cada depósito, incluidos aquellos que están menos saturados (Figura 5I-L), mientras que la segmentación incorrecta no recogerá estos depósitos (Figura 6J-L). Por lo tanto, es importante optimizar los métodos de tinción e imagen para evitar variaciones drásticas. Una forma de hacerlo es prestando especial atención a los pasos de permeabilización de la muestra durante la inmunotinción. Para optimizar la señal fluorescente, las células pueden ser lisadas antes de la fijación e inmunotinción para APOE, lo que resulta en una saturación uniforme y una mejor segmentación de los depósitos de APOE.
También se proporcionan imágenes segmentadas de células maduradas en una plataforma de cultivo que no sea una placa de 96 pocillos. El software LipidUNet se ejecutó en imágenes de células cultivadas en un pozo trans, y mientras que los depósitos de lípidos están umbralizados, también lo son los poros en la membrana del transpozo (Figura 6M-O). Debido a la similitud en forma y tamaño, el software LipidUNet en su forma actual enmascarará tanto los depósitos de lípidos como los poros transwell indiscriminadamente.
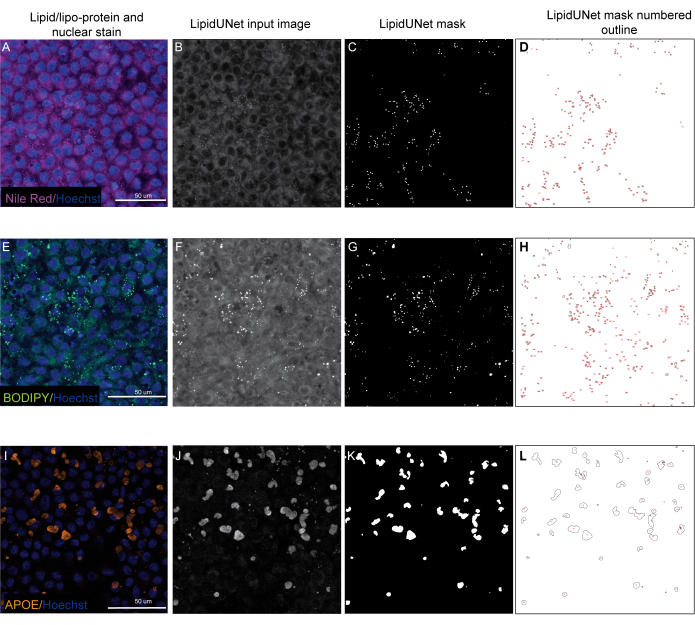
Figura 5: Resultados representativos. (A,E,I) El EPR chapado en 96 pocillos se tiñe con tinción nuclear de Hoechst (azul) y rojo del Nilo (magenta), BODIPY (verde) o APOE (naranja) y son las proyecciones de intensidad máxima de una pila Z. (B, F, J) Las imágenes de entrada en escala de grises para el software LipidUNet después del procesamiento de imágenes. (C, G, K) Máscaras generadas por LipidUNet, donde todos los depósitos están identificados correctamente. (D,H,L) Los contornos de cada partícula enmascarada están numerados. Estas etiquetas permiten conectar cada partícula de la imagen a una entrada en el spreadheet con los datos en bruto. (A-D) muestra la tinción del Rojo del Nilo, y el software es capaz de reconocer los depósitos contra el fondo con precisión a pesar de una señal más débil. (E-H) muestra un fuerte contraste entre la señal BODIPY y el fondo, lo cual es ideal. LipidUNet identifica correctamente cada depósito en la imagen. (I-L) muestra una fuerte señal APOE y representa la variabilidad de la saturación de la señal que a menudo se ve con esta tinción. No obstante, la segmentación de imágenes es capaz de identificar los bordes de cada depósito APOE. Haga clic aquí para ver una versión más grande de esta figura.
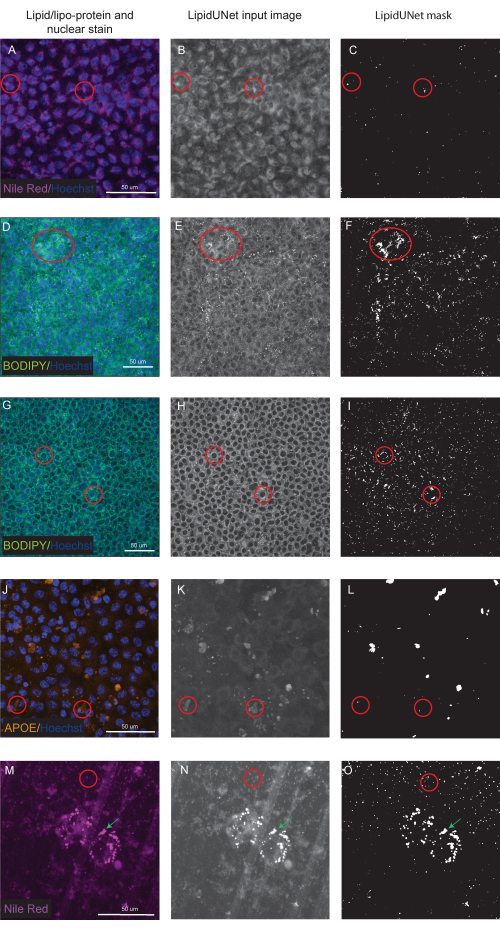
Figura 6: Resultados subóptimos. (A,D,G,J,M) El EPR de 96 pocillos se tiñe con tinción nuclear de Hoechst (azul) y rojo del Nilo (magenta), BODIPY (verde) o APOE (naranja) y son las proyecciones de intensidad máxima de una pila Z. (B, E, H, K, N) Las imágenes de entrada en escala de grises para el software LipidUNet después del procesamiento de imágenes. (C,F,I,L,O) Las máscaras incorrectas generadas por LipidUNet. Los círculos rojos indican dónde el software ha identificado incorrectamente un depósito de lípidos. (A-C) El procesamiento de Nile Red es incorrecto porque el software ha identificado la tinción de fondo como un depósito. Esto puede suceder más a menudo cuando hay un fondo alto pero pocos depósitos de lípidos en la imagen. Se muestran dos ejemplos de tinción BODIPY: una imagen de mala calidad debido a la tinción BODIPY débil (D-F) y (G - I) una señal BODIPY fuerte con fondo alto. En ambos casos, el software es incapaz de distinguir pequeños depósitos de lípidos circulares del anillo circular de fondo que rodea el núcleo. Si bien la tinción y las imágenes deben optimizarse para evitar estos errores, la versión más reciente de LipidUNet se ha mejorado en gran medida para estas imágenes. (J-L) Segmentación incorrecta de APOE. Dado que los depósitos son más variables en tamaño y saturación de señal, el software tiene dificultades para reconocer algunos depósitos. (M-O) RPE sembrado en un pozo trans y teñido con Rojo del Nilo. Aquí se muestra una rebanada de la pila Z con depósitos de lípidos de Nile Red y poros transwell. El software es incapaz de distinguir entre los dos, como se muestra por el círculo rojo que contiene poros transwell y la flecha verde que apunta a los depósitos de Nilo Red. Haga clic aquí para ver una versión más grande de esta figura.

Figura 7: Comparación de herramientas de máscara. (A, B, C) El EPR de 96 pocillos con cantidades variables de deposición de lípidos se identifica con rojo del Nilo (rojo). Las imágenes se enmascaran utilizando tres métodos de enmascaramiento comunes diferentes, Find Maxima, Max Entropy y Renyi Entropy, y se comparan con la máscara generada por LipidUNet. La imagen original se acompaña de un recuento manual de los depósitos de lípidos, mientras que las máscaras muestran los recuentos previstos por cada método de segmentación. La tasa de error media se calculó para cada método de segmentación utilizando la siguiente fórmula: media[(|Recuento previsto - Recuento manual|/Conteo manual) x 100]. La máscara generada por LipidUNet identifica con mayor precisión los depósitos de lípidos en las imágenes con deposición variable en comparación con otros métodos de enmascaramiento (tasas de error promedio: 23% LipidUnet, 1164% Find Maxima, 851% Max Entropy, 203% Renyi Entropy). Haga clic aquí para ver una versión más grande de esta figura.
| Componente | Número de gato | Stock Conc. | Final Conc. | Ml |
| MEM alfa | 12571-063 | NA | 500 | |
| Suplemento N2 | 17502-048 | NA | 1% | 5 |
| FBS inactivado por calor | SH30071.03 | NA | 5% | 25 |
| NMEM NEAA | 11140-050 | 10mM | 0,01 mM | 5 |
| Piruvato de sodio | 11360-070 | 100mM | 1mM | 5 |
| Penicilina-estreptomicina | 15140-122 | 10000u/mL | 100U/mL | 5 |
| Taurina | T4571 | 50mg/mL | 250ug/mL | 2.5 |
| Hidrocortisona | H6909 | 18.1mg/L | 20ug/L | 0.553 |
| T3 | T5516 | 20ug/L | 0.013ug/L | 0.33 |
| Volumen total, ml | 548.383 | |||
Tabla 1: Composición del reactivo RPE-MM. Una lista de reactivos y concentraciones óptimas para RPE-MM.
Discusión
Este protocolo proporciona un método para etiquetar, obtener imágenes y cuantificar eficientemente los depósitos de lípidos en modelos de enfermedades in vitro monogénicas y poligénicas para enfermedades oculares degenerativas. El software basado en IA, LipidUNet, se puede aplicar a tres marcadores lipídicos comunes, APOE, Nile Red y BODIPY, y proporciona un método rápido y automático para el análisis que permite que la cuantificación sea estándar e imparcial.
La principal limitación de LipidUNet es el hecho de que el conjunto de datos de entrenamiento para la IA se limitó a imágenes de aumento de 40x de células cultivadas en una placa de 96 pocillos. Como resultado del conjunto de imágenes de entrenamiento, LipidUNet, en su forma actual, se limita a analizar imágenes de aumento de 40x. El software se puede utilizar para analizar imágenes 40x de células cultivadas en otras superficies de cultivo además de una placa de 96 pocillos, pero se debe tener cuidado de examinar las máscaras de salida generadas para verificar el umbral preciso por parte del software. Se necesitarán más conjuntos de imágenes (con diferentes aumentos) para ampliar el alcance de las muestras / imágenes que puede analizar.
El protocolo tiene varios pasos críticos. En el paso del marcador lipídico, el usuario debe confirmar que su compuesto de etiquetado elegido (BODIPY, APOE, Nile Red) ha etiquetado su muestra de manera efectiva. Las células maduras del EPR a menudo están muy pigmentadas, lo que puede afectar la señal fluorescente de la inmunotinción de anticuerpos. Cuando la señal de fluorescencia es débil o cuando hay demasiada tinción de fondo, LipidUNet no puede discernir las gotas de lípidos con precisión. Por una razón similar, se deben usar los ajustes de adquisición seleccionados correctamente para el paso de creación automática de imágenes del protocolo. Si las imágenes adquiridas son de mala calidad, LipidUNet tendrá dificultades para enmascarar adecuadamente las imágenes y, por lo tanto, la cuantificación será inexacta (Figura 6A-L). Finalmente, el post-procesamiento de las imágenes es un paso importante, ya que LipidUNet tiene requisitos específicos para que el software funcione.
En comparación con los flujos de trabajo para el análisis de lípidos que utilizan umbrales manuales, o técnicas que implican umbrales automáticos en software como Fiji, LipidUNet ofrece una segmentación no sesgada y confiable a través de imágenes con deposición de lípidos variable, como se refleja en una pequeña tasa de error en la identificación de partículas lipídicas (Figura 7). El software permite al usuario la entrada de imágenes de entrenamiento adicionales, lo que permite el análisis de conjuntos de imágenes más allá de aquellos que utilizan un objetivo de aumento de 40x o incluso aquellos que utilizan un marcador lipídico diferente, como se describe en el protocolo. En el futuro, el software será entrenado para analizar imágenes 3D de modo que sea posible la cuantificación del volumen del depósito lipídico. Las enfermedades oculares degenerativas que implican la deposición de lípidos como un importante contribuyente a la patología son prevalentes, y se prevé que los casos aumenten a medida que la población de ancianos se expanda13. Los modelos precisos de la enfermedad y las herramientas de análisis eficientes, como hemos descrito en este protocolo, permitirán el desarrollo de nuevas intervenciones terapéuticas.
Divulgaciones
Sin divulgaciones.
Agradecimientos
Agradecemos al núcleo histológico del Instituto Nacional del Ojo (NEI) por el uso del sistema confocal Zeiss. Este trabajo fue apoyado por los fondos NEI IRP (número de subvención ZIA EY000533-04).
Materiales
| Name | Company | Catalog Number | Comments |
| 0.22 µm Steriflip filter system | EMD Millipore | SCGP00525 | |
| 1x Dulbecco's Phosphate Buffered Saline | Gibco | 14190-144 | |
| 3,3',5-Triiodo-L-thyronine | Sigma | T5516 | |
| Albumin Bovine, Fraction V | MP Biomedical | 160069 | |
| Alexa Fluor 555 rabbit anti-goat IgG (H+L) | Invitrogen | A21431 | APOE secondary antibody |
| APOE primary antibody | Millipore Sigma | AB947 | |
| BODIPY 493/503 | Invitrogen | D3922 | Protect from light |
| Complement competent human serum | Millipore Sigma | S1-LITER | |
| CTS N2 Supplement | Life Technologies | A13707-01 | |
| Fetal Bovine Serum | Hyclone | SH30071.03 | |
| Fluoromount-G | SouthernBiotech | 0100-01 | Slide mounting media |
| Glass Cover Slips #1 1/2 22 mm x 22 mm | Electron Microscopy Sciences | 72204-01 | |
| Glass Microscope Slide 25 mm x 75 mm- 1.2 mm Thick | Electron Microscopy Sciences | 71870-01 | |
| Hydrocortisone | Sigma | H0396 | |
| MEM Alpha | Life Technologies | 12571-063 | |
| MEM non-essential Amino Acids | Life Technologies | 11140 | |
| Nile Red | Sigma | 72485-100MG | Protect from light |
| Paraformaldehyde 16% Solution, EM Grade | Electron Microscopy Sciences | 15710 | |
| Penicillin-Strep | Life Technologies | 15140-148 | |
| Phosphate Buffered Saline 10x | Gibco | 70011-044 | |
| Rod Outer Segments (OS) | InVision Bioresources | 98740 | |
| Sodium bicarbonate | Sigma Aldrich | S5761 | |
| Sodium Pyruvate | Life Technologies | 11360-070 | |
| Sucrose | Sigma Aldrich | S1888 | |
| SYBR Green Master Mix | Bio-Rad | 1725274 | |
| Taurine | Sigma | T0625 | |
| Triton X-100 | Sigma | 9002-93-1 | |
| Tween 20 Ultrapure | Affymetrix | 9005-64-5 | |
| Vitronectin | Life Technologies | A14701SA | |
| Y-27632 dihydrochloride | R&D Systems | 1254 |
Referencias
- Kanow, M. A., et al. Biochemical adaptations of the retina and retinal pigment epithelium support a metabolic ecosystem in the vertebrate eye. eLife. 6, e28899(2017).
- Adijanto, J., et al. The retinal pigment epithelium utilizes fatty acids for ketogenesis. The Journal of Biological Chemistry. 289 (30), 20570-20582 (2014).
- Farnoodian, M., et al. Cell-autonomous lipid-handling defects in Stargardt iPSC-derived retinal pigment epithelium cells. Stem Cell Reports. 17 (11), 2438-2450 (2022).
- Curcio, C. A. Soft drusen in age-related macular degeneration: Biology and targeting via the oil spill strategies. Investigative Ophthalmology & Visual Science. 59 (4), AMD160-AMD181 (2018).
- Cankova, Z., Huang, J. D., Kruth, H. S., Johnson, M. Passage of low-density lipoproteins through Bruch's membrane and choroid. Experimental Eye Research. 93 (6), 947-955 (2011).
- Curcio, C. A., et al. Esterified and unesterified cholesterol in drusen and basal deposits of eyes with age-related maculopathy. Experimental Eye Research. 81 (6), 731-741 (2005).
- Miyagishima, K. J., et al. AMPK modulation ameliorates dominant disease phenotypes of CTRP5 variant in retinal degeneration. Communications Biology. 4 (1), 1360(2021).
- Sharma, R., et al. Epithelial phenotype restoring drugs suppress macular degeneration phenotypes in an iPSC model. Nature Communications. 12 (1), 7293(2021).
- Farnoodian, M., et al. Cell-autonomous lipid-handling defects in Stargardt iPSC-derived retinal pigment epithelium cells. Stem Cell Reports. 17 (11), 2438-2450 (2022).
- Hallam, D., et al. An induced pluripotent stem cell patient specific model of complement factor H (Y402H) polymorphism displays characteristic features of age-related macular degeneration and indicates a beneficial role for UV light exposure. Stem Cells (Dayton, Ohio). 35 (11), 2305-2320 (2017).
- Sharma, R., Bose, D., Montford, J., Ortolan, D., Bharti, K. Triphasic developmentally guided protocol to generate retinal pigment epithelium from induced pluripotent stem cells. STAR Protocols. 3 (3), 101582(2022).
- Issa, P. C., Barnard, A. R., Herrmann, P., Washington, I., MacLaren, R. E. Rescue of the Stargardt phenotype in Abca4 knockout mice through inhibition of vitamin A dimerization. Proceedings of the National Academy of Sciences of the United States of America. 112 (27), 8415-8420 (2015).
- GBD 2019 Blindness and Vision Impairment Collaborators. Blindness and Vision Impairment Collaborators. Causes of blindness and vision impairment in 2020 and trends over 30 years, and prevalence of avoidable blindness in relation to VISION 2020: the Right to Sight: an analysis for the Global Burden of Disease Study. Lancet Global Health. 9 (2), e144-e160 (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados