Method Article
הדמיה של מוקדי DNA חד-גדיליים בשלב G1 של מחזור התא
In This Article
Summary
הפרוטוקול הבא מציג זיהוי של מוקדי DNA חד-גדיליים בשלב G1 של מחזור התא תוך שימוש בסנכרון מחזור התא ואחריו צביעה אימונופלואורסצנטית RPA2.
Abstract
לדנ"א מסלולי תיקון תאיים ייעודיים המסוגלים להתמודד עם נגעים שעלולים לנבוע הן ממקורות אנדוגניים והן ממקורות אקסוגניים. תיקון DNA דורש שיתוף פעולה בין חלבונים רבים, האחראים לכסות מגוון רחב של משימות, החל מזיהוי ואיתות על נוכחות נגע DNA ועד לתיקון פיזי שלו. במהלך תהליך זה, עקבות של DNA חד-גדילי (ssDNA) נוצרים לעתים קרובות, אשר בסופו של דבר מתמלאים על ידי DNA פולימראזות. טבען של רצועות ssDNA אלה (הן מבחינת אורך והן מבחינת מספר), יחד עם הפולימראז שגויס כדי למלא פערים אלה, הם ספציפיים למסלול תיקון. ההדמיה של מסלולי ssDNA אלה יכולה לעזור לנו להבין את הדינמיקה המסובכת של מנגנוני תיקון דנ"א.
פרוטוקול זה מספק שיטה מפורטת להכנת תאים מסונכרנים G1 למדידת היווצרות מוקדי ssDNA בעת לחץ גנוטוקסי. באמצעות גישה אימונופלואורסצנטית קלה לניצול, אנו מדמיינים ssDNA על ידי צביעה עבור RPA2, מרכיב של חלבון השכפול ההטרוטרימרי קומפלקס A (RPA). RPA2 נקשר ומייצב מתווכי ssDNA המתעוררים בעקבות לחץ גנוטוקסי או שכפול כדי לשלוט בתיקון הדנ"א ובהפעלת מחסום הנזק לדנ"א. צביעת 5-Ethynyl-2'-deoxyuridine (EdU) משמשת להדמיה של שכפול DNA כדי לא לכלול תאים בשלב S. פרוטוקול זה מספק גישה חלופית לבדיקות הקונבנציונליות המבוססות על 5-bromo-2'-deoxyuridine (BrdU) והוא מתאים יותר לזיהוי מוקדי ssDNA מחוץ לשלב S.
Introduction
כדי לקיים חיים, תאים סוקרים ומתקנים דנ"א ללא הרף כדי לשמור על שלמותם הגנומית. תאים עלולים לצבור סוגים שונים של נזק לדנ"א עקב מקורות אנדוגניים (למשל, חמצון, אלקילציה, דהמינציה, שגיאות שכפול) ואקסוגניים (למשל, UV, קרינה מייננת) של גורמי עקה של DNA. כישלון לתקן נגעים אלה גורם אפופטוזיס, מעצר מחזור התא, או הזדקנות יכול להוביל למחלות1. נגעי DNA יכולים להיות מטופלים על ידי כל אחד ממסלולי תיקון ה- DNA העיקריים הבאים: DR (תיקון היפוך ישיר), אשר מתקן בעיקר בסיסים אלקילטים2; BER (תיקון כריתת בסיס), המכוון לשגיאות בסיסי DNA לא מגושמים ולשברי DNA חד-גדיליים (SSB)3; NER (תיקון כריתת נוקלאוטידים) תיקון נגעי DNA מגושמים ומעוותים סליל4; MMR (תיקון אי-התאמה) מתמקד בעיקר באי-התאמות DNA, לולאות החדרה/מחיקה (IDLs) ונזקי בסיס מסוימים5; NHEJ (חיבור קצה לא הומולוגי) ו- HRR (תיקון רקומבינציה הומולוגית) הפעילים שניהם בהפסקות DNA דו-גדיליות (DSBs)6; ו-TLS (translesion synthesis), שהוא מנגנון מעקף נגע DNA7. למרות שלמסלולים אלה יש ייחודיות מצע ברורה, יש ביניהם חפיפות מסוימות כדי להבטיח יתירות לתיקון יעיל. הבנת הפעולה של מסלולי תיקון הדנ"א השונים בשלבים שונים של מחזור התא היא חיונית מכיוון שגורמי תיקון דנ"א אלה יכולים לשמש מטרות חיוניות לגישות טיפוליות לטיפול בסרטן, הזדקנות והפרעות נוירולוגיות 8,9.
DNA חד-גדילי (ssDNA) נוצר לאורך מחזור התא עקב תיקון נגעי DNA שנוצרו על ידי גורמים אנדוגניים ואקסוגניים הפוגעים ב- DNA. בעקה גנוטוקסית, ssDNA נוצר בשפע בשלבי S ו-G2 שבהם HRR ו-MMR הם בעלי הפעילות הגבוהה ביותר שלהם וכאשר מנגנון השכפול נתקע או קורס כאשר הם נתקלים בנגעי DNA 6,10,11. מסלולי תיקון דנ"א אחרים (למשל, NHEJ/microhomology-mediated end joining (MMEJ)/Single strand annealing [SSA]) מייצרים גם הם ssDNA במהלך תיקון DSB12. עקבות ssDNA אלה נובעות בדרך כלל מכריתת DNA, המבוצעת על ידי אקסונוקלאזות כגון EXO1, DNA2 ו- CtIP במהלך HR ו- MMR, אנדונוקלאזות כגון XPF ו- XPG במהלך NER, או באמצעות הפעולה המשולבת של POLB ו- FEN1 במהלך BER 4,13,14,15,16,17,18,19 . הודות לעבודת מנגנון השכפול, עקבות ssDNA נוצרות גם כאשר מנחת המסוקים של הדנ"א משחרר את הדנ"א מול פולימראזות משוכפלות הקשורות ל-PCNA20. לעומת זאת, בשלב G1, היעדר HRR ושכפול DNA והפעילות המוגבלת של MMR מפחיתים את היקף עקבות ssDNA הנוצרים ולכן מאתגרים יותר לאיתור 10,11,21.
רצועות ssDNA תאיות הן מבנים רגישים מאוד שיש להגן עליהם כדי למנוע היווצרות DSB. זה מושג על ידי ציפוי עקבות ssDNA עם RPA. RPA הוא קומפלקס חלבונים הטרוטרימרי שופע המורכב מתת-יחידות מרובות (RPA1, RPA2 ו-RPA3, המכונים גם RPA70, RPA32 ו-RPA14, בהתאמה), אשר באים לידי ביטוי בכל מקום לאורך מחזורהתא 22. כל תת-יחידה של RPA מכילה תחום קושר DNA (DBD), המסוגל לקיים אינטראקציה עם 4-6 נוקלאוטידים, ותת-היחידות המשולבות יוצרות ליבת טרימריזציה יציבה. בסך הכל, RPA נקשר לכ-20-30 נוקלאוטידים בעלי זיקה תת-ננומולרית23,24.
שיטות קונבנציונליות משתמשות במיקרוסקופ אימונופלואורסצנטי (IF) כדי להמחיש מוקדי ssDNA על ידי תיוג 5-bromo-2'-deoxyuridine (BrdU) המשולב בדנ"א גנומי באמצעות נוגדני BrdU25. גישה זו מסתמכת על העובדה כי נוגדני BrdU יכולים לזהות BrdU רק ב- ssDNA25 חשוף. למרות שגישה זו פשוטה, היא מציגה גם מגבלות מסוימות. לדוגמה, תאים מטופלים מראש כדי לשלב BrdU לפני תחילת הניסוי, וזה לוקח זמן רב ויכול להפריע למשפיעים במורד הזרם. לכן, זיהוי ssDNA מבוסס BrdU מוגבל לתאים משוכפלים ולא ניתן להשתמש בו עבור תאים שקטים. זה לא כולל את היישום של שיטה זו כדי לחקור תיקון DNA בתאים שאינם משתכפלים למרות חשיבותה במספר מחלות כגון סרטן ניוון עצבי 5,26. בנוסף, מכיוון שהמבנים של BrdU ו-EdU דומים מאוד, רוב נוגדני BrdU מציגים תגובתיות צולבת כלפי EdU, שיש לקחת בחשבון כאשר מכוונים לניסויי תיוג כפול27. צביעת RPA שימשה בעבר להצגת מוקדי ssDNA בעיקר בתאי שלב S; עם זאת, כמה מאמרים השתמשו בו בהצלחה גם מחוץ לשלב S 28,29,30,31,32,33,34,35. הפרוטוקול הבא מנצל ביעילות את התכונות של RPA, ומאפשר הדמיה של מוקדי ssDNA בעקבות נזק לדנ"א בשלב G1 של מחזור התא (אם כי ניתן להשתמש בו בכל שלבי מחזור התא).
Protocol
1. תחזוקה של תאי אפיתל פיגמנט רשתית hTERT-אימורטליזציה (RPE1)
- שמור על קווי תאי RPE1 במדיום הנשר המעובד (DMEM) של Dulbecco בתוספת 10% סרום בקר עוברי מומת בחום (Hi-FBS) ו- 100 מיקרוגרם / מ"ל פניצילין-סטרפטומיצין (המכונה מעתה והלאה מדיום תרבית) באינקובטור לח עם 5% CO2 ב 37 ° C. לגידול שגרתי, יש לגדל תאי RPE1 בצלחת שטופלה בתרבית רקמה בקוטר 15 ס"מ ולפצל אותם כאשר מגיעים למפגש של 80-90% (~16-18 × 106 תאים למנה של 15 ס"מ).
- בעת פיצול, להסיר את המדיום ולשטוף את התאים עם 10 מ"ל של 1x פוספט buffered מלוחים (PBS).
- הוסף 3 מ"ל של 0.05% טריפסין-EDTA כדי לכסות את כל פני השטח של המנה. שמור על התאים בטמפרטורה של 37°C עם הטריפסין עד שהם מתנתקים.
- לאחר טריפסיניזציה, להשעות מחדש את התאים עם מדיום תרבית ולסובב אותם למטה ב 150 × גרם במשך 5 דקות בטמפרטורת החדר (RT, 22-25 ° C). הסר את supernatant בעדינות resuspend את התאים ב 10 מ"ל של מדיום תרבית.
- זרע 1.6-1.8 × 106 תאים לתוך צלחת חדשה 15 ס"מ (~ 1 מ"ל של השעיית התא).
הערה: כל עבודת תרבית הרקמה צריכה להיעשות תחת רמות בטיחות BSL-2. זמן הדגירה של טריפסיניזציה תלוי במפגש תאים. בדרך כלל, התהליך לוקח 2-3 דקות כדי להשלים עבור צלחת 90% confluent. תאים צריכים להיבדק לזיהום מיקופלסמה על בסיס קבוע עם ערכות זמינות מסחרית (ראה דוגמאות בטבלת החומרים).
2. siRNA מפיל את גן העניין (GOI)
- זרע 1.0 × 106 תאי RPE1 לתוך צלחת טיפול תרבית רקמה 10 ס"מ עם 10 מ"ל של מדיום תרבית ביום שלפני הטרנספקציה.
- ביום הטרנספקציה, מורכב siRNA. עבור צלחת של 10 ס"מ, השתמש בריכוז סופי של 20 ננומטר siRPA2 ו 12 μL של מגיב טרנספקציה מבוסס שומנים ב 500 μL של מדיום טרנספקציה בסרום נמוך. ערבבו בעדינות את כל הרכיבים על ידי הזזת הצינור ודגרו ב-RT (22-25°C) למשך 5 דקות.
- הוסיפו את תערובת ה-siRNA המורכבת לתאים בטיפה ודגרו על התאים עם ה-siRNA במשך 48 שעות.
3. סנכרון תאי RPE1 לשלב G0
- טריפסין את תאי RPE1 משלב 2.3 כמתואר בסעיף 1 (~2 × 106 תאים).
- מעבירים את מתלה התא לצינורות צנטריפוגות 15 מ"ל וצנטריפוגות אותו ב 150 × גרם, RT (22-25 ° C) למשך 5 דקות.
- הסר את supernatant ו resuspend את התאים ב 12 מ"ל של PBS. צנטריפוגה את התאים ב 150 × גרם ב RT (22-25 ° C) במשך 5 דקות. חזור על הסרת הסופרנאטנט והצנטריפוגה פעמיים.
- השהה מחדש את התאים ב- 10 מ"ל של DMEM ללא סרום בתוספת 100 מיקרוגרם / מ"ל פניצילין-סטרפטומיצין,1 מ"מ נתרן פירובט, 15 מ"מ HEPES, וצלחת אותם על צלחת תרבית רקמות 10 ס"מ.
הערה: אם התאים נוטים להתגבש, השהה אותם מחדש ב-1 מ"ל בלבד של DMEM ללא סרום וטפט אותם מעלה ומטה 5x באמצעות קצה P1000 כדי לעקור את הגושים לפני דילול התרחיף עד לנפח סופי של 10 מ"ל. - לאחר 24 שעות של רעב בסרום, הציגו את סבב ההשתקה השני באמצעות אותו הליך המתואר בסעיף 2 על ידי הוספת siRNA מורכב לתאים הרעבים בסרום.
- שמור את תאי RPE1 ב- DMEM ללא סרום למשך 72 שעות לפני שתמשיך לשחרור G1.
4. ציפוי כיסוי ושחרור תאים לשלב G1
- עקרו פינצטה עם 70% אתנול והניחו כיסוי זכוכית יחיד (קוטר 12 מ"מ ועובי #1.5 [0.17 מ"מ]) בבאר של צלחת 24 בארות.
- דללו את מטריצת ציפוי הוויטרונקטין עם PBS לקבלת ריכוז סופי של 10 מיקרוגרם/מ"ל. הוסף 500 μL של תמיסת ויטרונקטין לכל באר המכילה את הכיסויים ודגר במשך שעה אחת ב- RT.
- הסר את תמיסת הציפוי ושטוף את הכיסויים עם 1 מ"ל של PBS.
- נתקו את תאי RPE1 המורעבים בסרום מהצלחת המטופלת בתרבית רקמה בקוטר 10 ס"מ באמצעות 1 מ"ל של 0.05% טריפסין לאחר שטיפת PBS למשך דקה אחת בטמפרטורה של 37°C.
הערה: התאים מתנתקים הרבה יותר מהר לאחר רעב בסרום. היזהר בעת שטיפת התאים עם PBS והשתמש בזמני טריפסיניזציה קצרים. - כדי להשבית את הטריפסין, להשעות מחדש את תאי RPE1 בסך הכל 6 מ"ל של מדיום תרבית. הסר את הטריפסין הבלתי פעיל על ידי סיבוב התאים כלפי מטה באמצעות 150 × גרם ב- RT (22-25 ° C) למשך 5 דקות.
- להשהות מחדש את התאים ב 1 מ"ל של מדיום תרבית ולמדוד את מספר התא.
- זרע 4 × 104 תאי RPE1 על הכיסוי המצופה בסך הכל 500 μL של מדיום תרבית.
הערה: ודא שיכולת הכדאיות של התא גבוהה מ- 90% לפני שתמשיך לשלבים במורד הזרם. ניתן להעריך במהירות את כדאיות התא על ידי צביעה כחולה טריפאן במהלך שלב ספירת התא. - לאחר 6 שעות של ציפוי התאים למדיום התרבית, תאים משוחררי G0 יהיו בשלב G1 מוקדם. בצע ניסויים ב- G1 בחלון זה של 6-12 שעות לפני שהתאים מתחילים להיכנס לשלב S.
- לפני הצגת נזק לדנ"א, יש לפעום את התאים עם 10 μM 5-ethynyl-2'-deoxyuridine (EdU) למשך 30 דקות ב-37°C, מדולל במדיום תרבית.
- הסר את המדיום המכיל EdU ורדוף אחר התאים עם 10 מיקרומטר תימידין במשך 10 דקות ב 37 ° C כדי למנוע שילוב EdU שארית במהלך השראת נזק DNA.
- הסר את המדיום עם thymidine ולטפל בתאים עם 250 μM H2O2 במשך 1 שעות, מדולל במדיום תרבית.
5. צביעה immunofluorescence של ssDNA
- יש לשטוף תאים פעם אחת עם 1 מ"ל של PBS RT (22-25°C) כדי להסיר את רכיבי המדיום והסרום.
הערה: היו עדינים בעת שטיפת התאים כדי למנוע ניתוק וייבוש. אין לעבד בארות רבות בו זמנית. - טרום מיצוי: דגרו על התאים שנשטפו ב-1 מ"ל של מאגר מיצוי CSK (טבלה 1) למשך 5 דקות ב-RT (22-25°C).
הערה: מיצוי מקדים של CSK מסיר את כל החלבונים שאינם קשורים לכרומטין, כולל RPA2 מסיס.
אזהרה: Triton X-100 מזיק בבליעה ועלול לגרום לגירוי בעור ולנזק לעיניים. - הסר את מאגר CSK מהתאים ותקן אותם ישירות על ידי הוספת 0.5 מ"ל של תמיסת פרפורמלדהיד 3.6% (ב- PBS) המכילה 0.05% Triton X-100 למשך 10 דקות ב- RT (22-25 ° C).
זהירות: חשוב להכין 3.6% PFA ממלאי PFA 32% טרי. פרפורמלדהיד עלול לגרום לנזק חמור לעיניים, לגירוי בעור ולגירוי נשימתי. - שטפו את התאים פעם אחת עם 1 מ"ל PBS המכיל 0.05% Triton X-100 כדי להסיר את ה-PFA.
- חדירה נוספת של תאים באמצעות 1 מ"ל PBS המכיל 0.5% Triton X-100 למשך 15 דקות ב-RT (22-25°C).
- תגובת EdU click-IT להמחשת תאים משוכפלים (שלב S)
- הסר את תמיסת החדירה ושטוף את התאים פי 2 באמצעות 1 מ"ל של חיץ חוסם (טבלה 1).
אזהרה: אלבומין בסרום בקר (BSA) עלול לגרום לגירוי נשימתי. - הוסף 1 מ"ל של חיץ חוסם (טבלה 1) וניער בעדינות את הצלחת המכילה את הכיסוי למשך 10 דקות ב- RT (22-25 ° C).
- הסר את מאגר החסימה והוסף 500 μL של קוקטייל תגובת לחיצה המכיל פיקוליל אזיד 647 (טבלה 1). יש לדגור על החלקות הכיסוי במשך 30 דקות ב-RT (22-25°C) באמצעות נדנוד עדין ולבצע דגירות במורד הזרם בחושך.
הערה: בעת שימוש בנוגדני BrdU, השתמש בכמות כפולה (1 מ"ל) ובזמן (60 דקות) עבור תגובת הלחיצה כפי שהומלץ על ידי היצרן כדי להבטיח שהתגובה רוויה וה- EdU המשולב מסומן. זה מגביל את התגובה הצולבת של נוגדני BrdU27.
- הסר את תמיסת החדירה ושטוף את התאים פי 2 באמצעות 1 מ"ל של חיץ חוסם (טבלה 1).
- הסירו את תערובת תגובת הלחיצה ושטפו את התאים פעמיים עם PBS עם 0.05% Triton X-100 למשך 10 דקות ב-RT (22-25°C) (איור 1 ואיור 2).
- הוסף 1 מ"ל של חיץ חוסם ודגור ב- RT (22-25 ° C) למשך 30 דקות. לחלופין, שמור את התאים במאגר חוסם ב -4 מעלות צלזיוס למשך הלילה.
- יש למרוח נוגדן ראשוני (נוגד RPA2 חולדה, דילול 1:1,000) למשך שעתיים ב-RT (22-25°C) ב-250-500 מיקרוליטר של חיץ חוסם עם נדנוד עדין.
- שטפו את התאים פי 2 עם PBS המכיל 0.05% Triton X-100 כדי להסיר במהירות את רוב תמיסת הנוגדנים.
- המשך לשטוף את התאים במשך 3 x 10 דקות עם מאגר חוסם ב RT (22-25 ° C).
- יש למרוח נוגדן משני (אנטי-חולדה Alexa-488, דילול 1:1,000) ב-250-500 מיקרוליטר של חיץ חוסם ב-RT (22-25°C) למשך שעתיים עם נדנוד עדין.
- שטפו את התאים עם חיץ חוסם 2x כדי להסיר במהירות את רוב הנוגדן המשני. המשך לשטוף את התאים במשך 3 x 10 דקות עם PBS המכיל 0.05% Triton X-100 ב RT (22-25 ° C).
- כדי לנטרל את הגרעינים, יש לשטוף תאים פעם אחת עם PBS המכיל 0.05% Triton X-100 ו-1 μg/mL 4',6-diamidino-2-phenylindole (DAPI) למשך 10 דקות ב-RT (22-25°C). שטפו תאים פעם אחת עם PBS למשך 5 דקות ב-RT (22-25°C).
- הרכיבו את זכוכית הכיסוי על שקופיות מיקרוסקופ באמצעות 10 מיקרוליטר של אמצעי הרכבה/כיסוי. טבלו את הכיסויים במים מזוקקים לפני ההרכבה כדי להיפטר מגבישי מלח. דמיינו את השקופיות ביום המחרת ואחסנו אותן בטמפרטורה של 4°C למשך שבועות (איור 3).
6. רכישה וכימות של תמונות
- כדי לצלם תמונות, השתמש בכל מיקרוסקופ אפיפלואורסצנטי זמין המצויד בערכות סינון שגרתיות כדי לצלם ערוצי DAPI, FITC ו- Cy5 עם הגדלה של לפחות 60-63x, צמצם מספרי גבוה ומטרות שמן כדי להמחיש מוקדים גרעיניים.
הערה: עירור DAPI אופטימלי הוא ~ 359 ננומטר; עירור Alexa 488 הוא ~ 488 ננומטר; בעוד עירור Alexa 647 הוא ~ 647 ננומטר. - לניתוח תמונות, פתחו קובצי תמונה בפיג'י/ImageJ.
- הכינו מסכות גרעיניות באמצעות צביעת DAPI (איור 4A-F ווידאו משלים S1).
- פתח את תמונת DAPI.
- בחר תהליך | שפר את הניגודיות והגדר פיקסל רווי ל- 0.35.
- קליק תהליך | בינארי | המר למסיכה. בחר בינארי | מלא חורים ולחץ על נתח | לנתח חלקיקים. הגדר את הגודל ל - 10-Infinity.
- במנהל החזר ההשקעה, לחץ על הצג הכל.
- מציאת מוקדי RPA2 בגרעין (איור 4G,H ווידאו משלים S1)
- פתח את תמונת RPA2.
- בחר תהליך | מצא את מקסימה. הגדר את ההבלטה לערך המדגיש את מוקדי RPA2 (בין 500 ל- 750), ומפריד בינה לבין הרקע.
- לבסוף, לחץ על לחצן מדידה במנהל החזר ההשקעה.
- חשב את המספר הכולל של מוקדי ssDNA גרעיניים על ידי חלוקת הערך בעמודה RawinDen ב- 255 (הערך המרבי של עוצמת הפיקסלים בכל מוקד).
- בצע ניתוח סטטיסטי באמצעות כלי התוכנה הסטטיסטי המועדף.
הערה: אל תכלול את כל התאים החיוביים ל-EdU ואת מסכות ה-DAPI המפולחות באופן שגוי מהניתוח.
- הכינו מסכות גרעיניות באמצעות צביעת DAPI (איור 4A-F ווידאו משלים S1).
תוצאות
כדי להתגבר על המגבלות של זיהוי ssDNA ב- G1, השתמשנו ב- RPA2, אשר משפר הן את הספציפיות והן את העוצמה של זיהוי מוקדי ssDNA35. כדי להשיג סנכרון תאים מדויק, השתמשנו בתאי RPE1 שניתן להרעיב אותם בסרום ביעילות ולסנכרן אותם לשלב G0. לאחר מכן ניתן לגרום להם להיכנס מחדש למחזור התא על ידי הוספת סרום לאחר חסך בסרום. כדי לאשר את יעילות הסנכרון, תייגנו את התאים עם EdU ואת תכולת הדנ"א שלהם עם פרופידיום-יודי. בהמשך אספנו תוצאות איכותיות וכמותיות באמצעות ציטומטריית זרימה (איור משלים S1A). תרשימי הנקודות מראים כי לאחר 72 שעות של רעב בסרום, ~98% מהתאים נמצאים בשלב G0. לאחר הוספת מדיה המכילה סרום במשך 6 שעות, תאים נכנסים מחדש למחזור התא (כפי שניתן לראות על-ידי העלייה ברמות p27 באיור 1A), כאשר ~97% מהתאים נמצאים ב-G1, בעוד שיש להם רק <1% תאים בשלב S, <2% תאים בשלב G2 (איור משלים S1A). לאחר 20-28 שעות של הוספת סרום לתאים, הם עוברים בהדרגה דרך שלב S, כפי שניתן לראות בחלקות ציטומטריית הזרימה (איור משלים S1A). פרוטוקול סנכרון תאים זה נותן אוכלוסיית G1 טהורה ~ 97% (6 שעות לאחר הוספת סרום לאחר 72 שעות של רעב בסרום). כדי לאמת עוד יותר את יעילות הסנכרון, השווינו את הביטוי של סמני מחזור התא לאחר שחרור הסרום באמצעות קרישה מערבית (איור 1A ואיור משלים S1B), ובמקביל ביצענו בדיקת שילוב EdU כדי להמחיש שכפול דנ"א. צביעת EdU מדגישה גם את יעילות הסנכרון ואת היעדר שכפול הדנ"א בשלב G1 (איור 1B,C).
שיטות קונבנציונליות לזיהוי ssDNA בתאי יונקים מסתמכות על זיהוי BrdU ב- ssDNA. איור 2A מדגים שבטיפול ב-H2O2 וב-neocarzinostatin (NCS), ניתן היה לזהות את מוקדי BrdU רק בתאי שלב S, בעוד שלא ניתן היה לזהות מוקדי ssDNA בתאים שאינם בשלב S. צביעת נוגדני BrdU הראתה גם צביעת רקע גרעין בולטת שניתן היה לזהות בכל הגרעינים, ללא תלות בשלב מחזור התא או בטיפולים שיושמו. באמצעות פרוטוקול הקליק של EdU המתואר כאן, לא יכולנו לזהות לוקליזציה משותפת של מוקדי EdU ו-BrdU, כפי שניתן לראות בדגימות הלא מטופלות של איור 2A. כדי לשלול לחלוטין כל אות BrdU שעלה מתגובתיות צולבת, נמנענו מתיוג EdU ובמקום זאת השתמשנו בציקלין A2 כסמן S-G2. עם זאת, צביעת ציקלין A2 לא אפשרה מיצוי מוקדם של CSK, ובמצב זה לא ראינו מוקדי BrdU, אפילו לאחר עקה גנוטוקסית (איור משלים S2A). זה מדגיש את העובדה כי מיצוי מראש CSK הוא הכרחי עבור צביעת ssDNA מבוסס BrdU. כביקורת, בדקנו צביעת נוגדנים BrdU בתנאי דנטורציה. זה פותח את הדנ"א כדי לחשוף את BrdU המשולב, אשר מגלה כי BrdU שולב באופן אחיד (איור משלים S2B).
לעומת זאת, צביעת RPA2 מראה היווצרות מוקדים תלויי NCS ו-H2O2 לא רק בשלב S אלא גם בשלבים אחרים של מחזור התא (איור 2B). כביקורת, טיפלנו גם בתאים עם HU, אשר גורם רק להצטברות ssDNA בתאים שעברו שכפול. כצפוי, זיהינו עלייה באות רק בטיפול HU עם נוגדן RPA2 בתאים חיוביים ל- EdU, מה שמדגיש את הספציפיות של גישה זו. נוגדן RPA2 יכול גם לזהות היווצרות ssDNA טבעית במהלך שכפול בהיעדר עקה גנוטוקסית אקסוגנית (איור 2B). האופי הרגיש מאוד של נוגדן RPA2 הניע אותנו לנסות להשתמש בו בשלב G1 שבו צביעת BrdU קונבנציונלית לא הצליחה לזהות שום אות על עקה גנוטוקסית (איור משלים S2C). איור 3A מראה כי היווצרות מוקדי ssDNA בטיפול H2O2 הייתה ניתנת לזיהוי בעת שימוש בנוגדן אנטי-RPA2, אפילו ב-G1. חלה עלייה משמעותית במספר מוקדי RPA2 בגרעינים אלה לאחר טיפול H2O2 (איור 3B). מוקדים אלה היו ספציפיים ל-RPA2 מאחר שההשתקה של RPA2 ביטלה את אות ה-IF (איור 3A,B). איור 3C ואיור משלים S1C מראים את היעילות של השתקת RPA2 בתאים האלה. בהשוואה לשיטות קונבנציונליות, זיהוי מבוסס RPA2 של ssDNA הוא רגיש מאוד, ולכן ניתן להרחיב את יישומו לתאי פאזת G1.
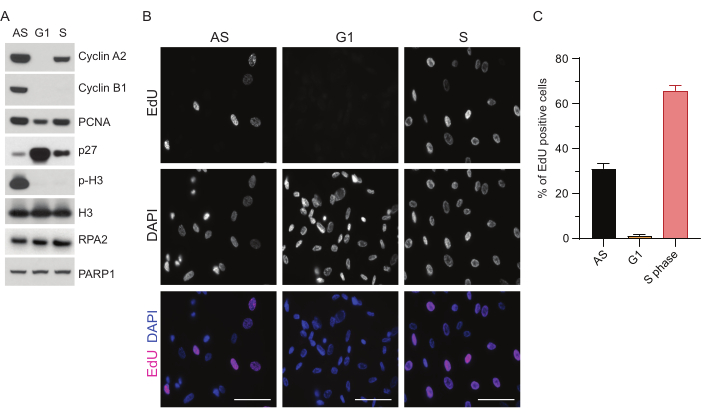
איור 1: יעילות הסנכרון של תאי RPE1 לאחר רעב בסרום. (A) כתמים חיסוניים מראים רמות חלבון מצוינות בתאי RPE1 מסונכרנים בשלב אסינכרוני, G1 ו-S. (B) תמונות מייצגות מציגות תאי RPE1 מסונכרנים אסינכרוניים, G1 ו-S שנחשפו ל-10 μM EdU במשך 30 דקות לפני הקיבוע והודמיינו על ידי תגובת Click-IT. DAPI שימש להכתמת DNA גרעיני. פסי קנה מידה = 50 מיקרומטר. (C) גרף מציג אחוז תאים חיוביים ל-EdU על פני כלל אוכלוסיית התאים שהוערכה על-ידי DAPI. שורת השגיאה מייצגת שגיאת תקן של ממוצע, והמספרים המנותחים של גרעינים היו: AS n = 219, G1 n = 630, S n = 437. קיצורים: RPE1 = hTERT-immortalized retinal pigment epithelial cells; AS = אסינכרוני; EdU = 5-ethynyl-2'-deoxyuridine; DAPI = 4',6-diamidino-2-phenylindole. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
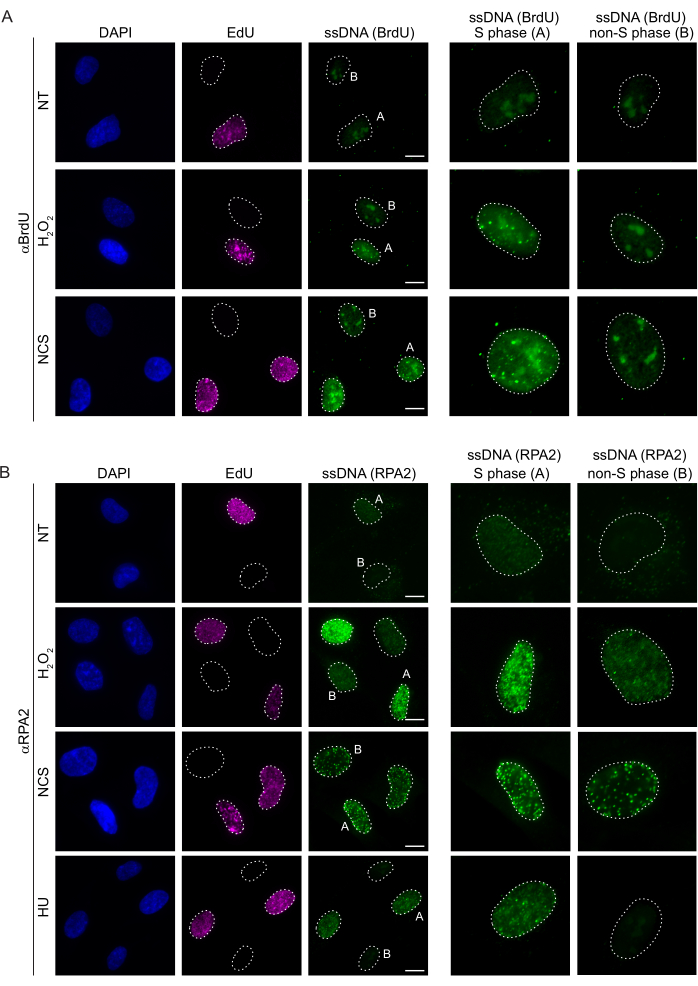
איור 2: זיהוי ssDNA עם נוגדן BrdU או נוגדן RPA2 על נזק לדנ"א. (A) תמונות מייצגות מדגימות מוקדי ssDNA באמצעות αBrdU (ירוק), תאי פאזת S מודגשים על-ידי EdU (סגול), ו-DAPI שימש להכתמת דנ"א גרעיני (כחול). תאי RPE1 נשמרו ב-BrdU של 10 מיקרומטר במשך 48 שעות לפני כל טיפול נוסף. לאחר 48 שעות, התאים היו פועמים עם 10 מיקרומטר EdU במשך 30 דקות ואחריו טיפול של H2O2 (250 מיקרומטר) במשך 1 שעה או Neocarzinostatin (0.5 מיקרוגרם / מ"ל) במשך 4 שעות. התאים תוקנו לאחר מיצוי מוקדם של CSK. קו מקווקו לבן מציין את הגבול של כל גרעין. סרגל קנה מידה = 5 מיקרומטר. לוחות מימין הם תמונות מוגדלות של גרעיני הפאזה S או הלא-S שצוינו. (B) תמונות מייצגות ממחישות מוקדי ssDNA באמצעות נוגדן αRPA2 (ירוק). תאי שלב S מודגשים על ידי EdU (סגול), ו- DAPI שימש כדי להכתים DNA גרעיני (כחול). תאי RPE1 היו פועמים עם 10 μM EdU במשך 30 דקות ואחריו 1 h H2O2 (250 μM), 4 שעות של Hydroxyurea (2 mM), או 4 שעות של NCS (0.5 מיקרוגרם / מ"ל). התאים תוקנו לאחר מיצוי מוקדם של CSK. קו מקווקו לבן מציין את הגבול של כל גרעין. סרגל קנה מידה = 10 מיקרומטר. לוחות מימין הם תמונות מוגדלות של גרעיני הפאזה S או הלא-S שצוינו. קיצורים: ssDNA = DNA חד-גדילי; BrdU = 5-bromo-2'-deoxyuridine; DAPI = 4',6-diamidino-2-phenylindole; RPE1 = hTERT-אימורטליזציה פיגמנט תאי אפיתל; EdU = 5-ethynyl-2'-deoxyuridine; NCS = Neocarzinostatin; HU = hydroxyurea. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
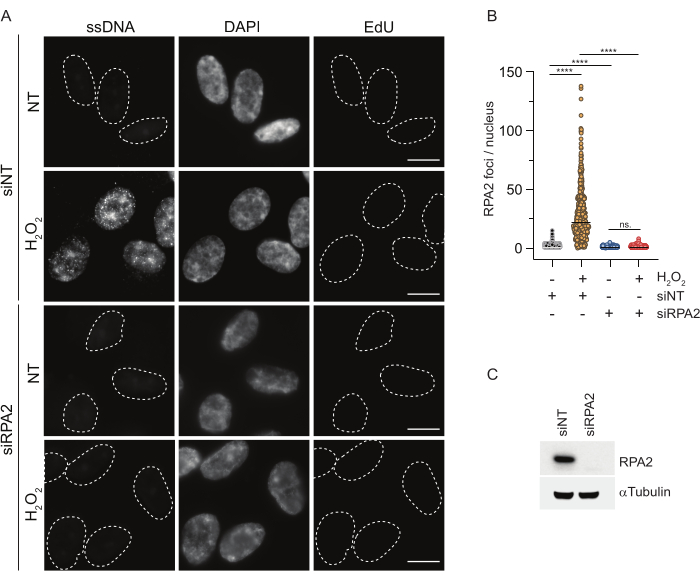
איור 3: זיהוי מוקדי ssDNA בשלב G1 באמצעות נוגדן RPA2. (A) תאי RPE1 הודבקו ב-siRNA המכוון ל-RPA2 או בבקרת siRNA שאינה ממוקדת, ולאחר מכן סונכרנו ב-G1 וסומנו בדופק עם 10 μM EdU במשך 30 דקות לפני שטיפלו בהם עם H2O2 (250 μM) למשך שעה אחת היכן שצוין. DAPI שימש להכתמת DNA גרעיני. התאים תוקנו לאחר מיצוי מוקדם של CSK. קו מקווקו לבן מציין את הגבול של כל גרעין. סרגל קנה מידה = 5 מיקרומטר. (B) המדידות למספר המוקדים/גרעין RPA2 בוצעו משני ניסויים בלתי תלויים. רק תאים שליליים EdU נלקחו בחשבון במהלך הניתוח. קווים מייצגים את הערך הממוצע בחלקות. בדיקת ANOVA לא פרמטרית (Kruskal-Wallis) בוצעה לניתוח סטטיסטי. הכוכבים מציינים P < 0.0001. מספר הגרעינים שנותחו היו: siNT no H2O2 n = 513, siNT H2O2 n = 603, siRPA2 no H2O2 n = 266, siRPA2 H2O2 n = 536. (C) היעילות של הפלת siRNA מוצגת באימונובלוטינג. קיצורים: siNT = בקרת siRNA שאינה ממוקדת; BrdU = 5-bromo-2'-deoxyuridine; DAPI = 4',6-diamidino-2-phenylindole; RPE1 = hTERT-אימורטליזציה פיגמנט תאי אפיתל; EdU = 5-ethynyl-2'-deoxyuridine. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.

איור 4: כימות מוקדי ssDNA באמצעות פיג'י. צעדים מפורטים בפיג'י המראים כיצד להעריך מספרי מוקדי RPA2 בגרעין. (א-ה) יצירת מסכה גרעינית באמצעות ערוץ DAPI. (פ-ח) סף לזיהוי מוקדי ssDNA גרעיניים בודדים מאות הרקע. קיצורים: ssDNA = DNA חד-גדילי; DAPI = 4',6-diamidino-2-phenylindole. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
| חיץ ציטו-שלד (CSK) | |
| צינורות pH 7.0 | 10 מ"מ |
| NaCl | 100 מ"מ |
| EDTA pH 8 | 1 מ"מ |
| MgCl2 | 3 מ"מ |
| די-סוכרוז | 300 מ"מ |
| טריטון X-100 | 0.20% |
| קוקטייל מעכב פוספטאז | 1 טבליה לכל 10 מ"ל |
| קוקטייל מעכב פרוטאז | 1 טבליה לכל 10 מ"ל |
| מדולל ב ddH2O | לא ישים |
| חיץ כביסה | |
| טריטון X-100 | 0.05% |
| מדולל ב-PBS | לא ישים |
| מאגר חדירות | |
| טריטון X-100 | 0.50% |
| מדולל ב-PBS | לא ישים |
| פתרון קיבוע | |
| פרפורמאלדהיד | 3.60% |
| טריטון X-100 | 0.05% |
| מדולל ב-PBS | לא ישים |
| מאגר חוסם | |
| אלבומין בסרום בקר (BSA) | 5% |
| טריטון X-100 | 0.10% |
| מדולל ב-PBS | לא ישים |
| קוקטייל התגובה Click-iT Plus | |
| מאגר תגובה אחד של Click-iT | 435 מ"ל |
| פתרון Alexa Fluor PCA | 5 מ"ל |
| CuSO4-נחושת הגנה premix | 10 מ"ל |
| 1x תוסף מאגר Click-iT | 50 מ"ל |
| עוצמת קול כוללת | 500 מ"ל |
טבלה 1: הרכב המאגרים המשמשים בפרוטוקול זה.
איור משלים S1. (A) תאי RPE1 סונכרנו לשלב G0 באמצעות רעב בסרום במשך 72 שעות, ולאחר מכן שוחררו לשלבים שונים של מחזור התא על-ידי החדרה מחדש של הסרום. תרשימי נקודות מראים תאים בשלב G0/G1, S או G2/M, כאשר השעות מציינות את הזמן לאחר הוספה מחדש של סרום לאחר רעב בסרום. תרשים מימין מציג את אחוז תאי G0/G1, S ו- G2/M בכל תנאי. ניתוח FACS בוצע באמצעות ערכת הפצת תאים זמינה מסחרית באמצעות EdU ופרופידיום יודיד על פי המלצות היצרן. (B) סריקות כתם מערבי לא חתוך עבור איור 1. מספרים מראים סמני משקל מולקולריים ב-kDa. PARP1 שימש כבקרת העמסה והועמס על הג'ל שפותח גם כנגד CCNA2, p27 (שהופשט עוד יותר עבור PCNA), ו-pH3 (S10) (הופשט עוד יותר עבור H3) על ידי חיתוך הממברנה. CCNB1 ו-RPA2 הועמסו על ג'ל נפרד, תוך שימוש באותה כמות של חלבון ליזט כדי להבטיח השוואה. (C) סריקות כתם מערבי לא חתוך עבור איור 3. מספרים מראים סמני משקל מולקולריים ב-kDa. קיצור: EdU = 5-ethynyl-2'-deoxyuridine. אנא לחץ כאן כדי להוריד קובץ זה.
איור משלים S2: (A) תמונות מייצגות ממחישות מוקדי ssDNA באמצעות נוגדן BrdU (ירוק); תאי שלב S מודגשים על-ידי ציקלין A2 (אדום); ו-DAPI שימש להכתמת דנ"א גרעיני (כחול). תאי RPE1 נשמרו ב-BrdU של 10 מיקרומטר במשך 48 שעות לפני המשך הטיפול. לאחר 48 שעות, תאים טופלו עם H2O2 (250 μM) במשך 1 שעות או Neocarzinostatin (0.5 מיקרוגרם / מ"ל) במשך 4 שעות לפני קיבוע. קו מקווקו לבן מציין את הגבול של כל גרעין. סרגל קנה מידה = 5 מיקרומטר. (B) צביעת BrdU של תאי RPE1 עם וללא מצב דנטורציה. תאי RPE1 אסינכרוניים טופלו מראש עם 10 μM BrdU במשך 48 שעות. סרגל קנה מידה = 10 מיקרומטר. (C) המדידות למספר המוקדים/גרעין BrdU בוצעו משני ניסויים עצמאיים בתאי RPE1 מסונכרנים G1. רק תאים שליליים EdU נלקחו בחשבון במהלך הניתוח. קווים מייצגים את הערך הממוצע בחלקות. בדיקת ANOVA לא פרמטרית (Kruskal-Wallis) בוצעה לניתוח סטטיסטי. האות 'ns' מצביעה על הבדל לא משמעותי. מספר הגרעינים שנותחו היו: NT n = 52, NCS n = 105, H2O2 n = 82. קיצורים: siNT = בקרת siRNA שאינה ממוקדת; BrdU = 5-bromo-2'-deoxyuridine; DAPI = 4',6-diamidino-2-phenylindole; RPE1 = hTERT-אימורטליזציה פיגמנט תאי אפיתל; NCS = Neocarzinostatin. אנא לחץ כאן כדי להוריד קובץ זה.
וידאו משלים S1: הקלטת מסך של ניתוח מוקדי RPA2 מבוסס פיג'י. אנא לחץ כאן כדי להוריד קובץ זה.
Discussion
שמירה על תרבית תאים בריאה ונטולת מיקופלסמה היא קריטית לכל הניסויים שתוארו לעיל. לתאי RPE1 יש חיבור חזק לכלי פלסטיק המטופלים בתרבית רקמות תחת אמצעי תרבית רגילים; עם זאת, מאפייני הקשירה שלהם פוחתים באופן משמעותי כאשר הם נשמרים בתנאים נטולי סרום. בנוסף, כדי לצלם תמונות ברזולוציה גבוהה של מוקדי ssDNA תחת מיקרוסקופ, התאים צריכים להיות מצופים על זכוכית כיסוי בעובי 0.17 מ"מ, שאינה הידרופילית מספיק כדי לתמוך בחיבור תקין של תאי RPE1. ללא תאים שטוחים כראוי ומפוזרים באופן שווה, זה מאתגר מאוד לדמיין מוקדי ssDNA בודדים. לכן, קריטי לבחור את חומר הציפוי המתאים (למשל, ויטרונקטין) ולהשאיר מספיק זמן (6-12 שעות) לתאים להתפשט ולהתחבר לאחר שחרורם לשלב G1.
חלק מאתגר בפרוטוקול הוא להשיג תאי RPE1 הומוגניים מסונכרנים G1. זה דורש שני שלבים קריטיים. ראשית, להרעבה יעילה בסרום, יש לבצע טריפסיניזציה, לשטוף ביסודיות עם PBS ולזרוע ישירות על צלחות תרבית רקמות חדשות באמצעות מדיה נטולת סרום. שטיפת התאים ישירות בכלי תרבית רקמה להסרת הסרום לא תניב סנכרון G0 יעיל. שנית, כאשר משחררים תאים לשלב G1, יש לבצע שוב טריפסינזציה ולזרוע אותם על צלחות תרבית רקמה טריות. באופן דומה, רק שינוי המדיום והוספת מדיום גידול המכיל סרום לתאים לא יביא לכניסה סינכרונית של G1. בנוסף, עבור כניסה נכונה של G1, צפיפות הזריעה של התאים על משקפי הכיסוי המצופים חייבת להיות ברמות מפגש מסוימות. בעוד סנכרון תאים מושלם הוא בדרך כלל בלתי ניתן להשגה, פרוטוקול סנכרון זה המתואר כאן נותן ~ 97% אוכלוסיית G1 טהורה. צפיפות הזריעה המומלצת עבור RPE1 על כיסוי בקוטר 12 מ"מ היא ~ 4 × 104 כדי לרכוש שדה ראייה הומוגני להדמיה, עם מפגש של כ -70%. צפיפות זריעה גבוהה יותר גורמת לתאים להתנתק ו"להתקלף" לאחר מיצוי CSK ותגרום לאות רקע גבוה יותר במהלך רכישת התמונה.
כדי להפחית כל אות רקע ולהשיג יחס אות לרעש חיובי, שטיפה יסודית לאחר דגירה ראשונית ומשנית של נוגדנים היא חיונית. מכיוון שיש ליישם שלבי שטיפה רבים, חיוני גם למנוע מהבאר להתייבש במהלך כל שלב כביסה. אנו ממזערים חפץ זה על ידי יישום מינימום של 0.05% Triton X-100 בכל שלבי הכביסה והדגירה. לאחר שהבארות התייבשו, התאים הציגו יחס אות לרעש שונה; זה מוביל לתבנית דמוית פסיפס מתחת למיקרוסקופ ועלול להפריע להערכה. רכישת תמונה Z-stack בשילוב עם deconvolution יכולה לסייע בלכידת מוקדים במישורי מוקד שונים כדי לשפר את הניתוח.
שיטות קונבנציונליות מסתמכות על זיהוי של BrdU משולב בתנאים שאינם דנטורציה. שיטות אלה, עם זאת, תלויות בטיפול מקדים של תאים עם מינונים גבוהים של BrdU לפחות 1-2 ימים (או זמן שווה ערך מחזור התא המלא בקו התא בשימוש) כדי להבטיח שילוב גנומי אחיד. באופן בלתי נמנע, שילוב נרחב של BrdU עלול לגרום להפרעה במחזור התא36. כדי להתמודד עם מגבלות אלה, שיטה זו משתמשת ב- RPA2 אנדוגני כדי לזהות מוקדי ssDNA. גישה זו אינה דורשת שילוב BrdU מונחה שכפול, ניתן להשתמש בה גם בתאים פוסט-מיטוטיים. מכיוון שאין צורך בשילוב נרחב של BrdU, הדבר חוסך זמן ומפחית את מורכבות הניסוי. על ידי שימוש בצביעת RPA2 כדי להמחיש ssDNA, אנו יכולים להשתמש ב-2′-deoxy-5-ethynyluridine (EdU) ובכימיית קליקים כדי לסמן שכפול DNA תוך הימנעות מתגובתיות צולבת אפשרית של נוגדני BrdU כנגד EdU 27,37,38. יש לנקוט בזהירות מיוחדת כדי להסוות כראוי את ה- EdU המשולב במהלך תגובת הלחיצה, כך שהנוגדנים BrdU לא יגיבו בהצלבה עם EdU27,39.
לבסוף, יתרון חשוב בשימוש ב-RPA2 במקום ב-BrdU הוא פשוט יחס אות לרעש מעולה בהשוואה לצביעת BrdU מחוץ לשלב S. מצאנו שצביעת BrdU ללא דנטורציה והיכולת שלה לדמיין ssDNA מוגבלת לשלב S אפילו בתאים משוכפלים (איור 2). נוגדן BrdU נקשר רק ל- BrdU החשוף מספיק במתיחות ssDNA. קשירה של חלבוני תיקון, כולל RPA2, למתיחות ssDNA עלולה לדכא או לעכב את החשיפה המספקת של BrdU ב- ssDNA. מצאנו גם כי מיצוי מראש של CSK נחוץ להדמיית ssDNA באמצעות נוגדן BrdU. הדבר אפשרי מכיוון שעקבות ssDNA אינן נגישות לנוגדן מבלי להסיר מהן רכיבי חלבון הקשורים קלות.
עם זאת, ישנן כמה מגבלות הקשורות לפרוטוקול זה. מגבלה של שימוש ב- RPA2 לזיהוי ssDNA היא הצורך לייעל את שלב החילוץ מראש של CSK. עודף RPA2 לא קשור חייב להישטף מהדנ"א לפני קיבוע התאים. מצד אחד, תת-מיצוי מוביל לרקע גבוה בגלל מקטע החלבון RPA2 שאינו קשור ל-ssDNA. מצד שני, מיצוי יתר יוביל לאובדן אות. עבור זיהוי BrdU, זה אינו משתנה מכיוון ש- BrdU משולב ביציבות ב- DNA ולא ניתן לשטוף אותו על ידי מיצוי מראש. לכן, יש לשקול בזהירות את זמן החילוץ מראש של CSK, את כמות Triton X-100 בחיץ, את הנפח ואת הטמפרטורה שבה מתבצע החילוץ מראש. חילוץ מוקדם של CSK גם מגביל את השימוש בגודל הגרעין כדי להבחין בין תאי G0/G1 לתאי S/G2.
בנוסף, איננו יכולים לשלול את האפשרות שחלק מהאות שמגיע מ-RPA2 מקורו בכך שהוא קשור לאינטראקטורים אחרים של חלבונים קושרי כרומטין. יש גם לקחת בחשבון את ספציפיות המין של נוגדן RPA2. הנוגדן המשמש בפרוטוקול זה יכול לזהות RPA2 של בני אדם, עכברים, חולדות, אוגרים וקופים. מגבלה נוספת של גישה זו היא שלא כל קווי התאים יכולים להיות מורעבים בסרום לסנכרון G0. רוב קווי התאים הסרטניים יכולים לעקוף את נקודות הביקורת של מחזור התא ולהתרבות אפילו במדיה נטולת נסיוב. למרות שרעב בסרום מועיל, מכיוון שהוא אינו גורם נזק לדנ"א, יש לעקוב בקפידה אחר יעילות סנכרון התאים שלהם כדי לוודא שמושגת העשרה נכונה של שלב מחזור התא. עבור תאים שאינם מגיבים לחסך בסרום, יש לשקול שיטות אחרות לסנכרון תאים (למשל, ניעור מיטוטי, עיכוב CDK1 למעצר G2, או טכניקות לא פולשניות כגון אילוטריציה צנטריפוגלית). שיטה אפשרית נוספת היא שימוש בהדמיה בתוכן גבוה כדי למדוד EdU ותכולת DNA גרעיני עבור פרופיל מחזור התא של תאים אסינכרוניים31. יש לשקול את ההשלכות של שימוש בשיטות סנכרון חלופיות כדי למנוע הפרעה לניתוח במורד הזרם. לדוגמה, השימוש בבלוק תימידין כפול או אפידיקולין, המשמש לעתים קרובות בספרות, יגרום ללחץ שכפול ולנזק לדנ"א40.
חקירת מנגנוני תיקון הדנ"א ממשיכה להוות מוקד דיון מרכזי בתחומי הסרטן והביולוגיה של התא. הפרוטוקול המוצג כאן מציע גישה רבת ערך להכנת תאים, המאפשרת הדמיה וניתוח כמותי של ssDNA עם חשיפה לחומרים מזיקים לדנ"א. יש לציין כי פרוטוקול זה מדגיש את השימוש בחלבון קושר ssDNA, RPA2, ומדגים את הספציפיות הגבוהה שלו כדי לדמיין כמויות קטנות של מוקדי ssDNA תוך הימנעות מתגובתיות צולבת לא רצויה בכל שלבי מחזור התא. השימוש ב-RPA2 מעניק יתרונות רבים, במיוחד עבור חוקרים השואפים לנתח תאים בשלב G1 של מחזור התא. פרוטוקול זה לוקח בחשבון מספר מגבלות ומטפל בחששות הקשורים להפרעות אותות, רעשי רקע לא רצויים ותגובתיות צולבת בעת שימוש בצביעת RPA2 או BrdU לזיהוי ssDNA.
Disclosures
למחברים אין אינטרסים מתחרים להצהיר.
Acknowledgements
המחברים מודים למישל פגאנו על תמיכתו ותובנותיו המועילות, לאשלי צ'וי ושרון קייסארי על קריאה ביקורתית של כתב היד, ולג'פרי אסטרדה ווילמה דיאז על תמיכתם המתמשכת. עבודה זו נתמכה על ידי תוספת גיוון למענק המכונים הלאומיים לבריאות GM136250.
Materials
| Name | Company | Catalog Number | Comments |
| Alpha-tubulin antibody | Sigma-Aldrich | T6074 | primary antibody (1:5,000) |
| Axio Observer Inverted Microscope | Zeiss | na | microscope |
| Bis-Tris Plus Mini Protein Gels, 4-12% | Invitrogen | NW04127BOX | Western Blot |
| Bovine Serum Albumin | Jackson ImmunoResearch | 001-000-162 | blocking |
| BrdU (5-Bromo-2'-deoxyuridine) | Sigma-Aldrich | B5002-100MG | nucleotide analogue |
| BrdU antibody BU1/75 | Abcam | ab6326 | primary antibody (1:500) |
| CellAdhere Dilution Buffer | Stemcell Technologies | 07183 | coating reagent |
| Click-iT Plus EdU Flow Cytometry Assay Kits | Invitrogen | C10632 | flow cytomery |
| Click-iT Plus EdU Cell Proliferation Kit for Imaging, Alexa Fluor 647 dye | Thermo Fisher Scientific | C10640 | click-reaction kit |
| cOmplete ULTRA Protease inhibitor tablets | Sigma-Aldrich | 5892791001 | reagent |
| Countess 3 Automated cell counter | Thermo Scientific | AMQAX2000 | cell counter |
| Coverslip | neuVitro | GG12PRE | tissue culture |
| Cyclin A2 antibody | Santa Cruz Biotechnology | sc-271682 | primary antibody (1:1,000) for IF and WB |
| Cyclin B1 antibody | Santa Cruz Biotechnology | sc-245 | primary antibody (1:5,000) |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D2650-100ML | vehicle control |
| DMEM, high glucose, with HEPES | Gibco | 12430051 | cell culture medium for RPE cells |
| DPBS, no calcium, no magnesium | Gibco | 14190144 | the PBS used throughout the protocol |
| D-Sucrose | Thermo Fisher Scientific | bp220-1 | reagent |
| Eclipse Ti2 Series Epifluorescent Microscope | Nikon | na | microscope |
| EdU (5-Ethynyl-2'-deoxyuridine) | Thermo Fisher Scientific | C10637 | nucleotide analog |
| Falcon 24-well plate | Corning | 351147 | tissue culture |
| Falcon Cell Culture Dishes 100 mm | Corning | 353003 | tissue culture |
| Fetal Bovine Serum, heat inactivated | Gibco | 16140071 | media supplement |
| Fiji (ImageJ) | NIH | version 1.54f | software and algorithms |
| FxCycle PI/RNase Staining Solution | Invitrogen | F10797 | PI staining |
| Goat anti-mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor Plus 555 | Thermo Fisher Scientific | A21422 | secondary antibody (1:1,000) |
| Goat anti-rat IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor Plus 488 | Thermo Fisher Scientific | A48262 | secondary antibody (1:1,000) |
| Histone H3 antibody | Abcam | ab1791 | primary antibody (1:10,000) |
| hTERT RPE1 | ATCC | CRL-3216 | cell line |
| Hydrochloric acid | Sigma-Aldrich | H1758-100ML | reagent |
| Hydrogen peroxide 30% soultion | Sigma-Aldrich | H1009-100ML | reagent |
| Hydroxyurea,98% powder | Sigma-Aldrich | H8627-5G | reagent |
| Invitrogen Ultra Pure 0.5 M EDTA pH 8.0 | Thermo Fisher Scientific | 15-575-020 | reagent |
| Lipfectamine RNAiMAX Transfection Reagent | Invitrogen | 13778150 | transfection reagent |
| Magnesium chloride solution 1 M | Sigma-Aldrich | M1028-100ML | reagent |
| MycoFluor | Thermo Fisher | M7006 | Mycoplasma Detection Kit |
| Neocarzinostatin from Streptomyces carzinostaticus | Sigma-Aldrich | N9162-100UG | reagent |
| NuPage MES SDS Running Buffer (20x) | Invitrogen | NP0002 | Western Blot |
| onTARGETplus Human RPA2 siRNA | Dharmacon | L-017058-01-0005 | siRNA |
| p27 antibody | BD Biosciences | 610241 | primary antibody (1:1,000) |
| Paraformaldehyde aqueous solution (32%) | Electron Microscopy Sciences | 50-980-494 | fixative |
| PARP1 antibody | Cell Signaling Technology | 9542S | primary antibody (1:1,000) |
| PCNA antibody | Cell Signaling Technology | 13110S | primary antibody (1:2,000) |
| Penicillin-Streptomycin | Gibco | 15140163 | media supplement |
| pH3 antibody | Cell Signaling Technology | 3377S | primary antibody (1:2,000) |
| PhosSTOP phosphatase inhibitor tablets | Sigma-Aldrich | 4906837001 | reagent |
| PIPES Buffer 0.5 M solution, pH 7.0 | Bioworld | 41620034-1 | reagent |
| Precision Plus Protein Kaleidoscope Prestained Protein Standards | Bio-Rad | 1610395 | Western Blot |
| Prism | GraphPad | version 10 | statistical analysis and graph |
| ProLong Diamond Antifade Mountant | Thermo Scientific | P36961 | mounting media |
| Reduced serum media (Opti-MEM) | Gibco | 31985070 | used for transfection |
| Rpa32/rpa2 antibody (mouse) | EMD Millipore | NA19L | primary antibody (1:1,000) for WB |
| Rpa32/rpa2 antibody (rat) | Cell Signaling Technology | 2208S | primary antibody (1:1,000) for IF |
| Sodium Chloride solution (5 M) | Sigma-Aldrich | S5150 | reagent |
| Sodium Pyruvate (100 mM) | Gibco | 11360070 | media supplement |
| Sodium tetraborate decahydrate | Sigma-Aldrich | B3535-500G | reagent |
| Thermo Scientific Pierce DAPI Nuclear Counterstain | Thermo Scientific | 62248 | nucleic acid stain |
| Thymidine,powder | Sigma-Aldrich | T1985-1G | reagent |
| Triton X-100 aqueous solution (10%) | Sigma-Aldrich | 11332481001 | detergent |
| Trypsin-EDTA (0.5%), no phenol red | Gibco | 1540054 | cell dissociation agent |
| Vitronectin XF | Stemcell Technologies | 07180 | coating reagent |
| ZE5 Cell Analyzer | Bio-Rad | na | flow cytomery |
References
- Hakem, R. DNA-damage repair; the good, the bad, and ugly. EMBO J. 27 (4), 589-605 (2008).
- Gutierrez, R., O'Connor, T. R. DNA direct reversal repair and alkylating agent drug resistance. Cancer Drug Resist. 4 (2), 414-423 (2021).
- Krokan, H. E., Bjoras, M. Base excision repair. Cold Spring Harb Perspect Biol. 5 (4), a012583 (2013).
- Marteijn, J. A., Lans, H., Vermeulen, W., Hoeijmakers, J. H. Understanding nucleotide excision repair and its roles in cancer and ageing. Nat Rev Mol Cell Biol. 15 (7), 465-481 (2014).
- Li, G. M. Mechanisms and functions of DNA mismatch repair. Cell Res. 18 (1), 85-98 (2008).
- Hustedt, N., Durocher, D. The control of DNA repair by the cell cycle. Nat Cell Biol. 19 (1), 1-9 (2016).
- Yang, W., Gao, Y. Translesion and repair DNA polymerases: diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Bhat, D. S., et al. Therapeutic disruption of RAD52-ssDNA complexation via novel drug-like inhibitors. NAR Cancer. 5 (2), zcad018 (2023).
- Gupta, P., Saha, B., Chattopadhyay, S., Patro, B. S. Pharmacological targeting of differential DNA repair, radio-sensitizes WRN-deficient cancer cells in vitro and in vivo. Biochem Pharmacol. 186, 114450 (2021).
- Pena-Diaz, J., et al. Noncanonical mismatch repair as a source of genomic instability in human cells. Mol Cell. 47 (5), 669-680 (2012).
- Schroering, A. G., Edelbrock, M. A., Richards, T. J., Williams, K. J. The cell cycle and DNA mismatch repair. Exp Cell Res. 313 (2), 292-304 (2007).
- Scully, R., Panday, A., Elango, R., Willis, N. A. DNA double-strand break repair-pathway choice in somatic mammalian cells. Nat Rev Mol Cell Biol. 20 (11), 698-714 (2019).
- Escribano-Diaz, C., et al. A cell cycle-dependent regulatory circuit composed of 53BP1-RIF1 and BRCA1-CtIP controls DNA repair pathway choice. Mol Cell. 49 (5), 872-883 (2013).
- Genschel, J., Modrich, P. Mechanism of 5'-directed excision in human mismatch repair. Mol Cell. 12 (5), 1077-1086 (2003).
- Hu, J., et al. Nucleotide excision repair in human cells: fate of the excised oligonucleotide carrying DNA damage in vivo. J Biol Chem. 288 (29), 20918-20926 (2013).
- Huertas, P., Jackson, S. P. Human CtIP mediates cell cycle control of DNA end resection and double strand break repair. J Biol Chem. 284 (14), 9558-9565 (2009).
- Keijzers, G., et al. Human exonuclease 1 (EXO1) regulatory functions in DNA replication with putative roles in cancer. Int J Mol Sci. 20 (1), 74 (2018).
- Symington, L. S. End resection at double-strand breaks: mechanism and regulation. Cold Spring Harb Perspect Biol. 6 (8), a016436 (2014).
- Liu, Y., et al. DNA polymerase beta and flap endonuclease 1 enzymatic specificities sustain DNA synthesis for long patch base excision repair. J Biol Chem. 280 (5), 3665-3674 (2005).
- Wold, M. S., Kelly, T. Purification and characterization of replication protein A, a cellular protein required for in vitro replication of simian virus 40 DNA. Proc Natl Acad Sci U S A. 85 (8), 2523-2527 (1988).
- Wienholz, F., Vermeulen, W., Marteijn, J. A. Amplification of unscheduled DNA synthesis signal enables fluorescence-based single cell quantification of transcription-coupled nucleotide excision repair. Nucleic Acids Res. 45 (9), e68 (2017).
- Wold, M. S. Replication protein A: a heterotrimeric, single-stranded DNA-binding protein required for eukaryotic DNA metabolism. Annu Rev Biochem. 66, 61-92 (1997).
- Chen, R., Wold, M. S. Replication protein A: single-stranded DNA's first responder: dynamic DNA-interactions allow replication protein A to direct single-strand DNA intermediates into different pathways for synthesis or repair. Bioessays. 36 (12), 1156-1161 (2014).
- Kang, Y., et al. Alteration of replication protein A binding mode on single-stranded DNA by NSMF potentiates RPA phosphorylation by ATR kinase. Nucleic Acids Res. 51 (15), 7936-7950 (2023).
- Kilgas, S., Kiltie, A. E., Ramadan, K. Immunofluorescence microscopy-based detection of ssDNA foci by BrdU in mammalian cells. STAR Protoc. 2 (4), 100978 (2021).
- Madabhushi, R., Pan, L., Tsai, L. H. DNA damage and its links to neurodegeneration. Neuron. 83 (2), 266-282 (2014).
- Liboska, R., Ligasova, A., Strunin, D., Rosenberg, I., Koberna, K. Most anti-BrdU antibodies react with 2'-deoxy-5-ethynyluridine -- the method for the effective suppression of this cross-reactivity. PLoS One. 7 (12), e51679 (2012).
- Biehs, R., et al. DNA double-strand break resection occurs during non-homologous end joining in G1 but is distinct from resection during homologous recombination. Mol Cell. 65 (4), 671-684.e5 (2017).
- Cruz-Garcia, A., Lopez-Saavedra, A., Huertas, P. BRCA1 accelerates CtIP-mediated DNA-end resection. Cell Rep. 9 (2), 451-459 (2014).
- Ercilla, A., et al. Physiological tolerance to ssDNA enables strand uncoupling during DNA replication. Cell Rep. 30 (7), 2416-2429.e7 (2020).
- Lezaja, A., et al. RPA shields inherited DNA lesions for post-mitotic DNA synthesis. Nat Commun. 12 (1), 3827 (2021).
- Mukherjee, B., Tomimatsu, N., Burma, S. Immunofluorescence-based methods to monitor DNA end resection. Methods Mol Biol. 1292, 67-75 (2015).
- Ochs, F., et al. 53BP1 fosters fidelity of homology-directed DNA repair. Nat Struct Mol Biol. 23 (8), 714-721 (2016).
- Raderschall, E., Golub, E. I., Haaf, T. Nuclear foci of mammalian recombination proteins are located at single-stranded DNA regions formed after DNA damage. Proc Natl Acad Sci U S A. 96 (5), 1921-1926 (1999).
- Forment, J. V., Walker, R. V., Jackson, S. P. A high-throughput, flow cytometry-based method to quantify DNA-end resection in mammalian cells. Cytometry A. 81 (10), 922-928 (2012).
- Mistrik, M., et al. Cells and stripes: A novel quantitative photo-manipulation technique. Sci Rep. 6, 19567 (2016).
- Aten, J. A., Bakker, P. J., Stap, J., Boschman, G. A., Veenhof, C. H. DNA double labelling with IdUrd and CldUrd for spatial and temporal analysis of cell proliferation and DNA replication. Histochem J. 24 (5), 251-259 (1992).
- Podgorny, O., Peunova, N., Park, J. H., Enikolopov, G. Triple S-phase labeling of dividing stem cells. Stem Cell Reports. 10 (2), 615-626 (2018).
- Cappella, P., Gasparri, F., Pulici, M., Moll, J. Cell proliferation method: click chemistry based on BrdU coupling for multiplex antibody staining. Curr Protoc Cytom. Chapter 7, (2008).
- Ligasova, A., Koberna, K. Strengths and weaknesses of cell synchronization protocols based on inhibition of DNA synthesis. Int J Mol Sci. 22 (19), 10759 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved