Method Article
在细胞周期的 G1 期可视化单链 DNA 病灶
摘要
以下方案介绍了利用细胞周期同步和RPA2免疫荧光染色检测细胞周期G1期的单链DNA病灶。
摘要
DNA具有专用的细胞修复途径,能够应对可能来自内源性和/或外源性来源的病变。DNA修复需要众多蛋白质之间的协作,负责涵盖广泛的任务,从识别DNA损伤的存在并发出信号到物理修复它。在此过程中,通常会产生单链 DNA (ssDNA) 的轨迹,最终由 DNA 聚合酶填充。这些ssDNA轨迹的性质(就长度和数量而言),以及为填补这些空白而募集的聚合酶,是修复途径特异性的。这些ssDNA轨迹的可视化可以帮助我们理解DNA修复机制的复杂动力学。
该协议为制备G1同步细胞以测量遗传毒性应激时ssDNA病灶的形成提供了详细的方法。使用易于利用的免疫荧光方法,我们通过染色 RPA2(异源三聚体复制蛋白 A 复合物 (RPA) 的组成部分)来可视化 ssDNA。RPA2 结合并稳定遗传毒性应激或复制时产生的 ssDNA 中间体,以控制 DNA 修复和 DNA 损伤检查点激活。5-乙炔基-2'-脱氧尿苷 (EdU) 染色用于可视化 DNA 复制以排除任何 S 期细胞。该方案为传统的非变性5-溴-2'-脱氧尿苷(BrdU)测定提供了一种替代方法,并且更适合检测S期外的ssDNA病灶。
引言
为了维持生命,细胞不断调查和修复DNA,以保持其基因组完整性。由于DNA应激源的内源性(例如氧化、烷基化、脱氨化、复制错误)和外源性(例如紫外线、电离照射)来源,细胞可能会积累各种类型的DNA损伤。未能修复这些病变会导致细胞凋亡、细胞周期停滞或衰老,并可能导致疾病1。DNA损伤可以通过以下任何一种主要的DNA修复途径来解决:DR(直接逆转修复),主要修复烷基化碱基2;BER(碱基切除修复),针对非大块 DNA 碱基错误和单链 DNA 断裂 (SSB)3;NER(核苷酸切除修复)矫正体积庞大、螺旋扭曲的 DNA 损伤4;MMR(错配修复)主要针对 DNA 错配、插入/缺失环 (IDL) 和某些碱基损伤5;NHEJ(非同源末端连接)和 HRR(同源重组修复)在双链 DNA 断裂 (DSB) 处均具有活性6;TLS(跨病变合成),这是一种DNA病变旁路机制7。尽管这些途径具有不同的底物特异性,但它们之间存在某些重叠,以确保冗余以实现有效修复。了解不同 DNA 修复途径在不同细胞周期阶段的作用至关重要,因为这些 DNA 修复因子可以作为治疗癌症、衰老和神经系统疾病的治疗方法的重要靶标 8,9。
单链 DNA (ssDNA) 是由于内源性和外源性 DNA 损伤剂产生的 DNA 损伤的修复而在整个细胞周期中产生的。在遗传毒性应激下,ssDNA 在 HRR 和 MMR 活性最高的 S 期和 G2 期大量生成,当遇到 DNA 损伤时复制机制停滞或崩溃时 6,10,11。其他 DNA 修复途径(例如,NHEJ/微同源介导的末端连接 (MMEJ)/单链退火 [SSA])也在 DSB 修复过程中产生 ssDNA12。这些 ssDNA 轨迹通常来自 DNA 切除,由 HR 和 MMR 期间的 EXO1、DNA2 和 CtIP 等核酸外切酶、NER 期间的 XPF 和 XPG 等核酸内切酶或 BER4、13、14、15、16、17、18、19 期间的 POLB 和 FEN1 的联合作用进行.由于复制机制的工作,当DNA解旋酶在PCNA结合的复制聚合酶20前面展开DNA时,也会产生ssDNA轨迹。相反,在 G1 期,HRR 和 DNA 复制的缺乏以及 MMR 的有限活性降低了生成的 ssDNA 轨迹的范围,因此检测更具挑战性 10,11,21。
细胞 ssDNA 轨道是高度敏感的结构,必须加以保护以避免 DSB 的形成。这是通过用 RPA 涂覆 ssDNA 轨道来实现的。RPA 是一种丰富的异源三聚体蛋白复合物,由多个亚基(RPA1、RPA2 和 RPA3,也称为 RPA70、RPA32 和 RPA14)组成,它们在整个细胞周期中普遍表达22。每个 RPA 亚基都包含一个 DNA 结合域 (DBD),能够与 4-6 个核苷酸相互作用,组合后的亚基形成稳定的三聚体化核心。RPA 总共与大约 20-30 个核苷酸结合,具有亚纳摩尔亲和力23,24。
常规方法使用免疫荧光 (IF) 显微镜,通过使用 BrdU 抗体标记掺入基因组 DNA 中的 5-溴-2'-脱氧尿苷 (BrdU) 来可视化 ssDNA 病灶25。这种方法依赖于这样一个事实,即 BrdU 抗体只能检测暴露的 ssDNA25 中的 BrdU。尽管这种方法很简单,但它也显示出某些局限性。例如,在实验开始前对细胞进行预处理以掺入BrdU,这很耗时,并且会干扰下游效应器。因此,基于BrdU的ssDNA检测仅限于复制细胞,不能用于静止细胞。这不包括该方法在非复制细胞中研究DNA修复的应用,尽管它在癌症和神经退行性变等多种疾病中很重要5,26。此外,由于 BrdU 和 EdU 的结构非常相似,大多数 BrdU 抗体对 EdU 表现出交叉反应性,这在进行双标记实验时必须考虑27。RPA染色以前主要用于显示S期细胞中的ssDNA病灶;然而,一些论文也成功地在S阶段28,29,30,31,32,33,34,35之外使用它。以下方案有效地利用了RPA的特性,允许在细胞周期的G1期(尽管它可用于所有细胞周期阶段)中DNA损伤后的ssDNA病灶的可视化。
研究方案
1. 维持 hTERT 永生化视网膜色素上皮细胞 (RPE1)
- 将 RPE1 细胞系维持在补充有 10% 热灭活胎牛血清 (Hi-FBS) 和 100 μg/mL 青霉素-链霉素(从现在开始称为培养基)的 Dulbecco 改良 Eagle 培养基 (DMEM) 中,在 37 °C 下含有 5% CO2 的加湿培养箱中。 对于常规培养,在 15 cm 组织培养处理的培养皿中培养 RPE1 细胞,并在达到 80-90% 汇合度时分裂(~16-18 ×每 15 cm 培养皿10 6 个细胞)。
- 分裂时,取出培养基并用 10 mL 1x 磷酸盐缓冲盐水 (PBS) 冲洗细胞。
- 加入 3 mL 0.05% 胰蛋白酶-EDTA 以覆盖培养皿的整个表面。用胰蛋白酶将细胞保持在37°C,直至它们分离。
- 胰蛋白酶消化后,用培养基重悬细胞,并在室温(RT,22-25°C)下以150× g 旋转5分钟。除去上清液,将细胞轻轻重悬于10mL培养基中。
- 将 1.6-1.8 ×10 6 个细胞接种到新的 15 cm 培养皿(~1 mL 细胞悬液)中。
注意:所有组织培养工作均应在 BSL-2 安全水平下进行。胰蛋白酶消化的孵育时间取决于细胞汇合度。通常,对于 90% 汇合板,该过程需要 2-3 分钟才能完成。应使用市售试剂盒定期筛查细胞 是否存在支原体 污染(参见 材料表中的示例)。
2. 目的基因 (GOI) 的 siRNA 敲低
- 在转染前一天,将 1.0 ×10 6 个 RPE1 细胞接种到 10 cm 组织培养处理的培养板中,加入 10 mL 培养基。
- 在转染当天,复合siRNA。对于 10 cm 板,在 500 μL 低血清转染培养基中使用终浓度为 20 nM siRPA2 和 12 μL 脂质转染试剂。轻弹试管轻轻混合所有组分,并在室温(22-25°C)下孵育5分钟。
- 将复合的siRNA混合物滴加到细胞中,并将细胞与siRNA一起孵育48小时。
3. RPE1细胞同步进入G0期
- 如第 1 节所述,从步骤 2.3 开始胰蛋白酶消化 RPE1 细胞(~2 × 106 个细胞)。
- 将细胞悬液转移到 15 mL 离心管中,并以 150 × g、室温 (22-25 °C) 离心 5 分钟。
- 除去上清液并将细胞重悬于 12 mL PBS 中。在室温(22-25°C)下以150× g 离心细胞5分钟。重复除去上清液并离心两次。
- 将细胞重悬于补充有 100 μg/mL 青霉素-链霉素、 1 mM 丙酮酸钠、15 mM HEPES 的 10 mL 无血清 DMEM 中,并将它们铺板到 10 cm 组织培养皿上。
注意:如果细胞倾向于结块,请将它们重悬于仅 1 mL 无血清的 DMEM 中,并使用 P1000 吸头上下移液 5 次以去除团块,然后将悬浮液稀释至最终体积为 10 mL。 - 血清饥饿 24 小时后,通过将复合的 siRNA 添加到血清饥饿细胞中,使用与第 2 节中描述的相同的程序引入第二轮沉默。
- 在进行G1释放之前,将RPE1细胞保持在无血清DMEM中72小时。
4. 盖玻片涂层并将细胞释放到 G1 期
- 用70%乙醇对镊子进行灭菌,并将单个玻璃盖玻片(直径12mm,#1.5厚度[0.17mm])放入24孔板的孔中。
- 用PBS稀释玻连蛋白包被基质,以获得10μg/mL的终浓度。将 500 μL 玻连蛋白溶液加入每个含有盖玻片的孔中,并在室温下孵育 1 小时。
- 去除包衣溶液并用 1 mL PBS 洗涤盖玻片。
- 在PBS在37°C下洗涤1分钟后,使用1mL0.05%胰蛋白酶从10cm组织培养的处理板中分离血清饥饿的RPE1细胞。
注意:血清饥饿后细胞分离得更快。用PBS洗涤细胞时要小心,并使用较短的胰蛋白酶消化时间。 - 为了灭活胰蛋白酶,将 RPE1 细胞重悬于总共 6 mL 的培养基中。通过在室温(22-25°C)下使用150× g 旋转细胞5分钟来除去灭活的胰蛋白酶。
- 将细胞重悬于 1 mL 培养基中并测量细胞数量。
- 将 4 ×10 4 个 RPE1 细胞接种到包被的盖玻片上,在总共 500 μL 的培养基中。
注意:在进行下游步骤之前,请确保细胞活力高于 90%。在细胞计数步骤中,可以通过台盼蓝染色快速评估细胞活力。 - 将细胞接种到培养基中 6 小时后,G0 释放的细胞将处于早期 G1 期。在细胞开始进入S期之前,在G1中进行6-12小时的实验。
- 在引入DNA损伤之前,用10μM5-乙炔基-2'-脱氧尿苷(EdU)在37°C下脉冲细胞30分钟,在培养基中稀释。
- 除去含有EdU的培养基,并在37°C下用10μM胸苷追逐细胞10分钟,以防止在DNA损伤诱导过程中剩余的EdU掺入。
- 用胸苷除去培养基,并用250μM H2O2处理细胞1小时,在培养基中稀释。
5. ssDNA的免疫荧光染色
- 用 1 mL RT (22-25 °C) PBS 洗涤细胞一次,以除去培养基和血清成分。
注意: 洗涤细胞时要轻柔,以免脱落和干燥。不要同时处理多个孔。 - 提取前:将洗涤后的细胞在室温(22-25°C)下在1mL CSK提取缓冲液(表1)中孵育5分钟。
注意:CSK预提取去除所有非染色质结合蛋白,包括可溶性RPA2。
注意: Triton X-100 如果吞咽是有害的,可能会引起皮肤刺激和眼睛损伤。 - 从细胞中除去CSK缓冲液,并通过在室温(22-25°C)下加入0.5mL含有0.05%Triton X-100的0.5mL含有0.05%Triton X-100的3.6%多聚甲醛溶液(在PBS中)直接固定它们。
注意:从32%的PFA原料中制备3.6%的PFA是很重要的。多聚甲醛可能会引起严重的眼睛损伤、皮肤刺激和呼吸道刺激。 - 用含有 0.05% Triton X-100 的 1 mL PBS 洗涤细胞一次以除去 PFA。
- 在室温(22-25°C)下,使用含有0.5%Triton X-100的1mL PBS进一步透化细胞15分钟。
- EdU click-IT 反应可视化复制细胞(S 期)
- 除去透化溶液,用1mL封闭缓冲液洗涤细胞2次(表1)。
注意:牛血清白蛋白 (BSA) 可能会引起呼吸道刺激。 - 加入1mL封闭缓冲液(表1),并在室温(22-25°C)下轻轻摇晃含有盖玻片的板10分钟。
- 取出封闭缓冲液,加入500μL含有吡啶酰叠氮化物647的点击反应混合物(表1)。使用轻轻摇动将盖玻片在室温(22-25°C)下孵育30分钟,并在黑暗中进行下游孵育。
注意:使用 BrdU 抗体时,按照制造商的建议,使用双倍的量 (1 mL) 和时间(60 分钟)进行点击反应,以确保反应饱和并标记掺入的 EdU。这限制了 BrdU 抗体的交叉反应性27。
- 除去透化溶液,用1mL封闭缓冲液洗涤细胞2次(表1)。
- 除去点击反应混合物,并在室温(22-25°C)下用含有0.05%Triton X-100的PBS洗涤细胞2次10分钟(图1 和 图2)。
- 加入 1 mL 封闭缓冲液,并在室温 (22-25 °C) 下孵育 30 分钟。或者,将细胞保持在4°C的封闭缓冲液中过夜。
- 在室温(22-25°C)下在250-500μL封闭缓冲液中轻轻摇动,施加一抗(抗RPA2大鼠,1:1,000稀释度)2小时。
- 用含有 0.05% Triton X-100 的 PBS 洗涤细胞 2 次,以快速除去大部分抗体溶液。
- 在室温(22-25°C)下用封闭缓冲液继续洗涤细胞3×10分钟。
- 在室温(22-25°C)下将二抗(抗大鼠Alexa-488,1:1,000稀释度)在250-500μL封闭缓冲液中轻轻摇晃2小时。
- 用封闭缓冲液洗涤细胞 2x,以快速除去大部分二抗。在室温(22-25°C)下继续用含有0.05%Triton X-100的PBS洗涤细胞3×10分钟。
- 为了对细胞核进行复染,用含有0.05%Triton X-100和1μg/ mL 4',6-二脒基-2-苯基吲哚(DAPI)的PBS在室温(22-25°C)下洗涤细胞10分钟。在室温(22-25°C)下用PBS洗涤细胞5分钟。
- 使用 10 μL 封固剂/盖玻片将盖玻片安装到显微镜载玻片上。在安装之前将盖玻片浸入蒸馏水中以去除任何盐晶体。第二天对载玻片进行成像,并将其在4°C下储存数周(图3)。
6. 图像采集和定量
- 要捕获图像,请使用任何配备常规滤光片组的可用落射荧光显微镜,以至少 60-63 倍的放大倍率、高数值孔径和油物镜对 DAPI、FITC 和 Cy5 通道进行成像,以可视化核病灶。
注意:最佳DAPI激发为~359 nm;Alexa 488 激发波长为 ~488 nm;而 Alexa 647 激发为 ~647 nm。 - 要进行图像分析,请在 Fiji/ImageJ 中打开图像文件。
- 使用DAPI染色制作核掩膜(图4A-F和补充视频S1)。
- 打开 DAPI 映像。
- 选择 "工艺"|"增强对比度 并将 饱和像素 设置为 0.35。
- 单击 "进程"|"二进制 |转换为蒙版。选择 二进制 |填充孔 洞,然后单击 分析 |分析颗粒。 将大小 设置为 10-Infinity。
- 在 ROI 管理器中,单击显示 全部。
- 在细胞核中寻找 RPA2 病灶(图 4G,H 和补充视频 S1)
- 打开 RPA2 映像。
- 选择 "流程"|"找到 Maxima.将 突出 显示 RPA2 焦点(介于 500 和 750 之间)的值,将其与背景分开。
- 最后,单击 Measure 按钮在 ROI 管理器。
- 通过将 RawinDen 列中的值除以 255 (每个焦点中像素强度的最大值)来计算核 ssDNA 病灶的总数。
- 使用首选的统计软件工具执行统计分析。
注意:从分析中排除所有 EdU 阳性细胞和未正确分割的 DAPI 掩模。
- 使用DAPI染色制作核掩膜(图4A-F和补充视频S1)。
结果
为了克服在 G1 中检测 ssDNA 的局限性,我们使用了 RPA2,它增强了 ssDNA 病灶检测的特异性和强度35。为了实现精确的细胞同步,我们使用了可以有效血清饥饿并同步到 G0 期的 RPE1 细胞。然后可以通过在血清剥夺后添加血清来诱导它们重新进入细胞周期。为了确认同步效率,我们用EdU标记细胞,用碘丙啶标记其DNA含量。我们通过流式细胞术进一步收集了定性和定量结果(补充图S1A)。点图显示,血清饥饿 72 小时后,~98% 的细胞处于 G0 期。加入含血清培养基6小时后,细胞重新进入细胞周期(如图1A中p27水平的增加所示),G1中具有~97%的细胞,而S期只有<1%的细胞,G2期有<2%的细胞(补充图S1A)。将血清添加到细胞中20-28小时后,它们逐渐通过S期,如流式细胞术图所示(补充图S1A)。该细胞同步方案给出 ~97% 纯 G1 群体(血清饥饿 72 小时后血清添加后 6 小时)。为了进一步验证同步效率,我们使用蛋白质印迹法(图1A和补充图S1B)比较血清释放后细胞周期标志物的表达,并同时进行EdU掺入测定以可视化DNA复制。EdU染色还突出了同步效率和G1期DNA复制的缺乏(图1B,C)。
检测哺乳动物细胞中 ssDNA 的常规方法依赖于检测 ssDNA 中的 BrdU。 图2A 表明,在H2O2 和新卡嗪抑素(NCS)处理下,BrdU病灶仅在S期细胞中检测到,而在非S期细胞中未检测到ssDNA病灶。BrdU抗体染色还显示出明显的核仁背景染色,可以在所有细胞核中检测到,与细胞周期阶段或应用的处理无关。使用此处描述的 EdU 点击协议,我们无法检测到共定位的 EdU 和 BrdU 病灶,这在 图 2A 的未经处理的样品中很明显。为了完全排除任何由交叉反应性引起的BrdU信号,我们避免了EdU标记,而是使用细胞周期蛋白A2作为S-G2标记物。然而,细胞周期蛋白 A2 染色不允许 CSK 预提取,在这种情况下,即使在遗传毒性应激之后,我们也没有看到任何 BrdU 病灶(补充图 S2A)。这凸显了CSK预提取对于基于抗BrdU的ssDNA染色是必要的。作为对照,我们在变性条件下测试了BrdU抗体染色。这打开了 DNA 以暴露掺入的 BrdU,这表明 BrdU 被均匀掺入(补充图 S2B)。
相比之下,RPA2染色不仅在S期,而且在其他细胞周期期显示NCS和H2O2依赖性病灶形成(图2B)。作为对照,我们还用胡处理细胞,这仅导致ssDNA在经历复制的细胞中积累。正如预期的那样,我们仅在EdU阳性细胞中检测到使用RPA2抗体进行胡处理时信号增加,这突出了这种方法的特异性。在没有外源性遗传毒性应激的情况下,RPA2 抗体还可以检测复制过程中自然发生的 ssDNA 形成(图 2B)。RPA2 抗体的高灵敏度促使我们尝试在 G1 期使用它,在 G1 期,传统的 BrdU 染色无法检测到遗传毒性应激的任何信号(补充图 S2C)。 图 3A 显示,当使用抗 RPA2 抗体时,即使在 G1 中,也可以检测到 H2O2 处理后 ssDNA 病灶的形成。H2O2 处理后,这些细胞核中的RPA2病灶数量显着增加(图3B)。这些病灶是 RPA2 特异性的,因为 RPA2 的沉默消除了 IF 信号(图 3A、B)。 图3C 和 补充图S1C 显示了这些细胞中RPA2沉默的效率。与传统方法相比,基于RPA2的ssDNA检测具有高度的灵敏度,因此其应用可以扩展到G1期细胞。
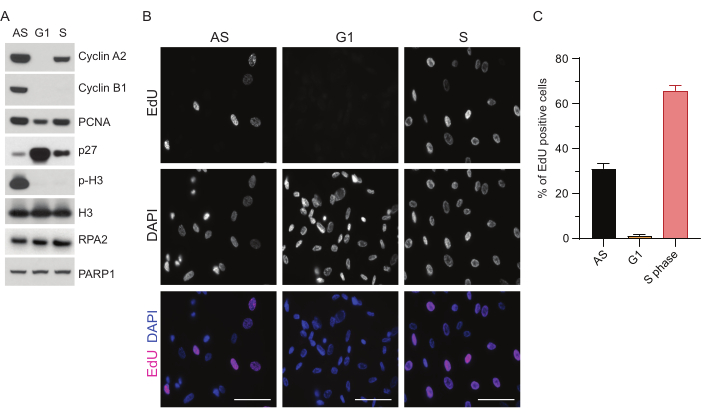
图 1:血清饥饿后 RPE1 细胞的同步效率。 (A) 免疫印迹显示异步、G1 和 S 期同步 RPE1 细胞中指示的蛋白质水平。(B) 代表性图像显示异步、G1 和 S 期同步的 RPE1 细胞,这些细胞在固定前暴露于 10 μM EdU 30 分钟并通过 Click-IT 反应可视化。DAPI用于对核DNA进行复染。比例尺 = 50 μm。 (C) 图表显示了 EdU 阳性细胞占 DAPI 评估的总细胞群的百分比。误差线表示平均值的标准误差,分析的原子核数如下:AS n = 219,G1 n = 630,S n = 437。缩写:RPE1 = hTERT永生化的视网膜色素上皮细胞;AS = 异步;EdU = 5-乙炔基-2'-脱氧尿苷;DAPI = 4',6-二脒基-2-苯基吲哚。 请点击这里查看此图的较大版本.
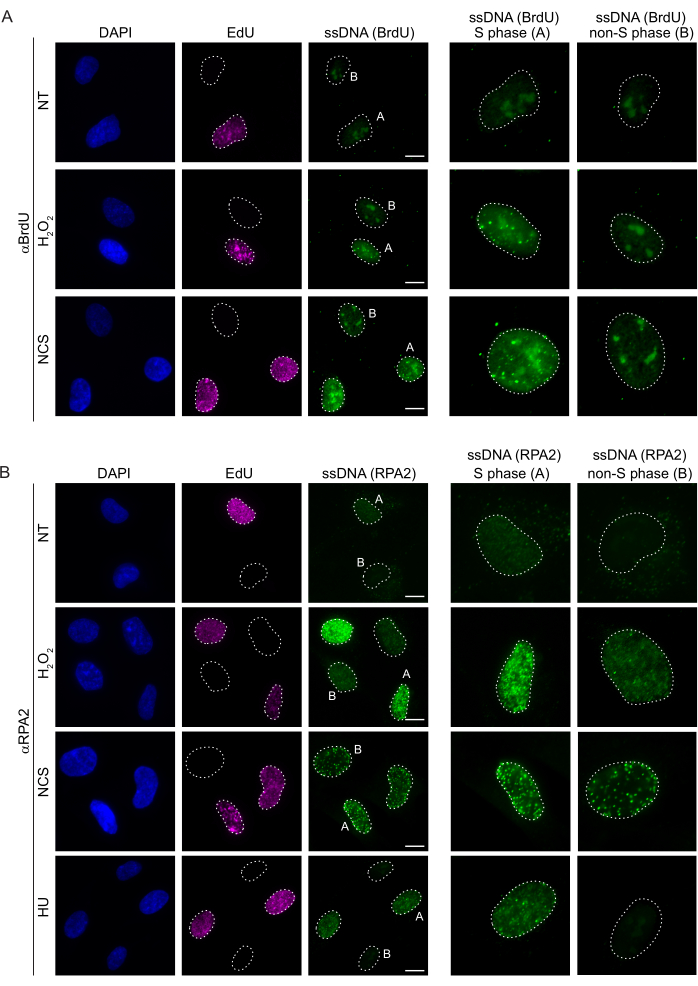
图 2:DNA 损伤时使用 BrdU 抗体或 RPA2 抗体进行 ssDNA 检测。 (A) 代表性图像使用 αBrdU(绿色)显示 ssDNA 病灶,S 期细胞由 EdU(紫色)突出显示,DAPI 用于对核 DNA 进行复染(蓝色)。在进行任何额外处理之前,将RPE1细胞保持在10μM BrdU中48小时。48 小时后,用 10 μM EdU 脉冲细胞 30 分钟,然后用 H2O2 (250 μM) 处理 1 小时或新卡齐诺抑素 (0.5 μg/mL) 4 小时。在CSK预提取后固定细胞。白色虚线表示每个原子核的边界。比例尺 = 5 μm。右侧的面板是指示的 S 相或非 S 相核的放大图像。(B) 代表性图像显示了使用 αRPA2 抗体(绿色)的 ssDNA 病灶。S期细胞由EdU(紫色)突出显示,DAPI用于对核DNA(蓝色)进行复染。用 10 μM EdU 脉冲 RPE1 细胞 30 分钟,然后用 1 小时 H2O2 (250 μM)、4 小时羟基脲 (2 mM) 或 4 小时 NCS (0.5 μg/mL)。在CSK预提取后固定细胞。白色虚线表示每个原子核的边界。比例尺 = 10 μm。右侧的面板是指示的 S 相或非 S 相核的放大图像。缩写:ssDNA = 单链 DNA;BrdU = 5-溴-2'-脱氧尿苷;DAPI = 4',6-二脒基-2-苯基吲哚;RPE1 = hTERT永生化的视网膜色素上皮细胞;EdU = 5-乙炔基-2'-脱氧尿苷;NCS = 新卡齐诺他汀;胡 = 羟基脲。 请点击这里查看此图的较大版本.
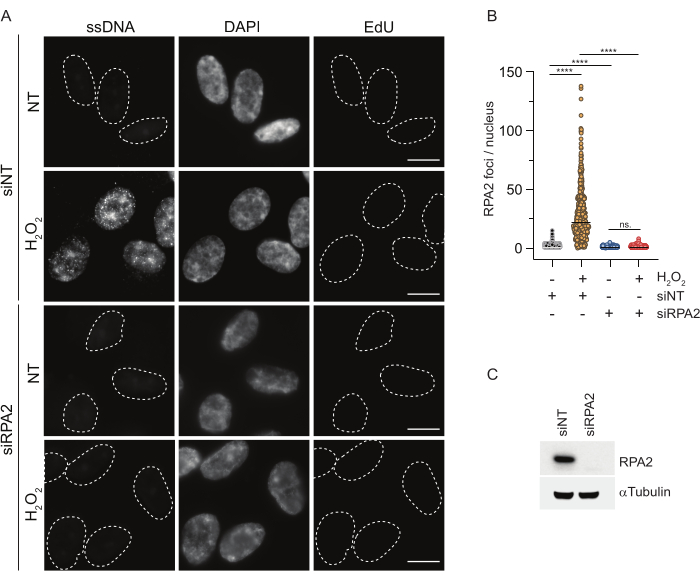
图 3:使用 RPA2 抗体检测 G1 期的 ssDNA 病灶。 (A) 用靶向 RPA2 或非靶向 siRNA 对照转染 RPE1 细胞,随后在 G1 中同步并用 10 μM EdU 脉冲标记 30 分钟,然后用 H2O2 (250 μM) 处理它们 1 小时。DAPI用于对核DNA进行复染。在CSK预提取后固定细胞。白色虚线表示每个原子核的边界。比例尺 = 5 μm。 (B) RPA2 病灶/细胞核数量的测量来自两个独立的实验。在分析过程中仅考虑了 EdU 阴性细胞。线表示图上的平均值。进行非参数方差分析(Kruskal-Wallis)进行统计分析。星星表示 P < 0.0001。分析的细胞核数如下:siNT no H2O2 n = 513,siNT H2O2 n = 603,siRPA2 no H2O2 n = 266,siRPA2 H2O2 n = 536。(C) siRNA 敲低的效率显示在免疫印迹中。缩写:siNT = 非靶向 siRNA 对照;BrdU = 5-溴-2'-脱氧尿苷;DAPI = 4',6-二脒基-2-苯基吲哚;RPE1 = hTERT永生化的视网膜色素上皮细胞;EdU = 5-乙炔基-2'-脱氧尿苷。 请点击这里查看此图的较大版本.

图4:使用斐济对ssDNA病灶进行定量。斐济的详细步骤,展示了如何评估细胞核中的RPA2病灶数量。(A-E)使用 DAPI 通道创建核掩模。(F-H)阈值从背景信号中识别单个核 ssDNA 病灶。缩写:ssDNA = 单链 DNA;DAPI = 4',6-二脒基-2-苯基吲哚。请点击这里查看此图的较大版本.
| 细胞骨架 (CSK) 缓冲液 | |
| 管道 pH 7.0 | 10 米 |
| 氯化钠 | 100 米 |
| 乙二胺四乙酸 pH 值 8 | 1 毫米 |
| 氯化镁2 | 3 毫米 |
| D-蔗糖 | 300毫米 |
| 海卫一X-100 | 0.20% |
| 磷酸酶抑制剂混合物 | 每 10 mL 1 片 |
| 蛋白酶抑制剂混合物 | 每 10 mL 1 片 |
| 在ddH2O中稀释 | 不适用 |
| 洗涤缓冲液 | |
| 海卫一X-100 | 0.05% |
| 在PBS中稀释 | 不适用 |
| 透化缓冲液 | |
| 海卫一X-100 | 0.50% |
| 在PBS中稀释 | 不适用 |
| 固定液 | |
| 多聚甲醛 | 3.60% |
| 海卫一X-100 | 0.05% |
| 在PBS中稀释 | 不适用 |
| 阻塞缓冲区 | |
| 牛血清白蛋白(BSA) | 5% |
| 海卫一X-100 | 0.10% |
| 在PBS中稀释 | 不适用 |
| Click-iT Plus 反应鸡尾酒 | |
| 1x Click-iT 反应缓冲液 | 435毫升 |
| Alexa Fluor PCA解决方案 | 5毫升 |
| CuSO4-铜保护剂预混料 | 10毫升 |
| 1x Click-iT 缓冲液添加剂 | 50毫升 |
| 总体积 | 500毫升 |
表 1:该协议中使用的缓冲液的组成。
补充图S1。 (A) 使用血清饥饿将 RPE1 细胞同步到 G0 期 72 小时,随后通过重新引入血清释放到不同的细胞周期阶段。点图显示处于 G0/G1、S 或 G2/M 期的细胞,其中小时表示血清饥饿后重新添加血清后的时间。右图显示了每种条件下 G0/G1、S 和 G2/M 细胞的百分比。根据制造商的建议,使用市售的 EdU 和碘化丙啶的细胞增殖试剂盒进行 FACS 分析。(B) 图 1 的未裁剪蛋白质印迹扫描。数字显示分子量标记,单位为kDa。PARP1 被用作上样对照,并通过切割膜上样到凝胶上,该凝胶也针对 CCNA2、p27(进一步剥离为 PCNA)和 pH3 (S10)(进一步剥离为 H3)开发。将CCNB1和RPA2上样到单独的凝胶上,使用相同量的蛋白质裂解物以确保可比性。(C) 图 3 的未裁剪蛋白质印迹扫描。数字显示分子量标记,单位为kDa。缩写:EdU = 5-乙炔基-2'-脱氧尿苷。 请点击此处下载此文件。
补充图S2:(A)使用BrdU抗体(绿色)的代表性图像显示了ssDNA病灶;S 期细胞由细胞周期蛋白 A2(红色)突出显示;DAPI用于对核DNA(蓝色)进行复染。在进一步治疗之前,将RPE1细胞保持在10μM BrdU中48小时。48小时后,用H2O2 (250μM)处理细胞1小时或新卡嗔他汀(0.5μg/ mL)4小时,然后固定。白色虚线表示每个原子核的边界。比例尺 = 5 μm。 (B) 有和没有变性条件的 RPE1 细胞的 BrdU 染色。异步 RPE1 细胞用 10 μM BrdU 预处理 48 小时。比例尺 = 10 μm。 (C) BrdU 病灶/细胞核数量的测量来自 G1 同步 RPE1 细胞中的两个独立实验。在分析过程中仅考虑了 EdU 阴性细胞。线表示图上的平均值。进行非参数方差分析(Kruskal-Wallis)进行统计分析。"ns"表示无显著差异。分析的原子核数如下:NT n = 52,NCS n = 105,H2O2 n = 82。缩写:siNT = 非靶向 siRNA 对照;BrdU = 5-溴-2'-脱氧尿苷;DAPI = 4',6-二脒基-2-苯基吲哚;RPE1 = hTERT永生化的视网膜色素上皮细胞;NCS = 新卡司他汀。 请点击此处下载此文件。
补充视频 S1:基于斐济的 RPA2 病灶分析的屏幕录制。请点击此处下载此文件。
讨论
保持健康、无支原体的细胞培养对于上述所有实验都至关重要。在正常培养基下,RPE1细胞与组织培养处理的塑料器皿有很强的附着力;然而,当保持在无血清条件下时,它们的结合特性会显着减弱。此外,为了在显微镜下捕获 ssDNA 病灶的高分辨率图像,需要将细胞接种到 0.17 mm 厚的盖玻片上,该盖玻片的亲水性不足以支持 RPE1 细胞的正确附着。如果没有适当扁平和均匀分布的细胞,可视化单个 ssDNA 病灶是非常具有挑战性的。因此,选择合适的包衣材料(例如玻连蛋白)并在释放细胞进入 G1 期后留出足够的时间(6-12 小时)让细胞扩散和附着至关重要。
该方案的一个具有挑战性的部分是获得均质的 G1 同步 RPE1 细胞。这需要两个关键步骤。首先,为了有效地进行血清饥饿,需要对细胞进行胰蛋白酶消化,用PBS彻底洗涤,然后使用无血清培养基直接接种到新的组织培养皿上。直接在组织培养皿中洗涤细胞以去除血清不会产生有效的 G0 同步。其次,当将细胞释放到 G1 期时,必须再次将细胞胰蛋白酶消化并接种到新鲜的组织培养板上。同样,仅更换培养基并向细胞中添加含血清的培养基不会导致同步进入 G1。此外,为了正确进入 G1,镀膜盖玻片上细胞的接种密度必须处于一定的汇合度水平。虽然完美的细胞同步通常是无法实现的,但这里描述的这种同步方案给出了~97%的纯G1群体。RPE1 在直径为 12 mm 的盖玻片上的推荐接种密度为 ~4 × 104 ,以获得均匀的成像视野,汇合度约为 70%。较高的接种密度会导致细胞在CSK提取后分离和"剥离",并在图像采集过程中产生更高的背景信号。
为了减少任何背景信号并获得有利的信噪比,在一抗和二抗孵育后彻底清洗至关重要。由于要应用许多洗涤步骤,因此在每个洗涤步骤中防止孔变干也很重要。我们通过在所有洗涤和孵育步骤中应用至少 0.05% 的 Triton X-100 来最大限度地减少这种伪影。一旦孔干涸,细胞的信噪比就会改变;这会导致显微镜下出现马赛克状图案,并可能干扰评估。Z-stack图像采集与反卷积相结合,可以帮助捕获不同焦平面的病灶,从而改善分析。
传统方法依赖于在非变性条件下检测掺入的BrdU。然而,这些方法依赖于用高剂量的BrdU对细胞进行至少1-2天的预处理(或相当于所用细胞系中一个完整的细胞周期的时间),以确保均匀的基因组掺入。不可取的是,广泛的 BrdU 掺入会导致细胞周期干扰36。为了解决这些局限性,该方法利用内源性 RPA2 检测 ssDNA 病灶。这种方法不需要复制驱动的BrdU掺入,它也可以用于有丝分裂后细胞。由于不需要大量掺入BrdU,因此可以节省时间并降低实验复杂性。通过使用 RPA2 染色来可视化 ssDNA,我们可以使用 2′-脱氧-5-乙炔基尿苷 (EdU) 和点击化学来标记 DNA 复制,同时避免针对 EdU27、37、38 的 BrdU 抗体可能出现交叉反应。在点击反应过程中,必须特别注意适当地掩盖掺入的 EdU,以便 BrdU 抗体不会与 EdU 发生交叉反应27,39。
最后,使用 RPA2 代替 BrdU 的一个重要好处是,与 S 期外的 BrdU 染色相比,具有更高的信噪比。我们发现,即使在复制细胞中,非变性BrdU染色及其可视化ssDNA的能力也仅限于S期(图2)。BrdU 抗体仅与 ssDNA 延伸中充分暴露的 BrdU 结合。修复蛋白(包括 RPA2)与 ssDNA 延伸的结合可能会抑制或阻碍 ssDNA 中 BrdU 的充分暴露。我们还发现,CSK预提取对于使用BrdU抗体进行ssDNA可视化是必要的。这是可能的,因为如果不从ssDNA轨迹中去除轻结合的蛋白质成分,抗体就无法访问ssDNA轨迹。
尽管如此,该协议仍存在一些限制。使用 RPA2 进行 ssDNA 检测的一个局限性是需要优化 CSK 预提取步骤。在固定细胞之前,必须从DNA中洗掉未结合的过量RPA2。一方面,由于 RPA2 蛋白组分不与 ssDNA 结合,欠提取导致高背景。另一方面,过度提取会导致信号丢失。对于BrdU检测,这不是一个变量,因为BrdU稳定地掺入DNA中,不能通过预提取洗掉。因此,必须仔细考虑CSK预萃取的时间,缓冲液中Triton X-100的量,体积和进行预萃取的温度。CSK 预提取还限制了使用细胞核大小来区分 G0/G1 细胞和 S/G2 细胞。
此外,我们不能排除来自RPA2的某些信号起源于它与其他染色质结合蛋白相互作用物结合的可能性。还必须考虑 RPA2 抗体的物种特异性。该协议中使用的抗体可以识别人、小鼠、大鼠、仓鼠和猴子 RPA2。这种方法的另一个局限性是,并非所有细胞系都可以进行血清饥饿以进行 G0 同步。大多数癌细胞系可以绕过细胞周期检查点,即使在血清剥夺的培养基中也能增殖。虽然血清饥饿是有益的,因为它不会导致DNA损伤,但必须仔细监测它们的细胞同步效率,以确保实现适当的细胞周期阶段富集。对于对血清剥夺无反应的细胞,必须考虑其他细胞同步方法(例如,有丝分裂甩除、CDK1 抑制 G2 阻滞或离心洗脱等非侵入性技术)。另一种可能的方法是使用高内涵成像来测量 EdU 和核 DNA 含量,用于异步细胞的细胞周期分析31。必须考虑使用替代同步方法防止干扰下游分析的影响。例如,使用文献中经常使用的双胸苷阻滞剂或阿菲二科林会导致复制应激和 DNA 损伤40。
DNA修复机制的研究仍然是癌症和细胞生物学领域讨论的焦点。这里介绍的方案为细胞的制备提供了一种有价值的方法,能够在暴露于DNA损伤剂时对ssDNA进行可视化和定量分析。值得注意的是,该协议强调了ssDNA结合蛋白RPA2的利用,证明了其高特异性,可以可视化少量ssDNA病灶,同时避免在所有细胞周期阶段产生不必要的交叉反应。使用 RPA2 具有许多优势,特别是对于旨在分析细胞周期 G1 期细胞的研究人员而言。该协议考虑了几个局限性,并解决了使用RPA2或BrdU染色检测ssDNA时与信号干扰,不需要的背景噪声和交叉反应性相关的问题。
披露声明
作者没有要声明的竞争利益。
致谢
作者感谢米歇尔·帕加诺(Michele Pagano)的支持和有益的见解,感谢阿什利·崔(Ashley Chui)和莎朗·凯萨里(Sharon Kaisari)对手稿的批判性阅读,以及杰弗里·埃斯特拉达(Jeffrey Estrada)和维尔玛·迪亚兹(Vilma Diaz)的持续支持。这项工作得到了美国国立卫生研究院资助GM136250多样性补充的支持。
材料
| Name | Company | Catalog Number | Comments |
| Alpha-tubulin antibody | Sigma-Aldrich | T6074 | primary antibody (1:5,000) |
| Axio Observer Inverted Microscope | Zeiss | na | microscope |
| Bis-Tris Plus Mini Protein Gels, 4-12% | Invitrogen | NW04127BOX | Western Blot |
| Bovine Serum Albumin | Jackson ImmunoResearch | 001-000-162 | blocking |
| BrdU (5-Bromo-2'-deoxyuridine) | Sigma-Aldrich | B5002-100MG | nucleotide analogue |
| BrdU antibody BU1/75 | Abcam | ab6326 | primary antibody (1:500) |
| CellAdhere Dilution Buffer | Stemcell Technologies | 07183 | coating reagent |
| Click-iT Plus EdU Flow Cytometry Assay Kits | Invitrogen | C10632 | flow cytomery |
| Click-iT Plus EdU Cell Proliferation Kit for Imaging, Alexa Fluor 647 dye | Thermo Fisher Scientific | C10640 | click-reaction kit |
| cOmplete ULTRA Protease inhibitor tablets | Sigma-Aldrich | 5892791001 | reagent |
| Countess 3 Automated cell counter | Thermo Scientific | AMQAX2000 | cell counter |
| Coverslip | neuVitro | GG12PRE | tissue culture |
| Cyclin A2 antibody | Santa Cruz Biotechnology | sc-271682 | primary antibody (1:1,000) for IF and WB |
| Cyclin B1 antibody | Santa Cruz Biotechnology | sc-245 | primary antibody (1:5,000) |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D2650-100ML | vehicle control |
| DMEM, high glucose, with HEPES | Gibco | 12430051 | cell culture medium for RPE cells |
| DPBS, no calcium, no magnesium | Gibco | 14190144 | the PBS used throughout the protocol |
| D-Sucrose | Thermo Fisher Scientific | bp220-1 | reagent |
| Eclipse Ti2 Series Epifluorescent Microscope | Nikon | na | microscope |
| EdU (5-Ethynyl-2'-deoxyuridine) | Thermo Fisher Scientific | C10637 | nucleotide analogue |
| Falcon 24-well plate | Corning | 351147 | tissue culture |
| Falcon Cell Culture Dishes 100 mm | Corning | 353003 | tissue culture |
| Fetal Bovine Serum, heat inactivated | Gibco | 16140071 | media supplement |
| Fiji (ImageJ) | NIH | version 1.54f | software and algorithms |
| FxCycle PI/RNase Staining Solution | Invitrogen | F10797 | PI staining |
| Goat anti-mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor Plus 555 | Thermo Fisher Scientific | A21422 | secondary antibody (1:1,000) |
| Goat anti-rat IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor Plus 488 | Thermo Fisher Scientific | A48262 | secondary antibody (1:1,000) |
| Histone H3 antibody | Abcam | ab1791 | primary antibody (1:10,000) |
| hTERT RPE1 | ATCC | CRL-3216 | cell line |
| Hydrochloric acid | Sigma-Aldrich | H1758-100ML | reagent |
| Hydrogen peroxide 30% soultion | Sigma-Aldrich | H1009-100ML | reagent |
| Hydroxyurea,98% powder | Sigma-Aldrich | H8627-5G | reagent |
| Invitrogen Ultra Pure 0.5 M EDTA pH 8.0 | Thermo Fisher Scientific | 15-575-020 | reagent |
| Lipfectamine RNAiMAX Transfection Reagent | Invitrogen | 13778150 | transfection reagent |
| Magnesium chloride solution 1 M | Sigma-Aldrich | M1028-100ML | reagent |
| MycoFluor | Thermo Fisher | M7006 | Mycoplasma Detection Kit |
| Neocarzinostatin from Streptomyces carzinostaticus | Sigma-Aldrich | N9162-100UG | reagent |
| NuPage MES SDS Running Buffer (20x) | Invitrogen | NP0002 | Western Blot |
| onTARGETplus Human RPA2 siRNA | Dharmacon | L-017058-01-0005 | siRNA |
| p27 antibody | BD Biosciences | 610241 | primary antibody (1:1,000) |
| Paraformaldehyde aqueous solution (32%) | Electron Microscopy Sciences | 50-980-494 | fixative |
| PARP1 antibody | Cell Signaling Technology | 9542S | primary antibody (1:1,000) |
| PCNA antibody | Cell Signaling Technology | 13110S | primary antibody (1:2,000) |
| Penicillin-Streptomycin | Gibco | 15140163 | media supplement |
| pH3 antibody | Cell Signaling Technology | 3377S | primary antibody (1:2,000) |
| PhosSTOP phosphatase inhibitor tablets | Sigma-Aldrich | 4906837001 | reagent |
| PIPES Buffer 0.5 M solution, pH 7.0 | Bioworld | 41620034-1 | reagent |
| Precision Plus Protein Kaleidoscope Prestained Protein Standards | Bio-Rad | 1610395 | Western Blot |
| Prism | GraphPad | version 10 | statistical analysis and graph |
| ProLong Diamond Antifade Mountant | Thermo Scientific | P36961 | mounting media |
| Reduced serum media (Opti-MEM) | Gibco | 31985070 | used for transfection |
| Rpa32/rpa2 antibody (mouse) | EMD Millipore | NA19L | primary antibody (1:1,000) for WB |
| Rpa32/rpa2 antibody (rat) | Cell Signaling Technology | 2208S | primary antibody (1:1,000) for IF |
| Sodium Chloride solution (5 M) | Sigma-Aldrich | S5150 | reagent |
| Sodium Pyruvate (100 mM) | Gibco | 11360070 | media supplement |
| Sodium tetraborate decahydrate | Sigma-Aldrich | B3535-500G | reagent |
| Thermo Scientific Pierce DAPI Nuclear Counterstain | Thermo Scientific | 62248 | nucleic acid stain |
| Thymidine,powder | Sigma-Aldrich | T1985-1G | reagent |
| Triton X-100 aqueous solution (10%) | Sigma-Aldrich | 11332481001 | detergent |
| Trypsin-EDTA (0.5%), no phenol red | Gibco | 1540054 | cell dissociation agent |
| Vitronectin XF | Stemcell Technologies | 07180 | coating reagent |
| ZE5 Cell Analyzer | Bio-Rad | na | flow cytomery |
参考文献
- Hakem, R. DNA-damage repair; the good, the bad, and ugly. EMBO J. 27 (4), 589-605 (2008).
- Gutierrez, R., O'Connor, T. R. DNA direct reversal repair and alkylating agent drug resistance. Cancer Drug Resist. 4 (2), 414-423 (2021).
- Krokan, H. E., Bjoras, M. Base excision repair. Cold Spring Harb Perspect Biol. 5 (4), a012583(2013).
- Marteijn, J. A., Lans, H., Vermeulen, W., Hoeijmakers, J. H. Understanding nucleotide excision repair and its roles in cancer and ageing. Nat Rev Mol Cell Biol. 15 (7), 465-481 (2014).
- Li, G. M. Mechanisms and functions of DNA mismatch repair. Cell Res. 18 (1), 85-98 (2008).
- Hustedt, N., Durocher, D. The control of DNA repair by the cell cycle. Nat Cell Biol. 19 (1), 1-9 (2016).
- Yang, W., Gao, Y. Translesion and repair DNA polymerases: diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Bhat, D. S., et al. Therapeutic disruption of RAD52-ssDNA complexation via novel drug-like inhibitors. NAR Cancer. 5 (2), zcad018(2023).
- Gupta, P., Saha, B., Chattopadhyay, S., Patro, B. S. Pharmacological targeting of differential DNA repair, radio-sensitizes WRN-deficient cancer cells in vitro and in vivo. Biochem Pharmacol. 186, 114450(2021).
- Pena-Diaz, J., et al. Noncanonical mismatch repair as a source of genomic instability in human cells. Mol Cell. 47 (5), 669-680 (2012).
- Schroering, A. G., Edelbrock, M. A., Richards, T. J., Williams, K. J. The cell cycle and DNA mismatch repair. Exp Cell Res. 313 (2), 292-304 (2007).
- Scully, R., Panday, A., Elango, R., Willis, N. A. DNA double-strand break repair-pathway choice in somatic mammalian cells. Nat Rev Mol Cell Biol. 20 (11), 698-714 (2019).
- Escribano-Diaz, C., et al. A cell cycle-dependent regulatory circuit composed of 53BP1-RIF1 and BRCA1-CtIP controls DNA repair pathway choice. Mol Cell. 49 (5), 872-883 (2013).
- Genschel, J., Modrich, P. Mechanism of 5'-directed excision in human mismatch repair. Mol Cell. 12 (5), 1077-1086 (2003).
- Hu, J., et al. Nucleotide excision repair in human cells: fate of the excised oligonucleotide carrying DNA damage in vivo. J Biol Chem. 288 (29), 20918-20926 (2013).
- Huertas, P., Jackson, S. P. Human CtIP mediates cell cycle control of DNA end resection and double strand break repair. J Biol Chem. 284 (14), 9558-9565 (2009).
- Keijzers, G., et al. Human exonuclease 1 (EXO1) regulatory functions in DNA replication with putative roles in cancer. Int J Mol Sci. 20 (1), 74(2018).
- Symington, L. S. End resection at double-strand breaks: mechanism and regulation. Cold Spring Harb Perspect Biol. 6 (8), a016436(2014).
- Liu, Y., et al. DNA polymerase beta and flap endonuclease 1 enzymatic specificities sustain DNA synthesis for long patch base excision repair. J Biol Chem. 280 (5), 3665-3674 (2005).
- Wold, M. S., Kelly, T. Purification and characterization of replication protein A, a cellular protein required for in vitro replication of simian virus 40 DNA. Proc Natl Acad Sci U S A. 85 (8), 2523-2527 (1988).
- Wienholz, F., Vermeulen, W., Marteijn, J. A. Amplification of unscheduled DNA synthesis signal enables fluorescence-based single cell quantification of transcription-coupled nucleotide excision repair. Nucleic Acids Res. 45 (9), e68(2017).
- Wold, M. S. Replication protein A: a heterotrimeric, single-stranded DNA-binding protein required for eukaryotic DNA metabolism. Annu Rev Biochem. 66, 61-92 (1997).
- Chen, R., Wold, M. S. Replication protein A: single-stranded DNA's first responder: dynamic DNA-interactions allow replication protein A to direct single-strand DNA intermediates into different pathways for synthesis or repair. Bioessays. 36 (12), 1156-1161 (2014).
- Kang, Y., et al. Alteration of replication protein A binding mode on single-stranded DNA by NSMF potentiates RPA phosphorylation by ATR kinase. Nucleic Acids Res. 51 (15), 7936-7950 (2023).
- Kilgas, S., Kiltie, A. E., Ramadan, K. Immunofluorescence microscopy-based detection of ssDNA foci by BrdU in mammalian cells. STAR Protoc. 2 (4), 100978(2021).
- Madabhushi, R., Pan, L., Tsai, L. H. DNA damage and its links to neurodegeneration. Neuron. 83 (2), 266-282 (2014).
- Liboska, R., Ligasova, A., Strunin, D., Rosenberg, I., Koberna, K. Most anti-BrdU antibodies react with 2'-deoxy-5-ethynyluridine -- the method for the effective suppression of this cross-reactivity. PLoS One. 7 (12), e51679(2012).
- Biehs, R., et al. DNA double-strand break resection occurs during non-homologous end joining in G1 but is distinct from resection during homologous recombination. Mol Cell. 65 (4), 671-684.e5 (2017).
- Cruz-Garcia, A., Lopez-Saavedra, A., Huertas, P. BRCA1 accelerates CtIP-mediated DNA-end resection. Cell Rep. 9 (2), 451-459 (2014).
- Ercilla, A., et al. Physiological tolerance to ssDNA enables strand uncoupling during DNA replication. Cell Rep. 30 (7), 2416-2429.e7 (2020).
- Lezaja, A., et al. RPA shields inherited DNA lesions for post-mitotic DNA synthesis. Nat Commun. 12 (1), 3827(2021).
- Mukherjee, B., Tomimatsu, N., Burma, S. Immunofluorescence-based methods to monitor DNA end resection. Methods Mol Biol. 1292, 67-75 (2015).
- Ochs, F., et al. 53BP1 fosters fidelity of homology-directed DNA repair. Nat Struct Mol Biol. 23 (8), 714-721 (2016).
- Raderschall, E., Golub, E. I., Haaf, T. Nuclear foci of mammalian recombination proteins are located at single-stranded DNA regions formed after DNA damage. Proc Natl Acad Sci U S A. 96 (5), 1921-1926 (1999).
- Forment, J. V., Walker, R. V., Jackson, S. P. A high-throughput, flow cytometry-based method to quantify DNA-end resection in mammalian cells. Cytometry A. 81 (10), 922-928 (2012).
- Mistrik, M., et al. Cells and stripes: A novel quantitative photo-manipulation technique. Sci Rep. 6, 19567(2016).
- Aten, J. A., Bakker, P. J., Stap, J., Boschman, G. A., Veenhof, C. H. DNA double labelling with IdUrd and CldUrd for spatial and temporal analysis of cell proliferation and DNA replication. Histochem J. 24 (5), 251-259 (1992).
- Podgorny, O., Peunova, N., Park, J. H., Enikolopov, G. Triple S-phase labeling of dividing stem cells. Stem Cell Reports. 10 (2), 615-626 (2018).
- Cappella, P., Gasparri, F., Pulici, M., Moll, J. Cell proliferation method: click chemistry based on BrdU coupling for multiplex antibody staining. Curr Protoc Cytom. Chapter 7, Unit7.34 (2008).
- Ligasova, A., Koberna, K. Strengths and weaknesses of cell synchronization protocols based on inhibition of DNA synthesis. Int J Mol Sci. 22 (19), 10759(2021).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。