A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
ביצוע מסך CRISPR רחב גנום במבחנה בתאי T של קולטן אנטיגן כימרי
In This Article
Summary
המאמר מתאר פרוטוקול ליישום מודל in vitro לתשישות כדי להשלים מסך נוקאאוט CRISPR כלל גנומי בתאי T קולטן אנטיגן כימרי בריא מתורם.
Abstract
טיפול תאי בקולטן אנטיגן כימרי T (CART) הוא צורה חדשנית של אימונותרפיה ממוקדת מטרה שחוללה מהפכה בטיפול בסרטן. עם זאת, התגובה העמידה נותרה מוגבלת. מחקרים אחרונים הראו כי הנוף האפיגנטי של תוצרי תאי CART טרום עירוי יכול להשפיע על התגובה לטיפול, ועריכת גנים הוצעה כפתרון. עם זאת, יש לעשות עבודה נוספת כדי לקבוע את אסטרטגיית עריכת הגנים האופטימלית. מסכי CRISPR רחבי גנום הפכו לכלים פופולריים הן לחקר מנגנוני התנגדות והן לאופטימיזציה של אסטרטגיות עריכת גנים. עם זאת, היישום שלהם על תאים ראשוניים מציב אתגרים רבים. כאן אנו מתארים שיטה להשלמת מסך נוקאאוט CRISPR כלל גנומי בתאי CART מתורמים בריאים. כמודל הוכחת היתכנות, תכננו שיטה זו כדי לחקור את התפתחות התשישות בתאי CART המכוונים לאנטיגן CD19. עם זאת, אנו מאמינים כי ניתן להשתמש בגישה זו כדי לטפל במגוון מנגנונים של עמידות לטיפול במבני CAR שונים ובמודלים של גידולים.
Introduction
טיפול תאי קולטן אנטיגן כימרי T (CART) הראה הצלחה מרשימה בטיפול בממאירויות של תאי B; עם זאת, התגובה העמידה מוגבלת ל-30-40%1,2,3,4,5. בעוד שחוקרים פיתחו ובדקו מספר גישות לטיפול במנגנוני עמידות לטיפול בתאי CART, כולל אופטימיזציה של תכנון CAR, עריכת גנים וטיפולים משולבים, התפתחות העמידות נותרה בלתי ידועה במידה רבה. לאחרונה, יש ראיות הולכות וגדלות לכך שפרופיל ביטוי הגנים הבסיסי של תוצרי תאי CART טרום עירוי הוא גורם חשוב הן לרעילות והן ליעילות של טיפול CART 6,7,8,9. ככזה, עריכת גנים במהלך ייצור תאי CART הפכה לגישה פופולרית לשיפור ההצלחה בטיפול בתאי CART.
לדוגמה, המעבדה שלנו הראתה בעבר כי שימוש בטכנולוגיית Cas9 של חזרות פלינדרומיות קצרות מקובצות במרווחים קבועים (CRISPR) כדי לחסל את הגן גרנולוציטים-מקרופאגים-גורם מגרה מושבה (GM-CSF) משפר את פעילות תאי CART ומפחית סימנים של רעילות הקשורה ל-CART 10,11,12. בנוסף, טיפול תאי מהונדס CRISPR נבדק בניסויים קליניים והראה יעילות ובטיחות13. יחד, זה מצביע על כך שעריכת גנים עם טכנולוגיית CRISPR יכולה לא רק לשפר את ההבנה שלנו של ביולוגיה של תאי CART אלא גם ליצור תוצרים של תאי CART הניתנים לתרגום.
מסכי CRISPR רחבי גנום הפכו לכלים פופולריים יותר ויותר במחקר הביולוגיה של הסרטן כדי להבין מנגנונים של עמידות לטיפולים. בטכניקה זו, עשרות אלפי רנ"א מנחה (gRNAs) מועברים למאגרי תאים כדי לעודד כניסה של gRNA אחד לכל תא14. לאחר מכן, תאים עוברים תנאים מעוררי לחץ שבהם תאים שהותמרו עם gRNA המכוונים לגנים חיוניים מתים, ותאים שהועתקו עם gRNA המכוונים לגנים מעכבים שורדים ומתרבים. באמצעות ריצוף הדור הבא, אנו יכולים לקבוע כיצד ייצוג gRNA משתנה בכל מסך הקריספר14.
עם זאת, קנה המידה וזמן הבחירה הנדרשים להפרעות גנים ברחבי הגנום יכולים להיות מאתגרים לביצוע בתאים ראשוניים, כמו תאי CART. לפיכך, קבוצות השתמשו במסכי CRISPR ממוקדים כדי להבין טוב יותר את מנגנוני ההתנגדות הטיפולית15,16. לעתים קרובות קל יותר להשלים מסכים ייעודיים בתאים ראשיים מכיוון שיש להם ספריות קטנות יותר הדורשות פחות תאים כדי להשיג ייצוג ספריה הולם. בעוד שמחקרים אלה שיפרו את הבנתנו את מנגנוני העמידות לטיפול בתאי CART, מסכים ממוקדים מציגים הטיה עקב הבחירה הידנית של מטרות גנים. מאמר זה מבקש לתאר שיטה להשלמת מסך CRISPR רחב גנום במבחנה בתאי CART מתורמים בריאים. ככזו, גישה זו של תפוקה גבוהה מאפשרת זיהוי יעיל ובלתי משוחד של מסלולים וגנים מרכזיים שניתן לערוך כדי לשפר את התגובות הטיפוליות 17,18,19.
בפרט, הפרוטוקול המתואר במאמר זה נועד להגביר את הבנת השדה של תשישות תאי CART על ידי השלמת מסך הנוקאאוט CRISPR רחב הגנום עם מודל in vitro לתשישות. תשישות היא גורל תאי CART לא מתפקד שהיה מעורב באי-תגובה לטיפול בתאי CART20,21,22. גורל תאי זה ידוע כמווסת אפיגנטית, והוא מאופיין בירידה בהתפשטות תאי CART, ירידה בייצור ציטוקינים משפיעים ועלייה בביטוי קולטנים מעכבים23. בספרות קודמת, עריכת גנים הצליחה למנוע התפתחות של תשישות על ידי ויסות מעלה או מטה של גני מפתח 24,25,26. בהתחשב הן בירידה ביכולת ההתרבות כאשר תאי CART מתרוקנים והן בראיות לכך שעריכת גנים יכולה למנוע את התרחשותו, מידלנו את מסך הנוקאאוט CRISPR שלנו בגנום חוץ גופי על גורל תאי זה. עם זאת, פרוטוקול זה עשוי להיות מתוקן בעתיד כדי לחקור מנגנונים אחרים של התנגדות לטיפול בתאי CART.
Protocol
חשוב לציין, הפרוטוקול המתואר להלן עוקב אחר הנחיות וקיבל אישור מוועדת הביקורת המוסדית של מאיו קליניק (IRB 18-005745) והוועדה לבטיחות ביולוגית מוסדית (IBC HIP00000252.43). כל עבודת תרבית תאים, כולל ייצור לנטיויראלי, צריכה להתבצע במכסה מנוע של תרבית תאים עם ציוד מגן אישי מתאים. בפרט, יש לבצע עבודה לנטי-ויראלית תחת אמצעי זהירות ברמת בטיחות ביולוגית 2 (BSL-2), כולל שימוש באקונומיקה של 10% לחיטוי פריטים לפני השלכה.
1. הגברה של ספריית CRISPR
- הגבירו את ספריית הקריספר בהתאם להמלצות היצרן. בקצרה, אלקטרופורס את ספריית הקריספר לארבעה אליציטוטים של תאים אלקטרו-מוכשרים על ידי השלמת הפעולות הבאות:
- הוסף 2 μL של 50 ng/μL פלסמיד ספריית CRISPR ל 25 μL של תאים electrocompetent בקובט 1.0 מ"מ.
- אלקטרופורציה של הדגימות באמצעות ההגדרות הבאות: 10 μF, 600 Ohms, 1,800 וולט וקבוע זמן בין 3.5 אלפיות השנייה ל-4.5 אלפיות השנייה.
- לשחזר 975 μL של מדיה התאוששות ולהעביר צינור עם 1 מ"ל נוסף של מדיה התאוששות.
- סובב את כל aliquots ב 250 סל"ד במשך 1 שעה ב 37 ° C.
- לוח את הטרנספורמציות:
- אגרו את כל האליציטוטים של תאים מחושמלים וערבבו היטב.
- צלחת כל התאים electroporated על ידי פיזור 400 μL של תערובת טרנספורמציה על צלחות מרק Luria שחוממו מראש 10 ס"מ (LB) עם 100 מיקרוגרם / מ"ל carbenicillin. מניחים את הצלחת באינקובטור ב 32 ° C למשך 14 שעות.
- קציר המושבות:
- פיפטה 1 מ"ל של מדיה LB + 100 מיקרוגרם / מ"ל carbenicillin על כל צלחת. מגרדים את המושבות מכל צלחת ואוספים אותן לתוך צינור חרוטי של 50 מ"ל.
- הוסף עוד 1 מ"ל של מדיה LB + 100 מיקרוגרם / מ"ל קרבניצלין לכל צלחת כדי לשטוף ולהבטיח התאוששות מלאה.
- בצע הכנה מקסימלית על ידי ביצוע ההוראות של ערכת maxi-prep.
- מאגר את ה- DNA פלסמיד שנוצר.
2. ריצוף הדור הבא לאימות ייצוג gRNA בסיסי בספריית CRISPR
הערה: פריימרים של NGS עבור ספריית CRISPR זו תוכננו בפרסום קודם27. שימוש בפריימר הפוך שונה לכל דגימה יבצע ברקוד לכל דגימה ויאפשר איגום דגימות במהלך הריצוף.
- הכן את הדגימות עבור PCR עם תערובת התגובה הבאה (נפח כולל = 50 μL): תערובת מאסטר PCR באיכות גבוהה, 2x: 25 μL; DNA תבנית מאוגמת, 0.5 מיקרוגרם: 1 μL; פריימר קדמי לספריית NGS, 10 מיקרומטר: 1.25 מיקרוליטר; פריימר הפוך לספריית NGS, 10 מיקרומטר: 1.25 מיקרוליטר; מים ללא DNase: 21.5 μL.
- בצע PCR עם תנאי הרכיבה הבאים: מחזור 1: דנטורציה ב 98 ° C למשך 3 דקות; מחזורים 2-23: denature ב 98 ° C עבור 10 שניות, anneal ב 63 ° C עבור 10 שניות, להאריך ב 72 ° C במשך 25 שניות; מחזור 24: הארכה ב-72°C למשך 2 דקות.
- לטהר את תגובת ה-PCR על ידי איגום תגובות ה-PCR לכל דגימה וטיהור מוצר ה-PCR בערכת טיהור PCR בהתאם להוראות היצרן.
- יש להריץ 3 מיקרוגרם של מוצר PCR עבור כל דגימה על ג'ל אגרוז 2% (w/v).
- הסר את מוצר ה- PCR (~ 260-270 bp) מהג'ל על ידי חילוץ ה- DNA מהג'ל באמצעות ערכת מיצוי ג'ל בהתאם להוראות היצרן. יש לשאוף ל-500 ננוגרם של דנ"א לפחות.
- אחסן את הדגימות שחולצו ב -20 ° C.
- בצעו ריצוף מהדור הבא (NGS) עם הדגימות וכוונו ל-100 קריאות לכל gRNA27.
- נתח את תוצאות הריצוף באמצעות צינור MAGeCK-VISPR וודא שיותר מ- 99.5% מה- gRNA בספרייה מיוצגים28.
3. ייצור ספריית CRISPR ולנטי-וירוסים לביטוי CAR
- הכן lentivirus כפי שתואר קודם10,29.
- יום 1: התחילו עם צלוחיות T175 של 80-90% תאי 293T שגדלו במצע D10 המורכב מ-10% נסיוב בקר עוברי (FBS) (v/v), 1% פניצילין-סטרפטומיצין-גלוטמין (PSG) (v/v), ותווך העיט המעובד של דולבקו. לפני שמתחילים, ריאגנטים טרנספקציה חמה ו- DNA לטמפרטורת החדר ולכמת את ה- DNA.
- עבור כל בקבוק 293T, סמן שני צינורות חרוטי 50 מ"ל A ו - B. הוסף 4.5 מ"ל של מדיום סרום מופחת לצינור A ולצינור B.
- בצינור מיקרוצנטריפוגה סטרילי של 1.5 מ"ל, מערבבים את הדנ"א הבא ומאפשרים לשבת במשך דקה אחת: 18 מיקרוגרם של pCMVR8.74 (gag, pol, tat, ו- rev), 7 מיקרוגרם של pMD2.G (VSV-G), ו- 15 מיקרוגרם של וקטור ביטוי לנטיוויראלי (ספריית קריספר A או וקטורים CAR19-28ζ).
- הוסף 129 μL של מגיב transfection לצינור A. מערבבים היטב צינור A על ידי מערבולת אצבע.
- מוסיפים את תערובת הדנ"א לצינור B. מערבבים היטב את צינור B על ידי מערבולת אצבע.
- הוסף 111 μL של מגיב קו-ליפידים נייטרלי לצינור B. מערבבים היטב את צינור B על ידי מערבולת אצבע.
- מוסיפים את תכולת צינור A לצינור B ומערבבים היטב על ידי מערבולת אצבע. יש לדגור על צינור B למשך 30 דקות בטמפרטורת החדר.
- לקראת סוף תקופת הדגירה בת 30 הדקות, הכינו את תאי 293T על ידי שאיפת המדיה הקיימת. הוסף 16 מ"ל של מדיה R10 המורכבת מ- 10% FBS (v/v), 1% PSG (v/v) ו- Roswell Park Memorial Institute (RPMI) 1640.
- לאחר תקופת הדגירה של 30 דקות, הוסף את התוכן של צינור B (כ 9 מ"ל) טיפה לבקבוק של 293T תאים. הניחו את הבקבוק וסובבו אותו בעדינות לפני הדגירה בטמפרטורה של 37°C, 5% CO2.
- ביום 2 (כ 24 שעות לאחר transfection), לאסוף lentivirus כדלקמן:
הערה: יש להכין מראש את כל הציוד והחומרים ולבצע את הפעולות הבאות על קרח כדי לשמור על קור הסופרנאטנט הנגיפי.- מעבירים את הסופרנאטנט מצלוחיות 293T הנגועות לצינורות חרוטיים מקוררים של 50 מ"ל.
- הוסף 30 מ"ל של חומרי הדפסה R10 שחוממו מראש לצלוחיות 293T.
- פסולת תאית גלולה על ידי צנטריפוגה של צינורות חרוטיים 50 מ"ל ב 900 × גרם במשך 10 דקות ב 4 ° C ברוטורים סגורים.
- לאחר הצנטריפוגה, מסננים את הסופרנאטנט עם מסנן 0.45 מיקרומטר. סובבו את התסנין באולטרה-צנטריפוגה בטמפרטורה של 112,700 × גרם ב-4°C למשך שעתיים.
- לאחר סיבוב של 2 שעות, שואפים את הסופרנאטנט עד שנשאר בערך 5 מ"ל. השהה מחדש את החלקיקים הנגיפיים על ידי פיפטוף עדין למעלה ולמטה פי 10. יש לאחסן את המתלה הנגיפי בטמפרטורה של 4°C למשך הלילה.
- ביום השלישי, לאסוף ולאחסן lentivirus כדלקמן:
- מעבירים את הסופרנאטנט מצלוחיות 293T הנגועות לצינורות חרוטיים מקוררים של 50 מ"ל.
- הוסיפו 30 מ"ל של 10% אקונומיקה לצלוחיות 293T.
- משחררים את הפסולת התאית בסופרנאטנט על ידי צנטריפוגה של צינורות חרוטי 50 מ"ל ב 900 × גרם במשך 10 דקות ב 4 ° C ברוטורים סגורים.
- לאחר הצנטריפוגה, מסננים את הסופרנאטנט עם מסנן 0.45 מיקרומטר. סובבו את התסנין באולטרה-צנטריפוגה במהירות של 112,700 × גרם ב-4°C למשך שעתיים.
- לאחר הצנטריפוגה של שעתיים, שאפו את הסופרנאטנט עד שיישאר כ-2 מ"ל, והשהו מחדש על ידי פיפטציה עדינה מעלה ומטה פי 10. לאחסן את supernatant ויראלי ב 300-500 μL aliquots ולשמור על -80 ° C.
4. חישוב של titer ויראלי עבור LENTIVIRUS מבטא CAR
הערה: רכב LENTIVIRUS היה TITERED כפי שתואר קודם10,12.
- ביום 1, הפעל וצלחת תאי T .
- בודד 1 × 106 תאי T ממרכזיות תורם בריא באמצעות ערכת בחירה שלילית.
- ספור את תאי ה- T.
- שטפו מספיק חרוזי CD3+/CD28+ כדי לעורר את תאי ה-T ביחס חרוז:תא של 3:1. לדוגמה, כדי לעורר 1 × 106 תאי T, לשטוף 3 × 106 חרוזים. לשם כך, שטפו את החרוזים על ידי השלמת 3x הבאים:
- השתמש בהפרדה מגנטית כדי להסיר את החרוזים מהמתלים.
- הסר את supernatant.
- הוסף 1000 μL של מדיה של תאי T (TCM) המורכבת מ-10% סרום אנושי (v/v), 1% PSG (v/v) ותווך תאים המטופוייטים.
הערה: יש לעקר TCM לפני השימוש על ידי סינון עם מסנן ואקום סטרילי 0.45 מיקרומטר ולאחר מכן עם מסנן ואקום סטרילי 0.22 מיקרומטר. - הוסף חרוזים לתרחיף תאי T ביחס חרוז:תא של 3:1.
- השהה מחדש את תאי T ל- 1 ×10 6 תאים/מ"ל ב- TCM.
- הוסף 100 μL של תאי T פעילים ל -10 בארות של צלחת 96 בארות. לדגור את הצלחת ב 37 ° C, 5% CO2.
- ביום השני, הוסף דילול של CAR lentivirus על ידי יצירת צלחת דילול כדלקמן:
- הוסף 100 μL של TCM לשבע בארות של צלחת 96 בארות.
- לבאר הראשונה, להוסיף 50 μL של וירוס עניין; מערבבים היטב.
- לדלל את הנגיף עוד יותר על ידי הוספת 50 μL מבאר 1 לבאר 2; מערבבים היטב.
- מוסיפים 50 μL מבאר 2 לבאר 3 וממשיכים בדפוס זה עד באר 7.
- לאחר ערבוב באר 7, להסיר 50 μL. ודא כי כל הבארות יש נפח כולל של 100 μL.
- הוסף 50μL מכל באר של צלחת הדילול לשבע הבארות הראשונות של צלחת תאי T מהיום הראשון. מערבבים היטב.
- לשלוש הבארות האחרונות של צלחת תא T, להוסיף 50 μL של TCM; מערבבים היטב. אחסנו את הצלחת באינקובטור בטמפרטורה של 37°C, 5% CO2.
- ביום 3, הזינו את התאים על ידי הוספת 100 מיקרוליטר של TCM לכל הבארות של צלחת תאי T; מערבבים היטב.
- ביום הרביעי, בצעו ציטומטריית זרימה כדי לזהות את אחוז התאים המבטאים CAR עם נוגדן שרשרת אנטי-קאפה, סמן חלופי לביטוי CAR, על ידי השלמת הפעולות הבאות:
- סובבו את צלחת תאי ה-T במהירות של 1,000 × גרם למשך 3 דקות ונטרלו.
- שטפו את הצלחת 3x עם חיץ נוגדנים נגד שרשרת קאפה (המורכב מאלבומין 4% בסרום בקר ב-PBS (w/v)) על ידי הוספת 200 μL של חיץ נוגדנים נגד שרשרת קאפה, סיבוב הצלחת ב-1,000 × גרם למשך 3 דקות, ודקאנט.
- הוסיפו נוגדן שרשרת ביוטין נגד קאפה לכל באר. יש לדגור במשך 45 דקות על נדנדה בטמפרטורה של 4 מעלות צלזיוס.
- שטפו את הצלחת 3x עם חיץ נוגדנים נגד שרשרת קאפה כמו בשלב 4.4.2.
- הכתימו את התאים לציטומטריית זרימה עם נוגדן סטרפטאבידין מצומד פלואורוכרום ונוגדן חי/מת. דגרו על הדגימות במשך 15 דקות בטמפרטורת החדר בחושך.
- שטפו את הדגימות פעם אחת במאגר זרימה (המורכב מ-2% FBS (v/v) ו-1% נתרן אזיד (v/v) ב-PBS) על ידי הוספת 200 μL של חיץ זרימה, סיבוב הצלחת ב-1,000 × גרם למשך 3 דקות ודקאנט.
- השהה מחדש ב 100 μL של חיץ זרימה והפעל על הציטומטר.
- בצע gating שלילי עם שלוש בארות untransmeded.
5. חישוב של titer ויראלי עבור lentivirus ספריית CRISPR
הערה: פרוטוקול זה מתאר מספר מינימלי של תאי T לטיטרציה של לנטיוירוס ספריית קריספר, אך ניתן להגדיל אותו כך שיתאים ליותר תאי T ולכן לנפחים גדולים יותר של וירוסים.
- ביום 0, הפעל את תאי ה- T על ידי הוספת 18 × 106 חרוזי CD3+/CD28+ (יחס חרוז 3:1) ל- 6 × 106 תאי T והבאת הנפח הסופי ל- 6 מ"ל עם TCM. Aliquot 0.5 מ"ל של השעיית תא T לתוך 12 בארות של צלחת 48 בארות. יש לאחסן באינקובטור בטמפרטורה של 37°C, 5% CO2.
- עשרים וארבע שעות לאחר מכן, ביום הראשון, הוסף וירוס CAR בריבוי הדבקה (MOI) של 3 לכל באר. מערבבים היטב על ידי פיפטינג למעלה ולמטה מספר פעמים. יש לאחסן באינקובטור בטמפרטורה של 37°C, 5% CO2.
- ארבעים ושמונה שעות מאוחר יותר, ביום השני, הוסף סדרה של נפחי וירוסי ספריית קריספר לתאי CART המופעלים על-ידי הוספת אחד מהנפחים הבאים של וירוס קריספר לכל באר (0 μL, 0 μL, 6.25 μL, 6.25 μL, 12.5 μL, 12.5 μL, 25 μL, 25 μL, 50 μL, 50 μL, 100 μL או 100 μL). מערבבים היטב על ידי פיפטינג למעלה ולמטה מספר פעמים. יש לאחסן באינקובטור בטמפרטורה של 37°C, 5% CO2.
- ביום השלישי, הוסף 1 מיקרוגרם של פורומיצין לכל מ"ל של מדיה למחצית הבארות, כולל באר אחת שקיבלה כל אחד מנפחי וירוס הקריספר הבאים (0 μL, 6.25 μL, 12.5 μL, 25 μL, 50 μL ו- 100 μL). הבארות האחרות לא יטופלו בפורומיצין.
- בימים 4-8, ספרו את תאי ה-T מדי יום והתאימו את צפיפות התאים הסופית ל-1 × 106 תאים/מ"ל עם 1 מיקרוגרם/מ"ל פורומיצין ב-TCM עבור בארות הטיפול בפורומיצין ועם TCM עבור הבארות הלא מטופלות.
- ביום 8, ספרו את מספר התאים החיים בכל באר וחשבו את יחידות הטרנסדוקציה/מ"ל של סופרנאטנט נגיפי באמצעות המשוואה הבאה:
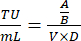
כאשר A: ספירת תאים חיים בהמרה ויראלית היטב; B: ספירת תאים חיים בבקרת ברירה ללא אנטיביוטיקה; V: נפח הנגיף המשמש להדבקה בכל באר; D: דילול קיפול. - באמצעות TU/mL המחושב היטב מבאר המציגה יעילות התמרה של כ -20%, חשב את נפח הנגיף הדרוש כדי להתמיר 1 × 106 תאים ב- MOI של 0.3 באמצעות הנוסחה הבאה:
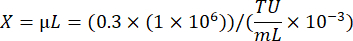
6. שלב ייצור תאי CART של מסך CRISPR (ימים 0 - 8)
הערה: הפרוטוקול הבא מתאר את השלמת מסך הקריספר בשכפול ביולוגי אחד. עם זאת, פרוטוקול זה אומת בעבר עם שלושה עותקים ביולוגיים ואנו ממליצים על שימוש בלפחות שלושה עותקים ביולוגיים עבור אחרים המשלימים פרוטוקולזה 30.
- יום 0: בודדו ועוררו תאי T מדם תורם בריא כפי שתואר קודם לכן10.
- בודד תאי דם חד-גרעיניים היקפיים (PBMCs) מדם תורם בריא שזהותו אינה מזוהה.
- הוסף 15 מ"ל של מדיום שיפוע צפיפות לצינור הפרדת שיפוע צפיפות.
- הוסף דם מדולל 1:2 לחלק העליון של צינור ההפרדה שיפוע הצפיפות. צנטריפוגה ב-1,200 × גרם למשך 10 דקות בטמפרטורת החדר.
הערה: יש לדלל את הדם במאגר בידוד (2% FBS (v/v) במי מלח חוצצי פוספט [PBS]). - מסכן מהשכבה העליונה לתוך צינור חרוטי חדש של 50 מ"ל ומכפיל את הנפח עם חיץ בידוד. צנטריפוגה את הדגימה ב 300 × גרם במשך 8 דקות ב 4 ° C. שאפו את הסופר-נאנט.
- השהה מחדש את גלולת התא עם מאגר בידוד וספור את PBMCs.
- בודד תאי T ממרכזיות באמצעות ערכת בחירה שלילית. בצע את הוראות היצרן עבור הערכה.
הערה: נניח התאוששות של 10-20% של תאי T מ- PBMCs. - לעורר תאי T עם חרוזי CD3+/CD28+ .
- ספור את תאי ה- T.
- Aliquot מספיק CD3+/CD28+ חרוזים כדי לעורר את תאי T ביחס חרוז:תא 3: 1. לדוגמה, כדי לעורר 110 × 106 תאי T, aliquot 330 × 106 חרוזים. שטפו את החרוזים פי 3 על ידי הפרדת החרוזים תחילה מהמתלים עם הפרדה מגנטית ולאחר מכן הוספת 1,000 מיקרוליטר TCM.
- הוסף את החרוזים למתלה תא T. השהה מחדש את תאי ה- T ל- 1 ×10 6 תאים/מ"ל ב- TCM.
- Aliquot תאי T בלוחות 6 בארות ליעילות ההמרה הטובה ביותר. הוסף 5 × 106 תאי T לכל באר. לדגור על התאים ב 37 ° C, 5% CO2 במשך 24 שעות.
- בודד תאי דם חד-גרעיניים היקפיים (PBMCs) מדם תורם בריא שזהותו אינה מזוהה.
- יום 1: בתנאי BSL-2+, הפשירו מספיק CAR lentivirus כדי להתמיר תאי T בריבוי זיהומים של 3.0. שברו בעדינות את רוזטות תאי ה-T לפני הוספת הכמות המתאימה של לנטי-וירוס מאוגד כדי להתמיר 5 × 106 תאי T ב-MOI של 3.0. לדגור על התאים ב 37 ° C, 5% CO2.
הערה: מאגר lentivirus מהפקות מרובות לתוך צינור אחד לפני הפיכת תאי T. זה מבטיח שכל תאי T יקבלו את אותו הטיפול. בנוסף, lentivirus צריך להיות מופשר רק פעם אחת. - יום 2: בתנאי BSL-2+, הפשירו מספיק CRISPR lentivirus כדי להתמיר תאי T ב-MOI של 0.3. שברו בעדינות את רוזטות תאי ה-T והשהו מחדש את תאי ה-T לפני הוספת הכמות המתאימה של לנטיוירוס מאוגד כדי להתמיר 5 × 106 תאי T ב-MOI של 0.3. לדגור על התאים ב 37 ° C, 5% CO2.
הערה: מאגר lentivirus מהפקות מרובות לתוך צינור אחד לפני הפיכת תאי T. זה מבטיח שכל תאי T יקבלו את אותו הטיפול. - ימים 3-5: השהה מחדש את תאי ה-CART כדי לפרק רוזטות ולספור את התאים. הביאו את ריכוז התאים הסופי ל-1 × 106 תאים/מ"ל ב-TCM עם 1 מיקרוגרם/מ"ל פורומיצין. לדגור על התאים ב 37 ° C, 5% CO2.
- יום 6: השהה מחדש את תאי ה- CART כדי לפרק רוזטות, הסר את חרוזי CD3+/CD28+ באמצעות הפרדה מגנטית, וספור את תאי ה- CART. הוסף TCM עם 1 מיקרוגרם / מ"ל puromycin כדי להביא את ריכוז התא הסופי ל 1 × 106 תאים / מ"ל ולדגור את התאים ב 37 ° C, 5% CO2. קבע ביטוי CAR באמצעות ציטומטריית זרימה כמתואר בשלב 4.4.
7. שלב בחירת gRNA של מסך CRISPR (ימים 8 - 22)
- יום 8: שמרו דגימות לריצוף הדור הבא (NGS) והתחילו את התרבות המשותפת.
- שטפו את תאי ה-CART 3x ב-TCM על ידי צנטריפוגה של הדגימות ב-300 × גרם למשך 5 דקות ב-4°C, שאיפת הסופר-נטנט והשעיית הדגימות ב-20 מ"ל TCM.
- ספרו את תאי ה-CART.
- שמור דגימות עבור NGS על-ידי השלמת הפעולות הבאות:
- Aliquot מספיק תאי CART בצינור חרוטי של 50 מ"ל כדי לשמור על ייצוג gRNA של 500 תאים לכל gRNA (למשל, aliquot בערך 33 × 10 6תאי CART אם משתמשים בספריית CRISPR עם 65,383 gRNA). צנטריפוגה את הדגימה ב 300 × גרם במשך 5 דקות ב 4 ° C.
- להשעות מחדש את גלולת התא ב 1 מ"ל של PBS ולהעביר צינור מיקרוצנטריפוגה 2 מ"ל. לשטוף את הצינור החרוטי 50 מ"ל עם 1 מ"ל של PBS ולהעביר את הכביסה לצינור מיקרוצנטריפוגה 2 מ"ל.
- צנטריפוגה הדגימות ב 500 × גרם במשך 5 דקות ב 4 ° C. הסר את supernatant. אחסנו את כדורי התא בטמפרטורה של -20°C.
- התחל את התרבית המשותפת על ידי הוספת תאי CART לתאי CD19+ JeKo-1 ביחס של 1:1. לדוגמה, הוסף 80 × 106 תאי CART ל- 80 × 106 תאי JeKo-1. הבא את הנפח הכולל של התרבות המשותפת ל -160 מ"ל עם TCM. לאחסן את הדגימות באינקובטור ב 37 ° C, 5% CO2.
הערה: אנו ממליצים להתחיל עם לפחות 80 × 106 תאי CART כדי לשמור על ייצוג הספרייה וכדי שיהיו מספיק תאים כדי לשמר את הדגימות ביום ה-15 וביום ה-22.
- יום 10: כדי לעורר מחדש את תאי ה-CART, אספו את התרביות המשותפת לתוך צינורות חרוטיים של 50 מ"ל וצנטריפוגו את הדגימות ב-300 × גרם למשך 5 דקות ב-4 מעלות צלזיוס. השהה מחדש כל דגימה ב- 80 מ"ל של TCM, והוסף 80 × 106 תאי JeKo-1 ב- 80 מ"ל של TCM. לאחסן את הדגימות באינקובטור ב 37 ° C, 5% CO2.
- ימים 12 ו-14: גירוי מחדש של תאי CART כמו בשלב 7.2.
- יום 15: לשמר את הדגימות עבור NGS ולעורר מחדש את תאי CART.
- בודד תאי CART מתרבית משותפת על ידי ספירת התאים תחילה ולאחר מכן שילוב ערכת ברירה חיובית עבור תאי T CD4+ עם ערכת ברירה חיובית עבור תאי T CD8+ כדי לבודד תאי T מתרביות משותפות CART-JeKo-1. ספור את תאי ה- T.
- שמור את הדגימות עבור NGS על ידי aliquoting 33 × 106 CART תאים בצינור חרוטי 50 מ"ל צנטריפוגה הדגימה ב 300 × גרם במשך 5 דקות ב 4 ° C. להשעות מחדש את הדגימה ב 1 מ"ל של PBS ולהעביר אותו צינור מיקרוצנטריפוגה 2 מ"ל. לשטוף את הצינור החרוטי 50 מ"ל עם 1 מ"ל של PBS ולהעביר את הכביסה לצינור מיקרוצנטריפוגה 2 מ"ל. צנטריפוגה הדגימות ב 500 × גרם במשך 5 דקות ב 4 °C (75 °F); להסיר את supernatant; ולאחסן את כדורי התא ב -20 ° C.
- הגדר תרביות משותפות כדי להמשיך את מודל ה- in vitro לתשישות על ידי הוספת 80 × 106 תאי CART ל- 80 × 106 תאי JeKo-1. לאחר מכן, להביא את הנפח הכולל של התרבות המשותפת ל 160 מ"ל עם TCM. לאחסן את הדגימות באינקובטור ב 37 ° C, 5% CO2.
- ימים 17, 19 ו-21: גירוי מחדש של תאי CART כמו בשלב 7.2.
- יום 22: שמרו את הדגימות עבור NGS.
- בודדו תאי CART מהתרבית המשותפת על ידי ספירה ראשונית של התאים. לאחר מכן, שלב ערכת בחירה חיובית עבור תאי T CD4+ עם ערכת ברירה חיובית עבור תאי T CD8+ כדי לבודד תאי T מהתרביות המשותפות CART-JeKo-1. ספור את תאי ה- T.
- כדי לשמר דגימות עבור NGS, חזור על שלב 7.4.2.
8. הכנת DNA גנומי לריצוף הדור הבא
הערה: כדי לשמור על כיסוי הולם, יש לבודד DNA גנומי (gDNA) מלפחות 33 × 106 תאי CART. בנוסף, יש להכין את כל gDNA מ 33 × 106 תאי CART לריצוף.
- בודדו gDNA מכדורי תאים קפואים באמצעות ערכת בידוד gDNA.
- לטהר את gDNA באמצעות משקעים אתנול.
- Prechill 100% EtOH עד -20°C.
- Aliquot דילל DNA באופן שווה למספר צינורות מיקרוצנטריפוגות (200-250 μL). הוסף את הדברים הבאים לכל aliquot: שני כרכים של 100% EtOH (500 μL עבור 250 μL aliquot או 400 μL עבור 200 μL); 0.1 נפח של 3 M נתרן אצטט (25 μL עבור 250 μL aliquot או 20 μL עבור 200 μL); ו-1 μL של 20 מ"ג/מ"ל גליקוגן. לדגור את הדגימות לילה ב -20 מעלות צלזיוס.
- ביום השני, סובבו את הדגימות במהירות של 13,000 × גרם במשך 20 דקות כדי לשפוך את הדנ"א המואץ. הסר את supernatant. שטפו את הכדוריות עם 1 מ"ל של 70% EtOH על ידי סיבוב ב 7,500 × גרם במשך 10 דקות.
- הסר שאריות EtOH לחלוטין. הניחו לכדוריות להתייבש במשך 30-45 דקות.
- להשהות את הכדוריות ב 50 μL של מים סטריליים.
- הכינו gDNA עבור NGS.
הערה: פריימרים של NGS עבור ספריית CRISPR זו המגבירים gRNA עם רצפי מתאם Illumina תוכננו בפרסום קודם27. שימוש בפריימר הפוך שונה לכל דגימה יבצע ברקוד לכל דגימה ויאפשר איגום דגימות במהלך הריצוף.- הכן את הדגימות עבור PCR עם תערובת התגובה הבאה (נפח כולל = 50 μL): תערובת מאסטר PCR באיכות גבוהה, 2x: 25 μL; DNA תבנית מאוגמת, 0.5 מיקרוגרם: 1 μL; פריימר קדמי לספריית NGS, 10 מיקרומטר: 1.25 מיקרוליטר; פריימר הפוך לספריית NGS, 10 מיקרומטר: 1.25 מיקרוליטר; מים ללא DNase: 21.5 μL.
- בצע PCR עם תנאי הרכיבה הבאים: מחזור 1: דנטורציה ב 98 ° C למשך 3 דקות; מחזורים 2-23: Denature 98 ° C עבור 10 שניות, anneal ב 63 ° C במשך 10 שניות, להאריך ב 72 ° C במשך 25 שניות; מחזור 24: הארכה ב-72°C למשך 2 דקות.
- אגרו את תגובות ה-PCR עבור כל דגימה וטיהרו את מוצר ה-PCR באמצעות ערכת טיהור PCR בהתאם להוראות היצרן.
- יש להריץ 3 מיקרוגרם של מוצר PCR עבור כל דגימה על ג'ל אגרוז 2% (w/v).
- הסר את מוצר ה- PCR (~ 260-270 bp) מהג'ל על ידי חילוץ ה- DNA מהג'ל באמצעות ערכת מיצוי ג'ל בהתאם להוראות היצרן. אחסן את הדגימות שחולצו ב -20 ° C.
- בצע ריצוף של הדור הבא (NGS) עם הדגימות. יש לשאוף ליותר מ-500 קריאות לכל gRNA בספרייה.
9. ניתוח תוצאות הרצף
- כדי לנתח NGS, השתמש בפלטפורמת MAGeCK-VISPR28. השתמש בהגדרות הסטנדרטיות עבור אלגוריתם הערכת הסבירות המרבית (MAGeCK-MLE). כדי להכין את קבצי הפלט לניתוח:
- הפעל את FASTQC כדי להעריך את איכות קובץ הרצף31.
- מזג קובצי רצף משויכים עבור כל דוגמה עם bbmerge32.
- נתח את הדגימות בפלטפורמת MAGeCK-VISPR באמצעות אלגוריתם MAGeCK-MLE. עבור ניתוח MAGeCK-MLE, נרמלו את כל ספירות ה-gRNA לרשימה של 1,000 gRNA שאינם ממוקדים.
- הערך את איכות קובץ הרצף על-ידי הערכת תוכן GC, איכות בסיס, קריאות רצף וקריאות ממופות.
- הערך את ההצלחה של שלב הלחץ הסלקטיבי של מסך הקריספר על ידי הערכת שינויים באינדקס ג'יני, אשכולות ניתוח רכיבים עיקריים (PCA) ומסלולים הקשורים לרשימת הגנים שנבחרו באופן שלילי32.
- חקור את הלהיטים המובילים על ידי ביצוע חיפושי ספרות וביצוע ניתוח מסלולים.
תוצאות
כדי לחקור גנים ומסלולים שניתן לערוך כדי לשפר את פעילות תאי ה-CART באופן בלתי מוטה, תכננו מסך נוקאאוט CRISPR רחב גנום במבחנה (איור 1). למסך זה שני שלבים: שלב ייצור תאי CART ושלב לחץ סלקטיבי. בשלב ייצור תאי CART, לפחות 110 ×10 6 תאי T מבודדים תחילה מ- PBMCs תורמים ברי...
Discussion
עריכת גנים הפכה לכלי רב עוצמה הן בהבנת מנגנוני ההתנגדות לטיפולים והן בעיצוב טיפולים חדשניים בתאי CART לשיפור תוחלת החיים והפעילות של תאי CART 16,17,26. בעוד שכמה אסטרטגיות לעריכת גנים הראו שיפורים בפעילות תאי CART הן במודלים פרה...
Disclosures
SSK היא ממציאה של פטנטים בתחום האימונותרפיה CAR המורשים לנוברטיס (באמצעות הסכם בין מאיו קליניק, אוניברסיטת פנסילבניה ונוברטיס), הומאניגן (באמצעות מאיו קליניק), מטאפורג' (באמצעות מאיו קליניק), מוסטנגביו (באמצעות מאיו קליניק), וטיפולים כימליים (באמצעות מאיו קליניק). CS, CMR ו-SSK הם ממציאים של פטנטים בעלי רישיון ל-Immix Biopharma. SSK מקבלת מימון מחקר מ-Kite, Gilead, Juno, BMS, Novartis, Humanigen, MorphoSys, Tolero, Sunesis/Viracta, LifEngine Animal Health Laboratories Inc. ו-Lentigen. SSK השתתפה בפגישות ייעוץ עם Kite/Gilead, Calibr, Luminary Therapeutics, Humanigen, Juno/BMS, Capstan Bio ו-Novartis. SSK כיהנה במועצת הבטיחות והניטור של נתונים עם Humanigen ו-Carisma. SSK פיטרה יועץ עבור Torque, Calibr, Novartis, Capstan Bio, BMS, Carisma ו- Humanigen. CMS ו- SSK הם ממציאי קניין רוחני שנבע מפרוטוקול זה.
Acknowledgements
מחקר זה מומן בחלקו על ידי מרכז מאיו קליניק לרפואה אישית (SSK), מרכז מאיו קליניק מקיף לסרטן (SSK), מרכז מאיו קליניק לביותרפיה רגנרטיבית (SSK), המכונים הלאומיים לבריאות K12CA090628 (SSK) ו- R37CA266344-01 (SSK), CA201127 המענקים של משרד ההגנה (SSK), קרן פרדולין (SSK) ושותפות מינסוטה לביוטכנולוגיה וגנומיקה רפואית (SSK). CMS נתמך על ידי Mayo Clinic Graduate School of Biomedical Sciences. סכימת מסך CRISPR (איור 1) נוצרה באמצעות BioRender.com (Siegler, L. (2022) https://BioRender.com/k71r054).
Materials
| Name | Company | Catalog Number | Comments |
| 293T cells | ATCC | CRL-3216 | Cells used for lentivirus production |
| Biotin ProteinL Antibody | GenScript | M00097 | anti-kappa chain antibody for CAR detection |
| Bovine Serum Albumin | Millipore Sigma | A7906 | |
| Carbenicillin disodium salt | Millipore Sigma | C1389-1G | Carbenicillin antibiotic |
| CD4 Isolation Beads | Miltenyi Biotec | 130-045-101 | |
| CD8 Isolation Beads | Miltenyi Biotec | 130-045-201 | |
| CTS (Cell Therapy Systems) Dynabeads CD3/CD28 | Gibco | 40203D | |
| Cytoflex | Beckman Coulter | NC2279958 | |
| DNase-Free Water | Invitrogen | AM9937 | |
| Dulbecco's modified eagle's medium (DMEM) | Corning | 10-017-CV | |
| Dulbecco's Phosphate-Buffered Saline | Gibco | 14190-144 | |
| EasySep Human T Cell Isolation Kit | STEMCELL Technologies | 17951RF | Negative isolation kit |
| Endura Electrocompetent Cells | Biosearch Technologies | 60242-1 | Electrocompetent cells with recovery medium |
| Ethanol | Millipore Sigma | E7023 | |
| Fetal bovine serum (FBS) | Corning | 35-010-CV | |
| GeCKO v2 CRISPR Knockout Pooled Library A | AddGene | 1000000048 | CRISPR library plasmid |
| Gene Pulser II | Bio-Rad | 165-2105 | Electroporator |
| Glycogen | Millipore Sigma | 10901393001 | |
| JeKo-1 | ATCC | CRL-3006 | CD19+ target cells |
| Lipofectamine 3000 Transfection Reagent | ThermoFisher Scientific | L3000075 | Transfection reagent kit with a transfection reagent (Lipofectamine 3000 Reagent) and a neutral co-lipid reagent (p3000) |
| LIVE/DEAD Aqua | Invitrogen | L34966 | |
| Lymphoprep | STEMCELL Technologies | 7851 | Density gradient medium |
| Machery-Nagel NucleoBond Xtra Maxi Kits | ThermoFisher Scientific | 12748412 | Maxi-prep kit |
| NEBNext High-Fidelity 2X PCR MasterMix | New England BioLabs | M0541S | High fidelity PCR mastermix |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985-070 | Reduced serum medium |
| pCMVR8.74 | AddGene | 22036 | Lentiviral packaging plasmid |
| Pennicillin-streptomycin-glutamine (100X) | Life Technologies | 10378-016 | |
| pMD2.G | AddGene | 12259 | VSV-G envelope expressing plasmid |
| Pooled Human AB Serum | Innovative Research | ISERABHI | |
| Puromycin | Millipore Sigma | P8833 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | Gek extraction kit |
| Qucik-DNA Midiprep Plus Kit | Zymo Research | D4075 | Kit used to isolate gDNA |
| RoboSep-S | STEMCELL Technologies | 21000 | Automated cell separator |
| Roswell Park Memorial Institute 1640 Medium (RPMI) | Gibco | 21870092 | |
| SepMate-50 | STEMCELL Technologies | 85450 | Density gradient separation tube |
| Sodium Acetate | Invitrogen | AM9740 | |
| Sodium Azide | Fisher Scientific | 71448-16 | |
| Streptavidin Antibody (PE) | BioLegend | 405203 | Secondary antibody used for CAR detection |
| T100 Thermal Cycler | Bio-Rad | 1861096 | |
| Ultracentrifuge (Optima XPN-80) | BeckmanCoulter | A99839 | |
| Vacuum Filter Systems, 0.22 µm | ThermoFisher Scientific | 567-0020 | |
| Vacuum Filter Systems, 0.45 µm | ThermoFisher Scientific | 165-0045 | |
| X-VIVO 15 Serum-Free Hematopoietic Cell Medium | Lonza | 04-418Q | Hematopoietic cell medium |
References
- Neelapu, S. S., et al. 5-year follow-up supports curative potential of axicabtagene ciloleucel in refractory large B-cell lymphoma (zuma-1). Blood. 141 (19), 2307-2315 (2023).
- Neelapu, S. S., et al. Axicabtagene ciloleucel CAR T-cell therapy in refractory large B-cell lymphoma. N Engl J Med. 377 (26), 2531-2544 (2017).
- Locke, F. L., et al. Long-term safety and activity of axicabtagene ciloleucel in refractory large B-cell lymphoma (zuma-1): A single-arm, multicentre, phase 1-2 trial. Lancet Oncol. 20 (1), 31-42 (2019).
- Maude, S. L., et al. Tisagenlecleucel in children and young adults with B-cell lymphoblastic leukemia. N Engl J Med. 378 (5), 439-448 (2018).
- Schuster, S. J., et al. Tisagenlecleucel in adult relapsed or refractory diffuse large B-cell lymphoma. N Engl J Med. 380 (1), 45-56 (2018).
- Bai, Z., et al. Single-cell antigen-specific landscape of CAR T infusion product identifies determinants of CD19-positive relapse in patients with all. Sci Adv. 8 (23), eabj2820 (2022).
- Fraietta, J. A., et al. Determinants of response and resistance to CD19 chimeric antigen receptor (CAR) T cell therapy of chronic lymphocytic leukemia. Nat Med. 24, 563-571 (2018).
- Gennert, D. G., et al. Dynamic chromatin regulatory landscape of human CAR T cell exhaustion. Proc Natl Acad Sci USA. 118 (30), e2104758118 (2021).
- Deng, Q., et al. Characteristics of anti-CD19 CAR T cell infusion products associated with efficacy and toxicity in patients with large B cell lymphomas. Nat Med. 26 (12), 1878-1887 (2020).
- Sterner, R. M., Cox, M. J., Sakemura, R., Kenderian, S. S. Using CRISPR/CAS9 to knock out GM-CSF in CAR-T cells. J Vis Exp. (149), e59629 (2019).
- Sterner, R. M., et al. GM-CSF inhibition reduces cytokine release syndrome and neuroinflammation but enhances CAR-T cell function in xenografts. Blood. 133 (7), 697-709 (2019).
- Cox, M. J., et al. GM-CSF disruption in CART cells modulates T cell activation and enhances CART cell anti-tumor activity. Leukemia. 36 (6), 1635-1645 (2022).
- Ottaviano, G., et al. Phase 1 clinical trial of CRISPR-engineered CAR19 universal T cells for treatment of children with refractory B cell leukemia. Sci Transl Med. 14 (668), eabq3010 (2022).
- Joung, J., et al. Genome-scale CRISPR-CAS9 knockout and transcriptional activation screening. Nat Protoc. 12, 828-863 (2017).
- Mccutcheon, S. R., et al. Transcriptional and epigenetic regulators of human CD8+ T cell function identified through orthogonal CRISPR screens. Nat Genet. 55, 2211-2223 (2023).
- Zhu, W., Kelly, C., Dagur, P., Dunbar, C. E., Cordes, S. CRISPR activation screen to optimize chimeric antigen receptor (CAR) T cell immunophenotype. Blood. 142 (Supplement 1), 4820-4820 (2023).
- Wang, D., et al. CRISPR screening of CAR T cells and cancer stem cells reveals critical dependencies for cell-based therapies. Cancer Discov. 11 (5), 1192-1211 (2021).
- Shifrut, E., et al. Genome-wide CRISPR screens in primary human T cells reveal key regulators of immune function. Cell. 175 (7), 1958-1971.e15 (2018).
- Belk, J. A., et al. Genome-wide crispr screens of T cell exhaustion identify chromatin remodeling factors that limit T cell persistence. Cancer Cell. 40 (7), 768-786.e7 (2022).
- Jiang, P., et al. Single-cell ATAC-seq maps the comprehensive and dynamic chromatin accessibility landscape of CAR-T cell dysfunction. Leukemia. 36 (11), 2656-2668 (2022).
- Rossi, J., et al. Preinfusion polyfunctional anti-CD19 chimeric antigen receptor T cells are associated with clinical outcomes in NHL. Blood. 132 (8), 804-814 (2018).
- Beider, K., et al. Molecular and functional signatures associated with CAR T cell exhaustion and impaired clinical response in patients with B cell malignancies. Cells. 11 (7), 1140 (2022).
- Wherry, E. J. T. cell exhaustion. Nat Immunol. 12, 492-499 (2011).
- Prinzing, B., et al. Deleting DNMT3A in CAR T cells prevents exhaustion and enhances antitumor activity. Sci Transl Med. 13, eabh0272 (2021).
- Lynn, R. C., et al. C-jun overexpression in CAR T cells induces exhaustion resistance. Nature. 576, 293-300 (2019).
- Tang, N., et al. TGF-beta inhibition via CRISPR promotes the long-term efficacy of CAR T cells against solid tumors. JCI Insight. 5 (4), e133977 (2020).
- Joung, J., et al. Genome-scale CRISPR-CAS9 knockout and transcriptional activation screening. Nat Protoc. 12 (4), 828-863 (2017).
- Li, W., et al. Quality control, modeling, and visualization of CRISPR screens with MAGeCK-VISPR. Genome Biol. 16, 281 (2015).
- Sakemura, R., et al. Targeting cancer-associated fibroblasts in the bone marrow prevents resistance to CART-cell therapy in multiple myeloma. Blood. 139 (26), 3708-3721 (2022).
- Stewart, C. M., et al. IL-4 drives exhaustion of CD8+ CART cells. Nat Commun. 15 (1), 7921 (2024).
- . FastQC: A quality control tool for high throughput sequence data Available from: https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010)
- . Bbmerge Available from: https://github.com/BioInfoTools/BBMap/blob/master/sh/bbmerge.sh (2019)
- Pattanayak, V., et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed CAS9 nuclease specificity. Nat Biotechnol. 31 (9), 839-843 (2013).
- Doench, J. G., et al. Rational design of highly active sgRNAs for CRISPR-CAS9-mediated gene inactivation. Nat Biotechnol. 32 (12), 1262-1267 (2014).
- Dong, M. B., et al. Systematic immunotherapy target discovery using genome-scale in vivo CRISPR screens in CD8 T cells. Cell. 178 (5), 1189-1204.e23 (2019).
- Zhou, P., et al. Single-cell CRISPR screens in vivo map T cell fate regulomes in cancer. Nature. 624 (7990), 154-163 (2023).
- Sutra Del Galy, A., et al. In vivo genome-wide CRISPR screens identify SOCS1 as intrinsic checkpoint of CD4+ TH1 cell response. Sci Immunol. 6 (66), eabe8219 (2021).
- Ramos, A., et al. Leukemia-intrinsic determinants of CAR-T response revealed by iterative in vivo genome-wide CRISPR screening. Nat Commun. 14 (1), 8048 (2023).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved