É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Realizando uma triagem nocaute CRISPR em todo o genoma in vitro em células T receptoras de antígeno quimérico
Neste Artigo
Resumo
O artigo descreve um protocolo para a aplicação de um modelo in vitro de exaustão para completar uma triagem de nocaute CRISPR em todo o genoma em células T receptoras de antígeno quimérico de doadores saudáveis.
Resumo
A terapia celular com receptor de antígeno quimérico T (CART) é uma forma inovadora de imunoterapia direcionada que revolucionou o tratamento do câncer. No entanto, a resposta duradoura continua a ser limitada. Estudos recentes mostraram que o cenário epigenético dos produtos celulares CART pré-infusão pode influenciar a resposta à terapia, e a edição de genes foi proposta como uma solução. No entanto, mais trabalho precisa ser feito para determinar a estratégia ideal de edição de genes. As telas CRISPR de todo o genoma tornaram-se ferramentas populares para investigar mecanismos de resistência e otimizar estratégias de edição de genes. No entanto, sua aplicação às células primárias apresenta muitos desafios. Aqui, descrevemos um método para completar uma tela de nocaute CRISPR em todo o genoma em células CART de doadores saudáveis. Como um modelo de prova de conceito, projetamos este método para investigar o desenvolvimento de exaustão em células CART direcionadas ao antígeno CD19. No entanto, acreditamos que essa abordagem pode ser usada para abordar uma variedade de mecanismos de resistência à terapia em diferentes construções de CAR e modelos de tumores.
Introdução
A terapia com células T do receptor de antígeno quimérico (CART) mostrou um sucesso impressionante no tratamento de malignidades de células B; no entanto, a resposta durável é limitada a 30-40%1,2,3,4,5. Embora os pesquisadores tenham desenvolvido e testado várias abordagens para abordar os mecanismos de resistência à terapia com células CART, incluindo a otimização do design do CAR, edição de genes e terapias combinadas, o desenvolvimento da resistência permanece amplamente desconhecido. Recentemente, tem havido evidências crescentes de que o perfil de expressão gênica basal de produtos de células CART pré-infusão é um determinante importante da toxicidade e eficácia da terapia CART 6,7,8,9. Como tal, a edição de genes durante a produção de células CART tornou-se uma abordagem popular para melhorar o sucesso da terapia celular CART.
Por exemplo, nosso laboratório mostrou anteriormente que o uso da tecnologia Cas9 de repetições palindrômicas curtas agrupadas regularmente interespaçadas (CRISPR) para eliminar o fator estimulador de colônias de granulócitos-macrófagos do gene (GM-CSF) melhora a atividade das células CART e reduz os sinais de toxicidade associada ao CART 10,11,12. Além disso, a terapia celular projetada por CRISPR foi testada em ensaios clínicos e mostrou eficácia e segurança13. Juntos, isso indica que a edição de genes com a tecnologia CRISPR pode não apenas melhorar nossa compreensão da biologia celular CART, mas também gerar produtos celulares CART traduzíveis.
As telas nocaute CRISPR em todo o genoma tornaram-se ferramentas cada vez mais populares na pesquisa da biologia do câncer para entender os mecanismos de resistência à terapêutica. Nessa técnica, dezenas de milhares de RNAs guia (gRNAs) são entregues a pools de células para estimular a entrada de um gRNA por célula14. Em seguida, as células passam por condições indutoras de pressão, onde as células transduzidas com gRNAs direcionados a genes essenciais morrem e as células transduzidas com gRNAs direcionados a genes inibitórios sobrevivem e proliferam. Usando o sequenciamento de última geração, podemos determinar como a representação do gRNA muda ao longo da tela CRISPR14.
No entanto, a escala e o tempo de seleção necessários para perturbações gênicas em todo o genoma podem ser difíceis de realizar em células primárias, como as células CART. Como tal, os grupos utilizaram telas CRISPR direcionadas para entender melhor os mecanismos de resistência terapêutica 15,16. As telas direcionadas geralmente são mais fáceis de concluir em células primárias porque têm bibliotecas menores que exigem menos células para obter uma representação adequada da biblioteca. Embora esses estudos tenham melhorado nossa compreensão dos mecanismos de resistência à terapia com células CART, as telas direcionadas introduzem um viés devido à seleção manual de alvos genéticos. Este artigo procura descrever um método para completar uma triagem de nocaute CRISPR em todo o genoma in vitro em células CART de doadores saudáveis. Como tal, essa abordagem de alto rendimento permite a identificação eficiente e imparcial das principais vias e genes que podem ser editados para melhorar as respostas terapêuticas 17,18,19.
Em particular, o protocolo descrito neste artigo foi projetado para aumentar a compreensão do campo sobre a exaustão de células CART, completando a tela de nocaute CRISPR em todo o genoma com um modelo in vitro de exaustão. A exaustão é um destino disfuncional das células CART que tem sido implicado na não resposta à terapia celular CART 20,21,22. Sabe-se que esse destino celular é regulado epigeneticamente e é caracterizado por uma diminuição na proliferação de células CART, uma diminuição na produção de citocinas efetoras e um aumento na expressão de receptores inibitórios23. Na literatura anterior, a edição de genes foi capaz de prevenir o desenvolvimento de exaustão pela regulação positiva ou negativa de genes-chave24 , 25 , 26 . Dada a diminuição da capacidade proliferativa à medida que as células CART se esgotam e a evidência de que a edição de genes pode prevenir sua ocorrência, modelamos nossa tela de nocaute CRISPR em todo o genoma in vitro nesse destino celular. No entanto, este protocolo pode ser alterado no futuro para explorar outros mecanismos de resistência à terapia com células CART.
Protocolo
É importante ressaltar que o protocolo descrito abaixo segue as diretrizes e recebeu a aprovação do Conselho de Revisão Institucional da Mayo Clinic (IRB 18-005745) e do Comitê Institucional de Biossegurança (IBC HIP00000252.43). Todo o trabalho de cultura celular, incluindo a produção de lentivírus, deve ser realizado em uma capa de cultura celular com equipamento de proteção individual adequado. Em particular, o trabalho lentiviral deve ser conduzido sob precauções de nível de biossegurança 2 (BSL-2), incluindo o uso de alvejante a 10% para desinfetar itens antes do descarte.
1. Amplificação da Biblioteca CRISPR
- Amplifique a biblioteca CRISPR de acordo com as recomendações do fabricante. Resumidamente, eletropore a biblioteca CRISPR em quatro alíquotas de células eletrocompetentes, completando o seguinte:
- Adicione 2 μL de plasmídeo da biblioteca CRISPR de 50 ng/μL a 25 μL de células eletrocompetentes em uma cubeta de 1,0 mm.
- Eletropore as amostras usando as seguintes configurações: 10 μF, 600 Ohms, 1.800 volts e uma constante de tempo entre 3.5 ms e 4.5 ms.
- Recupere em 975 μL de mídia de recuperação e transfira para um tubo com 1 mL adicional de mídia de recuperação.
- Girar todas as alíquotas a 250 rpm durante 1 h a 37 °C.
- Placa as transformações:
- Agrupe todas as alíquotas de células eletroporadas e misture bem.
- Plaquear todas as células eletroporadas espalhando 400 μL da mistura de transformação em placas pré-aquecidas de caldo Luria (LB) de 10 cm com 100 μg/mL de carbenicilina. Colocar a placa numa incubadora a 32 °C durante 14 h.
- Colha as colônias:
- Pipetar 1 ml de LB + 100 μg/ml de carbenicilina em cada placa. Raspe as colônias de cada placa e colete-as em um tubo cônico de 50 mL.
- Adicione mais 1 mL de meio LB + 100 μg/mL de carbenicilina a cada placa para lavar e garantir a recuperação completa.
- Execute uma maxi-prep seguindo as instruções de um kit de maxi-prep.
- Agrupe o DNA do plasmídeo resultante.
2. Sequenciamento de última geração para verificar a representação de gRNA de linha de base na biblioteca CRISPR
NOTA: Os primers NGS para esta biblioteca CRISPR foram projetados em uma publicação anterior27. O uso de um primer reverso diferente para cada amostra irá codificar cada amostra e permitir o agrupamento de amostras durante o sequenciamento.
- Preparar as amostras para PCR com a seguinte mistura de reacção (volume total = 50 μL): master mix de PCR de alta fidelidade, 2x: 25 μL; DNA molde combinado, 0,5 μg: 1 μL; Primário direto da biblioteca NGS, 10 μM: 1,25 μL; Primer reverso da biblioteca NGS, 10 μM: 1,25 μL; Água livre de DNase: 21,5 μL.
- Realizar PCR com as seguintes condições de ciclagem: ciclo 1: desnaturar a 98 °C durante 3 min; ciclos 2-23: desnaturar a 98 °C por 10 s, recozimento a 63 °C por 10 s, estender a 72 °C por 25 s; ciclo 24: estender a 72 °C por 2 min.
- Purifique a reação de PCR agrupando as reações de PCR para cada amostra e purificando o produto de PCR com um kit de purificação de PCR de acordo com as instruções do fabricante.
- Execute 3 μg do produto de PCR para cada amostra em um gel de agarose a 2% (p / v).
- Remova o produto de PCR (~ 260-270 bp) do gel extraindo o DNA do gel usando um kit de extração de gel de acordo com as instruções do fabricante. Apontar para pelo menos 500 ng de DNA.
- Armazenar as amostras extraídas a -20 °C.
- Realize o sequenciamento de próxima geração (NGS) com as amostras e aponte para 100 leituras por gRNA27.
- Analise os resultados do sequenciamento com o pipeline MAGeCK-VISPR e certifique-se de que mais de 99,5% dos gRNAs na biblioteca estejam representados28.
3. Produção de biblioteca CRISPR e lentivírus de expressão CAR
- Prepare o lentivírus conforme descrito anteriormente 10,29.
- Dia 1: Comece com frascos T175 de 80-90% de células 293T confluentes que cresceram em meio D10 composto por 10% de soro fetal bovino (FBS) (v/v), 1% de penicilina-estreptomicina-glutamina (PSG) (v/v) e meio de águia modificado de Dulbecco. Antes de começar, aqueça os reagentes de transfecção e o DNA à temperatura ambiente e quantifique o DNA.
- Para cada balão de 293T, rotular dois tubos cónicos de 50 ml A e B. Adicione 4,5 mL de meio sérico reduzido ao tubo A e ao tubo B.
- Em um tubo de microcentrífuga estéril de 1,5 mL, misture o seguinte DNA e deixe descansar por 1 min: 18 μg de pCMVR8.74 (gag, pol, tat e rev), 7 μg de pMD2.G (VSV-G) e 15 μg de vetor de expressão lentiviral (CRISPR biblioteca A ou vetores CAR19-28ζ).
- Adicione 129 μL de reagente de transfecção ao tubo A. Misture bem o tubo A por vórtice de dedo.
- Adicione a mistura de DNA ao tubo B. Misture bem o tubo B por vórtice de dedo.
- Adicione 111 μL de reagente colipídico neutro ao tubo B. Misture bem o tubo B por vórtice de dedo.
- Adicione o conteúdo do tubo A ao tubo B e misture bem por vórtice de dedo. Incubar o tubo B por 30 min em temperatura ambiente.
- No final do período de incubação de 30 minutos, prepare as células 293T aspirando o meio existente. Adicione 16 mL de mídia R10 composta por 10% de FBS (v/v), 1% de PSG (v/v) e Roswell Park Memorial Institute (RPMI) 1640.
- Após o período de incubação de 30 minutos, adicione o conteúdo do tubo B (aproximadamente 9 mL) gota a gota ao frasco de células 293T. Colocar o balão e rodá-lo suavemente antes de o incubar a 37 °C, 5% de CO2.
- No dia 2 (aproximadamente 24 h após a transfecção), colete lentivírus da seguinte forma:
NOTA: Pré-resfrie todos os equipamentos e materiais e execute o seguinte no gelo para manter o sobrenadante viral frio.- Transfira o sobrenadante dos frascos transfectados de 293T para tubos cônicos pré-resfriados de 50 mL.
- Adicione 30 mL de meio R10 pré-aquecido aos frascos 293T.
- Sedimentar os detritos celulares centrifugando os tubos cónicos de 50 ml a 900 × g durante 10 min a 4 °C em rotores fechados.
- Após centrifugação, filtrar o sobrenadante com um filtro de 0,45 μm. Centrifugar o filtrado numa ultracentrífuga a 112,700 × g a 4 °C durante 2 h.
- Após a centrifugação de 2 h, aspirar o sobrenadante até restarem aproximadamente 5 ml. Ressuspenda as partículas virais pipetando suavemente para cima e para baixo 10x. Conservar a suspensão viral a 4 °C durante a noite.
- No dia 3, colete e armazene lentivírus da seguinte forma:
- Transfira o sobrenadante dos frascos transfectados de 293T para tubos cônicos pré-resfriados de 50 mL.
- Adicione 30 mL de alvejante a 10% aos frascos 293T.
- Pulverizar os detritos celulares no sobrenadante centrifugando os tubos cónicos de 50 ml a 900 × g durante 10 min a 4 °C em rotores fechados.
- Após centrifugação, filtrar o sobrenadante com um filtro de 0,45 μm. Centrifugar o filtrado numa ultracentrífuga a 112,700 × g a 4 °C durante 2 h.
- Após a centrífuga de 2 h, aspire o sobrenadante até que restem aproximadamente 2 mL e ressuspenda pipetando suavemente para cima e para baixo 10x. Armazenar o sobrenadante viral em alíquotas de 300-500 μL e conservar a -80 °C.
4. Cálculo do título viral para lentivírus que expressa CAR
NOTA: O lentivírus CAR foi titulado conforme descrito anteriormente 10,12.
- No dia 1, ative e plaquee as células T.
- Isole 1 × 106 células T de PBMCs de doadores saudáveis usando um kit de seleção negativa.
- Conte as células T.
- Lave grânulos CD3 + / CD28 + suficientes para estimular as células T em uma proporção de 3: 1 grânulos: célula. Por exemplo, para estimular 1 × 106 células T, lave 3 × 106 contas. Para fazer isso, lave as contas completando os 3x a seguir:
- Use a separação magnética para remover os grânulos da suspensão.
- Remova o sobrenadante.
- Adicione 1000 μL de meio de células T (TCM) composto por 10% de soro humano (v/v), 1% de PSG (v/v) e meio de células hematopoiéticas.
NOTA: O TCM deve ser esterilizado antes do uso, filtrando com um filtro de vácuo estéril de 0.45 μm e, em seguida, com um filtro de vácuo estéril de 0.22 μm. - Adicione grânulos à suspensão de células T na proporção de 3:1 cordão:célula.
- Ressuspenda as células T a 1 × 106 células/mL em TCM.
- Adicione 100 μL de células T ativadas a 10 poços de uma placa de 96 poços. Incubar a placa a 37 °C, 5% CO2.
- No dia 2, adicione uma diluição de lentivírus CAR criando uma placa de diluição da seguinte forma:
- Adicione 100 μL de TCM a sete poços de uma placa de 96 poços.
- Ao primeiro poço, adicione 50 μL do vírus de interesse; misture bem.
- Diluir ainda mais o vírus em série adicionando 50 μL do poço 1 ao poço 2; misture bem.
- Adicione 50 μL do poço 2 ao poço 3 e continue este padrão até o poço 7.
- Depois de misturar o poço 7, remova 50 μL. Certifique-se de que todos os poços tenham um volume total de 100 μL.
- Adicionar 50 μL de cada alvéolo da placa de diluição aos primeiros sete alvéolos da placa de células T a partir do dia 1. Misture bem.
- Aos últimos três poços da placa de células T, adicione 50 μL de TCM; misture bem. Armazenar a placa numa incubadora a 37 °C, 5% CO2.
- No dia 3, alimente as células adicionando 100 μL de TCM a todos os poços da placa de células T; misture bem.
- No dia 4, realize citometria de fluxo para detectar a porcentagem de células que expressam CAR com um anticorpo de cadeia anti-kappa, um marcador substituto para a expressão de CAR, completando o seguinte:
- Gire a placa de células T a 1.000 × g por 3 min e transvase.
- Lave a placa 3x com tampão de anticorpo anti-cadeia kappa (composto por 4% de albumina de soro bovino em PBS (p/v)) adicionando 200 μL de tampão de anticorpo anti-cadeia kappa, girando a placa a 1.000 × g por 3 min e decantando.
- Adicione um anticorpo anti-cadeia kappa de biotina a cada poço. Incubar durante 45 min num balancim a 4 °C.
- Lave a placa 3x com tampão de anticorpo de cadeia kappa como na etapa 4.4.2.
- Cor das células para citometria de fluxo com um anticorpo estreptavidina conjugado com fluorocromo e um anticorpo vivo/morto. Incube as amostras por 15 min em temperatura ambiente no escuro.
- Lave as amostras uma vez em tampão de fluxo (composto por 2% de FBS (v/v) e 1% de azida de sódio (v/v) em PBS) adicionando 200 μL de tampão de fluxo, girando a placa a 1.000 × g por 3 min e decantando.
- Ressuspender em 100 μl de tampão de fluxo e passar no citômetro.
- Execute o bloqueio negativo com os três poços não transduzidos.
5. Cálculo do título viral para lentivírus da biblioteca CRISPR
NOTA: Este protocolo descreve um número mínimo de células T para titular o lentivírus da biblioteca CRISPR, mas pode ser ampliado para acomodar mais células T e, portanto, maiores volumes de vírus.
- No Dia 0, ative as células T adicionando 18 ×10 6 grânulos CD3 + / CD28 + (proporção 3: 1 grânulo: célula) a 6 × 106 células T e trazendo o volume final para 6 mL com TCM. Alíquota 0,5 mL da suspensão de células T em 12 poços de uma placa de 48 poços. Armazenar em uma incubadora a 37 °C, 5% CO2.
- Vinte e quatro horas depois, no Dia 1, adicione o vírus CAR em uma multiplicidade de infecção (MOI) de 3 a cada poço. Misture bem pipetando para cima e para baixo várias vezes. Armazenar em uma incubadora a 37 °C, 5% CO2.
- Quarenta e oito horas depois, no Dia 2, adicione uma série de volumes de vírus da biblioteca CRISPR às células CART ativadas, adicionando um dos seguintes volumes de vírus CRISPR a cada poço (0 μL, 0 μL, 6,25 μL, 6,25 μL, 12,5 μL, 12,5 μL, 25 μL, 25 μL, 50 μL, 50 μL, 100 μL ou 100 μL). Misture bem pipetando para cima e para baixo várias vezes. Armazenar em uma incubadora a 37 °C, 5% CO2.
- No dia 3, adicione 1 μg de puromicina por mL de meio à metade dos poços, incluindo um poço que recebeu cada um dos seguintes volumes de vírus CRISPR (0 μL, 6,25 μL, 12,5 μL, 25 μL, 50 μL e 100 μL). Os outros poços não serão tratados com puromicina.
- Dos dias 4 a 8, conte as células T diariamente e ajuste a densidade celular final para 1 × 106 células/mL com 1 μg/mL de puromicina em MTC para os poços de tratamento com puromicina e com MTC para os poços não tratados.
- No dia 8, conte o número de células vivas por poço e calcule as unidades de transdução/mL de sobrenadante viral usando a seguinte equação:
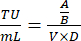
Onde A: Contagem de células vivas em poço transduzido viralmente; B: Contagem de células vivas em poço de controle sem seleção de antibióticos; V: Volume de vírus usado para infecção em cada poço; D: Dobra de diluição. - Usando o poço TU / mL calculado a partir de um poço mostrando aproximadamente 20% de eficiência de transdução, calcule o volume de vírus necessário para transduzir 1 × 106 células em um MOI de 0,3 usando a seguinte fórmula:
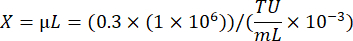
6. Fase de produção de células CART da tela CRISPR (dias 0 a 8)
NOTA: O protocolo a seguir descreve a conclusão da tela CRISPR em uma réplica biológica. No entanto, este protocolo foi previamente validado com três réplicas biológicas e recomendamos o uso de pelo menos três réplicas biológicas para outros que completem este protocolo30.
- Dia 0: Isole e estimule as células T do sangue de doadores saudáveis, conforme descrito anteriormente10.
- Isole as células mononucleares do sangue periférico (PBMCs) do sangue de um doador saudável não identificado.
- Adicione 15 mL de um meio de gradiente de densidade a um tubo de separação de gradiente de densidade.
- Adicione sangue diluído 1:2 na parte superior do tubo de separação do gradiente de densidade. Centrifugue a 1.200 × g por 10 min em temperatura ambiente.
NOTA: O sangue deve ser diluído com tampão de isolamento (2% FBS (v/v) em solução salina tamponada com fosfato [PBS]). - Transforme a camada superior em um novo tubo cônico de 50 mL e dobre o volume com tampão de isolamento. Centrifugar a amostra a 300 × g durante 8 min a 4 °C. Aspire o sobrenadante.
- Ressuspenda o pellet de célula com tampão de isolamento e conte os PBMCs.
- Isole as células T das PBMCs usando um kit de seleção negativa. Siga as instruções do fabricante para o kit.
NOTA: Suponha uma recuperação de 10-20% de células T de PBMCs. - Estimule as células T com esferas CD3+/CD28+ .
- Conte as células T.
- Alíquota de grânulos CD3 + / CD28 + suficientes para estimular as células T em uma proporção de 3: 1 grânulos: célula. Por exemplo, para estimular 110 × 106 células T, alíquota 330 × 106 contas. Lave os grânulos 3x separando-os primeiro da suspensão com separação magnética e, em seguida, adicionando 1.000 μL de TCM.
- Adicione as contas à suspensão de células T. Ressuspenda as células T a 1 × 106 células/mL em TCM.
- Alíquota das células T em placas de 6 poços para melhor eficiência de transdução. Adicione 5 × 106 células T a cada poço. Incubar as células a 37 °C, 5% de CO2 por 24 h.
- Isole as células mononucleares do sangue periférico (PBMCs) do sangue de um doador saudável não identificado.
- Dia 1: Em condições BSL-2+, descongele lentivírus CAR suficiente para transduzir células T em uma multiplicidade de infecção de 3,0. Quebre suavemente as rosetas de células T antes de adicionar a quantidade apropriada de lentivírus agrupado para transduzir 5 × 106 células T em um MOI de 3,0. Incubar as células a 37 °C, 5% CO2.
NOTA: Agrupe lentivírus de várias produções em um tubo antes de transduzir as células T. Isso garante que todas as células T recebam o mesmo tratamento. Além disso, o lentivírus deve ser descongelado apenas uma vez. - Dia 2: Sob condições BSL-2+, descongele lentivírus CRISPR suficiente para transduzir células T em um MOI de 0,3. Quebre suavemente as rosetas de células T e ressuspenda as células T antes de adicionar a quantidade apropriada de lentivírus agrupado para transduzir 5 × 106 células T em um MOI de 0,3. Incubar as células a 37 °C, 5% CO2.
NOTA: Agrupe lentivírus de várias produções em um tubo antes de transduzir as células T. Isso garante que todas as células T recebam o mesmo tratamento. - Dias 3 a 5: Ressuspenda as células CART para quebrar as rosetas e contar as células. Traga a concentração celular final para 1 × 106 células / mL em TCM com 1 μg / mL de puromicina. Incubar as células a 37 °C, 5% CO2.
- Dia 6: Ressuspenda as células CART para quebrar as rosetas, remova as esferas CD3+/CD28+ por meio de separação magnética e conte as células CART. Adicione TCM com 1 μg / mL de puromicina para trazer a concentração celular final para 1 × 106 células / mL e incube as células a 37 ° C, 5% CO2. Determinar a expressão de CAR com citometria de fluxo, conforme descrito no passo 4.4.
7. Fase de seleção de gRNA da tela CRISPR (dias 8 a 22)
- Dia 8: Preservar amostras para sequenciamento de próxima geração (NGS) e iniciar a co-cultura.
- Lave as células CART 3x em MTC centrifugando as amostras a 300 × g por 5 min a 4 ° C, aspirando o sobrenadante e ressuspendendo as amostras em 20 mL de TCM.
- Conte as células CART.
- Preserve amostras para NGS completando o seguinte:
- Alíquota de células CART suficientes em um tubo cônico de 50 mL para manter uma representação de gRNA de 500 células por gRNA (por exemplo, alíquota de aproximadamente 33 × 106 células CART se estiver usando uma biblioteca CRISPR com 65.383 gRNAs). Centrifugar a amostra a 300 × g durante 5 min a 4 °C.
- Ressuspenda o pellet celular em 1 mL de PBS e transfira para um tubo de microcentrífuga de 2 mL. Lave o tubo cônico de 50 mL com 1 mL de PBS e transfira a lavagem para o tubo de microcentrífuga de 2 mL.
- Centrifugar as amostras a 500 × g durante 5 min a 4 °C. Remova o sobrenadante. Armazene os pellets de células a -20 °C.
- Inicie a co-cultura adicionando células CART às células CD19+ JeKo-1 na proporção de 1:1. Por exemplo, adicione 80 × 106 células CART a 80 × 106 células JeKo-1. Traga o volume total da co-cultura para 160 mL com TCM. Armazenar as amostras numa incubadora a 37 °C, 5% de CO2.
NOTA: Recomendamos começar com pelo menos 80 × 106 células CART para manter a representação da biblioteca e ter células suficientes para preservar as amostras no Dia 15 e no Dia 22.
- Dia 10: Para reestimular as células CART, colete as co-culturas em tubos cônicos de 50 mL e centrifugue as amostras a 300 × g por 5 min a 4 ° C. Ressuspenda cada amostra em 80 mL de TCM e adicione 80 × 106 células JeKo-1 em 80 mL de TCM. Armazenar as amostras numa incubadora a 37 °C, 5% de CO2.
- Dias 12 e 14: Reestimule as células CART como na etapa 7.2.
- Dia 15: Preservar as amostras para NGS e reestimular as células CART.
- Isole as células CART da co-cultura contando primeiro as células e, em seguida, combinando um kit de seleção positiva para células T CD4 + com um kit de seleção positiva para células T CD8 + para isolar as células T das co-culturas CART-JeKo-1. Conte as células T.
- Preservar as amostras para o NGS aliquotando 33 × 106 células CART num tubo cónico de 50 ml e centrifugando a amostra a 300 × g durante 5 min a 4 °C. Ressuspenda a amostra em 1 mL de PBS e transfira-a para um tubo de microcentrífuga de 2 mL. Lave o tubo cônico de 50 mL com 1 mL de PBS e transfira a lavagem para o tubo de microcentrífuga de 2 mL. Centrifugar as amostras a 500 × g durante 5 min a 4 °C; remova o sobrenadante; e armazene os pellets de células a -20 °C.
- Configure co-culturas para continuar o modelo in vitro de exaustão, adicionando 80 × 106 células CART a 80 × 106 células JeKo-1. Em seguida, traga o volume total da co-cultura para 160 mL com TCM. Armazenar as amostras numa incubadora a 37 °C, 5% de CO2.
- Dias 17, 19 e 21: Reestimule as células CART como na etapa 7.2.
- Dia 22: Preserve as amostras para NGS.
- Isole as células CART da co-cultura contando primeiro as células. Em seguida, combine um kit de seleção positiva para células T CD4+ com um kit de seleção positiva para células T CD8+ para isolar células T das co-culturas CART-JeKo-1. Conte as células T.
- Para preservar amostras para NGS, repita a etapa 7.4.2.
8. Preparação de DNA genômico para sequenciamento de próxima geração
NOTA: Para manter a cobertura adequada, o DNA genômico (gDNA) deve ser isolado de pelo menos 33 × 106 células CART. Além disso, a totalidade do gDNA de 33 × 106 células CART deve ser preparada para sequenciamento.
- Isole o gDNA de pellets de células congeladas usando um kit de isolamento de gDNA.
- Purifique o gDNA por precipitação de etanol.
- Pré-resfriamento 100% EtOH a -20 °C.
- A alíquota eluía o DNA igualmente em vários tubos de microcentrífuga (200-250 μL). Adicionar a cada alíquota o seguinte: dois volumes de EtOH a 100% (500 μL para alíquota de 250 μL ou 400 μL para 200 μL); 0,1 volume de acetato de sódio 3 M (25 μL para alíquota de 250 μL ou 20 μL para 200 μL); e 1 μL de glicogênio 20 mg/mL. Incubar as amostras durante a noite a -20 °C.
- No segundo dia, gire as amostras a 13.000 × g por 20 min para pellet o DNA precipitado. Remova o sobrenadante. Lave os pellets com 1 mL de EtOH a 70% girando a 7.500 × g por 10 min.
- Remova completamente o EtOH residual. Deixe os pellets secarem por 30-45 min.
- Ressuspenda os pellets em 50 μL de água estéril.
- Prepare gDNA para NGS.
NOTA: Os primers NGS para esta biblioteca CRISPR que amplificam gRNAs com sequências adaptadoras Illumina foram projetados em uma publicação anterior27. O uso de um primer reverso diferente para cada amostra irá codificar cada amostra e permitir o agrupamento de amostras durante o sequenciamento.- Preparar as amostras para PCR com a seguinte mistura de reacção (volume total = 50 μL): master mix de PCR de alta fidelidade, 2x: 25 μL; DNA molde combinado, 0,5 μg: 1 μL; Primário direto da biblioteca NGS, 10 μM: 1,25 μL; Primer reverso da biblioteca NGS, 10 μM: 1,25 μL; Água livre de DNase: 21,5 μL.
- Realizar PCR com as seguintes condições de ciclagem: ciclo 1: desnaturar a 98 °C durante 3 min; ciclos 2-23: Desnaturar 98 °C por 10 s, recozer a 63 °C por 10 s, estender a 72 °C por 25 s; ciclo 24: estender a 72 °C por 2 min.
- Agrupe as reações de PCR para cada amostra e purifique o produto de PCR com um kit de purificação de PCR de acordo com as instruções do fabricante.
- Execute 3 μg do produto de PCR para cada amostra em um gel de agarose a 2% (p / v).
- Remova o produto de PCR (~ 260-270 bp) do gel extraindo o DNA do gel usando um kit de extração de gel de acordo com as instruções do fabricante. Armazenar as amostras extraídas a -20 °C.
- Realize o sequenciamento de próxima geração (NGS) com as amostras. Apontar para mais de 500 leituras por gRNA na biblioteca.
9. Análise dos resultados da sequência
- Para analisar o NGS, use a plataforma MAGeCK-VISPR28. Use as configurações padrão para o algoritmo de estimativa de máxima verossimilhança (MAGeCK-MLE). Para preparar os arquivos de saída para análise:
- Execute o FASTQC para avaliar a qualidade do arquivo de sequência31.
- Mescle arquivos de sequenciamento de extremidade emparelhada para cada amostra com bbmerge32.
- Analise as amostras na plataforma MAGeCK-VISPR com o algoritmo MAGeCK-MLE. Para a análise MAGeCK-MLE, normalize todas as contagens de gRNA para a lista de 1.000 gRNAs não direcionados.
- Avalie a qualidade do arquivo de sequenciamento avaliando o conteúdo do GC, a qualidade base, as leituras de sequenciamento e as leituras mapeadas.
- Avalie o sucesso da fase de pressão seletiva da tela CRISPR avaliando as mudanças no índice de Gini, agrupamento da análise de componentes principais (PCA) e vias associadas à lista de genes selecionados negativamente32.
- Interrogue os principais sucessos realizando pesquisas bibliográficas e realizando análises de caminhos.
Resultados
Para interrogar genes e vias que podem ser editados para melhorar a atividade das células CART de maneira imparcial, projetamos uma tela nocaute CRISPR em todo o genoma in vitro (Figura 1). Esta tela tem duas fases: uma fase de produção de células CART e uma fase de pressão seletiva. Na fase de produção de células CART, pelo menos 110 × 106 células T são primeiro isoladas de PBMCs de doadores saudáveis e ativadas com esferas C...
Discussão
A edição de genes tornou-se uma ferramenta poderosa tanto na compreensão dos mecanismos de resistência às terapias quanto no desenvolvimento de novas terapias com células CART para melhorar a longevidade e a atividade das células CART16 , 17 , 26 . Embora algumas estratégias de edição de genes tenham mostrado melhorias na atividade das células CART em modelos pré-clínicos e ensaios ...
Divulgações
A SSK é inventora de patentes no campo da imunoterapia CAR licenciadas para a Novartis (por meio de um acordo entre a Mayo Clinic, a Universidade da Pensilvânia e a Novartis), a Humanigen (por meio da Mayo Clinic), a Mettaforge (por meio da Mayo Clinic) e a MustangBio (por meio da Mayo Clinic) e a Chymal Therapeutics (por meio da Mayo Clinic). CS, CMR e SSK são inventores de patentes licenciadas para a Immix Biopharma. A SSK recebe financiamento de pesquisa da Kite, Gilead, Juno, BMS, Novartis, Humanigen, MorphoSys, Tolero, Sunesis/Viracta, LifEngine Animal Health Laboratories Inc. e Lentigen. A SSK participou de reuniões consultivas com Kite/Gilead, Calibr, Luminary Therapeutics, Humanigen, Juno/BMS, Capstan Bio e Novartis. SSK atuou no conselho de segurança e monitoramento de dados da Humanigen e da Carisma. A SSK cortou um consultor da Torque, Calibr, Novartis, Capstan Bio, BMS, Carisma e Humanigen. CMS e SSK são inventores da propriedade intelectual que resultou deste protocolo.
Agradecimentos
Este estudo foi parcialmente financiado pelo Mayo Clinic Center for Individualized Medicine (SSK), Mayo Clinic Comprehensive Cancer Center (SSK), Mayo Clinic Center for Regenerative Biotherapeutics (SSK), National Institutes of Health K12CA090628 (SSK) e R37CA266344-01 (SSK), Department of Defense grant CA201127 (SSK), Predolin Foundation (SSK) e Minnesota Partnership for Biotechnology and Medical Genomics (SSK). O CMS é apoiado pela Escola de Pós-Graduação em Ciências Biomédicas da Mayo Clinic. O esquema de tela CRISPR (Figura 1) foi criado com BioRender.com (Siegler, L. (2022) https://BioRender.com/k71r054).
Materiais
| Name | Company | Catalog Number | Comments |
| 293T cells | ATCC | CRL-3216 | Cells used for lentivirus production |
| Biotin ProteinL Antibody | GenScript | M00097 | anti-kappa chain antibody for CAR detection |
| Bovine Serum Albumin | Millipore Sigma | A7906 | |
| Carbenicillin disodium salt | Millipore Sigma | C1389-1G | Carbenicillin antibiotic |
| CD4 Isolation Beads | Miltenyi Biotec | 130-045-101 | |
| CD8 Isolation Beads | Miltenyi Biotec | 130-045-201 | |
| CTS (Cell Therapy Systems) Dynabeads CD3/CD28 | Gibco | 40203D | |
| Cytoflex | Beckman Coulter | NC2279958 | |
| DNase-Free Water | Invitrogen | AM9937 | |
| Dulbecco's modified eagle's medium (DMEM) | Corning | 10-017-CV | |
| Dulbecco's Phosphate-Buffered Saline | Gibco | 14190-144 | |
| EasySep Human T Cell Isolation Kit | STEMCELL Technologies | 17951RF | Negative isolation kit |
| Endura Electrocompetent Cells | Biosearch Technologies | 60242-1 | Electrocompetent cells with recovery medium |
| Ethanol | Millipore Sigma | E7023 | |
| Fetal bovine serum (FBS) | Corning | 35-010-CV | |
| GeCKO v2 CRISPR Knockout Pooled Library A | AddGene | 1000000048 | CRISPR library plasmid |
| Gene Pulser II | Bio-Rad | 165-2105 | Electroporator |
| Glycogen | Millipore Sigma | 10901393001 | |
| JeKo-1 | ATCC | CRL-3006 | CD19+ target cells |
| Lipofectamine 3000 Transfection Reagent | ThermoFisher Scientific | L3000075 | Transfection reagent kit with a transfection reagent (Lipofectamine 3000 Reagent) and a neutral co-lipid reagent (p3000) |
| LIVE/DEAD Aqua | Invitrogen | L34966 | |
| Lymphoprep | STEMCELL Technologies | 7851 | Density gradient medium |
| Machery-Nagel NucleoBond Xtra Maxi Kits | ThermoFisher Scientific | 12748412 | Maxi-prep kit |
| NEBNext High-Fidelity 2X PCR MasterMix | New England BioLabs | M0541S | High fidelity PCR mastermix |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985-070 | Reduced serum medium |
| pCMVR8.74 | AddGene | 22036 | Lentiviral packaging plasmid |
| Pennicillin-streptomycin-glutamine (100X) | Life Technologies | 10378-016 | |
| pMD2.G | AddGene | 12259 | VSV-G envelope expressing plasmid |
| Pooled Human AB Serum | Innovative Research | ISERABHI | |
| Puromycin | Millipore Sigma | P8833 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | Gek extraction kit |
| Qucik-DNA Midiprep Plus Kit | Zymo Research | D4075 | Kit used to isolate gDNA |
| RoboSep-S | STEMCELL Technologies | 21000 | Automated cell separator |
| Roswell Park Memorial Institute 1640 Medium (RPMI) | Gibco | 21870092 | |
| SepMate-50 | STEMCELL Technologies | 85450 | Density gradient separation tube |
| Sodium Acetate | Invitrogen | AM9740 | |
| Sodium Azide | Fisher Scientific | 71448-16 | |
| Streptavidin Antibody (PE) | BioLegend | 405203 | Secondary antibody used for CAR detection |
| T100 Thermal Cycler | Bio-Rad | 1861096 | |
| Ultracentrifuge (Optima XPN-80) | BeckmanCoulter | A99839 | |
| Vacuum Filter Systems, 0.22 µm | ThermoFisher Scientific | 567-0020 | |
| Vacuum Filter Systems, 0.45 µm | ThermoFisher Scientific | 165-0045 | |
| X-VIVO 15 Serum-Free Hematopoietic Cell Medium | Lonza | 04-418Q | Hematopoietic cell medium |
Referências
- Neelapu, S. S., et al. 5-year follow-up supports curative potential of axicabtagene ciloleucel in refractory large B-cell lymphoma (zuma-1). Blood. 141 (19), 2307-2315 (2023).
- Neelapu, S. S., et al. Axicabtagene ciloleucel CAR T-cell therapy in refractory large B-cell lymphoma. N Engl J Med. 377 (26), 2531-2544 (2017).
- Locke, F. L., et al. Long-term safety and activity of axicabtagene ciloleucel in refractory large B-cell lymphoma (zuma-1): A single-arm, multicentre, phase 1-2 trial. Lancet Oncol. 20 (1), 31-42 (2019).
- Maude, S. L., et al. Tisagenlecleucel in children and young adults with B-cell lymphoblastic leukemia. N Engl J Med. 378 (5), 439-448 (2018).
- Schuster, S. J., et al. Tisagenlecleucel in adult relapsed or refractory diffuse large B-cell lymphoma. N Engl J Med. 380 (1), 45-56 (2018).
- Bai, Z., et al. Single-cell antigen-specific landscape of CAR T infusion product identifies determinants of CD19-positive relapse in patients with all. Sci Adv. 8 (23), eabj2820 (2022).
- Fraietta, J. A., et al. Determinants of response and resistance to CD19 chimeric antigen receptor (CAR) T cell therapy of chronic lymphocytic leukemia. Nat Med. 24, 563-571 (2018).
- Gennert, D. G., et al. Dynamic chromatin regulatory landscape of human CAR T cell exhaustion. Proc Natl Acad Sci USA. 118 (30), e2104758118 (2021).
- Deng, Q., et al. Characteristics of anti-CD19 CAR T cell infusion products associated with efficacy and toxicity in patients with large B cell lymphomas. Nat Med. 26 (12), 1878-1887 (2020).
- Sterner, R. M., Cox, M. J., Sakemura, R., Kenderian, S. S. Using CRISPR/CAS9 to knock out GM-CSF in CAR-T cells. J Vis Exp. (149), e59629 (2019).
- Sterner, R. M., et al. GM-CSF inhibition reduces cytokine release syndrome and neuroinflammation but enhances CAR-T cell function in xenografts. Blood. 133 (7), 697-709 (2019).
- Cox, M. J., et al. GM-CSF disruption in CART cells modulates T cell activation and enhances CART cell anti-tumor activity. Leukemia. 36 (6), 1635-1645 (2022).
- Ottaviano, G., et al. Phase 1 clinical trial of CRISPR-engineered CAR19 universal T cells for treatment of children with refractory B cell leukemia. Sci Transl Med. 14 (668), eabq3010 (2022).
- Joung, J., et al. Genome-scale CRISPR-CAS9 knockout and transcriptional activation screening. Nat Protoc. 12, 828-863 (2017).
- Mccutcheon, S. R., et al. Transcriptional and epigenetic regulators of human CD8+ T cell function identified through orthogonal CRISPR screens. Nat Genet. 55, 2211-2223 (2023).
- Zhu, W., Kelly, C., Dagur, P., Dunbar, C. E., Cordes, S. CRISPR activation screen to optimize chimeric antigen receptor (CAR) T cell immunophenotype. Blood. 142 (Supplement 1), 4820-4820 (2023).
- Wang, D., et al. CRISPR screening of CAR T cells and cancer stem cells reveals critical dependencies for cell-based therapies. Cancer Discov. 11 (5), 1192-1211 (2021).
- Shifrut, E., et al. Genome-wide CRISPR screens in primary human T cells reveal key regulators of immune function. Cell. 175 (7), 1958-1971.e15 (2018).
- Belk, J. A., et al. Genome-wide crispr screens of T cell exhaustion identify chromatin remodeling factors that limit T cell persistence. Cancer Cell. 40 (7), 768-786.e7 (2022).
- Jiang, P., et al. Single-cell ATAC-seq maps the comprehensive and dynamic chromatin accessibility landscape of CAR-T cell dysfunction. Leukemia. 36 (11), 2656-2668 (2022).
- Rossi, J., et al. Preinfusion polyfunctional anti-CD19 chimeric antigen receptor T cells are associated with clinical outcomes in NHL. Blood. 132 (8), 804-814 (2018).
- Beider, K., et al. Molecular and functional signatures associated with CAR T cell exhaustion and impaired clinical response in patients with B cell malignancies. Cells. 11 (7), 1140 (2022).
- Wherry, E. J. T. cell exhaustion. Nat Immunol. 12, 492-499 (2011).
- Prinzing, B., et al. Deleting DNMT3A in CAR T cells prevents exhaustion and enhances antitumor activity. Sci Transl Med. 13, eabh0272 (2021).
- Lynn, R. C., et al. C-jun overexpression in CAR T cells induces exhaustion resistance. Nature. 576, 293-300 (2019).
- Tang, N., et al. TGF-beta inhibition via CRISPR promotes the long-term efficacy of CAR T cells against solid tumors. JCI Insight. 5 (4), e133977 (2020).
- Joung, J., et al. Genome-scale CRISPR-CAS9 knockout and transcriptional activation screening. Nat Protoc. 12 (4), 828-863 (2017).
- Li, W., et al. Quality control, modeling, and visualization of CRISPR screens with MAGeCK-VISPR. Genome Biol. 16, 281 (2015).
- Sakemura, R., et al. Targeting cancer-associated fibroblasts in the bone marrow prevents resistance to CART-cell therapy in multiple myeloma. Blood. 139 (26), 3708-3721 (2022).
- Stewart, C. M., et al. IL-4 drives exhaustion of CD8+ CART cells. Nat Commun. 15 (1), 7921 (2024).
- . FastQC: A quality control tool for high throughput sequence data Available from: https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010)
- . Bbmerge Available from: https://github.com/BioInfoTools/BBMap/blob/master/sh/bbmerge.sh (2019)
- Pattanayak, V., et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed CAS9 nuclease specificity. Nat Biotechnol. 31 (9), 839-843 (2013).
- Doench, J. G., et al. Rational design of highly active sgRNAs for CRISPR-CAS9-mediated gene inactivation. Nat Biotechnol. 32 (12), 1262-1267 (2014).
- Dong, M. B., et al. Systematic immunotherapy target discovery using genome-scale in vivo CRISPR screens in CD8 T cells. Cell. 178 (5), 1189-1204.e23 (2019).
- Zhou, P., et al. Single-cell CRISPR screens in vivo map T cell fate regulomes in cancer. Nature. 624 (7990), 154-163 (2023).
- Sutra Del Galy, A., et al. In vivo genome-wide CRISPR screens identify SOCS1 as intrinsic checkpoint of CD4+ TH1 cell response. Sci Immunol. 6 (66), eabe8219 (2021).
- Ramos, A., et al. Leukemia-intrinsic determinants of CAR-T response revealed by iterative in vivo genome-wide CRISPR screening. Nat Commun. 14 (1), 8048 (2023).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados