このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
キメラ抗原受容体T細胞におけるIn vitroゲノムワイドCRISPRノックアウトスクリーニングの実施
要約
この記事では、健康なドナーキメラ抗原受容体T細胞におけるゲノムワイドCRISPRノックアウトスクリーニングを完了するための消耗に対する in vitro モデルの適用に関するプロトコルについて説明しています。
要約
キメラ抗原受容体T(CART)細胞療法は、がんの治療に革命をもたらした革新的な標的免疫療法です。ただし、耐久性のある応答は依然として制限されています。最近の研究では、注入前のCART細胞製品のエピジェネティックな状況が治療への反応に影響を与える可能性があることが示されており、その解決策として遺伝子編集が提案されています。しかし、最適な遺伝子編集戦略を決定するためには、さらに多くの作業を行う必要があります。ゲノムワイドなCRISPRスクリーニングは、耐性のメカニズムを調査し、遺伝子編集戦略を最適化するための一般的なツールとなっています。しかし、初代細胞への応用には多くの課題があります。ここでは、健康なドナーのCART細胞でゲノムワイドなCRISPRノックアウトスクリーニングを完成させる方法について説明します。概念実証モデルとして、CD19抗原を標的とするCART細胞の枯渇の進行を調べるために、この手法を設計しました。しかし、このアプローチは、さまざまなCARコンストラクトや腫瘍モデルにおける治療に対する耐性のさまざまなメカニズムに対処するために使用できると考えています。
概要
キメラ抗原受容体T(CART)細胞療法は、B細胞悪性腫瘍の治療において目覚ましい成功を収めています。ただし、耐久性のある応答は30〜40%に制限されています1,2,3,4,5。研究者は、CAR設計の最適化、遺伝子編集、併用療法など、CART細胞療法に対する耐性のメカニズムに対処するためのいくつかのアプローチを開発およびテストしてきましたが、耐性の発生はほとんどわかっていません。最近、注入前CART細胞製品のベースライン遺伝子発現プロファイルが、CART療法の毒性と有効性の両方の重要な決定要因であるという証拠が増えています6,7,8,9。そのため、CART細胞産生中の遺伝子編集は、CART細胞治療の成功を向上させるための一般的なアプローチとなっています。
例えば、私たちの研究室では、遺伝子顆粒球マクロファージコロニー刺激因子(GM-CSF)をノックアウトするために、クラスター化された規則的な間隔を空けた短い回文反復(CRISPR)Cas9技術を使用すると、CART細胞の活性が向上し、CART関連毒性の兆候が減少することを示しました10,11,12。さらに、CRISPR改変細胞療法は臨床試験で検証されており、有効性と安全性が示されています13。これらを総合すると、CRISPR技術を用いた遺伝子編集は、CART細胞生物学の理解を深めるだけでなく、翻訳可能なCART細胞産物を作製できることを示しています。
ゲノムワイドなCRISPRノックアウトスクリーニングは、治療薬に対する耐性のメカニズムを理解するためのがん生物学研究において、ますます人気のあるツールとなっています。この手法では、数万のガイドRNA(gRNA)が細胞のプールに送達され、細胞1個につき1個のgRNAの侵入が促進される14。その後、細胞は圧力誘導状態に置かれ、必須遺伝子を標的とするgRNAを形質導入した細胞は死滅し、阻害遺伝子を標的とするgRNAを形質導入した細胞は生存して増殖します。次世代シーケンシングを用いることで、CRISPRスクリーニング全体でgRNAの表現がどのように変化するかを決定することができる14。
しかし、ゲノムワイドな遺伝子摂動に必要なスケールと選択時間は、CART細胞のような初代細胞で達成するのが難しい場合があります。そのため、グループは標的CRISPRスクリーニングを利用して、治療耐性のメカニズムをさらに理解しています15,16。ターゲットスクリーニングは、多くの場合、一次細胞では、適切なライブラリー表現を達成するために必要な細胞が少なくて済む小さなライブラリーを持っているため、簡単に完了できます。これらの研究により、CART細胞療法に対する耐性のメカニズムについての理解が深まりましたが、標的スクリーニングでは、遺伝子標的を手動で選択するため、バイアスが生じます。この記事では、健康なドナーのCART細胞でin vitroゲノムワイドCRISPRノックアウトスクリーニングを完了する方法について説明しようとしています。そのため、このハイスループットアプローチにより、治療反応を改善するために編集できる主要な経路と遺伝子を効率的かつ偏りなく同定することができます17,18,19。
特に、この記事で説明するプロトコルは、消耗のin vitroモデルを使用してゲノムワイドなCRISPRノックアウトスクリーニングを完了することにより、CART細胞の消耗に関する分野の理解を深めるように設計されています。疲労は、CART細胞療法に対する無反応に関与している機能不全のCART細胞運命である20,21,22。この細胞運命はエピジェネティックに制御されていることが知られており、CART細胞の増殖の減少、エフェクターサイトカイン産生の減少、および抑制性受容体の発現の増加を特徴としています23。先行文献では、遺伝子編集は、主要な遺伝子をアップレギュレーションまたはダウンレギュレーションすることによって、疲労の発生を防ぐことができた24,25,26。CART細胞が疲弊するにつれて増殖能力が低下することと、遺伝子編集がその発生を防ぐことができるという証拠の両方を考慮して、この細胞運命に基づいてin vitroゲノムワイドCRISPRノックアウトスクリーニングをモデル化しました。ただし、このプロトコルは、CART細胞療法に対する耐性の他のメカニズムを調査するために、将来修正される可能性があります。
プロトコル
重要なことに、以下に概説するプロトコルは、メイヨークリニックの治験審査委員会(IRB 18-005745)および施設バイオセーフティ委員会(IBC HIP00000252.43)のガイドラインに従い、承認を受けています。レンチウイルスの産生を含むすべての細胞培養作業は、適切な個人用保護具を備えた細胞培養フードで実施する必要があります。特に、レンチウイルスの作業は、廃棄前に10%漂白剤を使用してアイテムを消毒するなど、バイオセーフティレベル2(BSL-2)の予防措置の下で実施する必要があります。
1. CRISPRライブラリーの増幅
- メーカーの推奨に従ってCRISPRライブラリを増幅します。簡単に説明すると、以下を完了することにより、CRISPRライブラリをエレクトロポレートしてエレクトロコンピテントセルの4つのアリコートにします。
- 1.0 mmキュベット中の25 μLのエレクトロコンピテントセルに、2 μLの50 ng/μL CRISPRライブラリープラスミドを添加します。
- 10 μF、600 Ω、1,800 V、3.5 ms〜4.5 msの時定数の設定を使用して、サンプルをエレクトロポレートします。
- 975 μLのリカバリー培地で回収し、1 mLのリカバリー培地を追加したチューブに移します。
- すべてのアリコートを250 RPMで37°Cで1時間回転させます。
- 変換をプレートします。
- エレクトロポレーションセルのすべてのアリコートをプールし、よく混合します。
- 予熱した10 cmのLuriaブロス(LB)プレートに400 μLの形質転換ミックスを100 μg/mLのカルベニシリンで広げて、すべてのエレクトロポレーション細胞をプレート化します。プレートを32°Cのインキュベーターに14時間置きます。
- コロニーを収穫します。
- 各プレートに1 mLのLB培地+ 100 μg / mLのカルベニシリンをピペットで移します。コロニーを各プレートからこすり落とし、50mLのコニカルチューブに集めます。
- さらに1 mLのLB培地+100 μg/mLのカルベニシリンを各プレートに加え、洗浄して完全に回復させます。
- マキシプレップキットの指示に従って、マキシプレップを実行します。
- 得られたプラスミドDNAをプールします。
2. CRISPRライブラリーにおけるベースラインgRNAの発現を検証するための次世代シーケンシング
注:このCRISPRライブラリーのNGSプライマーは、以前の論文27で設計されています。サンプルごとに異なるリバースプライマーを使用すると、各サンプルがバーコード化され、シーケンシング中にサンプルをプールできます。
- 次の反応混合物(総容量= 50 μL)を使用してPCR用のサンプルを調製します:ハイフィデリティPCRマスターミックス、2x:25 μL;プールされたテンプレートDNA、0.5 μg:1 μL;NGSライブラリフォワードプライマー、10 μM:1.25 μL;NGSライブラリリバースプライマー、10 μM:1.25 μL;DNaseフリー水:21.5μL。
- 次のサイクリング条件でPCRを実行します:サイクル1:98°Cで3分間変性します。サイクル2-23:98°Cで10秒間変性、63°Cで10秒間アニール、72°Cで25秒間延長。サイクル24:72°Cで2分間延長します。
- 各サンプルのPCR反応をプールし、製造元の指示に従ってPCR精製キットでPCR産物を精製することにより、PCR反応を精製します。
- 各サンプルについて3 μgのPCR産物を2%(w/v)アガロースゲルで泳ぎます。
- PCR産物(~260-270 bp)をゲルから取り出し、製造元の指示に従ってゲル抽出キットを使用してゲルからDNAを抽出します。少なくとも500 ngのDNAを目指してください。
- 抽出したサンプルは-20°Cで保存します。
- サンプルを用いて次世代シーケンシング(NGS)を行い、gRNA27あたり100リードを目指します。
- MAGeCK-VISPRパイプラインでシーケンシング結果を解析し、ライブラリ内のgRNAの99.5%以上が表されていることを確認28。
3. CRISPRライブラリーとCAR発現レンチウイルスの作製
- 前述のようにレンチウイルスを調製します10,29。
- 1日目:10%ウシ胎児血清(FBS)(v / v)、1%ペニシリン-ストレプトマイシン-グルタミン(PSG)(v / v)、およびダルベッコの修飾ワシ培地で構成されるD10培地で増殖している80〜90%のコンフルエント293T細胞のT175フラスコから始めます。開始する前に、トランスフェクション試薬とDNAを室温まで温め、DNAを定量します。
- 293Tフラスコごとに、2本の50 mLコニカルチューブ A と Bを標識します。4.5 mLの還元血清培地をチューブ A とチューブ Bに加えます。.
- 滅菌済みの1.5 mL微量遠心チューブで、18 μgのpCMVR8.74(gag、pol、tat、およびrev)、7 μgのpMD2.G(VSV-G)、および15 μgのレンチウイルス発現ベクター(CRISPRライブラリーAまたはCAR19-28ζベクター)を1分間静置します。
- チューブ Aに129 μLのトランスフェクション試薬を添加します。チューブ A をフィンガーボルテックスでよくミックスします。
- DNA混合物をチューブ Bに加えます。チューブ B をフィンガーボルテックスでよく混ぜます。
- 111 μLの中性共脂質試薬をチューブ Bに加えます。チューブ B をフィンガーボルテックスでよく混ぜます。
- チューブ A の内容物をチューブ B に加え、フィンガーボルテックスでよく混ぜます。チューブ B を室温で30分間インキュベートします。
- 30分間のインキュベーション期間の終わりに向けて、既存の培地を吸引して293T細胞を調製します。10% FBS(v/v)、1% PSG(v/v)、およびロズウェルパークメモリアルインスティテュート(RPMI)1640で構成されるR10培地16 mLを16 mL加えます。
- 30分間のインキュベーション期間後、チューブ B の内容物(約9mL)を293T細胞のフラスコに滴下します。フラスコを下に置き、静かに回転させてから、37°C、5%CO2でインキュベートします。
- 2日目(トランスフェクションの約24時間後)に、次のようにレンチウイルスを収集します。
注意: すべての機器と材料を事前に冷やし、氷の上で以下を実行してウイルス上清を冷たく保ちます。- トランスフェクションした293Tフラスコから上清を予冷した50mLコニカルチューブに移します。
- 予熱したR10培地30 mLを293Tフラスコに加えます。
- 50 mLのコニカルチューブを900 × g で4°C、10分間遠心分離することにより、細胞の破片をペレット化します。
- 遠心分離後、0.45 μmフィルターで上清をろ過します。濾液を超遠心分離機で112,700 × g 、4°C、2時間回転させます。
- 2時間回転後、約5 mLが残るまで上清を吸引します。.ウイルス粒子を10倍に静かにピペッティングして再懸濁します。ウイルス懸濁液を4°Cで一晩保存してください。
- 3日目に、次のようにレンチウイルスを収集して保管します。
- トランスフェクションした293Tフラスコから上清を予冷した50mLコニカルチューブに移します。
- 293Tフラスコに10%漂白剤30mLを加えます。
- 50 mLのコニカルチューブを900 gで900 × g 、4°Cで10分間遠心分離することにより、上清中の細胞破片をペレット化します。
- 遠心分離後、0.45 μmフィルターで上清をろ過します。濾液を超遠心分離機で112,700 × g 、4°C、2時間回転させます。
- 2時間の遠心分離後、約2 mLが残るまで上清を吸引し、10回静かにピペッティングして再懸濁します。ウイルス上清を300〜500μLのアリコートに保存し、-80°Cに保ちます。
4. CAR発現レンチウイルスのウイルス力価の計算
注:CARレンチウイルスは、前述の10,12と同様に力価が測定されました。
- 1日目に、T細胞を活性化してプレートします。
- ネガティブセレクションキットを使用して、健康なドナーPBMCから1×106 T細胞を単離します。
- T細胞を数えます。
- T細胞を刺激するのに十分なCD3+/CD28+ ビーズを3:1のビーズ:セル比で洗浄してください。例えば、1 × 106 T細胞を刺激するには、3 × 106 ビーズを洗浄します。これを行うには、次の3回を完了してビーズを洗います。
- 磁気分離を使用して、懸濁液からビーズを取り除きます。
- 上清を取り除きます。
- 10%ヒト血清(v/v)、1%PSG(v/v)、造血細胞培地からなるT細胞培地(TCM)1000 μLを添加します。
注:TCMは、使用前に0.45μmの滅菌真空フィルターでろ過し、次に0.22μmの滅菌真空フィルターでろ過して滅菌する必要があります。 - T細胞懸濁液にビーズを3:1のビーズ:セル比で添加します。
- T細胞をTCMで1 × 106 細胞/mLに再懸濁します。
- 100 μLの活性化T細胞を96ウェルプレートの10ウェルに添加します。プレートを37°C、5%CO2でインキュベートします。
- 2日目に、次のように希釈プレートを作成して、CARレンチウイルスの希釈を追加します。
- 100 μLのTCMを96ウェルプレートの7ウェルに添加します。
- 最初のウェルに、目的のウイルス50μLを加えます。よく混ぜます。
- ウェル1からウェル2に50μLを添加して、ウイルスをさらに連続希釈します。よく混ぜます。
- ウェル2からウェル3に50μLを加え、このパターンをウェル7まで続けます。
- ウェル7を混合した後、50 μLを取り出し、すべてのウェルの総容量が100 μLであることを確認します。
- 希釈プレートの各ウェルから50μLを1日目のT細胞プレートの最初の7つのウェルに加えます。よく混ぜます。
- T細胞プレートの最後の3つのウェルに、50μLのTCMを加えます。よく混ぜます。プレートを37°C、5%CO2のインキュベーターに保管します。
- 3日目に、T細胞プレートのすべてのウェルに100μLのTCMを加えて細胞を給餌します。よく混ぜます。
- 4日目に、以下を完了することにより、CAR発現の代替マーカーである抗カッパ鎖抗体でCARを発現する細胞の割合を検出するためにフローサイトメトリーを実施します。
- T細胞プレートを1,000 × g で3分間スピンダウンし、デカントします。
- 200 μLの抗κ鎖抗体バッファーを加え、プレートを1,000 × g で3分間スピンダウンし、デカンテーションして、抗κ鎖抗体バッファー(PBS中の4%ウシ血清アルブミン(w / v)で構成)でプレートを3回洗浄します。
- ビオチン抗κ鎖抗体を各ウェルに添加します。ロッカー上で45分間インキュベートし、4°Cで保存します。
- ステップ4.4.2と同様に、プレートを抗κ鎖抗体バッファーで3回洗浄します。
- フローサイトメトリー用の細胞を、蛍光色素標識ストレプトアビジン抗体と生/死抗体で染色します。サンプルを暗所で室温で15分間インキュベートします。
- 200 μLのフローバッファーを加え、プレートを1,000 ×gで3分間スピンダウンし、デカンテーションして、フローバッファー(2% FBS(v / v)とPBS中の1%アジ化ナトリウム(v / v)で構成)でサンプルを一度洗浄します。
- 100 μLのフローバッファーに再懸濁し、サイトメーターで実行します。
- 3つの未形質導入ウェルでネガティブゲーティングを行います。
5. CRISPRライブラリーレンチウイルスのウイルス力価の計算
注:このプロトコルは、CRISPRライブラリーレンチウイルスを滴定するための最小数のT細胞を説明していますが、より多くのT細胞、したがって大量のウイルスに対応するためにスケールアップすることができます。
- 0日目に、18 × 106 CD3+/CD28+ ビーズ (ビーズ:細胞比 3:1) を 6 × 106 T細胞に添加し、TCM で最終容量を 6 mL にすることで T 細胞を活性化します。0.5 mLのT細胞懸濁液を48ウェルプレートの12ウェルに分注します。インキュベーターに37°C、5%CO2で保存します。
- 24時間後の 1日目に、感染の多重度(MOI)が3のCARウイルスをすべてのウェルに追加します。ピペッティングを数回上下させてよく混ぜます。インキュベーターに37°C、5%CO2で保存します。
- 48時間後の 2日目に、次のボリュームのCRISPRウイルスのいずれかを各ウェルに追加することにより、活性化CART細胞に一連のCRISPRライブラリウイルスボリュームを追加します(0 μL、0 μL、6.25 μL、6.25 μL、12.5 μL、12.5 μL、25 μL、25 μL、50 μL、50 μL、100 μL、または100 μL)。ピペッティングを数回上下させてよく混ぜます。インキュベーターに37°C、5%CO2で保存します。
- 3日目に、次のCRISPRウイルス容量(0 μL、6.25 μL、12.5 μL、25 μL、50 μL、100 μL)をそれぞれ投与したウェルを含む、培地1 mLあたり1 μgのピューロマイシンをウェルの半分に加えます。他のウェルはピューロマイシンで処理されません。
- 4 日目から 8 日目まで、毎日 T 細胞をカウントし、最終的な細胞密度を 1 × 106 細胞/mL に調整し、ピューロマイシン処理ウェルの場合は TCM に 1 μg/mL ピューロマイシンを使用し、未処理ウェルには TCM を使用します。
- 8日目に、ウェルあたりの生細胞の数をカウントし、次の式を使用してウイルス上清の形質導入単位/ mLを計算します。
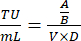
ここで、A:ウイルスに形質導入された生細胞数。B:抗生物質選択なしのコントロールウェルでの生細胞数。V:各ウェルの感染に使用されたウイルスの量。D:希釈倍率。 - 約 20% の形質導入効率を示すウェルから計算した TU/mL ウェルを使用して、次の式を使用して MOI 0.3 で 1 個× 10個の 6 個の細胞を形質導入するために必要なウイルスの量を計算します。
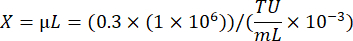
6. CRISPRスクリーニングのCART細胞産生段階(0日目から8日目)
注:以下のプロトコルは、1回の生物学的複製におけるCRISPRスクリーニングの完了を説明しています。しかし、このプロトコルは以前に3つの生物学的複製で検証されており、このプロトコル30を完了する他のものに対しては、少なくとも3つの生物学的複製を使用することを推奨する。
- 0日目:前述のように、健康なドナーの血液からT細胞を分離して刺激します10。
- 匿名化された健康なドナー血液から末梢血単核細胞(PBMC)を分離します。
- 15 mLの密度グラジエント培地を密度グラジエント分離チューブに加えます。
- 1:2 に希釈した血液を密度勾配分離チューブの上部に加えます。1,200 × g で室温で10分間遠心分離します。
注:血液は、分離バッファー(リン酸緩衝生理食塩水[PBS]中の2%FBS(v / v))で希釈する必要があります。. - 新しい50 mLコニカルチューブへの最上層からの脱落が不十分で、アイソレーションバッファーで容量が2倍になります。サンプルを300 × g で4°Cで8分間遠心分離します。 上清を吸引します。
- 細胞ペレットを分離バッファーで再懸濁し、PBMCをカウントします。
- ネガティブセレクションキットを使用してPBMCからT細胞を単離します。キットの製造元の指示に従ってください。
注:PBMCからのT細胞の10〜20%の回収を想定しています。 - CD3+/CD28+ ビーズでT細胞を刺激する。
- T細胞を数えます。
- T細胞を3:1のビーズ:セル比で刺激するのに十分なCD3+/CD28+ ビーズを分注します。例えば、110×106 T細胞を刺激するには、330×106 ビーズを分注する。まずビーズを磁気分離で懸濁液から分離し、次にTCM1,000μLを加えて、ビーズを3回洗浄します。
- T細胞懸濁液にビーズを添加します。T細胞をTCMで1 × 106 細胞/mLに再懸濁します。
- T細胞を6ウェルプレートに分注して、最高の形質導入効率を得ます。各ウェルに5×106 T細胞を添加します。細胞を37°C、5%CO2 で24時間インキュベートします。
- 匿名化された健康なドナー血液から末梢血単核細胞(PBMC)を分離します。
- 1日目:BSL-2+条件下で、感染の多様性が3.0のT細胞に形質導入するのに十分なCARレンチウイルスを解凍します。T細胞のロゼットを静かに砕いてから、プールしたレンチウイルスを適切な量に加え、MOI3.0で5×106 のT細胞を形質導入します。細胞を37°C、5%CO2でインキュベートします。
注:T細胞を形質導入する前に、複数のプロダクションからのレンチウイルスを1つのチューブにプールします。これにより、すべてのT細胞が同じ治療を受けることが保証されます。さらに、レンチウイルスは一度だけ解凍する必要があります。 - 2日目:BSL-2+条件下で、T細胞に形質導入するのに十分な量のCRISPRレンチウイルスをMOI0.3で解凍します。T細胞のロゼットを静かに分解し、T細胞を再懸濁してから、適切な量のプールされたレンチウイルスを添加して、MOI0.3で5個×10個の6 個のT細胞を形質導入します。細胞を37°C、5%CO2でインキュベートします。
注:T細胞を形質導入する前に、複数のプロダクションからのレンチウイルスを1つのチューブにプールします。これにより、すべてのT細胞が同じ治療を受けることが保証されます。 - 3 - 5日目: CART 細胞を再懸濁してロゼットを分解し、細胞をカウントします。最終細胞濃度をTCM中の106 細胞/mL×1 μg/mLピューロマイシンで1 6細胞/mLにします。細胞を37°C、5%CO2でインキュベートします。
- 6日目:CART細胞を再懸濁してロゼットを分解し、磁気分離によってCD3 + / CD28 + ビーズを除去し、CART細胞をカウントします。TCMを1 μg/mLピューロマイシンと添加して、最終細胞濃度を1 × 106 細胞/mLにし、細胞を37°C、5%CO2でインキュベートします。ステップ4.4で説明したフローサイトメトリーでCAR発現を測定します。
7. CRISPRスクリーニングのgRNA選択段階(8日目〜22日目)
- 8日目:次世代シーケンシング(NGS)用のサンプルを保存し、共培養を開始します。
- サンプルを300 × g 、4°Cで5分間遠心分離し、上清を吸引し、サンプルを20 mLのTCMに再懸濁することにより、CART細胞をTCMで3回洗浄します。
- CARTセルをカウントします。
- NGSのサンプルを保存するには、以下の手順を完了してください。
- gRNAあたり500個の細胞のgRNA表現を維持するために、50 mLの円錐チューブに十分なCART細胞を分注します(たとえば、65,383個のgRNAを含むCRISPRライブラリーを使用する場合は、約33個×10個の6 個のCART細胞を分注します)。サンプルを300 × g で4°Cで5分間遠心分離します。
- 細胞ペレットを1 mLのPBSに再懸濁し、2 mLの微量遠心チューブに移します。50 mLのコニカルチューブを1 mLのPBSで洗浄し、洗浄液を2 mLの微量遠心チューブに移します。
- サンプルを500 × g で4°Cで5分間遠心分離します。上清を取り除きます。セルペレットは-20°Cで保存します。
- CD19+ JeKo-1細胞にCART細胞を1:1の比率で加えることにより、共培養を開始します。たとえば、80 × 106 CART セルを 80 × 106 JeKo-1 セルに追加します。TCMで共培養の総量を160 mLにします。サンプルをインキュベーター(37°C、5% CO2)で保存します。
注:ライブラリ表現を維持し、15日目と22日目にサンプルを保存するのに十分な細胞を持つために、少なくとも80個×10個の6 CART細胞から始めることをお勧めします。
- 10日目:CART細胞を再刺激するために、共培養物を50 mLのコニカルチューブに集め、サンプルを300 × g で4°Cで5分間遠心分離します。各サンプルを80 mLのTCMに再懸濁し、80 mLのTCMに80 ×10 6 JeKo-1細胞を加えます。サンプルをインキュベーター(37°C、5% CO2)で保存します。
- 12日目 と 14日目:ステップ7.2のようにCART細胞を再刺激します。
- 15日目:NGS用のサンプルを保存し、CART細胞を再刺激します。
- 最初に細胞をカウントし、次にCD4+ T細胞のポジティブ選択キットとCD8+ T細胞のポジティブ選択キットを組み合わせて、CART-JeKo-1共培養からT細胞を分離することにより、共培養からCART細胞を分離します。T細胞を数えます。
- 50 mLのコニカルチューブに33個×10個の6 CART細胞を分注し、サンプルを300 × g で4°Cで5分間遠心分離することにより、NGS用のサンプルを保存します。サンプルを1 mLのPBSに再懸濁し、2 mLの微量遠心チューブに移します。50 mLのコニカルチューブを1 mLのPBSで洗浄し、洗浄液を2 mLの微量遠心チューブに移します。サンプルを500 × g で4°Cで5分間遠心分離します。上清を取り除きます。細胞ペレットを-20°Cで保存します。
- 80 × 106 CART細胞を80 106 JeKo-1細胞に添加することにより、消耗に対するin vitroモデルを継続×共培養を設定します。次に、TCMで共培養物の総容量を160 mLにします。サンプルをインキュベーター(37°C、5% CO2)で保存します。
- 17日目、19日目、 21日目:ステップ7.2のようにCART細胞を再刺激します。
- 22日目:NGS用のサンプルを保存します。
- 最初に細胞をカウントすることにより、共培養物からCART細胞を単離します。次に、CD4+ T細胞のポジティブ選択キットとCD8+ T細胞のポジティブ選択キットを組み合わせて、CART-JeKo-1共培養物からT細胞を単離します。T細胞を数えます。
- NGSのサンプルを保存するには、ステップ7.4.2を繰り返します。
8. 次世代シーケンシングのためのゲノムDNAの作製
注:十分なカバレッジを維持するために、ゲノムDNA(gDNA)は少なくとも33×106 CART細胞から単離する必要があります。さらに、33 ×10 6 CART細胞由来のgDNA全体をシーケンシング用に準備する必要があります。
- 凍結細胞ペレットからgDNAを単離するには、gDNA単離キットを使用します。
- エタノール沈殿によりgDNAを精製します。
- 100% EtOHを-20°Cまで予冷します。
- アリコートは、いくつかの微量遠心チューブ(200-250 μL)にDNAを均等に溶出しました。各アリコートに以下を追加します:100% EtOHの2つの容量(250 μLアリコートの場合は500 μL、200 μLの場合は400 μL)。0.1容量の3 M酢酸ナトリウム(250 μLアリコートの場合は25 μL、200 μLの場合は20 μL)。20 mg/mL グリコーゲン 1 μLサンプルを-20°Cで一晩インキュベートします。
- 2日目に、サンプルを13,000 × g で20分間遠心して、沈殿したDNAをペレット化します。上清を取り除きます。ペレットを70% EtOH1 mLで7,500 × g で10分間紳目して洗浄します。
- 残留EtOHを完全に除去します。ペレットを30〜45分間乾燥させます。
- ペレットを50μLの滅菌水に再懸濁します。
- NGS用のgDNAを調製します。
注:このCRISPRライブラリーのNGSプライマーは、Illuminaアダプター配列でgRNAを増幅するもので、以前の論文27で設計されています。サンプルごとに異なるリバースプライマーを使用すると、各サンプルがバーコード化され、シーケンシング中にサンプルをプールできます。- 次の反応混合物(総容量= 50 μL)を使用してPCR用のサンプルを調製します:ハイフィデリティPCRマスターミックス、2x:25 μL;プールされたテンプレートDNA、0.5 μg:1 μL;NGSライブラリフォワードプライマー、10 μM:1.25 μL;NGSライブラリリバースプライマー、10 μM:1.25 μL;DNaseフリー水:21.5μL。
- 次のサイクリング条件でPCRを実行します:サイクル1:98°Cで3分間変性します。サイクル2-23:変性98°Cで10秒間、63°Cで10秒間アニールし、72°Cで25秒間延長します。サイクル24:72°Cで2分間延長します。
- 各サンプルのPCR反応をプールし、製造元の指示に従ってPCR精製キットでPCR産物を精製します。
- 各サンプルについて3 μgのPCR産物を2%(w/v)アガロースゲルで泳ぎます。
- PCR産物(~260-270 bp)をゲルから取り出し、製造元の指示に従ってゲル抽出キットを使用してゲルからDNAを抽出します。抽出したサンプルは-20°Cで保存します。
- サンプルで次世代シーケンシング(NGS)を実行します。ライブラリ内のgRNAあたり500リード以上を目指します。
9. シーケンシング結果の解析
- NGSを解析するには、MAGeCK-VISPRプラットホーム28を使用します。最尤推定 (MAGeCK-MLE) アルゴリズムの標準設定を使用します。分析用に出力ファイルを準備するには、次のようにします。
- FASTQCを実行して、シーケンスファイルの品質を評価する31.
- bbmerge32 を使用して、各サンプルのペアエンド シーケンシング ファイルをマージします。
- MAGeCK-MLEアルゴリズムを使用して、MAGeCK-VISPRプラットフォームでサンプルを分析します。MAGeCK-MLE解析では、すべてのgRNAカウントを1,000の非ターゲティングgRNAのリストに正規化します。
- GCコンテンツ、ベース品質、シーケンシングリード、およびマッピングリードを評価することにより、シーケンシングファイルの品質を評価します。
- Gini指数、主成分分析(PCA)クラスタリング、および陰性に選択された遺伝子のリストに関連する経路の変化を評価することにより、CRISPRスクリーニングの選択圧段階の成功を評価する32。
- 文献検索とパスウェイ分析を行うことにより、上位のヒットを調査します。
結果
CART細胞の活性を偏りなく改善するために編集可能な遺伝子と経路を調べるために、in vitroゲノムワイドCRISPRノックアウトスクリーンを設計しました(図1)。このスクリーニングには、CART細胞産生期と選択圧期の2つのフェーズがあります。CART細胞の産生段階では、少なくとも110×106 T細胞が最初に健康なドナーPBMCから単離され、CD3
ディスカッション
遺伝子編集は、治療に対する耐性のメカニズムを理解するだけでなく、CART細胞の寿命と活性を改善するための新しいCART細胞療法を設計する上でも強力なツールとなっている16,17,26。一部の遺伝子編集戦略では、前臨床モデルと臨床試験の両方でCART細胞活性の改善が示されていますが、遺伝子編集...
開示事項
SSKは、ノバルティス社(メイヨークリニック、ペンシルベニア大学、ノバルティス社との契約)、ヒューマニゲン社(メイヨークリニック社)、メタフォージ社(メイヨークリニック社)、マスタングバイオ社(メイヨークリニック社)、カイマールセラピューティクス社(メイヨークリニック社)にライセンス供与されたCAR免疫療法分野の特許の発明者です。CS、CMR、およびSSKは、Immix Biopharmaにライセンス供与された特許の発明者です。SSKは、Kite、Gilead、Juno、BMS、Novartis、Humanigen、MorphoSys、Tolero、Sunesis/Viracta、LifEngine Animal Health Laboratories Inc.、Lentigenから研究資金を受けています。SSKは、Kite/Gilead、Calibr、Luminary Therapeutics、Humanigen、Juno/BMS、Capstan Bio、Novartisとのアドバイザリーミーティングに参加しています。SSKは、HumanigenおよびCarismaとともに、データの安全性および監視委員会の委員を務めてきました。SSKは、Torque、Calibr、Novartis、Capstan Bio、BMS、Carisma、Humanigenのコンサルタントを解雇しました。CMSとSSKは、このプロトコルから生まれた知的財産の発明者です。
謝辞
この研究は、Mayo Clinic Center for Individualized Medicine(SSK)、Mayo Clinic Comprehensive Cancer Center(SSK)、Mayo Clinic Center for Regenerative Biotherapeutics(SSK)、National Institutes of Health K12CA090628(SSK)およびR37CA266344-01(SSK)、Department of Defense grant CA201127(SSK)、Predolin Foundation(SSK)、およびMinnesota Partnership for Biotechnology and Medical Genomics(SSK)から一部資金提供を受けました。CMSは、メイヨークリニック生物医科学研究科の支援を受けています。CRISPRの画面図(図1)は、BioRender.com(Siegler, L. (2022) https://BioRender.com/k71r054)を用いて作成しました。
資料
| Name | Company | Catalog Number | Comments |
| 293T cells | ATCC | CRL-3216 | Cells used for lentivirus production |
| Biotin ProteinL Antibody | GenScript | M00097 | anti-kappa chain antibody for CAR detection |
| Bovine Serum Albumin | Millipore Sigma | A7906 | |
| Carbenicillin disodium salt | Millipore Sigma | C1389-1G | Carbenicillin antibiotic |
| CD4 Isolation Beads | Miltenyi Biotec | 130-045-101 | |
| CD8 Isolation Beads | Miltenyi Biotec | 130-045-201 | |
| CTS (Cell Therapy Systems) Dynabeads CD3/CD28 | Gibco | 40203D | |
| Cytoflex | Beckman Coulter | NC2279958 | |
| DNase-Free Water | Invitrogen | AM9937 | |
| Dulbecco's modified eagle's medium (DMEM) | Corning | 10-017-CV | |
| Dulbecco's Phosphate-Buffered Saline | Gibco | 14190-144 | |
| EasySep Human T Cell Isolation Kit | STEMCELL Technologies | 17951RF | Negative isolation kit |
| Endura Electrocompetent Cells | Biosearch Technologies | 60242-1 | Electrocompetent cells with recovery medium |
| Ethanol | Millipore Sigma | E7023 | |
| Fetal bovine serum (FBS) | Corning | 35-010-CV | |
| GeCKO v2 CRISPR Knockout Pooled Library A | AddGene | 1000000048 | CRISPR library plasmid |
| Gene Pulser II | Bio-Rad | 165-2105 | Electroporator |
| Glycogen | Millipore Sigma | 10901393001 | |
| JeKo-1 | ATCC | CRL-3006 | CD19+ target cells |
| Lipofectamine 3000 Transfection Reagent | ThermoFisher Scientific | L3000075 | Transfection reagent kit with a transfection reagent (Lipofectamine 3000 Reagent) and a neutral co-lipid reagent (p3000) |
| LIVE/DEAD Aqua | Invitrogen | L34966 | |
| Lymphoprep | STEMCELL Technologies | 7851 | Density gradient medium |
| Machery-Nagel NucleoBond Xtra Maxi Kits | ThermoFisher Scientific | 12748412 | Maxi-prep kit |
| NEBNext High-Fidelity 2X PCR MasterMix | New England BioLabs | M0541S | High fidelity PCR mastermix |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985-070 | Reduced serum medium |
| pCMVR8.74 | AddGene | 22036 | Lentiviral packaging plasmid |
| Pennicillin-streptomycin-glutamine (100X) | Life Technologies | 10378-016 | |
| pMD2.G | AddGene | 12259 | VSV-G envelope expressing plasmid |
| Pooled Human AB Serum | Innovative Research | ISERABHI | |
| Puromycin | Millipore Sigma | P8833 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | Gek extraction kit |
| Qucik-DNA Midiprep Plus Kit | Zymo Research | D4075 | Kit used to isolate gDNA |
| RoboSep-S | STEMCELL Technologies | 21000 | Automated cell separator |
| Roswell Park Memorial Institute 1640 Medium (RPMI) | Gibco | 21870092 | |
| SepMate-50 | STEMCELL Technologies | 85450 | Density gradient separation tube |
| Sodium Acetate | Invitrogen | AM9740 | |
| Sodium Azide | Fisher Scientific | 71448-16 | |
| Streptavidin Antibody (PE) | BioLegend | 405203 | Secondary antibody used for CAR detection |
| T100 Thermal Cycler | Bio-Rad | 1861096 | |
| Ultracentrifuge (Optima XPN-80) | BeckmanCoulter | A99839 | |
| Vacuum Filter Systems, 0.22 µm | ThermoFisher Scientific | 567-0020 | |
| Vacuum Filter Systems, 0.45 µm | ThermoFisher Scientific | 165-0045 | |
| X-VIVO 15 Serum-Free Hematopoietic Cell Medium | Lonza | 04-418Q | Hematopoietic cell medium |
参考文献
- Neelapu, S. S., et al. 5-year follow-up supports curative potential of axicabtagene ciloleucel in refractory large B-cell lymphoma (zuma-1). Blood. 141 (19), 2307-2315 (2023).
- Neelapu, S. S., et al. Axicabtagene ciloleucel CAR T-cell therapy in refractory large B-cell lymphoma. N Engl J Med. 377 (26), 2531-2544 (2017).
- Locke, F. L., et al. Long-term safety and activity of axicabtagene ciloleucel in refractory large B-cell lymphoma (zuma-1): A single-arm, multicentre, phase 1-2 trial. Lancet Oncol. 20 (1), 31-42 (2019).
- Maude, S. L., et al. Tisagenlecleucel in children and young adults with B-cell lymphoblastic leukemia. N Engl J Med. 378 (5), 439-448 (2018).
- Schuster, S. J., et al. Tisagenlecleucel in adult relapsed or refractory diffuse large B-cell lymphoma. N Engl J Med. 380 (1), 45-56 (2018).
- Bai, Z., et al. Single-cell antigen-specific landscape of CAR T infusion product identifies determinants of CD19-positive relapse in patients with all. Sci Adv. 8 (23), eabj2820 (2022).
- Fraietta, J. A., et al. Determinants of response and resistance to CD19 chimeric antigen receptor (CAR) T cell therapy of chronic lymphocytic leukemia. Nat Med. 24, 563-571 (2018).
- Gennert, D. G., et al. Dynamic chromatin regulatory landscape of human CAR T cell exhaustion. Proc Natl Acad Sci USA. 118 (30), e2104758118 (2021).
- Deng, Q., et al. Characteristics of anti-CD19 CAR T cell infusion products associated with efficacy and toxicity in patients with large B cell lymphomas. Nat Med. 26 (12), 1878-1887 (2020).
- Sterner, R. M., Cox, M. J., Sakemura, R., Kenderian, S. S. Using CRISPR/CAS9 to knock out GM-CSF in CAR-T cells. J Vis Exp. (149), e59629 (2019).
- Sterner, R. M., et al. GM-CSF inhibition reduces cytokine release syndrome and neuroinflammation but enhances CAR-T cell function in xenografts. Blood. 133 (7), 697-709 (2019).
- Cox, M. J., et al. GM-CSF disruption in CART cells modulates T cell activation and enhances CART cell anti-tumor activity. Leukemia. 36 (6), 1635-1645 (2022).
- Ottaviano, G., et al. Phase 1 clinical trial of CRISPR-engineered CAR19 universal T cells for treatment of children with refractory B cell leukemia. Sci Transl Med. 14 (668), eabq3010 (2022).
- Joung, J., et al. Genome-scale CRISPR-CAS9 knockout and transcriptional activation screening. Nat Protoc. 12, 828-863 (2017).
- Mccutcheon, S. R., et al. Transcriptional and epigenetic regulators of human CD8+ T cell function identified through orthogonal CRISPR screens. Nat Genet. 55, 2211-2223 (2023).
- Zhu, W., Kelly, C., Dagur, P., Dunbar, C. E., Cordes, S. CRISPR activation screen to optimize chimeric antigen receptor (CAR) T cell immunophenotype. Blood. 142 (Supplement 1), 4820-4820 (2023).
- Wang, D., et al. CRISPR screening of CAR T cells and cancer stem cells reveals critical dependencies for cell-based therapies. Cancer Discov. 11 (5), 1192-1211 (2021).
- Shifrut, E., et al. Genome-wide CRISPR screens in primary human T cells reveal key regulators of immune function. Cell. 175 (7), 1958-1971.e15 (2018).
- Belk, J. A., et al. Genome-wide crispr screens of T cell exhaustion identify chromatin remodeling factors that limit T cell persistence. Cancer Cell. 40 (7), 768-786.e7 (2022).
- Jiang, P., et al. Single-cell ATAC-seq maps the comprehensive and dynamic chromatin accessibility landscape of CAR-T cell dysfunction. Leukemia. 36 (11), 2656-2668 (2022).
- Rossi, J., et al. Preinfusion polyfunctional anti-CD19 chimeric antigen receptor T cells are associated with clinical outcomes in NHL. Blood. 132 (8), 804-814 (2018).
- Beider, K., et al. Molecular and functional signatures associated with CAR T cell exhaustion and impaired clinical response in patients with B cell malignancies. Cells. 11 (7), 1140 (2022).
- Wherry, E. J. T. cell exhaustion. Nat Immunol. 12, 492-499 (2011).
- Prinzing, B., et al. Deleting DNMT3A in CAR T cells prevents exhaustion and enhances antitumor activity. Sci Transl Med. 13, eabh0272 (2021).
- Lynn, R. C., et al. C-jun overexpression in CAR T cells induces exhaustion resistance. Nature. 576, 293-300 (2019).
- Tang, N., et al. TGF-beta inhibition via CRISPR promotes the long-term efficacy of CAR T cells against solid tumors. JCI Insight. 5 (4), e133977 (2020).
- Joung, J., et al. Genome-scale CRISPR-CAS9 knockout and transcriptional activation screening. Nat Protoc. 12 (4), 828-863 (2017).
- Li, W., et al. Quality control, modeling, and visualization of CRISPR screens with MAGeCK-VISPR. Genome Biol. 16, 281 (2015).
- Sakemura, R., et al. Targeting cancer-associated fibroblasts in the bone marrow prevents resistance to CART-cell therapy in multiple myeloma. Blood. 139 (26), 3708-3721 (2022).
- Stewart, C. M., et al. IL-4 drives exhaustion of CD8+ CART cells. Nat Commun. 15 (1), 7921 (2024).
- . FastQC: A quality control tool for high throughput sequence data Available from: https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010)
- . Bbmerge Available from: https://github.com/BioInfoTools/BBMap/blob/master/sh/bbmerge.sh (2019)
- Pattanayak, V., et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed CAS9 nuclease specificity. Nat Biotechnol. 31 (9), 839-843 (2013).
- Doench, J. G., et al. Rational design of highly active sgRNAs for CRISPR-CAS9-mediated gene inactivation. Nat Biotechnol. 32 (12), 1262-1267 (2014).
- Dong, M. B., et al. Systematic immunotherapy target discovery using genome-scale in vivo CRISPR screens in CD8 T cells. Cell. 178 (5), 1189-1204.e23 (2019).
- Zhou, P., et al. Single-cell CRISPR screens in vivo map T cell fate regulomes in cancer. Nature. 624 (7990), 154-163 (2023).
- Sutra Del Galy, A., et al. In vivo genome-wide CRISPR screens identify SOCS1 as intrinsic checkpoint of CD4+ TH1 cell response. Sci Immunol. 6 (66), eabe8219 (2021).
- Ramos, A., et al. Leukemia-intrinsic determinants of CAR-T response revealed by iterative in vivo genome-wide CRISPR screening. Nat Commun. 14 (1), 8048 (2023).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved