Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Réalisation d’un criblage CRISPR in vitro à l’échelle du génome dans des cellules T à récepteur d’antigène chimérique
Dans cet article
Résumé
L’article décrit un protocole pour l’application d’un modèle in vitro d’épuisement afin de réaliser un dépistage CRISPR knock-out à l’échelle du génome dans des cellules T à récepteur antigénique chimérique de donneur sain.
Résumé
La thérapie cellulaire à récepteur antigénique chimérique T (CART) est une forme innovante d’immunothérapie ciblée qui a révolutionné le traitement du cancer. Cependant, la réponse durable reste limitée. Des études récentes ont montré que le paysage épigénétique des produits cellulaires CART de pré-perfusion peut influencer la réponse au traitement, et l’édition de gènes a été proposée comme solution. Cependant, il reste encore du travail à faire pour déterminer la stratégie optimale d’édition génomique. Les cribles CRISPR à l’échelle du génome sont devenus des outils populaires pour étudier les mécanismes de résistance et optimiser les stratégies d’édition de gènes. Pourtant, leur application aux cellules primaires présente de nombreux défis. Nous décrivons ici une méthode permettant de réaliser un criblage CRISPR à l’échelle du génome dans des cellules CART provenant de donneurs sains. En tant que modèle de preuve de concept, nous avons conçu cette méthode pour étudier le développement de l’épuisement dans les cellules CART ciblant l’antigène CD19. Cependant, nous pensons que cette approche peut être utilisée pour aborder une variété de mécanismes de résistance au traitement dans différentes constructions CAR et modèles tumoraux.
Introduction
La thérapie cellulaire à récepteur T antigénique chimérique (CART) a montré un succès impressionnant dans le traitement des tumeurs malignes à cellules B ; Cependant, la réponse durable est limitée à 30-40 %1,2,3,4,5. Bien que les chercheurs aient développé et testé plusieurs approches pour s’attaquer aux mécanismes de résistance à la thérapie cellulaire CART, y compris l’optimisation de la conception des CAR, l’édition de gènes et les thérapies combinées, le développement de la résistance reste largement inconnu. Récemment, il y a eu de plus en plus de preuves que le profil d’expression génique de base des produits cellulaires CART avant la perfusion est un déterminant important de la toxicité et de l’efficacité du traitement CART 6,7,8,9. En tant que telle, l’édition génomique pendant la production de cellules CART est devenue une approche populaire pour améliorer le succès de la thérapie cellulaire CART.
Par exemple, notre laboratoire a précédemment montré que l’utilisation de la technologie Cas9 Cas9 pour éliminer le gène du facteur de stimulation des colonies de granulocytes-macrophages (GM-CSF) améliore l’activité des cellules CART et réduit les signes de toxicité associée à CART 10,11,12. De plus, la thérapie cellulaire conçue par CRISPR a été testée dans des essais cliniques et a montré son efficacité et son innocuité13. L’ensemble de ces éléments indique que l’édition génomique avec la technologie CRISPR peut non seulement améliorer notre compréhension de la biologie cellulaire CART, mais aussi générer des produits cellulaires CART traduisibles.
Les cribles CRISPR à l’échelle du génome sont devenus des outils de plus en plus populaires dans la recherche en biologie du cancer pour comprendre les mécanismes de résistance aux traitements. Dans cette technique, des dizaines de milliers d’ARNg guides (ARNg) sont délivrés à des pools de cellules pour encourager l’entrée d’un ARNg par cellule14. Ensuite, les cellules subissent des conditions induisant une pression où les cellules transduites avec des ARNg ciblant des gènes essentiels meurent et les cellules transduites avec des ARNg ciblant des gènes inhibiteurs survivent et prolifèrent. En utilisant le séquençage de nouvelle génération, nous pouvons déterminer comment la représentation de l’ARNg change dans l’écran CRISPR14.
Cependant, l’échelle et le temps de sélection nécessaires pour les perturbations génétiques à l’échelle du génome peuvent être difficiles à réaliser dans les cellules primaires, comme les cellules CART. À ce titre, les groupes ont utilisé des cribles CRISPR ciblés pour mieux comprendre les mécanismes de résistance thérapeutique15,16. Les criblages ciblés sont souvent plus faciles à réaliser dans les cellules primaires, car ils comportent des banques plus petites qui nécessitent moins de cellules pour obtenir une représentation adéquate des bibliothèques. Bien que ces études aient amélioré notre compréhension des mécanismes de résistance à la thérapie cellulaire CART, les criblages ciblés introduisent un biais dû à la sélection manuelle des cibles géniques. Cet article cherche à décrire une méthode pour réaliser un criblage CRISPR in vitro à l’échelle du génome dans des cellules CART provenant de donneurs sains. En tant que telle, cette approche à haut débit permet une identification efficace et impartiale des voies et des gènes clés qui peuvent être modifiés pour améliorer les réponses thérapeutiques 17,18,19.
En particulier, le protocole décrit dans cet article est conçu pour accroître la compréhension du domaine de l’épuisement des cellules CART en complétant le criblage CRISPR knock-out à l’échelle du génome avec un modèle in vitro de l’épuisement. L’épuisement est un destin dysfonctionnel des cellules CART qui a été impliqué dans la non-réponse à la thérapie cellulaire CART 20,21,22. Ce destin cellulaire est connu pour être régulé épigénétiquement, et il est caractérisé par une diminution de la prolifération des cellules CART, une diminution de la production de cytokines effectrices et une augmentation de l’expression des récepteurs inhibiteurs23. Dans la littérature antérieure, l’édition génomique a été capable d’empêcher le développement de l’épuisement en régulant à la hausse ou à la baisse les gènes clés 24,25,26. Compte tenu à la fois de la diminution de la capacité proliférative à mesure que les cellules CART s’épuisent et des preuves que l’édition de gènes peut empêcher son apparition, nous avons modélisé notre crible CRISPR in vitro à l’échelle du génome sur ce destin cellulaire. Cependant, ce protocole pourrait être modifié à l’avenir pour explorer d’autres mécanismes de résistance à la thérapie cellulaire CART.
Protocole
Il est important de noter que le protocole décrit ci-dessous suit les lignes directrices du Conseil d’examen institutionnel de la Mayo Clinic (IRB 18-005745) et du Comité de biosécurité institutionnel (IBC HIP00000252.43) et a reçu leur approbation. Tous les travaux de culture cellulaire, y compris la production de lentivirus, doivent être effectués dans une hotte de culture cellulaire avec un équipement de protection individuelle approprié. En particulier, les travaux sur les lentiviraux doivent être effectués dans le cadre de précautions de niveau de biosécurité 2 (BSL-2), y compris l’utilisation d’eau de Javel à 10 % pour désinfecter les articles avant de les éliminer.
1. Amplification de la bibliothèque CRISPR
- Amplifiez la bibliothèque CRISPR selon les recommandations du fabricant. En bref, électroporez la bibliothèque CRISPR en quatre aliquotes de cellules électrocompétentes en complétant ce qui suit :
- Ajouter 2 μL de plasmide de la banque CRISPR de 50 ng/μL à 25 μL de cellules électrocompétentes dans une cuvette de 1,0 mm.
- Électroporez les échantillons à l’aide des réglages suivants : 10 μF, 600 Ohms, 1 800 Volts et une constante de temps comprise entre 3,5 ms et 4,5 ms.
- Récupérer dans 975 μL de milieu de récupération et transférer dans un tube avec 1 mL supplémentaire de milieu de récupération.
- Faites pivoter toutes les aliquotes à 250 tr/min pendant 1 h à 37 °C.
- Plaquez les transformations :
- Regroupez toutes les aliquotes de cellules électroporées et mélangez bien.
- Plaquez toutes les cellules électroporées en étalant 400 μL du mélange de transformation sur des plaques de bouillon Luria (LB) préchauffées de 10 cm avec 100 μg/mL de carbénicline. Placez la plaque dans un incubateur à 32 °C pendant 14 h.
- Récolter les colonies :
- Pipeter 1 mL de support LB + 100 μg/mL de carbénicilline sur chaque plaque. Grattez les colonies de chaque assiette et rassemblez-les dans un tube conique de 50 ml.
- Ajouter 1 mL de milieu LB + 100 μg/mL de carbénicilline dans chaque assiette à laver et assurer une récupération complète.
- Effectuez une maxi-préparation en suivant les instructions d’un kit de maxi-préparation.
- Mettez en commun l’ADN plasmidique résultant.
2. Séquençage de nouvelle génération pour vérifier la représentation de base de l’ARNg dans la bibliothèque CRISPR
REMARQUE : Les amorces NGS de cette bibliothèque CRISPR ont été conçues dans une publication précédente27. L’utilisation d’une amorce inverse différente pour chaque échantillon permettra d’coder chaque échantillon à barres et de regrouper les échantillons pendant le séquençage.
- Préparez les échantillons pour la PCR avec le mélange réactionnel suivant (volume total = 50 μL) : mélange maître PCR haute fidélité, 2x : 25 μL ; ADN matrice regroupé, 0,5 μg : 1 μL ; amorce directe de la bibliothèque NGS, 10 μM : 1,25 μL ; amorce inverse de la banque NGS, 10 μM : 1,25 μL ; Eau sans DNase : 21,5 μL.
- Effectuer une PCR dans les conditions de cycle suivantes : cycle 1 : dénaturation à 98 °C pendant 3 min ; cycles 2 à 23 : dénaturation à 98 °C pendant 10 s, recuit à 63 °C pendant 10 s, extension à 72 °C pendant 25 s ; cycle 24 : étendre à 72 °C pendant 2 min.
- Purifiez la réaction PCR en regroupant les réactions PCR pour chaque échantillon et en purifiant le produit PCR avec un kit de purification PCR selon les instructions du fabricant.
- Analysez 3 μg du produit PCR pour chaque échantillon sur un gel d’agarose à 2 % (p/v).
- Retirez le produit PCR (~260-270 pb) du gel en extrayant l’ADN du gel à l’aide d’un kit d’extraction de gel conformément aux instructions du fabricant. Visez au moins 500 ng d’ADN.
- Conservez les échantillons extraits à -20 °C.
- Effectuez un séquençage de nouvelle génération (NGS) avec les échantillons et visez 100 lectures par ARNg27.
- Analysez les résultats de séquençage avec le pipeline MAGeCK-VISPR et assurez-vous que plus de 99,5 % des ARNg de la bibliothèque sont représentés28.
3. Production de lentivirus de la bibliothèque CRISPR et de lentivirus d’expression CAR
- Préparez le lentivirus comme décrit précédemment 10,29.
- Jour 1 : Commencez avec des flacons T175 de 80 à 90 % de cellules 293T confluentes qui ont été cultivées dans un milieu D10 composé de 10 % de sérum de veau fœtal (FBS) (v/v), de 1 % de pénicilline-streptomycine et de glutamine (PSG) (v/v) et de milieu d’aigle modifié de Dulbecco. Avant de commencer, réchauffez les réactifs de transfection et l’ADN à température ambiante et quantifiez l’ADN.
- Pour chaque fiole de 293T, étiqueter deux tubes coniques A et B de 50 mL. Ajouter 4,5 ml de milieu sérique réduit dans le tube A et le tube B.
- Dans un tube de microcentrifugation stérile de 1,5 mL, mélanger l’ADN suivant et laisser reposer pendant 1 min : 18 μg de pCMVR8.74 (gag, pol, tat et rev), 7 μg de pMD2.G (VSV-G) et 15 μg de vecteur d’expression lentiviral (banque CRISPR A ou vecteurs CAR19-28ζ).
- Ajouter 129 μL de réactif de transfection dans le tube A. Mélanger le tube A bien par vortex de doigt.
- Ajoutez le mélange d’ADN dans le tube B. Bien mélanger le tube B par vortex à doigts.
- Ajouter 111 μL de réactif colipidique neutre dans le tube B. Bien mélanger le tube B par vortex à doigts.
- Ajouter le contenu du tube A au tube B et bien mélanger par vortex au doigt. Incuber le tube B pendant 30 min à température ambiante.
- Vers la fin de la période d’incubation de 30 minutes, préparez les cellules 293T en aspirant le milieu existant. Ajouter 16 ml de milieu R10 composé de 10 % de FBS (v/v), de 1 % de PSG (v/v) et de Roswell Park Memorial Institute (RPMI) 1640.
- Après la période d’incubation de 30 minutes, ajouter le contenu du tube B (environ 9 mL) goutte à goutte dans le ballon de cellules 293T. Posez le ballon vers le bas et tournez-le doucement avant de l’incuber à 37 °C, 5 % de CO2.
- Le jour 2 (environ 24 h après la transfection), prélever le lentivirus comme suit :
REMARQUE : Prérefroidissez tout l’équipement et le matériel et effectuez les opérations suivantes sur de la glace pour garder le surnageant viral froid.- Transvaser le surnageant des fioles 293T transfectées dans des tubes coniques prérefroidis de 50 mL.
- Ajouter 30 ml de média R10 préchauffé dans les flacons de 293T.
- Granulez les débris cellulaires en centrifugeant les tubes coniques de 50 mL à 900 × g pendant 10 min à 4 °C dans des rotors fermés.
- Après la centrifugation, filtrez le surnageant avec un filtre de 0,45 μm. Faire tourner le filtrat dans une ultracentrifugeuse à 112 700 × g à 4 °C pendant 2 h.
- Après 2 h d’essorage, aspirer le surnageant jusqu’à ce qu’il reste environ 5 ml. Remettez en suspension les particules virales en pipetant doucement de haut en bas 10x. Conservez la suspension virale à 4 °C pendant la nuit.
- Le jour 3, prélever et stocker le lentivirus comme suit :
- Transvaser le surnageant des fioles 293T transfectées dans des tubes coniques prérefroidis de 50 mL.
- Ajouter 30 ml d’eau de Javel à 10 % dans les flacons de 293T.
- Granuler les débris cellulaires dans le surnageant en centrifugeant les tubes coniques de 50 mL à 900 × g pendant 10 min à 4 °C dans des rotors fermés.
- Après la centrifugation, filtrez le surnageant avec un filtre de 0,45 μm. Faire tourner le filtrat dans une ultracentrifugeuse à 112 700 × g à 4 °C pendant 2 h.
- Après la centrifugeuse de 2 h, aspirer le surnageant jusqu’à ce qu’il reste environ 2 mL, et remettre en suspension en pipetant doucement de haut en bas 10x. Stocker le surnageant viral dans des aliquotes de 300 à 500 μL et conserver à -80 °C.
4. Calcul du titre viral pour le lentivirus exprimant le CAR
REMARQUE : Le lentivirus CAR a été titré comme décrit précédemment10,12.
- Le jour 1, activez et mettez en plaque les lymphocytes T.
- Isolez 1 × 106 lymphocytes T de PBMC de donneurs sains à l’aide d’un kit de sélection négative.
- Comptez les lymphocytes T.
- Lavez suffisamment de billes CD3+/CD28+ pour stimuler les cellules T à un rapport perles :cellules de 3:1. Par exemple, pour stimuler 1 × 106 cellules T, lavez 3 × 106 perles. Pour ce faire, lavez les perles en complétant les 3 fois suivantes :
- Utilisez la séparation magnétique pour retirer les billes de la suspension.
- Retirez le surnageant.
- Ajoutez 1000 μL de milieu de cellules T (MTC) composé de 10 % de sérum humain (v/v), de 1 % de PSG (v/v) et de milieu cellulaire hématopoïétique.
REMARQUE : La MTC doit être stérilisée avant utilisation en filtrant avec un filtre à vide stérile de 0,45 μm, puis avec un filtre à vide stérile de 0,22 μm. - Ajoutez des billes à la suspension à cellules T à un rapport bille :cellule de 3:1.
- Remettre les lymphocytes T en suspension à 1 × 106 cellules/mL dans la MTC.
- Ajoutez 100 μL de cellules T activées dans 10 puits d’une plaque de 96 puits. Incuber la plaque à 37 °C, 5 % CO2.
- Le jour 2, ajouter une dilution du lentivirus CAR en créant une plaque de dilution comme suit :
- Ajoutez 100 μL de TCM dans sept puits d’une plaque de 96 puits.
- Au premier puits, ajouter 50 μL du virus d’intérêt ; Bien mélanger.
- Diluer davantage le virus en série en ajoutant 50 μL du puits 1 au puits 2 ; Bien mélanger.
- Ajoutez 50 μL du puits 2 au puits 3 et continuez ce schéma jusqu’au puits 7.
- Après avoir mélangé le puits 7, retirez 50 μL. Assurez-vous que tous les puits ont un volume total de 100 μL.
- Ajoutez 50 μL de chaque puits de la plaque de dilution aux sept premiers puits de la plaque de cellules T à partir du jour 1. Bien mélanger.
- Dans les trois derniers puits de la plaque de lymphocytes T, ajoutez 50 μL de TCM ; Bien mélanger. Conserver la plaque dans un incubateur à 37 °C, 5 % CO2.
- Le jour 3, nourrissez les cellules en ajoutant 100 μL de MTC dans tous les puits de la plaque de cellules T ; Bien mélanger.
- Le jour 4, effectuez une cytométrie en flux pour détecter le pourcentage de cellules exprimant le CAR avec un anticorps anti-chaîne kappa, un marqueur de substitution de l’expression du CAR, en complétant les opérations suivantes :
- Faites tourner la plaque de cellules T à 1 000 × g pendant 3 minutes et décantez.
- Laver la plaque 3 fois avec un tampon d’anticorps anti-chaîne kappa (composé de 4 % d’albumine sérique bovine dans du PBS (p/v)) en ajoutant 200 μL de tampon d’anticorps anti-chaîne kappa, en faisant tourner la plaque à 1 000 × g pendant 3 min et en décantantant.
- Ajoutez un anticorps anti-chaîne kappa à la biotine dans chaque puits. Incuber pendant 45 min sur une bascule à 4 °C.
- Lavez la plaque 3 fois avec un tampon d’anticorps anti-chaîne kappa comme à l’étape 4.4.2.
- Colorez les cellules pour la cytométrie en flux avec un anticorps anti-streptavidine conjugué au fluorochrome et un anticorps vivant/mort. Incuber les échantillons pendant 15 min à température ambiante dans l’obscurité.
- Laver les échantillons une fois dans un tampon d’écoulement (composé de 2 % de FBS (v/v) et de 1 % d’azoture de sodium (v/v) dans du PBS) en ajoutant 200 μL de tampon d’écoulement, en faisant tourner la plaque à 1 000 × g pendant 3 min et en transvasant.
- Remettre en suspension dans 100 μL de tampon d’écoulement et exécuter sur le cytomètre.
- Effectuez un gating négatif avec les trois puits non transduis.
5. Calcul du titre viral pour le lentivirus de la banque CRISPR
REMARQUE : Ce protocole décrit un nombre minimum de lymphocytes T pour titrer le lentivirus de la banque CRISPR, mais il peut être mis à l’échelle pour accueillir plus de lymphocytes T et donc des volumes plus élevés de virus.
- Le jour 0, activez les lymphocytes T en ajoutant 18 × 106 billes CD3+/CD28+ (rapport perles :cellules de 3:1) à 6 × 106 lymphocytes T et en portant le volume final à 6 mL avec TCM. Aliquote 0,5 mL de la suspension de cellules T dans 12 puits d’une plaque de 48 puits. Conserver dans un incubateur à 37 °C, 5 % CO2.
- Vingt-quatre heures plus tard, le jour 1, ajoutez le virus CAR à une multiplicité d’infection (MOI) de 3 à chaque puits. Bien mélanger en pipetant plusieurs fois de haut en bas. Conserver dans un incubateur à 37 °C, 5 % CO2.
- Quarante-huit heures plus tard, le jour 2, ajoutez une série de volumes de virus de la bibliothèque CRISPR aux cellules CART activées en ajoutant l’un des volumes suivants de virus CRISPR à chaque puits (0 μL, 0 μL, 6,25 μL, 6,25 μL, 12,5 μL, 12,5 μL, 25 μL, 25 μL, 50 μL, 50 μL, 50 μL, 100 μL ou 100 μL). Bien mélanger en pipetant plusieurs fois de haut en bas. Conserver dans un incubateur à 37 °C, 5 % CO2.
- Le jour 3, ajouter 1 μg de puromycine par mL de milieu dans la moitié des puits, y compris un puits qui a reçu chacun des volumes de virus CRISPR suivants (0 μL, 6,25 μL, 12,5 μL, 25 μL, 50 μL et 100 μL). Les autres puits ne seront pas traités avec de la puromycine.
- Du 4e au 8e jour, compter les lymphocytes T quotidiennement et ajuster la densité cellulaire finale à 1 × 106 cellules /mL avec 1 μg/mL de puromycine dans la MTC pour les puits de traitement de la puromycine et avec la MTC pour les puits non traités.
- Le jour 8, comptez le nombre de cellules vivantes par puits et calculez les unités de transduction/mL de surnageant viral à l’aide de l’équation suivante :
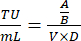
Où A : Le nombre de cellules vivantes est bien transduit par le virus ; B : Nombre de cellules vivantes dans le puits de contrôle sans sélection d’antibiotiques ; V : Volume de virus utilisé pour l’infection dans chaque puits ; D : Pli de dilution. - À l’aide du puits TU/mL calculé à partir d’un puits montrant une efficacité de transduction d’environ 20 %, calculez le volume de virus nécessaire pour transduire 1 × 106 cellules à un moment d’inertie de 0,3 en utilisant la formule suivante :
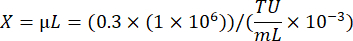
6. Phase de production de cellules CART de l’écran CRISPR (jours 0 à 8)
REMARQUE : Le protocole qui suit décrit la réalisation du criblage CRISPR dans une répétition biologique. Cependant, ce protocole a déjà été validé avec trois réplicats biologiques et nous recommandons l’utilisation d’au moins trois réplicats biologiques pour les autres qui complètent ce protocole30.
- Jour 0 : Isolez et stimulez les lymphocytes T du sang de donneur sain, comme décrit précédemment10.
- Isolez les cellules mononucléées du sang périphérique (PBMC) à partir de sang de donneur sain anonymisé.
- Ajouter 15 mL d’un milieu à gradient de densité dans un tube de séparation à gradient de densité.
- Ajoutez du sang dilué 1:2 dans le haut du tube de séparation à gradient de densité. Centrifuger à 1 200 × g pendant 10 min à température ambiante.
REMARQUE : Le sang doit être dilué avec un tampon d’isolement (2 % de FBS (v/v) dans une solution saline tamponnée au phosphate [PBS]). - Versez la couche supérieure dans un nouveau tube conique de 50 ml et doublez le volume avec un tampon d’isolement. Centrifuger l’échantillon à 300 × g pendant 8 min à 4 °C. Aspirez le surnageant.
- Remettez en suspension la pastille cellulaire avec un tampon d’isolement et comptez les PBMC.
- Isolez les lymphocytes T des PBMC à l’aide d’un kit de sélection négative. Suivez les instructions du fabricant pour le kit.
REMARQUE : Supposons une récupération de 10 à 20 % des lymphocytes T des PBMC. - Stimulez les lymphocytes T avec des billes CD3+/CD28+ .
- Comptez les lymphocytes T.
- Aliquote suffisamment de billes CD3+/CD28+ pour stimuler les lymphocytes T à un rapport perle :cellule de 3:1. Par exemple, pour stimuler 110 × 106 lymphocytes T, aliquote 330 × 106 billes. Lavez les billes 3 fois en les séparant d’abord de la suspension par séparation magnétique, puis en ajoutant 1 000 μL de TCM.
- Ajoutez les billes à la suspension à cellules T. Remettre les lymphocytes T en suspension à 1 × 106 cellules/mL dans la MTC.
- Aliquote des cellules T en plaques à 6 puits pour une meilleure efficacité de transduction. Ajouter 5 × 106 lymphocytes T dans chaque puits. Incuber les cellules à 37 °C, 5 % CO2 pendant 24 h.
- Isolez les cellules mononucléées du sang périphérique (PBMC) à partir de sang de donneur sain anonymisé.
- Jour 1 : Dans des conditions BSL-2+, décongeler suffisamment de lentivirus CAR pour transduire les lymphocytes T à une multiplicité d’infection de 3,0. Brisez doucement les rosettes des lymphocytes T avant d’ajouter la quantité appropriée de lentivirus mélangé pour transduire 5 × 106 lymphocytes T à un moment d’inertie de 3,0. Incuber les cellules à 37 °C, 5 % CO2.
REMARQUE : Regroupez les lentivirus de plusieurs productions dans un seul tube avant de transduire les lymphocytes T. Cela garantit que tous les lymphocytes T reçoivent le même traitement. De plus, le lentivirus ne doit être décongelé qu’une seule fois. - Jour 2 : Dans des conditions BSL-2+, décongeler suffisamment de lentivirus CRISPR pour transduire les lymphocytes T à un moment d’inertie de 0,3. Brisez doucement les rosettes des lymphocytes T et remettez les lymphocytes T en suspension avant d’ajouter la quantité appropriée de lentivirus regroupé pour transduire 5 × 106 lymphocytes T à un moment d’inertie de 0,3. Incuber les cellules à 37 °C, 5 % CO2.
REMARQUE : Regroupez les lentivirus de plusieurs productions dans un seul tube avant de transduire les lymphocytes T. Cela garantit que tous les lymphocytes T reçoivent le même traitement. - Jours 3 à 5 : Remettre les cellules CART en suspension pour briser les rosettes et compter les cellules. Porter la concentration cellulaire finale à 1 × 106 cellules/mL en MTC avec 1 μg/mL de puromycine. Incuber les cellules à 37 °C, 5 % CO2.
- Jour 6 : Remettre les cellules CART en suspension pour briser les rosettes, retirer les billes CD3+/CD28+ par séparation magnétique, et compter les cellules CART. Ajouter la MTC avec 1 μg/mL de puromycine pour porter la concentration cellulaire finale à 1 × 106 cellules/mL et incuber les cellules à 37 °C, 5 % CO2. Déterminer l’expression du CAR par cytométrie en flux comme décrit à l’étape 4.4.
7. Phase de sélection de l’ARNg du criblage CRISPR (jours 8 à 22)
- Jour 8 : Conserver les échantillons pour le séquençage de nouvelle génération (NGS) et commencer la co-culture.
- Laver les cellules CART 3x dans la MTC en centrifugeant les échantillons à 300 × g pendant 5 min à 4 °C, en aspirant le surnageant et en remettant les échantillons en suspension dans 20 mL de MTC.
- Comptez les cellules CART.
- Conservez les échantillons pour le NGS en procédant comme suit :
- Aliquote suffisamment de cellules CART dans un tube conique de 50 mL pour maintenir une représentation d’ARNg de 500 cellules par ARNg (par exemple, aliquote environ 33 × 106 cellules CART si vous utilisez une banque CRISPR avec 65 383 ARNg). Centrifuger l’échantillon à 300 × g pendant 5 min à 4 °C.
- Remettre la pastille cellulaire en suspension dans 1 mL de PBS et transférer dans un tube de microcentrifugation de 2 mL. Lavez le tube conique de 50 ml avec 1 ml de PBS et transférez le lavage dans le tube de microcentrifugation de 2 ml.
- Centrifuger les échantillons à 500 × g pendant 5 min à 4 °C. Retirez le surnageant. Conservez les granulés de cellule à -20 °C.
- Commencer la co-culture en ajoutant des cellules CART à des cellules CD19+ JeKo-1 selon un rapport de 1:1. Par exemple, ajoutez 80 cellules CART × 106 à 80 cellules × 106 JeKo-1. Portez le volume total de la co-culture à 160 mL avec la TCM. Conservez les échantillons dans un incubateur à 37 °C, 5 % de CO2.
REMARQUE : Nous vous recommandons de commencer avec au moins 80 cellules CART × 106 pour maintenir la représentation de la bibliothèque et avoir suffisamment de cellules pour préserver les échantillons au jour 15 et au jour 22.
- Jour 10 : Pour restimuler les cellules CART, recueillir les co-cultures dans des tubes coniques de 50 mL et centrifuger les échantillons à 300 × g pendant 5 min à 4 °C. Remettre chaque échantillon en suspension dans 80 mL de MTC et ajouter 80 × 106 cellules JeKo-1 dans 80 mL de MTC. Conservez les échantillons dans un incubateur à 37 °C, 5 % de CO2.
- Jours 12 et 14 : Restimulez les cellules CART comme à l’étape 7.2.
- Jour 15 : Conserver les échantillons pour le NGS et restimuler les cellules CART.
- Isolez les cellules CART de la coculture en comptant d’abord les cellules, puis en combinant un kit de sélection positive pour les lymphocytes T CD4+ avec un kit de sélection positive pour les lymphocytes T CD8+ afin d’isoler les lymphocytes T des co-cultures CART-JeKo-1. Comptez les lymphocytes T.
- Conservez les échantillons pour le NGS en aliquotant 33 × 10cellules 6 CART dans un tube conique de 50 mL et en centrifugeant l’échantillon à 300 × g pendant 5 min à 4 °C. Remettre l’échantillon en suspension dans 1 mL de PBS et le transférer dans un tube de microcentrifugation de 2 mL. Lavez le tube conique de 50 ml avec 1 ml de PBS et transférez le lavage dans le tube de microcentrifugation de 2 ml. Centrifuger les échantillons à 500 × g pendant 5 min à 4 °C ; retirer le surnageant ; et conservez les granulés de cellule à -20 °C.
- Mettre en place des co-cultures pour poursuivre le modèle in vitro de l’épuisement en ajoutant 80 × 10cellules CART à 80 ×10 cellules JeKo-1. Ensuite, porter le volume total de la co-culture à 160 mL avec la MTC. Conservez les échantillons dans un incubateur à 37 °C, 5 % de CO2.
- Jours 17, 19 et 21 : Restimulez les cellules CART comme à l’étape 7.2.
- Jour 22 : Conservez les échantillons pour le NGS.
- Isolez les cellules CART de la co-culture en comptant d’abord les cellules. Ensuite, combinez un kit de sélection positive pour les lymphocytes T CD4+ avec un kit de sélection positive pour les lymphocytes T CD8+ afin d’isoler les lymphocytes T des co-cultures CART-JeKo-1. Comptez les lymphocytes T.
- Pour conserver les échantillons pour le NGS, répétez l’étape 7.4.2.
8. Préparation de l’ADN génomique pour le séquençage de nouvelle génération
REMARQUE : Pour maintenir une couverture adéquate, l’ADN génomique (ADNg) doit être isolé d’au moins 33 × 106 cellules CART. De plus, l’intégralité de l’ADNg de 33 × 106 cellules CART doit être préparée pour le séquençage.
- Isolez l’ADNg à partir de pastilles cellulaires congelées à l’aide d’un kit d’isolement d’ADNg.
- Purifier l’ADNg par précipitation d’éthanol.
- Prérefroidissez 100 % EtOH à -20 °C.
- L’aliquote a élué l’ADN de manière égale dans plusieurs tubes de microcentrifugation (200-250 μL). Ajouter à chaque aliquote les éléments suivants : deux volumes d’EtOH à 100 % (500 μL pour une aliquote de 250 μL ou 400 μL pour 200 μL) ; 0,1 volume d’acétate de sodium 3 M (25 μL pour une aliquote de 250 μL ou 20 μL pour 200 μL) ; et 1 μL de glycogène à 20 mg/mL. Incuber les échantillons pendant la nuit à -20 °C.
- Le deuxième jour, faites tourner les échantillons à 13 000 × g pendant 20 minutes pour granuler l’ADN précipité. Retirez le surnageant. Lavez les granulés avec 1 ml d’EtOH à 70 % en les essorant à 7 500 × g pendant 10 min.
- Éliminez complètement l’EtOH résiduel. Laissez sécher les granulés pendant 30 à 45 minutes.
- Remettre les granulés en suspension dans 50 μL d’eau stérile.
- Préparez l’ADNg pour le NGS.
REMARQUE : Les amorces NGS pour cette banque CRISPR qui amplifient les ARNg avec des séquences adaptatrices Illumina ont été conçues dans une publication précédente27. L’utilisation d’une amorce inverse différente pour chaque échantillon permettra d’coder chaque échantillon à barres et de regrouper les échantillons pendant le séquençage.- Préparez les échantillons pour la PCR avec le mélange réactionnel suivant (volume total = 50 μL) : mélange maître PCR haute fidélité, 2x : 25 μL ; ADN matrice regroupé, 0,5 μg : 1 μL ; amorce directe de la bibliothèque NGS, 10 μM : 1,25 μL ; amorce inverse de la banque NGS, 10 μM : 1,25 μL ; Eau sans DNase : 21,5 μL.
- Effectuer une PCR dans les conditions de cycle suivantes : cycle 1 : dénaturation à 98 °C pendant 3 min ; cycles 2 à 23 : Dénaturation à 98 °C pendant 10 s, recuit à 63 °C pendant 10 s, extension à 72 °C pendant 25 s ; cycle 24 : étendre à 72 °C pendant 2 min.
- Regroupez les réactions PCR pour chaque échantillon et purifiez le produit PCR avec un kit de purification PCR selon les instructions du fabricant.
- Analysez 3 μg du produit PCR pour chaque échantillon sur un gel d’agarose à 2 % (p/v).
- Retirez le produit PCR (~260-270 pb) du gel en extrayant l’ADN du gel à l’aide d’un kit d’extraction de gel conformément aux instructions du fabricant. Conservez les échantillons extraits à -20 °C.
- Effectuez un séquençage de nouvelle génération (NGS) avec les échantillons. Visez plus de 500 lectures par ARNg dans la bibliothèque.
9. Analyse des résultats de séquençage
- Pour analyser le NGS, utilisez la plateforme MAGeCK-VISPR28. Utilisez les paramètres standard de l’algorithme d’estimation du maximum de vraisemblance (MAGeCK-MLE). Pour préparer les fichiers de sortie pour l’analyse :
- Exécutez FASTQC pour évaluer la qualité du fichierde séquence 31.
- Fusionnez les fichiers de séquençage par paires pour chaque échantillon avec bbmerge32.
- Analysez les échantillons de la plateforme MAGeCK-VISPR avec l’algorithme MAGeCK-MLE. Pour l’analyse MAGeCK-MLE, normalisez tous les nombres d’ARNg à la liste des 1 000 ARNg non ciblés.
- Évaluez la qualité des fichiers de séquençage en évaluant le contenu GC, la qualité de base, les lectures de séquençage et les lectures cartographiées.
- Évaluez le succès de la phase de pression sélective du criblage CRISPR en évaluant les changements dans l’indice de Gini, le regroupement par analyse en composantes principales (ACP) et les voies associées à la liste des gènes sélectionnés négativement32.
- Interrogez les meilleurs résultats en effectuant des recherches documentaires et en effectuant une analyse des parcours.
Résultats
Pour interroger les gènes et les voies qui peuvent être modifiés pour améliorer l’activité des cellules CART de manière non biaisée, nous avons conçu un crible CRISPR in vitro à l’échelle du génome (Figure 1). Ce crible comporte deux phases : une phase de production de cellules CART et une phase de pression sélective. Dans la phase de production de cellules CART, au moins 110 × 106 lymphocytes T sont d’abord isolés à ...
Discussion
L’édition génomique est devenue un outil puissant à la fois pour comprendre les mécanismes de résistance aux thérapies et pour concevoir de nouvelles thérapies cellulaires CART afin d’améliorer la longévité et l’activité des cellules CART 16,17,26. Bien que certaines stratégies d’édition génomique aient montré des améliorations de l’activité des cellules CART dans les ...
Déclarations de divulgation
SSK est l’inventeur de brevets dans le domaine de l’immunothérapie CAR qui sont concédés sous licence à Novartis (par le biais d’un accord entre la Mayo Clinic, l’Université de Pennsylvanie et Novartis), Humanigen (par l’intermédiaire de la Mayo Clinic), Mettaforge (par l’intermédiaire de la Mayo Clinic) et MustangBio (par l’intermédiaire de la Mayo Clinic), et Chymal therapeutics (par l’intermédiaire de la Mayo Clinic). CS, CMR et SSK sont des inventeurs de brevets concédés sous licence à Immix Biopharma. SSK reçoit des fonds de recherche de Kite, Gilead, Juno, BMS, Novartis, Humanigen, MorphoSys, Tolero, Sunesis/Viracta, LifEngine Animal Health Laboratories Inc. et Lentigen. SSK a participé à des réunions consultatives avec Kite/Gilead, Calibr, Luminary Therapeutics, Humanigen, Juno/BMS, Capstan Bio et Novartis. SSK a siégé au conseil de sécurité et de surveillance des données avec Humanigen et Carisma. SSK a licencié un consultant pour Torque, Calibr, Novartis, Capstan Bio, BMS, Carisma et Humanigen. CMS et SSK sont les inventeurs de la propriété intellectuelle qui a résulté de ce protocole.
Remerciements
Cette étude a été en partie financée par le Mayo Clinic Center for Individualized Medicine (SSK), le Mayo Clinic Comprehensive Cancer Center (SSK), le Mayo Clinic Center for Regenerative Biotherapeutics (SSK), les National Institutes of Health K12CA090628 (SSK) et R37CA266344-01 (SSK), le Department of Defense grant CA201127 (SSK), la Predolin Foundation (SSK) et le Minnesota Partnership for Biotechnology and Medical Genomics (SSK). CMS est soutenu par la Mayo Clinic Graduate School of Biomedical Sciences. Le schéma de l’écran CRISPR (Figure 1) a été créé avec BioRender.com (Siegler, L. (2022) https://BioRender.com/k71r054).
matériels
| Name | Company | Catalog Number | Comments |
| 293T cells | ATCC | CRL-3216 | Cells used for lentivirus production |
| Biotin ProteinL Antibody | GenScript | M00097 | anti-kappa chain antibody for CAR detection |
| Bovine Serum Albumin | Millipore Sigma | A7906 | |
| Carbenicillin disodium salt | Millipore Sigma | C1389-1G | Carbenicillin antibiotic |
| CD4 Isolation Beads | Miltenyi Biotec | 130-045-101 | |
| CD8 Isolation Beads | Miltenyi Biotec | 130-045-201 | |
| CTS (Cell Therapy Systems) Dynabeads CD3/CD28 | Gibco | 40203D | |
| Cytoflex | Beckman Coulter | NC2279958 | |
| DNase-Free Water | Invitrogen | AM9937 | |
| Dulbecco's modified eagle's medium (DMEM) | Corning | 10-017-CV | |
| Dulbecco's Phosphate-Buffered Saline | Gibco | 14190-144 | |
| EasySep Human T Cell Isolation Kit | STEMCELL Technologies | 17951RF | Negative isolation kit |
| Endura Electrocompetent Cells | Biosearch Technologies | 60242-1 | Electrocompetent cells with recovery medium |
| Ethanol | Millipore Sigma | E7023 | |
| Fetal bovine serum (FBS) | Corning | 35-010-CV | |
| GeCKO v2 CRISPR Knockout Pooled Library A | AddGene | 1000000048 | CRISPR library plasmid |
| Gene Pulser II | Bio-Rad | 165-2105 | Electroporator |
| Glycogen | Millipore Sigma | 10901393001 | |
| JeKo-1 | ATCC | CRL-3006 | CD19+ target cells |
| Lipofectamine 3000 Transfection Reagent | ThermoFisher Scientific | L3000075 | Transfection reagent kit with a transfection reagent (Lipofectamine 3000 Reagent) and a neutral co-lipid reagent (p3000) |
| LIVE/DEAD Aqua | Invitrogen | L34966 | |
| Lymphoprep | STEMCELL Technologies | 7851 | Density gradient medium |
| Machery-Nagel NucleoBond Xtra Maxi Kits | ThermoFisher Scientific | 12748412 | Maxi-prep kit |
| NEBNext High-Fidelity 2X PCR MasterMix | New England BioLabs | M0541S | High fidelity PCR mastermix |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985-070 | Reduced serum medium |
| pCMVR8.74 | AddGene | 22036 | Lentiviral packaging plasmid |
| Pennicillin-streptomycin-glutamine (100X) | Life Technologies | 10378-016 | |
| pMD2.G | AddGene | 12259 | VSV-G envelope expressing plasmid |
| Pooled Human AB Serum | Innovative Research | ISERABHI | |
| Puromycin | Millipore Sigma | P8833 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | Gek extraction kit |
| Qucik-DNA Midiprep Plus Kit | Zymo Research | D4075 | Kit used to isolate gDNA |
| RoboSep-S | STEMCELL Technologies | 21000 | Automated cell separator |
| Roswell Park Memorial Institute 1640 Medium (RPMI) | Gibco | 21870092 | |
| SepMate-50 | STEMCELL Technologies | 85450 | Density gradient separation tube |
| Sodium Acetate | Invitrogen | AM9740 | |
| Sodium Azide | Fisher Scientific | 71448-16 | |
| Streptavidin Antibody (PE) | BioLegend | 405203 | Secondary antibody used for CAR detection |
| T100 Thermal Cycler | Bio-Rad | 1861096 | |
| Ultracentrifuge (Optima XPN-80) | BeckmanCoulter | A99839 | |
| Vacuum Filter Systems, 0.22 µm | ThermoFisher Scientific | 567-0020 | |
| Vacuum Filter Systems, 0.45 µm | ThermoFisher Scientific | 165-0045 | |
| X-VIVO 15 Serum-Free Hematopoietic Cell Medium | Lonza | 04-418Q | Hematopoietic cell medium |
Références
- Neelapu, S. S., et al. 5-year follow-up supports curative potential of axicabtagene ciloleucel in refractory large B-cell lymphoma (zuma-1). Blood. 141 (19), 2307-2315 (2023).
- Neelapu, S. S., et al. Axicabtagene ciloleucel CAR T-cell therapy in refractory large B-cell lymphoma. N Engl J Med. 377 (26), 2531-2544 (2017).
- Locke, F. L., et al. Long-term safety and activity of axicabtagene ciloleucel in refractory large B-cell lymphoma (zuma-1): A single-arm, multicentre, phase 1-2 trial. Lancet Oncol. 20 (1), 31-42 (2019).
- Maude, S. L., et al. Tisagenlecleucel in children and young adults with B-cell lymphoblastic leukemia. N Engl J Med. 378 (5), 439-448 (2018).
- Schuster, S. J., et al. Tisagenlecleucel in adult relapsed or refractory diffuse large B-cell lymphoma. N Engl J Med. 380 (1), 45-56 (2018).
- Bai, Z., et al. Single-cell antigen-specific landscape of CAR T infusion product identifies determinants of CD19-positive relapse in patients with all. Sci Adv. 8 (23), eabj2820 (2022).
- Fraietta, J. A., et al. Determinants of response and resistance to CD19 chimeric antigen receptor (CAR) T cell therapy of chronic lymphocytic leukemia. Nat Med. 24, 563-571 (2018).
- Gennert, D. G., et al. Dynamic chromatin regulatory landscape of human CAR T cell exhaustion. Proc Natl Acad Sci USA. 118 (30), e2104758118 (2021).
- Deng, Q., et al. Characteristics of anti-CD19 CAR T cell infusion products associated with efficacy and toxicity in patients with large B cell lymphomas. Nat Med. 26 (12), 1878-1887 (2020).
- Sterner, R. M., Cox, M. J., Sakemura, R., Kenderian, S. S. Using CRISPR/CAS9 to knock out GM-CSF in CAR-T cells. J Vis Exp. (149), e59629 (2019).
- Sterner, R. M., et al. GM-CSF inhibition reduces cytokine release syndrome and neuroinflammation but enhances CAR-T cell function in xenografts. Blood. 133 (7), 697-709 (2019).
- Cox, M. J., et al. GM-CSF disruption in CART cells modulates T cell activation and enhances CART cell anti-tumor activity. Leukemia. 36 (6), 1635-1645 (2022).
- Ottaviano, G., et al. Phase 1 clinical trial of CRISPR-engineered CAR19 universal T cells for treatment of children with refractory B cell leukemia. Sci Transl Med. 14 (668), eabq3010 (2022).
- Joung, J., et al. Genome-scale CRISPR-CAS9 knockout and transcriptional activation screening. Nat Protoc. 12, 828-863 (2017).
- Mccutcheon, S. R., et al. Transcriptional and epigenetic regulators of human CD8+ T cell function identified through orthogonal CRISPR screens. Nat Genet. 55, 2211-2223 (2023).
- Zhu, W., Kelly, C., Dagur, P., Dunbar, C. E., Cordes, S. CRISPR activation screen to optimize chimeric antigen receptor (CAR) T cell immunophenotype. Blood. 142 (Supplement 1), 4820-4820 (2023).
- Wang, D., et al. CRISPR screening of CAR T cells and cancer stem cells reveals critical dependencies for cell-based therapies. Cancer Discov. 11 (5), 1192-1211 (2021).
- Shifrut, E., et al. Genome-wide CRISPR screens in primary human T cells reveal key regulators of immune function. Cell. 175 (7), 1958-1971.e15 (2018).
- Belk, J. A., et al. Genome-wide crispr screens of T cell exhaustion identify chromatin remodeling factors that limit T cell persistence. Cancer Cell. 40 (7), 768-786.e7 (2022).
- Jiang, P., et al. Single-cell ATAC-seq maps the comprehensive and dynamic chromatin accessibility landscape of CAR-T cell dysfunction. Leukemia. 36 (11), 2656-2668 (2022).
- Rossi, J., et al. Preinfusion polyfunctional anti-CD19 chimeric antigen receptor T cells are associated with clinical outcomes in NHL. Blood. 132 (8), 804-814 (2018).
- Beider, K., et al. Molecular and functional signatures associated with CAR T cell exhaustion and impaired clinical response in patients with B cell malignancies. Cells. 11 (7), 1140 (2022).
- Wherry, E. J. T. cell exhaustion. Nat Immunol. 12, 492-499 (2011).
- Prinzing, B., et al. Deleting DNMT3A in CAR T cells prevents exhaustion and enhances antitumor activity. Sci Transl Med. 13, eabh0272 (2021).
- Lynn, R. C., et al. C-jun overexpression in CAR T cells induces exhaustion resistance. Nature. 576, 293-300 (2019).
- Tang, N., et al. TGF-beta inhibition via CRISPR promotes the long-term efficacy of CAR T cells against solid tumors. JCI Insight. 5 (4), e133977 (2020).
- Joung, J., et al. Genome-scale CRISPR-CAS9 knockout and transcriptional activation screening. Nat Protoc. 12 (4), 828-863 (2017).
- Li, W., et al. Quality control, modeling, and visualization of CRISPR screens with MAGeCK-VISPR. Genome Biol. 16, 281 (2015).
- Sakemura, R., et al. Targeting cancer-associated fibroblasts in the bone marrow prevents resistance to CART-cell therapy in multiple myeloma. Blood. 139 (26), 3708-3721 (2022).
- Stewart, C. M., et al. IL-4 drives exhaustion of CD8+ CART cells. Nat Commun. 15 (1), 7921 (2024).
- . FastQC: A quality control tool for high throughput sequence data Available from: https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010)
- . Bbmerge Available from: https://github.com/BioInfoTools/BBMap/blob/master/sh/bbmerge.sh (2019)
- Pattanayak, V., et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed CAS9 nuclease specificity. Nat Biotechnol. 31 (9), 839-843 (2013).
- Doench, J. G., et al. Rational design of highly active sgRNAs for CRISPR-CAS9-mediated gene inactivation. Nat Biotechnol. 32 (12), 1262-1267 (2014).
- Dong, M. B., et al. Systematic immunotherapy target discovery using genome-scale in vivo CRISPR screens in CD8 T cells. Cell. 178 (5), 1189-1204.e23 (2019).
- Zhou, P., et al. Single-cell CRISPR screens in vivo map T cell fate regulomes in cancer. Nature. 624 (7990), 154-163 (2023).
- Sutra Del Galy, A., et al. In vivo genome-wide CRISPR screens identify SOCS1 as intrinsic checkpoint of CD4+ TH1 cell response. Sci Immunol. 6 (66), eabe8219 (2021).
- Ramos, A., et al. Leukemia-intrinsic determinants of CAR-T response revealed by iterative in vivo genome-wide CRISPR screening. Nat Commun. 14 (1), 8048 (2023).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon