Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Durchführung eines genomweiten In-vitro-CRISPR-Knockout-Screenings in chimären Antigenrezeptor-T-Zellen
In diesem Artikel
Zusammenfassung
Der Artikel beschreibt ein Protokoll für die Anwendung eines in vitro Modells zur Erschöpfung zur Vervollständigung eines genomweiten CRISPR-Knockout-Screenings in gesunden chimären Antigenrezeptor-T-Zellen von Spendern.
Zusammenfassung
Die chimäre Antigenrezeptor-T-Zelltherapie (CART) ist eine innovative Form der zielgerichteten Immuntherapie, die die Behandlung von Krebs revolutioniert hat. Das dauerhafte Ansprechverhalten bleibt jedoch begrenzt. Jüngste Studien haben gezeigt, dass die epigenetische Landschaft von CART-Zellprodukten vor der Infusion das Ansprechen auf die Therapie beeinflussen kann, und Gen-Editing wurde als Lösung vorgeschlagen. Es muss jedoch noch mehr Arbeit geleistet werden, um die optimale Gen-Editing-Strategie zu bestimmen. Genomweite CRISPR-Screenings sind zu beliebten Instrumenten geworden, um sowohl Resistenzmechanismen zu untersuchen als auch Gen-Editing-Strategien zu optimieren. Ihre Anwendung auf Primärzellen birgt jedoch viele Herausforderungen. Hier beschreiben wir eine Methode, um ein genomweites CRISPR-Knockout-Screening in CART-Zellen von gesunden Spendern durchzuführen. Als Proof-of-Concept-Modell haben wir diese Methode entwickelt, um die Entwicklung der Erschöpfung in CART-Zellen zu untersuchen, die auf das CD19-Antigen abzielen. Wir glauben jedoch, dass dieser Ansatz verwendet werden kann, um eine Vielzahl von Mechanismen der Therapieresistenz in verschiedenen CAR-Konstrukten und Tumormodellen zu untersuchen.
Einleitung
Die Zelltherapie mit chimären Antigenrezeptoren T (CART) hat beeindruckende Erfolge bei der Behandlung von B-Zell-Malignomen gezeigt. Das dauerhafte Ansprechverhalten ist jedoch auf 30-40%1,2,3,4,5 begrenzt. Während Forscher mehrere Ansätze entwickelt und getestet haben, um die Mechanismen der Resistenz gegen die CART-Zelltherapie zu untersuchen, einschließlich der Optimierung des CAR-Designs, der Genomeditierung und der Kombinationstherapien, ist die Entwicklung der Resistenz noch weitgehend unbekannt. In jüngster Zeit gibt es zunehmend Hinweise darauf, dass das Ausgangs-Genexpressionsprofil von CART-Zellprodukten vor der Infusion eine wichtige Determinante sowohl für die Toxizität als auch für die Wirksamkeit der CART-Therapie ist 6,7,8,9. Daher ist die Gen-Editierung während der CART-Zellproduktion zu einem beliebten Ansatz geworden, um den Erfolg der CART-Zelltherapie zu verbessern.
Zum Beispiel hat unser Labor zuvor gezeigt, dass die Verwendung der CRISPR-Cas9-Technologie (Clustered Regularly Interspaced Short Palindromic Repeats) zur Ausschaltung des Gens Granulozyten-Makrophagen-Kolonie-stimulierender Faktor (GM-CSF) die CART-Zellaktivität verbessert und die Anzeichen einer CART-assoziierten Toxizität reduziert 10,11,12. Darüber hinaus wurde die CRISPR-technische Zelltherapie in klinischen Studien getestet und hat Wirksamkeit und Sicherheit gezeigt13. Zusammengenommen deutet dies darauf hin, dass die Genomeditierung mit der CRISPR-Technologie nicht nur unser Verständnis der CART-Zellbiologie verbessern, sondern auch übersetzbare CART-Zellprodukte erzeugen kann.
Genomweite CRISPR-Knockout-Screens sind zu immer beliebteren Werkzeugen in der Krebsbiologieforschung geworden, um die Mechanismen der Resistenz gegen Therapeutika zu verstehen. Bei dieser Technik werden Zehntausende von Guide-RNAs (gRNAs) in Zellpools abgegeben, um den Eintritt einer gRNA pro Zelle zu fördern14. Dann werden die Zellen druckinduzierten Bedingungen ausgesetzt, bei denen Zellen, die mit gRNAs transduziert wurden, die auf essentielle Gene abzielen, absterben und Zellen, die mit gRNAs transduziert wurden, die auf inhibitorische Gene abzielen, überleben und sich vermehren. Mit Hilfe von Next-Generation-Sequencing können wir bestimmen, wie sich die gRNA-Repräsentation im gesamten CRISPR-Screenverändert 14.
Der Umfang und die Selektionszeit, die für genomweite Genstörungen erforderlich sind, können jedoch in Primärzellen, wie z. B. CART-Zellen, eine Herausforderung darstellen. Daher haben die Gruppen gezielte CRISPR-Screens verwendet, um die Mechanismen der therapeutischen Resistenz besser zu verstehen15,16. Zielraster sind in primären Zellen oft einfacher zu vervollständigen, da sie über kleinere Bibliotheken verfügen, die weniger Zellen benötigen, um eine angemessene Bibliotheksdarstellung zu erreichen. Während diese Studien unser Verständnis der Mechanismen der Resistenz gegen die CART-Zelltherapie verbessert haben, führen gezielte Screens aufgrund der manuellen Auswahl von Genzielen zu einer Verzerrung. In diesem Artikel wird versucht, eine Methode zu beschreiben, mit der ein in vitro genomweites CRISPR-Knockout-Screening in CART-Zellen von gesunden Spendern durchgeführt werden kann. Auf diese Weise ermöglicht dieser Hochdurchsatzansatz eine effiziente, unvoreingenommene Identifizierung von Schlüsselsignalen und Genen, die bearbeitet werden können, um das therapeutische Ansprechen zu verbessern 17,18,19.
Insbesondere soll das in diesem Artikel beschriebene Protokoll das Verständnis der Erschöpfung von CART-Zellen in diesem Bereich verbessern, indem das genomweite CRISPR-Knockout-Screening mit einem In-vitro-Modell für die Erschöpfung vervollständigt wird. Erschöpfung ist ein dysfunktionales Schicksal von CART-Zellen, das mit dem Nichtansprechen auf die CART-Zelltherapie in Verbindung gebracht wird 20,21,22. Es ist bekannt, dass dieses zelluläre Schicksal epigenetisch reguliert ist und durch eine Abnahme der CART-Zellproliferation, eine Abnahme der Effektorzytokinproduktion und eine Zunahme der Expression inhibitorischer Rezeptoren gekennzeichnetist 23. In der bisherigen Literatur war die Geneditierung in der Lage, die Entwicklung von Erschöpfung zu verhindern, indem sie Schlüsselgene entweder hoch- oder herunterregulierte 24,25,26. Angesichts der Abnahme der Proliferationsfähigkeit mit zunehmender Erschöpfung der CART-Zellen und der Evidenz, dass Gen-Editing ihr Auftreten verhindern kann, haben wir unseren in vitro genomweiten CRISPR-Knockout-Screen auf diesem zellulären Schicksal modelliert. Dieses Protokoll könnte jedoch in Zukunft geändert werden, um andere Mechanismen der Resistenz gegen die CART-Zelltherapie zu erforschen.
Protokoll
Wichtig ist, dass das unten beschriebene Protokoll den Richtlinien des Institutional Review Board (IRB 18-005745) der Mayo Clinic und des Institutional Biosafety Committee (IBC HIP00000252.43) folgt und von diesem genehmigt wurde. Alle Zellkulturarbeiten, einschließlich der lentiviralen Produktion, sollten in einer Zellkulturhaube mit geeigneter persönlicher Schutzausrüstung durchgeführt werden. Insbesondere sollten lentivirale Arbeiten unter Vorsichtsmaßnahmen der Biosicherheitsstufe 2 (BSL-2) durchgeführt werden, einschließlich der Verwendung von 10 % Bleichmittel zur Desinfektion von Gegenständen vor der Entsorgung.
1. Amplifikation der CRISPR-Bibliothek
- Erweitern Sie die CRISPR-Bibliothek gemäß den Empfehlungen des Herstellers. Kurz gesagt, elektropopolieren Sie die CRISPR-Bibliothek in vier Aliquots elektrokompetenter Zellen, indem Sie die folgenden Schritte ausführen:
- Geben Sie 2 μl 50 ng/μl CRISPR-Bibliotheksplasmid zu 25 μl elektrokompetenten Zellen in einer 1,0-mm-Küvette.
- Elektropotieren Sie die Proben mit den folgenden Einstellungen: 10 μF, 600 Ohm, 1.800 Volt und einer Zeitkonstante zwischen 3,5 ms und 4,5 ms.
- In 975 μl Rückgewinnungsmedium zurückgewinnen und in ein Röhrchen mit weiteren 1 ml Rückgewinnungsmedium übertragen.
- Alle Aliquoten 1 h lang bei 37 °C bei 250 U/min drehen.
- Plattieren Sie die Transformationen:
- Alle Aliquots der elektroporierten Zellen zusammenlegen und gut mischen.
- Alle elektroporierten Zellen werden mit 400 μl der Transformationsmischung auf vorgewärmte 10 cm Luria-Bouillonplatten (LB) mit 100 μg/ml Carbenicillin aufgetragen. Stellen Sie die Platte für 14 h bei 32 °C in einen Inkubator.
- Ernte der Völker:
- Pipettieren Sie 1 ml LB-Medium + 100 μg/ml Carbenicillin auf jede Platte. Kratzen Sie die Kolonien von jeder Platte ab und sammeln Sie sie in einem konischen 50-ml-Röhrchen.
- Geben Sie weitere 1 ml LB-Medium + 100 μg/ml Carbenicillin in jede Platte, um sie zu waschen und eine vollständige Wiederherstellung zu gewährleisten.
- Führen Sie eine Maxi-Vorbereitung durch, indem Sie den Anweisungen eines Maxi-Vorbereitungs-Kits folgen.
- Bündeln Sie die resultierende Plasmid-DNA.
2. Next-Generation-Sequencing zur Verifizierung der gRNA-Repräsentation zu Studienbeginn in der CRISPR-Bibliothek
HINWEIS: NGS-Primer für diese CRISPR-Bibliothek wurden in einer früheren Veröffentlichungentwickelt 27. Die Verwendung eines anderen Reverse-Primers für jede Probe führt zu einem Barcode für jede Probe und ermöglicht das Pooling von Proben während der Sequenzierung.
- Bereiten Sie die Proben für die PCR mit folgendem Reaktionsgemisch vor (Gesamtvolumen = 50 μL): High-Fidelity-PCR-Mastermix, 2x: 25 μL; gepoolte Template-DNA, 0,5 μg: 1 μL; Vorwärtsprimer der NGS-Bibliothek, 10 μM: 1,25 μL; Reverse-Primer der NGS-Bibliothek, 10 μM: 1,25 μL; DNasefreies Wasser: 21,5 μL.
- PCR unter den folgenden Zyklusbedingungen durchführen: Zyklus 1: Denaturierung bei 98 °C für 3 Minuten; Zyklen 2-23: Denaturierung bei 98 °C für 10 s, Glühen bei 63 °C für 10 s, Ausdehnen bei 72 °C für 25 s; Zyklus 24: Bei 72 °C für 2 min verlängern.
- Reinigen Sie die PCR-Reaktion, indem Sie die PCR-Reaktionen für jede Probe zusammenfassen und das PCR-Produkt mit einem PCR-Aufreinigungskit gemäß den Anweisungen des Herstellers aufreinigen.
- Führen Sie 3 μg des PCR-Produkts für jede Probe auf ein 2 % (w/v) Agarosegel auf.
- Entfernen Sie das PCR-Produkt (~260-270 bp) aus dem Gel, indem Sie die DNA aus dem Gel extrahieren, indem Sie ein Gel-Extraktionskit gemäß den Anweisungen des Herstellers verwenden. Streben Sie mindestens 500 ng DNA an.
- Lagern Sie die extrahierten Proben bei -20 °C.
- Führen Sie mit den Proben eine Next-Generation-Sequenzierung (NGS) durch und streben Sie 100 Reads pro gRNA27 an.
- Analysieren Sie die Sequenzierungsergebnisse mit der MAGeCK-VISPR-Pipeline und stellen Sie sicher, dass mehr als 99,5 % der gRNAs in der Bibliothek repräsentiert sind28.
3. Herstellung von CRISPR-Bibliothek und CAR-Expressions-Lentiviren
- Bereiten Sie das Lentivirus wie zuvor beschrieben vor10,29.
- Tag 1: Beginnen Sie mit T175-Kolben mit 80-90% konfluenten 293T-Zellen, die in D10-Medien gewachsen sind, die aus 10% fötalem Rinderserum (FBS) (v/v), 1% Penicillin-Streptomycin-Glutamin (PSG) (v/v) und Dulbeccos modifiziertem Adlermedium bestehen. Bevor Sie beginnen, erwärmen Sie Transfektionsreagenzien und DNA auf Raumtemperatur und quantifizieren Sie die DNA.
- Für jeden 293-t-Kolben werden zwei konische 50-ml-Röhrchen A und B etikettiert. Geben Sie 4,5 ml reduziertes Serummedium in die Sonde A und in die Sonde B.
- Mischen Sie in einem sterilen 1,5-ml-Mikrozentrifugenröhrchen die folgende DNA und lassen Sie sie 1 Minute lang ruhen: 18 μg pCMVR8.74 (gag, pol, tat und rev), 7 μg pMD2.G (VSV-G) und 15 μg lentiviraler Expressionsvektor (CRISPR-Bibliothek A oder CAR19-28ζ-Vektoren).
- 129 μl Transfektionsreagenz in Röhrchen A geben. Tube A durch Fingerwirbeln gut mischen.
- Gib das DNA-Gemisch in Röhrchen B. Tube B durch Fingerwirbeln gut mischen.
- 111 μl neutrales Co-Lipid-Reagenz in Röhrchen B geben. Tube B durch Fingerwirbeln gut mischen.
- Den Inhalt von Röhrchen A in Röhrchen B geben und durch Fingerwirbeln gut vermischen. Röhrchen B 30 min bei Raumtemperatur inkubieren.
- Gegen Ende der 30-minütigen Inkubationszeit bereiten Sie die 293T-Zellen vor, indem Sie das vorhandene Medium aspirieren. Fügen Sie 16 ml R10-Medium hinzu, das aus 10 % FBS (v/v), 1 % PSG (v/v) und Roswell Park Memorial Institute (RPMI) 1640 besteht.
- Nach Ablauf der 30-minütigen Inkubationszeit wird der Inhalt von Röhrchen B (ca. 9 ml) tropfenweise in den Kolben mit 293T-Zellen gegeben. Der Kolben wird abgesetzt und vorsichtig gedreht, bevor er bei 37 °C und 5 % CO 2 inkubiertwird.
- Am Tag 2 (ca. 24 h nach der Transfektion) sammeln Sie Lentiviren wie folgt:
HINWEIS: Kühlen Sie alle Geräte und Materialien vor und führen Sie die folgenden Schritte auf Eis durch, um den Virusüberstand kalt zu halten.- Der Überstand aus den transfizierten 293T-Kolben wird in vorgekühlte konische 50-ml-Röhrchen überführt.
- Geben Sie 30 ml vorgewärmtes R10-Medium in die 293T-Kolben.
- Pelletieren Sie zelluläre Ablagerungen durch Zentrifugieren der konischen 50-ml-Röhrchen bei 900 × g für 10 min bei 4 °C in geschlossenen Rotoren.
- Nach dem Zentrifugieren wird der Überstand mit einem 0,45 μm Filter filtriert. Das Filtrat in einer Ultrazentrifuge bei 112.700 × g bei 4 °C für 2 h schleudern.
- Nach dem 2-stündigen Schleudern aspirieren Sie den Überstand, bis noch etwa 5 ml übrig sind. Resuspendieren Sie die Viruspartikel, indem Sie 10x sanft auf und ab pipettieren. Lagern Sie die Virussuspension über Nacht bei 4 °C.
- Sammeln und lagern Sie an Tag 3 das Lentivirus wie folgt:
- Der Überstand aus den transfizierten 293T-Kolben wird in vorgekühlte konische 50-ml-Röhrchen überführt.
- Geben Sie 30 ml 10%iges Bleichmittel in die 293T-Kolben.
- Die zellulären Trümmer im Überstand werden pelletiert, indem die konischen 50-ml-Röhrchen bei 900 × g für 10 min bei 4 °C in geschlossenen Rotoren zentrifugiert werden.
- Nach dem Zentrifugieren wird der Überstand mit einem 0,45 μm Filter filtriert. Das Filtrat in einer Ultrazentrifuge bei 112.700 × g bei 4 °C für 2 h schleudern.
- Nach der 2-stündigen Zentrifuge aspirieren Sie den Überstand, bis etwa 2 ml übrig sind, und resuspendieren Sie ihn, indem Sie ihn vorsichtig 10x auf und ab pipettieren. Lagern Sie den Virusüberstand in 300-500 μl Aliquoten und halten Sie ihn bei -80 °C.
4. Berechnung des Virustiters für CAR-exprimierende Lentiviren
HINWEIS: Das CAR-Lentivirus wurde wie zuvor beschrieben titert10,12.
- An Tag 1 T-Zellen aktivieren und plattieren.
- Isolieren Sie 1 × 106 T-Zellen aus gesunden Spender-PBMCs mit einem Negativ-Selektionskit.
- Zählen Sie die T-Zellen.
- Waschen Sie genügend CD3+/CD28+ -Kügelchen, um die T-Zellen in einem Verhältnis von 3:1 zu stimulieren. Um beispielsweise 1 × 106 T-Zellen zu stimulieren, waschen Sie 3 × 106 Kügelchen. Waschen Sie dazu die Perlen, indem Sie die folgenden 3x ausführen:
- Verwenden Sie eine magnetische Trennung, um die Kügelchen aus der Suspension zu entfernen.
- Entfernen Sie den Überstand.
- Fügen Sie 1000 μl T-Zell-Medium (TCM) hinzu, das aus 10 % Humanserum (v/v), 1 % PSG (v/v) und hämatopoetischem Zellmedium besteht.
HINWEIS: TCM sollte vor der Verwendung durch Filtrieren mit einem 0,45 μm sterilen Vakuumfilter und dann mit einem 0,22 μm sterilen Vakuumfilter sterilisiert werden. - Fügen Sie der T-Zell-Suspension Kügelchen in einem Verhältnis von 3:1 Kügelchen:Zelle hinzu.
- Resuspendieren Sie die T-Zellen auf 1 × 106 Zellen/ml in der TCM.
- Geben Sie 100 μl aktivierte T-Zellen in 10 Vertiefungen einer 96-Well-Platte. Die Platte wird bei 37 °C und 5 % CO2 inkubiert.
- Fügen Sie an Tag 2 eine Verdünnung des CAR-Lentivirus hinzu, indem Sie eine Verdünnungsplatte wie folgt erstellen:
- Geben Sie 100 μl TCM in sieben Vertiefungen einer 96-Well-Platte.
- Geben Sie in die erste Vertiefung 50 μl des gewünschten Virus; Gut mischen.
- Verdünnen Sie das Virus seriell weiter, indem Sie 50 μl aus Vertiefung 1 bis Vertiefung 2 hinzufügen. Gut mischen.
- Fügen Sie 50 μl von Vertiefung 2 bis Vertiefung 3 hinzu und setzen Sie dieses Muster bis Vertiefung 7 fort.
- Entfernen Sie nach dem Mischen von Vertiefung 7 50 μl. Stellen Sie sicher, dass alle Vertiefungen ein Gesamtvolumen von 100 μl haben.
- Geben Sie 50 μl aus jeder Vertiefung der Verdünnungsplatte in die ersten sieben Vertiefungen der T-Zellplatte von Tag 1. Gut mischen.
- Zu den letzten drei Vertiefungen der T-Zellplatte werden 50 μl TCM gegeben; Gut mischen. Lagern Sie die Platte in einem Inkubator bei 37 °C, 5 % CO2.
- Füttern Sie die Zellen an Tag 3, indem Sie 100 μl TCM in alle Vertiefungen der T-Zellplatte geben. Gut mischen.
- Führen Sie an Tag 4 eine Durchflusszytometrie durch, um den Prozentsatz der Zellen, die CAR exprimieren, mit einem Anti-Kappa-Ketten-Antikörper, einem Surrogatmarker für die CAR-Expression, zu bestimmen, indem Sie die folgenden Schritte ausführen:
- Die T-Cell-Platte bei 1.000 × g 3 min eindrehen und dekantieren.
- Waschen Sie die Platte 3x mit Anti-Kappa-Ketten-Antikörperpuffer (bestehend aus 4 % Rinderserumalbumin in PBS (w/v)), indem Sie 200 μl Anti-Kappa-Ketten-Antikörperpuffer hinzufügen, die Platte 3 Minuten lang bei 1.000 × g abdrehen und dekantieren.
- Geben Sie einen Biotin-Anti-Kappa-Ketten-Antikörper in jede Vertiefung. 45 min auf einer Wippe bei 4 °C inkubieren.
- Waschen Sie die Platte 3x mit Anti-Kappa-Ketten-Antikörperpuffer wie in Schritt 4.4.2.
- Färben Sie die Zellen für die Durchflusszytometrie mit einem Fluorochrom-konjugierten Streptavidin-Antikörper und einem lebenden/toten Antikörper. Inkubieren Sie die Proben 15 Minuten lang bei Raumtemperatur im Dunkeln.
- Waschen Sie die Proben einmal in einem Flow-Puffer (bestehend aus 2 % FBS (v/v) und 1 % Natriumazid (v/v) in PBS), indem Sie 200 μl Flow-Puffer hinzufügen, die Platte 3 Minuten lang bei 1.000 × g herunterdrehen und dekantieren.
- In 100 μl Durchflusspuffer resuspendieren und auf dem Zytometer laufen lassen.
- Führen Sie ein negatives Gating mit den drei nicht transduzierten Wells durch.
5. Berechnung des Virustiters für das Lentivirus der CRISPR-Bibliothek
HINWEIS: Dieses Protokoll beschreibt eine Mindestanzahl von T-Zellen für die Titration des Lentivirus der CRISPR-Bibliothek, kann jedoch hochskaliert werden, um mehr T-Zellen und damit höhere Virusvolumina aufzunehmen.
- Aktivieren Sie an Tag 0 die T-Zellen, indem Sie 18 × 106 CD3+/CD28+ Kügelchen (3:1 Kügelchen:Zell-Verhältnis) zu 6 × 106 T-Zellen hinzufügen und das endgültige Volumen mit TCM auf 6 ml bringen. Aliquotieren Sie 0,5 mL der T-Zell-Suspension in 12 Vertiefungen einer 48-Well-Platte. In einem Inkubator bei 37 °C, 5 % CO2 lagern.
- Vierundzwanzig Stunden später, an Tag 1, geben Sie das CAR-Virus mit einer Multiplizität der Infektion (MOI) von 3 in jede Vertiefung. Gut mischen, indem Sie mehrmals auf und ab pipettieren. In einem Inkubator bei 37 °C, 5 % CO2 lagern.
- Achtundvierzig Stunden später, an Tag 2, fügen Sie den aktivierten CART-Zellen eine Reihe von Virusvolumina der CRISPR-Bibliothek hinzu, indem Sie in jede Vertiefung eines der folgenden Volumina des CRISPR-Virus geben (0 μL, 0 μL, 6,25 μL, 6,25 μL, 12,5 μL, 12,5 μL, 25 μL, 25 μL, 50 μL, 50 μL, 100 μL oder 100 μL). Gut mischen, indem Sie mehrmals auf und ab pipettieren. In einem Inkubator bei 37 °C, 5 % CO2 lagern.
- Geben Sie an Tag 3 1 μg Puromycin pro ml Medium in die Hälfte der Vertiefungen, einschließlich einer Vertiefung, die jedes der folgenden CRISPR-Virusvolumina (0 μl, 6,25 μl, 12,5 μl, 25 μl, 50 μl und 100 μl) aufnahm. Die anderen Vertiefungen werden nicht mit Puromycin behandelt.
- Von Tag 4 bis 8 zählen Sie die T-Zellen täglich und passen Sie die endgültige Zelldichte auf 1 × 106 Zellen/ml mit 1 μg/ml Puromycin in der TCM für die Puromycin-Behandlungsvertiefungen und mit TCM für die unbehandelten Vertiefungen an.
- Zählen Sie an Tag 8 die Anzahl der lebenden Zellen pro Vertiefung und berechnen Sie die Transduktionseinheiten/ml des Virusüberstands anhand der folgenden Gleichung:
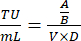
Dabei A: Anzahl lebender Zellen in viral gut transduziert; B: Anzahl lebender Zellen in einer Kontrollvertiefung ohne Antibiotika-Selektion; V: Virusvolumen, das für die Infektion in jeder Vertiefung verwendet wird; D: Verdünnungsfalte. - Berechnen Sie unter Verwendung der TU/ml-Vertiefung, die aus einer Vertiefung mit einer Transduktionseffizienz von etwa 20 % berechnet wurde, das Virusvolumen, das für die Transduktion von 1 × 106 Zellen bei einem MOI von 0,3 erforderlich ist, indem Sie die folgende Formel verwenden:
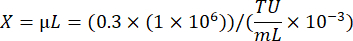
6. Produktionsphase der CART-Zellen des CRISPR-Screens (Tage 0 - 8)
HINWEIS: Das folgende Protokoll beschreibt die Fertigstellung des CRISPR-Screenings in einem biologischen Replikat. Dieses Protokoll wurde jedoch zuvor mit drei biologischen Replikaten validiert, und wir empfehlen die Verwendung von mindestens drei biologischen Replikaten für andere, die dieses Protokoll vervollständigen30.
- Tag 0: Isolierung und Stimulation von T-Zellen aus gesundem Spenderblut, wie zuvor beschrieben10.
- Isolieren Sie mononukleäre Zellen des peripheren Blutes (PBMCs) aus anonymisiertem gesundem Spenderblut.
- Geben Sie 15 ml eines Dichtegradientenmediums in ein Dichtegradienten-Trennröhrchen.
- Geben Sie 1:2 verdünntes Blut auf die Oberseite des Trennröhrchens mit Dichtegradienten. Bei 1.200 × g 10 min bei Raumtemperatur zentrifugieren.
HINWEIS: Das Blut sollte mit Isolationspuffer (2% FBS (v/v) in phosphatgepufferter Kochsalzlösung [PBS]) verdünnt werden. - Die oberste Schicht in ein neues konisches 50-ml-Röhrchen ablegen und das Volumen mit Isolationspuffer verdoppeln. Die Probe wird bei 300 × g 8 min bei 4 °C zentrifugiert. Aspirieren Sie den Überstand.
- Resuspendieren Sie das Zellpellet mit Isolationspuffer und zählen Sie die PBMCs.
- Isolieren Sie T-Zellen aus PBMCs mit einem Negativ-Selektionskit. Befolgen Sie die Anweisungen des Herstellers für das Kit.
HINWEIS: Gehen Sie von einer 10-20%igen Rückgewinnung von T-Zellen aus PBMCs aus. - Stimulieren Sie T-Zellen mit CD3+/CD28+ Kügelchen.
- Zählen Sie die T-Zellen.
- Aliquotieren Sie genügend CD3+/CD28+ -Kügelchen, um die T-Zellen in einem Kügelchen-Zell-Verhältnis von 3:1 zu stimulieren. Um beispielsweise 110 × 106 T-Zellen zu stimulieren, aliquotieren Sie 330 × 106 Kügelchen. Waschen Sie die Kügelchen 3x, indem Sie die Kügelchen zuerst mit magnetischer Trennung aus der Suspension trennen und dann 1.000 μl TCM hinzufügen.
- Geben Sie die Kügelchen in die T-Zell-Suspension. Resuspendieren Sie die T-Zellen auf 1 × 106 Zellen/ml in der TCM.
- Aliquotieren Sie die T-Zellen in 6-Well-Platten, um die beste Transduktionseffizienz zu erzielen. 5 × 106 T-Zellen in jede Vertiefung geben. Inkubieren Sie die Zellen bei 37 °C, 5 % CO2 für 24 h.
- Isolieren Sie mononukleäre Zellen des peripheren Blutes (PBMCs) aus anonymisiertem gesundem Spenderblut.
- Tag 1: Unter BSL-2+-Bedingungen genügend CAR-Lentiviren auftauen, um T-Zellen bei einer Infektionsvielfalt von 3,0 zu transduzieren. Brechen Sie die T-Zell-Rosetten vorsichtig auf, bevor Sie die entsprechende Menge an gepooltem Lentivirus hinzufügen, um 5 × 106 T-Zellen bei einem MOI von 3,0 zu transduzieren. Inkubieren Sie die Zellen bei 37 °C, 5 % CO2.
HINWEIS: Lentiviren aus mehreren Produktionen vor der Transduktion von T-Zellen in ein Röhrchen poolen. Dadurch wird sichergestellt, dass alle T-Zellen die gleiche Behandlung erhalten. Außerdem sollte das Lentivirus nur einmal aufgetaut werden. - Tag 2: Tauen Sie unter BSL-2+-Bedingungen genügend CRISPR-Lentiviren auf, um T-Zellen bei einem MOI von 0,3 zu transduzieren. Brechen Sie die T-Zell-Rosetten vorsichtig auf und resuspendieren Sie die T-Zellen, bevor Sie die entsprechende Menge an gepooltem Lentivirus hinzufügen, um 5 × 106 T-Zellen bei einem MOI von 0,3 zu transduzieren. Inkubieren Sie die Zellen bei 37 °C, 5 % CO2.
HINWEIS: Lentiviren aus mehreren Produktionen vor der Transduktion von T-Zellen in ein Röhrchen poolen. Dadurch wird sichergestellt, dass alle T-Zellen die gleiche Behandlung erhalten. - Tag 3 - 5: Resuspendieren Sie die CART-Zellen, um Rosetten aufzubrechen und die Zellen zu zählen. Bringen Sie die endgültige Zellkonzentration auf 1 × 106 Zellen/ml in der TCM mit 1 μg/ml Puromycin. Inkubieren Sie die Zellen bei 37 °C, 5 % CO2.
- Tag 6: Resuspendieren Sie die CART-Zellen, um Rosetten aufzubrechen, entfernen Sie die CD3+/CD28+ -Kügelchen durch magnetische Trennung und zählen Sie die CART-Zellen. Fügen Sie TCM mit 1 μg/ml Puromycin hinzu, um die endgültige Zellkonzentration auf 1 × 106 Zellen/ml zu erhöhen, und inkubieren Sie die Zellen bei 37 °C, 5 % CO2. Bestimmen Sie die CAR-Expression mit Durchflusszytometrie, wie in Schritt 4.4 beschrieben.
7. gRNA-Auswahlphase des CRISPR-Screenings (Tage 8 - 22)
- Tag 8: Bewahren Sie die Proben für das Next-Generation-Sequencing (NGS) auf und beginnen Sie mit der Co-Kultur.
- Waschen Sie die CART-Zellen 3x in der TCM, indem Sie die Proben bei 300 × g für 5 min bei 4 °C zentrifugieren, den Überstand aspirieren und die Proben in 20 mL TCM resuspendieren.
- Zählen Sie die CART-Zellen.
- Bewahren Sie Proben für NGS auf, indem Sie die folgenden Schritte ausführen:
- Aliquotieren Sie genügend CART-Zellen in einem konischen 50-ml-Röhrchen, um eine gRNA-Repräsentation von 500 Zellen pro gRNA aufrechtzuerhalten (z. B. aliquotieren Sie etwa 33 × 106 CART-Zellen, wenn eine CRISPR-Bibliothek mit 65.383 gRNAs verwendet wird). Die Probe wird bei 300 × g 5 min bei 4 °C zentrifugiert.
- Das Zellpellet wird in 1 ml PBS resuspendiert und in ein 2 ml-Mikrozentrifugenröhrchen überführt. Waschen Sie das konische 50-ml-Röhrchen mit 1 mL PBS und übertragen Sie die Wäsche auf das 2-ml-Mikrozentrifugenröhrchen.
- Die Proben werden bei 500 × g 5 min bei 4 °C zentrifugiert. Entfernen Sie den Überstand. Lagern Sie die Zellpellets bei -20 °C.
- Beginnen Sie die Co-Kultur, indem Sie CART-Zellen zu CD19+ JeKo-1-Zellen im Verhältnis 1:1 hinzufügen. Fügen Sie z. B. 80 × 106 CART-Zellen zu 80 × 106 JeKo-1-Zellen hinzu. Bringen Sie das Gesamtvolumen der Co-Kultur mit TCM auf 160 ml. Lagern Sie die Proben in einem Inkubator bei 37 °C, 5 % CO2.
HINWEIS: Wir empfehlen, mit mindestens 80 × 106 CART-Zellen zu beginnen, um die Bibliotheksdarstellung zu erhalten und genügend Zellen zu haben, um die Proben an Tag 15 und Tag 22 zu erhalten.
- Tag 10: Um die CART-Zellen zu restimulieren, sammeln Sie die Co-Kulturen in konische 50-ml-Röhrchen und zentrifugieren Sie die Proben bei 300 × g für 5 min bei 4 °C. Resuspendieren Sie jede Probe in 80 mL TCM und fügen Sie 80 × 106 JeKo-1-Zellen in 80 ml TCM hinzu. Lagern Sie die Proben in einem Inkubator bei 37 °C, 5 % CO2.
- Tag 12 und 14: Restimulieren Sie die CART-Zellen wie in Schritt 7.2.
- Tag 15: Bewahren Sie die Proben für NGS auf und stimulieren Sie die CART-Zellen.
- Isolieren Sie CART-Zellen aus Co-Kulturen, indem Sie zuerst die Zellen zählen und dann ein Positiv-Selektionskit für CD4+ T-Zellen mit einem Positiv-Selektionskit für CD8+ T-Zellen kombinieren, um T-Zellen aus den CART-JeKo-1-Co-Kulturen zu isolieren. Zählen Sie die T-Zellen.
- Die Proben werden für NGS konserviert, indem 33 × 106 CART-Zellen in einem konischen 50-ml-Röhrchen aliquotiert und die Probe bei 300 × g für 5 min bei 4 °C zentrifugiert wird. Resuspendieren Sie die Probe in 1 mL PBS und überführen Sie sie in ein 2 mL Mikrozentrifugenröhrchen. Waschen Sie das konische 50-ml-Röhrchen mit 1 mL PBS und übertragen Sie die Wäsche auf das 2-ml-Mikrozentrifugenröhrchen. Die Proben werden bei 500 × g für 5 min bei 4 °C zentrifugiert; Entfernen Sie den Überstand; und lagern Sie die Zellpellets bei -20 °C.
- Richten Sie Co-Kulturen ein, um das In-vitro-Modell für die Erschöpfung fortzusetzen, indem Sie 80 × 106 CART-Zellen zu 80 × 106 JeKo-1-Zellen hinzufügen. Bringen Sie dann das Gesamtvolumen der Co-Kultur mit TCM auf 160 ml. Lagern Sie die Proben in einem Inkubator bei 37 °C, 5 % CO2.
- Tage 17, 19 und 21: Restimulieren Sie CART-Zellen wie in Schritt 7.2.
- Tag 22: Bewahren Sie die Proben für NGS auf.
- Isolieren Sie CART-Zellen aus der Co-Kultur, indem Sie zuerst die Zellen zählen. Kombinieren Sie dann ein Positiv-Selektionskit für CD4+ T-Zellen mit einem Positiv-Selektionskit für CD8+ T-Zellen, um T-Zellen aus den CART-JeKo-1-Cokulturen zu isolieren. Zählen Sie die T-Zellen.
- Um Proben für NGS zu konservieren, wiederholen Sie Schritt 7.4.2.
8. Vorbereitung der genomischen DNA für die Sequenzierung der nächsten Generation
HINWEIS: Um eine angemessene Abdeckung zu gewährleisten, sollte genomische DNA (gDNA) aus mindestens 33 × 106 CART-Zellen isoliert werden. Zusätzlich sollte die Gesamtheit der gDNA von 33 × 106 CART-Zellen für die Sequenzierung vorbereitet werden.
- Isolieren Sie gDNA aus gefrorenen Zellpellets mit einem gDNA-Isolationskit.
- Reinigen Sie die gDNA durch Ethanolfällung.
- 100% EtOH auf -20 °C vorkühlen.
- Aliquot eluierte DNA gleichmäßig in mehrere Mikrozentrifugenröhrchen (200-250 μL). Zu jedem Aliquot wird Folgendes hinzugefügt: zwei Volumina mit 100 % EtOH (500 μl für 250 μl aliquot oder 400 μl für 200 μl); 0,1 Volumen von 3 M Natriumacetat (25 μl für 250 μl aliquot oder 20 μl für 200 μl); und 1 μl 20 mg/ml Glykogen. Die Proben über Nacht bei -20 °C inkubieren.
- Am zweiten Tag werden die Proben 20 Minuten lang bei 13.000 × g geschleudert, um die gefällte DNA zu pelletieren. Entfernen Sie den Überstand. Waschen Sie die Pellets mit 1 mL 70% EtOH, indem Sie sie 10 Minuten lang bei 7.500 × g schleudern.
- Restliches EtOH vollständig entfernen. Lassen Sie die Pellets 30-45 Minuten trocknen.
- Resuspendieren Sie die Pellets in 50 μl sterilem Wasser.
- Bereiten Sie gDNA für NGS vor.
HINWEIS: NGS-Primer für diese CRISPR-Bibliothek, die gRNAs mit Illumina-Adaptersequenzen amplifizieren, wurden in einer früheren Veröffentlichungentworfen 27. Die Verwendung eines anderen Reverse-Primers für jede Probe führt zu einem Barcode für jede Probe und ermöglicht das Pooling von Proben während der Sequenzierung.- Bereiten Sie die Proben für die PCR mit folgendem Reaktionsgemisch vor (Gesamtvolumen = 50 μL): High-Fidelity-PCR-Mastermix, 2x: 25 μL; gepoolte Template-DNA, 0,5 μg: 1 μL; Vorwärtsprimer der NGS-Bibliothek, 10 μM: 1,25 μL; Reverse-Primer der NGS-Bibliothek, 10 μM: 1,25 μL; DNasefreies Wasser: 21,5 μL.
- PCR unter den folgenden Zyklusbedingungen durchführen: Zyklus 1: Denaturierung bei 98 °C für 3 Minuten; Zyklen 2-23: Denaturierung 98 °C für 10 s, Glühen bei 63 °C für 10 s, Ausfahren bei 72 °C für 25 s; Zyklus 24: Bei 72 °C für 2 min verlängern.
- Fassen Sie die PCR-Reaktionen für jede Probe zusammen und reinigen Sie das PCR-Produkt mit einem PCR-Aufreinigungskit gemäß den Anweisungen des Herstellers.
- Führen Sie 3 μg des PCR-Produkts für jede Probe auf ein 2 % (w/v) Agarosegel auf.
- Entfernen Sie das PCR-Produkt (~260-270 bp) aus dem Gel, indem Sie die DNA aus dem Gel extrahieren, indem Sie ein Gel-Extraktionskit gemäß den Anweisungen des Herstellers verwenden. Lagern Sie die extrahierten Proben bei -20 °C.
- Führen Sie mit den Proben eine Next-Generation-Sequenzierung (NGS) durch. Streben Sie mehr als 500 Reads pro gRNA in der Bibliothek an.
9. Analyse der Sequenzierungsergebnisse
- Um NGS zu analysieren, verwenden Sie die MAGeCK-VISPR-Plattform28. Verwenden Sie die Standardeinstellungen für den Algorithmus zur Maximum-Likelihood-Schätzung (MAGeCK-MLE). So bereiten Sie die Ausgabedateien für die Analyse vor:
- Führen Sie FASTQC aus, um die Qualität der Sequenzdatei31 zu bewerten.
- Führen Sie Paired-End-Sequenzierungsdateien für jedes Sample mit bbmerge32 zusammen.
- Analysieren Sie die Proben auf der MAGeCK-VISPR-Plattform mit dem MAGeCK-MLE-Algorithmus. Normalisieren Sie für die MAGeCK-MLE-Analyse alle gRNA-Zählungen auf die Liste von 1.000 gRNAs, die nicht auf das Ziel abzielen.
- Bewerten Sie die Qualität der Sequenzierungsdatei, indem Sie den GC-Inhalt, die Basisqualität, die Sequenzierungs-Reads und die zugeordneten Reads bewerten.
- Bewerten Sie den Erfolg der selektiven Druckphase des CRISPR-Screenings durch Bewertung von Veränderungen des Gini-Index, der Clusterbildung der Hauptkomponentenanalyse (PCA) und der Signalwege, die mit der Liste negativ ausgewählter Gene verbunden sind32.
- Hinterfragen Sie die Top-Treffer, indem Sie Literaturrecherchen durchführen und Pfadanalysen durchführen.
Ergebnisse
Um Gene und Signalwege zu untersuchen, die bearbeitet werden können, um die Aktivität von CART-Zellen unvoreingenommen zu verbessern, haben wir einen in vitro genomweiten CRISPR-Knockout-Screen entwickelt (Abbildung 1). Dieses Sieb besteht aus zwei Phasen: einer Produktionsphase für CART-Zellen und einer selektiven Druckphase. In der Phase der Herstellung von CART-Zellen werden zunächst mindestens 110 × 106 T-Zellen aus gesunden Spen...
Diskussion
Die Genom-Editierung ist zu einem leistungsfähigen Werkzeug geworden, um sowohl die Mechanismen der Resistenz gegen Therapien zu verstehen als auch neuartige CART-Zelltherapien zu entwickeln, um die Langlebigkeit und Aktivität von CART-Zellen zu verbessern 16,17,26. Während einige Gen-Editing-Strategien sowohl in präklinischen Modellen als auch in klinischen Studien Verbesserungen der CART-Z...
Offenlegungen
SSK ist ein Erfinder von Patenten auf dem Gebiet der CAR-Immuntherapie, die an Novartis (durch eine Vereinbarung zwischen der Mayo Clinic, der University of Pennsylvania und Novartis), Humanigen (über die Mayo Clinic), Mettaforge (über die Mayo Clinic) und MustangBio (über die Mayo Clinic) sowie an Chymal Therapeutics (über die Mayo Clinic) lizenziert sind. CS, CMR und SSK sind Erfinder von Patenten, die an Immix Biopharma lizenziert sind. SSK erhält Forschungsgelder von Kite, Gilead, Juno, BMS, Novartis, Humanigen, MorphoSys, Tolero, Sunesis/Viracta, LifEngine Animal Health Laboratories Inc. und Lentigen. SSK hat an Beratungsgesprächen mit Kite/Gilead, Calibr, Luminary Therapeutics, Humanigen, Juno/BMS, Capstan Bio und Novartis teilgenommen. SSK war bei Humanigen und Carisma im Ausschuss für Datensicherheit und -überwachung tätig. SSK hat einen Berater für Torque, Calibr, Novartis, Capstan Bio, BMS, Carisma und Humanigen getrennt. CMS und SSK sind Erfinder des geistigen Eigentums, das aus diesem Protokoll hervorgegangen ist.
Danksagungen
Diese Studie wurde teilweise vom Mayo Clinic Center for Individualized Medicine (SSK), dem Mayo Clinic Comprehensive Cancer Center (SSK), dem Mayo Clinic Center for Regenerative Biotherapeutics (SSK), den National Institutes of Health K12CA090628 (SSK) und R37CA266344-01 (SSK), dem Department of Defense Grant CA201127 (SSK), der Predolin Foundation (SSK) und der Minnesota Partnership for Biotechnology and Medical Genomics (SSK) finanziert. CMS wird von der Mayo Clinic Graduate School of Biomedical Sciences unterstützt. Das CRISPR-Screen-Schema (Abbildung 1) wurde mit BioRender.com erstellt (Siegler, L. (2022) https://BioRender.com/k71r054).
Materialien
| Name | Company | Catalog Number | Comments |
| 293T cells | ATCC | CRL-3216 | Cells used for lentivirus production |
| Biotin ProteinL Antibody | GenScript | M00097 | anti-kappa chain antibody for CAR detection |
| Bovine Serum Albumin | Millipore Sigma | A7906 | |
| Carbenicillin disodium salt | Millipore Sigma | C1389-1G | Carbenicillin antibiotic |
| CD4 Isolation Beads | Miltenyi Biotec | 130-045-101 | |
| CD8 Isolation Beads | Miltenyi Biotec | 130-045-201 | |
| CTS (Cell Therapy Systems) Dynabeads CD3/CD28 | Gibco | 40203D | |
| Cytoflex | Beckman Coulter | NC2279958 | |
| DNase-Free Water | Invitrogen | AM9937 | |
| Dulbecco's modified eagle's medium (DMEM) | Corning | 10-017-CV | |
| Dulbecco's Phosphate-Buffered Saline | Gibco | 14190-144 | |
| EasySep Human T Cell Isolation Kit | STEMCELL Technologies | 17951RF | Negative isolation kit |
| Endura Electrocompetent Cells | Biosearch Technologies | 60242-1 | Electrocompetent cells with recovery medium |
| Ethanol | Millipore Sigma | E7023 | |
| Fetal bovine serum (FBS) | Corning | 35-010-CV | |
| GeCKO v2 CRISPR Knockout Pooled Library A | AddGene | 1000000048 | CRISPR library plasmid |
| Gene Pulser II | Bio-Rad | 165-2105 | Electroporator |
| Glycogen | Millipore Sigma | 10901393001 | |
| JeKo-1 | ATCC | CRL-3006 | CD19+ target cells |
| Lipofectamine 3000 Transfection Reagent | ThermoFisher Scientific | L3000075 | Transfection reagent kit with a transfection reagent (Lipofectamine 3000 Reagent) and a neutral co-lipid reagent (p3000) |
| LIVE/DEAD Aqua | Invitrogen | L34966 | |
| Lymphoprep | STEMCELL Technologies | 7851 | Density gradient medium |
| Machery-Nagel NucleoBond Xtra Maxi Kits | ThermoFisher Scientific | 12748412 | Maxi-prep kit |
| NEBNext High-Fidelity 2X PCR MasterMix | New England BioLabs | M0541S | High fidelity PCR mastermix |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985-070 | Reduced serum medium |
| pCMVR8.74 | AddGene | 22036 | Lentiviral packaging plasmid |
| Pennicillin-streptomycin-glutamine (100X) | Life Technologies | 10378-016 | |
| pMD2.G | AddGene | 12259 | VSV-G envelope expressing plasmid |
| Pooled Human AB Serum | Innovative Research | ISERABHI | |
| Puromycin | Millipore Sigma | P8833 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | Gek extraction kit |
| Qucik-DNA Midiprep Plus Kit | Zymo Research | D4075 | Kit used to isolate gDNA |
| RoboSep-S | STEMCELL Technologies | 21000 | Automated cell separator |
| Roswell Park Memorial Institute 1640 Medium (RPMI) | Gibco | 21870092 | |
| SepMate-50 | STEMCELL Technologies | 85450 | Density gradient separation tube |
| Sodium Acetate | Invitrogen | AM9740 | |
| Sodium Azide | Fisher Scientific | 71448-16 | |
| Streptavidin Antibody (PE) | BioLegend | 405203 | Secondary antibody used for CAR detection |
| T100 Thermal Cycler | Bio-Rad | 1861096 | |
| Ultracentrifuge (Optima XPN-80) | BeckmanCoulter | A99839 | |
| Vacuum Filter Systems, 0.22 µm | ThermoFisher Scientific | 567-0020 | |
| Vacuum Filter Systems, 0.45 µm | ThermoFisher Scientific | 165-0045 | |
| X-VIVO 15 Serum-Free Hematopoietic Cell Medium | Lonza | 04-418Q | Hematopoietic cell medium |
Referenzen
- Neelapu, S. S., et al. 5-year follow-up supports curative potential of axicabtagene ciloleucel in refractory large B-cell lymphoma (zuma-1). Blood. 141 (19), 2307-2315 (2023).
- Neelapu, S. S., et al. Axicabtagene ciloleucel CAR T-cell therapy in refractory large B-cell lymphoma. N Engl J Med. 377 (26), 2531-2544 (2017).
- Locke, F. L., et al. Long-term safety and activity of axicabtagene ciloleucel in refractory large B-cell lymphoma (zuma-1): A single-arm, multicentre, phase 1-2 trial. Lancet Oncol. 20 (1), 31-42 (2019).
- Maude, S. L., et al. Tisagenlecleucel in children and young adults with B-cell lymphoblastic leukemia. N Engl J Med. 378 (5), 439-448 (2018).
- Schuster, S. J., et al. Tisagenlecleucel in adult relapsed or refractory diffuse large B-cell lymphoma. N Engl J Med. 380 (1), 45-56 (2018).
- Bai, Z., et al. Single-cell antigen-specific landscape of CAR T infusion product identifies determinants of CD19-positive relapse in patients with all. Sci Adv. 8 (23), eabj2820 (2022).
- Fraietta, J. A., et al. Determinants of response and resistance to CD19 chimeric antigen receptor (CAR) T cell therapy of chronic lymphocytic leukemia. Nat Med. 24, 563-571 (2018).
- Gennert, D. G., et al. Dynamic chromatin regulatory landscape of human CAR T cell exhaustion. Proc Natl Acad Sci USA. 118 (30), e2104758118 (2021).
- Deng, Q., et al. Characteristics of anti-CD19 CAR T cell infusion products associated with efficacy and toxicity in patients with large B cell lymphomas. Nat Med. 26 (12), 1878-1887 (2020).
- Sterner, R. M., Cox, M. J., Sakemura, R., Kenderian, S. S. Using CRISPR/CAS9 to knock out GM-CSF in CAR-T cells. J Vis Exp. (149), e59629 (2019).
- Sterner, R. M., et al. GM-CSF inhibition reduces cytokine release syndrome and neuroinflammation but enhances CAR-T cell function in xenografts. Blood. 133 (7), 697-709 (2019).
- Cox, M. J., et al. GM-CSF disruption in CART cells modulates T cell activation and enhances CART cell anti-tumor activity. Leukemia. 36 (6), 1635-1645 (2022).
- Ottaviano, G., et al. Phase 1 clinical trial of CRISPR-engineered CAR19 universal T cells for treatment of children with refractory B cell leukemia. Sci Transl Med. 14 (668), eabq3010 (2022).
- Joung, J., et al. Genome-scale CRISPR-CAS9 knockout and transcriptional activation screening. Nat Protoc. 12, 828-863 (2017).
- Mccutcheon, S. R., et al. Transcriptional and epigenetic regulators of human CD8+ T cell function identified through orthogonal CRISPR screens. Nat Genet. 55, 2211-2223 (2023).
- Zhu, W., Kelly, C., Dagur, P., Dunbar, C. E., Cordes, S. CRISPR activation screen to optimize chimeric antigen receptor (CAR) T cell immunophenotype. Blood. 142 (Supplement 1), 4820-4820 (2023).
- Wang, D., et al. CRISPR screening of CAR T cells and cancer stem cells reveals critical dependencies for cell-based therapies. Cancer Discov. 11 (5), 1192-1211 (2021).
- Shifrut, E., et al. Genome-wide CRISPR screens in primary human T cells reveal key regulators of immune function. Cell. 175 (7), 1958-1971.e15 (2018).
- Belk, J. A., et al. Genome-wide crispr screens of T cell exhaustion identify chromatin remodeling factors that limit T cell persistence. Cancer Cell. 40 (7), 768-786.e7 (2022).
- Jiang, P., et al. Single-cell ATAC-seq maps the comprehensive and dynamic chromatin accessibility landscape of CAR-T cell dysfunction. Leukemia. 36 (11), 2656-2668 (2022).
- Rossi, J., et al. Preinfusion polyfunctional anti-CD19 chimeric antigen receptor T cells are associated with clinical outcomes in NHL. Blood. 132 (8), 804-814 (2018).
- Beider, K., et al. Molecular and functional signatures associated with CAR T cell exhaustion and impaired clinical response in patients with B cell malignancies. Cells. 11 (7), 1140 (2022).
- Wherry, E. J. T. cell exhaustion. Nat Immunol. 12, 492-499 (2011).
- Prinzing, B., et al. Deleting DNMT3A in CAR T cells prevents exhaustion and enhances antitumor activity. Sci Transl Med. 13, eabh0272 (2021).
- Lynn, R. C., et al. C-jun overexpression in CAR T cells induces exhaustion resistance. Nature. 576, 293-300 (2019).
- Tang, N., et al. TGF-beta inhibition via CRISPR promotes the long-term efficacy of CAR T cells against solid tumors. JCI Insight. 5 (4), e133977 (2020).
- Joung, J., et al. Genome-scale CRISPR-CAS9 knockout and transcriptional activation screening. Nat Protoc. 12 (4), 828-863 (2017).
- Li, W., et al. Quality control, modeling, and visualization of CRISPR screens with MAGeCK-VISPR. Genome Biol. 16, 281 (2015).
- Sakemura, R., et al. Targeting cancer-associated fibroblasts in the bone marrow prevents resistance to CART-cell therapy in multiple myeloma. Blood. 139 (26), 3708-3721 (2022).
- Stewart, C. M., et al. IL-4 drives exhaustion of CD8+ CART cells. Nat Commun. 15 (1), 7921 (2024).
- . FastQC: A quality control tool for high throughput sequence data Available from: https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010)
- . Bbmerge Available from: https://github.com/BioInfoTools/BBMap/blob/master/sh/bbmerge.sh (2019)
- Pattanayak, V., et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed CAS9 nuclease specificity. Nat Biotechnol. 31 (9), 839-843 (2013).
- Doench, J. G., et al. Rational design of highly active sgRNAs for CRISPR-CAS9-mediated gene inactivation. Nat Biotechnol. 32 (12), 1262-1267 (2014).
- Dong, M. B., et al. Systematic immunotherapy target discovery using genome-scale in vivo CRISPR screens in CD8 T cells. Cell. 178 (5), 1189-1204.e23 (2019).
- Zhou, P., et al. Single-cell CRISPR screens in vivo map T cell fate regulomes in cancer. Nature. 624 (7990), 154-163 (2023).
- Sutra Del Galy, A., et al. In vivo genome-wide CRISPR screens identify SOCS1 as intrinsic checkpoint of CD4+ TH1 cell response. Sci Immunol. 6 (66), eabe8219 (2021).
- Ramos, A., et al. Leukemia-intrinsic determinants of CAR-T response revealed by iterative in vivo genome-wide CRISPR screening. Nat Commun. 14 (1), 8048 (2023).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten