Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Realización de un cribado de knockout CRISPR en todo el genoma in vitro en células T receptoras de antígenos quiméricos
En este artículo
Resumen
El artículo describe un protocolo para la aplicación de un modelo in vitro de agotamiento para completar un cribado de knockout de CRISPR en todo el genoma en células T receptoras de antígenos quiméricos de donantes sanos.
Resumen
La terapia celular con receptor de antígeno quimérico T (CART) es una forma innovadora de inmunoterapia dirigida que ha revolucionado el tratamiento del cáncer. Sin embargo, la respuesta duradera sigue siendo limitada. Estudios recientes han demostrado que el panorama epigenético de los productos de células CART antes de la infusión puede influir en la respuesta a la terapia, y la edición genética se ha propuesto como una solución. Sin embargo, se necesita más trabajo para determinar la estrategia óptima de edición de genes. Las pantallas CRISPR de todo el genoma se han convertido en herramientas populares tanto para investigar los mecanismos de resistencia como para optimizar las estrategias de edición de genes. Sin embargo, su aplicación a las células primarias presenta muchos desafíos. Aquí describimos un método para completar un cribado de knockout de CRISPR en todo el genoma en células CART de donantes sanos. Como modelo de prueba de concepto, diseñamos este método para investigar el desarrollo del agotamiento en las células CART dirigidas al antígeno CD19. Sin embargo, creemos que este enfoque se puede utilizar para abordar una variedad de mecanismos de resistencia a la terapia en diferentes construcciones de CAR y modelos tumorales.
Introducción
La terapia con células del receptor de antígeno quimérico T (CART) ha demostrado un éxito impresionante en el tratamiento de las neoplasias malignas de células B; Sin embargo, la respuesta duradera se limita al 30-40%1,2,3,4,5. Si bien los investigadores han desarrollado y probado varios enfoques para abordar los mecanismos de resistencia a la terapia celular CART, incluida la optimización del diseño de CAR, la edición de genes y las terapias combinadas, el desarrollo de resistencia sigue siendo en gran medida desconocido. Recientemente, ha habido cada vez más evidencia de que el perfil de expresión génica basal de los productos de células CART antes de la infusión es un determinante importante tanto de la toxicidad como de la eficacia de la terapia CART 6,7,8,9. Como tal, la edición de genes durante la producción de células CART se ha convertido en un enfoque popular para mejorar el éxito de la terapia celular CART.
Por ejemplo, nuestro laboratorio demostró previamente que el uso de la tecnología Cas9 de repeticiones palindrómicas cortas agrupadas regularmente interespaciadas (CRISPR) para eliminar el gen factor estimulante de colonias de granulocitos y macrófagos (GM-CSF) mejora la actividad de las células CART y reduce los signos de toxicidad asociada a CART 10,11,12. Además, la terapia celular modificada con CRISPR se ha probado en ensayos clínicos y ha demostrado eficacia y seguridad13. En conjunto, esto indica que la edición de genes con tecnología CRISPR no solo puede mejorar nuestra comprensión de la biología celular CART, sino también generar productos celulares CART traducibles.
Las pantallas knockout CRISPR de todo el genoma se han convertido en herramientas cada vez más populares en la investigación de la biología del cáncer para comprender los mecanismos de resistencia a las terapias. En esta técnica, decenas de miles de ARN guía (ARNg) se administran a grupos de células para fomentar la entrada de un ARNg por célula14. Luego, las células se someten a condiciones inductoras de presión en las que las células transducidas con ARNg dirigidos a genes esenciales mueren y las células transducidas con ARNg dirigidos a genes inhibidores sobreviven y proliferan. Mediante el uso de la secuenciación de próxima generación, podemos determinar cómo cambia la representación del ARNg a lo largo de la pantalla CRISPR14.
Sin embargo, la escala y el tiempo de selección necesarios para las perturbaciones genéticas en todo el genoma pueden ser difíciles de lograr en las células primarias, como las células CART. Por ello, los grupos han utilizado cribados CRISPR dirigidos para comprender mejor los mecanismos de la resistencia terapéutica 15,16. Las pantallas de destino suelen ser más fáciles de completar en las celdas primarias porque tienen bibliotecas más pequeñas que requieren menos celdas para lograr una representación adecuada de la biblioteca. Si bien estos estudios han mejorado nuestra comprensión de los mecanismos de resistencia a la terapia celular CART, los cribados dirigidos introducen un sesgo debido a la selección manual de las dianas genéticas. Este artículo busca describir un método para completar un cribado CRISPR in vitro de todo el genoma en células CART de donantes sanos. Como tal, este enfoque de alto rendimiento permite la identificación eficiente e imparcial de vías y genes clave que se pueden editar para mejorar las respuestas terapéuticas 17,18,19.
En particular, el protocolo descrito en este artículo está diseñado para aumentar la comprensión del campo sobre el agotamiento de las células CART al completar la pantalla de knockout de CRISPR en todo el genoma con un modelo in vitro para el agotamiento. El agotamiento es un destino disfuncional de las células CART que se ha implicado en la falta de respuesta a la terapia con células CART 20,21,22. Se sabe que este destino celular está regulado epigenéticamente y se caracteriza por una disminución en la proliferación de células CART, una disminución en la producción de citocinas efectoras y un aumento en la expresión de receptores inhibidores23. En la literatura previa, la edición genética ha sido capaz de prevenir el desarrollo del agotamiento mediante la regulación positiva o negativa de genes clave 24,25,26. Dada la disminución de la capacidad proliferativa a medida que las células CART se agotan y la evidencia de que la edición genética puede prevenir su aparición, modelamos nuestra pantalla CRISPR knockout in vitro de todo el genoma en este destino celular. Sin embargo, este protocolo podría modificarse en el futuro para explorar otros mecanismos de resistencia a la terapia celular CART.
Protocolo
Es importante destacar que el protocolo que se describe a continuación sigue las pautas y ha recibido la aprobación de la Junta de Revisión Institucional de Mayo Clinic (IRB 18-005745) y el Comité Institucional de Bioseguridad (IBC HIP00000252.43). Todos los trabajos de cultivo celular, incluida la producción lentiviral, deben realizarse en una campana de cultivo celular con el equipo de protección personal adecuado. En particular, el trabajo lentiviral debe realizarse bajo precauciones de nivel de bioseguridad 2 (BSL-2), incluido el uso de lejía al 10% para desinfectar los artículos antes de desecharlos.
1. Amplificación de la biblioteca CRISPR
- Amplifique la biblioteca CRISPR de acuerdo con las recomendaciones del fabricante. Brevemente, electropore la biblioteca CRISPR en cuatro alícuotas de celdas electrocompetentes completando lo siguiente:
- Añada 2 μL de plásmido de biblioteca CRISPR de 50 ng/μL a 25 μL de células electrocompetentes en una cubeta de 1,0 mm.
- Electropore las muestras utilizando los siguientes ajustes: 10 μF, 600 ohmios, 1.800 voltios y una constante de tiempo entre 3,5 ms y 4,5 ms.
- Recupere en 975 μL de medio de recuperación y transfiéralo a un tubo con 1 mL adicional de medio de recuperación.
- Gire todas las alícuotas a 250 RPM durante 1 h a 37 °C.
- Placa de las transformaciones:
- Agrupe todas las alícuotas de las celdas electroporadas y mezcle bien.
- Coloque todas las células electroporadas esparciendo 400 μL de la mezcla de transformación en placas de caldo Luria (LB) precalentadas de 10 cm con 100 μg/mL de carbenicilina. Coloque la placa en una incubadora a 32 °C durante 14 h.
- Cosecha las colonias:
- Pipetear 1 mL de medio LB + 100 μg/mL de carbenicilina en cada placa. Raspe las colonias de cada plato y recójalas en un tubo cónico de 50 ml.
- Agregue otro 1 mL de medio LB + 100 μg/mL de carbenicilina a cada placa para lavar y asegurar una recuperación completa.
- Realiza una maxi-preparación siguiendo las instrucciones de un kit de maxi-preparación.
- Agrupa el ADN plasmídico resultante.
2. Secuenciación de nueva generación para verificar la representación basal del ARNg en la biblioteca CRISPR
NOTA: Los cebadores NGS para esta biblioteca CRISPR han sido diseñados en una publicación anterior27. El uso de un cebador inverso diferente para cada muestra mostrará un código de barras de cada muestra y permitirá la agrupación de muestras durante la secuenciación.
- Prepare las muestras para PCR con la siguiente mezcla de reacción (volumen total = 50 μL): mezcla maestra de PCR de alta fidelidad, 2x: 25 μL; ADN molde agrupado, 0,5 μg: 1 μL; cebador delantero de la biblioteca NGS, 10 μM: 1,25 μL; cebador inverso de la biblioteca NGS, 10 μM: 1,25 μL; Agua libre de DNasa: 21,5 μL.
- Realice PCR con las siguientes condiciones de ciclo: ciclo 1: desnaturalización a 98 °C durante 3 min; ciclos 2-23: desnaturalización a 98 °C durante 10 s, recocido a 63 °C durante 10 s, extensión a 72 °C durante 25 s; ciclo 24: extender a 72 °C durante 2 min.
- Purifique la reacción de PCR agrupando las reacciones de PCR para cada muestra y purificando el producto de PCR con un kit de purificación de PCR de acuerdo con las instrucciones del fabricante.
- Coloque 3 μg del producto de PCR para cada muestra en un gel de agarosa al 2% (p/v).
- Retire el producto de PCR (~ 260-270 bp) del gel extrayendo el ADN del gel mediante el uso de un kit de extracción de gel de acuerdo con las instrucciones del fabricante. Apunta a tener al menos 500 ng de ADN.
- Almacene las muestras extraídas a -20 °C.
- Realice la secuenciación de próxima generación (NGS) con las muestras y apunte a 100 lecturas por ARNg27.
- Analice los resultados de la secuenciación con la tubería MAGeCK-VISPR y asegúrese de que más del 99,5% de los ARNg de la biblioteca estén representados28.
3. Producción de lentivirus de la biblioteca CRISPR y de expresión CAR
- Prepare lentivirus como se describió anteriormente 10,29.
- Día 1: Comience con frascos T175 de 80-90% de células 293T confluentes que han estado creciendo en medios D10 compuestos por un 10% de suero fetal bovino (FBS) (v/v), un 1% de penicilina-estreptomicina-glutamina (PSG) (v/v) y medio de águila modificado de Dulbecco. Antes de comenzar, caliente los reactivos de transfección y el ADN a temperatura ambiente y cuantifique el ADN.
- Para cada matraz de 293T, etiquete dos tubos cónicos A y B de 50 mL. Añadir 4,5 mL de medio sérico reducido al tubo A y al tubo B.
- En un tubo de microcentrífuga estéril de 1,5 ml, mezcle el siguiente ADN y déjelo reposar durante 1 minuto: 18 μg de pCMVR8.74 (gag, pol, tat y rev), 7 μg de pMD2.G (VSV-G) y 15 μg de vector de expresión lentiviral (vectores de la biblioteca CRISPR A o CAR19-28ζ).
- Añadir 129 μL de reactivo de transfección al tubo A. Mezcle el tubo A bien mediante vórtice con el dedo.
- Agregue la mezcla de ADN al tubo B. Mezcle bien el tubo B mediante vórtice con los dedos.
- Añadir 111 μL de reactivo colipídico neutro al tubo B. Mezcle bien el tubo B mediante vórtice con los dedos.
- Agregue el contenido del tubo A al tubo B y mezcle bien con el vórtice de los dedos. Incubar el tubo B durante 30 min a temperatura ambiente.
- Hacia el final del período de incubación de 30 minutos, prepare las células 293T aspirando los medios existentes. Agregue 16 mL de medio R10 compuesto por 10% de FBS (v/v), 1% de PSG (v/v) y Roswell Park Memorial Institute (RPMI) 1640.
- Después del período de incubación de 30 minutos, agregue el contenido del tubo B (aproximadamente 9 mL) gota a gota al matraz de células 293T. Coloque el matraz en el suelo y gírelo suavemente antes de incubarlo a 37 °C, 5% de CO2.
- El día 2 (aproximadamente 24 horas después de la transfección), recoja lentivirus de la siguiente manera:
NOTA: Enfríe previamente todo el equipo y los materiales y realice lo siguiente con hielo para mantener frío el sobrenadante viral.- Transfiera el sobrenadante de los matraces 293T transfectados a tubos cónicos preenfriados de 50 mL.
- Agregue 30 mL de medios R10 precalentados a los matraces 293T.
- Pellet restos celulares centrifugando los tubos cónicos de 50 mL a 900 × g durante 10 min a 4 °C en rotores cerrados.
- Después de centrifugar, filtre el sobrenadante con un filtro de 0,45 μm. Centrifugar el filtrado en una ultracentrífuga a 112.700 × g a 4 °C durante 2 h.
- Después de las 2 h de centrifugado, aspire el sobrenadante hasta que queden aproximadamente 5 mL. Vuelva a suspender las partículas virales pipeteando suavemente hacia arriba y hacia abajo 10 veces. Almacene la suspensión viral a 4 °C durante la noche.
- El día 3, recoja y almacene lentivirus de la siguiente manera:
- Transfiera el sobrenadante de los matraces 293T transfectados a tubos cónicos preenfriados de 50 mL.
- Añadir 30 mL de lejía al 10% a los matraces 293T.
- Granular los restos celulares en el sobrenadante centrifugando los tubos cónicos de 50 mL a 900 × g durante 10 min a 4 °C en rotores cerrados.
- Después de centrifugar, filtre el sobrenadante con un filtro de 0,45 μm. Centrifugar el filtrado en una ultracentrífuga a 112.700 × g a 4 °C durante 2 h.
- Después de la centrífuga de 2 h, aspire el sobrenadante hasta que queden aproximadamente 2 mL y vuelva a suspender pipeteando suavemente hacia arriba y hacia abajo 10 veces. Almacene el sobrenadante viral en alícuotas de 300-500 μL y manténgalo a -80 °C.
4. Cálculo del título viral para lentivirus que expresan CAR
NOTA: El lentivirus de la CAR se tituló como se describió anteriormente10,12.
- El día 1, active y coloque placas de células T.
- Aísle 1 × 106 células T de PBMC de donantes sanos utilizando un kit de selección de negativos.
- Cuenta las células T.
- Lave suficientes perlas CD3+/CD28+ para estimular las células T en una proporción de 3:1 perla:célula. Por ejemplo, para estimular 1 × 106 células T, lave 3 × 106 perlas. Para ello, lava las cuentas completando las siguientes 3x:
- Utilice la separación magnética para quitar las cuentas de la suspensión.
- Retire el sobrenadante.
- Agregue 1000 μL de medio de células T (TCM) compuesto por 10% de suero humano (v/v), 1% de PSG (v/v) y medio de células hematopoyéticas.
NOTA: El TCM debe esterilizarse antes de su uso filtrando con un filtro de vacío estéril de 0,45 μm y luego con un filtro de vacío estéril de 0,22 μm. - Agregue perlas a la suspensión de celdas T en una proporción de perlas de 3:1.
- Resuspender las células T a 1 × 106 células/mL en TCM.
- Agregue 100 μL de células T activadas a 10 pocillos de una placa de 96 pocillos. Incubar la placa a 37 °C, 5% de CO2.
- El día 2, agregue una dilución de lentivirus CAR creando una placa de dilución de la siguiente manera:
- Agregue 100 μL de TCM a siete pocillos de una placa de 96 pocillos.
- Al primer pocillo, agregue 50 μL del virus de interés; mezclar bien.
- Diluir aún más el virus en serie añadiendo 50 μL del pocillo 1 al pocillo 2; mezclar bien.
- Agregue 50 μL desde el pocillo 2 hasta el pocillo 3 y continúe este patrón hasta el pocillo 7.
- Después de mezclar el pocillo 7, retire 50 μL. Asegúrese de que todos los pocillos tengan un volumen total de 100 μL.
- Agregue 50 μL de cada pocillo de la placa de dilución a los primeros siete pocillos de la placa de células T a partir del día 1. Mezclar bien.
- A los últimos tres pocillos de la placa de células T, agregue 50 μL de TCM; mezclar bien. Almacene la placa en una incubadora a 37 °C, 5% de CO2.
- En el día 3, alimente las células añadiendo 100 μL de MTC a todos los pocillos de la placa de células T; mezclar bien.
- El día 4, realice una citometría de flujo para detectar el porcentaje de células que expresan CAR con un anticuerpo anti-cadena kappa, un marcador sustituto de la expresión de CAR, completando lo siguiente:
- Gire la placa de células T a 1.000 × g durante 3 min y decante.
- Lave la placa 3 veces con tampón de anticuerpos anti-cadena kappa (compuesto por un 4% de albúmina sérica bovina en PBS (p/v)) añadiendo 200 μL de tampón de anticuerpos anti-cadena kappa, girando la placa a 1.000 × g durante 3 min y decantando.
- Agregue un anticuerpo anti-cadena kappa de biotina a cada pocillo. Incubar durante 45 min en un balancín a 4 °C.
- Lave la placa 3 veces con tampón de anticuerpos anti-cadena kappa como en el paso 4.4.2.
- Tiñir las células para la citometría de flujo con un anticuerpo de estreptavidina conjugado con fluorocromo y un anticuerpo vivo/muerto. Incubar las muestras durante 15 minutos a temperatura ambiente en la oscuridad.
- Lavar las muestras una vez en tampón de flujo (compuesto por 2% de FBS (v/v) y 1% de azida de sodio (v/v) en PBS) añadiendo 200 μL de tampón de flujo, girando la placa a 1.000 × g durante 3 min, y decantando.
- Vuelva a suspender en 100 μL de tampón de flujo y ejecute en el citómetro.
- Realice la compuerta negativa con los tres pozos no transducidos.
5. Cálculo del título viral para el lentivirus de la biblioteca CRISPR
NOTA: Este protocolo describe un número mínimo de lentivirus de la biblioteca CRISPR, pero se puede ampliar para acomodar más linfocitos T y, por lo tanto, mayores volúmenes de virus.
- En el día 0, active las células T añadiendo 18 × 106 perlas CD3+/CD28+ (relación 3:1 perla:célula ) a 6 linfocitos T × 106 y llevando el volumen final a 6 mL con la MTC. Alícuota de 0,5 mL de la suspensión de la célula T en 12 pocillos de una placa de 48 pocillos. Almacenar en incubadora a 37 °C, 5% CO2.
- Veinticuatro horas más tarde, el día 1, agregue el virus CAR a una multiplicidad de infección (MOI) de 3 a cada pocillo. Mezcle bien pipeteando hacia arriba y hacia abajo varias veces. Almacenar en incubadora a 37 °C, 5% CO2.
- Cuarenta y ocho horas más tarde, el día 2, agregue una serie de volúmenes de virus de la biblioteca CRISPR a las células CART activadas agregando uno de los siguientes volúmenes de virus CRISPR a cada pocillo (0 μL, 0 μL, 6,25 μL, 6,25 μL, 12,5 μL, 12,5 μL, 25 μL, 25 μL, 50 μL, 50 μL, 100 μL o 100 μL). Mezcle bien pipeteando hacia arriba y hacia abajo varias veces. Almacenar en incubadora a 37 °C, 5% CO2.
- El día 3, agregue 1 μg de puromicina por ml de medio a la mitad de los pocillos, incluido un pocillo que recibió cada uno de los siguientes volúmenes del virus CRISPR (0 μL, 6,25 μL, 12,5 μL, 25 μL, 50 μL y 100 μL). Los otros pocillos no se tratarán con puromicina.
- A partir de los días 4 a 8, cuente las células T diariamente y ajuste la densidad celular final a 1 × 106 células/mL con 1 μg/mL de puromicina en MTC para los pocillos de tratamiento con puromicina y con MTC para los pocillos no tratados.
- En el día 8, cuente el número de células vivas por pocillo y calcule las unidades de transducción/ml del sobrenadante viral utilizando la siguiente ecuación:
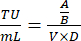
Donde A: Recuento de células vivas en pocillo transducido viralmente; B: Recuento de células vivas en el pozo de control sin selección de antibióticos; V: Volumen de virus utilizado para la infección en cada pocillo; D: Pliegue de dilución. - Usando el pocillo TU/mL calculado a partir de un pocillo que muestra aproximadamente un 20% de eficiencia de transducción, calcule el volumen de virus necesario para transducir 1 × 106 células a un MOI de 0,3 utilizando la siguiente fórmula:
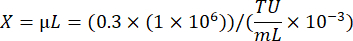
6. Fase de producción de células CART de la pantalla CRISPR (días 0 - 8)
NOTA: El protocolo que sigue describe la finalización de la pantalla CRISPR en una réplica biológica. Sin embargo, este protocolo ha sido validado previamente con tres réplicas biológicas y recomendamos el uso de al menos tres réplicas biológicas para otras que completen este protocolo30.
- Día 0: Aislar y estimular las células T de la sangre sana de un donante como se ha descrito anteriormente10.
- Aislar células mononucleares de sangre periférica (PBMC) de sangre de donantes sanos no identificados.
- Agregue 15 mL de un medio de gradiente de densidad a un tubo de separación de gradiente de densidad.
- Agregue sangre diluida 1:2 en la parte superior del tubo de separación en gradiente de densidad. Centrifugar a 1.200 × g durante 10 min a temperatura ambiente.
NOTA: La sangre debe diluirse con tampón de aislamiento (2% FBS (v/v) en solución salina tamponada con fosfato [PBS]). - Suministre la capa superior en un nuevo tubo cónico de 50 mL y duplique el volumen con tampón de aislamiento. Centrifugar la muestra a 300 × g durante 8 min a 4 °C. Aspirar el sobrenadante.
- Vuelva a suspender el pellet de celda con tampón de aislamiento y cuente las PBMC.
- Aísle las células T de las PBMC utilizando un kit de selección de negativos. Siga las instrucciones del fabricante para el kit.
NOTA: Suponga una recuperación del 10-20% de las células T de las PBMC. - Estimula las células T con perlas CD3+/CD28+ .
- Cuenta las células T.
- Alícuota suficientes perlas CD3+/CD28+ para estimular las células T en una proporción de 3:1 perla:célula. Por ejemplo, para estimular 110 × 106 células T, alícuota 330 × 106 perlas. Lave las perlas 3 veces separando primero las perlas de la suspensión con separación magnética y luego agregando 1,000 μL de TCM.
- Agregue las perlas a la suspensión de células T. Resuspender las células T a 1 × 106 células/mL en la MTC.
- Alícuota las células T en placas de 6 pocillos para una mejor eficiencia de transducción. Agregue 5 × 106 células T a cada pocillo. Incubar las células a 37 °C, 5% de CO2 durante 24 h.
- Aislar células mononucleares de sangre periférica (PBMC) de sangre de donantes sanos no identificados.
- Día 1: En condiciones BSL-2+, descongele suficiente lentivirus CAR para transducir células T a una multiplicidad de infección de 3.0. Rompa suavemente las rosetas de células T antes de agregar la cantidad adecuada de lentivirus agrupado para transducir 5 × 106 células T a un MOI de 3.0. Incubar las células a 37 °C, 5% de CO2.
NOTA: Agrupe el lentivirus de múltiples producciones en un tubo antes de transducir las células T. Esto asegura que todas las células T reciban el mismo tratamiento. Además, el lentivirus solo debe descongelarse una vez. - Día 2: En condiciones BSL-2+, descongele suficiente lentivirus CRISPR para transducir células T a un MOI de 0,3. Rompa suavemente las rosetas de células T y vuelva a suspender las células T antes de agregar la cantidad adecuada de lentivirus agrupado para transducir 5 × 106 células T a un MOI de 0,3. Incubar las células a 37 °C, 5% de CO2.
NOTA: Agrupe el lentivirus de múltiples producciones en un tubo antes de transducir las células T. Esto asegura que todas las células T reciban el mismo tratamiento. - Días 3 - 5: Vuelva a suspender las células CART para romper las rosetas y contar las células. Llevar la concentración celular final a 1 × 106 células/mL en TCM con 1 μg/mL de puromicina. Incubar las células a 37 °C, 5% de CO2.
- Día 6: Vuelva a suspender las células CART para romper las rosetas, retire las perlas CD3+/CD28+ mediante separación magnética y cuente las células CART. Añadir MTC con 1 μg/mL de puromicina para llevar la concentración celular final a 1 × 106 células/mL e incubar las células a 37 °C, 5% CO2. Determine la expresión de CAR con citometría de flujo como se describe en el paso 4.4.
7. Fase de selección de ARNg del cribado CRISPR (días 8 - 22)
- Día 8: Preservar las muestras para la secuenciación de próxima generación (NGS) y comenzar el cocultivo.
- Lavar las células CART 3 veces en MTC centrifugando las muestras a 300 × g durante 5 min a 4 °C, aspirando el sobrenadante y resuspendiendo las muestras en 20 mL de MTC.
- Cuente las celdas CART.
- Conserve las muestras para NGS completando lo siguiente:
- Alícuota suficientes células CART en un tubo cónico de 50 mL para mantener una representación de ARNg de 500 células por ARNg (por ejemplo, alícuota de aproximadamente 33 × 106 células CART si se utiliza una biblioteca CRISPR con 65.383 ARNg). Centrifugar la muestra a 300 × g durante 5 min a 4 °C.
- Vuelva a suspender el pellet de celda en 1 mL de PBS y transfiéralo a un tubo de microcentrífuga de 2 mL. Lave el tubo cónico de 50 mL con 1 mL de PBS y transfiera el lavado al tubo de microcentrífuga de 2 mL.
- Centrifugar las muestras a 500 × g durante 5 min a 4 °C. Retire el sobrenadante. Almacene los gránulos de celda a -20 °C.
- Comience el cocultivo añadiendo células CART a las células CD19+ JeKo-1 en una proporción de 1:1. Por ejemplo, agregue 80 × 106 celdas CART a 80 × 106 celdas JeKo-1. Llevar el volumen total del cocultivo a 160 mL con TCM. Almacene las muestras en una incubadora a 37 °C, 5% de CO2.
NOTA: Recomendamos comenzar con al menos 80 × 106 celdas CART para mantener la representación de la biblioteca y tener suficientes celdas para preservar las muestras en el día 15 y el día 22.
- Día 10: Para reestimular las células CART, recoja los cocultivos en tubos cónicos de 50 ml y centrifugue las muestras a 300 × g durante 5 min a 4 °C. Vuelva a suspender cada muestra en 80 mL de MTC y agregue 80 × 106 células JeKo-1 en 80 mL de MTC. Almacene las muestras en una incubadora a 37 °C, 5% de CO2.
- Días 12 y 14: Reestimule las células CART como en el paso 7.2.
- Día 15: Preservar las muestras para NGS y reestimular las células CART.
- Aísle las células CART del cocultivo contando primero las células y luego combinando un kit de selección positiva para las células T CD4+ con un kit de selección positiva para las células T CD8+ para aislar las células T de los cocultivos CART-JeKo-1. Cuenta las células T.
- Conserve las muestras para NGS alícuotas de 33 × 106 células CART en un tubo cónico de 50 ml y centrifugando la muestra a 300 × g durante 5 min a 4 °C. Vuelva a suspender la muestra en 1 mL de PBS y transfiérala a un tubo de microcentrífuga de 2 mL. Lave el tubo cónico de 50 mL con 1 mL de PBS y transfiera el lavado al tubo de microcentrífuga de 2 mL. Centrifugar las muestras a 500 × g durante 5 min a 4 °C; Retire el sobrenadante; y almacene los gránulos de celda a -20 °C.
- Establezca cocultivos para continuar con el modelo in vitro de agotamiento mediante la adición de 80 × 106 células CART a 80 × 106 células JeKo-1. A continuación, llevar el volumen total del cocultivo a 160 mL con TCM. Almacene las muestras en una incubadora a 37 °C, 5% de CO2.
- Días 17, 19 y 21: Reestimule las células CART como en el paso 7.2.
- Día 22: Preservar las muestras para NGS.
- Aísle las células CART del cocultivo contando primero las células. A continuación, combine un kit de selección positiva para linfocitos T CD4+ con un kit de selección positiva para linfocitos T CD8+ para aislar linfocitos T de los cocultivos CART-JeKo-1. Cuenta las células T.
- Para preservar las muestras para NGS, repita el paso 7.4.2.
8. Preparación de ADN genómico para la secuenciación de nueva generación
NOTA: Para mantener una cobertura adecuada, el ADN genómico (ADNg) debe aislarse de al menos 33 × 106 células CART. Además, la totalidad del ADNg de 33 × 106 células CART debe prepararse para la secuenciación.
- Aísle el ADNg de gránulos de células congeladas mediante el uso de un kit de aislamiento de ADNg.
- Purificar el ADNg a través de la precipitación de etanol.
- Preenfriar 100% EtOH a -20 °C.
- Alícuota eluyó el ADN por igual en varios tubos de microcentrífuga (200-250 μL). Añádase a cada alícuota lo siguiente: dos volúmenes de EtOH al 100% (500 μL para 250 μL de alícuota o 400 μL para 200 μL); 0,1 volumen de acetato de sodio 3 M (25 μL para 250 μL de alícuota o 20 μL para 200 μL); y 1 μL de 20 mg/mL de glucógeno. Incubar las muestras durante la noche a -20 °C.
- El segundo día, centrifugar las muestras a 13.000 × g durante 20 minutos para granular el ADN precipitado. Retire el sobrenadante. Lavar los pellets con 1 mL de EtOH al 70% centrifugando a 7.500 × g durante 10 min.
- Elimine por completo el EtOH residual. Deje que los gránulos se sequen durante 30-45 min.
- Vuelva a suspender los pellets en 50 μL de agua estéril.
- Prepare el ADNg para NGS.
NOTA: Los cebadores NGS para esta biblioteca CRISPR que amplifican los ARNg con secuencias adaptadoras de Illumina se han diseñado en una publicación anterior27. El uso de un cebador inverso diferente para cada muestra mostrará un código de barras de cada muestra y permitirá la agrupación de muestras durante la secuenciación.- Prepare las muestras para PCR con la siguiente mezcla de reacción (volumen total = 50 μL): mezcla maestra de PCR de alta fidelidad, 2x: 25 μL; ADN molde agrupado, 0,5 μg: 1 μL; cebador delantero de la biblioteca NGS, 10 μM: 1,25 μL; cebador inverso de la biblioteca NGS, 10 μM: 1,25 μL; Agua libre de DNasa: 21,5 μL.
- Realice PCR con las siguientes condiciones de ciclo: ciclo 1: desnaturalización a 98 °C durante 3 min; ciclos 2-23: Desnaturalizar a 98 °C durante 10 s, recocido a 63 °C durante 10 s, extender a 72 °C durante 25 s; ciclo 24: extender a 72 °C durante 2 min.
- Agrupe las reacciones de PCR para cada muestra y purifique el producto de PCR con un kit de purificación de PCR de acuerdo con las instrucciones del fabricante.
- Coloque 3 μg del producto de PCR para cada muestra en un gel de agarosa al 2% (p/v).
- Retire el producto de PCR (~ 260-270 bp) del gel extrayendo el ADN del gel mediante el uso de un kit de extracción de gel de acuerdo con las instrucciones del fabricante. Almacene las muestras extraídas a -20 °C.
- Realice la secuenciación de próxima generación (NGS) con las muestras. Apunta a más de 500 lecturas por ARNg en la biblioteca.
9. Análisis de los resultados de la secuenciación
- Para analizar NGS, utilice la plataforma MAGeCK-VISPR28. Utilice la configuración estándar para el algoritmo de estimación de máxima verosimilitud (MAGeCK-MLE). Para preparar los archivos de salida para el análisis:
- Ejecute FASTQC para evaluar la calidad del archivo de secuencia31.
- Combine los archivos de secuenciación de extremos emparejados para cada muestra con bbmerge32.
- Analice las muestras en la plataforma MAGeCK-VISPR con el algoritmo MAGeCK-MLE. Para el análisis MAGeCK-MLE, normalice todos los recuentos de ARNg a la lista de 1.000 ARNg no dirigidos.
- Evalúe la calidad de los archivos de secuenciación mediante la evaluación del contenido de GC, la calidad base, las lecturas de secuenciación y las lecturas asignadas.
- Evaluar el éxito de la fase de presión selectiva del cribado CRISPR mediante la evaluación de los cambios en el índice de Gini, el agrupamiento del análisis de componentes principales (PCA) y las vías asociadas con la lista de genes seleccionados negativamente32.
- Interrogue a los principales éxitos mediante la realización de búsquedas bibliográficas y el análisis de rutas.
Resultados
Para interrogar los genes y las vías que se pueden editar para mejorar la actividad de las células CART de manera imparcial, diseñamos una pantalla CRISPR knockout in vitro de todo el genoma (Figura 1). Esta criba tiene dos fases: una fase de producción de celdas CART y una fase de presión selectiva. En la fase de producción de células CART, al menos 110 × 106 células T se aíslan primero de las PBMC de donantes sanos y se activa...
Discusión
La edición genética se ha convertido en una herramienta poderosa tanto para comprender los mecanismos de resistencia a las terapias como para diseñar nuevas terapias con células CART para mejorar la longevidad y la actividad de las células CART 16,17,26. Si bien algunas estrategias de edición de genes han mostrado mejoras en la actividad de las células CART tanto en modelos preclínicos c...
Divulgaciones
SSK es inventor de patentes en el campo de la inmunoterapia con CAR que están licenciadas a Novartis (a través de un acuerdo entre Mayo Clinic, la Universidad de Pensilvania y Novartis), Humanigen (a través de Mayo Clinic), Mettaforge (a través de Mayo Clinic) y MustangBio (a través de Mayo Clinic) y Chymal therapeutics (a través de Mayo Clinic). CS, CMR y SSK son inventores de patentes licenciadas a Immix Biopharma. SSK recibe fondos de investigación de Kite, Gilead, Juno, BMS, Novartis, Humanigen, MorphoSys, Tolero, Sunesis/Viracta, LifEngine Animal Health Laboratories Inc. y Lentigen. SSK ha participado en reuniones de asesoramiento con Kite/Gilead, Calibr, Luminary Therapeutics, Humanigen, Juno/BMS, Capstan Bio y Novartis. SSK ha formado parte de la junta de seguridad y supervisión de datos con Humanigen y Carisma. SSK ha despedido a una consultora para Torque, Calibr, Novartis, Capstan Bio, BMS, Carisma y Humanigen. CMS y SSK son inventores de la propiedad intelectual que resultó de este protocolo.
Agradecimientos
Este estudio fue financiado en parte por el Centro de Medicina Personalizada de Mayo Clinic (SSK), el Centro Oncológico Integral de Mayo Clinic (SSK), el Centro de Bioterapéutica Regenerativa de Mayo Clinic (SSK), los Institutos Nacionales de Salud K12CA090628 (SSK) y R37CA266344-01 (SSK), el Departamento de Defensa CA201127 de subvenciones (SSK), la Fundación Predolin (SSK) y la Asociación de Minnesota para la Biotecnología y la Genómica Médica (SSK). Los CMS cuentan con el apoyo de la Escuela de Graduados en Ciencias Biomédicas de Mayo Clinic. El esquema de pantalla CRISPR (Figura 1) se creó con BioRender.com (Siegler, L. (2022) https://BioRender.com/k71r054).
Materiales
| Name | Company | Catalog Number | Comments |
| 293T cells | ATCC | CRL-3216 | Cells used for lentivirus production |
| Biotin ProteinL Antibody | GenScript | M00097 | anti-kappa chain antibody for CAR detection |
| Bovine Serum Albumin | Millipore Sigma | A7906 | |
| Carbenicillin disodium salt | Millipore Sigma | C1389-1G | Carbenicillin antibiotic |
| CD4 Isolation Beads | Miltenyi Biotec | 130-045-101 | |
| CD8 Isolation Beads | Miltenyi Biotec | 130-045-201 | |
| CTS (Cell Therapy Systems) Dynabeads CD3/CD28 | Gibco | 40203D | |
| Cytoflex | Beckman Coulter | NC2279958 | |
| DNase-Free Water | Invitrogen | AM9937 | |
| Dulbecco's modified eagle's medium (DMEM) | Corning | 10-017-CV | |
| Dulbecco's Phosphate-Buffered Saline | Gibco | 14190-144 | |
| EasySep Human T Cell Isolation Kit | STEMCELL Technologies | 17951RF | Negative isolation kit |
| Endura Electrocompetent Cells | Biosearch Technologies | 60242-1 | Electrocompetent cells with recovery medium |
| Ethanol | Millipore Sigma | E7023 | |
| Fetal bovine serum (FBS) | Corning | 35-010-CV | |
| GeCKO v2 CRISPR Knockout Pooled Library A | AddGene | 1000000048 | CRISPR library plasmid |
| Gene Pulser II | Bio-Rad | 165-2105 | Electroporator |
| Glycogen | Millipore Sigma | 10901393001 | |
| JeKo-1 | ATCC | CRL-3006 | CD19+ target cells |
| Lipofectamine 3000 Transfection Reagent | ThermoFisher Scientific | L3000075 | Transfection reagent kit with a transfection reagent (Lipofectamine 3000 Reagent) and a neutral co-lipid reagent (p3000) |
| LIVE/DEAD Aqua | Invitrogen | L34966 | |
| Lymphoprep | STEMCELL Technologies | 7851 | Density gradient medium |
| Machery-Nagel NucleoBond Xtra Maxi Kits | ThermoFisher Scientific | 12748412 | Maxi-prep kit |
| NEBNext High-Fidelity 2X PCR MasterMix | New England BioLabs | M0541S | High fidelity PCR mastermix |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985-070 | Reduced serum medium |
| pCMVR8.74 | AddGene | 22036 | Lentiviral packaging plasmid |
| Pennicillin-streptomycin-glutamine (100X) | Life Technologies | 10378-016 | |
| pMD2.G | AddGene | 12259 | VSV-G envelope expressing plasmid |
| Pooled Human AB Serum | Innovative Research | ISERABHI | |
| Puromycin | Millipore Sigma | P8833 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | Gek extraction kit |
| Qucik-DNA Midiprep Plus Kit | Zymo Research | D4075 | Kit used to isolate gDNA |
| RoboSep-S | STEMCELL Technologies | 21000 | Automated cell separator |
| Roswell Park Memorial Institute 1640 Medium (RPMI) | Gibco | 21870092 | |
| SepMate-50 | STEMCELL Technologies | 85450 | Density gradient separation tube |
| Sodium Acetate | Invitrogen | AM9740 | |
| Sodium Azide | Fisher Scientific | 71448-16 | |
| Streptavidin Antibody (PE) | BioLegend | 405203 | Secondary antibody used for CAR detection |
| T100 Thermal Cycler | Bio-Rad | 1861096 | |
| Ultracentrifuge (Optima XPN-80) | BeckmanCoulter | A99839 | |
| Vacuum Filter Systems, 0.22 µm | ThermoFisher Scientific | 567-0020 | |
| Vacuum Filter Systems, 0.45 µm | ThermoFisher Scientific | 165-0045 | |
| X-VIVO 15 Serum-Free Hematopoietic Cell Medium | Lonza | 04-418Q | Hematopoietic cell medium |
Referencias
- Neelapu, S. S., et al. 5-year follow-up supports curative potential of axicabtagene ciloleucel in refractory large B-cell lymphoma (zuma-1). Blood. 141 (19), 2307-2315 (2023).
- Neelapu, S. S., et al. Axicabtagene ciloleucel CAR T-cell therapy in refractory large B-cell lymphoma. N Engl J Med. 377 (26), 2531-2544 (2017).
- Locke, F. L., et al. Long-term safety and activity of axicabtagene ciloleucel in refractory large B-cell lymphoma (zuma-1): A single-arm, multicentre, phase 1-2 trial. Lancet Oncol. 20 (1), 31-42 (2019).
- Maude, S. L., et al. Tisagenlecleucel in children and young adults with B-cell lymphoblastic leukemia. N Engl J Med. 378 (5), 439-448 (2018).
- Schuster, S. J., et al. Tisagenlecleucel in adult relapsed or refractory diffuse large B-cell lymphoma. N Engl J Med. 380 (1), 45-56 (2018).
- Bai, Z., et al. Single-cell antigen-specific landscape of CAR T infusion product identifies determinants of CD19-positive relapse in patients with all. Sci Adv. 8 (23), eabj2820 (2022).
- Fraietta, J. A., et al. Determinants of response and resistance to CD19 chimeric antigen receptor (CAR) T cell therapy of chronic lymphocytic leukemia. Nat Med. 24, 563-571 (2018).
- Gennert, D. G., et al. Dynamic chromatin regulatory landscape of human CAR T cell exhaustion. Proc Natl Acad Sci USA. 118 (30), e2104758118 (2021).
- Deng, Q., et al. Characteristics of anti-CD19 CAR T cell infusion products associated with efficacy and toxicity in patients with large B cell lymphomas. Nat Med. 26 (12), 1878-1887 (2020).
- Sterner, R. M., Cox, M. J., Sakemura, R., Kenderian, S. S. Using CRISPR/CAS9 to knock out GM-CSF in CAR-T cells. J Vis Exp. (149), e59629 (2019).
- Sterner, R. M., et al. GM-CSF inhibition reduces cytokine release syndrome and neuroinflammation but enhances CAR-T cell function in xenografts. Blood. 133 (7), 697-709 (2019).
- Cox, M. J., et al. GM-CSF disruption in CART cells modulates T cell activation and enhances CART cell anti-tumor activity. Leukemia. 36 (6), 1635-1645 (2022).
- Ottaviano, G., et al. Phase 1 clinical trial of CRISPR-engineered CAR19 universal T cells for treatment of children with refractory B cell leukemia. Sci Transl Med. 14 (668), eabq3010 (2022).
- Joung, J., et al. Genome-scale CRISPR-CAS9 knockout and transcriptional activation screening. Nat Protoc. 12, 828-863 (2017).
- Mccutcheon, S. R., et al. Transcriptional and epigenetic regulators of human CD8+ T cell function identified through orthogonal CRISPR screens. Nat Genet. 55, 2211-2223 (2023).
- Zhu, W., Kelly, C., Dagur, P., Dunbar, C. E., Cordes, S. CRISPR activation screen to optimize chimeric antigen receptor (CAR) T cell immunophenotype. Blood. 142 (Supplement 1), 4820-4820 (2023).
- Wang, D., et al. CRISPR screening of CAR T cells and cancer stem cells reveals critical dependencies for cell-based therapies. Cancer Discov. 11 (5), 1192-1211 (2021).
- Shifrut, E., et al. Genome-wide CRISPR screens in primary human T cells reveal key regulators of immune function. Cell. 175 (7), 1958-1971.e15 (2018).
- Belk, J. A., et al. Genome-wide crispr screens of T cell exhaustion identify chromatin remodeling factors that limit T cell persistence. Cancer Cell. 40 (7), 768-786.e7 (2022).
- Jiang, P., et al. Single-cell ATAC-seq maps the comprehensive and dynamic chromatin accessibility landscape of CAR-T cell dysfunction. Leukemia. 36 (11), 2656-2668 (2022).
- Rossi, J., et al. Preinfusion polyfunctional anti-CD19 chimeric antigen receptor T cells are associated with clinical outcomes in NHL. Blood. 132 (8), 804-814 (2018).
- Beider, K., et al. Molecular and functional signatures associated with CAR T cell exhaustion and impaired clinical response in patients with B cell malignancies. Cells. 11 (7), 1140 (2022).
- Wherry, E. J. T. cell exhaustion. Nat Immunol. 12, 492-499 (2011).
- Prinzing, B., et al. Deleting DNMT3A in CAR T cells prevents exhaustion and enhances antitumor activity. Sci Transl Med. 13, eabh0272 (2021).
- Lynn, R. C., et al. C-jun overexpression in CAR T cells induces exhaustion resistance. Nature. 576, 293-300 (2019).
- Tang, N., et al. TGF-beta inhibition via CRISPR promotes the long-term efficacy of CAR T cells against solid tumors. JCI Insight. 5 (4), e133977 (2020).
- Joung, J., et al. Genome-scale CRISPR-CAS9 knockout and transcriptional activation screening. Nat Protoc. 12 (4), 828-863 (2017).
- Li, W., et al. Quality control, modeling, and visualization of CRISPR screens with MAGeCK-VISPR. Genome Biol. 16, 281 (2015).
- Sakemura, R., et al. Targeting cancer-associated fibroblasts in the bone marrow prevents resistance to CART-cell therapy in multiple myeloma. Blood. 139 (26), 3708-3721 (2022).
- Stewart, C. M., et al. IL-4 drives exhaustion of CD8+ CART cells. Nat Commun. 15 (1), 7921 (2024).
- . FastQC: A quality control tool for high throughput sequence data Available from: https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010)
- . Bbmerge Available from: https://github.com/BioInfoTools/BBMap/blob/master/sh/bbmerge.sh (2019)
- Pattanayak, V., et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed CAS9 nuclease specificity. Nat Biotechnol. 31 (9), 839-843 (2013).
- Doench, J. G., et al. Rational design of highly active sgRNAs for CRISPR-CAS9-mediated gene inactivation. Nat Biotechnol. 32 (12), 1262-1267 (2014).
- Dong, M. B., et al. Systematic immunotherapy target discovery using genome-scale in vivo CRISPR screens in CD8 T cells. Cell. 178 (5), 1189-1204.e23 (2019).
- Zhou, P., et al. Single-cell CRISPR screens in vivo map T cell fate regulomes in cancer. Nature. 624 (7990), 154-163 (2023).
- Sutra Del Galy, A., et al. In vivo genome-wide CRISPR screens identify SOCS1 as intrinsic checkpoint of CD4+ TH1 cell response. Sci Immunol. 6 (66), eabe8219 (2021).
- Ramos, A., et al. Leukemia-intrinsic determinants of CAR-T response revealed by iterative in vivo genome-wide CRISPR screening. Nat Commun. 14 (1), 8048 (2023).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados