Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Kimerik Antijen Reseptörü T Hücrelerinde İn Vitro Genom Çapında CRISPR Nakavt Taraması Gerçekleştirme
Bu Makalede
Özet
Makale, sağlıklı donör kimerik antijen reseptörü T hücrelerinde genom çapında bir CRISPR nakavt ekranını tamamlamak için tükenme için bir in vitro modelin uygulanması için bir protokolü açıklamaktadır.
Özet
Kimerik antijen reseptörü T (CART) hücre tedavisi, kanser tedavisinde devrim yaratan yenilikçi bir hedefe yönelik immünoterapi şeklidir. Bununla birlikte, kalıcı yanıt sınırlı kalmaktadır. Son çalışmalar, preinfüzyon CART hücre ürünlerinin epigenetik manzarasının tedaviye yanıtı etkileyebileceğini göstermiştir ve gen düzenlemesi bir çözüm olarak önerilmiştir. Bununla birlikte, optimal gen düzenleme stratejisini belirlemek için daha fazla çalışma yapılması gerekmektedir. Genom çapında CRISPR ekranları, hem direnç mekanizmalarını araştırmak hem de gen düzenleme stratejilerini optimize etmek için popüler araçlar haline geldi. Yine de birincil hücrelere uygulanmaları birçok zorluk ortaya çıkarmaktadır. Burada, sağlıklı donörlerden alınan CART hücrelerinde genom çapında bir CRISPR nakavt ekranını tamamlamak için bir yöntem açıklıyoruz. Bir kavram kanıtı modeli olarak, CD19 antijenini hedefleyen CART hücrelerinde tükenme gelişimini araştırmak için bu yöntemi tasarladık. Bununla birlikte, bu yaklaşımın farklı CAR yapılarında ve tümör modellerinde tedaviye karşı çeşitli direnç mekanizmalarını ele almak için kullanılabileceğine inanıyoruz.
Giriş
Kimerik antijen reseptörü T (CART) hücre tedavisi, B hücreli malignitelerin tedavisinde etkileyici bir başarı göstermiştir; Bununla birlikte, dayanıklı yanıt %30-40 ile sınırlıdır1,2,3,4,5. Araştırmacılar, CAR tasarımının optimizasyonu, gen düzenleme ve kombinasyon tedavileri dahil olmak üzere CART hücre tedavisine direnç mekanizmalarını ele almak için çeşitli yaklaşımlar geliştirmiş ve test etmiş olsalar da, direncin gelişimi büyük ölçüde bilinmemektedir. Son zamanlarda, preinfüzyon CART hücre ürünlerinin başlangıç gen ekspresyon profilinin, CART tedavisinin 6,7,8,9 hem toksisitesinin hem de etkinliğinin önemli bir belirleyicisi olduğuna dair artan kanıtlar olmuştur. Bu nedenle, CART hücre üretimi sırasında gen düzenleme, CART hücre tedavisinin başarısını artırmak için popüler bir yaklaşım haline gelmiştir.
Örneğin, laboratuvarımız daha önce gen granülosit-makrofaj koloni uyarıcı faktörü (GM-CSF) devre dışı bırakmak için kümelenmiş düzenli aralıklı kısa palindromik tekrarlar (CRISPR) Cas9 teknolojisinin kullanılmasının CART hücre aktivitesini iyileştirdiğini ve CART ile ilişkili toksisite belirtilerini azalttığını göstermiştir 10,11,12. Ek olarak, CRISPR ile tasarlanmış hücre tedavisi klinik çalışmalarda test edilmiş ve etkinlik ve güvenlik göstermiştir13. Birlikte, bu, CRISPR teknolojisi ile gen düzenlemenin yalnızca CART hücre biyolojisi anlayışımızı geliştirmekle kalmayıp, aynı zamanda çevrilebilir CART hücre ürünleri de üretebileceğini göstermektedir.
Genom çapında CRISPR nakavt ekranları, terapötiklere direnç mekanizmalarını anlamak için kanser biyolojisi araştırmalarında giderek daha popüler araçlar haline geldi. Bu teknikte, hücre başına bir gRNA'nın girişini teşvik etmek için on binlerce kılavuz RNA (gRNA) hücre havuzlarına verilir14. Daha sonra hücreler, temel genleri hedefleyen gRNA'larla dönüştürülen hücrelerin öldüğü ve inhibitör genleri hedefleyen gRNA'larla dönüştürülen hücrelerin hayatta kaldığı ve çoğaldığı basınca neden olan koşullara tabi tutulur. Yeni nesil dizileme kullanarak, gRNA temsilinin CRISPR ekranı14 boyunca nasıl değiştiğini belirleyebiliriz.
Bununla birlikte, genom çapında gen bozulmaları için gereken ölçek ve seçim süresinin, CART hücreleri gibi birincil hücrelerde gerçekleştirilmesi zor olabilir. Bu nedenle, gruplar terapötik dirençmekanizmalarını daha iyi anlamak için hedefli CRISPR ekranlarını kullanmışlardır 15,16. Hedeflenen ekranların birincil hücrelerde tamamlanması genellikle daha kolaydır, çünkü yeterli kitaplık gösterimi elde etmek için daha az hücre gerektiren daha küçük kitaplıklara sahiptirler. Bu çalışmalar, CART hücre tedavisine direnç mekanizmaları hakkındaki anlayışımızı geliştirmiş olsa da, hedefe yönelik ekranlar, gen hedeflerinin manuel olarak seçilmesi nedeniyle bir önyargı ortaya koymaktadır. Bu makale, sağlıklı donörlerden alınan CART hücrelerinde in vitro genom çapında bir CRISPR nakavt ekranını tamamlamak için bir yöntemi tanımlamayı amaçlamaktadır. Bu nedenle, bu yüksek verimli yaklaşım, terapötik yanıtları iyileştirmek için düzenlenebilen temel yolların ve genlerin verimli, tarafsız bir şekilde tanımlanmasına olanak tanır 17,18,19.
Özellikle, bu makalede açıklanan protokol, genom çapında CRISPR nakavt ekranını tükenme için bir in vitro modelle tamamlayarak alanın CART hücre tükenmesi konusundaki anlayışını artırmak için tasarlanmıştır. Tükenme, CART hücre tedavisine yanıt vermemeyle ilişkilendirilen işlevsiz bir CART hücre kaderidir 20,21,22. Bu hücresel kaderin epigenetik olarak düzenlendiği bilinmektedir ve CART hücre proliferasyonunda bir azalma, efektör sitokin üretiminde bir azalma ve inhibitör reseptörlerin ekspresyonunda bir artış ile karakterize edilir23. Önceki literatürde, gen düzenleme, anahtar genleri yukarı veya aşağı regüle ederek tükenme gelişimini önleyebilmiştir 24,25,26. Hem CART hücreleri tükendikçe proliferatif kabiliyetteki azalma hem de gen düzenlemenin oluşumunu önleyebileceğine dair kanıtlar göz önüne alındığında, in vitro genom çapında CRISPR nakavt ekranımızı bu hücresel kader üzerinde modelledik. Bununla birlikte, bu protokol gelecekte CART hücre tedavisine karşı diğer direnç mekanizmalarını araştırmak için değiştirilebilir.
Protokol
Daha da önemlisi, aşağıda özetlenen protokol, Mayo Clinic'in Kurumsal İnceleme Kurulu (IRB 18-005745) ve Kurumsal Biyogüvenlik Komitesi'nin (IBC HIP00000252.43) yönergelerini takip eder ve bunlardan onay almıştır. Lentiviral üretim de dahil olmak üzere tüm hücre kültürü çalışmaları, uygun kişisel koruyucu ekipmana sahip bir hücre kültürü başlığında gerçekleştirilmelidir. Özellikle, lentiviral çalışma, bertaraf edilmeden önce öğeleri dezenfekte etmek için %2 çamaşır suyu kullanımı da dahil olmak üzere biyogüvenlik seviye 2 (BSL-10) önlemleri altında yapılmalıdır.
1. CRISPR Kütüphanesinin Güçlendirilmesi
- CRISPR kitaplığını üreticinin tavsiyelerine göre yükseltin. Kısaca, aşağıdakileri tamamlayarak CRISPR kütüphanesini dört elektroyetkin hücre alikotuna elektrostatik hale getirin:
- 1.0 mm'lik bir küvette 25 μL elektroyetkin hücreye 2 μL 50 ng/μL CRISPR kütüphane plazmidi ekleyin.
- Aşağıdaki ayarları kullanarak numuneleri elektrostatik hale getirin: 10 μF, 600 Ohm, 1.800 volt ve 3,5 ms ile 4,5 ms arasında bir zaman sabiti.
- 975 μL geri kazanım ortamında geri kazanın ve ek 1 mL geri kazanım ortamı içeren bir tüpe aktarın.
- Tüm alikotları 250 °C'de 1 saat boyunca 37 RPM'de döndürün.
- Dönüşümleri plakalayın:
- Elektroporasyonlu hücrelerin tüm alikotlarını bir araya getirin ve iyice karıştırın.
- 400 μL dönüşüm karışımını 100 μg / mL karbenisilin ile önceden ısıtılmış 10 cm Luria suyu (LB) plakalarına yayarak tüm elektroporasyonlu hücreleri plakalayın. Plakayı 14 saat boyunca 32 ° C'de bir inkübatöre yerleştirin.
- Kolonileri hasat edin:
- Her plakaya 1 mL LB ortamı + 100 μg/mL karbenisilin pipetleyin. Kolonileri her plakadan kazıyın ve 50 mL'lik konik bir tüpe toplayın.
- Yıkamak ve tam iyileşme sağlamak için her plakaya 1 mL LB ortamı + 100 μg/mL karbenisilin daha ekleyin.
- Bir maksi hazırlık kitinin talimatlarını izleyerek bir maksi hazırlık yapın.
- Elde edilen plazmit DNA'sını bir araya getirin.
2. CRISPR kütüphanesinde temel gRNA temsilini doğrulamak için yeni nesil dizileme
NOT: Bu CRISPR kütüphanesi için NGS primerleri, önceki bir yayındatasarlanmıştır 27. Her numune için farklı bir ters astar kullanmak, her numuneyi barkodlayacak ve sıralama sırasında numunelerin havuzlanmasına izin verecektir.
- Numuneleri aşağıdaki reaksiyon karışımıyla PCR için hazırlayın (toplam hacim = 50 μL): yüksek kaliteli PCR ana karışımı, 2x: 25 μL; havuzlanmış şablon DNA, 0.5 μg: 1 μL; NGS kütüphanesi ileri astar, 10 μM: 1.25 μL; NGS kütüphanesi ters astar, 10 μM: 1.25 μL; DNaz içermeyen su: 21,5 μL.
- Aşağıdaki döngü koşullarıyla PCR gerçekleştirin: döngü 1: 98 °C'de 3 dakika denatüre; döngü 2-23: 98 °C'de 10 saniye denatüre etme, 63 °C'de 10 saniye tavlama, 72 °C'de 25 saniye uzatma; döngü 24: 72 °C'de 2 dakika uzatın.
- Her numune için PCR reaksiyonlarını bir araya getirerek ve PCR ürününü üreticinin talimatlarına göre bir PCR saflaştırma kiti ile saflaştırarak PCR reaksiyonunu saflaştırın.
- %2 (a/h) agaroz jel üzerinde her numune için 3 μg PCR ürünü çalıştırın.
- Üreticinin talimatlarına göre bir jel ekstraksiyon kiti kullanarak DNA'yı jelden çıkararak PCR ürününü (~ 260-270 bp) jelden çıkarın. En az 500 ng DNA hedefleyin.
- Ekstrakte edilen numuneleri -20 °C'de saklayın.
- Örneklerle yeni nesil dizileme (NGS) gerçekleştirin ve gRNA27 başına 100 okuma hedefleyin.
- Dizileme sonuçlarını MAGeCK-VISPR boru hattı ile analiz edin ve kitaplıktaki gRNA'ların %99,5'inden fazlasının temsil edildiğinden emin olun28.
3. CRISPR kütüphanesi ve CAR ekspresyonu lentivirüslerinin üretimi
- Lentivirüsü daha önce tarif edildiği gibi hazırlayın10,29.
- 1. Gün: %10 fetal sığır serumu (FBS) (h/h), %1 penisilin-streptomisin-glutamin (PSG) (h/h) ve Dulbecco'nun modifiye kartal besiyerinden oluşan D10 ortamında büyüyen %80-90 birleşik 293T hücresinin T175 şişeleri ile başlayın. Başlamadan önce, transfeksiyon reaktiflerini ve DNA'yı oda sıcaklığına ısıtın ve DNA'yı ölçün.
- Her 293T şişe için iki adet 50 mL konik tüp A ve B'yi etiketleyin. Tüp A'ya ve tüp B'ye 4.5 mL indirgenmiş serum ortamı ekleyin.
- Steril 1.5 mL'lik bir mikrosantrifüj tüpünde, aşağıdaki DNA'yı karıştırın ve 1 dakika bekletin: 18 μg pCMVR8.74 (gag, pol, tat ve rev), 7 μg pMD2.G (VSV-G) ve 15 μg lentiviral ekspresyon vektörü (CRISPR kütüphanesi A veya CAR19-28ζ vektörleri).
- Tüp A'ya 129 μL transfeksiyon reaktifi ekleyin. Tüp A'yı parmak girdabı ile karıştırın.
- DNA karışımını B tüpüne ekleyin. Tüp B'yi parmak girdabı ile iyice karıştırın.
- Tüp B'ye 111 μL nötr ko-lipid reaktifi ekleyin. Tüp B'yi parmak girdabı ile iyice karıştırın.
- Tüp A'nın içeriğini tüp B'ye ekleyin ve parmak girdabı ile iyice karıştırın. Tüp B'yi oda sıcaklığında 30 dakika inkübe edin.
- 30 dakikalık inkübasyon süresinin sonuna doğru, mevcut besiyerini aspire ederek 293T hücrelerini hazırlayın. %10 FBS (v/v), %1 PSG (v/v) ve Roswell Park Memorial Institute (RPMI) 1640'tan oluşan 16 mL R10 ortamı ekleyin.
- 30 dakikalık inkübasyon süresinden sonra, tüp B'nin içeriğini (yaklaşık 9 mL) damla damla 293T hücreli şişeye ekleyin. Şişeyi yere yatırın ve 37 °C, %5 CO2'de inkübe etmeden önce hafifçe döndürün.
- 2. Günde (transfeksiyondan yaklaşık 24 saat sonra), lentivirüsü aşağıdaki gibi toplayın:
NOT: Tüm ekipman ve malzemeleri önceden soğutun ve viral süpernatanı soğuk tutmak için buz üzerinde aşağıdakileri gerçekleştirin.- Süpernatanı transfekte edilmiş 293T şişelerden önceden soğutulmuş 50 mL konik tüplere aktarın.
- 293T şişelere 30 mL önceden ısıtılmış R10 ortamı ekleyin.
- 50 mL'lik konik tüpleri kapalı rotorlarda 4 °C'de 10 dakika boyunca 900 × g'da santrifüjleyerek pelet hücresel artıkları.
- Santrifüjlemeden sonra, süpernatanı 0,45 μm'lik bir filtre ile filtreleyin. Süzüntüyü 112.700 × g'da bir ultrasantrifüjde 4 ° C'de 2 saat döndürün.
- 2 saatlik dönüşten sonra, süpernatanı yaklaşık 5 mL kalana kadar aspire edin. 10 kat yukarı ve aşağı hafifçe pipetleyerek viral partikülleri yeniden süspanse edin. Viral süspansiyonu gece boyunca 4 °C'de saklayın.
- 3. Günde, lentivirüsü aşağıdaki gibi toplayın ve saklayın:
- Süpernatanı transfekte edilmiş 293T şişelerden önceden soğutulmuş 50 mL konik tüplere aktarın.
- 293T şişelere 30 mL %10 çamaşır suyu ekleyin.
- 50 mL konik tüpleri kapalı rotorlarda 4 ° C'de 10 dakika boyunca 900 × g'da santrifüjleyerek süpernatanttaki hücresel kalıntıları peletleyin.
- Santrifüjlemeden sonra, süpernatanı 0,45 μm'lik bir filtre ile filtreleyin. Süzüntüyü 112.700 × g'da 4 ° C'de 2 saat boyunca bir ultrasantrifüjde döndürün.
- 2 saatlik santrifüjün ardından, süpernatanı yaklaşık 2 mL kalana kadar aspire edin ve 10 kat yukarı ve aşağı hafifçe pipetleyerek yeniden süspanse edin. Viral süpernatanı 300-500 μL'lik alikotlarda saklayın ve -80 ° C'de tutun.
4. CAR eksprese eden lentivirüs için viral titrenin hesaplanması
NOT: CAR lentivirus daha önce tarif edildiği gibititremiştir 10,12.
- 1. Günde, T hücrelerini aktive edin ve plakalayın.
- Negatif bir seçim kiti kullanarak sağlıklı donör PBMC'lerden 1 ×10 6 T hücresini izole edin.
- T hücrelerini sayın.
- T hücrelerini 3: 1 boncuk: hücre oranında uyarmak için yeterli miktarda CD3 + / CD28 + boncuklarını yıkayın. Örneğin, 1 × 106 T hücresini uyarmak için 3 × 106 boncuk yıkayın. Bunu yapmak için, aşağıdaki 3x'i tamamlayarak boncukları yıkayın:
- Boncukları süspansiyondan çıkarmak için manyetik ayırma kullanın.
- Süpernatanı çıkarın.
- % 10 insan serumu (v / v),% 1 PSG (h / v) ve hematopoietik hücre ortamından oluşan 1000 μL T hücresi ortamı (TCM) ekleyin.
NOT: TCM, kullanımdan önce 0.45 μm steril vakum filtresi ve ardından 0.22 μm steril vakum filtresi ile filtrelenerek sterilize edilmelidir. - T hücresi süspansiyonuna 3: 1 boncuk: hücre oranında boncuklar ekleyin.
- TCM'de T hücrelerini 1 × 106 hücre / mL'ye yeniden süspanse edin.
- 96 oyuklu bir plakanın 10 oyuğuna 100 μL aktif T hücresi ekleyin. Plakayı 37 °C, %5 CO2'de inkübe edin.
- 2. Günde, aşağıdaki gibi bir seyreltme plakası oluşturarak bir CAR lentivirus seyreltmesi ekleyin:
- 96 oyuklu bir plakanın yedi oyuğuna 100 μL TCM ekleyin.
- İlk kuyucuğa, ilgilenilen virüsün 50 μL'sini ekleyin; İyice karıştırın.
- Kuyu 1'den kuyu 2'ye 50 μL ekleyerek virüsü seri olarak daha da seyreltin; İyice karıştırın.
- Kuyu 2'den kuyu 3'e 50 μL ekleyin ve bu deseni kuyu 7'ye kadar devam ettirin.
- İyice 7 karıştırdıktan sonra 50 μL çıkarın. Tüm kuyucukların toplam hacminin 100 μL olduğundan emin olun.
- 1. Günden itibaren seyreltme plakasının her bir oyuğundan T hücresi plakasının ilk yedi oyuğuna 50μL ekleyin. İyice karıştırın.
- T hücresi plakasının son üç oyuğuna 50 μL TCM ekleyin; İyice karıştırın. Plakayı 37 °C, %5 CO2'de bir inkübatörde saklayın.
- 3. Günde, T hücre plakasının tüm kuyucuklarına 100 μL TCM ekleyerek hücreleri besleyin; İyice karıştırın.
- 4. Günde, aşağıdakileri tamamlayarak CAR ekspresyonu için bir vekil belirteç olan bir anti-kappa zincir antikoru ile CAR eksprese eden hücrelerin yüzdesini tespit etmek için akış sitometrisi yapın:
- T hücresi plakasını 1.000 × g'da 3 dakika boyunca döndürün ve boşaltın.
- Plakayı 3x anti-kappa zincir antikor tamponu ile 200 μL anti-kappa zincir antikor tamponu ekleyerek, plakayı 3 dakika boyunca 1.000 × g'da döndürerek ve boşaltarak anti-kappa zincir antikor tamponu (PBS'de (a/h)% 4 sığır serum albümininden oluşur) yıkayın.
- Her oyuğa bir biyotin anti-kappa zincir antikoru ekleyin. 4 ° C'de bir külbütör üzerinde 45 dakika inkübe edin.
- Plakayı 3x'i adım 4.4.2'deki gibi anti-kappa zincir antikor tamponu ile yıkayın.
- Hücreleri akış sitometrisi için florokrom konjuge streptavidin antikoru ve canlı/ölü antikor ile boyayın. Numuneleri karanlıkta oda sıcaklığında 15 dakika inkübe edin.
- Numuneleri bir kez akış tamponunda (PBS'de %2 FBS (h/v) ve PBS'de %1 sodyum azidden (h/v) oluşur) 200 μL akış tamponu ekleyerek, plakayı 3 dakika boyunca 1.000 × g'da aşağı döndürerek ve boşaltarak yıkayın.
- 100 μL akış tamponunda yeniden süspanse edin ve sitometre üzerinde çalıştırın.
- Dönüştürülmemiş üç kuyu ile negatif geçit gerçekleştirin.
5. CRISPR kütüphanesi lentivirüsü için viral titrenin hesaplanması
NOT: Bu protokol, CRISPR kütüphanesi lentivirüsünü titre etmek için minimum sayıda T hücresi tanımlar, ancak daha fazla T hücresi ve dolayısıyla daha yüksek hacimlerde virüs barındıracak şekilde ölçeklendirilebilir.
- 0. Günde, 6 × 106 T hücresine 18 × 10 6 CD3 + / CD28 + boncuk (3: 1 boncuk: hücre oranı) ekleyerek ve son hacmi TCM ile 6 mL'ye getirerek T hücrelerini aktive edin. T hücresi süspansiyonunun 0.5 mL'sini 48 oyuklu bir plakanın 12 oyuğuna alikot edin. 37 °C,% 5 CO2'de bir inkübatörde saklayın.
- Yirmi dört saat sonra, 1. günde, her kuyucuğa 3 enfeksiyon çokluğunda (MOI) CAR virüsü ekleyin. Birkaç kez yukarı ve aşağı pipetleyerek iyice karıştırın. 37 °C,% 5 CO2'de bir inkübatörde saklayın.
- Kırk sekiz saat sonra, 2. Günde, her bir oyuğa aşağıdaki CRISPR virüsü hacimlerinden birini ekleyerek aktive edilmiş CART hücrelerine bir dizi CRISPR kütüphane virüsü hacmi ekleyin (0 μL, 0 μL, 6.25 μL, 6.25 μL, 12.5 μL, 12.5 μL, 25 μL, 25 μL, 50 μL, 50 μL, 100 μL veya 100 μL). Birkaç kez yukarı ve aşağı pipetleyerek iyice karıştırın. 37 °C,% 5 CO2'de bir inkübatörde saklayın.
- 3. Günde, aşağıdaki CRISPR virüs hacimlerinin her birini (0 μL, 6.25 μL, 12.5 μL, 25 μL, 50 μL ve 100 μL) alan bir kuyucuk da dahil olmak üzere, oyukların yarısına mL ortam başına 1 μg puromisin ekleyin. Diğer kuyucuklar puromisin ile muamele edilmeyecektir.
- 4-8. Günler arasında, T hücrelerini günlük olarak sayın ve nihai hücre yoğunluğunu, puromisin tedavi kuyucukları için TCM'de 1 μg / mL puromisin ve tedavi edilmemiş kuyucuklar için TCM ile 1 ×10 6 hücre / mL'ye ayarlayın.
- 8. Günde, kuyucuk başına canlı hücre sayısını sayın ve aşağıdaki denklemi kullanarak viral süpernatantın transdüksiyon birimlerini / mL'sini hesaplayın:
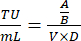
Nerede A: Viral olarak transdüksiyona uğrayan kuyuda canlı hücre sayısı; B: Antibiyotik seçimi olmayan kontrol kuyusunda canlı hücre sayımı; V: Her kuyucukta enfeksiyon için kullanılan virüs hacmi; D: Seyreltme katlama. - Yaklaşık% 20 iletim verimliliği gösteren bir kuyudan hesaplanan TU / mL kuyusunu kullanarak, aşağıdaki formülü kullanarak 0.3'lük bir MOI'de 1 × 106 hücreyi dönüştürmek için gereken virüs hacmini hesaplayın:
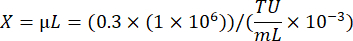
6. CRISPR ekranının CART hücre üretim aşaması (0 - 8 Gün)
NOT: Aşağıdaki protokol, CRISPR ekranının tek bir biyolojik kopyada tamamlanmasını açıklar. Bununla birlikte, bu protokol daha önce üç biyolojik kopya ile doğrulanmıştır ve bu protokolü tamamlayan diğerleri için en az üç biyolojik kopyanın kullanılmasını öneririz30.
- 0. Gün: Daha önce tarif edildiği gibi sağlıklı donör kanından T hücrelerini izole edin ve uyarın10.
- Periferik kan mononükleer hücrelerini (PBMC'ler) tanımlanmamış sağlıklı donör kanından izole edin.
- Bir yoğunluk gradyan ayırma tüpüne 15 mL'lik bir yoğunluk gradyan ortamı ekleyin.
- Yoğunluk gradyan ayırma tüpünün üstüne 1: 2 seyreltilmiş kan ekleyin. Oda sıcaklığında 10 dakika boyunca 1.200 × g'da santrifüjleyin.
NOT: Kan, izolasyon tamponu ile seyreltilmelidir (fosfat tamponlu salin [PBS] içinde% 2 FBS (h/h)). - Üst tabakayı yeni bir 50 mL konik tüpe boşaltın ve izolasyon tamponu ile hacmi iki katına çıkarın. Numuneyi 300 × g'da 4 °C'de 8 dakika santrifüjleyin. Süpernatanı aspire edin.
- Hücre peletini izolasyon tamponu ile yeniden süspanse edin ve PBMC'leri sayın.
- Negatif bir seçim kiti kullanarak T hücrelerini PBMC'lerden izole edin. Kit için üreticinin talimatlarına uyun.
NOT: PBMC'lerden T hücrelerinin %10-20 oranında geri kazanıldığını varsayalım. - T hücrelerini CD3 + / CD28 + boncukları ile uyarın.
- T hücrelerini sayın.
- T hücrelerini 3: 1 boncuk: hücre oranında uyarmak için yeterli CD3 + / CD28 + boncuğu alın. Örneğin, 110 × 106 T hücresini uyarmak için, alikot 330 × 106 boncuk. Boncukları önce manyetik ayırma ile süspansiyondan ayırarak ve ardından 1.000 μL TCM ekleyerek boncukları 3x yıkayın.
- Boncukları T hücresi süspansiyonuna ekleyin. TCM'de T hücrelerini 1 × 106 hücre / mL'ye yeniden süspanse edin.
- En iyi transdüksiyon verimliliği için T hücrelerini 6 oyuklu plakalarda alikotlayın. Her kuyucuğa 5 × 106 T hücresi ekleyin. Hücreleri 37 ° C'de,% 5 CO2'de 24 saat inkübe edin.
- Periferik kan mononükleer hücrelerini (PBMC'ler) tanımlanmamış sağlıklı donör kanından izole edin.
- 1. Gün: BSL-2 + koşulları altında, 3.0'lık bir enfeksiyon çokluğunda T hücrelerini dönüştürmek için yeterli CAR lentivirüsünü çözün. 3.0'lık bir MOI'de 5 × 106 T hücresini dönüştürmek için uygun miktarda havuzlanmış lentivirüs eklemeden önce T hücresi rozetlerini nazikçe parçalayın. Hücreleri 37 ° C,% 5 CO2'de inkübe edin.
NOT: T hücrelerini dönüştürmeden önce birden fazla üretimden gelen lentivirüsü tek bir tüpte toplayın. Bu, tüm T hücrelerinin aynı tedaviyi almasını sağlar. Ek olarak, lentivirüs sadece bir kez çözülmelidir. - 2. Gün: BSL-2+ koşulları altında, 0.3'lük bir MOI'de T hücrelerini dönüştürmek için yeterli CRISPR lentivirüsünü çözün. 0.3'lük bir MOI'de 5 ila 10 6 T hücresini dönüştürmek için uygun miktarda havuzlanmış lentivirüs eklemeden önce T hücresi rozetlerini nazikçe parçalayın ×T hücrelerini yeniden süspanse edin. Hücreleri 37 ° C,% 5 CO2'de inkübe edin.
NOT: T hücrelerini dönüştürmeden önce birden fazla üretimden gelen lentivirüsü tek bir tüpte toplayın. Bu, tüm T hücrelerinin aynı tedaviyi almasını sağlar. - 3 - 5. Günler: Rozetleri parçalamak ve hücreleri saymak için CART hücrelerini yeniden askıya alın. 1 μg / mL puromisin ile TCM'de son hücre konsantrasyonunu 1 × 106 hücre / mL'ye getirin. Hücreleri 37 ° C,% 5 CO2'de inkübe edin.
- 6. Gün: Rozetleri parçalamak için CART hücrelerini yeniden askıya alın, manyetik ayırma yoluyla CD3+/CD28+ boncuklarını çıkarın ve CART hücrelerini sayın. Son hücre konsantrasyonunu 1 × 106 hücre / mL'ye getirmek için 1 μg / mL puromisin ile TCM ekleyin ve hücreleri 37 ° C,% 5 CO2'de inkübe edin. Adım 4.4'te açıklandığı gibi akış sitometrisi ile CAR ekspresyonunu belirleyin.
7. CRISPR ekranının gRNA seçim aşaması (8 - 22. günler)
- 8. Gün: Yeni nesil dizileme (NGS) için örnekleri koruyun ve ortak kültüre başlayın.
- Numuneleri 300 × g'da 4 ° C'de 5 dakika santrifüjleyerek, süpernatanı aspire ederek ve numuneleri 20 mL TCM'de yeniden süspanse ederek CART hücrelerini TCM'de 3x yıkayın.
- CART hücrelerini sayın.
- Aşağıdakileri tamamlayarak NGS için örnekleri koruyun:
- gRNA başına 500 hücrenin gRNA temsilini korumak için 50 mL'lik konik bir tüpte yeterli sayıda CART hücresi bulundurun (örneğin, 65.383 gRNA'lı bir CRISPR kütüphanesi kullanılıyorsa, yaklaşık 33 × 106 CART hücresi). Numuneyi 300 × g'da 4 °C'de 5 dakika santrifüjleyin.
- Hücre peletini 1 mL PBS'de yeniden süspanse edin ve 2 mL'lik bir mikrosantrifüj tüpüne aktarın. 50 mL konik tüpü 1 mL PBS ile yıkayın ve yıkamayı 2 mL mikrosantrifüj tüpüne aktarın.
- Numuneleri 500 × g'da 4 °C'de 5 dakika santrifüjleyin. Süpernatanı çıkarın. Hücre peletlerini -20 °C'de saklayın.
- CD19 + JeKo-1 hücrelerine 1: 1 oranında CART hücreleri ekleyerek ko-kültürü başlatın. Örneğin, 80 × 106 JeKo-1 hücresine 80 × 106 CART hücresi ekleyin. TCM ile ko-kültürün toplam hacmini 160 mL'ye getirin. Numuneleri 37 °C, %5 CO2'de bir inkübatörde saklayın.
NOT: Kitaplık temsilini sürdürmek ve örnekleri 15. ve 22. günlerde korumak için yeterli hücreye sahip olmak için en az 80 ×10 6 CART hücresiyle başlamanızı öneririz.
- 10. Gün: CART hücrelerini yeniden uyarmak için, ko-kültürleri 50 mL konik tüplere toplayın ve numuneleri 300 × g'da 4 ° C'de 5 dakika boyunca santrifüjleyin. Her numuneyi 80 mL TCM'de yeniden süspanse edin ve 80 mL TCM'ye 80 × 106 JeKo-1 hücresi ekleyin. Numuneleri 37 °C, %5 CO2'de bir inkübatörde saklayın.
- 12 . ve 14. günler: Adım 7.2'deki gibi CART hücrelerini yeniden uyarın.
- 15. Gün: Numuneleri NGS için koruyun ve CART hücrelerini yeniden uyarın.
- Önce hücreleri sayarak ve daha sonra CD4 + T hücreleri için pozitif bir seçim kitini CD8 + T hücreleri için pozitif bir seçim kiti ile birleştirerek CART hücrelerini CART-JeKo-1 ko-kültürlerinden izole edin. T hücrelerini sayın.
- 50 mL'lik konik bir tüpte 33 × 106 CART hücresini ayırarak ve numuneyi 300 × g'da 4 °C'de 5 dakika santrifüjleyerek numuneleri NGS için koruyun. Numuneyi 1 mL PBS içinde yeniden süspanse edin ve 2 mL'lik bir mikrosantrifüj tüpüne aktarın. 50 mL konik tüpü 1 mL PBS ile yıkayın ve yıkamayı 2 mL mikrosantrifüj tüpüne aktarın. Numuneleri 500 × g'da 4 °C'de 5 dakika santrifüjleyin; süpernatanı çıkarın; ve hücre peletlerini -20 °C'de saklayın.
- 80 × 10 6 JeKo-1 hücresine 8×0 106 CART hücresi ekleyerek tükenme için in vitro modele devam etmek için ko-kültürler ayarlayın. Ardından, TCM ile ko-kültürün toplam hacmini 160 mL'ye getirin. Numuneleri 37 °C, %5 CO2'de bir inkübatörde saklayın.
- 17, 19 ve 21. günler: Adım 7.2'deki gibi CART hücrelerini yeniden uyarın.
- 22. Gün: Numuneleri NGS için saklayın.
- Önce hücreleri sayarak CART hücrelerini ko-kültürden izole edin. Daha sonra, T hücrelerini CART-JeKo-1 ko-kültürlerinden izole etmek için CD4 + T hücreleri için pozitif bir seçim kitini CD8 + T hücreleri için pozitif bir seçim kiti ile birleştirin. T hücrelerini sayın.
- Örnekleri NGS için korumak için adım 7.4.2'yi tekrarlayın.
8. Yeni nesil dizileme için genomik DNA'nın hazırlanması
NOT: Yeterli kapsama alanını korumak için, genomik DNA (gDNA) en az 33 × 106 CART hücresinden izole edilmelidir. Ek olarak, 33 × 106 CART hücresinden gelen gDNA'nın tamamı dizileme için hazırlanmalıdır.
- Bir gDNA izolasyon kiti kullanarak donmuş hücre peletlerinden gDNA'yı izole edin.
- Etanol çökeltme yoluyla gDNA'yı saflaştırın.
- %100 EtOH'yi -20 °C'ye kadar soğutun.
- Aliquot, DNA'yı birkaç mikrosantrifüj tüpüne (200-250 μL) eşit olarak ayrıştırdı. Her bir alikota aşağıdakileri ekleyin: iki hacim% 100 EtOH (250 μL alikot için 500 μL veya 200 μL için 400 μL); 0.1 hacim 3 M sodyum asetat (250 μL alikot için 25 μL veya 200 μL için 20 μL); ve 1 μL 20 mg / mL glikojen. Numuneleri gece boyunca -20 °C'de inkübe edin.
- İkinci gün, çökelmiş DNA'yı peletlemek için numuneleri 13.000 × g'da 20 dakika döndürün. Süpernatanı çıkarın. Peletleri 1 mL %70 EtOH ile 7.500 × g'da 10 dakika döndürerek yıkayın.
- Kalan EtOH'yi tamamen çıkarın. Peletlerin kurumasını bekleyin 30-45 dakika.
- Peletleri 50 μL steril suda tekrar süspanse edin.
- NGS için gDNA'yı hazırlayın.
NOT: gRNA'ları Illumina adaptör dizileri ile çoğaltan bu CRISPR kütüphanesi için NGS primerleri, önceki bir yayındatasarlanmıştır 27. Her numune için farklı bir ters astar kullanmak, her numuneyi barkodlayacak ve sıralama sırasında numunelerin havuzlanmasına izin verecektir.- Numuneleri aşağıdaki reaksiyon karışımıyla PCR için hazırlayın (toplam hacim = 50 μL): yüksek kaliteli PCR ana karışımı, 2x: 25 μL; havuzlanmış şablon DNA, 0.5 μg: 1 μL; NGS kütüphanesi ileri astar, 10 μM: 1.25 μL; NGS kütüphanesi ters astar, 10 μM: 1.25 μL; DNaz içermeyen su: 21,5 μL.
- Aşağıdaki döngü koşullarıyla PCR gerçekleştirin: döngü 1: 98 °C'de 3 dakika denatüre; döngü 2-23: 10 saniye boyunca 98 ° C'de denatüre, 10 saniye boyunca 63 ° C'de tavlama, 25 saniye boyunca 72 ° C'de uzatın; döngü 24: 72 °C'de 2 dakika uzatın.
- Her numune için PCR reaksiyonlarını bir araya getirin ve PCR ürününü üreticinin talimatlarına göre bir PCR saflaştırma kiti ile saflaştırın.
- %2 (a/h) agaroz jel üzerinde her numune için 3 μg PCR ürünü çalıştırın.
- Üreticinin talimatlarına göre bir jel ekstraksiyon kiti kullanarak DNA'yı jelden çıkararak PCR ürününü (~ 260-270 bp) jelden çıkarın. Ekstrakte edilen numuneleri -20 °C'de saklayın.
- Örneklerle yeni nesil dizileme (NGS) gerçekleştirin. Kütüphanede gRNA başına 500'den fazla okuma hedefleyin.
9. Sıralama sonuçlarının analizi
- NGS'yi analiz etmek için MAGeCK-VISPR platformunukullanın 28. En yüksek olabilirlik tahmini (MAGeCK-MLE) algoritması için standart ayarları kullanın. Çıktı dosyalarını analize hazırlamak için:
- Sıra dosyası kalitesini değerlendirmek için FASTQC'yi çalıştırın31.
- bbmerge32 ile her örnek için eşleştirilmiş uçlu sıralama dosyalarını birleştirin.
- MAGeCK-MLE algoritması ile MAGeCK-VISPR platformundaki numuneleri analiz edin. MAGeCK-MLE analizi için, tüm gRNA sayımlarını hedeflenmeyen 1.000 gRNA listesine normalleştirin.
- GC içeriğini, temel kaliteyi, sıralama okumalarını ve eşlenmiş okumaları değerlendirerek sıralama dosyası kalitesini değerlendirin.
- Gini indeksindeki değişiklikleri, temel bileşen analizi (PCA) kümelemesini ve negatif olarak seçilen genler listesiyle ilişkili yolları değerlendirerek CRISPR ekranının seçici basınç fazının başarısını değerlendirin32.
- Literatür taramaları yaparak ve yol analizi yaparak en iyi hitleri sorgulayın.
Sonuçlar
CART hücre aktivitesini tarafsız bir şekilde iyileştirmek için düzenlenebilen genleri ve yolları sorgulamak için, in vitro genom çapında bir CRISPR nakavt ekranı tasarladık (Şekil 1). Bu ekranın iki aşaması vardır: bir CART hücresi üretim aşaması ve seçici bir basınç aşaması. CART hücre üretim aşamasında, en az 110 × 106 T hücresi önce sağlıklı donör PBMC'lerden izole edilir ve CD3 + / CD28 ...
Tartışmalar
Gen düzenleme, hem tedavilere direnç mekanizmalarını anlamada hem de CART hücrelerinin ömrünü ve aktivitesini iyileştirmek için yeni CART hücre tedavileri tasarlamada güçlü bir araç haline gelmiştir 16,17,26. Bazı gen düzenleme stratejileri hem klinik öncesi modellerde hem de klinik çalışmalarda CART hücre aktivitesinde iyileşmeler göstermiş olsa da, gen düzenleme stra...
Açıklamalar
SSK, Novartis'e (Mayo Clinic, Pennsylvania Üniversitesi ve Novartis arasındaki bir anlaşma yoluyla), Humanigen'e (Mayo Clinic aracılığıyla), Mettaforge'a (Mayo Clinic aracılığıyla) ve MustangBio'ya (Mayo Clinic aracılığıyla) ve Chymal therapeutics'e (Mayo Clinic aracılığıyla) lisanslı CAR immünoterapi alanındaki patentlerin mucididir. CS, CMR ve SSK, Immix Biopharma'ya lisanslı patentlerin mucitleridir. SSK, Kite, Gilead, Juno, BMS, Novartis, Humanigen, MorphoSys, Tolero, Sunesis/Viracta, LifEngine Hayvan Sağlığı Laboratuvarları A.Ş. ve Lentigen'den araştırma fonu almaktadır. SSK, Kite/Gilead, Calibr, Luminary Therapeutics, Humanigen, Juno/BMS, Capstan Bio ve Novartis ile danışma toplantılarına katılmıştır. SSK, Humanigen ve Carisma ile veri güvenliği ve izleme kurulunda görev almıştır. SSK, Torque, Calibr, Novartis, Capstan Bio, BMS, Carisma ve Humanigen için bir danışman ayırdı. CMS ve SSK, bu protokolden kaynaklanan fikri mülkiyetin mucitleridir.
Teşekkürler
Bu çalışma kısmen Mayo Clinic Bireyselleştirilmiş Tıp Merkezi (SSK), Mayo Clinic Kapsamlı Kanser Merkezi (SSK), Mayo Clinic Rejeneratif Biterapötikler Merkezi (SSK), Ulusal Sağlık K12CA090628 Enstitüleri (SSK) ve R37CA266344-01 (SSK), Savunma Bakanlığı hibe CA201127 (SSK), Predolin Vakfı (SSK) ve Minnesota Biyoteknoloji ve Tıbbi Genomik Ortaklığı (SSK) tarafından finanse edilmiştir. CMS, Mayo Clinic Biyomedikal Bilimler Enstitüsü tarafından desteklenmektedir. CRISPR ekran şeması (Şekil 1) BioRender.com (Siegler, L. (2022) https://BioRender.com/k71r054) ile oluşturulmuştur.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 293T cells | ATCC | CRL-3216 | Cells used for lentivirus production |
| Biotin ProteinL Antibody | GenScript | M00097 | anti-kappa chain antibody for CAR detection |
| Bovine Serum Albumin | Millipore Sigma | A7906 | |
| Carbenicillin disodium salt | Millipore Sigma | C1389-1G | Carbenicillin antibiotic |
| CD4 Isolation Beads | Miltenyi Biotec | 130-045-101 | |
| CD8 Isolation Beads | Miltenyi Biotec | 130-045-201 | |
| CTS (Cell Therapy Systems) Dynabeads CD3/CD28 | Gibco | 40203D | |
| Cytoflex | Beckman Coulter | NC2279958 | |
| DNase-Free Water | Invitrogen | AM9937 | |
| Dulbecco's modified eagle's medium (DMEM) | Corning | 10-017-CV | |
| Dulbecco's Phosphate-Buffered Saline | Gibco | 14190-144 | |
| EasySep Human T Cell Isolation Kit | STEMCELL Technologies | 17951RF | Negative isolation kit |
| Endura Electrocompetent Cells | Biosearch Technologies | 60242-1 | Electrocompetent cells with recovery medium |
| Ethanol | Millipore Sigma | E7023 | |
| Fetal bovine serum (FBS) | Corning | 35-010-CV | |
| GeCKO v2 CRISPR Knockout Pooled Library A | AddGene | 1000000048 | CRISPR library plasmid |
| Gene Pulser II | Bio-Rad | 165-2105 | Electroporator |
| Glycogen | Millipore Sigma | 10901393001 | |
| JeKo-1 | ATCC | CRL-3006 | CD19+ target cells |
| Lipofectamine 3000 Transfection Reagent | ThermoFisher Scientific | L3000075 | Transfection reagent kit with a transfection reagent (Lipofectamine 3000 Reagent) and a neutral co-lipid reagent (p3000) |
| LIVE/DEAD Aqua | Invitrogen | L34966 | |
| Lymphoprep | STEMCELL Technologies | 7851 | Density gradient medium |
| Machery-Nagel NucleoBond Xtra Maxi Kits | ThermoFisher Scientific | 12748412 | Maxi-prep kit |
| NEBNext High-Fidelity 2X PCR MasterMix | New England BioLabs | M0541S | High fidelity PCR mastermix |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985-070 | Reduced serum medium |
| pCMVR8.74 | AddGene | 22036 | Lentiviral packaging plasmid |
| Pennicillin-streptomycin-glutamine (100X) | Life Technologies | 10378-016 | |
| pMD2.G | AddGene | 12259 | VSV-G envelope expressing plasmid |
| Pooled Human AB Serum | Innovative Research | ISERABHI | |
| Puromycin | Millipore Sigma | P8833 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | Gek extraction kit |
| Qucik-DNA Midiprep Plus Kit | Zymo Research | D4075 | Kit used to isolate gDNA |
| RoboSep-S | STEMCELL Technologies | 21000 | Automated cell separator |
| Roswell Park Memorial Institute 1640 Medium (RPMI) | Gibco | 21870092 | |
| SepMate-50 | STEMCELL Technologies | 85450 | Density gradient separation tube |
| Sodium Acetate | Invitrogen | AM9740 | |
| Sodium Azide | Fisher Scientific | 71448-16 | |
| Streptavidin Antibody (PE) | BioLegend | 405203 | Secondary antibody used for CAR detection |
| T100 Thermal Cycler | Bio-Rad | 1861096 | |
| Ultracentrifuge (Optima XPN-80) | BeckmanCoulter | A99839 | |
| Vacuum Filter Systems, 0.22 µm | ThermoFisher Scientific | 567-0020 | |
| Vacuum Filter Systems, 0.45 µm | ThermoFisher Scientific | 165-0045 | |
| X-VIVO 15 Serum-Free Hematopoietic Cell Medium | Lonza | 04-418Q | Hematopoietic cell medium |
Referanslar
- Neelapu, S. S., et al. 5-year follow-up supports curative potential of axicabtagene ciloleucel in refractory large B-cell lymphoma (zuma-1). Blood. 141 (19), 2307-2315 (2023).
- Neelapu, S. S., et al. Axicabtagene ciloleucel CAR T-cell therapy in refractory large B-cell lymphoma. N Engl J Med. 377 (26), 2531-2544 (2017).
- Locke, F. L., et al. Long-term safety and activity of axicabtagene ciloleucel in refractory large B-cell lymphoma (zuma-1): A single-arm, multicentre, phase 1-2 trial. Lancet Oncol. 20 (1), 31-42 (2019).
- Maude, S. L., et al. Tisagenlecleucel in children and young adults with B-cell lymphoblastic leukemia. N Engl J Med. 378 (5), 439-448 (2018).
- Schuster, S. J., et al. Tisagenlecleucel in adult relapsed or refractory diffuse large B-cell lymphoma. N Engl J Med. 380 (1), 45-56 (2018).
- Bai, Z., et al. Single-cell antigen-specific landscape of CAR T infusion product identifies determinants of CD19-positive relapse in patients with all. Sci Adv. 8 (23), eabj2820 (2022).
- Fraietta, J. A., et al. Determinants of response and resistance to CD19 chimeric antigen receptor (CAR) T cell therapy of chronic lymphocytic leukemia. Nat Med. 24, 563-571 (2018).
- Gennert, D. G., et al. Dynamic chromatin regulatory landscape of human CAR T cell exhaustion. Proc Natl Acad Sci USA. 118 (30), e2104758118 (2021).
- Deng, Q., et al. Characteristics of anti-CD19 CAR T cell infusion products associated with efficacy and toxicity in patients with large B cell lymphomas. Nat Med. 26 (12), 1878-1887 (2020).
- Sterner, R. M., Cox, M. J., Sakemura, R., Kenderian, S. S. Using CRISPR/CAS9 to knock out GM-CSF in CAR-T cells. J Vis Exp. (149), e59629 (2019).
- Sterner, R. M., et al. GM-CSF inhibition reduces cytokine release syndrome and neuroinflammation but enhances CAR-T cell function in xenografts. Blood. 133 (7), 697-709 (2019).
- Cox, M. J., et al. GM-CSF disruption in CART cells modulates T cell activation and enhances CART cell anti-tumor activity. Leukemia. 36 (6), 1635-1645 (2022).
- Ottaviano, G., et al. Phase 1 clinical trial of CRISPR-engineered CAR19 universal T cells for treatment of children with refractory B cell leukemia. Sci Transl Med. 14 (668), eabq3010 (2022).
- Joung, J., et al. Genome-scale CRISPR-CAS9 knockout and transcriptional activation screening. Nat Protoc. 12, 828-863 (2017).
- Mccutcheon, S. R., et al. Transcriptional and epigenetic regulators of human CD8+ T cell function identified through orthogonal CRISPR screens. Nat Genet. 55, 2211-2223 (2023).
- Zhu, W., Kelly, C., Dagur, P., Dunbar, C. E., Cordes, S. CRISPR activation screen to optimize chimeric antigen receptor (CAR) T cell immunophenotype. Blood. 142 (Supplement 1), 4820-4820 (2023).
- Wang, D., et al. CRISPR screening of CAR T cells and cancer stem cells reveals critical dependencies for cell-based therapies. Cancer Discov. 11 (5), 1192-1211 (2021).
- Shifrut, E., et al. Genome-wide CRISPR screens in primary human T cells reveal key regulators of immune function. Cell. 175 (7), 1958-1971.e15 (2018).
- Belk, J. A., et al. Genome-wide crispr screens of T cell exhaustion identify chromatin remodeling factors that limit T cell persistence. Cancer Cell. 40 (7), 768-786.e7 (2022).
- Jiang, P., et al. Single-cell ATAC-seq maps the comprehensive and dynamic chromatin accessibility landscape of CAR-T cell dysfunction. Leukemia. 36 (11), 2656-2668 (2022).
- Rossi, J., et al. Preinfusion polyfunctional anti-CD19 chimeric antigen receptor T cells are associated with clinical outcomes in NHL. Blood. 132 (8), 804-814 (2018).
- Beider, K., et al. Molecular and functional signatures associated with CAR T cell exhaustion and impaired clinical response in patients with B cell malignancies. Cells. 11 (7), 1140 (2022).
- Wherry, E. J. T. cell exhaustion. Nat Immunol. 12, 492-499 (2011).
- Prinzing, B., et al. Deleting DNMT3A in CAR T cells prevents exhaustion and enhances antitumor activity. Sci Transl Med. 13, eabh0272 (2021).
- Lynn, R. C., et al. C-jun overexpression in CAR T cells induces exhaustion resistance. Nature. 576, 293-300 (2019).
- Tang, N., et al. TGF-beta inhibition via CRISPR promotes the long-term efficacy of CAR T cells against solid tumors. JCI Insight. 5 (4), e133977 (2020).
- Joung, J., et al. Genome-scale CRISPR-CAS9 knockout and transcriptional activation screening. Nat Protoc. 12 (4), 828-863 (2017).
- Li, W., et al. Quality control, modeling, and visualization of CRISPR screens with MAGeCK-VISPR. Genome Biol. 16, 281 (2015).
- Sakemura, R., et al. Targeting cancer-associated fibroblasts in the bone marrow prevents resistance to CART-cell therapy in multiple myeloma. Blood. 139 (26), 3708-3721 (2022).
- Stewart, C. M., et al. IL-4 drives exhaustion of CD8+ CART cells. Nat Commun. 15 (1), 7921 (2024).
- . FastQC: A quality control tool for high throughput sequence data Available from: https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010)
- . Bbmerge Available from: https://github.com/BioInfoTools/BBMap/blob/master/sh/bbmerge.sh (2019)
- Pattanayak, V., et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed CAS9 nuclease specificity. Nat Biotechnol. 31 (9), 839-843 (2013).
- Doench, J. G., et al. Rational design of highly active sgRNAs for CRISPR-CAS9-mediated gene inactivation. Nat Biotechnol. 32 (12), 1262-1267 (2014).
- Dong, M. B., et al. Systematic immunotherapy target discovery using genome-scale in vivo CRISPR screens in CD8 T cells. Cell. 178 (5), 1189-1204.e23 (2019).
- Zhou, P., et al. Single-cell CRISPR screens in vivo map T cell fate regulomes in cancer. Nature. 624 (7990), 154-163 (2023).
- Sutra Del Galy, A., et al. In vivo genome-wide CRISPR screens identify SOCS1 as intrinsic checkpoint of CD4+ TH1 cell response. Sci Immunol. 6 (66), eabe8219 (2021).
- Ramos, A., et al. Leukemia-intrinsic determinants of CAR-T response revealed by iterative in vivo genome-wide CRISPR screening. Nat Commun. 14 (1), 8048 (2023).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır