Method Article
Il pomodoro / GFP-FLP / FRT Metodo per Live Imaging del Mosaico adulti
In questo articolo
Riepilogo
Il metodo Tomato / GFP-FLP / FRT comporta la visualizzazione cellule fotorecettrici mosaico nel vivere Drosophila. Può essere usato per seguire singoli destini delle cellule fotorecettrici nella retina per giorni o settimane. Questo metodo è ideale per gli studi di degenerazione retinica e di malattie neurodegenerative o lo sviluppo delle cellule fotorecettori.
Abstract
L'occhio Drosophila è ampiamente usato come modello per studi di sviluppo e degenerazione neuronale. Con il potente tecnica mitotico ricombinazione, eleganti schermi genetici basati su analisi clonale hanno portato alla identificazione di vie di segnalazione coinvolte nello sviluppo dell'occhio e fotorecettori (PR) la differenziazione nelle fasi larvali. Descriviamo qui il metodo Tomato / GFP-FLP / FRT, che può essere utilizzato per una rapida analisi clonale negli occhi della vita adulta Drosophila. Cellule fotorecettrici fluorescenti vengono esposte con la tecnica cornea neutralizzazione, sulla retina con cloni mosaico generati mediante ricombinazione flipase-mediata. Questo metodo presenta alcuni importanti vantaggi rispetto classica sezionamento istologica della retina: può essere utilizzato per screening ad alto rendimento e si è dimostrato un metodo efficace per identificare i fattori che regolano la sopravvivenza e la funzione PR. Può essere usato per analisi cinetiche di PR degenerazione nella stessa anim soggiornoAl diverse settimane, per dimostrare la necessità di geni specifici per la sopravvivenza PR o funzione nella mosca adulta. Questo metodo è anche utile per affrontare problemi di autonomia cella mutanti sviluppo, come quelli in cui l'istituzione della polarità cellulare planare è interessato.
Introduzione
La retina Drosophila è composto da circa 800 unità ommatidial (Figura 1A), organizzati appunto, con un asse ben definito di polarità (Figura 1B). Ogni unità contiene 20 celle: otto cellule fotorecettrici (PRS) e 12 cellule accessorie, comprese le cellule dei coni, cellule pigmentate e setole cellule 1,2. Due classi di PR si distinguono in base al tipo di rodopsina (Rh) esprimono. I sei PR esterne (R1-6) esprimono Th1 e sono disposte in un modello trapezoidale (Figura 1C). Nel centro del trapezio, i due PR interni (R7/R8) esprimono quattro possibili tipi di rodopsina (RH3, RH4, RH5 o RH6) e sono organizzati in modo tale che R7 si trova sulla cima di R8 3.
Più di 2.500 geni sono coinvolti nella morfogenesi dell'occhio Drosophila 4, che si è dimostrato un modello molto potente per studi di un ampio pannello di processi, compresi svi occhioluppo, PR reclutamento, la differenziazione, la polarità planare delle cellule, morfogenesi, la sopravvivenza, l'apoptosi e trasduzione visiva 5-9.
I ricercatori che lavorano sugli occhi Drosophila hanno, nel corso di molti anni, le tecniche di imaging della retina e la realizzazione di schermi genetici sistematici sviluppato. Il modo più semplice per l'immagine della retina adulta è quello di esaminare la cornea in un animale immobilizzato (Figura 1A). La struttura della cornea può essere visualizzata con precisione da microscopia elettronica a scansione ed è stato utilizzato in uno screening genetico su larga scala dal consorzio URCFG ( http://www.bruinfly.ucla.edu ) 10. Questo è un approccio molto efficace per la visualizzazione di cambiamenti globali morfologiche nella struttura corneale, come rugosità degli occhi o lucentezza, spesso indotte da mutazioni in geni che controllano i primi passi nello sviluppo o la vitalità cellulare. Tuttavia, visualizzazione globale della cornea non è sufficiente di identificare i fattori che regolano PR rhabdomere morfogenesi o adulto PR vitalità e funzione. Tali analisi richiedono un esame approfondito più di integrità PR, basato sulla microscopia a contrasto di fase di sezioni in resina semi-sottili tangenziali della retina 11-13. Questa tecnica è adatta per analisi di mosaico, in cui PR mutanti possono essere identificati dalla loro mancanza di pigmento rosso 14,15.
Cloni mutanti mosaico possono anche essere visualizzati in dissezioni tutto il montaggio della pupa o adulto retina, sulla base della loro mancanza di proteina fluorescente verde segnale (GFP) sulla fluorescenza microscopia 16,17. Queste due tecniche sono molto utili, ma entrambi sono-lavoro e che richiede tempo e non sono quindi adatti per lo screening su larga scala. Noi e altri abbiamo sviluppato l'uso di tecniche cornea neutralizzazione per l'imaging di PR producono proteine fluorescenti 18,19, per facilitare l'analisi rapida PR. Con questa tecnica, mutantecloni possono essere identificati sulla base della autofluorescenza della (w +) pigmento rosso in mosaico adulti Drosophila cloni. Tuttavia, questo metodo non fornisce la risoluzione unicellulare necessaria per combattere autonomia cella emette 19. Abbiamo superato questo problema sviluppando il metodo di pomodoro / GFP-FLP / FRT, che combina l'imaging di proteine fluorescenti da cornea neutralizzazione con ricombinazione mitotica 20. Questo metodo consente la-throughput elevato, rapida e precisa identificazione delle PR mutanti in cloni di mosaico, con risoluzione unicellulare ( http://www.ens-lyon.fr/LBMC/ApoDrosoDatabase/ ). E 'adatto per l'uso nelle analisi cinetiche, per questi singoli SPE in un periodo di settimane nel vivere Drosophila. Descriviamo qui il metodo Tomato / GFP-FLP / FRT e suggerimenti per il suo utilizzo in semplici, analisi cinetiche degli occhi mosaico.
Protocollo
1. Attraversando con linee di pomodoro / GFP-FLP / FRT
La generazione di mosche trasportano il clone mutante richiede un unico incrocio tra una linea mutante-FRT trasporto e il corrispondente Tomato / GFP-FLP / FRT linea di volo.
Questo lavoro ha utilizzato la raccolta BruinFly di mutazioni FRT-ricombinati ( http://www.bruinfly.ucla.edu ). Quattro linee Tomato / GFP-FLP / FRT possono essere utilizzati, portando FRT Th1-tdtomatoninaC su ogni braccio del 2 ° e 3 ° cromosomi in combinazione con fonti di flipase (ey-FLP) e l'espressione di GFP in tutti i PR esterne (Th1-Gal4 UAS -GFP) 20:
- 2L braccio: linea # 43345, P {ry [+ t7.2] = RH1-GAL4} 1, P {ry [+ t7.2] = ey-FLP.N} 2, w [*]; P {w [+ mC] = ninaE-tdTomato-ninaC} 2L P {ry [+ t7.2] = neoFRT} 40A; P {w [+ mC] = UAS-GFP-ninaC} 3
- 2R braccio: linea # 43346, P {ry [+ t7.2] = RH1-GAL4} 1, P {ry [+ t7.2] = ey-FLP.N} 2, w [*]; P {ry [t7.2 +] = neoFRT} 42D P{W [+ mC] = ninaE-tdTomato-ninaC} 2R; P {w [+ mC] = UAS-GFP-ninaC} 3
- 3L braccio: linea # 43347, P {ry [+ t7.2] = RH1-GAL4} 1, P {ry [+ t7.2] = ey-FLP.N} 2, w [*]; P {w [+ mC] = UAS-GFP-ninaC} 2; P {w [+ mC] = ninaE-tdTomato-ninaC} 3L P {ry [+ t7.2] = neoFRT} 80B/TM6B, Tb [1]
- 3R braccio: linea # 43348, P {ry [+ t7.2] = RH1-GAL4} 1, P {ry [+ t7.2] = ey-FLP.N} 2, w [*]; P {w [+ mC] = UAS-GFP-ninaC} 2; P {ry [+ t7.2] = neoFRT} 82B P {w [+ mC] = ninaE-tdTomato-ninaC} 3R/TM6B, Tb [1]
Queste quattro linee sono disponibili presso il magazzino centrale Bloomington Drosophila. Hanno fornito una fonte di FLP nell'occhio sviluppo per la generazione di cloni mitotiche 21. La wild-type cromosoma trasporta una sequenza FRT e RH1-tdTomato ninaC costrutto che codifica per la proteina fluorescente rossa tdTomato, come marcatore per la wild-type e PRS esterni eterozigoti. In questo costrutto, gli ultimi 41 amminoacidi della isoforma p174 del ninaC (né inattivazione né attivazione C) gene era appended in-frame al C-terminale della sequenza tdTomato. La coda C-terminale dell'isoforma p174 ninaC è responsabile per la localizzazione rhabdomeral della proteina 22 e permessi migliore visualizzazione in tempo reale di PR fluorescenti rossi con la tecnica cornea neutralizzazione. Il promotore RH1 è il minimo 234 bp (-152 +82) promotore RH1. Il marcatore fluorescente verde GFP è espressa da tutte le PR esterne, a causa della presenza del RH1-GAL4 (promotore 3kb) e UAS-GFP ninaC costrutti. Incroci tra un titolo mosca portatrice di una mutazione sul cromosoma x FRT (FRT-x) e le linee di pomodoro / GFP-FLP / FRT produce una prole con il seguente genotipo (esempio di mutazione su 2L): RH1-Gal4, ey-FLP ; FRT40A, RH1-tdTomato ninaC / FRT40A-x; UAS-GFP ninaC. In queste mosche, cellule mutanti omozigoti sono generati per ricombinazione mitotica nel disco occhio sviluppando e si dividono per generare un clone di PR mutanti omozigoti (Figura 2A). Omosessualecellule mutanti zygous possono essere identificati perché esprimono solo la proteina fluorescente verde (GFP), mentre la wild-type circostante e cellule eterozigoti esprimono sia tdTomato e GFP (Figure 2B e B ").
2. Preparazione Drosophila per fotorecettore Visualization
Per visualizzare PR dalla tecnica cornea neutralizzazione, immobilizzare linea su piastre di agarosio.
- Preparare una bottiglia di lavaggio riempita con acqua a 4 ° C distillata e posizionarlo sul ghiaccio.
- Sciogliere agarosio regolare in acqua (1,2-1,5% agarosio in 100 ml di acqua) mediante riscaldamento in un forno a microonde. Porre il pallone in un bagno d'acqua a 55 ° C e lasciare che la soluzione di agarosio raffreddare a questa temperatura. Mantenere l'agarosio a 55 ° C fino all'utilizzo.
- Anestetizzare le mosche con CO 2 per almeno 1 min.
- Versare la soluzione di agarosio calda (a 55 ° C) in una capsula di Petri (35 x 10 mm, 1,37 x 0,39 in) e posizionare immediatamentediatamente la Drosophila anestetizzato sul agarosio. Per i principianti, a partire da 10 mosche per capsula di Petri è ragionevole. Un'antenna più grande Petri (60 x 15 mm, 2.362 x 0,59 in) può anche essere usato.
- Sotto un microscopio da dissezione, orientare la Drosophila su un lato con una pinza, spingere un'ala in agarosio, tale che l'ala e metà del corpo sono incorporati nel agarosio (Figure 3A-3A ""). Attaccare l'altra ala sulla superficie del agarosio. Nota: Se le mosche non sono incorporati abbastanza profondamente nel agarosio, la mosca sarà libero di muovere la testa durante l'imaging del PR, e questo si tradurrà in immagini sfocate. L'orientamento dell'occhio sarà anche disturbato. Può essere difficile far precipitare il Drosophila in agarosio, a causa sia della concentrazione di agarosio o la sua temperatura. E 'più facile incorporare mosche in agarosio più concentrato, ma se l'agarosio è troppo concentrato, la mosca può affondare. Se l'agarosio è troppo freddo, può essere difficult per incorporare al volo, ma una temperatura troppo alta può danneggiare la cornea.
- Mettere la piastra di Petri su ghiaccio e consentire l'agarosio solidificare.
- Con pinze, orientare la testa in modo che un occhio è esposto l'obiettivo ad immersione (Figura 3A 'e 3A ""). Generalmente, un occhio può essere considerato ben orientata al microscopio da dissezione quando il pseudopupil (visualizzata come un punto nero) è al centro dell'occhio. Un orientamento ottimale dell'occhio massimizza la larghezza del campo ommatidial in cui i PR sono focalizzati. Lo scopo della fase di orientamento è per individuare la zona degli occhi con il più ampio campo di PR focalizzate, di solito al centro dell'occhio. Questa fase è utile per rimuovere eventuali gambe o aristae coprire l'occhio e per prevenire la visualizzazione.
- Coprire la mosca con acqua ghiacciata e lasciare la piastra di Petri in ghiaccio fino visualizzazione. Acqua ghiacciata mantiene in linea anestetizzati.
3. VisualizzazioneFotorecettori sotto il microscopio
Per la visualizzazione di PR attraverso la cornea, questo protocollo utilizza un microscopio verticale dotato di un obiettivo acqua con una lunga distanza di lavoro (W N-Achroplan 40X/0.75).
- Porre la capsula di Petri coperta con acqua ghiacciata su un vetrino sul palco del microscopio (figure 3 aC). La piastra di Petri può essere incollato al vetrino, rendendo possibile spostarlo su uniformemente con le viti micrometriche.
- Immergere l'obiettivo ad immersione nell'acqua della scatola di Petri (Figure 3B'-3C '). Posizionare la testa della mosca sotto il fascio di eccitazione (iniziare con il filtro GFP, per esempio). Spostare il palco su e giù finché il raggio converge sull'occhio. Quando l'occhio è al giusto livello, tende a riflettere la luce di eccitazione.
- Guardare attraverso l'oculare per la fluorescenza. Quando l'occhio è posizionato al centro del campo di vista, concentrare sotto lacornea per visualizzare i fotorecettori fluorescenti.
Nota 1: Se si guarda attraverso l'oculare, può essere a disagio per distinguere le parti del corpo della mosca, in particolare la parte distale del ventre e la testa, per esempio. In questi casi, si dovrebbe guardare direttamente allo stadio per riposizionare la Drosophila.
Nota 2: fluorescenza classica o microscopia confocale possono essere utilizzati. La microscopia confocale offre immagini con meno di sfondo e un più ampio campo di PR mirate della microscopia classica. Un esempio di set-up è un LSM510 microscopio confocale (Zeiss) con un obiettivo di acqua 40X. Risultati migliori si ottengono aprendo il foro del microscopio confocale più ampio di quanto normalmente raccomandato per l'obiettivo. Ad esempio, utilizzare un valore di 204, anziché il valore predefinito 98 per l'apertura del foro.
Nota 3: i livelli più elevati di w + pigmentazione dell'occhio ridurre la fluorescenza di fondo.
4. Time-corso visualizzazione di cellule fotorecettrici
Cura è necessaria quando si segue il destino di un individuo PR all'interno dello stesso occhio per un periodo di tempo.
- Ridurre la temperatura del agarosio a 45 ° C, per massimizzare la sopravvivenza Drosophila. L'agarosio solidifica molto rapidamente a questa temperatura. Solo uno Drosophila deve essere usato per ogni piatto Petri, per garantire che le mosche non mescolati durante osservazioni.
- Sistematicamente posizionare in linea sullo stesso lato, per la visualizzazione dello stesso occhio. Ciò può essere ottenuto prendendo al volo anestetizzato dal flacone da un'ala e mettendo la Drosophila sul agarosio. Orientare sempre l'occhio allo stesso modo, se possibile, rendendo più facile trovare cloni di PR che sono stati visti prima.
- Identificare i cloni sulla base della loro forma al microscopio confocale. Il campo visivo è spesso situato vicino al clone di interesse. In such casi, l'occhio dovrebbe essere riorientata sott'acqua, per centrare il campo visivo sul clone di interesse, basato sulla polarità dell'occhio (Figura 4).
5. Recuperare mosche dopo Visualization
- Rimuovere l'acqua dalla piastra di Petri.
- Estrarre delicatamente la Drosophila fuori dal agarosio con una pinza.
- Essiccare la Drosophila su un tessuto.
- Riportare la Drosophila in una fiala, assicurando che non si blocca nel cibo. Sono ammessi al volo per venire attorno a 25 ° C.
Risultati
Il metodo Tomato / GFP-FLP / FRT può essere usato per studiare l'effetto di una mutazione o di espressione ectopica sullo sviluppo e la sopravvivenza di PR nella retina Drosophila. E 'rapido, che lo rende ideale per scopi di screening, come recentemente dimostrato 20. La presenza di PR mutanti accanto alla wild-type PR nello stesso occhio rende facile rilevare i difetti associati PR mutanti e di affrontare la questione dell'autonomia delle cellule.
I difetti di sviluppo osservati nella nostra analisi colpiti vari processi, tra cui il reclutamento di PR, la morfogenesi e la polarità planare delle cellule (PCP) stabilimento (Figura 5). Sette in contumacia è necessario per PR assunzioni, in particolare per R7 uno dei PR interiore. La perdita di Seven nei risultati contumacia la perdita del PR interiore e alcuni PR esterni (Figura 5A). Testa granulare (GRH) è coinvolto nella creazione PCP e alcune ommatidi sono inverted in GRH cloni mutanti (Figura 5B). Questo metodo permette di identificare i PR mutanti in ommatidia mosaico, facilitando l'identificazione delle PR in cui è richiesto il gene per la corretta acquisizione del PCP. Abbiamo dimostrato che è necessario che GRH per la corretta acquisizione del PCP in R3 precursori 20. Infatti, abbiamo osservato che, nel ommatidia invertita, R4, che ha origine anormalmente dal precursore R3, era sempre mutante. Briciole (CRBs) è una proteina di membrana apicale richiesto per la morfogenesi rhabdomere 23. La perdita di CRBs nei CRBs 11A22 risultati mutanti in irregolari, PR più o meno grandi (Figura 5C).
Questo metodo può anche essere usato per seguire il destino dei singoli PR nell'età adulta (Figura 6). E 'ideale per studi di neurodegenerazione, perché un gruppo di PR omozigote mutante può essere visualizzato e lo stesso gruppo possono essere trovate nella stessa occhio delstesso volare per un periodo di giorni o settimane. E 'quindi possibile determinare quali PR sopravvivono e che sono destinate a morire, perché ogni clone ha una forma unica che può essere riconosciuto quando l'occhio è posizionato al microscopio a fluorescenza. Poiché la retina è polarizzata, è possibile orientare la retina ritrovare lo stesso clone, sulla base della sua forma. Abbiamo usato questo metodo per dimostrare che FATP k10307 mutazione induce degenerazione progressiva PR in età adulta (Figura 6, 24). Questo metodo di visualizzazione può essere utilizzato anche per analizzare autonomia cella. In FATP k10307 retina mosaico, perdita di PR è stata limitata a FATP PR mutanti, PRS wild-type è influenzato. Il requisito di FATP per vitalità PR è quindi cellula autonoma. Siamo stati anche in grado di salvare il FATP mutante PR da reexpressing wild-type FATP con un driver RH1-Gal4 e un costrutto UAS-FATP (Figura 7). Ilpossibilità di realizzare tali esperimenti soccorso è uno dei vantaggi del metodo Tomato / GFP-FLP / FRT sopra analisi clonale basato su sezioni istologiche di retina resina incorporato. Infatti, rilevamento clonale con il metodo pomodoro / GFP-FLP / FRT non è influenzato dall'uso di P (UAS, w +) costrutti transgenici, mentre la pigmentazione rosso associato con il mini-bianco gene (w +) copre l'occhio con pigmento rosso , mascherando cloni mosaico in sezioni istologiche.
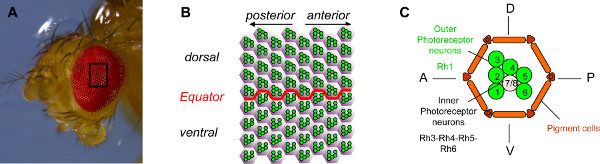
Figura 1. Organizzazione degli occhi Drosophila. (A) fotografia di un occhio Drosophila prese con uno stereomicroscopio. L'occhio Drosophila è composto da circa 800 ommatidia. (B) Schema di un campo di 64 ommatidi al centro dell'occhio. I sei neuroni fotorecettori esterno (PRS) di ogni ommatidium,in verde, sono organizzati in un trapezio stereotipata che punta ai poli più vicini dell'occhio. Hanno quindi puntano in direzioni opposte nel ventrale e la parte dorsale dell'occhio e sono l'immagine speculare secondo il confine tra le due parti, che si chiama l'equatore. (C) Schema di un ommatidium. Ogni ommatidium contiene otto PR. PR R1 a R6, in verde, sono i PR esterne. Essi esprimono la molecola rhodopsin1 fotosensibile (Th1) e sono l'equivalente dei bastoncelli di mammifero. Il PRS interni, R7 e R8, sono mostrati in grigio (R7 si trova sulla cima della R8). Il PRS interni esprimono rodopsina 3, 4, 5 o 6 e sono l'equivalente delle cellule cono di mammifero. Per semplicità, solo il rhabdomere, la parte sensibile alla luce del PR, viene mostrato per ogni PR. Clicca qui per ingrandire la figura .
Figura 2. Generazione e visualizzazione di cloni PR mosaico nell'occhio Drosophila con il metodo del pomodoro / GFP-FLP / FRT. (A) Schema della generazione di cloni di mosaico nel metodo Tomato / GFP-FLP / FRT. Cloni mosaico sono generati mediante ricombinazione mitotica, causa l'espressione del flipase (sotto il controllo del promotore eyeless) durante lo sviluppo dell'occhio e le sequenze FRT. Dopo la ricombinazione della sequenza FRT e divisione cellulare, mutante omozigote, omozigote wild-type e cellule eterozigoti può essere generata da una cella eterozigoti. Queste cellule si dividono di nuovo a formare cloni mosaico di PR negli occhi degli adulti. L'identificazione di cellule mutanti è facilitata inserendo un costrutto che esprime la proteina fluorescente rossa tdTomato nel wild-type cromosoma, etichettare wild-type e cellule eterozigoti. (B-B'') Visualizzazione di cloni PR mosaico con tmetodo da lui Tomato / GFP-FLP / FRT. Il metodo Tomato / GFP-FLP / FRT riunisce ricombinazione mitotica, cornea neutralizzazione e microscopia confocale. Tutti PR esprimono la proteina fluorescente GFP verde, che facilitano la visualizzazione del PRS con cornea neutralizzazione e la microscopia confocale (B). PR possono essere visualizzati a livello di singola cellula. Cloni mutanti mosaico sono generati mediante ricombinazione mitotica e possono essere identificati dall'assenza della proteina fluorescente rossa tdTomato (B '). Di conseguenza, sulle immagini unite, PR omozigoti appaiono in verde, mentre la wild-type e eterozigoti PR sono di colore giallo (B''). Clicca qui per ingrandire la figura .
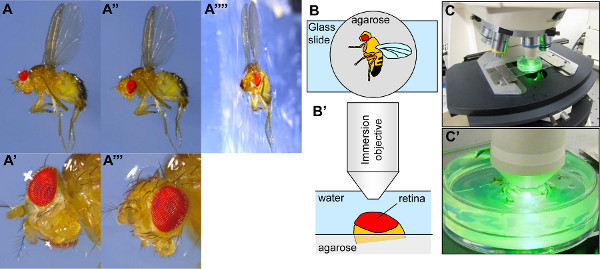
Figura 3. Impostazione della Drosophila per visualizatione cornea neutralizzazione. (A-A'''') Fotografie di una Drosophila immobilizzati su un piatto riempito di agarosio. Nel pannello A'''', un pezzo di agarosio contenente il fly è stata tagliata per scattare una fotografia del profilo. La Drosophila è mezza incorporato nel agarosio, con l'ala destra all'interno del agarosio e la sinistra bloccato sulla superficie agarosio (A, A'', A''''). Quando la Drosophila è incorporato nel agarosio, la testa è posizionata frequentemente mal per la visualizzazione dell'occhio (A, A '). La testa deve quindi essere riorientato con una pinza tali che il centro dell'occhio è rivolto verso l'alto (A'', A'' '). (B-B') diagramma schematico che mostra un Drosophila immobilizzato su una piastra di agarosio e il posizionamento del Drosophila sotto l'obiettivo del microscopio. (C-C ') Le fotografie della regolazione della Drosophila sul palco delmicroscopio sotto il suo obiettivo. Clicca qui per ingrandire la figura .
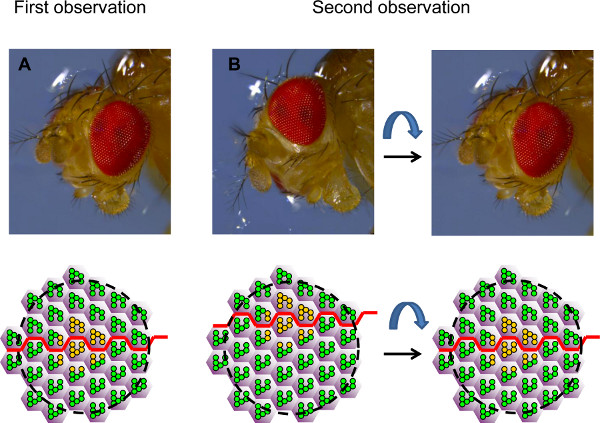
Figura 4. Orientamento dell'occhio per l'analisi time-course. La figura mostra le fotografie di una testa di Drosophila in due diversi orientamenti ei campi corrispondenti di ommatidia visti dalla cornea neutralizzazione, rappresentati come diagrammi schematici. (A) Durante la prima osservazione, l'occhio è posto tale che un clone di tipo selvaggio o eterozigoti PR (in giallo) viene visualizzato al centro del campo, a livello dell'equatore (linea rossa). (B) Nella seconda osservazione, l'occhio spesso non è esattamente lo stesso orientamento. Così, lo stesso gruppo di PR può essere ritrovato, ma non è possibile visualizzare l'esatto stesso campo (a sinistra). L'occhio deve essere riorientata per lo stesso campo da visualizzare nuovamente. La posizione dell'equatore può essere utilizzato come guida. In questo esempio, sappiamo che il campo osservato è troppo ventrale e che l'occhio deve quindi essere ruotato ventralmente per posizionare l'equatore nel mezzo del campo osservato di nuovo, come nella prima osservazione.
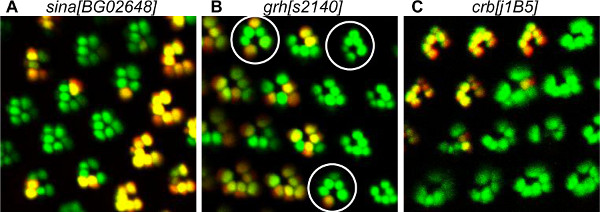
Figura 5. Esempi di PR difetti di sviluppo individuati dal metodo Tomato / GFP-FLP / FRT. (A) Visualizzazione di mosaico sina [BG02648] PR mutanti, mostrando la perdita di PR interne. Nel ommatidia mutante, PR esterni sono raggruppati insieme per l'assenza di quella interna PR R7. Infatti, sina è noto per essere richiesto per il reclutamento PR interiore. (B) Visualizzazione di GRH mosaico [s2140] PR mutanti che presentano difetti di polarità. Il mosaico mutante ommatidi, SURRondato da un cerchio, puntano nella direzione opposta da wild-type ommatidia, indicando una inversione dorso-ventrale. Uno studio dettagliato con il metodo del pomodoro / GFP-FLP / FRT ha dimostrato che GRH è stato richiesto nel precursore R3 per la corretta acquisizione di ommatidium polarità 20. (C) Visualizzazione di mosaico CRB [j1B5] PR mutanti, mostrando deformati PR mutante omozigote. Crb è noto per essere richiesto per morfogenesi rhabdomere.
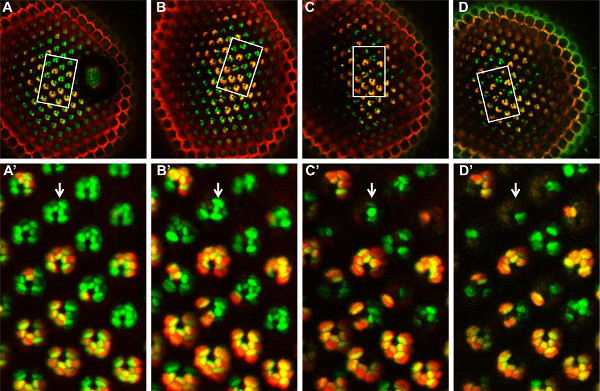
Figura 6. Analisi tempo-corso di FATP mosaico [k10307] PR mutanti con il metodo di pomodoro / GFP-FLP / FRT, più di 14 giorni. FATP Un mosaico [k10307] retina mutante si osserva il giorno 1 (A, A '), giorno 4 ( B, B '), il giorno 8 (C, C') e il giorno 14 (D, D ') dopo la schiusa. Lo stesso gruppo di PR mosaico può essere fond ad ogni tempo, nel campo osservato (rettangolo bianco, A, B, C, D). In questo campo di PR (A ', B', C ', D'), PR mutanti omozigoti sono etichettati in verde, mentre PR wild-type eterozigoti ed omozigoti sono etichettati in giallo. Dal giorno 4 (B ') in poi, i PR mutante omozigote iniziano a scomparire, indicando che il FATP [k10307] mutazione induce una progressiva degenerazione di questi PR. Clicca qui per ingrandire la figura .
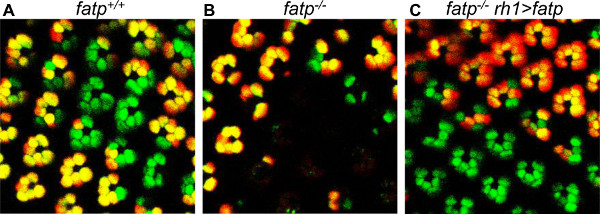
Figura 7. Salvataggio di FATP [k10307] PR mutanti con un mini-bianco-contabile-FATP esprimere costruire. PRS mosaico sono visualizzate con il metodo del pomodoro / GFP-FLP / FRT in 15 giorni di età mosche. (A) Visualizzazione di controllo mosaico PR. (B) Visualization di mosaico FATP PR mutanti, mostrando la perdita di PR mutanti. (C) Visualizzazione del mosaico FATP PR mutanti in una mosca reexpressing FATP in PR esterno (rh1> FATP). Il PRS mutanti sono salvati. La presenza di un mini-bianco gene e la pigmentazione occhio rosso associato in rh1> condizioni FATP non altera pomodoro o fluorescenza GFP o rilevamento clonale.
Discussione
L'occhio Drosophila è stato ampiamente utilizzato per decifrare le vie di segnalazione che regolano lo sviluppo, la proliferazione e la sopravvivenza. Nei primi anni 1990, un gran numero di schermi genetici sono stati effettuati per identificare i percorsi necessari durante le fasi iniziali dello sviluppo PR 25-27. L'efficienza degli schermi genetici è stata aumentata notevolmente con la tecnica di ricombinazione mitotica, che consente di testare perdita-di-funzione mutazioni nei cloni mosaico 21. Quindi, il ruolo delle mutazioni letali embrionali potrebbe essere testato sistematicamente in cloni mutanti omozigoti negli occhi di mosche che erano altrimenti eterozigoti. La maggior parte delle proiezioni mosaico si sono concentrati sui primi PR reclutamento, la differenziazione, la proiezione assonale o morfogenesi che si verificano nelle larve o pupe di sviluppo 10,25-31. Ad oggi, solo una schermata mosaico ha indagato i meccanismi che regolano la funzione adulto PR, attraverso il monitoraggio della risposta visiva tramite electroretinOgram Registrazioni 32. Con il metodo Pomodoro / GFP-FLP / FRT e la possibilità di effettuare analisi andamento nel tempo a vivere animali mutanti, abbiamo progettato un nuovo metodo per identificare i fattori che regolano la vitalità PR adulti e funzionamento 20,24.
Il metodo Tomato / GFP-FLP / FRT ha diversi importanti vantaggi rispetto al sezionamento istologico classico occhi di resina-embedded. In primo luogo, questo metodo è molto più veloce, più economico e più facile da eseguire rispetto al metodo istologico, a condizione che un microscopio a fluorescenza dotato di un adeguato obiettivo di immersione acqua è disponibile (vedi Tabella di reagenti e attrezzature). In secondo luogo, il metodo di pomodoro / GFP-FLP / FRT può essere utilizzato in combinazione con l'espressione del transgene, come l'espressione di P (UAS, w +), portando un mini-bianco transgene. In contrasto con sezioni istologiche, in cui viene utilizzato w + per il rilevamento clonale, nel sistema Pomodoro / GFP-FLP / FRT, P (UAS, w +) non interferisce con clonale detection basato sulla proteina fluorescente Pomodoro. Utilizzando P (UAS-FATP, w +) mosche transgeniche, siamo stati in grado di visualizzare in soccorso di PR mutanti FATP (Figura 7). In terzo luogo, un trabocchetto chiave di tutti i tipi di analisi clonale, compresa quella basata sulla Tomato / GFP-FLP / FRT, è l'ambiguità del genotipo di PR perduti. Infatti, può essere chiaro se un PR è assente a causa della mancanza di una funzione gene specifico oa causa di una cella effetto non autonomo, in particolare al confine del clone mutante. Il metodo Tomato / GFP-FLP / FRT aggira questo problema per degenerazione rendendo possibile seguire wild-type e mutanti PR nello stesso animale su periodi di diverse settimane. Siamo stati in grado di seguire il destino dei singoli PR di cinetica analisi su diversi FATP mosche mutanti (Figura 6). Lo stesso clone può essere riconosciuto in un determinato animale in tempi diversi, a causa della forma precisa delle wild-type circostanti cloni. Siamo stati in grado di mostrare inequivocabilely che tutte le PR mancanti erano mutante per FATP, indicando che il ruolo di mutanti FATP in PR è la cellula-autonoma. Pertanto, monitorando la perdita di PR in una analisi cinetica, è possibile determinare il genotipo di PR in modelli di adulto-inizio degenerazione. Infine, il metodo di pomodoro / GFP-FLP / FRT è dimostrato molto potente per l'identificazione dei fattori che regolano la costituzione del PCP 20. La possibilità di aver realizzato un gran numero di ommatidia mosaico per un fenotipo polarità senza la necessità di sezioni, facilita la rapida determinazione del requisito PR per la creazione di PCP. Tuttavia, analisi più fine dell'integrità PR richiederebbe taglio tradizionale seguita da contrasto di fase o di microscopie elettroniche.
In conclusione, il metodo di pomodoro / GFP-FLP / FRT apre nuove possibilità per lo screening mosaico efficace per identificare i fattori che regolano i processi di sviluppo di pubbliche relazioni e funzioni adulti di PR, come la visrisposta manuale e la vitalità.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
PLATIM e Droso-Tools strutture del UMS3444 Biosciences, Lione, Francia. La ricerca di BM è stata sostenuta da sovvenzioni dal Fondation pour la Recherche Médicale dal CNRS (ATIP) e l'ANR-12-BSV1-0019-01. PD è stato supportato da Retina Francia associazione e l'Ecole Normale Supérieure di Lione (Francia). CL è stato supportato da La Ligue Nationale contre le Cancer Association.
Materiali
| Name | Company | Catalog Number | Comments |
| Reagent | |||
| Agarose | Euromedex | D5-E | Regular agarose is used to immobilize the flies |
| Petri dish | BD Falcon | 353001 | 35 x 10 mm, 1.37 x 0.39 inch |
| Cold-ice water | To maintain the flies anesthetized | ||
| Equipment | |||
| Dissecting microscope | Dutcher | SMZ645 | |
| Water Bath | Julabo | 9550102 | To keep the agarose solution at warm temperature |
| 40X Water objective | Zeiss | 420967-9900-000 | Water dipping objective for the confocal microscope |
Riferimenti
- Hardie, R. C., Ottoson, D. . Sensory physiology 5. 5, 1-79 (1985).
- Wolff, T., Ready, D. F. Cell death in normal and rough eye mutants of Drosophila. Development. 113, 825-839 (1991).
- Rister, J., Desplan, C. The retinal mosaics of opsin expression in invertebrates and vertebrates. Dev Neurobiol. 71, 1212-1226 (2011).
- Halder, G., Callaerts, P., Gehring, W. J. Induction of ectopic eyes by targeted expression of the eyeless gene in Drosophila. Science. 267, 1788-1792 (1995).
- Mollereau, B., Domingos, P. M. Photoreceptor differentiation in Drosophila: from immature neurons to functional photoreceptors. Dev Dyn. 232, 585-592 (2005).
- Mollereau, B. Cell death: what can we learn from flies? Editorial for the special review issue on Drosophila apoptosis. Apoptosis. 14, 929-934 (2009).
- Charlton-Perkins, M., Brown, N. L., Cook, T. A. The lens in focus: a comparison of lens development in Drosophila and vertebrates. Mol Genet Genomics. 286, 189-213 (2011).
- Jenny, A. Planar cell polarity signaling in the Drosophila eye. Curr Top Dev Biol. 93, 189-227 (2010).
- Montell, C. Drosophila visual transduction. Trends Neurosci. 35, 356-363 (2012).
- Chen, J., et al. Discovery-based science education: functional genomic dissection in Drosophila by undergraduate researchers. PLoS Biol. 3, e59 (2005).
- Tomlinson, A., Ready, D. F. Neuronal differentiation in the Drosophila ommatidium. Dev Biol. 120, 366-376 (1987).
- Mendes, C. S., et al. ER stress protects from retinal degeneration. Embo J. 28, 1296-1307 (2009).
- Jenny, A. Preparation of adult Drosophila eyes for thin sectioning and microscopic analysis. J Vis Exp. , (2011).
- Mollereau, B., et al. Two-step process for photoreceptor formation in Drosophila. Nature. 412, 911-913 (2001).
- Domingos, P. M., et al. Spalt transcription factors are required for R3/R4 specification and establishment of planar cell polarity in the Drosophila eye. Development. 131, 5695-5702 (2004).
- Mendes, C. S., et al. Cytochrome c-d regulates developmental apoptosis in the Drosophila retina. EMBO Rep. 7, 933-939 (2006).
- Domingos, P. M., et al. Regulation of R7 and R8 differentiation by the spalt genes. Dev Biol. 273, 121-133 (2004).
- Mollereau, B., et al. A green fluorescent protein enhancer trap screen in Drosophila photoreceptor cells. Mech Dev. 93, 151-160 (2000).
- Pichaud, F., Desplan, C. A new visualization approach for identifying mutations that affect differentiation and organization of the Drosophila ommatidia. Development. 128, 815-826 (2001).
- Gambis, A., Dourlen, P., Steller, H., Mollereau, B. Two-color in vivo imaging of photoreceptor apoptosis and development in Drosophila. Dev Biol. 351, 128-134 (2011).
- Golic, K. G. Site-specific recombination between homologous chromosomes in Drosophila. Science. 252, 958-961 (1991).
- Porter, J. A., Hicks, J. L., Williams, D. S., Montell, C. Differential localizations of and requirements for the two Drosophila ninaC kinase/myosins in photoreceptor cells. J Cell Biol. 116, 683-693 (1992).
- Johnson, K., Grawe, F., Grzeschik, N., Knust, E. Drosophila crumbs is required to inhibit light-induced photoreceptor degeneration. Curr Biol. 12, 1675-1680 (2002).
- Dourlen, P., et al. Drosophila fatty acid transport protein regulates rhodopsin-1 metabolism and is required for photoreceptor neuron survival. PLoS Genet. 8, e1002833 (2012).
- Mlodzik, M., Hiromi, Y., Weber, U., Goodman, C. S., Rubin, G. M. The Drosophila seven-up gene, a member of the steroid receptor gene superfamily, controls photoreceptor cell fates. Cell. 60, 211-224 (1990).
- Gaul, U., Chang, H., Choi, T., Karim, F., Rubin, G. M. Identification of ras targets using a genetic approach. Ciba Found Symp. 176, 85-92 (1993).
- Wassarman, D. A., Therrien, M., Rubin, G. M. The Ras signaling pathway in Drosophila. Curr Opin Genet Dev. 5, 44-50 (1995).
- Janody, F., et al. A mosaic genetic screen reveals distinct roles for trithorax and polycomb group genes in Drosophila eye development. Genetics. 166, 187-200 (2004).
- Legent, K., Steinhauer, J., Richard, M., Treisman, J. E. A screen for X-linked mutations affecting Drosophila photoreceptor differentiation identifies Casein kinase 1alpha as an essential negative regulator of wingless signaling. Genetics. 190, 601-616 (2012).
- Newsome, T. P., Asling, B., Dickson, B. J. Analysis of Drosophila photoreceptor axon guidance in eye-specific mosaics. Development. 127, 851-860 (2000).
- Berger, J., et al. Systematic identification of genes that regulate neuronal wiring in the Drosophila visual system. PLoS Genet. 4, e1000085 (2008).
- Bayat, V., et al. Mutations in the mitochondrial methionyl-tRNA synthetase cause a neurodegenerative phenotype in flies and a recessive ataxia (ARSAL) in humans. PLoS Biol. 10, e1001288 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon