È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Procedura per l'Adaptive Laboratorio evoluzione dei microrganismi Utilizzando un chemostat
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui, vi presentiamo un protocollo per ottenere adattativa evoluzione laboratorio di microrganismi in condizioni che utilizzano la cultura chemostat. Inoltre, l'analisi genomica del ceppo evoluto è discusso.
Abstract
Natural evolution involves genetic diversity such as environmental change and a selection between small populations. Adaptive laboratory evolution (ALE) refers to the experimental situation in which evolution is observed using living organisms under controlled conditions and stressors; organisms are thereby artificially forced to make evolutionary changes. Microorganisms are subject to a variety of stressors in the environment and are capable of regulating certain stress-inducible proteins to increase their chances of survival. Naturally occurring spontaneous mutations bring about changes in a microorganism's genome that affect its chances of survival. Long-term exposure to chemostat culture provokes an accumulation of spontaneous mutations and renders the most adaptable strain dominant. Compared to the colony transfer and serial transfer methods, chemostat culture entails the highest number of cell divisions and, therefore, the highest number of diverse populations. Although chemostat culture for ALE requires more complicated culture devices, it is less labor intensive once the operation begins. Comparative genomic and transcriptome analyses of the adapted strain provide evolutionary clues as to how the stressors contribute to mutations that overcome the stress. The goal of the current paper is to bring about accelerated evolution of microorganisms under controlled laboratory conditions.
Introduzione
I microrganismi possono sopravvivere e di adattarsi ad ambienti diversi. Sotto stress grave, l'adattamento può avvenire attraverso l'acquisizione di fenotipi benefici da mutazioni genomiche casuali e la successiva selezione positiva 1-3. Pertanto, le cellule microbiche in grado di adattarsi cambiando metabolica o reti di regolazione per una crescita ottimale, che viene definito "l'evoluzione adattativa". Recenti importanti tendenze microbici, come focolai di superbatteri e la comparsa di ceppi microbici resistenti, sono strettamente correlate a adattivo evoluzione in condizioni di stress. In condizioni di laboratorio definite, siamo in grado di studiare i meccanismi di evoluzione molecolare e anche controllare la direzione di evoluzione microbica per varie applicazioni. Diversamente organismi multicellulari, organismi unicellulari sono adatti all'evoluzione laboratorio adattativa (ALE) per i seguenti motivi: si rigenerano rapidamente, mantengono grandi popolazioni, ed è facile per creare e mantenere homambienti ogeneous. In combinazione con i recenti progressi nelle tecniche di sequenziamento del DNA e le tecnologie high-throughput, ALE permette l'osservazione diretta dei cambiamenti genomiche che portano a modifiche normative sistemici. dinamiche mutazionale e una diversità della popolazione sono anche osservabili. Strategie di ingegneria genetica può essere determinata dall'analisi di ceppi ALE 4,5.
Cultura chemostat è un metodo utilizzato per ottenere cellule allo stato stazionario e aumentare la produttività nei processi di fermentazione 6. terreno fresco viene aggiunto e brodo di coltura è raccolta durante il processo (quest'ultimo include medio e biomasse). Cultura chemostat A lungo termine, però, cambia la produttività dello stato stazionario della cultura e provoca l'accumulo di mutazioni spontanee e selezione durante la coltura (Figura 1a). Sotto varie pressioni selettive (fattori di stress), l'accumulo di mutazioni è migliorata. Un graduale aumento dello stress in un lungo periodo chemostat prevede una selezione continua di mutazioni che funzionano contro i fattori di stress indicati, quali temperatura, pH, pressione osmotica, nutrienti fame, ossidazione, prodotti tossici, ecc transfer Colony da un supporto solido e trasferimento seriale da un mezzo liquido (ripetuto cultura batch) permettono anche ai ricercatori di ottenere microrganismi evoluti (Figura 1B e 1C). Anche se la cultura chemostat richiede metodi complicati, la piscina della diversità (numero di repliche e dimensione della popolazione) è superiore a quello ottenuto con il trasferimento della colonia e le tecniche di trasferimento seriale. L'esposizione di stress stabile alle singole celle e diminuito variazione di stato cellulare durante la coltura chemostat (stato stazionario) sono altri benefici di ALE rispetto alle tecniche di coltura a base di batch. ALE indotta da stress di Escherichia coli sottoposti a condizioni succinato alto è introdotto in questo articolo.
iles / ftp_upload / 54446 / 54446fig1.jpg "/>
Figura 1: Metodi di evoluzione adattativa di laboratorio (A) chemostat;. (B) trasferimento seriale; (C) trasferimento colonia. Le figure principali illustrare il concetto di metodi per ALE, e le figure in basso mostrano il numero di cellule che sono cresciuti durante ALE. Cliccate qui per vedere una versione più grande di questa figura.
Protocollo
1. Preparazione dell'apparecchiatura
- Ottenere un barattolo chemostat (150-250 ml) o una beuta (250 ml) contenente una luce di ingresso ed una luce di uscita. Collegare le porte con tubo in silicone che consente portate di 10-100 ml / h. Facoltativamente, utilizzare una presa d'aria, una porta di uscita dell'aria, e le porte di ingresso e uscita a temperatura controllata.
- Ottenere un dispositivo adatto per il barattolo chemostat che prevede l'agitazione e la temperatura di controllo (o utilizzare un incubatore agitazione rotante).
- Ottenere due pompe peristaltiche per erogare mezzo fresco e raccogliere la cultura.
- Ottenere un vaso serbatoio (10-20 L) contenente una porta di uscita media e una porta di ingresso dell'aria.
- Ottenere tubo in silicone adatto per il tasso di diluizione (ad esempio, ID 0,8 mm campo di portata 0,06-36 ml / min; L / S 13 tubi).
2. Preparazione medio e sterilizzazione
- Media iniziale
- Sciogliere 0,3 g di glucosio, 0,08 g NH 4Cl, 0,05 g di NaCl, 0,75 g di Na 2 HPO 4 · 2H 2 O, e 0,3 g KH 2 PO 4 in 90 ml di acqua distillata (DW) in un vaso chemostato.
- Sigillare il vaso chemostat insieme con il tubo con fascette. Non sigillare la presa d'aria.
- Sterilizzare il vaso chemostat in autoclave a 121 ° C per 15 min. Dopo la sterilizzazione, conservare il barattolo chemostat a temperatura ambiente.
- Sciogliere 0,02 g MgSO 4 · 7H 2 O, 0,01 g CaCl 2, e 0,1 mg di tiamina in 10 ml DW (soluzione A).
- soluzione Filter A utilizzando una siringa e un filtro a siringa pre-sterilizzato (un filtro da 0,45 micron pori).
- Aggiungere la soluzione A filtra al barattolo chemostat.
- Lo stress medio
- Sciogliere 30 g di glucosio, 8 g di NH 4 Cl, 5 g di NaCl, 75 g di Na 2 HPO 4 · 2H 2 O, 30 g KH 2 PO 4, e 300 g di disodio succinato esaidrato (Na 2 · succinato · 6H 2 O; il fattore di stress utilizzato in questo esperimento) in 9,9 L DW in un barattolo serbatoio.
- Sigillare il vaso serbatoio con il tubo con fascette. Non sigillare la presa d'aria.
- Sterilizzare il vaso serbatoio in autoclave a 121 ° C per 15 min. Dopo la sterilizzazione, conservare il vaso a temperatura ambiente.
- Sciogliere 2 g MgSO 4 · 7H 2 O, 1 g CaCl 2, e 10 mg di tiamina in 100 ml DW (soluzione A).
- Filtro soluzione A con una siringa e un filtro a siringa pre-sterilizzato (un filtro da 0,45 micron pori).
- Aggiungere la soluzione A filtra al barattolo serbatoio.
- Asetticamente collegare il tubo di silicone sterilizzato al barattolo serbatoio e collegare le pompe peristaltiche.
- -Stress elevato Media
- Preparare il mezzo come nella sezione 2.2, ma con una maggiore concentrazione di stress (cioè, 3-5 g / L elevato nell'adattamento succinato).
Nota: Questo protocollo è per l'adattamento ad una sollecitazione thin possono essere consegnati attraverso il mezzo. Nel caso di stress fisici quali temperatura, agitazione, o l'illuminazione, la coltivazione dovrebbe essere progettato di conseguenza.
- Preparare il mezzo come nella sezione 2.2, ma con una maggiore concentrazione di stress (cioè, 3-5 g / L elevato nell'adattamento succinato).
3. La coltivazione iniziale
- Inoculare una singola colonia di wild-tipo E. coli in una provetta da 15 ml contenente 4 ml di terreno iniziale.
- Incubare la provetta in un incubatore agitazione per 12 ore a 37 ° C e 220 rpm.
- Trasferire asetticamente 1 ml di precoltura al barattolo chemostat.
- Incubare il vaso chemostat, prevedendo aerazione (aria 50 ml / min) e l'agitazione (200 rpm), a 37 ° C per 6 ore.
4. Lo stress Adattamento
- Asetticamente collegare l'estremità del tubo in silicone dalle pompe al vaso chemostat.
- Avviare la pompa di scarico (10 ml / h o superiore) e raccogliere la cultura.
Nota: La coltura deve essere in fase esponenziale, tipicamente 4-8 ore dopo la coltivazione iniziale. - Check la densità ottica (600 nm) della cultura dal tubo di uscita.
- Avviare la pompa di aspirazione (10 ml / h, corrispondente ad un tasso di diluizione 0,1 hr -1).
- Controllare la densità ottica della coltura a 600 nm dalla presa tubo ogni 24 ore.
- Azionare il chemostat per 96 ore (fatturato 9,6 volte) o più. Se la densità ottica è stabile, scambiare il serbatoio contenente il mezzo high-stress. Se la densità ottica è inferiore a 0.2, fermare la pompa di aspirazione alimentazione per 6 ore. Riavviare la pompa di aspirazione e verificare che la densità ottica è di 0,2.
- Gradualmente aumentare la concentrazione del fattore stressante cambiando ad un serbatoio contenente una concentrazione di stress elevato.
- Prelevare campioni della cultura adattato ogni volta che raggiunge un traguardo (ad esempio, un ceppo atto a 100 g / L di stress succinato), e memorizzare per ulteriori analisi genomica.
- Per la conservazione del campione, mescolare il campione di coltura (0,5 ml) con un sterilizzato 80% glicerolo solution (0,5 ml) e conservarlo a -80 ° C.
Nota: Se il microrganismo acquisisce la capacità di degradare il fattore di stress durante la procedura ALE, la concentrazione di stress nella giara di fermentazione non è la stessa di quella nel serbatoio fresco.
5. Single-colonia isolamento del ceppo stress-adattato
- Preparare medio piastra di agar (1,6% agar) contenente lo stesso stress e alla stessa concentrazione del mezzo.
- Piatto cultura uscita (0,1 ml) dalla chemostat, e incubare a 37 ° C per 16 ore.
- Raccogliere singole colonie dalla piastra con uno stuzzicadenti sterile e inoculare in provette da 15 ml contenenti la stessa stress e alla stessa concentrazione media come nella chemostato e incubare per 6 ore.
- Trasferire 1 ml di brodo di coltura in un pallone da 250 ml Erlenmeyer contenente 50 ml di terreno. Harvest 0,5 ml di brodo di coltura ogni 1 ora, e misurare la densità ottica a 600 nm. Confrontare il tasso di crescita del strai adattaton a quella del ceppo selvatico dato il fattore di stress.
Risultati
Per l'alta-succinato adattamento stress, la wild-type E. coli W3110 ceppo è stato coltivato in un chemostato a D = 0,1 hr -1 per 270 giorni (figura 2).
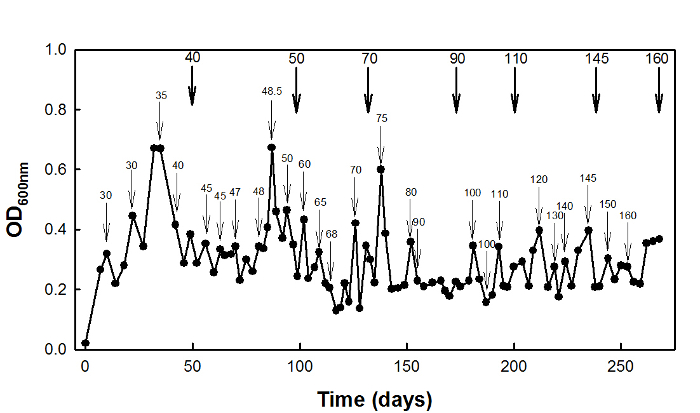
Figura 2: High-succinato sforzo di adattamento di E. coli W3110 con la cultura chemostat. frecce sottili indicano l'ora in cui è stata aumentata la concentrazione di ...
Discussione
I microrganismi sono in grado di adattarsi a quasi tutti gli ambienti a causa del loro rapido tasso di crescita e la diversità genetica. Adaptive evoluzione di laboratorio permette di evolvere microrganismi in condizioni progettati, che fornisce un modo di selezionare gli organismi individuali harboring mutazioni spontanee che sono favorevoli nelle condizioni date.
La tecnica chemostat è più robusto per realizzare evoluzione artificialmente guidato di tecniche di trasferimento per i segue...
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
This study was financially supported by the Korean Ministry of Science, ICT and Future Planning (Intelligent Synthetic Biology Center program 2012M3A6A8054887). P. Kim was supported by a fellowship from the Catholic University of Korea (2015).
Materiali
| Name | Company | Catalog Number | Comments |
| Mini-chemostat fermentor | Biotron Inc. | - | manufactured by special order |
| silicon tubing | Cole-Parmer | Masterflex L/S 13 | tubing size can be varied depending on the dilution rate and the size of fermentor jar. |
| reservoir jar | Bellco | Media storage bottle | 20 L |
| chemicals | Sigma-Aldrich | - | reagent grade |
| glucose | Sigma-Aldrich | G5767 | ACS reagent |
| NH4Cl | Sigma-Aldrich | A9434 | for molecular biology, suitable for cell culture, ≥99.5% |
| NaCl | Sigma-Aldrich | 746398 | ACS reagent, ≥99% |
| Na2HPO4·2H2O | Sigma-Aldrich | 4272 | 98.5-101% |
| KH2PO4 | Sigma-Aldrich | 795488 | ACS reagent, ≥99% |
| MgSO4·7H2O | Sigma-Aldrich | 230391 | ACS reagent, ≥98% |
| CaCl2 | Sigma-Aldrich | 793639 | ACS reagent, ≥96% |
| thiamine·HCl | Sigma-Aldrich | T4625 | reagent grade, ≥99% |
| Na2·succinate·6H2O | Sigma-Aldrich | S2378 | ReagentPlus, ≥99% |
Riferimenti
- Rando, O. J., Verstrepen, K. J. Timescales of genetic and epigenetic inheritance. Cell. 128, 655-668 (2007).
- Kim, H. J., et al. Short-term differential adaptation to anaerobic stress via genomic mutations by Escherichia coli strains K-12 and B lacking alcohol dehydrogenase. Front Microbiol. 5, 476 (2014).
- Mendizabal, I., Keller, T. E., Zeng, J., Yi, S. V. Epigenetics and evolution. Integr Comp Biol. 54, 31-42 (2014).
- Lee, J. Y., Seo, J., Kim, E. S., Lee, H. S., Kim, P. Adaptive evolution of Corynebacterium glutamicum resistant to oxidative stress and its global gene expression profiling. Biotechnol Lett. 35, 709-717 (2013).
- Lee, J. Y., et al. Artificial oxidative stress-tolerant Corynebacterium glutamicum. AMB Express. 4, 15 (2014).
- Narang, A. The steady states of microbial growth on mixtures of substitutable substrates in a chemostat. J Theor Biol. 190, 241-261 (1998).
- Kwon, Y. D., Kim, S., Lee, S. Y., Kim, P. Long-term continuous adaptation of Escherichia coli to high succinate stress and transcriptome analysis of the tolerant strain. J Biosci Bioeng. 111, 26-30 (2011).
- Barrick, J. E., Lenski, R. E. Genome dynamics during experimental evolution. Nat Rev Genet. 14, 827-839 (2013).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25, 2078-2079 (2009).
- McKenna, A., et al. The Genome Analysis Toolkit: a MapReduce framework for analyzing next-generation DNA sequencing data. Genome Res. 20, 1297-1303 (2010).
- Deatherage, D. E., Barrick, J. E. Identification of mutations in laboratory-evolved microbes from next-generation sequencing data using breseq. Methods Mol Biol. 1151, 165-188 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon