Method Article
Dissezione e macchiatura immunofluorescente del corpo di fungo e del fotoricettore neuroni nel cervello adulto Drosophila melanogaster
In questo articolo
Riepilogo
Questo protocollo descrive la dissezione e immunostaining del adulto Drosophila melanogaster tessuti del cervello. In particolare, questo protocollo evidenzia l'uso di Drosophila fungo del fotoricettore e corpo neuroni come sottoinsiemi di un neurone di esempio che possono essere utilizzati con precisione per scoprire i principi generali alla base di molti aspetti dello sviluppo neuronale.
Abstract
Sviluppo del sistema nervoso comporta una serie sequenziale di eventi che sono coordinati da diverse vie di segnalazione e reti di regolazione. Molte delle proteine coinvolte in queste vie sono evolutivamente conservate tra mammiferi e altri eucarioti, come moscerino della frutta Drosophila melanogaster, suggerendo l'esistano di principi organizzativi simili durante lo sviluppo di questi organismi. D'importanza, Drosophila è stato usato estesamente per identificare i meccanismi cellulari e molecolari che regolano processi che sono necessari nei mammiferi, compresa la neurogenesi, differenziazione, orientamento assonale e sinaptogenesi. Mosche sono anche stati utilizzati con successo per modellare una varietà di malattie umane neurodevelopmental. Qui descriviamo un protocollo per la microdissezione passo dopo passo, fissazione e immunofluorescente localizzazione delle proteine all'interno del cervello di Drosophila adulto. Questo protocollo si concentra su due popolazioni neuronali di esempio, i neuroni del corpo del fungo e fotorecettori retinici e include passaggi facoltativi per la traccia di neuroni individuali corpo fungo usando analisi di mosaico con una tecnica di reprensibile Cell Marker (MARCM). Dati di esempio da cervelli wild-type e mutanti sono mostrati insieme con una breve descrizione di un criterio di punteggio per orientamento axonal difetti. Mentre questo protocollo mette in evidenza due anticorpi ben consolidati per indagare la morfologia del corpo del fungo e i neuroni del fotoricettore, altre regioni del cervello di Drosophila e la localizzazione delle proteine all'interno di altre regioni del cervello può anche essere studiato usando questo protocollo.
Introduzione
Anche se il sistema nervoso di Drosophila è inferiore a quello degli esseri umani e roditori, la sua complessità fornisce un modello potente, socievole per comprendere meglio i suoi omologhi dei vertebrati. In molti casi, i genomi di vertebrati e mosche codificano proteine molto simili che dettano i meccanismi di sviluppo del sistema nervoso. Infatti, molti dei geni necessari per lo sviluppo neuronale nei vertebrati sono ortologhi in mosche, compresi quelli coinvolti in vie di segnalazione che controllo patterning, neurogenesi e orientamento axonal1,2,3 ,4,5. Ad esempio, netrina è un ligando necessario per una guida assonale in mammiferi e d. melanogaster6,7,8. Mentre netrina era originariamente isolato da pulcino embrionali cervello tessuto6, studi successivi hanno rivelato che netrina svolge un ruolo conservato durante lo sviluppo del sistema nervoso centrale embrionale (CNS) in Drosophila8. Altri studi hanno utilizzato schermi genetici nell'embrionale della drosofila CNS per identificare conservati ligandi e recettori necessari per path-finding in Drosophila e vertebrati9.
Mentre la Mosca embrionale CNS è stata utilizzata ampiamente in passato per identificare ligandi, i recettori e proteine di segnalazione intracellulare necessarie per orientamento axonal8,9, lavoro recente ha studiato i modi in cui molte delle Queste proteine controllano anche decisioni di path-finding durante le fasi successive di sviluppo. In particolare, indagine del corpo del fungo (MB) e sviluppo di neurone dei fotorecettori retinici (Figura 1) ha fornito la comprensione nei meccanismi che path-finding controllo, formazione della sinapsi, potatura dell'assone e molti altri aspetti di un neurone sviluppo10,11,12,13,14,15,16,17. I neuroni del fotoricettore connettersi retina Vola in regioni del cervello adulto chiamato lamina e midollo e sono critico per l'inoltro di informazioni visive al cervello (Recensito da18,19), mentre i neuroni del corpo del fungo sono centralmente situato nel cervello volo e sono necessari per apprendimento e memoria20,21. Sia i neuroni del fotoricettore e i neuroni intrinseci dei corpi del fungo, chiamati cellule di Kenyon, utilizzano meccanismi di evolutivamente conservati Consiglio axonal diffusibile e contatto-dipendente per trovare loro obiettivi post-sinaptiche. Oltre ad essere visibile in mosche adulte, del fotoricettore e MB neuroni possono anche essere direttamente visualizzati in larve e pupe con anticorpi o geni reporter22,23,24,25. La possibilità di visualizzare facilmente questi due insiemi di neuroni in diversi momenti dello sviluppo ha promosso il loro uso come modelli superbe per molti aspetti dello sviluppo neuronale.
Oltre ad essere utilizzato come un modello per capire i meccanismi dello sviluppo normale del sistema nervoso, studi recenti hanno dimostrato che le mosche possono anche servire come modelli accurati di un'ampia varietà di malattie umane, compreso la sindrome di X Fragile (FXS)26 , Disabilità intellettuale (ID)27,28,29,30,31e gli altri32. Ad esempio, per studiare la funzione molecolare di ZC3H14, un gene recentemente collegato alla disabilità intellettuale umano, abbiamo creato un modello fly di ID utilizzando un allele nullo della Mosca dell'ortologo di ZC3H14, chiamato Nab230. Manca Nab2 mosche hanno memoria gravi menomazioni ed estesa code di poli (a), ricapitolare quello che si osserva in pazienti umani o paziente derivato cellula linee33,34. D'importanza, mosche carente Nab2 anche visualizzare severa del cervello morfologia difetti nei loro corpi fungo adulto34, simile a ciò che si osserva nelle mosche privi del gene di sindrome FXS, FMR135. Così, mosche possono servire come un importante organismo modello per studiare lo sviluppo normale del cervello e malattie che interrompono.
Infine, l'accessibilità dei metodi ad alto rendimento per monitorare il comportamento, combinato con la vasta gamma di strumenti genetici disponibili, rendono Drosophila organismo modello di scelta per l'identificazione di regioni del cervello che controllano i comportamenti complessi, come apprendimento e memoria, sonno, corteggiamento, sete e altri36,37,38,39. Uno strumento particolarmente utile che è al centro di «strumenti» di un genetista volare è il sistema GAL4/UAS (Figura 2). Questo sistema40,41 utilizza espressione specifica del tessuto dell'attivatore trascrizionale Gal4 per aumentare l'espressione di geni o transgeni a valle di una sequenza di attivazione a Monte (UAS). Le modifiche di questo sistema hanno permesso che i ricercatori, ad esempio, controllare con precisione l'eccitabilità dei neuroni specifici42,43, iperesprimere o colpo specifici geni di interesse44, 45, l'analisi del calcio in tempo reale dinamica nel vivo46ed esprimere geni reporter per contrassegnare un neurone lignaggi41. La combinazione del sistema GAL4/UAS con ricombinazione mitotica anche permesso la creazione di analisi del mosaico con un indicatore di cella reprensibile (MARCM) sistema12,47. MARCM è stato usato estesamente nell'analisi di singoli neuroni per identificare i componenti di segnalazione cellulari necessari per orientamento axonal12,47. Anche se queste e altre tecniche hanno fornito un numero di preziose informazioni sui meccanismi cellulari necessari per il funzionamento del sistema nervoso, la maggior parte richiedono che il cervello di Drosophila in primo luogo essere sezionato; rimozione accurata del cervello è necessario per mantenere corrette cervello modelli di morfologia e connessione tra regioni del cervello. Il seguente protocollo utilizza fungo neuroni del fotoricettore e corpo comepopolazioni neuronali di esempio come si guida l'utente attraverso la dissezione e cervello di Drosophila immunofluorescenza colorazione dell'adulto.
Protocollo
1. genetica di drosophila melanogaster e procedure facoltative di Shock termico
- Una volta mosche sono stati attraversati e progenie F1 hanno covato, ottenere femmine e/o i maschi del genotipo appropriato. A seconda della regione di cervello indagata, mosche dovrebbe essere raccolti ogni giorno e separati per sesso in modo che si possono distinguere più facilmente modelli di età-dipendente e/o sessualmente dimorphic della connettività cerebrale.
Nota: Facoltativo: se mosche vengono utilizzati per l'analisi di mosaico con un indicatore di cella reprensibile (MARCM) analisi 12 , 47, embrioni, larve o pupe dovrebbero essere scioccato a 37 ° C per 30-45 minuti a calore indurre la ricombinazione mitotica. Per impostare la destinazione specifiche regioni dei corpi fungo per analisi MARCM, scossa di calore dovrebbe essere cronometrato in base alla pianificazione determinata da 12 e descritta nella Figura 5 B. Se utilizzando MARCM per indagare regioni del cervello diverso dai corpi del fungo, esperimenti pilota dovrebbero essere eseguiti per determinare lo stadio ottimo per essere calore-scioccato. Mentre la procedura riportata di seguito vengono scritti riguardo alla obtecta Vola, Farata adulti possono anche essere sezionati utilizzando la procedura una volta tolto il caso pupal.
2. Dissezione stazione preparazione
- posizione lo stereomicroscopio e sorgente luminosa con attaccato il goosenecks ottica fibra su un grande benchtop. Per favorire i movimenti di mano ferma e ridurre la mano " agitare " mentre di dissezione, è essenziale che uno spazio adeguato di resto di mano ed il braccio è disponibile intorno al microscopio. Assicurarsi che ci sia circa 8-10 pollici su entrambi i lati del microscopio e 4-6 pollici tra la base del microscopio e bordo del banco.
- Riempimento 2 o 3 pozzi di un vetro piatto 9-pozzo o 3-pozzetti con 1,0 mL di tampone PTN (0.1 M sodio fosfato tampone pH 7.2, tensioattivo non ionico di 0,1%, vedi tabella materiali per componenti di buffer completo) e posto accanto alla stazione della dissezione su ghiaccio. Appena dissecati cervelli saranno trasferiti a questo piatto e conservati fino alla fase di fissazione.
- Nota: se la formazione immagine diretta è necessaria, buffer di dissezione dovrebbe essere 1x tampone Phosphate Buffered Saline (PBS) o HL3 48. Se è richiesta la localizzazione intracellulare della proteina, PBS utilizzabile anche come un buffer alternativo per la dissezione e la fissazione. A seguito di fissazione, permeabilizzazione delle membrane cellulari quindi va eseguito con PTN lavaggi contenenti 0,3% o 0,1% detergente nonionico.
- Se PBS viene usato per dissezioni e la fissazione, puntali per pipette dovrebbero essere risciacquata almeno una volta con un buffer contenente detersivo (ad esempio PTN) per impedire che cervelli attaccare verso le punte di pipetta di plastica durante il trasferimento per microcentrifuga.
- Usando un vuoto 35 mm vetro o plastica di Petri, costruire un piatto di dissezione contenente un elastomero di silicone. Brevemente, miscelare i componenti in elastomero secondo il produttore ' indicazioni s, versare in piatti 35 mm e lasciarlo polimerizzare durante la notte su una superficie piana. Elastomero contenente piatti di dissezione dovrebbe essere utilizzato per proteggere le punte fini della dissezione del forcipe, che possono essere facilmente danneggiato se avviene il contatto tra la pinza e una superficie più dura, come ad esempio un piatto di vetro. Acquistiamo inoltre regolarmente piatti rivestito di silicone commercialmente disponibile da rivenditori online. Per aumentare il contrasto durante le dissezioni, sono particolarmente utili in silicone elastomero dissezione piatti contenenti carbone inattivato (e così colorata di nero).
3. Procedura di dissezione del cervello adulto
- Anesthetize 3-5 giorni adulti vecchio d. melanogaster con CO 2 o utilizzando ghiaccio. Se si utilizza il ghiaccio, posto nel flaconcino contenente mosche upside-down (spina fine giù) in un secchio di ghiaccio per ~ 5 min ponendo il flacone in ghiaccio capovolta impedisce Vola da diventando presentata nel cibo. Una volta che sono stati anestetizzati mosche, posto le mosche su una seduta di pad o capsula di Petri metallo fredda nel ghiaccio o in CO 2 emettitori volare pad. Se la dissezione cervelli per analizzare la neurodegenerazione, mosche più grandi possono anche essere utilizzati.
- Mettere una piccola quantità (150-200 µ l) di PTN al centro del piatto la dissezione utilizzando una pipetta o una pipetta p200 per creare un " bolla " di PTN. Posizionare il piatto di dissezione sotto lo stereomicroscopio e regolare l'illuminazione e concentrarsi in modo che la bolla di PTN riempie il campo visivo ed è uniformemente illuminata.
- Manipolare mosche in modo che siano " pancia fino " (cioè, lato ventrale verso l'alto) mentre giaceva sul metallo o CO 2 pad.
- Utilizzando una coppia di pinze #5, afferrare l'addome di una Mosca per essere sezionato e tenere in mano al volo, immergere completamente nel PTN sul piatto da dissezione.
Nota: Per il resto del protocollo, tutte le fasi devono essere eseguite mentre la testa è sommerso nel PTN. - Utilizzando un secondo paio di pinze #5, afferrare la base della proboscide Vola e tirare le due paia di pinze a pezzi per staccare la testa di Mosca dal corpo. Scartare l'addome e torace. Durante questo passaggio, è fondamentale che la testa non è rilasciata e ha permesso a galleggiare sulla superficie del PTN. Una volta che la testa è mobile, può essere molto difficile da afferrare nuovamente senza schiacciare il cervello.
Nota: Utilizzando questo metodo, le connessioni tra il cervello e il midollo ventrale del nervo sono reciso. Se sono necessarie connessioni intatte tra queste regioni del SNC, un protocollo di dissezione alternativo, ad esempio 49 , 50, dovrebbe essere seguito. Se la proboscide si stacca dalla testa Vola prima la testa viene rimossa, ci sarà un buco dove era la proboscide. In questo caso, afferrare la testa di Mosca al bordo del foro vicino a un occhio. Poi rimuovere la testina utilizzando una quantità moderata di forza tirando le due paia di pinze distanti l'uno da altra. Occasionalmente, quando la testa viene rimossa dal corpo, i resti del cavo dell'intestino e/o ventrale del nervo attaccato alla testa e deve essere rimosso prima di continuare la dissezione. - Mentre una coppia di pinze afferra la proboscide, la seconda coppia dovrebbe afferrare il bordo mediale dell'occhio proprio vola. Tirare lentamente, il forcipe distanti l'uno da altra. Questo passaggio deve essere eseguito con una piccola quantità di forza laterale costante. Le pinze si muovono lentamente distanti l'uno da altra, la proboscide dovrebbe tirare lontano dalla testa e creare un foro centrale nella cuticola testa. Scartare la proboscide con la prima coppia di pinze senza rilasciare la parte mediale dell'occhio destro dalla seconda coppia.
Nota: L'adulto d. melanogaster cervello è nella caudale (cioè, posteriore/rear) regione della testa vola. Così, afferrare quella regione della testa dovrebbe essere evitato. Idealmente, solo la rostrale (cioè, frontale) porzione della testa vicino alla retina mediale dovrebbe essere colto direttamente con il forcipe. Il cervello e la trachea associato dovrebbe essere visibile attraverso il foro centrale nella cuticola. In questo momento, qualsiasi thread filante bianco della trachea che sporge dal buco può essere rimosso ed eliminato. - Con la seconda coppia di pinze, afferrare il bordo mediale della retina sinistra (al bordo del foro centrale nella cuticola testa). Per rimuovere le retine e cuticola associata, tirare lentamente il forcipe lontani uno da altro ad un angolo di 180 °. Come la retina si dissocia dal lobo ottico sottostante, si dovrebbe sentire una leggera diminuzione in tensione. Procedere lentamente per evitare di strappare il lobo ottico.
Nota: Separare il forcipe troppo rapidamente durante questo passaggio potrebbe causare la lacerazione dei the ottica lobo o rottura delle strutture del corpo del fungo. Occasionalmente, la cuticola verrà rimossa ma pezzi della retina rimarrà attaccati al lobo ottico. Se i corpi fungo di imaging, non è completamente necessario rimuovere l'intera retina. Tuttavia, analisi di altre regioni del cervello (come stimolo di neuroni retinici dei lobi del cervello ottica) possono richiedere la retina per essere completamente rimosso come descritto da 48 , 51.
Nota: Come la retina lentamente è separata dal lobo ottico sottostante del cervello, il lobo ottico dovrebbe essere osservabile come una struttura opaca bianca ricoperta di bianco, filante trachea. Una volta tolta una retina, può essere scartato. Quando si analizza il pathfinding dei neuroni retinici, si dovrebbe prestare attenzione particolare durante questa fase per evitare danni al lobo ottico. Protocollo addizionale focalizzata sulla dissezione e formazione immagine diretta dei neuroni del fotoricettore è inoltre disponibili 48. - Ora, rimuovere con attenzione tanto della trachea visibile possibile. La trachea può già contenere o poi riempire con aria, causando il cervello per galleggiare e, potenzialmente, essere perso durante i successivi passaggi di immunostaining. Per rimuovere la trachea, prenderlo fuori il cervello utilizzando una coppia molto forte di forcipe #5.
- Rimuovere la retina rimanente e la cuticola circostante utilizzando entrambe le paia di pinze per afferrare la regione mediale della retina sinistra vola. Attentamente e strappare la retina a metà per rimuovere i pezzi della retina e della cuticola. In alcuni casi, rimuovere la cuticola rimanente senza schiacciare il cervello si rivela particolarmente impegnativi. In questi casi, abbiamo trovato che i restanti fili del cavo ventrale del nervo invece possono essere afferrati da un paio di pinze mentre l'altra coppia di pinze è utilizzata per rimuovere con cautela l'ultimo della cuticola.
- Utilizzando una pipetta p200, spostare dissecati cervelli in un pozzetto del piatto 9 o 3 pozzetti contenenti PTN. Cervelli dello stesso genotipo dovrebbero essere raggruppati nello stesso bene e tenuti su ghiaccio. Tessuto cerebrale dovrebbe essere risolto entro un'ora della dissezione. Cervello può essere fissato in piccoli lotti e se è necessario un più grande numero dei cervelli in pool. Nella maggior parte dei casi, un ricercatore esperto può solitamente sezionare un cervello e trasferirlo nella vaschetta di vetro in circa 3-5 min
4. Fissazione e procedura di macchiatura immunofluorescente
- utilizzando una pipetta p200, transfer cervelli sezionati dal 9-pozzetto a un tubo del microcentrifuge 0,5 mL riempito con 0,5 mL di paraformaldeide 4% diluito nel PTN. Cervelli di almeno 10-15 dello stesso genotipo possono essere combinati in una microcentrifuga. Tutti i passaggi rimanenti (fino al montaggio dei cervelli sui vetrini) sono stati completati in provette per microcentrifuga da 0,5 ml.
Attenzione: Paraformaldeide (PFA) deve essere gestita in una cappa aspirante. PFA rifiuti dovrebbero essere salvato e smaltiti correttamente. 20% paraformaldeide acquistato in fiale di vetro può essere aliquotati in microcentrifuga e conservati a-20 ° C fino a quando necessario. - All'interno di una cappa aspirante, incubare cervelli in paraformaldeide al 4% per 20 min con velocità lenta a dondolo a temperatura ambiente.
- a seguito di fissazione, consentire cervelli a depositarsi sul fondo del tubo del microcentrifuge da gravità.
Nota: In alcuni casi, il cervello può aderire al lato del tubo del microcentrifuge. In questo caso, è di solito utile per ruotare lateralmente il tubo tra il dito indice e il pollice o per toccare molto delicatamente il tubo in panchina per promuovere affondamento dei cervelli. - Rimuovi fissativo utilizzando un p1000 Pipettare ed eseguire due " veloce " lava con 500 µ l di PTN, permettendo i cervelli a depositarsi sul fondo del tubo del microcentrifuge tra i lavaggi. Durante questi lavaggi rapidi, una volta che tutti i cervelli si sono insediati per gravità verso il basso del tubo del microcentrifuge, il PTN possono essere immediatamente scambiati per buffer di fresco; nessun tempo di lavaggio supplementare è necessario.
Nota: In genere, lasciando il buffer supplementare nel tubo è preferibile rischiare la rimozione del cervello. L'esame attento della punta della pipetta è spesso richiesto per garantire che nessun cervello è stati erroneamente rimossi dal tubo. Se cervelli sono stati accidentalmente pipettato nella punta, dispensare loro nuovamente dentro il tubo del microcentrifuge, attendere per i cervelli di stabilirsi e quindi continuare a rimuovere qualsiasi PTN supplementare che rimane. - Dopo l'ultimo lavaggio rapido, è necessario utilizzare una pipetta p1000 per eseguire tre " lungo " lava: aggiungere 500 µ l di PTN e lavaggio per 20 min a temperatura ambiente su un bilanciere/nutator. Tutte le future " lungo " lavaggi dovrebbero essere per 20 min.
Nota: Seguendo questi lavaggi, fissata cervelli può essere conservato durante la notte a 4 ° C nel PTN. - Rimuovere l'ultimo lavaggio usando una pipetta p1000 e incubare cervelli su un bilanciere o nutator a temperatura ambiente in 0,5 mL di soluzione [PTN + 5% di siero di capra normale (NGS)] per almeno 30 min a temperatura ambiente bloccante.
- Anticorpi secondari capra verrà utilizzato nei passaggi successivi protocollo. Se verranno utilizzati anticorpi secondari da un'altra specie, normale del siero da tale specie (anziché NGS) deve essere utilizzato nelle soluzioni di blocco e l'anticorpo.
- Utilizzando una pipetta p1000, rimuovere la soluzione bloccante e aggiungere l'anticorpo primario diluito in PTN (PTN + 5% NGS + anticorpo primario diluito). Quando si utilizza un anticorpo primario per la prima volta, la diluizione ottima di tale anticorpo dovrebbe essere determinata empiricamente.
Nota: Per visualizzare il corpo del fungo neuroni, sono in genere utilizzati anticorpi riconoscendo Fas2. Questi anticorpi sono disponibili da Banca inerente allo sviluppo di ibridomi studi (DSHB) come anticorpo 1 4 e dovrebbe essere diluito 01:20 in PTN + 5% NGS.
Nota: Per visualizzare i neuroni del fotoricettore, anticorpi che riconoscono chaoptin sono tipicamente utilizzati. Gli anticorpi Chaoptin sono disponibili da DSHB come anticorpo 24B10 e dovrebbe essere diluito 01:20 in PTN + 5% NGS.
Nota: Il processo di fissazione in genere Elimina fluorescenza da proteina fluorescente come proteina fluorescente verde (GFP). Di conseguenza, quando si utilizza MARCM per analizzare axonal Guida di singoli neuroni di MB, usare un anticorpo riconoscendo GFP. - Incubare cervelli in soluzione di anticorpo primario su un bilanciere/nutator per 2-3 notti a 4 ° C.
- Dopo incubazione con gli anticorpi primari, consentire cervelli depositano alla parte inferiore del tubo del microcentrifuge e quindi rimuovere la soluzione di anticorpo primario.
- Utilizzando una pipetta p1000, eseguire 2 " veloce " lavaggi e 3 " lungo " 20 min si lava con 0,5 mL di PTN come descritto sopra in punti 4.4 e 4.5, attentamente permettendo cervelli per depositarsi per gravità verso il basso del tubo del microcentrifuge tra ogni nello stato di Washington
- Cervelli Incubare per 3 ore a temperatura ambiente con anticorpi secondari con etichetta fluorescente appropriati. Gli anticorpi secondari sono solitamente diluiti in 0,5 mL di PTN + 5% NGS ad una concentrazione di 1: 200.
Nota: Una volta aggiunti fluorescenti anticorpi secondari, cervello dovrebbe essere tenuto all'oscuro per il resto dell'esperimento.
Nota: In caso residuali resti di fluorescenza della GFP, durante l'esecuzione di analisi MARCM si consiglia di utilizzare un secondario anticorpo marcato con un fluoroforo avendo lunghezze d'onda di eccitazione/emissione simili come GFP (ad es., Fluoroscein isothyocyanate (FITC) o Alexa488). - Dopo incubazione anticorpo secondario, consentire cervelli depositano alla parte inferiore del tubo del microcentrifuge e rimuovere la soluzione di anticorpo secondario.
- Eseguire 2 " veloce " lavaggi e 3 " lungo " 20 min si lava con 0,5 mL di PTN come descritto sopra in punti 4.4 e 4.5, attentamente permettendo cervelli per depositarsi sul fondo del tubo del microcentrifuge tra ogni nello stato di Washington
- Seguendo il terzo " lungo " 20 min lavaggio, uso una pipetta p200 per rimuovere quanto più buffer possibile. Mezzo di montaggio
- aggiungere 75 µ l di antislittamento fluorescente ai cervelli. Pipetta cervelli e mezzo di montaggio in una volta il puntale della pipetta per mescolare. Non capovolgere la provetta dal cervello potrebbe diventare bloccato sul tappo o i lati del tubo del microcentrifuge.
Nota: A seguito di sospensione del cervello per mezzo di montaggio, tubi possono essere avvolto in carta stagnola per rallentare il fluoroforo tempra e conservati a 4 ° C durante la notte. Se necessario, cervelli possono essere conservati per diversi giorni a 4 ° C, ma idealmente dovrebbe essere montati sui vetrini presto.
5. Adulto di d. melanogaster cervelli su vetrini da microscopio e la formazione immagine di montaggio
- costruire un " ponte " diapositiva. Posizione due " base " coprioggetti circa 1 cm di distanza su una diapositiva di carica positiva. Assicurarsi che il lato carico positivamente della diapositiva è rivolto verso l'alto. Aderire i coprioggetti alla diapositiva con smalto per unghie, come mostrato in Figura 3 A. È solitamente utile sigillare i tre bordi esterni di ogni base coprioggetto con smalto per unghie per garantire mezzi di montaggio non stoppino sotto questi vetrini base. Lasciare asciugare completamente la smalto per unghie (10-15 min) prima di procedere.
- Mettere il vetrino sotto lo stereomicroscopio e dispensare la soluzione di supporti di montaggio contenente i cervelli sezionati nello spazio tra i due vetrini coprioggetti. Per fornire più contrasto, è utile manovrare le luci a collo di cigno, affinché siano paralleli con la parte superiore del banco.
- Rimuovi extra montaggio supporti dalla diapositiva utilizzando una pipetta, facendo attenzione a non pipettare i cervelli fuori della diapositiva. Mezzi di montaggio supplementare via di
- stoppino. In tal modo il cervello per essere posizionato più precisamente durante il passaggio successivo.
- Usando un paio di pinze e lo stereomicroscopio, posizionare i cervelli sulla diapositiva in un modello di griglia con lobi antennali fronte up.
- Inserire un vetrino coprioggetto (il " ponte ") sopra i cervelli ( Figura 3 A). Utilizzare smalto per unghie per sigillare i lati dello slip cover ponte in cui vengono a contatto del " base " vetrini coprioggetto.
- Lentamente utilizzando una pipetta p200, riempire la cavità di centro sotto il ponte con mezzi di montaggio fresco ( Figura 3 B). Depositare una goccia alla volta sul bordo aperto del centro coprivetrino e lasciare che i supporti di montaggio per stoppino sotto il vetrino coprioggetto ponte centro. Continuare fino a quando tutta la cavità è riempita con montanti, poi sigillare la parte superiore e inferiore con smalto per unghie chiare.
- Una volta asciutto lo smalto per unghie, le diapositive di immagini immediatamente o conservare in una scatola a tenuta di luce stretto scivolo a -20 ° C.
- Entro una settimana, immagine cervelli utilizzando un microscopio confocale a scansione laser con laser di eccitazione e filtrare cubi appropriati agli anticorpi fluorescenti secondari selezionati. Immagini di Z-stack dei neuroni del corpo del fungo sono in genere ottenuti utilizzando X 20 o 40 X obiettivi. Imaging dei neuroni dei fotorecettori retinici possono richiedere maggiore ingrandimento.
Risultati
Il metodo sopra descritto permette di visualizzare affidabili e riproducibili di praticamente qualsiasi regione del cervello di Drosophila adulto. Qui ci siamo concentrati sul fungo organismi e i neuroni del fotoricettore, ma altri studi hanno utilizzato metodi simili per visualizzare regioni cerebrali come la pars intercerebralis52, orologio neuroni53,54e antenne Lobo proiezione neuroni55, tra molti altri. D'importanza, questa tecnica può essere utilizzata per visualizzare sia le strutture del cervello intero nonché singoli neuroni all'interno di quelle strutture utilizzando tecniche quali MARCM12,47. Figura 4, Figura 5e Figura 6 mostrano diversi tipi di dati da corpi del fungo e del fotoricettore neuroni nel cervello adulto che può essere generato usando questa tecnica di dissezione e immunostaining.
In primo luogo, il metodo sopra descritto può essere utilizzato per visualizzare direttamente la morfologia del corpo del fungo utilizzando gli anticorpi che riconoscono 2 Fasciculin (Fas2) o geni reporter espresse nel corpo del fungo neuroni34. Come mostrato in Figura 4A e Figura 4B, Fas2 anticorpi possono essere utilizzati per visualizzare il α, β e (in misura minore) lobi γ dei corpi fungo in adulto Drosophila cervelli. Fas2 è una proteina di adesione cellula-cellula necessaria per fascicolazione del neurone ed è espresso ad alti livelli nei α e β lobi23,56,57, rendendolo un indicatore affidabile e consolidato di questi funghi neuroni del corpo. In particolare, il corpo dell'elissoide a forma circolare, situato nella regione centrale del cervello adulto possa anche essere visualizzato usando questo anticorpo (Figura 4A).
Difetti di proteine di axonal guida spesso causano di corpo del fungo in modo incompleto penetranti fenotipi mutanti15,34,35. Pertanto, nella maggior parte dei casi, diversi dozzina cervelli dovrebbero essere imaged e analizzati. Ad esempio, gli assoni di lobo fungo corpo β di Nab2 null mosche impropriamente attraversano la linea mediana del cervello. Questo fenotipo di "fusione" lobo "crossing over" o β è osservato tipicamente in ~ 80% di adulto Nab2 null Vola, ma è principalmente assente dal selvaggio-tipo controlli e può essere classificata come lieve, moderata o completa la fusione34. La "fusione" dei lobi β attraverso il midline risultati dalla proiezione controlaterale non corretta degli assoni nell'opposto emisfero del cervello. Come mostrato nella Figura 4C e dettagliate in34,35, leggera fusione si riferisce ai lobi β collegati da un "filo sottile di fibre Fas2-positive," mentre moderata fusione si riferisce a più lobi sostanzialmente collegato β che mostrano uno spessore leggermente in diminuzione del lobo al midline. Fusione completa (o "estreme") si riferisce ai lobi β che sono completamente collegati e non mostrano nessuna diminuzione di spessore del lobo o Fas2 colorazione al midline. La portata della fusione di corpo del fungo β del lobo può essere quantificata e visualizzata come dimostrato a 34,35 , o come una tabella indicante la percentuale di cervelli mostrando ogni tipo di difetto di morfologia.
Oltre alla colorazione con gli anticorpi Fas2, il MARCM tecnica12,47,48 utilizzabile anche per visualizzare le decisioni di orientamento dell'assone di singoli neuroni GFP+ entro i lobi di corpo del fungo. MARCM utilizza ricombinazione mitotica durante lo sviluppo per creare singoli neuroni o clonale gruppi di neuroni, contrassegnati con GFP (Figura 5A). MARCM fornisce un modo per generare un piccolo numero di neuroni che sono totalmente privi di una proteina all'interno di una Mosca altrimenti eterozigotica. Come tale, questa tecnica è stato particolarmente utile per analizzare le funzioni di guida assonale di proteine che sono anche essenziali per la vitalità generale organismal12,47. Neuroni null omozigotici contrassegnati con GFP possono essere confrontati direttamente per controllare i neuroni contrassegnati con GFP in una priorità bassa genetica di selvaggio-tipo. Inoltre, a seconda di quando lo sviluppo di larve o pupe sono scioccato di calore, diverse classi di corpo del fungo neuroni possono essere mirati (Figura 5B).
Un esempio del tipo di dati generati utilizzando questa tecnica è illustrato nella Figura 5C, dove il selvaggio-tipo (cioè, controllo) MARCM cloni vengono generati come esempio. Per generare i singoli neuroni del corpo del fungo GFP+ illustrati nella Figura 5C, lo sviluppo di F1 larve inizialmente sono state alloggiate a 25 ° C. Circa il 5-6 giorni dopo la schiusa larvale (ALH), pupe erano calore scioccato per 30 min a 37 ° C e poi tornò a 25 ° C fino a sfarfallamento. Seguito da cova adulto, cervelli sono stati sezionati nel PTN, corretti e contemporaneamente incubati con anticorpi che riconoscono Fas2 (4 1, diluito 01:20 in PTN) e GFP (diluito 1: 500 nel PTN). Cervelli erano quindi incubati con anticorpi secondari e montati su slitte come descritto sopra. Le cellule GFP+ sono state identificate e imaging utilizzando un microscopio confocale a scansione laser e proiezioni di massima intensità sono state create utilizzando ImageJ. Come dimostrato nella Figura 5C, controllo GFP+ neuroni generati nei lobi α e β colocalize con Fas2 e terminare prima di contattare la linea mediana che separa i due emisferi del cervello di Drosophila . Un singolo assone progetti anteriormente, si biforca e quindi entrambi progetti dorsalmente e medialmente rispetto alla forma il α e β lobi. Parecchi studi precedenti hanno usato questa tecnica per indagare se determinate proteine sono necessari per studiare la divisione cellulare in modo autonomo per molti aspetti della assonogenesi, tra cui estensione, path-finding, ramificazione, o potatura10, 11,12,13,14,15,16,17,34.
Oltre i corpi del fungo, le decisioni di pathfinding axonal dei neuroni dei fotorecettori retinici (R-cellule) possono essere visualizzate anche utilizzando il metodo di dissezione descritto sopra (e anche del de di metodo di dissezionecome descritto nel riferimento48). Ogni ommatidi all'interno dell'occhio di Mosca contiene 8 neuroni del fotoricettore che possono essere classificati in tre gruppi (Figura 1): cellule di R1-R6, che proiettano assoni alla lamina superficiale del cervello lobo ottico; Cellule R7, che proiettano assoni al livello più profondo di M6 del midollo; e le cellule di R8, che proiettano assoni per lo strato intermedio di M3 del midollo19,58. Il modello di proiezione di ciascuna classe di fotorecettore è stato studiato estesamente e insieme questi neuroni sono stati usati con successo come modello per comprendere le vie di segnalazione coinvolte nell'assone Guida19,59. D'importanza, tutti gli assoni dei fotorecettori sono facilmente visualizzabili nei cervelli di Drosophila dissecati immunostaining dell'adulto, pupa, o tessuti larvali usando un anticorpo che riconosce chaoptin, una cella in superficie glicoproteina24, 60 , 61. geni reporter che esprimono GFP o β-galattosidasi in ogni tipo di cellula R (R1-R6, R7 e R8 cellule) inoltre sono state costruite e può essere utilizzati per visualizzare ogni classe di fotorecettore62. Poiché ogni tipo di cellula R termina in un livello diverso del lobo ottico in via di sviluppo, questi giornalisti hanno permesso un confronto dettagliato fra le vie di segnalazione necessarie per ogni tipo di cellula cercare correttamente la sua destinazione. Nella Figura 6è riportato un esempio dei modelli di espressione prodotta da due di questi geni reporter in combinazione con la localizzazione di chaoptin (che può essere utilizzato come marcatore di tutti i neuroni del fotoricettore). Per visualizzare i fotorecettori R7, cervelli contenente il gene reporter R7 specifiche Rhodopsin4-LacZ erano dissecati e macchiati con gli anticorpi chaoptin e β-galattosidasi. Rhodopsin 4 (Rh4) è espressa in particolare in un sottoinsieme di R7 cellule63; il promotore Rh4 pertanto può essere utilizzato per guidare l'espressione del gene reporter in solo queste cellule. Come previsto, le cellule R7 terminano nello strato più profondo M6 del midollo (indicato con una linea gialla tratteggiata nella figura 6). Allo stesso modo, i cervelli esprimendo la β-galattosidasi da promotore del Rhodopsin 6 (Rh6), che si esprime in particolare nel R8 fotorecettori63, sono stati sezionati e immunostained utilizzando anticorpi che riconoscono chaoptin e Β-galattosidasi. Come illustrato nella Figura 6, R8 fotorecettori terminato a livello R3 dellamidollo. Anche se non mostrato qui, Rh1-LacZ utilizzabile anche per visualizzare la terminazione di R1-R6 fotorecettori nella lamina.
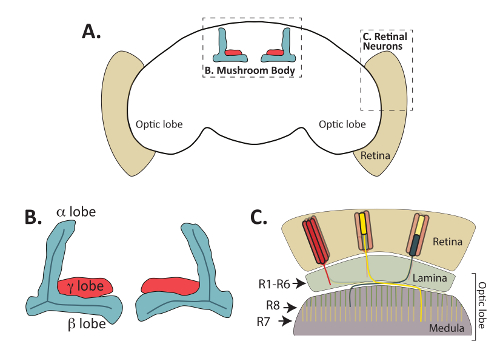
Figura 1: L'adulto cervello di Drosophila melanogaster è costituito da regioni funzionalmente distinte ma interconnessione. (A), un contorno dell'adulto di d. melanogaster cervello è mostrato, evidenziando i corpi del fungo in posizione centrale e i neuroni dei fotorecettori retinici periferici. (B) diagramma allargata dei fasci assone corpo fungo adulto (chiamato anche lobi) che vengono riconosciuti dagli anticorpi Fas2. I neuroni intrinseci dei corpi del fungo, chiamati cellule di Kenyon, proiettano assoni anteriormente da dorsalmente trovano corpi cellulari (corpi cellulari omessi da questo diagramma) e formano strutture distinte del lobo. Nel cervello adulto, la forma γ lobi (mostrato in rosso) da un singolo fascio mediale degli assoni, mentre la dorsale α e β mediale lobi (mostrato in blu) forma da un singolo assone che moltiplica durante il processo di path-finding. Α ' / β' neuroni sviluppano lobi axonal che parzialmente si sovrappongono con quelli dei neuroni α/β, ma non sono riconosciuti dagli anticorpi Fas2 e vengono omessi da questo diagramma. (C) neuroni retinici sono fondamentali per l'inoltro di informazioni visive dalla retina al lobo ottico. Progetto di assoni da corpi cellulari situato nella retina (beige) e formare connessioni con le cellule bersaglio post-sinaptico nella lamina o midollo. R1-R6 del fotoricettore cellule (mostrate in rosso) formare specifiche connessioni con le cellule nello strato esterno del lobo ottico, chiamato la lamina. R7 del fotoricettore cellule sinapsi (giallo) con target nello strato M6 del midollo, mentre le cellule R8 (mostrate in verde) proiettano assoni al livello M3 leggermente più superficiale del midollo. Parte C adattato da permesso da Macmillan Publishers Ltd: Nature Neuroscience (riferimento64, copyright (2011)). Clicca qui per visualizzare una versione più grande di questa figura.
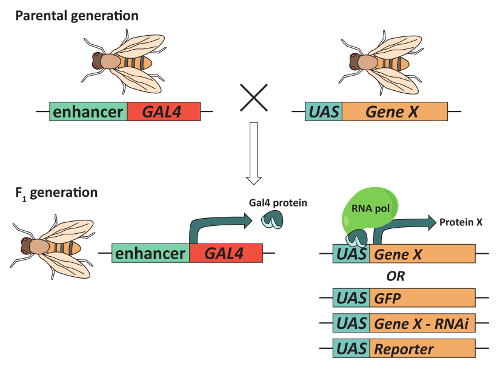
Figura 2: Sistema GAL4/UAS the può essere utilizzato per l'espressione genica mirata. Per ottenere le mosche che esprimono un gene di interesse ("Gene X") in un modello specifico del tessuto, mosche devono contenere un transgene esprimenti la proteina attivatore trascrizionale Gal4 sotto il controllo di un enhancer specifico del tessuto e un transgene contenente il DNA Gal4 associazione di sequenza (chiamato la sequenza di attivazione a Monte o UAS) adiacente al Gene X. in genere, questa combinazione si ottiene accoppiamento parentale mosche contenenti ognuno un transgene e selezionando per la progenie di1 F che contengono entrambi. Nella F1 generazione risultante, Gal4 sarà espresso e legheranno l'UAS per attivare la trascrizione del Gene X in un modo specifico di tessuto. D'importanza, diverse contenenti UAS transgeni utilizzabile in combinazione con la stessa GAL4 "driver". Ad esempio, si può abbinare un transgene GFP espressa, un transgene che abbatte i espressione di un gene di interesse utilizzando l'interferenza del RNA o un transgene UAS che produce un reporter UAS-contenente un transgene esprimendo Gal4 nei corpi del fungo (MBs). Clicca qui per visualizzare una versione più grande di questa figura.
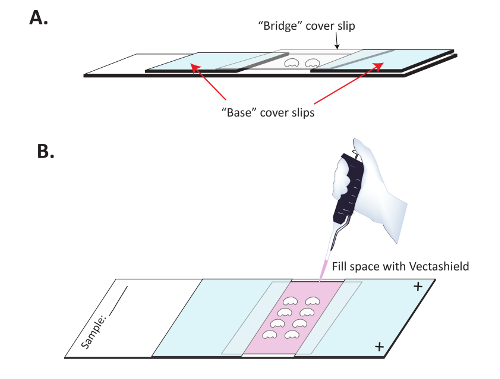
Figura 3 : Cervelli in preparazione per l'imaging di immunofluorescenza di montaggio. (A) cervelli sono montati su vetrini SuperFrost Plus con una copertura del ponte per impedire l'appiattimento dei cervelli. Due vetrini "base" sono rispettate una diapositiva caricati positivamente Superfrost Plus con smalto per unghie trasparente e dissecati cervelli vengono poi poste sulla diapositiva tra di loro. Un "ponte" chiaro vetrini coprioggetto è posizionato sopra i cervelli e rispettato la base vetrini coprioggetto. (B) una volta asciugato lo smalto per unghie, Vectashield lentamente è dispensato sotto il ponte per preservare la fluorescenza dell'anticorpo secondario. Smalto per unghie trasparente viene quindi utilizzato per sigillare la parte superiore e inferiore del vetrino coprioggetto "ponte". Clicca qui per visualizzare una versione più grande di questa figura.
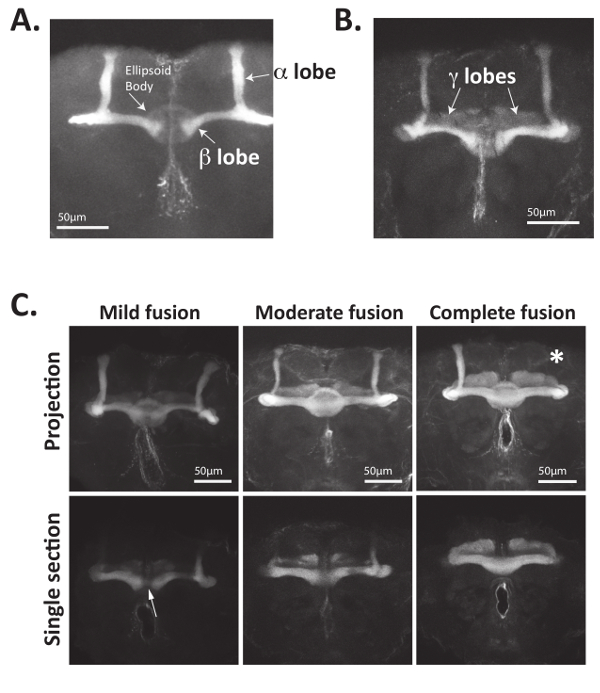
Figura 4: Gli assoni di corpo del fungo possono essere visualizzati chiaramente usando gli anticorpi che riconoscono Fas2. (A) adulto Drosophila cervelli sono stati sezionati, corretti e incubati con anticorpi che riconoscono Fas2. Gli anticorpi primari sono stati riconosciuti utilizzando anticorpi secondari capra-anti-mouse Alexa488-accoppiato e cervelli sono stati montati sui vetrini caricati positivamente e imaging utilizzando un microscopio confocale di scansione laser. Bisymmetrically trova fungo somatocita corpi si estendono gli assoni anteriormente, biforcano e formano fasci di assoni (chiamati anche lobi). Gli assoni che formano la proiezione di dorsalmente α lobi e medialmente proiettando i lobi β express Fas2. Criticamente, lobi β del selvaggio-tipo mosche terminano prima la linea mediana del cervello. Il corpo dell'elissoide centrale possa anche essere visualizzato utilizzando 4 1 anticorpo. (B) medialmente proiettando γ lobo assoni del corpo del fungo della drosofila adulto anche esprimere Fas2 e possono essere visualizzati utilizzando 4 1 anticorpo. In genere, l'espressione di Fas2 in assoni lobo γ è inferiore a quello dei lobi α e β. (C) il corpo del fungo assoni dei cervelli Nab2-null (genotipo: Nab2ex3/Nab2ex3) mis-progetto controlaterale e spesso mancano i lobi. Nab2-null cervelli erano sezionati, corretti e macchiati con 4 1 anticorpo per visualizzare il corpo del fungo lobi α, β e γ. Proiezioni di intensità massima dello stack Z anche come singole sezioni ottiche concentrate sulla regione del midline sono mostrate. Mentre gli assoni corpo fungo selvaggio-tipo β lobo raramente attraversano la linea mediana del cervello, Nab2 null cervelli hanno diverse quantità di mis-proiezione attraverso il midline nell'emisfero cerebrale controlaterale, conseguente lobi β "fuso". Come definito in riferimenti34,35, fusione lieve si intende < lobi β con solo parecchi "fili" degli assoni attraversano la linea mediana, moderata fusion si riferisce a situazioni dove i neuroni del lobo β positivo Fas2 attraversano il lobo del midline ma β di cervello Larghezza al midline è diminuita e fusione completa si riferisce a situazioni dove non c'è nessuna riduzione di spessore lobo β come i lobi attraversano la linea mediana del cervello. Poiché il corpo dell'elissoide esprime anche Fas2, sezioni ottiche mostrando il midline sono spesso più utile nel visualizzare fusione lobo β. In particolare, lobi mancanti sono osservati anche frequentemente (indicato qui con l'asterisco bianco). Dati in parte C viene utilizzati nella quantificazione dei fenotipi di corpo del fungo da riferimento34, con permesso. Clicca qui per visualizzare una versione più grande di questa figura.
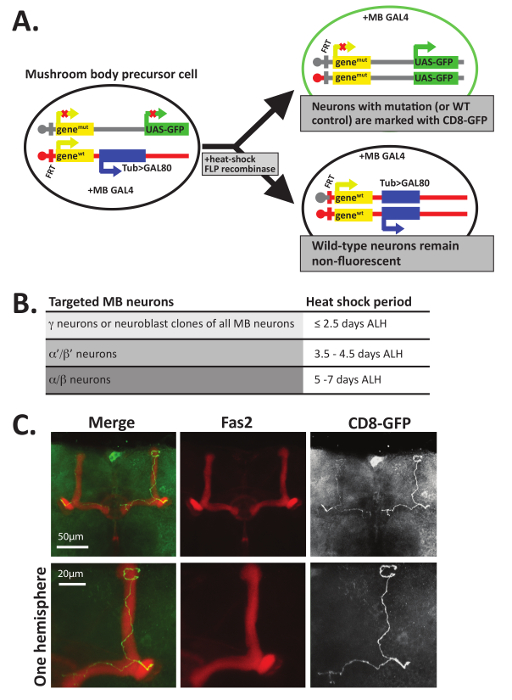
Figura 5 : MARCM può essere utilizzato per visualizzare gli assoni dei neuroni singoli. (A) analisi di mosaico con un'indicatore di cella reprensibile (MARCM) usi FLP ricombinasi-mediata ricombinazione mitotica presso siti FRT per creare due cellule figlie distinte. In questo esempio, tutte le celle contengono una proteina di GAL4 corpo-specific del fungo su un cromosoma separato. A seguito di ricombinazione mitotica durante la divisione cellulare, una cellula figlia (in alto) esprime GFP (o la membrana associato CD8-GFP) e contiene due alleli del mutante di un gene di interesse, mentre l'altra cellula figlia (in basso) eredita due alleli di wild type (WT) e una transgene esprimenti la proteina Gal80 utilizzando un promotore di tubulina. Gal80 inibisce Gal4, così le cellule che producono Gal80 saranno non fluorescenti e dovrebbero essere sia eterozigote o omozigote wild type. Solo quelle cellule che sono GFP+ conterrà due copie dell'allele del mutante. Si noti che viene visualizzato solo uno dei diversi metodi per la generazione di cellule GFP+ che contengono anche due alleli del mutante di un gene di interesse; 12,45,56 per altri esempi, vedere. (B) poiché lo sviluppo dei neuroni del corpo del fungo comincia con i neuroni γ, allora α'/ β' neuroni e termina con i neuroni α/β, ogni classe del neurone può essere selettivamente mirati e visualizzate da scioccante in diversi momenti dello sviluppo di calore. Come descritto in 12, una scossa di calore di 40 min a 37 ° C che si verifica ≤ 2.5 giorni dopo la schiusa larvale (ALH) riguarderà specificamente γ; neuroni, mentre scossa di calore che si verifica di 3,5-4,5 giorni ALH (nella fase larvale L3 tardiva) si rivolgerà α'/ β' neuroni e una tale o di scossa di calore ccurs tra 5-7 giorni ALH (durante lo sviluppo pupa) saranno destinati a neuroni α/β. Individuali degli assoni di selvaggio-tipo (C) sono stati visualizzati utilizzando MARCM. Pupe di selvaggio-tipo ~ 5-6 giorno vecchio (genotipo: hsFLP, UAS-CD8-GFP; FRT82B, UAS-CD8-GFP/FRT82B, vasca > Gal80; OK107-Gal4/+) rimasti scioccati a 37 ° C per 40 min per indurre la ricombinazione mitotica calore. Cervelli sono stati sezionati, corretti e macchiati con anticorpi che riconoscono la GFP (1: 500) e Fas2 (01:20). Cervelli erano quindi incubati con anticorpi secondari fluorescente contrassegnati e visualizzati da microscopia confocal. In questi dati di esempio da un genotipo di "controllo", le cellule positive generate utilizzando MARCM GFP sono selvaggio-tipo e invece di contenere due alleli di genemut , contengono due alleli di geneWT . Lobi β corpo del fungo (visualizzati usando anticorpi che riconoscono Fas2) terminare prima di raggiungere la linea mediana del cervello; Sono presenti anche i neuroni del lobo α. Per visualizzare maggiori dettagli, una zoomata in vista di un singolo emisfero è mostrato nella riga inferiore delle immagini. Parte C è stata adattata con il permesso di riferimento34. Clicca qui per visualizzare una versione più grande di questa figura.
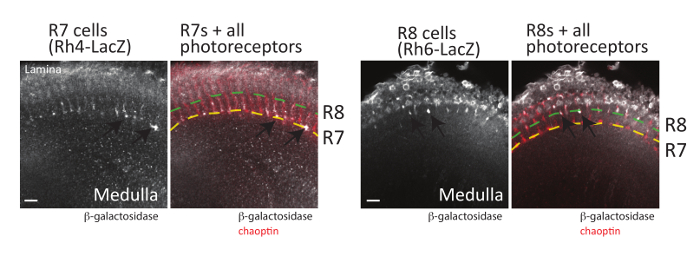
Figura 6 : Gli assoni dei fotorecettori possono anche essere visualizzati. Adulto cervelli esprime β-galattosidasi in fotorecettori R7 (a sinistra, utilizzando Rh4-LacZ) o R8 fotorecettori (a destra, utilizzando Rh6-LacZ) sono stati sezionati, corretti e incubate con anticorpi che riconoscono la β-galattosidasi o chaoptin. Cervelli erano quindi incubati con anticorpi secondari fluorescente contrassegnati e visualizzati da microscopia confocale a scansione laser. Singole sezioni ottiche sono indicate da ogni cervello. Sulla sinistra, possono essere visto diversi assoni dei fotorecettori di R7 (frecce) che termina con lo strato più profondo di M6 del midollo (indicato dalla linea tratteggiata gialla). Sulla destra, R8 assoni dei fotorecettori (frecce) terminano nello strato esterno M3 del midollo (indicato dalla linea verde tratteggiata). Poiché R7 fotorecettori express entrambi Rh3 o Rh4 e R8 fotorecettori express Rh5 o Rh663, tutti i fotorecettori R7 e R8 non possono essere visualizzati utilizzando un singolo gene reporter LacZ . Barre di scala = 10 µm. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Il metodo di dissezione e visualizzazione sopra descritto può essere utilizzato in un'ampia varietà di immunostaining e vive applicazioni di imaging. Ci hanno delineato un protocollo di immunostaining generale e hanno messo in evidenza un modo in cui MARCM può essere utilizzato per visualizzare axonal morfologia dei neuroni individuali del corpo del fungo. Inoltre, queste procedure generali possono essere utilizzate anche per altre regioni del cervello in fissa di imaging o adulto appena dissecata cervelli48,65. Formazione immagine diretta può fornire un approccio più efficiente quando si analizzano i modelli di espressione di enhancer trap, geni reporter o MiMIC linee66, ad esempio. Per evitare che cattura artefatti dalla morte delle cellule durante la formazione immagine diretta di GFP in tessuto unfixed, cervelli dovrebbero essere sezionati in 1x Phosphate Buffered Saline (PBS) o HL3 media48, montati su slitte di ponte in 1X PBS (o HL3) e fotografati in meno di 15-20 min cura Occorre inoltre di rimuovere tanto trachea come possibile dal cervello dissecato prima di formazione immagine, poiché può interferire con la visualizzazione della fluorescenza di GFP.
Mentre colorazione condizioni per valutare la morfologia dei neuroni del fotoricettore e corpo del fungo sono relativamente ben consolidata24,61,67, la localizzazione di una proteina specifica di interesse in queste cellule tipi possono richiedere ottimizzazione e risoluzione dei problemi completa. In particolare, diversi passaggi critici, tra cui la diluizione dell'anticorpo, blocco buffer componente concentrazione e la fissazione, dovrebbero essere ottimizzati per generare risultati più riproducibili e affidabili. In primo luogo, deve essere determinata la concentrazione ottimale di anticorpo primario. Anche se commercialmente disponibili anticorpi hanno spesso una diluizione consigliata (e spesso inizialmente iniziamo utilizzando quella concentrazione), questi valori sono raramente determinati empiricamente sui tessuti della drosofila . Di conseguenza, testare una serie di diluizioni di anticorpo primario che gamma sopra e sotto suggerimento iniziale del produttore spesso si traduce in colorazione più specifica. Mentre la diluizione di anticorpo primario può avere un effetto significativo sulla macchiatura di precisione e deve essere determinata con precisione, i cervelli di tempo vengono incubati in anticorpo primario contenente soluzioni possono variare notevolmente con scarso effetto sul risultato complessivo. Ad esempio, abbiamo osservato risultati simili quando incubando sezionati cervelli con l'anticorpo primario di anticorpo Fas2 per due, tre o anche quattro giorni a 4 ° C. Anche se si consiglia di almeno due notti, abbiamo anche incubate cervelli in soluzione di anticorpo primario per una sola notte e ottenuto macchiatura riproducibile. D'importanza, limitando la quantità di tempo cervelli sono incubati nell'anticorpo secondario a 3 h a temperatura ambiente (o una notte a 4 ° C) aiuta il legame non specifico limite di anticorpi secondari al tessuto cerebrale.
In secondo luogo, può essere necessario utilizzare altri reagenti "blocchi" per eliminare il segnale di fondo indesiderato. Le opzioni includono l'aumento della concentrazione di NGS a ~ 10%, l'aggiunta di 1-10% dell'albumina di siero bovino (BSA) per le soluzioni Anticorpo bloccante sia primario e secondario, o pre-adsorbimento di anticorpi primari durante la notte a 4 ° C con embrioni della drosofila (Vedi riferimento68, punto 2.9, passaggio 3).
Mentre abbiamo scritto il protocollo sopra utilizzando PTN del buffer suggerito per tutti i fissazione, lavaggio e fasi di incubazione dell'anticorpo, fissazione del tessuto in altri buffer (ad esempio PLP e PEM, elencati nella Tabella materiali) può provocare profonde differenze in potenza del segnale. Ad esempio, studi precedenti hanno dimostrato che la ciclina E è inosservabile in dischi immaginali larvale quando i tessuti sono stati corretti nel tradizionale paraformaldeide 4%, diluito in PBS, ma è chiaramente visibile quando i tessuti sono stati corretti nel buffer di PLP (Ken Moberg, personali comunicazione e riferimento69). Oltre alle modifiche nel buffer componenti, alterare il tempo e la temperatura per il fissaggio del tessuto possa anche significativamente influenzare immunofluorescente macchiatura e può essere determinate empiricamente se necessario. In generale, tempo di fissazione deve essere sufficientemente lungo per consentire sufficiente reticolazione delle componenti cellulari e manutenzione a lungo termine della morfologia cellulare globale pur essendo limitato abbastanza per impedire over-reticolazione e "seppellire" degli epitopi della proteina. Pertanto, quando inizialmente ottimizzazione delle condizioni di colorazione per un anticorpo primario appena acquisita, di solito limitare il tempo di fissazione a circa 20 min e spesso risolvono i tessuti alle temperature più fredde.
Diversi controlli dovrebbero essere incluso in qualsiasi esperimento di immunofluorescenza per studiare la specificità degli anticorpi primari e secondari così come l'effetto di transgeni/genetico sul fenotipo osservato. Per accertare se l'anticorpo primario utilizzato specificamente riconosce la proteina d'interesse, tessuto da mosche carenti questa proteina e/o tessuto che overexpressing la proteina dovrebbe essere incluso come controlli. L'aggiunta di proteine in eccesso di antigene purificato può anche essere incluso per determinare se l'anticorpo primario riconosce altri epitopi nel cervello volo. Infine, qualsiasi segnale fluorescente presente quando gli anticorpi primari vengono omessi rappresenta il livello di legame non specifico dagli anticorpi secondari selezionati.
Diversi controlli importanti dovrebbero essere inclusi anche per valutare il contributo del background genetico o la presenza di transgeni per intensità di colorazione o morfologia neuronale. Ad esempio, le mosche utilizzate nell'esperimento MARCM nella Figura 5 contengono transgeni multipli (hsFLP, UAS-CD8-GFP, FRT82B, vasca > GAL80 e OK107-GAL4), ognuno dei quali dovrebbe essere analizzato separatamente per gli effetti sulla morfologia del corpo del fungo. A una morfologia molto minimo, fungo corpo di mosche che contiene OK107-GAL4 sia UAS-CD8-GFP dovrebbero essere analizzati. Può anche essere necessario valutare l'effetto di scossa di calore e produzione ricombinasi Flp sullo sviluppo del corpo del fungo analizzando Vola contenente sia hsFLP e FRT82B. Anche se MARCM può fornire la comprensione significativa in se una data proteina controlla autonomamente axonal di orientamento, il numero di controlli necessari per rendere con precisione questa conclusione può essere un limite minore di questa tecnica. Meno complicati esperimenti, controlli simili sono altresì applicabili. Ad esempio, prendere un esperimento dove il sistema GAL4/UAS viene utilizzato in combinazione con un transgene di RNAi per atterramento espressione di una proteina di interesse in tutti i neuroni. In questo esperimento, almeno due transgeni sarà presente: un pan-neuronale GAL4 driver, ad esempio di Elav-GAL4 e il transgene UAS-RNAi . La morfologia dei neuroni del corpo del fungo in mosche contenenti ciascuno di questi transgeni da solo dovrebbe essere studiata oltre alla condizione sperimentale dove mosche harbor sia transgeni al volo stesso.
Infine, per determinare se la proteina di interesse è espresso nei neuroni di corpo del fungo è solitamente necessario cervelli co-macchia con gli anticorpi al Fas2. In alternativa, le proteine fluorescenti come GFP, RFP o la membrana associato CD8-GFP possono anche essere espressa utilizzando il sistema GAL4/UAS e utilizzato in combinazione con anticorpi che riconoscono la proteina di interesse. Poiché Fas2 riconosce solo gli assoni scanditi che formano i lobi α e β del corpo del fungo, l'uso di GAL4-driven, membrana-limita CD8-GFP è stato particolarmente utile per la marcatura di neuroni del corpo del fungo.
Difetti di axonal guida spesso non sono 100% con liquidi penetranti e anche cervello dello stesso genotipo potrebbe mostrare qualche variabilità. Pertanto, quando l'analisi appena generato alleli mutanti per difetti nel corpo del fungo o path-finding del fotoricettore, dovrebbe essere utilizzata una combinazione di alleli differenti e approcci. Maggior parte degli studi nella letteratura utilizzare vari metodi per indagare se una proteina svolge un ruolo delle cellule-autonoma nel controllo Guida assonale: i) analisi di pathfinding difetti in omozigoti mosche null (preferibilmente utilizzando diversi nugli alleli ll), ii) analisi di pathfinding difetti nelle mosche manca la proteina di interesse solo in neuroni (di solito tramite RNAi), iii) analisi MARCM dei difetti di path-finding e iv) esperimenti dove il gene è riespresso nei neuroni null omozigotica di salvataggio mosche. Idealmente, dovrebbero essere analizzati diversi dozzina cervelli al genotipo per difetti nella morfologia neuronale.
Sebbene il protocollo qui descritto si concentra principalmente su immunofluorescente localizzazione delle proteine all'interno del tessuto fisso, diverse applicazioni future di questa tecnica sono state sviluppate immagine dal vivo al tessuto di cervello. Una volta cervelli vengono sezionati (solitamente in terreno di coltura delle cellule), quindi possono essere coltivate per diversi giorni a 25 ° C. Questi metodi di coltura ex vivo stanno sviluppandi per studiare una vasta gamma di processi biologici, quali attività di proteina che promuove la rigenerazione assonale dopo lesione70,71, intracellulare segnalazione dinamica72e lo sviluppo neuronale73.
Divulgazioni
Gli autori dichiarano di non avere nessun concorrenti interessi finanziari.
Riconoscimenti
Vorremmo ringraziare Changhui Pak e Alysia Vrailas Mortimer per inizialmente insegnamento SMK la tecnica di dissezione del cervello. Ringraziamo anche i membri del gruppo di Ken Moberg lab, soprattutto Chris turni, per leggere criticamente il manoscritto. Anticorpi che riconoscono Fas2 (1 4) e chaoptin (24B10) sono stati ottenuti da Developmental Studies Hybridoma Bank. Anticorpo 24B10 è stato depositato per il DSHB da Seymour Benzer e Nansi Colley24,60,61, anticorpo 1 4 è stato depositato per il DSHB da Corey Goodman 22,23,56. Stock di volare sono stati ottenuti dal centro di Stock di Bloomington. Desideriamo anche ringraziare l'Ohio Agricultural Research e Development Center (OARDC) Classifica MCIC Imaging Center per l'utilizzo del microscopio confocale all'immagine i cervelli in figura 4A e B. SMK è sostenuta da una sovvenzione NICHD (1 R15 HD084241-01A1).
Materiali
| Name | Company | Catalog Number | Comments |
| Microdissection forceps/tweezers | Ted Pella | 505-NM | |
| Sylgard dishes | Living Systems Instrumentation | DD-50-S-BLK | Available from amazon.com |
| Fas2 Antibody | Developmental Studies Hybridoma Bank | 1D4 | |
| Chaoptin Antibody | Developmental Studies Hybridoma Bank | 24B10 | |
| GFP Antibody | Aves Lab | GFP-1010 | |
| Alexa488 goat anti-mouse secondary antibody | ThermoFisher | A-11001 | |
| Alexa488 goat anti-chicken secondary antibody | ThermoFisher | A-11039 | |
| Alexa647 goat anti-mouse secondary antibody | ThermoFisher | A-21236 | |
| 20% paraformaldehyde | Electron Microscope Services | RT15713 | |
| VectaShield | Vector Labs | H-1000 | |
| SuperFrost Plus Slides | ThermoFisher | 99-910-01 | |
| Coverslips | ThermoFisher | 12-553-454 | |
| Na Phosphate Buffer monobasic | Sigma | S3139 | |
| Na phosphate Buffer dibasic | Sigma | S3264 | |
| Triton X 100 | Sigma | X100-100ml | |
| fingernail polish | Electron Microscope Services (EMS) | 72180 | |
| stereomicroscope | Leica S6D with KL300 LED light source | ||
| 9-well dish (spot plate) | VWR | 89090-482 | |
| nutator/rocker | Fisher | 22-363-152 or 88-861-041 | |
| 35mm dish | Genesee Scientific | 32-103 | |
| Sylgard | Fisher | 50-366-794 | |
| Kimwipe | Fisher | 06-666 | |
| Name | Company | Catalog Number | Comments |
| Potential Fixation Buffers | |||
| PTN Buffer | 0.1M NaPhosphate, pH 7.2, 0.1% Triton-X-100, Typically make up 0.5 L of 0.1M NaPhosphate buffer and aliquote 50ml at a time as needed | ||
| PLP buffer | 2% paraformaldehyde, 0.01M NaI04, 0.075M Lysine, 0.037M NaPO4, pH 7.2, Dissolve 0.36 g lysine in 10 ml H2O + 7.5 ml 0.1 M NaH2PO4 pH 7.2 + 2.5 ml 0.1 M Na2HPO4 on ice. Immediately before use, mix 15 ml of this buffered lysine solution with 50 mg NaIO4 (sodium periodate) + 2ml of the 20% high grade paraformaldehyde (EMS) + 3ml H2O | ||
| PEM buffer | 0.1M PIPES pH 7.0, 2mM MgS04, 1mM EGTA, This buffer can be conveniently made as a 2x stock and diluted with 8% paraformaldehyde (PFA) to give a final concentration of 4% PFA | ||
| Name | Company | Catalog Number | Comments |
| Fly Stocks available from Bloomington | |||
| elav (c155)-GAL4 | BL458 | Pan-neuronal GAL4 driver | |
| w*;;;OK107-GAL4 | BL 854 | GAL4 driver for all mushroom body neurons (OK107-GAL4 insertion is on the 4th chromosome) | |
| y(1), w(67c23); c739-GAL4 | BL 7362 | GAL4 driver for alpha and beta lobes (on 2nd chromosome) | |
| y(1), w(67c23); c739-GAL4, UAS-CD8-GFP | BL 64305 | GAL4 driver for alpha and beta lobes, also contains UAS-CD8-GFP | |
| w*; 201Y-GAL4 | BL 4440 | GAL4 driver for primarily the gamma lobes of mushroom body (on 2nd chromosome) | |
| y(1), w(67c23); 201Y-GAL4, UAS-CD8-GFP | BL 64296 | GAL4 driver for mushroom body gamma lobes, also contains UAS-CD8-GFP | |
| w*, elav (c155)-GAL4, hsFLP; FRTG13, Tub>Gal80/CyO | BL 5145 | MARCM stock, contains FRT site and GAL80 on 2nd chromosome | |
| w*, elav (c155)-GAL4, hsFLP, UAS-CD8-GFP | BL5146 | MARCM stock, contains hsFLP, pan-neuronal GAL4, and CD8-GFP on X chromosome | |
| y(1), w*, hsFLP, UAS-CD8-GFP;;FRT82B, Tub>GAL80/TM3, Sb(1);OK107-GAL4 | BL 44408 | MARCM stock for flipping 3rd chromosome | |
| y(1), w*, hsFLP, UAS-CD8-GFP;FRT40A, Tub>GAL80;OK107-GAL4 | BL44406 | MARCM stock for flipping 2nd chromosome | |
| w*, hsFLP, tub>GAL80, FRT19A; UAS-CD8-GFP/CyO;;OK107-GAL4 | BL 44407 | MARCM stock for flipping X chromosome | |
| y(1), w*; UAS-CD8-GFP/CyO | BL 5137 | GFP labels cell surface (CD8 is a transmembrane protein) | |
| y(1), w*; FRTG13, UAS-CD8-GFP | BL 5139 | MARCM stock, contains FRT site and CD8-GFP on 2nd chromosome | |
| y(1), w*, hsFLP, UAS-CD8-GFP; Pin(1)/CyO | BL 28832 | MARCM stock, contains hsFLP and CD8-GFP on X chromosome | |
| w*; FRTG13, Tub>GAL80 | BL 5140 | MARCM stock, contains FRT site and GAL80 on 2nd chromosome | |
| y(1), w*;; FRT82B, Tub>GAL80 | BL 5135 | MARCM stock, contains FRT site and GAL80 on 3rd chromosome |
Riferimenti
- Reichert, H. Evolutionary conservation of mechanisms for neural regionalization, proliferation and interconnection in brain development. Biol Lett. 5 (1), 112-116 (2009).
- Bellen, H. J., Tong, C., Tsuda, H. 100 years of Drosophila research and its impact on vertebrate neuroscience: a history lesson for the future. Nat Rev Neurosci. 11 (7), 514-522 (2010).
- Bier, E. Drosophila, the golden bug, emerges as a tool for human genetics. Nat Rev Genet. 6 (1), 9-23 (2005).
- Oortveld, M. A., et al. Human intellectual disability genes form conserved functional modules in Drosophila. PLoS Genet. 9 (10), 1003911 (2013).
- Sanchez-Soriano, N., Tear, G., Whitington, P., Prokop, A. Drosophila as a genetic and cellular model for studies on axonal growth. Neural Dev. 2, 9 (2007).
- Serafini, T., et al. The netrins define a family of axon outgrowth-promoting proteins homologous to C. elegans UNC-6. Cell. 78 (3), 409-424 (1994).
- Kennedy, T. E., Serafini, T., de la Torre, J. R., Tessier-Lavigne, M. Netrins are diffusible chemotropic factors for commissural axons in the embryonic spinal cord. Cell. 78 (3), 425-435 (1994).
- Harris, R., Sabatelli, L. M., Seeger, M. A. Guidance cues at the Drosophila CNS midline: identification and characterization of two Drosophila Netrin/UNC-6 homologs. Neuron. 17 (2), 217-228 (1996).
- Seeger, M., Tear, G., Ferres-Marco, D., Goodman, C. S. Mutations affecting growth cone guidance in Drosophila: genes necessary for guidance toward or away from the midline. Neuron. 10 (3), 409-426 (1993).
- Huberman, A. D., Clandinin, T. R., Baier, H. Molecular and cellular mechanisms of lamina-specific axon targeting. Cold Spring Harb Perspect Biol. 2 (3), 001743 (2010).
- Hattori, D., et al. Dscam diversity is essential for neuronal wiring and self-recognition. Nature. 449 (7159), 223-227 (2007).
- Lee, T., Luo, L. Mosaic analysis with a repressible cell marker for studies of gene function in neuronal morphogenesis. Neuron. 22 (3), 451-461 (1999).
- Reynaud, E., et al. Guidance of Drosophila Mushroom Body Axons Depends upon DRL-Wnt Receptor Cleavage in the Brain Dorsomedial Lineage Precursors. Cell Rep. 11 (8), 1293-1304 (2015).
- Reuter, J. E., et al. A mosaic genetic screen for genes necessary for Drosophila mushroom body neuronal morphogenesis. Development. 130 (6), 1203-1213 (2003).
- Ng, J. Wnt/PCP proteins regulate stereotyped axon branch extension in Drosophila. Development. 139 (1), 165-177 (2012).
- Lai, Y. W., et al. Drosophila microRNA-34 Impairs Axon Pruning of Mushroom Body gamma Neurons by Downregulating the Expression of Ecdysone Receptor. Sci Rep. 6, 39141 (2016).
- Watts, R. J., Hoopfer, E. D., Luo, L. Axon pruning during Drosophila metamorphosis: evidence for local degeneration and requirement of the ubiquitin-proteasome system. Neuron. 38 (6), 871-885 (2003).
- Borst, A., Helmstaedter, M. Common circuit design in fly and mammalian motion vision. Nat Neurosci. 18 (8), 1067-1076 (2015).
- Clandinin, T. R., Zipursky, S. L. Making connections in the fly visual system. Neuron. 35 (5), 827-841 (2002).
- Schurmann, F. W. Fine structure of synaptic sites and circuits in mushroom bodies of insect brains. Arthropod Struct Dev. 45 (5), 399-421 (2016).
- Heisenberg, M. Mushroom body memoir: from maps to models. Nat Rev Neurosci. 4 (4), 266-275 (2003).
- Hummel, T., Krukkert, K., Roos, J., Davis, G., Klambt, C. Drosophila Futsch/22C10 is a MAP1B-like protein required for dendritic and axonal development. Neuron. 26 (2), 357-370 (2000).
- Grenningloh, G., Rehm, E. J., Goodman, C. S. Genetic analysis of growth cone guidance in Drosophila: fasciclin II functions as a neuronal recognition molecule. Cell. 67 (1), 45-57 (1991).
- Fujita, S. C., Zipursky, S. L., Benzer, S., Ferrus, A., Shotwell, S. L. Monoclonal antibodies against the Drosophila nervous system. Proc Natl Acad Sci U S A. 79 (24), 7929-7933 (1982).
- Zipursky, S. L., Venkatesh, T. R., Teplow, D. B., Benzer, S. Neuronal development in the Drosophila retina: monoclonal antibodies as molecular probes. Cell. 36 (1), 15-26 (1984).
- Wan, L., Dockendorff, T. C., Jongens, T. A., Dreyfuss, G. Characterization of dFMR1, a Drosophila melanogaster homolog of the fragile X mental retardation protein. Mol Cell Biol. 20 (22), 8536-8547 (2000).
- Androschuk, A., Al-Jabri, B., Bolduc, F. V. From Learning to Memory: What Flies Can Tell Us about Intellectual Disability Treatment. Front Psychiatry. 6, 85 (2015).
- Bolduc, F. V., Tully, T. Fruit flies and intellectual disability. Fly (Austin). 3 (1), 91-104 (2009).
- van der Voet, M., Nijhof, B., Oortveld, M. A., Schenck, A. Drosophila models of early onset cognitive disorders and their clinical applications. Neurosci Biobehav Rev. 46, 326-342 (2014).
- Pak, C., et al. Mutation of the conserved polyadenosine RNA binding protein, ZC3H14/dNab2, impairs neural function in Drosophila and humans. Proc Natl Acad Sci U S A. 108 (30), 12390-12395 (2011).
- Gatto, C. L., Broadie, K. Drosophila modeling of heritable neurodevelopmental disorders. Curr Opin Neurobiol. 21 (6), 834-841 (2011).
- van Alphen, B., van Swinderen, B. Drosophila strategies to study psychiatric disorders. Brain Res Bull. 92, 1-11 (2013).
- Kelly, S. M., et al. A conserved role for the zinc finger polyadenosine RNA binding protein, ZC3H14, in control of poly(A) tail length. RNA. 20 (5), 681-688 (2014).
- Kelly, S. M., et al. The Drosophila ortholog of the Zc3h14 RNA binding protein acts within neurons to pattern axon projection in the developing brain. Dev Neurobiol. 76 (1), 93-106 (2016).
- Michel, C. I., Kraft, R., Restifo, L. L. Defective neuronal development in the mushroom bodies of Drosophila fragile X mental retardation 1 mutants. J Neurosci. 24 (25), 5798-5809 (2004).
- Yamamoto, D., Koganezawa, M. Genes and circuits of courtship behaviour in Drosophila males. Nat Rev Neurosci. 14 (10), 681-692 (2013).
- Busto, G. U., Cervantes-Sandoval, I., Davis, R. L. Olfactory learning in Drosophila. Physiology (Bethesda). 25 (6), 338-346 (2010).
- Ueno, T., et al. Identification of a dopamine pathway that regulates sleep and arousal in Drosophila. Nat Neurosci. 15 (11), 1516-1523 (2012).
- Vogelstein, J. T., et al. Discovery of brainwide neural-behavioral maps via multiscale unsupervised structure learning. Science. 344 (6182), 386-392 (2014).
- Brand, A. H., Perrimon, N. Targeted gene expression as a means of altering cell fates and generating dominant phenotypes. Development. 118 (2), 401-415 (1993).
- Duffy, J. B. GAL4 system in Drosophila: a fly geneticist's Swiss army knife. Genesis. 34 (1-2), 1-15 (2002).
- Hamada, F. N., et al. An internal thermal sensor controlling temperature preference in Drosophila. Nature. 454 (7201), 217-220 (2008).
- Hodge, J. J. Ion channels to inactivate neurons in Drosophila. Front Mol Neurosci. 2, 13 (2009).
- Neumuller, R. A., Perrimon, N. Where gene discovery turns into systems biology: genome-scale RNAi screens in Drosophila. Wiley Interdiscip Rev Syst Biol Med. 3 (4), 471-478 (2011).
- Ni, J. Q., et al. A Drosophila resource of transgenic RNAi lines for neurogenetics. Genetics. 182 (4), 1089-1100 (2009).
- Seelig, J. D., et al. Two-photon calcium imaging from head-fixed Drosophila during optomotor walking behavior. Nat Methods. 7 (7), 535-540 (2010).
- Lee, T., Luo, L. Mosaic analysis with a repressible cell marker (MARCM) for Drosophila neural development. Trends Neurosci. 24 (5), 251-254 (2001).
- Williamson, W. R., Hiesinger, P. R. Preparation of developing and adult Drosophila brains and retinae for live imaging. J Vis Exp. (37), (2010).
- Boerner, J., Godenschwege, T. A. Whole mount preparation of the adult Drosophila ventral nerve cord for giant fiber dye injection. J Vis Exp. (52), (2011).
- Sweeney, S. T., Hidalgo, A., de Belle, J. S., Keshishian, H. Dissection of adult Drosophila brains. Cold Spring Harb Protoc. 2011 (12), 1472-1474 (2011).
- Tito, A. J., Cheema, S., Jiang, M., Zhang, S. A Simple One-step Dissection Protocol for Whole-mount Preparation of Adult Drosophila Brains. J Vis Exp. (118), (2016).
- Liu, Y., Liao, S., Veenstra, J. A., Nassel, D. R. Drosophila insulin-like peptide 1 (DILP1) is transiently expressed during non-feeding stages and reproductive dormancy. Sci Rep. 6, 26620 (2016).
- Shafer, O. T., Helfrich-Forster, C., Renn, S. C., Taghert, P. H. Reevaluation of Drosophila melanogaster's neuronal circadian pacemakers reveals new neuronal classes. J Comp Neurol. 498 (2), 180-193 (2006).
- Rieger, D., Shafer, O. T., Tomioka, K., Helfrich-Forster, C. Functional analysis of circadian pacemaker neurons in Drosophila melanogaster. J Neurosci. 26 (9), 2531-2543 (2006).
- Hillebrand, J., et al. The Me31B DEAD-Box Helicase Localizes to Postsynaptic Foci and Regulates Expression of a CaMKII Reporter mRNA in Dendrites of Drosophila Olfactory Projection Neurons. Front Neural Circuits. 4, 121 (2010).
- Goodman, C. S., Davis, G. W., Zito, K. The many faces of fasciclin II: Genetic analysis reveals multiple roles for a cell adhesion molecule during the generation of neuronal specificity. Cold Spring Harb Symp Quant Biol. 62, 479-491 (1997).
- Fushima, K., Tsujimura, H. Precise control of fasciclin II expression is required for adult mushroom body development in Drosophila. Dev Growth Differ. 49 (3), 215-227 (2007).
- Prokop, A., Meinertzhagen, I. A. Development and structure of synaptic contacts in Drosophila. Semin Cell Dev Biol. 17 (1), 20-30 (2006).
- Hadjieconomou, D., Timofeev, K., Salecker, I. A step-by-step guide to visual circuit assembly in Drosophila. Curr Opin Neurobiol. 21 (1), 76-84 (2011).
- Van Vactor, D. Adhesion and signaling in axonal fasciculation. Curr Opin Neurobiol. 8 (1), 80-86 (1998).
- Reinke, R., Krantz, D. E., Yen, D., Zipursky, S. L. Chaoptin, a cell surface glycoprotein required for Drosophila photoreceptor cell morphogenesis, contains a repeat motif found in yeast and human. Cell. 52 (2), 291-301 (1988).
- Tahayato, A., et al. Otd/Crx, a dual regulator for the specification of ommatidia subtypes in the Drosophila retina. Dev Cell. 5 (3), 391-402 (2003).
- Cook, T., Desplan, C. Photoreceptor subtype specification: from flies to humans. Semin Cell Dev Biol. 12 (6), 509-518 (2001).
- Hakeda-Suzuki, S., et al. Goal collaborates with Flamingo in conferring synaptic-layer specificity in the visual system. Nat Neurosci. 14 (3), 314-323 (2011).
- Wu, J. S., Luo, L. A protocol for mosaic analysis with a repressible cell marker (MARCM) in Drosophila. Nat Protoc. 1 (6), 2583-2589 (2006).
- Venken, K. J., et al. MiMIC: a highly versatile transposon insertion resource for engineering Drosophila melanogaster genes. Nat Methods. 8 (9), 737-743 (2011).
- Crittenden, J. R., Skoulakis, E. M., Han, K. A., Kalderon, D., Davis, R. L. Tripartite mushroom body architecture revealed by antigenic markers. Learn Mem. 5 (1-2), 38-51 (1998).
- Muller, H. A. Immunolabeling of embryos. Methods Mol Biol. 420, 207-218 (2008).
- Baker, N. E., Li, K., Quiquand, M., Ruggiero, R., Wang, L. H. Eye development. Methods. 68 (1), 252-259 (2014).
- Ayaz, D., et al. Axonal injury and regeneration in the adult brain of Drosophila. J Neurosci. 28 (23), 6010-6021 (2008).
- Fang, Y., Bonini, N. M. Axon degeneration and regeneration: insights from Drosophila models of nerve injury. Annu Rev Cell Dev Biol. 28, 575-597 (2012).
- Tomchik, S. M., Davis, R. L. Dynamics of learning-related cAMP signaling and stimulus integration in the Drosophila olfactory pathway. Neuron. 64 (4), 510-521 (2009).
- Rabinovich, D., Mayseless, O., Schuldiner, O. Long term ex vivo culturing of Drosophila brain as a method to live image pupal brains: insights into the cellular mechanisms of neuronal remodeling. Front Cell Neurosci. 9, 327 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon