Method Article
Cultura e dosaggio delle popolazioni di caenorhabditis elegans a grande scala
In questo articolo
Riepilogo
Per utilizzare Caenorhabditis elegans (C. elegans) nella ricerca omica, è necessario un metodo per generare grandi popolazioni di vermi in cui un singolo campione può essere misurato su piattaforme per analisi comparative. Qui viene presentato un metodo per la cultura delle popolazioni C. elegans su tavole di coltura su larga scala (LSCPs) e per documentare la crescita della popolazione.
Abstract
Caenorhabditis elegans (C. elegans) è stato e rimane un prezioso organismo modello per studiare biologia dello sviluppo, invecchiamento, neurobiologia e genetica. L'ampio corpus di lavori su C. elegans lo rende un candidato ideale per integrarsi negli studi su animali interi a grande popolazione per sezionare le componenti biologiche complesse e le loro relazioni con un altro organismo. Per utilizzare i C. elegans nella ricerca collaborativa sull'economia, è necessario un metodo per generare grandi popolazioni di animali in cui un singolo campione può essere suddiviso e analizzato su diverse piattaforme per analisi comparative.
Qui viene presentato un metodo per la coltura e raccogliere un'abbondante popolazione di Elegans a fase mista C. su una piastra di coltura su larga scala (LSCP) e i successivi dati fenotipici. Questa pipeline produce un numero sufficiente di animali per raccogliere dati fenotipici e di popolazione, insieme a tutti i dati necessari per gli esperimenti di -omica (ad esempio, genomica, trascrittomica, proteomica e metabolomica). Inoltre, il metodo LSCP richiede una manipolazione minima per gli animali stessi, meno tempo di preparazione dell'utente, fornisce uno stretto controllo ambientale e garantisce che la manipolazione di ciascun campione sia coerente durante tutto lo studio per la riproducibilità complessiva. Infine, vengono presentati i metodi per documentare le dimensioni della popolazione e la distribuzione della popolazione delle fasi di vita di C. elegans in un dato LSCP.
Introduzione
C. elegans è un piccolo nematode libero che si trova in tutto il mondo in una varietà di habitat naturali1. La sua relativa facilità di crescita, il tempo di generazione rapida, il sistema di riproduzione e il corpo trasparente lo rendono un potente organismo modello che è stato ampiamente studiato in biologia dello sviluppo, invecchiamento, neurobiologia e genetica2,3. Il copioso lavoro su C. elegans lo rende un candidato principale da utilizzare negli studi in omica per collegare in modo completo i fenotipi con componenti biologici complessi e le loro relazioni in un dato organismo.
Per utilizzare C. elegans nella ricerca collaborativa sull'omica, è necessario un metodo per generare grandi popolazioni miste di animali in cui un singolo campione può essere suddiviso e utilizzato su piattaforme e strumenti diversi per analisi comparative. La creazione di una pipeline per generare un campione di questo tipo richiede una forte consapevolezza della dieta, dell'ambiente, dello stress, della struttura della popolazione e della gestione e raccolta dei campioni. Pertanto, è fondamentale integrare le condizioni di coltivazione standard e riproducibili nelle tubazioni su larga scala. Nella ricerca C. elegans, due metodi tradizionali sono utilizzati per la coltura dei vermi: piatti di Agar Petri e coltura liquida4.
Storicamente, quando sono necessarie grandi quantità di C. elegans, vengono coltivate in coltura liquida4. I passaggi coinvolti nella generazione di una grande popolazione di vermi nella coltura liquida richiedono più passaggi di manipolazione che spesso includono la sincronizzazione della candeggina con cuticole gravide gravide per adulti, rilasciando embrioni per raggiungere la dimensione della popolazione desiderata. Tuttavia, quando si utilizza la sincronizzazione della candeggina, la crescita della popolazione dipende dall'inizio delle dimensioni del censimento e, pertanto, ha effetti sulla crescita successiva e sul numero di abitanti. Inoltre, i ceppi di C. elegans variano nella sensibilità della cuticola, nel tempo di esposizione e nella risposta allo stress alla sincronizzazione della candeggina, rendendo difficile il dosaggio di molti ceppi allavolta 5,6,7,8,9.
Inoltre, la crescita dei vermi nella coltura liquida richiede un paio di passaggi di trasferimento in quanto spesso si consiglia di coltivare solo una generazione di vermi prima della raccolta perché il sovraffollamento può facilmente verificarsi se coltivato per più generazioni e portare alla formazione di dauer nonostante la presenza dicibo 10. La formazione di Dauer avviene attraverso piccole molecole di segnalazione come gli ascarosides, spesso indicati come "feromoni dauer"11,12,13,14, vengono rilasciati in mezzi liquidi ed effetto la crescita della popolazione. Inoltre, la crescita di grandi popolazioni di vermi in coltura liquida porta all'accumulo eccessivo di batteri nella coltura, creando difficoltà quando è necessario un campione pulito per i test fenotipiche a valle. Infine, quando una coltura liquida viene contaminata, è più difficile da mantenere poiché le spore fungine o le cellule batteriche sono facilmente disperse in tutto il supporto15.
L'altro metodo tradizionale di coltivazione di C. elegans è sui piatti agar Petri. Le piastre Petri disponibili in commercio consentono di coltivare facilmente più generazioni di vermi a stadio misto senza i rapidi effetti del sovraffollamento e dell'elevata formazione di dauer come si vede nelle colture liquide. Tuttavia, uno svantaggio per la crescita dei vermi sulle tradizionali piastre di Agar Petri è che la più grande piastra di Petri disponibile in commercio non produce grandi popolazioni di vermi per uno studio di -omica senza aggiungere in una fase di sincronizzazione della candeggina. In sintesi, coltivare popolazioni a fasi miste di C. elegans su piatti agar Petri è più adatto per raccogliere dati -omics, ma abbiamo richiesto un metodo per generare popolazioni molto grandi senza cultoe liquida.
Qui presentiamo un metodo per la cultura e raccogliamo grandi popolazioni di C. elegans a fase mista su lastre di coltura su larga scala (LSCP). La raccolta di campioni attraverso questa pipeline produce abbastanza campioni per raccogliere dati fenotipici e di popolazione, insieme a tutti i dati necessari per gli esperimenti di -omica(ad esempio,genomica, trascrittomica, proteomica e metabolomica). Inoltre, il metodo LSCP richiede una manipolazione minima degli animali, meno tempo di preparazione dell'utente, fornisce uno stretto controllo ambientale e garantisce che la manipolazione di ciascun campione sia coerente durante tutto lo studio per la riproducibilità complessiva.
Protocollo
1. Sterilizzare l'LSCP e le attrezzature
- Preparare gli LSP in vetro mediante lavaggio a mano, seguito dal lavaggio dei piatti e dalla successiva autoclave per garantire che le vetrerie siano esiste da contaminanti prima di iniziare l'esperimento. Conservare gli LSP autoclavati in un luogo asciutto e pulito fino all'uso.
NOTA: Assicurarsi che gli LSP siano sicuri per lavastoviglie e autoclave. Assicurarsi che i coperchi LSCP siano lavabili in lavastoviglie. - Preparare i coperchi LSCP mediante lavaggio a mano seguito da lavaggio dei piatti. Conservare i coperchi LSCP in un cestino pulito fino a quando necessario.
- Il giorno in cui Nematode Growth Media Agarose (NGMA) è preimposta, pulire i coperchi LSCP con una soluzione di candeggina al 10% due volte, seguita dal 70% di etanolo. Una volta sbiancato con candeggina al 10% e etanolo al 70%, tenere i coperchi LSCP in un bidone pulito nella cappa di flusso laminare dove verrà prepatta la NGMA.
2. Preparare l'agarosio dei mezzi di crescita del nematode (NGMA)
- Preparare NGMA combinando i seguenti reagenti in un pallone autoclavato da 2 L Erlenmeyer con barra di agitazione su una piastra di agitazione: 2,5 g di peptone, 3 g di NaCl, 7 g di agarrose, 10 g di agar e 975 mL di acqua sterile16. Assicurarsi che il volume totale sia uguale a 1 L. Nastro un tappo di lamina al pallone.
NOTA: Le fasi di preparazione per l'NGMA come descritto qui produrranno abbastanza materiale per 2,5 LSP. Il protocollo può essere adattato alle dimensioni del batch LSCP necessarie in un determinato esperimento. - Autoclave su ciclo liquido a 121 °C e 21 p.s.i. per 45 min.
- Accendere il bagno d'acqua e impostare su 50 °C. Portare l'NGMA autoclavato al bagno d'acqua per raffreddare a 50 °C.
- Portare il pallone 2 L Erlenmeyer di NGMA nel cofano o nello spazio pulito e mbiare su una piastra di agitazione. Utilizzare un termometro per tenere traccia della temperatura NGMA.
- Dopo che l'NGMA ha raggiunto i 50 °C, aggiungere quanto segue nell'ordine indicato con una pipetta monouso sterile all'interno della cappa o nello spazio pulito: 25 mL di 1 M KH2PO4 (tampone fosfato K), 1 mL di colesterolo (5 mg/mL in etanolo), 1 mL di etanolo 1 M CaCl2, 1 mL di 1 M MgSO4, 1 mL di nystatin (10mg/mL) e 1 mL di streptomicina (100 mg/mL)16.
- Versare 400 mL di NGMA in un LSCP di vetro sterile, profondo circa 1,3 cm, consentire all'LSCP di solidificarsi su una superficie piana nel cofano e posizionare il coperchio in lamina autoclavato sul LSCP.
- Una volta impostato l'agar, rimuovere il foglio e posizionare un coperchio pulito a tenuta d'aria su LSCP e passare a 4 °C per la conservazione. Conservare NGMA in LSCP a 4 °C fino all'uso e all'uso entro 5 giorni.
3. Generare cibo E. coli per NGMA su LSCP
- Per generare una fonte alimentare stabile, generare lotti di HT115 (DE3) E. coli utilizzando un concetto di media di piccoli lotti coerente con il teorema del limitecentrale 17. Conservare a -80 °C. Quando necessario tirare le scorte batteriche E. coli da -80 °C al disgelo18.
NOTA: In questo protocollo, le scorte batteriche di E. coli sono state coltivate in un bioreattore. Alla fine della crescita della cultura, la cultura fu diluita 1:50, e l'OD600 misurato era 0,4. Pertanto, la cultura aveva un OD600 effettivo di 20. I batteri sono stati pellettati, pesati e rimorsi in mezzo K ad una concentrazione di 0,5 g/mL (peso umido), trasferiti in aliquote da 2 mL e congelati19.
4. Prato batterico su NGMA
- Portare gli LSP NGMA da 4 °C a temperatura ambiente (RT) per diverse ore prima di diffondere il prato batterico per consentire all'intero LSCP di raggiungere RT.
- Estrarre le scorte batteriche E. coli necessarie da -80 °C al disgelo18.
- Diluire le scorte batteriche E. coli con 2 mL di K-medium sterile per raggiungere 0,5 g di E. coli in 4 mL per NGMA LSCP. Pipetta con cura 4 mL di E. coli nel mezzo del NGMA LSCP.
- Utilizzare uno spargitore sterile per diffondere batteri in un rettangolo lasciando circa 3,8 cm di stanza intorno ai bordi del NGMA E. coli libero.
- Lasciare l'NGMA LSCP con E. coli nel cofano con il ventilatore in 1 h per assicurarsi che la sospensione E. coli si ascili completamente.
- Una volta che il prato batterico è asciutto, spingere il coperchio saldamente e conservare a 4 °C fino a quando non viene utilizzato.
5. Chunk worm per ridurre lo stress e la variabilità dell'età tra i campioni
- Striature di vermi da un calcio di verme congelato a una piastra di 6 cm appenaseminata 4. Questo piatto fungerà da piatto "master chunk".
NOTA: Chunking è un metodo ottimale per trasferire vermi da un ceppo omozigoto20. Se un ceppo è eterozigote o deve essere mantenuto raccogliendo e accoppiandosi, il chunking non è consigliabile. Potrebbe essere necessario ottimizzare la frequenza di chunking a seconda dei genotipi di worm utilizzati, della temperatura scelta per la crescita e dei passaggi a valle. - Dopo che la piastra del pezzo principale è piena di adulti gravidi sani (circa 3 giorni) con un sacco di prato E. coli ancora presente, seguire le linee guida standard di chunking C. elegans come descritto in WormBook per produrre quattro piatti a blocchitotali 4.
- Conservare tutte le piastre di blocco in una stanza a temperatura controllata (CT) a 20 °C, salvo diversa indicazione per la crescita.
NOTA: Se gli utenti di questo protocollo non hanno accesso a una sala CT come descritto qui, si consiglia di utilizzare un piccolo incubatore in cui la temperatura può essere controllata o una stanza designata in cui le condizioni ambientali possono essere controllate il più possibile. Se nessuna di queste opzioni alternative è disponibile, si noti che la variazione nella crescita del campione potrebbe essere maggiore. - Una volta che molti adulti gravidi sono osservati nellapiastra del 4 ° pezzo, passare al passaggio 6.
6. Avvistare gli adulti gravidi sbiancati su LSCP
NOTA: Questa tecnica di sbiancamento viene utilizzata per sradicare la maggior parte dei contaminanti e sciogliere la cuticola degli ermafroditi rilasciando embrioni dal verme adulto. La soluzione di candeggina si immergerrà nell'NGMA prima della schiusa degli embrioni.
- Portare gli LSCP su RT per diverse ore prima di avvistare i vermi sbiancanti.
- Preparare un rapporto 7:2:1 di ddH2O : candeggina : 5 M NaOH. Rendi fresca questa soluzione di ipoclorito alcalino poco prima dell'uso.
NOTA: Utilizzare lo stesso stock di candeggina e NaOH per tutta la durata di un determinato esperimento per evitare effetti del lotto di candeggina. La candeggina utilizzata in questo protocollo era l'ipoclorito di sodio al 5-10%. - Accendi un bruciatore Bunsen e accendi un piccone di vermi prima di procedere. Raccogliere e. coli fresco su un piccone sterile dal bordo del prato batterico sulla LSCP.
- Scegli un singolo adulto gravide dal piatto di 4° pezzo per lo sbiancamento spot.
- Pipetta 5 μL della soluzione alcalina di ipoclorito in un angolo dell'LSCP lontano dal prato E. coli.
- Posizionare l'adulto gravid raccolto nella soluzione di ipoclorito alcalino da 5 μL. Tocca il nematode per aiutare a interrompere la cuticola e rilasciare le uova.
- Ripetere i passaggi da 6,4 a 6,6 per un totale di 4x e posizionare 5 adulti gravidi uniformemente intorno al prato di E. coli. Scegli tutti e 5 gli adulti gravidi dalla stessa piastra a 4pezzi per assicurarti che individui quasi geneticamente isogenici vengano aggiunti a un determinato campione.
- Riposizionare il coperchio sull'LSCP.
- Ripetere i passaggi per tutti gli LSCP.
7. Crescita del verme nella stanza a temperatura controllata (CT)
- Dopo lo sbiancamento spot, posizionare il coperchio saldamente sull'LSCP e posizionarlo nella sala CT impostata a 20 °C con flusso d'aria costante e un fotoperiodo 12L:12D (luce 12 ore e oscurità 12 ore).
- Si noti l'ora e la posizione in cui il campione è stato collocato nella sala CT.
NOTA: La posizione all'interno della stanza deve sempre essere documentata per registrare eventuali differenze ambientali che i campioni che potrebbero potenzialmente incontrare durante la crescita. Una volta che il campione è nella stanza CT, dovrebbe rimanere nel punto assegnato indisturbato. Non aprire il coperchio dell'LSCP nella sala CT per ridurre la possibilità di contaminazione. - Porta l'LSCP al microscopio, al di fuori della sala CT, per osservare la crescita e la densità della popolazione.
NOTA: Ogni ceppo e campione di C. elegans varierà nella sua crescita, quindi monitora attentamente i campioni. Sebbene si raccomandi di non disturbare la crescita della LSCP mentre si trova nella sala CT, gli LSCP sono stati trasportati fuori dalla sala CT e i coperchi sono stati aperti ogni 2 giorni per monitorare la crescita del campione. Togliere i coperchi sigillati dai LSCP ogni 2 giorni consente inoltre a O2 di fluire nell'LSCP. - Prima della raccolta assicurarsi che l'LSCP sia diventato pieno di una grande popolazione di vermi. Utilizzare i seguenti criteri per decidere se l'LSCP è pronto per essere raccolto.
- Assicurarsi che l'LSCP sia pieno di vermi adulti gravidi.
- Assicurarsi che la piastra contenga una grande dimensione della popolazione (cioè, i vermi coprono l'intera superficie dell'agar).
- Assicurarsi che la piastra non abbia molte uova sulla superficiedell'agar (cioè, ilnumero massimo di vermi dovrebbe essere nato).
- Assicurarsi che la piastra abbia da minimo a nessun E. coli rimasto, indicando che i vermi muoiono di fame e genererebbero larve di dauer se lasciati sul piatto per altri due giorni.
NOTA: Sebbene la maggior parte degli LSP sia pronta per la raccolta tra 10 e 20 giorni, a seconda della ceppo e del campione, controllare frequentemente ogni LSCP al momento di stabilire questo protocollo per determinare i normali tempi di raccolta.
- Pulire guanti e area con il 70% di etanolo tra la manipolazione di LSCP per evitare la contaminazione incrociata tra ceppi.
8. Raccolta del campione LSCP
- Accendere e lasciare raffreddare la centrifuga a 4 °C prima della raccolta dei campioni.
- Preparare tre tubi conici da 50 ml con 50 ml di soluzione M9 per LSCP da raccogliere.
- Etichettare un tubo conico da 15 ml per LSCP.
NOTA: Tutti i passaggi di centrifugazione vengono eseguiti nel tubo conico da 15 ml, perché i vermi tendono a pellet bene in questi tubi. - Versare 50 ml di soluzione M9 (da un tubo conico da 50 ml nel passaggio 8.2) sulla superficie LSCP e ruotare intorno per assicurarsi che M9 copra l'intera superficie NGMA.
- Mentre M9 si trova sulla superficie LSCP, innescare una pipetta sierologica sterile con M9.
NOTA: Adescando la pipetta sierologica sterile con M9, questo assicura che meno vermi si attacchino all'interno della pipetta di plastica, prevenendo la perdita del campione. - Inclinare l'LSCP in modo che M9 e la popolazione di vermi si riunisci in un angolo dell'LSCP.
NOTA: La miscela di soluzione M9 e vermi dell'LSCP sarà definita la "sospensione del verme" nelle fasi a valle. - Utilizzando una pipetta sierologica innescata con un pipettor automatico, sospensione del verme pipetta e posto nel tubo conico originale da 50 ml. Una volta raccolti 50 ml di sospensione del verme, posizionare il tubo conico su un rocker per interrompere i grumi batterici e i detriti.
- Ripetere i passaggi da 8,4 a 8,7 raccogliendo 150 mL di sospensione worm per LSCP.
- Trasferire 15 ml di sospensione del verme, da uno dei tre tubi conici da 50 ml, versando in un tubo conico etichettato da 15 ml messo da parte nella fase 8.3. Centrifugare il tubo conico da 15 ml a 884 x g per 1 min a 4 °C. La maggior parte dei vermi si pellet nella parte inferiore del tubo.
- Aspirare il supernatante assicurandosi di non disturbare il pellet di verme.
- Continuare ad aggiungere circa 13 ml di sospensione del verme allo stesso tubo conico da 15 ml ripetendo i passaggi 8.9 e 8.10 fino a quando non vengono consumati tutti i 150 ml di sospensione del verme. Invertire il tubo e disturbare il pellet tra le centrifugazioni per lavare e aspirare il più possibile batteri e detriti.
NOTA: In questa fase, il contenuto di tutti e tre i tubi conici da 50 ml viene condensato in un unico tubo da 15 ml. - Aggiungere 10 ml di M9 pulito al tubo conico da 15 ml e agitare il pellet di verme invertendo. Centrifugare il tubo conico da 15 ml a 884 x g per 1 min a 4 °C. Aspirare il supernatante assicurandosi di non disturbare il pellet di verme. Ripeti due volte.
NOTA: Se c'è una grande quantità di detriti o batteri nel campione, ripetere il passaggio 8.12 fino a quando il campione non è pulito. - Una volta pulito il campione, aggiungere ddH2O al pellet di verme per un totale di 10 mL di ddH2O e vermi. Agitare il pellet di verme invertendo. Muoversi rapidamente nel passaggio 9.1, poiché i vermi devono rimanere in ddH2O per 5 minuti o meno per evitare stress osmotico.
NOTA: La sospensione del pellet di verme in ddH2O è il solvente preferito per i passaggi a valle. I vermi possono essere sospesi in altri solventi o tamponi se sono compatibili con un determinato flusso di lavoro sperimentale.
9. Stimare la dimensione della popolazione
NOTA: spostarsi rapidamente tra i passaggi da 9.1 a 9.7. La miscela di ddH2O e vermi del passaggio 8.13 è indicata come il "campione di verme" nei passaggi successivi.
- Prima di pipettare il campione di verme, punta della pipetta primaria da utilizzare con M9 per evitare che i vermi si attacchino all'interno della pipetta di plastica prevenendo la perdita del campione e riducendo la variazione di conteggio.
- Prendere un'aliquota di 100 μL di campione di verme e diluirla in 900 μL di M9. Mescolare bene e fare una diluizione seriale (1:10, 1:100, 1:1000). Ripetere questo passaggio due volte per ottenere un totale di tre set di repliche di aliquote.
NOTA: i vermi pipettanti possono causare un'elevata variabilità nel numero di abitanti campione. Assicurarsi che il campione di worm sia omogeneo prima di pipeggiare l'aliquota desiderata. - Impostare il tubo conico da 15 ml su un rocker per continuare a spostare la coltura mentre vengono conteggiate le aliquote.
- Assicurarsi che il campione di verme sia ben miscelato e omogeneo. Pipetta 5 μL dal campione di verme 1:10, erogarla su una diapositiva di microscopia e contare il numero di vermi. Se questo numero è minore di circa 50 worm, conta anche le diluizioni 1:100 e 1:1000. Se è superiore a 50, passare alla successiva diluizione seriale.
NOTA: se troppi worm non possono essere conteggiati con precisione, utilizzare invece la successiva diluizione seriale per il conteggio. - Contare ogni replica aliquota di ogni diluizione 3x. Al termine del conteggio, per la maggior parte delle culture, saranno documentati 9 conteggitotali (cioè3 conteggi totali per ogni replica aliquota).
- Media dei conteggi di diluizione per determinare la dimensione stimata della popolazione del campione di worm. Questi conteggi di diluizione determineranno il volume del campione di worm necessario per creare la dimensione aliquota desiderata per i passaggi -omics.
NOTA: In questo esperimento sono state generate aliquote di circa 200.000 worm a stadio misto. Inoltre, un'aliquota di circa 50.000 vermi a stadio misto è stata accantonata per lo smistamento in un grande citometro a flusso di particelle (descritto nel passaggio 10). - Una volta che il campione di verme è stato diviso in aliquote appropriate, congelare flash nell'azoto liquido e conservare il campione a -80 °C.
NOTA: Non congelare l'aliquota destinata alla citometria a flusso di particelle di grandi dimensioni.
10. Campione di preparazione (opzionale) per la citometria a flusso di particelle di grandi dimensioni
NOTA: I passaggi 10, 11 e 12 sono il metodo preferito dagli autori per registrare la crescita del campione(cioè, la dimensione dellapopolazione e la distribuzione della popolazione delle fasi del ciclo di vita di C. elegans) e determinare il successo di una cultura. Gli utenti di questo protocollo possono sostituire i passaggi 10, 11 e 12 opzionali con le proprie metriche di successo della crescita. I passaggi 10, 11 e 12 sono descritti qui per due motivi; in primo luogo, in modo che gli utenti che hanno apparecchiature utilizzate nei passaggi 10, 11 e 12 possano replicare questi passaggi e, in secondo luogo, per mostrare la convalida di questo metodo di crescita. Il passaggio 9 precedente fornisce una buona stima del numero totale di worm per determinare le dimensioni delle aliquote e il passaggio 10 è una metrica più quantitativa per stimare il numero e la distribuzione della popolazione dei vermi in un determinato campione.
- Portare l'aliquota di circa 50.000 vermi a stadio misto (messi da parte nella fase 9.6) fino a 10 mL di volume totale in soluzione M9.
- Creare una soluzione composta da 1 mg/mL di E. coli e una diluizione di 1:50 di 0,5 μM di microsfere fluorescenti rosse19.
- Aggiungere 200 μL di questa soluzione ai 10 mL di vermi a stadio misto in M9 e incubare mentre si dondola per 20 minuti.
- Dopo 20 minuti, centrifugare il tubo conico da 15 ml a 884 x g per 1 min a 4 °C.
- Aspirare il supernatante assicurandosi di non disturbare il pellet di verme.
- Lavare il pellet di vermi due volte con la soluzione M9 per eliminare i batteri in eccesso e le microsfere fluorescenti rosse.
- Aggiungere 5 mL di M9 al pellet di verme e assicurarsi che il pellet abbia un aspetto pulito. Se il pellet è pulito, aggiungere 5 mL di M9 con azide di sodio da 50 mM sia per raddrizzare che per uccidere i vermi per un conteggio e undimensionamento accurati 21.
- Documentare l'ora e la data in cui l'azide di sodio viene aggiunto al campione.
- Mettere da parte il campione sul rocker fino a quando necessario per la citometria a flusso di particelle di grandi dimensioni.
NOTA: L'azide di sodio è noto per influenzare la fisiologia del nematode(cioè,la lunghezza del corpo, il metabolismo e la termotoleranza). Pertanto, è fondamentale notare il tempo in cui i vermi sono esposti all'azide di sodio poiché molti di questi effetti fisiologici si verificano nel giro dipochi minuti 22. A causa degli effetti fisiologici noti dell'azide di sodio sui vermi, questo trattamento influenzerà la qualità dell'immagine a valle e dovrebbe essere considerato.
11. (facoltativo) Documentare la distribuzione della popolazione e la preparazione di una piastra da 384 pozzi per l'imaging
NOTA: Il passaggio 11 utilizza un grande citometro a flusso di particelle (LPFC). La conoscenza di base di un LPFC è assunta in questo protocollo. Altri metodi possono essere sostituiti per documentare la crescita e la distribuzione della popolazione dei campioni. I passaggi documentati qui sono per gli utenti che prevedono di utilizzare un LPFC nella pipeline23.
- Accendere, pulire e innescare l'LPFC e consentire ai laser di riscaldarsi per 1 h prima di ordinare i campioni.
- Dopo che il laser si è riscaldato, apri il profilo e la scala "Histogram" a un Time of Flight (TOF) del 2050.
- Aggiungere un'area bar all' "Istogramma" che si estende su un intervallo TOF di 100. La prima regione bar copre un TOF di 50-150.
- Continuare a creare venti aree bar ciascuna su un intervallo TOF di 100. Queste aree bar si estenderanno su tutta la gamma TOF dal 50 al 2050. Cfr. tabella complementare 1 per le regioni esatte da utilizzare in tutta la distribuzione tof.
- Salvate questo Istogramma impostato come"Esperimento " da utilizzare nelle future esecuzioni LPFC.
- Selezionare la piastra calibrata a 384 po' o calibrare lo strumento su una piastra da 384 po' per erogare oggetti.
- Una volta nel modello di piastra calibrato da 384 pozzi, impostare il modello per erogare 20 oggetti recintato in quattro pozzi (quattro repliche tecniche di ogni regione recintata) per ciascuna delle 20 regioni a barre create durante i passaggi 11.3-4. Vedere la tabella complementare 2 per un layout di esempio su come erogare vermi nella piastra a 384 pozzi.
- Trasferire il campione dal passo 10.9 in un tubo conico da 50 ml e aggiungere una soluzione M9 aggiuntiva per ottenere circa 40 mL di volume totale.
- Iniziare a ordinare automaticamente il campione in base ai parametri impostati nel passaggio 11.7 mescolando continuamente il campione per evitare di depositare e erogare simultaneamente oggetti dal campione nella piastra calibrata a 384 poggia.
NOTA: assicurarsi che la portata dell'LPFC sia operativa tra 15-20 oggetti al secondo e specificare che non vengano ordinate doppie. - Una volta che l'intero campione è stato ordinato e il numero massimo di regioni recintato è stato erogato nella piastra da 384 pozzi, togliere il campione dall'LPFC e lo strumento pulito.
NOTA: quando vengono raggiunte regioni TOF più grandi, potrebbe diventare difficile continuare a riempire la piastra da 384 pozzi a causa del basso numero di eventi in quella regione TOF. Riempi il maggior numero possibile di regioni recintate per avere la migliore idea di dove le fasi di vita di C. elegans rientrano nella distribuzione LPFC prima di esaurirsi. - Posizionare una pellicola di tenuta sopra la piastra da 384 poggia-poggia fino a quando non viene stampata.
NOTA: Lastra di immagine il più rapidamente possibile dopo lo smistamento perché i campioni vengono trattati con azidedi sodio 22. Le microsfere fluorescenti rosse possono essere viste nei file di dati LPFC raccolti(ad esempio i dati PH Red nel file di testo di output) in base al livello di fluorescenza rossa emesso in ogni oggetto ordinato per aiutare a identificare quali oggetti sono vermi vivi, worm morti, dauer ospazzatura 24.
12. (opzionale) Imaging piastra da 384 pozzi
NOTA: Il passaggio 12 utilizza un microscopio micro confocale per la lettura delle lastre. In questo protocollo si presuppone la conoscenza di base di un microscopio microfocale. Altri metodi possono essere sostituiti per documentare la crescita e la distribuzione della popolazione dei campioni.
- Utilizzo di un microscopio microfocale a lettura piastra con lente 20x.
- Aprite lascheda " Objective and Camera" e impostate sulla modalità "10x Plan ApoLambda".
- Aprite lascheda " Camera Binning" e impostate su "2".
- Apri la scheda "Siti da visitare su piastra" e imposta su "4" siti per pozzo e "sovrapporsi ai siti del 10%" per unire le immagini in seguito.
- Apritela scheda" Wavelength " e impostate su "Brightfield 1".
- Aprire la scheda "Illuminazione" e impostare su "Luce trasmessa, campione luminoso".
- Posizionare la piastra a 384 pozzi al microscopio e impostare la "Z Stack" su "Calculate Offset" e trovare il piano focale corretto per i campioni nella piastra a 384 pozzi.
- Eseguire la piastra da 384 pozzi sul microscopio micro confocale raccogliendo quattro immagini per pozzo.
- Monta le quattro immagini insieme per creare un'immagine per pozzo.
Risultati
La crescita di C. elegans utilizzando il metodo LSCP produce in media circa 2,4 milioni di vermi a stadio misto per campione nell'corso di 12,2 giorni. La crescita di C. elegans con il metodo LSCP consente agli utenti di generare grandi popolazioni a stadio misto di C. elegans con poca manipolazione e manipolazione degli animali, ideale per studi di economia su larga scala ( Figura1). Una volta che un LSCP è diventato pieno di vermi adulti, ha raggiunto una grande dimensione della popolazione e ha meno batteri minimi, gli utenti possono raccogliere e stimare le dimensioni della popolazione. Questo punto può anche servire da controllo di qualità valutando se la popolazione è sufficiente da utilizzare in una pipeline -omica (Figura 2). Le dinamiche della popolazione dipendono dal ceppo stesso,dal comportamento del ceppo (cioè, i ceppi scavatori tendevano ad avere un minore recupero deivermi) e dal successodella crescita (cioè, contaminazione). Il metodo LSCP è stato testato su 15 ceppi di C. elegans contenenti una miscela di mutanti del Caenorhabditis Genetics Center (CGC) e cendr (Caenorhabditis elegans Natural Diversity Resource)25. I genotipi di ceppo sono descritti nella tabella complementare 3.
Il metodo LSCP ha prodotto dimensioni della popolazione da circa 94.500 a 9.290.000. La dimensione media della popolazione all'interno del ceppo di riferimento, PD1074, e tra i ceppi era di circa 2,4 milioni di vermi (Figura 3). Non sono state riscontrate differenze significative nelle dimensioni stimate della popolazione tra i ceppi di C. elegans nel corso di una media di 12,2 giorni di crescita LSCP(figura 4). I PD1074 LSCP hanno impiegato tra i 10 e i 14 giorni per crescere fino a raggiungere una popolazione a fasi miste. Il tempo medio di crescita nel PD1074 è stato di 10 giorni. La tensione in crescita più lenta è cresciuta per un massimo di 20 giorni e la varietà in più rapida crescita è cresciuta per un minimo di 10 giorni(figura 4).
Pertanto, utilizzando questo metodo LSCP, gli utenti possono facilmente integrare nuovi ceppi di interesse in uno studio con poca conoscenza dei tempi di sviluppo e delle competenze di base. Si noti che i ceppi e i fenotipi che devono essere mantenuti mediante prelievo, hanno difetti di fecondità, sono eterozigoti o hanno difetti di crescita potrebbero non funzionare bene in questa pipeline.
La citometria a flusso di particelle di grandi dimensioni e l'imaging di campioni consentono agli utenti di documentare la distribuzione della popolazione. Un'ampia varietà di piattaforme può essere utilizzata per misurare la crescita demografica di successo.
Per le misurazioni riproducibili dell'omica, è importante coltivare colture coerenti. Le metriche della riproducibilità delle impostazioni cultura sono il numero di worm e una distribuzione delle dimensioni coerente per un determinato ceppo. Mostriamo la distribuzione del campione per il ceppo di riferimento, PD1074 – una variante del ceppo originale N2 Bristol, utilizzando le immagini al microscopio lPFC23,26 e micro confocale come proxy per il successo della crescita. Poiché i vermi sono stati misurati dallo stadio L1 fino all'adulto gravid sulla distribuzione LPFC (Figura 5), nell'imaging successivo (Figura 6) e nella variazione della distribuzione della popolazione tra i campioni (Figura 7), possiamo vedere che questa pipeline ha generato una popolazione a fase mista di C. elegans.
Per dare un'occhiata più da vicino alla distribuzione della popolazione dei nostri campioni a fase mista, abbiamo esaminato la distribuzione di 35 LSCP PD1074 osservando la percentuale di vermi che rientrano in ogni regione durante l'intera distribuzione del tempo di volo (TOF)(cioè, lunghezzacorporea)(Figura 7A,B).
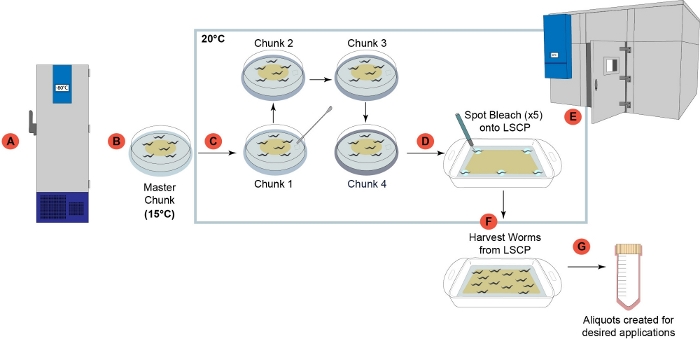
Figura 1: Panoramica della pipeline di crescita worm LSCP. (A) Una volta ricevuti in laboratorio, tutti i ceppi sono stati preparati e congelati per la conservazione a lungo termine a -80 °C2. (B) Una piastra "master chunk" è stata preparata da un maga di verme congelato e conservata a 15 °C per essere utilizzata per non più di un mese. (C) Ogni campione ha subito quattro fasi successive di chunking per ridurre lo stress generazionale prima di crescere sulla LSCP. (D) 5 singoli adulti gravidi sono stati prelevati dalla piastra "chunk 4" 6 cm nella fase (D) e sbiancati in cinque aree dell'LSCP. (E) L'LSCP è stato collocato in una stanza a temperatura controllata e coltivato a 20 °C fino a quando l'LSCP non era pieno di vermi adulti, ha raggiunto una grande dimensione della popolazione e ha lasciato batteri minimi. (F) La popolazione di vermi è stata raccolta e raccolta per le fasi a valle. (G) Le aliquote sono state create dall'LSCP e sono state congelate per le applicazioni desiderate a valle. Clicca qui per visualizzare una versione più grande di questa figura.
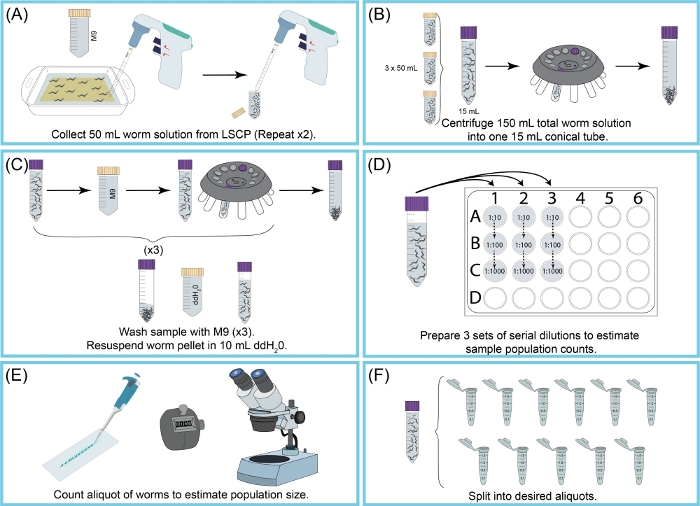
Figura 2: Panoramica della raccolta e della stima delle dimensioni della popolazione da parte della LSCP. (A) 50 mL di M9 sono stati utilizzati per lavare i vermi dalla superficie NGMA. La sospensione del verme è stata pipettata in un tubo conico da 50 ml. Il passaggio (A) è stato ripetuto due volte. (B)15 ml di sospensione del verme sono stati versati in un nuovo tubo conico da 15 ml. I vermi venivano pellettizzazione centrifugando. M9 + detriti sono stati aspirati senza disturbare il pellet di verme. Il passo (B) è stato ripetuto fino a quando non sono stati raccolti tutti i 150 mL di sospensione del verme. (C) Il pellet di verme è stato lavato e centrifugato tre volte con M9 per eliminare i detriti rimanenti. Una volta pulito il campione, il pellet di verme è stato rimorsinato in 10 mL di ddH2O. (D) È stata creata una diluizione seriale del campione per stimare le dimensioni della popolazione di vermi. Sono stati utilizzati i fattori di diluizione che consentivano di contare accuratamente i vermi. I fattori di diluizione utilizzati sono cambiati a seconda delle dimensioni della popolazione dell'LSCP. (E) Una volta scelti i fattori di diluizione, tutti i vermi di tutte e tre le repliche aliquote di tale diluizione sono stati pipettati su uno scivolo pulito e i vermi sono stati conteggiati al microscopio di sezionazione. (F) Il campione è stato suddiviso in aliquote di dimensioni appropriate. Clicca qui per visualizzare una versione più grande di questa figura.
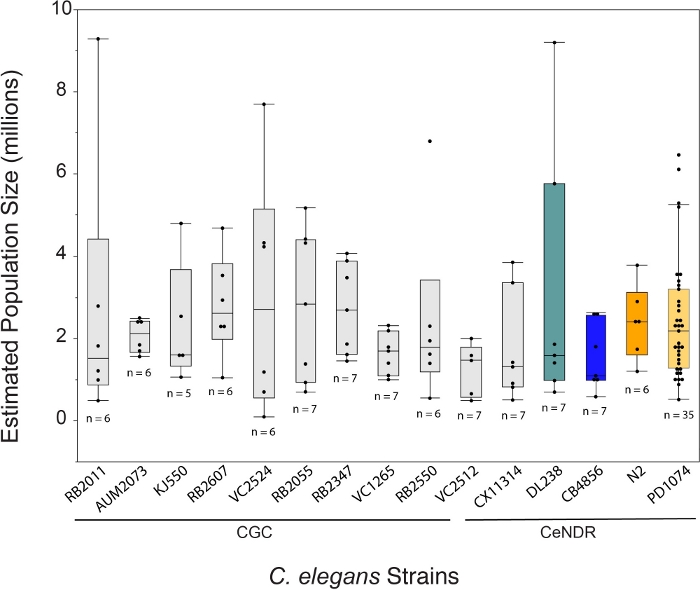
Figura 3: Il metodo LSCP ha generato in media una popolazione di 2,4 milioni di worm a stadio misto. L'LSCP produce dimensioni della popolazione nella popolazione più piccola che si è in crescita a circa 94.500 e con la più grande crescita della popolazione a circa 9.290.000. La dimensione media della popolazione in tutti i ceppi era di 2,4 milioni di vermi. Le barre sotto i nomi dei ceppo C. elegans indicano se un ceppo è un mutante CGC o un isolato naturale CeNDR. Per ogni ceppo viene visualizzata la dimensione del campione LSCP. Sono stati eseguiti confronti per tutte le coppie che utilizzano il test HSD di Tukey. Non sono state osservate differenze significative tra le dimensioni stimate della popolazione tra i ceppi di C. elegans (F(14.108) = 0,7, p = 0,77). Le barre colorate indicano i display a colori standard per la rispettiva rappresentazione dello sforzo C. elegans. Clicca qui per visualizzare una versione più grande di questa figura.
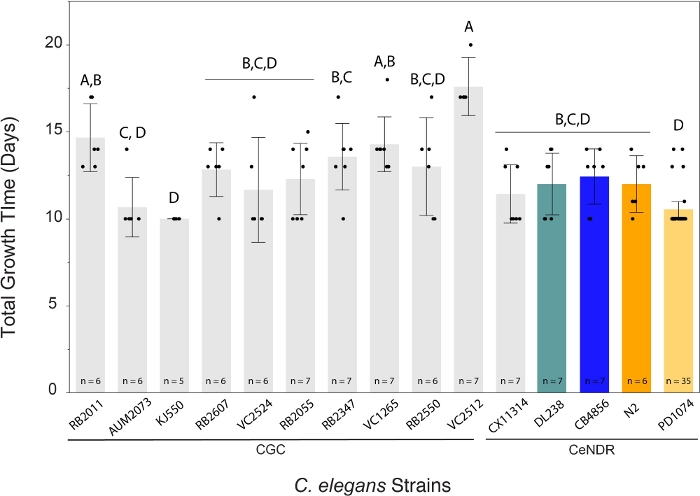
Figura 4: Il metodo LSCP ha generato grandi popolazioni miste di worm in 10-20 giorni. Un dato C. elegans LSCP crebbe fino a quando il campione era pieno di vermi adulti, raggiunse una grande dimensione della popolazione e aveva un prato batterico minimo rimasto. Gli LSCP hanno impiegato tra i 10 e i 20 giorni per crescere fino a raggiungere una popolazione a fasi miste, a seconda del ceppo. Il tempo medio di crescita tra i ceppi è stato di 12,2 giorni. Per ogni ceppo viene visualizzata la dimensione del campione LSCP. Ogni barra di errore è stata costruita utilizzando 1 deviazione standard dalla media. I livelli non collegati dalla stessa lettera sono significativamente diversi. Confronti per tutte le coppie che utilizzano il test HSD di Tukey. Una differenza significativa è stata riscontrata nella quantità di tempo di crescita sull'LSCP necessaria tra i ceppi di C. elegans (F(14.108) = 8,8, p < 0,0001*). Le barre colorate indicano i display a colori standard per la rispettiva rappresentazione dello sforzo C. elegans. Clicca qui per visualizzare una versione più grande di questa figura.
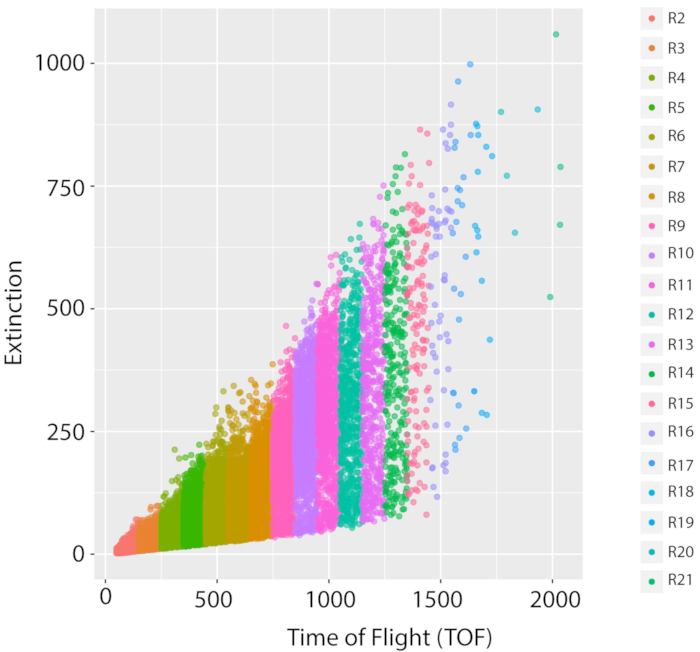
Figura 5: Misurazione mista della popolazione e della crescita del ceppo di riferimento di tipo selvatico, PD1074. Una distribuzione rappresentativa LPFC di una crescita LSCP del ceppo di riferimento di tipo selvaggio, una variante del ceppo originale N2 Bristol (PD1074) documenta la distribuzione delle dimensioni e il conteggio degli eventi di una popolazione a fase mista. L'asse x visualizza la lunghezza (Tempo di volo, TOF) dei worm ordinati. L'asse y mostra la densità ottica (estinzione ottica, EXT) dei vermi ordinati. Ogni punto dati è un worm documentato nell'esempio. Ogni area TOF utilizzata per l'analisi delle immagini viene visualizzata in un colore diverso. Sono state create venti regioni TOF (R2 – R21) che vanno da un TOF di 50 al 2050. I dettagli su ciascuna regione tof sono disponibili nella tabella complementare 1. Clicca qui per visualizzare una versione più grande di questa figura.
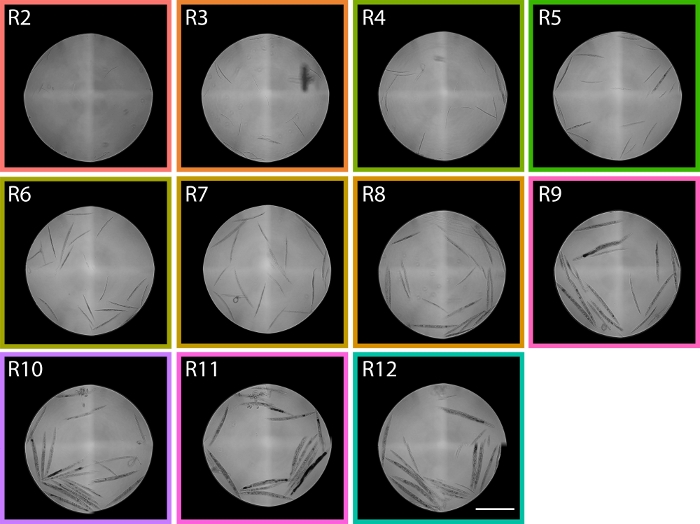
Figura 6: Le immagini dei worm ordinati dalle regioni TOF che vanno da R2 a R12 mostrano la distribuzione LPFC PD1074. Nella regione R2, i vermi L1 possono essere identificati e nella regione R9 vengono identificati adulti prevalentemente gravidi, che coprono i due estremi larvali dello sviluppo dandoci regioni approssimative all'interno della distribuzione citometrica del flusso di dove sono previsti stadi nella distribuzione. La barra di scala rappresenta 1 mm. Le immagini rappresentative sono state prese dalla distribuzione LPFC visualizzata nella figura 5e le caselle colorate corrispondono alle regioni della figura 5. Clicca qui per visualizzare una versione più grande di questa figura.
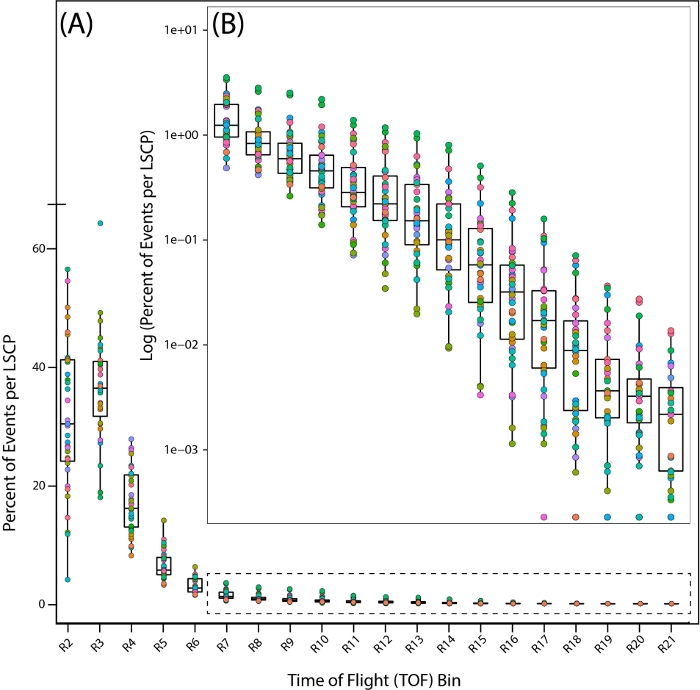
Figura 7: Distribuzione della popolazione attraverso le regioni del tempo di volo (TOF) nel ceppo di riferimento di tipo selvaggio, PD1074. Distribuzione dei worm nell'intera regione TOF che mostra le regioni in cui sono stati trovati i worm. Ogni PD1074 LSCP è rappresentato come un singolo colore. (A) L'asse x mostra le venti regioni TOF (R2 – R21) osservate e conteggiate per l'LSCP, che visualizzano l'intera distribuzione delle dimensioni. L'asse y mostra la percentuale di worm di un dato LSCP con una dimensione corporea che è caduta in una data regione TOF. (B) Poiché una frazione minore della popolazione di vermi si trova tra le regioni R7-R21, il registro della percentuale di vermi che rientrava in ciascuna regione è stato preso per visualizzare la distribuzione della popolazione. Nell'asse x vengono visualizzate le regioni TOF R7-R21. L'asse y visualizza il registro della percentuale di worm di un determinato LSCP con una dimensione corporea che è caduta in una determinata regione TOF. Clicca qui per visualizzare una versione più grande di questa figura.
Figura complementare 1: Temperatura media giornaliera (°C) delle condizioni di crescita in cui l'LSCP è stato coltivato e gestito. Le temperature riportate della stanza a temperatura controllata (CT) sono state documentate e raccolte durante l'arco di sei mesi di crescita e raccolta del campione. La temperatura media giornaliera è riportata qui. Non sono state osservate differenze significative tra la temperatura in cui l'LCSP è cresciuto durante la durata del progetto (F(5,24) = 2,59, p = 0,0524). L'intera differenza di temperatura si è estendeta non superiore a 0,003 °C per tutta la durata di sei mesi della crescita e della generazione del campione. Clicca qui per scaricare questa cifra.
Tabella complementare 1: Regioni gated TOF utilizzate per ordinare i vermi in piastre da 384 pozza per l'imaging. Le aree binned sono state create per coprire un TOF di 100 in tutta la distribuzione TOF dal 50 al 2050. Le regioni recintate possono essere modificate e ottimizzate in base alle proprie esigenze. Ogni area TOF utilizzata per l'analisi delle immagini viene visualizzata in un colore diverso. Clicca qui per scaricare questa tabella.
Tabella supplementare 2: modello di piastra a 384 pozzi di regioni TOF e layout di replica. Ogni campione è stato ordinato in una piastra da 384 po', per l'imaging. Sono state create quattro repliche per ogni area selezionata per l'ordinamento. Le regioni recintate possono essere modificate e ottimizzate in base alle proprie esigenze. Cfr. tabella complementare 1 per specifiche regioni recinta create e utilizzate in questo protocollo. Ogni area TOF utilizzata per l'analisi delle immagini viene visualizzata in un colore diverso. Clicca qui per scaricare questa tabella.
Tabella complementare 3: I ceppi di C. elegans utilizzati in questo protocollo contengono una miscela di ceppi di CGC e CeNDR. La tensione, il genotipo, la sorgente di deformazione e i dettagli sono descritti in questa tabella. Clicca qui per scaricare questa tabella.
Discussione
Una varietà di navi può essere utilizzata come LSCP. In questo protocollo è stata utilizzata una teglia standard in vetro. Gli LSPC in uso avevano dimensioni esterne di 35,56 x 20,32 cm, dimensioni interne di 27,94 x 17,78 cm e circa 4,45 cm di profondità e erano dotati di un coperchio montato. Pertanto, la quantità di batteri qui utilizzati è stata ottimizzata per un LSCP con le dimensioni di cui sopra per produrre una grande popolazione di vermi a stadio misto. Il volume e la concentrazione batterica possono essere regolati per soddisfare le esigenze sperimentali.
La contaminazione da muffe, funghi o altre fonti batteriche può verificarsi in qualsiasi fase del metodo LSCP, quindi maneggiare i campioni con cura. Prima di iniziare qualsiasi fase del protocollo, assicurarsi che lo spazio di lavoro sia pulito con il 70% di etanolo e il 10% di candeggina. Se disponibile, trattare le aree usate con luce UV per 30 minuti e accendere un filtro dell'aria HEPA 30 minuti prima di iniziare ogni passaggio.
Facendo crescere l'LSCP in un ambiente controllato ( cioè , in una sala CT impostata a 20 °C),l'utentepuò monitorare più facilmente la crescita del campione e documentare la potenziale contaminazione. Se la superficie della LSCP viene contaminata, tagliare la contaminazione quando possibile e lasciare che il campione continui a crescere o scartare il campione se la contaminazione non è possibile controllare. È imperativo affrontare rapidamente la contaminazione per ridurre la crescita indesiderata e garantire che non superi i vermi per le risorse.
Questo metodo è pensato per coloro che vogliono coltivare culture di popolazione mista su larga scala di C. elegans. Sebbene sia possibile coltivare popolazioni sincronizzate di vermi sull'LSCP come fatto su piastre petri disponibili in commercio e in coltura liquida, gli autori non hanno testato questa opzione. Inoltre, se gli utenti desiderano coltivare in media più di 2,4 milioni di worm in un determinato campione, si consiglia un metodo diverso4. Il successo della crescita dipende dalla tensione in fase di elaborazione nella pipeline. Gli autori sono stati in grado di far crescere con successo popolazioni di circa 2,4 milioni di vermi in almeno cinque repliche biologiche di 15 ceppi di Elegans C. , indicando che il metodo è robusto.
Prima di iniziare l'esperimento si noti che l'età e la salute di un dato verme possono influenzare la fecondità e il conseguente tempo di crescita della popolazione. Assicurarsi che i worm siano mantenuti in condizioni di salute con uno stress minimo prima di essere utilizzati in questa pipeline. Si presume che campioni di stock siano stati creati, congelati e conservati a -80 °C per ridurre la deriva genetica nel tempo.
A seconda delle esigenze di un dato esperimento, il numero di adulti gravidi in partenza su un LSCP può essere modificato. La modifica del numero di adulti gravidi in partenza sulla LSCP cambierà il tasso di crescita e quindi il tempo di raccolta. Cinque adulti gravidi sono usati per seminare ogni LSCP per i seguenti motivi: (1) Era necessario un modo semplice, veloce ed efficiente per seminare molti ceppi di C. elegans sui LSCP contemporaneamente e (2) per ridurre le differenze di età tra gli adulti gravidi raccolti che potrebbero portare all'eterogeneità della crescita.
Questo metodo consente all'utente di raccogliere grandi popolazioni di vermi con tutte le fasi del ciclo di vita presenti. Con i metodi attuali disponibili, la raccolta di campioni su larga scala di C. elegans richiede la sincronizzazione della candeggina per ottenere il numero di worm desiderati per il lavoro a valle. Dato questo approccio, ora si può coltivare il maggior numero possibile di vermi in fermentatori o colture liquide su larga scala senza le difficoltà associate alla sincronizzazione della candeggina e a più passaggi di movimentazione. Il nostro protocollo consente di indirizzare i ceppi di interesse in modo efficiente, utilizzare un tempo di gestione minimo per far crescere il campione stesso e isolare le fasi dei worm o della popolazione in base alle esigenze nelle pipeline downstream.
Un LPFC è stato utilizzato come strumento per documentare la distribuzione e le dimensioni della popolazione in un dato LSCP. L'LPFC utilizzato è un sistema a flusso continuo che analizza, ordina e distribuisce i worm in base alle loro dimensioni (TOF) e alla densità ottica. Quando un dato worm passa attraverso la cella di flusso, il rilevatore di perdita di luce assiale cattura la quantità di luce del segnale bloccata da un laser a stato solido da 488 nm per il periodo di tempo necessario a passare un verme, dando all'utente il TOF e la densità ottica del worm. Le ottiche e i rivelatori di raccolta della fluorescenza possono anche essere utilizzati per massimizzare la sensibilità e la raccolta della fluorescenza su ciascun campione. I parametri di raccolta LPFC variano in base allo strumento. Gli utenti possono utilizzare una varietà di piattaforme per acquisire le dimensioni del worm e non si limitano all'utilizzo di questo protocollo se un LPFC non è disponibile.
Gli autori stanno usando campioni coltivati nel metodo qui descritto per identificare metaboliti sconosciuti in vari ceppi di C. elegans tramite cromatografia liquida - spettrometria di massa, spettroscopia NMR e sequenziamento dell'RNA. Gli autori prevedono di continuare a utilizzare questo metodo per la crescita di campioni in questa pipeline con una varietà di ceppi C. elegans in quanto nuovi ceppi di interesse possono essere facilmente elaborati utilizzando questa pipeline.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo i membri dell'Edison Lab per le utili discussioni e feedback su questo manoscritto; in particolare, B.M. Garcia. Alcuni ceppi sono stati forniti dal CGC, che è finanziato da NIH Office of Research Infrastructure Programs (P40 OD010440), e CeNDR, che è finanziato da NSF Living Collections CSBR 1930382. Questo lavoro è stato sostenuto da una sovvenzione del NIH (U2CES030167).
Materiali
| Name | Company | Catalog Number | Comments |
| 10 mL Sterile Serological Pipettes | VWR | 89130-898 | |
| 10 ul pipette tips | VWR | 89079-438 | |
| 100 ul pipette tips | VWR | 89079-442 | |
| 1000 mL Graduated Cylinder | VWR | 10124-380 | |
| 1000 ul pipette tips | VWR | 89079-488 | |
| 15 mL conical tubes | VWR | 89039-668 | |
| 190 Proof Ethanol | VWR | 89125-166 | |
| 2 L Wide Neck Erlenmeyer Flask | VWR | 75804-654 | |
| 50 mL conical tubes | VWR | 75874-294 | |
| Agar | Sigma | 05040-100G | |
| Agarose | Sigma | A9539-500G | |
| BVC Control G Fluid Aspiration System | Vacuubrand | ||
| Calcium Chloride | Sigma | 449709-10G | |
| Cholesterol | Sigma | C3045-25G | |
| Clorox Bleach | VWR | 89414-502 | |
| Conviron Control Temperature Room | Conviron | https://www.conviron.com/environmental-rooms | |
| Corning Low Volume 384 Well Black with Clear Flat Bottom Polystyrene TC-Treated Microplate | VWR | 89089-866 | |
| Fisher Scientific Accuspin 3R | Fisher | ||
| Flat-Bottom 24-Well Plate | VWR | 29443-952 | |
| Honeywell True HEPA Purifier 465 sq ft. | Home Depot | 204390560 | |
| HT115 E. coli (DE3) | CGC | HT115(DE3) | https://cgc.umn.edu/strain/HT115(DE3) |
| Kimwipes | VWR | 470224-038 | |
| Large Scale Culture Plate (LSCP) | Pyrex | 1090948 | Pyrex 2-quart Glass Baking Dish with Red Lid |
| Magnesium Sulfate | Sigma | C86677-25G | |
| MgSO4 | VWR | 97062-998 | |
| Microscope Plain Slides | VWR | 16004-422 | |
| Millipore Filter | Millipore | 1.11727.2500 | |
| Molecular Devices ImageXpress | Molecular Devices | Model Number:IXMConfocal | https://www.moleculardevices.com/products/cellular-imaging-systems/high-content-imaging/imagexpress-micro-confocal#gref , Authors used MetaXpress Software Version 6.5.4.532 |
| Nystatin (10mg/mL) | Sigma | N6261-25MU | |
| Peptone | Sigma | P7750-100G | |
| Petri Dishes (6 cm) | VWR | 25384-092 | |
| Pipette Controller | VWR | 613-4180 | |
| Potassium Chloride | Fisher | P217-3 | |
| Potassium Phosphate Monobasic | VWR | 0781-500G | |
| Potasssium Hydroxide | Fisher | P250-500 | |
| Red Fluroscent Microspheres | Polysciences | 19507-5 | |
| Sodium Chloride | Sigma | 746398-500G | |
| Sodium Hydroxide | Fisher | 111357 | |
| Sodium Phosphate Dibasic Anhydrous | Fisher | BP332-500 | |
| Standard Gilson Pipette Set | Gilson | FA10002M, FA10004M, FA10006M | |
| Streptomycin (100mg/mL) | Sigma | S6501-25G | |
| Union Biometrica COPAS BioSorter | Union Biometrica | https://www.unionbio.com/biosorter/ , authors used: Flow Pilot software version 1.6.1.3. |
Riferimenti
- Félix, M. A., Braendle, C. The natural history of Caenorhabditis elegans. Current Biology. 20 (22), 965-969 (2010).
- Corsi, A. K. A Transparent window into biology: A primer on Caenorhabditis elegans. WormBook. , 1-31 (2015).
- Brenner, S. The genetics of behaviour. British Medical Bulletin. 29 (3), 269-271 (1973).
- Stiernagle, T. Maintenance of C. elegans. WormBook: The Online Review of C. elegans Biology. , (2006).
- Xiong, H., Pears, C., Woollard, A. An enhanced C. elegans based platform for toxicity assessment. Scientific Reports. 7 (1), 9839 (2017).
- Loer, C. M., et al. Cuticle integrity and biogenic amine synthesis in Caenorhabditis elegans require the cofactor tetrahydrobiopterin (BH4). Genetics. 200 (1), 237-253 (2015).
- Li, Y., Paik, Y. K. A potential role for fatty acid biosynthesis genes during molting and cuticle formation in Caenorhabditis elegans. BMB Reports. 44 (4), 285-290 (2011).
- Meli, V. S., Osuna, B., Ruvkun, G., Frand, A. R. MLT-10 Defines a Family of DUF644 and Proline-rich Repeat Proteins Involved in the Molting Cycle of Caenorhabditis elegans. Molecular Biology of the Cell. 21 (10), 1648-1661 (2010).
- Fritz, J. A., Behm, C. A. CUTI-1: A novel tetraspan protein involved in C. elegans CUTicle formation and epithelial integrity. PloS One. 4 (4), 5117 (2009).
- Golden, J., Riddle, D. A pheromone influences larval development in the nematode Caenorhabditis elegans. Science. 218 (4572), 578-580 (1982).
- Jeong, P. Y., et al. Chemical structure and biological activity of the Caenorhabditis elegans dauer-inducing pheromone. Nature. 433 (7025), 541-545 (2005).
- Srinivasan, J., et al. A blend of small molecules regulates both mating and development in Caenorhabditis elegans. Nature. 454 (7208), 1115-1118 (2008).
- Kaplan, F., et al. Ascaroside Expression in Caenorhabditis elegans Is Strongly Dependent on Diet and Developmental Stage. PLoS One. 6 (3), 17804 (2011).
- Golden, J., Riddle, D. A pheromone influences larval development in the nematode Caenorhabditis elegans. Science. 218 (4572), 578-580 (1982).
- Çelen, &. #. 3. 0. 4. ;., Doh, J. H., Sabanayagam, C. R. Effects of liquid cultivation on gene expression and phenotype of C. elegans. BMC Genomics. 19 (1), 562 (2018).
- Andersen, E. C., Bloom, J. S., Gerke, J. P., Kruglyak, L. A Variant in the Neuropeptide Receptor npr-1 is a Major Determinant of Caenorhabditis elegans Growth and Physiology. PLoS Genetics. 10 (2), 1004156 (2014).
- Rosenblatt, M. A central limit theorem and a strong mixing condition. Proceedings of the National Academy of Sciences. 42 (1), 43-47 (1956).
- Boyd, W. A., Smith, M. V., Freedman, J. H. Caenorhabditis elegans as a model in developmental toxicology. Methods in Molecular Biology. 889, 15-24 (2012).
- Nika, L., Gibson, T., Konkus, R., Karp, X. Fluorescent Beads Are a Versatile Tool for Staging Caenorhabditis elegans in Different Life Histories. Genes, Genomes, Genetics. 6 (7), 1923-1933 (2016).
- Denver, D. R., Morris, K., Streelman, J. T., Kim, S. K., Lynch, M., Thomas, W. K. The transcriptional consequences of mutation and natural selection in Caenorhabditis elegans. Nature Genetics. 37 (5), 544-548 (2005).
- Massie, M. R., Lapoczka, E. M., Boggs, K. D., Stine, K. E., White, G. E. Exposure to the metabolic inhibitor sodium azide induces stress protein expression and thermotolerance in the nematode Caenorhabditis elegans. Cell Stress & Chaperones. 8 (1), 1-7 (2003).
- Kevin, S., Pulak, R., Strange, K. Techniques for Analysis, Sorting, and Dispensing of C. elegans on the COPAS Flow-Sorting System. C. elegans. Methods in Molecular Biology. 351, 275-286 (2006).
- Lee, D., et al. Selection and gene flow shape niche-associated variation in pheromone response. Nature Ecology & Evolution. 3 (10), 1455-1463 (2019).
- Cook, D. E., Zdraljevic, S., Roberts, J. P., Andersen, E. C. CeNDR, the Caenorhabditis elegans natural diversity resource. Nucleic Acids Research. 45, 650-657 (2016).
- Nika, L., Gibson, T., Konkus, R., Karp, X. Fluorescent Beads Are a Versatile Tool for Staging Caenorhabditis elegans in Different Life Histories. Genes, Genomes, Genetics. 6 (7), 1923-1933 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon