È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Cromatografia di esclusione dimensionale per analizzare l'eterogeneità delle vescicole della membrana esterna batterica
In questo articolo
Riepilogo
Le vescicole batteriche svolgono un ruolo importante nella patogenesi e hanno promettenti applicazioni biotecnologiche. L'eterogeneità delle vescicole complica l'analisi e l'uso; pertanto, è necessario un metodo semplice e riproducibile per separare le varie dimensioni delle vescicole. Qui, dimostriamo l'uso della cromatografia di esclusione dimensionale per separare vescicole eterogenee prodotte da Aggregatibacter actinomycetemcomitans.
Abstract
La parete cellulare dei batteri Gram-negativi è costituita da una membrana interna (citoplasmatica) ed esterna (OM), separata da un sottile strato di peptidoglicano. Durante la crescita, la membrana esterna può sanguinare per formare vescicole sferiche della membrana esterna (OMV). Questi OMV sono coinvolti in numerose funzioni cellulari tra cui la consegna del carico alle cellule ospiti e la comunicazione con le cellule batteriche. Recentemente, il potenziale terapeutico delle OMV ha iniziato a essere esplorato, compreso il loro uso come vaccini e veicoli per la somministrazione di farmaci. Sebbene gli OMV siano derivati dall'OM, è stato a lungo apprezzato che il carico lipidico e proteico dell'OMV differisce, spesso in modo significativo, da quello dell'OM. Più recentemente, sono state scoperte prove che i batteri possono rilasciare più tipi di OMV e esistono prove che le dimensioni possono influire sul meccanismo del loro assorbimento da parte delle cellule ospiti. Tuttavia, gli studi in questo settore sono limitati dalle difficoltà nel separare in modo efficiente le OMV di dimensioni eterogenee. La centrifugazione a gradiente di densità (DGC) è stata tradizionalmente utilizzata per questo scopo; tuttavia, questa tecnica richiede molto tempo ed è difficile da scalare. La cromatografia ad esclusione dimensionale (SEC), d'altra parte, è meno ingombrante e si presta al necessario scale-up futuro per l'uso terapeutico delle OMV. Qui, descriviamo un approccio SEC che consente la separazione riproducibile di vescicole di dimensioni eterogenee, utilizzando come caso di prova, OMV prodotti da Aggregatibacter actinomycetemcomitans, che variano in diametro da meno di 150 nm a più di 350 nm. Dimostriamo la separazione tra OMV "grandi" (350 nm) e OMV "piccole" (<150 nm), verificate mediante diffusione dinamica della luce (DLS). Raccomandiamo tecniche basate su SEC rispetto a tecniche basate su DGC per la separazione di vescicole di dimensioni eterogenee grazie alla sua facilità d'uso, riproducibilità (incluso user-to-user) e possibilità di scale-up.
Introduzione
I batteri Gram-negativi rilasciano vescicole derivate dalla loro membrana esterna, le cosiddette vescicole di membrana esterna (OMV), durante la crescita. Questi OMV svolgono un ruolo importante nella comunicazione cellula-cellula, sia tra batteri e ospite che tra cellule batteriche, trasportando una serie di importanti biomolecole, tra cui DNA / RNA, proteine, lipidi e peptidoglicani1,2. In particolare, il ruolo delle OMV nella patogenesi batterica è stato ampiamente studiato a causa del loro arricchimento in alcuni fattori di virulenza e tossine3,4,5,6,7,8,9,10,11.
È stato riportato che le OMV variano in dimensioni da 20 a 450 nm, a seconda dei batteri genitori e dello stadio di crescita, con diversi tipi di batteri che rilasciano OMV di dimensioni eterogenee8,12,13,14,che differiscono anche nella loro composizione proteica e nel meccanismo di ingresso delle cellule ospiti12. H. pylori ha rilasciato OMV di diametro compreso tra 20 e 450 nm, con gli OMV più piccoli contenenti una composizione proteica più omogenea rispetto agli OMV più grandi. È importante sottolineare che le due popolazioni di OMV sono state osservate per essere internalizzate dalle cellule ospiti attraverso diversi meccanismi12. Inoltre, abbiamo dimostrato che l'Aggregatibacter actinomycetemcomitans rilascia una popolazione di piccole OMV (<150 nm) insieme a una popolazione di OMV di grandi dimensioni (>350 nm), con gli OMV contenenti una quantità significativa di una tossina proteica secreta, leucotossina (LtxA)15. Mentre il ruolo dell'eterogeneità OMV nei processi cellulari è chiaramente importante, le difficoltà tecniche nel separare e analizzare popolazioni distinte di vescicole hanno limitato questi studi.
Oltre alla loro importanza nella patogenesi batterica, gli OMV sono stati proposti per l'uso in una serie di applicazioni biotecnologiche, tra cui come vaccini e veicoli per la somministrazione di farmaci16,17,18,19,20. Per il loro uso traslazionale in tali approcci, è necessaria una preparazione pulita e monodispersa di vescicole. Pertanto, sono necessari metodi di separazione efficaci ed efficienti.
Più comunemente, la centrifugazione a gradiente di densità (DGC) viene utilizzata per separare popolazioni di vescicole di dimensioni eterogenee dai detriti cellulari, compresi i flagelli e le proteine secrete21; il metodo è stato anche segnalato come approccio per separare sottopopolazioni OMV di dimensioni eterogenee12,13,14. Tuttavia, DGC richiede molto tempo, è inefficiente e altamente variabile da utente a utente22 e, pertanto, non è l'ideale per lo scale-up. Al contrario, la cromatografia di esclusione dimensionale (SEC) rappresenta un approccio scalabile, efficiente e coerente per purificare gli OMV21,23,24. Abbiamo scoperto che una colonna SEC lunga (50 cm), a flusso gravitazionale, riempita con un mezzo di filtrazione in gel è sufficiente per purificare e separare in modo efficiente le sottopopolazioni di OMV. In particolare, abbiamo utilizzato questo approccio per separare gli OMV di A. actinomicetimmcomitani in sottopopolazioni "grandi" e "piccole", nonché per rimuovere la contaminazione da proteine e DNA. La purificazione è stata completata in meno di 4 ore ed è stata completata la completa separazione delle sottopopolazioni OMV e la rimozione dei detriti.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Preparazione dei buffer
- Per preparare il tampone di lavaggio ELISA, aggiungere 3,94 g di Tris-base, 8,77 g di NaCl e 1 g di albumina sierica bovina (BSA) a 1 L di acqua deionizzata (DI). Aggiungere 500 μL di polisorbato-20. Regolare il pH a 7,2 utilizzando HCl o NaOH.
- Per preparare il buffer di blocco, aggiungere 3,94 g di Tris-base, 8,77 g di NaCl e 10 g di BSA. Aggiungere 500 μL di polisorbato-20 a 1 L di acqua DI. Regolare il pH a 7,2 utilizzando HCl o NaOH.
- Per preparare il tampone di eluizione (PBS), aggiungere 8,01 g di NaCl, 2,7 g di KCl, 1,42 g di Na2HPO4e 0,24 g di KH2POda 4 a 1 L di acqua DI. Regolare il pH a 7,4 utilizzando HCl o NaOH.
NOTA: Una soluzione 10x di questo tampone può essere prodotta e diluita con acqua DI secondo necessità.
2. Preparazione del campione OMV
- Crescere le cellule di A. actinomycetemcomitans fino alla fase esponenziale tardiva (densità ottica a 600 nm di 0,7). Pellet le celle centrifugando due volte a 10.000 x g a 4 °C per 10 min. Filtrare il surnatante attraverso un filtro da 0,45 μm.
- Concentrare il surnatante privo di batteri utilizzando filtri cut-off a peso molecolare da 50 kDa. Ultracentrifificare la soluzione concentrata a 105.000 x g a 4 °C per 30 min.
- Ripresa del pellet in PBS e ultracentrifuga di nuovo (105.000 x g a 4 °C per 30 min.) Rispendare il pellet in 2 ml di PBS.
3. Imballaggio della colonna S-1000
- Mescolare la bottiglia di terreno filtrante gel con un'asta di vetro e versare in una bottiglia di vetro il volume necessario per riempire la colonna, più circa il 50% in eccesso (circa 135 ml). Lascia riposare queste perle fino a quando non si sono depositate, quindi decantare il liquido in eccesso. Spese di sospensione delle perle nel tampone di eluizione, in modo che la soluzione finale sia circa il 70% (in volume) di gel, il 30% di tampone. Degassare la soluzione sotto vuoto.
- Montare la colonna di vetro verticalmente usando un supporto ad anello e riempire con un tampone di eluizione per bagnare le pareti della colonna. Scaricare il tampone fino a quando non rimane solo circa 1 cm di buffer nella colonna.
- Senza creare bolle, pipettare con cura le perle nella colonna, riempiendo la colonna verso l'alto. Continuare a drenare il buffer in eccesso durante questo processo. Assicurati di non lasciare che le perle si depositino completamente prima di aggiungere ulteriori perle nella parte superiore della colonna. La colonna deve essere imballata ad un'altezza di circa 2 cm sotto il fondo del serbatoio della colonna.
4. Caricamento del campione e raccolta delle frazioni
- Degassare il tampone di eluizione sotto vuoto. Lavare la colonna con due volumi di colonne (180 ml) di tampone di eluizione.
- Consentire al buffer rimanente di entrare completamente nella colonna. Una volta che il tampone ha raggiunto la parte superiore dello strato di gel, pipettare con cura un campione di 2 ml contenente OMV (ad una concentrazione lipidica di circa 100 - 200 nmol / L) sulla superficie delle perle, facendo attenzione a non disturbare nessuna delle perle nella parte superiore della colonna. Lasciare che il campione entri completamente nel gel, cioè quando nessun liquido rimane sopra lo strato di gel.
- Aggiungere con attenzione e lentamente il tampone di eluizione sulla parte superiore della colonna di gel. Non disturbare lo strato superiore del gel, in quanto ciò causerà la diluizione del campione.
- Posizionare un singolo tubo da 50 ml sotto la colonna e aprire la colonna. Raccogli i primi 20 ml dell'eluente. Aggiungere un ulteriore tampone di eluizione nella parte superiore della colonna, con attenzione, se necessario, per garantire che la colonna non sia mai asciutta.
- Posizionare una serie di tubi da 1,5 ml sotto la colonna. Avviare la colonna e raccogliere una serie di campioni da 1 ml in ogni tubo. Mentre i campioni vengono raccolti, continuare ad aggiungere il buffer di eluizione nella parte superiore della colonna, se necessario. Ripetere fino a quando non sono state raccolte 96 frazioni. Arrestare la colonna.
NOTA: i campioni devono essere conservati a -20 °C per la conservazione a lungo termine o a 4 °C per la conservazione a breve termine fino a ulteriori analisi. - Per pulire la colonna, eseguire un volume di colonna (90 ml) di 0,1 M NaOH attraverso la colonna. Eseguire due volumi di colonne (180 ml) di buffer di eluizione attraverso la colonna.
5. Analisi del campione
- Per misurare la concentrazione lipidica in ogni frazione, pipettare 50 μL di ciascuna frazione in un singolo pozzettino di una piastra a 96 pozzetti. A ciascun pozzo, aggiungere 2,5 μL di colorante lipofilo. Incubare per 15 s. Misurare l'intensità della fluorescenza su un lettore di piastre con una lunghezza d'onda di eccitazione di 515 nm e una lunghezza d'onda di emissione di 640 nm. Per calcolare la frazione di tutti i lipidi in ciascun campione, sommare tutte le intensità di emissione e dividere ogni singola intensità per il totale.
- Per misurare la concentrazione di una particolare proteina, pipettare 100 μL di ogni frazione in un singolo pozzetto di una immunopiastra ELISA. Incubare a 25 °C per 3 ore.
- Decantare i campioni. Aggiungere 200 μL di tampone di lavaggio ELISA a ciascun pozzesso e decantare. Ripeti quattro volte per un totale di cinque lavaggi.
- Aggiungere 200 μL di tampone bloccante a ciascun pozzo e incubare per 1 ora a 25 °C. Travasare.
- Incubare piastre con tampone bloccante da 100 μL più anticorpo primario (1:10.000 per anticorpi purificati; 1:10 per anticorpi non purificati) durante la notte a 4 °C. Travasare.
- Aggiungere 200 μL di tampone di lavaggio ELISA a ciascun pozzesso e decantare. Ripeti quattro volte per un totale di cinque lavaggi.
- Aggiungere 100 μL di tampone di lavaggio ELISA più anticorpo secondario (1:30.000) a ciascun pozzezze. Incubare per 1 ora a 25 °C.
- Aggiungere 200 μL di tampone di lavaggio ELISA a ciascun pozzesso e decantare. Ripeti quattro volte per un totale di cinque lavaggi.
- Aggiungere 100 μL della soluzione monosezione da 3,3',5,5'-tetrametilbenzidina (TMB) e incubare per 15-30 minuti o fino a quando non si sviluppa un colore blu. Arrestare la reazione TMB con 50 μL della soluzione di arresto.
- Su un lettore di piastre, leggere l'assorbanza di ciascun pozzo a una lunghezza d'onda di 450 nm.
- Per misurare la concentrazione totale di proteine, registrare l'assorbanza a una lunghezza d'onda di 280 nm (A280) di ciascuna frazione, utilizzando uno spettrofotometro UV-vis.
Uno schema del protocollo è mostrato nella Figura 1.
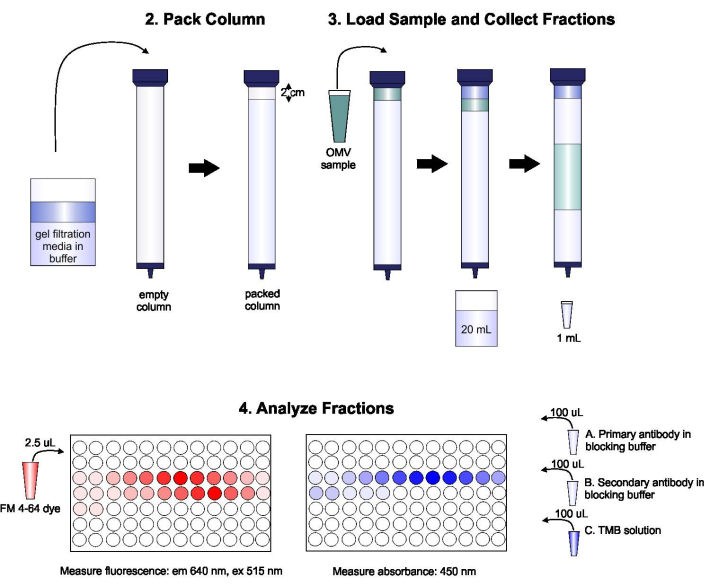
Figura 1: Schema della procedura SEC. La colonna è imballata con un mezzo filtrante in gel degassato con cura per evitare bolle e discontinuità, quindi lavata con due volumi di colonna di tampone di eluizione. Successivamente, il campione viene accuratamente pipettato sulla parte superiore del gel, senza interrompere l'imballaggio del gel. La colonna viene aperta ed eseguita fino a quando il campione non entra completamente nel gel. A questo punto, il buffer viene posizionato nella parte superiore della colonna e vengono raccolti i primi 20 ml di eluato. Successivamente, viene raccolta una serie di frazioni da 1 ml. Queste frazioni vengono quindi poste in una piastra a 96 pozzetti o in una piastra immuno a 96 pozzetti per l'analisi del contenuto lipidico e proteico. Fare clic qui per visualizzare una versione più grande di questa figura.
Access restricted. Please log in or start a trial to view this content.
Risultati
La Figura 2 mostra i risultati rappresentativi di questo metodo. Gli OMV prodotti da A. actinomycetemcomitans ceppo JP2 sono stati purificati per la prima volta dal surnatante di coltura utilizzando l'ultracentrifugazione15. In precedenza abbiamo scoperto che questo ceppo produce due popolazioni di OMV, una con diametri di circa 300 nm e una con diametri di circa 100 nm15. Per separare queste popolazioni OMV, abbiamo purificato il camp...
Access restricted. Please log in or start a trial to view this content.
Discussione
Qui, abbiamo fornito un protocollo per la separazione semplice, veloce e riproducibile delle sottopopolazioni OMV batteriche. Sebbene la tecnica sia relativamente semplice, ci sono alcuni passaggi che devono essere eseguiti con estrema attenzione per garantire che si verifichi una separazione efficiente nella colonna. In primo luogo, è essenziale che il gel venga caricato nella colonna con attenzione e lentamente per evitare bolle d'aria. Abbiamo osservato che lasciare il gel a temperatura ambiente per diverse ore prima...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno conflitti di interesse da segnalare.
Riconoscimenti
Questo lavoro è stato finanziato dalla National Science Foundation (1554417) e dal National Institutes of Health (DE027769).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 1-Step Ultra TMB-ELISA | Thermo Scientific | 34028 | |
| Amicon 50 kDa filters | Millipore Sigma | UFC905024 | |
| Bovine Serum Albumin (BSA) | Fisher Scientific | BP9704-100 | |
| ELISA Immuno Plates | Thermo Scientific | 442404 | |
| FM 4-64 | Thermo Scientific | T13320 | 1.5 x 50 cm |
| Glass Econo-Column | BioRad | 7371552 | |
| Infinite 200 Pro Plate Reader | Tecan | ||
| Potassium Chloride (KCl) | Amresco (VWR) | 0395-500G | |
| Potassium Phosphate Monobasic Anhydrous (KH2PO4) | Amresco (VWR) | 0781-500G | |
| Sephacryl S-1000 Superfine | GE Healthcare | 17-0476-01 | |
| Sodium Chloride (NaCl) | Fisher Chemical | S271-3 | |
| Sodium Phosphate Dibasic Anhydrous (Na2HPO4) | Amresco (VWR) | 0404-500G | |
| Tris Base | VWR | 0497-1KG | |
| Tween(R) 20 | Acros Organics | 23336-2500 |
Riferimenti
- Kuehn, M. J., Kesty, N. C. Bacterial outer membrane vesicles and the host-pathogen interaction. Genes and Development. 19, 2645-2655 (2005).
- Kulp, A., Kuehn, M. J. Biological functions and biogenesis of secreted bacterial outer membrane vesicles. Annual Reviews Microbiology. 64, 163-184 (2010).
- Kato, S., Kowashi, Y., Demuth, D. R. Outer membrane-like vesicles secreted by Actinobacillus actinomycetemcomitans are enriched in leukotoxin. Microbial Pathogenesis. 32 (1), 1-13 (2002).
- Nice, J. B., et al. Aggregatibacter actinomycetemcomitans leukotoxin is delivered to host cells in an LFA-1-independent manner when associated with outer membrane vesicles. Toxins. 10 (10), 414(2018).
- Haurat, M. F., et al. Selective sorting of cargo proteins into bacterial membrane vesicles. Journal of Biological Chemistry. 286 (2), 1269-1276 (2011).
- Horstman, A. L., Kuehn, M. J. Enterotoxigenic Escherichia coli secretes active heat-labile enterotoxin via outer membrane vesicles. The Journal of Biological Chemistry. 275 (17), 12489-12496 (2000).
- Wai, S. N., et al. Vesicle-mediated export and assembly of pore-forming oligomers of the Enterobacterial ClyA cytotoxin. Cell. 115, 25-35 (2003).
- Balsalobre, C., et al. Release of the Type I secreted α-haemolysin via outer membrane vesicles from Escherichia coli. Molecular Microbiology. 59 (1), 99-112 (2006).
- Donato, G. M., et al. Delivery of Bordetella pertussis adenylate cyclase toxin to target cells via outer membrane vesicles. FEBS Letters. 586, 459-465 (2012).
- Kim, Y. R., et al. Outer membrane vesicles of Vibrio vulnificus deliver cytolysin-hemolysin VvhA into epithelial cells to induce cytotoxicity. Biochemical and Biophysical Research Communications. 399, 607-612 (2010).
- Maldonado, R., Wei, R., Kachlany, S. C., Kazi, M., Balashova, N. V. Cytotoxic effects of Kingella kingae outer membrane vesicles on human cells. Microbial Pathogenesis. 51 (1-2), 22-30 (2011).
- Turner, L., et al. Helicobacter pylori outer membrane vesicle size determines their mechanisms of host cell entry and protein content. Frontiers in Immunology. 9, 1466(2018).
- Zavan, L., Bitto, N. J., Johnston, E. L., Greening, D. W., Kaparakis-Liaskos, M. Helicobacter pylori growth stage determines the size, protein composition, and preferential cargo packaging of outer membrane vesicles. Proteomics. 19 (1-2), 1800209(2019).
- Rompikuntal, P. K., et al. Perinuclear localization of internalized outer membrane vesicles carrying active cytolethal distending toxin from Aggregatibacter actinomycetemcomitans. Infections and Immunity. 80 (1), 31-42 (2012).
- Nice, J. B., et al. Aggregatibacter actinomycetemcomitans leukotoxin is delivered to host cells in an LFA-1-independent manner when associated with outer membrane vesicles. Toxins. 10 (10), 414(2018).
- Stevenson, T. C., et al. Immunization with outer membrane vesicles displaying conserved surface polysaccharide antigen elicits broadly antimicrobial antibodies. Proceedings of the National Academy of Sciences. 115 (14), 3106-3115 (2018).
- Gujrati, V., et al. Bioengineered bacterial outer membrane vesicles as cell-specific drug-delivery vehicles for cancer therapy. ACS Nano. 8 (2), 1525-1537 (2014).
- Huang, W., et al. Development of novel nanoantibiotics using an outer membrane vesicle-based drug efflux mechanism. Journal of Controlled Release. 317, 1-22 (2020).
- Chen, D. J., et al. Delivery of foreign antigens by engineered outer membrane vesicle vaccines. Proceedings of the National Academy of Sciences. 107 (7), 3099-3104 (2010).
- Chen, L., et al. Outer membrane vesicles displaying engineered glycotopes elicit protective antibodies. Proceedings of the National Academy of Sciences. 113 (26), 3609-3618 (2016).
- Singorenko, P. D., et al. Isolation of membrane vesicles from prokaryotes: A technical and biological comparison reveals heterogeneity. Journal of Extracellular Vesicles. 6 (1), 1324731(2017).
- Zeringer, E., Barta, T., Li, M., Vlassov, A. V. Strategies for isolation of exosomes. Cold Spring Harbor Protocols. 2015 (4), 319-323 (2015).
- Benedikter, B. J., et al. Ultrafiltration combined with size exclusion chromatography efficiently isolates extracellular vesicles from cell culture media for compositional and functional studies. Science Reports. 7 (1), 15297(2017).
- Mol, E. A., Goumans, M. J., Doevendans, P. A., Sluijter, J. P. G., Vader, P. Higher functionality of extracellular vesicles isolated using size-exclusion chromatography compared to ultracentrifugation. Nanomedicine. 13 (6), 2061-2065 (2017).
- Lally, E. T., Golub, E. E., Kieba, I. R. Identification and immunological characterization of the domain of Actinobacillus actinomycetemcomitans leukotoxin that determines its specificity for human target cells. Journal of Biological Chemistry. 269 (49), 31289-31295 (1994).
- Chang, E. H., Giaquinto, P., Huang, J., Balashova, N. V., Brown, A. C. Epigallocatechin gallate inhibits leukotoxin release by Aggregatibacter actinomycetemcomitans by promoting association with the bacterial membrane. Molecular Oral Microbiology. 35 (1), 29-39 (2020).
- Klimentová, J., Stulík, J. Methods of isolation and purification of outer membrane vesicles from gram-negative bacteria. Microbiological Research. 170, 1-9 (2015).
- Dauros Singorenko, P., et al. Isolation of membrane vesicles from prokaryotes: a technical and biological comparison reveals heterogeneity. Journal of Extracellular Vesicles. 6 (1), 1324731(2017).
- Monguió-Tortajada, M., Gálvez-Montón, C., Bayes-Genis, A., Roura, S., Borràs, F. E. Extracellular vesicle isolation methods: rising impact of size-exclusion chromatography. Cellular and Molecular Life Sciences. 76 (12), 2369-2382 (2019).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon