È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Strumenti microfluidici per sondare le interazioni fungine-microbiche a livello cellulare
In questo articolo
Riepilogo
A causa dell'opacità del suolo, le interazioni tra i suoi microbi costituenti non possono essere facilmente visualizzate con risoluzione cellulare. Qui vengono presentati due strumenti microfluidici, che offrono nuove opportunità per studiare le interazioni fungo-microbiche. I dispositivi sono versatili e semplici da usare, consentendo un elevato controllo spaziotemporale e immagini ad alta risoluzione a livello cellulare.
Abstract
I funghi filamentosi sono abitanti di successo del suolo e svolgono un ruolo importante negli ecosistemi del suolo, come nella decomposizione della materia organica e inorganica, nonché nella regolazione dei livelli di nutrienti. Lì trovano anche numerose opportunità di interagire con una varietà di altri microbi come batteri o altri funghi. Studiare le interazioni fungine a livello cellulare, tuttavia, può essere difficile a causa della natura del suolo simile a una scatola nera. Sono in fase di sviluppo nuovi strumenti microfluidici per lo studio delle interazioni fungine; sono evidenziate due piattaforme progettate per studiare le interazioni batterico-fungine e fungino-fungine. All'interno di questi microcanali, le interazioni fungo-microbiche possono essere monitorate in ambienti fisico-chimici controllati a una risoluzione temporale e spaziale più elevata di quanto fosse possibile in precedenza. L'applicazione di questi strumenti ha prodotto numerose nuove intuizioni biologiche, come l'osservazione dell'attaccamento polare batterico alle ife o la rivelazione di antagonismi fungino-fungini non caratterizzati. Una caratteristica fondamentale di queste metodologie riguarda la facilità d'uso di questo strumento da parte di non esperti, producendo tecnologie altamente traducibili per l'uso nei laboratori di microbiologia.
Introduzione
Il suolo è un ambiente eccezionalmente diversificato contenente un'abbondanza di microrganismi che sono strumentali ai cicli del carbonio e del fosforo 1,2. I funghi filamentosi sono una componente importante di numerosi ecosistemi come decompositori di materia organica e inorganica e possono migliorare la nutrizione delle piante attraverso la formazione di relazioni simbiotiche 3,4. All'interno del suolo, i funghi interagiscono dinamicamente con una moltitudine di microbi come altri funghi5, batteri6, virus7 e nematodi8. Queste interazioni hanno conseguenze significative per la salute del suolo e delle piante. Tuttavia, a causa della mancanza di sistemi sperimentali appropriati in grado di eseguire l'imaging di microrganismi interagenti ad alta risoluzione, molti rimangono indefiniti.
Le indagini riguardanti le interazioni batterico-fungine (BFI) e le interazioni fungo-fungine (FFI) hanno preziose applicazioni in una vasta gamma di campi, tra cui gli antimicrobici in medicina e gli agenti di controllo biologico in agricoltura. Ad esempio, il fungo Coprinopsis cinerea produce il peptide copsina, che ha dimostrato di esibire attività antibatterica contro il patogeno umano Listeria monocytogenes9. Allo stesso modo, il composto di derivazione fungina, griseofulvina, è ampiamente usato come trattamento per le infezioni fungine umane ed è inoltre in grado di inibire la crescita del fungo patogeno della pianta Alternaria solani10,11. Diversi ceppi del batterio Bacillus subtilis che abita nel suolo hanno anche dimostrato di essere efficaci agenti di biocontrollo del patogeno vegetale fungino Rhizoctonia solani12,13. Tuttavia, a causa delle limitazioni associate alle metodologie tradizionali, i BFI e gli FFA sono poco compresi a livello di singole cellule.
Gli studi convenzionali in genere esplorano BFI e FFI su macroscala usando placche di agar con due o più specie in confronto. La loro interazione è valutata misurando i tassi di crescita e la produzione di metaboliti delle specie in confronto 14,15,16; tuttavia, questa metodologia è risolta solo a livello di colonia. Per studiare le interazioni a livello cellulare, gli inoculanti batterici e fungini possono essere coltivati su vetrini per microscopio rivestiti di agar che vengono poi ripresi al microscopio17. Tuttavia, può essere difficile seguire una singola ifa usando vetrini per microscopio a causa della mancanza di confinamento, il che significa che le immagini time-lapse sono più difficili da ottenere. Inoltre, l'opportunità di confinare spazialmente altri microrganismi all'interno di regioni definite del micelio fungino o di creare ambienti chimici definiti che possono essere perturbati, ad esempio, non è possibile in tali configurazioni. La natura "scatola nera" del suolo aggiunge anche alla complessità dello studio delle interazioni fungo-microbiche a livello di singole cellule18. Osservando le specie interagenti lontano dall'incredibile diversità del microbioma del suolo, è possibile valutare il modo esatto in cui i singoli membri interagiscono. Pertanto, vi è una continua necessità di piattaforme versatili che consentano l'imaging ad alta risoluzione a cella singola di BFI e FFA.
Le tecnologie microfluidiche, i cosiddetti sistemi lab-on-a-chip, forniscono una piattaforma ideale per lo studio di BFI e FONI a livello di singole celle. Il campo della microfluidica, originato da tecnologie sviluppate per l'analisi chimica e la microelettronica, è stato adottato dalle scienze biologiche19. Le tecnologie microfluidiche regolano piccoli volumi di fluidi all'interno di una rete su misura di canali miniaturizzati, aventi almeno una dimensione su scala micrometrica, e il loro uso nella ricerca biologica si sta espandendodi 20. In particolare, sono stati sviluppati dispositivi microfluidici per esaminare la crescita di funghi filamentosi 21,22,23,24,25,26,27,28,29,30. Uno dei vantaggi dell'utilizzo di questa tecnologia è che il confinamento delle ife e la distribuzione dei nutrienti all'interno di microcanali assomiglia più da vicino alla struttura dell'ambiente del suolo rispetto ai metodi convenzionali di agar31. Recentemente, le piattaforme microfluidiche sono state utilizzate per studiare le interazioni tra neutrofili umani e patogeni fungini32, batteri e radici vegetali33, nonché funghi e nematodi34,35.
Uno dei molti vantaggi dell'utilizzo della microfluidica per lo studio delle interazioni microbiche include il controllo specifico dell'ambiente dei microcanali. Ad esempio, i regimi di flusso laminare possono essere sfruttati per generare gradienti di concentrazione definiti, il che è particolarmente utile quando si esamina la chemiotassi batterica36. Un altro vantaggio è che la natura trasparente del poli(dimetilsilossano) (PDMS), un polimero elastomerico economico e biocompatibile comunemente usato nella produzione di dispositivi microfluidici, facilita l'imaging ad alta risoluzione di singole cellule utilizzando la microscopia a campo luminoso e fluorescenza37. Allo stesso modo, il confinamento dei microbi all'interno di microcanali significa che possono essere eseguiti esperimenti time-lapse che tracciano singole cellule, consentendo di registrare e quantificare le singole risposte cellulari37. Infine, poiché i dispositivi microfluidici possono essere progettati per essere facili da usare, possono essere facilmente utilizzati da non esperti38.
Approfondire la conoscenza delle interazioni tra microrganismi che vivono nel suolo è importante per migliorare le pratiche di gestione sostenibile degli ecosistemi che mantengono la biodiversità e per mitigare l'impatto dei cambiamenti climatici sugli ambienti terrestri39. Pertanto, lo sviluppo di nuovi strumenti microfluidici è fondamentale per espandere la comprensione dei funghi e delle loro interazioni a livello cellulare. Il protocollo qui si concentrerà su due dispositivi microfluidici prodotti per lo studio di BFI40 e FFI41 come rappresentato nella Figura 1.

Figura 1: Rappresentazione visiva e schematica dei dispositivi di interazione batterico-fungina (BFI) e di interazione fungo-fungino (FFI). (A) Immagine del dispositivo BFI. Una spina miceliale è posizionata all'ingresso di un'estremità dei microcanali per consentire la crescita ifale nel dispositivo. L'ingresso batterico si trova all'estremità opposta. Barra della scala = 5 mm. (B) Panoramica schematica del dispositivo BFI, raffigurante il posizionamento delle prese batteriche e la direzione della crescita ifale attraverso i microcanali di interazione. I canali sono profondi 10 μm, larghi 100 μm e lunghi 7 mm, con 28 canali di osservazione in totale. (C) Saggio di confronto su piastra di agar tra Coprinopsis cinerea e Bacillus subtilis NCIB 3610, barra di scala = 20 mm (sinistra). Immagini al microscopio che mostrano l'interazione tra C. cinerea e B. subtilis NCIB 3610 all'interno del microcanale (centrale e destro), cioè l'attaccamento polare dei batteri alle ife fungine. Barra della scala = 25 μm (al centro) e 10 μm (a destra). (D) Immagine del dispositivo FFI legato a una capsula di Petri con fondo di vetro, doppia inoculata con tappi miceliali. Barra della scala = 1 cm. (E) Panoramica schematica del dispositivo FFI. Due spine inoculanti fungine vengono introdotte nelle prese d'ingresso alle due estremità del dispositivo, consentendo l'esplorazione ifale dei microcanali. I canali di controllo sono collegati a un solo ingresso fungino e hanno un canale senza uscita, impedendo le interazioni tra i funghi di prova. I canali di interazione collegano entrambe le insenature fungine e consentono interazioni ifali tra i soggetti del test all'interno del microcanale. Ogni canale di interazione è costituito da 18 sezioni a forma di diamante, che misurano una lunghezza totale di 8,8 mm (490 x 430 μm per diamante), 10 μm di profondità e hanno una regione di collegamento tra ciascun diamante di 20 μm. I tipi di canale sono duplicati, barre di scala = 1 mm. (F) Zona di interazione tra due fronti ifali in avvicinamento, che crescono da estremità opposte del canale di interazione interconnesso. Immagine al microscopio a contrasto di fase, barra di scala = 250 μm. I pannelli in questa figura sono stati modificati da Stanley et al., 2014 (A-C)40 e Gimeno et al., 2021 (D-F)41. Fare clic qui per visualizzare una versione più grande di questa figura.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Nota : un riepilogo delle procedure descritte in questo protocollo sono rappresentate visivamente nella Figura 2.
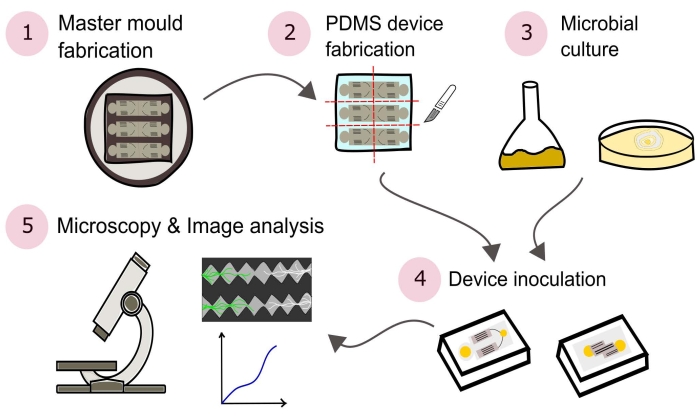
Figura 2: Rappresentazione schematica della metodologia presentata composta da cinque sezioni principali dettagliate in questo protocollo. I progetti dei dispositivi vengono creati utilizzando un software CAD (Computer Aided Design) e uno stampo master prodotto con fotolitografia (1). Questo viene utilizzato per fondere il poli(dimetilsilossano) (PDMS), che viene poi tagliato a dadini in lastre e legato a piastre di Petri con fondo di vetro per formare i dispositivi microfluidici (2). I microbi da includere nello studio vengono coltivati (3) e utilizzati per inoculare i dispositivi (4). Le interazioni sono studiate al microscopio e quantificate utilizzando tecniche di analisi delle immagini (5). Fare clic qui per visualizzare una versione più grande di questa figura.
1. Fabbricazione dello stampo principale
- Produzione di maschere fotografiche
- Genera progetti di dispositivi microfluidici utilizzando il software CAD (Computer Aided Design). Le dimensioni dei dispositivi presentati sono riportate nella Figura 1 e maggiori dettagli sulle caratteristiche specifiche del design sono elencati in modo completo nelle rispettive pubblicazioni40,41.
- Esportare il file di progettazione CAD utilizzando un formato appropriato (ad esempio, .dwg, .dxf). Stampare una maschera fotolitografica su pellicola inviando il file di progettazione CAD esportato a un fornitore commerciale per la stampa.
- Fotolitografia
NOTA: i seguenti passaggi devono essere eseguiti all'interno di un ambiente privo di polvere e controllato dalla luce, come una cappa a flusso laminare o una camera bianca. Le condizioni sperimentali qui fornite sono fornite come guida e dovrebbero essere ottimizzate internamente. Gli autori raccomandano di cercare una formazione specifica e di consultare i protocollistabiliti 42.- Preparare un wafer di silicio da 100 mm cuocendo in forno a 200 °C per 2 ore. Spin-coat il wafer di silicio con fotoresist SU-8 2010, puntando ad uno spessore target di 10 μm, utilizzando le seguenti condizioni: 500 rpm per 10 s (accelerazione 100 rpm/s) e 3.000 rpm per 45 s (accelerazione 300 rpm/s).
ATTENZIONE: il fotoresist SU-8 è pericoloso, fare attenzione durante la manipolazione e prevenire l'inalazione e il contatto con la pelle. È infiammabile, potenzialmente cancerogeno e tossico per l'ambiente. - Cuocere il wafer di silicio rivestito a 95 °C per 2,5 minuti (cottura morbida). Esporre il fotoresiste alla luce ultravioletta (UV), utilizzando la maschera fotolitografica a pellicola e una dose di energia di 140 mJ/cm2 a una lunghezza d'onda di 365 nm utilizzando un allineatore a maschera.
- Cuocere il wafer di silicio rivestito a 95 °C per 3,5 minuti (cottura post esposizione). Immergere e agitare il wafer di silicio in una soluzione di sviluppo per 3 minuti per rivelare le strutture microfabbricate rimuovendo il fotoresist non esposto.
ATTENZIONE: la soluzione per sviluppatori può essere infiammabile, prendere le precauzioni appropriate durante la manipolazione e l'archiviazione. - Risciacquare con una nuova soluzione per sviluppatori per 10 s. Risciacquare con alcool isopropilico per 10 s e asciugare all'aria. Utilizzare aria compressa filtrata per garantire che le strutture siano completamente asciutte. Misurare l'altezza delle strutture SU-8, ad esempio utilizzando un profilometro.
- Silanizzare ogni stampo master con 50 μL di clorotrimetilsilano applicando una pressione di vuoto di 50 mbar per 2 ore. Gli autori osservano che la ri-silanizzazione dello stampo principale non è risultata necessaria.
ATTENZIONE: Il clorotrimetilsilano è una sostanza pericolosa. Indossare adeguati dispositivi di protezione individuale (DPI) e maneggiare con cura. Evitare il contatto con la pelle e gli occhi e prevenire l'inalazione. Tenere lontano da fonti di accensione e utilizzare in un'area ben ventilata.
- Preparare un wafer di silicio da 100 mm cuocendo in forno a 200 °C per 2 ore. Spin-coat il wafer di silicio con fotoresist SU-8 2010, puntando ad uno spessore target di 10 μm, utilizzando le seguenti condizioni: 500 rpm per 10 s (accelerazione 100 rpm/s) e 3.000 rpm per 45 s (accelerazione 300 rpm/s).
2. Fabbricazione del dispositivo
NOTA: i seguenti passaggi devono essere eseguiti all'interno di un ambiente privo di polvere, ad esempio una cappa a flusso laminare.
- Preparazione di lastre di poli(dimetilsilossano) (PDMS)
- Preparare circa 40 g di PDMS mescolando accuratamente la base e l'agente polimerizzante in un rapporto 10:1 usando una spatola in un bicchiere di plastica pulito. Degassare la miscela per rimuovere tutte le bolle d'aria posizionando il bicchiere di plastica contenente il PDMS in una camera a vuoto (pressione del vuoto = 50 mbar) per 1 ora.
- Fissare lo stampo principale in un supporto di plastica utilizzando nastro trasparente. Pulire utilizzando aria filtrata compressa per rimuovere eventuali particelle di polvere.
NOTA: In alternativa, il foglio di alluminio può essere modellato attorno a una capsula di Petri di vetro e quindi utilizzato per ospitare lo stampo principale e contenere il PDMS43. - Versare la miscela PDMS sul centro dello stampo master, assicurandosi che sia su una superficie piana e lasciare depositare.
NOTA: La miscela PDMS deve essere versata il più vicino possibile alla superficie dello stampo master e un flusso continuo mantenuto per ridurre al minimo l'introduzione di bolle d'aria. Le bolle d'aria possono essere rimosse dirigendo l'aria compressa sulla bolla o estraendole usando un ago sottile. - Coprire liberamente lo stampo principale con un coperchio di plastica per evitare che le particelle di polvere si depositino sulla superficie del PDMS. Trasferire lo stampo master in forno e polimerizzare durante la notte a 70 °C.
- Togliere lo stampo master dal forno e lasciare raffreddare. Staccare il PDMS polimerizzato dallo stampo master e dal telaio in plastica, avendo cura di evitare di danneggiare lo stampo master / PDMS.
- Posizionare del nastro trasparente sui microcanali in rilievo nel PDMS per mantenere una superficie priva di polvere. Assicurarsi che il nastro venga rimosso prima dell'incollaggio.
- Tagliare il PDMS in lastre (cioè, se più dispositivi sono inclusi nel progetto sullo stampo principale, molti possono essere fabbricati da una singola fusione) come indicato dal progetto utilizzando una ghigliottina montata o una lama di rasoio. Quando si taglia l'apertura laterale della lastra BFI PDMS, assicurarsi che i microcanali siano completamente aperti (Figura 1A). Per la lastra FFI PDMS, assicurarsi che ogni angolo sia tagliato per consentirgli di adattarsi alla capsula di Petri con fondo di vetro mostrata nella Figura 1D.
- Punzonare i fori di ingresso / uscita desiderati in base al design del dispositivo. Utilizzare una fresa di precisione per perforare fori di ingresso di 3,18 mm e 4,75 mm per i dispositivi BFI e FFI esemplari, rispettivamente.
- Incollaggio di lastre PDMS per creare dispositivi
NOTA: Le seguenti fasi di lavaggio (2.2.1-2.2.2) utilizzano un pulitore ad ultrasuoni riempito con acqua purificata (ddH2O) a 37 kHz. Il lavaggio delle lastre PDMS aiuta a migliorare il successo dell'incollaggio44 e a ridurre il rischio di contaminazione. Per manipolare le lastre PDMS, utilizzare pinze pulite e sollevare utilizzando i fori di ingresso per evitare danni ai microcanali o alla superficie del dispositivo.- Immergere le lastre PDMS in 0,5 M NaOH e sonicare per 5 min. Risciacquare con ddH2O. Trasferire lastre PDMS in soluzione di etanolo al 70% e sonicare per 5 minuti. Risciacquare con ddHsterile 20.
- Immergere le lastre PDMS in ddH2O sterile e sonicare per 5 minuti. Rimuovere le lastre PDMS dal ddH2O sterile, asciugare con aria compressa filtrata e metterle in una capsula di Petri quadrata sterile.
- Mettere la capsula di Petri quadrata contenente le lastre PDMS in un forno a 70 °C per 1 ora ad asciugare. Togliere dal forno e lasciare raffreddare in un ambiente privo di polvere. Eliminare la polvere dalla superficie delle lastre PDMS utilizzando nastro adesivo e/o aria compressa filtrata.
- Attivare le superfici delle lastre PDMS e delle piastre di Petri con fondo in vetro da incollare utilizzando un pulitore al plasma con le seguenti impostazioni: pressione del vuoto 0,75 mbar, potenza 50%, tempo di rivestimento 1 min. Posizionare le superfici da attivare (e successivamente incollare) rivolte verso l'alto nel detergente al plasma.
- Rimuovere le lastre PDMS e le piastre di Petri con fondo di vetro dal detergente al plasma e legare delicatamente le superfici attivate in contatto conforme l'una con l'altra. Legare le lastre BFI e FFI PDMS alle piastre di Petri con fondo di vetro da 35 mm e 50 mm di diametro (spessore del vetro 0,17 mm), rispettivamente.
NOTA: Fare attenzione a non applicare troppa pressione durante l'incollaggio, in quanto ciò può causare il collasso dei microcanali. - Verificare il successo dell'incollaggio semplicemente cercando di estrarre la lastra PDMS dalla piastra di Petri con fondo di vetro con una pinzetta. Visualizza i dispositivi a occhio o utilizzando la microscopia generica per garantire che non ci siano collassi degli ingressi o dei microcanali di inoculazione.
NOTA: per le condizioni sature (ad esempio, condizioni sature di acqua e/o ricche di sostanze nutritive), includere il punto 2.2.7 del protocollo. Se sono richieste condizioni di insaturo d'acqua, procedere al passaggio 2.2.8. I dispositivi possono essere riempiti con acqua o supporti. - Riempire i dispositivi immediatamente dopo l'incollaggio pipettando 100 μL della soluzione desiderata per il dispositivo BFI (ingresso batterico e apertura laterale) o 30 μL di fluido in ciascun ingresso (60 μL totali) per il dispositivo FFI. Se sono presenti bolle d'aria, queste si dissipano circa 10 minuti dopo il riempimento poiché pdms è poroso.
- Aggiungere ddH2O sterile (~100-200 μL) nella capsula di Petri per mantenere l'umidità.
3. Coltura microbica
NOTA: Le seguenti fasi forniscono una procedura microbiologica generale per la coltura fungina e batterica e devono essere eseguite in condizioni sterili (ad esempio, utilizzando una fiamma o un armadietto di sicurezza microbiologica) appropriato per il livello di contenimento richiesto per i microbi desiderati. Esempi specifici sono forniti alla fine di ogni sezione per una specie di interesse.
- Coltura fungina
- Preparare il terreno di coltura desiderato integrato con agar. Autoclave del mezzo a 121 °C per 15 min. Lasciare raffreddare il mezzo a 50 °C e versare in piastre di Petri da 9 cm di diametro, mantenendo condizioni sterili.
- Utilizzare una piralide di sughero per rimuovere un tappo di agar di 4 mm di diametro contenente micelio da una colonia di frigoriferi del ceppo fungino desiderato per attivare l'isolato. Questo è condotto per garantire una crescita standardizzata e vigorosa del fungo prima dell'inoculazione del dispositivo.
NOTA: I microbi possono anche essere attivati da uno stock di glicerolo, cioè isolati fungini conservati su tappi di agar in soluzione di glicerolo al 50% a -70 °C41. - Posizionare il lato del tappo con micelio a contatto con la superficie dell'agar al centro della capsula di Petri non inoculata. Sostituire il coperchio sulla parte superiore della capsula di Petri e sigillare prima di incubare alla temperatura appropriata per la deformazione desiderata per il periodo di tempo richiesto, in genere, circa 3 o 4 giorni.
NOTA: Esempio di condizioni di coltura per Trichoderma rossicum: Estratto di malto agar, incubato a 25 °C al buio per 48 ore.
- Coltura batterica
- Strisciare l'isolato batterico desiderato dal brodo di origine (ad esempio, brodo di glicerolo o singola colonia dalla piastra di agar) su una piastra di agar per ottenere singole colonie batteriche e garantire l'assenza di contaminazione45. Sigillare la piastra con pellicola.
- Incubare ad una temperatura e durata specifiche per l'isolato di interesse fino a quando non si osservano singole colonie.
- Preparare il brodo di coltura desiderato. Ad esempio, aggiungere 10 g di triptone, 10 g di NaCl e 5 g di estratto di lievito per 1 L di ddH2O per preparare il terreno LB per la coltura di B. subtilis. Autoclave del mezzo a 121 °C per 15 min.
- Lasciare raffreddare il mezzo a temperatura ambiente. Aggiungere il mezzo a un pallone di coltura sterile all'interno di un ambiente sterile.
- Toccare una singola colonia batterica dalla piastra di agar usando un anello di inoculazione sterile. Trasferire l'anello inoculato nel terreno di coltura sterile toccando brevemente il liquido con il ciclo.
- Sigillare il matraccio con un coperchio sterile o un foglio e metterlo all'interno di un'incubatrice vibrante durante la notte utilizzando le impostazioni appropriate per le specie selezionate.
NOTA: Esempio di condizioni di coltura per B. subtilis: i) coltura liquida - crescita aerobica a 37 °C a 200 giri/min in terreno LB e ii) coltura su piastra - temperatura ambiente su piastra lb agar. Fare riferimento ai documenti FFI / BFI 40,41 per ulteriori dettagli sulla coltivazione di diversi ceppi fungini.
4. Inoculazione del dispositivo
NOTA: I seguenti passaggi devono avvenire all'interno di una cappa a flusso laminare utilizzando apparecchiature sterili.
- Inoculazione fungina
- Utilizzare una piralide di sughero sterilizzata (ø = 4 mm) per rimuovere un tappo di agar dalla colonia alla periferia di una coltura di 3 giorni (passaggio 3.1). Assicurarsi che il fronte ifale in crescita rimanga intatto.
- Introdurre il tappo nell'ingresso fungino, micelio lato verso il basso, con la direzione di crescita del fronte ifale orientato verso le aperture dei microcanali per favorire l'infiltrazione ifale dei canali.
- Ripetere i passaggi 4.1.1-4.1.2 per la seconda specie fungina (se si utilizza il dispositivo FFI), introducendo la spina nell'ingresso opposto. Se si utilizza il dispositivo BFI, omettere questo passaggio e continuare con il passaggio 4.1.4.
- Sigillare la capsula di Petri con pellicola trasparente e incubare a 25-28 °C al buio fino all'inizio dell'imaging. Determinare il tempo di incubazione pre-imaging in base all'evento biologico previsto da osservare, ad esempio i confronti fungo-fungino e il tasso di crescita delle specie incluse all'interno del dispositivo.
- Inoculazione batterica
- Diluire i batteri da una coltura notturna (fase 3.2) in un rapporto 1:25 utilizzando lo stesso terreno di coltura come descritto nel punto 3.2.3. Coltura per 3 ore a 37 °C.
- Lavare i batteri pellettizzando la coltura utilizzando una centrifuga a 2000 x g per 10 minuti. Scartare il surnatante e risospese le cellule nel volume desiderato di soluzione di cloruro di sodio allo 0,9% p/v.
- Centrifugare di nuovo per ottenere un pellet. Scartare il surnatante e risospese le cellule in mezzo liquido (ad esempio, C. cinerea mezzo minimo a un OD600 di 1). Ottimizzare il valore OD600 per il ceppo batterico in questione.
- Rimuovere il dispositivo BFI dall'incubatore e aprirlo in un ambiente sterile. Pipetta 10 μL di sospensione nell'ingresso batterico.
NOTA: Ottimizzare i tempi esatti di inoculazione per le interazioni batterico-fungine in questione. Ad esempio, introdurre batteri nel dispositivo BFI 18 ore di inoculazione post-fungina se si utilizza C. cinerea. - Sigillare la capsula di Petri con una pellicola trasparente e incubare a 25 °C al buio fino all'inizio dell'imaging. Conservare il dispositivo in posizione verticale.
5. Microscopia e analisi delle immagini
- Microscopia
NOTA: Il ricercatore dovrebbe selezionare il metodo appropriato di imaging in accordo con la natura dell'esperimento da condurre, ad esempio un'epifluorescenza a campo largo invertita o una microscopia confocale. Qui è stata fornita una panoramica generale, poiché i dettagli specifici dipenderanno dagli attributi della configurazione della microscopia scelta.- Accendere il computer del microscopio, il corpo principale del microscopio (se applicabile), la fotocamera, l'incubatore a temperatura controllata e le sorgenti luminose. Assicurarsi che il microscopio sia stato impostato correttamente, ad esempio, l'illuminazione Köhler è stata applicata correttamente per un'illuminazione uniforme del campione. Avviare il pacchetto software di imaging.
NOTA: quando si utilizza un incubatore a temperatura controllata, è importante consentire alla temperatura del microscopio di equilibrarsi per diverse ore prima di iniziare un esperimento. - Montare il dispositivo microfluidico nell'inserto del palco. Assicurarsi che il dispositivo sia ben fissato, cioè con nastro adesivo, per evitare di spostare il dispositivo durante il movimento attivo della fase.
- Acquisire immagini dei dispositivi inoculati, ad esempio esperimenti a punto singolo o time-lapse. Le specifiche di imaging complete relative agli esperimenti condotti con i dispositivi BFI e FFI sono fornite nelle rispettive pubblicazioni di cui sopra40,41.
NOTA: le immagini Brightfield sono state acquisite utilizzando la microscopia a contrasto di fase per visualizzare la proliferazione ifale attraverso i canali di crescita utilizzando il software di messa a fuoco automatica e ingrandimento 10x, 0,30 NA (apertura numerica) o ingrandimento 20x, obiettivi da 0,45 NA. L'eccitazione delle proteine reporter fluorescenti è stata ottenuta utilizzando un motore di luce a diodi emettitori di luce ad alta potenza con lunghezze d'onda specifiche per il fluoroforo. - Esportare le immagini in un formato adatto per la successiva elaborazione delle immagini. Ad esempio, .tiff.
- Accendere il computer del microscopio, il corpo principale del microscopio (se applicabile), la fotocamera, l'incubatore a temperatura controllata e le sorgenti luminose. Assicurarsi che il microscopio sia stato impostato correttamente, ad esempio, l'illuminazione Köhler è stata applicata correttamente per un'illuminazione uniforme del campione. Avviare il pacchetto software di imaging.
- Analisi delle immagini
NOTA: Gli autori raccomandano Fiji46 come strumento per l'analisi delle immagini, ma sono disponibili altri pacchetti software. Di seguito sono riportati esempi di analisi delle immagini condotte utilizzando Fiji dalle pubblicazioni sui dispositivi BFI e FFI presentate. Questi passaggi sono specifici per un Mac e possono differire leggermente se si utilizza un PC.- Misurazioni del tasso di crescita ifale
NOTA: Questo metodo è stato utilizzato nel manoscritto BFI40 per misurare i tassi di crescita delle singole ife.- Scarica, installa e avvia Fiji. Importate la sequenza di immagini da un esperimento time-lapse selezionando File > Importa sequenza > immagine. Individuare la cartella in cui sono archiviati i dati e selezionare Apri. Nella finestra Opzioni sequenza selezionare Preferenze, quindi OK.
- Selezionate l'icona Linea retta dalla barra degli strumenti principale. Posizionare l'inizio della linea retta sulla punta della punta ifale crescente facendo clic e quindi trascinando contemporaneamente il cursore in un altro punto all'interno della finestra. Apparirà una linea gialla con tre caselle che indicano l'inizio, il punto medio e la fine della linea.
- Passare al fotogramma successivo della sequenza di immagini tenendo premuti CTRL e >. Posiziona l'estremità della linea retta sulla punta dell'ifa crescente selezionando e trascinando la casella quadrata nella posizione corretta.
- Tieni premuti Ctrl e M per misurare la lunghezza della linea in pixel. Apparirà una finestra Risultati con i dati misurati. Definire i dati visualizzati nella finestra Risultati come segue: fare clic sulla finestra Risultati , quindi selezionare Risultati > Imposta misurazioni.
- Passare al fotogramma successivo della sequenza di immagini tenendo premuto CTRL e quindi >. Posiziona l'inizio della linea retta sulla punta dell'ifa crescente selezionando e trascinando la casella quadrata nella posizione corretta.
- Tenere premuto CTRL e quindi M per misurare la lunghezza della linea in pixel. Apparirà una finestra Risultati con i dati misurati.
- Ripetere i passaggi 5.2.1.5-5.2.1.6 fino al termine della misurazione della crescita dell'ifa in pixel.
- Selezionare tutti i dati nella finestra Risultati . Copia e incolla in un altro programma software, ad esempio un foglio di calcolo, per elaborare i dati. Traccia la crescita ifale (in pixel o micrometri) in funzione del tempo e calcola i tassi di crescita medi. Eseguire almeno tre repliche biologiche per esperimento.
- Misure di intensità di fluorescenza
NOTA: Questo metodo è stato utilizzato nella pubblicazione FFI41 per valutare la variazione dell'intensità di fluorescenza nelle ife di Fusarium graminearum 8/1-wt-GFP a contatto con Clonostachys rosea 016 in funzione del tempo.- Scarica, installa e avvia Fiji. Importate la sequenza di immagini da un esperimento time-lapse selezionando File > Importa sequenza > immagine. Individuare la cartella in cui sono archiviati i dati e selezionare Apri. Nella finestra Opzioni sequenza selezionare Preferenze , quindi OK.
- Specificare una regione di interesse (ROI) per misurare l'intensità assoluta di fluorescenza di un'ifa utilizzando lo strumento rettangolare, situato nella barra degli strumenti principale. La dimensione del quadrato può essere definita esattamente come segue: Modifica > Selezione > Specifica; il ROI può anche essere salvato per riferimento futuro in ROI Manager selezionando Modifica > selezione > Aggiungi al manager.
- Misurare l'intensità assoluta di fluorescenza (valore di grigio medio) all'interno del ROI definito per ogni immagine nell'intera sequenza o pila di immagini come segue: Image > Stacks > Measure Stack. La finestra Risultati si aprirà automaticamente una volta che tutte le immagini nello stack sono state elaborate.
NOTA: i dati visualizzati nella finestra Risultati possono essere definiti come segue: fare clic sulla finestra Risultati e quindi selezionare Risultati > Imposta misurazioni. Assicurarsi che l'opzione Valore grigio medio sia stata selezionata. - Selezionare tutti i dati nella finestra Risultati . Copia e incolla in un altro programma software, ad esempio un foglio di calcolo, per tracciare le intensità assolute di fluorescenza del ROI specificato in funzione del tempo.
- Ripetere i passaggi 5.2.2.2-5.2.2.4 per raccogliere le misurazioni assolute dell'intensità di fluorescenza per ciascun ROI, cioè sull'ifa di interesse, accanto all'ifa di interesse o all'interno del canale di controllo corrispondente.
- Calcolare le opportune intensità di fluorescenza relativa in unità arbitrarie (AU), ad esempio dividendo l'intensità di fluorescenza assoluta del ROI [ifa di interesse] per l'intensità assoluta di fluorescenza del ROI [canale di controllo]. Consultare la pubblicazione FFI41 per dettagli più specifici.
- Eseguire almeno tre repliche biologiche per esperimento e tracciare le intensità di fluorescenza relative in funzione del tempo.
- Misurazioni del tasso di crescita ifale
Access restricted. Please log in or start a trial to view this content.
Risultati
I risultati rappresentativi sono presentati dai dispositivi esemplari BFI40 e FFI41. Le misurazioni del tasso di crescita ifale possono essere facilmente ottenute utilizzando questi dispositivi in combinazione con tecniche di microscopia di base. La Figura 3A-B illustra le interazioni batterico-fungine tra C. cinerea hyphae e B. subtilis NCIB 3610. La presenza di B. subtilis arresta l...
Access restricted. Please log in or start a trial to view this content.
Discussione
Questo articolo presenta un protocollo per lo studio delle interazioni fungo-microbiche utilizzando la microfluidica del canale. Gli autori mirano a dimostrare la versatilità di questi dispositivi e incoraggiare l'adattamento per soddisfare gli interessi del ricercatore. Utilizzando i dispositivi esemplari BFI e FFI, le interazioni fungine-microbiche possono essere studiate in modo più dettagliato di quanto precedentemente accessibile. Rimuovendo la complessità di fondo e l'eterogeneità del suolo, moderando la cresci...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non dichiarano interessi concorrenti.
Riconoscimenti
Riconosciamo il sostegno finanziario del Dipartimento di Bioingegneria dell'Imperial College di Londra e del Leverhulme Trust (Research Grant Reference: RPG-2020-352).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Agar | Difco Laboratories | 214010 | Used to solidify culture medium for bacterial and fungal cultivation within Petri dishes |
| Aluminum foil | Fisher Scientific Ltd | 11759408 | |
| AutoCAD 2021 | Autodesk, USA | ||
| Autoclave (VX-75) | Systec | ||
| Centrifuge (5810R) | Eppendorf | ||
| Chlorotrimethysilane | Merck Life Sciences | 386529 | CAUTION: Chlorotrimethylsilane is a hazardous substance. Wear appropriate PPE and handle with care. Avoid contact with skin and eyes and prevent inhalation. Keep away from sources of ignition and use in a well-ventilated area. |
| Cork borer | SLS | COR1000 | |
| Developer solution (mr-Dev 600) | Microresist Technologies | CAUTION: mr-Dev 600 developer solution is flammable | |
| Erlenmeyer flasks | VWR | 214-1108 | e.g. 200 mL; choose size to suit your exact needs |
| Ethanol (70% v/v) | Fisher Scientific Ltd | E/0650DF/15 | Diluted from 99.8% (Analytical Reagent Grade) |
| Fiji | ImageJ | Exemplar software package for imaging processing | |
| Filtered, compressed air | Available as standard in most labs. Altervatively, an oil-free compressor with air regulator can be used. | ||
| Flat-headed wafer tweezers | SLS | INS5026 | |
| Forceps | Fisher Scientific Ltd | 10008051 | Bent, sharp |
| Glass bottom petri dish | World Precision Instruments | FD35-100 | 35 mm |
| Glass bottom petri dish | World Precision Instruments | FD5040-100 | 50 mm |
| Glass crystallisation dishes | VWR | 216-1865 | Used for washing of PDMS slabs |
| Glass crystallisation dishes | VWR | 216-1866 | Used in the development of master moulds |
| Glass media bottles | Fisher Scientific Ltd | 15456113 | e.g. 250 mL; choose size to suit your exact needs |
| Glass syringe (Hamilton) | Fisher Scientific Ltd | 10625251 | Used for dispensing chlorotrimethylsilane |
| Hot plate (HP 160 III BM) | SAWATEC | ||
| Inoculation loop | VWR | COPA175CS01 | |
| Isopropyl alcohol | Sigma-Aldrich | W292907 | |
| Laminar flow hood | Air Science (PCR) | Exemplar laminar flow hood used for device fabrication | |
| LB medium | Fisher Scientific Ltd | BP9723-500 | Exemplar nutrient broth for bacterial overnight culture |
| Light emitting diode light engine (LedHUB) | Omicron-Laserage Laserprodukte GmbH | Exemplar light source that can be used for imaging fungal-microbial interactions (fluorescence) | |
| MA6 Ultraviolet mask aligner | Suss Microtec | ||
| Malt extract | VWR | 84618 | Used to make exemplar fungal culture medium (Malt extract agar) |
| Mask Writer | Applied Materials | 4700DP | Example of a mask writer which can be used to print photo-mask for photolithography |
| Master mould plastic mount | 3D-printed bespoke holder manufactured in-house | ||
| Microbiological safety cabinet (BioMat2) | Contained Air Solutions | Exemplar MSC used for microbial culture and device inoculation | |
| Milli-Q purified water | Available as standard in biology labs. | ||
| NaOH | Fisher Scientific Ltd | BP359-500 | |
| NIS-Elements Advanced Research imaging software | Nikon | Exemplar software package for image acquisition | |
| NIS-Elements Free Viewer | Nikon | Exemplar software package for viewing acquired images | |
| Oven (Binder BD115) | Fisher Scientific Ltd | 15602126 | Used for curing poly(dimethylsiloxane)(PDMS) |
| Oven (CLO-2AH-S) | KOYO | Used for preparing silicon wafers | |
| Parafilm | Bemis | HS234526B | transparent film |
| Petri dishes, square sterile | Fisher Scientific Ltd | 11708573 | 120.5 mm |
| Petri dishes, sterile | Fisher Scientific Ltd | 15370366 | 90 mm |
| Photolithography mask | Micro Lithography Services Ltd. UK | ||
| Plasma cleaner (Zepto) | Diener Electronic | 100012601 | |
| Plastic cup | Semadeni | 8323 | |
| Plastic spatula | Semadeni | 3340 | |
| Portable precision balance (OHAUS Scout) | Fisher Scientific Ltd | 15519631 | Used for weighing PDMS, media components etc. |
| Precision cutter | Syneo | HS1251135P1183 | Cutting edge diameter: 3.18 mm |
| Precision cutter | Syneo | HS1871730P1183S | Cutting edge diameter: 4.75 mm |
| Profilometer | Bruker | Dektak XT-stylus | |
| Razor blades | Häberle Labortechnik | 9156110 | |
| Refridgerator | Haden | 4-6 °C | |
| Retiga R1 CCD camera | Qimaging | Exemplar camera that can be used for imaging fungal-microbial interactions | |
| Scotch magic tape | Office Depot | 3969954 | 19 mm invisible tape; clear tape |
| Shaking incubator (Cole-Parmer SI500) | Fisher Scientific Ltd | 10257954 | |
| Silicon wafer | Inseto | 100 mm | |
| Soda lime glass plate | Inseto | 125 mm x 125 mm x 2 mm. Used to hold photolithography mask in mask aligner | |
| Sodium chloride | Sigma-Aldrich | S7653 | |
| Spincoater | SAWATEC | SM-180-BM | |
| SU-8 2010 photoresist | MicroChem | CAUTION: SU-8 photoresist is hazardous, take care when handling and prevent inhalation and contact with skin. Flammable, potentially carcinogenic and toxic to the environment. | |
| Sylgard 184 elastomer kit | VWR | 634165S | Used for the preparation of poly(dimethylsiloxane)(PDMS) devices |
| Temperature controlled incubator | Okolab | Exemplar incubator that can be used for imaging fungal-microbial interactions | |
| Ti2-E inverted epifluorescence microscope | Nikon | MEA54000 | Exemplar microscope that can be used for imaging fungal-microbial interactions |
| Ultrasonic cleaner S-Line | Fisher Scientific Ltd | FB15050 | |
| Vacuum desiccator | Fisher Scientific Ltd | 10528861 | Silianisation and PDMS degassing should be conducted in separate desiccators |
| x10/0.3 NA CFI Plan Fluor DL objective lens | Nikon | MRH20105 | Exemplar objective lens that can be used for imaging fungal-microbial interactions |
| x20/0.45 NA CFI Plan Fluor DL objective lens | Nikon | MRH48230 | Exemplar objective lens that can be used for imaging fungal-microbial interactions |
Riferimenti
- Zhu, Y. -G., Miller, R. M. Carbon cycling by arbuscular mycorrhizal fungi in soil-plant systems. Trends in Plant Science. 8 (9), 407-409 (2003).
- Dai, Z., et al. Long-term nutrient inputs shift soil microbial functional profiles of phosphorus cycling in diverse agroecosystems. The ISME Journal. 14 (3), 757-770 (2020).
- Op De Beeck, M., et al. Regulation of fungal decomposition at single-cell level. The ISME Journal. 14 (4), 896-905 (2020).
- Bender, S. F., et al. Symbiotic relationships between soil fungi and plants reduce N2O emissions from soil. The ISME Journal. 8 (6), 1336-1345 (2014).
- Dullah, S., et al. Melanin production and laccase mediated oxidative stress alleviation during fungal-fungal interaction among basidiomycete fungi. IMA Fungus. 12 (1), 33(2021).
- Deveau, A., et al. Bacterial-fungal interactions: ecology, mechanisms and challenges. FEMS Microbiology Reviews. 42 (3), 335-352 (2018).
- Bian, R., et al. Facilitative and synergistic interactions between fungal and plant viruses. Proceedings of the National Academy of Sciences of the United States of America. 117 (7), 3779-3788 (2020).
- Jiang, X., Xiang, M., Liu, X. Nematode-trapping fungi. Microbiology Spectrum. 5 (1), (2017).
- Essig, A., et al. a novel peptide-based fungal antibiotic interfering with the peptidoglycan synthesis. Journal of Biological Chemistry. 289 (50), 34953-34964 (2014).
- Tang, H. -Y., Zhang, Q., Li, H., Gao, J. -M. Antimicrobial and allelopathic metabolites produced by Penicillium brasilianum. Natural Product Research. 29 (4), 345-348 (2015).
- Bai, Y. -B., et al. Antifungal activity of griseofulvin derivatives against phytopathogenic fungi In vitro and In vivo and three-dimensional quantitative structure-activity relationship analysis. Journal of Agricultural and Food Chemistry. 67 (22), 6125-6132 (2019).
- Solanki, M. K., et al. Characterization of antagonistic-potential of two Bacillus strains and their biocontrol activity against Rhizoctonia solani in tomato. Journal of Basic Microbiology. 55 (1), 82-90 (2015).
- Jamali, H., Sharma, A., Srivastava, A. K. Biocontrol potential of Bacillus subtilis RH5 against sheath blight of rice caused by Rhizoctonia solani. Journal of Basic Microbiology. 60 (3), 268-280 (2020).
- Válková, H., Novotný, Č, Malachová, K., Šlosarčíková, P., Fojtík, J. Effect of bacteria on the degradation ability of Pleurotus ostreatus. Science of The Total Environment. 584-585, 1114-1120 (2017).
- Leyva-Rojas, J. A., Coy-Barrera, E., Hampp, R. Interaction with soil bacteria affects the growth and amino acid content of Piriformospora indica. Molecules. 25 (3), Basel, Switzerland. 572(2020).
- Dullah, S., et al. Fungal interactions induce changes in hyphal morphology and enzyme production. Mycology. 12 (4), 279-295 (2021).
- Marfetán, J. A., Romero, A. I., Folgarait, P. J. Pathogenic interaction between Escovopsis weberi and Leucoagaricus sp.: mechanisms involved and virulence levels. Fungal Ecology. 17, 52-61 (2015).
- Cortois, R., De Deyn, G. B. The curse of the black box. Plant and Soil. 350 (1), 27-33 (2012).
- Whitesides, G. M. The origins and the future of microfluidics. Nature. 442 (7101), 368-373 (2006).
- Sackmann, E. K., Fulton, A. L., Beebe, D. J. The present and future role of microfluidics in biomedical research. Nature. 507 (7491), 181-189 (2014).
- Hanson, K. L., et al. Fungi use efficient algorithms for the exploration of microfluidic networks. Small. 2 (10), 1212-1220 (2006).
- Held, M., Edwards, C., Nicolau, D. V. Probing the growth dynamics of Neurospora crassa with microfluidic structures. Fungal Biology. 115 (6), 493-505 (2011).
- Thomson, D. D., et al. Contact-induced apical asymmetry drives the thigmotropic responses of Candida albicans hyphae. Cellular Microbiology. 17 (3), 342-354 (2015).
- Lee, K. K., Labiscsak, L., Ahn, C. H., Hong, C. I. Spiral-based microfluidic device for long-term time course imaging of Neurospora crassa with single nucleus resolution. Fungal Genetics and Biology. 94, 11-14 (2016).
- Asenova, E., Lin, H. Y., Fu, E., Nicolau, D. V., Nicolau, D. V. Optimal fungal space searching algorithms. IEEE Transactions on NanoBioscience. 15 (7), 613-618 (2016).
- Soufan, R., et al. Pore-scale monitoring of the effect of microarchitecture on fungal growth in a two-dimensional soil-like micromodel. Frontiers in Environmental Science. 6, (2018).
- Uehling, J. K., et al. Microfluidics and metabolomics reveal symbiotic bacterial-fungal interactions between Mortierella elongata and Burkholderia include metabolite exchange. Frontiers in Microbiology. 10, 2163(2019).
- Millet, L. J., et al. Increasing access to microfluidics for studying fungi and other branched biological structures. Fungal Biology and Biotechnology. 6 (8), 1-14 (2019).
- Baranger, C., Fayeulle, A., Le Goff, A. Microfluidic monitoring of the growth of individual hyphae in confined environments. Royal Society Open Science. 7 (8), 191535(2020).
- Aleklett, K., Ohlsson, P., Bengtsson, M., Hammer, E. C. Fungal foraging behaviour and hyphal space exploration in micro-structured Soil Chips. The ISME Journal. 15 (6), 1782-1793 (2021).
- Aleklett, K., et al. Build your own soil: exploring microfluidics to create microbial habitat structures. The ISME Journal. 12 (2), 312-319 (2018).
- Ellett, F., Jorgensen, J., Frydman, G. H., Jones, C. N., Irimia, D. Neutrophil interactions stimulate evasive hyphal branching by Aspergillus fumigatus. PLOS Pathogens. 13 (1), 1006154(2017).
- Massalha, H., Korenblum, E., Malitsky, S., Shapiro, O. H., Aharoni, A. Live imaging of root-bacteria interactions in a microfluidics setup. Proceedings of the National Academy of Sciences of the United States of America. 114 (17), 4549-4554 (2017).
- Schmieder, S. S., et al. Bidirectional propagation of signals and nutrients in fungal networks via specialized hyphae. Current Biology. 29 (2), 217-228 (2019).
- Tayyrov, A., Stanley, C. E., Azevedo, S., Künzler, M. Combining microfluidics and RNA-sequencing to assess the inducible defensome of a mushroom against nematodes. BMC Genomics. 20 (1), 243(2019).
- Stanley, C. E., Grossmann, G., Casadevall i Solvas, X., deMello, A. J. Soil-on-a-Chip: microfluidic platforms for environmental organismal studies. Lab on a Chip. 16 (2), 228-241 (2016).
- Stanley, C. E., vander Heijden, M. G. A. Microbiome-on-a-Chip: new frontiers in plant-microbiota research. Trends in Microbiology. 25 (8), 610-613 (2017).
- Ortseifen, V., Viefhues, M., Wobbe, L., Grünberger, A. Microfluidics for biotechnology: bridging gaps to foster microfluidic applications. Frontiers in Bioengineering & Biotechnology. 8, 589074(2020).
- Jansson, J. K., Hofmockel, K. S. The soil microbiome-from metagenomics to metaphenomics. Current Opinion in Microbiology. 43, 162-168 (2018).
- Stanley, C. E., et al. Probing bacterial-fungal interactions at the single cell level. Integrative Biology (Camb). 6 (10), 935-945 (2014).
- Gimeno, A., et al. A versatile microfluidic platform measures hyphal interactions between Fusarium graminearum and Clonostachys rosea in real-time. Communications Biology. 4 (1), 262(2021).
- Duffy, D. C., McDonald, J. C., Schueller, O. J. A., Whitesides, G. M. Rapid prototyping of microfluidic systems in poly(dimethylsiloxane). Analytical Chemistry. 70 (23), 4974-4984 (1998).
- Stanley, C. E., et al. Fabrication and use of the dual-flow-RootChip for the imaging of Arabidopsis roots in asymmetric microenvironments. Bio-protocol. 8 (18), 3010(2018).
- Choi, C. -H., Lee, H., Weitz, D. A. Rapid patterning of PDMS microfluidic device wettability using syringe-vacuum-induced segmented flow in nonplanar geometry. ACS Applied Materials & Interfaces. 10 (4), 3170-3174 (2018).
- Sanders, E. R. Aseptic laboratory techniques: plating methods. Journal of Visualized Experiments. (63), e3064(2012).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Harting, R., et al. Pseudomonas strains induce transcriptional and morphological changes and reduce root colonization of Verticillium spp. Frontiers in Microbiology. 12, 652468(2021).
- Boenisch, M. J. Structural and molecular characterisation of the penetration process of Fusarium graminearum during Fusarium head blight infection. , Staats-und Universitätsbibliothek Hamburg Carl von Ossietzky. (2013).
- Eynck, C., Koopmann, B., Grunewaldt-Stoecker, G., Karlovsky, P., von Tiedemann, A. Differential interactions of Verticillium longisporum and V. dahliae with Brassica napus detected with molecular and histological techniques. European Journal of Plant Pathology. 118 (3), 259-274 (2007).
- Ghanem, N., Stanley, C. E., Harms, H., Chatzinotas, A., Wick, L. Y. Mycelial effects on phage retention during transport in a microfluidic platform. Environmental Science & Technology. 53 (20), 11755-11763 (2019).
- Alrifaiy, A., Lindahl, O. A., Ramser, K. Polymer-based microfluidic devices for pharmacy, biology and tissue engineering. Polymers. 4 (3), 1349-1398 (2012).
- Duncombe, T. A., Tentori, A. M., Herr, A. E. Microfluidics: reframing biological enquiry. Nature Reviews Molecular Cell Biology. 16 (9), 554-567 (2015).
- Hoelzle, D., et al. Microfluidic device design, fabrication, and testing protocols. Protocol Exchange. , (2015).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon