Method Article
Imaging di mtHyPer7, un biosensore raziometrico per il perossido mitocondriale, in cellule di lievito viventi
In questo articolo
Riepilogo
Il perossido di idrogeno (H 2 O2 ) è sia una fonte di danno ossidativo che una molecola di segnalazione. Questo protocollo descrive come misurare l'H 2 O2mitocondriale utilizzando HyPer7 mirato ai mitocondri (mtHyPer7), un biosensore raziometrico geneticamente codificato, nel lievito vivente. Descrive in dettaglio come ottimizzare le condizioni di imaging ed eseguire analisi quantitative cellulari e subcellulari utilizzando software disponibili gratuitamente.
Abstract
La disfunzione mitocondriale, o alterazione funzionale, si trova in molte malattie e condizioni, tra cui disturbi neurodegenerativi e muscoloscheletrici, cancro e normale invecchiamento. Qui, viene descritto un approccio per valutare la funzione mitocondriale nelle cellule di lievito viventi a risoluzioni cellulari e subcellulari utilizzando un biosensore raziometrico geneticamente codificato, minimamente invasivo. Il biosensore, HyPer7 mirato ai mitocondri (mtHyPer7), rileva il perossido di idrogeno(H 2 O2 ) nei mitocondri. È costituito da una sequenza di segnale mitocondriale fusa a una proteina fluorescente permutata circolarmente e dal dominio H 2 O2-responsivodi una proteina batterica OxyR. Il biosensore viene generato e integrato nel genoma del lievito utilizzando un sistema CRISPR-Cas9 marker-free, per un'espressione più coerente rispetto ai costrutti trasmessi da plasmidi.
mtHyPer7 è quantitativamente mirato ai mitocondri, non ha alcun effetto rilevabile sul tasso di crescita del lievito o sulla morfologia mitocondriale e fornisce una lettura quantitativa per H2 O2 mitocondriale in condizioni di crescita normali e dopo esposizione a stress ossidativo. Questo protocollo spiega come ottimizzare le condizioni di imaging utilizzando un sistema di microscopio confocale a disco rotante ed eseguire analisi quantitative utilizzando un software disponibile gratuitamente. Questi strumenti consentono di raccogliere ricche informazioni spazio-temporali sui mitocondri sia all'interno delle cellule che tra le cellule di una popolazione. Inoltre, il flusso di lavoro qui descritto può essere utilizzato per convalidare altri biosensori.
Introduzione
I mitocondri sono organelli cellulari eucariotici essenziali ben noti per la loro funzione nella produzione di ATP attraverso la fosforilazione ossidativa e il trasporto di elettroni1. Inoltre, i mitocondri sono siti per l'immagazzinamento del calcio, la sintesi di lipidi, amminoacidi, acidi grassi e cluster ferro-zolfo e la trasduzione del segnale 2,3. All'interno delle cellule, i mitocondri formano una rete dinamica con morfologia e distribuzione caratteristiche, che varia a seconda del tipo di cellula e dello stato metabolico. Inoltre, sebbene i mitocondri possano fondersi e dividersi, non tutti i mitocondri di una cellula sono equivalenti. Numerosi studi hanno documentato l'eterogeneità funzionale dei mitocondri all'interno delle singole cellule in attributi quali il potenziale di membrana e lo stato ossidativo 4,5,6. Questa variazione nella funzione mitocondriale è dovuta in parte al danno all'organello da mutazioni del mtDNA (che si verificano a un tasso più elevato rispetto al DNA nucleare) e al danno ossidativo da specie reattive dell'ossigeno (ROS) generate sia all'interno che all'esterno dell'organello 7,8,9. Il danno all'organello è mitigato da meccanismi di controllo della qualità mitocondriale che riparano il danno o eliminano i mitocondri danneggiati in modo irreparabile10.
Il perossido di idrogeno (H 2 O2 ) è una specie reattiva dell'ossigeno che è fonte di danno ossidativo alle proteine cellulari, agli acidi nucleici e ai lipidi. Tuttavia, H 2 O2funge anche da molecola di segnalazione che regola le attività cellulari attraverso l'ossidazione reversibile dei tioli nelle proteine bersaglio11,12. H2 O2 è prodotto da elettroni che fuoriescono dalla catena di trasporto degli elettroni mitocondriali e da enzimi specifici, come la NADPH ossidasi e le monoamino ossidasi 13,14,15,16,17,18,19,20. È anche inattivato da sistemi antiossidanti, compresi quelli a base di tioredossina e glutatione21,22,23. Pertanto, l'analisi dei livelli mitocondriali di H 2 O2è fondamentale per comprendere il ruolo di questo metabolita nella normale funzione mitocondriale e cellulare e in condizioni di stress ossidativo.
L'obiettivo generale di questo protocollo è quello di rilevare l'H 2O 2 mitocondriale utilizzando un biosensore raziometrico H2O2 geneticamente codificato, HyPer7, mirato all'organello (mtHyPer7). mtHyPer7 è una chimera costituita dalla sequenza del segnale mitocondriale di ATP9 (la presequenza Su9), una forma circolarmente permutata di proteina fluorescente verde (GFP), e dal dominio di legameH 2 O2 della proteina OxyR di Neisseria meningitidis24 (Figura 1). Nella GFP permutata circolarmente, i termini N e C della GFP nativa si fondono e si formano nuovi termini vicino al cromoforo, che conferiscono una maggiore mobilità alla proteina e una maggiore labilità delle sue caratteristiche spettrali rispetto alla GFP25 nativa. L'interazione del dominio OxyR di mtHyPer7 con H 2 O2 è ad alta affinità, H 2 O 2selettiva e porta all'ossidazione reversibile dei residui di cisteina conservati e alla formazione di ponti disolfuro. I cambiamenti conformazionali associati all'ossidazione di OxyR vengono trasferiti alla GFP permutata circolarmente in mtHyPer7, il che si traduce in uno spostamento spettrale nel massimo di eccitazione del cromoforo mtHyPer7 da 405 nm nello stato ridotto a 488 nm nello stato H 2 O2-ossidato 26. Pertanto, il rapporto di fluorescenza da mtHyPer7 in risposta all'eccitazione a 488 nm rispetto a 405 nm riflette l'ossidazione della sonda da parte di H 2 O2.
Idealmente, un biosensore dovrebbe fornire una lettura quantitativa in tempo reale, assoluta e quantitativa della sua molecola bersaglio. Purtroppo, però, questo non è sempre possibile nelle misurazioni del mondo reale. Nel caso di sensori di ossidazione, come mtHyPer7, la lettura in tempo reale è influenzata dalla velocità di riduzione del ponte disolfuro. I sistemi di riduzione utilizzati dai biosensori ROS differiscono e possono alterare drasticamente la dinamica di risposta della sonda, come dimostrato dal confronto tra HyPer7, ridotto dal sistema della tioredossina, e roGFP2-Tsa2ΔCR, ridotto dal glutatione nel citosol27 del lievito. Quindi, per trarre una conclusione sulla concentrazione relativa diH 2 O2 dai rapporti mtHyPer7, si deve assumere che il sistema di riduzione mantenga una capacità costante durante l'esperimento. Nonostante queste considerazioni, HyPer7 e le relative sonde sono state utilizzate in vari contesti per ottenere informazioni su H 2 O2nelle cellule viventi 25,28,29.
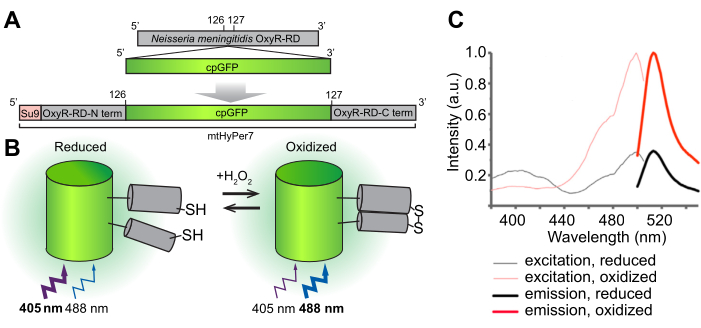
Figura 1: Progettazione, meccanismo molecolare e spettri di eccitazione/emissione del biosensore H 2 O2 mtHyPer7. (A) La sonda mtHyPer7 è derivata inserendo GFP permutata circolarmente nel dominio OxyR-RD di Neisseria meningitidis. Contiene la sequenza di targeting mitocondriale della subunità 9 dell'ATP sintasi di Neurospora crassa (Su9). (B) Illustrazione del meccanismo di rilevamento H 2 O2di mtHyPer7. L'ossidazione delle cisteine nel dominio RD aumenta l'emissione di fluorescenza all'eccitazione a 488 nm e diminuisce l'emissione prodotta dall'eccitazione a 405 nm. (C) Spettri di eccitazione e di emissione di HyPer7 in forme ossidate e ridotte. Questa figura è ristampata con il permesso di Pak et al.24. Abbreviazioni: GFP = proteina fluorescente verde; cpGFP = GFP permutata circolarmente. Fare clic qui per visualizzare una versione più grande di questa figura.
Questo imaging raziometrico di mtHyPer7 offre importanti vantaggi per la quantificazione mitocondriale di H 2 O224,27; Fornisce un controllo interno per la concentrazione della sonda. Inoltre, lo spostamento del picco di eccitazione prodotto dall'esposizionea H 2 O 2 non è completo, anche in concentrazioni saturanti di H 2 O 2. Pertanto, l'imaging del rapporto può aumentare la sensibilità incorporando due punti spettrali nell'analisi.
La sonda mtHyPer7 utilizzata qui ha un'affinità molto elevata per H2 O2 e una sensibilità relativamente bassa al pH24 ed è stata indirizzata con successo ai mitocondri30 di Caenorhabditis elegans. Questa proteina è stata utilizzata anche nel lievito27,31. Tuttavia, studi precedenti si basavano sull'espressione plasmatica di mtHyPer7, che si traduce in variabilità da cellula a cellula nell'espressione della sonda27. Inoltre, il costrutto descritto in questo protocollo è stato integrato in una regione conservata e priva di geni sul cromosoma X32 utilizzando un approccio basato su CRISPR per l'integrazione senza marcatori. L'espressione del gene biosensore integrato è controllata anche dal forte promotore costitutivo di TEF1 (Figura 2). Di conseguenza, c'è un'espressione più stabile e coerente del biosensore nelle popolazioni di cellule di lievito rispetto a quella osservata utilizzando l'espressione del biosensore trasmessa da plasmidi e le cellule che portano il biosensore possono essere propagate senza la necessità di terreni selettivi.
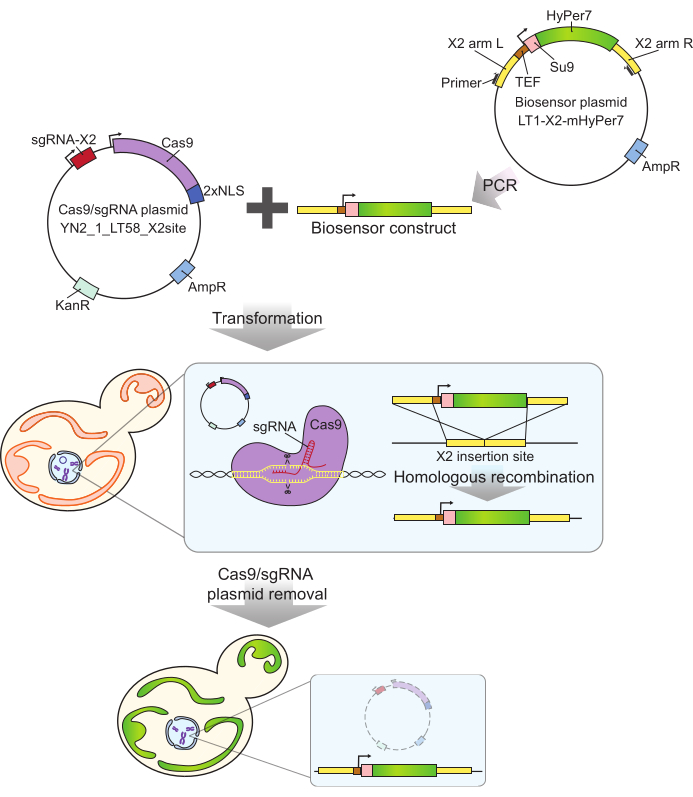
Figura 2: Generazione di cellule che esprimono mtHyPer7 mediante CRISPR. Il costrutto del biosensore contenente Cas9 e plasmide contenente sgRNA (YN2_1_LT58_X2site) e mtHyPer7 amplificato con PCR viene introdotto nelle cellule di lievito in gemmazione mediante trasformazione in acetato di litio. Il sito di inserzione privo di geni sul cromosoma X (X2) viene riconosciuto e tagliato dalla proteina Cas9 con l'sgRNA e il biosensore viene integrato nel genoma mediante ricombinazione omologa. Dopo l'identificazione dei trasformanti di successo mediante screening microscopico, PCR delle colonie e sequenziamento, il plasmide Cas9 viene rimosso (curato) mediante crescita in terreni non selettivi. Abbreviazioni: sgRNA = RNA guida singola; TEF = fattore di potenziamento della trascrizione. Fare clic qui per visualizzare una versione più grande di questa figura.
Infine, mtHyPer7 offre vantaggi rispetto ad altri biosensori ROS. Ad esempio, i coloranti organici utilizzati per rilevare i ROS (ad esempio, diidroetidio [DHE]2 e MitoSOX3) possono produrre colorazioni irregolari o non specifiche e sono spesso somministrati in solventi come etanolo o dimetilsolfossido, che richiedono controlli aggiuntivi per gli effetti del solvente. Un'altra classe di biosensori ROS sono i biosensori basati sul trasferimento di energia a risonanza di fluorescenza (FRET) (ad esempio, Redoxfluor per lo stato redox cellulare4 e i sensori di perossido HSP-FRET5, OxyFRET 6 e PerFRET6). Queste sonde sono geneticamente codificate e altamente sensibili in linea di principio e possono essere quantitativamente indirizzate ai mitocondri utilizzando sequenze di segnale mitocondriale ben caratterizzate. Tuttavia, ci sono sfide nell'uso di sonde basate su FRET, tra cui lo sfondo dovuto all'eccitazione incrociata e al bleed-through, e requisiti rigorosi per la vicinanza e l'orientamento dei fluorofori affinché si verifichi FRET33,34. Inoltre, le sonde FRET sono costituite da due proteine fluorescenti che richiedono costrutti più grandi per l'espressione nelle cellule di interesse rispetto alle sonde a spostamento spettrale. Il protocollo qui descritto è stato sviluppato per sfruttare i punti di forza del biosensore basato su HyPer7 e per utilizzare questa sonda compatta, raziometrica, ad alta affinità, geneticamente codificata per l'imaging quantitativo del perossido nei mitocondri nel lievito vivente.
Protocollo
1. Generazione del plasmide biosensore, integrazione nel genoma del lievito e valutazione del targeting di mtHyPer7 ai mitocondri e degli effetti sulla morfologia mitocondriale, sui tassi di crescita cellulare o sulla sensibilità allo stress ossidativo
NOTA: Fare riferimento al file supplementare 1, alla tabella supplementare S1 , alla tabella supplementare S2 e alla tabella supplementare S3 per la costruzione di plasmidi di biosensori, ceppi, plasmidi e primer, rispettivamente, utilizzati per la costruzione e la caratterizzazione di biosensori. Vedere la Tabella dei materiali per i dettagli relativi a tutti i materiali, i reagenti e gli strumenti utilizzati in questo protocollo.
- Amplificare il costrutto del biosensore dal plasmide del biosensore utilizzando i primer Y290 e Y291 (Figura 2) tramite la reazione a catena della polimerasi (PCR).
- Miscelare 500 ng del plasmide Cas9/RNA guida (YN2_1_LT58_X2site32) con 50 μL del prodotto PCR della fase 1.1 (il costrutto del biosensore). Trasformare la miscela in lievito in gemmazione utilizzando il metodo 35 dell'acetato di litio e selezionare i trasformanti su piastre YPD contenenti 200 mg/mL di G418.
- Screening dei trasformanti candidati mediante amplificazione PCR del DNA genomico utilizzando i primer Y292 e Y293. Per i trasformatori che recano un inserto delle dimensioni previste (positivo: 3,5 kb; negativo: 0,3 kb), sequenziare la regione inserita per un'ulteriore convalida.
- Valutare l'effetto del biosensore sulla crescita e sulla funzione mitocondriale (Figura 3). Se il biosensore sembra influenzare la funzione delle cellule o dei mitocondri, cercare di ridurre al minimo l'effetto cambiando il promotore, il sito di integrazione o il ceppo parentale.
- Determinare l'effetto del costrutto del biosensore sul tasso di crescita in presenza e in assenza di un fattore di stress ossidativo (ad esempio, paraquat), come descritto in precedenza36.
- Diluire le celle in fase logaritmica a una densità ottica di 600 nm (OD600) di 0,0035 in 200 μL di terreno corrispondente con e senza trattamento in ciascun pozzetto di una piastra a fondo piatto a 96 pozzetti. Misurare la densità ottica della coltura ogni 20 minuti per 72 ore utilizzando un lettore di piastre. Calcolare il tasso di crescita massimo (pendenza) come la variazione massima della OD in un intervallo di 240 minuti durante il percorso di 72 ore.
- Visualizzare le cellule mediante microscopia a fluorescenza e valutare la luminosità e la morfologia mitocondriale come descritto in precedenza37.
- Far crescere le cellule fino alla fase intermedia, colorare i mitocondri con MitoTracker Red da 250 nM per 30 minuti e lavare due volte prima dell'imaging. Acquisizione di pile Z di 6 μm di profondità a intervalli di 0,3 μm su un microscopio a fluorescenza ad ampio campo o confocale e ispezione visiva. Cerca i mitocondri che formano strutture tubulari allungate.
- Determinare l'effetto del costrutto del biosensore sul tasso di crescita in presenza e in assenza di un fattore di stress ossidativo (ad esempio, paraquat), come descritto in precedenza36.
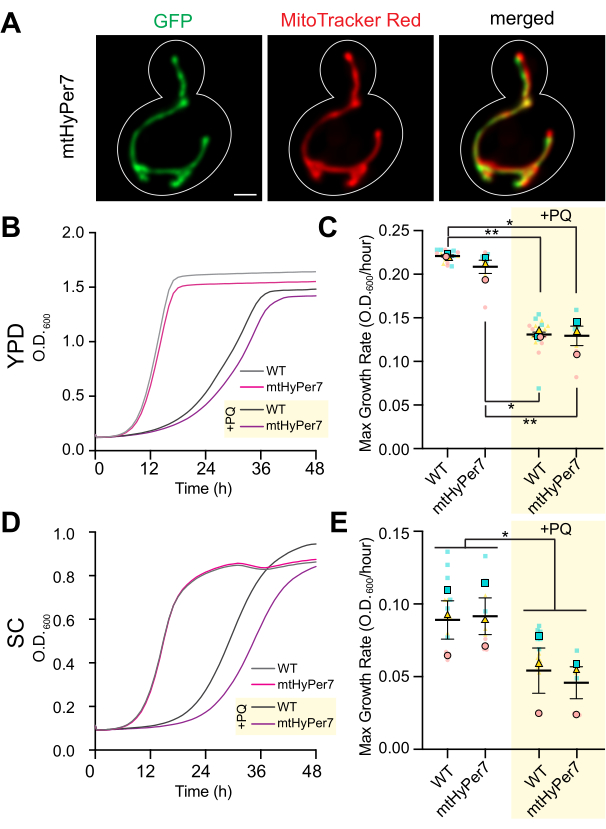
Figura 3: mtHyPer7 è mirato ai mitocondri e non influenza la morfologia mitocondriale, la crescita cellulare o la sensibilità allo stress ossidativo. (A) Morfologia mitocondriale in cellule di lievito viventi che esprimono mtHyPer7. Pannello di sinistra: mtHyPer7 visualizzato con eccitazione a 488 nm. Pannello centrale: mitocondri marcati con 250 nM MitoTracker Red. Pannello di destra: immagini unite. Vengono mostrate le proiezioni di massima intensità. Il contorno della cella viene visualizzato in bianco. Barra della scala = 1 μm. (B,C) Curve di crescita e tasso di crescita massimo delle cellule wild-type e delle cellule che esprimono mtHyPer7 cresciute in presenza (+PQ) o assenza di paraquat 2,5 mM nei terreni YPD. (D,E) Curve di crescita e tasso di crescita massimo di cellule wild-type e che esprimono mtHyPer7 cresciute in presenza (+PQ) o assenza di paraquat 2,5 mM in terreni SC. Tutte le curve di crescita sono la media di tre repliche indipendenti. I tassi di crescita massimi sono rappresentati come media ± errore standard della media (SEM). L'analisi della curva di crescita è stata eseguita diluendo le cellule in fase mid-log a un OD 600 di 0,0035 in200 μL di terreno corrispondente in ciascun pozzetto di una piastra a fondo piatto a 96 pozzetti. L'OD della coltura è stato misurato ogni 20 minuti per 72 ore utilizzando un lettore di piastre. Ogni ceppo/condizione è stato placcato in triplice copia ed è stato tracciato il tasso di crescita medio. Il tasso di crescita massimo (pendenza) è stato calcolato utilizzando le variazioni di OD in un intervallo di 240 minuti nel corso di 72 ore. Abbreviazioni: WT = wild type; PQ = paraquat. Fare clic qui per visualizzare una versione più grande di questa figura.
2. Coltura cellulare e preparazione per l'imaging (Figura 4)
- Propagare le cellule in mezzo sintetico fino alla fase mid-log per l'imaging. Una coltura in fase mid-log da 5 mL fornisce cellule sufficienti per quattro o cinque vetrini e un totale di >100 cellule gemmate per l'analisi.
- Al mattino del giorno prima dell'esperimento, preparare precolture liquide inoculando 5 mL di terreno sintetico completo (SC) in una provetta a fondo conico da 50 mL con una singola colonia di cellule di lievito.
- Incubare la precoltura in un agitatore orbitale a 200 rpm e 30 °C per 6-8 h. Misurare l'OD 600 della precoltura, che dovrebbe essere nella fase logaritmica intermedia: ~0,5-1 × 107 cellule/mL, OD600 ~0,1-0,3.
- Preparare una coltura in fase intermedia utilizzando la precoltura per l'imaging il giorno successivo. Calcolare la quantità di precoltura necessaria per una coltura in fase intermedia dopo la crescita notturna (8-16 ore). Quando le cellule vengono coltivate in terreno SC, il tempo di raddoppio è di ~2 ore. Pertanto, il volume (V) di precoltura per l'inoculazione di una coltura notturna da 5 mL viene calcolato utilizzando l'equazione (1):
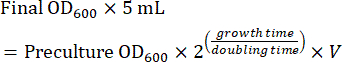 (1)
(1) - Inoculare 5 mL di terreno SC in una provetta a fondo conico da 50 mL con la quantità di precoltura calcolata al punto 2.1.3. Coltivare in uno shaker orbitale a 200 rpm e 30 °C per 8-16 h.
- Il giorno dell'imaging, verificare che la coltura generata nel passaggio 2.1.4 si trovi nella fase mid-log (OD600 ~0,1-0,3). Concentrare le cellule da 1 mL della coltura in fase mid-log mediante centrifugazione a 6.000 × g per 30 s. Rimuovere il surnatante, lasciando 10-20 μL del surnatante nella provetta. Risospendere il pellet cellulare mescolando delicatamente con il terreno residuo utilizzando una micropipetta.
- Utilizzare un soffiatore d'aria o un fanno privo di lanugine per rimuovere la polvere da un vetrino da microscopio e aggiungere 1,8 μL di sospensione cellulare al vetrino. Coprire le celle con un vetrino coprioggetti #1,5 (170 μm di spessore), abbassando lentamente il vetrino coprioggetti ad angolo per evitare l'introduzione di bolle.
- Immagine immediata ed eliminazione del vetrino dopo 10 minuti di imaging.
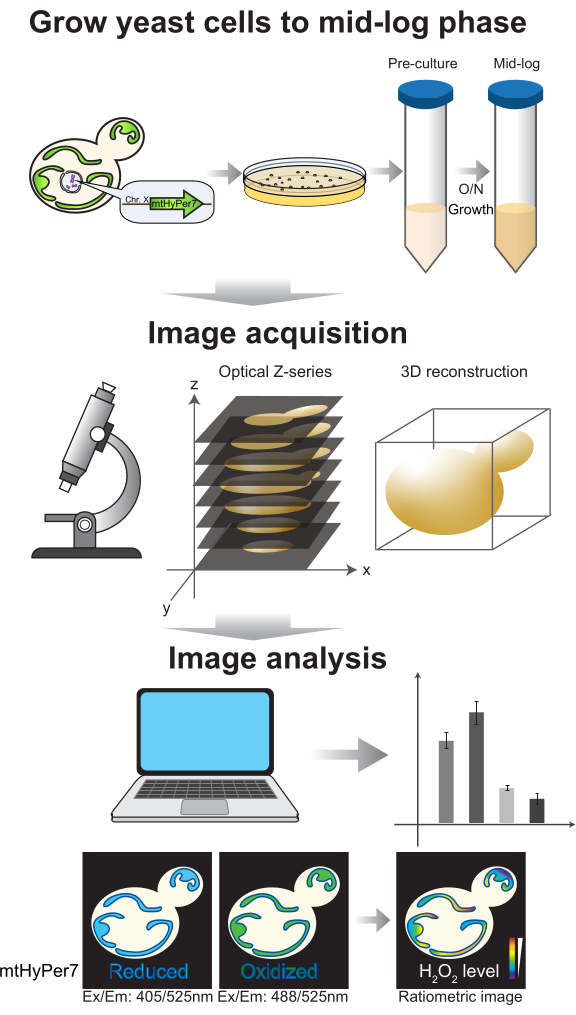
Figura 4: Crescita cellulare e imaging. Le cellule di lievito in gemmazione che esprimono mtHyPer7 vengono coltivate fino alla fase di mid-log. Le immagini della serie Z vengono raccolte mediante microscopia confocale a disco rotante e quindi sottoposte a ricostruzione e analisi 3D. Vedere le sezioni 2-3 del protocollo. Fare clic qui per visualizzare una versione più grande di questa figura.
3. Ottimizzazione delle condizioni di imaging e della raccolta delle immagini (Figura 5)
- Selezionare una modalità di imaging. Per il sezionamento ottico senza post-elaborazione, è preferibile uno strumento confocale a disco rotante. Tuttavia, se il segnale è basso o se questo strumento non è disponibile, utilizzare l'imaging a campo largo, assicurandosi che le immagini a campo largo siano deconvolute per rimuovere la sfocatura fuori fuoco, come descritto in precedenza38.
- Selezionare l'ottica. Utilizzare un obiettivo ad immersione in olio ad alta apertura numerica, come 100x/1,45 Plan-Apochromat.
- Selezionare le lunghezze d'onda di eccitazione ed emissione. Per l'imaging confocale a disco rotante, utilizzare un'eccitazione laser a 405 nm e 488 nm e un filtro di emissione GFP standard. Per l'imaging ad ampio campo, utilizzare un diodo a emissione luminosa (LED) o l'eccitazione della lampada, ma assicurarsi che la configurazione del filtro consenta la variazione dell'eccitazione mentre l'emissione passa attraverso il filtro GFP standard.
NOTA: Ciò può essere ottenuto, ad esempio, rimuovendo il filtro di eccitazione da un cubo GFP e utilizzando un LED o una ruota portafiltri per selezionare l'eccitazione. - Selezionare il tempo di esposizione e l'intensità dell'illuminazione.
- Stabilire condizioni di acquisizione che producano un segnale prontamente rilevabile e una risoluzione accettabile in ciascun canale di fluorescenza. Ad esempio, su un microscopio confocale a disco rotante con una fotocamera sCMOS, utilizzare il binning 2 x 2, una potenza laser del 20-40% e un'esposizione di 200-600 ms.
- Esaminare l'istogramma dell'immagine. In un'immagine a 12 bit (4.096 possibili livelli di grigio), assicurarsi che la gamma dinamica totale dei valori dei pixel sia di almeno diverse centinaia di livelli di grigio, senza saturazione (Figura 5A). Inoltre, assicurati che l'intervallo sia superiore di un ordine di grandezza rispetto al livello di rumore. Calcolare il livello di disturbo come deviazione standard dei valori dei pixel in un'area priva di celle di un'immagine, misurato come descritto al punto 4.1.3.1.
- Test per il fotosbiancamento o lo stress mitocondriale indotto dall'imaging nelle condizioni di imaging selezionate. Se si osserva un eccessivo fotosbiancamento o un aumento dell'H2 O2 mitocondriale, ridurre la potenza del laser e aumentare l'esposizione o il binning.
- Raccogliere una serie di immagini time-lapse, senza ritardi tra le acquisizioni, per valutare gli effetti delle condizioni di imaging sulla stabilità del segnale e sullo stress ossidativo nei mitocondri. Utilizzando il software di acquisizione del microscopio o ImageJ, misurare il valore medio dei pixel in ciascun canale di fluorescenza per confermare la stabilità del segnale (variazione del <5%; Figura 5B). Se l'esperimento non prevede l'imaging time-lapse, assicurarsi che la fluorescenza sia stabile su due o tre pile Z ripetute (25-35 esposizioni).
NOTA: Una diminuzione della fluorescenza di entrambi i canali può indicare fotosbiancamento; tuttavia, una diminuzione della fluorescenza eccitata a 405 nm accompagnata da un aumento del canale eccitato a 488 nm può indicare un aumento dell'H 2 O2mitocondriale e dello stress ossidativo indotto dall'imaging nell'organello.
- Raccogliere una serie di immagini time-lapse, senza ritardi tra le acquisizioni, per valutare gli effetti delle condizioni di imaging sulla stabilità del segnale e sullo stress ossidativo nei mitocondri. Utilizzando il software di acquisizione del microscopio o ImageJ, misurare il valore medio dei pixel in ciascun canale di fluorescenza per confermare la stabilità del segnale (variazione del <5%; Figura 5B). Se l'esperimento non prevede l'imaging time-lapse, assicurarsi che la fluorescenza sia stabile su due o tre pile Z ripetute (25-35 esposizioni).
- Acquisisci immagini. Se si raccolgono pile Z, assicurarsi che l'intervallo Z sia lo stesso per tutte le immagini nel set di dati e includere l'intera cella. Per il lievito in gemmazione, immagine con un intervallo Z di 0,5 μm e una profondità totale della pila di 6 μm. Acquisisci immagini a luce trasmessa per documentare i confini delle celle.
- Analizza le immagini manualmente o in modo semiautomatico tramite gli script disponibili nel file supplementare 2 e in https://github.com/theresaswayne/biosensor, utilizzando la distribuzione Fiji di ImageJ39 e il software statistico R (Figura 6). Nelle istruzioni del software, i comandi di menu gerarchici sono visualizzati in grassetto con "|", che indica una sequenza di selezioni di menu. Le opzioni e i pulsanti sono visualizzati in grassetto.
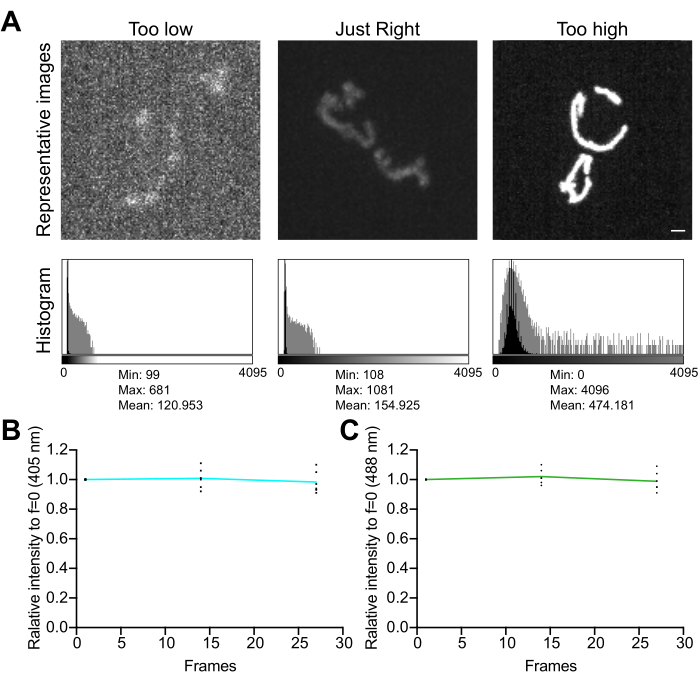
Figura 5: Ottimizzazione dell'imaging . (A) Valutazione delle immagini e degli istogrammi per il corretto intervallo di intensità. Vengono mostrate le proiezioni di massima intensità delle immagini confocali del disco rotante. Gli istogrammi sono mostrati sia con un asse Y lineare (nero) che con un asse Y logaritmico (grigio) per illustrare la gamma dinamica dell'immagine. Pannelli di sinistra: un'immagine rumorosa con bassa intensità e gamma dinamica. Pannelli centrali: un'immagine con una gamma dinamica accettabile (~1.000 livelli di grigio) e intensità. Pannelli di destra: un'immagine con contrasto impropriamente migliorato per produrre una saturazione eccessiva. Barra della scala = 1 μm. (B,C) Analisi del fotosbiancamento di mtHyPer7. Le pile Z successive sono state raccolte senza ritardi, le pile Z sono state sommate e l'intensità media dei mitocondri è stata misurata e normalizzata al primo punto temporale. Vengono mostrati i risultati dei primi tre punti temporali (27 esposizioni). (B) Lunghezza d'onda di eccitazione: 405 nm. (C) Lunghezza d'onda di eccitazione: 488 nm. I valori mostrati sono le medie di tre cellule di ciascuno dei tre studi indipendenti. Fare clic qui per visualizzare una versione più grande di questa figura.
4. Analisi semiautomatica con script
- Genera e misura le immagini del rapporto utilizzando uno script di analisi del biosensore (ad esempio, biosensor.ijm o biosensor-image-subtraction.ijm).
- Selezionare lo script da utilizzare. Il protocollo per l'utilizzo di ogni script è simile; Eventuali differenze saranno specificate nel testo.
- biosensor.ijm: in questo script, lo sfondo e il rumore vengono corretti utilizzando aree dell'immagine selezionate dall'utente, valori fissi o nessuna sottrazione. Selezionare i metodi per la correzione dello sfondo e del rumore in modo indipendente.
- biosensor-image-subtraction.ijm: In questo script, lo sfondo e il rumore sono entrambi gestiti dallo stesso metodo selezionato dall'utente, che può essere uno dei seguenti: immagine vuota, area selezionata dall'utente all'interno dell'immagine, valore fisso o nessuna correzione.
- In Figi, aprire un'immagine dello stack Z multicanale acquisita nel passaggio 3.5. Aprire il file di script di analisi del biosensore desiderato. Eseguire lo script facendo clic su Esegui nella finestra Editor script . Nella finestra di dialogo visualizzata, immettere le informazioni richieste.
- Selezionare i numeri di canale del numeratore e del denominatore per il calcolo del rapporto. Per mtHyPer7, il numeratore è il canale eccitato a 488 nm e il denominatore è il canale eccitato a 405 nm. Selezionare il numero del canale dell'immagine a luce trasmessa, se presente, o 0 se non è presente.
- Selezionare il metodo di sottrazione dello sfondo desiderato tra le quattro opzioni seguenti. Se lo sfondo è relativamente uniforme, scegliete Seleziona un'area dell'immagine, che misura direttamente il livello di sfondo nell'immagine. Se lo sfondo varia in modo sostanziale sul campo, utilizzare lo script biosensor-image-subtraction.ijm e selezionare Immagine vuota (per raccogliere un'immagine vuota per la correzione, acquisire una pila Z multicanale con condizioni di acquisizione identiche in un campo visivo privo di celle o da un vetrino realizzato con terreno di coltura privo di cellule). Il valore fisso consente l'immissione di un valore di sfondo misurato in precedenza. Nessuno lascia lo sfondo non corretto, ma può ridurre la precisione della misurazione.
- Selezionare il metodo di sottrazione del rumore desiderato. Il valore del rumore viene utilizzato come soglia inferiore sulle immagini del canale mascherate sottratte dallo sfondo per ridurre l'effetto della variazione casuale nella lettura del rilevatore. Optare per Seleziona un'area dell'immagine o Valore fisso per consentire l'immissione di un livello di rumore misurato in precedenza, che di solito funziona bene poiché i livelli di rumore sono costanti se le condizioni di imaging vengono mantenute costanti. In alternativa, scegliere Nessuno e utilizzare un valore pari a 1 come soglia inferiore, che può aumentare la variabilità delle misurazioni.
- Selezionare un algoritmo di soglia per rilevare i mitocondri in modo accurato e coerente; Si consiglia Otsu o MaxEntropy . Idealmente, utilizzare lo stesso algoritmo per tutte le immagini in un esperimento, ma garantire il riconoscimento accurato dei mitocondri. Utilizzare un metodo di soglia diverso se si verificano cambiamenti nella morfologia durante un esperimento.
- Selezionare il numero di aree di interesse (ROI) per cella. Ad esempio, se si misurano le differenze madre-bocciolo, selezionare 2.
- Selezionare la cartella di output in cui verranno salvate le immagini delle misurazioni e dei rapporti.
- Segui le istruzioni per la correzione dello sfondo e del rumore.
- Selezione dell'area (se applicabile): se è stata scelta l'opzione Seleziona un'area dell'immagine per la misurazione dello sfondo o del rumore, seguire le istruzioni per disegnare un'area di sfondo (al di fuori di eventuali celle o artefatti fluorescenti) utilizzando lo strumento ROI rettangolare. Dopo aver disegnato l'area, fare clic su OK.
- Immissione di valori fissi (se applicabile): se è stato scelto un valore fisso per la misurazione del rumore di fondo o del rumore, seguire le istruzioni per immettere i valori di fondo e/o di rumore per ciascun canale di fluorescenza.
- Immagine di riferimento vuota (se applicabile): nello script biosensor-image-subtraction.ijm, se è stata scelta l'opzione Immagine vuota per la correzione dello sfondo o del rumore, seguire le istruzioni per selezionare un file di immagine vuoto.
- Segna il ROI per la misurazione. Nelle colture in fase intermedia, limitare l'analisi alle cellule gemmate.
- Disegna le ROI corrispondenti a singole cellule o regioni subcellulari in base all'immagine in campo chiaro. Non è necessario che il ROI corrisponda esattamente al contorno della cellula, poiché verranno misurati solo i mitocondri con soglia all'interno del ROI. In alternativa, apri un set di ROI salvato in precedenza: in Gestione ROI, fai clic su Altro, quindi seleziona il file ROI.
- Premere T dopo aver creato ogni ROI per aggiungere il ROI selezionato a ROI Manager. In Gestione ROI, selezionare Mostra tutto per documentare le celle contrassegnate. Ogni area aggiunta verrà visualizzata come elemento numerato nell'elenco ROI Manager. Se si analizza più di un ROI per cellula (ad esempio, madre e gemma), contrassegnare i ROI nello stesso ordine per ogni cellula analizzata.
- Dopo aver aggiunto tutte le ROI desiderate a Gestione ROI, fare clic su OK nella finestra di dialogo Contrassegna celle .
- Scegli il formato della tabella delle misure. Nella finestra di dialogo MultiMisura visualizzata, selezionare Misura tutte le sezioni. Selezionare l'opzione Una riga per sezione per produrre una tabella con il formato desiderato. Usa il process_multiROI_tables. Script R per elaborare le tabelle create con l'opzione Una riga per sezione ; non selezionare Aggiungi risultati. Ripetere questo passaggio 3 volte (per la misurazione delle immagini del numeratore [488], del denominatore [405] e del rapporto [488/405]).
- Lo script salverà i file di output nella cartella selezionata nel passaggio 4.1.2.6.
- Selezionare lo script da utilizzare. Il protocollo per l'utilizzo di ogni script è simile; Eventuali differenze saranno specificate nel testo.
- Calcolo dei rapporti medi ponderati per ogni regione o cella
- Utilizzando i risultati ottenuti nel passaggio precedente, calcolare i rapporti medi ponderati per ciascuna regione o cella utilizzando l'equazione (2). Calcola i rapporti "pixelwise" o "regionwise" (vedi la discussione per i dettagli).
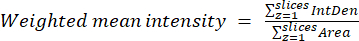 (2)
(2)
NOTA: i valori di area e densità integrata (IntDen) includono solo i pixel all'interno dei mitocondri con soglia. Questo calcolo può essere automatizzato utilizzando il process_multiROI_tables. Script R e software statistico R.
- Utilizzando i risultati ottenuti nel passaggio precedente, calcolare i rapporti medi ponderati per ciascuna regione o cella utilizzando l'equazione (2). Calcola i rapporti "pixelwise" o "regionwise" (vedi la discussione per i dettagli).
- Genera un'immagine con rapporto colorato. Poiché i cambiamenti di tonalità (colore) sono più evidenti all'occhio umano rispetto ai cambiamenti di intensità, convertire il valore del rapporto in una scala di colori per una più facile interpretazione visiva. Lo script colorize_ratio_image.ijm colorerà un'immagine con rapporto mascherato.
- In Figi, aprire un'immagine dello stack Z del rapporto generata nel passaggio 4.1.6. Aprire il file di script colorize_ratio_image.ijm. Eseguire lo script facendo clic su Esegui nella finestra Editor script . Nella finestra di dialogo visualizzata, immettere le informazioni richieste.
- Metodo di colorazione: nell'opzione Non modulato , tutti i pixel mitocondriali appaiono alla stessa luminosità. Tuttavia, alcune immagini possono apparire rumorose poiché sia i pixel deboli che quelli luminosi contribuiscono al rapporto dell'immagine. Per ridurre questo effetto, utilizzare l'opzione Intensità modulata .
- Valore minimo e massimo visualizzato: questi valori controllano l'intervallo di valori del rapporto che verranno colorati. Scegliere valori vicini ai valori medi minimo e massimo osservati in un esperimento (in base ai rapporti medi ponderati calcolati nel passaggio 4.2.1). All'interno di un esperimento, assicurarsi di acquisire tutte le immagini con condizioni di imaging identiche e visualizzare tutte le immagini con valori minimi e massimi identici.
- Modalità di proiezione: le pile Z vengono visualizzate come proiezioni per mostrare l'intera popolazione mitocondriale. La proiezione viene creata prima della colorazione. Selezionare Max per una proiezione di intensità massima (mantenendo il rapporto massimo e i valori di intensità) o Media per una proiezione di intensità media.
- Cartella di output: selezionare la cartella in cui salvare le immagini colorate.
- Se è stata selezionata l'opzione Non modulato , selezionare una combinazione di colori (tabella di ricerca; LUT) nella finestra di dialogo visualizzata. Utilizza le LUT RGB Fire o Rainbow integrate in Fiji o qualsiasi LUT desiderata nel formato .lut di ImageJ (importa da file LUT). Si consiglia la LUT Rainbow Smooth40 (inclusa nel file supplementare 2 e su GitHub).
- Lo script salva i file di output nella cartella selezionata nel passaggio 4.3.1.4. Questi includono l'immagine del rapporto colorato (Color_RGB.tif) e l'immagine del rapporto colorato con una barra di calibrazione che mostra la corrispondenza tra i valori del rapporto e il colore dell'immagine (Color_with_bar.tif).
- In Figi, aprire un'immagine dello stack Z del rapporto generata nel passaggio 4.1.6. Aprire il file di script colorize_ratio_image.ijm. Eseguire lo script facendo clic su Esegui nella finestra Editor script . Nella finestra di dialogo visualizzata, immettere le informazioni richieste.
5. Analisi manuale
NOTA: questo approccio richiede più tempo, ma consente flessibilità nella pre-elaborazione e nell'impostazione della soglia.
- Corretto per lo sfondo.
- Imposta le opzioni nelle Figi.
- Fare clic su Analizza | Impostare le misure. Nella finestra di dialogo visualizzata, selezionare le caselle Area, Media, DevdStandard, IntDen e Visualizza etichetta. Impostare le cifre decimali su 3.
- Fare clic su Modifica | Opzioni | Ingresso/Uscita. Nella finestra di dialogo visualizzata, selezionare le caselle Salva numeri di riga e Salva intestazioni di colonna.
- Per aumentare la sensibilità del calcolo del rapporto, sottrarre lo sfondo da ciascun canale di fluorescenza. Se lo sfondo è relativamente uniforme, misurare l'intensità media di un'area priva di celle in un'immagine e sottrarre questo valore dall'intera immagine (passaggio 5.1.2.1). In alternativa, se lo sfondo varia in modo sostanziale sul campo, utilizzare un'immagine vuota per la correzione (passaggio 5.1.2.2.).
- Per sottrarre una regione selezionata dall'utente, fare clic su Immagine | Colore | Dividi i canali e tieni aperte tutte le immagini dei canali, poiché saranno necessarie in seguito. Per ogni canale di fluorescenza, disegna un ROI sullo sfondo (all'esterno di qualsiasi cella) e fai clic su Analizza | Misurare, oppure, per le pile, fare clic su Immagine | Pile | Pila di misure. Osservare il valore medio del ROI visualizzato nella tabella Risultati . Fare clic su Modifica | Selezionare Nessuno, quindi Elabora | Matematica | Sottrai. Nella finestra di dialogo visualizzata, immettere il valore medio di sfondo misurato, arrotondato all'intero più vicino.
NOTA: l'arrotondamento impedisce errori con operazioni binarie in un secondo momento. - Per sottrarre un'immagine vuota da un file di dati, raccogliere un'immagine vuota da un campo visivo completamente privo di celle o da un vetrino preparato con terreno di coltura privo di cellule. Fare clic su Elabora | Calcolatore di immagini. Nella finestra di dialogo visualizzata, impostare l'operazione su Sottrai e impostare Immagine1 e Immagine2 rispettivamente sull'immagine della cella e sull'immagine vuota. Selezionare Crea nuova finestra e Risultato a 32 bit.
- Per sottrarre una regione selezionata dall'utente, fare clic su Immagine | Colore | Dividi i canali e tieni aperte tutte le immagini dei canali, poiché saranno necessarie in seguito. Per ogni canale di fluorescenza, disegna un ROI sullo sfondo (all'esterno di qualsiasi cella) e fai clic su Analizza | Misurare, oppure, per le pile, fare clic su Immagine | Pile | Pila di misure. Osservare il valore medio del ROI visualizzato nella tabella Risultati . Fare clic su Modifica | Selezionare Nessuno, quindi Elabora | Matematica | Sottrai. Nella finestra di dialogo visualizzata, immettere il valore medio di sfondo misurato, arrotondato all'intero più vicino.
- Imposta le opzioni nelle Figi.
- Per limitare l'analisi ai mitocondri, eseguire la segmentazione. Poiché ogni canale può cambiare intensità a seconda dello stato del biosensore, utilizzare la somma dei due canali per definire l'area dei mitocondri in modo coerente.
- Per creare un'immagine di somma, fare clic su Elabora | Calcolatore di immagini. Nella finestra di dialogo visualizzata, impostare l'operazione su Aggiungi e impostare Immagine1 e Immagine2 sui due canali fluorescenti sottratti dallo sfondo in qualsiasi ordine. Seleziona Crea nuova finestra e Risultato a 32 bit e attendi che appaia un'immagine di somma.
- Impostare la soglia per definire i mitocondri sull'immagine della somma.
- Clicca sull'immagine | Regola | Soglia. Nella finestra Soglia visualizzata, seleziona Sfondo scuro e Istogramma pila. Impostare Method sull'algoritmo desiderato (ad esempio, Otsu o MaxEntropy). Utilizzare la soglia automatica per la riproducibilità, ma se nessun metodo automatico è adatto, regolare manualmente la soglia.
NOTA: Idealmente, lo stesso algoritmo dovrebbe essere utilizzato per tutte le immagini di un esperimento, ma i cambiamenti nella morfologia durante un esperimento potrebbero richiedere un metodo di soglia diverso in alcune condizioni. - Quando la soglia è soddisfacente, fare clic su Applica. Nella finestra di dialogo visualizzata, scegliere Converti in maschera. Nella finestra di dialogo visualizzata, seleziona Sfondo nero e lascia deselezionate le altre caselle. Salvare l'immagine della maschera risultante per la valutazione dell'accuratezza della segmentazione.
- Clicca sull'immagine | Regola | Soglia. Nella finestra Soglia visualizzata, seleziona Sfondo scuro e Istogramma pila. Impostare Method sull'algoritmo desiderato (ad esempio, Otsu o MaxEntropy). Utilizzare la soglia automatica per la riproducibilità, ma se nessun metodo automatico è adatto, regolare manualmente la soglia.
- Convertire i valori della maschera da 0 e 255 a 0 e 1, rispettivamente, facendo clic su Elabora | Matematica | Dividere. Impostare il valore su 255 e, quando richiesto, selezionare l'opzione per elaborare tutte le immagini.
- Applicare la maschera ai canali di fluorescenza sottratti dallo sfondo facendo clic su Elabora | Calcolatore di immagini. Nella finestra di dialogo visualizzata, impostare l'operazione su Moltiplica. Impostate Image1 e Image2 rispettivamente sul canale del numeratore e sulla maschera. Seleziona Crea nuova finestra e Risultato a 32 bit; Ripetere la moltiplicazione della maschera con il canale del denominatore.
- Prepara ogni canale mascherato per il calcolo del rapporto impostando i pixel di sfondo su NaN ("non un numero").
- Selezionare il canale del numeratore mascherato e fare clic su Immagine | Regola | Soglia. Nella finestra Soglia , fare clic su Imposta, quindi nella finestra di dialogo visualizzata, impostare il valore minimo sul livello di rumore calcolato o su 1 e lasciare il valore massimo invariato.
- Nella finestra Soglia , fare clic su Applica. Nella finestra di dialogo successiva, scegliere Imposta su NaN. Ripetere l'operazione per il canale del denominatore mascherato.
- Genera l'immagine del rapporto.
- Fare clic su Elabora | Calcolatore di immagini. Nella finestra di dialogo visualizzata, impostare l'operazione su Dividi. Impostate Image1 e Image2 rispettivamente sui canali del numeratore mascherato e del denominatore sottratti dallo sfondo. Per mtHyPer7, il numeratore è la forma ossidata eccitata a 488 nm e il denominatore è la forma ridotta eccitata a 405 nm. Pertanto, rapporti più alti indicano H 2O2 più alti.
- Selezionare Crea nuova finestra e Risultato a 32 bit. Salvate l'immagine del rapporto per l'analisi.
- Contrassegnare e quantificare le aree di interesse. Ogni cellula o regione subcellulare viene selezionata e memorizzata come ROI in ROI Manager. I ROI non devono corrispondere perfettamente ai contorni delle cellule, poiché verranno misurate solo le regioni dei mitocondri mascherate.
- Utilizzando l'immagine a luce trasmessa per prevenire le distorsioni, creare ROI corrispondenti alle singole cellule (o regioni subcellulari). Nelle colture in fase intermedia, limitare l'analisi alle cellule di lievito portatrici di gemme, che sono attivamente impegnate nella divisione cellulare. Premere T dopo aver creato ogni ROI per aggiungerlo a ROI Manager. Selezionare Mostra tutto per documentare le celle contrassegnate.
- Fai clic sull'immagine del rapporto creata sopra e, in Gestione ROI, fai clic su Altro ... | MultiMisura. Nella finestra di dialogo visualizzata, selezionare Misura tutte le sezioni. Non selezionare Aggiungi risultati. Selezionare l'opzione Una riga per sezione per produrre una tabella con il formato desiderato. Il process_multiROI_tables. Lo script R elaborerà le tabelle create con l'opzione Una riga per sezione nel software statistico R.
- Salvare i risultati. Fare clic sulla finestra Risultati , quindi su File | Salvare e salvare la tabella dei risultati in formato .csv o .xls. Impostare il nome del file in modo che corrisponda al nome dell'immagine.
- Salva il ROI. In Gestione ROI, assicurati innanzitutto che non sia selezionata alcuna ROI: fai clic su Deseleziona, quindi su Altro | Salva. Apri le ROI salvate insieme all'immagine originale nelle Figi per incrociare le misurazioni con l'immagine.
- Calcolare i rapporti medi ponderati per cella o regione utilizzando i risultati ottenuti nel passaggio precedente e l'equazione (3). Calcola i rapporti "pixelwise" o "regionwise" (vedi la discussione per i dettagli).
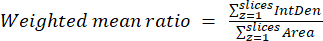 (3)
(3)
NOTA: i valori dell'area e della densità integrata includono solo i pixel all'interno dei mitocondri con soglia. Questo calcolo può essere automatizzato utilizzando il process_multiROI_tables. R nel software statistico R. - Genera un'immagine con rapporto colorato.
NOTA: Le immagini colorate possono essere non modulate, in cui tutti i pixel mitocondriali appaiono alla stessa luminosità, o modulate in intensità, in cui l'intensità dei pixel nell'immagine originale viene utilizzata per impostare le intensità nell'immagine colorata.- Per produrre un'immagine colorata non modulata:
- Aprire l'immagine del rapporto generata sopra. Clicca sull'immagine | Duplica per generare una copia dell'immagine, quindi chiudi l'immagine originale.
- Se l'immagine è una pila Z, selezionare una singola sezione o generare una proiezione Z per visualizzare tutti i mitocondri. Utilizzare una proiezione di intensità massima (mantenendo il rapporto massimo e i valori di intensità in corrispondenza di ciascuna coordinata pixel XY) o una proiezione di intensità media.
- Clicca sull'immagine | Tabelle di ricerca e impostare la LUT sulla combinazione di colori desiderata (ad esempio, Rainbow RGB o Fire [disponibile per impostazione predefinita in ImageJ]). In alternativa, fare clic su File | Importare la LUT e selezionare la LUT desiderata nel formato .lut di ImageJ, ad esempio Rainbow Smooth40 (incluso nel file supplementare 2 e su GitHub). Assicurati che la LUT abbia un colore scuro assegnato al valore 0.
- Impostare il contrasto del display facendo clic su Immagine | Regola | Luminosità/Contrasto. Nella finestra Luminosità/Contrasto, fare clic su Imposta e, nella finestra di dialogo visualizzata, immettere i valori desiderati per i valori Minimo e Massimo visualizzati. Per massimizzare le differenze di colore osservate, impostarle approssimativamente al minimo e al massimo ottenuti in tutte le immagini. Impostare tutte le immagini di un esperimento sugli stessi livelli di contrasto.
NOTA: Non fare clic su Applica, poiché ciò modificherà i valori dei pixel e impedirà la generazione di una barra di calibrazione accurata nel passaggio successivo. - Aggiungi una barra di calibrazione del colore facendo clic su Analizza | Strumenti | Barra di calibrazione. Nella finestra di dialogo che appare, selezionare l'opzione Sovrapponi per rimuovere la barra, se lo si desidera, facendo clic su Immagine | Sovrapposizione | Rimuovi sovrapposizione.
- Se lo si desidera, aggiungere una barra di scala facendo clic su Analizza | Strumenti | Barra della scala. Nella finestra di dialogo visualizzata, impostare le dimensioni, la posizione e il colore desiderati per la barra. Selezionare l'opzione Sovrapponi per mantenere il colore della barra indipendentemente dalla LUT utilizzata.
- Generare un'immagine RGB per la pubblicazione facendo clic su Immagine | Sovrapposizione | Appiattire. Salvate l'immagine risultante.
NOTA: questa immagine è solo per la presentazione o la pubblicazione. I valori di intensità vengono alterati, quindi non può essere misurato. Anche le barre vengono bruciate in modo permanente sull'immagine.
- Per creare un'immagine a intensità modulata:
- Clicca sull'immagine | Duplica per generare una copia dell'immagine, quindi chiudi l'originale.
- Se l'immagine è una pila Z, selezionare una singola sezione o generare una proiezione Z per visualizzare tutti i mitocondri.
- Impostare il contrasto di visualizzazione dell'immagine in rapporto facendo clic su Immagine | Regola | Luminosità/Contrasto, quindi nella finestra Luminosità/Contrasto, fare clic su Imposta. Nella finestra di dialogo visualizzata, immettere i valori desiderati per i valori Minimo e Massimo visualizzati. Per massimizzare le differenze di colore osservate, impostarle approssimativamente sul minimo e sul massimo ottenuti in tutte le immagini e impostare tutte le immagini in un esperimento sugli stessi livelli di contrasto. Fare clic su Applica per ridimensionare i valori dei pixel.
- Per creare una barra di calibrazione, duplicare l'immagine migliorata e generare una barra passando a Analizza | Strumenti | Barra di calibrazione. Converti l'immagine e la sovrapposizione in formato RGB facendo clic su Immagine | Sovrapposizione | Appiattire. Se lo si desidera, incollare questa barra sull'immagine RGB a modulazione di intensità ottenuta al punto 5.6.2.12 per la presentazione o la pubblicazione.
- Crea una nuova immagine con la stessa larghezza e altezza dell'immagine facendo clic su File | Nuovo | Immagine.... Nella finestra di dialogo che si apre, impostare i parametri come segue: Tipo: RGB; Riempire con: Nero; Larghezza e altezza: 9larghezza e altezza dell'immagine); Fette: 1.
- Converti la nuova pila in una pila di immagini HSB (tonalità, saturazione, luminosità) facendo clic su Immagine | Tipologia | Stack HSB.
- Fare clic sull'immagine con rapporto di contrasto regolato accedendo a Modifica | Seleziona tutto, quindi Modifica | Copia. Quindi, fai clic sullo stack HSB, seleziona la prima sezione (Tonalità) e fai clic su Modifica | Incolla per trasferire i valori del rapporto.
- Selezionare la sezione 2 (Saturazione) dello stack HSB. Impostare il colore di primo piano su bianco passando a Modifica | Opzioni | Colori. Fare clic su Modifica | Seleziona tutto, quindi Modifica | Riempi e, nella finestra di dialogo visualizzata, selezionare No per riempire di bianco solo la sezione corrente.
- Apri l'immagine dei dati grezzi e dividi i canali facendo clic su Immagine | Colore | Canali divisi.
- Generare la media dei due canali di rapporto utilizzando il Calcolatore di immagini e facendo clic su Elabora | Calcolatore di immagini. Nella finestra di dialogo visualizzata, impostare l'operazione su Media e impostare Immagine1 e Immagine2 rispettivamente sulle immagini del numeratore e del denominatore. Seleziona Crea nuova finestra.
- Fai clic sull'immagine media creata sopra navigando su Modifica | Seleziona tutto, quindi Modifica | Copia. Quindi, fare clic sullo stack HSB, selezionare la terza sezione (Valore) e fare clic su Modifica | Incolla per trasferire i valori di intensità.
- Converti lo stack HSB nello spazio colore RGB facendo clic su Immagine | Tipologia | Colore RGB. Salvate l'immagine risultante.
- Per produrre un'immagine colorata non modulata:
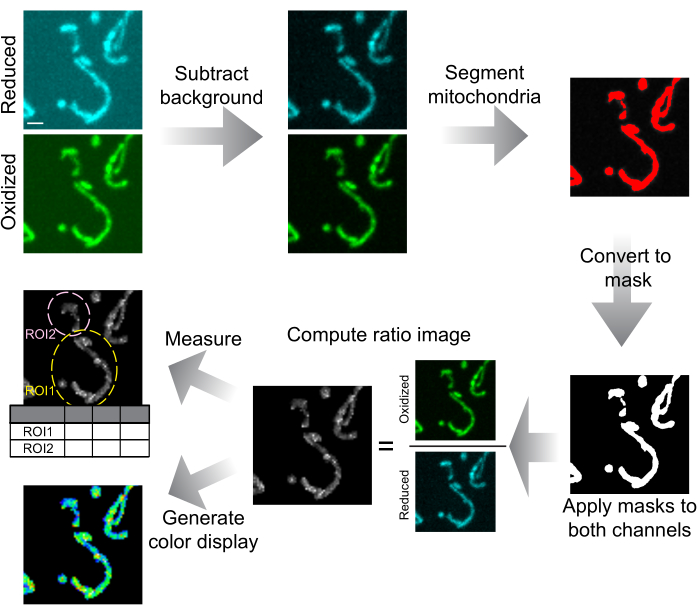
Figura 6: Schema di analisi e presentazione delle immagini. Le immagini confocali del disco rotante vengono prima sottratte sullo sfondo. I mitocondri vengono segmentati in base alla soglia e convertiti in maschere per ogni fetta. Queste maschere vengono applicate a entrambi i canali e le immagini mascherate vengono utilizzate per calcolare l'immagine del rapporto. I ROI vengono disegnati per misurare il rapporto mtHyPer7 nelle cellule o nelle regioni subcellulari. È inoltre possibile generare immagini con rapporto colorato per la presentazione dei dati. Barra della scala = 1 μm. Abbreviazione: ROI = regione di interesse. Fare clic qui per visualizzare una versione più grande di questa figura.
Risultati
Per confermare che mtHyPer7 è una sonda adatta per valutare l'H 2 O2mitocondriale, è stata valutata la localizzazione di mtHyPer7 e il suo effetto sui mitocondri del lievito e sulla salute delle cellule. Per valutare il targeting di mtHyPer7, i mitocondri sono stati colorati con il colorante vitale specifico per i mitocondri MitoTracker Red nelle cellule di lievito di controllo e nel lievito che esprime mtHyPer7. Utilizzando la colorazione MitoTracker Red, i mitocondri sono stati risolti come lunghe strutture tubolari che si allineavano lungo l'asse madre-gemma e si accumulavano nella punta della gemma e nella punta della cellula madre distale alla gemma41. La morfologia mitocondriale era simile nelle cellule di controllo e nelle cellule esprimenti mtHyPer7. Inoltre, mtHyPer7 è co-localizzato con i mitocondri colorati di rosso di MitoTracker (Figura 3A). Pertanto, mtHyPer7 è stato mirato in modo efficiente e quantitativo ai mitocondri senza influenzare la normale morfologia o distribuzione mitocondriale.
Successivamente, sono stati eseguiti ulteriori esperimenti di convalida per valutare l'effetto di mtHyPer7 sulla fitness cellulare e sulla sensibilità allo stress ossidativo nei mitocondri. I tassi di crescita delle cellule che esprimono mtHyPer7 in terreni ricchi o sintetici a base di glucosio (YPD o SC, rispettivamente) sono simili a quelli delle cellule wild-type non trasformate (Figura 3B,D). Per valutare i possibili effetti sullo stress ossidativo nei mitocondri, il lievito di controllo e che esprime mtHyPer7 è stato trattato con bassi livelli di paraquat, una piccola molecola redox-attiva che si accumula nei mitocondri e provoca elevati livelli di superossido nell'organello24,27. Se l'espressione di mtHyPer7 induce stress ossidativo nei mitocondri o protegge i mitocondri dallo stress ossidativo, allora le cellule che esprimono mtHyPer7 dovrebbero mostrare una maggiore o minore sensibilità al trattamento con paraquat, rispettivamente. Il trattamento con bassi livelli di paraquat ha portato a una diminuzione dei tassi di crescita del lievito. Inoltre, i tassi di crescita delle cellule wild-type e delle cellule che esprimono mtHyPer7 in presenza di paraquat erano simili (Figura 3C,E). Pertanto, l'espressione del biosensore non ha creato stress cellulari significativi nelle cellule di lievito in gemmazione né alterato la sensibilità del lievito allo stress ossidativo mitocondriale.
Data l'evidenza che mtHyPer7 è adatto per lo studio dell'H 2 O 2 mitocondriale nel lievito in gemmazione, è stato testato se mtHyPer7 potesse rilevare H 2 O 2 mitocondriale nel lievito wild-type in fase intermedia e i cambiamenti nell'H 2 O 2 mitocondriale indotti dall'aggiunta esterna di H 2 O 2. È stato eseguitoun esperimento di titolazione H 2 O2 ed è stato misurato il rapporto medio ossidato:ridotto (O/R) mtHyPer7 nei mitocondri.
Gli script di analisi automatizzata hanno prodotto diversi file di output.
L'immagine del rapporto (ratio.tif): una pila Z costituita dal rapporto delle pile corrette per lo sfondo a 488 nm e 405 nm. Questo stack può essere visualizzato nelle Figi senza ulteriori elaborazioni; Tuttavia, si consiglia di migliorare il contrasto e/o colorare l'immagine come descritto nei passaggi 4.3 o 5.6 del protocollo prima della visualizzazione.
L'immagine della maschera (mask.tif): una pila Z costituita dalle aree mitocondriali soglie utilizzate per l'analisi. Questa immagine deve essere utilizzata per valutare l'accuratezza della soglia.
I ROI utilizzati per l'analisi (ROIs.zip): quando questo file viene aperto nelle Figi, insieme all'immagine di origine, i ROI vengono sovrapposti all'immagine per incrociare la tabella dei risultati e l'immagine di origine. Ogni ROI viene rinominata con un numero di cella e un numero di ROI.
Tabelle di misurazione (NumResults.csv, DenomResults.csv, Results.csv) contenenti rispettivamente l'area, la media e la densità integrata dei mitocondri con soglia per ciascuna fetta nelle immagini del numeratore, del denominatore e del rapporto. Nelle sezioni in cui i mitocondri erano assenti o sfocati, viene registrato NaN.
Un file di log (Log.txt) che documenta le opzioni utilizzate per la correzione dello sfondo e del rumore, la soglia e il calcolo del rapporto.
Il rapporto O/R di mtHyPer7 ha mostrato una risposta dose-dipendente alla concentrazione di H 2 O 2, che ha raggiunto un plateau a 1-2 mM di H 2 O 2aggiunti esternamente (Figura 7). Sorprendentemente, il rapporto HyPer7 era più basso nelle cellule esposte a 0,1 mM H 2 O2rispetto alle cellule di controllo, sebbene questa differenza non fosse statisticamente significativa. Una spiegazione per questo fenomeno può essere una risposta all'ormesi, in cui l'esposizione a un basso livello di stressor può indurre risposte allo stress, come meccanismi antiossidanti, che a loro volta riducono la quantità di ROS rilevabili dalla sonda. Livelli più elevati di fattori di stress, al contrario, possono sopraffare le risposte interne allo stress e comportare una lettura più elevata di HyPer7.
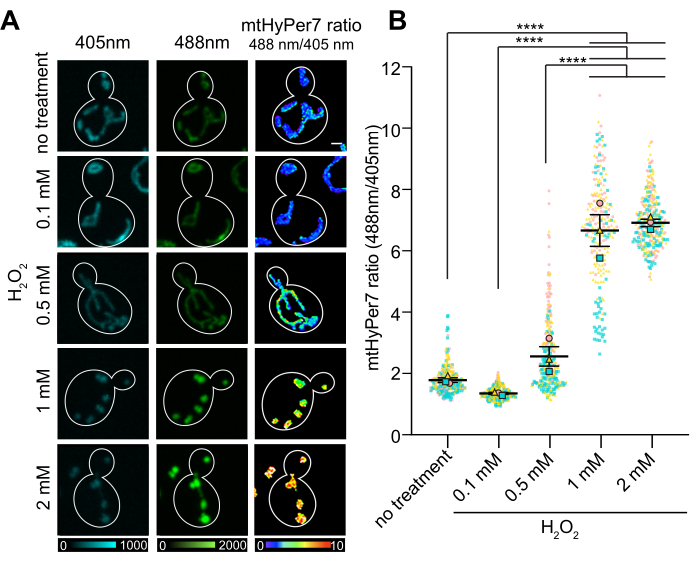
Figura 7: Risposta di mtHyPer7 all'aggiunta esterna di H 2 O 2. (A) Proiezione massima di immagini eccitate a 405 nm e 488 nm e proiezione di intensità media delle immagini raziometriche di mtHyPer7 in cellule in fase logaritmica media esposte a diverse concentrazioni di H 2 O 2. Lo pseudocolore indica il rapporto mtHyPer7 ossidato:ridotto (scala in basso). Barra della scala = 1 μm. Il contorno della cella è mostrato in bianco; n > 100 celle per condizione. (B) Quantificazione del rapporto mtHyPer7 ossidato:ridotto in cellule di lievito in gemmazione trattate con diverse concentrazioni di H2 O2 . Vengono mostrati i mezzi di cinque prove indipendenti, con simboli di forma e colore diversi per ogni prova. Media ± SEM del rapporto mtHyPer7 ossidato:ridotto: 1,794 ± 0,07627 (nessun trattamento), 1,357 ± 0,03295 (0,1 mM), 2,571 ± 0,3186 (0,5 mM), 6,693 ± 0,5194 (1 mM), 7,017 ± 0,1197 (2 mM). p < 0,0001 (ANOVA unidirezionale con test di confronto multiplo di Tukey). I valori di p sono indicati come: ****p < 0,0001. Fare clic qui per visualizzare una versione più grande di questa figura.
Infine, studi precedenti hanno rivelato che i mitocondri più in forma, che sono più ridotti e contengono meno superossido, sono ereditati preferenzialmente dalle cellule figlie del lievito 4 e che le catalasi citosoliche sono trasportate e attivate nelle cellule figliedel lievito42,43,44,45. Questi studi documentano l'ereditarietà asimmetrica dei mitocondri durante la divisione delle cellule di lievito e un ruolo per questo processo nella forma fisica e nella durata della vita delle cellule figlie, nonché l'asimmetria dell'età madre-figlia. Per verificare se esistono differenze in H2 O2 tra madri e gemme, mtHyPer7 è stato misurato in gemme e cellule madri. Sono state osservate differenze nell'H 2O 2 mitocondriale all'interno delle cellule di lievito e una diminuzione sottile ma statisticamente significativa nella lettura del biosensore H 2 O 2 è stata rilevata nei mitocondri nel germoglio rispetto a quelli nella cellula madre (Figura 8). Questi risultati sono coerenti con i risultati precedenti secondo cui i mitocondri nel germoglio sono meglio protetti dallo stress ossidativo. Forniscono anche la documentazione che mtHyPer7 può fornire una lettura quantitativa per H 2 O2mitocondriale con risoluzione cellulare e subcellulare nel lievito in gemmazione.
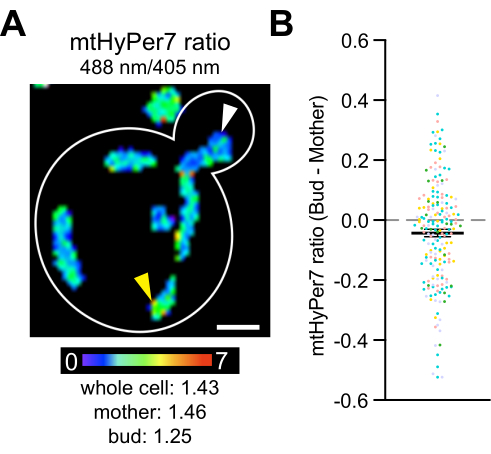
Figura 8: Il livello mitocondrialedi H 2 O2 è più basso nella cellula figlia. (A) Proiezione massima di un'immagine raziometrica di mtHyPer7 in una cella rappresentativa. Lo pseudocolore indica il rapporto mtHyPer7 ossidato:ridotto (scala in basso). Le differenze subcellulari nel rapporto sono evidenti (punte di freccia). Barra della scala = 1 μm. Contorno della cella: bianco. (B) Differenza tra il rapporto mtHyPer7 nella gemma e nella cellula madre. Per ogni singola cella, il valore del rapporto madre è stato sottratto dal valore del rapporto di gemma e tracciato come un punto. n = 193 cellule raggruppate da cinque esperimenti indipendenti, mostrate con simboli di colore diverso per ogni esperimento. Media ± SEM della differenza tra il rapporto mtHyPer7 nella gemma e nelle cellule madri: -0,04297 ± 0,01266. p = 0,0008 (test t accoppiato) Fare clic qui per visualizzare una versione più grande di questa figura.
Tabella supplementare S1: Ceppi utilizzati in questo studio. Elenco dei ceppi di lievito utilizzati. Fare clic qui per scaricare il file.
Tabella supplementare S2: Plasmidi utilizzati in questo studio. Elenco dei plasmidi utilizzati. Fare clic qui per scaricare il file.
Tabella supplementare S3: Primer utilizzati in questo studio. Elenco dei primer utilizzati. Fare clic qui per scaricare il file.
File supplementare 1: Protocollo per la costruzione di plasmidi biosensori. Fare clic qui per scaricare il file.
File supplementare 2: Script per l'analisi automatizzata. Per ogni script vengono forniti file di input e file di output di esempio. Fare clic qui per scaricare il file.
Discussione
In questo protocollo, viene descritto un metodo per l'utilizzo di mtHyPer7 come biosensore per valutare l'H 2O2 mitocondriale in cellule di lievito viventi in gemmazione. Il biosensore è costruito utilizzando un metodo basato su CRISPR e introdotto in una regione gene-free conservata nel genoma del lievito senza l'uso di marcatori selezionabili. Rispetto ai biosensori a base di plasmidi, quelli integrati sono espressi in tutte le cellule e a livelli coerenti, fornendo risultati di quantificazione più affidabili. Non vengono utilizzati marcatori selezionabili per generare cellule che esprimono mtHyPer7, il che consente l'uso di una gamma più ampia di background di ceppi e facilita la modificazione genetica delle cellule che esprimono biosensori. La proteina mtHyPer7 è correttamente indirizzata ai mitocondri senza effetti evidenti sulla morfologia mitocondriale, sulla funzione, sulla distribuzione o sui tassi di crescita cellulare. mtHyPer7 mostra una risposta dose-dipendente all'aggiunta esterna di H2 O2 . Inoltre, mtHyPer7 è in grado di riportare l'eterogeneità della qualità mitocondriale con risoluzione subcellulare. Infine, l'utilizzo di un microscopio confocale a disco rotante rispetto alla microscopia ad ampio campo per l'imaging di biosensori mirati ai mitocondri provoca meno fotosbiancamento dei fluorofori e genera immagini ad alta risoluzione per l'analisi delle differenze subcellulari.
Limiti e approcci alternativi
Questo metodo non è adatto per l'imaging di cellule per più di 10 minuti, poiché le cellule si asciugheranno sotto il vetrino coprioggetto. Per l'imaging a lungo termine, è meglio utilizzare il metodo46 del tampone di agar o immobilizzare le cellule in una piastra di coltura con fondo di vetro riempita con terreno SC.
La scelta del biosensore deve essere guidata dalla concentrazione del bersaglio in condizioni sperimentali. Se la sensibilità di HyPer7 è troppo alta, si consiglia una versione diversa di HyPer, come HyPer3 o HyPerRed47,48. Tuttavia, va notato che altre sonde HyPer sono più sensibili al pH. Per una maggiore sensibilità, potrebbe essere più appropriata la roGFP a base di perossiredossina (roGFP2Tsa2ΔCR)27.
Lo stato stazionario di ossidazione del sensore H 2 O2è legato sia ai tassi di ossidazione che a quelli di riduzione. Il tasso di ossidazione dei biosensori è causato principalmente daH 2 O2 , ma il tasso di riduzione dipende dai sistemi di riduzione antiossidante attivi nella cellula e nell'organello. È stato dimostrato che HyPer7 è prevalentemente ridotto dal sistema della tioredossina nel citosol del lievito e la sua riduzione è più rapida di quella di roGFP2Tsa2ΔCR27. Pertanto, i diversi meccanismi di riduzione e le dinamiche di risposta della sonda dovrebbero essere presi in considerazione quando si interpretanole misurazioni dei biosensori H 2 O2. In particolare, per dedurre i livelli di H 2 O2dalla lettura del biosensore, si deve presumere che il sistema di riduzione mantenga una capacità costante durante l'esperimento. In alternativa agli script qui descritti, è stata resa disponibile gratuitamente una varietà di altri software per l'analisi dei sensori redox49.
Passaggi critici
Con qualsiasi biosensore, è fondamentale dimostrare che il biosensore stesso non influisce sul processo misurato. Pertanto, è importante confrontare la crescita e la morfologia mitocondriale dei ceppi in ciascuna condizione sperimentale. In questo caso, la morfologia mitocondriale viene valutata utilizzando MitoTracker Red, che colora i mitocondri in modo dipendente dal potenziale di membrana. Tuttavia, il confronto dei mitocondri nelle cellule non trasformate e trasformate in biosensori può essere realizzato colorando con tetrametilrodammina estere metilico (TMRM), un colorante vitale mitocondriale alternativo sensibile al potenziale di membrana, o MitoTracker Green, che colora i mitocondri indipendentemente dal potenziale di membrana. Se si sospettano effetti deleteri, può essere utile ridurre il livello di espressione o modificare il sito di integrazione.
Anche la convalida del comportamento dose-risposta della sonda e il rapporto segnale/rumore della tecnica di imaging sono essenziali per raccogliere risultati affidabili. Se la variabilità all'interno di un gruppo supera la variabilità tra i gruppi, le differenze diventano difficili da rilevare. La variabilità intragruppo può derivare da una reale variazione della popolazione o dal rumore nel processo di rilevamento. I passaggi chiave per aumentare il rapporto segnale/rumore sono l'acquisizione dell'immagine (intervallo di valori dei pixel e rumore), la sottrazione dello sfondo e la soglia.
Gli effetti del rumore possono anche essere ridotti durante le fasi di calcolo. L'approccio più semplice consiste nel calcolare l'intensità media ponderata dalle misurazioni dell'immagine del rapporto (Risultati.csv), dove ogni pixel rappresenta il rapporto locale tra le efficienze di eccitazione. Questo produce un rapporto "pixelwise". Tuttavia, se il rapporto segnale/rumore dell'immagine è basso, è possibile ottenere risultati più affidabili calcolando l'intensità media ponderata per un ROI sia nel canale del numeratore che in quello del denominatore, e quindi calcolando il rapporto tra queste due medie ponderate (rapporto "regionwise").
Per selezionare un metodo di soglia, il comando Figi Immagine | Regola | Auto Threshold può essere utilizzato per provare automaticamente tutti i metodi Fiji incorporati. Per valutare la segmentazione (soglia), una maschera salvata viene convertita in una selezione facendo clic su Modifica | Selezione | Crea selezione, aggiunto a ROI Manager (premendo T) e quindi attivato sul file immagine raw. Se i mitocondri non vengono rilevati adeguatamente, è necessario tentare un metodo di segmentazione diverso.
Quando si confrontano le immagini, è essenziale acquisire tutte le immagini con condizioni di imaging identiche, nonché visualizzare tutte le immagini con un miglioramento del contrasto identico.
Il movimento mitocondriale deve essere preso in considerazione quando si ottimizzano le condizioni di imaging. Se i mitocondri si muovono in modo significativo tra l'eccitazione a 405 e 488 nm, l'immagine del rapporto non sarà accurata. Si consiglia di mantenere il tempo di esposizione <500 ms e di modificare l'eccitazione con il metodo più veloce disponibile (ad es. un impulso di trigger o un filtro sintonizzabile acusto-ottico). Quando si acquisisce una pila Z, entrambe le eccitazioni devono essere eseguite per ogni fase Z prima di passare alla fase Z successiva.
Per la visualizzazione dei risultati, i cambiamenti di tonalità (colore) sono più evidenti all'occhio umano rispetto ai cambiamenti di intensità. Pertanto, il valore del rapporto viene convertito in una scala di colori per facilitare l'interpretazione visiva. Le immagini colorate possono essere non modulate, in cui tutti i pixel mitocondriali appaiono alla stessa luminosità, o modulate in intensità, in cui l'intensità dei pixel nell'immagine originale viene utilizzata per impostare le intensità nell'immagine colorata.
Modifica e risoluzione dei problemi
In alternativa alla conferma della funzione mitocondriale mediante la sfida con il paraquat, le cellule possono essere replicate o inoculate in fonti di carbonio fermentabili e non fermentabili.
Per la sottrazione di sfondo, la sottrazione di sfere rotolanti (passando a Elaborazione | Sottrai sfondo...) Può anche essere utilizzato per rimuovere la non uniformità dell'illuminazione. È necessario assicurarsi che la presenza di celle non alteri lo sfondo sottratto (selezionando l'opzione Crea sfondo ed esaminando il risultato).
In sintesi, la sonda mtHyPer7 fornisce un metodo coerente e minimamente invasivo per mettere in relazione lo stato morfologico e funzionale dei mitocondri del lievito nelle cellule viventi e consente lo studio di un importante fattore di stress cellulare e molecola di segnalazione in un sistema modello geneticamente trattabile e facilmente accessibile.
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse.
Riconoscimenti
Gli autori ringraziano Katherine Filpo Lopez per l'assistenza tecnica esperta. Questo lavoro è stato sostenuto da sovvenzioni del National Institutes of Health (NIH) (GM122589 e AG051047) a LP.
Questi studi hanno utilizzato la risorsa condivisa di microscopia confocale e specializzata dell'Herbert Irving Comprehensive Cancer Center della Columbia University, finanziata in parte attraverso il NIH/NCI Cancer Center Support Grant P30CA013696.
Materiali
| Name | Company | Catalog Number | Comments |
| 100x/1.45 Plan Apo Lambda objective lens | Nikon | MRD01905 | |
| Adenine sulfate | Sigma-Aldrich | A9126 | |
| Bacto Agar | BD Difco | DF0145170 | |
| Bacto Peptone | BD Difco | DF0118170 | |
| Bacto Tryptone | BD Difco | DF211705 | |
| Bacto Yeast Extract | BD Difco | DF0127179 | |
| BamHI | New England Biolabs | R0136S | |
| BglII | New England Biolabs | R0144S | |
| Carbenicilin | Sigma-Aldrich | C1389 | |
| Carl Zeiss Immersol Immersion Oil | Carl Zeiss | 444960 | |
| Dextrose (D-(+)-Glucose) | Sigma-Aldrich | G8270 | |
| E. cloni 10G chemical competent cell | Bioserch Technologies | 60108 | |
| FIJI | NIH | Schindelin et al 2012 | |
| G418 (Geneticin) | Sigma-Aldrich | A1720 | |
| GFP emission filter | Chroma | 525/50 | |
| Gibson assembly | New England Biolabs | E2611 | |
| Graphpad Prism 7 | GraphPad | https://www.graphpad.com/scientific-software/prism/ | |
| H2O2 (stable) | Sigma-Aldrich | H1009 | |
| HO-pGPD-mito-roGFP-KanMX6-HO | Pon Lab | JYE057/EP41 | Liao et al 20201 |
| Incubator Shaker | New Brunswick Scientific | E24 | |
| KAPA HiFi PCR kit | Roche Sequencing and Life Science, Kapa Biosystems, Wilmington, MA | KK1006 | |
| L-arginine hydrochloride | Sigma-Aldrich | A8094 | |
| laser | Agilent | 405 and 488 nm | |
| L-histidine hydrochloride | Sigma-Aldrich | H5659 | |
| L-leucine | Sigma-Aldrich | L8912 | |
| L-lysine hydrochloride | Sigma-Aldrich | L8662 | |
| L-methionine | Sigma-Aldrich | M9625 | |
| L-phenylalanine | Sigma-Aldrich | P5482 | |
| L-tryptophan | Sigma-Aldrich | T8941 | |
| L-tyrosine | Sigma-Aldrich | T8566 | |
| mHyPer7 plasmid | This study | JYE116 | |
| Microscope coverslips | ThermoScientific | 3406 | #1.5 (170 µm thickness) |
| Microscope slides | ThermoScientific | 3050 | |
| MitoTracker Red CM-H2Xros | ThermoFisherScientific | M7513 | |
| NaCl | Sigma-Aldrich | S9888 | |
| NEBuilder HiFi Assembly Master Mix | New England Biolabs | E2621 | |
| Nikon Elements | Nikon | Microscope acquisition software | |
| Nikon Ti Eclipse inverted microscope | Nikon | ||
| Paraquat (Methyl viologen dichloride hydrate) | Sigma-Aldrich | Cat. 856177 | |
| RStudio | Posit.co | Free desktop version | |
| Spectrophotometer | Beckman | BU530 | |
| Stagetop incubator | Tokai Hit | INU | |
| Uracil | Sigma-Aldrich | U1128 | |
| Yeast nitrogen base (YNB) containing ammonium sulfate without amino acids | BD Difco | DF0919073 | |
| YN2_1_LT58_X2site | Addgene | 177705 | Pianale et al 2021 |
| Zyla 4.2 sCMOS camera | Andor |
Riferimenti
- vander Bliek, A. M., Sedensky, M. M., Morgan, P. G. Cell biology of the mitochondrion. Genetics. 207 (3), 843-871 (2017).
- McBride, H. M., Neuspiel, M., Wasiak, S. Mitochondria: more than just a powerhouse. Current Biology. 16 (14), 551-560 (2006).
- Shi, R., Hou, W., Wang, Z. -. Q., Xu, X. Biogenesis of iron-sulfur clusters and their role in DNA metabolism. Frontiers in Cell and Developmental Biology. 9, 735678 (2021).
- McFaline-Figueroa, J. R., et al. Mitochondrial quality control during inheritance is associated with lifespan and mother-daughter age asymmetry in budding yeast. Aging Cell. 10 (5), 885-895 (2011).
- Higuchi-Sanabria, R., et al. Mitochondrial anchorage and fusion contribute to mitochondrial inheritance and quality control in the budding yeast Saccharomyces cerevisiae. Molecular Biology of the Cell. 27 (5), 776-787 (2016).
- Higuchi-Sanabria, R., et al. Role of asymmetric cell division in lifespan control in Saccharomyces cerevisiae. FEMS Yeast Research. 14 (8), 1133-1146 (2014).
- Lam, Y. T., Aung-Htut, M. T., Lim, Y. L., Yang, H., Dawes, I. W. Changes in reactive oxygen species begin early during replicative aging of Saccharomyces cerevisiae cells. Free Radical Biology & Medicine. 50 (8), 963-970 (2011).
- Laun, P., et al. Aged mother cells of Saccharomyces cerevisiae show markers of oxidative stress and apoptosis. Molecular Microbiology. 39 (5), 1166-1173 (2001).
- Doudican, N. A., Song, B., Shadel, G. S., Doetsch, P. W. Oxidative DNA damage causes mitochondrial genomic instability in Saccharomyces cerevisiae. Molecular and Cellular Biology. 25 (12), 5196-5204 (2005).
- Roca-Portoles, A., Tait, S. W. G. Mitochondrial quality control: from molecule to organelle. Cellular and Molecular Life Sciences. 78 (8), 3853-3866 (2021).
- Sies, H., Berndt, C., Jones, D. P. Oxidative stress. Annual Review of Biochemistry. 86, 715-748 (2017).
- Sies, H., Jones, D. P. Reactive oxygen species (ROS) as pleiotropic physiological signalling agents. Nature Reviews Molecular Cell Biology. 21 (7), 363-383 (2020).
- Imlay, J. A., Fridovich, I. Assay of metabolic superoxide production in Escherichia coli. The Journal of Biological Chemistry. 266 (11), 6957-6965 (1991).
- Fridovich, I. Mitochondria: are they the seat of senescence. Aging Cell. 3 (1), 13-16 (2004).
- Quinlan, C. L., Perevoshchikova, I. V., Hey-Mogensen, M., Orr, A. L., Brand, M. D. Sites of reactive oxygen species generation by mitochondria oxidizing different substrates. Redox Biology. 1 (1), 304-312 (2013).
- Griendling, K. K., Minieri, C. A., Ollerenshaw, J. D., Alexander, R. W. Angiotensin II stimulates NADH and NADPH oxidase activity in cultured vascular smooth muscle cells. Circulation Research. 74 (6), 1141-1148 (1994).
- Griendling, K. K., Sorescu, D., Ushio-Fukai, M. NAD(P)H oxidase: role in cardiovascular biology and disease. Circulation Research. 86 (5), 494-501 (2000).
- Edmondson, D. E., Binda, C., Wang, J., Upadhyay, A. K., Mattevi, A. Molecular and mechanistic properties of the membrane-bound mitochondrial monoamine oxidases. Biochemistry. 48 (20), 4220-4230 (2009).
- Ramsay, R. R., Singer, T. P. The kinetic mechanisms of monoamine oxidases A and B. Biochemical Society Transactions. 19 (1), 219-223 (1991).
- Ramsay, R. R. Kinetic mechanism of monoamine oxidase A. Biochemistry. 30 (18), 4624-4629 (1991).
- Handy, D. E., Loscalzo, J. Redox regulation of mitochondrial function. Antioxidants & Redox Signaling. 16 (11), 1323-1367 (2012).
- Wood, Z. A., Schröder, E., Robin Harris, J., Poole, L. B. Structure, mechanism and regulation of peroxiredoxins. Trends in Biochemical Sciences. 28 (1), 32-40 (2003).
- Slade, L., et al. Examination of the superoxide/hydrogen peroxide forming and quenching potential of mouse liver mitochondria. Biochimica et Biophysica Acta. General Subjects. 1861 (8), 1960-1969 (2017).
- Pak, V. V., et al. Ultrasensitive genetically encoded indicator for hydrogen peroxide identifies roles for the oxidant in cell migration and mitochondrial function. Cell Metabolism. 31 (3), 642-653 (2020).
- Topell, S., Hennecke, J., Glockshuber, R. Circularly permuted variants of the green fluorescent protein. FEBS Letters. 457 (2), 283-289 (1999).
- Belousov, V. V., et al. Genetically encoded fluorescent indicator for intracellular hydrogen peroxide. Nature Methods. 3 (4), 281-286 (2006).
- Kritsiligkou, P., Shen, T. K., Dick, T. P. A comparison of Prx- and OxyR-based H2O2 probes expressed in S. cerevisiae. The Journal of Biological Chemistry. 297 (1), 100866 (2021).
- Baird, G. S., Zacharias, D. A., Tsien, R. Y. Circular permutation and receptor insertion within green fluorescent proteins. Proceedings of the National Academy of Sciences. 96 (20), 11241-11246 (1999).
- Abedi, M. R., Caponigro, G., Kamb, A. Green fluorescent protein as a scaffold for intracellular presentation of peptides. Nucleic Acids Research. 26 (2), 623-630 (1998).
- Onukwufor, J. O., et al. A reversible mitochondrial complex I thiol switch mediates hypoxic avoidance behavior in C. elegans. Nature Communications. 13 (1), 2403 (2022).
- Vega, M., et al. Antagonistic effects of mitochondrial matrix and intermembrane space proteases on yeast aging. BMC Biology. 20 (1), 160 (2022).
- Torello Pianale, L., Rugbjerg, P., Olsson, L. Real-time monitoring of the yeast intracellular state during bioprocesses with a toolbox of biosensors. Frontiers in Microbiology. 12, 802169 (2022).
- Imani, M., Mohajeri, N., Rastegar, M., Zarghami, N. Recent advances in FRET-based biosensors for biomedical applications. Analytical Biochemistry. 630, 114323 (2021).
- Zadran, S., et al. Fluorescence resonance energy transfer (FRET)-based biosensors: visualizing cellular dynamics and bioenergetics. Applied Microbiology and Biotechnology. 96 (4), 895-902 (2012).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Liao, P. -. C., Wolken, D. M. A., Serrano, E., Srivastava, P., Pon, L. A. Mitochondria-associated degradation pathway (MAD) function beyond the outer membrane. Cell Reports. 32 (2), 107902 (2020).
- Higuchi-Sanabria, R., Swayne, T. C., Boldogh, I. R., Pon, L. A. Live-cell imaging of mitochondria and the actin cytoskeleton in budding yeast. Methods in Molecular Biology. 1365, 25-62 (2016).
- Liao, P. -. C., Yang, E. J., Pon, L. A. Live-cell imaging of mitochondrial redox state in yeast cells. STAR Protocols. 1 (3), 100160 (2020).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Collins, T. J. ImageJ for microscopy. BioTechniques. 43, 25-30 (2007).
- Chazotte, B. Labeling mitochondria with MitoTracker dyes. Cold Spring Harbor Protocols. 2011 (8), 990-992 (2011).
- Aguilaniu, H., Gustafsson, L., Rigoulet, M., Nyström, T. Asymmetric inheritance of oxidatively damaged proteins during cytokinesis. Science. 299 (5613), 1751-1753 (2003).
- Erjavec, N., Larsson, L., Grantham, J., Nyström, T. Accelerated aging and failure to segregate damaged proteins in Sir2 mutants can be suppressed by overproducing the protein aggregation-remodeling factor Hsp104p. Genes & Development. 21 (19), 2410-2421 (2007).
- Erjavec, N., Cvijovic, M., Klipp, E., Nyström, T. Selective benefits of damage partitioning in unicellular systems and its effects on aging. Proceedings of the National Academy of Sciences. 105 (48), 18764-18769 (2008).
- Erjavec, N., Nyström, T. Sir2p-dependent protein segregation gives rise to a superior reactive oxygen species management in the progeny of Saccharomyces cerevisiae. Proceedings of the National Academy of Sciences. 104 (26), 10877-10881 (2007).
- Davidson, R., Liu, Y., Gerien, K. S., Wu, J. Q. Real-time visualization and quantification of contractile ring proteins in single living cells. Methods in Molecular Biology. 1369, 9-23 (2016).
- Bilan, D. S., et al. HyPer-3: a genetically encoded H2O2 probe with improved performance for ratiometric and fluorescence lifetime imaging. ACS Chemical Biology. 8 (3), 535-542 (2013).
- Ermakova, Y. G., et al. Red fluorescent genetically encoded indicator for intracellular hydrogen peroxide. Nature Communications. 5 (1), 5222 (2014).
- Fricker, M. D. Quantitative redox imaging software. Antioxidants & Redox Signaling. 24 (13), 752-762 (2016).
- Yang, E. J., Pernice, W. M., Pon, L. A. A role for cell polarity in lifespan and mitochondrial quality control in the budding yeast Saccharomyces cerevisiae. iSCIENCE. 25 (3), 103957 (2022).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon