È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Modellazione realistica delle membrane utilizzando miscele lipidiche complesse negli studi di simulazione
In questo articolo
Riepilogo
La diversità dei lipidi di membrana nella struttura e nella composizione è un importante contributo ai processi cellulari e può essere un marcatore di malattia. Le simulazioni di dinamica molecolare ci permettono di studiare le membrane e le loro interazioni con le biomolecole a risoluzione atomistica. Qui forniamo un protocollo per costruire, eseguire e analizzare sistemi a membrana complessi.
Abstract
I lipidi sono elementi costitutivi strutturali delle membrane cellulari; Le specie lipidiche variano tra gli organelli cellulari e tra gli organismi. Questa varietà si traduce in diverse proprietà meccaniche e strutturali nella membrana che hanno un impatto diretto sulle molecole e sui processi che si verificano in questa interfaccia. La composizione lipidica è dinamica e può servire a modulare i processi di segnalazione cellulare. Gli approcci computazionali sono sempre più utilizzati per prevedere le interazioni tra biomolecole e fornire informazioni molecolari agli osservabili sperimentali. La dinamica molecolare (MD) è una tecnica basata sulla meccanica statistica che prevede il movimento degli atomi in base alle forze che agiscono su di essi. Le simulazioni MD possono essere utilizzate per caratterizzare l'interazione delle biomolecole. Qui, introduciamo brevemente la tecnica, delineiamo i passaggi pratici per i principianti che sono interessati a simulare i doppi strati lipidici, dimostriamo il protocollo con un software adatto ai principianti e discutiamo le alternative, le sfide e le considerazioni importanti del processo. In particolare, sottolineiamo l'importanza dell'utilizzo di miscele lipidiche complesse per modellare una membrana cellulare di interesse per catturare gli ambienti idrofobici e meccanici appropriati nella simulazione. Discutiamo anche alcuni esempi in cui la composizione e le proprietà della membrana modulano le interazioni dei doppi strati con altre biomolecole.
Introduzione
I lipidi sono i principali costituenti delle membrane, che forniscono confini per le cellule e consentono la compartimentazione intracellulare 1,2,3. I lipidi sono anfifilici, con un gruppo di teste polari e due code di acidi grassi idrofobici; Questi si auto-assemblano in un doppio strato per ridurre al minimo il contatto delle catene idrofobiche con l'acqua 3,4. Varie combinazioni di gruppi di teste idrofile e code idrofobiche danno luogo a diverse classi di lipidi nelle membrane biologiche, come glicerofosfosfolipidi, sfingolipidi e steroli (Figura 1)1,5,6. I glicerofosfolipidi sono elementi costitutivi primari delle membrane cellulari eucariotiche composte da glicerofosfato, acidi grassi a catena lunga e gruppi di testa a basso peso molecolare7. La nomenclatura dei lipidi si basa sulle differenze nei gruppi di teste; esempi includono fosfatidil-colina (PC), fosfatidil-etanolammina (PE), fosfatidil-serina (PS), fosfatidil-glicerolo (PG), fosfatidil-inositolo (PI) o acido fosfatidico non modificato (PA)5,6. Per quanto riguarda le code idrofobiche, la lunghezza e il grado di saturazione variano, insieme alla struttura della spina dorsale. Le combinazioni possibili sono numerose, dando vita a migliaia di specie lipidiche nelle cellule dei mammiferi6. I cambiamenti nella composizione lipidica della membrana portano a diverse proprietà meccaniche e strutturali della membrana che influiscono sull'attività sia delle proteine integrali di membrana che delle proteine periferiche 2,6.
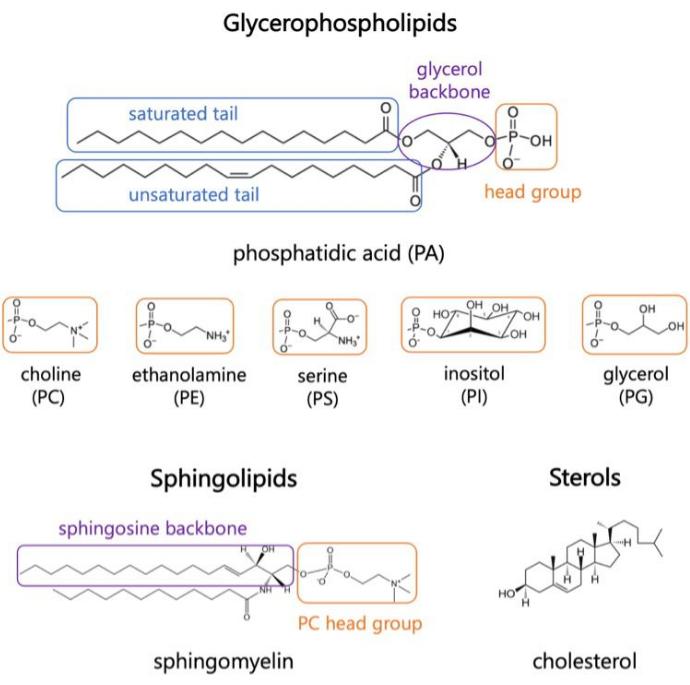
Figura 1. Strutture lipidiche rappresentative. Le code degli acidi grassi sono mostrate in riquadri blu, i gruppi di teste lipidiche comuni in arancione e le spine dorsali dei campioni in viola. Fare clic qui per visualizzare una versione più grande di questa figura.
I lipidi sono attori attivi nei processi cellulari, nell'attivazione delle proteine nelle cascate di segnalazione e nell'omeostasi delle cellule sane 8,9. Le dinamiche lipidiche alterate sono il risultato di un'infezione o possono essere marcatori di patogenesi della malattia 10,11,12,13,14,15. Come barriere per la cellula, lo studio dei lipidi di membrana e del loro ruolo nella permeazione di piccole molecole è di rilevanza per i sistemi di somministrazione dei farmaci e i meccanismi di rottura della membrana16,17. La diversità chimica e i diversi rapporti delle specie lipidiche tra organelli, tessuti e organismi danno origine a complesse dinamiche di membrana2. È quindi importante mantenere queste caratteristiche negli studi di modellizzazione dei doppi strati lipidici, soprattutto quando l'obiettivo di uno studio è quello di esaminare le interazioni di altre biomolecole con la membrana. Le specie lipidiche da considerare in un modello dipendono dall'organismo e dal compartimento cellulare di interesse. Ad esempio, i lipidi PG sono importanti per il trasferimento di elettroni nella bateria fotosintetica18, mentre i lipidi fosforilati dell'inositolo (PIP) sono i principali attori nella dinamica della membrana plasmatica (PM) e nelle cascate di segnalazione nelle cellule di mammifero 19,20. All'interno della cellula, il PM, il reticolo endoplasmatico (ER), il Golgi e le membrane mitocondriali contengono abbondanze lipidiche uniche che influenzano la loro funzione. Ad esempio, l'ER è il fulcro della biogenesi lipidica e trasporta il colesterolo verso il PM e il Golgi; contiene un'elevata diversità lipidica con abbondanza di PC e PE, ma un basso contenuto di steroli, che favorisce la fluidità della membrana21,22,23,24. Al contrario, il PM incorpora centinaia e persino migliaia di specie lipidiche a seconda dell'organismo25, contiene alti livelli di sfingolipidi e colesterolo che gli conferiscono una rigidità caratteristica rispetto ad altre membrane della cellula24. L'asimmetria dei lembi deve essere presa in considerazione per membrane come il PM, che ha un foglietto esterno ricco di sfingomielina, PC e colesterolo e un foglietto interno ricco di PE, PI e PS che sono importanti per le cascate di segnalazione24. Infine, la diversità lipidica induce anche la formazione di micro-domini che differiscono per impacchettamento e ordine interno, noti come zattere lipidiche24,26; Questi mostrano un'asimmetria laterale, si ipotizza che svolgano un ruolo importante nella segnalazione cellulare26 e sono difficili da studiare a causa della loro natura transitoria.
Tecniche sperimentali come la fluoroscopia, la spettroscopia e sistemi di membrane modello come le vescicole unilamellari giganti (GUV) sono state utilizzate per studiare le interazioni delle biomolecole con le membrane. Tuttavia, la natura complessa e dinamica dei componenti coinvolti è difficile da catturare con i soli metodi sperimentali. Ad esempio, ci sono limitazioni sull'imaging dei domini transmembrana delle proteine, sulla complessità delle membrane utilizzate in tali studi e sull'identificazione di stati intermedi o transitori durante il processo di interesse27,28,29. Dall'avvento della simulazione molecolare di monostrati e doppi strati lipidici negli anni '8029, i sistemi lipidico-proteici e le loro interazioni possono ora essere quantificati a livello molecolare. La simulazione della dinamica molecolare (MD) è una tecnica computazionale comune che prevede il movimento delle particelle in base alle loro forze intermolecolari. Un potenziale di interazione additiva descrive le interazioni legate e non legate tra le particelle del sistema30. L'insieme di parametri utilizzati per modellare queste interazioni è chiamato campo di forza di simulazione (FF). Questi parametri sono ottenuti da calcoli ab initio, calcoli semi-empirici e meccanici quantistici e ottimizzati per riprodurre dati provenienti da esperimenti di diffrazione di raggi X ed elettroni, NMR, infrarossi, Raman e spettroscopia neutronica, tra gli altri metodi31.
Le simulazioni MD possono essere utilizzate per studiare sistemi a vari livelli di risoluzione32,33,34. I sistemi che mirano a caratterizzare specifiche interazioni biomolecolari, legami idrogeno e altri dettagli ad alta risoluzione vengono studiati con simulazioni all-atom (AA). Al contrario, le simulazioni a grana grossa (CG) raggruppano gli atomi in gruppi funzionali più grandi per ridurre i costi computazionali ed esaminare le dinamiche su scala più ampia33. Tra questi due ci sono le simulazioni di atomi uniti (UA), in cui gli atomi di idrogeno sono combinati con i rispettivi atomi pesanti per accelerare il calcolo33,35. Le simulazioni MD sono un potente strumento per l'esplorazione della dinamica delle membrane lipidiche e delle loro interazioni con altre molecole e possono servire a fornire meccanismi a livello molecolare per i processi di interesse all'interfaccia di membrana. Inoltre, le simulazioni MD possono servire a restringere gli obiettivi sperimentali e prevedere le proprietà macromolecolari di un dato sistema sulla base di interazioni microscopiche.
In breve, dato un insieme di coordinate iniziali, velocità e un insieme di condizioni come temperatura e pressione costanti, le posizioni e le velocità di ciascuna particella vengono calcolate attraverso l'integrazione numerica del potenziale di interazione e della legge del moto di Newton. Questa operazione viene ripetuta in modo iterativo, generando così una traiettoria di simulazione30. Questi calcoli vengono eseguiti con un motore MD; tra i vari pacchetti open-source, GROMACS36 è uno dei motori più comunemente usati e quello che descriviamo qui. Include anche strumenti per l'analisi e la costruzione delle coordinate iniziali dei sistemi da simulare37. Altri motori MD includono NAMD38; CHARMM39 e AMBER40, che l'utente può selezionare a propria discrezione in base alle prestazioni computazionali di un determinato sistema. È fondamentale visualizzare le traiettorie durante la simulazione, nonché per l'analisi e l'interpretazione dei risultati. Sono disponibili una varietà di strumenti; qui discutiamo la dinamica molecolare visiva (VMD) che offre un'ampia gamma di funzionalità, tra cui la visualizzazione tridimensionale (3-D) con metodi di disegno e colorazione espansivi, la visualizzazione dei dati volumetrici, la costruzione, la preparazione e l'analisi delle traiettorie dei sistemi di simulazione MD e la creazione di filmati di traiettorie senza limiti sulle dimensioni del sistema, se la memoria è disponibile41,42,43.
L'accuratezza della dinamica prevista tra i componenti del sistema è direttamente influenzata dal FF scelto per la propagazione della traiettoria. Gli sforzi di parametrizzazione empirica di FF sono perseguiti da pochi gruppi di ricerca. I FF più consolidati e comuni per MD includono CHARMM39, AMBER 40, Martini44, OPLS 45 e SIRAH 46. Il campo di forza47 dell'additivo CHARMM36 di tutti gli atomi (C36) è ampiamente utilizzato per l'AA MD dei sistemi a membrana, in quanto riproduce accuratamente i dati strutturali sperimentali. È stato originariamente sviluppato dalla comunità CHARMM ed è compatibile con più motori MD come GROMACS e NAMD. Nonostante i miglioramenti tra i FF comuni, c'è uno sforzo continuo per migliorare i set di parametri per consentire previsioni che riproducano fedelmente le osservabili sperimentali, guidato dall'interesse per particolari sistemi di studio48,49.
Una sfida quando si simulano le membrane lipidiche è determinare la lunghezza della traiettoria di simulazione. Ciò dipende in gran parte dalle metriche da analizzare e dal processo che si intende caratterizzare. Tipicamente, le miscele lipidiche complesse richiedono più tempo per raggiungere l'equilibrio, poiché più specie devono avere abbastanza tempo per diffondersi sul piano della membrana e raggiungere un'organizzazione laterale stabile. Si dice che una simulazione è in equilibrio quando la proprietà di interesse ha raggiunto un plateau e fluttua intorno a un valore costante. E' pratica comune ottenere almeno 100-200 ns di traiettoria equilibrata per effettuare opportune analisi statistiche sulle proprietà e interazioni di interesse. È comune eseguire simulazioni di sola membrana tra 200-500 ns, a seconda della complessità della miscela lipidica e della domanda di ricerca. Le interazioni proteina-lipidi richiedono in genere tempi di simulazione più lunghi, tra 500 e 2000 ns. Alcuni approcci per accelerare il campionamento e le dinamiche osservabili con i sistemi a membrana sono: (i) il modello mimetico di membrana altamente mobile (HMMM), che sostituisce i carboni finali dei lipidi nella membrana con solvente organico per accelerare il campionamento50; e (ii) il ripartizionamento della massa dell'idrogeno (HMR), che combina una frazione delle masse degli atomi pesanti all'interno di un sistema con quelle degli atomi di idrogeno per consentire l'uso di una simulazione più ampia del timestep51.
Il seguente protocollo illustra un approccio adatto ai principianti per costruire, eseguire e analizzare modelli di membrana realistici utilizzando AA MD. Data la natura delle simulazioni MD, è necessario eseguire più traiettorie per tenere conto della riproducibilità e di una corretta analisi statistica dei risultati. La prassi corrente prevede l'esecuzione di almeno tre repliche per ogni sistema di interesse. Una volta che le specie lipidiche sono state selezionate per l'organismo e il processo di interesse, i passaggi di base per costruire, eseguire e analizzare una traiettoria di simulazione di un sistema di sola membrana sono delineati e riassunti nella Figura 2.
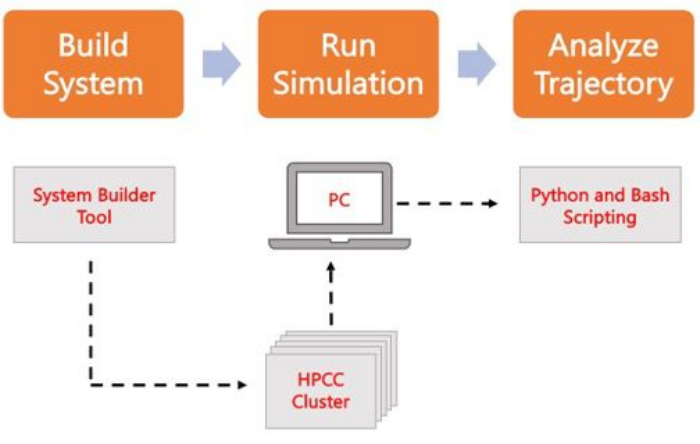
Figura 2. Schema per l'esecuzione di simulazioni MD. Le caselle arancioni corrispondono alle tre fasi principali descritte nel protocollo. Di seguito è riportato il flusso di lavoro del processo di simulazione. Durante la configurazione del sistema, il sistema contenente le coordinate iniziali di un sistema a membrana solvatato viene costruito con un generatore di input di sistema come CHARMM-GUI Membrane Builder. Dopo aver trasferito i file di input in un cluster di calcolo ad alte prestazioni, la traiettoria di simulazione viene propagata utilizzando un motore MD, ad esempio GROMACS. L'analisi della traiettoria può essere eseguita sul cluster di computer o su una stazione di lavoro locale insieme alla visualizzazione. L'analisi viene quindi eseguita, utilizzando pacchetti con codice di analisi incorporato come GROMACS e VMD, oppure utilizzando script Bash o varie librerie Python. Fare clic qui per visualizzare una versione più grande di questa figura.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Costruire le coordinate del sistema
- Passare a CHARMM-GUI.org (C-GUI) utilizzando un browser Web. Nel menu in alto, vai a Generatore di input, quindi seleziona Generatore di membrane dalle opzioni verticali sul lato sinistro dello schermo.
- Per creare un bilayer, selezionare Bilayer Builder.
NOTA: gli utenti per la prima volta devono attivare il proprio account gratuito prima di creare il primo set di coordinate. - Selezionare Sistema solo membrana. Salvare l'ID JOB generato per recuperare il sistema e continuare dal punto in cui si era interrotto durante il processo, se necessario.
- È possibile visualizzare i sistemi durante ogni fase del processo di costruzione facendo clic su Visualizza struttura nella casella situata nella parte superiore della pagina o scaricando il file PDB risultante. Fai attenzione ai componenti mancanti, agli errori nelle specie lipidiche di input selezionate o nelle dimensioni del cerotto.
- Selezionare i componenti del sistema.
- Scegliere l'opzione Lipidi eterogenei , anche se si costruisce un doppio strato monocomponente; quindi selezionare un tipo di Riquadro rettangolare .
- Seleziona 45 molecole d'acqua per lipide per l'opzione di idratazione; Questo è sufficiente per garantire un doppio strato completamente idratato.
- Impostare la lunghezza di XY in base al numero di componenti lipidici. Quindi, selezionare il numero di lipidi da includere per ciascuna specie lipidica determinata prima del modello. Per il caso di studio discusso nella sezione successiva, è stato costruito un modello di membrana con 600 lipidi distribuiti simmetricamente in due foglietti. Per modellare l'ER delle cellule eucariotiche è stata utilizzata una miscela di 336 lipidi DOPC, 132 DPPE, 60 CHOL, 72 POPI per il modello PI; e 330 lipidi DOPC, 126 DPPE, 54 CHOL, 66 POPI e 24 DOPS per il modello PI-PS.
NOTA: C-GUI fornisce una libreria di strutture lipidiche tra cui scegliere; Clicca sulle immagini accanto al nome della specie per la sua struttura chimica. - Digitare il numero desiderato di molecole nel foglietto superiore e inferiore nelle due caselle accanto al nome del lipide. Per il caso di studio, è auspicabile una composizione simmetrica della membrana: assicurarsi che non vi siano errori sul numero ineguagliato di lipidi nel foglietto superiore e nel foglio inferiore. Se si desidera un'asimmetria, assicurarsi che il numero totale di lipidi in ogni foglietto sia corretto. Per i dettagli sulla costruzione di doppi strati asimmetrici, fare riferimento al lavoro di Park et al.52,53.
- Vai all'inizio dell'elenco delle specie lipidiche e fai clic sul pulsante Mostra le informazioni di sistema. Assemblare i componenti e completare il sistema.
- Selezionare l'opzione per includere gli ioni neutralizzanti utilizzando l'algoritmo basato sulla distanza per una convergenza più rapida54.
- Lasciare la concentrazione predefinita della soluzione di KCl a 0,15 mM. Si tratta di una tipica concentrazione salina per rendere neutra la scatola di simulazione per i doppi strati di membrana.
NOTA: Se è necessario utilizzare una concentrazione diversa, assicurarsi di fare clic sul pulsante Calcola composizione solvente dopo averlo modificato.
- Selezionare le condizioni e le impostazioni di simulazione.
- Selezionare CHARMM36m come opzione FF; È comunemente usato per simulazioni di lipidi e proteine, ma l'utente può selezionare altre opzioni discusse nell'introduzione.
- Selezionare GROMACS come motore MD per ottenere file di input di esempio nel formato corrispondente.
NOTA: GROMACS è consigliato per i nuovi utenti perché dispone di più risorse online, tutorial e forum per il supporto. L'utente può scegliere tra più motori MD per esplorare le opzioni in termini di simulazione, prestazioni e sintassi del codice. - Selezionare l'insieme NPT (Constant Particle-Pressure-Temperature), di gran lunga l'insieme dinamico più utilizzato nella simulazione dei doppi strati lipidici.
- Impostare la temperatura e la pressione in Kelvin e bar rispettivamente a 303 K e 1 bar. È tipico impostare la temperatura tra 298 K e 310 K per lo studio dei processi biologici per garantire un doppio strato nello stato di disordine liquido.
NOTA: La temperatura dipende dalle condizioni del processo da simulare e può essere modificata secondo necessità. A seconda delle specie lipidiche nel modello, impostare la temperatura in modo che sia al di sopra della temperatura di transizione dei componenti lipidici puri prima di eseguire la simulazione.
- Scaricare i file risultanti e trasferirli nel cluster di computer.
- Visualizza il sistema finale su un software a tua scelta, come VMD o PyMol, e ispeziona la corretta configurazione.
NOTA: È bene controllare, ad esempio, che ci sia abbastanza acqua intorno alla membrana in modo che i lipidi non interagiscano con gli atomi dell'immagine durante la simulazione, e una corretta impostazione del foglietto (un doppio strato senza spazio o acqua in mezzo).
- Visualizza il sistema finale su un software a tua scelta, come VMD o PyMol, e ispeziona la corretta configurazione.
2. Esecuzione di simulazioni MD
- Caricare e decomprimere i file da C-GUI sul cluster di elaborazione. Passare alla directory Gromacs . Creare uno script di invio di rilassamento.
- Seguire le linee guida del cluster per il formato di uno script di invio.
- Copiare i comandi elencati fino a poco sopra il commento # Production nel file README nello script di invio.
NOTA: Questa impostazione predefinita di C-GUI è un loop che esegue un rilassamento del sistema in 6 fasi. Se si desidera un protocollo diverso e consolidato, modificarlo per leggere le coordinate appena compilate e scaricate dalla C-GUI.
- Copiare i comandi elencati fino a poco sopra il commento # Production nel file README nello script di invio.
- Inviare lo script di rilassamento e verificare che tutti i file di output siano stati scaricati per tutti i passaggi prima di passare all'esecuzione di produzione. Al termine, verificare la presenza dei seguenti file di output da GROMACS, generati durante l'esecuzione in 6 passaggi: *.log, *.tpr, *.gro, *.edr, *.trr / *.xtc
- Creare uno script di esecuzione dell'ambiente di produzione.
- Usare come modello uno degli esempi di comandi gmx grompp e gmx mdrun di uno qualsiasi dei passaggi di rilassamento.
- Prima di utilizzare lo script, assicurarsi di creare un file *.mdp che contenga opzioni di simulazione simili a quelle del file step7_production.mdp fornito.
NOTA: Le opzioni predefinite fornite sono standard per le simulazioni a membrana; Le variazioni sono elencate in nm e il tempo è espresso in picosecondi o numero di passi (picosecondi / passo temporale di integrazione). Aggiornare gli nsteps in modo che vengano eseguiti fino alla lunghezza di simulazione desiderata (che è uguale a dt * nsteps) e nst[x,v,f]out per aggiornare la frequenza di salvataggio dei dati in numero di passaggi di integrazione. Per il case study, impostare nsteps su 250.000.000 per una lunghezza di simulazione di 500 ns (tempo di simulazione/passo di integrazione = 500.000 ps / 0,002ps) e nst[x,v,f]out su 50.000 per salvare i dati ogni 100 ps
- Prima di eseguire la simulazione vera e propria, eseguire studi di benchmark per determinare l'uso ottimale delle risorse.
- Eseguire il sistema per 1-2 ns utilizzando un numero diverso di nodi di calcolo.
NOTA: Il caso di studio ER è stato presentato sul cluster di calcolo ad alte prestazioni55 del centro UB per la ricerca computazionale (CCR) per 2 ns, dove le prestazioni sono state testate per 1-10 nodi. - Confrontare le prestazioni in ns/giorno per ogni impostazione per determinare le risorse ottimali per l'esecuzione. È pratica comune selezionare il numero di nodi che determinano il 75%-80% delle prestazioni massime.
- Eseguire il sistema per 1-2 ns utilizzando un numero diverso di nodi di calcolo.
- Eseguire il ciclo di produzione.
- Eseguire ogni sistema in triplice copia per garantire la riproducibilità e condurre analisi statistiche sui dati.
- Estendere la traiettoria in base ai benchmark se il tempo di coda consentito per l'invio nel cluster di elaborazione si esaurisce. Usate i comandi gmx convert-tpr, quindi gmx mdrun per continuare la raccolta delle traiettorie.
NOTA: Le opzioni sono descritte nella documentazione GROMACS online (https://manual.gromacs.org/). - Per un sistema di sola membrana, esaminare se il sistema ha raggiunto l'equilibrio calcolando l'area per lipide nel tempo. In caso contrario, estendere la traiettoria di simulazione.
3. Analisi della traiettoria
- Visualizzare il sistema prima di eseguire l'analisi per determinare le molecole di interesse e la parte della traiettoria destinata alla caratterizzazione.
- Comprimere i file di traiettoria grezzi (*.trr) modificando il formato del file in *.xtc e/o saltando i fotogrammi per ridurre le dimensioni del file e facilitare un trasferimento più efficiente alla stazione locale per la visualizzazione e l'analisi.
NOTA: Per i sistemi a membrana di grandi dimensioni, si può scegliere di rimuovere l'acqua dalla traiettoria per ridurre ulteriormente le dimensioni del file. Questo può essere fatto con file di indice su GROMACS, script TCL su VMD o librerie Python come MDAnalysis e MDTraj. - Eseguire le analisi scelte durante la parte equilibrata della traiettoria, come determinato dalla serie temporale area per lipide.
NOTA: Fare riferimento alla discussione per maggiori dettagli sulle tipiche analisi a membrana e su come eseguirle.
Access restricted. Please log in or start a trial to view this content.
Risultati
Per illustrare l'uso del protocollo e i risultati che si possono ottenere, viene discusso uno studio comparativo per modelli di membrana per il reticolo endoplasmatico (ER). I due modelli in questo studio erano (i) il modello PI, che contiene le prime quattro specie lipidiche trovate nel reticolo endoplasmatico, e (ii) il modello PI-PS, che ha aggiunto le specie lipidiche anioniche fosfatidilserina (PS). Questi modelli sono stati successivamente utilizzati in uno studio su una proteina virale e su come interagisce con la...
Access restricted. Please log in or start a trial to view this content.
Discussione
Le tecniche sperimentali possono visualizzare le biomolecole ad alta risoluzione utilizzando la microscopia crioelettronica (cryo-EM)58, le tecniche di fluorescenza e la microscopia a forza atomica (AFM)59. Tuttavia, è difficile catturare l'interazione e la dinamica delle interazioni molecolari che sono alla base dei percorsi biologici, della patogenesi della malattia e della somministrazione terapeutica a livello atomico o amminoacidico. In questo articolo, sono state dis...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno interessi concorrenti da divulgare.
Riconoscimenti
Gli autori ringraziano Jinhui Li e Ricardo X. Ramirez per le loro traiettorie di simulazione e le discussioni durante la stesura di questo manoscritto. O.C. è stato sostenuto dalla University at Buffalo Presidential Fellowship e dal National Institute of Health's Initiative for Maximizing Student Development Training Grant 1T32GM144920-01 assegnato a Margarita L. Dubocovich (PI).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Anaconda3 | Anaconda Inc (Python & related libraries) | N/A | |
| CHARMM-GUI.org | Im lab, Lehigh University | N/A | |
| GROMACS | GROMACS development team | N/A | |
| Linux HPC Cluster | UB CCR | N/A | |
| MATLAB | MathWorks | N/A | |
| VMD | Theoretical and Computational Biophysics Group | N/A |
Riferimenti
- Vanni, S., Riccardi, L., Palermo, G., De Vivo, M. Structure and Dynamics of the Acyl Chains in the Membrane Trafficking and Enzymatic Processing of Lipids. Accounts of Chemical Research. 52 (11), 3087-3096 (2019).
- Harayama, T., Riezman, H. Understanding the diversity of membrane lipid composition. Nature Reviews Molecular Cell Biology. 19 (5), 281-296 (2018).
- Tanaka, M. Comprehensive Biophysics. Edward, H. . E. gelman , Elsevier. 261-272 (2012).
- Bruce Alberts, A. J., Julian Lewis,, Martin Raff,, Keith Roberts,, Peter Walter, Molecular Biology of the Cell. , Garland Science. (2002).
- Watson, H. Biological membranes. Essays in Biochemistry. 59, 43-69 (2015).
- Coskun, Ü, Simons, K. Cell Membranes: The Lipid Perspective. Structure. 19 (11), 1543-1548 (2011).
- Biobased Surfactants (Second Edition) eds. Douglas G, H. ayes, Daniel, K. Y., Solaiman,, Richard, D. , AOCS Press. 515-529 (2019).
- González-Rubio, P., Gautier, R., Etchebest, C., Fuchs, P. F. J. Amphipathic-Lipid-Packing-Sensor interactions with lipids assessed by atomistic molecular dynamics. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1808, 2119-2127 (2011).
- Halbleib, K., et al. Activation of the Unfolded Protein Response by Lipid Bilayer Stress. Molecular Cell. 67, 673-684 (2017).
- Andreasen, M., Lorenzen, N., Otzen, D. Interactions between misfolded protein oligomers and membranes: A central topic in neurodegenerative diseases. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1848 (9), 1897-1907 (2015).
- Calianese, D. C., Birge, R. B. Biology of phosphatidylserine (PS): basic physiology and implications in immunology, infectious disease, and cancer. Cell Commununication and Signaling. 18 (1), 41(2020).
- Nieto-Garai, J. A., Contreras, F. X., Arboleya, A., Lorizate, M. Role of Protein-Lipid Interactions in Viral Entry. Advanced Biology. 6, 2101264(2022).
- Mazzon, M., Mercer, J. Lipid interactions during virus entry and infection. Cell Microbiology. 16, 1493-1502 (2014).
- Colombelli, C., Aoun, M., Tiranti, V. Defective lipid metabolism in neurodegeneration with brain iron accumulation (NBIA) syndromes: not only a matter of iron. Journal of Inherited Metabolic Disease. 38 (1), 123-136 (2015).
- Saini-Chohan, H. K., Mitchell, R. W., Vaz, F. M., Zelinski, T., Hatch, G. M. Delineating the role of alterations in lipid metabolism to the pathogenesis of inherited skeletal and cardiac muscle disorders: Thematic Review Series: Genetics of Human Lipid Diseases. Journal of Lipid Research. 53 (1), 4-27 (2012).
- Martinotti, C., Ruiz-Perez, L., Deplazes, E., Mancera, R. L. Molecular Dynamics Simulation of Small Molecules Interacting with Biological Membranes. ChemPhysChem. 21 (14), 1486-1514 (2020).
- Li, J., Kalyanram, P., Rozati, S., Monje-Galvan, V., Gupta, A. Interaction of Cyanine-D112 with Binary Lipid Mixtures: Molecular Dynamics Simulation and Differential Scanning Calorimetry Study. ACS Omega. 7 (11), 9765-9774 (2022).
- Nagy, L., et al. Protein/Lipid Interaction in the Bacterial Photosynthetic Reaction Center: Phosphatidylcholine and Phosphatidylglycerol Modify the Free Energy Levels of the Quinones. Biochemistry. 43 (40), 12913-12923 (2004).
- Ramirez, R. X., Campbell, O., Pradhan, A. J., Atilla-Gokcumen, G. E., Monje-Galvan, V. Modeling the molecular fingerprint of protein-lipid interactions of MLKL on complex bilayers. Frontiers in Chemistry. 10, (2023).
- Dondelinger, Y., et al. MLKL Compromises Plasma Membrane Integrity by Binding to Phosphatidylinositol Phosphates. Cell Reports. 7 (4), 971-981 (2014).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews Molecular Cell Biology. 9 (2), 112-124 (2008).
- van Meer, G., de Kroon, A. I. P. M. Lipid map of the mammalian cell. Journal of Cell Science. 124 (1), 5(2011).
- Lee, H. R., Lee, G. Y., You, D. G., Kim, H. K., Young, D. Y. Hepatitis C virus p7 induces membrane permeabilization by interacting with phosphatidylserine. International Journal of Molecular Sciences. 21 (3), 897(2020).
- Casares, D., Escribá, P. V., Rosselló, C. A. Membrane Lipid Composition: Effect on Membrane and Organelle Structure, Function and Compartmentalization and Therapeutic Avenues. International Journal of Molecular Sciences. 20 (9), 2167(2019).
- Marrink, S. J., et al. Computational Modeling of Realistic Cell Membranes. Chemical Reviews. 119 (9), 6184-6226 (2019).
- Janmey, P. A., Kinnunen, P. K. J. Biophysical properties of lipids and dynamic membranes. Trends in Cell Biology. 16 (10), 538-546 (2006).
- Brémaud, E., Favard, C., Muriaux, D. Deciphering the Assembly of Enveloped Viruses Using Model Lipid Membranes. Membranes. 12, 441(2022).
- Campbell, O., Monje-Galvan, V. Protein-driven membrane remodeling: Molecular perspectives from Flaviviridae infections. Biophysical Journal. 122 (11), 1890-1899 (2022).
- Loschwitz, J., Olubiyi, O. O., Hub, J. S., Strodel, B., Poojari, C. S. Computer simulations of protein-membrane systems. Progress in molecular biology and translational science. 170, 273-403 (2020).
- Shell, M. S. Thermodynamics and Statistical Mechanics: An Integrated ApproachCambridge Series in Chemical Engineering. Scott Shell, M. , Cambridge University Press. 21-49 (2015).
- Yang, J., et al. Molecular Dynamic Simulation of Ni-Al Alloy-H2O Reactions Using the ReaxFF Reactive Force Field. ACS Omega. 8 (11), 9807-9814 (2023).
- Ingólfsson, H. I., Arnarez, C., Periole, X., Marrink, S. J. Computational 'microscopy' of cellular membranes. Journal of Cell Science. 129 (2), 257-268 (2016).
- Klauda, J. B. Perspective: Computational modeling of accurate cellular membranes with molecular resolution. The Journal of Chemical Physics. 149 (22), 220901(2018).
- Chavent, M., Duncan, A. L., Sansom, M. S. P. Molecular dynamics simulations of membrane proteins and their interactions: from nanoscale to mesoscale. Current Opinion in Structural Biology. 40, 8-16 (2016).
- Khakbaz, P., Monje-Galvan, V., Zhuang, X., Klauda, J. B. Biogenesis of Fatty Acids, Lipids and Membranes. Otto Geiger, , Springer International Publishing. 1-19 (2017).
- Abraham, M. J., et al. GROMACS: High performance molecular simulations through multi-level parallelism from laptops to supercomputers. SoftwareX. 1, 19-25 (2015).
- Lemkul, J. A. From Proteins to Perturbed Hamiltonians: A Suite of Tutorials for the GROMACS-2018 Molecular Simulation Package. Living Journal of Computational Molecular Science. 1 (1), 5068(2018).
- Phillips, J. C., et al. Scalable molecular dynamics on CPU and GPU architectures with NAMD. The Journal of Chemical Physics. 153 (4), 044130(2020).
- Klauda, J. B., et al. Update of the CHARMM All-Atom Additive Force Field for Lipids: Validation on Six Lipid Types. The Journal of Physical Chemistry B. 114 (23), 7830-7843 (2010).
- Wang, J., Wolf, R. M., Caldwell, J. W., Kollman, P. A., Case, D. A. Development and testing of a general amber force field. Journal of Computational Chemistry. 25 (9), 1157-1174 (2004).
- John Stone, A. A., et al. Using VMD. , http://csbmb.beckman.illinois.edu/BIOP586C/vmd-tutorial-2011.pdf (2011).
- Humphrey, W., Dalke, A., Schulten, K. VMD: Visual molecular dynamics. Journal of Molecular Graphics. 14 (1), 33-38 (1996).
- Hsin, J., Arkhipov, A., Yin, Y., Stone, J. E., Schulten, K. Using VMD: An Introductory Tutorial. Current Protocols in Bioinformatics. 24 (1), 5.7.1-5.7.48 (2008).
- Souza, P. C. T., et al. Martini 3: a general purpose force field for coarse-grained molecular dynamics. Nature Methods. 18 (4), 382-388 (2021).
- Jorgensen, W. L., Maxwell, D. S., Tirado-Rives, J. Development and Testing of the OPLS All-Atom Force Field on Conformational Energetics and Properties of Organic Liquids. Journal of the American Chemical Society. 118 (45), 11225-11236 (1996).
- Machado, M. R., et al. The SIRAH 2.0 Force Field: Altius, Fortius, Citius. Journal of Chemical Theory and Computation. 15 (4), 2719-2733 (2019).
- Huang, J., et al. CHARMM36m: an improved force field for folded and intrinsically disordered proteins. Nature Methods. 14 (1), 71-73 (2017).
- Mu, J., Liu, H., Zhang, J., Luo, R., Chen, H. F. Recent Force Field Strategies for Intrinsically Disordered Proteins. Journal of Chemical Information and Modeling. 61 (3), 1037-1047 (2021).
- Inakollu, V. S. S., Geerke, D. P., Rowley, C. N., Yu, H. Polarisable force fields: what do they add in biomolecular simulations. Current Opinion in Structural Biology. 61, 182-190 (2020).
- Ohkubo, Y. Z., et al. Accelerating Membrane Insertion of Peripheral Proteins with a Novel Membrane Mimetic Model. Biophysical Journal. 102 (9), 2130-2139 (2012).
- Hopkins, C. W., Le Grand, S., Walker, R. C., Roitberg, A. E. Long-Time-Step Molecular Dynamics through Hydrogen Mass Repartitioning. Journal of Chemical Theory and Computation. 11 (4), 1864-1874 (2015).
- Park, S., Beaven, A. H., Klauda, J. B., Im, W. How Tolerant are Membrane Simulations with Mismatch in Area per Lipid between Leaflets. Journal of Chemical Theory and Computation. 11 (7), 3466-3477 (2015).
- Park, S., Im, W., Pastor, R. W. Developing initial conditions for simulations of asymmetric membranes: a practical recommendation. Biophysical Journal. 120 (22), 5041-5059 (2021).
- Wu, E. L., et al. CHARMM-GUI Membrane Builder toward realistic biological membrane simulations. Journal of Computational Chemistry. 35 (27), 1997-2004 (2014).
- Center for Computational Research, U.a.B.. CCR Facility Description. , https://ubir.buffalo.edu/xmlui/handle/10477/79221 (2019).
- Piggot, T. J., Allison, J. R., Sessions, R. B., Essex, J. W. On the Calculation of Acyl Chain Order Parameters from Lipid Simulations. Journal of Chemical Theory and Computation. 13 (11), 5683-5696 (2017).
- Li, J., Monje-Galvan, V. Effect of Glycone Diversity on the Interaction of Triterpenoid Saponins and Lipid Bilayers. ACS Applied Bio Materials. , (2023).
- Renaud, J. P., et al. Cryo-EM in drug discovery: achievements, limitations and prospects. Nature Reviews Drug Discovery. 17 (7), 471-492 (2018).
- Ando, T., Uchihashi, T., Kodera, N. High-Speed AFM and Applications to Biomolecular Systems. Annual Review of Biophysics. 42 (1), 393-414 (2013).
- Martínez, L., Andrade, R., Birgin, E. G., Martínez, J. M. PACKMOL: A package for building initial configurations for molecular dynamics simulations. Journal of Computational Chemistry. 30 (13), 2157-2164 (2009).
- Jewett, A. I., et al. Moltemplate: A Tool for Coarse-Grained Modeling of Complex Biological Matter and Soft Condensed Matter Physics. Journal of Molecular Biology. 433 (11), 166841(2021).
- Jo, S., Kim, T., Iyer, V. G., Im, W. CHARMM-GUI: A web-based graphical user interface for CHARMM. Journal of Computational Chemistry. 29 (11), 1859-1865 (2008).
- Polêto, M. D., Lemkul, J. A. Integration of experimental data and use of automated fitting methods in developing protein force fields. Communications Chemistry. 5 (1), 38(2022).
- Hynninen, A. P., Crowley, M. F. New faster CHARMM molecular dynamics engine. Journal of Computational Chemistry. 35 (5), 406-413 (2014).
- Kim, S. Issues on the Choice of a Proper Time Step in Molecular Dynamics. Physics Procedia. 53, 60-62 (2014).
- Grubmüller, H., Heller, H., Windemuth, A., Schulten, K. Generalized Verlet Algorithm for Efficient Molecular Dynamics Simulations with Long-range Interactions. Molecular Simulation. 6 (1-3), 121-142 (1991).
- Darden, T., York, D., Pedersen, L. Particle mesh Ewald: An N·log(N) method for Ewald sums in large systems. Journal of Chemical Physics. 98 (12), 10089-10092 (1993).
- Hepatitis C. , https://www.who.int/news-room/fact-sheets/detail/hepatitis-c (2021).
- Braun, E., et al. Best Practices for Foundations in Molecular Simulations [Article v1.0]. Living Journal of Computational Molecular Science. 1 (1), 5957(2018).
- Moradi, S., Nowroozi, A., Shahlaei, M. Shedding light on the structural properties of lipid bilayers using molecular dynamics simulation: a review study. RSC Advances. 9 (8), 4644-4658 (2019).
- Monje-Galvan, V., Klauda, J. B. Modeling Yeast Organelle Membranes and How Lipid Diversity Influences Bilayer Properties. Biochemistry. 54 (45), 6852-6861 (2015).
- Michaud-Agrawal, N., Denning, E. J., Woolf, T. B., Beckstein, O. MDAnalysis: A toolkit for the analysis of molecular dynamics simulations. Journal of Computational Chemistry. 32 (10), 2319-2327 (2011).
- Gowers, R., et al. MDAnalysis: A Python Package for the Rapid Analysis of Molecular Dynamics Simulations. SciPy. , (2016).
- McGibbon, R. obert T., et al. MDTraj: A Modern Open Library for the Analysis of Molecular Dynamics Trajectories. Biophysical Journal. 109 (8), 1528-1532 (2015).
- Fortunato, M. E., Colina, C. M. pysimm: A python package for simulation of molecular systems. SoftwareX. 6, 7-12 (2017).
- Scherer, M. K., et al. PyEMMA 2: A Software Package for Estimation, Validation, and Analysis of Markov Models. Journal of Chemical Theory and Computation. 11 (11), 5525-5542 (2015).
- Song, W., et al. PyLipID: A Python Package for Analysis of Protein-Lipid Interactions from Molecular Dynamics Simulations. Journal of Chemical Theory and Computation. 18 (2), 1188-1201 (2022).
- Monje-Galvan, V., Klauda, J. B. Peripheral membrane proteins: Tying the knot between experiment and computation. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1858 (7, Part B), 1584-1593 (2016).
- Monje-Galvan, V., Voth, G. A. Binding mechanism of the matrix domain of HIV-1 gag on lipid membranes. eLife. 9, e58621(2020).
- Wang, B., Guo, C. Concentration-Dependent Effects of Cholesterol on the Dimerization of Amyloid-β Peptides in Lipid Bilayers. ACS Chemical Neuroscience. 13 (18), 2709-2718 (2022).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon