このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
細菌のための2つの成分規制システムを遺伝子標的を決定するために、DNA親和性精製チップ(DAP-チップ)方法
要約
このビデオの記事は、遺伝子標的と2成分系の応答調節因子の結合部位を決定するためのin vitroのマイクロアレイベースの方法を説明します。
要約
このようなChIPチップインビボ方法において 、転写因子のためのグローバルな遺伝子標的を決定するために使用される十分に確立された技術である。しかし、彼らは特徴づけられていない活性化条件で、細菌の2成分の規制制度を模索中使用が制限されている。このようなシステムは、ユニークな信号の存在下で活性化する場合にのみ転写を調節する。これらの信号はしばしば不明であるので、このビデオ資料に記載のインビトロマイクロアレイベースの方法は、遺伝子標的および応答調節因子の結合部位を決定するために使用することができる。このDNA-アフィニティー精製チップ法は、配列決定されたゲノムを有する任意の生物の任意の精製されたレギュレータに使用されてもよい。プロトコルは、精製されたタグが付いたタンパク質は、カスタムタイリングアレイへのDNAとハイブリダイゼーションの蛍光標識に続いて、タンパク質に結合したDNAを剪断ゲノムDNAに結合して、親和性浄化することができますが含まれます。仕するためのアッセイを最適化するために使用され得る工程の前にCレギュレータはまた、記載されている。アレイデータの分析によって生成されたピークは、次いで、実験的に検証される結合部位モチーフを予測するために使用される。モチーフの予測は、さらに密接に関連した種にオルソロガス応答調節因子の遺伝子標的を決定するために使用することができる。私たちは、遺伝子標的を決定し、サイトのモチーフを結合し、このような環境細菌デスルホビブリオ尋常 Hildenborough中sigma54依存応答レギュレータDVU3023機能を予測することにより、この方法の適用可能性を実証する。
概要
生存し繁殖する細菌の能力は、それらが知覚とその環境における摂動に応答することができるどの程度に決定的に依存し、これは順番に、それらのシグナル伝達系に依存する。信号システム細菌エンコードの数は、その「微生物のIQ」と呼ばれているとその環境の変動と複数の信号を感知する能力を微調整その応答1の両方を示している可能性があり。二成分シグナル伝達系(TCS)細菌によって使用される最も普及しているシグナル伝達系であり、それらは外部信号を感知し、エフェクターレスポンスレギュレーター(RR)2リン酸化を介して送信するヒスチジンキナーゼ(HK)からなる。 RRは、出力領域の様々なため、異なるエフェクターのモードを持つことができますが、最も一般的な応答は、ドメイン1のDNA 結合を介した転写調節です。信号が感知され、VASの対応する機能のTCSsのT大半は不明のままである。
このようなChIPチップ等のインビボ方法は、日常的に、転写のゲノム結合部位の決定のために使用されているが3因子活性化状態または信号が既知である場合、それらは、細菌の二成分系資源レコードのために使用することができる。多くの場合、TCSをアクティブに環境手がかりは、その遺伝子標的よりも決定することが困難です。ここに記載したインビトロマイクロアレイに基づくアッセイは、効果的かつ迅速に遺伝子標的を決定し、のTCSsの機能を予測するために使用することができる。このアッセイは、RRがリン酸化され、従って、アセチルリン酸4などの小分子供与体を用いてインビトロで活性化することができるという事実を利用する。
この方法では、DNA-親和性精製チップ( 図1)DAP-チップという、目的のRR遺伝子はE.でHisタグにクローニングする大腸菌 、そして続いて精製タグを付けたタンパク質を双方向に許可されているNDゲノムDNAをせん断した。タンパク質に結合したDNAは、その後、アフィニティー精製、濃縮し、入力DNAによって濃縮されて増幅され、蛍光標識されたが、一緒にプールし、カスタム目的の生物( 図1)に対して行われるタイリングアレイにハイブリダイズ。マイクロアレイ実験は、アーチファクトを受けやすく、したがって、追加のステップは、アッセイを最適化するのに用いられる。一つのそのような工程は、( 図2のワークフローを参照)を電気泳動移動度シフトアッセイ(EMSA)を用いて、研究中のRR毎にターゲットを決定しようとすることである。その後、ゲノムDNAおよびDAPステップに結合した後、タンパク質結合及び投入DNAは、このように最適な結合条件を確認し、正のターゲットは入力割合と比較して、タンパク質に結合した画分に濃縮されているかどうかを確認するために定量PCRによって調べられるRR( 図2)。アレイハイブリダイゼーションの後、データは、ゲノム遺伝子座タンパク質hを示す高強度信号のピークを見つけるために分析される広告が結合した。関数が得られた遺伝子標的に基づいて、RRのために予測することができる。標的ゲノム遺伝子座を、次いで、実験的にEMSAを( 図2)を用いて検証される結合部位モチーフを予測するために使用される。 RRのための機能的予測および遺伝子標的は、次いで密接に( 図2)と類似の結合モチーフをそれらのゲノムをスキャンすることによってオーソロガスRRをコードする種が関連するように拡張することができる。 DAPチップ方式は、以前に何もありませんでしたTCSのための豊富な情報を提供することができます。この方法はまた、タンパク質を精製することができ、DNA結合条件を決定することができる場合、任意の転写調節のために使用され、ゲノム配列を有する関心対象の任意の生物に利用可能することができる。
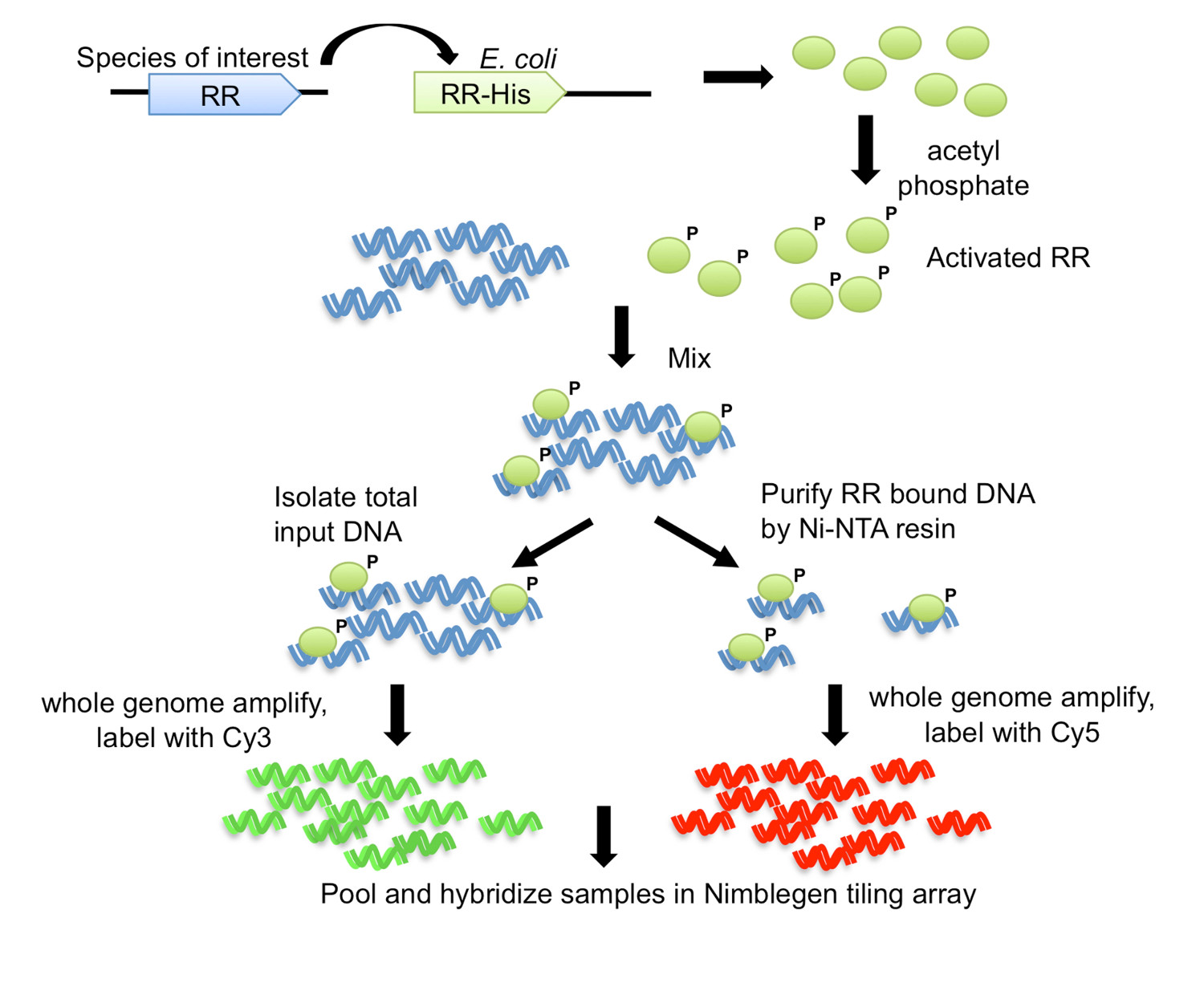
図1。のDNAアフィニティー精製したチップ(DAP-チップ)戦略7。目的の生物からのRR遺伝子を大腸菌にカルボキシ末端Hisタグでクローン化される大腸菌発現株。精製されたHisタグ付きタンパク質は、アセチルリン酸とリン酸化により活性化され、剪断されたゲノムDNAと混合する。残りは、Ni-NTA樹脂を用いてアフィニティー精製を行っている間の結合反応のアリコートを、入力DNAとして保存される。入力およびRR-結合したDNAは、増幅全ゲノムであり、それぞれCy3およびCy5で標識。標識されたDNAを一緒にプールし、次いで、遺伝子標的を決定するために分析されたタイリングアレイにハイブリダイズされる。図は、修正され、7からクリエイティブコモンズライセンスを使用して復刻。
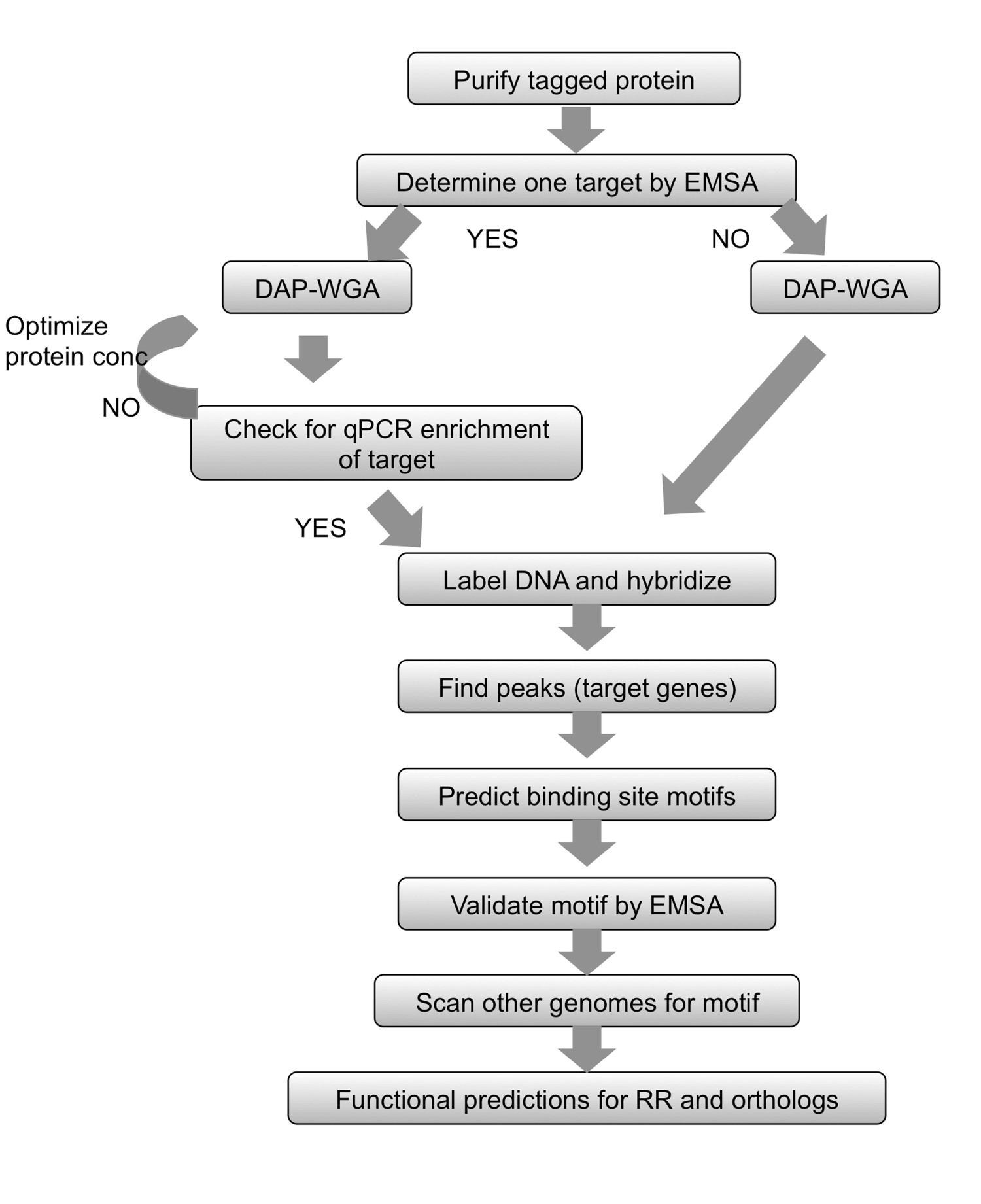
図2。ワークフローの概要。任意の精製されたタグが付いprotei用nは、EMSAを使用してターゲットを決定することから始めます。許可したゲノムDNAを結合して、DNA-アフィニティ精製するタンパク質(DAP)、全ゲノム(WGA)濃縮し、入力DNAを増幅する。遺伝子標的が既知である場合、公知の標的は、タンパク質結合画分中に濃縮されることを確実にするために定量PCRを使用する。標的が決定できなかった場合には、DNAの標識およびアレイハイブリダイゼーションに直接進む。定量PCRによる濃縮が観察されなかった場合には、結合タンパク質のgDNAと異なるタンパク質の量を使用してDAP-WGAステップを繰り返す。ピークを見つけ、遺伝子をターゲットにマップするために、配列解析を使用してください。結合部位モチーフを予測するために、標的遺伝子の上流領域を使用してください。 EMSAをを使用して実験的にモチーフを検証します。研究中のRRのオルソログをコードする近縁種のゲノムをスキャンし、同様にそれらの種で標的遺伝子を予測するモチーフを使用してください。得られた遺伝子標的に基づいて、RRおよびそのオーソログの生理学的機能を予測することができる。図は、創造的なCを使用して修正して復刻ommonsは7からライセンス。
プロトコル
注意:以下のプロトコルは、細菌デスルホビブリオ尋常 HildenboroughからのRR DVU3023の遺伝子標的の決定のために調整されています。これは、目的の任意の他の転写調節因子に適合させることができる。
1。クローン、精製RR
- Dから、RR遺伝子、特にDVU3023のクローンを作成する尋常 Hildenborough 大腸菌発現ベクターへの遺伝子がC末端Hisタグおよび発現がT7プロモーターの制御下にあるようにする。
注:いくつかのクローニング方法を用いてもよいし、研究者によって決定される。代替的な親和性タグを使用することもできる。 - Eに発現構築物を変換大腸菌 BL21(DE3)発現株。
- 37℃でBL21発現株の1リットル育つ対数期中期では、タンパク質発現を誘導するために0.5mmのIPTGを追加します。 24時間室温で成長を続けています。
- 10マイルのための5000×gで遠心分離細胞4℃でのN溶解緩衝液に再懸濁細胞(20mMのリン酸ナトリウム、pH7.4、500mMのNaCl、40 mMイミダゾール、1 mg / mlのリゾチーム、1×ヌクレアーゼベンゾナーゼ)。 4℃で、フレンチプレスを使用して細胞を溶解
- 4℃で30分間15,000 xgで遠心ライセート0.45μmのシリンジフィルターを使用してフィルタ。
- 洗浄緩衝液10ml(20mMのリン酸ナトリウム、pH7.4、500mMのNaCl、40 mMイミダゾール)を用いたFPLC機器上で1ミリリットルのNi Sepharoseカラムを洗浄する。
- カラムの溶解液をロードします。洗浄緩衝液20mlでカラムを洗浄。
- 0〜100%溶出緩衝液(20mMのリン酸ナトリウム、pH7.4、500mMのNaCl、500mMイミダゾール)の勾配を用いて溶出DVU3023。
- 脱塩カラム上で溶出した画分をロードし、脱塩緩衝液(20mMのリン酸ナトリウムpH7.4、100mMのNaCl)で洗浄した。
- タンパク質を濃縮する高い分子量カットオフ遠心フィルター中で遠心タンパク質サンプル。 -20℃で50%、店舗タンパク質に0.1 mMおよびグリセロールにDTTを追加
注:精製方法が検討されて各タンパク質について個別に最適化する必要がある。
2。電気泳動移動度シフトアッセイ(EMSA)を使用して、RRの遺伝子ターゲットを決定します
- PCRは、D.を使用して、候補標的遺伝子DVU3025の上流400bpの領域を増幅尋常 Hildenborough鋳型としてゲノムDNA及び未標識フォワードプライマーおよびリバース5'-ビオチン標識プライマー。
注:多くの場合、RRは、自分の遺伝子/オペロンの上流領域と結合する、またはそれらが近位にコードされる遺伝子を調節することができる。候補標的遺伝子を選択するためのヒント。 RRは、他の種のオルソログがある場合は、周辺に保存されている遺伝子を探します。別の方法は、それ自体がsigma54に依存しているのRRのためのレギュロンの予測( 例えば 、RegPrecise 5)、または予測sigma54依存性プロモーター6に基づいて選択する候補遺伝子が含まれています。 - 400 bは切り出し、1%アガロースゲル上でPCR産物を実行するP大型商品、およびゲル抽出キットをきれいに使用してDNAを精製。
- 10 mMトリス塩酸、pH 7.5、50mMのKCl、5mMのMgCl 2、1mMのDTT、25%グリセロールおよび1μg/ mlのポリdI.dC(非ビオチン化DNA基質100 fmolのとの混合DVU3023タンパク質(0.5ピコモル)固有の競合DNA)を20μlの総容量。また、コントロールとして、任意のタンパク質を含まない反応を設定します。 20分間のベンチで、室温で反応をインキュベートする。
- 注:これらは記載されている標準的な条件および他の反応成分は、目的のRRに応じて添加してもよい。活性化が望まれる場合、反応(多くの場合、RRは活性化せず、インビトロで DNAに結合するであろう)に50mMのアセチルリン酸を加える。
- 30分間、100Vで0.5×TBE緩衝液中プレキャストミニ6%ポリアクリルアミド0.5×TBEゲルを事前に実行してください。 gの上の結合反応や反応の負荷18μlに5倍のローディングバッファーを5μl(0.1%ブロモフェノールブルー、0.1%キシレンシア、1X TBE 30%グリセロール)を追加エル。 2時間、100Vで動作します。
注:ゲルの大部分がバッファによって絶縁されているように、ランニングバッファーで電気泳動システム、外側バッファ室を充填する、タンパク質-DNA複合体の解離をもたらすことができる過熱を回避するために。あるいは、ゲルを4℃で実行されてもよい - ゲルの大きさに充電ナイロン膜をカットし、少なくとも10分間0.5×TBEでそれを浸します。カセットからゲルを取り除く。ゲルオフ任意の尾根をカットし、0.5×TBEに浸した2厚手の濾紙の間にゲル及び膜を挟むと、セミドライブロッティング装置内に入れ、30分間20 Vで動作します。
- 商業紫外線架橋剤本機器内膜を配置し、3分までの時間を設定します。
注:膜は今、次のステップに進む前に、数日間室温で乾燥保存することができる。 - ストレプトアビジン - 西洋ワサビperoxidaを採用し、市販の化学発光検出キットを使用して、製造業者の説明書に従ってブロットを開発するためのそれ自体のコンジュゲート。
- 画像コンピュータを使用してブロットは、CCD搭載カメラにフックアップし、RRはテストされているDNAと結合することを示すRRの存在下で、DNA基質の移動度の変化を探します。
注:タンパク質-DNAシフト反応の特異性は見ずれを解消または減少させるべきである非標識競合DNAの過剰に含むことによって試験することができる。
3。ゲノムDNA-タンパク質が結合した後、ターゲット濃縮を確認
- ゲノムDNA-タンパク質結合反応
- 間に2秒のギャップを1秒9の低振幅パルスでマイクロチップを使用して1.5mlマイクロチューブに(濃度100〜200 ngの/μLで)100μlのゲノムDNAを超音波処理することによって、500塩基対の平均サイズにせん断ゲノムDNA、それぞれ。
注:チューブの側面へのDNA飛散した場合、すべての3パルスの後内容をスピンダウン。使用される正確な条件使用超音波処理装置機器によって異なります。剪断されたDNA断片は100〜1000 bpの範囲であり得るが、平均サイズが400〜600 bpの範囲内であるべきである。あまりにも多くのせん断が少なく完全な結合部位をもたらし、どちら多すぎたり少なすぎたり、せん断、下流の全ゲノム増幅工程の間に、ライブラリの準備に影響を与えることがあります。 - 10mMのトリス-HCl、pH7.5、1mMのDTT、50mMのKCl、5mMのMgCl 2、25%グリセロール、及び50mMのアセチルリン酸にRRタンパク質(DVU3023 0.5ピコモル)で剪断したゲノムDNAの2〜3μgのを混ぜる。 30分間25℃で反応をインキュベートする。転送インプットDNAとして1.5mlチューブやラベルに、この反応物10μl。
注:アセチルホスフェートは、in vitroリン酸化によりタンパク質を活性化するために添加される。リン酸化は、DNA結合を刺激するが、多くのRRも活性化することなく、7 in vitroで DNAを結合。反応に添加されるタンパク質の量は、タンパク質の活性プレップに依存するであろう。 EMSAを選ぶために用いることができるこの金額をマイズ。
- 間に2秒のギャップを1秒9の低振幅パルスでマイクロチップを使用して1.5mlマイクロチューブに(濃度100〜200 ngの/μLで)100μlのゲノムDNAを超音波処理することによって、500塩基対の平均サイズにせん断ゲノムDNA、それぞれ。
- 親和性は、タンパク質結合DNAを精製
- 0.6ミリリットルマイクロチューブへのNi-NTAアガロース樹脂の30を添加する。下部に樹脂を収集するために1分間100×gで遠心分離し、上澄みを除去します。 100洗浄緩衝液(10mMのトリス-HCl、pH7.5、5mMのMgCl 2、50mMのKCl、25%グリセロール)μlの、フリック管が混合する、2分間100×gで遠心分離を追加する。上清を取り除きます。
- 洗浄したNi-NTA樹脂への結合反応の残りの90μLを加え、30分間ロータリーシェーカーでインキュベートする。
- 2分間100×gで遠心し、上清(非結合DNA)を削除します。 、洗浄緩衝液100μl加え内容物を混合するチューブをフリックし、2分間100×gで遠心分離します。上清を取り除きます。さらに2回の洗浄ステップを繰り返します。
- 樹脂に溶出緩衝液35μlの(20mMリン酸ナトリウム緩衝液pH8、500mMのNaCl、500mMイミダゾール)を添加し、ボルテックスで混和する。室温で5分間ベンチ上でインキュベート。 2分間100×gで遠心分離する。タンパク質結合DNA画などの新しい1.5mlチューブやラベルに上清を移します。
- また、入力されたDNAに溶出バッファー35μlを加える。
- 入力およびPCR精製キットを用いてタンパク質結合DNA画分を精製する。
- 全ゲノムは、入力およびタンパク質結合DNA試料を増幅する。
- PCRチューブを分離するための入力と、タンパク質に結合したDNAの10を添加する。 10倍の断片化バッファーの1μL、ライブラリ調製緩衝液の2液を追加し、各サンプルライブラリ安定化溶液の1μL。ボルテックスでよく混ぜる。 2分間95℃でサーモサイクラー、氷上で寒さの熱。
注:DNAの開始量は、増幅バイアスを導入することを避けるために、少なくとも10 NGにする必要があります。 - 、ライブラリの準備の酵素1μl加え、ピペッティングにより混合し、16°C/20分でサーマルサイクラー中で24°C/20分、20分、75℃/ 5分のために37℃でインキュベート、および4℃で保持
- 各チューブに、水47.5μL、10倍の増幅マスターミックス7.5μL、およびポリメラーゼの5μlを加える。 94°C/15秒を20サイクル、65℃/ 5分続く95℃/ 3分、で十分と熱混ぜる。 4℃で反応を開催
- PCR精製キットを用いて増幅したDNAサンプルを精製し、分光光度法によりDNA濃度を測定する。
- PCRチューブを分離するための入力と、タンパク質に結合したDNAの10を添加する。 10倍の断片化バッファーの1μL、ライブラリ調製緩衝液の2液を追加し、各サンプルライブラリ安定化溶液の1μL。ボルテックスでよく混ぜる。 2分間95℃でサーモサイクラー、氷上で寒さの熱。
- 定量PCRを用いたタンパク質に結合したDNA中の目的の濃縮を確認します。
- デザイン定量PCRプライマー任意の自由に利用できるプライマー設計ソフトウェアを使用して、EMSA検証済み標的遺伝子(DVU3025)の上流領域の200塩基対を増幅する。
- 各プライマーセットで各DNAテンプレートの三重qPCR反応をセットアップします。各プライマーセットのためのマスターミックスを調製する。 1Xマスターミックスは、2X SYBRグリーン定量PCRミックスを10μL、18μLの合計に対する各プライマーおよび水0.5μMが含まれています。アリコートを96ウェルPCRプレートのウェルあたりマスターミックス18μlの。
- DilutE増幅され、精製された入力およびタンパク質結合DNAサンプル5 NGに/水でμlで、そしてDNAテンプレートとして使用しています。ウェルにDNAテンプレートの2を添加する。
- 200 XG / 1分で遠心分離器でプレートをスピンダウン、超明確な定量PCRシールフィルムでプレートをシール。リアルタイム定量PCR機にプレートを置きます。サイクル関連する定量PCRを使用したソフトウェアを次のように95℃/ 1分、95°C/10秒を40サイクル、59°C/15秒、70°C/35秒。
- また、関係のない遺伝子(ネガティブコントロール)の上流領域を増幅するプライマーを用いた三連qPCR反応を設定します。
- インプットDNAとは、タンパク質に結合したDNAのC Tの値を引いΔCTを計算します。 2ΔCTなどのタンパク質に結合したDNA中の標的遺伝子の倍の濃縮を計算します。
注意:ターゲット上流領域は、入力DNA対DNA、タンパク質結合に富んでいた場合、それは結合反応および親和性精製をSUCCであったことを示しているessful。 4に進みます。対象上流領域の濃縮が観察されなかった場合には、タンパク質の異なる量の結合反応(ステップ3.1)を繰り返します。
4。DNA標識およびアレイハイブリダイゼーション
- Cy5でCy3及びエンリッチDNAとラベル投入DNA
OTE:Cy3およびCy5色素は、光に敏感であり、ケアは最小限に露光を維持するために注意する必要があります。- 40μlの1μgのDNAを混ぜて9量体をCy3/Cy5-labeled、水で80μlに音量を調整します。
- 熱は、(サーモサイクラー中)暗所で10分間98℃で変性する。 2分間氷上に急速冷却。
- よく混ぜ、それぞれの反応に、5のdNTP、および8μLの水、および中(暗所で2時間、37℃でインキュベート- 2μLクレノウポリメラーゼ(50,000 U / mlの3'-5'-エキソ)を追加サーモサイクラー)。
- 0.5 M.への反応およびNaCl溶液を停止するために50 mMのEDTAを追加
- 1.5mlチューブCONTに転送サンプル0.9体積のイソプロパノールをaining、10分間、暗所でインキュベートし、10分間12,000 xgで遠心。ペレットをCy5標識DNAに対するCy3標識DNAや青、ピンクでなければなりません。
- 2分間12000×gで80%エタノール(500μL)と遠心でペレットを洗浄。暗闇の中で5〜10分間空気乾燥ペレット。
注意:ペレットは-20℃で保存することができる - 25μlの水にペレットを再懸濁。分光光度法でDNA濃度を測定します。
- プール一緒に6μgの暗闇の中で弱火で遠心機で1.5ミリリットルチューブ、真空乾燥中のCy3及びCy5標識DNA(それが透明であれば遠心機の蓋をカバー)のそれぞれ。
注:ペレットをハイブリダイゼーションのための準備ができるまで-20℃で保存してもよい。
- マイクロアレイハイブリダイゼーション
- C.を使用し、42℃に温度を設定する前に3-4時間にハイブリダイゼーションシステムを回す
- 1×溶液を11.8μlの2×Hybridiを含むようなハイブリダイゼーション溶液のマスターミックスを調製zationバッファー、4.7μlのハイブリダイゼーション成分A、および0.5μLアライメントオリゴ。
- 5μlの水にペレットを再懸濁します。サンプルに、このミックスを13μLを加える。 15秒間の渦は、5分間乾燥した水浴中で95℃でインキュベートする。ロードするための準備ができるまで、ハイブリダイゼーションシステムに42℃でサンプルを保管してください。
- 製造業者のプロトコルに従って、マイクロアレイスライドミキサ組立体を準備する。
- ハイブリダイゼーションシステム内のミキサー·スライドアセンブリを配置します。充填ポートへの負荷の試料16μLを、接着フィルム付きのポートをシールシステム内に混入オンにし、42℃で16〜20時間ハイブリダイズ
- 水中で10倍市販の緩衝液に希釈して1×洗浄緩衝液I(250ml)中、II(50ml)およびIII(50ml)に調製し、各々にDTTを1mMに入れる。暖かいバッファiが42℃
- 精力的にSHながら、暖かいバッファI.ピールミキサーオフを含む皿の内側解体ツールにミキサー·スライド、および場所をスライドさせ手で解体ツールをaking。
- 洗浄バッファーI 50mlで容器にスライドを置き、手で2分間激しく振る。
- 50ミリリットル洗浄緩衝液IIを第二の容器への移送スライド、手で1分間激しく振る。
- 転送50ミリリットル洗浄緩衝液IIIと第三の容器にスライドさせ、手で15秒間激しく振る。
- すぐにペーパータオルの上のスライドの縁をブロットし、スライドラックに配置します。スライドを乾燥させるために2分間200×gで遠心分離する。 、スライドケース内でホイルでそれをラップし、デシケーター中で保管してください。
- 配列をスキャンする
- 測定器の取扱説明書に従ってマイクロアレイスキャナー内でスライドを置きます。
- 532ナノメートル= Cy3および635 Nmは= Cy5のような波長を設定するためにスキャナソフトウェアを使用し、350と400の間の初期の光電子増倍向上。
- スライド上のアレイを配置するスライドをプレビューします。スキャンのアレイ領域を選択します。
- 配列をスキャンし、アレイの機能は主に黄色であるように、光電子増倍設定を調整します。ヒストグラムは、赤と緑の重畳曲線や、できるだけお互いに近いが表示されるはずです。曲線は飽和時の10 -5正規化されたカウントの上に終了する必要があります。
- 別々に両方の532と635 nmの画像を保存します。
- 配列データ解析
- アレイ解析ソフトに画像をインポートします。ソフトウェアを使用して、(Cy3およびCy5イメージのため)、各アレイ用の2組のレポート(。ペア)を作成します。ペアファイルを使用してスケーリングログ2比ファイルを作成します。 500塩基対のスライディングウィンドウを使用してピークを検索するために、ログ2比ファイルを使用します。遺伝子標的の上流領域にピーク遺伝子座をマップします。
- 陽性標的(EMSAを用いて決定し、この例では、定量PCR、DVU3025によって確認)がトップピーク内に現れるかどうかを確認します。
注意:正のターゲットはトップヒットの中でされていない場合は、それが可能であることconsi下RRディレーティングは、いくつかの遺伝子標的を有し、DAP-チップを複製行うことができ、すべての複製に共通するピークがターゲットリストを生成するために使用することができる。
5。結合部位モチーフ予測と検証
- DAP-チップ分析によって生成されたトップの遺伝子標的のための上流領域(400 bp)のために配列を取得します。モチーフを予測するオンライン微生物(meme.nbcr.net)を介してこれらの配列にMEMEを適用します。
注:このようなsigma54依存調節因子としてエンハンサー結合タンパク質が開始部位の上流に数百塩基対に結合することができる。他の転写調節因子のために、このような上流200塩基対より短い領域が十分であろう。 - デザイントップと両端に10拠点が隣接して予測結合部位モチーフが含まれている下の鎖のDNAオリゴマー。ビオチン化上の鎖の5 '注文。
- 10 mMトリス塩酸、pH 8.0,1 mMのEDTA、および50mM NaCl中に1:1.5の比率で上部と下部の鎖オリゴを混合PCRチューブ中の20μlの総反応容量。 25℃までゆっくりと冷却に続いてサーマルサイクラー中で5分間95℃に加熱する、 10倍に希釈し、EMSAのために、野生型のdsDNA基質として使用しています。
- 上記と同じ手順を使用して修飾された基質を準備したが、設計上のオリゴマーは、結合モチーフの保存された塩基中4-6置換を実施する。
- RR及び野生型または修飾基質との結合反応を設定し、ステップ2で説明したようにEMSAを使用して調べます。
- 注意:RRは、野生型の基板ではなく、変更された1を結合する場合に予測モチーフが検証されます。
その他関連細菌種のモチーフの6。保全
- DVU3023のオルソログが含まれている関心のゲノムを選択します。 NCBIウェブサイトからのゲノム配列および注釈ファイルを入手します。
- POSを構築するために、DAPチップターゲットからのモチーフ配列を使用するスクリプトを書くことが、Perlなどのプログラミング言語を使用してくださいition加重マトリックスや他の配列決定されたゲノムに存在する類似のモチーフを獲得するために行列を使用しています。
結果
上記の方法は、 デスルホビブリオ尋常 Hildenborough 7細菌を減らすモデル硫酸でRRの全体的な遺伝子ターゲットを特定するために適用した。この生物は、それが感知して応答することができる信号の多種多様を示し、70以上のRRで表されるのTCSsが多数ある。 インビボでは 、これらの信号システムの機能について解析し、それらの信号ので、実行するのは難しいので、それら...
ディスカッション
ここで説明DAPチップ方式が正常つが代表的結果として示されているのビブリオ尋常 Hildenborough 7におけるいくつかのRRのための遺伝子標的を決定するために使用された。 RR DVU3023、候補遺伝子ターゲットを選択することは簡単でした。 DVU3025は、RR遺伝子のすぐ下流に位置し、RRおよび標的遺伝子は、いくつかのビブリオ種において保存され、さらに、予測DVU3025 sigma54依存性...
開示事項
著者らは、開示することは、特別の利害関係はありません。
謝辞
私たちは、ビデオ撮影の準備で彼女の助けと技術を実証するためのエイミー·チェンに感謝します。エニグマが行ったこの作品:遺伝子および分子集合体(http://enigma.lbl.gov)と生態系とネットワークの統合、ローレンスバークレー国立研究所の科学重点領域プログラム、科学局によってサポートされていました、生物のOfficeと契約番号DE-AC02-05CH11231下、米国エネルギー省の環境研究、。
資料
| Name | Company | Catalog Number | Comments |
| Name of Material/Equipment | Company | Catalog number | Comments |
| HisTrapFF column (Ni-Sepharose column) | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5255-01 | |
| Akta explorer (FPLC instrument) | GE Lfe Sciences, Pittsburgh, PA, USA | ||
| HiPrep 26/10 Desalting column | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5087-01 | |
| Qiaquick Gel extraction kit | Qiagen Inc, Valencia, CA, USA | 28704 | |
| Biotin-labeled oligonucleotides | Integrated DNA Technologies | N/A | |
| 6% polyacrylamide-0.5X TBE precast mini DNA retardation gel | Life Technologies, Grand Island, NY, USA | EC63652BOX | Alternately, you can pour your own gel. |
| Nylon membrane | EMD Millipore, Billerica, MA, USA | INYC00010 | |
| Trans-Blot SD Semi-dry electrophoretic transfer cell | Biorad, Hercules, CA, USA | 170-3940 | |
| Extra thick blot paper, 8 x 13.5 cm | Biorad, Hercules, CA, USA | 170-3967 | |
| UV crosslinker Model XL-1000 | Fisher Scientific | 11-992-89 | |
| Nucleic Acid chemiluminescent detection kit (Pierce) | Thermo fisher Scientific, Rockford, IL, USA | 89880 | |
| Ni-NTA agarose resin | Qiagen Inc, Valencia, CA, USA | 30210 | |
| GenomePlex Whole genome amplification kit (Fragmentation buffer, library preparation buffer, library stabilization solution, library preparation enzyme, 10X amplification master mix, WGA polymerase ) | Sigma-Aldrich, St. Louis, MO, USA | WGA2-50RXN | |
| Nanodrop ND-1000 | Thermo Scientific, Wilmington, DE, USA | For quantitation of DNA | |
| Perfecta Sybr Green SuperMix, with ROX | Quanta biosciences | 95055-500 | Any Sybr Green PCR mix may be used |
| PlateMax Ultra clear heat sealing film for qPCR | Axygen | ||
| 96 well clear low profile PCR microplate | Life Technologies, Grand Island, NY, USA | PCR-96-LP-AB-C | |
| Applied Biosystems StepOne Plus Real time PCR system | Life Technologies, Grand Island, NY, USA | 4376600 | Any real time PCR system may be used |
| Qiaquick PCR purification kit | Qiagen Inc, Valencia, CA, USA | 28104 | Any PCR clean up kit may be used |
| Cy3/Cy5-labeled nonamers | Trilink biotechnologies, San Diego, CA, USA | N46-0001, N46-0002 | |
| Klenow polymerase 50,000U/ml, 3'-5' exo- | New England Biolabs, Ipswich, MA | M0212M | |
| Hybridization system | Roche-Nimblegen, Madison, WI, USA | N/A | This company no longer makes arrays or related items, so alternate sources such as Agilent or Affymetrix will need to be used, |
| Custom printed microarrays and mixers | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Hybridization kit (2X Hybridization buffer, Hybridization component A, Alignment oligo) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Wash buffer kit (10X Wash buffer I, II, III, 1 M DTT) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| GenePix 4200A microarray scanner | Molecular Devices, Sunnyvale CA, USA | This model has been replaced by superior ones | |
| GenePix Pro microarray software | Molecular Devices, Sunnyvale CA, USA | ||
| Nimblescan v.2.4, ChIP-chip analysis software | Roche-Nimblegen, Madison, WI, USA | N/A | |
参考文献
- Galperin, M. Y. Diversity of structure and function of response regulator output domains. Curr Opin Microbiol. 13, 150-159 (2010).
- Casino, P., Rubio, V., Marina, A. The mechanism of signal transduction by two-component systems. Curr Opin Struct Biol. 20, 763-771 (2010).
- Aparicio, O., Geisberg, J. V., Struhl, K. Chromatin immunoprecipitation for determining the association of proteins with specific genomic sequences in vivo. Curr Protoc Cell Biol. 17 (17), (2004).
- McCleary, W. R., Stock, J. B. Acetyl phosphate and the activation of two-component response regulators. J Biol Chem. 269, 31567-31572 (1994).
- Novichkov, P. S., et al. RegPrecise web services interface: programmatic access to the transcriptional regulatory interactions in bacteria reconstructed by comparative genomics. Nucleic Acids Res. 40, 604-608 (2012).
- Studholme, D. J., Buck, M., Nixon, T. Identification of potential sigma(N)-dependent promoters in bacterial genomes. Microbiology. 146 (12), 3021-3023 (2000).
- Rajeev, L., et al. Systematic mapping of two component response regulators to gene targets in a model sulfate reducing bacterium. Genome Biol. 12, 99 (2011).
- Crooks, G. E., Hon, G., Chandonia, J. M., Brenner, S. E. WebLogo: a sequence logo generator. Genome Res. 14, 1188-1190 (2004).
- Barbieri, C. M., Mack, T. R., Robinson, V. L., Miller, M. T., Stock, A. M. Regulation of response regulator autophosphorylation through interdomain contacts. J Biol Chem. 285, 32325-32335 (2010).
- Bourret, R. B. Receiver domain structure and function in response regulator proteins. Curr Opin Microbiol. 13, 142-149 (2010).
- Gao, R., Stock, A. M. Molecular strategies for phosphorylation-mediated regulation of response regulator activity. Curr Opin Microbiol. 13, 160-167 (2010).
- Hung, D. C., et al. Oligomerization of the response regulator ComE from Streptococcus mutans is affected by phosphorylation. J Bacteriol. 194, 1127-1135 (2012).
- Ladds, J. C., et al. The response regulator Spo0A from Bacillus subtilis is efficiently phosphorylated in Escherichia coli. FEMS Microbiol Lett. 223, 153-157 (2003).
- Wen, Y., Feng, J., Scott, D. R., Marcus, E. A., Sachs, G. Involvement of the HP0165-HP0166 two-component system in expression of some acidic-pH-upregulated genes of Helicobacter pylori. J Bacteriol. 188, 1750-1761 (2006).
- Liu, X., Noll, D. M., Lieb, J. D., Clarke, N. D. DIP-chip: rapid and accurate determination of DNA-binding specificity. Genome Res. 15, 421-427 (2005).
- Gossett, A. J., Lieb, J. D. DNA Immunoprecipitation (DIP) for the Determination of DNA-Binding Specificity. CSH Protoc. 2008, (2008).
- Mascher, T., et al. The Streptococcus pneumoniae cia regulon: CiaR target sites and transcription profile analysis. J Bacteriol. 185, 60-70 (2003).
- Park, P. J. ChIP-seq: advantages and challenges of a maturing technology. Nat Rev Genet. 10, 669-680 (2009).
- Lefrancois, P., Zheng, W., Snyder, M. ChIP-Seq using high-throughput DNA sequencing for genome-wide identification of transcription factor binding sites. Methods Enzymol. 470, 77-104 (2010).
- Ho, J. W., et al. ChIP-chip versus ChIP-seq: lessons for experimental design and data analysis. BMC Genomics. 12, 134 (2011).
- Liu, X., Lee, C. K., Granek, J. A., Clarke, N. D., Lieb, J. D. Whole-genome comparison of Leu3 binding in vitro and in vivo reveals the importance of nucleosome occupancy in target site selection. Genome Res. 16, 1517-1528 (2006).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved