Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
DNA-Affinitäts-gereinigten Chip (DAP-Chip)-Methode, um Gene für bakterielle Ziele fest, Zwei-Komponenten-Systeme Regulatory
In diesem Artikel
Zusammenfassung
Dieses Video Artikel beschreibt ein in vitro-Mikroarray-basierte Methode, um die Gen-Targets und Bindungsstellen für Zweikomponentensystem Antwortregulatoren bestimmen.
Zusammenfassung
In-vivo-Methoden wie Chip-Chip sind etablierte Techniken zur globalen Zielgene für Transkriptionsfaktoren zu bestimmen. Von begrenztem Nutzen sind sie jedoch in der Erforschung bakteriellen Zwei-Komponenten-Regulationssysteme mit charakterisierter Aktivierungsbedingungen. Solche Systeme regulieren die Transkription nur, wenn in Gegenwart von eindeutigen Signalen aktiviert. Da diese Signale oft unbekannt sind, können in diesem Video-Artikel beschriebene in vitro-Mikroarray-basierten Verfahren verwendet werden, um Gen-Targets und Bindungsstellen für Antwortregulatoren zu bestimmen. Diese DNA-Affinitäts-gereinigten-Chip-Verfahren kann für jede gereinigte Regler in jedem Organismus mit einem sequenzierten Genom verwendet werden. Das Protokoll beinhaltet wodurch die gereinigte markierte Protein zu scher genomische DNA binden und Affinitätsreinigung des proteingebundenen DNA, gefolgt von der fluoreszierenden Markierung der DNA und Hybridisierung an eine individuelle Unterteilung Array. Vorhergehenden Schritte, die verwendet werden, um den Test zu optimieren Spezifikationen werden könnenc Regler werden ebenfalls beschrieben. Die von der Array-Daten-Analyse erzeugten Peaks werden verwendet, um Bindungsstelle Motive, die dann experimentell validiert werden vorherzusagen. Die Vorhersagen Motiv kann weiter verwendet werden, um Gen-Targets orthologer Antwortregulatoren in nahe verwandten Arten zu bestimmen. Wir zeigen die Anwendbarkeit dieser Methode durch Bestimmung der Gen-Targets und-Bindungsstelle Motive und damit die Vorhersage der Funktion für ein sigma54 abhängige Antwortregulator DVU3023 in der Umwelt Bakterium Desulfovibrio vulgaris Hildenborough.
Einleitung
Die Fähigkeit der Bakterien zu überleben und zu gedeihen ist kritisch davon abhängig, wie gut sie in der Lage zu erkennen und zu reagieren, um Störungen in ihrer Umgebung sind, und diese wiederum ist abhängig von der Signalübertragungssysteme. Die Anzahl der Signalanlagen ein Bakterium kodiert wurde seine "mikrobielle IQ" bezeichnet und kann ein Hinweis sowohl Variabilität der seine Umwelt und seine Fähigkeit, mehrere Signale zu erfassen und eine Feinabstimmung seiner Antwort 1 sein. Zwei-Komponenten-Signalübertragungssystemen (ASR) sind die am weitesten verbreiteten Signalsysteme von Bakterien verwendet werden, und sie bestehen aus einem Histidin-Kinase (HK), der das externe Signal erfasst und überträgt über die Phosphorylierung an ein Effektor-Antwortregulator (RR) 2 bestehen. RR kann eine Vielzahl von Ausgangsdomänen und damit unterschiedliche Effektor-Modi, aber die häufigste Antwort Transkriptionsregulation über eine DNA-bindende Domäne 1. Die erfassten Signale und die entsprechenden Funktionen des Vast Mehrheit der FKS vor unbekannt.
Obwohl in vivo-Methoden wie Chip-Chip werden routinemäßig für die Bestimmung von genomischen Bindungsstellen von Transkriptionsfaktoren 3 verwendet wird, kann sie nur für bakterielle Zweikomponentensystem-Ressourceneinträge verwendet, wenn die Aktivierungsbedingungen oder Signale bekannt sind. Oft sind die Umweltreize, die einen TCS aktivieren sind härter als ihre Gen-Targets zu bestimmen. Die in vitro-Mikroarray-basierten hier beschriebene Assay kann verwendet werden, um die Gen-Targets wirksam und schnell zu bestimmen und vorherzusagen Funktionen FKS werden. Dieser Assay nutzt die Tatsache, dass RRS phosphoryliert und somit mit kleinen Molekülen wie Acetyl Geber Phosphat 4 in vitro aktiviert.
Bei diesem Verfahren, genannt DAP-DNA-Chip für affinitätsgereinigtem-Chip (1), das RR-Gen von Interesse mit einem His-Tag kloniert, in E. coli, und eine anschließend gereinigt markierte Protein wird bi erlaubtnd genomische DNA geschert. Die an Protein gebundene DNA wird dann durch Affinitätschromatographie-Aufreinigung angereichert werden die angereicherten und Input-DNA amplifiziert, fluoreszenzmarkierten, gepoolt und auf ein Array, das individuelle Fliesen auf den Organismus von Interesse (1) hergestellt wird, hybridisiert. Mikroarray-Experimenten unterliegen Artefakte und daher zusätzliche Schritte verwendet werden, um den Assay zu optimieren. Ein solcher Schritt ist, zu versuchen, ein Ziel zu bestimmen, unter der RR-Studie unter Verwendung von elektrophoretischen Mobilitätsverschiebungstests (EMSA) (siehe Arbeitsablauf in Fig. 2). Dann, nach der Bindung an genomische DNA und die DAP Schritte, die proteingebundene und die Input-DNA durch qPCR untersucht, ob die positive Ziel in der proteingebundenen Fraktion gegenüber dem Eingangsfraktion angereichert, wodurch bestätigt optimaler Bindungsbedingungen für die RR (Fig. 2). Anordnung nach Hybridisierung werden die Daten analysiert, um Peaks höherer Intensität anzeigendes Signal genomischen Loci zu finden, wo das Protein had gebunden. Funktionen für die RR auf der Grundlage der vorhergesagten Gen-Targets erhalten werden. Die Ziel-Loci verwendet werden, um Bindungsstelle Motive, die dann experimentell mit EMSA (Abbildung 2) bestätigt werden vorherzusagen. Die Funktionsvorhersagen und Gen-Targets für die RR kann dann verlängert werden, um eng verwandte Arten, die orthologen RRs kodieren durch das Scannen dieser Genome für ähnliche Bindungsmotive (Abbildung 2). Der DAP-Chip-Verfahren kann eine Fülle von Informationen für ein TCS wo vorher da war keiner zu liefern. Das Verfahren kann auch für jede Transkriptionsregulator verwendet werden, wenn das Protein gereinigt und DNA-Bindungsbedingungen bestimmt werden können, und für jeden Organismus von Interesse mit einer Genomsequenz vorhanden.
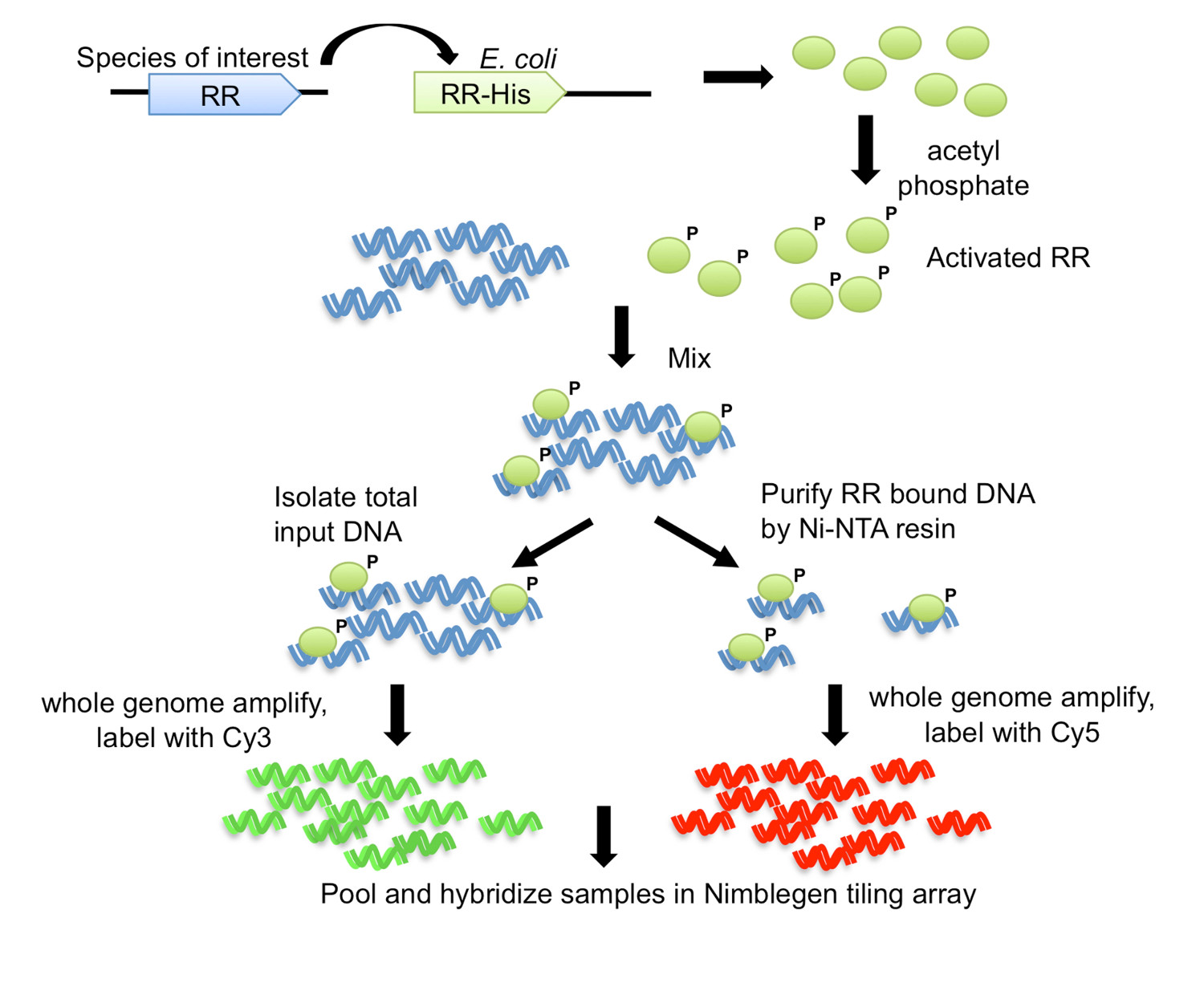
Abbildung 1. Die DNA-Affinitäts-gereinigten-Chip-(DAP-Chip)-Strategie 7. Die RR-Gens aus dem Organismus von Interesse ist mit einem Carboxy-terminalen His-Tag in einen E. kloniert coli Expressionsstamm. Gereinigte His-markierte Protein durch Phosphorylierung mit Acetylphosphat aktiviert und mit geschnittenen genomischen DNA gemischt. Ein Aliquot der Bindungsreaktion als Eingangs DNA gespeichert, während der Rest der Affinitätsreinigung unter Verwendung von Ni-NTA-Harz unterzogen. Der Eingang und der RR-gebundene DNA sind ganze Genom amplifiziert und mit Cy3 bzw. Cy5 markiert. Die markierte DNA wird zusammen gebündelt und zu einem Kachelanordnung, die dann analysiert wird, um die Gen-Targets zu bestimmen, hybridisiert. Abbildung modifiziert und neu aufgelegt mit der Creative-Commons-Lizenz von 7.
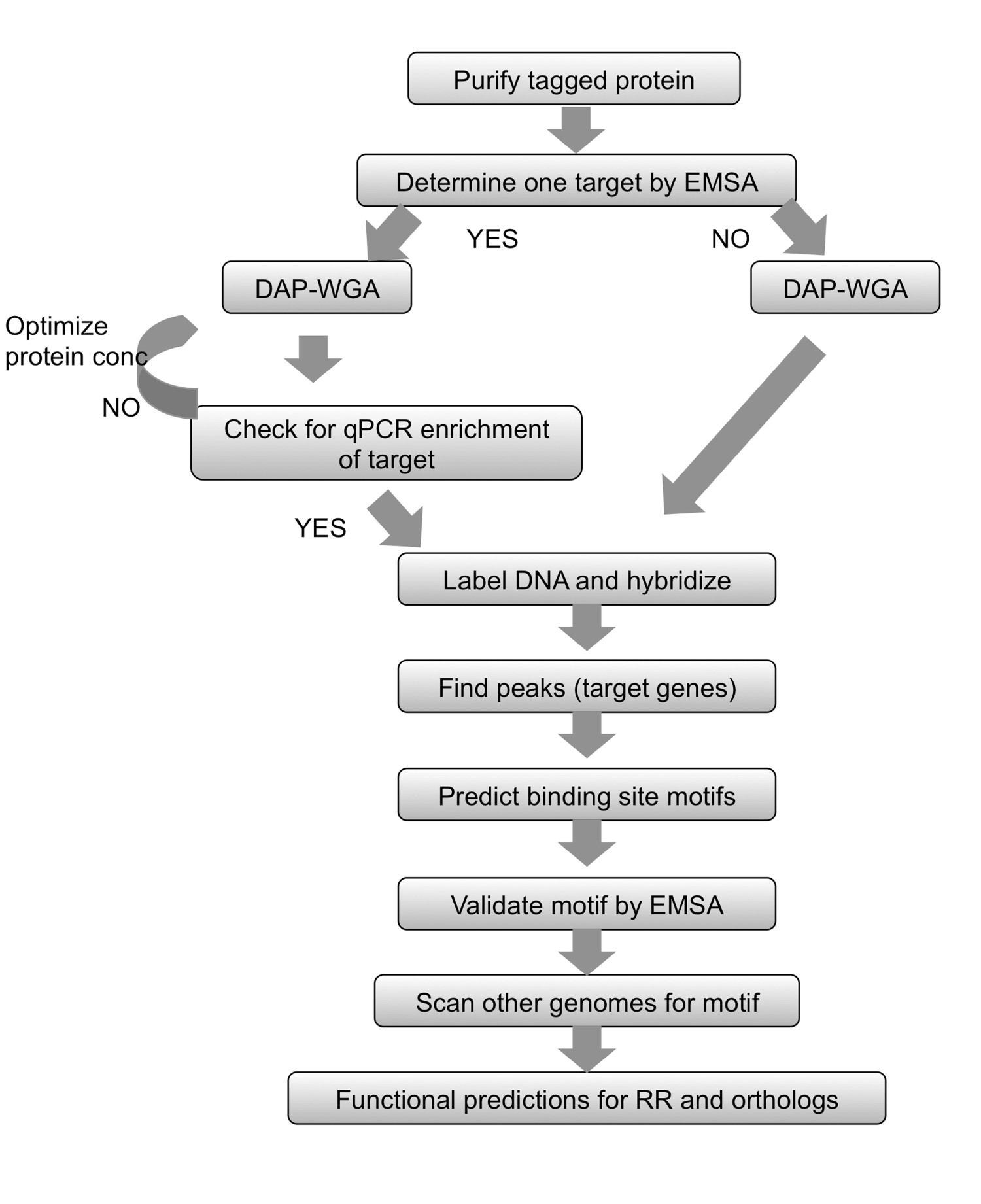
Abbildung 2. Zusammenfassung der Workflow. Für gereinigt getaggt protein, beginnen mit der Bestimmung eines Ziel mittels EMSA. Lassen Protein, um genomische DNA und DNA-Affinität-Entschlackung (DAP) und des gesamten Genoms binden verstärken (WGA) angereichert und die Input-DNA. Wenn ein Ziel-Gen bekannt ist, verwenden qPCR um sicherzustellen, daß der bekannte Target in der proteingebundenen Fraktion angereichert. Wenn kein Ziel ermittelt werden konnte, gehen Sie direkt zur DNA-Markierung und Array-Hybridisierung. Wenn Anreicherung durch qPCR konnte nicht beobachtet werden, wiederholen Sie den Protein-gDNA-Bindung und DAP-WGA Schritte mit unterschiedlichen Proteinmengen. Verwenden Array-Analyse Spitzen finden und sie den Zielgene. Verwenden Sie die vorgelagerten Bereiche des Zielgene zu Bindungsstelle Motive vorherzusagen. Bestätigen Sie die Motive experimentell unter Verwendung EMSAs. Verwenden Sie das Motiv, die Genome verwandter Arten Orthologe des RR-Studie unter Codierung zu scannen, und vorherzusagen, Gene in diesen Zielarten als gut. Basierend auf den Gen-Targets erhalten werden, kann die physiologische Funktion des RR und ihre Orthologe vorhergesagt werden. Abbildung modifiziert und neu aufgelegt mit der kreativen commons Lizenz von 7.
Protokoll
Hinweis: Die unten Protokoll wird für die Bestimmung von Gen-Ziele der RR DVU3023 aus dem Bakterium Desulfovibrio vulgaris Hildenborough zugeschnitten. Es können zu jedem anderen Transkriptionsregulator von Interesse angepasst werden.
1. Clone und Entschlacken RR
- Klonen Sie die RR-Gen, insbesondere DVU3023, aus D. vulgaris Hilden in einem Escherichia coli-Expressionsvektor, so dass das Gen C-terminal His-markiert und Expression unter der Kontrolle eines T7-Promotors.
Hinweis: Mehrere Klonierungsverfahren kann verwendet werden und wird durch den Forscher bestimmt. Alternative Affinitäts-Tags können auch verwendet werden. - Den Ausdruck in E. konstruieren coli BL21 (DE3) Expressionsstamm.
- Aufwachsen 1 L des Expressionsstamm BL21 bei 37 ° C. In der Mitte der log-Phase, fügen IPTG 0,5 mM bis Proteinexpression induzieren. Weiterhin Wachstum bei Raumtemperatur für 24 Stunden.
- Zentrifuge Zellen bei 5000 × g für 10 Meilenn bei 4 ° C Die Zellen in Lysepuffer (20 mM Natriumphosphat, pH 7,4, 500 mM NaCl, 40 mM Imidazol, 1 mg / ml Lysozym, 1x Nuklease Benzonase). Lysieren Zellen unter Verwendung eines Französisch-Presse bei 4 ° C.
- Zentrifuge Lysats bei 15.000 × g für 30 min bei 4 ° C. Filter mit einem 0,45 um Spritzenfilter.
- Auf einem FPLC-Gerät unter Verwendung von 10 ml Waschpuffer (20 mM Natriumphosphat, pH 7,4, 500 mM NaCl, 40 mM Imidazol) waschen ein 1-ml-Ni-Sepharose-Säule.
- Laden Lysat auf die Säule. Mit 20 ml Waschpuffer waschen Spalte.
- Eluat DVU3023 Verwendung eines Gradienten von 0-100% Elutionspuffer (20 mM Natriumphosphat, pH 7,4, 500 mM NaCl, 500 mM Imidazol).
- Laden eluierten Fraktionen auf eine Entsalzungssäule und Waschen mit einem Entsalzungspuffer (20 mM Natriumphosphat pH 7,4, 100 mM NaCl).
- Zentrifuge Proteinprobe in einem Hochmolekulargewichts-Cutoff-Filter-Zentrifuge, um das Protein zu konzentrieren. Hinzufügen DTT und 0,1 mM und Glycerin 50% Protein und Speicher bei -20 ° C.
Hinweis: Reinigungsverfahren müssen individuell für jede untersuchte Protein optimiert werden.
2. Bestimmen Gene Ziel für RR Mit elektrophoretische Mobilität Shift-Assay (EMSA)
- PCR amplifizieren 400 bp-Region stromaufwärts von der Kandidatenzielgens DVU3025 mit D. vulgaris Hilden genomischer DNA als Matrize und einen vorderen unmarkierten Primer und ein Rückwärts 5'-Biotin-markiertem Primer.
Hinweis: Tipps, um einen Kandidaten Zielgens wählen: Oft RRs binden die vorgelagerten Bereiche des eigenen Gen / Operon, oder sie können proximal codierten Gene regulieren. Wenn der RR hat Orthologe in anderen Spezies, suchen nach Genen, die in der Nachbarschaft konserviert sind. Alternative Methoden gehören die Auswahl von Kandidatengenen auf Basis Regulon Vorhersagen (zB RegPrecise 5) oder die Vorhersage sigma54-abhängigen Promotoren 6 für RRs, die sich sigma54 abhängig sind. - Führen Sie das PCR-Produkt auf einem 1% Agarose-Gel, schneiden Sie die 400 bp große Produkt und reinigen die DNA mit einem Gel Extraktion Kit bereinigen.
- Mischen DVU3023 Protein (0,5 pmol) mit 100 fmol der biotinylierten DNA-Substrat in 10 mM Tris-HCl, pH 7,5, 50 mM KCl, 5 mM MgCl 2, 1 mM DTT, 25% Glycerin und 1 &mgr; g / ml Poly dI.dC (Nicht spezifischen Kompetitor-DNA) in einem Gesamtvolumen von 20 pl. Auch eine Reaktion ohne Protein als Kontrolle gesetzt. Inkubiere die Reaktion bei Raumtemperatur auf der Bank 20 min.
- Anmerkung: Dies sind aufgelistet und in Abhängigkeit von der RR von Interesse können auch andere Reaktionskomponenten zugesetzt werden Standardbedingungen. Wenn die Aktivierung gewünscht, 50 mM Acetyl-Phosphat zu der Reaktion (oft RRs wird DNA in vitro zu binden, ohne Aktivierung).
- Vorlauf eine Fertig mini 6% Polyacrylamid-Gel in 0,5 x TBE 0,5 x TBE-Puffer bei 100 V für 30 min. In 5 ul 5x Ladepuffer (0,1% Bromphenolblau, 0,1% Xylencyanol, 30% Glycerin in 1x TBE) auf die Bindungsreaktionen und Last 18 ul der Reaktionen auf die gel. Führen Sie bei 100 V für 2 Stunden.
Hinweis: Um eine Überhitzung, die Dissoziation des Protein-DNA-Komplexe führen kann, füllen die äußere Pufferkammer in der Elektrophorese-System mit Laufpuffer, so dass die Mehrheit des Gels wird durch Puffer isoliert vermeiden. Alternativ kann das Gel bei 4 ° C ausgeführt werden, - Schnitt eine geladene Nylonmembran für die Größe des Gels und genießen sie in 0,5 x TBE für mindestens zehn Minuten. Entfernen Sie das Gel der Kassette. Schneiden Sie keine Kämme aus dem Gel und Sandwich-Gel und die Membran zwischen zwei in 0,5 x TBE getränkt dicke Filterpapiere und legen in einem Halbtrocken Blotapparatur und laufen bei 20 V für 30 min.
- Legen Sie die Membran in einem kommerziellen UV-Licht Vernetzungsinstrument und die Zeit auf 3 min.
Hinweis: Die Membran kann nun trocken bei Raumtemperatur für mehrere Tage, bevor zum nächsten Schritt gespeichert werden. - Verwenden Sie einen handelsüblichen Chemilumineszenznachweis-Kit, das eine Streptavidin-Meerrettich peroxida beschäftigtse-Konjugat, das Blot nach Herstellerangaben zu entwickeln.
- Bild der Blot unter Verwendung eines Computers angeschlossen an eine CCD-Kamera ausgestattet ist und nach einer Verschiebung in der Mobilität des DNA-Substrats in Gegenwart des RR, die anzeigt, dass der RR bindet die DNA getestet.
Hinweis: Die Spezifität der DNA-Protein-Verschiebung kann, indem in die Reaktions einen Überschuss an unmarkiertem Kompetitor-DNA, die Beseitigung oder Verringerung der Verschiebung gesehen sollte getestet werden.
3. Überprüfen Target Enrichment nach Genomic DNA-Protein-Bindung
- Genomic DNA-Protein-Bindungsreaktion
- Scher genomischen DNA auf eine durchschnittliche Größe von 500 bp durch Beschallen 100 &mgr; l genomische DNA (bei Konzentrationen von 100-200 ng / &mgr; l) in einem 1,5 ml Mikrozentrifugenröhrchen unter Verwendung einer Mikrospitze mit 9 Impulsen niedriger Amplitude von 1 Sekunde, 2 Sekunden Lücke zwischen jeden.
Hinweis: Wenn die DNA spritzt an den Seiten der Röhre, Spin-Down der Inhalt nach jeweils 3 Impulse. Die genauen Bedingungen verwendetwird mit dem Ultraschallgerät Instrument variieren. Die gescherten DNA-Fragmente können von 100-1000 bp liegen, aber die mittlere Größe innerhalb von 400-600 bp. Zu viel Scher kann in weniger intakte Bindungsstellen führen und entweder zu viel oder zu wenig Scher Bibliothek wird während der Herstellung nachgelagerten Amplifikation des gesamten Genoms Schritte beeinflussen. - Mischen 2-3 ug genomische DNA geschert mit RR-Protein (0,5 pmol DVU3023) in 10 mM Tris-HCl pH 7,5, 1 mM DTT, 50 mM KCl, 5 mM MgCl 2, 25% Glycerin und 50 mM Acetyl-Phosphat. Reaktionen Inkubieren bei 25 ° C für 30 min. Übertragungs 10 ul dieser Reaktion auf eine 1,5-ml-Röhrchen und Etikett als Eingangs DNA.
Hinweis: Acetyl-Phosphat zugesetzt wird, um das Protein durch in vitro-Phosphorylierung zu aktivieren. Die Phosphorylierung stimuliert die DNA-Bindung, aber viele RRs binden auch DNA in vitro 7 ohne Aktivierung. Die Proteinmenge der Reaktion zugegeben wird auf die Aktivität des Proteins prep abhängen. Die EMSA kann verwendet werden, um zu entscheiden seinimize Betrag.
- Scher genomischen DNA auf eine durchschnittliche Größe von 500 bp durch Beschallen 100 &mgr; l genomische DNA (bei Konzentrationen von 100-200 ng / &mgr; l) in einem 1,5 ml Mikrozentrifugenröhrchen unter Verwendung einer Mikrospitze mit 9 Impulsen niedriger Amplitude von 1 Sekunde, 2 Sekunden Lücke zwischen jeden.
- Affinity reinigen die Protein-gebundener DNA
- Hinzufügen von 30 &mgr; l Ni-NTA-Agarose-Harz zu einem 0,6 ml Mikrozentrifugenröhrchen. Zentrifugieren bei 100 g für 1 min, um das Harz am Boden zu sammeln, und den Überstand entfernen. 100 ul Waschpuffer (10 mM Tris-HCl, pH 7,5, 5 mM MgCl 2, 50 mM KCl, 25% Glycerin), Flick das Rohr zu mischen und Zentrifuge bei 100 × g für 2 min. Überstand entfernen.
- Die restlichen 90 &mgr; l der Bindungsreaktion zu der gewaschenen Ni-NTA-Harz und Inkubieren in einem Rundschüttler für 30 min.
- Zentrifugieren bei 100 g für 2 min, und entfernen Sie den Überstand (ungebundene DNA). 100 l Waschpuffer, Flick das Rohr, um den Inhalt zu mischen und Zentrifuge bei 100 g für 2 min. Überstand entfernen. Wiederholen Sie den Waschschritt noch zweimal.
- Mit 35 &mgr; l Elutionspuffer (20 mM Natriumphosphatpuffer, pH 8, 500 mM NaCl, 500 mM Imidazol) zum Harz und vortexen. Inkubieren auf der Bank bei RT für 5 min. Zentrifugieren bei 100 g für 2 min. Den Überstand in ein neues 1,5-ml-Röhrchen und Etikett wie das Protein-gebundenen DNA-Fraktion.
- Fügen auch 35 ul des Elutionspuffers mit dem Eingang DNA.
- Reinige den Eingangs-und die Protein-gebundenen DNA-Fraktionen unter Verwendung eines PCR-Reinigungskits.
- Gesamte Genom verstärken die Ein-und Protein-gebundener DNA-Proben.
- Werden 10 ul der Eingangs-und Protein-gebundenen DNA-PCR-Röhrchen zu trennen. 1 ul 10x Fragmentation-Puffer, 2 ul Bibliothek Präparationspuffer und 1 ul Bibliothek Stabilisierungslösung zu jeder Probe. Durch Vortexen mischen. Hitze in einem Thermocycler bei 95 ° C für 2 min, und Chill auf Eis.
Hinweis: Der Ausgangsbetrag der DNA sollte mindestens 10 ng um zu vermeiden, die Einführung Verstärkung Bias sein. - 1 ul der Bibliothek Vorbereitung Enzyme, mischen durch Pipettieren, und Inkubation im Thermocycler bei 16 ° C/20 min, 24 ° C/20 min, 37 ° C für 20 min, 75 ° C / 5 min, Und halten bei 4 ° C
- Zu jedem Röhrchen hinzugefügt 47,5 &mgr; l Wasser, 7,5 ul 10x-Amplifikation Master-Mix und 5 ul Polymerase. Gut mischen und Wärme bei 95 ° C / 3 min, gefolgt von 20 Zyklen von 94 ° C/15 sec, 65 ° C / 5 min. Halten Reaktionen bei 4 ° C
- Reinige die amplifizierte DNA-Proben unter Verwendung eines PCR-Reinigungs-Kit und DNA-Konzentration spektrophotometrisch messen.
- Werden 10 ul der Eingangs-und Protein-gebundenen DNA-PCR-Röhrchen zu trennen. 1 ul 10x Fragmentation-Puffer, 2 ul Bibliothek Präparationspuffer und 1 ul Bibliothek Stabilisierungslösung zu jeder Probe. Durch Vortexen mischen. Hitze in einem Thermocycler bei 95 ° C für 2 min, und Chill auf Eis.
- Stellen Sie sicher, Zielanreicherung in der Protein-gebundener DNA mit Hilfe der qPCR
- Design-qPCR-Primer 200 bp der stromaufwärts gelegenen Bereich der EMSA-prüft Zielgens (DVU3025) mit jedem frei zur Verfügung Primer-Design-Software zu verstärken.
- Bis dreifach qPCR Reaktionen für jede DNA-Vorlage mit jedem Primer-Set ein. Bereiten Sie ein Master-Mix für jedes Primer-Set. 1x Master-Mix enthält 10 ul 2x SYBR Green qPCR-Mix, 0,5 uM von jedem Primer und Wasser auf ein Gesamtvolumen von 18 ul. Aliquot 18 ul des Master-Mix pro Vertiefung einer 96-Well-PCR-Platte.
- DILUTe das verstärkte und gereinigt Eingangs-und Protein-gebundener DNA-Proben zu 5 ng / ul mit Wasser und verwenden als DNA-Vorlage. In 2 ul DNA-Vorlage in die Vertiefungen.
- Verschließen Sie die Platte mit einem extrem klare qPCR Siegelfolie, Spin-down-Platte in einer Zentrifuge bei 200 xg / 1 min. Zeigen Platte in Echtzeit qPCR Maschine. Zyklus wie folgt unter Verwendung der zugehörigen Software qPCR: 95 ° C / 1 min, 40 Zyklen von 95 º C/10 sec, 59 º C/15 sec, 70 º C/35 sec.
- Auch bis dreifacher Ausfertigung qPCR-Reaktionen mit Primern gesetzt zu den vorgelagerten Regionen eines nicht verwandten Gen (Negativkontrolle) zu verstärken.
- Berechnen &Dgr; T durch Subtrahieren C T-Wert von proteingebundenen DNA von dem der Ausgangs-DNA. Berechnen fache Anreicherung des Zielgens in der DNA-Protein-gebundenen als 2 &Dgr; C T.
Hinweis: Wenn der Ziel upstream-Region wurde in der Protein-gebundener DNA gegen den Eingang DNA angereichert ist, zeigt dies, dass die Bindungsreaktionen und Affinitätsreinigung waren successful. Fahren Sie mit Schritt 4 fort. Wenn Anreicherung der Ziel-Region stromaufwärts wurde nicht beobachtet, wiederholen Sie die Bindungsreaktionen (Schritt 3.1) mit verschiedenen Proteinmenge.
4. DNA Labeling-und Array-Hybridisierung
- Label-Eingabe-DNA mit Cy3 und Cy5-DNA mit Enriched
Anmerkung: Cy3-und Cy5-Farbstoffe sind lichtempfindlich und sollte darauf geachtet werden Lichtexposition auf ein Minimum zu halten.- Mischen Sie 1 ug DNA mit 40 ul Cy3/Cy5-labeled 9-mers und die Lautstärke anpassen, um 80 ul mit Wasser.
- Hitze denaturiert bei 98 ° C für 10 min im Dunkeln (in einem Thermocycler). Schnellkühl auf Eis für 2 min.
- Add 2 ul Klenow-Polymerase (3'-5'-exo - 50000 U / ml), 5 mM dNTPs und 8 &mgr; l Wasser zu jedem Reaktions, gut mischen, und bei 37 ° C für 2 h im Dunkeln (in ein Thermocycler).
- In EDTA bis 50 mM, um die Reaktion und NaCl-Lösung auf 0,5 M. stoppen
- Über Proben zu 1,5 ml Röhrchen containing 0,9 Volumen Isopropanol, Inkubation im Dunkeln für 10 min und Zentrifuge bei 12.000 g für 10 min. Das Pellet sollte rosa für Cy3-markierte DNA und blau für Cy5-markierte DNA sein.
- Waschen Sie das Pellet mit 80% Ethanol (500 ul) und Zentrifugation bei 12.000 × g für 2 min. Klima trockenen Pellets für 5-10 min im Dunkeln.
Hinweis: Die Pellets können bei -20 ° C gelagert werden - Pellet in 25 ul Wasser. Messen Sie die DNA-Konzentration spektrophotometrisch.
- Pool zusammen 6 ug jeder der Cy3-und Cy5-markierte DNA in einem 1,5-ml-Röhrchen und Vakuumtrocken in einer Zentrifuge bei schwacher Hitze in der Dunkelheit (decken den Zentrifugendeckel, wenn es transparent ist).
Hinweis: Pellets kann bei -20 ° C bis bereit für die Hybridisierung gespeichert werden.
- Microarray-Hybridisierung
- Drehen Sie den Hybridisierungssystem auf 3-4 Stunden vor der Verwendung und die Temperatur auf 42 ° C
- Bereiten Sie eine Hybridisierungslösung Master-Mix, so dass 1x Lösung enthält 11,8 ul 2x Hybridirung Puffer, 4,7 ul Hybridisierung Komponente A und 0,5 ul Ausrichtung Oligo.
- Resuspendieren der Pellets in 5 ul Wasser. Add 13 ul dieser Mischung auf die Probe. Vortex für 15 Sekunden, Inkubation bei 95 ° C in einem Trocken Bad für 5 min. Halten Proben bei 42 ° C in der Hybridisierungssystem bis zum Laden bereit.
- Vorbereitung der Mikroarray-Objektträger-Mischeranordnung gemäß dem Protokoll des Herstellers.
- Setzen Sie den Mixer-Schlittenanordnung innerhalb des Hybridisierungssystems. Last 16 ul Probe in die Einfüllöffnung abdichten Ports mit einer Klebefolie, drehen Mischen auf in dem System, und hybridisieren für 16-20 h bei 42 ° C.
- Bereiten 1x Waschpuffer I (250 ml), II (50 ml) und III (50 ml) durch Verdünnen der 10x kommerziell verfügbaren Puffer in Wasser, und jeweils 1 mM DTT hinzufügen. Warme Puffer I auf 42 ° C
- Schieben Sie die Mixer-Folie in einem Demontagewerkzeug, Ort und in einer Schale mit warmem Puffer I. Peel der Mischer aus, während kräftig shnter den Demontagewerkzeug von Hand.
- Zeigen Sie die Folie in einen Behälter mit 50 ml Waschpuffer I und kräftig schütteln für 2 min mit der Hand.
- Transferschlitten in einen zweiten Behälter mit 50 ml Waschpuffer II und kräftig schütteln für 1 min mit der Hand.
- Transferschlitten in einen dritten Behälter mit 50 ml Waschpuffer III und kräftig schütteln für 15 Sekunden von Hand.
- Blot schnell die Ränder der Folie auf ein Papiertuch und legen in einer Dia-Rack. Zentrifugieren bei 200 g für 2 min, um die Diashow zu trocknen. Legen Sie innerhalb Dia Fall, wickeln Sie es mit Folie, und speichern Sie in einem Exsikkator.
- Scannen des Array
- Den Objektträger in der Microarray-Scanner nach den Anweisungen des Instruments.
- Verwenden Sie eine Scanner-Software, um die Wellenlängen 532 nm = Cy3 und Cy5 = 635 nm eingestellt, und die ersten Photomultiplier Gewinne zwischen 350 und 400.
- Vorschau auf die Folie, um das Array auf der Folie zu finden. Wählen Sie das Array-Bereich für das Scannen.
- Scannen Sie den Array und stellen die Photomultiplier-Einstellungen, so dass die Array-Funktionen sind meist gelb. Das Histogramm sollte die rote und grüne Kurven überlagert oder so nah wie möglich beieinander zu zeigen. Die Kurven sollten über den 10 -5 normalisierte zählt bei Sättigung beenden.
- Sparen Sie separat beide 532 und 635 nm Bilder.
- Array-Datenanalyse
- Importieren Sie die Bilder in ein Array-Analyse-Software. Mit der Software erstellen Sie zwei Paar Berichte (. Paar) für jedes Array (für Cy3 und Cy5-Bilder). Neues skalierte Protokoll 2-Verhältnis Dateien mit den Paar-Dateien. Verwenden Sie log 2 Verhältnis Dateien für Spitzen mit einem Schiebefenster von 500 bp suchen. Karte des Spitzen Loci zu den vorgelagerten Bereiche des Gen-Targets.
- Prüfen Sie, ob der positive Ziel (bestimmt mit EMSA und qPCR, DVU3025 in diesem Beispiel bestätigt) innerhalb der oberen Spitzen erscheint.
Hinweis: Wenn das positive Ziel ist nicht unter den Top-Hits, ist es möglich, dass die RR unter consideration mehrere Gen-Targets und replizieren DAP-Chips durchgeführt werden kann und die Spitzen, die für alle Wiederholungen verwendet werden, um eine Zielliste erzeugt.
5. Binding Site Motiv Vorhersage und Validierung
- Rufen Sie Sequenzen für Upstream-Regionen (400 bp) für die Top-Gen-Targets wie DAP-Chip-Analyse erzeugt. Bewerben MEME dieser Sequenzen durch Mikroben Online (meme.nbcr.net), um Motive vorherzusagen.
Hinweis: Enhancer-Bindungsproteine wie sigma54 abhängigen Regler können mehrere hundert Basenpaare stromaufwärts von der Startstelle zu binden. Für andere Transkriptionsregulatoren eine kürzere Region wie 200 bp stromaufwärts ausreichen. - Design-Ober-und Unter DNA-Oligomere, die die vorhergesagte Bindungsstelle Motiv von 10 Basen an beiden Enden flankiert enthalten. Bestellen Sie den oberen Strang 5 'biotinyliert.
- Mischen der oberen und unteren Strang Oligos Verhältnis 1:1,5 in 10 mM Tris-HCl, pH 8,0, 1 mM EDTA und 50 mM NaCl inein Gesamtreaktionsvolumen von 20 ul in PCR-Röhrchen. Hitze auf 95 ° C für 5 min in einem thermischen Cycler, gefolgt von langsamem Abkühlen auf 25 ° C. Verdünnen Sie das Zehnfache und die Verwendung als Wildtyp-dsDNA-Substrat für die EMSA.
- Vorbereitung modifizierten Substraten unter Verwendung der gleichen Schritte wie oben, aber Design Oligomere 4-6 Substitutionen in der konservierten Basen der Bindungsmotiv tragen.
- Richten Sie Bindungsreaktionen mit dem RR und dem Wildtyp-oder modifizierte Substrate und untersuchen mittels EMSA, wie in Schritt 2 beschrieben.
- Hinweis: Die vorhergesagte Motiv wird validiert, wenn die RR bindet den Wildtyp-Substrat, aber nicht die modifizierte ein.
6. Erhaltung der Motif in anderen verwandten Bakterienarten
- Wählen Genome von Interesse, dass Orthologe DVU3023 enthalten. Erhalten Folge und Anmerkungsdateien für den Genomen von der NCBI-Website.
- Verwenden Sie eine Programmiersprache wie Perl-Skripte, die die Motiv-Sequenzen aus der DAP-Chip-Ziele verwenden, um eine pos bauen schreibenition gewichteten Matrix und verwenden Sie die Matrix, in anderen sequenzierten Genomen vorhanden ähnliche Motive punkten.
Ergebnisse
Das oben genannte Verfahren wurde angewendet, um die globalen Gen-Targets der RRS in der Modellsulfatreduzierenden Bakteriums Desulfovibrio vulgaris Hildenborough 7 zu bestimmen. Dieser Organismus besitzt eine große Anzahl von FKS über 70 RRs dargestellt, was auf die Vielzahl von möglichen Signalen, die es erfaßt und darauf reagiert. In-vivo-Analysen der Funktionen dieser Signalanlagen schwer, da ihre Signale führen und somit ihre Aktivierungsbedingungen sind unbekannt. Hier wurde die D...
Diskussion
Die hier beschriebene DAP-Chip-Methode wurde erfolgreich eingesetzt, um die Gen-Targets für mehrere RRS in Desulfovibrio vulgaris Hildenborough 7, von denen eine hier als repräsentatives Ergebnis gezeigt zu bestimmen. Für RR DVU3023, die Wahl eines Kandidaten-Gen Ziel war einfach. DVU3025 ist unmittelbar stromabwärts von der RR-Gen und die RR-und Zielgene werden in mehreren Desulfovibrio-Arten konserviert sind, und zusätzlich eine vorhergesagte DVU3025 sigma54-abhängigen Promotor. Die ...
Offenlegungen
Die Autoren haben keinen Interessenkonflikt offen zu legen.
Danksagungen
Wir danken Amy Chen für ihre Hilfe bei der Vorbereitung der Videodreh und zur Demonstration der Technik. Diese Arbeit von ENIGMA durchgeführt: Ökosysteme und Netzwerke integriert mit Gene und molekulare Baugruppen (http://enigma.lbl.gov), ein Scientific Focus Area Programm am Lawrence Berkeley National Laboratory, wurde durch das Amt für Wissenschaft, Amt für Biologische und unterstützt Umweltforschung, des US Department of Energy unter Vertrag Nr. DE-AC02-05CH11231.
Materialien
| Name | Company | Catalog Number | Comments |
| Name of Material/Equipment | Company | Catalog number | Comments |
| HisTrapFF column (Ni-Sepharose column) | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5255-01 | |
| Akta explorer (FPLC instrument) | GE Lfe Sciences, Pittsburgh, PA, USA | ||
| HiPrep 26/10 Desalting column | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5087-01 | |
| Qiaquick Gel extraction kit | Qiagen Inc, Valencia, CA, USA | 28704 | |
| Biotin-labeled oligonucleotides | Integrated DNA Technologies | N/A | |
| 6% polyacrylamide-0.5X TBE precast mini DNA retardation gel | Life Technologies, Grand Island, NY, USA | EC63652BOX | Alternately, you can pour your own gel. |
| Nylon membrane | EMD Millipore, Billerica, MA, USA | INYC00010 | |
| Trans-Blot SD Semi-dry electrophoretic transfer cell | Biorad, Hercules, CA, USA | 170-3940 | |
| Extra thick blot paper, 8 x 13.5 cm | Biorad, Hercules, CA, USA | 170-3967 | |
| UV crosslinker Model XL-1000 | Fisher Scientific | 11-992-89 | |
| Nucleic Acid chemiluminescent detection kit (Pierce) | Thermo fisher Scientific, Rockford, IL, USA | 89880 | |
| Ni-NTA agarose resin | Qiagen Inc, Valencia, CA, USA | 30210 | |
| GenomePlex Whole genome amplification kit (Fragmentation buffer, library preparation buffer, library stabilization solution, library preparation enzyme, 10X amplification master mix, WGA polymerase ) | Sigma-Aldrich, St. Louis, MO, USA | WGA2-50RXN | |
| Nanodrop ND-1000 | Thermo Scientific, Wilmington, DE, USA | For quantitation of DNA | |
| Perfecta Sybr Green SuperMix, with ROX | Quanta biosciences | 95055-500 | Any Sybr Green PCR mix may be used |
| PlateMax Ultra clear heat sealing film for qPCR | Axygen | ||
| 96 well clear low profile PCR microplate | Life Technologies, Grand Island, NY, USA | PCR-96-LP-AB-C | |
| Applied Biosystems StepOne Plus Real time PCR system | Life Technologies, Grand Island, NY, USA | 4376600 | Any real time PCR system may be used |
| Qiaquick PCR purification kit | Qiagen Inc, Valencia, CA, USA | 28104 | Any PCR clean up kit may be used |
| Cy3/Cy5-labeled nonamers | Trilink biotechnologies, San Diego, CA, USA | N46-0001, N46-0002 | |
| Klenow polymerase 50,000U/ml, 3'-5' exo- | New England Biolabs, Ipswich, MA | M0212M | |
| Hybridization system | Roche-Nimblegen, Madison, WI, USA | N/A | This company no longer makes arrays or related items, so alternate sources such as Agilent or Affymetrix will need to be used, |
| Custom printed microarrays and mixers | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Hybridization kit (2X Hybridization buffer, Hybridization component A, Alignment oligo) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Wash buffer kit (10X Wash buffer I, II, III, 1 M DTT) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| GenePix 4200A microarray scanner | Molecular Devices, Sunnyvale CA, USA | This model has been replaced by superior ones | |
| GenePix Pro microarray software | Molecular Devices, Sunnyvale CA, USA | ||
| Nimblescan v.2.4, ChIP-chip analysis software | Roche-Nimblegen, Madison, WI, USA | N/A | |
Referenzen
- Galperin, M. Y. Diversity of structure and function of response regulator output domains. Curr Opin Microbiol. 13, 150-159 (2010).
- Casino, P., Rubio, V., Marina, A. The mechanism of signal transduction by two-component systems. Curr Opin Struct Biol. 20, 763-771 (2010).
- Aparicio, O., Geisberg, J. V., Struhl, K. Chromatin immunoprecipitation for determining the association of proteins with specific genomic sequences in vivo. Curr Protoc Cell Biol. 17 (17), (2004).
- McCleary, W. R., Stock, J. B. Acetyl phosphate and the activation of two-component response regulators. J Biol Chem. 269, 31567-31572 (1994).
- Novichkov, P. S., et al. RegPrecise web services interface: programmatic access to the transcriptional regulatory interactions in bacteria reconstructed by comparative genomics. Nucleic Acids Res. 40, 604-608 (2012).
- Studholme, D. J., Buck, M., Nixon, T. Identification of potential sigma(N)-dependent promoters in bacterial genomes. Microbiology. 146 (12), 3021-3023 (2000).
- Rajeev, L., et al. Systematic mapping of two component response regulators to gene targets in a model sulfate reducing bacterium. Genome Biol. 12, 99 (2011).
- Crooks, G. E., Hon, G., Chandonia, J. M., Brenner, S. E. WebLogo: a sequence logo generator. Genome Res. 14, 1188-1190 (2004).
- Barbieri, C. M., Mack, T. R., Robinson, V. L., Miller, M. T., Stock, A. M. Regulation of response regulator autophosphorylation through interdomain contacts. J Biol Chem. 285, 32325-32335 (2010).
- Bourret, R. B. Receiver domain structure and function in response regulator proteins. Curr Opin Microbiol. 13, 142-149 (2010).
- Gao, R., Stock, A. M. Molecular strategies for phosphorylation-mediated regulation of response regulator activity. Curr Opin Microbiol. 13, 160-167 (2010).
- Hung, D. C., et al. Oligomerization of the response regulator ComE from Streptococcus mutans is affected by phosphorylation. J Bacteriol. 194, 1127-1135 (2012).
- Ladds, J. C., et al. The response regulator Spo0A from Bacillus subtilis is efficiently phosphorylated in Escherichia coli. FEMS Microbiol Lett. 223, 153-157 (2003).
- Wen, Y., Feng, J., Scott, D. R., Marcus, E. A., Sachs, G. Involvement of the HP0165-HP0166 two-component system in expression of some acidic-pH-upregulated genes of Helicobacter pylori. J Bacteriol. 188, 1750-1761 (2006).
- Liu, X., Noll, D. M., Lieb, J. D., Clarke, N. D. DIP-chip: rapid and accurate determination of DNA-binding specificity. Genome Res. 15, 421-427 (2005).
- Gossett, A. J., Lieb, J. D. DNA Immunoprecipitation (DIP) for the Determination of DNA-Binding Specificity. CSH Protoc. 2008, (2008).
- Mascher, T., et al. The Streptococcus pneumoniae cia regulon: CiaR target sites and transcription profile analysis. J Bacteriol. 185, 60-70 (2003).
- Park, P. J. ChIP-seq: advantages and challenges of a maturing technology. Nat Rev Genet. 10, 669-680 (2009).
- Lefrancois, P., Zheng, W., Snyder, M. ChIP-Seq using high-throughput DNA sequencing for genome-wide identification of transcription factor binding sites. Methods Enzymol. 470, 77-104 (2010).
- Ho, J. W., et al. ChIP-chip versus ChIP-seq: lessons for experimental design and data analysis. BMC Genomics. 12, 134 (2011).
- Liu, X., Lee, C. K., Granek, J. A., Clarke, N. D., Lieb, J. D. Whole-genome comparison of Leu3 binding in vitro and in vivo reveals the importance of nucleosome occupancy in target site selection. Genome Res. 16, 1517-1528 (2006).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten