JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
세균에 대한 두 개의 구성 요소 규제 시스템을 유전자 표적을 확인하는 DNA 친화력 - 정제 칩 (DAP-칩) 방법
요약
이 비디오 문서는 두 개의 구성 요소 시스템 응답 레귤레이터 유전자 표적 및 결합 부위를 결정하기 위해 시험 관내 마이크로 어레이 기반의 방법을 설명한다.
초록
이러한 칩 칩으로 생체 방법에서 전사 인자 글로벌 유전자 목표를 결정하는 데 사용 잘 확립 기술은 다음과 같습니다. 그러나, 그들은에 uncharacterized 활성화 조건 세균성 두 구성 요소 규제 시스템을 탐험에 사용이 제한됩니다. 독특한 신호들의 존재하에 활성화 된 때, 그러한 시스템에만 전사를 조절한다. 이들 신호는 종종 알 수 있기 때문에,이 비디오 문서에 설명 된 시험 관내 마이크로 어레이 기반의 방법은 유전자 표적 및 응답 레귤레이터 결합 부위를 결정하기 위하여 사용될 수있다. 이것은 DNA-친 화성 정제 칩 방법은 시퀀스 된 게놈 어떤 유기체 어떠한 레귤레이터 정제에 사용될 수있다. 프로토콜은 정제 태그 단백질은 사용자 정의 타일 배열에 DNA와 하이브리드의 형광 표지 다음에 단백질 바인딩 DNA를 깎인 게놈 DNA에 결합하고 친화력 정화 할 수 있도록 포함한다. 이 사양에 대한 분석을 최적화하기 위해 사용될 수있다 같이 전일C 규제도 설명되어 있습니다. 어레이 데이터 분석에 의해 생성 된 피크 후 실험적으로 검증되는 결합 부위 모티프를 예측하는 데 사용된다. 모티프 예측 더욱 밀접한 관련 종 orthologous 반응 조절제의 유전자 표적을 결정하기 위하여 사용될 수있다. 우리는 유전자의 목표를 결정하고 사이트의 모티브를 결합함으로써 환경 박테리아 Desulfovibrio 심상 성 Hildenborough에서 sigma54 의존 응답 레귤레이터 DVU3023의 기능을 예측하여이 방법의 적용 가능성을 보여줍니다.
서문
생존하고 번성하는 박테리아의 능력들은 인식하고 그들의 환경에서 섭동에 응답 할 수있다 얼마나 잘 매우 의존적이며, 이것은 차례로 자신의 신호 전달 체계에 의존한다. 경보 시스템 박테리아 인코딩의 수는 "미생물 IQ"라고되어 그 환경의 변화와 여러 신호를 감지 할 수있는 능력과 미세 조정의 응답 1 두의 표시가 될 수 있습니다. 두 성분의 신호 전달 시스템 (TCS)은 세균에 의해 사용되는 가장 널리 시그널링 시스템이며, 그들은 외부 신호를 감지하여 음향 응답 레귤레이터 (RR) 2 인산화를 통해 송신 히스티딘 키나아제 (HK)으로 구성된다. 의 RR은 출력 영역의 다양하므로 서로 다른 음향 모드를 가질 수 있지만, 가장 일반적인 반응은 도메인 1 DNA 결합을 통해 전사 조절이다. 감지 신호 및 VAS의 대응 기능TCSS의 t의 대부분은 알 수없는 남아 있습니다.
이러한 칩 - 칩과 같은 생체 내 방법으로 일상적 전사는 3 요소 게놈 결합 부위의 측정에 사용되지만, 활성화 조건 또는 신호가 알려져있는 경우, 그들은 단지 세균이 성분계 RR들에 대해 사용될 수있다. 종종 TCS를 활성화 환경 단서는 자신의 유전자를 대상으로보다 결정하기 어렵습니다. 시험 관내 마이크로 어레이는 기재 기반 분석을 효과적으로 빠르게 유전자 표적을 결정하고 TCSS의 기능을 예측하기 위해 사용될 수있다. 상기 분석 된 RR가 인산화 따라서 아세틸 포스페이트 4와 같은 작은 분자를 사용 도너 시험 관내에서 활성화 될 수 있다는 사실을 이용한다.
이 방법에서는, DNA 친화력 - 정제 - 칩 (그림 1)에 대한 DAP 칩 이름, 관심의 RR 유전자는 E.에서 그의 태그로 복제됩니다 대장균 및 이후 정제 태그 단백질은 양방향으로 허용된다차 게놈 DNA를 전단한다. 단백질 - 결합 DNA가 다음 친화력 정화에 충실 풍부한 입력 DNA가 증폭, 형광 함께 풀링 및 사용자 관심의 유기체 (그림 1)에 만든 기와 배열에 하이브리드 레이블. 마이크로 어레이 실험 유물로 될 수 있으며, 따라서 추가 단계 분석을 최적화하기 위해 사용된다. 하나의 단계는 (그림 2에서 워크 플로우를 참조) 전기 이동성 이동 분석 (EMSA)를 사용하여 연구에서 RR에 대한 하나의 목표를 결정하려고하는 것입니다. 그런 다음, 게놈 DNA와 DAP 단계에 바인딩 다음, 단백질 - 결합 및 입력 DNA 따라서 최적의 바인딩 상태를 확인한, 긍정적 인 목표는 입력 부분에 상대적으로 단백질 - 결합 부분에 농축되어 있는지 qPCR에 의해 검사된다 RR (그림 2). 어레이 혼성화 후, 데이터는 게놈 궤적을 나타내는 높은 강도 신호의 피크를 찾기 위해 분석된다 여기서 단백질 H광고는 바인딩. 함수 얻어진 유전자 표적에 근거 RR 대해 예측 될 수있다. 대상 게놈 유전자 좌는 다음 실험적 EMSAs (그림 2)를 사용하여 유효성을 검사 결합 부위의 모티브를 예측하는 데 사용됩니다. RR의 기능 예측 및 유전자 목표는 밀접하게 유사한 결합 모티프에 대한 사람들의 게놈 (그림 2)를 스캔하여 orthologous의 RR을 인코딩 종 관련 확장 할 수있다. DAP 칩 방법은 이전에 아무도 없었다 TCS에 대한 풍부한 정보를 제공 할 수 있습니다. 상기 방법은 또한 단백질이 정제 될 수 있으며 DNA 결합 상태가 결정될 수 있으면 어떠한 전사 조절을 위해 사용하고, 게놈 서열과 관심있는 유기체에 해당 될 수있다.
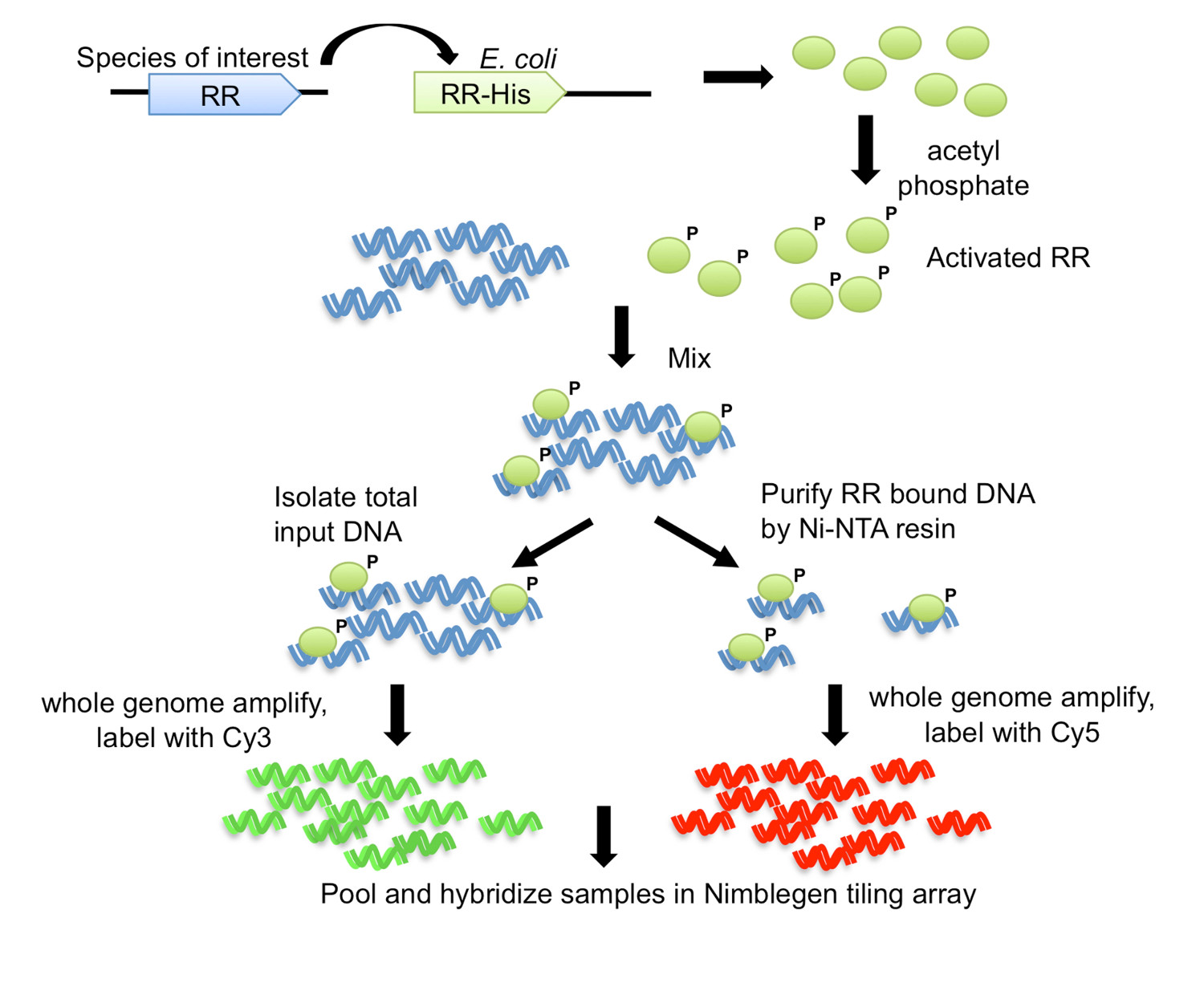
그림 1. DNA 친화력 - 정제 - 칩 (DAP-칩) 전략 7. 그 유기체에서 RR 유전자는 E.에 카르복시 말단 그의 태그로 복제됩니다 대장균 발현 균주. 정제 그의 태그가 단백질 아세틸 포스페이트 인산화에 의해 활성화하고, 쉬어 된 게놈 DNA와 혼합된다. 나머지는 니켈-NTA 수지를 사용하여 친 화성 정제를 실시하면서 결합 반응의 분취 량은, 입력 DNA로 저장된다. 입력 및 RR 바인딩 된 DNA는 증폭 된 전체 게놈 있으며, 각각를 Cy3 및 Cy5에,로 표지. 표지 된 DNA를 함께 풀링하고 유전자 표적을 결정하기 위해 분석된다 타일링 어레이에 혼성화된다. 그림 수정 및 크리에이티브 커먼즈 라이센스를 7에서 사용하는 재판.
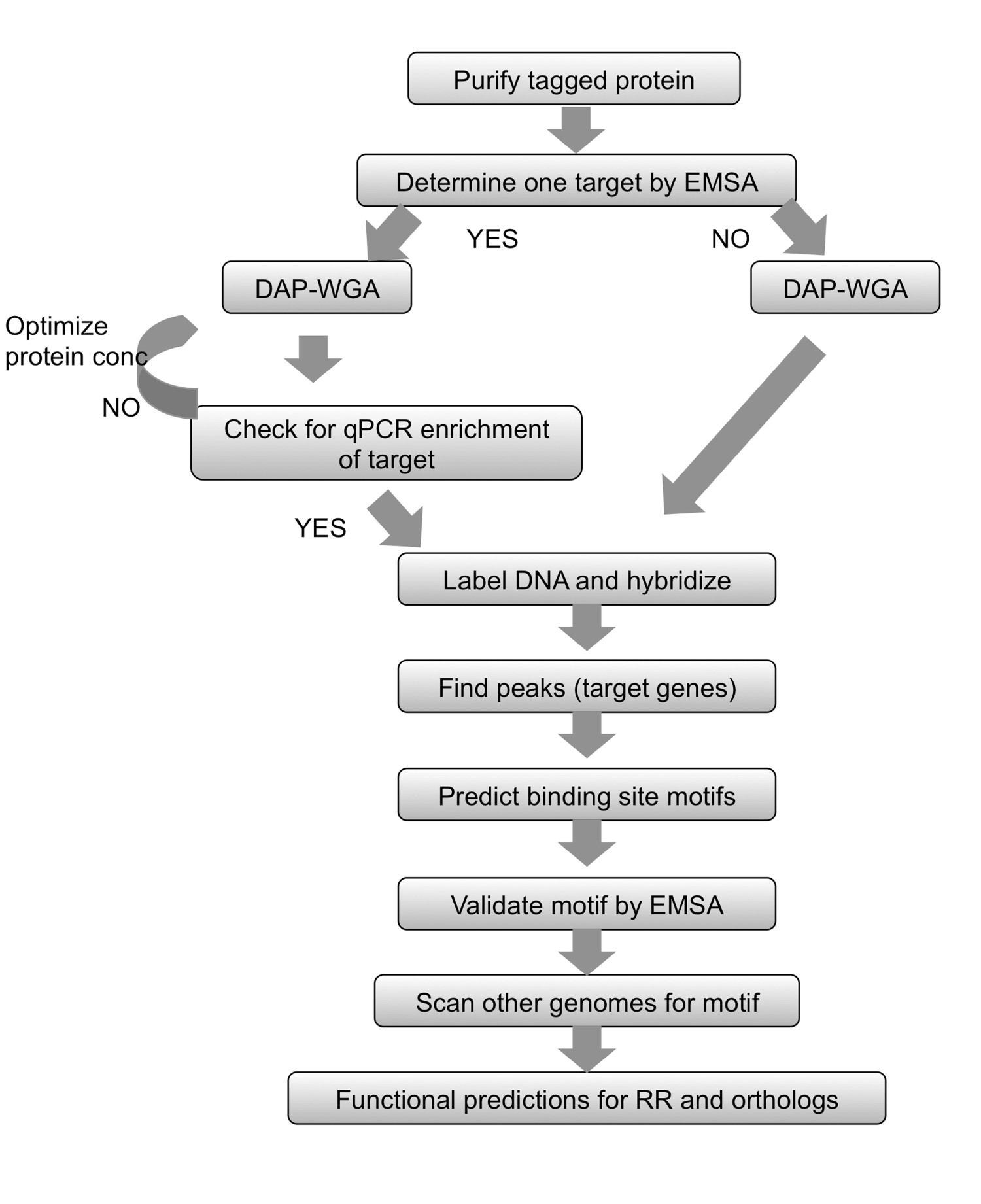
워크 플로의 그림 2. 요약. 모든 정제 태그 PROTEI 위해N, EMSA를 이용하여 대상을 결정함으로써 시작한다. (WGA) 농축 및 입력 DNA 증폭 게놈 DNA 다음 DNA 친화력 - 정화 (DAP) 및 전체 게놈을 결합하는 단백질을 허용합니다. 표적 유전자가 알려진 경우, 알려진 타겟 단백질 - 결합 분획에 농축되도록 qPCR에를 사용한다. 어떤 대상을 판별 할 수 있다면, DNA 라벨 및 배열 하이브리드로 직접 진행합니다. qPCR에 의한 농축이 관찰 할 수없는 경우, 결합 단백질 - gDNA를 다른 단백질의 양을 사용하여 DAP-WGA의 단계를 반복합니다. 피크를 찾아 유전자를 대상에 매핑하는 배열 분석을 사용합니다. 결합 부위 모티프를 예측하는 표적 유전자의 상류 영역을 사용한다. 실험적으로 EMSAs를 사용하여 주제를 유효성을 검사합니다. 연구에서 RR의 orthologs를 인코딩 관련 종의 게놈을 스캔 모티브를 사용하고,뿐만 아니라 그 종을 대상으로 유전자를 예측하고있다. 얻어진 유전자 타겟에 기초하여, RR과 orthologs의 생리적 기능은 예측 될 수있다. 그림 수정하고 창조적 인 C를 사용하여 다시 인쇄ommons 7에서 라이센스.
프로토콜
참고 : 아래의 프로토콜이 박테리아 Desulfovibrio 심상 성 Hildenborough에서 RR의 DVU3023의 유전자 표적의 결정에 맞게 조정됩니다. 그것은 또한 임의의 다른 전사 조절에 적용될 수있다.
1. 복제 및 순화 RR
- RR 유전자, D.에서 구체적으로 DVU3023를 복제 불가리스 Hildenborough 대장균 발현 벡터에 유전자가 C-말단 그의-태그와식이 T7 프로모터의 제어하에되도록.
참고 : 몇 클로닝 방법을 사용할 수 있고 연구원에 의해 결정된다. 다른 친화도 태그도 사용될 수있다. - 식 E.로 구성 변환 대장균 BL21 (DE3) 식을 변형.
- 37 ° C에서 BL21 식 변형의 1 L를 성장 중반 로그 단계에서, 단백질의 발현을 유도하기 위해 0.5 mm로 IPTG를 추가합니다. 실온에서 24 시간 동안 성장을 계속한다.
- 10 마일 5,000 XG에 원심 분리기 세포4 ℃에서 N 용해 완충액에서 재현 탁 셀 (20 mM의 나트륨 포스페이트, pH 7.4의 500 mM의 염화나트륨, 40 mM의 이미 다졸, 1 ㎎ / ㎖ 리소자임, 1X 클레아 벤조). 4 ℃에서 프랑스의 프레스를 사용하여 세포를 Lyse
- 4 ℃에서 30 분 동안 15,000 XG에 원심 분리기 해물 0.45 μm의 주사기 필터를 사용하여 필터링 할 수 있습니다.
- 세척 버퍼 10 ML (20 MM의 인산 나트륨, 산도 7.4, 500 mM의 NaCl을, 40 mM의 이미 다졸)를 사용하여 FPLC 악기에 1 ㎖ 니켈 세 파로스 칼럼을 씻으십시오.
- 열 해물을로드합니다. 세척 버퍼 20 ㎖로 컬럼을 씻으십시오.
- 0-100 % 용출 완충액 구배 (20 mM의 나트륨 포스페이트, pH 7.4의 500 mM의 NaCl을 500 mM의 이미 다졸)를 사용하여 용리 DVU3023.
- 탈염 컬럼에서 용출 분수를로드하고 탈염 버퍼 (20 MM의 인산 나트륨의 pH 7.4, 100 mM의 염화나트륨)로 세척.
- 단백질을 농축하는 고 분자량 컷오프 필터 원심 분리기에서 단백질 샘플. -20 ° C에서 50 % 및 저장 단백질 0.1 ㎜, 글리세롤에 DTT 추가
주 : 정제 방법이 연구에서 각각의 단백질에 대해 개별적으로 최적화해야합니다.
2. 전기 영동 이동성 이동 분석 (EMSA)를 사용하여 RR에 대한 유전자의 대상을 결정
- PCR은 D.를 사용하여 후보 타겟 유전자 DVU3025 400 bp의 상류 영역을 증폭 불가리스 Hildenborough 주형으로서 게놈 DNA 및 비 표지 정방향 프라이머 및 역방향 5'-바이오틴 - 표지 된 프라이머.
참고 : 종종 RR이 자신의 유전자 / 오페론의 상류 지역을 결합하거나 근위부를 인코딩 유전자를 조절할 수 있습니다 후보 표적 유전자를 선택하는 팁. RR은 다른 종의 orthologs이있는 경우, 지역에 보존되어 유전자를 찾습니다. 다른 방법은 자신이 sigma54 - 의존의 RR에 대한 귤론 예측 (예를 들어, RegPrecise 5), 또는 예측 sigma54 의존 발기인 6에 따라 선택하는 후보 유전자 (가) 있습니다. - B (400)를 잘라, 1 % 아가 로즈 겔상에서 PCR 생성물을 실행할P 크기의 제품 및 겔 추출 키트를 정리하여 DNA를 정화.
- 10 mM 트리스 염산, 산도 7.5, 50 밀리미터의 KCl, 5 mM의 MgCl2를 2, 1 mM의 DTT, 25 % 글리세롤, 1 ㎍ / ㎖의 폴리 dI.dC (비에 바이오틴 DNA 기판의 100 fmol으로 DVU3023 단백질 (0.5 pmol의)을 혼합 20 μL의 총 부피의 별 경쟁 DNA). 또한 제어와 같은 단백질이없는 반응을 설정합니다. 20 분 동안 벤치에 실온에서 반응을 품어.
- 참고 :이 나열과 다른 반응 구성 요소가 관심의 RR에 따라 추가 될 수있다 표준 조건입니다. 활성화가 요구되는 경우, 반응물 (종종의 RR이 활성화하지 않고 시험 관내에서 DNA를 결합한다)에 50 mM의 아세틸 포스페이트를 추가한다.
- 30 분 동안 100 V에서 0.5 배의 TBE 버퍼에 프리 캐스트 미니 6 % 폴리 아크릴 아미드 - 0.5 배의 TBE 젤을 사전 실행합니다. G에 대한 결합 반응과 반응의 부하 18 μL에 배 로딩 버퍼의 5 μL (파란색 0.1 % 브로 모 페놀, 0.1 % 자일 렌 cyanol, 1X TBE 30 % 글리세롤)를 추가엘. 2 시간 동안 100 V에서 실행합니다.
주 : 겔의 대부분이 버퍼에 의해 절연되도록 버퍼를 실행하여 전기 시스템에서 외부 버퍼 챔버를 채우는, 단백질-DNA 복합체의 탈퇴로 이어질 수있는 과열을 방지하기 위해. 또는, 겔은 4 ℃에서 실행할 수 있습니다 - 젤의 크기에 충전 된 나일론 막 잘라 적어도 10 분 동안 0.5 배의 TBE에 담근. 카세트에서 젤을 제거합니다. 젤 떨어져있는 능선을 잘라 0.5 배의 TBE에 담가 두 개의 두꺼운 여과지 사이에 젤과 막 사이에, 그리고 반 건조 블로 팅 장치 내부에 배치하고 30 분 동안 20 V에서 실행합니다.
- 상업 자외선 가교제 악기 내부에 멤브레인을 놓고 3 분으로 시간을 설정합니다.
참고 : 멤브레인 이제 다음 단계로 진행하기 전에 몇 일 동안 실온에서 건조 저장 될 수있다. - 스트렙 타비 딘 - 말 무 과산화를 채용 시판 화학 발광 검출 키트를 사용제조업체의 지침에 따라 오점을 개발하기 위해 자체의 어원.
- 이미지를 컴퓨터를 사용하여 얼룩은 CCD를 탑재 카메라에 매여 및 RR 테스트중인 DNA 바인딩을 나타냅니다 RR의 존재의 DNA 기판 이동의 변화를 찾습니다.
주 : 단백질-DNA 시프트의 특이 반응에 제거하거나 볼 시프트를 줄여야 비 표지 경쟁자 DNA의 과잉을 포함하여 시험 될 수있다.
3. 게놈 DNA-단백질 바인딩 후 대상 농축 확인
- 게놈 DNA-단백질 결합 반응
- 그 사이에 2 초 간격으로, 1 초 9 낮은 진폭의 펄스와 마이크로 팁을 사용하여 1.5 ML의 microfuge 튜브 (농도 100 ~ 200 NG / μL에서) 100 ㎕의 게놈 DNA를 초음파 처리하여 500 bp의 평균 크기 전단 게놈 DNA 각.
참고 : 튜브의 측면에 DNA를 뿌려 놓은 것, 모든 3 펄스 후 내용을 아래로 회전합니다. 실 사용 조건사용되는 초음파 분쇄기 악기와 다를 수 있습니다. 전단 된 DNA 단편 100-1000 bp의 범위 일 수 있지만, 보통 크기가 400-600 염기쌍 내에 있어야한다. 너무 많은 전단 적은 그대로 결합 부위에 발생할 수 있습니다, 너무 많이 또는 너무 작은 전단 하나가 하류 전체 게놈 증폭 단계에서 라이브러리 준비에 영향을 미칠 것입니다. - 10 mM 트리스 - 염산 pH를 7.5, 1 mM의 DTT, 50 mM의 KCl을, 5 mM의 MgCl2를, 25 % 글리세롤 및 50 mM의 아세틸 인산 RR 단백질 (DVU3023 0.5 pmol의)로 깎인 게놈 DNA의 2-3 μg 섞는다. 30 분 동안 25 ° C에서 반응을 품어. 입력 DNA로 1.5 ML 튜브 및 라벨에 대한 반응의 양도 10 μL.
참고 : 아세틸 인산이 체외 인산화 단백질을 활성화하기 위해 추가됩니다. 인산화 바인딩 DNA를 자극하지만, 많은 RR이 또한 활성화 7 않고 체외에서 DNA를 결합한다. 반응물에 첨가 단백질의 양은 단백질 프렙의 활성에 의존 할 것이다. EMSA이 선택하는 데 사용할 수 있습니다이 금액을 imize.
- 그 사이에 2 초 간격으로, 1 초 9 낮은 진폭의 펄스와 마이크로 팁을 사용하여 1.5 ML의 microfuge 튜브 (농도 100 ~ 200 NG / μL에서) 100 ㎕의 게놈 DNA를 초음파 처리하여 500 bp의 평균 크기 전단 게놈 DNA 각.
- 선호도는 단백질 바인딩 DNA를 정화
- 0.6 ML의 microfuge 튜브에 니켈 - NTA 아가로 오스 수지의 30 μl를 추가합니다. 하단의 수지를 수집하기 위해 1 분 100 XG에 원심 분리기 및 뜨는을 제거합니다. 100 세척 버퍼 (10 mM 트리스-HCl, pH7.5, 5 mM의 MgCl2를, 50 mM의 KCl을, 25 % 글리세롤)의 μL, 영화 튜브를 혼합하고, 2 분 100 XG에 원심 분리기를 추가합니다. 뜨는을 제거합니다.
- 세척 니켈 - NTA 수지에 결합 반응의 나머지 90 μl를 추가하고 30 분 동안 회전 진탕 배양한다.
- 2 분 100 XG에서 원심 분리하고, 상층 액 (언 바운드 DNA)를 제거합니다. , 세척 버퍼 100 μl를 추가 내용을 혼합 튜브를 가볍게, 2 분 100 XG에 원심 분리기. 뜨는을 제거합니다. 두 번 더 세척 단계를 반복합니다.
- 수지에 용출 버퍼의 35 μL (20 MM의 인산 나트륨 완충액 pH를 8, 500 mM의 NaCl을, 500 mM의 이미 다졸)를 추가하고 소용돌이로 교반하여 혼합한다. 5 분간 RT에서 벤치에 부화. 2 분 100 XG에 원심 분리기. 단백질 바인딩 된 DNA의 일부로서 새로운 1.5 ML 튜브 및 라벨에 뜨는을 전송합니다.
- 또한 입력 DNA에 용출 버퍼의 35 μl를 추가합니다.
- 입력 및 PCR 정제 키트를 사용하여 단백질 - 결합 DNA 분획을 정제 하였다.
- 전체 게놈은 입력 및 단백질 - 결합 DNA 샘플을 증폭한다.
- 입력 및 PCR 튜브를 분리하는 단백질 - 결합 DNA의 10 μl를 추가합니다. 배 분열 버퍼의 1 μL, 도서관 준비 버퍼 2 μL, 각각의 샘플 라이브러리 안정화 용액 1 μl를 추가합니다. 소용돌이로 교반하여 잘 섞는다. 2 분, 그리고 얼음에 진정을위한 95 ° C에서 열 순환기에 열.
주 : DNA의 시작 양이 증폭 바이어스를 도입하는 것을 방지하기 위해 적어도 10 NG이어야한다. - , 도서관 준비 효소 1 μl를 추가 피펫으로 혼합하고, 16 ° C/20 분에 열 자전거 타는 사람에 24 ° C/20 분, 20 분, 75 ° C / 5 분에게 37 ° C를 품어4 ° C에서 홀드
- 각 튜브에 물 47.5 μL, 10 배 증폭 마스터 믹스의 7.5 μL, 중합 효소의 5 μl를 추가합니다. 94 ° C/15 초 20주기, 65 ° C / 5 분 뒤에 95 ° C / 3 분,에 잘 열 섞는다. 4 ℃에서 반응을 잡고
- PCR 정제 키트를 사용하여 증폭 된 DNA 샘플을 정화하고 분광 DNA 농도를 측정한다.
- 입력 및 PCR 튜브를 분리하는 단백질 - 결합 DNA의 10 μl를 추가합니다. 배 분열 버퍼의 1 μL, 도서관 준비 버퍼 2 μL, 각각의 샘플 라이브러리 안정화 용액 1 μl를 추가합니다. 소용돌이로 교반하여 잘 섞는다. 2 분, 그리고 얼음에 진정을위한 95 ° C에서 열 순환기에 열.
- qPCR에를 사용하여 단백질 - 결합 DNA의 대상 농축 확인
- 디자인 qPCR에 프라이머 모든 자유롭게 사용할 수 프라이머 디자인 소프트웨어를 사용하여 EMSA 검증 표적 유전자 (DVU3025)의 상류 지역의 200 bp의 증폭합니다.
- 각각의 프라이머 세트 각 DNA 템플릿에 대한 세중의 qPCR 반응을 설정합니다. 각각의 프라이머 세트에 대한 마스터 믹스를 준비합니다. 1X 마스터 믹스 2 배 SYBR 녹색 qPCR에 믹스 10 μL, 18 μL의 총에 각각의 프라이머와 물을 0.5 μM이 포함되어 있습니다. 96 - 웰 PCR 플레이트의 웰 당 마스터 믹스의 분취 액 18 μL.
- Dilut전자 증폭 5 NG에 정제 입력 및 단백질 - 결합 DNA 샘플 / 물 μL와 DNA 템플릿으로 사용합니다. 우물에 DNA 템플릿 2 μl를 추가합니다.
- , 매우 명확한 qPCR의 밀봉 필름 플레이트를 밀봉 200 XG / 1 분에서 원심 분리기에 플레이트 스핀 다운. 실시간 qPCR의 기계에 판을 놓습니다. 사이클 관련 qPCR에 소프트웨어를 사용하여 다음과 같이 95 ° C / 1 분, 95 ° C/10 초, 40 회, 59 ° C/15 초, 70 ° C/35 초.
- 또한 관련이없는 유전자 (대조군)의 상류 지역을 증폭하는 프라이머와 세중의 qPCR 반응을 설정합니다.
- 입력 DNA의에서 단백질 - 결합 DNA의 C의 T 값을 빼서 ΔC T를 계산합니다. 2 ΔC T와 같은 단백질 바인딩 된 DNA의 표적 유전자의 배 농축을 계산합니다.
참고 : 대상 상류 영역이 입력 DNA 대 단백질 - 결합 DNA를 농축 한 경우는 결합 반응과 선호도 정화 SUCC 없음을 나타냅니다essful. 4 단계로 진행합니다. 대상 상류 지역의 농축이 관찰되지 않은 경우, 단백질의 서로 다른 양의 결합 반응 (3.1 단계)를 반복합니다.
4. DNA 라벨링 및 배열 하이브리드
- Cy5에와 Cy3에 충실 DNA와 라벨 입력 DNA
OTE :를 Cy3와 Cy5의 염료는 빛에 민감하고주의를 최소한으로 빛의 노출을 유지하기 위해주의해야한다.- 40 μL와 혼합 1 μg의 DNA는 9-MERS를 Cy3/Cy5-labeled 물 80 μL에 볼륨을 조절합니다.
- 열 (열 순환기에) 어둠 속에서 10 분 동안 98 ° C에서 변성. 2 분 동안 얼음에 빠른 진정.
- 잘 섞어, 각 반응에, 5 밀리미터의 dNTPs, 8 μL의 물, 그리고에 (어둠 속에서 2 시간 동안 37 ° C에서 알을 품다 - 2 μL 클레 나우 중합 효소 (50,000 U / ㎖ 3'-5'-엑소)를 추가 열 순환기).
- 0.5 M.에 대한 반응과 NaCl 용액을 중지하는 50 mM EDTA에 추가
- 1.5 ML 튜브 (계속)로 전송 샘플10 분 동안 12,000 XG에서 0.9 이소프로판올의 양, 10 분 동안 어둠 속에서 부화, 원심 분리기를 aining. 펠릿은 Cy5에 표지 된 DNA에 대한 Cy3로 표지 된 DNA와 블루 핑크해야한다.
- 2 분 동안 12,000 XG에서 80 % 에탄올 (500 μL) 및 원심 분리기와 펠렛을 씻으십시오. 어둠 속에서 5 ~ 10 분간 자연 건조 펠릿.
참고 : 펠렛은 -20 ℃에서 저장 될 수있다 - 25 μL의 물을 Resuspend 펠릿. 분광 광도계로 DNA 농도를 측정한다.
- 풀 함께 6 μg 어둠 속에서 낮은 열에서 원심 분리기에서 1.5 ML 튜브 진공 건조의를 Cy3 및 Cy5에 표지 DNA (투명 경우 원심 분리기 뚜껑을 포함)의 각.
참고 : 펠렛 하이브리드에 대한 준비가 될 때까지 -20 ° C에서 저장 될 수있다.
- 마이크로 어레이 혼성화
- C.를 사용하여 42 ℃로 온도를 설정하기 전에 3 ~ 4 시간에 하이브리드 시스템을 켭니다
- 1X 솔루션은 11.8 ㎕의 2 배 Hybridi이 포함되어 있는지 등 하이브리드 솔루션의 마스터 믹스를 준비합니다를 sync_len 버퍼, 4.7 ㎕의 하이브리드 구성 요소 A, 0.5 ㎕의 정렬 올리고.
- 5 μL 물에 알약을 재현 탁. 샘플이 믹스의 13 μl를 추가합니다. 15 초 동안 소용돌이, 5 분 동안 건조 욕조에 95 ℃에서 배양한다. 로딩을위한 준비가 될 때까지 하이브리드 시스템의 42 ° C에서 샘플을 보관하십시오.
- 제조 업체의 프로토콜에 따라, 마이크로 어레이 슬라이드 믹서 어셈블리를 준비합니다.
- 하이브리드 시스템 내에서 믹서 슬라이드 어셈블리를 놓습니다. 충전 포트에로드 샘플의 16 μl를, 접착 필름과 포트를 밀봉 시스템에 혼합 설정하고, 42 ℃에서 16 ~ 20 시간 동안 하이브리드
- 물에 10 배 상업적으로 이용 가능한 버퍼를 희석하여 1X 세척 버퍼 I (250 ㎖), II (50 ㎖), III (50 ㎖)를 준비하고 각각에 DTT가 1 ㎜에 추가합니다. 따뜻한 버퍼 I는 42 ° C.
- 따뜻한 버퍼 I. 벗기고 믹서를 포함하는 그릇 안에 분해 도구로 믹서 슬라이드, 장소를 밀어 적극적으로 쉬 동안손으로 분해 도구를 aking.
- 세척 버퍼 I 50 ㎖로 용기에 슬라이드를 삽입하고 손으로 2 분 동안 격렬하게 흔들어.
- 전송 50 ㎖ 세척 버퍼 II와 두 번째 용기에 슬라이드와 손으로 1 분 동안 격렬하게 흔들어.
- 전송 50 ㎖ 세척 버퍼 III와 세 번째 용기에 밀어 넣고 손으로 15 초 동안 격렬하게 흔들어.
- 빨리 종이 타월에 슬라이드의 가장자리를 얼룩 및 슬라이드 랙에 배치합니다. 슬라이드를 건조 2 분 200 XG에 원심 분리기. 슬라이드 케이스 내에서 호일로 포장하고, 데시 케이 터에 저장합니다.
- 배열 스캔
- 악기의 지시에 따라 마이크로 어레이 스캐너에서 슬라이드를 놓습니다.
- 532 나노 미터 = Cy3에 635 ㎚ 인 = Cy5에로 파장을 설정하는 스캐너의 소프트웨어를 사용하고, 350 및 400 사이의 최초의 광전자 증 배관의 이익.
- 슬라이드의 배열을 찾을 슬라이드를 미리 볼 수 있습니다. 스캔 어레이 영역을 선택합니다.
- 배열을 검색하고 배열 기능은 대부분 노란색이라는 광전자 증 배관의 설정 등의 조정. 히스토그램은 빨강과 녹색 겹쳐 곡선 또는 가능한 한 서로 근접을 표시해야합니다. 곡선은 포화 상태에 10-5 정규화 카운트 위에 종료해야합니다.
- 개별적으로 모두 532 및 635 nm의 이미지를 저장합니다.
- 어레이 데이터 분석
- 배열 분석 소프트웨어로 이미지를 가져옵니다. 소프트웨어를 사용하여, (를 Cy3와 Cy5에 이미지에 대한) 각 어레이에 두 개의 쌍 보고서 (. 쌍)을 만듭니다. 쌍 파일을 사용하여 스케일 로그 2 비율 파일을 만듭니다. 500 BP의 슬라이딩 창을 사용하여 피크를 검색 할 로그 2 비율 파일을 사용합니다. 유전자 표적의 상류 지역에 피크 궤적을지도.
- 긍정적 인 대상 (EMSA을 사용하여 결정하고이 예에서 qPCR에, DVU3025에 의해 확인)이 최고 피크 내에 표시되었는지 확인합니다.
주 : 양성 대상 베스트 히트 구비하지 않으면,이 가능한지 consi 아래 RR배급 제도 여러 유전자 표적을 가지며 칩 DAP 복제 수행 될 수 있으며, 모든 복제물에 공통 피크가 목표리스트를 생성하는데 사용될 수있다.
5. 결합 부위 주제 예측 및 검증
- DAP 칩 분석에 의해 발생함에 따라 유전자 표적을 위해 상류 지역 (400 BP)에 대한 시퀀스를 검색합니다. 모티브를 예측하기 위해 온라인 (meme.nbcr.net) 미생물을 통해 이러한 순서에 MEME을 적용합니다.
참고 사항 : sigma54 의존 규제로 증강 결합 단백질이 시작 사이트의 상류 수백 개의 염기쌍을 바인딩 할 수 있습니다. 다른 전사 조절기를 들어, 200 BP와 같은 짧은 지역은 상류에 충분합니다. - 디자인 상단과 양쪽 끝에서 10 기지에 의해 측면 예측 결합 부위 모티브를 포함하는 바닥 가닥 DNA 올리고머. 비오틴 상단 가닥 5 '를 주문하십시오.
- 10 mM 트리스 염산, 산도 8.0, 1 ㎜ EDTA, 50 mM의 염화나트륨의에서 1:1.5 비율의 상단과 하단 가닥 올리고 혼합PCR 튜브에 20 μl의 총 반응 부피. 열을 25 ℃로 천천히 냉각 다음에 열 자전거 타는 사람에서 5 분, 95 ° C에서 열 배 희석하고 EMSA에 대한 야생형 dsDNA 기판으로 사용합니다.
- 상기와 동일한 절차를 사용하여 수정 기판을 준비하지만, 설계 올리고머 결합 모티프의 보존 된 염기에서 4-6의 치환을 수행하는.
- RR 및 야생형 또는 수정 기판과 결합 반응을 설정하고 2 단계에서 설명 된대로 EMSA를 사용하여 검사합니다.
- 참고 : RR 야생 형 기판 있지만 수정을 결합하면 예상 모티브가 확인됩니다.
기타 관련 세균 종의 주제 6. 보존
- DVU3023의 orthologs를 포함하는 관심의 게놈을 선택합니다. NCBI 사이트에서 게놈에 대한 순서와 주석 파일을 얻습니다.
- POS를 구축하기 위해 DAP 칩 대상에서 모티브 시퀀스를 사용하는 스크립트를 작성하는 등의 Perl과 같은 프로그래밍 언어를 사용하여ition 가중 행렬과 다른 염기 서열을 유전체에 존재하는 유사한 주제를 점수 행렬을 사용합니다.
결과
상기 방법은 Desulfovibrio 불가리스 Hildenborough 7 박테리아를 감소 모델 황산에서의 RR 해외 유전자 표적을 결정하기 위해 적용 하였다. 이 유기체가 그것을 감지에 응답 가능한 신호의 다양한 나타내는 70의 RR로 표현 TCSS의 다수가 있습니다. 생체 이러한 경보 시스템의 기능을 분석하고 자신의 신호 때문에 수행하기 어렵고 따라서 자신의 활성화 조건입니다 알 수 있습니다. 여기...
토론
여기에 설명 된 칩 DAP 방법은 성공적 일이 대표적인 결과로 게재되어있는 Desulfovibrio 불가리스 Hildenborough 7 몇개의 RR 대한 유전자 표적을 결정하는 데 사용되었다. RR의 DVU3023 들어, 후보 유전자 표적을 선택하는 것은 수월했다. DVU3025은 RR 유전자의 바로 하류에 위치하고 있으며, RR 및 대상 유전자는 여러 Desulfovibrio 종에서 보존하고, 또한 DVU3025는 예측 sigma54 의존 발기인 있습?...
공개
저자는 공개 할 관심의 충돌이 없습니다.
감사의 말
우리는 비디오 촬영을 위해 준비하는 그녀의 도움과 기술을 보여 에이미 첸 감사합니다. 수수께끼에 의해 수행이 작품 : 유전자 및 분자 조립 (http://enigma.lbl.gov)와 생태계 및 네트워크 통합은, 로렌스 버클리 국립 연구소의 과학적인 초점 영역 프로그램은 과학, 생물의 사무실의 사무실에 의해 지원되었다 계약 번호 DE-AC02-05CH11231에서 미국 에너지 국의 환경 연구.
자료
| Name | Company | Catalog Number | Comments |
| Name of Material/Equipment | Company | Catalog number | Comments |
| HisTrapFF column (Ni-Sepharose column) | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5255-01 | |
| Akta explorer (FPLC instrument) | GE Lfe Sciences, Pittsburgh, PA, USA | ||
| HiPrep 26/10 Desalting column | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5087-01 | |
| Qiaquick Gel extraction kit | Qiagen Inc, Valencia, CA, USA | 28704 | |
| Biotin-labeled oligonucleotides | Integrated DNA Technologies | N/A | |
| 6% polyacrylamide-0.5X TBE precast mini DNA retardation gel | Life Technologies, Grand Island, NY, USA | EC63652BOX | Alternately, you can pour your own gel. |
| Nylon membrane | EMD Millipore, Billerica, MA, USA | INYC00010 | |
| Trans-Blot SD Semi-dry electrophoretic transfer cell | Biorad, Hercules, CA, USA | 170-3940 | |
| Extra thick blot paper, 8 x 13.5 cm | Biorad, Hercules, CA, USA | 170-3967 | |
| UV crosslinker Model XL-1000 | Fisher Scientific | 11-992-89 | |
| Nucleic Acid chemiluminescent detection kit (Pierce) | Thermo fisher Scientific, Rockford, IL, USA | 89880 | |
| Ni-NTA agarose resin | Qiagen Inc, Valencia, CA, USA | 30210 | |
| GenomePlex Whole genome amplification kit (Fragmentation buffer, library preparation buffer, library stabilization solution, library preparation enzyme, 10X amplification master mix, WGA polymerase ) | Sigma-Aldrich, St. Louis, MO, USA | WGA2-50RXN | |
| Nanodrop ND-1000 | Thermo Scientific, Wilmington, DE, USA | For quantitation of DNA | |
| Perfecta Sybr Green SuperMix, with ROX | Quanta biosciences | 95055-500 | Any Sybr Green PCR mix may be used |
| PlateMax Ultra clear heat sealing film for qPCR | Axygen | ||
| 96 well clear low profile PCR microplate | Life Technologies, Grand Island, NY, USA | PCR-96-LP-AB-C | |
| Applied Biosystems StepOne Plus Real time PCR system | Life Technologies, Grand Island, NY, USA | 4376600 | Any real time PCR system may be used |
| Qiaquick PCR purification kit | Qiagen Inc, Valencia, CA, USA | 28104 | Any PCR clean up kit may be used |
| Cy3/Cy5-labeled nonamers | Trilink biotechnologies, San Diego, CA, USA | N46-0001, N46-0002 | |
| Klenow polymerase 50,000U/ml, 3'-5' exo- | New England Biolabs, Ipswich, MA | M0212M | |
| Hybridization system | Roche-Nimblegen, Madison, WI, USA | N/A | This company no longer makes arrays or related items, so alternate sources such as Agilent or Affymetrix will need to be used, |
| Custom printed microarrays and mixers | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Hybridization kit (2X Hybridization buffer, Hybridization component A, Alignment oligo) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Wash buffer kit (10X Wash buffer I, II, III, 1 M DTT) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| GenePix 4200A microarray scanner | Molecular Devices, Sunnyvale CA, USA | This model has been replaced by superior ones | |
| GenePix Pro microarray software | Molecular Devices, Sunnyvale CA, USA | ||
| Nimblescan v.2.4, ChIP-chip analysis software | Roche-Nimblegen, Madison, WI, USA | N/A | |
참고문헌
- Galperin, M. Y. Diversity of structure and function of response regulator output domains. Curr Opin Microbiol. 13, 150-159 (2010).
- Casino, P., Rubio, V., Marina, A. The mechanism of signal transduction by two-component systems. Curr Opin Struct Biol. 20, 763-771 (2010).
- Aparicio, O., Geisberg, J. V., Struhl, K. Chromatin immunoprecipitation for determining the association of proteins with specific genomic sequences in vivo. Curr Protoc Cell Biol. 17 (17), (2004).
- McCleary, W. R., Stock, J. B. Acetyl phosphate and the activation of two-component response regulators. J Biol Chem. 269, 31567-31572 (1994).
- Novichkov, P. S., et al. RegPrecise web services interface: programmatic access to the transcriptional regulatory interactions in bacteria reconstructed by comparative genomics. Nucleic Acids Res. 40, 604-608 (2012).
- Studholme, D. J., Buck, M., Nixon, T. Identification of potential sigma(N)-dependent promoters in bacterial genomes. Microbiology. 146 (12), 3021-3023 (2000).
- Rajeev, L., et al. Systematic mapping of two component response regulators to gene targets in a model sulfate reducing bacterium. Genome Biol. 12, 99 (2011).
- Crooks, G. E., Hon, G., Chandonia, J. M., Brenner, S. E. WebLogo: a sequence logo generator. Genome Res. 14, 1188-1190 (2004).
- Barbieri, C. M., Mack, T. R., Robinson, V. L., Miller, M. T., Stock, A. M. Regulation of response regulator autophosphorylation through interdomain contacts. J Biol Chem. 285, 32325-32335 (2010).
- Bourret, R. B. Receiver domain structure and function in response regulator proteins. Curr Opin Microbiol. 13, 142-149 (2010).
- Gao, R., Stock, A. M. Molecular strategies for phosphorylation-mediated regulation of response regulator activity. Curr Opin Microbiol. 13, 160-167 (2010).
- Hung, D. C., et al. Oligomerization of the response regulator ComE from Streptococcus mutans is affected by phosphorylation. J Bacteriol. 194, 1127-1135 (2012).
- Ladds, J. C., et al. The response regulator Spo0A from Bacillus subtilis is efficiently phosphorylated in Escherichia coli. FEMS Microbiol Lett. 223, 153-157 (2003).
- Wen, Y., Feng, J., Scott, D. R., Marcus, E. A., Sachs, G. Involvement of the HP0165-HP0166 two-component system in expression of some acidic-pH-upregulated genes of Helicobacter pylori. J Bacteriol. 188, 1750-1761 (2006).
- Liu, X., Noll, D. M., Lieb, J. D., Clarke, N. D. DIP-chip: rapid and accurate determination of DNA-binding specificity. Genome Res. 15, 421-427 (2005).
- Gossett, A. J., Lieb, J. D. DNA Immunoprecipitation (DIP) for the Determination of DNA-Binding Specificity. CSH Protoc. 2008, (2008).
- Mascher, T., et al. The Streptococcus pneumoniae cia regulon: CiaR target sites and transcription profile analysis. J Bacteriol. 185, 60-70 (2003).
- Park, P. J. ChIP-seq: advantages and challenges of a maturing technology. Nat Rev Genet. 10, 669-680 (2009).
- Lefrancois, P., Zheng, W., Snyder, M. ChIP-Seq using high-throughput DNA sequencing for genome-wide identification of transcription factor binding sites. Methods Enzymol. 470, 77-104 (2010).
- Ho, J. W., et al. ChIP-chip versus ChIP-seq: lessons for experimental design and data analysis. BMC Genomics. 12, 134 (2011).
- Liu, X., Lee, C. K., Granek, J. A., Clarke, N. D., Lieb, J. D. Whole-genome comparison of Leu3 binding in vitro and in vivo reveals the importance of nucleosome occupancy in target site selection. Genome Res. 16, 1517-1528 (2006).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유