È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
DNA purificato per affinità Chip (DAP-chip) metodo per determinare obiettivi del gene per batterica, due sistemi di regolamentazione dei componenti
In questo articolo
Riepilogo
In questo articolo video descrive un metodo microarray basato in vitro per determinare gli obiettivi del gene e siti di legame per due regolatori di risposta del sistema dei componenti.
Abstract
Nei metodi in vivo come ChIP-chip sono tecniche comunemente utilizzate per determinare obiettivi del gene globali per i fattori di trascrizione. Tuttavia, essi sono di uso limitato a esplorare batteriche due sistemi di regolazione dei componenti con condizioni di attivazione non caratterizzate. Tali sistemi regolano la trascrizione solo quando attivato in presenza di segnali unici. Poiché questi segnali sono spesso sconosciuti, il metodo vitro microarray basato in descritto in questo articolo video può essere utilizzata per determinare obiettivi del gene e siti di legame per i regolatori di risposta. Questo metodo-chip DNA purificato per affinità può essere utilizzato per qualsiasi regolatore purificata in qualsiasi organismo con un genoma sequenziato. Il protocollo prevede che permette la proteina taggato purificata da associare alla tosato DNA genomico e poi affinità purificazione del DNA alle proteine, seguita dalla marcatura fluorescente del DNA e ibridazione di una matrice di affiancamento personalizzato. Precedente passaggi che possono essere utilizzati per ottimizzare il dosaggio per specifiregolatori c sono anche descritti. I picchi generati dall'analisi dei dati della matrice vengono utilizzati per prevedere motivi sito di legame, che vengono poi validate sperimentalmente. Le previsioni motivi possono essere ulteriormente utilizzati per determinare obiettivi del gene di regolatori di risposta ortologhi in specie strettamente correlate. Dimostriamo l'applicabilità di questo metodo determinando gli obiettivi gene e vincolante motivi sito e prevedere quindi la funzione di un regolatore di risposta DVU3023 sigma54-dipendente nel batterio ambientale Desulfovibrio vulgaris Hildenborough.
Introduzione
La capacità dei batteri di sopravvivere e prosperare è criticamente dipendente da come sono in grado di percepire e rispondere alle perturbazioni nel loro ambiente, e questo a sua volta dipende dalla loro sistemi di trasduzione del segnale. Il numero dei sistemi di segnalamento un batterio codifica è stato chiamato il suo "IQ microbica" e può essere un'indicazione di entrambi variabilità del suo ambiente e della sua capacità di percepire segnali multipli e mettere a punto la sua risposta 1. Due sistemi componenti di trasduzione del segnale (TCS) sono i sistemi di segnalazione più diffusi utilizzati dai batteri, e sono costituiti da una istidina chinasi (HK) che rileva il segnale esterno e trasmette tramite fosforilazione di un regolatore risposta effettrice (RR) 2. RR può avere una varietà di domini di uscita e quindi diverse modalità effettrici, ma la risposta più comune è la regolazione trascrizionale tramite un dominio di legame al DNA 1. I segnali rilevati e le corrispondenti funzioni dei vast maggioranza dei STC rimangono sconosciute.
Sebbene i metodi in vivo come ChIP-chip sono abitualmente utilizzati per la determinazione dei siti di legame di fattori di trascrizione genomica 3, possono essere utilizzati solo per batteriche RR sistema a due componenti se sono note le condizioni attivanti o segnali. Spesso i segnali ambientali che attivano una TCS sono più difficili da determinare rispetto ai loro obiettivi del gene. Il microarray in vitro saggio basato qui descritto può essere utilizzato per determinare efficacemente e rapidamente i geni bersaglio e prevedere funzioni di STC. Questo saggio sfrutta il fatto che RR possono essere fosforilati e quindi attivate in vitro utilizzando piccoli donatori molecola come acetil fosfato 4.
In questo metodo, denominato DAP-chip per DNA-affinità-chip purificata (Figura 1), il gene RR di interesse viene clonato con His-tag in E. coli, e una proteina marcata successivamente purificata è consentito bind a tranciata DNA genomico. Il DNA alle proteine viene poi arricchita da affinità di purificazione, il DNA arricchito e l'ingresso sono amplificati, fluorescente, in pool insieme e ibridato in una matrice di piastrelle che è su misura per l'organismo di interesse (Figura 1). Esperimenti microarray sono soggette ad artefatti e quindi passaggi aggiuntivi sono impiegati per ottimizzare il dosaggio. Un passo è tentare di determinare un obiettivo per il RR sotto studio mediante saggi di spostamento della mobilità elettroforetica (EMSA) (vedi workflow in figura 2). Poi, dopo il legame al DNA genomico e le fasi DAP, il DNA legato alle proteine e di ingresso vengono esaminate da qPCR per vedere se il bersaglio positivo è arricchito nella frazione legata alle proteine rispetto alla frazione di ingresso, confermando così condizioni ottimali di legame per l' RR (Figura 2). Dopo matrice ibridazione, i dati vengono analizzati per individuare picchi di segnale di maggiore intensità che indicano loci genomici dove la proteina had vincolato. Le funzioni possono essere previsti per l'RR base agli obiettivi genica ottenuti. I loci genomici destinazione vengono utilizzati per prevedere motivi sito di legame, che vengono poi sperimentalmente validati utilizzando EMSAs (Figura 2). Le previsioni funzionali e obiettivi del gene per la RR possono poi essere estesi alle specie che codificano RR ortologhe mediante la scansione di questi genomi per motivi di legame simili (Figura 2), strettamente legato. Il metodo DAP-chip in grado di fornire una serie di informazioni per una TCS dove prima non c'era nessuno. Il metodo può anche essere usato per qualsiasi regolatore trascrizionale se la proteina può essere purificata e DNA condizioni di legame può essere determinata, e per qualsiasi organismo di interesse con una sequenza genomica disponibili.
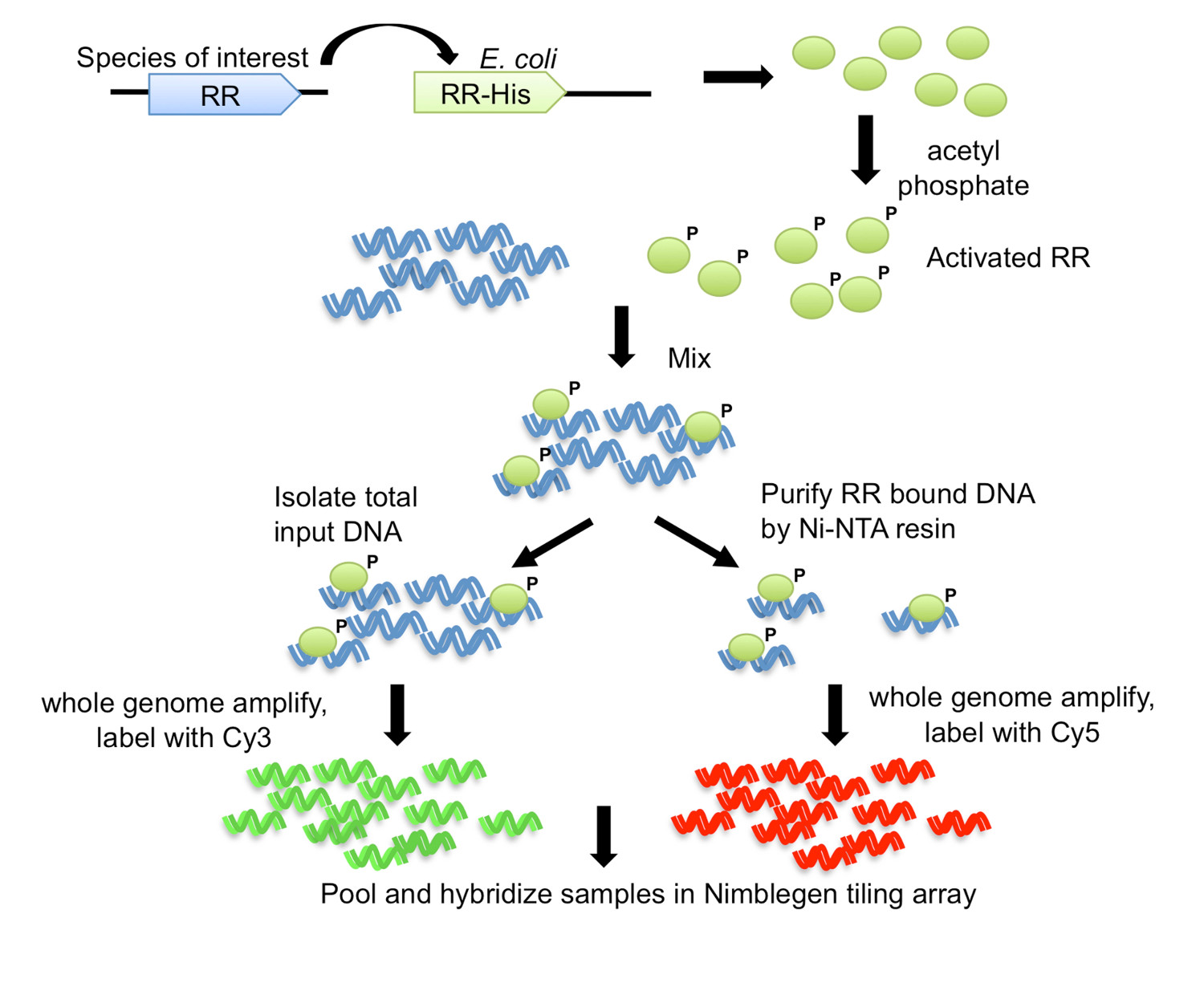
Figura 1. Il chip DNA purificato per affinità (DAP-chip) strategia 7. Il gene RR dall'organismo di interesse viene clonato con carbossi-terminale His-tag in un E. ceppo di espressione coli. Purificato la proteina His-tagged viene attivata dalla fosforilazione con acetil fosfato, e miscelato con tranciato DNA genomico. Un'aliquota della reazione di legame viene salvato come DNA di ingresso, mentre il resto è sottoposto a purificazione per affinità utilizzando resina Ni-NTA. L'ingresso e il DNA RR-bound sono intero genoma amplificato, ed etichettati con Cy3 e Cy5, rispettivamente. Il DNA marcato viene raggruppate e ibridato a una matrice piastrelle, che viene poi analizzato per determinare gli obiettivi del gene. Figura modificato e ristampato utilizzando la licenza Creative Commons da 7.
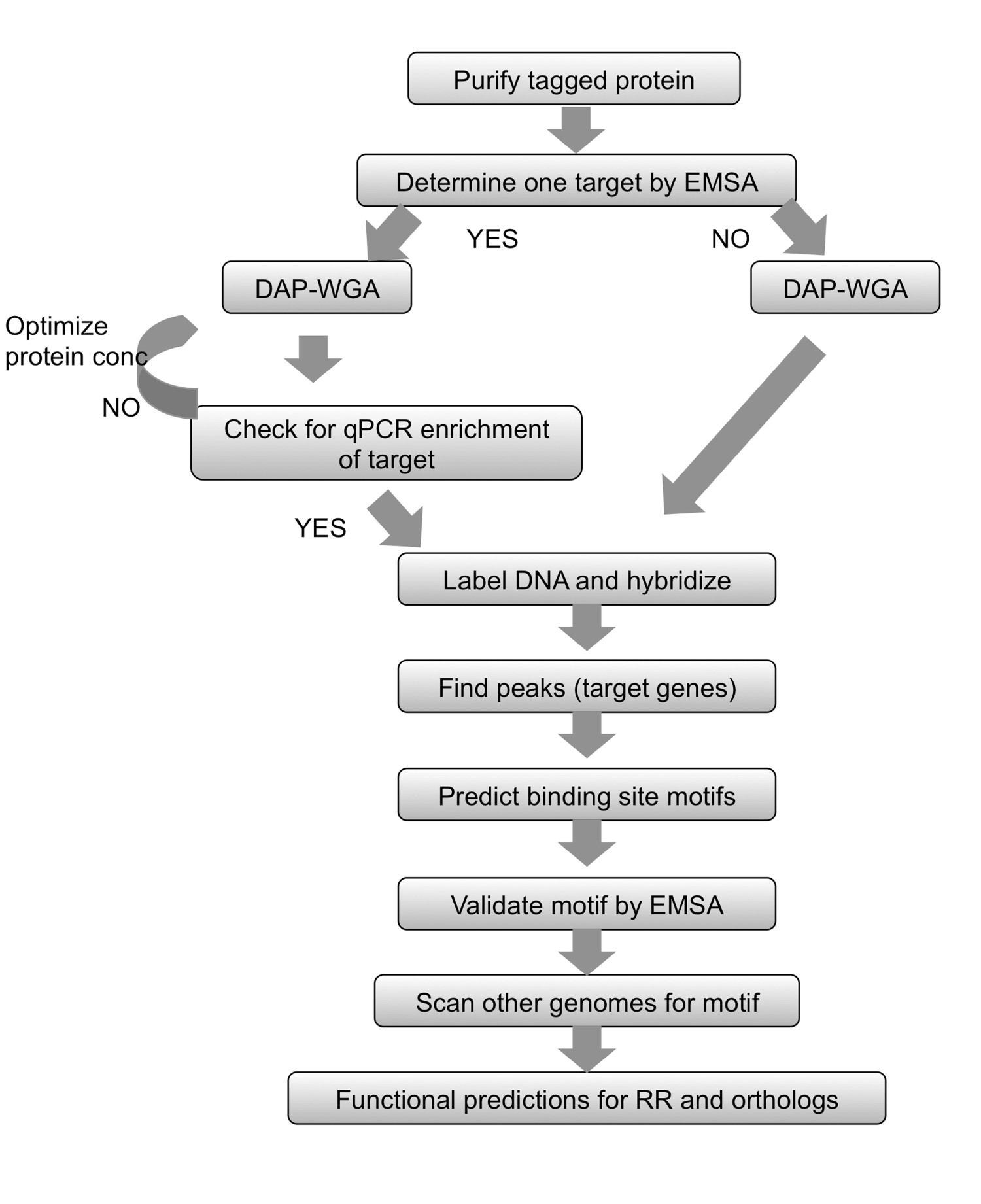
Figura 2. Sintesi dei flussi di lavoro. Per qualsiasi Protei taggato purificaton, iniziare determinando un obiettivo utilizzando EMSA. Lasciare proteina di legare il DNA genomico e poi DNA-affinità-Purify (DAP) e l'intero genoma di amplificare (WGA) e il DNA ingresso arricchito. Se un gene bersaglio è noto, utilizzare qPCR per garantire che il bersaglio noto è arricchito nella frazione legata alle proteine. Se nessun obiettivo potrebbe essere determinato, procedere direttamente alla etichettatura del DNA e la matrice di ibridazione. Se non poteva essere osservato arricchimento da qPCR, quindi ripetere il legame proteina-gDNA e gradini DAP-WGA utilizzando diverse quantità di proteine. Utilizzare l'analisi di matrice per individuare e mappare i picchi loro di geni bersaglio. Utilizzare le regioni a monte dei geni bersaglio per prevedere motivi sito di legame. Convalidare i motivi sperimentalmente utilizzando EMSAs. Utilizzare il motivo per eseguire la scansione del genoma di specie affini che codificano ortologhi del RR in fase di studio, e prevedere i geni mirati in quelle specie. Sulla base degli obiettivi del gene ottenuti, la funzione fisiologica della RR e suoi ortologhi può essere previsto. Figura modificato e ristampato con il c creativaommons licenza da 7.
Protocollo
Nota: Il protocollo seguito è su misura per la determinazione degli obiettivi del gene della DVU3023 RR dal batterio Desulfovibrio vulgaris Hildenborough. Può essere adattata a qualsiasi altro regolatore trascrizionale di interesse.
1. Clone e Purify RR
- Clonare il gene RR, in particolare DVU3023, da D. vulgaris Hildenborough in un Escherichia coli vettore di espressione tale che il gene è C-terminale His-tag e di espressione è sotto il controllo di un promotore T7.
Nota: Diversi metodi di clonazione possono essere utilizzati e sono determinati dal ricercatore. Si possono utilizzare anche i tag di affinità alternativi. - Trasforma l'espressione costrutto in E. coli BL21 (DE3) ceppo espressione.
- Crescere 1 L di ceppo espressione BL21 a 37 ° C. Alla fase mid-log, aggiungere IPTG 0,5 mM per indurre l'espressione della proteina. Continua crescita a temperatura ambiente per 24 ore.
- Centrifugare le cellule a 5.000 xg per 10 kmn a 4 ° C. Risospendere le cellule in tampone di lisi (20 mM sodio fosfato, pH 7,4, NaCl 500 mM, imidazolo 40 mM, 1 mg / ml lisozima, 1x benzonasi nucleasi). Cellule Lisare utilizzando una pressa francese a 4 ° C.
- Centrifugare lisato a 15.000 xg per 30 min a 4 ° C. Filtrare con un filtro siringa da 0,45 micron.
- Lavare la colonna 1 ml Ni Sepharose su uno strumento FPLC utilizzando 10 ml di tampone di lavaggio (fosfato di sodio 20 mM, pH 7,4, NaCl 500 mM, imidazolo 40 mM).
- Caricare lisato sulla colonna. Lavare colonna con 20 ml di tampone di lavaggio.
- Eluire DVU3023 utilizzando un gradiente di 0-100% tampone di eluizione (20 mM fosfato di sodio, pH 7,4, NaCl 500 mM, imidazolo 500 mM).
- Caricare frazioni eluite su una colonna desalificazione, e lavare con un tampone di dissalazione (20 mM sodio fosfato pH 7,4, 100 mM NaCl).
- Centrifugare campione di proteine in un peso di taglio del filtro alta centrifuga molecolare di concentrare la proteina. Aggiungere DTT 0,1 mM e glicerolo al 50% e proteine conservare a -20 ° C.
Nota: I metodi di purificazione dovranno essere ottimizzati singolarmente per ogni proteina studiata.
2. Determinare gene target per RR Uso Electrophoretic Mobility Maiusc Assay (EMSA)
- PCR amplifica 400 bp regione a monte del DVU3025 candidato gene bersaglio, utilizzando D. vulgaris Hildenborough DNA genomico come modello, e un primer marcato in avanti e una retromarcia 5'-biotina marcata con primer.
Nota: Consigli per scegliere un candidato gene bersaglio: Spesso RR legano le regioni a monte del proprio gene / operone, oppure possono regolare i geni prossimale codificati. Se la RR ha ortologhi in altre specie, per cercare i geni che sono conservati nel quartiere. Metodi alternativi comprendono la scelta di geni candidati sulla base di previsioni regulone (ad esempio, RegPrecise 5), o previsione promotori sigma54-dipendente per 6 RR che sono essi stessi sigma54-dipendente. - Eseguire il prodotto di PCR su un gel di agarosio 1%, tagliare il 400 bprodotto di dimensioni p, e purificare il DNA utilizzando un gel di estrazione ripulire kit.
- Mescolare proteina DVU3023 (0,5 pmol) con 100 fmol di substrato DNA biotinilato in 10 mM Tris HCl, pH 7.5, 50 mM KCl, 5 mM MgCl 2, 1 mM DTT, 25% glicerolo e 1 mg / ml poly dI.dC (non -specifico concorrente DNA) in un volume totale di 20 microlitri. Anche impostare una reazione senza alcuna proteina come controllo. Incubare le reazioni a temperatura in panchina ambiente per 20 min.
- Nota: Queste sono condizioni standard elencati e gli altri componenti di reazione possono essere aggiunti a seconda della RR di interesse. Se l'attivazione è desiderato, aggiungere 50 mM fosfato acetil alla reazione (spesso RR legheranno DNA in vitro senza attivazione).
- Pre-eseguire un prefabbricato mini 6% poliacrilammide-TBE 0.5x gel in tampone TBE 0,5 x a 100 V per 30 min. Aggiungere 5 ml di tampone 5x carico (0,1% blu di bromofenolo, 0,1% xilene cyanol, 30% glicerolo in 1x TBE) per le reazioni di legame e di carico di 18 ml di reazioni accede al gel. Eseguire a 100 V per 2 ore.
Nota: Per evitare il surriscaldamento, che può portare alla dissociazione dei complessi proteina-DNA, riempire la camera di transito esterno nel sistema di elettroforesi con tampone di corsa tale che la maggior parte del gel è isolato dal tampone. In alternativa, il gel può essere eseguito a 4 ° C. - Tagliare una membrana di nylon caricata alla dimensione del gel e immergerlo in 0,5 x TBE per almeno dieci minuti. Rimuovere il gel dalla cassetta. Tagliare tutte le creste il gel e panino il gel e la membrana tra due filtri di carta spessa imbevuti di 0,5 x TBE, e posto all'interno di un apparato di blotting semi-dry e correre a 20 V per 30 min.
- Posizionare la membrana all'interno di uno strumento di luce UV reticolante commerciali e impostare il tempo di 3 min.
Nota: La membrana può ora essere conservato asciutto a temperatura ambiente per alcuni giorni prima di procedere alla fase successiva. - Utilizzare un kit di rivelazione chemiluminescente disponibile in commercio che impiega una streptavidina-rafano perossidazionese coniugato a sviluppare il blot secondo le istruzioni del produttore.
- Immagine blot utilizzando un computer collegato a una telecamera CCD attrezzato e cercare un cambiamento nella mobilità substrato DNA in presenza del RR, che indica che il RR lega il DNA in esame.
Nota: La specificità del turno proteina-DNA può essere testato includendo nella reazione un eccesso di DNA non marcato concorrente, che dovrebbe eliminare o ridurre lo spostamento visto.
3. Verificare arricchimento target dopo la DNA-proteina Genomic Binding
- Genomic DNA-proteina reazione di legame
- Shear DNA genomico ad una dimensione media di 500 bp da sonicating 100 microlitri di DNA genomico (a concentrazioni 100-200 ng / mL) in una provetta da 1,5 ml microcentrifuga utilizzando un micropunte con 9 bassi impulsi con ampiezza di 1 sec, con 2 sec divario tra ciascuno.
Nota: Se gli schizzi di DNA ai lati del tubo, rallentano il contenuto dopo ogni 3 impulsi. Le condizioni esatte usatevarierà con lo strumento utilizzato sonicatore. I frammenti di DNA tranciati possono variano da 100-1000 bp, ma la dimensione media dovrebbero essere all'interno di 400-600 bp. Troppo taglio può comportare un minor numero di siti di legame intatti, e troppo o troppo poco taglio interesserà preparazione biblioteca durante l'intera procedura di amplificazione del genoma a valle. - Mescolare 2-3 ug di DNA genomico tranciata con proteine RR (0,5 pmol di DVU3023) in 10 mM Tris-HCl pH 7,5, 1 mM DTT, 50 mM KCl, 5 mM MgCl 2, 25% glicerolo, 50 mM e acetil fosfato. Incubare le reazioni a 25 ° C per 30 min. Trasferire 10 ml di questa reazione in una provetta da 1,5 ml ed etichetta come DNA ingresso.
Nota: acetil fosfato viene aggiunto per attivare la proteina mediante fosforilazione in vitro. La fosforilazione stimola legame al DNA, ma molti RR legano anche il DNA in vitro senza attivazione 7. La quantità di proteina aggiunto alla reazione dipenderà attività della proteina prep. L'EMSA può essere utilizzato per scegliereimize questo importo.
- Shear DNA genomico ad una dimensione media di 500 bp da sonicating 100 microlitri di DNA genomico (a concentrazioni 100-200 ng / mL) in una provetta da 1,5 ml microcentrifuga utilizzando un micropunte con 9 bassi impulsi con ampiezza di 1 sec, con 2 sec divario tra ciascuno.
- Affinity purificare il DNA legata alle proteine
- Aggiungere 30 ml di resina Ni-NTA agarosio in una provetta da microcentrifuga 0,6 ml. Centrifugare a 100 xg per 1 minuto per raccogliere la resina nella parte inferiore, e rimuovere il surnatante. Aggiungere 100 microlitri di tampone di lavaggio (10 mM Tris-HCl, pH 7,5, 5 mM MgCl2, 50 mM KCl, 25% glicerolo), flick provetta per miscelare e centrifugare a 100 xg per 2 minuti. Rimuovere il surnatante.
- Aggiungere i restanti 90 microlitri della reazione di legame alla resina lavata Ni-NTA e incubare in un agitatore rotante per 30 min.
- Centrifugare a 100 xg per 2 minuti e rimuovere il surnatante (DNA non legato). Aggiungere 100 microlitri di tampone di lavaggio, sfiora il tubo per miscelare il contenuto, e centrifugare a 100 xg per 2 minuti. Rimuovere il surnatante. Ripetere la fase di lavaggio per altre due volte.
- Aggiungere 35 ml di tampone di eluizione (tampone fosfato di sodio 20 mM pH 8, NaCl 500 mM, imidazolo 500 mm) per la resina e mescolare nel vortex. Incubare in panchina a temperatura ambiente per 5 min. Centrifugare a 100 xg per 2 minuti. Trasferire il surnatante in una nuova provetta da 1,5 ml ed etichetta come frazione del DNA legata alle proteine.
- Anche aggiungere 35 ml di tampone di eluizione al DNA ingresso.
- Purificare l'ingresso e le frazioni di DNA legati alle proteine utilizzando un kit di purificazione PCR.
- Intero genoma amplificare l'input e campioni di DNA legati alle proteine.
- Aggiungere 10 ml di ingresso e legata alle proteine DNA per separare provette PCR. Aggiungere 1 ml di 10x tampone di frammentazione, 2 ml di tampone Biblioteca Preparazione e 1 ml di soluzione Biblioteca di stabilizzazione per ogni campione. Mescolare bene nel vortex. Calore in un termociclatore a 95 ° C per 2 min, e freddo su ghiaccio.
Nota: La quantità di DNA di partenza dovrebbe essere di almeno 10 ng per evitare di introdurre distorsioni amplificazione. - Aggiungere 1 ml di Biblioteca preparato enzimatico, mescolare pipettando, e incubare in termociclatore a 16 ° C/20 min, 24 ° C/20 min, 37 ° C per 20 min, 75 ° C / 5 min, E tenuta a 4 ° C.
- Per ogni provetta, aggiungere 47,5 ml di acqua, 7,5 ml di 10x amplificazione master mix, e 5 ml di polimerasi. Mescolare bene e scaldare a 95 ° C / 3 min, seguiti da 20 cicli di 94 ° C/15 sec, 65 ° C / 5 min. Tenere reazioni a 4 ° C.
- Purificare i campioni di DNA amplificati usando un kit di purificazione PCR e misurare la concentrazione di DNA spettrofotometricamente.
- Aggiungere 10 ml di ingresso e legata alle proteine DNA per separare provette PCR. Aggiungere 1 ml di 10x tampone di frammentazione, 2 ml di tampone Biblioteca Preparazione e 1 ml di soluzione Biblioteca di stabilizzazione per ogni campione. Mescolare bene nel vortex. Calore in un termociclatore a 95 ° C per 2 min, e freddo su ghiaccio.
- Verificare arricchimento target nel DNA legata alle proteine mediante qPCR
- Primer design qPCR per amplificare 200 bp della regione a monte del gene target EMSA-verificato (DVU3025) utilizzando qualsiasi software di progettazione di primer liberamente disponibile.
- Impostare reazioni qPCR triplicato per ogni modello di DNA con ogni set di primer. Preparare una master mix per ogni set di primer. 1x master mix contenente 10 ml di miscela 2x SYBR Green qPCR, 0,5 mM di ciascun primer e acqua per un totale di 18 microlitri. Aliquota 18 microlitri della master mix per pozzetto di una piastra PCR da 96 pozzetti.
- Dilute il amplificati e ingresso purificata e campioni di DNA legati alle proteine di 5 ng / mL con acqua e utilizzare come modello il DNA. Aggiungere 2 ml di DNA templato ai pozzetti.
- Sigillare la piastra con un ultra trasparente pellicola di tenuta qPCR, spin down piastra in una centrifuga a 200 xg / 1 min. Posizionare la piastra in una macchina qPCR in tempo reale. Ciclo come segue utilizzando il software associato qPCR: 95 ° C / 1 min, e 40 cicli di 95 ° C/10 sec, 59 ° C/15 sec, e 70 ° C/35 sec.
- Anche istituito reazioni qPCR triplicato con primer per amplificare regioni a monte di un gene estraneo (controllo negativo).
- Calcola ΔC T sottraendo C valore T di legame proteico DNA da quello del DNA ingresso. Calcola arricchimento piega di gene bersaglio nel DNA legata alle proteine come 2 ΔC T.
Nota: Se la regione a monte obiettivo è stato arricchito nel legata alle proteine DNA rispetto al DNA ingresso, indica che le reazioni di legame e purificazione per affinità erano successful. Passare al punto 4. Se non è stato osservato un arricchimento della regione a monte di destinazione, ripetere le reazioni di legame (passo 3,1) con diverse quantità di proteine.
4. Etichettatura DNA e ibridazione Array
- Etichetta DNA di ingresso con il DNA Cy3 e arricchito con Cy5
ota: Cy3 e Cy5 coloranti sono sensibili alla luce e la cura devono essere prese per mantenere l'esposizione della luce al minimo.- Mescolare 1 mg DNA con 40 microlitri Cy3/Cy5-labeled 9-mers e regolare il volume a 80 ml con acqua.
- Denaturare calore a 98 ° C per 10 minuti al buio (in un termociclatore). Freddo rapido in ghiaccio per 2 min.
- Aggiungere 2 microlitri Klenow polimerasi (3'-5'-exo -, 50.000 U / ml), 5 mM dNTPs, e 8 microlitri di acqua a ciascuna reazione, mescolare bene, e incubare a 37 ° C per 2 ore al buio (in un termociclatore).
- Aggiungere 50 mM EDTA per bloccare la reazione e la soluzione di NaCl allo 0,5 M.
- Campioni Trasferire 1,5 ml provette containing 0,9 volume di isopropanolo, incubare al buio per 10 minuti, e centrifugare a 12.000 xg per 10 min. Il pellet deve essere rosa per il DNA Cy3 marcato e blu per il DNA Cy5 marcato.
- Lavare pellet con etanolo 80% (500 microlitri) e centrifugare a 12.000 xg per 2 min. Pellets all'aria per 5-10 minuti nel buio.
Nota: Le pastiglie possono essere conservati a -20 ° C. - Risospendere pellet in 25 microlitri di acqua. Misurare la concentrazione di DNA spettrofotometricamente.
- Pool insieme 6 mg ciascuno di DNA-Cy3 e Cy5 marcato in una provetta da 1,5 ml e vuoto a secco in una centrifuga a fuoco al buio (coprire il coperchio della centrifuga se è trasparente).
Nota: Le pastiglie possono essere conservati a -20 ° C fino al momento ibridazione.
- Microarray Ibridazione
- Accendere il sistema di ibridazione in 3-4 ore prima di usare e impostare la temperatura a 42 ° C.
- Preparare una soluzione di ibridazione master mix tale soluzione contiene 11,8 microlitri 1x 2x Hybriditampone zione, 4,7 microlitri Ibridazione componente A, e 0,5 microlitri allineamento oligo.
- Risospendere il pellet in 5 ml di acqua. Aggiungere 13 ml di questa miscela al campione. Vortex per 15 sec, incubare a 95 ° C in un bagno a secco per 5 min. I campioni a 42 ° C nel sistema ibridazione fino pronto per il caricamento.
- Preparare il microarray gruppo otturatore, mixer, secondo il protocollo del produttore.
- Posizionare il gruppo miscelatore slitta all'interno del sistema di ibridazione. Carico 16 ml di campione nella porta di riempimento, sigillare le porte con un film adesivo, mescolando a girare nel sistema, e ibridare per 16-20 ore a 42 ° C.
- Preparare 1x I tamponi di lavaggio (250 ml), II (50 ml) e III (50 ml) diluendo i 10x buffer disponibili commercialmente in acqua, e ad ogni aggiungi DTT 1 mM. Buffer caldo I a 42 ° C.
- Far scorrere il mixer-slide in uno strumento di smontaggio, e posto all'interno di un recipiente contenente calda Buffer I. Sbucciare la mixer off, mentre vigorosamente shaking lo strumento di smontaggio a mano.
- Mettere il vetrino in un contenitore con 50 ml di tampone di lavaggio I e agitare vigorosamente per 2 minuti a mano.
- Trasferimento di scivolare in un secondo contenitore con tampone di 50 ml di lavaggio II e agitare vigorosamente per 1 min a mano.
- Trasferimento diapositiva in un terzo contenitore con 50 ml di Tampone di lavaggio III, e agitare energicamente per 15 secondi a mano.
- Asciugare rapidamente i bordi del vetrino su un tovagliolo di carta, e inserire in un rack scorrevole. Centrifugare a 200 xg per 2 minuti per asciugare il vetrino. Collocare all'interno caso diapositiva, avvolgere con pellicola e riporre in un essiccatore.
- Scansione Array
- Posizionare la diapositiva all'interno dello scanner microarray secondo le istruzioni dello strumento.
- Utilizzare un software dello scanner per impostare le lunghezze d'onda 532 nm = Cy3 e 635 nm = Cy5, e gli utili fotomoltiplicatori iniziali tra i 350 ei 400.
- Visualizzare l'anteprima della diapositiva per individuare la matrice sulla diapositiva. Selezionare la regione di matrice per la scansione.
- Scansione matrice e regolare le impostazioni fotomoltiplicatori tale che le caratteristiche di matrice sono per lo più giallo. L'istogramma dovrebbe mostrare le curve rosse e verdi sovrapposte o il più vicino possibile l'una all'altra. Le curve devono terminare sopra i 10 -5 conteggi normalizzati a saturazione.
- Salvare separatamente entrambi i 532 e 635 nm immagini.
- Array Analisi dei dati
- Importare le immagini in un software di analisi serie. Utilizzando il software, creare due relazioni (coppia. Coppia) per ogni array (per le immagini Cy3 e Cy5). Crea scalate log 2 file di rapporto utilizzando i file coppia. Utilizzare log 2 file di rapporto per la ricerca di picchi utilizzando una finestra scorrevole di 500 bp. Mappa loci vetta alle regioni a monte dei geni bersaglio.
- Controllare se il target positivo (determinato mediante EMSA e confermato da qPCR, DVU3025 in questo esempio) compare tra le prime vette.
Nota: Se il bersaglio positivo non è tra i primi successi, è possibile che la RR in consideration ha diversi obiettivi del gene e replicare DAP-chip può essere condotta e picchi comuni a tutte le repliche può essere utilizzato per generare un elenco di destinazione.
5. Binding Pronostico sito Motif e Validazione
- Recuperare sequenze per le regioni a monte (400 bp) per i target gene migliori come generato da analisi DAP-chip. Applicare MEME su queste sequenze attraverso microbi Online (meme.nbcr.net) per prevedere motivi.
Nota: Enhancer proteine leganti quali regolatori sigma54-dipendenti possono associare diverse centinaia di paia di basi a monte del sito di inizio. Per altri regolatori trascrizionali, una regione inferiore come 200 bp a monte sarà sufficiente. - Top Progettazione e inferiore filo oligomeri di DNA che contengono il motivo del sito di legame predetto affiancato da 10 basi su entrambe le estremità. Ordinare il filo top 5 'biotinilato.
- Mescolare le superiore e inferiore oligo filo in rapporto 1:1,5 in 10 mM Tris HCl, pH 8.0, 1 mM EDTA, NaCl e 50 mmun volume totale di reazione di 20 microlitri in tubi PCR. Riscaldare a 95 ° C per 5 minuti in un termociclatore, seguito da un raffreddamento lento a 25 ° C. Diluire dieci volte e utilizzare come wild-type substrato dsDNA per EMSA.
- Preparare substrati modificati utilizzando la stessa procedura di cui sopra, ma oligomeri di progettazione per portare 4-6 sostituzioni nelle basi conservati del motivo di legame.
- Impostare le reazioni di legame con la RR e il tipo selvatico o substrati modificati ed esaminare con EMSA come descritto al punto 2.
- Nota: Il motivo previsto è convalidata se il RR lega il tipo di supporto di natura, ma non quello modificato.
6. Conservazione della Motif in altre specie batteriche correlati
- Selezionare genomi di interesse che contengono ortologhi di DVU3023. Ottenere file di sequenze e annotazioni per i genomi dal sito NCBI.
- Utilizzare un linguaggio di programmazione come Perl per scrivere script che utilizzeranno le sequenze motivi dagli obiettivi DAP-chip per costruire un posmatrice pesata ition e usare la matrice di segnare motivi simili presenti in altri genomi sequenziati.
Risultati
Il metodo di cui sopra è stato applicato per determinare gli obiettivi del gene globali delle RR del modello solfato riducendo batterio Desulfovibrio vulgaris Hildenborough 7. Questo organismo ha un gran numero di STC rappresentata da oltre 70 RR, indicando l'ampia varietà di possibili segnali che rileva e risponde. In vivo analisi sulle funzioni di questi sistemi di segnalamento sono difficili da eseguire in quanto i loro segnali e quindi le condizioni attivanti sono sconosciute. Qui ...
Discussione
Il metodo DAP-chip qui descritto è stato utilizzato con successo per determinare gli obiettivi del gene per diversi RR in Desulfovibrio vulgaris Hildenborough 7 di cui una è indicata qui come risultato rappresentativo. Per RR DVU3023, la scelta di un obiettivo gene candidato era semplice. DVU3025 si trova immediatamente a valle del gene RR, ed i geni RR e di destinazione sono conservati in diverse specie Desulfovibrio, e inoltre DVU3025 ha un promotore sigma54-dipendente predetto. L'EM...
Divulgazioni
Gli autori non hanno alcun conflitto di interesse a divulgare.
Riconoscimenti
Ringraziamo Amy Chen per il suo aiuto nella preparazione per le riprese video e per dimostrare la tecnica. Questo lavoro condotto da ENIGMA: Ecosistemi e reti integrate con i geni e assembly molecolari (http://enigma.lbl.gov), un programma di messa a fuoco Scientifico Area presso il Lawrence Berkeley National Laboratory, è stato supportato da Office of Science, Ufficio biologiche e ricerca ambientale, del Dipartimento dell'Energia degli Stati Uniti nell'ambito del contratto n ° DE-AC02-05CH11231.
Materiali
| Name | Company | Catalog Number | Comments |
| Name of Material/Equipment | Company | Catalog number | Comments |
| HisTrapFF column (Ni-Sepharose column) | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5255-01 | |
| Akta explorer (FPLC instrument) | GE Lfe Sciences, Pittsburgh, PA, USA | ||
| HiPrep 26/10 Desalting column | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5087-01 | |
| Qiaquick Gel extraction kit | Qiagen Inc, Valencia, CA, USA | 28704 | |
| Biotin-labeled oligonucleotides | Integrated DNA Technologies | N/A | |
| 6% polyacrylamide-0.5X TBE precast mini DNA retardation gel | Life Technologies, Grand Island, NY, USA | EC63652BOX | Alternately, you can pour your own gel. |
| Nylon membrane | EMD Millipore, Billerica, MA, USA | INYC00010 | |
| Trans-Blot SD Semi-dry electrophoretic transfer cell | Biorad, Hercules, CA, USA | 170-3940 | |
| Extra thick blot paper, 8 x 13.5 cm | Biorad, Hercules, CA, USA | 170-3967 | |
| UV crosslinker Model XL-1000 | Fisher Scientific | 11-992-89 | |
| Nucleic Acid chemiluminescent detection kit (Pierce) | Thermo fisher Scientific, Rockford, IL, USA | 89880 | |
| Ni-NTA agarose resin | Qiagen Inc, Valencia, CA, USA | 30210 | |
| GenomePlex Whole genome amplification kit (Fragmentation buffer, library preparation buffer, library stabilization solution, library preparation enzyme, 10X amplification master mix, WGA polymerase ) | Sigma-Aldrich, St. Louis, MO, USA | WGA2-50RXN | |
| Nanodrop ND-1000 | Thermo Scientific, Wilmington, DE, USA | For quantitation of DNA | |
| Perfecta Sybr Green SuperMix, with ROX | Quanta biosciences | 95055-500 | Any Sybr Green PCR mix may be used |
| PlateMax Ultra clear heat sealing film for qPCR | Axygen | ||
| 96 well clear low profile PCR microplate | Life Technologies, Grand Island, NY, USA | PCR-96-LP-AB-C | |
| Applied Biosystems StepOne Plus Real time PCR system | Life Technologies, Grand Island, NY, USA | 4376600 | Any real time PCR system may be used |
| Qiaquick PCR purification kit | Qiagen Inc, Valencia, CA, USA | 28104 | Any PCR clean up kit may be used |
| Cy3/Cy5-labeled nonamers | Trilink biotechnologies, San Diego, CA, USA | N46-0001, N46-0002 | |
| Klenow polymerase 50,000U/ml, 3'-5' exo- | New England Biolabs, Ipswich, MA | M0212M | |
| Hybridization system | Roche-Nimblegen, Madison, WI, USA | N/A | This company no longer makes arrays or related items, so alternate sources such as Agilent or Affymetrix will need to be used, |
| Custom printed microarrays and mixers | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Hybridization kit (2X Hybridization buffer, Hybridization component A, Alignment oligo) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Wash buffer kit (10X Wash buffer I, II, III, 1 M DTT) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| GenePix 4200A microarray scanner | Molecular Devices, Sunnyvale CA, USA | This model has been replaced by superior ones | |
| GenePix Pro microarray software | Molecular Devices, Sunnyvale CA, USA | ||
| Nimblescan v.2.4, ChIP-chip analysis software | Roche-Nimblegen, Madison, WI, USA | N/A | |
Riferimenti
- Galperin, M. Y. Diversity of structure and function of response regulator output domains. Curr Opin Microbiol. 13, 150-159 (2010).
- Casino, P., Rubio, V., Marina, A. The mechanism of signal transduction by two-component systems. Curr Opin Struct Biol. 20, 763-771 (2010).
- Aparicio, O., Geisberg, J. V., Struhl, K. Chromatin immunoprecipitation for determining the association of proteins with specific genomic sequences in vivo. Curr Protoc Cell Biol. 17 (17), (2004).
- McCleary, W. R., Stock, J. B. Acetyl phosphate and the activation of two-component response regulators. J Biol Chem. 269, 31567-31572 (1994).
- Novichkov, P. S., et al. RegPrecise web services interface: programmatic access to the transcriptional regulatory interactions in bacteria reconstructed by comparative genomics. Nucleic Acids Res. 40, 604-608 (2012).
- Studholme, D. J., Buck, M., Nixon, T. Identification of potential sigma(N)-dependent promoters in bacterial genomes. Microbiology. 146 (12), 3021-3023 (2000).
- Rajeev, L., et al. Systematic mapping of two component response regulators to gene targets in a model sulfate reducing bacterium. Genome Biol. 12, 99 (2011).
- Crooks, G. E., Hon, G., Chandonia, J. M., Brenner, S. E. WebLogo: a sequence logo generator. Genome Res. 14, 1188-1190 (2004).
- Barbieri, C. M., Mack, T. R., Robinson, V. L., Miller, M. T., Stock, A. M. Regulation of response regulator autophosphorylation through interdomain contacts. J Biol Chem. 285, 32325-32335 (2010).
- Bourret, R. B. Receiver domain structure and function in response regulator proteins. Curr Opin Microbiol. 13, 142-149 (2010).
- Gao, R., Stock, A. M. Molecular strategies for phosphorylation-mediated regulation of response regulator activity. Curr Opin Microbiol. 13, 160-167 (2010).
- Hung, D. C., et al. Oligomerization of the response regulator ComE from Streptococcus mutans is affected by phosphorylation. J Bacteriol. 194, 1127-1135 (2012).
- Ladds, J. C., et al. The response regulator Spo0A from Bacillus subtilis is efficiently phosphorylated in Escherichia coli. FEMS Microbiol Lett. 223, 153-157 (2003).
- Wen, Y., Feng, J., Scott, D. R., Marcus, E. A., Sachs, G. Involvement of the HP0165-HP0166 two-component system in expression of some acidic-pH-upregulated genes of Helicobacter pylori. J Bacteriol. 188, 1750-1761 (2006).
- Liu, X., Noll, D. M., Lieb, J. D., Clarke, N. D. DIP-chip: rapid and accurate determination of DNA-binding specificity. Genome Res. 15, 421-427 (2005).
- Gossett, A. J., Lieb, J. D. DNA Immunoprecipitation (DIP) for the Determination of DNA-Binding Specificity. CSH Protoc. 2008, (2008).
- Mascher, T., et al. The Streptococcus pneumoniae cia regulon: CiaR target sites and transcription profile analysis. J Bacteriol. 185, 60-70 (2003).
- Park, P. J. ChIP-seq: advantages and challenges of a maturing technology. Nat Rev Genet. 10, 669-680 (2009).
- Lefrancois, P., Zheng, W., Snyder, M. ChIP-Seq using high-throughput DNA sequencing for genome-wide identification of transcription factor binding sites. Methods Enzymol. 470, 77-104 (2010).
- Ho, J. W., et al. ChIP-chip versus ChIP-seq: lessons for experimental design and data analysis. BMC Genomics. 12, 134 (2011).
- Liu, X., Lee, C. K., Granek, J. A., Clarke, N. D., Lieb, J. D. Whole-genome comparison of Leu3 binding in vitro and in vivo reveals the importance of nucleosome occupancy in target site selection. Genome Res. 16, 1517-1528 (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon