Method Article
マイクロパターンと定量イメージングを用いた哺乳類細胞の新しい空間組織のマッピング
要約
ここで提示する方法は、マイクロパターニングと定量イメージングを使用して、哺乳類培養における空間的組織を明らかにする。この技術は、標準的な細胞生物学研究室で確立することが容易であり、インビトロでのパターニングを研究するための扱いやすいシステムを提供しています。
要約
生物学の基本的な目標は、開発中にパターンがどのように出現するかを理解することです。いくつかのグループは、幹細胞がマイクロパターンに空間的に限定されている場合にインビトロでパターニングを達成できることを示しており、したがって、生物学的原理の基本原理を識別するユニークな機会を提供する実験モデルを設定する。組織。
ここでは、方法論の独自の実装について説明します。標準的な細胞生物学研究室での確立を容易にするために、特殊な装置の必要性を減らすためにフォトパターニング技術を適応させました。また、標準的な形状や大きさのコロニー内の細胞のサブ集団の優先位置を正確に測定するために、自由でオープンソースで簡単に画像解析フレームワークを導入しました。この方法は、一見乱雑な細胞集団においても、パターニングイベントの存在を明らかにすることを可能にする。この手法は定量的な洞察を提供し、特定のパターニングプロセス上の環境の影響(物理的な手がかりや内因性シグナル伝達など)を切り離すために使用できます。
概要
哺乳類系では、パターニングは細胞の集団挙動の緊急特性であり、適切な手がかりが細胞1、2、3、4に提供されれば、パターンはインビトロで形成され得る。5,6.インビトロで自己組織化する細胞の本質的能力を明らかにする1つの方法は、定義された形状とサイズ7、8、9、10のグループ/コロニーを形成するために細胞を強制することです.これを可能にする技術は、マイクロパターニング11である。マイクロパターニングにより、細胞外マトリックス(ECM)分子が表面に堆積する位置を正確に定義することが可能になります。これにより、セルが付着できる場所が決まり、セルが空間的に整理する方法が制御されます。
マイクロパターニングは、多数の用途を持つ技術であり、例えば、マイクロパターニングは、分化12の前に初期条件の標準化を可能にする。重要なことに、マイクロパターン化は細胞コロニーの大きさ、形状、間隔を容易に制御することを可能にし、この特性は、モルフォゲンまたは物理的な手がかりに細胞の集合的応答を尋問することを目的とした実験を考案するために使用することができる7,8,10歳,13歳,14歳,15歳,16歳,17.
いくつかのマイクロパターニング方法が開発されている11.フォトパターニング技術は、おそらく18を確立するための最も簡単な方法です。これらのアプローチはまた、単一細胞18、19、20の形状を制御するために使用することができるので、精度の利点を持っています。しかし、彼らはまた、一般的に標準的な生物学の実験室で容易に利用できないスピンコーター、プラズマ室およびUVO(UV-オゾン)クリーナーを含む高価な専門装置を必要とします。技術の採用を容易にするために、我々はUVOランプだけを必要とするプロトコルを適応させました。私たちは、はさみでカットしたり、所望のフォーマットに穴パンチで切断することができる市販のプラスチックスライドから始まります。
マイクロパターンの重要な有用性の1つは、複数の反復にわたって個々のコロニーを比較するためにコロニーを標準化する能力です。これにより、これらのコロニー内のパターン形成がどの程度再現可能であるかを尋ね、パターニングプロセスの堅牢性に影響を与える因子を探索することができる。重要なのは、複数の標準化されたコロニーにわたる「平均化された」パターンの定量化は、それ以外の場合には明らかでないパターン化プロセスを明らかにすることもできる。標準化されたコロニーのパターニングを定量化できる利点は、理想的には単一細胞レベルでタンパク質発現を正確に測定できることにあります。しかし、マイクロパターン上の細胞は密集していることが多く、高精度でセグメント化することが困難です。また、セルは 2 次元ではなく 3 次元で編成されることが多く、セグメンテーション中に 3 次元 (3D) 情報を検出して保存することは困難です。セルが正常にセグメント化されると、結果のデータセットからパターニング情報を抽出するための計算方法が必要になります。
これらの問題を克服するためにセグメンテーションと画像解析ツールを開発しました。この分析方法は、フリーでオープンソースのソフトウェアのみを使用し、実装するコマンドラインやプログラミングの知識を必要としません。ここでの方法を説明するために、早期分化ブラチュリー(Tbra)21,22のマーカーを自発的に発現するマウス胚性幹(mES)細胞を用いる。明らかな空間配置は視覚的に検出できませんが、この方法はコロニー内のT+細胞の優先的な位置決めのマップを作成することを可能にします。我々はまた、Tbraパターン化がId1を発現する細胞の優先局在化の欠如と対照的であることを示し、骨形態形成タンパク質(BMP)経路23の直接読み出しである。また、この方法の現在の制限と、この手法を他の実験システムに適合させる方法についても議論する。
プロトコル
注: メソッドの概要は、図1に示されています。
1. マスクデザイン
- Azioune et al.18に記載されているガイドラインに従ってフォトマスクを設計します。この調査で使用したソフトウェアおよびマスクの製造元を参照する場合は、材料の表を参照してください。
注: 1 つのマスクで複数のジオメトリ、サイズ、または図形間の間隔を作成できます。UVランプは49までの異なった設計(2 cm x 2 cmの切りくずを仮定する)を含むことができる15 cmのフォトマスクを合わせることができる。
2. マイクロパターン製造手順
- 必要な材料を準備します。
- リン酸緩衝生理食生(PBS)に0.1%ポロクサマー407(10mLの10mg)の溶液を調べ、室温でシェーカーに残します。ポロクサマー407は溶解するのに約20分かかります。
- 10cm角のペトリ皿の底に実験室フィルム(材料の表を参照)を置きます。これは、マトリックス堆積のためのチャンバとして使用されます。
- フォトマスクの表面を100%アセトンで洗浄し、次に100%イソプロパノールで、最後にddH2Oで清掃します。可能であれば、フォトマスクを空気乾燥するか、清潔なペーパータオルでマスクを乾かしてください。
- フォトマスクとまったく同じサイズの正方形、硬質、不透明なプラスチック片を準備します(後で「ホルダー」と呼ばれます)。
注:これは、照明ステップ中にフォトマスクに接触してプラスチックカバースリップを維持するために使用されます。 - UVOランプをオンにし、10分間ウォーミングイルミネーションを実行します。
- フォトパターンチップを作成します。
注:任意のサイズのチップを作成するために手順を適応させることが可能です。わかりやすくするために、ここでは 12 mm の丸いマイクロパターンチップを生成する手順について説明します。- 12 mm の穴パンチを使用して、疎水性プラスチック スライドをカットして 12 mm のラウンド カバースリップを作成し、きれいな新しいペトリ皿に入れます。
注意:これは表面処理に損傷を与える可能性があるため、プラスチックの表面との皮膚接触を避けるために、常に手袋を使用してください。 - ピンセットでカバースリップから保護フィルムを慎重に取り外します。
注: 種まき手順 (セクション 3) 中にチップ上のセルの配置に影響を与える可能性があるため、プラスチック表面を損傷しないようにしてください。 - フォトマスクを清潔で安定した表面(例えば、フォトマスクボックス)に置き、クロム側を上向きに置き、目的のチップ設計の位置にddH2 Oの2 μLドロップを追加します。
- ddH2Oのドロップにカバースリップを置き、穏やかに押します。
注:フォトマスクに面するプラスチック側が、前の手順で取り外したフィルムによって保護された側であることを確認します。 - プラスチック製のスライドの上にホルダーを置き、フォトマスクに接触するプラスチック片を維持するためにクランプでこのサンドイッチを慎重に固定します。
注:プラスチック製のスライドがフォトマスクの表面と接触して完全に維持されるように、クランプをプラスチックスライドの位置にできるだけ近づけます。 - アセンブリを光源から約2cmのUVOランプに入れ、10分間点灯します。
注:光の電力は、チップがソースから2cmの距離に置かれると、波長254nmで6 mW/cm2と推定されます。 - 底面にフォトマスクでサンドイッチを保持し、サンドイッチを分解しながらスライドが動き回るのを防ぐために、片手で圧力を維持しながらクランプを慎重に取り外します。ホルダーを取り外し、すべてのプラスチック片がマスクの上にあり、ホルダーにくっついないようにします。
- チップの上にddH2Oを追加し、フォトマスクからチップをそっと取り外します。
メモ:プラスチックチップがフォトマスクに貼り付いている場合は、チップが突然取り外された場合に備えて、チップの上にピンセットを少し上に保持しながら、プラスチックピペットチップを使用してチップを取り外します。 - 最後に、マトリックス蒸着室内にフォトパターンチップを配置します。
メモ:チップの照らされた側面が上向きであることを確認します。
- 12 mm の穴パンチを使用して、疎水性プラスチック スライドをカットして 12 mm のラウンド カバースリップを作成し、きれいな新しいペトリ皿に入れます。
- マトリックスを堆積します。
注:このセクションのすべての手順は、組織培養フードで行う必要があります。- ポロクサマー407溶液を0.22 μmポリエテルスルホン(PES)フィルターで濾過します。
- 無菌濾過ポロクサマー407とゼラチンの1mg/mLの500 μg/mLを混合してECMコーティング溶液を調出します。
注: その他の ECM 分子の可能性のある追加情報については、表 1も参照してください。 - 各照らされたチップに200 μLのコーティング溶液を加えます。実験室用フィルムは、落下がチップの外に落ちるのを防ぎます。
- 蒸発を制限し、一晩4°Cでチップと一緒に置くためにddH2Oで満たされた3センチペトリ皿を追加します。
3. 播種手順
注:以下に説明するステップは、標準的なmESC培地を用いてCGR8マウス胚性幹細胞(mESC)24用に最適化されています(材料の表も参照)。ただし、原則として、任意の細胞型に対して手順を適応させることが可能です。また、マウス胚性幹細胞の従来の細胞培養は、広範な文書化が他の場所で見出すことができるとしてここに記載されていないことにも注意してください25.
- コーティング溶液を吸引し、滅菌PBSで少なくとも5分間チップを2回インキュベートします。
- その間、温暖培地中に5.5 x 105セル/mLの細胞懸濁液を調用する。
- 各チップ上の細胞懸濁液のピペ200 μL(約100,000セル/cm2)。
- 播種室を閉じ、細胞をインキュベーターに1時間付着させたままにする。
- 1時間後、マルチウェルプレート(チップ数に応じて4ウェルまたは24ウェルプレート)のウェルを500μL/ウェルの温かい媒体で充填し、チップを無菌ピンセットでプレートに移します。
- 非付着細胞を取り外すためにプレートを激しく振ります。培地を吸引し、すぐに新鮮な暖かい媒体に置き換えます。顕微鏡下でパターニングが見えるかどうかを確認します(図2a)。
注: mESC または他のマトリックスタンパク質以外の細胞株を使用する場合は、接着時間を最適化する必要がある場合があります (表 2も参照)。 - 図 2a に示すようにパターンがはっきりと見えるまで、この手順を繰り返します。
注: この手順は重要であり、手順の成功を決定します。強すぎる洗浄手順は、細胞を剥離し、対照的に不十分な洗浄は、パターンの間に付着した細胞を残す可能性がある(図2c,d)。
4. 固定
注:培養中の48時間後、細胞はパターンの形状に厳密に従う密集したコロニーを形成する必要があります(図2bに示すように)。
- プレートにチップを残し、媒体の約90%を取り出し、チップが乾燥するのを防ぐのに十分な培地を残します。
注:チップがアーティファクトの汚れを避け、表面からの細胞の剥離を防ぐために、チップが決して乾燥しないようにすることが重要です。接着パターン間のチップ表面の疎水性のために、チップは脱濡れする傾向を持ち得る。この段階では、固定剤は、大きなドーム型コロニーがチップから切り離される可能性があります。ワッシュは非常に穏やかで、理想的には、チップに直接ではなく、井戸の側面に液体をピペッティングすることによって行われるべきです。 - ウェルあたり少なくとも500 μLのパラホルムアルデヒド(PFA)ベースの固定溶液をウェルごとに追加し、10分間インキュベートします。
注:コロニーが特に厚く(5セル層以上)に見える場合は、固定時間を20分に調整する必要がある場合があります。 - 固定後、洗浄液(0.01%ポロクサマー407付PBS)で3回洗浄する。洗浄液中に希釈された50mM NH4Clを用いた余分な洗浄は、残留PFA架橋活性をクエンチするためにインターカレータされてもよい。
- ブロッキング溶液で少なくとも30分間サンプルをインキュベートします。
注:この段階では、サンプルは染色の約1週間前に4°Cで保存されてもよい。その場合は、蒸発を防ぐために、実験室用フィルムでプレートを密封してください。
5. 免疫染色
- 10cm角ペトリ皿の底に実験室用フィルムのシートを配置することにより、染色室を準備します。
- 抗体溶液を調記します(この記事で使用される抗体と希釈のリストについては、表3を参照)。
- 上向きの細胞を支える側でチップを染色室に入れ、直ちに100μLの一次抗体溶液をチップ上に加えます。
注意:この段階では、チップはステップ4.1のように簡単に濡れ落ちるべきではありません。しかし、チップが乾燥しないことが重要であるため、注意が必要です。複数のチップを処理する必要がある場合は、各チップにステップ 5.3 を順番に適用します。 - 室温で回転プラットフォーム上で1時間インキュベートします。
注:細胞が大きな3D構造を形成している場合、サンプルの均一な染色を可能にするために、より長いインキュベーション時間が必要な場合があります。インキュベーション時間は24時間まで増加してもよい。しかし、染色室は水で満たされた3cmの皿を含む必要があり、染色室は蒸発を防ぐために実験室フィルムで密封されなければなりません。 - 新鮮なマルチウェルプレートにチップを移し、洗浄液で3回洗浄します。
- ステップ5.3および5.4に記載されているように、二次抗体を使用してインキュベーションを行う。
- 任意の標準的な取り付け媒体(例えば、Mowiol)の20 μLを使用して顕微鏡スライドにチップを取り付けます。
6. イメージング
注:イメージ投射は標準的な共焦点顕微鏡で行うことができる。ここでは、後続の定量分析に十分な画質を確保するための推奨事項のみを提供します。
注意: オペレータのバイアスを避けるために、画像へのコロニーは核エンベロープ信号を使用してのみ選択する必要があります(コロニーがパターンの形状に正しく従っているかどうかを確認します)。顕微鏡の設定を調整する場合を除き、目的のマーカーの信号をチェックすることは避けてください。
- 集録ビットの深さが 12 ビットまたは 16 ビットであることを確認します。
- 各画像チャンネルのダイナミックレンジを最大化するために、適切な設定を特定します。特に、画像のクリッピングは避けてください。
- マイクロパターンの自己蛍光をイメージするチャネルを含めます(図3参照)。
- 画像サイズとズーム係数を調整して、X 軸と Y 軸で 0.1 ~ 0.6 μm のボクセル サイズと Z 軸の 0.2 ~ 2 μm の範囲を取得します。
注:例えば、本研究では、40倍の目的(1.3に等しい数値絞り)、デジタルズームのない1024 x 1024ピクセルの画像サイズ、および0.5 μmのZステップサイズを持つ反転走査共焦点顕微鏡を使用しました。その結果、ボクセルサイズは0.38 μm x 0.38 μm x 0.5 μmでした。 - コロニーごとに、Z 軸に沿った最小位置と最大位置を定義して、コロニー全体が取得されるようにします。信号が低い平面を少なくとも 1 つ、コロニーの上下に含める必要があります。
- Z スタックの向きが常に取得されていることを確認します (常に上から下へ、または常に下から上に)
- スキャン速度、画像解像度、フレーム平均、検出器のゲインを調整して、画質と撮影時間の最適な値を特定します。その指標として、図3に示すように1つのコロニーに対する撮像時間は約2~3分であった。
注意:すべての画像は、同等にするために、同じ目的と取得設定で同じ顕微鏡で取得する必要があります。 - 取得の最後に、すべてのイメージを保存し、各イメージが表す実験条件を識別するために一意の命名規則を割り当てます。
注:例として図 4を参照してください。画像は、BioFormats 26でサポートされている任意の形式に保存されることがあります。コロニーが視野よりも大きい場合、ImageJのステッチプラグインは27を使用することができる。また、ステッチの場合は、イルミネーションロールオフ補正が必要になる場合があります。
7. 画像解析
注: この手順で推奨されるコンピュータの仕様は、16 GB RAM、マルチコア 3.33 GHz CPU、および少なくとも 50 GB のディスク領域 (イメージされたチップの数に応じて) です。ソフトウェアは、Linux、WindowsとMacOS上でテストされています。PickCellsは、複雑な多次元画像における細胞の集合的組織の分析に特化したグラフィックユーザーインターフェイスを備えたクロスプラットフォーム画像解析アプリケーションです(Blin et al.,)。PickCells に関する詳細情報と、ここで説明した特定のモジュールのドキュメントについては、オンラインで見つけることができます: https://pickcellslab.frama.io/docs/。また、我々はソフトウェアを改善し続けるにつれて、インターフェイスが変更されることに注意してください。インターフェイスが図またはビデオに示されているものと異なる場合は、オンラインマニュアルを参照してください。
- オンラインで入手できるドキュメントに従って PickCells をインストールして実行します。
- 画像を読み込み、提供された情報の正確性を確認します (図4-1)。
- 各チャンネルの名前を文書化します。
- Nessysモジュール28(図4-2)を用いた核エンベロープ信号に基づくセグメント核と、生成されたセグメント化された画像の名前を付けるために使用される接頭辞(例えば「核」)を提供する。
注: 使用方法とパラメータ調整に関するドキュメントは、https://framagit.org/pickcellslab/nessysにあります。 - セグメンテーション エディタ モジュール (図 4-3)を使用して、必要に応じてセグメンテーションを検査および編集する
注: 何らかの理由でセグメンテーションプロセスで満足のいく結果が得られなかった場合は、データベース フォルダ内のイメージを手動で削除し、MetaModelビューの「セグメンテーション結果」ノードも削除します。次に、手順 7.4 と 7.5 を繰り返します。画像の小さなサブセットのみのセグメンテーションのみが満足のいく結果を提供しない場合は、Nessys スタンドアロン アプリケーション (7.4 のリンクを参照) を使用し、"障害のあるイメージ' のセグメンテーションを試み、データベース フォルダ内の対応するファイルを置き換えます。 - 基本的なセグメンテーションモジュールを使用してパターン自動蛍光信号をセグメント化する(図4-4)
- 生成されたセグメント化されたイメージの名前に使用するプレフィックス ("パターン" など) を指定します。
- 自己蛍光信号を含むチャネルを選択します。
- ノイズリダクションを適用します。一般に、カーネルサイズが 10 x 10 x 0.5 ボクセルのガウスフィルタを使用すると、満足のいく結果が生じます。
- 下限しきい値を設定して、背景が青色で表示され、前景が白く表示されるようにします。また、上限しきい値を最大値に設定して、最終結果 (赤い領域) から高い強度が除外されないようにします。
- 最後の手順でスキップを選択します。
- [終了]をクリックし、すべてのイメージが処理されるまで待ちます。
- 核に関しては、セグメンテーションエディタモジュール(図4-5)を使用して、必要に応じてセグメンテーション結果を視覚的に検査し、修正できるようになりました。
- 核オブジェクトを作成し、基本的なオブジェクトフィーチャを計算します。
- メイン インターフェイスの左側にあるタスク バーから組み込み機能モジュールを起動します (図 4-6)。
- 楕円体フィッターパネルとサーフェスエクストラクターパネルを閉じて、基本フィーチャーパネルのみを開いたままにします。
- オブジェクトタイプとしてNucleusを選択し、ステップ7.4で示す接頭辞を「セグメント化された画像」に選択します。
- 「計算」を押して、すべてのイメージが処理されるまで待ちます。
注: この手順の後、核セグメンテーションを再度編集することはできません。
- パターン オブジェクトを作成し、基本的なオブジェクト フィーチャを計算します。手順 7.8.1 から 7.8.4 を繰り返し、今回のみ[カスタムタイプをオブジェクトタイプとして]を選択し、セグメント化されたイメージの場合はステップ 7.6.1 で指定したプレフィックスを選択します。
注: この手順を行った後、パターンセグメンテーションを再度編集することはできません。 - 各核が属するイメージの名前を核属性として格納します。
- [データ > 新しい属性]をクリックし、ポップアップ ダイアログで[Nucleus]を選択し、[OK] をクリックします。
- ノードに接続されている他のオブジェクトからデータを収集するを選択し、[次へ]をクリックします。
- 左側のパネルで[イメージ]を選択し、パス定義パネルの[完了]フラグの下にある尋問マークをダブルクリックして、イメージノードをパスのターゲットとして設定します。
- 左側のパネルで[使用可能な属性]ペインを展開し、name 属性を選択します。
- [縮小] 操作ペインを展開し、[1つを取得]を選択し、[変更]ボタンをクリックして [次へ]をクリックします。
- 「画像名」と入力し、タブキーを押して「OK」をクリックします。
- 核オブジェクトに「正規化座標」属性を作成します(図4-7)。
- ステップ 7.10.1 から 7.10.6 に適応して、パターンの大セントロイドの座標を核属性として格納します。この新しい属性に名前を付けます"パターン座標"。
- 次に、[データ > 新しい属性]をクリックし、[Nucleus] をクリックし、[OK] をクリックします。
- [ノードの空間ベクトルと方向ベクトルの間に関数を定義する] を選択し、[次へ]をクリックします。
- 関数の種類の場合は、配列操作を選択し、V1 で項目ベクトルを選択し、[高度]を選択し、V2 で項目ベクトルを選択し、パターン調整e を選択します。
- [次へ]をクリックし、[名前]フィールドに「正規化された座標」と入力し、[終了]をクリックします。
- タブ別の値ファイルにデータをエクスポートします。
8. R分析
- Rstudio をダウンロードしてインストールします。
注: ソフトウェア情報とダウンロード リンクは、https://www.rstudio.com/で入手できます。 - この分析に必要な R スクリプトをダウンロードします。
注: スクリプトは GitLab リポジトリからダウンロードできます: https://framagit.org/pickcellslab/hexmapr。 - Rstudioを開けろ
注: スクリプトを初めて実行する場合は、必要な R パッケージ (ggplot2 とスケール) をインストールします。 - Rstudio から binnedmap_template を開きます。R スクリプト。
- 作業ディレクトリをソース ファイルの場所に設定します。
- 図 5に示すように、スクリプトを特定のデータセットに適合させる手順に従います。
- スクリプトを実行して密度マップを生成します。
結果
ここで説明するフォトパターニング法は、培養細胞を定義された形状およびサイズのコロニーに正確に整理することを可能にする。この手順の成功は、図2aに示すように、付着細胞がフォトマスクの設計に従ってクラスタリングされるため、細胞播種手順(ステップ3.7)の直後に明らかにされるべきである。細胞播種後1時間で、個々のパターンは完全に結合しない(パターンあたり数個の細胞のみ)が、細胞が経つにつれて増殖するにつれて、パターンは接着面の外側のごく少数の細胞で完全に植民地化される(図2b)。カルチャの正確な外観は、セルラインに依存します。例えば、mESCフォームドーム型コロニー10.セルシード後にパターニングが1~2時間明確でないチップは、手順の失敗を示します(図2c,d)。
大きくて厚いコロニーは、均質に染色するのが難しい場合があります。抗体の浸透を改善することができるので、細胞を1ステップ(セクション4)で固定し、透過することを提案する。これは、選択された固定液が洗剤を含む理由です。図3は、免疫染色後に予想される蛍光シグナルを示す。明るい Id1 陽性細胞は、コロニーの密な領域 (明るい NPC 領域) 内に見つかります (図 3A)。このようなヒントは、抗体染色手順の品質を評価するのに有用である。また、本手法で作成されたマイクロパターンは自己蛍光である。この信号(図3A、B左ほとんどの画像)は、解析段階でコロニーを互いに空間的に登録し、図5に示す結果を作成するのに役立ちます。自己蛍光信号は、サンプルが405 nmレーザーで励起され、このチャネルがこの目的のために染色せずに残されるべきであるときに一般的に最も明るいです。図 3は、異なる形状のパターンにセルを正確に拘束する方法も示しています。
イメージングデータの分析は、私たちの研究室で開発されたフリーでオープンソースのソフトウェアであるPickCellsで行われます(Blin et al.,)。このソフトウェアには、共焦点イメージを読み取り、並べ替える画像解析モジュール (図 4-1)、セグメント化 (図4-2、4-4)およびセグメント化されたオブジェクト (図 4-3,4-5)を含み、オブジェクトを計算します。座標や平均強度などのフィーチャ (図 4-6)とデータをエクスポートします (図4-7,4-8)。重要なのは、マイクロパターンで増殖した細胞などの細胞の密度が高く不均一な集団に特に適する、Nessys28と呼ばれる堅牢な核セグメンテーション法を開発したことです(図3)。図 4-2は、個々のセルに一意の色の同一性が正確に与えられる Nessys モジュールの代表的な出力を示しています。最小限の編集のみが必要ですが、ユーザーがそう判断した場合に編集が可能です (図 4-3)。最後に PickCells には、データを視覚化するための多くの視覚化モジュールが用意されています。図4-6に例を示します: リング状のコロニーは、Z 軸に沿った位置に従って核が色分けされた 3D でレンダリングされます。PickCells で解析が検証されると、図 530に示すように、(https://framagit.org/pickcellslab/hexmapr) で使用できるスクリプトを使用して、R で空間マップを作成するデータをエクスポートできます。
我々は最近、小さい(30,000 μm 2)ディスクまたは楕円マイクロパターン上のmESCの空間的閉じ込めが中皮マーカーTbra10を発現する細胞の亜集団のパターン化を導くであることを示した。したがって、ここで我々の方法を説明するために、Tbraのパターニングは、より大きなコロニー(90,000 μm2)におけるBMPシグナル伝達の影響を受ける可能性があるかどうかを尋ねます。図 5Aは、mESC が大きなディスク マイクロパターンで成長する場合、Tbra+ セルがパターンの周辺(Tbra+ 密度マップ)に優先的に制限され、ローカルセル密度が最も低いことを示しています(図 5Aの左側の青いマップを参照)。).このトブラのパターン化は、平均トブラ強度の地図によって確認される。
これらのデータは、メソッドがサブビジュアル情報を明らかにできることを示しています。実際、図3から、1つのコロニーの目視検査は、Tbra発現における空間的組織の任意の形態を同定するのに十分ではない。これは、図5Aの右端のパネルに定量化され示されるコロニー変動に対する重要なコロニーによって特に説明される。
この手法は、T パターニングがこのコンテキストで BMP シグナリングによって駆動されていないことを示す可能性のある Id1 (BMP シグナリングのターゲット) に対して検出可能なパターニングが存在しないことも示しています。
マイクロパターン化により、コロニーにほぼすべての所望のジオメトリを強制的に採用させることができます。これは、システムがさまざまなジオメトリにどのように応答するかを調える場合に特に便利です。たとえば、コロニーの中心にモルフォゲン勾配が蓄積すると、コロニーに穴が開くと、この勾配が乱れる場合があります。興味深いことに、我々はまだあまり堅牢ではない方法で、リングマイクロパターン上のパターンを観察します(図5B)。
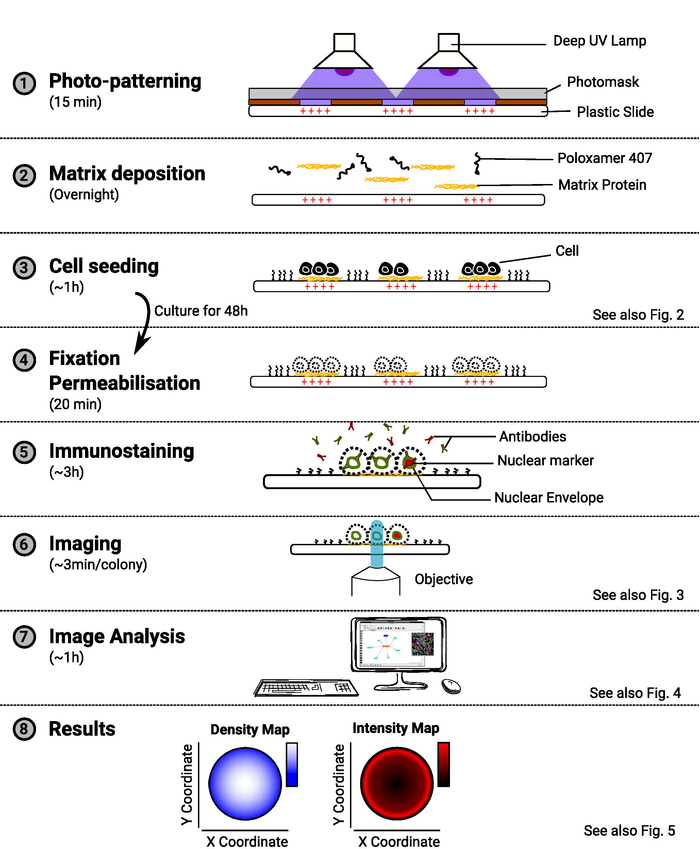
図 1: メソッドの概要。メソッドの主な手順を示す図。各ステップについて、タスクの名前で推定時間が示され、回路図は手順の目的を示します。関連する図形への参照も、使用可能な場合に提供されます。この図のより大きなバージョンを表示するには、ここをクリックしてください。
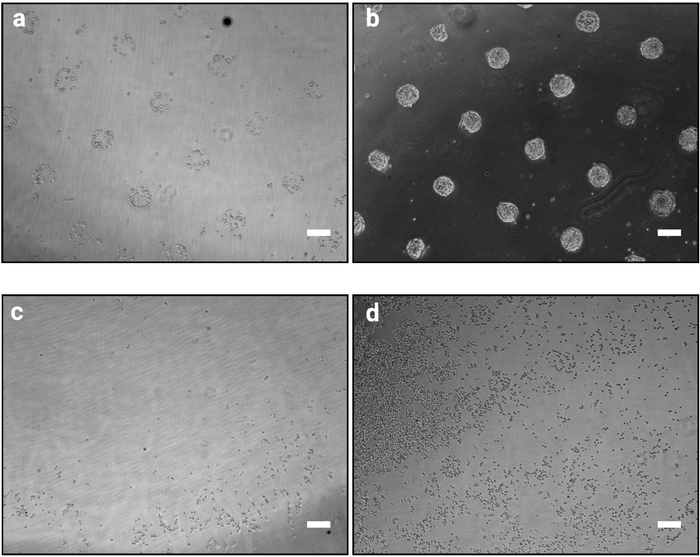
図2:培養外観1時間及び48時間後に細胞をマイクロパターンに播種した。マイクロパターンに播種されたmESCのブライトフィールド画像。(a) シード後1時間の予想細胞組織は、パターンを明確に識別可能にすべきである。(b) 48時間後の予想結果。mESC は増殖し、パターンシェイプに厳密に限定されています。(c-d)可能な非最適な結果は、スライドの周辺部(c)を除いてプラスチックに付着する細胞が非常に少ないか、またはパターン(d)の間に付着する細胞である。トラブルシューティング ガイドについては、表 2を参照してください。この図のより大きなバージョンを表示するには、ここをクリックしてください。
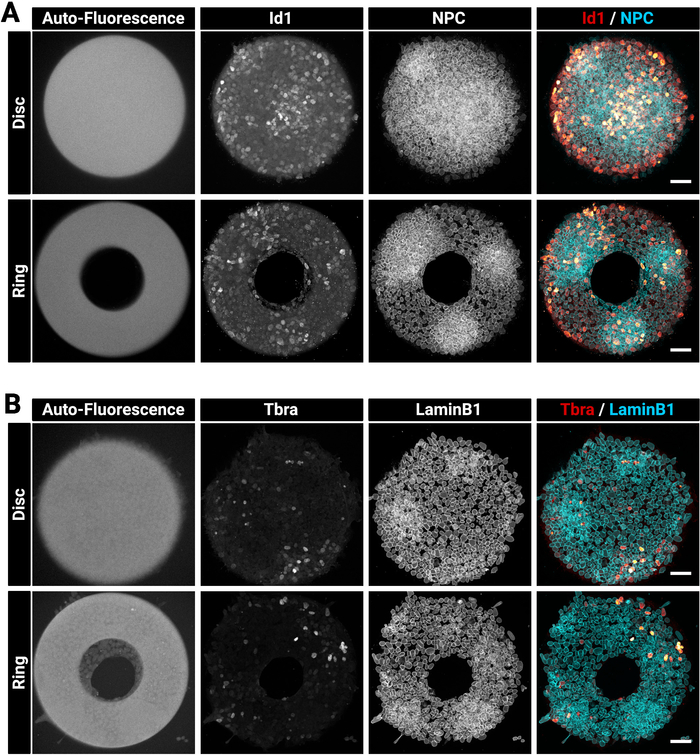
図3:マイクロパターン上で成長した免疫染色コロニーの代表的な共焦点画像。
(A)Id1および核細孔複合体または(B)トブラおよびラミンB1に対する免疫蛍光後の代表的なmESCコロニー。染色ごとに、ディスクマイクロパターン上で成長したコロニーとリングマイクロパターン上で成長したコロニーが示されている。個々のチャンネルは、グレースケール画像として提供されます。マイクロパターンの透明な自動蛍光信号(405nmレーザー励起)に注目してください。スケールバーは50 μmを表し、この図のより大きなバージョンを表示するにはここをクリックしてください。
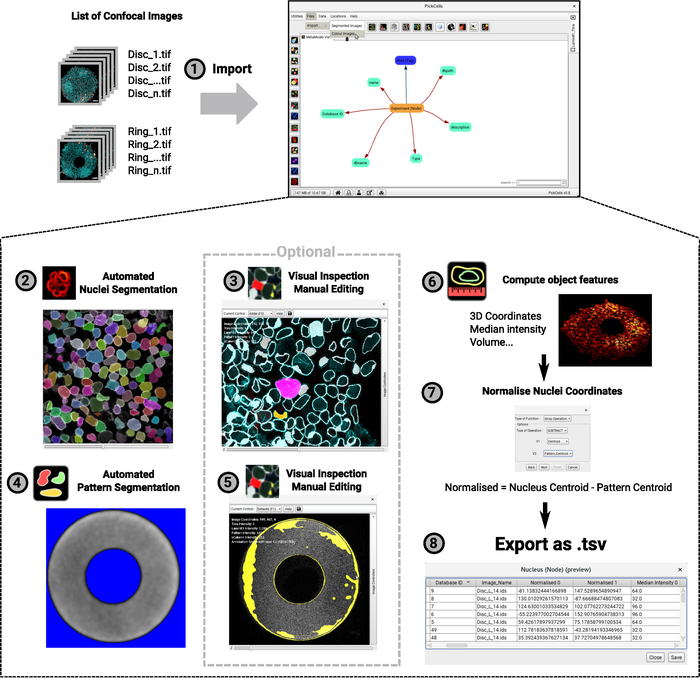
図4:画像解析手順のフローチャート。3D 共焦点イメージのリストは、分析のために PickCellsにインポートされます (1)。この例では、2 つの異なる形状 (図 2のようにディスクとリング) を使用した実験を示しています。イメージの命名規則は左側に、PickCells インターフェイスは右側に表示されます。次に、Nessys モジュールを使用して、自動的に核 (2) をセグメント化します。スクリーンショットでは、個々の核には、正確なセグメンテーションを示す一意の色が与えられます。パターンの自己蛍光もセグメント化され、今回は「基本セグメンテーション」モジュール(4)を使用する。背景は青と白の信号で表示され、パターン形状として定義されます。次に、セグメント化された図形を視覚的に検査して正確なセグメンテーションを確認し、必要に応じてセグメンテーション エディタ モジュール(3-5)を使用して編集します。スクリーンショットには、検出された図形の輪郭が表示されます。ピンクと黄色の図形が編集されました。最後に、オブジェクト フィーチャが計算され、後で R (6-7)に処理されるファイルにエクスポートされます。3D ビューとしてレンダリングされたコロニーのスクリーンショットが提供されます(6) .手順 2 ~ 6 では、この記事の執筆時点で PickCells インターフェイスで見つかったアイコンがステップ インデックスの横に表示されます。この図のより大きなバージョンを表示するには、ここをクリックしてください。
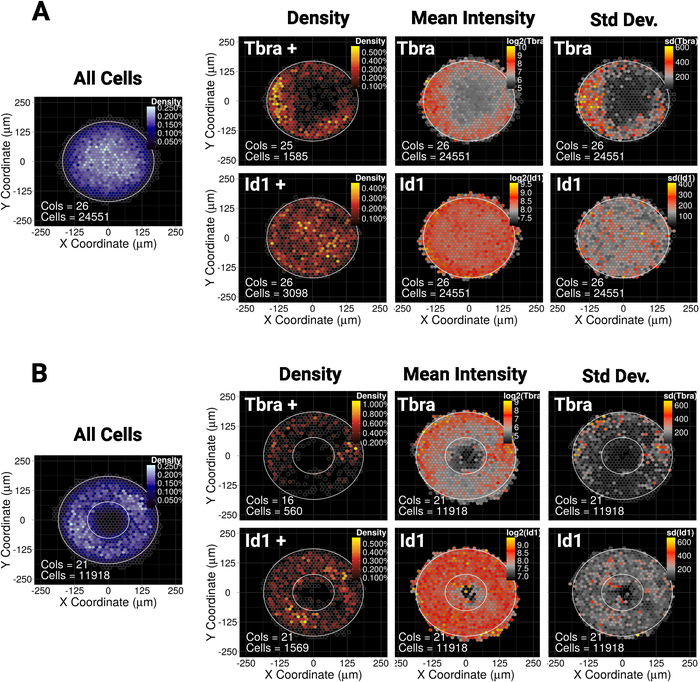
図5:2つの異なる転写因子とマイクロパターン形状の代表的な結果
(A)ディスク形状のマイクロパターンまたは(B)リング形状のマイクロパターン上で48時間成長したmESC用のビン化された空間マップ。各マイクロパターン形状について、細胞表現型に関係なく、セル密度のマップが青色のカラースケールで左側に表示されます。次に、各マーカー(一番上の行のTbraと一番下の行のId1)に対して、左から右に3つの異なるマップが提供されます:セルのみを表現するマーカーのセル密度マップ(しきい値ベースの解析)、平均マーカー強度のマップ(log2)とマップマーカー強度の標準偏差。強度は、任意の蛍光単位として与えられる。マップごとに、マイクロパターンの形状が白いアウトラインとして表示されます。この図のより大きなバージョンを表示するには、ここをクリックしてください。
| ECM タイプ | ゼラチン | フィブロネクチン | 地下膜マトリックス | |
| 濃度 | ECM濃度 | 1 mg/mL | 20 μg/mL | 200 μg/mL |
| ポロクサマー 407 濃度 | 500 μg/mL | 400 μg/mL | 1 mg/mL | |
| テスト済み | mESC | うん | うん | うん |
| mEpiSC | 違います | うん | うん | |
| 血清フリー培地 | 違います | うん | うん |
表1:ポロクサマー407およびECMの濃度を試験した。この表は、当社のラボでテストしたECMおよびポロクサマー407の濃度の概要を示しています。ECM/ポロクサマー407の組み合わせごとに、パターニングが成功した細胞タイプと、培養に血清が含まれているかどうかが示される。mESC=マウス胚性幹細胞、mEpiSC=マウスエピブラスト幹細胞。
| 手順 | 観測 | 考えられる問題 | ソリューション |
| マイクロパターニング | 低セルの添付ファイル | 不適切なECM/ポロクサマー307濃度比 | ECM/ポロクサマー307濃度比を高める |
| セルの接続時間が短すぎます | インキュベーション時間を増やして、細胞がパターンに適切に付着するのに十分な時間を与えます(ステップ3.4)。この工程を最適化するために、顕微鏡で細胞をチェックすると、細胞が付着し始めたことを示す細胞形態の変化を検出するのに役立ちます。 | ||
| 洗い過ぎ(ステップ3.6) | 培地を交換する場合は、チップに直接媒体をピペッティングすることは避けてください。代わりに、代わりに井戸の壁にメディアを穏やかにピペ | ||
| 細胞はパターンの間に付着する | 不適切なECM/ポロクサマー307濃度比 | ECM/ポロクサマー407濃度比を下げ | |
| セルの接続時間が長すぎます | インキュベーション時間を短縮します(ステップ3.4)。 | ||
| 洗い物が無効(ステップ3.6) | プレートを激しく振ることは、通常、過剰に細胞を剥離するのに十分である。チップに強く付着する傾向がある細胞タイプの場合、チップに直接ピペッティングすると、結果が向上する可能性があります。特に、このステップの後に培地内にセルが浮遊していないことを確認するために、洗い出しの数を増やすことも役立ちます。 | ||
| セルがパターンシェイプに厳密に従わない | 「互換性のない」セルタイプとパターンジオメトリ | フォトマスクに追加する複数のジオメトリ/サイズを計画/設計し、特定のパターン形状とセルタイプに最適なパターンサイズをテストおよび識別できるようにします。ディスカッションの「制限事項」セクションを参照してください。 | |
| 非最適なフォトパターニングは、これは自己蛍光信号の鮮明さを観察することによって診断することができる。パターン境界は図 2 のようにシャープに見える必要があります。パターンの境界線がぼやけているように見える場合は、写真のパターニングステップを改善する必要があります。 | ぼやけたパターンエッジは、プラスチックスライドが照明ステップ中にマスクの表面に十分に近づいていなかったことを示します。フォトマスクにスライドを保持しているピースが均一であり、照明手順中にアセンブリに一定の十分な圧力が加えられるようにします。 | ||
| 染色 | 非均質な染色 | 抗体インキュベーション時間が短すぎる | 抗体のインキュベーション時間を増やす(室温で最大24時間) |
| 取り付け手順中に平坦化されたコロニー | チャンフリードやサイトゥーチャンバーなどのチャンムやサイトゥーチャンバーなどのチャンバーにマイクロパターンスライドを取り付け、細胞を取り付けることなく免疫染色とイメージングの両方を行います。これにより、コロニーの 3D 構造が維持されます。 | ||
| 染色手順中にコロニーを切り離す | チップのデウェット | 十分な培地を残すか、2ピペットを使用し、1つは培地を取り除き、もう1つは新鮮な溶液を追加します。 | |
| コロニーは顕微鏡の下でせこく見える | 顕微鏡スライドにチップを取り付けている間、コロニーは引き起された | チップを取り付けるときには、非常に穏やかです。あるいは、カムライドやサイトゥーチャンバーなどのチャンム室にマイクロパターンスライドを取り付け、細胞を取り付けることなく免疫染色とイメージングの両方を行う。これはまた、コロニーの超構造を保持します。 |
表 2: トラブルシューティング ガイド。次の表に、最適でない結果の概要を示します。問題の潜在的な原因は、推奨される解決策と共に一覧表示されます。
ディスカッション
ここでは、細胞培養における新たなパターン化を解析する方法について述べている。セルコロニーの形状とサイズを標準化するために簡略化されたマイクロパターン化アプローチを用い、これらのコロニー内のパターンの検出と定量を可能にする画像解析ツールとRスクリプトを紹介する。
我々が提案するパイプラインは、著者が市販のマイクロパターンを使用して培養条件に焦点を当てた以前に公開された方法31とある程度類似しており、ESCコロニーにおける再生可能な生殖層層形成を得る。インビトロにおける初期の胃発生事象の研究。我々の目的は、細胞の集合的な組織が統計的分析後にのみ明らかになる可能性があるインビトロでのパターン形成の発見のための一般化可能なパイプラインを提供することに焦点を当てています。このため、複数のコロニーに対して3D空間における核位置の正確な識別と解析を可能にする堅牢な画像解析ワークフローを提供します(「パターン検出方法の利点と限界」セクションも参照)。ディスカッション)。また、長期的に市販のソリューションに代わる、より柔軟で安価な代替手段を提供するシンプルな社内マイクロパターニングアプローチを開発することにしました。
最後に、この原稿の改訂中に、Rスクリプトと同様のインビトロでのパターニング分析のための新しいパッケージが32をリリースされたことに注意してください。この新しいパッケージは、高スループットイメージングプラットフォームから得ることができる入力としてセル機能のテーブルを受け入れます。私たちは、プロトコルのステップ7で生成された核機能のテーブルは、この可能性をテストしていませんが、原則としてこの新しいパッケージへの入力として役立つと考えています。
他のセルタイプおよびコロニージオメトリへのメソッドの適応性
我々は、血清の存在下で多能性細胞の培養における中皮転写因子の出現を研究する文脈でこのアプローチを提示する。しかし、この方法は他の細胞型および無血清培養物に容易に適応可能であるが、ECM/ポロクサマー407濃度を最適化する必要があるかもしれない(トラブルシューティングガイドの場合は表1、表2を参照)。この方法はまた、マイクロパターンのより大きいまたは小さいサイズに、およびユーザーの必要性に応じて形状の広い範囲に適応させることができる。ただし、この方法を確立する際には、すべての形状/セル型の組み合わせが最適であるとは考えていないことに注意してください。例えば、mESCは高レベルのE-カドヘリン33,34を発現し、これらの細胞がECMを欠いている領域にまたがる集合的構造を形成することを可能にする。これらのセルは、鋭い角度を持つジオメトリや、パターンに小さな穴を含むジオメトリに厳密に従っていません。例えば、図3Bのリング上で、セルが中央領域を植民地化する過程にあることに注意してください。私たちの手の中で、より小さな中央領域は、mESCがリング状のコロニーを形成することを強制しませんでした。したがって、選択したセルタイプに適した最適なサイズと曲率をテストおよび識別できるようにフォトマスクを設計しながら、形状の多様性を含めることを強くお勧めします。
考慮すべきもう一つの重要な要因は、実験の長さと細胞の増殖速度です。一部の急速に増殖する細胞タイプ(多能性細胞を含む)では、何日もマイクロパターン上の細胞を維持することは困難な場合があります(mESCの場合は3日間が最大です)。また、マイクロパターン上の細胞の播種は、常にすべてのコロニーに最適に発生するとは限らないので、スペアを持つためにコロニーの過剰を播種することをお勧めします。
パターン検出方法の利点と限界
この方法の特に利点の1つは、複数の反復コロニーからの画像解析結果を組み合わせることによって「平均化された」パターンを検出する能力である(図5)。これは、個々のコロニーの検査から明らかでないパターニングイベントを明らかにすることができます。この「平均化」アプローチの欠点は、小さなスポットや狭いストライプなど、特定のタイプの反復パターンを見逃す可能性があることです。しかし、これらのタイプのパターンは、代わりに慎重に選択されたパターンサイズ8の組み合わせで明らかにされてもよい。また、ここで説明する画像解析パイプラインは、単一細胞とコロニー分解能の両方で定量的なデータを提供し、コロニー間変動のレベルを調査したり、複数でネイバー解析を実行したりする可能性を提供します。スケール10.
平均化方法のもう一つの重要な利点は、検出チャネルの利用可能な蛍動管によって制限されることなく、多くのマーカーの優先位置をマッピングする機会を提供することです。実際、ここで示した作品では分化のマーカーが2つだけ利用されていますが、コロニーを標準化し、「平均化」パターンを抽出できるため、異なるコロニーセットの分布マップを順番に比較することが可能になります。マーカーの一般的な空間的関係を明らかにします。
さらに、我々の焦点は分化のマーカーを研究することにあったが、分析方法は核マーカーが利用できる他の生物学的プロセスを研究するために拡張することができる。例えば、蛍光ユビキチン化細胞サイクル指標35(FUCCI)を含む細胞株のマイクロパターン化は、コロニーレベルの幾何学がグループ内の細胞周期事象にどのように影響するかを研究することを可能にするであろう。
今後の方向性
この方法は中程度のスループット画像解析に適していますが、画像取得は現在完全に自動化されていないので、非常に大規模な実験では制限される可能性があります。コロニーの定期的な配置は、単一細胞平均20のために開発されたものと同様に、完全に自動化された取得ルーチンを作成することを可能にする必要があります。しかし、コロニーの画像化に必要なフィールドのサイズが大きく、モザイク化が必要な場合があり、コロニーが3次元であるため、関連するコロニーのみをイメージングすることにより、データセットのサイズと取得時間の両方を短縮することが非常に望ましい。したがって、将来の取り組みは、関連するコロニーを識別し、各サンプルにイメージング座標を適応させる「インテリジェント」顕微鏡の開発に専念する可能性があります。これにより、時間と労力を削減するだけでなく、潜在的なオペレータのバイアスを防ぐことができます。
また、ユーザーが実行する必要があるステップの数を減らすことで、分析パイプラインをより効率的に行うこともできます。パイプライン構築メカニズムを構築し、Rを当社のソフトウェアに直接統合する計画があります(コードリポジトリの問題トラッカー[https://framagit.org/groups/pickcellslab/-/issues]のpickcells-api#3とpickcells-rjava#1も参照してください)。分析手順を完全に自動化すると、時間と労力が削減され、潜在的なユーザー エラーが制限されます。
最後に、我々の分析方法は、細胞パターンの動的性質をまだ完全に捕捉していないことに注意してください。一部の限られた動的情報は、スナップショットイメージ8、10、36の時系列を調べることによって抽出することができる。しかし、パターニングがどのように出現するかをよりよく理解したい場合は、細胞集団の歴史を記録できることが非常に望ましいです。1つの制限は、3D密度の細胞集団における個々の細胞の正確な追跡が依然として非常に困難な課題37であるということです。我々の細胞検出方法は、核エンベロープを使用し、密で重なり合う細胞集団28に特によく行います。核封筒のライブレポーターは、容易に入手可能である28、38およびマイクロパターニング技術の1つの利点は、細胞が長期イメージング中に視野の外に移動するのを防ぐために使用され得る。全体的に見て、最近確立されたツール28、39、40の組み合わせを使用して細胞の自動追跡が達成可能であり、これが根本的な新しい洞察をもたらすべきであると確信しています。自己組織化の原則。
開示事項
著者は開示するものが何もない
謝辞
この作品は、ヘンリー・ウェルカム卿の博士後期フェローシップ(WT100133からG.B.)、ウェルカム・トラスト・シニア・フェローシップ(WT103789AIA to S.L.)、ウェルカム・トラスト博士課程の学生シップ(108906/Z/15/ZからD.W.)によって資金提供されました。また、フォトパターニング技術の適応に関する彼のアドバイスに対して、マヌエル・テリー博士に感謝しています。
資料
| Name | Company | Catalog Number | Comments |
| 2-mercaptoethanol | Gibco | 31350-010 | handle in fume hood |
| 1× Master quartz anti-reflective chromium photomask | Toppan Photomask | Custom design | |
| 24-well plates | Corning | 3526 | |
| 4-well plates | Nunclon Delta | 176740 | |
| 5% Donkey Serum | Sigma | D9663 | |
| Anti Nuclear pore complex | Abcam | ab24609 | Mouse monoclonal, use 1:1000 |
| Anti-Id1 | Biocheck | 37-2 | Rabbit polyclonal, use 1:200 |
| Anti-Lamin B1 | Abcam | ab16048 | Rabbit polyclonal, use 1:1000 |
| Anti-Tbra | R&D | AF2085 | Goat ployclonal, use 1:400 |
| Blocking Solution | NA | NA | 0.1% TritonX-100 0.01% Pluronic F-127 5% Donkey Serum 0.003% Sodium Azide In PBS |
| CGR8 mESC | NA | NA | Reference: Mountford et al. PNAS, 1994 |
| Fixation solution | NA | NA | 4% PFA diluted in washing solution |
| Foetal bovine serum | Gibco | 10270-106 | Serum batches must be tested to ensure compatibility with ESC maintenance |
| Gelatin | Sigma | G1890 | |
| Glasgow Minimum Essential Medium | Sigma | G5154 | |
| Laboratory Film | VWR | 291-1212 | |
| Layout Editor Software | LayoutEditor | NA | https://layouteditor.com/ |
| Layout Editor Software | Klayout | NA | https://www.klayout.de/ |
| L-glutamine | Gibco | 25030-024 | |
| LIF | Millipore | ESG1107 | |
| MEM NEAA | Gibco | 11140-035 | |
| mESC Culture medium | NA | NA | GMEM 10% FCS 100 U/ml LIF 100 nM 2-mercaptoethanol 1× non-essential amino acids, 2 mM L- glutamine 1 mM sodium pyruvate |
| NH4Cl 50mM | Sigma | 9718 | |
| Paraformaldehyde | Sigma | 158127 | CAUTION: Toxic, handle undiluted stocks in fume hood and wear protective equipment |
| PBS (immunostaining) | Sigma | P4417 | Dilute in 200ml of ddH2O |
| PBS (tissue culture) | Gibco | 11140-035 | |
| Plastic slides | Ibidi | IB-10813 | |
| Poloxamer 407 | Sigma | P2443 | |
| ProLong Gold Antifade Mountant | Molecular Probes | P36930 | |
| Sodium Azide | Sigma | 8591 | CAUTION: Toxic, handle in fume hood and wear protective equipment |
| Sodium pyruvate | Gibco | 11360070 | |
| TritonX-100 | Sigma | T8532 | |
| Trypsin | Gibco | 25200056 | |
| UVO cleaner | Jetlight, USA | 42-220 | CAUTION: Follow manufacturer’s safety recommendations |
| Washing Solution | NA | NA | 0.1% TritonX-100 0.01% Pluronic F-127 In PBS |
参考文献
- Sasai, Y. Cytosystems dynamics in self-organization of tissue architecture. Nature. 493 (7432), 318-326 (2013).
- Turner, D. A., Baillie-Johnson, P., Martinez Arias, A. Organoids and the genetically encoded self-assembly of embryonic stem cells. BioEssays. 38 (2), 181-191 (2016).
- Simunovic, M., Brivanlou, A. H. Embryoids, organoids and gastruloids: new approaches to understanding embryogenesis. Development. 144 (6), 976-985 (2017).
- Tewary, M., Shakiba, N., Zandstra, P. W. Stem cell bioengineering: building from stem cell biology. Nature Reviews. Genetics. 19 (10), 595-614 (2018).
- Shahbazi, M. N., Zernicka-Goetz, M. Deconstructing and reconstructing the mouse and human early embryo. Nature Cell Biology. 20 (8), 878(2018).
- Laurent, J., et al. Convergence of microengineering and cellular self-organization towards functional tissue manufacturing. Nature Biomedical Engineering. 1 (12), 939(2017).
- McBeath, R., Pirone, D. M., Nelson, C. M., Bhadriraju, K., Chen, C. S. Cell shape, cytoskeletal tension, and RhoA regulate stem cell lineage commitment. Developmental Cell. 6 (4), 483-495 (2004).
- Tewary, M., et al. A stepwise model of Reaction-Diffusion and Positional-Information governs self-organized human peri-gastrulation-like patterning. Development. , (2017).
- Warmflash, A., Sorre, B., Etoc, F., Siggia, E. D., Brivanlou, A. H. A method to recapitulate early embryonic spatial patterning in human embryonic stem cells. Nature Methods. 11 (8), 847-854 (2014).
- Blin, G., Wisniewski, D., Picart, C., Thery, M., Puceat, M., Lowell, S. Geometrical confinement controls the asymmetric patterning of brachyury in cultures of pluripotent cells. Development. 145 (18), (2018).
- Micropatterning in cell biology. Pt. A. , Elsevier. Amsterdam. (2014).
- Bauwens, C. L., et al. Control of human embryonic stem cell colony and aggregate size heterogeneity influences differentiation trajectories. Stem Cells. 26 (9), Dayton, Ohio. 2300-2310 (2008).
- Heemskerk, I., Burt, K., Miller, M., Chabra, S., Guerra, M. C., Warmflash, A. Morphogen dynamics control patterning in a stem cell model of the human embryo. bioRxiv. , 202366(2017).
- Nemashkalo, A., Ruzo, A., Heemskerk, I., Warmflash, A. Morphogen and community effects determine cell fates in response to BMP4 signaling in human embryonic stem cells. Development. 144 (17), 3042-3053 (2017).
- Peerani, R., Onishi, K., Mahdavi, A., Kumacheva, E., Zandstra, P. W. Manipulation of signaling thresholds in “engineered stem cell niches” identifies design criteria for pluripotent stem cell screens. PloS One. 4 (7), e6438(2009).
- Peerani, R., et al. Niche-mediated control of human embryonic stem cell self-renewal and differentiation. The EMBO journal. 26 (22), 4744-4755 (2007).
- Etoc, F., et al. A Balance between Secreted Inhibitors and Edge Sensing Controls Gastruloid Self-Organization. Developmental Cell. 39 (3), 302-315 (2016).
- Azioune, A., Carpi, N., Tseng, Q., Théry, M., Piel, M. Protein micropatterns: A direct printing protocol using deep UVs. Methods in Cell Biology. 97, 133-146 (2010).
- Théry, M. Micropatterning as a tool to decipher cell morphogenesis and functions. Journal of Cell Science. 123 (Pt 24), 4201-4213 (2010).
- Degot, S., et al. Improved Visualization and Quantitative Analysis of Drug Effects Using Micropatterned Cells. JoVE (Journal of Visualized Experiments). (46), e2514(2010).
- Beddington, R. S. P., Rashbass, P., Wilson, V. Brachyury - a gene affecting mouse gastrulation and early organogenesis. Development. 116 (Supplement), 157-165 (1992).
- Wilkinson, D. G., Bhatt, S., Herrmann, B. G. Expression pattern of the mouse T gene and its role in mesoderm formation. Nature. 343 (6259), 657-659 (1990).
- Hollnagel, A., Oehlmann, V., Heymer, J., Rüther, U., Nordheim, A. Id Genes Are Direct Targets of Bone Morphogenetic Protein Induction in Embryonic Stem Cells. Journal of Biological Chemistry. 274 (28), 19838-19845 (1999).
- Mountford, P., et al. Dicistronic targeting constructs: reporters and modifiers of mammalian gene expression. Proceedings of the National Academy of Sciences of the United States of America. 91 (10), 4303-4307 (1994).
- Smith, A. G. Culture and differentiation of embryonic stem cells. Journal of tissue culture methods. 13 (2), 89-94 (1991).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Preibisch, S., Saalfeld, S., Tomancak, P. Globally optimal stitching of tiled 3D microscopic image acquisitions. Bioinformatics. 25 (11), 1463-1465 (2009).
- Blin, G., Sadurska, D., Migueles, R. P., Chen, N., Watson, J. A., Lowell, S. Nessys: a novel method for accurate nuclear segmentation in 3D. bioRxiv. , 502872(2018).
- Weiswald, L. -B., Guinebretière, J. -M., Richon, S., Bellet, D., Saubaméa, B., Dangles-Marie, V. In situ protein expression in tumour spheres: development of an immunostaining protocol for confocal microscopy. BMC cancer. 10, 106(2010).
- R Core Team. R: A Language and Environment for Statistical Computing. , R Foundation for Statistical Computing. Vienna, Austria. Available from: http://www.R-project.org/ (2013).
- Deglincerti, A., et al. Self-organization of human embryonic stem cells on micropatterns. Nature Protocols. 11 (11), 2223-2232 (2016).
- Ostblom, J., Nazareth, E. J. P., Tewary, M., Zandstra, P. W. Context-explorer: Analysis of spatially organized protein expression in high-throughput screens. PLOS Computational Biology. 15 (1), e1006384(2019).
- Larue, L., Ohsugi, M., Hirchenhain, J., Kemler, R. E-cadherin null mutant embryos fail to form a trophectoderm epithelium. Proceedings of the National Academy of Sciences. 91 (17), 8263-8267 (1994).
- Pieters, T., van Roy, F. Role of cell-cell adhesion complexes in embryonic stem cell biology. J Cell Sci. 127 (12), 2603-2613 (2014).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132 (3), 487-498 (2008).
- Morgani, S. M., Metzger, J. J., Nichols, J., Siggia, E. D., Hadjantonakis, A. -K. Micropattern differentiation of mouse pluripotent stem cells recapitulates embryo regionalized cell fate patterning. eLife. 7, e32839(2018).
- Ulman, V., et al. An objective comparison of cell-tracking algorithms. Nature Methods. 14 (12), 1141(2017).
- Moir, R. D., Yoon, M., Khuon, S., Goldman, R. D. Nuclear Lamins a and B1. The Journal of Cell Biology. 151 (6), 1155-1168 (2000).
- McDole, K., et al. In Toto Imaging and Reconstruction of Post-Implantation Mouse Development at the Single-Cell Level. Cell. 175 (3), 859-876 (2018).
- Stegmaier, J., et al. Real-Time Three-Dimensional Cell Segmentation in Large-Scale Microscopy Data of Developing Embryos. Developmental Cell. 36 (2), 225-240 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved