Method Article
Kartierung der Emergent Spatial Organization of Mammalian Cells mit Mikromustern und quantitativer Bildgebung
In diesem Artikel
Zusammenfassung
Die hier vorgestellte Methode verwendet Mikromuster ungemilzt mit quantitativer Bildgebung, um die räumliche Organisation innerhalb von Säugetierkulturen aufzuzeigen. Die Technik ist in einem Standardlabor für Zellbiologie leicht zu etablieren und bietet ein beschreibbares System zur Untersuchung von Mustern in vitro.
Zusammenfassung
Ein grundlegendes Ziel in der Biologie ist es zu verstehen, wie Muster während der Entwicklung entstehen. Mehrere Gruppen haben gezeigt, dass Musterung in vitro erreicht werden kann, wenn Stammzellen räumlich auf Mikromuster beschränkt sind, wodurch experimentelle Modelle erstellt werden, die einzigartige Möglichkeiten bieten, die Grundprinzipien biologischer Organisation.
Hier beschreiben wir unsere eigene Umsetzung der Methodik. Wir haben eine Fotomustertechnik angepasst, um den Bedarf an spezialtechnischem Gerät zu reduzieren, um die Etablierung der Methode in einem Standardlabor für Zellbiologie zu erleichtern. Wir haben auch ein freies, Open-Source- und einfach zu installierendes Bildanalyse-Framework entwickelt, um die bevorzugte Positionierung von Unterpopulationen von Zellen innerhalb von Kolonien mit Standardformen und -größen genau zu messen. Diese Methode ermöglicht es, die Existenz von Musterereignissen auch in scheinbar unorganisierten Populationen von Zellen aufzudecken. Die Technik liefert quantitative Erkenntnisse und kann verwendet werden, um Einflüsse der Umwelt (z. B. physikalische Hinweise oder endogene Signalisierung) auf einen bestimmten Musterprozess zu entkoppeln.
Einleitung
In Säugetiersystemen ist die Musterung eine auftauchende Eigenschaft des kollektiven Verhaltens von Zellen, und so können Muster in vitro bilden, wenn den Zellen1,2,3,4 , 5 , 6. Eine Möglichkeit, die intrinsische Fähigkeit der Zellen zu offenbaren, sich in vitroselbst zu organisieren, besteht darin, die Zellen zu zwingen, Gruppen/Kolonien einer definierten Form und Größe 7,8,9,10 zu bilden. . Eine Technik, die dies ermöglicht, ist Mikromusterung11. Die Mikromusterung ermöglicht es, genau zu definieren, an welcher Stelle extrazelluläre Matrixmoleküle (ECM) auf einer Oberfläche abgelagert werden. Dies wiederum bestimmt, wo die Zellen haften können und steuert daher, wie Zellen räumlich organisiert werden.
Micropatterning ist eine Technik mit zahlreichen Anwendungen, z.B. Mikromusterung ermöglicht die Standardisierung von Ausgangsbedingungen vor Differenzierung12. Wichtig ist, dass mikropatterning es ermöglicht, die Größe, Form und den Abstand von Zellkolonien leicht zu kontrollieren, und diese Eigenschaft kann verwendet werden, um Experimente zu entwickeln, die darauf abzielen, die kollektive Reaktion der Zellen auf Morphogen oder physikalische Hinweise zu verhören7 , 8 , 10 , 13 , 14 , 15 , 16 , 17.
Es wurden mehrere Mikromusterverfahren entwickelt11. Photopatterning-Techniken sind vielleicht die einfachsten Methoden, um18zu etablieren. Diese Ansätze haben auch den Vorteil der Präzision, da sie verwendet werden können, um die Form einzelner Zellen18,19,20zu steuern. Sie erfordern jedoch auch teure Spezialgeräte wie einen Spincoater, eine Plasmakammer und einen UVO (UV-Ozon) Reiniger, die in der Regel in Standard-Biologielaboratorien nicht ohne weiteres verfügbar sind. Um die Einführung der Technik zu erleichtern, haben wir das Protokoll angepasst, um nur die UVO-Lampe zu benötigen. Wir gehen von handelsüblichen Kunststoffschlitten aus, die mit einer Schere oder mit einem Lochschlag in das gewünschte Format geschnitten werden können.
Ein wichtiger Nutzen von Mikromustern ist die Fähigkeit, Kolonien zu standardisieren, um einzelne Kolonien über mehrere Replikationen hinweg zu vergleichen. Dies macht es möglich zu fragen, inwieweit die Musterbildung innerhalb dieser Kolonien reproduzierbar ist, und Faktoren zu erforschen, die die Robustheit des Musterprozesses beeinflussen. Wichtig ist, dass die Quantifizierung von "durchschnittlichen" Mustern in mehreren standardisierten Kolonien auch Musterprozesse aufzeigen kann, die sonst nicht erkennbar wären. Der Vorteil, die Musterung auf standardisierten Kolonien quantifizieren zu können, hängt davon ab, dass man die Proteinexpression genau messen kann, idealerweise auf Einzelzellebene. Allerdings sind Zellen auf Mikromustern oft dicht gepackt, was eine Segmentierung mit hoher Genauigkeit erschwert. Zellen organisieren sich auch oft in drei statt zwei Dimensionen, und es kann schwierig sein, dreidimensionale (3D) Informationen während der Segmentierung zu erkennen und zu bewahren. Nachdem Zellen erfolgreich segmentiert wurden, werden Berechnungsmethoden zum Extrahieren von Musterinformationen aus den resultierenden Datasets benötigt.
Wir haben Segmentierungs- und Bildanalyse-Tools entwickelt, um diese Probleme zu überwinden. Diese Analysemethode verwendet nur freie und Open-Source-Software und erfordert keine Kenntnisse der Befehlszeile oder Programmierung zu implementieren. Um die Methode hier zu veranschaulichen, verwenden wir Maus embryonale Stammzellen (mES), die spontan einen Marker der frühen Differenzierung Brachyury (Tbra)21,22ausdrücken. Obwohl keine scheinbare räumliche Anordnung visuell nachweisbar ist, ermöglicht die Methode die Erstellung einer Karte der bevorzugten Positionierung von T+-Zellen in Kolonien. Wir zeigen auch, dass Tbra-Musterung im Gegensatz zum Fehlen einer bevorzugten Lokalisierung der Zellen, die Id1 exdrücken, eine direkte Auslesung des Knochenmorphogenetischen Proteins (BMP) Signalweg23. Wir diskutieren auch die aktuellen Einschränkungen der Methode und wie diese Technik an andere experimentelle Systeme angepasst werden kann.
Protokoll
ANMERKUNG: Eine Übersicht über die Methode finden Sie in Abbildung 1.
1. Maskendesign
- Entwerfen Sie die Fotomaske nach den richtlinien beschrieben in Azioune et al.18. Eine Referenz an den Software- und Maskenhersteller, der für diese Studie verwendet wird, finden Sie in der Tabelle der Materialien.
HINWEIS: Mehrere Geometrien, Größen oder Abstände zwischen Shapes können auf einer Maske erstellt werden. Die UV-Lampe kann eine 15 cm Fotomaske passen, die bis zu 49 verschiedene Designs enthalten kann (unter der Annahme von 2 cm x 2 cm Späne).
2. Mikromusterherstellungsverfahren
- Bereiten Sie die notwendigen Materialien vor.
- Bereiten Sie eine Lösung von 0,1% Poloxamer 407 (10 mg für 10 ml) in Phosphat gepufferter Saline (PBS) vor und lassen Sie ihn bei Raumtemperatur auf einem Shaker ab. Der Poloxamer 407 dauert ca. 20 min, um sich aufzulösen.
- Lay Laborfilm (siehe Tabelle der Materialien) in der Unterseite einer 10 cm quadratischen Petrischale. Dies wird als Kammer für die Matrixabscheidung verwendet.
- Reinigen Sie die Oberfläche der Photomaske, zuerst mit 100% Aceton, dann mit 100% Isopropanol und schließlich mit ddH2O. Wenn möglich, trocknen Sie die Fotomaske an die Luft oder trocknen Sie die Maske auf andere Weise mit sauberem Papiertuch.
- Bereiten Sie ein quadratisches, starres und undurchsichtiges Stück Kunststoff mit der exakt gleichen Größe wie die Fotomaske (später als "Halter" bezeichnet).
HINWEIS: Dies wird verwendet, um Kunststoffabdeckungen in Kontakt mit der Fotomaske während des Beleuchtungsschritts zu halten. - Schalten Sie die UVO-Lampe ein und führen Sie eine wärmende Beleuchtung für 10 min aus.
- Erstellen Sie fotomusterte Chips.
HINWEIS: Es ist möglich, das Verfahren anzupassen, um Chips jeder gewünschten Größe zu erstellen. Der Einfachheit halber beschreiben wir hier das Verfahren zur Erzeugung eines 12 mm runden Mikromusterchips.- Schneiden Sie mit einem 12-mm-Lochloch hydrophobe Kunststoffschlitten, um 12 mm runde Abdeckungen zu erstellen und in eine saubere neue Petrischale zu legen.
VORSICHT: Verwenden Sie jederzeit Handschuhe, um Hautkontakt mit der Oberfläche des Kunststoffs zu vermeiden, da dies die Oberflächenbehandlung beschädigen kann. - Entfernen Sie den Schutzfilm vorsichtig mit einer Pinzette von den Abdeckungen.
HINWEIS: Vermeiden Sie eine Beschädigung der Kunststoffoberfläche, da dies die Platzierung der Zellen auf dem Chip während des Aussaatvorgangs beeinflussen kann (Abschnitt 3). - Legen Sie die Fotomaske auf eine saubere und stabile Oberfläche (z. B. Fotomaskenbox), Chromseite nach oben, und fügen Sie an der Position des gewünschten Chipdesigns einen Tropfen ddH2O von 2 l hinzu.
- Legen Sie einen Deckelrutsch auf den Tropfen von ddH2O und drücken Sie vorsichtig.
HINWEIS: Stellen Sie sicher, dass die Kunststoffseite, die der Fotomaske zusehen ist, die Seite ist, die durch die Folie geschützt wurde, die im vorherigen Schritt entfernt wurde. - Legen Sie den Halter auf die Kunststoffschlitten und fixieren Sie dieses Sandwich sorgfältig mit Klemmen, um die Kunststoffteile in Kontakt mit der Fotomaske zu halten.
HINWEIS: Stellen Sie die Klemmen so nah wie möglich an die Position der Kunststoffschlitten, um sicherzustellen, dass die Kunststoffschlitten perfekt in Kontakt mit der Oberfläche der Fotomaske gehalten werden. - Legen Sie die Baugruppe in der UVO-Lampe ca. 2 cm von der Lichtquelle ab und leuchten Sie 10 min.
HINWEIS: Die Leistung des Lichts wird auf 6 mW/cm2 bei einer Wellenlänge von 254 nm geschätzt, wenn der Chip in einem Abstand von 2 cm von der Quelle platziert wird. - Halten Sie das Sandwich mit der Fotomaske an der Unterseite und entfernen Sie vorsichtig die Klemmen, während Sie den Druck mit einer Hand beibehalten, um zu verhindern, dass sich die Dias beim Zerlegen des Sandwiches bewegen. Entfernen Sie den Halter, um sicherzustellen, dass sich alle Kunststoffteile noch auf der Maske befinden und nicht am Halter kleben.
- Fügen Sie ddH2O auf die Chips und lösen Sie die Chips vorsichtig von der Fotomaske.
HINWEIS: Wenn der Kunststoffchip an der Fotomaske klebt, lösen Sie den Chip mit einer Kunststoff-Pipettespitze, um den Chip zu drücken, während Sie eine Pinzette leicht über dem Chip halten, falls sich der Chip plötzlich löst. - Platzieren Sie schließlich die fotogemusterten Späne in der Matrix-Depositionskammer.
HINWEIS: Stellen Sie sicher, dass die beleuchtete Seite des Chips nach oben zeigt.
- Schneiden Sie mit einem 12-mm-Lochloch hydrophobe Kunststoffschlitten, um 12 mm runde Abdeckungen zu erstellen und in eine saubere neue Petrischale zu legen.
- Hinterlegen Sie die Matrix.
HINWEIS: Alle Eingriffe in diesem Abschnitt sollten in einer Gewebekulturhaube durchgeführt werden.- Filtern Sie die Poloxamer 407-Lösung durch einen 0,22 m Polyethersulfon (PES)-Filter.
- Bereiten Sie die ECM-Beschichtungslösung vor, indem Sie 500 g/ml sterilgefilterten Poloxamer 407 und 1 mg/ml Gelatine mischen.
HINWEIS: Weitere Informationen zu anderen möglichen ECM-Molekülen finden Sie auch in Tabelle 1. - Fügen Sie jedem beleuchteten Chip 200 l der Beschichtungslösung hinzu. Der Laborfilm verhindert, dass der Tropfen außerhalb des Chips fällt.
- Fügen Sie eine 3 cm Petrischale gefüllt mit ddH2O hinzu, um die Verdunstung zu begrenzen und mit dem Chip über Nacht bei 4 °C zu platzieren.
3. Seeding-Verfahren
HINWEIS: Die unten beschriebenen Schritte wurden für embryonale CGR8-Mausstammzellen (mESC)24 mit Standard-mESC-Medium optimiert (siehe auch Materialtabelle). Grundsätzlich ist es jedoch möglich, das Verfahren für jeden Zelltyp anzupassen. Beachten Sie auch, dass die konventionelle Zellkultur von embryonalen Stammzellen der Maus hier nicht beschrieben wird, da eine umfangreiche Dokumentation an anderer Stelle zu finden ist25.
- Die Beschichtungslösung ansaugen und die Chips zweimal für mindestens 5 min mit sterilem PBS inkubieren.
- In der Zwischenzeit eine Zellsuspension von 5,5 x 105 Zellen/ml in warmem Medium vorbereiten.
- Pipetten Sie 200 l Zellsuspension auf jeden Chip (ca. 100.000 Zellen/cm2).
- Schließen Sie die Säkammer und lassen Sie die Zellen für 1 h im Inkubator haften.
- Nach 1 h die Brunnen einer Mehrbrunnenplatte (4-well- oder 24-Well-Platte je nach Anzahl der Chips) mit 500 l/Well warmes Medium füllen und die Chips mit steriler Pinzette in die Platte geben.
- Schütteln Sie die Platte kräftig, um nicht anhaftende Zellen zu lösen. Das Medium ansaugen und sofort durch frisches warmes Medium ersetzen. Überprüfen Sie unter dem Mikroskop, ob die Musterung sichtbar ist (Abbildung 2a).
HINWEIS: Die Haftzeit muss möglicherweise bei der Verwendung anderer Zelllinien als mESC oder anderer Matrixproteine optimiert werden (siehe auch Tabelle 2). - Wiederholen Sie diesen Schritt, bis Muster deutlich sichtbar werden, wie in Abbildung 2adargestellt.
HINWEIS: Dieser Schritt ist entscheidend und bestimmt den Erfolg des Verfahrens. Ein zu intensives Waschverfahren kann die Zellen ablösen, im Gegensatz dazu kann unzureichendes Waschen dazu führen, dass Zellen zwischen den Mustern verbunden bleiben (Abbildung 2c,d).
4. Fixierung
HINWEIS: Nach 48 h in der Kultur sollten die Zellen dichte Kolonien bilden, die streng der Form der Muster folgen (wie in Abbildung 2bdargestellt).
- Lassen Sie die Späne in der Platte, entfernen Sie 90% des Mediums, so dass gerade genug Medium, um zu verhindern, dass die Späne trocknen.
HINWEIS: Es ist wichtig, dass der Chip nie trocknet, um Flecken zu vermeiden und eine Zellablösung von der Oberfläche zu verhindern. Aufgrund der Hydrophobie der Spanoberfläche zwischen den Klebstoffmustern kann der Chip eine Neigung zur Entnässe nässen. In diesem Stadium kann das Fixativ dazu führen, dass sich große gewölbte Kolonien vom Chip lösen. Washes sollte sehr sanft sein, idealerweise durch Pipettieren von Flüssigkeit auf der Seite des Brunnens und nicht direkt auf den Chip durchgeführt. - Fügen Sie pro Bohrplatz mindestens 500 l Paraformaldehyd (PFA)-basierte Fixierungslösung hinzu und inkubieren Sie 10 min.
HINWEIS: Wenn Kolonien besonders dick erscheinen (mehr als 5 Zellschichten), kann es notwendig sein, die Fixierungszeit auf 20 min einzustellen. - Nach der Fixierung 3 Mal mit der Waschlösung waschen (PBS mit 0,01% Poloxamer 407). Eine zusätzliche Wäsche mit 50 mM NH4Cl in Waschlösung verdünnt kann interkaliert werden, um Rest PFA Vernetzung Aktivität zu löschen.
- Inkubieren Sie die Proben für mindestens 30 min in Blockierlösung.
HINWEIS: In diesem Stadium können Proben etwa eine Woche vor der Färbung bei 4 °C gelagert werden. Wenn ja, versiegeln Sie die Platte mit Laborfolie, um Verdunstung zu verhindern.
5. Immunostaining
- Bereiten Sie eine Färbekammer vor, indem Sie ein Blatt Laborfolie in den Boden einer 10 cm quadratischen Petrischale legen.
- Bereiten Sie Antikörperlösungen vor (siehe Tabelle 3 für eine Liste der in diesem Artikel verwendeten Antikörper und Verdünnungen).
- Legen Sie den Chip in die Färbekammer mit der Seite, die die nach oben gerichteten Zellen stützt, und fügen Sie sofort 100 l primäre Antikörperlösung auf den Chip.
VORSICHT: In diesem Stadium sollten Späne nicht einfach entnässen wie in Schritt 4.1. Dennoch sollte darauf geachtet werden, dass die Späne nicht trocknen. Wenn mehrere Chips verarbeitet werden müssen, wenden Sie Schritt 5.3 auf jeden Chip sequenziell an. - 1 h auf einer rotierenden Plattform bei Raumtemperatur inkubieren.
HINWEIS: Wenn die Zellen große 3D-Strukturen gebildet haben, kann eine längere Inkubationszeit erforderlich sein, um eine gleichmäßige Färbung der Probe zu ermöglichen. Die Inkubationszeit kann bis zu 24 h erhöht werden. Die Färbekammer muss jedoch eine 3 cm große, mit Wasser gefüllte Schale enthalten und die Färbekammer muss mit Laborfolie versiegelt werden, um verdunsten zu verhindern. - Die Chips in eine frische Multiwell-Platte geben und 3 Mal mit der Waschlösung waschen.
- Inkubation mit sekundären Antikörpern durchführen, wie in Schritt 5.3 und 5.4 beschrieben.
- Montieren Sie den Chip auf einem Mikroskopschlitten mit 20 L eines beliebigen Standard-Montagemediums (z. B. Mowiol).
6. Bildgebung
HINWEIS: Die Bildgebung kann mit einem Standard-Konfokalmikroskop durchgeführt werden. Hier geben wir nur Empfehlungen, um eine Bildqualität zu gewährleisten, die für die anschließende quantitative Analyse ausreicht.
VORSICHT: Um eine Verzerrung des Betreibers zu vermeiden, sollten die Kolonien zum Abbild nur mit dem nuklearen Umschlagsignal ausgewählt werden (um zu sehen, ob eine Kolonie der Form des Musters richtig folgt). Vermeiden Sie es, das Signal von Markern von Interesse zu überprüfen, außer beim Anpassen der Mikroskopeinstellungen.
- Stellen Sie sicher, dass die Erfassungsbittiefe 12 oder 16 Bit beträgt.
- Identifizieren Sie die entsprechenden Einstellungen, um den Dynamikbereich für jeden Bildkanal zu maximieren. Vermeiden Sie insbesondere Bildausschnitte.
- Fügen Sie einen Kanal ein, um die Mikromuster-Autofluoreszenz abzubilden (siehe Abbildung 3).
- Passen Sie die Bildgröße und den Zoomfaktor an, um Voxelgrößen zwischen 0,1 und 0,6 m in den x- und y-Achsen und zwischen 0,2 und 2 m in der Z-Achse zu erhalten.
HINWEIS: In dieser Studie haben wir beispielsweise ein invertiertes Raster-Konfokalmikroskop mit einem 40-fachen Objektiv (numerische Blende gleich 1,3), einer Bildgröße von 1024 x 1024 Pixeln ohne digitaler Zoom und einer Z-Schritt-Größe von 0,5 m verwendet. Daraus ergab sich eine Voxelgröße von 0,38 x 0,38 x 0,5 m. - Definieren Sie für jede Kolonie die minimale und maximale Position entlang der Z-Achse, um sicherzustellen, dass die gesamte Kolonie erworben wird. Mindestens ein Flugzeug mit niedrigem bis keinem Signal sollte unterhalb und oberhalb der Kolonie enthalten sein.
- Stellen Sie sicher, dass Z-Stack-Ausrichtungen konsistent erfasst werden (entweder immer von oben nach unten oder immer von unten nach oben)
- Passen Sie die Scangeschwindigkeit, die Bildauflösung, die Bildmittelung und die Detektorgewinne an, um ein Optimum zwischen Bildqualität und Bildzeit zu ermitteln. Als Indikation betrug die Bildzeit für eine Kolonie, wie in Abbildung 3 dargestellt, etwa 2–3 min. Führen Sie die Bildaufnahme durch.
VORSICHT: Alle Bilder müssen, um vergleichbar zu sein, mit demselben Mikroskop mit den gleichen Objektiv- und Erfassungseinstellungen aufgenommen werden. - Speichern Sie am Ende der Erfassung alle Bilder, und weisen Sie eine eindeutige Namenskonvention zu, um die experimentelle Bedingung zu identifizieren, die jedes Bild darstellt.
HINWEIS: Siehe Abbildung 4 als Beispiel, diese Konvention wird später während des Analyseverfahrens verwendet. Beachten Sie, dass Bilder in jedem Format gespeichert werden können, das von BioFormats 26unterstützt wird. Wenn Kolonien größer als das Sichtfeld sind, kann das Näh-Plugin von ImageJ verwendet werden27. Beachten Sie auch, dass im Falle von Nähten, Beleuchtung Roll-off-Korrektur erforderlich sein kann.
7. Bildanalyse
HINWEIS: Die empfohlenen Computerspezifikationen für dieses Verfahren sind: 16 GB RAM, eine Multi-Core-CPU mit 3,33 GHz und mindestens 50 GB Speicherplatz (oder mehr abhängig von der Anzahl der chips, die abgebildet wurden). Die Software wurde unter Linux, Windows und MacOS getestet. PickCells ist eine plattformübergreifende Bildanalyseanwendung mit einer grafischen Benutzeroberfläche, die sich der Analyse der kollektiven Organisation der Zellen in komplexen mehrdimensionalen Bildern widmet (Blin et al., in Vorbereitung). Bitte beachten Sie, dass weitere Informationen zu PickCells sowie Dokumentation zu den hier genannten Modulen online zu finden sind: https://pickcellslab.frama.io/docs/. Beachten Sie auch, dass die Schnittstelle änderungen kann, während wir die Software ständig verbessern. Wenn sich die Benutzeroberfläche von der Abbildung oder dem Video unterscheidet, lesen Sie bitte das Online-Handbuch.
- Installieren und führen Sie PickCells entsprechend der online verfügbaren Dokumentation aus.
- Importieren Sie Bilder und überprüfen Sie die Richtigkeit der bereitgestellten Informationen (Abbildung 4-1).
- Dokumentieren Sie den Namen der einzelnen Kanäle.
- Segmentkerne, die auf dem Kernhüllensignal basieren, mit dem Nessys-Modul28 (Abbildung 4-2) und stellen ein Präfix (z. B. Nuklei) zur Verfügung, mit dem die generierten segmentierten Bilder bezeichnet werden.
HINWEIS: Dokumentationen zu Nutzungs- und Parameteranpassungen finden Sie unter https://framagit.org/pickcellslab/nessys. - Bei Bedarf Segmentierungen überprüfen und bearbeiten Sie sie mit dem Segmentierungseditormodul (Abbildung 4-3)
HINWEIS: Wenn der Segmentierungsprozess aus irgendeinem Grund keine zufriedenstellenden Ergebnisse lieferte, löschen Sie manuell Bilder im Datenbankordner und auch den Knoten "Segmentierungsergebnis" in der MetaModel-Ansicht. Wiederholen Sie dann Schritt 7.4 und 7.5. Wenn die Segmentierung nur einer kleinen Teilmenge von Bildern keine zufriedenstellenden Ergebnisse lieferte, verwenden Sie die eigenständige Nessys-Anwendung (siehe Link in 7.4), versuchen Sie die Segmentierung auf den "fehlerhaften Bildern" und ersetzen Sie die entsprechende Datei im Datenbankordner). - Segmentieren des Muster-Autofluoreszenzsignals mit dem Basissegmentierungsmodul (Abbildung 4-4)
- Geben Sie ein Präfix (z. B. "Muster" an), das zum Benennen der generierten segmentierten Bilder verwendet wird.
- Wählen Sie den Kanal aus, der das Autofluoreszenzsignal enthält.
- Anwenden von Rauschunterdrückung; In der Regel mit einem Gaußschen Filter mit einer Kernelgröße von 10 x 10 x 0,5 Voxel liefert zufriedenstellende Ergebnisse.
- Legen Sie den unteren Schwellenwert so fest, dass der Hintergrund blau angezeigt wird, während der Vordergrund weiß erscheint. Legen Sie auch den oberen Schwellenwert auf den Maximalwert fest, um zu vermeiden, dass hohe Intensitäten vom Endergebnis ausgeschlossen werden (rote Bereiche).
- Wählen Sie überspringen für den letzten Schritt aus.
- Klicken Sie auf Fertigstellen und warten Sie, bis alle Bilder verarbeitet sind.
- Was die Kerne betrifft, so können die Segmentierungsergebnisse nun mit dem Segmentierungseditormodul (Abbildung4-5) visuell überprüft und ggf. korrigiert werden.
- Erstellen Sie Kernobjekte und berechnen Sie grundlegende Objekt-Features.
- Starten Sie das Modul für systeminterne Features in der Taskleiste auf der linken Seite der Hauptschnittstelle (Abbildung 4-6).
- Schließen Sie die Bedienfelder Ellipsoid Fitter und Surface Extractor, um nur das Bedienfeld "Grundlegende Funktionen" offen zu halten.
- Wählen Sie Nucleus als Objekttyp und wählen Sie das Präfix in Schritt 7.4 für "segmentierte Bilder".
- Drücken Sie Compute und warten Sie, bis alle Bilder verarbeitet wurden.
HINWEIS: Nach diesem Schritt ist es nicht mehr möglich, die Kernsegmentierungen erneut zu bearbeiten.
- Erstellen Sie Musterobjekte, und berechnen Sie grundlegende Objekt-Features. Wiederholen Sie die Schritte 7.8.1 bis 7.8.4, nur dieses Mal wählen Sie Benutzerdefinierter Typ als Objekttyp und das Präfix in Schritt 7.6.1 für segmentierte Bilder.
HINWEIS: Nach diesem Schritt ist es nicht mehr möglich, die Mustersegmentierungen erneut zu bearbeiten. - Speichern Sie den Namen des Bildes, zu dem jeder Kern als Kernattribut gehört.
- Klicken Sie auf Daten > Neues Attribut und wählen Sie Nucleus im Popup-Dialog aus und klicken Sie auf Ok.
- Wählen Sie Daten von anderen Objekten sammeln aus, die mit dem Knoten verbunden sind, und klicken Sie auf Weiter.
- Wählen Sie im linken Bereich Bild aus, und doppelklicken Sie dann auf das Verhörzeichen unter dem Finish-Flag im Pfaddefinitionsfenster, um den Bildknoten als Ziel des Pfads festzulegen.
- Erweitern Sie den Bereich Verfügbare Attribute im linken Bereich, und wählen Sie das Name-Attribut aus.
- Erweitern Sie den Vorgangsbereich "Reduzierung", und wählen Sie "Einsabrufen" aus, klicken Sie dann auf die Schaltfläche "Ändern" und klicken Sie auf Weiter.
- Geben Sie "Bildname" ein, drücken Sie die Tabulatortaste und klicken Sie auf OK.
- Erstellen Sie ein Attribut "normalisierte Koordinate" in Kernobjekten (Abbildung 4-7).
- Passen Sie die Schritte 7.10.1 an 7.10.6 an, um die Koordinaten des Musterzentroids als Kernattribut zu speichern. Benennen Sie dieses neue Attribut "Pattern Coordinate".
- Klicken Sie dann auf Daten > Neues Attribut, wählen Sie Nucleus und klicken Sie auf Ok.
- Wählen Sie Eine Funktion zwischen räumlichen oder gerichteten Vektoren des Knotens definieren aus, und klicken Sie auf Weiter.
- Wählen Sie für Funktionstyp Arrayoperation aus, für V1 Elementvektor und dann Centroidaus, und für V2 wählen Sie Elementvektor und dann Pattern Coordinate aus.
- Klicken Sie auf Weiter, geben Sie "Normalisierte Koordinate" in das Feld Name ein, und klicken Sie auf Fertig stellen.
- Exportieren Sie die Daten in eine registerkartengetrennte Wertdatei.
8. R-Analyse
- Rstudio herunterladen und installieren.
HINWEIS: Softwareinformationen und Download-Links finden Sie unter https://www.rstudio.com/. - Laden Sie die für diese Analyse erforderlichen R-Skripte herunter.
HINWEIS: Skripte können aus dem GitLab-Repository heruntergeladen werden: https://framagit.org/pickcellslab/hexmapr. - Öffnen Sie Rstudio.
HINWEIS: Wenn Sie die Skripte zum ersten Mal ausführen, installieren Sie die erforderlichen R-Pakete (ggplot2 und scales). - Öffnen Sie in Rstudio die binnedmap_template. R-Skript.
- Legen Sie das Arbeitsverzeichnis auf den Speicherort der Quelldatei fest.
- Befolgen Sie die Anweisungen im Skript, um das Skript an ein bestimmtes Dataset anzupassen, um räumliche Karten zu erhalten, wie in Abbildung 5dargestellt.
- Führen Sie das Skript aus, um Dichtezuordnungen zu generieren.
Ergebnisse
Die hier beschriebene Fotomustermethode ermöglicht es, kultivierte Zellen präzise in Kolonien definierter Formen und Größen zu organisieren. Der Erfolg dieses Verfahrens sollte unmittelbar nach dem Zellsäverfahren (Schritt 3.7) deutlich erkennbar sein, da sich die haftenden Zellen entsprechend dem Fotomaskendesign wie in Abbildung 2azeigen. Bei 1 h nach der Zellaussaat sind einzelne Muster möglicherweise nicht vollständig konfluent (nur wenige Zellen pro Muster), jedoch werden die Muster mit nur sehr wenigen Zellen außerhalb der Klebeflächen vollständig kolonisiert (Abbildung 2b). Das genaue Erscheinungsbild der Kultur ist zelllinienabhängig. Zum Beispiel bilden mESC gewölbte Kolonien10. Ein Chip, bei dem die Musterung 1 bis 2 h nach dem Aussäen der Zelle nicht klar ist, weist auf einen Fehler der Prozedur hin (Abbildung 2c,d).
Große und dicke Kolonien können manchmal schwierig sein, homogen zu färben. Wir empfehlen, die Zellen in einem Schritt zu fixieren und zu permeabilisieren (Abschnitt 4), da dies die Antikörperdurchdringung verbessern kann29. Aus diesem Grund enthält die gewählte Fixierlösung ein Waschmittel. Abbildung 3 zeigt das Fluoreszenzsignal, das nach der Immunfärbung erwartet wird. Beachten Sie, dass helle Id1-positive Zellen in dichten Regionen (helle NPC-Regionen) der Kolonien gefunden werden (Abbildung 3A). Hinweise wie diese sind nützlich, um die Qualität des Antikörperfärbeverfahrens zu bewerten. Beachten Sie auch, dass die Mikromuster, die mit der vorliegenden Technik erstellt wurden, autofluoreszierend sind. Dieses Signal (Abbildung3A, B links die meisten Bilder) ist während der Analysephase nützlich, um Kolonien räumlich miteinander zu registrieren und die in Abbildung 5dargestellten Ergebnisse zu erstellen. Das Autofluoreszenzsignal ist in der Regel das hellste, wenn die Probe mit einem 405 nm Laser angeregt wird und dieser Kanal sollte zu diesem Zweck ohne Färbung belassen werden. Abbildung 3 zeigt auch, wie die Zellen genau auf Muster verschiedener Formen beschränkt sind.
Die Analyse von Bildgebungsdaten erfolgt in PickCells, einer kostenlosen und Open-Source-Software, die in unserem Labor entwickelt wurde (Blin et al., in Vorbereitung). Diese Software enthält die Bildanalysemodule zum Lesen und Sortieren konfokaler Bilder (Abbildung 4-1), zum Segmentieren (Abbildung 4-2,4-4) und zur Kuratierung segmentierter Objekte (Abbildung 4-3,4-5), um Objekte zu berechnen. Wie Koordinaten oder durchschnittse Intensität (Abbildung 4-6) und zum Exportieren der Daten (Abbildung 4-7,4-8). Wichtig ist, dass wir eine robuste nukleare Segmentierungsmethode namens Nessys28 entwickelt haben, die sich besonders für dichte und heterogene Populationen von Zellen eignet, wie Zellen, die auf Mikromustern angebaut werden (Abbildung 3). Abbildung 4 -2 zeigt eine repräsentative Ausgabe des Nessys-Moduls, bei der jede einzelne Zelle genau eine eindeutige Farbidentität erhält. Nur eine minimale Bearbeitung sollte notwendig sein, aber die Bearbeitung ist machbar, sollte sich der Benutzer so entscheiden (Abbildung 4-3). Schließlich bietet PickCells eine Reihe von Visualisierungsmodulen, um die Daten zu visualisieren. Ein Beispiel ist in Abbildung 4-6angegeben: ein ringförmiges Kolonie wird in 3D gerendert, wo Kerne entsprechend ihrer Position entlang der Z-Achse farbkodiert sind. Nachdem die Analyse in PickCells validiert wurde, können Daten exportiert werden, um die räumlichen Karten in R mit den unter (https://framagit.org/pickcellslab/hexmapr) verfügbaren Skripts zu erstellen, wie in Abbildung 530dargestellt.
Wir haben vor kurzem gezeigt, dass die räumliche Einschließung von mESC auf kleine (30.000 m2) Disc- oder Ellipsenmikromuster die Musterung einer Subpopulation von Zellen leitet, die den mesodermalen Marker Tbra10exezieren. Um unsere Methode hier zu veranschaulichen, fragen wir, ob die Musterung von Tbra durch BMP-Signalisierung in größeren Kolonien beeinflusst werden kann (90.000 m2). Abbildung 5A zeigt, dass Tbra+-Zellen bevorzugt auf die Peripherie des Musters beschränkt sind (Tbra+-Dichtekarte), wobei die lokale Zelldichte die niedrigste ist (siehe die blaue Karte links in Abbildung 5A). ). Diese Musterung von Tbra wird durch die Karte der mittleren Tbra-Intensität bestätigt.
Diese Daten zeigen, dass die Methode subvisuelle Informationen offenbaren kann. Tatsächlich reicht die visuelle Inspektion einer Kolonie aus Abbildung 3nicht aus, um irgendeine Form der räumlichen Organisation im Tbra-Ausdruck zu identifizieren. Dies erklärt sich insbesondere durch die wichtige Kolonie-Kolonie-Variabilität, die quantifiziert und im rechten Panel von Abbildung 5Adargestellt wird.
Die Technik zeigt auch, dass keine nachweisbare Musterung für Id1 (ein Ziel der BMP-Signalisierung) vorhanden ist, was darauf hindeuten kann, dass die T-Musterung in diesem Zusammenhang nicht durch BMP-Signalisierung gesteuert wird.
Micropatterning ermöglicht es, Kolonien zu zwingen, fast jede gewünschte Geometrie zu übernehmen. Dies ist besonders nützlich, um zu befragen, wie das System auf verschiedene Geometrien reagiert. Zum Beispiel können wir begründen, dass, wenn sich ein Morphogengradient in der Mitte der Kolonie aufbaut, die Schaffung eines Lochs in der Kolonie diesen Gradienten stören würde. Interessanterweise beobachten wir immer noch Muster auf einem Ringmikromuster, wenn auch in einer weniger robusten Weise (Abbildung 5B).
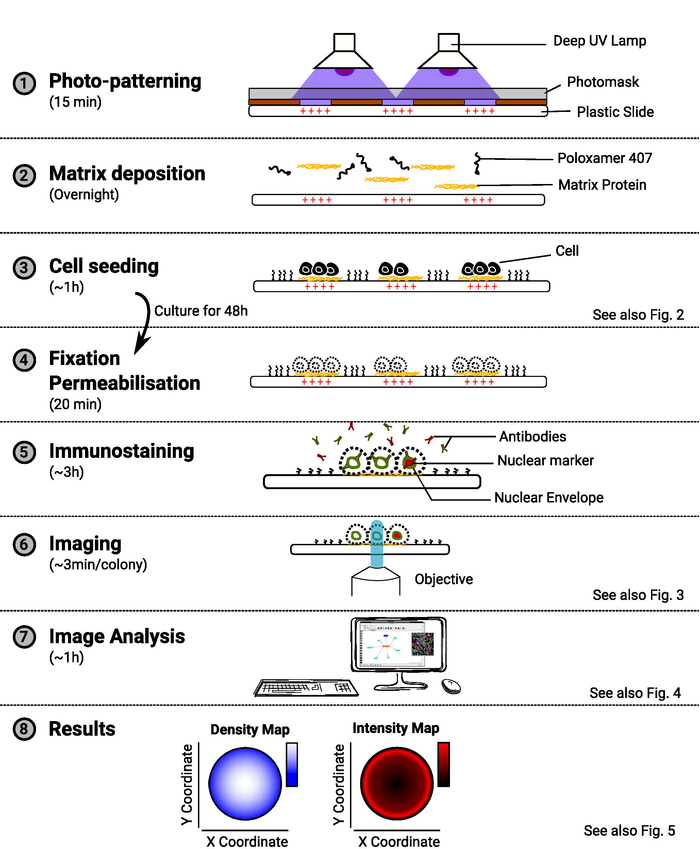
Abbildung 1: Übersicht über die Methode. Das Diagramm mit den wichtigsten Schritten der Methode. Für jeden Schritt wird die geschätzte Zeitunterweise unter dem Namen der Aufgabe angegeben, und ein Schaltplan veranschaulicht den Zweck des Verfahrens. Ein Hinweis auf eine relevante Zahl wird ebenfalls bereitgestellt, wenn verfügbar ist. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
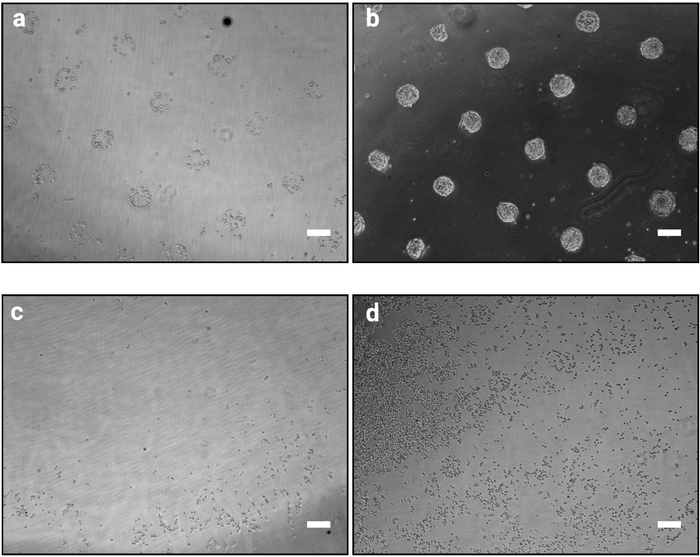
Abbildung 2: Kulturbild 1 h und 48 h nach dem Aussäen der Zellen auf Mikromustern. Hellfeldbilder von mESC, die auf Mikromustern gesät wurden. (a) Erwartete Zellorganisation 1 h nach dem Aussaat, Muster sollten klar identifizierbar sein. (b) Erwartetes Ergebnis nach 48 h Der Kulturen. mESC haben sich vermehrt und sind immer noch streng auf die Musterformen beschränkt. (c–d) Mögliche nicht-optimale Ergebnisse, entweder sehr wenige Zellen haften auf den Kunststoff außer an der Peripherie des Dias (c) oder Zellen haften zwischen den Mustern (d). Eine Anleitung zur Fehlerbehebung finden Sie in Tabelle 2. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
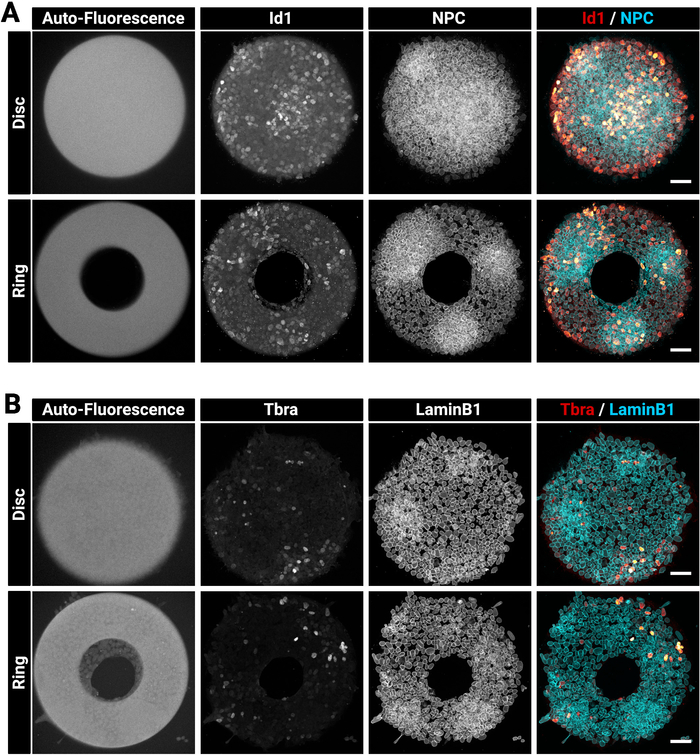
Abbildung 3: Repräsentative konfokale Bilder von immunbefleckten Kolonien, die auf Mikromustern angebaut werden.
Repräsentative mESC-Kolonien nach Immunfluoreszenz für (A) Id1 und Nuclear Pore Complex oder (B) Tbra und LaminB1. Für jede Färbung wird eine Kolonie gezeigt, die auf einem Scheibenmikromuster und eine Kolonie, die auf einem Ringmikromuster angebaut wird, angebaut wird. Einzelne Kanäle werden als Graustufenbilder bereitgestellt. Beachten Sie das klare Autofluoreszenzsignal des Mikromusters (405 nm Laseranregung). Die Maßstabsleiste steht für 50 m. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
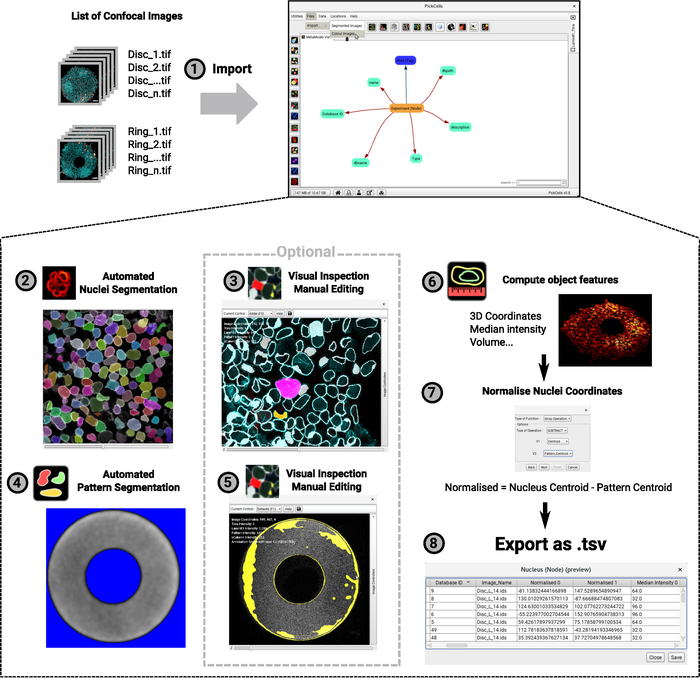
Abbildung 4: Flussdiagramm des Bildanalyseverfahrens. Eine Liste von konfokalen 3D-Bildern wird zur Analyse in PickCells importiert (1). Dieses Beispiel zeigt ein Experiment mit zwei unterschiedlichen Formen (Scheiben und Ringe wie in Abbildung 2). Die Bildbenennungskonvention wird auf der linken Seite und die PickCells-Schnittstelle auf der rechten Seite angezeigt. Dann wird das Nessys-Modul verwendet, um Kerne automatisch zu segmentieren (2). Im Screenshot erhält jeder einzelne Kern eine eindeutige Farbe, die eine genaue Segmentierung anzeigt. Die Autofluoreszenz des Musters wird dieses Mal auch mit dem Modul "Basissegmentierung" (4) segmentiert. Hintergrund wird in blau und weiß Signal wird als Musterform definiert werden. Segmentierte Shapes werden dann visuell überprüft, um eine genaue Segmentierung zu gewährleisten, und bei Bedarf mithilfe des Segmentierungs-Editormoduls (3–5)bearbeitet. Die Screenshots zeigen die Umrisse der erkannten Shapes. Die rosa und gelben Formen wurden bearbeitet. Schließlich werden Objekt-Features berechnet und in eine Datei exportiert, die später in R (6–7)verarbeitet werden soll. Ein Screenshot einer Kolonie, die als 3D-Ansicht gerendert wird, wird bereitgestellt (6). Für die Schritte 2 bis 6 werden die Symbole, die zum Zeitpunkt des Schreibens dieses Artikels in der PickCells-Schnittstelle gefunden wurden, neben dem Schrittindex angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
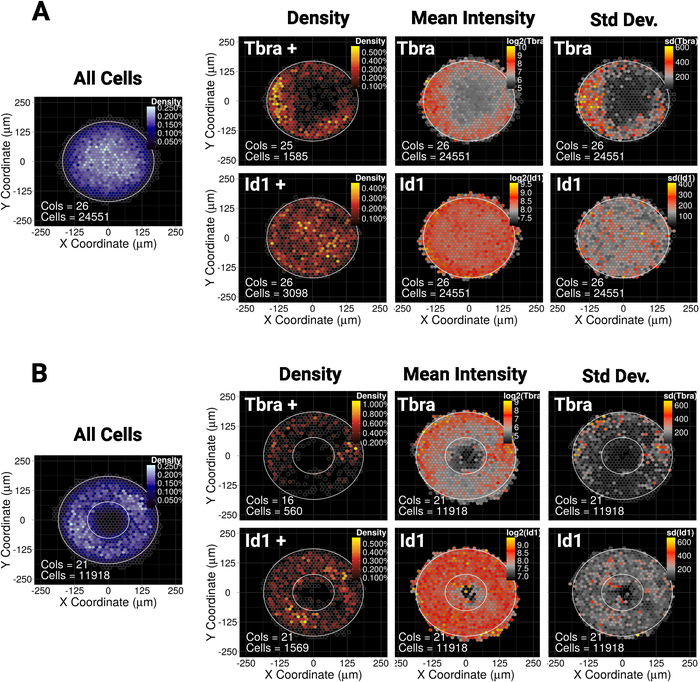
Abbildung 5: Repräsentative Ergebnisse für zwei verschiedene Transkriptionsfaktoren und Mikromusterformen
Binned Raumkarte für mESC gewachsen für 48 h auf (A) Scheiben geformte Mikromuster oder (B) ringförmige Mikromuster. Für jede Mikromusterform wird die Karte der Zelldichte, unabhängig vom Zellphänotyp, auf der linken Seite mit einer blauen Farbskala dargestellt. Dann werden für jeden Marker (Tbra in der oberen Zeile und Id1 in der unteren Zeile) drei verschiedene Karten bereitgestellt, von links nach rechts: Zelldichtekarte nur von Marker-Exsetierenden Zellen (schwellenbasierte Analyse), die Karte der durchschnittlichen Markerintensität (log2) und die Karte des Standardabweichung der Markerintensität. Intensitäten werden als beliebige Fluoreszenzeinheiten angegeben. Für jede Karte wird die Mikromusterform als weißer Umriss angegeben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| ECM-Typ | gelatine | Fibronektin | Basement Membrane Matrix | |
| konzentration | ECM-Konzentration | 1 mg/ml | 20 g/ml | 200 g/ml |
| Poloxamer 407 Konzentration | 500 g/ml | 400 g/ml | 1 mg/ml | |
| Getestet mit | mESC | ja | ja | ja |
| mEpiSC | Nein | ja | ja | |
| Serumfreies Medium | Nein | ja | ja |
Tabelle 1: Getestete Konzentrationen von Poloxamer 407 und ECM. Diese Tabelle gibt einen Überblick über die Konzentrationen von ECM und Poloxamer 407, die wir in unserem Labor getestet haben. Für jede ECM/Poloxamer 407-Kombination wird der Zelltyp, für den die Musterung erfolgreich durchgeführt wurde, sowie gezeigt, ob die Kultur Serum enthielt oder nicht. mESC = mausembryonale Stammzelle, mEpiSC = Maus-Epiblast-Stammzelle.
| verfahren | beobachtung | Mögliches Problem | lösung |
| Mikromusterung | Niedrige Zellanhaftung | ungeeignetes ECM/Poloxamer 307 Konzentrationsverhältnis | Erhöhung des Konzentrationsverhältnisses ECM/Poloxamer 307 |
| Zellanhaftungszeit zu kurz | Erhöhen Sie die Inkubationszeit, um den Zellen genügend Zeit zu geben, um sich richtig an Muster zu halten (Schritt 3.4). Um diesen Schritt zu optimieren, kann die Überprüfung der Zellen unter dem Mikroskop helfen, eine Änderung der Zellmorphologie zu erkennen, die darauf hinweist, dass die Zellen begonnen haben, sich zu haften. | ||
| Zu intensiv wärst es (Schritt 3.6) | Vermeiden Sie beim Austausch des Mediums das Pipettiermedium direkt auf die Chips. Stattdessen pfeifen Sie das Medium vorsichtig an den Wänden des Brunnens | ||
| Zellen haften zwischen den Mustern | ungeeignetes ECM/Poloxamer 307 Konzentrationsverhältnis | Verringern des Konzentrationsverhältnisses ECM/Poloxamer 407 | |
| Zellanhaftungszeit zu lang | Verringern Sie die Inkubationszeit (Schritt 3.4). | ||
| Wähes unwirksam (Schritt 3.6) | Das kräftige Schütteln der Platte reicht in der Regel aus, um die Zellen im Übermaß zu lösen. Bei Zelltypen, die dazu neigen, stark am Chip zu haften, kann das Pipettieren direkt auf den Chip das Ergebnis verbessern. Die Erhöhung der Anzahl der Wass kann auch helfen, insbesondere um sicherzustellen, dass nach diesem Schritt keine Zelle im Medium schweben bleibt. | ||
| Zellen folgen nicht strikt der Musterform | "Inkompatibler" Zelltyp und Mustergeometrie | Planen/Entwerfen mehrerer Geometrien/Größen, die der Fotomaske hinzugefügt werden sollen, um die optimale Mustergröße für eine bestimmte Musterform und einen bestimmten Zelltyp testen und identifizieren zu können. Bitte lesen Sie den Abschnitt "Einschränkungen" in der Diskussion | |
| Nicht-optimale Foto-Musterung, kann dies durch Beobachtung der Schärfe des Autofluoreszenzsignals diagnostiziert werden. Mustergrenzen sollten scharf aussehen wie in Abb. 2. Wenn die Ränder der Muster verschwommen erscheinen, muss der Fotomusterschritt verbessert werden. | Verschwommene Musterkanten weisen darauf hin, dass der Kunststoffschlitten während des Beleuchtungsschritts nicht nah genug an der Oberfläche der Maske war. Stellen Sie sicher, dass die Teile, die die Dias an der Fotomaske halten, gleichmäßig sind und dass während des Beleuchtungsvorgangs ein konstanter und ausreichender Druck auf die Baugruppe ausgeübt wird. | ||
| Färbung | Nicht homogene Färbung | Antikörper-Inkubationszeit zu kurz | Erhöhung der Inkubationszeit von Antikörpern (bis zu 24h bei Raumtemperatur) |
| Kolonien während des Montagevorgangs abgeflacht | Montieren Sie Mikromusterschlitten in einer Kammer wie Chamlide oder Cytoo-Kammern, um sowohl Immunfärbung als auch Bildgebung durchzuführen, ohne dass die Zellen montiert werden müssen. Dies wird die Kolonien 3D-Struktur besser erhalten. | ||
| Ablösen von Kolonien während des Färbevorgangs | Entnässtung des Chips | Lassen Sie genügend Medium oder verwenden Sie 2 Pipetten, eine, um das Medium zu entfernen und die andere, um die frische Lösung hinzuzufügen | |
| Kolonien erscheinen unter dem Mikroskop | Kolonien wurden bei der Montage des Chips auf dem Mikroskop-Dia geerbt | Seien Sie sehr sanft bei der Montage der Chips. Alternativ können Mikromuster in einer Kammer wie Chamlide oder Cytoochambers montiert werden, um sowohl immunstainierend als auch bildgebungsbildn zu sein, ohne dass die Zellen montiert werden müssen. Dies bewahrt auch die Kolonie Ultrastruktur. |
Tabelle 2: Leitfaden zur Fehlerbehebung. Diese Tabelle gibt einen Überblick über mögliche suboptimale Ergebnisse. Die möglichen Ursachen der Probleme werden auch zusammen mit empfohlenen Lösungen aufgelistet.
Diskussion
Hier beschreiben wir eine Methode zur Analyse der entstehenden Musterung in Zellkulturen. Ein vereinfachter Mikromusteransatz wird verwendet, um die Form und Größe von Zellkolonien zu standardisieren, und wir präsentieren Bildanalyse-Tools und R-Skripte, die die Erkennung und Quantifizierung von Mustern innerhalb dieser Kolonien ermöglichen.
Die Pipeline, die wir vorschlagen, ähnelt in gewissem Maße mit einer zuvor veröffentlichten Methode31, bei der sich die Autoren auf die Kulturbedingungen konzentrieren, indem sie kommerziell erhältliche Mikromuster verwenden, um reproduzierbare Keimschichtbildung in ESC-Kolonien für die Untersuchung von frühen Gastrulationsereignissen in vitro. Unser Ziel ist es, eine verallgemeinerbare Pipeline für die Entdeckung der Musterbildung in vitro bereitzustellen, in der die kollektive Organisation der Zellen erst nach statistischer Analyse sichtbar wird. Aus diesem Grund bieten wir einen robusten Bildanalyse-Workflow, der die genaue Identifizierung und Analyse der Kernposition im 3D-Raum über mehrere Kolonien ermöglicht (siehe auch den Abschnitt "Vorteil und Einschränkungen der Mustererkennungsmethode" Diskussion). Wir haben auch beschlossen, einen einfachen internen Mikromusteransatz zu entwickeln, der langfristig eine flexiblere und kostengünstigere Alternative zu kommerziell erhältlichen Lösungen bietet, von denen wir hoffen, dass sie für die Gemeinschaft nützlich sein werden.
Schließlich stellen wir fest, dass während der Überarbeitung dieses Manuskripts ein neues Paket zur Analyse von Mustern in vitro ähnlich unseren R-Skripten veröffentlicht wurde32. Dieses neue Paket akzeptiert Tabellen mit Zell-Features als Eingabe, die von Bildplattformen mit hohem Durchsatz abgerufen werden kann. Wir glauben, dass die Tabelle der Kernfunktionen, die in Schritt 7 unseres Protokolls erzeugt wurden, im Prinzip als Input zu diesem neuen Paket dienen könnte, obwohl wir diese Möglichkeit nicht selbst getestet haben.
Anpassungsfähigkeit der Methode an andere Zelltypen und Koloniegeometrien
Wir stellen diesen Ansatz im Zusammenhang mit der Untersuchung der Entstehung von mesodermalen Transkriptionsfaktoren in Kulturen pluripotenter Zellen in Gegenwart von Serum dar. Das Verfahren ist jedoch leicht an andere Zelltypen und an serumfreie Kulturen anpassbar, auch wenn es notwendig sein kann, die Konzentrationen von ECM/Poloxamer 407 zu optimieren (siehe Tabelle 1 für getestete Konzentrationen und Tabelle 2 für eine Fehlerbehebungsanleitung). Die Methode kann auch an größere oder kleinere Größen von Mikromustern und an eine breite Palette von Formen je nach den Bedürfnissen des Benutzers angepasst werden. Beim Festlegen der Methode ist es jedoch wichtig, sich bewusst zu sein, dass nicht alle Form-/Zelltypkombinationen optimal sind. Beispielsweise exprimiert mESC hohe Konzentrationen von E-Cadherin33,34, die es diesen Zellen ermöglichen, kollektive Strukturen zu bilden, die Bereiche ohne ECM umfassen. Diese Zellen folgen nicht streng Geometrien mit scharfen Winkeln oder enthalten kleine Löcher im Muster. Beachten Sie beispielsweise, dass die Zellen auf dem Ring von Abbildung 3Bgerade dabei sind, den zentralen Bereich zu kolonisieren. In unseren Händen zwang ein kleinerer zentraler Bereich mESC nicht, ringförmige Kolonien zu bilden. Es wird daher dringend empfohlen, bei der Gestaltung der Fotomaske eine Vielzahl von Geometrien einzubeziehen, um die optimalen Größen und Krümmungen testen und identifizieren zu können, die für den Zelltyp der Wahl geeignet sind.
Ein weiterer wichtiger Faktor, der zu berücksichtigen ist, ist die Länge des Experiments und die Proliferationsrate der Zellen. Für einige sich schnell ausbreitende Zelltypen (einschließlich pluripotenter Zellen) kann es schwierig sein, Zellen auf Mikromustern über viele Tage zu halten (für mESC sind drei Tage ein Maximum). Auch die Aussaat von Zellen auf Mikromustern tritt nicht immer optimal für jede Kolonie auf, so dass es ratsam ist, einen Überschuss an Kolonien auszusäen, um Ersatzteile zu haben.
Vorteile und Einschränkungen der Mustererkennungsmethode
Ein besonderer Vorteil der Methode ist die Fähigkeit, "gemittelte" Muster zu erkennen, indem Bildanalyseergebnisse aus mehreren Replizierungskolonien kombiniert werden (Abbildung 5). Dies kann Musterereignisse aufdecken, die bei der Inspektion einzelner Kolonien nicht ersichtlich sind. Ein Nachteil dieses "Mittelungsansatzes" besteht darin, dass er bestimmte Arten von sich wiederholenden Mustern vermissen kann, z. B. kleine Flecken oder schmale Streifen. Diese Mustertypen können jedoch stattdessen mit einer Kombination sorgfältig ausgewählter Mustergrößen8aufgedeckt werden. Außerdem bietet die hier beschriebene Bildanalyse-Pipeline quantitative Daten sowohl bei der Auflösung von Einzelzellen als auch bei der Kolonieauflösung, die die Möglichkeit bieten, den Grad der variabel envariabelen Variabilität zwischen den Kolonien zu untersuchen (Abbildung 5) oder eine Nachbarschaftsanalyse an mehreren Skalen10.
Ein weiterer wichtiger Vorteil der Mittelungsmethode besteht darin, dass sie die Möglichkeit bietet, die bevorzugte Position vieler Marker zu kartieren, ohne durch verfügbare Fluorophore von Detektionskanälen eingeschränkt zu werden. Obwohl wir in der hier vorgestellten Arbeit nur zwei Unterscheidungsmarker verwenden, ermöglicht es die Fähigkeit, Kolonien zu standardisieren und "gemittelte" Muster zu extrahieren, die Verteilungskarten aus verschiedenen Gruppen zusammen zu vergleichen, um die , um die verallgemeinerten räumlichen Beziehungen von Markern zueinander aufzudecken.
Obwohl unser Fokus hier auf der Untersuchung von Differenzierungsmarkern lag, kann die Analysemethode erweitert werden, um andere biologische Prozesse zu untersuchen, für die kerntechnische Marker zur Verfügung stehen. Beispielsweise würde die Mikromusterung einer Zelllinie, die einen Fluoreszenz-Ubiquitinationszellzyklusindikator35 (FUCCI) enthält, es ermöglichen zu untersuchen, wie die Geometrie auf Kolonieebene Zellzyklusereignisse in der Gruppe beeinflussen kann.
Zukünftige Richtungen
Die Methode ist für die Bildanalyse mit mittlerem Durchsatz geeignet, die Bildaufnahme ist jedoch derzeit nicht vollständig automatisiert und kann für sehr große Experimente eingeschränkt werden. Die regelmäßigen Arrangements von Kolonien sollten es ermöglichen, vollständig automatisierte Erfassungsroutinen zu schaffen, ähnlich dem, was für Einzelzellen im Durchschnitt20entwickelt wurde. Da jedoch die Größe des Feldes, das zum Abbilden einer Kolonie erforderlich ist, groß ist und möglicherweise Mosaiken erfordert, und da Kolonien dreidimensional sind, ist es sehr wünschenswert, sowohl die Datensatzgröße als auch die Erfassungszeit zu reduzieren, indem nur relevante Kolonien abgebildet werden. Daher könnten künftige Anstrengungen unternommen werden, um ein "intelligentes" Mikroskop zu entwickeln, das relevante Kolonien identifizieren und bildgebende Koordinaten an jede Probe anpassen kann. Dies wird nicht nur Zeit und Aufwand reduzieren, sondern auch potenzielle Voreingenommenheit der Betreiber verhindern.
Die Analysepipelines können auch effizienter gestaltet werden, indem die Anzahl der Schritte, die der Benutzer ausführen muss, reduziert wird. Wir haben Pläne, einen Pipeline-Building-Mechanismus zu bauen und R direkt in unsere Software zu integrieren (siehe auch Pickcells-api-3 und Pickcells-rjava-1 im Issue Tracker unserer Code-Repositorys [https://framagit.org/groups/pickcellslab/-/issues]). Die vollständige Automatisierung des Analyseverfahrens reduziert Zeit und Aufwand und begrenzt potenzielle Benutzerfehler.
Schließlich stellen wir fest, dass unsere Analysemethode die dynamische Natur der zellulären Musterung noch nicht vollständig erfasst. Einige begrenzte dynamische Informationen können extrahiert werden, indem eine Zeitreihe von Snapshot-Bildern8,10,36untersucht wird. Die Möglichkeit, die Geschichte der Zellpopulation aufzuzeichnen, ist jedoch sehr wünschenswert, wenn wir besser verstehen wollen, wie Muster entstehen. Eine Einschränkung ist, dass die genaue Verfolgung einzelner Zellen in einer 3D-dichte Zellpopulation eine sehr anspruchsvolle Aufgabe bleibt37. Unsere Zelldetektionsmethode verwendet die Kernhülle und schneidetbesonders gut bei dichten und überlappenden Zellpopulationen 28 ab. Live-Reporter der nuklearen Hülle sind leicht verfügbar28,38 und ein Vorteil der Mikromustertechnik ist, dass es verwendet werden kann, um zu verhindern, dass sich die Zellen während der Langzeitbildgebung außerhalb des Sichtfeldes bewegen. Insgesamt sind wir zuversichtlich, dass die automatisierte Nachverfolgung von Zellen mit einer Kombination von kürzlich etablierten Werkzeugen28,39,40 erreichbar sein wird und dass dies neue Einblicke in die grundlegenden Prinzipien der Selbstorganisation.
Offenlegungen
Die Autoren haben nichts zu verraten
Danksagungen
Diese Arbeit wurde durch ein Sir Henry Wellcome Post-Doctoral Fellowship (WT100133 to G.B.), ein Wellcome Trust Senior Fellowship (WT103789AIA to S.L.) und ein Wellcome Trust PhD Studentship to (108906/Z/15/Z to D.W.) finanziert. Wir danken auch Dr. Manuel Thery für seinen Rat zur Anpassung der Fotomustertechnik.
Materialien
| Name | Company | Catalog Number | Comments |
| 2-mercaptoethanol | Gibco | 31350-010 | handle in fume hood |
| 1× Master quartz anti-reflective chromium photomask | Toppan Photomask | Custom design | |
| 24-well plates | Corning | 3526 | |
| 4-well plates | Nunclon Delta | 176740 | |
| 5% Donkey Serum | Sigma | D9663 | |
| Anti Nuclear pore complex | Abcam | ab24609 | Mouse monoclonal, use 1:1000 |
| Anti-Id1 | Biocheck | 37-2 | Rabbit polyclonal, use 1:200 |
| Anti-Lamin B1 | Abcam | ab16048 | Rabbit polyclonal, use 1:1000 |
| Anti-Tbra | R&D | AF2085 | Goat ployclonal, use 1:400 |
| Blocking Solution | NA | NA | 0.1% TritonX-100 0.01% Pluronic F-127 5% Donkey Serum 0.003% Sodium Azide In PBS |
| CGR8 mESC | NA | NA | Reference: Mountford et al. PNAS, 1994 |
| Fixation solution | NA | NA | 4% PFA diluted in washing solution |
| Foetal bovine serum | Gibco | 10270-106 | Serum batches must be tested to ensure compatibility with ESC maintenance |
| Gelatin | Sigma | G1890 | |
| Glasgow Minimum Essential Medium | Sigma | G5154 | |
| Laboratory Film | VWR | 291-1212 | |
| Layout Editor Software | LayoutEditor | NA | https://layouteditor.com/ |
| Layout Editor Software | Klayout | NA | https://www.klayout.de/ |
| L-glutamine | Gibco | 25030-024 | |
| LIF | Millipore | ESG1107 | |
| MEM NEAA | Gibco | 11140-035 | |
| mESC Culture medium | NA | NA | GMEM 10% FCS 100 U/ml LIF 100 nM 2-mercaptoethanol 1× non-essential amino acids, 2 mM L- glutamine 1 mM sodium pyruvate |
| NH4Cl 50mM | Sigma | 9718 | |
| Paraformaldehyde | Sigma | 158127 | CAUTION: Toxic, handle undiluted stocks in fume hood and wear protective equipment |
| PBS (immunostaining) | Sigma | P4417 | Dilute in 200ml of ddH2O |
| PBS (tissue culture) | Gibco | 11140-035 | |
| Plastic slides | Ibidi | IB-10813 | |
| Poloxamer 407 | Sigma | P2443 | |
| ProLong Gold Antifade Mountant | Molecular Probes | P36930 | |
| Sodium Azide | Sigma | 8591 | CAUTION: Toxic, handle in fume hood and wear protective equipment |
| Sodium pyruvate | Gibco | 11360070 | |
| TritonX-100 | Sigma | T8532 | |
| Trypsin | Gibco | 25200056 | |
| UVO cleaner | Jetlight, USA | 42-220 | CAUTION: Follow manufacturer’s safety recommendations |
| Washing Solution | NA | NA | 0.1% TritonX-100 0.01% Pluronic F-127 In PBS |
Referenzen
- Sasai, Y. Cytosystems dynamics in self-organization of tissue architecture. Nature. 493 (7432), 318-326 (2013).
- Turner, D. A., Baillie-Johnson, P., Martinez Arias, A. Organoids and the genetically encoded self-assembly of embryonic stem cells. BioEssays. 38 (2), 181-191 (2016).
- Simunovic, M., Brivanlou, A. H. Embryoids, organoids and gastruloids: new approaches to understanding embryogenesis. Development. 144 (6), 976-985 (2017).
- Tewary, M., Shakiba, N., Zandstra, P. W. Stem cell bioengineering: building from stem cell biology. Nature Reviews. Genetics. 19 (10), 595-614 (2018).
- Shahbazi, M. N., Zernicka-Goetz, M. Deconstructing and reconstructing the mouse and human early embryo. Nature Cell Biology. 20 (8), 878(2018).
- Laurent, J., et al. Convergence of microengineering and cellular self-organization towards functional tissue manufacturing. Nature Biomedical Engineering. 1 (12), 939(2017).
- McBeath, R., Pirone, D. M., Nelson, C. M., Bhadriraju, K., Chen, C. S. Cell shape, cytoskeletal tension, and RhoA regulate stem cell lineage commitment. Developmental Cell. 6 (4), 483-495 (2004).
- Tewary, M., et al. A stepwise model of Reaction-Diffusion and Positional-Information governs self-organized human peri-gastrulation-like patterning. Development. , (2017).
- Warmflash, A., Sorre, B., Etoc, F., Siggia, E. D., Brivanlou, A. H. A method to recapitulate early embryonic spatial patterning in human embryonic stem cells. Nature Methods. 11 (8), 847-854 (2014).
- Blin, G., Wisniewski, D., Picart, C., Thery, M., Puceat, M., Lowell, S. Geometrical confinement controls the asymmetric patterning of brachyury in cultures of pluripotent cells. Development. 145 (18), (2018).
- Micropatterning in cell biology. Pt. A. , Elsevier. Amsterdam. (2014).
- Bauwens, C. L., et al. Control of human embryonic stem cell colony and aggregate size heterogeneity influences differentiation trajectories. Stem Cells. 26 (9), Dayton, Ohio. 2300-2310 (2008).
- Heemskerk, I., Burt, K., Miller, M., Chabra, S., Guerra, M. C., Warmflash, A. Morphogen dynamics control patterning in a stem cell model of the human embryo. bioRxiv. , 202366(2017).
- Nemashkalo, A., Ruzo, A., Heemskerk, I., Warmflash, A. Morphogen and community effects determine cell fates in response to BMP4 signaling in human embryonic stem cells. Development. 144 (17), 3042-3053 (2017).
- Peerani, R., Onishi, K., Mahdavi, A., Kumacheva, E., Zandstra, P. W. Manipulation of signaling thresholds in “engineered stem cell niches” identifies design criteria for pluripotent stem cell screens. PloS One. 4 (7), e6438(2009).
- Peerani, R., et al. Niche-mediated control of human embryonic stem cell self-renewal and differentiation. The EMBO journal. 26 (22), 4744-4755 (2007).
- Etoc, F., et al. A Balance between Secreted Inhibitors and Edge Sensing Controls Gastruloid Self-Organization. Developmental Cell. 39 (3), 302-315 (2016).
- Azioune, A., Carpi, N., Tseng, Q., Théry, M., Piel, M. Protein micropatterns: A direct printing protocol using deep UVs. Methods in Cell Biology. 97, 133-146 (2010).
- Théry, M. Micropatterning as a tool to decipher cell morphogenesis and functions. Journal of Cell Science. 123 (Pt 24), 4201-4213 (2010).
- Degot, S., et al. Improved Visualization and Quantitative Analysis of Drug Effects Using Micropatterned Cells. JoVE (Journal of Visualized Experiments). (46), e2514(2010).
- Beddington, R. S. P., Rashbass, P., Wilson, V. Brachyury - a gene affecting mouse gastrulation and early organogenesis. Development. 116 (Supplement), 157-165 (1992).
- Wilkinson, D. G., Bhatt, S., Herrmann, B. G. Expression pattern of the mouse T gene and its role in mesoderm formation. Nature. 343 (6259), 657-659 (1990).
- Hollnagel, A., Oehlmann, V., Heymer, J., Rüther, U., Nordheim, A. Id Genes Are Direct Targets of Bone Morphogenetic Protein Induction in Embryonic Stem Cells. Journal of Biological Chemistry. 274 (28), 19838-19845 (1999).
- Mountford, P., et al. Dicistronic targeting constructs: reporters and modifiers of mammalian gene expression. Proceedings of the National Academy of Sciences of the United States of America. 91 (10), 4303-4307 (1994).
- Smith, A. G. Culture and differentiation of embryonic stem cells. Journal of tissue culture methods. 13 (2), 89-94 (1991).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Preibisch, S., Saalfeld, S., Tomancak, P. Globally optimal stitching of tiled 3D microscopic image acquisitions. Bioinformatics. 25 (11), 1463-1465 (2009).
- Blin, G., Sadurska, D., Migueles, R. P., Chen, N., Watson, J. A., Lowell, S. Nessys: a novel method for accurate nuclear segmentation in 3D. bioRxiv. , 502872(2018).
- Weiswald, L. -B., Guinebretière, J. -M., Richon, S., Bellet, D., Saubaméa, B., Dangles-Marie, V. In situ protein expression in tumour spheres: development of an immunostaining protocol for confocal microscopy. BMC cancer. 10, 106(2010).
- R Core Team. R: A Language and Environment for Statistical Computing. , R Foundation for Statistical Computing. Vienna, Austria. Available from: http://www.R-project.org/ (2013).
- Deglincerti, A., et al. Self-organization of human embryonic stem cells on micropatterns. Nature Protocols. 11 (11), 2223-2232 (2016).
- Ostblom, J., Nazareth, E. J. P., Tewary, M., Zandstra, P. W. Context-explorer: Analysis of spatially organized protein expression in high-throughput screens. PLOS Computational Biology. 15 (1), e1006384(2019).
- Larue, L., Ohsugi, M., Hirchenhain, J., Kemler, R. E-cadherin null mutant embryos fail to form a trophectoderm epithelium. Proceedings of the National Academy of Sciences. 91 (17), 8263-8267 (1994).
- Pieters, T., van Roy, F. Role of cell-cell adhesion complexes in embryonic stem cell biology. J Cell Sci. 127 (12), 2603-2613 (2014).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132 (3), 487-498 (2008).
- Morgani, S. M., Metzger, J. J., Nichols, J., Siggia, E. D., Hadjantonakis, A. -K. Micropattern differentiation of mouse pluripotent stem cells recapitulates embryo regionalized cell fate patterning. eLife. 7, e32839(2018).
- Ulman, V., et al. An objective comparison of cell-tracking algorithms. Nature Methods. 14 (12), 1141(2017).
- Moir, R. D., Yoon, M., Khuon, S., Goldman, R. D. Nuclear Lamins a and B1. The Journal of Cell Biology. 151 (6), 1155-1168 (2000).
- McDole, K., et al. In Toto Imaging and Reconstruction of Post-Implantation Mouse Development at the Single-Cell Level. Cell. 175 (3), 859-876 (2018).
- Stegmaier, J., et al. Real-Time Three-Dimensional Cell Segmentation in Large-Scale Microscopy Data of Developing Embryos. Developmental Cell. 36 (2), 225-240 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten