Method Article
網膜芽細胞腫に対するリキッドバイオプシーとしての房水:明確な角膜穿刺とゲノム解析
要約
房水は、眼球外転移のリスクがあるために in vivo で生検できない網膜芽細胞腫、眼内癌に対する高収量のリキッドバイオプシーです。ここでは、明確な角膜穿刺を介して房水を安全に抽出する方法と、予後バイオマーカーを特定するためのゲノム解析の手順を示します。
要約
網膜芽細胞腫に対するリキッドバイオプシープラットフォームの適用には、臨床的に重要な潜在的有用性があります。これらの患者では直接腫瘍生検が禁止されていることを考えると、有用性が期待できます。房水(AH)は腫瘍とは別の区画に形成されますが、同じ眼腔内に囲まれています。したがって、これは、この疾患のリキッドバイオプシーまたは腫瘍生検の代理として使用できる、眼特異的な腫瘍ゲノム情報の豊富なソースです。この原稿では、透明な角膜穿刺により網膜芽細胞腫の眼からAHを安全に抽出するための方法論を詳しく説明しています。さらに、無細胞DNAの単離と精製、次世代シーケンシング、体細胞コピー数変更(SCNA)解析、 RB1 一塩基変異体(SNV)変異の同定、腫瘍分画推定などのゲノム解析のステップを示します。AH リキッドバイオプシー プラットフォームの分析前、分析、および初期の臨床的妥当性が評価されました。ただし、制限がないわけではありません。これらは主に、アッセイの特定のステップに必要なセルフリーDNAの量の結果です。網膜芽細胞腫について現在調査中の他の血液ベースのリキッドバイオプシープラットフォームと比較して、AHベースのプラットフォームは、眼から抽出できる生体液の量(したがってDNAの量)によって制限されます。利点は、AHが目特異的であることです。ここで説明するプラットフォームは、2つのメカニズム(SCNAと RB1 SNV)を介してAH内の循環腫瘍DNAを検出するという点で独特であり、腫瘍ゲノム情報の同定に対する感度が高くなります。AHリキッドバイオプシーは、網膜芽細胞腫患者の精密腫瘍学に直接臨床応用できる可能性があり、AHは各眼の腫瘍に特異的であるため、両側性疾患の患者にとって特に重要です。このプラットフォームを他の眼腫瘍の患者にも適用する研究が進行中です。
概要
網膜芽細胞腫(RB)は、まれでユニークながんです。乳幼児の発育中の網膜に発生する原発性眼内悪性腫瘍ですが、世界で年間約7000例しか発生しておらず、そのうち約250〜300例が米国にいます。先進国では患者の生存率は98%に近づいていますが1、臨床的に国際眼内RB分類(IIRC)2グループD/EまたはAJCC cT2b/3に分類される進行眼の眼の生存率ははるかに低いです。これらの進行した眼の多くは、主に(最初の治療として)または二次的に(グローブサルベージ療法の試みが失敗した後)核出されます。眼腫瘍学の現在の臨床診療では、RB患者の診断、眼の生存の予後、または治療モニタリングを支援するために現在臨床的に使用されている腫瘍由来の眼特異的分子バイオマーカーはありません。これは主に、眼球外腫瘍の広がりに対する懸念からRBでの直接腫瘍生検が禁止されているため、腫瘍組織が摘出眼からの分子および遺伝学的分析にのみ利用できるという事実によるものです3,4,5,6,7,8,9 .この禁止のため、RB腫瘍遺伝学およびそれらの臨床的相関に関する以前のレトロスペクティブ研究は、除核眼から得られた腫瘍組織の分析のみに限定されていた10,11,12,13。したがって、診断時および眼球救助療法全体で利用できる客観的な腫瘍由来の分子データが不足していました。これにより、in vivo腫瘍生物学の理解と、これらの腫瘍が治療中に分子レベルでどのように変化するかだけでなく、これらの若いがん患者に対して個別化された眼特異的なゲノムベースの治療計画を開発する能力も制限されています。
生検の禁止は別として、この癌の別のユニークな側面は、ほとんどの腫瘍が細胞周期を調節するRB1腫瘍抑制遺伝子の双対立遺伝子の喪失によって開始されることです。発達中の網膜は、この喪失に対して絶妙に敏感です。患者の 60% では、RB1 遺伝子のこの両対立遺伝子の不活性化は、体細胞の喪失としてのみ網膜で発生し、片側性疾患を引き起こします。しかし、患者の40%では、最初のRB1変異が生殖細胞系で起こり、その後、網膜で2回目の「ヒット」が起こります。これらの小児では、両眼に影響を与える複数の腫瘍がしばしば存在します。最後に、腫瘍の非常に小さなサブセット(<2%)は、RB1遺伝子に変異を伴わないMYCN増幅によって駆動されているようである。MYCNによる腫瘍はほぼ例外なく治療に失敗し、核摘出術を必要とするが、現在のところ、診断時にこの侵攻性腫瘍サブタイプを特定する明確で客観的な方法はない14,15。さらに、眼内RB腫瘍活動のモニタリングは、治療を行う眼腫瘍医による各眼の画像診断と臨床観察にほぼ独占的に依存しています。診断、予後、または治療全体を通じて眼特有の腫瘍動態をモニターするためのゴールドスタンダードの客観的で定量的な手段はありません。RBにはこのような特有の限界があるため、このがんに対するリキッドバイオプシープラットフォームの見通しは魅力的です。リキッドバイオプシーでは、体液を使用して無細胞DNAを単離し、配列決定を行い、循環腫瘍DNA(ctDNA)として知られる腫瘍由来のDNAであるかどうかを判断します。血清は他のがんについても研究されていますが、血清に含まれるRB ctDNAは眼特異的ではなく、両側性疾患に苦しむ患者の40%に明確な制限があります。さらに、進行した眼内または転移性疾患の設定でのみ記載されており、一般に腫瘍の割合が低い(<5%)16。
患者と家族が直面しているこれらの実際の臨床問題に対処するために、2017年に、房水(AH、目の前にある透明な液体)が、RB17を評価するためのリキッドバイオプシーとして、または腫瘍生検の代理として使用できる高収量のctDNA源であることを示しました。18、19。現在までに200を超えるサンプルで、サンプルの>95%でctDNAが同定され、これには進行度の低いIIRCグループA、B、およびCの眼、および両側の患者の眼の間のゲノムの違いが含まれています18,19。このDNAは、積極的に治療を受けている眼または未治療の眼における関連する分子バイオマーカーの検出のために、安全かつ効果的に単離することができる18,20,21。AHリキッドバイオプシープラットフォームは、腫瘍形成を開始する診断用RB1病原性多様体または一次MYCN増幅の同定にも使用できます21,22。重要なことに、我々は予後的な分子的特徴、すなわち、MYCN増幅の中央値または限局性MYCN増幅に対して≥1.5の振幅を持つ染色体6p増加の存在を同定したが、これは眼球の切除を必要とする眼内腫瘍制御の喪失の可能性が16.5増加することと関連している18,20,21.最後に、AHのctDNA腫瘍画分(TFx)の変化は、より高いレベルが活動性疾患と相関し、レベルの低下が陽性の治療反応と関連しているため、治療反応と相関することが実証されています23。これらのアプリケーションとその潜在的な臨床的有用性を考慮して、AH生検と評価の方法論を説明したいと考えました。これには、サンプル取得のための明確な角膜穿刺や、ゲノム解析、特にcfDNAライブラリの構築とシーケンシング、SCNA振幅測定、RB1病原性バリアントの同定、TFx計算のためのプロトコールが含まれます。
プロトコル
この研究は、ロサンゼルス小児病院と南カリフォルニア大学の治験審査委員会の承認の下で実施されており、ヘルシンキ宣言の信条に準拠しています。書面によるインフォームドコンセントは、常にすべての参加者の法定後見人から得られます。
AHリキッドバイオプシーのワークフローの概略図を 図1に示します。
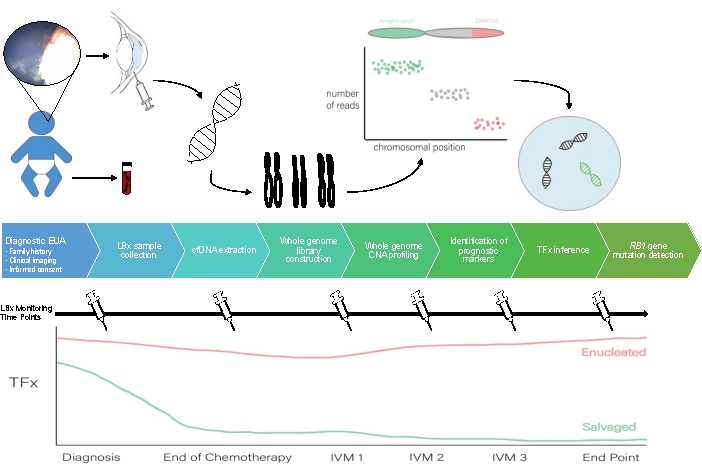
図1:AHリキッドバイオプシーが本明細書に記載されているプロトコルに付随するワークフロー。 EUA、麻酔下での検査;LBX、リキッドバイオプシー;cfDNA、無細胞DNA;CNA、コピー番号の変更;TFx、腫瘍画分;IVM、硝子体内メルファラン。 この図の拡大版を表示するには、ここをクリックしてください。
1.外科的処置:
注:この手順は、RB患者の臨床評価のための麻酔下(EUA)での定期検査中に行われます。AHを抽出するための穿刺手順は、眼科手術の標準的なトレーニングを完了した訓練を受けた眼科医のみが行う必要があります。
- 患者様の選択と選択基準は以下の通りです。
- チャンバーが形成され、腫瘍、滑膜、または白内障水晶体から浅くなることなく透明であることを確認してください。
注:一部の高度な目では、診断時にチャンバーが浅すぎてAHを安全に抽出できません。このような場合は、全身化学療法または動脈内化学療法の最初のサイクルが終了するまで待つことをお勧めします。主要な腫瘍が縮小すると、通常、心室は深くなり、安全なアプローチが可能になります。 - すべての構造がはっきりと見えること、および圧力が<22 mm Hgであることを確認して、圧力力学によるチャンバーの急激な浅瀬がないようにします。これはすべての時点で当てはまりますが、診断時にAHを採取する場合は特に重要です。
- 腫瘍による前眼部の直接的な関与がないことを確認し、これは直接視覚化と超音波生体顕微鏡法によって評価されます。この選択基準は、通常、EUA中に特定されます。
- チャンバーが形成され、腫瘍、滑膜、または白内障水晶体から浅くなることなく透明であることを確認してください。
- 患者が適切であると判断され、同意したら、患者を手術室に連れて行きます。RB患者の臨床ケアのためのEUAのルーチンとして、主治医の麻酔科医が麻酔を開始できるようにします。
- 患者が挿管され、鎮静され、EUAに関連するすべての臨床検査、画像診断、および局所治療ニーズ(レーザーや凍結療法など)が完了したら、リキッドバイオプシープラットフォームのAHを抽出します。通常の滅菌方法で目を準備し、ドレープします。これは、循環看護師または手術医が完了することができます。
- EUAの前に、施設で日常的にEUAに使用される拡張液を手術眼に置きます。.
注:ここでは、2.5%のフェニレフリン、1%のシクロペントラート、および1%のトロピカミドからなる併用点眼薬が使用されます。.ただし、EUAの拡張に使用される標準的な滴で十分です。AHを抽出するためだけに目を拡張する必要はありません。 - 5%ベタジン滴を手術眼に置き、スポンジを使用して中心から末梢に動かして塗布します。手術の準備には、上まぶたと下まぶたの縁を含めます。ベタジンを使用して外科的準備を3回完了し、少なくとも2分待ってから続行します。.
- 滅菌済みの青いタオルを患者の頭にかぶせ、頭の下に押し込んで落下しないようにします。まつげと眼窩周囲を滅菌済みの青いタオルまたはガーゼで乾かします。
- 手術医にスクラブをしてもらい、ガウンを着用し、手袋を着用してください。
- 滅菌済みの青いカーテンで患者を覆い、手術用の目を露出させます。滅菌フィールドを最適に視覚化するために、滅菌蓋鏡を配置します。
注: 子供は EUA の全身麻酔下にあるため、当施設では追加の局所麻酔は必要ありません。 - 機関のプロトコルに従ってタイムアウトを実行します。ここでは、EUAに対して最初のタイムアウトが行われ、手順の前に目を確認するために2回目のタイムアウトが行われます。
- EUAの前に、施設で日常的にEUAに使用される拡張液を手術眼に置きます。.
- 手術用顕微鏡を使用して、1 ccの注射器で32 Gの針で0.1 mLのAHを抽出し、透明な角膜穿刺を行います。
- 手順を開始する前に、滅菌バランス塩溶液(塩化ナトリウム0.64%、塩化カリウム0.075%、塩化カルシウム二水和物0.048%、塩化マグネシウム六水和物0.03%、酢酸ナトリウム三水和物0.39%、クエン酸ナトリウム二水和物0.17%、水酸化ナトリウムおよび/または塩酸(pHを調整するため)、および注射用水を含む各mL)で眼を濡らして、角膜の潤滑を維持します。これは標準的な商用製剤です。
- 32 G針が1 ccシリンジにルアーロックされていること、およびシリンジに圧力がかかっていないことを確認します(使用前にシリンジのプランジャーを数回出し入れします)。
- その後、針を辺縁部の透明な角膜に通し(穿刺の標準として垂直)、末梢拡張した虹彩の前房内に留まります。
- 抽出中は、針先を虹彩の前方に面取り、顕微鏡で直接視覚化します。必要に応じて、針が角膜を通過するときに0.12鉗子で目を安定させます。
注:針先と眼の構造との間に接触してはいけません。それは中前房の末梢虹彩の上に留まるべきです。 - AH0.1mLを抽出します。シリンジプランジャーは、外科医の利き手ではない手で(ただし練習が必要です)、または訓練を受けた助手が針を動かさずに操作します。針先の直接の視覚化を失わないでください。目の解剖学的構造を優先し、必要に応じてAHを少なくして、チャンバーが形成されたままになるようにします。
- 抽出後、前房から針をそっと取り外します。チャンバーが形成されたままであるが、わずかに浅いことを確認します。圧力は柔らかいですが、生理学的なものです。針を目から取り外したら、滅菌水で目をたっぷりと浴びます。このステップでは、水が細胞を溶解するため、安全対策として、滅菌水がバラス塩溶液よりも好まれます。
- 針の部位に漏れがないか調べます。漏れがある場合は、綿の先端アプリケーターを使用して注入部位に30秒間穏やかな圧力をかけてから、漏れがないか再度確認してください。必要に応じて、このプロセスを繰り返します。
注:このアプローチでは、漏れを止めるためにより侵襲的な方法を使用する必要はありませんでした。この手順の後に、化学療法の硝子体内注射が続くこともあります。このメソッドペーパーでは、プロトコルは主要な手順としてAH抽出に焦点を当てています。
- 針の部位に漏れがないか調べます。漏れがある場合は、綿の先端アプリケーターを使用して注入部位に30秒間穏やかな圧力をかけてから、漏れがないか再度確認してください。必要に応じて、このプロセスを繰り返します。
- 漏れがなければ、手順は完了したと見なされます。患者から滅菌ドレープを取り外し、顔からベタジンを洗浄します。
- 針先をシリンジから慎重に取り外し、滅菌キャップを使用してルアーロックでサンプルを閉じ、サンプルを失わないようにし、シリンジに適切にラベルを付けます。
注:これは、子供が抜管されている間に、臨床研究コーディネーターまたは外科医が行う必要があります。- 検体抽出後、サンプルをキャップ付きシリンジに-80°Cで保存します。サンプルをドライアイスの上に置いて、手術室から適切な冷凍庫に輸送することができます。
- 患者が回復に送られる前に、外科医の好みに応じて抗生物質の点眼薬を1滴手術用眼球の表面に置きます。ここでは、ステロイドと抗生物質の組み合わせ点眼薬が使用され、眼にはパッチが当てられていません。
- 手順が完了すると、担当の麻酔科医が適切な抜管を確認します。患者を回復に導きます。
- 施設の術後プロトコルに従って患者を自宅に退院させます。
- 目を包帯や保護カバーで覆う必要がないことを患者に伝えます。.
- 患者には、お風呂やプールに頭を24時間完全に浸さないように、また両親には患者(子供)の目をこすらないように伝えてください。患者の活動に他の制限はありません。
- 患者に、穿刺による痛みはありそうにないが、EUA中に関連する手順による痛みがある可能性があることを伝えます。患者は、外科医が提案するように、不快感がある場合にアセトアミノフェンまたはイブプロフェンを服用することができます。
2. cfDNAの単離と精製
- AH抽出後72時間以内にDNAの単離とプロセシングを行います。
- AHを室温で解凍します。サンプルをこまめにチェックし、解凍完了後すぐに抽出に向けて動きます。
- AHからcfDNAを抽出し、メーカーの取扱説明書に従ってcfDNA単離精製キット(詳細については 材料表 を参照)を使用して、50 μLのAVE溶液(キットに含まれています)に溶出します。
3. 次世代シーケンシング(NGS)と品質管理(QC)
- DNAライブラリーシーケンシングキット( 資料表を参照)を使用して、メーカーの取扱説明書に従ってシーケンシング用のDNAライブラリを構築します。DNAライブラリシーケンシングキットのマニュアルに詳述されているように、14サイクルのポリメラーゼ連鎖反応(PCR)でライブラリDNAを増幅します。
注:最終ライブラリは、ゲノム解析のための最終シーケンシングの1か月前まで-20°Cで保存できます。 - シングルエンド50サイクルまたはペアエンド150サイクルのプロトコルに従って、製造元のプロトコルに従って、NGSプラットフォーム( 材料の表を参照)上のDNAライブラリをシーケンシングします。
注:他のNGSプラットフォームは、原則として機能します(ただし、ここでは試していません)、シーケンシングフローセルに適したアダプターを使用します。 - 診断時または一次核摘出時に採取されたAHサンプルに対してQCを実行します。
注:他の時点で採取したサンプルについては、DNA質量が低すぎて以下に説明する手順の検出範囲外となるため、QCは実行できません。- 抽出したDNAをそれぞれ1 μLを、DNA定量アッセイおよびサイズプロファイリングアッセイ( 材料表を参照)に、メーカーのプロトコルに従って使用します。
注:ピークは約300 bpである必要があります。150 bpより短い場合、サンプルは損なわれています。QC に合格すべきサンプルの例については 補足ファイル 1A を、合格すべきでないサンプルの例については 補足ファイル 1B を参照してください。
- 抽出したDNAをそれぞれ1 μLを、DNA定量アッセイおよびサイズプロファイリングアッセイ( 材料表を参照)に、メーカーのプロトコルに従って使用します。
4. 体細胞コピー数改変(SCNA)データ解析
- NGS(セクション3)から得られたリードをヒトゲノム(hg19、Genome Reference Consortium GRCh37、University of California Santa Cruz Genome Browser database)24,25にアライナーでマッピングします。
- PCRの重複を削除します(samtools rmdup26)。
- 参照ゲノムから各ビンのグアニン塩基とシトシン塩基の割合を計算することにより、グアニン-シトシン含有量を正規化します。サンプルプログラムはBaslan et al.27に記載されています。
- DNAセグメントのコピー数を取得するには、ゲノムを5000個の可変長ビンに分割し、各ビンに含まれるリードの相対数を計算します。
- 参照フリーのlog2比を使用して、コピー数の推定値を決定します。正常な常染色体のウィンドウ数の中央値を取ります。
- DNAcopy (Bioconductor28) による循環バイナリ セグメンテーションを使用してセグメンテーションを実行します。
- ベースラインから 20% の偏向 (log2 比 = 0) で SCNA を正と定義し、log2 比 ≤ 0.2 (0.87 以下の比で欠失を定義) での損失と log2 比 0.2 ≥ での増加 (1.15 以上の比で増幅を定義) と定義します。
注:これらの閾値は、以前に確立されたリキッドバイオプシー分析27,29と一致しています。
5. AHサンプルの RB1 変異解析
- さらに、全ゲノムライブラリをそれぞれ500 ngに増幅して、メーカーのプロトコルに従って突然変異検出のためのキャプチャベースのターゲットNGSを実現します。
- NGSプラットフォームのペアエンド150 bpまたは50 bpシングルエンドシーケンシングを、キャプチャーしたライブラリに対して、メーカーのプロトコルごとに>100倍まで実行します。
- NGS データを処理します。この研究では、CHLA個別化医療センターのbcbioパイプラインに基づく社内パイプラインが利用されています30。
- Atropos31を使用してアダプターと低品質塩基から生のfastqデータをトリミングし、BWA-MEMとNovoAlign(v3)32,33を使用してヒトGRCh37リファレンスにアライメントします。
- FreeBayes34 で重複をマークし、生殖細胞変異体を決定します。
注:FreeBayesバリアント呼び出しツールでは、サンプルゲノムの各位置が参照ゲノムと比較され、対立遺伝子カウントが数学的にモデル化されて、遺伝子型尤度測定値34が得られる。 - Ensembl Variant Effect Predictor (v96)35 を使用して、5.3.4-5.3.5 の手順に従ってバリアント注釈を行います。
- VarDictを使用して、AHまたは対になった正常な血液サンプルがない腫瘍の体細胞変異体を決定する(すなわち、生殖細胞変異体に盲検化されている)36。
- 領域の連続変異体が≤3%の代替対立遺伝子を有する場合、ヘテロ接合性の喪失と呼べる。
注:市販のゲノム解析ソフトウェアは、上記のパラメータとともに使用することもできます。
6. cfDNA TFxの測定
- ichorCNAソフトウェア(CNAベースのTFx推定ソフトウェア)を使用して、配列決定された各AH cfDNAサンプルのTFxを推定します37。
注:これは、cfDNA TFx38を測定するための血液ベースのリキッドバイオプシーで使用される標準ソフトウェアです。AHでのソフトウェアの使用は、以前に公開されています21,23。 - ゲノムプロファイルと対応するTFx溶液を個別にレビューして、各サンプルのTFx推定値が適切であることを確認します39。
結果
2つの眼(症例33および47)からの包括的な結果を以下に示します。症例数は、比較のために以前の出版物と一致しています 18,20,21。治療を行った医師は、治療中のAHリキッドバイオプシーの結果を知らされませんでした。すべての治療決定は無作為化されておらず、以前に発表されたルーチンの標準治療に従って行われました40,41。臨床転帰データは、最終解析までゲノムデータとは分離されたままでした。提示された結果の生データは、対応する著者からのリクエストに応じて入手できます。NIHの資金提供により、データはNIHゲノムデータ共有ポリシーによって管理され、将来的にはNIH指定データリポジトリ(dbGAP)を介して他の研究者が利用できるようになります。また、責任著者からのリクエストにより入手することもできます。
症例33と47はどちらもIIRCグループD2の眼で、臨床症状は非常に類似していました。したがって、彼らは両親の決定により眼球救助療法を受け、眼科医および腫瘍内科医の治療、特に症例47の全身化学療法と症例33の動脈内化学療法で治療されました。現在受け入れられている臨床予後に基づくと、これら 2 人の患者の眼に対するグローブサルベージの予測成功は同じであったでしょう: グループ D の 65%-70% は平均ですが、これは治療センターによって異なります 41.しかし、このセンターでの腫瘍指向分子プロファイリングからこれまでに収集されたグループDのデータに基づくと、地球サルベージの予測成功率は、症例47(AHで6p増加が特定されていない)で72%、症例33(6p増加が特定されている)で9%になる18,20,21。これを以下に示します。
症例47は、診断時にAHリキッドバイオプシープラットフォームを使用してSCNAおよびSNVの検出に成功した眼の例であり、縦断的に治療反応に対応するTFxの傾向も示しています。患者は、生後 15 か月で 14 mm x 9 mm の IIRC グループ D、球硝子体播種を伴うステージ cT2b RB を発症した女性です。彼女は、定期的な臨床血清白血球検査によって決定された RB1 生殖細胞変異に対して陰性でした。AHから、診断時のAHではRB SCNAの1qゲインと6pゲインが同定され、さらに他の2つの非再発性RB SCNAの7p損失と13qゲインが同定されました(図2)。注目すべきは、6pのゲインの振幅が1.2であり、中央値に対する1.5の比率≥振幅のみが予後不良の前兆を示していることを示していることです。焦点 MYCN 増幅がなく、6pゲインが1.5の閾値を下回ったことを考えると、分子特性に基づく治療によるサルベージの予後は高かった。診断時に採取した同じAHサンプルを RB1 病原性多様体の検出についても評価したところ、 RB1 遺伝子内のSNV c.958C>T, p.Arg320*が明らかになり、変異対立遺伝子頻度は87.01%(95%信頼区間、79.7%-94.6%)であった。この患者は、カルボプラチン、エトポシド、ビンクリスチン (CEV) の 6 サイクルで治療され、疾患の退行が見られましたが、3 回の連続した硝子体内メルファラン (IVM) 注射を必要とする持続的な球状および粉塵のような播種が示されました。IVM治療中、AHサンプルA-C(それぞれ2週間間隔)は、ゲノムプロファイルの完全な正常化、TFxの減少、およびDNA濃度の減少を示しました。これらはすべて、疾患の臨床的退行と同時に行われました(図3)。診断後、TFx値は残りの治療で検出限界の5%未満にとどまりました。19カ月の追跡調査で、眼球は安定しており、腫瘍の再発や眼外への疾患の広がりはなかった。
これは、ケース 33 とは対照的です。この生後 22 か月の男性は、11 mm x 18 mm の網膜腫瘤と粉塵型の硝子体播種を伴う全体的に同様の臨床症状を示し、片側の IIRC グループ D、ステージ cT2b と一致していました。彼はまた、定期的な臨床血清白血球検査によって決定されたRB1生殖細胞変異についても陰性でした。診断時に採取したAHは、RB SCNAの1qゲイン、6pゲイン(この場合、振幅は中央値に対して1.5の比率)、および16q損失と焦点6q損失を示しました(図4A)。予後分子プロファイルに基づくと、この眼球は、6pの存在≥1.5の振幅に基づいて、核摘出の確率が16.5増加しました。このサンプルでは、RB1遺伝子の全長を完全にカバーしているにもかかわらず、RB1 SNVは同定されませんでした。RB1 SNVが同定されない理由の1つは、原発性MYCN駆動腫瘍にあり、RB1変異の同時発生が必ずしも予想されるとは限らない14,15,42,43,44,45。しかし、症例33では、AHサンプルまたはその除核腫瘍組織のいずれにおいてもMYCN増幅の証拠は示されませんでした(図5)。RB1 SNVの結果が陰性であることのより可能性の高い説明は、初期の腫瘍形成がエピジェネティックな調節不全(例えば、プロモーターのメチル化)46,47によって引き起こされたということであり、これはRBの既知の現象であり、これは本明細書に記載のアッセイでは同定されないであろう。
この患者に対する初期治療は、メルファランによる合計4サイクルの動脈内化学療法と、それに続く持続的な硝子体播種による4回のIVM注射でした。IVM療法中に3つのAHサンプル(それぞれ4週間間隔)が採取され、診断時に存在したのと同じ3つのSCNAが示されました(図5)。TFx値は、硝子体内の活動性腫瘍種子を反映して原発腫瘍体積が減少しているにもかかわらず、治療全体を通じて高いままでした。これは、TFxが眼の全体的な病状をどのように表しているかを示しています。診断から6か月後、活動性の疾患が持続したため、眼球を摘出しました。その時点で腫瘍組織から得られたゲノムプロファイルは、診断時に得られたAHサンプルと92.81%一致することを示しました(図4B)。
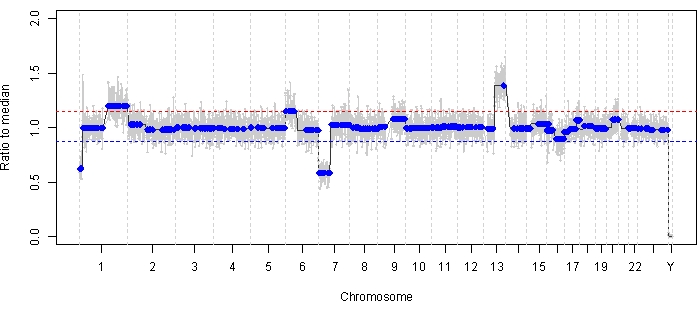
図2:症例47の診断時のゲノムプロファイル。 診断時に採取したAHでは、再発性の高いRB SCNAの1qゲインと6pゲイン、および非再発性のRB SCNAの7p損失と13qゲインが同定された。赤い線は利益のしきい値を表し、青い線は損失のしきい値を表します。特に、6pゲインの振幅は中央値に対して<1.5倍であり、これは予後不良の前兆となる分子シグネチャの閾値を下回っています。したがって、眼球救助のための陰性バイオマーカーの欠如に基づいて、この眼は治療に反応すると予測されます。 この図の拡大版を表示するには、ここをクリックしてください。
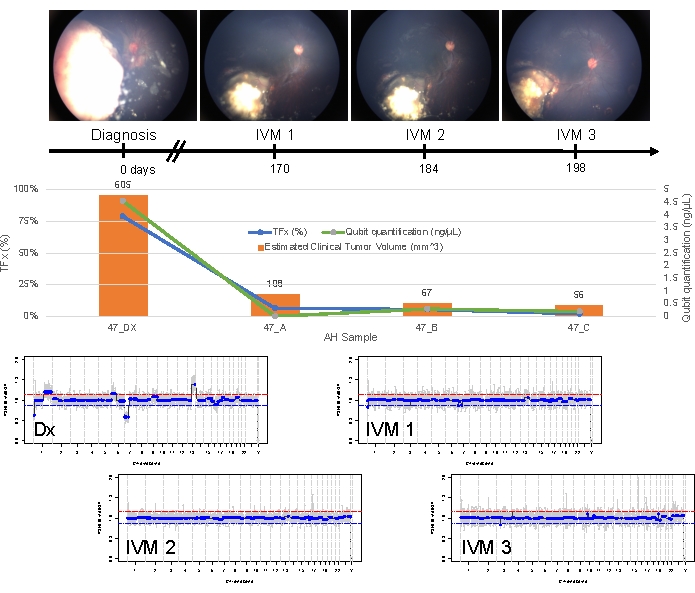
図3:ケース47の縦断的情報。 これには、眼底写真、cfDNA 定量化、B スキャン測定からの推定臨床腫瘍体積、ゲノムプロファイル、および AH がサンプリングされた各臨床時点 (Dx = 診断、A = IVM1、B = IVM2、C = IVM3) の TFx 推定が含まれます。この眼は治療に反応し、19カ月の追跡調査でサルベージされたままであった。cfDNA量の減少は経時的に観察され、診断時に得られた最高の収量を示す以前に発表された研究と一致していました。TFxは、播種の解決と主要な網膜腫瘍体積の減少を反映して、治療中にも減少しました(腫瘍体積は各臨床時点のオレンジ色のバーの上に示されています)。この疾患の臨床退行で予想されたように、ゲノムプロファイルも正規化されました。ゲノムプロファイルでは、赤い線はゲインの閾値を表し、青い線は損失の閾値を表しています。この図は、Xu, L. et al.21の許可を得て転載されています。 この図の拡大版を表示するには、ここをクリックしてください。
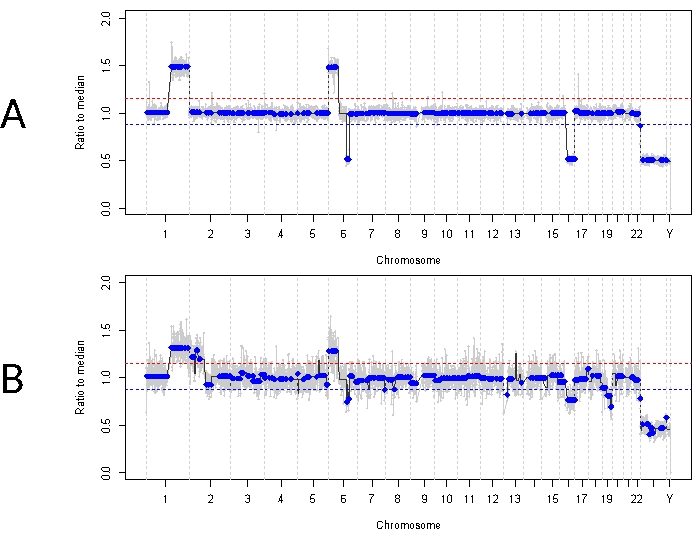
図4:ケース33のゲノムプロファイル。 赤い線はゲインの閾値を表し、青い線は症例33の診断時の損失(A)ゲノムプロファイルの閾値を表しています。AHでは、診断時に、焦点の6q損失に加えて、再発性の高いRB SCNAの1qゲイン、6pゲイン、および16q損失が同定されました。特に、6pゲインの振幅は中央値に対して1.5倍であり、予後不良の分子バイオマーカーを示しています。したがって、この分子シグネチャーに基づいて、この眼は治療失敗の可能性が著しく高いと予測されます。(B)除核腫瘍組織から得られたゲノムプロファイルで、診断時にAHから得られたプロファイルと非常に一致していた。正常な網膜組織との混ざり合いにより、腫瘍組織からのSCNAは、希釈されたTFxのためにAHと比較して低い振幅を示す ことができます。
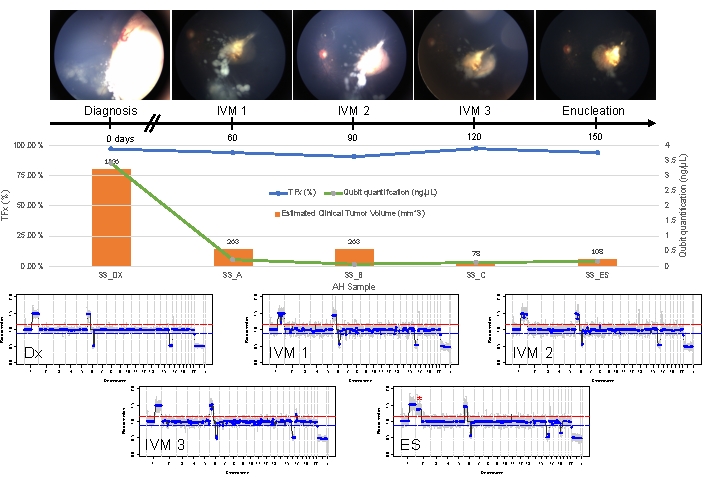
図5:ケース33の縦断的情報。 これには、眼底写真、無細胞DNA定量、B-スキャン測定からの推定臨床腫瘍体積(腫瘍体積は各臨床時点のオレンジ色のバーの上に示されます)、ゲノムプロファイル、およびAHがサンプリングされた各臨床時点のTFx推定が含まれます(Dx =診断、A = IVM1、B = IVM2、C = IVM3、SE =二次核摘出)。この眼は治療に反応せず、最終的には二次核摘出術(ES)が必要でした。これは、根尖腫瘍の再発を伴う持続的な活発な播種によるものでした。CfDNAの量は経時的に減少し、診断時に得られる最高の収量(1qゲイン、6pゲイン、16q損失、および焦点6q損失)を示す以前に発表された研究と一致しています。しかし、TFxは治療全体を通じて高いままであり、これは腫瘍由来のcfDNAをAHに放出する持続的な播種の結果です。ゲノムプロファイルは一貫しており、診断時に存在していたものと同じ3つのSCNAが示されました。ESで得られたAHでは、新たに大規模な2p増加(*)と19q損失が見られ、頂端腫瘍再発時のクローン性進化が示唆された。ゲノムプロファイルでは、赤い線はゲインの閾値を表し、青い線は損失の閾値を表しています。この図は、Xu, L. et al.21の許可を得て転載されています。 この図の拡大版を表示するには、ここをクリックしてください。
補足ファイル1: 例:(A)品質管理に合格すべきサンプルで、ピークが約300 bp、(B)品質管理に合格すべきでないサンプルで、ピークが約150 bpです 。このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
透明な角膜穿刺は、眼科における複数の診断および/または治療適応症に対して一般的に行われる手順です。特にRBについては、注射部位48への逆流を防ぐために注射前に眼圧を下げるための標準的な硝子体内化学療法注射プロトコルの一部です。一般的な手順であるにもかかわらず、リスクがまったくないわけではありません。以前の定説は、RBがアクティブな目に針を入れてはいけないというものでした。このドグマは、過去10年間で進化し、最初はパトリシア・シェベス・バリオスによる注射アデノウイルスベクター療法49の形成的研究を通じて、続いてフランシス・ムニエによるRBでの眼球注射の漸進的安全性向上方法48を通じて進化しました。この研究は、AHの抽出がこの癌のリキッドバイオプシー、または腫瘍生検の代理として使用される道を開きました。私たちは現在、プラットフォームの有用性と診断時に採取されたAHの予備的な安全性結果を報告する最初のセンターです。本明細書に記載の方法の一部は、RBに対するAH抽出の患者選択および手続き的側面の安全性を確保することを意図している。第一に、針は前房にのみ入ることができ、虹彩の瘢痕化や白内障を引き起こし、外科医が腫瘍を監視する能力を制限する可能性があるため、虹彩や水晶体に接触してはいけません。針が硝子体腔に入らないように(IVMの場合のように化学療法の送達と組み合わせない限り)、腫瘍に接触しないことが最も重要です。これは仮想的に腫瘍の播種と疾患の眼外拡大のリスクを高めます3,4,5。これらの構造は両方とも前房の後方にあり、水晶体と虹彩によって前房から分離されています。眼の構造への損傷を避けるためには、針を常にベベルアップし、末梢虹彩の上に置き、常に手術用顕微鏡を使用して直接視覚化することが重要です。針の部位からAHが軽微に漏れるリスクがあります。これが発生した場合は、綿の先端アプリケーターを介して穏やかな圧力で解決します。利用可能な最小のゲージ針を使用し、針が横方向のせん断なしに同じトラクトからゆっくりと引き抜かれるようにすることで、このリスクを減らすことができます。前房のわずかな浅さが予想されますが、心房は虹彩角膜に触れることなく形成されたままであり、眼圧は柔らかく、生理学的である必要があります。.
ゲノム解析に関しては、いくつかの重要なステップが関係しています。最も重要なのは、目から抽出した少量のサンプルの取り扱いです。凍結と解凍のサイクル50を繰り返すことで起こり得るDNAの分解を防ぐために、サンプルが処理前にずっと凍結されたままであることが重要である。抽出後すぐにサンプルをドライアイスに置き、-80°Cのフリーザーに移すことで、これを確実に行うことができます。サンプルの処理が開始されると、品質管理ステップは、cfDNAが高品質のライブラリに構築されることを保証することにより、トラブルシューティングの主な機会となります。DNA定量およびDNAフラグメントサイズプロファイリングアッセイを使用すると、一次核出術中または診断時に採取したサンプルに対して品質管理を行うことができます。これらのサンプルにはcfDNAの収量が高いためです18,21。得られたピークが約300 bpの場合、NGSプラットフォームによってcfDNAが認識されることが保証されます。得られたピークが 150 bp より短く、検出されたフラグメントの大部分がライブラリー構築プライマーまたはアダプターオリゴであることを示している場合、サンプルは損なわれており、SCNA についてさらに処理しないでください。しかし、数百のサンプルを処理した経験では、リードアライメント率が不十分なため、品質管理のために除去する必要があるのは5%未満です20。この手順は、検体の収集、取り扱い、保管、および処理のために標準化した方法とともに、AHリキッドバイオプシープラットフォームの分析前の有効性を示しています。
AHリキッドバイオプシーは、RB1病原性変異およびSCNAを正確かつ確実に検出する能力に基づいて、分析的妥当性も確立されており、AHサンプルから生成されたゲノムプロファイルと対応する腫瘍組織18,20,21,22,23との平均一致率は一貫して>95%である。このプラットフォームの分析的有効性は実証されていますが、制限がないわけではありません。RB1変異解析は、診断時または一次核摘出時に治療を受けていない眼で最も頻繁に得られるcfDNAが>10ngのサンプルでのみ実施できる18,21。これは、診断時または一次核出術時と比較して、積極的に治療を受けている眼からのAHサンプルに存在するcfDNAの濃度が低いためです。追加の制約は、SCNAが5%未満のTFxでは検出できないため、腫瘍負荷が大幅に減少した眼の疾患のモニタリングが妨げられることです。私たちの経験では、これは硝子体内化学療法治療に反応する眼で最も一般的に発生します23。
AH中のcfDNAのTFxを決定するために、CNAベースのTFx推定ソフトウェアが利用されます。このソフトウェアは、リキッドバイオプシーにおけるTFx計算のための標準的で受け入れられたツールであり、そのアルゴリズムは詳細に説明されています38,51。簡単に言うと、CNAベースのTFx推定ソフトウェアは、隠れマルコフモデルを利用して、シーケンシングされたcfDNA内の大規模なSCNAを予測します。TFx推定は、各遺伝子座におけるサブクローナリティおよび倍数性の違いを考慮しながら、SCNAの存在に基づいて導出され、これらから、CNAベースのTFx推定ソフトウェアは、最適なTFxソリューションを選択する38。ただし、CNAベースのTFx推定ソフトウェアに固有の制限は、TFxがサンプル中のSCNAの存在に基づいて計算されることです。したがって、SCNAのない(すなわち、フラットなゲノムプロファイルを持つ)サンプル中のTFxを決定することはできない38。以前に実証されたように、すべてのRB腫瘍がSCNA14、15、18、21、23、45を有するわけではない。したがって、CNAベースのTFx推定ソフトウェアによって決定される非常に低いTFxは、1)AHサンプルに測定可能な腫瘍由来のcfDNAが含まれていないか、2)腫瘍cfDNAが存在するが、SCNAが不足しているためCNAベースのTFx推定ソフトウェアでは検出できないことを意味します38。SNVのバリアント対立遺伝子画分(VAF)は、TFxの代用です。このため、SCNAを持たないRBeyesでも縦方向のTFxモニタリングを受けられるように、VAFに基づくRB1ベースのTFxパイプラインの開発に取り組んでいます。原発性MYCN駆動腫瘍14、15、42、43、44、45を除くすべてのRB腫瘍がRB1遺伝子に体細胞変異を含んでいることを考えると、SCNAに依存しないパイプラインは、本明細書で提示されたリキッドバイオプシープロトコルの適用を広げるであろう。さらに、SNVは5%未満のTFxまで監視できるため、プラットフォームの感度が向上します。
ここで説明するAHリキッドバイオプシープラットフォームは、RBの文献に存在する唯一のリキッドバイオプシープラットフォームではありませんが、注目すべきは、水性を腫瘍DNAの濃縮源として初めて説明し、これまでに発表された研究に基づいて、任意の生体液中の2つのメカニズム(SCNAとRB1 SNV)を介したctDNAの検出を初めて説明したことです。2つの方法でctDNAを検出する能力により、AHリキッドバイオプシーは文献の他のプラットフォームよりも高い感度を持っています。例えば、別のグループは、RB1 SNVs 52を用いてAH中のRB ctDNAを検出することに成功しました。ただし、このプラットフォームは、SNV の先験的な知識に基づくターゲット NGS リードに依存していました。対照的に、この原稿で詳述されているプラットフォームは、ノンバイアスWGSを利用しており、SCNAおよびSNVを検出する能力を与えています。血液ベースのリキッドバイオプシーも試みられていますが、得られたcfDNAは一貫してSCNA検出の閾値を下回っています。これは眼球サルベージの可能性の予後です(現時点では、RB1 SNVは眼のサルベージの予後を示すことが示されていません。 ただし、転移性疾患の検出には進化する役割がある可能性があります)19。Kothariらは、RB患者の血漿中のRB1 SNVを記載したが、除核を必要とする進行した眼内疾患の患者のみであった16。ここで説明するAHリキッドバイオプシープラットフォームは、シーディング19,21の存在がなくても、進行度の低い眼のAHでctDNAを検出する能力を持っています。さらに、血液ベースのリキッドバイオプシーは、血清から単離されたctDNAが両側RBの場合に両眼から分離され得るため、眼特異的ではありません。これにより、血液ベースのプラットフォームの臨床的有用性は、特にRB症例の40%を占める両側性患者では制限されますが、AHは眼特異的なままであり、SNVレベルとSCNAレベルの両方で眼間の不均一性を示すことができます53。
RBの分野におけるAHリキッドバイオプシーの重要性は最も重要です。AHリキッドバイオプシーは、積極的に治療を受けている眼の腫瘍内動態をよりよく理解する機会を提供するだけでなく、患者のケアを改善する可能性も秘めています。先行研究に基づき、MYCN増幅または中央値に対して≥1.5の振幅比を持つ染色体6pゲインの存在に基づく分子シグネチャーを確立し、これは、除核を必要とする治療失敗の可能性が16.5倍増加する予後である18,20,21。診断時にこの知識があれば、臨床医は適切な治療オプションと現在の治療法による眼球救助の可能性について家族に助言しやすくなります。RBのAHリキッドバイオプシープラットフォームの臨床的有効性は確立されていますが 17,18,19,20,21,22,23、現在、研究のみが承認されています。AHリキッドバイオプシーを臨床的に実施してRBの患者ケアを指示する前に、より大規模な前向き多施設研究が必要です。いずれにせよ、AHリキッドバイオプシーは、RBだけでなく、他の眼腫瘍に対しても、将来的に精密な腫瘍学を可能にする可能性を秘めています。
開示事項
Jesse Berry、Liya Xu、James Hicksは、眼科疾患の診断および予後評価のためのAqueous Humor Cell-Free DNAと題する特許出願を提出しました。それ以外の場合、著者は潜在的な利益相反を報告していません。
謝辞
この研究は、次の情報源によって支援されました:NIH賞K08CA232344のNCI(J.L.ベリーに)。ヒュンダイ ホープ オン ホイールズ RGA012351 (J. L. ベリーへ);小児眼がんトラスト(JLベリーへ);アメリカ癌協会IRG-16-181-57(JLベリーへ);ライト財団(J.L.ベリーとM.E.キムへ);テンプル騎士団アイ財団(JLベリーへ);ラリー&セリア・モー財団(J.L.ベリーへ);The Institute for Families, Inc., Children's Hospital Los Angeles (J. L. Berry);Research to Prevent Blindness(すべて)からの無制限の部門助成金。NCI P30CA014089(すべて);ヴィッキー・ジョセフ研究基金(P.クーンへ);キャロル・ヴァシリアディス研究基金(P.クーンへ);USCドーンシーフカレッジオブレターズ、アーツアンドサイエンス(P.クーンへ)。
資料
| Name | Company | Catalog Number | Comments |
| 1 cc syringe | surgical grade, whatever available in hospital | ||
| 32 G needle | surgical grade, whatever available in hospital | ||
| Aligner | Authors use Bowtie2 (http://bowtie-bio.sourceforge.net/bowtie2/index.shtml) but other aligners such as BWA or GRCh38 will also work | ||
| Atropos | generic term: adapter remover. https://atropos.readthedocs.io/en/latest/index.html# | ||
| Bioanalyzer High Sensitivity DNA Kit | Agilent | 5067-4626 | generic term: DNA fragment size profiling assay |
| BWA-MEM | generic term: long sequence aligner. http://bio-bwa.sourceforge.net/bwa.shtml | ||
| DNAcopy | Bioconductor | generic term: DNA copy number data analysis. https://bioconductor.org/packages/release/bioc/html/DNAcopy.html | |
| dsDNA High Sensitivity Assay | Qubit | Q32851 | generic term: DNA quantification assay |
| FreeBayes | generic term: sequence variant determiner. https://github.com/freebayes/freebayes | ||
| ichorCNA software | generic term: CNA-based TFx estimation. https://github.com/broadinstitute/ichorCNA | ||
| Illumina platform | Illumina | generic term: NGS platform; please note that other NGS platforms will work in principle, but have not been trialed by these authors | |
| NovoAlign (v3) | Novocraft | generic term: mapping of short reads onto reference genome. http://www.novocraft.com/products/novoalign/ | |
| QIAamp Circulating Nucleic Acid Kit | Qiagen | 55114 | generic term: cfDNA isolation and purification kit |
| QIAseq Ultralow Input Library Kit | Qiagen | 180492 | generic term: DNA library sequencing kit |
| Samtools rmdup | generic term: tool to remove duplicate reads. http://www.htslib.org/doc/samtools-rmdup.html | ||
| VarDict | generic term: variant caller. https://github.com/AstraZeneca-NGS/VarDict | ||
| Variant Effect Predictor | Ensembl | generic term: variant effect determinator. https://uswest.ensembl.org/info/docs/tools/vep/index.html |
参考文献
- Fernandes, A. G., Pollock, B. D., Rabito, F. A. Retinoblastoma in the United States: A 40-year incidence and survival analysis. Journal of Pediatric Ophthalmology & Strabismus. 55 (3), 182-188 (2018).
- Linn, A. M. Intraocular retinoblastoma: the case for a new group classification. Ophthalmology Clinics of North America. 18 (1), 41-53 (2005).
- Shields, J. A., Shields, C. L., Ehya, H., Eagle, R. C., De Potter, P. Fine-needle aspiration biopsy of suspected intraocular tumors. The 1992 Urwick lecture. Ophthalmology. 100 (11), 1677-1684 (1993).
- Karcioglu, Z. A., Gordon, R. A., Karcioglu, G. L. Tumor seeding in ocular fine needle aspiration biopsy. Ophthalmology. 92 (12), 1763-1767 (1985).
- Karcioglu, Z. A. Fine needle aspiration biopsy (FNAB) for retinoblastoma. Retina. 22 (6), 707-710 (2002).
- Eide, N., Syrdalen, P., Walaas, L., Hagmar, B. Fine needle aspiration biopsy in selecting treatment for inconclusive intraocular disease. Acta Ophthalmologica Scandinavica. 77 (4), 448-452 (1999).
- Eide, N., Walaas, L. Fine-needle aspiration biopsy and other biopsies in suspected intraocular malignant disease: a review. Acta Ophthalmologica. 87 (6), 588-601 (2009).
- Eriksson, O., Hagmar, B., Ryd, W. Effects of fine-needle aspiration and other biopsy procedures on tumor dissemination in mice. Cancer. 54 (1), 73-78 (1984).
- Ali, M. J., Honavar, S. G., Vemuganti, G. K., Singh, A. D. Fine needle aspiration biopsy of retinal tumors. Monographs in Clinical Cytology. 21, 72-81 (2012).
- Di Nicolantonio, F. a. l., et al. The chemosensitivity profile of retinoblastoma. Recent Results in Cancer Research. 161, 73-80 (2003).
- Francis, J. H., et al. Efficacy and toxicity of second-course ophthalmic artery chemosurgery for retinoblastoma. Ophthalmology. 122 (5), 1016-1022 (2015).
- Francis, J. H. Efficacy and toxicity of intravitreous chemotherapy for retinoblastoma: Four-year experience. Ophthalmology. 124 (4), 488-495 (2017).
- Francis, J. H., et al. Current treatment of bilateral retinoblastoma: The impact of intraarterial and intravitreous chemotherapy. Neoplasia. 20 (8), 757-763 (2018).
- Rushlow, D. E., et al. Characterisation of retinoblastomas without RB1 mutations: genomic, gene expression, and clinical studies. The Lancet Oncology. 14 (4), 327-334 (2013).
- Afshar, A. R., et al. Next-generation sequencing of retinoblastoma identifies pathogenic alterations beyond RB1 inactivation that correlate with aggressive histopathologic features. Ophthalmology. 127 (6), 804-813 (2020).
- Kothari, P. Cell-free DNA profiling in retinoblastoma patients with advanced intraocular disease: An MSKCC experience. Cancer Medicine. 9 (17), 6093-6101 (2020).
- Berry, J. L., et al. Potential of aqueous humor as a surrogate tumor biopsy for retinoblastoma. JAMA Ophthalmology. 135 (11), 1221-1230 (2017).
- Berry, J. L., et al. Genomic cfDNA analysis of aqueous humor in retinoblastoma predicts eye salvage: the surrogate tumor biopsy for retinoblastoma. Molecular Cancer Research. 16 (11), 1701-1712 (2018).
- Berry, J. L., et al. Aqueous humor is superior to blood as a liquid biopsy for retinoblastoma. Ophthalmology. 127 (4), 552-554 (2020).
- Xu, L., et al. Chromosome 6p amplification in aqueous humor cell-free DNA Is a prognostic biomarker for retinoblastoma ocular survival. Molecular Cancer Research. 18 (8), 1166-1175 (2020).
- Xu, L., et al. Establishing the clinical utility of ctDNA analysis for diagnosis, prognosis, and treatment monitoring of retinoblastoma: The aqueous humor liquid biopsy. Cancers. 13 (6), (2021).
- Xu, L. Simultaneous identification of clinically relevant RB1 mutations and copy number alterations in aqueous humor of retinoblastoma eyes. Ophthalmic Genetics. 41 (6), 526-532 (2020).
- Polski, A., et al. Longitudinal aqueous humor sampling reflects treatment response in retinoblastoma patients. Investigative Ophthalmology & Visual Science. 61 (7), 1394 (2020).
- . Genome Browser Available from: https://genome.ucsc.edu/cgi-bin/hgTracks?db=hg19&lastVirtModeType=default&lastVirtModeExtraState=&virtModeType=default&3A15578261%_2D15621068&hgsid=1135765621_qIuA0U (2021)
- Navarro Gonzalez, J., et al. The UCSC genome browser database: 2021 update. Nucleic Acids Research. 49 (1), 1046-1057 (2021).
- Li, H. A statistical framework for SNP calling, mutation discovery, association mapping and population genetical parameter estimation from sequencing data. Bioinformatics. 27 (21), 2987-2993 (2011).
- Baslan, T., et al. Genome-wide copy number analysis of single cells. Nature Protocols. 7 (6), 1024-1041 (2012).
- Huber, W., et al. Orchestrating high-throughput genomic analysis with Bioconductor. Nature Methods. 12 (2), 115-121 (2015).
- Baslan, T., et al. Erratum: Genome-wide copy number analysis of single cells. Nature Protocols. 11 (3), 1024-1041 (2016).
- Didion, J. P., Martin, M., Collins, F. S. Atropos: specific, sensitive, and speedy trimming of sequencing reads. PeerJ. 5, 3720 (2017).
- Li, H. Aligning sequence reads, clone sequences and assembly contigs with BWA-MEM. arXiv. , (2013).
- Garrison, E., Marth, G. Haplotype-based variant detection from short-read sequencing. arXiv. , (2012).
- McLaren, W., et al. The ensembl variant effect predictor. Genome Biology. 17 (1), 122 (2016).
- Lai, Z., et al. VarDict: a novel and versatile variant caller for next-generation sequencing in cancer research. Nucleic Acids Research. 44 (11), 108 (2016).
- . GitHub Available from: https://github.com/broadinstitute/ichorCNA (2017)
- Adalsteinsson, V. A., et al. Scalable whole-exome sequencing of cell-free DNA reveals high concordance with metastatic tumors. Nature Communications. 8 (1), 1-13 (2017).
- . GitHub Available from: https://github.com/broadinstitute/ichorCNA/wiki/Interpreting-ichorCNA-results (2017)
- Berry, J. L., et al. Long-term outcomes of Group D retinoblastoma eyes during the intravitreal melphalan era. Pediatric Blood & Cancer. 64 (12), (2017).
- Berry, J. L., et al. Long-term outcomes of Group D eyes in bilateral retinoblastoma patients treated with chemoreduction and low-dose IMRT salvage. Pediatric Blood & Cancer. 60 (4), 688-693 (2013).
- Francis, J. H., et al. Molecular changes in retinoblastoma beyond RB1: Findings from next-generation sequencing. Cancers (Basel). 13 (1), (2021).
- Schwermer, M., et al. Comprehensive characterization of RB1 mutant and MYCN amplified retinoblastoma cell lines. Experimental Cell Research. 375 (2), 92-99 (2019).
- Davies, H. R. Whole-genome sequencing of retinoblastoma reveals the diversity of rearrangements disrupting RB1 and uncovers a treatment-related mutational signature. Cancers. 13 (4), 754 (2021).
- Lillington, D. M., et al. Comparative genomic hybridization of 49 primary retinoblastoma tumors identifies chromosomal regions associated with histopathology, progression, and patient outcome. Genes, Chromosomes and Cancer. 36 (2), 121-128 (2003).
- Greger, V., et al. Frequency and parental origin of hypermethylated RB1 alleles in retinoblastoma. Human Genetics. 94 (5), 491-496 (1994).
- Raizis, A. M., et al. DNA hypermethylation/boundary control loss identified in retinoblastomas associated with genetic and epigenetic inactivation of the RB1 gene promoter. Epigenetics. 1-15, (2020).
- Munier, F. L., et al. Profiling safety of intravitreal injections for retinoblastoma using an anti-reflux procedure and sterilisation of the needle track. British Journal of Ophthalmology. 96 (8), 1084-1087 (2012).
- Chévez-Barrios, P., et al. Response of retinoblastoma with vitreous tumor seeding to adenovirus-mediated delivery of thymidine kinase followed by ganciclovir. Journal of Clinical Oncology. 23 (31), 7927-7935 (2005).
- Shao, W., Khin, S., Kopp, W. C. Characterization of effect of repeated freeze and thaw cycles on stability of genomic DNA using pulsed field gel electrophoresis. Biopreservation and Biobanking. 10 (1), 4-11 (2012).
- Choudhury, A. D., et al. Tumor fraction in cell-free DNA as a biomarker in prostate cancer. JCI Insight. 3 (21), (2018).
- Gerrish, A., et al. Non-invasive diagnosis of retinoblastoma using cell-free DNA from aqueous humour. British Journal of Ophthalmology. 103 (5), 721-724 (2019).
- Wong, E. Y., Xu, L., Shen, L. Inter-eye genomic heterogeneity in bilateral retinoblastoma via aqueous humor liquid biopsy. NPJ Precis Oncol. 5 (1), 73 (2021).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved