Method Article
Humor aquoso como biópsia líquida para retinoblastoma: paracentese clara da córnea e análise genômica
Neste Artigo
Resumo
O humor aquoso é uma biópsia líquida de alto rendimento para retinoblastoma, câncer intraocular que não pode ser biopsiado in vivo devido ao risco de disseminação extraocular. Aqui, um método para extrair com segurança o humor aquoso por meio de paracentese clara da córnea e etapas para análise genômica para identificar biomarcadores prognósticos são apresentados.
Resumo
Há uma utilidade clínica potencial significativa para a aplicação de uma plataforma de biópsia líquida para retinoblastoma, uma vez que a biópsia direta do tumor é proibida nesses pacientes. O humor aquoso (AH) se forma em um compartimento separado do tumor, mas é encerrado no mesmo espaço ocular. Assim, é uma fonte enriquecida de informações genômicas tumorais específicas do olho que podem ser usadas como uma biópsia líquida ou substituta da biópsia tumoral para esta doença. Este manuscrito detalha uma metodologia para extrair com segurança o AH dos olhos do retinoblastoma por meio de paracentese clara da córnea. Além disso, são apresentadas as etapas para análise genômica, incluindo isolamento e purificação de DNA livre de células, sequenciamento de próxima geração, análise de alteração do número de cópias somáticas (SCNA), identificação de mutação de variante de nucleotídeo único (SNV) RB1 e estimativa de fração tumoral. A validade pré-analítica, analítica e clínica precoce da plataforma de biópsia líquida AH foi avaliada; no entanto, não é sem limitações. Estes são em grande parte uma consequência da quantidade de DNA livre de células que é necessária para certas etapas do ensaio. Em comparação com outras plataformas de biópsia líquida baseadas em sangue atualmente sob investigação para retinoblastoma, uma plataforma baseada em AH é limitada pelo volume de biofluido (e, portanto, pela quantidade de DNA) que pode ser extraído do olho; o benefício é que a HA é específica para os olhos. A plataforma discutida aqui é única, pois detecta DNA tumoral circulante no AH por meio de dois mecanismos (SCNAs e RB1 SNVs), produzindo uma maior sensibilidade para identificar informações genômicas tumorais. A biópsia líquida de HA tem potencial para aplicação clínica direta em oncologia de precisão para pacientes com retinoblastoma, com particular importância para pacientes com doença bilateral, pois a HA é específica para os tumores em cada olho. Há pesquisas em andamento com aplicações desta plataforma também em pacientes com outros tumores oculares.
Introdução
O retinoblastoma (RB) é um câncer raro e único. Embora seja a malignidade intraocular primária mais comum que se forma na retina em desenvolvimento de bebês e crianças pequenas, existem apenas cerca de 7000 casos no mundo anualmente, e aproximadamente 250-300 deles estão nos Estados Unidos. Embora a sobrevida do paciente se aproxime de 98%em países desenvolvidos1, a sobrevida ocular para olhos avançados, que são clinicamente classificados como International Intraocular RB Classification (IIRC)2 Grupo D/E ou AJCC cT2b/3, é muito menor. Muitos desses olhos avançados são enucleados principalmente (como primeiro tratamento) ou secundariamente (após tentativas fracassadas de terapia de salvamento do globo). Na prática clínica atual da oncologia ocular, não há biomarcadores moleculares específicos do olho derivados do tumor que sejam usados clinicamente para auxiliar no diagnóstico, prognóstico para sobrevida ocular ou monitoramento do tratamento de pacientes com RB. Isso se deve, em grande parte, ao fato de que o tecido tumoral só está disponível para análises moleculares e genéticas de olhos enucleados, pois a biópsia direta do tumor em RB é proibida devido à preocupação com a disseminação do tumor extraocular 3,4,5,6,7,8,9 . Devido a essa proibição, estudos retrospectivos anteriores sobre a genética tumoral RB e suas correlações clínicas foram restritos à análise de tecido tumoral obtido apenas de olhos enucleados 10,11,12,13. Portanto, tem havido uma falta de dados moleculares derivados de tumores objetivos disponíveis no diagnóstico e durante a terapia de recuperação ocular. Isso limitou não apenas a compreensão da biologia do tumor in vivo e as maneiras pelas quais esses tumores mudam em nível molecular ao longo da terapia, mas também a capacidade de desenvolver planos de tratamento personalizados, específicos para os olhos e baseados em genômica para esses jovens pacientes com câncer.
Além da proibição da biópsia, outro aspecto único desse câncer é que a maioria dos tumores é iniciada pela perda bialélica do gene supressor de tumor RB1, que modula o ciclo celular. A retina em desenvolvimento é extremamente sensível a essa perda. Em 60% dos pacientes, essa inativação bialélica do gene RB1 ocorre na retina apenas como perda somática e resulta em doença unilateral. No entanto, em 40% dos pacientes, a mutação RB1 inicial ocorre na linha germinativa, seguida por um segundo 'golpe' na retina. Nessas crianças, muitas vezes há vários tumores afetando ambos os olhos. Finalmente, um subconjunto muito pequeno (<2%) de tumores parece ser impulsionado pela amplificação de MYCN sem mutações no gene RB1. Embora os tumores orientados por MYCN falhem quase universalmente no tratamento e exijam enucleação, atualmente não há uma maneira clara e objetiva de identificar esse subtipo de tumor agressivo no diagnóstico14,15. Além disso, o monitoramento da atividade do tumor RB intraocular depende quase exclusivamente de imagens e observações clínicas de cada olho pelo oncologista ocular assistente. Não há objetivo padrão-ouro, meios quantitativos de diagnóstico, prognóstico ou maneira de monitorar a dinâmica tumoral específica do olho durante o tratamento. Devido a essas limitações únicas para RB, a perspectiva de uma plataforma de biópsia líquida para esse câncer é atraente. As biópsias líquidas utilizam fluidos corporais para isolar e sequenciar o DNA livre de células para determinar se é derivado do tumor, conhecido como DNA tumoral circulante (ctDNA). Embora o soro seja pesquisado para outros tipos de câncer, o ctDNA RB encontrado no soro não é específico para os olhos, o que tem limitações claras para 40% dos pacientes que sofrem de doença bilateral. Além disso, só foi descrita no contexto de doença intraocular ou metastática avançada, geralmente com baixa fração tumoral (<5%)16.
Em um esforço para resolver esses problemas clínicos reais enfrentados por pacientes e familiares, em 2017, demonstramos que o humor aquoso (AH, o fluido claro na frente do olho) é uma fonte de alto rendimento de ctDNA que pode ser usada como uma biópsia líquida - ou mais ainda como um substituto para a biópsia do tumor - para avaliar o RB17, 18,19. Com mais de 200 amostras até o momento, foi identificado ctDNA em >95% das amostras, incluindo olhos menos avançados dos grupos A, B e C do IIRC, bem como diferenças genômicas entre os olhos em pacientes bilaterais18,19. Este DNA pode ser isolado com segurança e eficácia para detecção de biomarcadores moleculares relevantes em olhos que estão ativamente em tratamento ou virgens de tratamento 18,20,21. A plataforma de biópsia líquida AH também pode ser usada para identificar as variantes patogênicas diagnósticas de RB1 ou amplificação primária de MYCN que inicia a tumorigênese21,22. É importante ressaltar que identificamos uma assinatura molecular prognóstica - a presença de ganho do cromossomo 6p com uma amplitude de ≥1,5 para a amplificação mediana ou focal do MYCN - que está associada a um aumento de 16,5 na probabilidade de perda do controle do tumor intraocular que requer remoção do olho 18,20,21. Finalmente, foi demonstrado que as alterações na fração tumoral do ctDNA (TFx) na HA se correlacionam com a resposta terapêutica, pois níveis mais altos estão correlacionados com a doença ativa e níveis decrescentes estão associados à resposta positiva ao tratamento23. Dadas essas aplicações e sua potencial utilidade clínica, queríamos delinear a metodologia para biópsia e avaliação de HA. Isso inclui paracentese clara da córnea para aquisição de amostras e o protocolo para análise genômica, especificamente construção e sequenciamento da biblioteca cfDNA, determinação da amplitude SCNA, identificação da variante patogênica RB1 e cálculo de TFx.
Protocolo
Esta pesquisa é conduzida sob a aprovação do Conselho de Revisão Institucional do Hospital Infantil de Los Angeles e da Universidade do Sul da Califórnia e segue os princípios da Declaração de Helsinque. O consentimento informado por escrito é sempre obtido dos responsáveis legais de todos os participantes.
Um esquema para o fluxo de trabalho de biópsia líquida de AH pode ser visto na Figura 1.
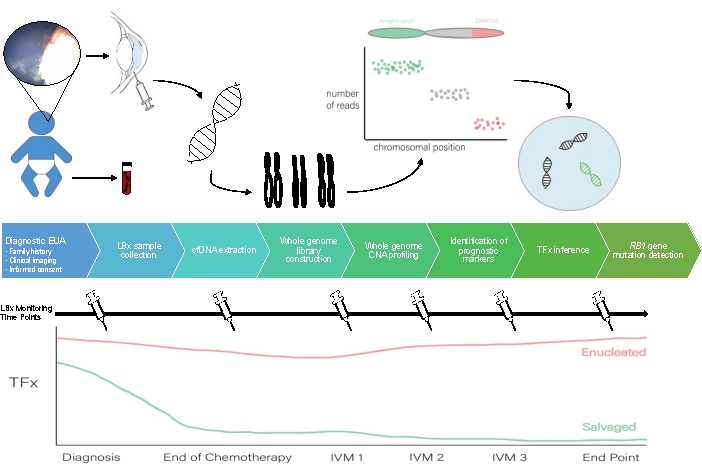
Figura 1: Fluxo de trabalho para a biópsia líquida de HA para acompanhar o protocolo aqui descrito. EUA, exame sob anestesia; LBX, biópsia líquida; cfDNA, DNA livre de células; CNA, alteração do número do exemplar; TFx, fração tumoral; MIV, melfalano intravítreo. Clique aqui para ver uma versão maior desta figura.
1. Procedimento cirúrgico:
NOTA: Este procedimento é realizado durante o exame de rotina sob anestesia (EUA) para avaliação clínica de pacientes com RB. O procedimento de paracentese para extrair HA só deve ser feito por um cirurgião oftalmológico treinado que tenha concluído o treinamento padrão em cirurgia ocular.
- Os critérios de seleção e inclusão dos pacientes são os seguintes.
- Certifique-se de que a câmara esteja formada e limpa, sem rasos do tumor, sinéquias ou cristalino cataractoso.
NOTA: Em alguns olhos avançados, a câmara é muito rasa no diagnóstico para extrair o AH com segurança. Nesses casos, recomenda-se esperar até depois do primeiro ciclo de quimioterapia sistêmica ou intra-arterial. Uma vez que o tumor principal encolhe, a câmara geralmente se aprofunda, o que permite uma abordagem segura. - Certifique-se de que haja uma visão clara de todas as estruturas e que a pressão seja de <22 mm Hg para que não haja um raso rápido da câmara devido à dinâmica da pressão. Isso é verdade para todos os momentos, mas particularmente crítico quando a HA é tomada no momento do diagnóstico.
- Certifique-se de que não haja envolvimento direto do segmento anterior pelo tumor, que é avaliado por visualização direta e biomicroscopia ultrassônica. Esse critério de inclusão é geralmente identificado durante a AUE.
- Certifique-se de que a câmara esteja formada e limpa, sem rasos do tumor, sinéquias ou cristalino cataractoso.
- Uma vez que o paciente tenha sido considerado apropriado e consentido, leve-o para a sala de cirurgia. Permitir que o anestesiologista assistente inicie a anestesia como é rotina para EUA para o atendimento clínico de pacientes com RB.
- Uma vez que o paciente esteja intubado e sedado, e todos os exames clínicos, de imagem e quaisquer necessidades terapêuticas locais (por exemplo, laser ou crioterapia) pertencentes à AUE estejam completos, extraia o AH para a plataforma de biópsia líquida. Prepare e cubra o olho da maneira estéril usual. Isso pode ser feito pela enfermeira circulante ou pelo cirurgião operacional.
- Antes da AUE, coloque as gotas dilatadoras rotineiramente utilizadas na instituição para uma AUE no olho cirúrgico.
NOTA: Aqui, são usados colírios combinados que consistem em 2,5% de fenilefrina, 1% de ciclopentolato e 1% de tropicamida. No entanto, quaisquer gotas padrão usadas para dilatação para EUA são suficientes; o olho não precisa ser dilatado simplesmente para extrair o AH. - Coloque gota de betadina a 5% no olho cirúrgico e aplique com uma esponja começando do centro e movendo-se perifericamente. Inclua as margens palpebrais superior e inferior no preparo cirúrgico. Complete a preparação cirúrgica com betadina três vezes e aguarde pelo menos dois minutos antes de prosseguir.
- Coloque uma toalha azul estéril sobre a cabeça do paciente e coloque-a sob a cabeça para evitar que caia. Seque os cílios e a área periorbital com uma toalha ou gaze azul estéril.
- Deixe o cirurgião se esfregar, vestir um avental e usar luvas.
- Cubra o paciente com cortinas azuis estéreis, deixando o olho cirúrgico exposto. Coloque um espéculo de tampa estéril para uma visualização ideal do campo estéril.
NOTA: Anestesia tópica adicional não é necessária em nossa instituição, pois a criança está sob anestesia geral para a AUE. - Execute o tempo limite de acordo com o protocolo institucional. Aqui, um tempo limite inicial é feito para o EUA e um segundo tempo limite para verificar o(s) olho(s) antes do procedimento.
- Antes da AUE, coloque as gotas dilatadoras rotineiramente utilizadas na instituição para uma AUE no olho cirúrgico.
- Usando um microscópio cirúrgico, execute uma paracentese corneana clara com a extração de 0,1 mL de AH com uma agulha de 32 G em uma seringa de 1 cc.
- Antes de iniciar o procedimento, molhe o olho com uma solução salina estéril balanceada (cada mL contendo cloreto de sódio 0,64%, cloreto de potássio 0,075%, cloreto de cálcio di-hidratado 0,048%, cloreto de magnésio hexa-hidratado 0,03%, acetato de sódio tri-hidratado 0,39%, citrato de sódio di-hidratado 0,17%, hidróxido de sódio e / ou ácido clorídrico (para ajustar o pH) e água para injeção) para manter a lubrificação da córnea; Esta é uma preparação comercial padrão.
- Certifique-se de que a agulha de 32 G está bloqueada na seringa de 1 cc e que não há pressão na seringa (movendo o êmbolo da seringa para dentro e para fora várias vezes antes de usar).
- Em seguida, passe a agulha pela córnea clara no limbo (perpendicular a ela, como é padrão para paracentese) e permaneça dentro da câmara anterior sobre a íris periférica dilatada.
- Mantenha o chanfro da ponta da agulha anteriormente à íris sob visualização direta através do microscópio durante a extração. Se preferir, estabilize o olho com 0,12 pinça à medida que a agulha passa pela córnea.
NOTA: Nunca deve haver contato entre a ponta da agulha e qualquer estrutura ocular; Deve permanecer sobre a íris periférica na câmara anteroântica. - Extraia 0,1 mL de AH. Manipule o êmbolo da seringa com a mão não dominante do cirurgião (mas com prática) ou por um assistente treinado sem mover a agulha. Não perca a visualização direta da ponta da agulha. Priorize a anatomia do olho e tome menos AH, se necessário, para garantir que a câmara permaneça formada.
- Após a extração, remova suavemente a agulha da câmara anterior. Certifique-se de que a câmara permaneça formada, mas ligeiramente rasa; A pressão será suave, mas fisiológica. Depois que a agulha for removida do olho, banhe-o copiosamente com água estéril; A água estéril é preferível a uma solução salina balanceada nesta etapa como medida de segurança, porque a água lisa as células.
- Examine o local da agulha quanto a vazamentos. Se houver vazamento, use um aplicador de ponta de algodão para aplicar uma leve pressão no local da injeção por 30 segundos antes de verificar novamente se há vazamento. Repita o processo, se necessário.
NOTA: O uso de métodos mais invasivos para interromper qualquer vazamento nunca foi necessário com essa abordagem. Às vezes, esse procedimento é seguido por injeção intravítrea de quimioterapia; para este artigo de métodos, o protocolo se concentra na extração de AH como procedimento primário.
- Examine o local da agulha quanto a vazamentos. Se houver vazamento, use um aplicador de ponta de algodão para aplicar uma leve pressão no local da injeção por 30 segundos antes de verificar novamente se há vazamento. Repita o processo, se necessário.
- Uma vez que não há vazamento, o procedimento é considerado concluído. Remova as cortinas estéreis do paciente e limpe a betadina do rosto.
- Remova cuidadosamente a ponta da agulha da seringa, use uma tampa estéril para fechar a amostra via luer lock para não perder nenhuma amostra e rotule a seringa adequadamente.
NOTA: Isso deve ser feito pelo coordenador de pesquisa clínica ou pelo cirurgião enquanto a criança está sendo extubada.- Após a extração da amostra, armazenar a amostra em uma seringa tampada a -80 °C; As amostras podem ser colocadas em gelo seco para serem transportadas da sala de cirurgia para um freezer apropriado.
- Coloque uma única gota de colírio antibiótico de acordo com a preferência do cirurgião na superfície do olho cirúrgico antes que o paciente seja enviado para recuperação. Aqui, colírios combinados de esteróides e antibióticos são usados e o olho não é tapado.
- Uma vez concluído o procedimento, o anestesiologista responsável garante a extubação adequada. Traga o paciente para a recuperação.
- Dê alta ao paciente para casa seguindo o protocolo pós-operatório institucional.
- Informe ao paciente que o olho não precisa ser coberto com nenhum curativo ou cobertura protetora.
- Informar o paciente para não mergulhar completamente a cabeça em uma banheira ou piscina por 24 horas, e os pais para não esfregar o olho do paciente (criança); Não há outras limitações na atividade do paciente.
- Informe ao paciente que é improvável que ele tenha dor da paracentese, mas pode ter dor de procedimentos associados durante a AUE. O paciente pode tomar paracetamol ou ibuprofeno em caso de algum desconforto, conforme sugerido pelo cirurgião.
2. isolamento e purificação do cfDNA
- Realize o isolamento e processamento do DNA dentro de 72 horas após a extração do AH.
- Descongele AH em temperatura ambiente. Verifique a amostra com frequência e avance para a extração imediatamente após a conclusão do descongelamento.
- Extraia o cfDNA do AH e eluia-o em 50 μL de solução AVE (incluída no kit) usando o kit de isolamento e purificação do cfDNA (consulte a Tabela de Materiais para obter detalhes) de acordo com o manual de instruções do fabricante.
3. Sequenciamento de próxima geração (NGS) e controle de qualidade (QC)
- Construa as bibliotecas de DNA para sequenciamento usando o kit de sequenciamento de biblioteca de DNA (consulte a Tabela de Materiais) de acordo com o manual de instruções do fabricante. Amplifique o DNA da biblioteca com 14 ciclos de reação em cadeia da polimerase (PCR), conforme detalhado no manual do kit de sequenciamento da biblioteca de DNA.
NOTA: As bibliotecas finais podem ser armazenadas a -20 °C até 1 mês antes do sequenciamento final para análise genômica. - Sequencie as bibliotecas de DNA na plataforma NGS (consulte a Tabela de Materiais) de acordo com o protocolo do fabricante seguindo o protocolo de 50 ciclos de extremidade única ou protocolo de 150 ciclos de extremidade emparelhada.
NOTA: Outras plataformas NGS funcionarão em princípio (embora não tenham sido testadas aqui), com adaptadores adequados para a célula de fluxo de sequenciamento. - Realize o controle de qualidade para quaisquer amostras de AH que foram coletadas no momento do diagnóstico ou enucleação primária.
NOTA: O CQ não pode ser realizado para amostras coletadas em outros momentos, pois a massa de DNA será muito baixa e fora da faixa de detecção para as etapas descritas abaixo.- Use 1 μL do DNA extraído para o ensaio de quantificação de DNA e o ensaio de perfil de tamanho (consulte a Tabela de Materiais) de acordo com os protocolos do fabricante.
NOTA: O pico deve ser em torno de 300 pb. Se for menor que 150 pb, a amostra foi comprometida. Consulte o arquivo suplementar 1A para obter um exemplo de uma amostra que deve passar no controle de qualidade e o arquivo suplementar 1B para obter um exemplo de uma amostra que não deve.
- Use 1 μL do DNA extraído para o ensaio de quantificação de DNA e o ensaio de perfil de tamanho (consulte a Tabela de Materiais) de acordo com os protocolos do fabricante.
4. Análise de dados de alteração do número de cópias somáticas (SCNA)
- Mapeie as leituras obtidas do NGS (seção 3) para o genoma humano (hg19, Genome Reference Consortium GRCh37, banco de dados do navegador do genoma da Universidade da Califórnia em Santa Cruz) 24 , 25 com um alinhador.
- Remova as duplicatas de PCR (samtools rmdup26).
- Normalize o conteúdo de guanina-citosina calculando a porcentagem de bases de guanina e citosina em cada compartimento do genoma de referência. Um programa de amostra é descrito em Baslan et al.27.
- Para obter números de cópias de segmentos de DNA, divida o genoma em 5000 caixas de comprimento variável e, em seguida, calcule o número relativo de leituras contidas em cada caixa.
- Use taxas de log2 sem referência para determinar as estimativas do número de cópias. Pegue a contagem média da janela de cromossomos autossômicos normais.
- Realize a segmentação usando segmentação binária circular com DNAcopy (Bioconductor28).
- Defina SCNAs como positivos com 20% de deflexão da linha de base (razão log2 = 0), o que significa que perdas em razões log2 ≤ 0,2 (proporção de 0,87 ou inferior define uma deleção) e ganhos em taxas log2 ≥ 0,2 (proporção de 1,15 ou superior define uma amplificação).
NOTA: Esses limiares são consistentes com análises de biópsia líquida previamente estabelecidas27,29.
5. Análise mutacional RB1 de amostras de AH
- Além disso, amplifique todas as bibliotecas do genoma para 500 ng cada para NGS direcionado baseado em captura para detecção de mutação de acordo com o protocolo do fabricante.
- Execute o sequenciamento de extremidade única de 150 bp ou 50 bp de plataforma NGS nas bibliotecas capturadas para >100x por protocolo do fabricante.
- Processe os dados NGS. Neste estudo, um pipeline interno baseado no pipeline bcbio no CHLA Center for Personalized Medicine é utilizado30.
- Corte os dados brutos do fastq de adaptadores e bases de baixa qualidade com o Atropos31 e alinhe-os à referência humana GRCh37 com BWA-MEM e NovoAlign (v3)32,33.
- Marque duplicatas com FreeBayes34 e determine a variante da linha germinativa.
NOTA: Nas ferramentas de chamada de variantes do FreeBayes, cada posição do genoma da amostra é comparada ao genoma de referência e as contagens de alelos são modeladas matematicamente para obter as medidas de verossimilhança do genótipo34. - Realize a anotação de variantes com o Ensembl Variant Effect Predictor (v96)35 seguindo as etapas 5.3.4-5.3.5.
- Use VarDict para determinar variantes somáticas na HA ou tumor sem a amostra de sangue normal pareada (ou seja, cega para a variante da linha germinativa)36.
- Chame de perda de heterozigosidade se as variantes contínuas de uma região tiverem alelos alternativos de ≤3%.
NOTA: O software comercial de análise genômica também pode ser usado com os parâmetros indicados acima.
6. Determinação de cfDNA TFx
- Estime TFx para cada amostra sequenciada de cfDNA AH usando o software ichorCNA (software de estimativa de TFx baseado em CNA) 37 .
NOTA: Este é o software padrão usado em biópsias líquidas baseadas em sangue para determinar cfDNA TFx38. O uso do software na HA foi publicado anteriormente21,23. - Revise individualmente os perfis genômicos e as soluções TFx correspondentes para verificar se a estimativa de TFx para cada amostra é apropriada39.
Resultados
Resultados abrangentes de dois olhos (casos 33 e 47) são apresentados abaixo. Os números de casos permanecem consistentes com publicações anteriores para fins de comparação 18,20,21. Os médicos assistentes desconheciam os resultados da biópsia líquida de HA durante a terapia. Todas as decisões de tratamento foram não randomizadas e tomadas de acordo com o padrão de tratamento de rotina publicado anteriormente40,41. Os dados dos desfechos clínicos permaneceram separados dos dados genômicos até a análise final. Os dados brutos dos resultados apresentados estão disponíveis mediante solicitação ao autor correspondente. Devido ao financiamento do NIH, os dados são regidos pela política de compartilhamento de dados genômicos do NIH e estarão disponíveis para outros pesquisadores por meio de um repositório de dados designado pelo NIH de acesso controlado (dbGAP) no futuro; Também está disponível mediante solicitação do autor correspondente.
Os casos 33 e 47 são ambos olhos do Grupo D2 do IIRC que tiveram apresentações clínicas muito semelhantes. Assim, eles foram tratados com terapia de salvamento do globo ocular por decisão dos pais e tratamento de oncologistas oculares e médicos - especificamente com quimioterapia sistêmica para o caso 47 e quimioterapia intra-arterial para o caso 33. Com base no prognóstico clínico atualmente aceito que se baseia no grupo IIRC do olho2, o sucesso previsto do salvamento do globo ocular para os olhos desses dois pacientes teria sido o mesmo: 65%-70% para o Grupo D é uma média, embora isso varie de acordo com o centro de tratamento41. No entanto, com base nos dados do Grupo D coletados até agora do perfil molecular direcionado ao tumor neste centro, o sucesso previsto do salvamento do globo ocular seria de 72% para o caso 47 (sem ganho de 6p identificado no AH) e 9% para o caso 33 (com ganho de 6p identificado)18,20,21. Isso é demonstrado abaixo.
O caso 47 é um exemplo de um olho com detecção bem-sucedida de SCNA e SNV usando a plataforma de biópsia líquida AH no momento do diagnóstico, juntamente com tendências de TFx correspondentes à resposta ao tratamento longitudinalmente. A paciente é do sexo feminino que se apresentou aos 15 meses de idade com uma RB de 14 mm x 9 mm IIRC Grupo D, estágio cT2b com semeadura vítrea esférica. Ela foi negativa para mutação germinativa RB1 , conforme determinado por testes clínicos de leucócitos séricos de rotina. A partir da HA, foram identificados ganhos 1q e 6p nos SCNAs RB no momento do diagnóstico, além de outros dois RB SCNAs não altamente recorrentes 7p loss e 13q gain (Figura 2). É importante notar que a amplitude do ganho de 6p foi de 1,2, e apenas amplitudes de ≥ proporção de 1,5 para a mediana demonstraram pressagiar um prognóstico ruim. Dado que não houve amplificação focal do MYCN e o ganho de 6p estava abaixo do limite de 1,5, o prognóstico para resgate com tratamento baseado em características moleculares foi alto. A mesma amostra de AH coletada no diagnóstico também foi avaliada para detecção de variantes patogênicas RB1 , que revelaram o SNV c.958C>T, p.Arg320* dentro do gene RB1 com uma frequência de alelos variantes de 87,01% (intervalo de confiança de 95%, 79,7%-94,6%). Este paciente foi tratado com seis ciclos de Carboplatina, Etoposídeo e Vincristina (CEV) com regressão da doença, mas demonstrou semeadura persistente em forma de esfera e poeira que exigiu três injeções sequenciais de melfalano intravítreo (IVM). Durante o tratamento com IVM, AS AMOSTRAS DE AH A-C (cada uma separada por duas semanas) demonstraram uma normalização completa do perfil genômico, diminuição do TFx e diminuição da concentração de DNA - tudo concomitante com a regressão clínica da doença (Figura 3). Após o diagnóstico, os valores de TFx permaneceram abaixo do limite de detecção de 5% pelo restante do tratamento. Aos 19 meses de acompanhamento, o olho permaneceu estável, sem recorrência do tumor ou disseminação extraocular da doença.
Isso contrasta com o caso 33. Este homem de 22 meses de idade teve uma apresentação clínica geral semelhante com uma massa retiniana de 11 mm x 18 mm e semeadura vítrea do tipo poeira, consistente com IIRC unilateral Grupo D, estágio cT2b. Ele também foi negativo para mutação germinativa RB1, conforme determinado por testes clínicos de leucócitos séricos de rotina. A HA tomada no momento do diagnóstico demonstrou ganho de 1q de SCNAs RB, ganho de 6p (neste caso com uma amplitude de 1,5 razão para a mediana) e perda de 16q juntamente com perda focal de 6q (Figura 4A). Com base no perfil molecular prognóstico, este olho teve uma chance aumentada de 16,5 de enucleação com base na presença de amplitude de 6p ≥ 1,5. Nenhum SNV RB1 foi identificado nesta amostra, apesar da cobertura total do comprimento total do gene RB1. Uma razão pela qual um SNV RB1 pode não ser identificado é em tumores primários causados por MYCN, onde uma mutação RB1 concomitante nem sempre é esperada 14,15,42,43,44,45. No entanto, o caso 33 não demonstrou evidência de amplificação do MYCN em nenhuma de suas amostras de AH ou em seu tecido tumoral enucleado (Figura 5). Uma explicação mais provável para um resultado negativo de RB1 SNV é que a tumorigênese inicial foi impulsionada por desregulação epigenética (por exemplo, metilação do promotor) 46 , 47 , um fenômeno conhecido em RB que não seria identificado pelo ensaio aqui descrito.
O tratamento inicial para este paciente foi de quatro ciclos totais de quimioterapia intra-arterial com melfalano, seguidos por quatro injeções de IVM devido à disseminação vítrea persistente. Três amostras de HA (cada uma separada por quatro semanas) foram obtidas durante a terapia com MIV e demonstraram os mesmos três SCNAs que estavam presentes no momento do diagnóstico (Figura 5). Os valores de TFx permaneceram altos durante todo o tratamento, apesar da diminuição do volume do tumor primário refletindo as sementes tumorais ativas no vítreo. Isso demonstra como o TFx é representativo do estado geral da doença no olho. Seis meses após o diagnóstico, devido à doença ativa persistente, o olho foi enucleado. O perfil genômico obtido do tecido tumoral naquele momento demonstrou 92,81% de concordância com a amostra de AH obtida no momento do diagnóstico (Figura 4B).
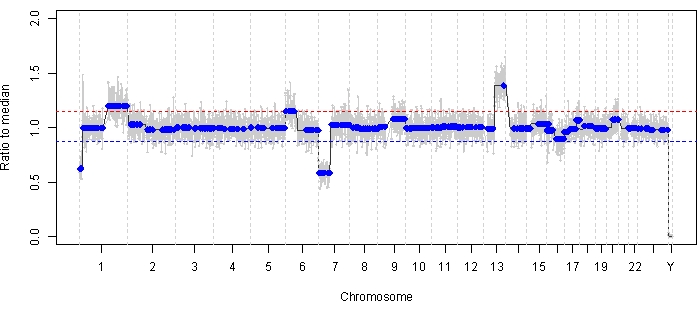
Figura 2: Perfil genômico no momento do diagnóstico para o caso 47. Ganho de 1q e ganho de 6p de SCNAs RB altamente recorrentes, juntamente com perda de 7p e ganho de 13q de SCNAs RB não altamente recorrentes, foram identificados no AH tomado no momento do diagnóstico. A linha vermelha representa o limite para um ganho, enquanto a linha azul representa o limite para uma perda. Notavelmente, a amplitude do ganho de 6p foi de < proporção de 1,5 para a mediana, que está abaixo do limite da assinatura molecular que pressagia um prognóstico ruim. Assim, com base na falta de biomarcadores negativos para o salvamento do olho, esse olho seria previsto para responder à terapia. Clique aqui para ver uma versão maior desta figura.
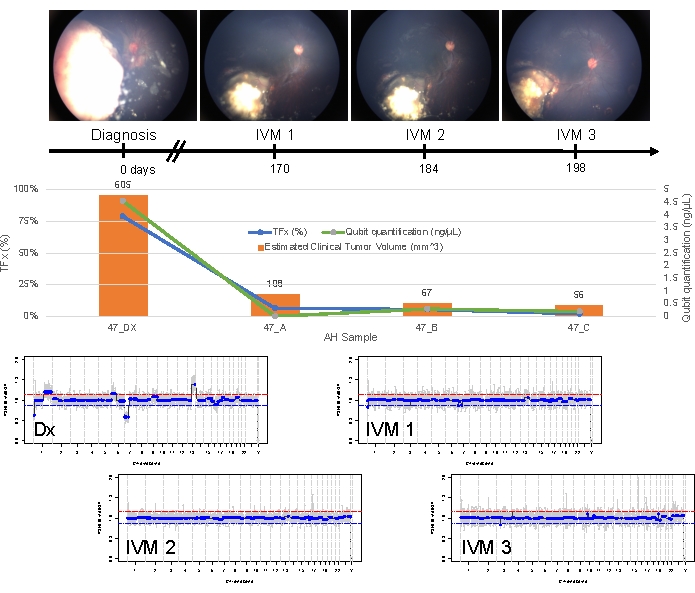
Figura 3: Informações longitudinais para o caso 47. Isso inclui fotos de fundo de olho, quantificação de cfDNA, volume clínico estimado do tumor a partir de medições de B-scan, perfis genômicos e estimativas de TFx para cada ponto de tempo clínico em que a HA foi amostrada (Dx = diagnóstico, A = IVM1, B = IVM2, C = IVM3). Este olho respondeu ao tratamento e permaneceu recuperado aos 19 meses de acompanhamento. Uma diminuição na quantidade de cfDNA foi observada ao longo do tempo, consistente com estudos publicados anteriormente mostrando o maior rendimento obtido no diagnóstico. O TFx também diminuiu ao longo do tratamento, refletindo a resolução da semeadura e a diminuição do volume do tumor principal da retina (os volumes tumorais são indicados acima da barra laranja de cada ponto de tempo clínico). Como esperado com a regressão clínica da doença, os perfis genômicos também se normalizaram. Nos perfis genômicos, a linha vermelha representa o limite para um ganho, enquanto a linha azul representa o limite para uma perda. Esta figura foi reimpressa com permissão de Xu, L. et al.21. Clique aqui para ver uma versão maior desta figura.
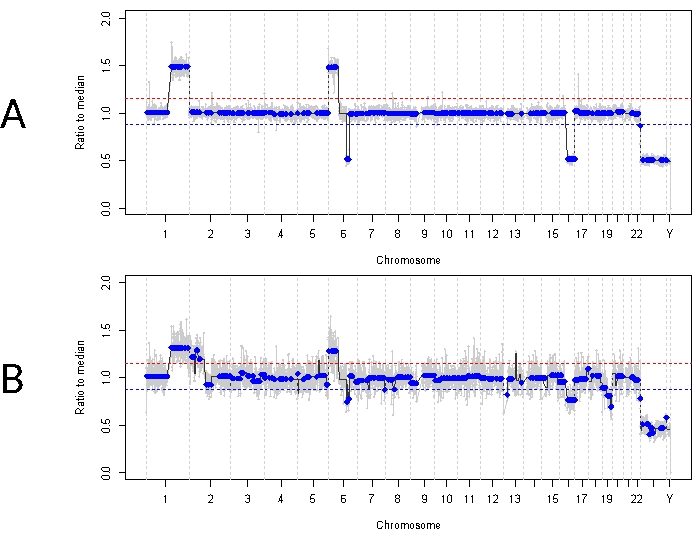
Figura 4: Perfis genômicos para o caso 33. A linha vermelha representa o limite para um ganho, enquanto a linha azul representa o limite para uma perda (A) Perfil genômico no diagnóstico para o caso 33. Ganhos de 1q, 6p e 16q de RB SCNAs altamente recorrentes foram identificados na HA no momento do diagnóstico, além da perda focal de 6q. Notavelmente, a amplitude do ganho de 6p foi de 1,5 em relação à mediana, indicando um biomarcador molecular de mau prognóstico. Assim, com base nessa assinatura molecular, preveríamos que esse olho tinha uma probabilidade significativamente maior de falha do tratamento. (B) Perfil genômico obtido de tecido tumoral enucleado, que foi altamente concordante com o perfil obtido de HA no diagnóstico. Devido à mistura com o tecido retiniano normal, os SCNAs do tecido tumoral podem demonstrar menor amplitude em comparação com o AH devido ao TFx diluído. Clique aqui para ver uma versão maior desta figura.
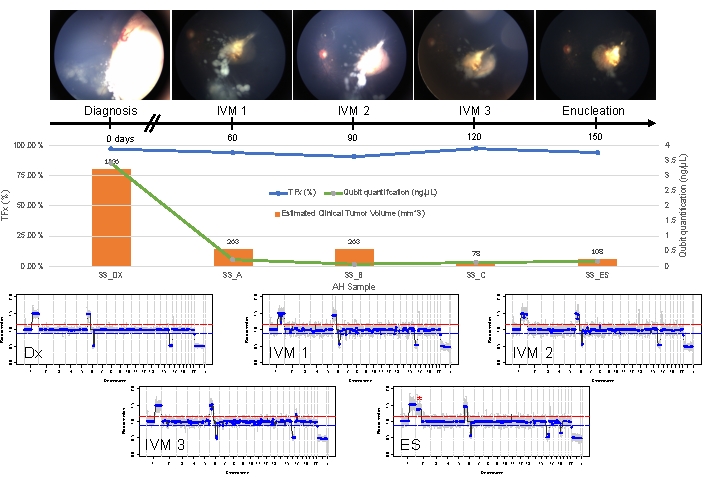
Figura 5: Informações longitudinais para o Caso 33. Isso inclui fotos de fundo de olho, quantificação de DNA livre de células, volume clínico estimado do tumor a partir de medições de B-scan (os volumes tumorais são indicados acima da barra laranja de cada ponto de tempo clínico), perfis genômicos e estimativas de TFx para cada ponto de tempo clínico em que a HA foi amostrada (Dx = diagnóstico, A = IVM1, B = IVM2, C = IVM3, SE = enucleação secundária). Este olho não respondeu ao tratamento, necessitando de enucleação secundária (ES). Isso ocorreu devido à semeadura persistentemente ativa acompanhada por uma recorrência do tumor apical. A quantidade de CfDNA diminuiu ao longo do tempo, consistente com os estudos publicados anteriormente que mostraram o maior rendimento obtido no diagnóstico (ganho de 1q, ganho de 6p, perda de 16q e perda focal de 6q). No entanto, o TFx permaneceu alto durante todo o tratamento, um produto da semeadura persistente que ainda libera cfDNA derivado do tumor no AH. Os perfis genômicos foram consistentes e mostraram os mesmos três SCNAs que estavam presentes no momento do diagnóstico. No AH obtido no ES, um novo ganho de 2p em larga escala (*) e perda de 19q foram observados, sugerindo evolução clonal no momento da recorrência do tumor apical. Nos perfis genômicos, a linha vermelha representa o limite para um ganho, enquanto a linha azul representa o limite para uma perda. Esta figura foi reimpressa com permissão de Xu, L. et al.21. Clique aqui para ver uma versão maior desta figura.
Arquivo suplementar 1: Um exemplo de (A) uma amostra que deve passar no controle de qualidade, com o pico em torno de 300 pb e (B) uma amostra que não deve passar no controle de qualidade, com o pico em torno de 150 pb. Clique aqui para baixar este arquivo.
Discussão
A paracentese clara da córnea é um procedimento realizado comumente para múltiplas indicações diagnósticas e/ou terapêuticas em oftalmologia. Especificamente para RB, faz parte do protocolo padrão de injeção de quimioterapia intravítrea para diminuir a pressão intraocular antes da injeção para evitar refluxo para o local da injeção48. Apesar de ser um procedimento comum, não é totalmente isento de riscos; o dogma anterior era que uma agulha nunca deveria entrar em um olho com RB ativo. Esse dogma evoluiu na última década, primeiro por meio do trabalho formativo de Patricia Chévez-Barrios sobre terapia de vetor adenoviral injetado49, seguido por métodos progressivos de segurança aprimorada para injeções oculares em RB por Francis Munier48. Este trabalho abriu caminho para que a extração do AH seja usada como biópsia líquida, ou substituta da biópsia tumoral, para esse câncer. Agora somos o primeiro centro a relatar a utilidade da plataforma e os resultados preliminares de segurança da HA tomada no momento do diagnóstico. Parte dos métodos aqui descritos destina-se a garantir a segurança da seleção do paciente e os aspectos processuais da extração de AH para RB. Em primeiro lugar, as agulhas só podem entrar na câmara anterior e não devem entrar em contato com a íris ou o cristalino, pois isso pode causar cicatrizes na íris ou catarata que limitam a capacidade do cirurgião de monitorar o tumor. É muito importante que a agulha nunca entre na cavidade vítrea (a menos que combinada com a administração de quimioterapia, como é o caso da MIV), ou entre em contato com o tumor, pois isso hipoteticamente eleva o risco de disseminação do tumor e extensão extraocular da doença 3,4,5. Ambas as estruturas são posteriores à câmara anterior e separadas dela pelo cristalino e pela íris. Para evitar danos a quaisquer estruturas oculares, é importante manter o bisel da agulha sempre levantado, acima da íris periférica, e sempre sob visualização direta usando o microscópio cirúrgico. Existe o risco de vazamento menor do AH do local da agulha; Se isso ocorrer, ele será resolvido com uma leve pressão por meio de um aplicador de ponta de algodão. Usar a menor agulha de calibre disponível e garantir que a agulha seja extraída lentamente do mesmo trato sem qualquer cisalhamento lateral diminuirá esse risco. Embora seja esperado um leve raso da câmara anterior, a câmara deve permanecer formada sem toque da córnea da íris e a pressão intraocular deve ser suave, mas fisiológica.
No que diz respeito às análises genômicas, existem várias etapas críticas envolvidas. O mais importante é o manuseio da amostra de pequeno volume uma vez extraída do olho; é crucial que a amostra permaneça congelada o tempo todo antes do processamento para evitar a degradação do DNA que pode ocorrer com ciclos repetidos de congelamento e descongelamento50. Garantir que as amostras sejam colocadas em gelo seco imediatamente após a extração e transferidas para um freezer a -80 °C ajuda a garantir isso. Uma vez que as amostras tenham começado a ser processadas, as etapas de controle de qualidade são a principal oportunidade para solucionar problemas, garantindo que o cfDNA seja construído em bibliotecas de alta qualidade. Usando a quantificação de DNA e o ensaio de perfil de tamanho de fragmento de DNA, o controle de qualidade pode ser realizado em amostras coletadas durante a enucleação primária ou no momento do diagnóstico devido ao maior rendimento de cfDNA presente nessas amostras18,21. Quando o pico obtido é em torno de 300 pb, isso garante que o cfDNA será reconhecido pela plataforma NGS. Se o pico obtido for inferior a 150 pb, indicando que a maioria dos fragmentos detectados são primers de construção de biblioteca ou oligos adaptadores, a amostra foi comprometida e não deve ser processada posteriormente para SCNAs. No entanto, em nossa experiência de processamento de centenas de amostras, menos de 5% precisam ser removidos para controle de qualidade devido à baixa taxa de alinhamento de leituras20. Juntamente com os métodos que padronizamos para coleta, manuseio, armazenamento e processamento de amostras, este procedimento demonstra a validade pré-analítica da plataforma de biópsia líquida AH.
A biópsia líquida de AH também estabeleceu validade analítica com base em sua capacidade de detectar com precisão e confiabilidade mutações patogênicas RB1 e SCNAs, com concordâncias médias consistentemente >95% entre os perfis genômicos gerados a partir de amostras de AH e tecido tumoral correspondente 18,20,21,22,23. Apesar da validade analítica demonstrada da plataforma, ela não é isenta de limitações. A análise mutacional RB1 só pode ser realizada em amostras com >10 ng de cfDNA, que são mais frequentemente obtidas em olhos virgens de tratamento no momento do diagnóstico ou da enucleação primária18,21. Isso se deve à menor concentração de cfDNA presente nas amostras de HA de olhos que estão ativamente em tratamento em comparação com a HA no diagnóstico ou no momento da enucleação primária. Uma restrição adicional é que os SCNAs não podem ser detectados em TFxs abaixo de 5%, o que impede o monitoramento da doença em olhos em que a carga tumoral caiu significativamente; Em nossa experiência, isso ocorre mais comumente em olhos responsivos ao tratamento quimioterápico intravítreo23.
Para determinar o TFx do cfDNA no AH, é utilizado um software de estimativa de TFx baseado em CNA. Este software é uma ferramenta padrão e aceita para o cálculo de TFx em biópsias líquidas, e seu algoritmo foi descrito em detalhes38,51. Resumidamente, o software de estimativa TFx baseado em CNA prevê SCNAs em larga escala dentro do cfDNA sequenciado utilizando um modelo de Markov oculto. As estimativas de TFx são derivadas com base na presença de SCNAs, levando em conta as diferenças na subclonalidade e ploidia em cada locus e, a partir delas, o software de estimativa de TFx baseado em CNA escolhe uma solução de TFx ideal38. No entanto, uma limitação inerente ao software de estimativa de TFx baseado em CNA é que o TFx é calculado com base na presença de SCNAs em uma amostra; portanto, não é possível determinar TFx em amostras sem SCNAs (ou seja, com perfis genômicos planos)38. Como foi demonstrado anteriormente, nem todos os tumores RB apresentam SCNAs 14,15,18,21,23,45. Portanto, um TFx muito baixo, conforme determinado pelo software de estimativa de TFx baseado em CNA, significa que 1) a amostra de AH não contém cfDNA derivado de tumor mensurável ou 2) o cfDNA do tumor está presente, mas é indetectável pelo software de estimativa de TFx baseado em CNA devido à falta de SCNAs38. A fração alélica variante (VAF) para SNV é um substituto para TFx. Por esse motivo, estamos trabalhando para desenvolver um pipeline TFx baseado em RB1 baseado em VAF para permitir que os olhos RB sem SCNAs também recebam monitoramento longitudinal de TFx. Dado que todos os tumores RB, com exceção dos tumores primários 14,15,42,43,44,45, contêm mutações somáticas no gene RB1, um pipeline não dependente de SCNAs ampliaria a aplicação do protocolo de biópsia líquida aqui apresentado. Além disso, como os SNVs podem ser monitorados para TFxs abaixo de 5%, isso aumentaria a sensibilidade de nossa plataforma.
A plataforma de biópsia líquida AH descrita aqui não é a única plataforma de biópsia líquida que existe na literatura para RB, mas, notavelmente, é a primeira a descrever o aquoso como uma fonte enriquecida de DNA tumoral e a primeira a descrever a detecção de ctDNA por meio de dois mecanismos (SCNAs e RB1 SNVs) em qualquer biofluido com base no trabalho publicado até o momento. Com a capacidade de detectar ctDNA de duas maneiras, a biópsia líquida de AH tem uma sensibilidade maior do que outras plataformas na literatura. Por exemplo, outro grupo detectou com sucesso o ctDNA RB no AH com SNVs RB1 52. No entanto, esta plataforma dependia de leituras NGS direcionadas com base no conhecimento a priori do SNV. Em contraste, a plataforma detalhada neste manuscrito utiliza WGS não tendencioso, dando-lhe a capacidade de detectar SCNAs e SNVs. Biópsias líquidas baseadas em sangue também foram tentadas, embora o cfDNA obtido tenha estado consistentemente abaixo do limite para detecção de SCNA, que é o prognóstico para a probabilidade de salvamento ocular (neste momento, os SNVs RB1 não demonstraram ser prognósticos para salvamento ocular, no entanto, pode haver um papel evolutivo para a detecção de doença metastática)19. Kothari e col. descreveram SNVs RB1 no plasma de pacientes com RB, mas apenas naqueles com doença intraocular avançada que requer enucleação16. A plataforma de biópsia líquida de AH aqui descrita tem a capacidade de detectar ctDNA na HA de olhos menos avançados, mesmo sem a presença de semeadura19,21. Além disso, as biópsias líquidas baseadas em sangue não são específicas para os olhos, pois o ctDNA isolado do soro pode ser de ambos os olhos em casos de RB bilateral. Isso limita a utilidade clínica das plataformas baseadas no sangue, particularmente em pacientes bilaterais que representam 40% dos casos de RB, enquanto a HA permanece específica para os olhos e pode demonstrar heterogeneidade interocular nos níveis de SNV e SCNA53.
A importância da biópsia líquida de AH para o campo de RB é fundamental. A biópsia líquida AH não apenas oferece a oportunidade de entender melhor a dinâmica intratumoral em olhos que estão ativamente em terapia, mas também tem o potencial de melhorar o atendimento ao paciente. Com base em estudos anteriores, estabelecemos uma assinatura molecular - baseada na presença de amplificação de MYCN ou ganho do cromossomo 6p com uma amplitude de proporção de ≥ 1,5 para a mediana - que é prognóstica para uma probabilidade 16,5 vezes maior de falha do tratamento que requer enucleação 18,20,21. Com esse conhecimento no momento do diagnóstico, os médicos estariam mais aptos a aconselhar as famílias sobre as opções de tratamento apropriadas e a probabilidade de salvamento ocular com a terapêutica atual. Embora a validade clínica da plataforma de biópsia líquida AH para RB tenha sido estabelecida 17,18,19,20,21,22,23, ela está atualmente aprovada apenas para pesquisa; estudos multicêntricos prospectivos maiores são necessários antes que a biópsia líquida de HA possa ser implementada clinicamente para ajudar a direcionar o atendimento ao paciente para RB. Independentemente disso, a biópsia líquida AH tem o potencial de permitir oncologia de precisão no futuro, não apenas para RB, mas também para outros tumores oculares.
Divulgações
Jesse Berry, Liya Xu e James Hicks entraram com um pedido de patente intitulado DNA livre de células de humor aquoso para avaliação diagnóstica e prognóstica de doenças oftálmicas. Caso contrário, os autores não relatam nenhum potencial conflito de interesses.
Agradecimentos
Esta pesquisa foi apoiada pelas seguintes fontes: NCI do NIH Award K08CA232344 (para JL Berry); Hyundai Hope on Wheels RGA012351 (para J. L. Berry); Fundo de Câncer de Olho Infantil (para JL Berry); Sociedade Americana do Câncer IRG-16-181-57 (para J. L. Berry); Fundação Wright (para J.L. Berry e M.E. Kim); Fundação de Olhos dos Cavaleiros Templários (para JL Berry); A Fundação Larry e Celia Moh (para JL Berry); Instituto para Famílias, Inc., Hospital Infantil de Los Angeles (JL Berry); uma bolsa departamental irrestrita da Research to Prevent Blindness (todas); O NCI P30CA014089 (todos); Fundo de Pesquisa Vicky Joseph (para P. Kuhn); Fundo de Pesquisa Carol Vassiliadis (para P. Kuhn); e Faculdade de Letras, Artes e Ciências da USC Dornsife (para P. Kuhn).
Materiais
| Name | Company | Catalog Number | Comments |
| 1 cc syringe | surgical grade, whatever available in hospital | ||
| 32 G needle | surgical grade, whatever available in hospital | ||
| Aligner | Authors use Bowtie2 (http://bowtie-bio.sourceforge.net/bowtie2/index.shtml) but other aligners such as BWA or GRCh38 will also work | ||
| Atropos | generic term: adapter remover. https://atropos.readthedocs.io/en/latest/index.html# | ||
| Bioanalyzer High Sensitivity DNA Kit | Agilent | 5067-4626 | generic term: DNA fragment size profiling assay |
| BWA-MEM | generic term: long sequence aligner. http://bio-bwa.sourceforge.net/bwa.shtml | ||
| DNAcopy | Bioconductor | generic term: DNA copy number data analysis. https://bioconductor.org/packages/release/bioc/html/DNAcopy.html | |
| dsDNA High Sensitivity Assay | Qubit | Q32851 | generic term: DNA quantification assay |
| FreeBayes | generic term: sequence variant determiner. https://github.com/freebayes/freebayes | ||
| ichorCNA software | generic term: CNA-based TFx estimation. https://github.com/broadinstitute/ichorCNA | ||
| Illumina platform | Illumina | generic term: NGS platform; please note that other NGS platforms will work in principle, but have not been trialed by these authors | |
| NovoAlign (v3) | Novocraft | generic term: mapping of short reads onto reference genome. http://www.novocraft.com/products/novoalign/ | |
| QIAamp Circulating Nucleic Acid Kit | Qiagen | 55114 | generic term: cfDNA isolation and purification kit |
| QIAseq Ultralow Input Library Kit | Qiagen | 180492 | generic term: DNA library sequencing kit |
| Samtools rmdup | generic term: tool to remove duplicate reads. http://www.htslib.org/doc/samtools-rmdup.html | ||
| VarDict | generic term: variant caller. https://github.com/AstraZeneca-NGS/VarDict | ||
| Variant Effect Predictor | Ensembl | generic term: variant effect determinator. https://uswest.ensembl.org/info/docs/tools/vep/index.html |
Referências
- Fernandes, A. G., Pollock, B. D., Rabito, F. A. Retinoblastoma in the United States: A 40-year incidence and survival analysis. Journal of Pediatric Ophthalmology & Strabismus. 55 (3), 182-188 (2018).
- Linn, A. M. Intraocular retinoblastoma: the case for a new group classification. Ophthalmology Clinics of North America. 18 (1), 41-53 (2005).
- Shields, J. A., Shields, C. L., Ehya, H., Eagle, R. C., De Potter, P. Fine-needle aspiration biopsy of suspected intraocular tumors. The 1992 Urwick lecture. Ophthalmology. 100 (11), 1677-1684 (1993).
- Karcioglu, Z. A., Gordon, R. A., Karcioglu, G. L. Tumor seeding in ocular fine needle aspiration biopsy. Ophthalmology. 92 (12), 1763-1767 (1985).
- Karcioglu, Z. A. Fine needle aspiration biopsy (FNAB) for retinoblastoma. Retina. 22 (6), 707-710 (2002).
- Eide, N., Syrdalen, P., Walaas, L., Hagmar, B. Fine needle aspiration biopsy in selecting treatment for inconclusive intraocular disease. Acta Ophthalmologica Scandinavica. 77 (4), 448-452 (1999).
- Eide, N., Walaas, L. Fine-needle aspiration biopsy and other biopsies in suspected intraocular malignant disease: a review. Acta Ophthalmologica. 87 (6), 588-601 (2009).
- Eriksson, O., Hagmar, B., Ryd, W. Effects of fine-needle aspiration and other biopsy procedures on tumor dissemination in mice. Cancer. 54 (1), 73-78 (1984).
- Ali, M. J., Honavar, S. G., Vemuganti, G. K., Singh, A. D. Fine needle aspiration biopsy of retinal tumors. Monographs in Clinical Cytology. 21, 72-81 (2012).
- Di Nicolantonio, F. a. l., et al. The chemosensitivity profile of retinoblastoma. Recent Results in Cancer Research. 161, 73-80 (2003).
- Francis, J. H., et al. Efficacy and toxicity of second-course ophthalmic artery chemosurgery for retinoblastoma. Ophthalmology. 122 (5), 1016-1022 (2015).
- Francis, J. H. Efficacy and toxicity of intravitreous chemotherapy for retinoblastoma: Four-year experience. Ophthalmology. 124 (4), 488-495 (2017).
- Francis, J. H., et al. Current treatment of bilateral retinoblastoma: The impact of intraarterial and intravitreous chemotherapy. Neoplasia. 20 (8), 757-763 (2018).
- Rushlow, D. E., et al. Characterisation of retinoblastomas without RB1 mutations: genomic, gene expression, and clinical studies. The Lancet Oncology. 14 (4), 327-334 (2013).
- Afshar, A. R., et al. Next-generation sequencing of retinoblastoma identifies pathogenic alterations beyond RB1 inactivation that correlate with aggressive histopathologic features. Ophthalmology. 127 (6), 804-813 (2020).
- Kothari, P. Cell-free DNA profiling in retinoblastoma patients with advanced intraocular disease: An MSKCC experience. Cancer Medicine. 9 (17), 6093-6101 (2020).
- Berry, J. L., et al. Potential of aqueous humor as a surrogate tumor biopsy for retinoblastoma. JAMA Ophthalmology. 135 (11), 1221-1230 (2017).
- Berry, J. L., et al. Genomic cfDNA analysis of aqueous humor in retinoblastoma predicts eye salvage: the surrogate tumor biopsy for retinoblastoma. Molecular Cancer Research. 16 (11), 1701-1712 (2018).
- Berry, J. L., et al. Aqueous humor is superior to blood as a liquid biopsy for retinoblastoma. Ophthalmology. 127 (4), 552-554 (2020).
- Xu, L., et al. Chromosome 6p amplification in aqueous humor cell-free DNA Is a prognostic biomarker for retinoblastoma ocular survival. Molecular Cancer Research. 18 (8), 1166-1175 (2020).
- Xu, L., et al. Establishing the clinical utility of ctDNA analysis for diagnosis, prognosis, and treatment monitoring of retinoblastoma: The aqueous humor liquid biopsy. Cancers. 13 (6), (2021).
- Xu, L. Simultaneous identification of clinically relevant RB1 mutations and copy number alterations in aqueous humor of retinoblastoma eyes. Ophthalmic Genetics. 41 (6), 526-532 (2020).
- Polski, A., et al. Longitudinal aqueous humor sampling reflects treatment response in retinoblastoma patients. Investigative Ophthalmology & Visual Science. 61 (7), 1394 (2020).
- . Genome Browser Available from: https://genome.ucsc.edu/cgi-bin/hgTracks?db=hg19&lastVirtModeType=default&lastVirtModeExtraState=&virtModeType=default&3A15578261%_2D15621068&hgsid=1135765621_qIuA0U (2021)
- Navarro Gonzalez, J., et al. The UCSC genome browser database: 2021 update. Nucleic Acids Research. 49 (1), 1046-1057 (2021).
- Li, H. A statistical framework for SNP calling, mutation discovery, association mapping and population genetical parameter estimation from sequencing data. Bioinformatics. 27 (21), 2987-2993 (2011).
- Baslan, T., et al. Genome-wide copy number analysis of single cells. Nature Protocols. 7 (6), 1024-1041 (2012).
- Huber, W., et al. Orchestrating high-throughput genomic analysis with Bioconductor. Nature Methods. 12 (2), 115-121 (2015).
- Baslan, T., et al. Erratum: Genome-wide copy number analysis of single cells. Nature Protocols. 11 (3), 1024-1041 (2016).
- Didion, J. P., Martin, M., Collins, F. S. Atropos: specific, sensitive, and speedy trimming of sequencing reads. PeerJ. 5, 3720 (2017).
- Li, H. Aligning sequence reads, clone sequences and assembly contigs with BWA-MEM. arXiv. , (2013).
- Garrison, E., Marth, G. Haplotype-based variant detection from short-read sequencing. arXiv. , (2012).
- McLaren, W., et al. The ensembl variant effect predictor. Genome Biology. 17 (1), 122 (2016).
- Lai, Z., et al. VarDict: a novel and versatile variant caller for next-generation sequencing in cancer research. Nucleic Acids Research. 44 (11), 108 (2016).
- . GitHub Available from: https://github.com/broadinstitute/ichorCNA (2017)
- Adalsteinsson, V. A., et al. Scalable whole-exome sequencing of cell-free DNA reveals high concordance with metastatic tumors. Nature Communications. 8 (1), 1-13 (2017).
- . GitHub Available from: https://github.com/broadinstitute/ichorCNA/wiki/Interpreting-ichorCNA-results (2017)
- Berry, J. L., et al. Long-term outcomes of Group D retinoblastoma eyes during the intravitreal melphalan era. Pediatric Blood & Cancer. 64 (12), (2017).
- Berry, J. L., et al. Long-term outcomes of Group D eyes in bilateral retinoblastoma patients treated with chemoreduction and low-dose IMRT salvage. Pediatric Blood & Cancer. 60 (4), 688-693 (2013).
- Francis, J. H., et al. Molecular changes in retinoblastoma beyond RB1: Findings from next-generation sequencing. Cancers (Basel). 13 (1), (2021).
- Schwermer, M., et al. Comprehensive characterization of RB1 mutant and MYCN amplified retinoblastoma cell lines. Experimental Cell Research. 375 (2), 92-99 (2019).
- Davies, H. R. Whole-genome sequencing of retinoblastoma reveals the diversity of rearrangements disrupting RB1 and uncovers a treatment-related mutational signature. Cancers. 13 (4), 754 (2021).
- Lillington, D. M., et al. Comparative genomic hybridization of 49 primary retinoblastoma tumors identifies chromosomal regions associated with histopathology, progression, and patient outcome. Genes, Chromosomes and Cancer. 36 (2), 121-128 (2003).
- Greger, V., et al. Frequency and parental origin of hypermethylated RB1 alleles in retinoblastoma. Human Genetics. 94 (5), 491-496 (1994).
- Raizis, A. M., et al. DNA hypermethylation/boundary control loss identified in retinoblastomas associated with genetic and epigenetic inactivation of the RB1 gene promoter. Epigenetics. 1-15, (2020).
- Munier, F. L., et al. Profiling safety of intravitreal injections for retinoblastoma using an anti-reflux procedure and sterilisation of the needle track. British Journal of Ophthalmology. 96 (8), 1084-1087 (2012).
- Chévez-Barrios, P., et al. Response of retinoblastoma with vitreous tumor seeding to adenovirus-mediated delivery of thymidine kinase followed by ganciclovir. Journal of Clinical Oncology. 23 (31), 7927-7935 (2005).
- Shao, W., Khin, S., Kopp, W. C. Characterization of effect of repeated freeze and thaw cycles on stability of genomic DNA using pulsed field gel electrophoresis. Biopreservation and Biobanking. 10 (1), 4-11 (2012).
- Choudhury, A. D., et al. Tumor fraction in cell-free DNA as a biomarker in prostate cancer. JCI Insight. 3 (21), (2018).
- Gerrish, A., et al. Non-invasive diagnosis of retinoblastoma using cell-free DNA from aqueous humour. British Journal of Ophthalmology. 103 (5), 721-724 (2019).
- Wong, E. Y., Xu, L., Shen, L. Inter-eye genomic heterogeneity in bilateral retinoblastoma via aqueous humor liquid biopsy. NPJ Precis Oncol. 5 (1), 73 (2021).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados