Method Article
Kammerwasser als Flüssigbiopsie beim Retinoblastom: Klare Hornhautparazentese und genomische Analyse
In diesem Artikel
Zusammenfassung
Das Kammerwasser ist eine Flüssigbiopsie mit hoher Ausbeute für Retinoblastom, intraokularen Krebs, der aufgrund des Risikos einer extraokulären Ausbreitung nicht in vivo biopsiert werden kann. Hierin werden ein Verfahren zur sicheren Extraktion von Kammerwasser mittels klarer Hornhautparazentese und Schritte zur genomischen Analyse zur Identifizierung prognostischer Biomarker vorgestellt.
Zusammenfassung
Es besteht ein erheblicher potenzieller klinischer Nutzen für die Anwendung einer Flüssigbiopsieplattform für Retinoblastome, da eine direkte Tumorbiopsie bei diesen Patienten verboten ist. Das Kammerwasser (AH) bildet sich in einem vom Tumor getrennten Kompartiment, ist aber im selben Augenraum eingeschlossen. Somit ist es eine angereicherte Quelle für augenspezifische tumorale genomische Informationen, die als Flüssigbiopsie oder Surrogat für eine Tumorbiopsie für diese Krankheit verwendet werden können. Dieses Manuskript beschreibt eine Methode zur sicheren Extraktion des AH aus Retinoblastom-Augen mittels klarer Hornhautparazentese. Darüber hinaus werden die Schritte für die Genomanalyse vorgestellt, einschließlich der Isolierung und Reinigung zellfreier DNA, der Sequenzierung der nächsten Generation, der Analyse der somatischen Kopienzahlveränderung (SCNA), der Identifizierung von RB1-Einzelnukleotidvarianten (SNV) und der Schätzung der Tumorfraktion. Die präanalytische, analytische und frühe klinische Validität der AH-Flüssigbiopsieplattform wurde bewertet; Es ist jedoch nicht ohne Einschränkungen. Diese sind weitgehend eine Folge der Menge an zellfreier DNA, die für bestimmte Schritte des Assays benötigt wird. Im Vergleich zu anderen blutbasierten Flüssigbiopsieplattformen, die derzeit für Retinoblastome untersucht werden, ist eine AH-basierte Plattform durch das Volumen der Bioflüssigkeit (und damit die Menge an DNA), die aus dem Auge extrahiert werden kann, begrenzt. Der Vorteil ist, dass AH augenspezifisch ist. Die hier diskutierte Plattform ist insofern einzigartig, als sie zirkulierende Tumor-DNA in der AH über zwei Mechanismen (SCNAs und RB1-SNVs ) nachweist, was zu einer höheren Sensitivität bei der Identifizierung tumoraler genomischer Informationen führt. Die AH-Flüssigbiopsie hat das Potenzial für eine direkte klinische Anwendung in der Präzisionsonkologie für Retinoblastom-Patienten, mit besonderer Bedeutung für Patienten mit bilateraler Erkrankung, da die AH spezifisch für die Tumoren in jedem Auge ist. Es gibt laufende Forschungen mit Anwendungen dieser Plattform auch bei Patienten mit anderen Augentumoren.
Einleitung
Das Retinoblastom (RB) ist eine seltene und einzigartige Krebserkrankung. Obwohl es sich um die häufigste primäre intraokulare Malignität handelt, die sich in der sich entwickelnden Netzhaut von Säuglingen und Kleinkindern bildet, gibt es weltweit jährlich nur etwa 7000 Fälle, davon etwa 250 bis 300 in den Vereinigten Staaten. Obwohl die Überlebensrate der Patienten in den Industrieländernbei 98 % liegt 1, ist das okuläre Überleben für fortgeschrittene Augen, die klinisch als International Intraocular RB Classification (IIRC)2 Group D/E oder AJCC cT2b/3 klassifiziert sind, weitaus niedriger. Viele dieser fortgeschrittenen Augen werden entweder primär (als erste Behandlung) oder sekundär (nach fehlgeschlagenen Versuchen einer Bulbus-Salvationstherapie) enukleiert. In der aktuellen klinischen Praxis der okulären Onkologie gibt es keine tumorabgeleiteten, augenspezifischen molekularen Biomarker, die derzeit klinisch eingesetzt werden, um die Diagnose, die Prognose des Augenüberlebens oder die Behandlungsüberwachung von Patienten mit RB zu unterstützen. Dies ist zum großen Teil darauf zurückzuführen, dass Tumorgewebe nur für molekulare und genetische Analysen von enukleierten Augen zur Verfügung steht, da eine direkte Tumorbiopsie bei RB aus Sorge vor einer extraokulären Tumorausbreitung verboten ist 3,4,5,6,7,8,9 . Aufgrund dieses Verbots beschränkten sich bisherige retrospektive Studien zur Genetik von RB-Tumoren und ihren klinischen Korrelationen nur auf die Analyse von Tumorgewebe, das aus enukleierten Augen gewonnen wurde 10,11,12,13. Daher fehlte es an objektiven, vom Tumor abgeleiteten molekularen Daten, die bei der Diagnose und während der gesamten augenrettenden Therapie verfügbar waren. Dies hat nicht nur das Verständnis der In-vivo-Tumorbiologie und der Art und Weise, wie sich diese Tumoren auf molekularer Ebene während der Therapie verändern, eingeschränkt, sondern auch die Fähigkeit, personalisierte, augenspezifische, genombasierte Behandlungspläne für diese jungen Krebspatienten zu entwickeln.
Neben dem Verbot der Biopsie ist ein weiterer einzigartiger Aspekt dieses Krebses, dass die meisten Tumoren durch den biallelischen Verlust des RB1-Tumorsuppressorgens ausgelöst werden, das den Zellzyklus moduliert. Die sich entwickelnde Netzhaut reagiert äußerst empfindlich auf diesen Verlust. Bei 60% der Patienten tritt diese bi-allelische Inaktivierung des RB1-Gens in der Netzhaut nur als somatischer Verlust auf und führt zu einer einseitigen Erkrankung. Bei 40 % der Patienten tritt die anfängliche RB1-Mutation jedoch in der Keimbahn auf, gefolgt von einem zweiten "Treffer" in der Netzhaut. Bei diesen Kindern gibt es oft mehrere Tumoren, die beide Augen betreffen. Schließlich scheint eine sehr kleine Untergruppe (<2%) der Tumoren durch MYCN-Amplifikation ohne Mutationen im RB1-Gen angetrieben zu werden. Während MYCN-getriebene Tumoren fast durchweg bei der Behandlung versagen und eine Enukleation erforderlich sind, gibt es derzeit keine klare, objektive Möglichkeit, diesen aggressiven Tumorsubtyp bei der Diagnosezu identifizieren 14,15. Darüber hinaus stützt sich die Überwachung der intraokularen RB-Tumoraktivität fast ausschließlich auf die Bildgebung und klinische Beobachtungen jedes Auges durch den behandelnden Augenonkologen. Es gibt keinen objektiven, quantitativen Goldstandard für die Diagnose, Prognose oder Möglichkeit, die augenspezifische Tumordynamik während der gesamten Behandlung zu überwachen. Aufgrund dieser einzigartigen Einschränkungen für RB ist die Aussicht auf eine Flüssigbiopsieplattform für diesen Krebs verlockend. Bei Flüssigbiopsien werden Körperflüssigkeiten verwendet, um zellfreie DNA zu isolieren und zu sequenzieren, um festzustellen, ob sie vom Tumor stammt, die als zirkulierende Tumor-DNA (ctDNA) bezeichnet wird. Während das Serum für andere Krebsarten erforscht wird, ist die im Serum enthaltene RB-ctDNA nicht augenspezifisch, was für die 40% der Patienten, die an einer bilateralen Erkrankung leiden, klare Einschränkungen hat. Darüber hinaus wurde sie nur im Rahmen einer fortgeschrittenen intraokularen oder metastasierten Erkrankung beschrieben, die im Allgemeinen eine niedrige Tumorfraktion (<5 %) aufweist16.
In dem Bemühen, diese realen klinischen Probleme anzugehen, mit denen Patienten und Angehörige konfrontiert sind, haben wir 2017 gezeigt, dass Kammerwasser (AH, die klare Flüssigkeit vor dem Auge) eine ertragreiche Quelle für ctDNA ist, die als Flüssigbiopsie – oder mehr noch als Ersatz für eine Tumorbiopsie – zur Beurteilung von RB17 verwendet werden kann. 18,19. Mit bisher über 200 Proben wurde ctDNA in >95 % der Proben identifiziert, einschließlich weniger fortgeschrittener IIRC-Augen der Gruppen A, B und C sowie genomische Unterschiede zwischen den Augen bei bilateralen Patienten18,19. Diese DNA kann sicher und effektiv isoliert werden, um relevante molekulare Biomarker in Augen nachzuweisen, die sich aktiv in Behandlung befinden oder nicht vorbehandelt wurden 18,20,21. Die AH-Flüssigbiopsieplattform kann auch zur Identifizierung der diagnostischen pathogenen RB1-Varianten oder der primären MYCN-Amplifikation verwendet werden, die die Tumorgenese einleitet21,22. Wichtig ist, dass wir eine prognostische molekulare Signatur identifiziert haben – das Vorhandensein eines Chromosomengewinns von 6p mit einer Amplitude von ≥1,5 zur medianen oder fokalen MYCN-Amplifikation –, die mit einer um 16,5 erhöhten Wahrscheinlichkeit für einen Verlust der intraokularen Tumorkontrolle verbunden ist, der die Entfernung des Auges erfordert 18,20,21. Schließlich konnte gezeigt werden, dass Veränderungen der ctDNA-Tumorfraktion (TFx) in der AH mit dem therapeutischen Ansprechen korrelieren, da höhere Spiegel mit der aktiven Erkrankung korrelieren und abnehmende Spiegel mit einem positiven Ansprechen auf die Behandlung verbunden sind23. Angesichts dieser Anwendungen und ihres potenziellen klinischen Nutzens wollten wir die Methodik für die AH-Biopsie und -Bewertung skizzieren. Dazu gehören die klare Hornhautparazentese für die Probenentnahme und das Protokoll für die Genomanalyse, insbesondere der Aufbau und die Sequenzierung von cfDNA-Bibliotheken, die Bestimmung der SCNA-Amplitude, die Identifizierung pathogener RB1-Varianten und die TFx-Berechnung.
Protokoll
Diese Forschung wird mit Genehmigung des Children's Hospital Los Angeles und des University of Southern California Institutional Review Board durchgeführt und entspricht den Grundsätzen der Deklaration von Helsinki. Eine schriftliche Einwilligungserklärung wird immer von den Erziehungsberechtigten aller Teilnehmer eingeholt.
Ein Schema für den Arbeitsablauf der AH-Flüssigbiopsie ist in Abbildung 1 zu sehen.
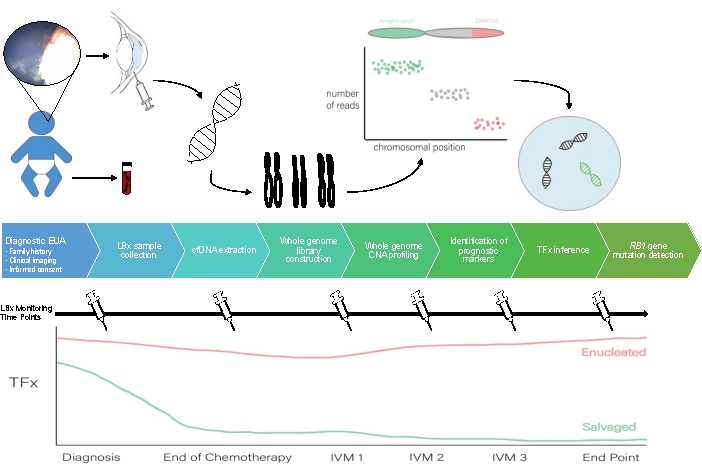
Abbildung 1: Arbeitsablauf für die AH-Flüssigbiopsie zur Begleitung des hierin beschriebenen Protokolls. EUA, Untersuchung unter Narkose; LBX, Flüssigbiopsie; cfDNA, zellfreie DNA; CNA, Änderung der Kopiennummer; TFx, Tumorfraktion; IVM, intravitreales Melphalan. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
1. Chirurgischer Eingriff:
HINWEIS: Dieses Verfahren wird während der Routineuntersuchung unter Narkose (EUA) zur klinischen Beurteilung von Patienten mit RB durchgeführt. Das Parazenteseverfahren zur Extraktion von AH sollte nur von einem ausgebildeten Augenchirurgen durchgeführt werden, der eine Standardausbildung in Augenchirurgie absolviert hat.
- Die Kriterien für die Patientenauswahl und den Einschluss lauten wie folgt.
- Stellen Sie sicher, dass die Kammer geformt und frei ist, ohne durch den Tumor, Synechien oder eine Kataraktlinse flach zu werden.
HINWEIS: Bei einigen fortgeschrittenen Augen ist die Kammer bei der Diagnose zu flach, um die AH sicher zu extrahieren. In diesen Fällen wird empfohlen, bis nach dem ersten Zyklus der systemischen oder intraarteriellen Chemotherapie zu warten. Sobald der Haupttumor schrumpft, vertieft sich die Kammer in der Regel, was einen sicheren Zugang ermöglicht. - Stellen Sie sicher, dass eine freie Sicht auf alle Strukturen vorhanden ist und dass der Druck <22 mm Hg beträgt, damit es aufgrund der Druckdynamik nicht zu einem schnellen Schrumpfen der Kammer kommt. Dies gilt für alle Zeitpunkte, ist aber besonders kritisch, wenn die AH bei der Diagnose eingenommen wird.
- Stellen Sie sicher, dass es keine direkte Beteiligung des vorderen Augenabschnitts durch den Tumor gibt, was durch direkte Visualisierung und Ultraschallbiomikroskopie beurteilt wird. Dieses Einschlusskriterium wird in der Regel während der EUA festgelegt.
- Stellen Sie sicher, dass die Kammer geformt und frei ist, ohne durch den Tumor, Synechien oder eine Kataraktlinse flach zu werden.
- Sobald der Patient für angemessen befunden wurde und eingewilligt hat, bringen Sie den Patienten in den Operationssaal. Erlauben Sie dem behandelnden Anästhesisten, die Anästhesie einzuleiten, wie es bei der EUA für die klinische Versorgung von Patienten mit RB üblich ist.
- Sobald der Patient intubiert und sediert ist und alle klinischen Untersuchungen, Bildgebungen und alle lokalen therapeutischen Bedürfnisse (z. B. Laser oder Kryotherapie) in Bezug auf die EUA abgeschlossen sind, extrahieren Sie die AH für die Flüssigbiopsieplattform. Bereiten Sie das Auge vor und drapieren Sie es in gewohnt steriler Weise. Dies kann von der zirkulierenden Krankenschwester oder dem operierenden Chirurgen durchgeführt werden.
- Platzieren Sie vor der EUA die dilatierenden Tropfen, die routinemäßig in der Einrichtung für eine EUA verwendet werden, in das chirurgische Auge.
HINWEIS: Hier kommen kombinierte Augentropfen bestehend aus 2,5 % Phenylephrin, 1 % Cyclopentolat und 1 % Tropicamid zum Einsatz. Die Standardtropfen, die für die Dilatation der EUA verwendet werden, sind jedoch ausreichend; das Auge muss nicht nur erweitert werden, um das AH zu extrahieren. - Geben Sie 5% Betadin-Tropfen in das chirurgische Auge und tragen Sie ihn mit einem Schwamm auf, beginnend in der Mitte und peripher bewegend. Beziehen Sie obere und untere Lidränder in die chirurgische Vorbereitung ein. Schließen Sie die chirurgische Vorbereitung dreimal mit Betadin ab und warten Sie dann mindestens zwei Minuten, bevor Sie fortfahren.
- Legen Sie ein steriles blaues Handtuch über den Kopf des Patienten und stecken Sie es unter den Kopf, damit es nicht herunterfällt. Trocknen Sie die Wimpern und den periorbitalen Bereich mit einem sterilen blauen Handtuch oder einer Gaze.
- Lassen Sie den Operateur einschrubben, ziehen Sie einen Kittel an und ziehen Sie Handschuhe an.
- Decken Sie den Patienten mit sterilen blauen Vorhängen ab und lassen Sie das chirurgische Auge frei. Platzieren Sie ein steriles Deckelspekulum für eine optimale Visualisierung des sterilen Feldes.
HINWEIS: Eine zusätzliche topische Anästhesie ist in unserer Einrichtung nicht erforderlich, da das Kind für die EUA unter Vollnarkose steht. - Führen Sie eine Auszeit gemäß dem institutionellen Protokoll durch. Hier wird eine erste Auszeit für die EUA und eine zweite Auszeit durchgeführt, um das/die Auge(n) vor dem Eingriff zu überprüfen.
- Platzieren Sie vor der EUA die dilatierenden Tropfen, die routinemäßig in der Einrichtung für eine EUA verwendet werden, in das chirurgische Auge.
- Führen Sie mit einem Operationsmikroskop eine klare Hornhautparazentese mit der Extraktion von 0,1 ml AH mit einer 32-G-Nadel auf einer 1-cm³-Spritze durch.
- Befeuchten Sie das Auge vor Beginn des Eingriffs mit einer sterilen, ausgewogenen Salzlösung (jeder ml enthält Natriumchlorid 0,64 %, Kaliumchlorid 0,075 %, Calciumchloriddihydrat 0,048 %, Magnesiumchloridhexahydrat 0,03 %, Natriumacetattrihydrat 0,39 %, Natriumcitratdihydrat 0,17 %, Natriumhydroxid und/oder Salzsäure (zur Einstellung des pH-Werts) und Wasser für Injektionszwecke), um die Hornhautschmierung aufrechtzuerhalten; Dies ist ein handelsübliches Präparat.
- Stellen Sie sicher, dass die 32-G-Nadel mit der Luer-Verriegelung an der 1-cm³-Spritze befestigt ist und dass kein Druck in der Spritze entsteht (indem Sie den Kolben der Spritze vor der Anwendung mehrmals ein- und ausschieben).
- Führen Sie anschließend die Nadel durch die klare Hornhaut am Limbus (senkrecht dazu, wie es bei der Parazentese üblich ist) und bleiben Sie in der vorderen Kammer über der peripheren erweiterten Iris.
- Halten Sie die Nadelspitze während der Extraktion unter direkter Visualisierung über das Mikroskop nach oben zur Iris. Falls gewünscht, stabilisieren Sie das Auge mit einer Pinzette von 0,12, während die Nadel durch die Hornhaut fährt.
HINWEIS: Es sollte niemals Kontakt zwischen der Nadelspitze und einer Okularstruktur geben. Es sollte über der peripheren Iris in der mittleren Vorderkammer verbleiben. - Extrahieren Sie 0,1 mL AH. Manipulieren Sie den Spritzenkolben mit der nicht dominanten Hand des Chirurgen (aber mit Übung) oder durch einen geschulten Assistenten, ohne die Nadel zu bewegen. Verlieren Sie nicht die direkte Visualisierung der Nadelspitze. Priorisieren Sie die Anatomie des Auges und nehmen Sie bei Bedarf weniger AH, um sicherzustellen, dass die Kammer gebildet bleibt.
- Entfernen Sie nach der Extraktion vorsichtig die Nadel aus der vorderen Kammer. Stellen Sie sicher, dass die Kammer geformt, aber leicht flach bleibt; Der Druck wird sanft, aber physiologisch sein. Sobald die Nadel aus dem Öhr entfernt ist, baden Sie das Auge reichlich mit sterilem Wasser; Steriles Wasser wird in diesem Schritt als Sicherheitsmaßnahme einer balancierten Salzlösung vorgezogen, da Wasser die Zellen lysiert.
- Untersuchen Sie die Nadelstelle auf Undichtigkeiten. Wenn es eine Leckage gibt, üben Sie mit einem Wattestäbchen-Applikator 30 Sekunden lang leichten Druck auf die Injektionsstelle aus, bevor Sie erneut auf Leckagen prüfen. Wiederholen Sie den Vorgang bei Bedarf.
HINWEIS: Der Einsatz invasiverer Methoden, um Leckagen zu stoppen, war bei diesem Ansatz nie erforderlich. Manchmal folgt auf dieses Verfahren eine intravitreale Injektion einer Chemotherapie; Für dieses Methodenpapier konzentriert sich das Protokoll auf die AH-Extraktion als primäres Verfahren.
- Untersuchen Sie die Nadelstelle auf Undichtigkeiten. Wenn es eine Leckage gibt, üben Sie mit einem Wattestäbchen-Applikator 30 Sekunden lang leichten Druck auf die Injektionsstelle aus, bevor Sie erneut auf Leckagen prüfen. Wiederholen Sie den Vorgang bei Bedarf.
- Sobald keine Leckage mehr auftritt, gilt der Vorgang als abgeschlossen. Entfernen Sie die sterilen Abdecktücher vom Patienten und reinigen Sie das Betadin vom Gesicht.
- Entfernen Sie vorsichtig die Nadelspitze von der Spritze, verschließen Sie die Probe mit einer sterilen Kappe über den Luer-Lock, um keine Probe zu verlieren, und beschriften Sie die Spritze entsprechend.
HINWEIS: Dies sollte vom Koordinator der klinischen Forschung oder dem Chirurgen durchgeführt werden, während das Kind extubiert wird.- Nach der Probenentnahme ist die Probe in einer Spritze mit Verschluss bei -80 °C zu lagern. Die Proben können auf Trockeneis gelegt werden, um sie vom Operationssaal in einen geeigneten Gefrierschrank zu transportieren.
- Geben Sie je nach Präferenz des Chirurgen einen einzelnen Tropfen antibiotischer Augentropfen auf die Oberfläche des chirurgischen Auges, bevor der Patient zur Genesung geschickt wird. Hier werden kombinierte Steroid-Antibiotika-Augentropfen verwendet, und das Auge wird nicht geflickt.
- Sobald der Eingriff abgeschlossen ist, sorgt der behandelnde Anästhesist für eine ordnungsgemäße Extubation. Bringen Sie den Patienten zur Genesung.
- Entlassen Sie den Patienten nach Hause nach dem institutionellen postoperativen Protokoll.
- Informieren Sie den Patienten, dass das Auge nicht mit einem Verband oder einer Schutzhülle abgedeckt werden muss.
- Informieren Sie den Patienten, dass er seinen Kopf 24 Stunden lang nicht vollständig in eine Badewanne oder ein Schwimmbad eintauchen darf, und die Eltern, das Auge des Patienten (Kindes) nicht zu reiben. Es gibt keine weiteren Einschränkungen der Patientenaktivität.
- Informieren Sie den Patienten, dass es unwahrscheinlich ist, dass er Schmerzen durch die Parazentese hat, aber während der EUA Schmerzen durch damit verbundene Eingriffe haben könnte. Der Patient kann Paracetamol oder Ibuprofen einnehmen, wenn er Beschwerden hat, wie vom Chirurgen empfohlen.
2. cfDNA-Isolierung und -Reinigung
- Führen Sie die DNA-Isolierung und -Verarbeitung innerhalb von 72 Stunden nach der AH-Extraktion durch.
- AH bei Raumtemperatur auftauen. Überprüfen Sie die Probe regelmäßig und gehen Sie sofort nach Abschluss des Auftauens zur Extraktion über.
- Extrahieren Sie cfDNA aus AH und eluieren Sie sie in 50 μl AVE-Lösung (im Kit enthalten) unter Verwendung des cfDNA-Isolations- und Aufreinigungskits (siehe Materialtabelle für Details) gemäß der Bedienungsanleitung des Herstellers.
3. Next Generation Sequencing (NGS) und Qualitätskontrolle (QC)
- Erstellen Sie die DNA-Bibliotheken für die Sequenzierung mit dem DNA-Bibliotheks-Sequenzierungskit (siehe Materialtabelle) gemäß der Bedienungsanleitung des Herstellers. Amplifizieren Sie die DNA-Bibliothek mit 14 Zyklen der Polymerase-Kettenreaktion (PCR), wie im Handbuch des DNA-Bibliotheks-Sequenzierungskits beschrieben.
HINWEIS: Die endgültigen Bibliotheken können bis zu 1 Monat vor der endgültigen Sequenzierung für die genomische Analyse bei -20 °C gelagert werden. - Sequenzieren Sie die DNA-Bibliotheken auf der NGS-Plattform (siehe Materialtabelle) gemäß dem Protokoll des Herstellers nach dem Single-End-Protokoll mit 50 Zyklen oder dem Paired-End-Protokoll mit 150 Zyklen.
HINWEIS: Andere NGS-Plattformen funktionieren prinzipiell (obwohl hier nicht ausprobiert), mit geeigneten Adaptern für die Sequenzierflusszelle. - Führen Sie eine Qualitätskontrolle für alle AH-Proben durch, die zum Zeitpunkt der Diagnose oder der primären Enukleation entnommen wurden.
HINWEIS: Die Qualitätskontrolle kann nicht für Proben durchgeführt werden, die zu anderen Zeitpunkten entnommen wurden, da die DNA-Masse zu gering ist und für die unten beschriebenen Schritte außerhalb des Nachweisbereichs liegt.- Verwenden Sie jeweils 1 μl der extrahierten DNA für den DNA-Quantifizierungs-Assay und den Size-Profiling-Assay (siehe Materialtabelle) gemäß den Protokollen des Herstellers.
HINWEIS: Der Spitzenwert sollte bei etwa 300 bp liegen. Wenn sie kürzer als 150 bp ist, wurde die Probe kompromittiert. Siehe Zusatzdatei 1A für ein Beispiel für eine Stichprobe, die die Qualitätskontrolle bestehen sollte, und Zusatzdatei 1B für ein Beispiel für eine Stichprobe, die dies nicht tun sollte.
- Verwenden Sie jeweils 1 μl der extrahierten DNA für den DNA-Quantifizierungs-Assay und den Size-Profiling-Assay (siehe Materialtabelle) gemäß den Protokollen des Herstellers.
4. Datenanalyse der somatischen Kopienzahländerung (SCNA)
- Ordnen Sie die aus NGS erhaltenen Reads (Abschnitt 3) dem menschlichen Genom zu (hg19, Genome Reference Consortium GRCh37, University of California Santa Cruz Genome Browser database)24,25 mit einem Aligner.
- Entfernen Sie die PCR-Duplikate (samtools rmdup26).
- Normalisieren Sie den Guanin-Cytosin-Gehalt, indem Sie den Prozentsatz an Guanin- und Cytosinbasen in jedem Bin aus dem Referenzgenom berechnen. Ein Beispielprogramm ist in Baslan et al.27 beschrieben.
- Um die Kopienzahlen der DNA-Segmente zu erhalten, unterteilen Sie das Genom in 5000 Bins mit variabler Länge und berechnen Sie dann die relative Anzahl der in jedem Bin enthaltenen Reads.
- Verwenden Sie referenzfreie log2-Verhältnisse, um Schätzungen der Kopienzahl zu bestimmen. Nehmen Sie die mediane Fensterzahl normaler autosomaler Chromosomen.
- Führen Sie die Segmentierung durch zirkuläre binäre Segmentierung mit DNAcopy (Bioconductor28) durch.
- Definieren Sie SCNAs als positiv bei einer Abweichung von 20 % vom Ausgangswert (log2-Verhältnis = 0), d. h. Verluste bei log2-Verhältnissen ≤ 0,2 (Verhältnis von 0,87 oder niedriger definiert eine Deletion) und Gewinne bei log2-Verhältnissen ≥ 0,2 (Verhältnis von 1,15 oder höher definiert eine Verstärkung).
HINWEIS: Diese Schwellenwerte stimmen mit den zuvor etablierten Flüssigbiopsieanalysenüberein 27,29.
5. RB1-Mutationsanalyse von AH-Proben
- Darüber hinaus können Sie die gesamten Genombibliotheken auf jeweils 500 ng amplifizieren, um ein auf Capture basierendes zielgerichtetes NGS für den Mutationsnachweis gemäß dem Protokoll des Herstellers zu ermöglichen.
- Führen Sie eine NGS-Plattform-Paired-End-Sequenzierung mit 150 bp oder 50 bp Single-End-Sequenzierung für die erfassten Bibliotheken bis zum >100-fachen pro Herstellerprotokoll durch.
- Verarbeiten Sie die NGS-Daten. In dieser Studie wird eine hauseigene Pipeline auf Basis der bcbio-Pipeline am CHLA Center for Personalized Medicine verwendet30.
- Schneiden Sie die fastq-Rohdaten von Adaptern und minderwertigen Basen mit Atropos31 ab und richten Sie sie mit BWA-MEM und NovoAlign (v3)32,33 an die humane GRCh37-Referenz aus.
- Markieren Sie Duplikate mit FreeBayes34 und bestimmen Sie anschließend die Keimbahnvariante.
HINWEIS: In FreeBayes-Tools zur Aufrufen von Varianten wird jede Position des Probengenoms mit dem Referenzgenom verglichen und die Allelzahlen werden mathematisch modelliert, um die Genotyp-Wahrscheinlichkeitsmaße34 zu erhalten. - Führen Sie die Variantenannotation mit dem Ensembl Variant Effect Predictor (v96)35 durch, indem Sie die Schritte 5.3.4 bis 5.3.5 befolgen.
- Verwenden Sie VarDict, um somatische Varianten in der AH oder im Tumor ohne die gepaarte normale Blutprobe (d. h. verblindete Keimbahnvariante) zu bestimmen36.
- Rufen Sie den Verlust der Heterozygotie auf, wenn die kontinuierlichen Varianten einer Region ≤3% alternative Allele aufweisen.
HINWEIS: Kommerzielle Genomanalysesoftware kann auch mit den oben genannten Parametern verwendet werden.
6. Bestimmung der cfDNA TFx
- Schätzen Sie TFx für jede sequenzierte AH-cfDNA-Probe mit der ichorCNA-Software (CNA-basierte TFx-Schätzungssoftware)37.
HINWEIS: Dies ist die Standardsoftware, die in blutbasierten Flüssigbiopsien zur Bestimmung von cfDNA TFx38 verwendet wird. Die Verwendung der Software auf AH wurde bereits veröffentlicht21,23. - Überprüfen Sie die genomischen Profile und die entsprechenden TFx-Lösungen einzeln, um sicherzustellen, dass die TFx-Schätzung für jede Probe angemessen ist39.
Ergebnisse
Im Folgenden finden Sie umfassende Ergebnisse von zwei Augen (Fälle 33 und 47). Die Fallzahlen stimmen zu Vergleichszwecken mit früheren Veröffentlichungen überein 18,20,21. Die behandelnden Ärzte wurden während der Therapie für die Ergebnisse der AH-Flüssigbiopsie verblindet. Alle Behandlungsentscheidungen wurden nicht randomisiert und gemäß der routinemäßigen Standardbehandlung getroffen, die zuvor veröffentlicht wurde40,41. Die Daten zu den klinischen Ergebnissen blieben bis zur endgültigen Analyse von den genomischen Daten getrennt. Rohdaten der vorgestellten Ergebnisse sind auf Anfrage beim entsprechenden Autor erhältlich. Aufgrund der NIH-Finanzierung unterliegen die Daten der NIH-Richtlinie zum Austausch genomischer Daten und werden in Zukunft anderen Forschern über ein NIH Designated Data Repository (dbGAP) mit kontrolliertem Zugriff zur Verfügung stehen. Es ist auch auf Anfrage beim korrespondierenden Autor erhältlich.
Bei den Fällen 33 und 47 handelt es sich um Augen der IIRC-GruppeD 2, die sehr ähnliche klinische Erscheinungsbilder aufwiesen. So wurden sie auf Wunsch der Eltern mit einer Globe-Salvaging-Therapie und mit Augen- und Medizinonkologen behandelt – insbesondere mit einer systemischen Chemotherapie für Fall 47 und einer intraarteriellen Chemotherapie für Fall 33. Basierend auf der derzeit akzeptierten klinischen Prognose, die sich auf die IIRC-Gruppe des Auges2 stützt, wäre der prognostizierte Erfolg der Globe-Salvage für die Augen dieser beiden Patienten derselbe gewesen: 65%-70% für Gruppe D ist ein Durchschnitt, obwohl dies je nach Behandlungszentrum variiert41. Basierend auf den Daten der Gruppe D, die bisher aus tumorgerichteten molekularen Profilen in diesem Zentrum gesammelt wurden, würde der prognostizierte Erfolg der Globe-Salvage jedoch 72 % für Fall 47 (ohne identifizierten 6p-Gewinn in der AH) und 9 % für Fall 33 (mit identifiziertem 6p-Gewinn) betragen18,20,21. Dies wird im Folgenden veranschaulicht.
Fall 47 ist ein Beispiel für ein Auge mit erfolgreichem SCNA- und SNV-Nachweis unter Verwendung der AH-Flüssigbiopsie-Plattform zum Zeitpunkt der Diagnose, zusammen mit TFx-Trends, die dem longitudinalen Ansprechen auf die Behandlung entsprechen. Bei der Patientin handelt es sich um eine Frau, die sich im Alter von 15 Monaten mit einem 14 mm x 9 mm IIRC Gruppe D, Stadium cT2b RB mit Kugelglasaussaat vorstellte. Sie war negativ für eine RB1-Keimbahnmutation , die durch routinemäßige klinische Serumleukozytentests festgestellt wurde. Von der AH wurden bei der Diagnose RB SCNAs 1q-Gewinn und 6p-Gewinn in der AH identifiziert, zusätzlich zu zwei anderen nicht hoch rezidivierenden RB-SCNAs mit 7p-Verlust und 13q-Gewinn (Abbildung 2). Bemerkenswert ist, dass die Amplitude der 6p-Verstärkung 1,2 betrug und nur Amplituden von ≥ Verhältnis von 1,5 zum Median eine schlechte Prognose anzeigen. Angesichts der Tatsache, dass es keine fokale MYCN-Amplifikation gab und die 6p-Verstärkung unter der Schwelle von 1,5 lag, war die Prognose für die Rettung mit einer Behandlung auf der Grundlage molekularer Merkmale hoch. Dieselbe AH-Probe, die bei der Diagnose entnommen wurde, wurde auch auf den Nachweis von RB1-pathogenen Varianten untersucht, die das SNV c.958C>T, p.Arg320* innerhalb des RB1-Gens mit einer Variantenallelfrequenz von 87,01 % (95 %-Konfidenzintervall, 79,7 %–94,6 %) aufwiesen. Dieser Patient wurde mit sechs Zyklen Carboplatin, Etoposid und Vincristin (CEV) mit Rückbildung der Erkrankung behandelt, zeigte jedoch eine persistierende kugel- und staubartige Aussaat, die drei aufeinanderfolgende intravitreale Melphalan-Injektionen (IVM) erforderte. Während der IVM-Behandlung zeigten die AH-Proben A-C (jeweils im Abstand von zwei Wochen) eine vollständige Normalisierung des genomischen Profils, eine Abnahme des TFx und eine Abnahme der DNA-Konzentration – alles gleichzeitig mit einer klinischen Rückbildung der Erkrankung (Abbildung 3). Nach der Diagnose blieben die TFx-Werte für den Rest der Behandlung unter der Nachweisgrenze von 5 %. Nach 19 Monaten Nachbeobachtung blieb das Auge stabil, ohne dass es zu einem Tumorrezidiv oder einer extraokulären Ausbreitung der Erkrankung kam.
Dies steht im Gegensatz zu Fall 33. Dieser 22 Monate alte Mann hatte ein insgesamt ähnliches klinisches Erscheinungsbild mit einer Netzhautmasse von 11 mm x 18 mm und einer staubartigen Glaskörperaussaat, was mit der einseitigen IIRC-Gruppe D, Stadium cT2b, übereinstimmt. Er war auch negativ für eine RB1-Keimbahnmutation, die durch routinemäßige klinische Serumleukozytentests festgestellt wurde. Die bei der Diagnose gemessene AH zeigte einen RB-SCNA-Gewinn von 1q, einen 6p-Gewinn (in diesem Fall mit einer Amplitude von 1,5 zum Median) und einen 16q-Verlust zusammen mit einem fokalen 6q-Verlust (Abbildung 4A). Basierend auf dem prognostischen molekularen Profil hatte dieses Auge eine um 16,5 erhöhte Wahrscheinlichkeit einer Enukleation basierend auf dem Vorhandensein von 6p ≥ einer Amplitude von 1,5. In dieser Probe wurde kein RB1-SNV identifiziert, obwohl die gesamte Länge des RB1-Gens vollständig abgedeckt wurde. Ein Grund, warum eine RB1-SNV möglicherweise nicht identifiziert werden kann, ist bei primären MYCN-getriebenen Tumoren, bei denen eine gleichzeitige RB1-Mutation nicht immer erwartet wird 14,15,42,43,44,45. Fall 33 zeigte jedoch keine Hinweise auf eine MYCN-Amplifikation in einer seiner AH-Proben oder seinem enukleierten Tumorgewebe (Abbildung 5). Eine wahrscheinlichere Erklärung für ein negatives RB1-SNV-Ergebnis ist, dass die anfängliche Tumorgenese durch eine epigenetische Dysregulation (z. B. Methylierung des Promotors) angetrieben wurde46,47, ein bekanntes Phänomen bei RB, das durch den hier beschriebenen Assay nicht identifiziert werden könnte.
Die Erstbehandlung dieser Patientin bestand aus insgesamt vier Zyklen intraarterieller Chemotherapie mit Melphalan, gefolgt von vier IVM-Injektionen aufgrund einer persistierenden Glaskörperaussaat. Drei AH-Proben (jeweils vier Wochen voneinander entfernt) wurden während der IVM-Therapie entnommen und zeigten die gleichen drei SCNAs, die bei der Diagnose vorhanden waren (Abbildung 5). Die TFx-Werte blieben während der gesamten Behandlung hoch, obwohl das abnehmende Primärtumorvolumen die aktiven Tumorseeds im Glaskörper widerspiegelte. Dies zeigt, dass TFx repräsentativ für den Gesamterkrankungszustand des Auges ist. Sechs Monate nach der Diagnose wurde das Auge aufgrund einer persistierenden aktiven Erkrankung enukleiert. Das genomische Profil, das zu diesem Zeitpunkt aus dem Tumorgewebe gewonnen wurde, zeigte eine Übereinstimmung von 92,81 % mit der AH-Probe, die zum Zeitpunkt der Diagnose erhalten wurde (Abbildung 4B).
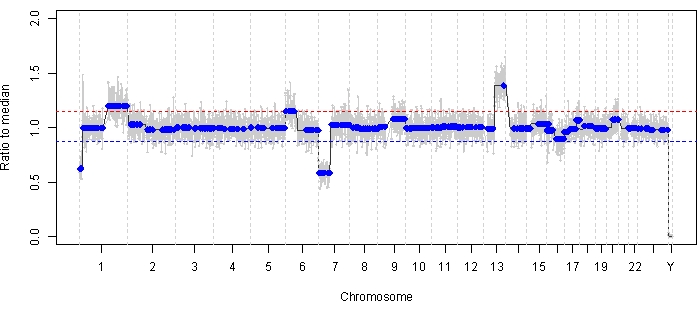
Abbildung 2: Genomisches Profil bei der Diagnose für Fall 47. Hochrezidivierende RB SCNAs 1q-Gewinn und 6p-Gewinn sowie nicht hochrezidivierende RB SCNAs 7p-Verlust und 13q-Gewinn wurden in der zum Zeitpunkt der Diagnose gemessenen AH identifiziert. Die rote Linie stellt die Schwelle für einen Gewinn dar, während die blaue Linie die Schwelle für einen Verlust darstellt. Bemerkenswert ist, dass die Amplitude der 6p-Verstärkung ein Verhältnis von <1,5 zum Median hatte, was unter der Schwelle der molekularen Signatur liegt, die auf eine schlechte Prognose hindeutet. Aufgrund des Mangels an negativen Biomarkern für die Rettung des Auges würde also vorhergesagt, dass dieses Auge auf die Therapie anspricht. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
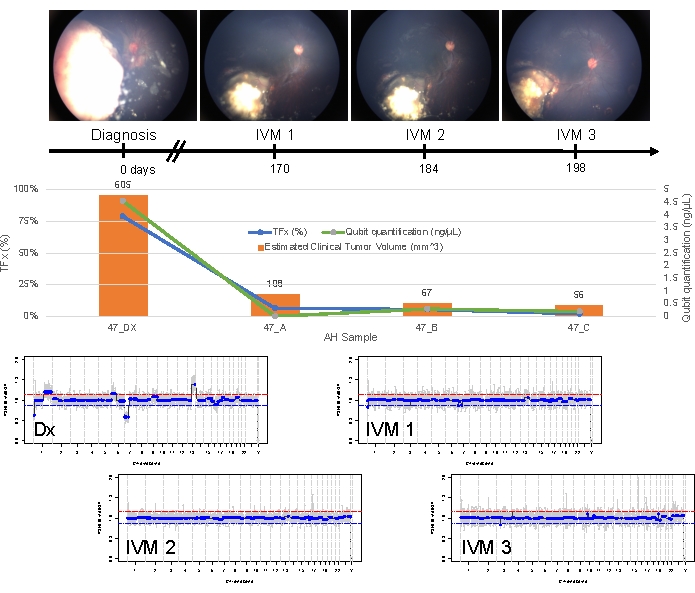
Abbildung 3: Längsschnittinformationen für Fall 47. Dazu gehören Fundusfotos, cfDNA-Quantifizierung, geschätztes klinisches Tumorvolumen aus B-Scan-Messungen, genomische Profile und TFx-Schätzungen für jeden klinischen Zeitpunkt, zu dem AH entnommen wurde (Dx = Diagnose, A = IVM1, B = IVM2, C = IVM3). Dieses Auge sprach auf die Behandlung an und blieb nach 19 Monaten Nachbeobachtung gerettet. Im Laufe der Zeit wurde eine Abnahme der cfDNA-Menge beobachtet, was mit zuvor veröffentlichten Studien übereinstimmt, die die höchste Ausbeute bei der Diagnose zeigten. Die TFx-Toxizität nahm während der Behandlung ebenfalls ab, was die Auflösung der Aussaat und die Abnahme des Haupttumorvolumens der Netzhaut widerspiegelt (das Tumorvolumen wird über dem orangefarbenen Balken jedes klinischen Zeitpunkts angezeigt). Wie bei der klinischen Rückbildung der Krankheit zu erwarten, normalisierten sich auch die genomischen Profile. In den genomischen Profilen stellt die rote Linie die Schwelle für einen Gewinn dar, während die blaue Linie die Schwelle für einen Verlust darstellt. Diese Abbildung wurde mit Genehmigung von Xu, L. et al.21 nachgedruckt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
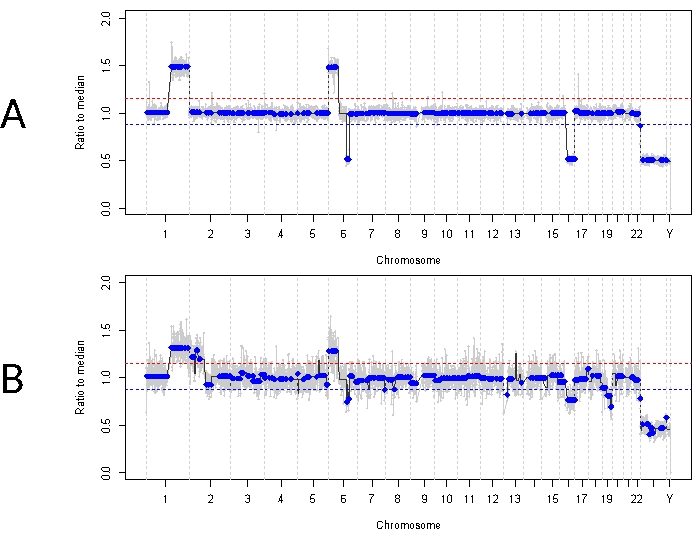
Abbildung 4: Genomische Profile für Fall 33. Die rote Linie stellt die Schwelle für einen Gewinn dar, während die blaue Linie die Schwelle für einen Verlust darstellt (A) Genomisches Profil bei der Diagnose für Fall 33. Zusätzlich zum fokalen 6q-Verlust wurden bei der Diagnose hochrezidivierende RB-SCNAs mit 1q-Verstärkung, 6p-Verstärkung und 16q-Verlust in der AH identifiziert. Bemerkenswert ist, dass die Amplitude des 6p-Gewinns ein Verhältnis von 1,5 zum Median hatte, was auf einen schlechten prognostischen molekularen Biomarker hinweist. Basierend auf dieser molekularen Signatur würden wir also vorhersagen, dass dieses Auge eine signifikant erhöhte Wahrscheinlichkeit für ein Versagen der Behandlung aufweist. (B) Genomisches Profil aus enukleiertem Tumorgewebe, das bei der Diagnose in hohem Maße mit dem von AH erhaltenen Profil übereinstimmte. Aufgrund der Vermischung mit normalem Netzhautgewebe können SCNAs aus Tumorgewebe aufgrund von verdünntem TFx eine geringere Amplitude im Vergleich zu AH aufweisen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
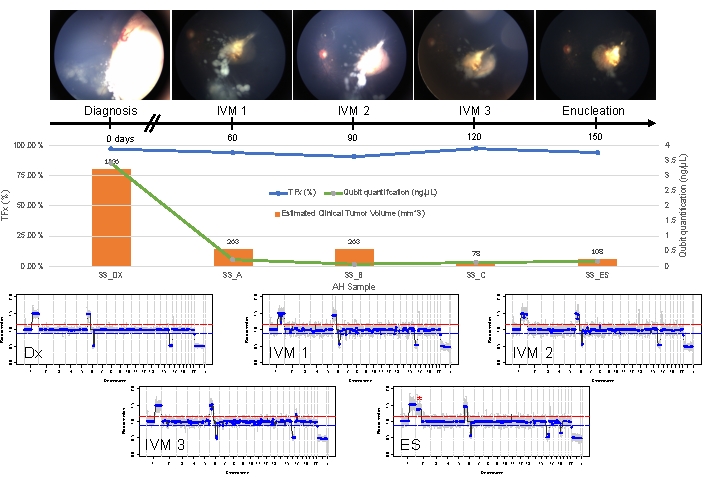
Abbildung 5: Längsschnittinformationen für Fall 33. Dazu gehören Fundusfotos, zellfreie DNA-Quantifizierung, geschätztes klinisches Tumorvolumen aus B-Scan-Messungen (Tumorvolumina werden über dem orangefarbenen Balken jedes klinischen Zeitpunkts angezeigt), genomische Profile und TFx-Schätzungen für jeden klinischen Zeitpunkt, zu dem AH entnommen wurde (Dx = Diagnose, A = IVM1, B = IVM2, C = IVM3, SE = sekundäre Enukleation). Dieses Auge sprach nicht auf die Behandlung an, so dass schließlich eine sekundäre Enukleation (ES) erforderlich war. Dies war auf eine anhaltend aktive Aussaat zurückzuführen, die mit einem apikalen Tumorrezidiv einherging. Die CfDNA-Menge nahm im Laufe der Zeit ab, was mit den zuvor veröffentlichten Studien übereinstimmt, die die höchste Ausbeute bei der Diagnose zeigten (1q-Gewinn, 6p-Gewinn, 16q-Verlust und fokaler 6q-Verlust). Der TFx-Spiegel blieb jedoch während der gesamten Behandlung hoch, ein Produkt der persistenten Aussaat, bei der immer noch tumorabgeleitete cfDNA in die AH abgegeben wird. Die genomischen Profile waren konsistent und zeigten die gleichen drei SCNAs, die bei der Diagnose vorhanden waren. In der an ES erhaltenen AH wurde ein neuer großflächiger 2p-Gewinn (*) und ein 19q-Verlust beobachtet, was auf eine klonale Evolution zum Zeitpunkt des apikalen Tumorrezidivs hindeutet. In den genomischen Profilen stellt die rote Linie die Schwelle für einen Gewinn dar, während die blaue Linie die Schwelle für einen Verlust darstellt. Diese Abbildung wurde mit Genehmigung von Xu, L. et al.21 nachgedruckt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Datei 1: Ein Beispiel für (A) eine Probe, die die Qualitätskontrolle bestehen sollte, mit einem Peak von etwa 300 bp und (B) eine Probe, die die Qualitätskontrolle nicht bestehen sollte, mit einem Peak von etwa 150 bp. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Die klare Hornhautparazentese ist ein Verfahren, das häufig bei mehreren diagnostischen und/oder therapeutischen Indikationen in der Augenheilkunde durchgeführt wird. Speziell für RB ist es Teil des standardmäßigen intravitrealen Chemotherapie-Injektionsprotokolls, um den Augeninnendruck vor der Injektion zu senken und einen Rückfluss zur Injektionsstellezu verhindern 48. Obwohl es sich um ein gängiges Verfahren handelt, ist es nicht ganz ohne Risiko. Bisheriges Dogma war, dass eine Nadel niemals in ein Auge mit aktiver RB eindringen sollte. Dieses Dogma hat sich in den letzten zehn Jahren weiterentwickelt, zunächst durch die prägende Arbeit von Patricia Chévez-Barrios zur injizierten adenoviralen Vektortherapie49, gefolgt von progressiven Methoden mit verbesserter Sicherheit für okuläre Injektionen bei RB durch Francis Munier48. Diese Arbeit hat den Weg für die Extraktion des AH geebnet, die als Flüssigbiopsie oder Ersatz für eine Tumorbiopsie für diesen Krebs verwendet werden kann. Wir sind jetzt das erste Zentrum, das über den Nutzen der Plattform und die vorläufigen Sicherheitsergebnisse von AH berichtet, die zum Zeitpunkt der Diagnose erhoben wurden. Ein Teil der hierin beschriebenen Verfahren soll die Sicherheit der Patientenauswahl und verfahrenstechnische Aspekte der AH-Extraktion für RB gewährleisten. Erstens können Nadeln nur in die Vorderkammer eindringen und sollten nicht mit der Iris oder Linse in Berührung kommen, da dies zu Irisnarben oder Katarakt führen kann, die die Fähigkeit des Chirurgen, den Tumor zu überwachen, einschränken. Es ist äußerst wichtig, dass die Nadel niemals in die Glaskörperhöhle eindringt (es sei denn, sie wird mit einer Chemotherapie kombiniert, wie es bei der IVM der Fall ist) oder den Tumor berührt, da dies hypothetisch das Risiko einer Tumoraussaat und einer extraokulären Ausdehnung der Erkrankung erhöht 3,4,5. Beide Strukturen befinden sich hinter der Vorderkammer und sind von dieser durch die Linse und die Iris getrennt. Um Schäden an den Augenstrukturen zu vermeiden, ist es wichtig, die Nadelfase immer oben, über der peripheren Iris und immer unter direkter Visualisierung mit dem Operationsmikroskop zu halten. Es besteht die Gefahr eines geringfügigen Austretens des AH aus der Nadelstelle; In diesem Fall löst es sich mit sanftem Druck über einen Wattestäbchen-Applikator auf. Die Verwendung der kleinsten verfügbaren Nadel und die Sicherstellung, dass die Nadel langsam und ohne seitliche Scherung aus demselben Trakt gezogen wird, verringert dieses Risiko. Obwohl eine leichte Seflachung der Vorderkammer zu erwarten ist, sollte die Kammer ohne Berührung der Hornhaut gebildet bleiben, und der Augeninnendruck sollte weich, aber physiologisch sein.
Bei der Genomanalyse sind mehrere kritische Schritte erforderlich. Das Wichtigste ist die Handhabung der kleinvolumigen Probe, sobald sie aus dem Auge entnommen wurde. Es ist von entscheidender Bedeutung, dass die Probe vor der Verarbeitung die gesamte Zeit über eingefroren bleibt, um einen DNA-Abbau zu verhindern, der bei wiederholten Einfrier- und Auftauzyklen auftreten kann50. Dies kann sichergestellt werden, wenn die Proben unmittelbar nach der Extraktion auf Trockeneis gelegt und in einen -80 °C-Gefrierschrank umgefüllt werden. Sobald die Proben mit der Verarbeitung begonnen haben, sind Qualitätskontrollschritte die wichtigste Möglichkeit zur Fehlerbehebung, indem sichergestellt wird, dass die cfDNA in hochwertigen Bibliotheken aufgebaut wird. Mittels DNA-Quantifizierung und DNA-Fragmentgrößenprofil-Assay kann eine Qualitätskontrolle an Proben durchgeführt werden, die während der primären Enukleation oder zum Zeitpunkt der Diagnose entnommen wurden, da in diesen Proben eine höhere Ausbeute an cfDNA vorhanden ist18,21. Wenn der erreichte Peak bei etwa 300 bp liegt, garantiert dies, dass die cfDNA von der NGS-Plattform erkannt wird. Wenn der erhaltene Peak kürzer als 150 bp ist, was darauf hindeutet, dass es sich bei der Mehrzahl der nachgewiesenen Fragmente um Bibliotheksaufbauprimer oder Adapteroligos handelt, ist die Probe kompromittiert und sollte nicht weiter für SCNAs verarbeitet werden. Nach unserer Erfahrung mit der Verarbeitung von Hunderten von Proben müssen jedoch weniger als 5 % für die Qualitätskontrolle entfernt werden, da das Reads-Alignment-Verhältnis20 schlecht ist. Zusammen mit den Methoden, die wir für die Entnahme, Handhabung, Lagerung und Verarbeitung von Proben standardisiert haben, zeigt dieses Verfahren die präanalytische Validität der AH-Flüssigbiopsie-Plattform.
Die AH-Flüssigbiopsie hat auch eine nachgewiesene analytische Validität, die auf ihrer Fähigkeit basiert, pathogene RB1-Mutationen und SCNAs genau und zuverlässig zu erkennen, mit mittleren Konkordanzen von konstant >95 % zwischen genomischen Profilen, die aus AH-Proben und entsprechendem Tumorgewebe generiert wurden 18,20,21,22,23. Trotz der nachgewiesenen analytischen Validität der Plattform ist sie nicht ohne Einschränkungen. Die RB1-Mutationsanalyse kann nur an Proben mit >10 ng cfDNA durchgeführt werden, die zum Zeitpunkt der Diagnose oder der primären Enukleation am häufigsten bei behandlungsnaiven Augen gewonnen werden18,21. Dies ist auf die geringere Konzentration an cfDNA in AH-Proben von Augen, die aktiv behandelt werden, im Vergleich zu AH bei der Diagnose oder zum Zeitpunkt der primären Enukleation zurückzuführen. Eine zusätzliche Einschränkung besteht darin, dass SCNAs bei TFxs unter 5 % nicht nachgewiesen werden können, was die Überwachung von Erkrankungen bei Augen verhindert, bei denen die Tumorlast signifikant gesunken ist. Unserer Erfahrung nach tritt dies am häufigsten bei Augen auf, die auf eine intravitreale Chemotherapie ansprechen23.
Um die TFx der cfDNA in der AH zu bestimmen, wird eine CNA-basierte TFx-Schätzsoftware verwendet. Diese Software ist ein standardisiertes und akzeptiertes Werkzeug für die TFx-Berechnung in Flüssigbiopsien, und ihr Algorithmus wurde ausführlich beschrieben38,51. Kurz gesagt, die CNA-basierte TFx-Schätzsoftware sagt großräumige SCNAs innerhalb sequenzierter cfDNA unter Verwendung eines versteckten Markov-Modells voraus. TFx-Schätzungen werden auf der Grundlage des Vorhandenseins von SCNAs abgeleitet, wobei Unterschiede in der Subklonalität und Ploidie an jedem Locus berücksichtigt werden, und aus diesen wählt die CNA-basierte TFx-Schätzsoftware eine optimale TFx-Lösung38 aus. Eine inhärente Einschränkung von CNA-basierter TFx-Schätzsoftware besteht jedoch darin, dass TFx auf der Grundlage des Vorhandenseins von SCNAs in einer Stichprobe berechnet wird. daher ist es nicht in der Lage, TFx in Proben ohne SCNAs (d. h. mit flachen genomischen Profilen) zu bestimmen38. Wie bereits gezeigt wurde, weisen nicht alle RB-Tumoren die SCNAs 14,15,18,21,23,45 auf. Daher bedeutet ein sehr niedriger TFx-Wert, wie er von einer CNA-basierten TFx-Schätzsoftware bestimmt wird, dass entweder 1) die AH-Probe keine messbare tumorbasierte cfDNA enthält oder 2) Tumor-cfDNA vorhanden ist, aber von der CNA-basierten TFx-Schätzsoftware aufgrund eines Mangels an SCNAs38 nicht nachweisbar ist. Die variante Allelfraktion (VAF) für SNV ist ein Surrogat für TFx. Aus diesem Grund arbeiten wir an der Entwicklung einer RB1-basierten TFx-Pipeline auf Basis von VAF, um auch RB-Augen ohne SCNAs eine longitudinale TFx-Überwachung zu ermöglichen. Angesichts der Tatsache, dass alle RB-Tumoren, mit Ausnahmen für primäre MYCN-getriebene Tumoren 14,15,42,43,44,45, somatische Mutationen im RB1-Gen enthalten, würde eine Pipeline, die nicht von SCNAs abhängig ist, die Anwendung des hier vorgestellten Flüssigbiopsieprotokolls erweitern. Da SNVs auf TFxs unter 5 % überwacht werden können, würde dies die Empfindlichkeit unserer Plattform erhöhen.
Die hier beschriebene AH-Flüssigbiopsieplattform ist nicht die einzige Flüssigbiopsieplattform, die in der Literatur für RB existiert, aber bemerkenswerterweise ist sie die erste, die das Wässrige als angereicherte Quelle für Tumor-DNA beschreibt, und die erste, die den Nachweis von ctDNA über zwei Mechanismen (SCNAs und RB1-SNVs) in einer Bioflüssigkeit beschreibt, basierend auf bisher veröffentlichten Arbeiten. Mit der Fähigkeit, ctDNA auf zwei Arten nachzuweisen, hat die AH-Flüssigbiopsie eine höhere Empfindlichkeit als andere Plattformen in der Literatur. Zum Beispiel hat eine andere Gruppe erfolgreich RB-ctDNA in der AH mit RB1-SNVs nachgewiesen 52. Diese Plattform stützte sich jedoch auf gezielte NGS-Reads, die auf a priori Wissen über die SNV basierten. Im Gegensatz dazu verwendet die in diesem Manuskript beschriebene Plattform ein unvoreingenommenes WGS, was ihr die Möglichkeit verleiht, SCNAs und SNVs zu erkennen. Es wurden auch blutbasierte Flüssigbiopsien versucht, obwohl die erhaltene cfDNA durchweg unter dem Schwellenwert für den SCNA-Nachweis lag, der die Prognose für die Wahrscheinlichkeit einer okulären Rettung darstellt (zu diesem Zeitpunkt wurde nicht gezeigt, dass RB1-SNVs für die Augenrettung prognostisch sind, Es könnte jedoch eine sich entwickelnde Rolle bei der Erkennung von metastasierenden Erkrankungen geben)19. Kothari et al. beschrieben RB1-SNVs im Plasma von RB-Patienten, aber nur solche mit einer fortgeschrittenen intraokularen Erkrankung, die eine Enukleation erforderte16. Die hier beschriebene AH-Flüssigbiopsie-Plattform ist in der Lage, ctDNA in der AH von weniger fortgeschrittenen Augen nachzuweisen, auch ohne das Vorhandensein von Seeding19,21. Darüber hinaus sind blutbasierte Flüssigbiopsien nicht augenspezifisch, da die aus dem Serum isolierte ctDNA bei bilateraler RB aus beiden Augen stammen kann. Dies schränkt den klinischen Nutzen blutbasierter Plattformen ein, insbesondere bei bilateralen Patienten, die 40 % der RB-Fälle ausmachen, während die AH augenspezifisch bleibt und sowohl auf der SNV- als auch auf der SCNA-Ebene eine Heterogenität zwischen den Augen aufweisen kann53.
Die Bedeutung der AH-Flüssigbiopsie für das Gebiet der RB ist von größter Bedeutung. Die AH-Flüssigbiopsie bietet nicht nur die Möglichkeit, die intratumorale Dynamik bei Augen, die sich aktiv einer Therapie unterziehen, besser zu verstehen, sondern hat auch das Potenzial, die Patientenversorgung zu verbessern. Basierend auf früheren Studien haben wir eine molekulare Signatur etabliert – basierend auf dem Vorhandensein einer MYCN-Amplifikation oder einer Chromosomen-6p-Verstärkung mit einer Amplitude von ≥1,5 Verhältnis zum Median –, die für eine 16,5-fach erhöhte Wahrscheinlichkeit eines Behandlungsversagens prognostisch ist, die eine Enukleation erforderlich macht 18,20,21. Mit diesem Wissen zum Zeitpunkt der Diagnose wären Ärzte besser in der Lage, Familien über geeignete Behandlungsoptionen und die Wahrscheinlichkeit einer Augenrettung mit den aktuellen Therapeutika zu beraten. Obwohl die klinische Validität der AH-Flüssigbiopsie-Plattform für RB nachgewiesen wurde 17,18,19,20,21,22,23, ist sie derzeit nur für die Forschung zugelassen; größere prospektive multizentrische Studien sind erforderlich, bevor die AH-Flüssigbiopsie klinisch eingesetzt werden kann, um die Patientenversorgung bei RB zu unterstützen. Unabhängig davon hat die AH-Flüssigbiopsie das Potenzial, in Zukunft eine Präzisionsonkologie zu ermöglichen, nicht nur bei RB, sondern auch bei anderen Augentumoren.
Offenlegungen
Jesse Berry, Liya Xu und James Hicks haben eine Patentanmeldung mit dem Titel Aqueous Humor Cell-Free DNA for Diagnostic and Prognostic Evaluation of Ophthalmic Disease eingereicht. Ansonsten berichten die Autoren von keinem potenziellen Interessenkonflikt.
Danksagungen
Diese Forschung wurde durch die folgenden Quellen unterstützt: NCI des NIH Award K08CA232344 (an J. L. Berry); Hyundai Hope on Wheels RGA012351 (mit J. L. Berry); Childhood Eye Cancer Trust (an J.L. Berry); American Cancer Society IRG-16-181-57 (an J. L. Berry); Wright Foundation (an J.L. Berry und M.E. Kim); Knights Templar Eye Foundation (für J.L. Berry); Die Larry and Celia Moh Foundation (an J. L. Berry); Das Institut für Familien, Inc., Kinderkrankenhaus Los Angeles (J. L. Berry); ein uneingeschränkter Zuschuss des Fachbereichs von Research to Prevent Blindness (alle); Das NCI P30CA014089 (alle); Vicky Joseph Research Fund (an P. Kuhn); Carol Vassiliadis Research Fund (an P. Kuhn); und USC Dornsife College of Letters, Arts and Sciences (an P. Kuhn).
Materialien
| Name | Company | Catalog Number | Comments |
| 1 cc syringe | surgical grade, whatever available in hospital | ||
| 32 G needle | surgical grade, whatever available in hospital | ||
| Aligner | Authors use Bowtie2 (http://bowtie-bio.sourceforge.net/bowtie2/index.shtml) but other aligners such as BWA or GRCh38 will also work | ||
| Atropos | generic term: adapter remover. https://atropos.readthedocs.io/en/latest/index.html# | ||
| Bioanalyzer High Sensitivity DNA Kit | Agilent | 5067-4626 | generic term: DNA fragment size profiling assay |
| BWA-MEM | generic term: long sequence aligner. http://bio-bwa.sourceforge.net/bwa.shtml | ||
| DNAcopy | Bioconductor | generic term: DNA copy number data analysis. https://bioconductor.org/packages/release/bioc/html/DNAcopy.html | |
| dsDNA High Sensitivity Assay | Qubit | Q32851 | generic term: DNA quantification assay |
| FreeBayes | generic term: sequence variant determiner. https://github.com/freebayes/freebayes | ||
| ichorCNA software | generic term: CNA-based TFx estimation. https://github.com/broadinstitute/ichorCNA | ||
| Illumina platform | Illumina | generic term: NGS platform; please note that other NGS platforms will work in principle, but have not been trialed by these authors | |
| NovoAlign (v3) | Novocraft | generic term: mapping of short reads onto reference genome. http://www.novocraft.com/products/novoalign/ | |
| QIAamp Circulating Nucleic Acid Kit | Qiagen | 55114 | generic term: cfDNA isolation and purification kit |
| QIAseq Ultralow Input Library Kit | Qiagen | 180492 | generic term: DNA library sequencing kit |
| Samtools rmdup | generic term: tool to remove duplicate reads. http://www.htslib.org/doc/samtools-rmdup.html | ||
| VarDict | generic term: variant caller. https://github.com/AstraZeneca-NGS/VarDict | ||
| Variant Effect Predictor | Ensembl | generic term: variant effect determinator. https://uswest.ensembl.org/info/docs/tools/vep/index.html |
Referenzen
- Fernandes, A. G., Pollock, B. D., Rabito, F. A. Retinoblastoma in the United States: A 40-year incidence and survival analysis. Journal of Pediatric Ophthalmology & Strabismus. 55 (3), 182-188 (2018).
- Linn, A. M. Intraocular retinoblastoma: the case for a new group classification. Ophthalmology Clinics of North America. 18 (1), 41-53 (2005).
- Shields, J. A., Shields, C. L., Ehya, H., Eagle, R. C., De Potter, P. Fine-needle aspiration biopsy of suspected intraocular tumors. The 1992 Urwick lecture. Ophthalmology. 100 (11), 1677-1684 (1993).
- Karcioglu, Z. A., Gordon, R. A., Karcioglu, G. L. Tumor seeding in ocular fine needle aspiration biopsy. Ophthalmology. 92 (12), 1763-1767 (1985).
- Karcioglu, Z. A. Fine needle aspiration biopsy (FNAB) for retinoblastoma. Retina. 22 (6), 707-710 (2002).
- Eide, N., Syrdalen, P., Walaas, L., Hagmar, B. Fine needle aspiration biopsy in selecting treatment for inconclusive intraocular disease. Acta Ophthalmologica Scandinavica. 77 (4), 448-452 (1999).
- Eide, N., Walaas, L. Fine-needle aspiration biopsy and other biopsies in suspected intraocular malignant disease: a review. Acta Ophthalmologica. 87 (6), 588-601 (2009).
- Eriksson, O., Hagmar, B., Ryd, W. Effects of fine-needle aspiration and other biopsy procedures on tumor dissemination in mice. Cancer. 54 (1), 73-78 (1984).
- Ali, M. J., Honavar, S. G., Vemuganti, G. K., Singh, A. D. Fine needle aspiration biopsy of retinal tumors. Monographs in Clinical Cytology. 21, 72-81 (2012).
- Di Nicolantonio, F. a. l., et al. The chemosensitivity profile of retinoblastoma. Recent Results in Cancer Research. 161, 73-80 (2003).
- Francis, J. H., et al. Efficacy and toxicity of second-course ophthalmic artery chemosurgery for retinoblastoma. Ophthalmology. 122 (5), 1016-1022 (2015).
- Francis, J. H. Efficacy and toxicity of intravitreous chemotherapy for retinoblastoma: Four-year experience. Ophthalmology. 124 (4), 488-495 (2017).
- Francis, J. H., et al. Current treatment of bilateral retinoblastoma: The impact of intraarterial and intravitreous chemotherapy. Neoplasia. 20 (8), 757-763 (2018).
- Rushlow, D. E., et al. Characterisation of retinoblastomas without RB1 mutations: genomic, gene expression, and clinical studies. The Lancet Oncology. 14 (4), 327-334 (2013).
- Afshar, A. R., et al. Next-generation sequencing of retinoblastoma identifies pathogenic alterations beyond RB1 inactivation that correlate with aggressive histopathologic features. Ophthalmology. 127 (6), 804-813 (2020).
- Kothari, P. Cell-free DNA profiling in retinoblastoma patients with advanced intraocular disease: An MSKCC experience. Cancer Medicine. 9 (17), 6093-6101 (2020).
- Berry, J. L., et al. Potential of aqueous humor as a surrogate tumor biopsy for retinoblastoma. JAMA Ophthalmology. 135 (11), 1221-1230 (2017).
- Berry, J. L., et al. Genomic cfDNA analysis of aqueous humor in retinoblastoma predicts eye salvage: the surrogate tumor biopsy for retinoblastoma. Molecular Cancer Research. 16 (11), 1701-1712 (2018).
- Berry, J. L., et al. Aqueous humor is superior to blood as a liquid biopsy for retinoblastoma. Ophthalmology. 127 (4), 552-554 (2020).
- Xu, L., et al. Chromosome 6p amplification in aqueous humor cell-free DNA Is a prognostic biomarker for retinoblastoma ocular survival. Molecular Cancer Research. 18 (8), 1166-1175 (2020).
- Xu, L., et al. Establishing the clinical utility of ctDNA analysis for diagnosis, prognosis, and treatment monitoring of retinoblastoma: The aqueous humor liquid biopsy. Cancers. 13 (6), (2021).
- Xu, L. Simultaneous identification of clinically relevant RB1 mutations and copy number alterations in aqueous humor of retinoblastoma eyes. Ophthalmic Genetics. 41 (6), 526-532 (2020).
- Polski, A., et al. Longitudinal aqueous humor sampling reflects treatment response in retinoblastoma patients. Investigative Ophthalmology & Visual Science. 61 (7), 1394 (2020).
- . Genome Browser Available from: https://genome.ucsc.edu/cgi-bin/hgTracks?db=hg19&lastVirtModeType=default&lastVirtModeExtraState=&virtModeType=default&3A15578261%_2D15621068&hgsid=1135765621_qIuA0U (2021)
- Navarro Gonzalez, J., et al. The UCSC genome browser database: 2021 update. Nucleic Acids Research. 49 (1), 1046-1057 (2021).
- Li, H. A statistical framework for SNP calling, mutation discovery, association mapping and population genetical parameter estimation from sequencing data. Bioinformatics. 27 (21), 2987-2993 (2011).
- Baslan, T., et al. Genome-wide copy number analysis of single cells. Nature Protocols. 7 (6), 1024-1041 (2012).
- Huber, W., et al. Orchestrating high-throughput genomic analysis with Bioconductor. Nature Methods. 12 (2), 115-121 (2015).
- Baslan, T., et al. Erratum: Genome-wide copy number analysis of single cells. Nature Protocols. 11 (3), 1024-1041 (2016).
- Didion, J. P., Martin, M., Collins, F. S. Atropos: specific, sensitive, and speedy trimming of sequencing reads. PeerJ. 5, 3720 (2017).
- Li, H. Aligning sequence reads, clone sequences and assembly contigs with BWA-MEM. arXiv. , (2013).
- Garrison, E., Marth, G. Haplotype-based variant detection from short-read sequencing. arXiv. , (2012).
- McLaren, W., et al. The ensembl variant effect predictor. Genome Biology. 17 (1), 122 (2016).
- Lai, Z., et al. VarDict: a novel and versatile variant caller for next-generation sequencing in cancer research. Nucleic Acids Research. 44 (11), 108 (2016).
- . GitHub Available from: https://github.com/broadinstitute/ichorCNA (2017)
- Adalsteinsson, V. A., et al. Scalable whole-exome sequencing of cell-free DNA reveals high concordance with metastatic tumors. Nature Communications. 8 (1), 1-13 (2017).
- . GitHub Available from: https://github.com/broadinstitute/ichorCNA/wiki/Interpreting-ichorCNA-results (2017)
- Berry, J. L., et al. Long-term outcomes of Group D retinoblastoma eyes during the intravitreal melphalan era. Pediatric Blood & Cancer. 64 (12), (2017).
- Berry, J. L., et al. Long-term outcomes of Group D eyes in bilateral retinoblastoma patients treated with chemoreduction and low-dose IMRT salvage. Pediatric Blood & Cancer. 60 (4), 688-693 (2013).
- Francis, J. H., et al. Molecular changes in retinoblastoma beyond RB1: Findings from next-generation sequencing. Cancers (Basel). 13 (1), (2021).
- Schwermer, M., et al. Comprehensive characterization of RB1 mutant and MYCN amplified retinoblastoma cell lines. Experimental Cell Research. 375 (2), 92-99 (2019).
- Davies, H. R. Whole-genome sequencing of retinoblastoma reveals the diversity of rearrangements disrupting RB1 and uncovers a treatment-related mutational signature. Cancers. 13 (4), 754 (2021).
- Lillington, D. M., et al. Comparative genomic hybridization of 49 primary retinoblastoma tumors identifies chromosomal regions associated with histopathology, progression, and patient outcome. Genes, Chromosomes and Cancer. 36 (2), 121-128 (2003).
- Greger, V., et al. Frequency and parental origin of hypermethylated RB1 alleles in retinoblastoma. Human Genetics. 94 (5), 491-496 (1994).
- Raizis, A. M., et al. DNA hypermethylation/boundary control loss identified in retinoblastomas associated with genetic and epigenetic inactivation of the RB1 gene promoter. Epigenetics. 1-15, (2020).
- Munier, F. L., et al. Profiling safety of intravitreal injections for retinoblastoma using an anti-reflux procedure and sterilisation of the needle track. British Journal of Ophthalmology. 96 (8), 1084-1087 (2012).
- Chévez-Barrios, P., et al. Response of retinoblastoma with vitreous tumor seeding to adenovirus-mediated delivery of thymidine kinase followed by ganciclovir. Journal of Clinical Oncology. 23 (31), 7927-7935 (2005).
- Shao, W., Khin, S., Kopp, W. C. Characterization of effect of repeated freeze and thaw cycles on stability of genomic DNA using pulsed field gel electrophoresis. Biopreservation and Biobanking. 10 (1), 4-11 (2012).
- Choudhury, A. D., et al. Tumor fraction in cell-free DNA as a biomarker in prostate cancer. JCI Insight. 3 (21), (2018).
- Gerrish, A., et al. Non-invasive diagnosis of retinoblastoma using cell-free DNA from aqueous humour. British Journal of Ophthalmology. 103 (5), 721-724 (2019).
- Wong, E. Y., Xu, L., Shen, L. Inter-eye genomic heterogeneity in bilateral retinoblastoma via aqueous humor liquid biopsy. NPJ Precis Oncol. 5 (1), 73 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten