Method Article
망막모세포종에 대한 액체 생검으로서의 수액: 맑은 각막 천자 및 게놈 분석
요약
수액은 안구 외 전이의 위험으로 인해 생체 내에서 생검할 수 없는 안구내암인 망막모세포종에 대한 고수율 액체 생검입니다. 본 연구에서는 맑은 각막 천자를 통해 수액을 안전하게 추출하는 방법과 예후 바이오마커를 확인하기 위한 유전체 분석 단계를 제시한다.
초록
망막모세포종에 대한 액체 생검 플랫폼의 적용은 이러한 환자에서 직접 종양 생검이 금지되어 있다는 점을 감안할 때 상당한 잠재적 임상적 유용성이 있습니다. 수액(AH)은 종양과 분리된 구획에 형성되지만 동일한 안구 공간 내에 둘러싸여 있습니다. 따라서 이 질병에 대한 종양 생검에 대한 액체 생검 또는 대체물로 사용할 수 있는 눈 특이적 종양 게놈 정보의 풍부한 소스입니다. 이 원고는 투명한 각막 천자를 통해 망막모세포종 눈에서 AH를 안전하게 추출하는 방법론을 자세히 설명합니다. 또한 cell-free DNA 분리 및 정제, 차세대 염기서열분석, 체세포 복제 수 변경(SCNA) 분석, RB1 단일 뉴클레오티드 변이체(SNV) 돌연변이 식별 및 종양 분획 추정을 포함한 게놈 분석 단계를 제시합니다. AH 액체 생검 플랫폼의 사전 분석, 분석 및 초기 임상 유효성이 평가되었습니다. 그러나 제한이 없는 것은 아닙니다. 이는 주로 분석의 특정 단계에 필요한 cell-free DNA의 양에 기인합니다. 현재 망막모세포종에 대해 연구 중인 다른 혈액 기반 액체 생검 플랫폼과 비교했을 때, AH 기반 플랫폼은 눈에서 추출할 수 있는 생체 유체의 양(따라서 DNA의 양)에 의해 제한됩니다. 장점은 AH가 눈에만 적용된다는 것입니다. 여기에서 논의된 플랫폼은 두 가지 메커니즘(SCNA 및 RB1 SNV)을 통해 AH에서 순환하는 종양 DNA를 검출하여 종양 게놈 정보를 식별하는 데 더 높은 감도를 제공한다는 점에서 독특합니다. AH 액체 생검은 망막모세포종 환자의 정밀 종양학에 직접 임상적으로 적용할 수 있는 잠재력을 가지고 있으며, AH가 각 눈의 종양에 특이적이기 때문에 양측 질환 환자에게 특히 중요합니다. 다른 안구 종양이 있는 환자에게도 이 플랫폼을 적용하는 것에 대한 연구가 진행 중입니다.
서문
망막모세포종(RB)은 희귀하고 독특한 암입니다. 영유아의 발달 중인 망막에서 형성되는 가장 흔한 원발성 안구 내 악성 종양이지만, 전 세계적으로 매년 약 7,000건의 사례가 발생하며 그 중 약 250-300건이 미국에서 발생합니다. 선진국에서는 환자 생존율이 98%에 육박하지만1, 임상적으로 IIRC(International Intraocular RB Classification)2 Group D/E 또는 AJCC cT2b/3으로 분류되는 진행성 안구의 안구 생존율은 훨씬 낮습니다. 이러한 진행된 눈의 대부분은 1차적으로(1차 치료로) 또는 2차적으로(지구 구조 요법 시도가 실패한 후) 적출됩니다. 현재 안구 종양학의 임상 실습에는 RB 환자의 진단, 안구 생존에 대한 예후 또는 치료 모니터링을 돕기 위해 현재 임상적으로 사용되는 종양에서 유래한 눈 특이적 분자 바이오마커가 없습니다. 이는 안구 외 종양 확산에 대한 우려로 인해 RB의 직접 종양 생검이 금지되어 있기 때문에 종양 조직이 적출된 눈의 분자 및 유전자 분석에만 사용할 수 있다는 사실에 기인합니다 3,4,5,6,7,8,9 . 이러한 금지 조항으로 인해, RB 종양 유전학 및 임상적 상관관계에 대한 이전의 후향적 연구는 적출된 눈에서 얻은 종양 조직의 분석에만 국한되었다 10,11,12,13. 따라서 진단 시와 안구 치료 전반에 걸쳐 사용할 수 있는 객관적인 종양 유래 분자 데이터가 부족했습니다. 이로 인해 생체 내 종양 생물학에 대한 이해와 이러한 종양이 치료 전반에 걸쳐 분자 수준에서 변화하는 방식에 대한 이해뿐만 아니라 이러한 젊은 암 환자를 위한 개인화되고 눈에 특화된 게놈 기반 치료 계획을 개발할 수 있는 능력도 제한되었습니다.
생검이 금지되는 것 외에도, 이 암의 또 다른 독특한 측면은 대부분의 종양이 세포주기를 조절하는 RB1 종양 억제 유전자의 이중 대립형질 소실에 의해 시작된다는 것입니다. 발달 중인 망막은 이러한 손실에 매우 민감합니다. 환자의 60%에서 RB1 유전자의 이중 대립유전자 불활성화는 망막에서 체세포 손실로만 발생하며 편측성 질환을 초래합니다. 그러나 환자의 40%에서 초기 RB1 돌연변이가 생식세포에서 발생하고 그 후 망막에서 두 번째 '타격'이 발생합니다. 이 아이들의 경우 양쪽 눈에 영향을 미치는 여러 종양이 있는 경우가 많습니다. 마지막으로, 종양의 매우 작은 하위 집합(<2%)은 RB1 유전자의 돌연변이 없이 MYCN 증폭에 의해 주도되는 것으로 보입니다. MYCN에 의한 종양은 거의 보편적으로 치료에 실패하고 적출이 필요하지만, 현재로서는 진단 시 이 공격적인 종양 아형을 확인할 수 있는 명확하고 객관적인 방법이 없습니다14,15. 또한, 안구 내 RB 종양 활성에 대한 모니터링은 거의 전적으로 안구 종양 전문의의 각 눈에 대한 영상 및 임상 관찰에 의존합니다. 진단, 예후 또는 치료 전반에 걸쳐 안구 특이적 종양 역학을 모니터링할 수 있는 객관적이고 정량적인 수단은 없습니다. RB에 대한 이러한 고유한 한계로 인해 이 암에 대한 액체 생검 플랫폼의 전망은 매력적입니다. 액체 생검은 체액을 사용하여 cell-free DNA를 분리하고 염기서열을 분석하여 순환 종양 DNA(ctDNA)로 알려진 종양 유래 DNA인지 확인합니다. 혈청은 다른 암에 대한 연구가 이루어지고 있지만, 혈청에서 발견되는 RB ctDNA는 눈에 특이적이지 않아 양측성 질환을 앓고 있는 40%의 환자에게 명확한 한계가 있습니다. 또한, 일반적으로 종양 분율(<5%)이 낮은, 진행성 안구 내 또는 전이성 질환의 상황에서만 설명되었습니다16.
환자와 가족이 직면한 이러한 실제 임상 문제를 해결하기 위한 노력의 일환으로, 2017년에 우리는 수액(AH, 눈 앞의 맑은 액체)이 RB17을 평가하기 위해 액체 생검 또는 종양 생검의 대체물로 사용할 수 있는 고수율 ctDNA 공급원임을 입증했습니다. 18,19. 현재까지 200개 이상의 샘플에서 샘플의 >95%에서 ctDNA가 확인되었으며, 여기에는 덜 진행된 IIRC 그룹 A, B 및 C 눈과 양측 환자의 눈 간 게놈 차이가 포함됩니다18,19. 이 DNA는 활발하게 치료를 받고 있거나 치료가 없는 눈에서 관련 분자 바이오마커를 검출하기 위해 안전하고 효과적으로 분리할 수 있습니다 18,20,21. AH 액체 생검 플랫폼은 종양 형성을 시작하는 진단용 RB1 병원성 변이체 또는 1차 MYCN 증폭을 식별하는 데에도 사용할 수 있습니다21,22. 중요한 것은, 예후 분자 서명(염색체 6p 이득의 존재, 진폭이 중앙값 또는 국소 MYCN 증폭에 대해 ≥1.5)을 확인했으며, 이는 안구 내 종양 조절 상실 가능성이 16.5 증가하여 안구 제거가 필요한 경우와 관련이 있다는 것입니다 18,20,21. 마지막으로, AH에서 ctDNA 종양 분율(TFx)의 변화는 치료 반응과 상관관계가 있는 것으로 입증되었는데, 이는 수치가 높을수록 활동성 질환과 상관관계가 있고, 수치가 감소하면 긍정적인 치료 반응과 관련이 있기 때문이다23. 이러한 응용 분야와 잠재적인 임상적 유용성을 감안할 때, 우리는 AH 생검 및 평가를 위한 방법론을 설명하고자 했습니다. 여기에는 샘플 수집을 위한 명확한 각막 천자 및 게놈 분석을 위한 프로토콜, 특히 cfDNA 라이브러리 구성 및 염기서열 분석, SCNA 진폭 결정, RB1 병원성 변이 식별 및 TFx 계산이 포함됩니다.
프로토콜
이 연구는 Children's Hospital Los Angeles와 University of Southern California Institutional Review Board의 승인 하에 수행되었으며 헬싱키 선언의 원칙을 준수합니다. 서면 동의는 항상 모든 참가자의 법적 보호자로부터 얻습니다.
AH 액체 생검 워크플로우에 대한 개략도는 그림 1에서 볼 수 있습니다.
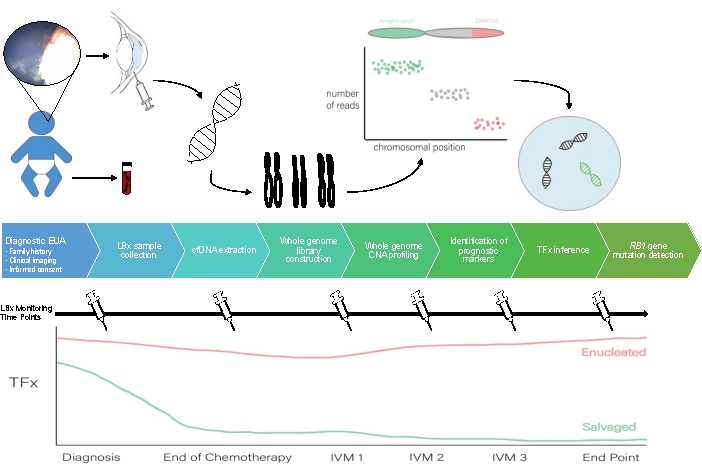
그림 1: 본 문서에 설명된 프로토콜에 수반되는 AH 액체 생검을 위한 워크플로우. EUA, 마취 하에 검사; LBX, 액체 생검; cfDNA, 무세포 DNA; CNA, 복제 수 변경; TFx, 종양 분율; IVM, 유리체강내 멜팔란. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
1. 외과 절차:
참고: 이 절차는 RB 환자의 임상 평가를 위해 마취(EUA) 하에서 정기 검사 중에 수행됩니다. AH를 추출하기 위한 천자 시술은 안과 수술에 대한 표준 교육을 이수한 훈련된 안과 의사만 수행해야 합니다.
- 환자 선택 및 포함 기준은 다음과 같습니다.
- 챔버가 종양, synechiae 또는 백내장 수정체에서 얕아지지 않고 형성되고 투명한지 확인합니다.
참고: 일부 고급 눈에서는 진단 시 챔버가 너무 얕아서 AH를 안전하게 추출할 수 없습니다. 이러한 경우, 전신 또는 동맥 내 화학 요법의 첫 번째 주기가 끝날 때까지 기다리는 것이 좋습니다. 일단 주종양이 줄어들면 일반적으로 챔버가 깊어지므로 안전한 접근이 가능합니다. - 모든 구조가 명확하게 보이고 압력이 <22mm Hg인지 확인하여 압력 역학으로 인해 챔버가 급격히 얕아지지 않도록 합니다. 이는 모든 시점에 해당되지만 진단 시 AH를 측정할 때 특히 중요합니다.
- 종양에 의한 전방 분절의 직접적인 침범이 없는지 확인하고, 이는 직접 시각화 및 초음파 생체 현미경 검사로 평가됩니다. 이 포함 기준은 일반적으로 EUA 중에 식별됩니다.
- 챔버가 종양, synechiae 또는 백내장 수정체에서 얕아지지 않고 형성되고 투명한지 확인합니다.
- 환자가 적절하다고 판단되고 동의하면 환자를 수술실로 데려옵니다. 담당 마취과 의사가 RB 환자의 임상 치료를 위해 EUA의 일상적으로 마취를 시작할 수 있도록 합니다.
- 환자에게 삽관 및 진정제를 투여하고 EUA와 관련된 모든 임상 검사, 영상 및 국소 치료 요구(예: 레이저 또는 냉동 요법)가 완료되면 액체 생검 플랫폼용 AH를 추출합니다. 일반적인 멸균 방식으로 눈을 준비하고 드레이프하십시오. 이것은 순환 간호사 또는 수술 외과의가 완료할 수 있습니다.
- EUA를 실시하기 전에, EUA를 위해 기관에서 일상적으로 사용하는 확장 점안액을 수술용 눈에 삽입하십시오.
참고: 여기에서는 2.5% 페닐에프린, 1% 사이클로펜톨레이트 및 1% 트로피카마이드로 구성된 복합 안약이 사용됩니다. 그러나 EUA의 확장에 사용되는 표준 점안액은 무엇이든 충분합니다. 단순히 AH를 추출하기 위해 눈을 확장할 필요는 없습니다. - 베타딘 방울 5%를 수술용 눈에 놓고 스펀지를 사용하여 중앙에서 시작하여 주변부로 움직여 바릅니다. 수술 준비에 상부 및 하부 눈꺼풀 가장자리를 포함시킵니다. 베타딘을 사용하여 수술 준비를 3회 완료한 후 최소 2분 후에 진행하십시오.
- 멸균된 파란색 수건을 환자의 머리에 씌우고 머리가 떨어지지 않도록 머리 아래에 밀어 넣습니다. 멸균된 파란색 수건이나 거즈로 속눈썹과 안와 주위 부위를 말리십시오.
- 수술 의사가 몸을 문지르고, 가운을 입고, 장갑을 끼게 하십시오.
- 멸균된 파란색 커튼으로 환자를 덮고 수술 눈이 노출되도록 합니다. 멸균 영역의 최적 시각화를 위해 멸균 뚜껑 검경을 배치합니다.
참고: 아이가 EUA에 대해 전신 마취를 받고 있으므로 본 기관에서는 추가 국소 마취가 필요하지 않습니다. - 기관 프로토콜에 따라 타임아웃을 수행합니다. 여기에서 EUA에 대한 초기 시간 초과가 수행되고 절차 전에 눈을 확인하기 위해 두 번째 시간 초과가 수행됩니다.
- EUA를 실시하기 전에, EUA를 위해 기관에서 일상적으로 사용하는 확장 점안액을 수술용 눈에 삽입하십시오.
- 작동 현미경을 사용하여 1cc 주사기에 32G 바늘로 0.1mL의 AH를 추출하여 투명 각막 천자를 수행합니다.
- 시술을 시작하기 전에 멸균 균형 잡힌 소금 용액(각 mL에는 염화나트륨 0.64%, 염화칼륨 0.075%, 염화칼슘 이수화물 0.048%, 염화마그네슘 육수화물 0.03%, 아세트산나트륨 삼수화물 0.39%, 구연산나트륨 이수화물 0.17%, 수산화나트륨 및/또는 염산(pH 조절용) 및 주사용 물 포함)으로 눈을 적셔 각막 윤활을 유지합니다. 이것은 표준 상업용 준비입니다.
- 32G 바늘이 1cc 주사기에 루어 록되어 있고 주사기에 압력이 없는지 확인합니다(사용하기 전에 주사기의 플런저를 여러 번 안팎으로 움직여).
- 그 후, 엿굵기의 투명한 각막을 통해 바늘을 통과시키고(천자의 표준인 것처럼 수직) 말초 팽창된 홍채 위의 전방(anterior chamber) 내에 머뭅니다.
- 발치하는 동안 바늘 끝이 홍채 앞쪽에 비스듬히 올라가 현미경을 통해 직접 시각화할 수 있도록 합니다. 원하는 경우 바늘이 각막을 통과할 때 0.12 겸자로 눈을 안정시킵니다.
알림: 바늘 끝과 안구 구조 사이에 접촉이 없어야 합니다. 그것은 중앙 전방의 주변 홍채 위에 남아 있어야 합니다. - AH 0.1mL를 추출합니다. 주사기 플런저를 외과의가 주로 사용하지 않는 손으로(연습을 통해) 또는 바늘을 움직이지 않고 훈련된 조수가 조작합니다. 바늘 끝의 직접적인 시각화를 잃지 마십시오. 눈의 해부학적 구조를 우선시하고 필요한 경우 AH를 줄여 안방이 형성된 상태를 유지하도록 합니다.
- 발치 후 전방에서 바늘을 부드럽게 제거합니다. 챔버가 형성되어 있지만 약간 얕은 상태로 유지되는지 확인하십시오. 압력은 부드럽지만 생리적일 것입니다. 눈에서 바늘을 떼어내면 멸균수로 눈을 충분히 목욕시킵니다. 이 단계에서는 물이 세포를 용해시키기 때문에 안전 조치로 발라시멘트 염 용액보다 멸균 물이 선호됩니다.
- 바늘 부위에 누출이 있는지 검사하십시오. 누출이 있는 경우 면봉 어플리케이터를 사용하여 주입 부위에 30초 동안 부드러운 압력을 가한 후 누출 여부를 다시 확인하십시오. 필요한 경우 이 과정을 반복합니다.
참고: 이 접근 방식에서는 누출을 막기 위해 보다 침습적인 방법을 사용할 필요가 없었습니다. 때때로 이 절차 후에는 화학 요법의 유리체강 내 주사가 뒤따릅니다. 이 방법 논문에서 프로토콜은 기본 절차로 AH 추출에 중점을 둡니다.
- 바늘 부위에 누출이 있는지 검사하십시오. 누출이 있는 경우 면봉 어플리케이터를 사용하여 주입 부위에 30초 동안 부드러운 압력을 가한 후 누출 여부를 다시 확인하십시오. 필요한 경우 이 과정을 반복합니다.
- 누출이 없으면 절차가 완료된 것으로 간주됩니다. 환자의 멸균 커튼을 제거하고 얼굴의 베타딘을 청소합니다.
- 주사기에서 바늘 끝을 조심스럽게 제거하고, 멸균 캡을 사용하여 샘플이 손실되지 않도록 루어 잠금 장치를 통해 샘플을 닫고, 주사기에 적절하게 라벨을 붙입니다.
참고: 이것은 아이가 발관하는 동안 임상 연구 코디네이터 또는 외과의가 수행해야 합니다.- 검체 추출 후 -80 °C의 캡이 있는 주사기에 샘플을 보관하십시오. 샘플을 드라이아이스에 담아 수술실에서 적절한 냉동고로 운반할 수 있습니다.
- 환자를 회복 실로 보내기 전에 외과의가 선호하는 수술 대상자에 항생제 안약 한 방울을 떨어뜨립니다. 여기에서는 스테로이드-항생제 조합 안약을 사용하며 안대는 하지 않습니다.
- 절차가 완료되면 담당 마취과 의사가 적절한 발관을 확인합니다. 환자를 회복으로 데려가십시오.
- 기관의 수술 후 프로토콜에 따라 환자를 집으로 퇴원시킵니다.
- 환자에게 눈을 붕대나 보호 덮개로 가릴 필요가 없다고 알립니다.
- 환자에게는 24시간 동안 욕조나 수영장에 머리를 완전히 담그지 말고, 부모에게는 환자(아이)의 눈을 비비지 말라고 알립니다. 환자 활동에는 다른 제한이 없습니다.
- 환자에게 천자로 인한 통증은 없을 것 같지만 EUA 기간 동안 관련 절차로 인해 통증이 있을 수 있음을 알립니다. 환자는 불편함이 있을 경우 외과 의사의 제안에 따라 아세트아미노펜 또는 이부프로펜을 복용할 수 있습니다.
2. cfDNA 분리 및 정제
- AH 추출 후 72시간 이내에 DNA 분리 및 처리를 수행합니다.
- 실온에서 AH를 해동합니다. 샘플을 자주 확인하고 해동이 완료된 직후 추출로 이동하십시오.
- AH에서 cfDNA를 추출하고 제조업체의 사용 설명서에 따라 cfDNA 분리 및 정제 키트(자세한 내용은 재료 표 참조)를 사용하여 50μL의 AVE 용액(키트에 포함)으로 용리합니다.
3. 차세대 염기서열분석(NGS) 및 품질 관리(QC)
- 제조업체의 사용 설명서에 따라 DNA 라이브러리 염기서열분석 키트( 재료 표 참조)를 사용하여 염기서열분석을 위한 DNA 라이브러리를 구성합니다. DNA 라이브러리 염기서열분석 키트 설명서에 자세히 설명된 대로 14주기의 중합효소 연쇄 반응(PCR)으로 라이브러리 DNA를 증폭합니다.
참고: 최종 라이브러리는 게놈 분석을 위한 최종 염기서열분석 1개월 전까지 -20°C에서 보관할 수 있습니다. - 단일 엔드 50 사이클 또는 쌍단 150 사이클 프로토콜에 따라 제조업체의 프로토콜에 따라 NGS 플랫폼( 재료 표 참조)에서 DNA 라이브러리를 염기서열분석합니다.
참고: 다른 NGS 플랫폼은 원칙적으로(여기서는 시도되지 않음) 염기서열분석 플로우 셀에 적합한 어댑터와 함께 작동합니다. - 진단 또는 1차 적출 시 채취한 모든 AH 샘플에 대해 QC를 수행합니다.
참고: DNA 질량이 너무 낮아 아래 설명된 단계의 검출 범위를 벗어나므로 다른 시점에서 채취한 샘플에 대해서는 QC를 수행할 수 없습니다.- 제조업체의 프로토콜에 따라 DNA 정량화 분석 및 크기 프로파일링 분석( 재료 표 참조)에 각각 1μL의 추출된 DNA를 사용합니다.
참고: 피크는 약 300bp여야 합니다. 150bp 미만이면 샘플이 손상된 것입니다. QC를 통과해야 하는 샘플의 예는 보충 파일 1A 를 참조하고, 그렇지 않아야 하는 샘플의 예는 보충 파일 1B 를 참조하십시오.
- 제조업체의 프로토콜에 따라 DNA 정량화 분석 및 크기 프로파일링 분석( 재료 표 참조)에 각각 1μL의 추출된 DNA를 사용합니다.
4. 체세포 복제 수 변경(SCNA) 데이터 분석
- NGS(섹션 3)에서 얻은 판독값을 정렬기를 사용하여 인간 게놈(hg19, Genome Reference Consortium GRCh37, University of California Santa Cruz Genome Browser database)24,25에 매핑합니다.
- PCR 중복을 제거합니다(samtools rmdup26).
- 참조 게놈에서 각 bin에 있는 guanine 및 cytosine 염기의 백분율을 계산하여 guanine-cytosine 함량을 정규화합니다. 샘플 프로그램은 Baslan et al.27에 설명되어 있습니다.
- DNA 세그먼트 복제 번호를 얻으려면 게놈을 5000개의 가변 길이 bin으로 나눈 다음 각 bin에 포함된 상대적인 판독 수를 계산합니다.
- 참조가 없는 log2 비율을 사용하여 복제 수 추정치를 결정합니다. 정상 상염색체 염색체의 중앙값 창 수를 취하십시오.
- DNAcopy(Bioconductor28)를 사용한 원형 이진 분할을 사용하여 분할을 수행합니다.
- SCNA를 기준선에서 20% 편향(log2 비율 = 0)에서 양수로 정의하며, 이는 log2 비율이 0.2(0.87 이하의 비율이 삭제를 정의함)≤ 손실과 0.2(1.15 이상의 비율이 증폭을 정의함)≥ 이득을 의미합니다.
참고: 이러한 임계값은 이전에 확립된 액체 생검 분석27,29와 일치합니다.
5. AH 샘플의 RB1 돌연변이 분석
- 또한 제조업체의 프로토콜에 따라 돌연변이 검출을 위한 캡처 기반 표적 NGS를 위해 전체 게놈 라이브러리를 각각 500ng로 증폭합니다.
- 캡처된 라이브러리에서 NGS 플랫폼 페어링 말단 150bp 또는 50bp 단일 말단 염기서열분석을 제조업체 프로토콜당 >100배까지 수행합니다.
- NGS 데이터를 처리합니다. 본 연구에서는 CHLA Center for Personalized Medicine의 bcbio 파이프라인을 기반으로 한 사내 파이프라인을 활용하였다30.
- Atropos31을 사용하여 어댑터 및 저품질 염기에서 원시 fastq 데이터를 트리밍하고 BWA-MEM 및 NovoAlign(v3)32,33을 사용하여 human GRCh37 참조에 정렬합니다.
- FreeBayes34 로 중복을 표시한 다음 생식계열 변형을 결정합니다.
참고: FreeBayes 변이 호출 도구에서 샘플 게놈의 각 위치를 참조 게놈과 비교하고 대립유전자 수를 수학적으로 모델링하여 유전자형 우도 측정값34을 얻습니다. - 5.3.4-5.3.5 단계에 따라 Ensembl Variant Effect Predictor (v96)35 를 사용하여 변형 주석을 수행합니다.
- VarDict를 사용하여 쌍을 이룬 정상 혈액 샘플(즉, 생식계열 변이에 대한 맹검)이 없는 AH 또는 종양의 체세포 변이를 확인합니다36.
- 한 영역의 연속 변이가 ≤3%의 대체 대립유전자를 가지고 있는 경우 이형접합성의 상실을 호출합니다.
참고: 상용 유전체 분석 소프트웨어는 위에 표시된 매개변수와 함께 사용할 수도 있습니다.
6. cfDNA TFx의 결정
- ichorCNA 소프트웨어(CNA 기반 TFx 추정 소프트웨어)를 사용하여 각 염기서열 AH cfDNA 샘플에 대한 TFx를 추정합니다37.
참고: 이것은 cfDNA TFx38을 결정하기 위한 혈액 기반 액체 생검에 사용되는 표준 소프트웨어입니다. AH에서 소프트웨어를 사용하는 것은 이전에발표되었습니다 21,23. - 게놈 프로파일과 해당 TFx 용액을 개별적으로 검토하여 각 샘플에 대한 TFx 추정치가 적절한지 확인합니다39.
결과
두 눈(사례 33 및 47)의 포괄적인 결과는 다음과 같습니다. 사례 수는 비교 목적으로 이전 간행물과 일치합니다 18,20,21. 치료 의사는 치료 중 AH 액체 생검 결과를 못했습니다. 모든 치료 결정은 무작위 배정되지 않았으며 이전에 발표된 일상적인 치료 표준에 따라 이루어졌습니다40,41. 임상 결과 데이터는 최종 분석이 있을 때까지 게놈 데이터와 분리되어 있었습니다. 제시된 결과의 원시 데이터는 교신 저자의 요청에 따라 제공됩니다. NIH 자금 지원으로 인해 데이터는 NIH 게놈 데이터 공유 정책의 적용을 받으며 향후 통제된 액세스 NIH 지정 데이터 저장소(dbGAP)를 통해 다른 연구자가 사용할 수 있습니다. 또한 교신 저자의 요청을 통해 사용할 수 있습니다.
사례 33과 47은 모두 IIRC 그룹 D2 눈으로 매우 유사한 임상 증상을 보였습니다. 따라서 그들은 부모의 결정에 따라 구제 요법을 받았고, 안과 및 종양 전문의, 특히 사례 47에 대해서는 전신 화학요법을, 사례 33에 대해서는 동맥내 화학요법을 받았다. 2번 눈의 IIRC 그룹에 의존하는 현재 인정된 임상적 예측에 기초하여, 이 두 환자의 눈에 대한 지구 구제의 예상 성공률은 동일했을 것이다: 그룹 D의 경우 65%-70%가 평균이지만, 이는치료 센터 41에 따라 다르다. 그러나 이 센터의 종양 유도 분자 프로파일링에서 지금까지 수집된 Group D 데이터를 기반으로 지구 구제의 예상 성공률은 사례 47(AH에서 6p 이득이 확인되지 않음)의 경우 72%, 사례 33(6p 이득이 확인됨)의 경우 9%입니다18,20,21. 이것은 아래에 설명되어 있습니다.
사례 47은 진단 시 AH 액체 생검 플랫폼을 사용하여 SCNA 및 SNV를 성공적으로 검출한 안구의 예이며, 치료 반응에 종단적으로 대응하는 TFx 경향을 보여줍니다. 환자는 생후 15개월에 14mm x 9mm IIRC Group D, stage cT2b RB와 구 유리체 파종을 동반한 암컷입니다. 그녀는 정기적인 임상 혈청 백혈구 검사에서 결정된 RB1 생식세포 돌연변이에 대해 음성이었습니다. AH에서, 진단 시 AH에서 RB SCNAs 1q gain 및 6p gain이 확인되었으며, 다른 두 개의 non-highly recurrent RB SCNAs 7p loss 및 13q gain이 확인되었습니다(그림 2). 주목할 점은 6p gain의 진폭이 1.2였으며 중앙값에 대한 ≥ 1.5 비율의 진폭만이 좋지 않은 예후를 예고하는 것으로 나타났습니다. 국소 MYCN 증폭이 없었고 6p gain이 1.5 임계값 미만이었다는 점을 감안할 때, 분자 특징에 기반한 치료를 통한 구제의 예후는 높았습니다. 진단 시 채취한 동일한 AH 샘플에서 RB1 병원성 변이체를 검출하기 위해 평가한 결과, RB1 유전자 내에서 변이 대립유전자 빈도가 87.01%(95% 신뢰 구간, 79.7%–94.6%)인 SNV c.958C>T, p.Arg320*이 밝혀졌습니다. 이 환자는 질병의 퇴행과 함께 Carboplatin, Etoposide 및 Vincristine(CEV)을 6주기로 투여받았지만 3번의 순차적 유리체강내 멜팔란(IVM) 주사가 필요한 지속적인 구 및 먼지 같은 파종을 보여주었습니다. IVM 처리 중 AH 샘플 A-C(각각 2주씩 분리)는 게놈 프로파일의 완전한 정상화, TFx 감소 및 DNA 농도 감소를 보여주었으며, 이 모든 것은 질병의 임상적 퇴행과 동시에 나타났습니다(그림 3). 진단 후 TFx 값은 나머지 치료 기간 동안 검출 한계인 5% 미만으로 유지되었습니다. 19개월의 추적 관찰 결과, 눈은 종양 재발이나 질병의 안구 외 전이 없이 안정적으로 유지되었습니다.
이는 사례 33과 대조됩니다. 이 22개월 된 수컷은 11mm x 18mm 망막 종괴와 분진형 유리체 파종으로 전반적으로 유사한 임상 증상을 보였으며, 이는 편측성 IIRC 그룹 D, cT2b 병기와 일치했습니다. 그는 또한 일상적인 임상 혈청 백혈구 검사에서 결정된 RB1 생식세포 돌연변이에 대해 음성이었습니다. 진단 시 측정한 AH는 RB SCNA의 1q gain, 6p gain(이 경우 진폭이 중앙값 대비 1.5 비율) 및 16q loss와 focal 6q loss를 보여주었습니다(그림 4A). 예후 분자 프로파일에 기초하여, 이 눈은 6p ≥ 1.5 진폭의 존재에 기초하여 적출 확률이 16.5 증가했다. RB1 유전자의 전체 길이에 대한 전체 범위에도 불구하고 이 샘플에서 RB1 SNV는 확인되지 않았습니다. RB1 SNV가 확인되지 않는 한 가지 이유는 원발성 MYCN 유발 종양에서 RB1 돌연변이가 항상 예상되지 않기 때문입니다 14,15,42,43,44,45. 그러나 사례 33에서는 AH 샘플이나 적출된 종양 조직에서 MYCN 증폭의 증거가 나타나지 않았습니다(그림 5). 음성 RB1 SNV 결과에 대한 더 그럴듯한 설명은 초기 종양 형성이 후성유전학적 조절 장애(예: 프로모터의 메틸화)에 의해 주도되었다는 것이며, 46,47 이는 RB에서 알려진 현상이며, 이는 본원에 기술된 분석에 의해 확인되지 않을 것입니다.
이 환자에 대한 초기 치료는 멜팔란을 이용한 동맥내 화학요법을 총 4회 투여한 후 지속적인 유리체 파종으로 인해 4번의 IVM 주사를 투여하는 것이었습니다. IVM 요법 중에 3개의 AH 샘플(각각 4주 간격으로 분리)을 얻었으며 진단 시 존재했던 것과 동일한 3개의 SCNA를 보여주었습니다(그림 5). TFx 값은 유리체의 활성 종양 종자를 반영하는 원발성 종양 부피 감소에도 불구하고 치료 전반에 걸쳐 높게 유지되었습니다. 이는 TFx가 눈의 전반적인 질병 상태를 어떻게 나타내는지 보여줍니다. 진단 후 6개월 후, 지속적인 활동성 질환으로 인해 눈이 적출되었습니다. 당시 종양 조직에서 얻은 게놈 프로파일은 진단 당시 얻은 AH 샘플과 92.81% 일치함을 보여주었습니다(그림 4B).
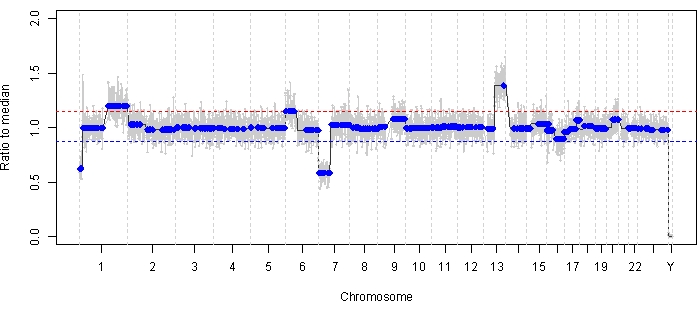
그림 2: 사례 47 진단 시 게놈 프로필. 진단 시 실시한 AH에서 고재발성 RB SCNA인 1q gain 및 6p gain과 함께 non-highly recurrent RB SCNAs 7p loss 및 13q gain이 확인되었습니다. 빨간색 선은 이득에 대한 임계값을 나타내고 파란색 선은 손실에 대한 임계값을 나타냅니다. 특히 6p 이득의 진폭은 중앙값 대비 <1.5 비율로 좋지 않은 예후를 예고하는 분자 서명의 임계값보다 낮았습니다. 따라서 안구 구제에 대한 음성 바이오마커가 없기 때문에 이 눈은 치료에 반응할 것으로 예측됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
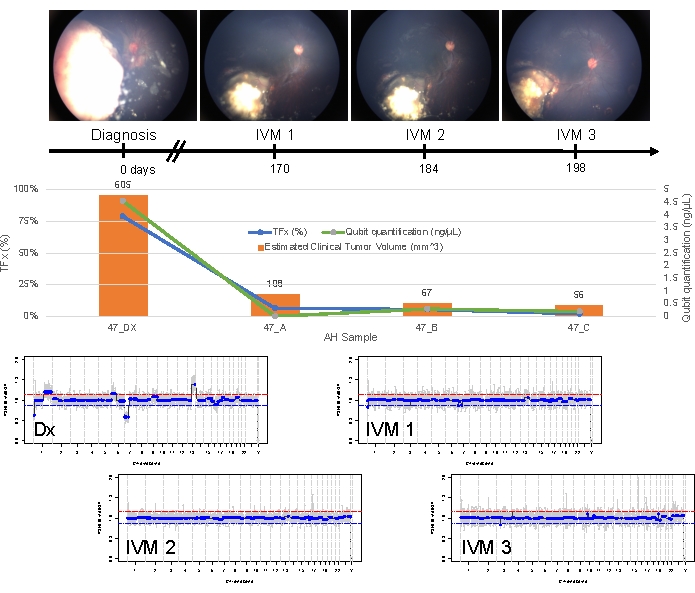
그림 3: 사례 47에 대한 종단 정보. 여기에는 안저 사진, cfDNA 정량화, B 스캔 측정에서 추정된 임상 종양 부피, 게놈 프로필 및 AH가 샘플링된 각 임상 시점(Dx = 진단, A = IVM1, B = IVM2, C = IVM3)에 대한 TFx 추정이 포함됩니다. 이 눈은 치료에 반응하여 19개월의 추적 관찰에서 회복된 상태를 유지했습니다. 시간이 지남에 따라 cfDNA 양의 감소가 관찰되었으며, 이는 진단 시 가장 높은 수율을 보여주는 이전에 발표된 연구와 일치합니다. TFx는 또한 치료 전반에 걸쳐 감소했는데, 이는 시딩(seeding)의 해상도와 주요 망막 종양 용적(종양 용적량은 각 임상 시점의 주황색 막대 위에 표시되어 있음)의 감소를 반영합니다. 질병의 임상적 퇴행에서 예상했던 대로, 게놈 프로파일도 정상화되었습니다. 게놈 프로필에서 빨간색 선은 이득에 대한 임계값을 나타내고 파란색 선은 손실에 대한 임계값을 나타냅니다. 이 그림은 Xu, L. et al.21의 허가를 받아 재인쇄되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
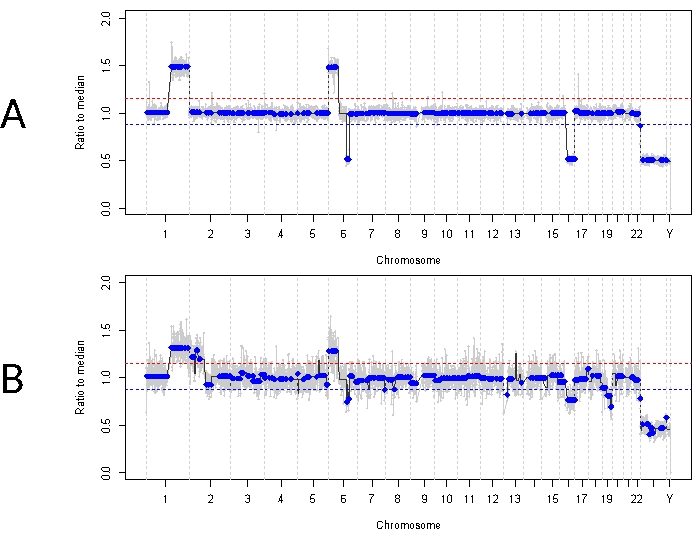
그림 4: 사례 33의 게놈 프로파일. 빨간색 선은 이득에 대한 임계값을 나타내고, 파란색 선은 사례 33에 대한 진단 시 손실(A) 게놈 프로파일에 대한 임계값을 나타냅니다. 진단 시 AH에서 초점 6q 손실과 더불어 고도로 재발하는 RB SCNAs 1q gain, 6p gain 및 16q loss가 확인되었습니다. 특히 6p gain의 진폭은 중앙값에 대한 1.5 비율로 예후가 좋지 않은 분자 바이오마커를 나타냅니다. 따라서 이 분자 시그니처를 기반으로 이 눈이 치료 실패 가능성을 크게 높였다는 것을 예측할 수 있습니다. (B) 진단 시 AH로부터 얻은 프로필과 매우 일치하는 적핵된 종양 조직에서 얻은 게놈 프로필. 정상 망막 조직과 혼합하기 때문에 종양 조직의 SCNA는 희석된 TFx로 인해 AH에 비해 더 낮은 진폭을 나타낼 수 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
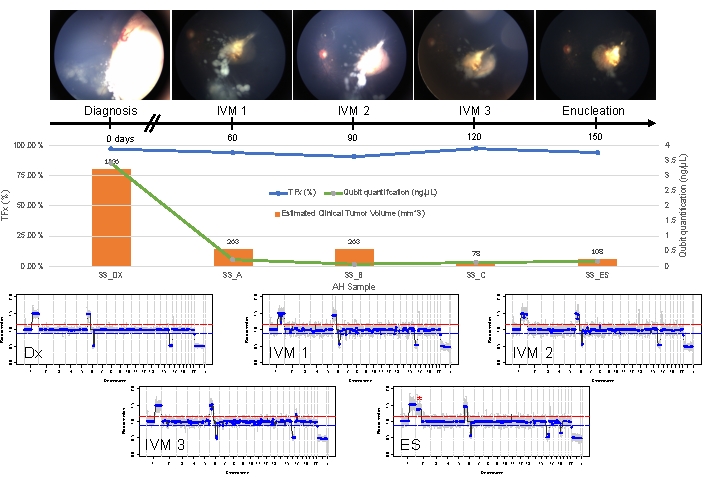
그림 5: 사례 33에 대한 종단 정보. 여기에는 안저 사진, cell-free DNA 정량화, B 스캔 측정에서 추정된 임상 종양 부피(종양 부피는 각 임상 시점의 주황색 막대 위에 표시됨), 게놈 프로필 및 AH가 샘플링된 각 임상 시점에 대한 TFx 추정(Dx = 진단, A = IVM1, B = IVM2, C = IVM3, SE = 2차 적출)이 포함됩니다. 이 눈은 치료에 반응하지 않아 결국 2차 적출술(ES)이 필요했습니다. 이것은 정점 종양 재발을 동반한 지속적으로 활동적인 파종 때문이었습니다. CfDNA의 양은 시간이 지남에 따라 감소했으며, 이는 진단 시 얻은 가장 높은 수율(1q gain, 6p gain, 16q loss 및 focal 6q loss)을 보여주는 이전에 발표된 연구와 일치합니다. 그러나 TFx는 치료 내내 높은 수준을 유지했는데, 이는 여전히 종양 유래 cfDNA를 AH로 흘려보내는 지속적인 파종의 산물입니다. 게놈 프로파일은 일관되었으며 진단 시 존재했던 것과 동일한 3개의 SCNA를 보여주었습니다. ES에서 얻은 AH에서 새로운 대규모 2p gain(*) 및 19q loss가 관찰되어 정점 종양 재발 시 클론 진화를 시사했습니다. 게놈 프로필에서 빨간색 선은 이득에 대한 임계값을 나타내고 파란색 선은 손실에 대한 임계값을 나타냅니다. 이 그림은 Xu, L. et al.21의 허가를 받아 재인쇄되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 파일 1: 예를 들면, (A) 피크가 약 300 bp인 경우 품질 관리를 통과해야 하는 샘플이고, (B) 약 150 bp의 피크로 품질 관리를 통과해서는 안 되는 샘플입니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
Clear corneal paracentesis는 안과에서 여러 진단 및/또는 치료 적응증을 위해 일반적으로 수행되는 절차입니다. 특히 RB의 경우, 주사 부위로의 역류를 방지하기 위해 주사 전에 안압을 낮추는 것이 표준 유리체강내 화학요법 주사 프로토콜의 일부입니다48. 일반적인 절차임에도 불구하고 위험이 전혀 없는 것은 아닙니다. 이전의 신조는 활동성 RB가 있는 눈에 바늘이 들어가서는 안 된다는 것이었습니다. 이 교리는 지난 10년 동안 발전해 왔는데, 처음에는 Patricia Chévez-Barrios의 주입 아데노바이러스 벡터 요법에 대한 형성 연구(formive work)를 통해49 그 다음에는 FrancisMunier48에 의한 RB의 안구 주사에 대한 점진적 안전성 강화 방법을 통해 발전했습니다. 이 연구는 AH 추출이 이 암에 대한 액체 생검 또는 종양 생검의 대리물로 사용될 수 있는 길을 열었습니다. 우리는 이제 플랫폼의 유용성과 진단 당시 취한 AH의 예비 안전 결과를 보고하는 첫 번째 센터입니다. 본 명세서에 기술된 방법의 일부는 RB에 대한 AH 추출의 환자 선택 및 절차적 측면의 안전성을 보장하기 위한 것입니다. 첫째, 바늘은 전방으로만 들어갈 수 있으며 홍채 흉터나 수정체에 닿으면 외과의가 종양을 모니터링할 수 있는 능력을 제한하는 홍채 흉터나 백내장을 유발할 수 있으므로 접촉해서는 안 됩니다. 바늘이 유리체강에 들어가지 않거나(IVM의 경우와 같이 화학요법 전달과 병용되지 않는 한) 종양과 접촉하지 않는 것이 가장 중요한데, 이는 종양 파종 및 질병의 안구 외 확장 위험을 가설적으로 증가시키기 때문입니다 3,4,5. 이 두 구조는 모두 전방의 뒤쪽에 있으며 수정체와 홍채에 의해 분리되어 있습니다. 안구 구조의 손상을 방지하려면 바늘이 항상 주변 홍채 위에 있고 수술용 현미경을 사용하여 항상 직접 시각화할 수 있도록 하는 것이 중요합니다. 바늘 부위에서 AH가 경미하게 누출될 위험이 있습니다. 이 경우 면 팁 어플리케이터를 통해 부드러운 압력으로 해결됩니다. 사용 가능한 가장 작은 게이지 바늘을 사용하고 측면 전단 없이 동일한 기관에서 바늘을 천천히 추출하면 이러한 위험을 줄일 수 있습니다. 전방의 약간 얕음이 예상되지만, 안방은 홍채 각막의 접촉 없이 형성된 상태를 유지해야 하며, 안압은 부드럽지만 생리학적이어야 합니다.
게놈 분석과 관련하여 몇 가지 중요한 단계가 있습니다. 가장 중요한 것은 눈에서 추출한 소량의 샘플을 처리하는 것입니다. 반복적인 동결 및 해동 주기로 발생할 수 있는 DNA 분해를 방지하기 위해 처리 전에 샘플이 전체 냉동 상태를 유지하는 것이 중요합니다50. 추출 직후 샘플을 드라이아이스에 넣고 -80°C 냉동고로 옮기면 이를 보장하는 데 도움이 됩니다. 시료 처리가 시작되면 품질 관리 단계는 cfDNA가 고품질 라이브러리로 구성되도록 보장하여 문제를 해결할 수 있는 주요 기회입니다. DNA 정량화 및 DNA 단편 크기 프로파일링 분석을 사용하면 이러한 샘플에 존재하는 cfDNA의 수율이 더 높기 때문에 1차 적출 중 또는 진단 시 채취한 샘플에 대해 품질 관리를 수행할 수 있습니다18,21. 얻어진 피크가 약 300bp일 때 이는 cfDNA가 NGS 플랫폼에 의해 인식될 것임을 보장합니다. 얻어진 피크가 150bp 미만인 경우, 이는 검출된 단편의 대다수가 라이브러리 구성 프라이머 또는 어댑터 올리고임을 나타내는 경우, 샘플이 손상된 것이므로 SCNA에 대해 더 이상 처리해서는 안 됩니다. 그러나 수백 개의 샘플을 처리한 경험에 비추어 볼 때, 낮은 판독 정렬 비율20으로 인해 품질 관리를 위해 제거해야 하는 시료는 5% 미만입니다. 검체 채취, 취급, 보관 및 처리를 위해 표준화한 방법과 함께 이 절차는 AH 액체 생검 플랫폼의 사전 분석 유효성을 보여줍니다.
AH 액체 생검은 또한 RB1 병원성 돌연변이 및 SCNA를 정확하고 신뢰할 수 있게 검출할 수 있는 능력을 기반으로 분석적 타당성을 확립했으며, AH 샘플에서 생성된 게놈 프로파일과 해당 종양 조직 간의 평균 일치율이 일관되게 >95%입니다 18,20,21,22,23. 플랫폼의 입증된 분석적 타당성에도 불구하고 제한이 없는 것은 아닙니다. RB1 돌연변이 분석은 >10ng의 cfDNA가 있는 샘플에 대해서만 수행할 수 있으며, 이는 진단 또는 1차 적출 시 치료 경험이 없는 눈에서 가장 자주 얻어집니다18,21. 이는 활발하게 치료를 받고 있는 눈의 AH 샘플에 존재하는 cfDNA의 농도가 진단 시 또는 1차 적출 시점의 AH에 비해 낮기 때문입니다. 또 다른 제약은 TFxs에서 SCNA가 5% 미만으로 검출될 수 없다는 것인데, 이는 종양 부담이 현저히 떨어진 눈의 질병 모니터링을 방해합니다. 본 연구의 경험에 비추어 볼 때, 이러한 현상은 유리체강내 화학요법 치료에 반응하는 눈에서 가장 흔하게 발생한다23.
AH에서 cfDNA의 TFx를 결정하기 위해 CNA 기반 TFx 추정 소프트웨어가 사용됩니다. 이 소프트웨어는 액체 생검에서 TFx 계산을 위한 표준적이고 승인된 도구이며 해당 알고리즘은자세히 설명되어 있습니다 38,51. 간단히 말해서, CNA 기반 TFx 추정 소프트웨어는 숨겨진 Markov 모델을 사용하여 염기서열 분석된 cfDNA 내에서 대규모 SCNA를 예측합니다. TFx 추정은 각 유전자 자리에서 subclonality 및 ploidy의 차이를 고려하면서 SCNA의 존재를 기반으로 도출되며, 이를 통해 CNA 기반 TFx 추정 소프트웨어는 최적의 TFx 솔루션을 선택합니다38. 그러나 CNA 기반 TFx 추정 소프트웨어의 고유한 한계는 TFx가 샘플에 SCNA가 있는지 기반으로 계산된다는 것입니다. 따라서 SCNA가 없는 샘플(즉, 평평한 게놈 프로필)에서 TFx를 측정할 수 없습니다38. 이전에 입증된 바와 같이, 모든 RB 종양이 SCNA 14,15,18,21,23,45를 가지고 있는 것은 아닙니다. 그러므로, CNA 기반 TFx 추정 소프트웨어에 의해 결정된 매우 낮은 TFx는 1) AH 샘플에 측정 가능한 종양 유래 cfDNA가 없거나 2) 종양 cfDNA가 존재하지만 SCNA가 부족하여 CNA 기반 TFx 추정 소프트웨어로 검출할 수 없음을 의미합니다38. SNV에 대한 변이 대립유전자 분율(VAF)은 TFx의 대용품입니다. 이러한 이유로 SCNA가 없는 RB eye도 종방향 TFx 모니터링을 받을 수 있도록 VAF를 기반으로 RB1 기반 TFx 파이프라인을 개발하기 위해 노력하고 있습니다. 원발성 MYCN 유발 종양 14,15,42,43,44,45를 제외한 모든 RB 종양이 RB1 유전자에 체세포 돌연변이를 포함하고 있다는 점을 감안할 때, SCNA에 의존하지 않는 파이프라인은 본 명세서에 제시된 액체 생검 프로토콜의 적용을 확대할 것입니다. 또한 SNV는 TFxs를 5% 미만으로 모니터링할 수 있기 때문에 플랫폼의 감도가 향상됩니다.
여기에 기술된 AH 액체 생검 플랫폼은 RB에 대한 문헌에 존재하는 유일한 액체 생검 플랫폼은 아니지만, 특히 종양 DNA의 농축 공급원으로 수성을 설명한 최초의 플랫폼이며, 현재까지 발표된 연구를 기반으로 모든 생체 유체에서 두 가지 메커니즘(SCNA 및 RB1 SNV)을 통한 ctDNA 검출을 설명한 최초의 플랫폼입니다. 두 가지 방법으로 ctDNA를 검출할 수 있는 AH 액체 생검은 문헌의 다른 플랫폼보다 민감도가 높습니다. 예를 들어, 다른 그룹은 RB1 SNVs(52)를 가진 AH에서 RB ctDNA를 성공적으로 검출하였다. 그러나 이 플랫폼은 SNV에 대한 선험적 지식을 기반으로 한 표적 NGS 판독에 의존했습니다. 이와는 대조적으로, 이 원고에 자세히 기술된 플랫폼은 비편향 WGS를 사용하여 SCNA 및 SNV를 검출할 수 있는 기능을 제공합니다. 혈액 기반 액체 생검도 시도되었지만, 얻은 cfDNA는 안구 구제 가능성에 대한 예후인 SCNA 검출 임계값 미만이었지만(현재 RB1 SNV는 안구 구제에 대한 예후가 있는 것으로 나타나지 않았습니다. 그러나 전이성 질환의 감지를 위한 역할이 진화하고 있을 수 있음)19. Kothari 등은 RB 환자의 혈장에서 RB1 SNV를 설명했지만, 적출이 필요한 진행성 안구 내 질환이 있는 환자만 해당한다고 설명했다16. 여기에 설명된 AH 액체 생검 플랫폼은 시딩(seeding)이 없어도 덜 발달된 눈의 AH에서 ctDNA를 검출할 수 있는 능력을 가지고 있습니다19,21. 또한, 혈액 기반 액체 생검은 양측 RB의 경우 혈청에서 분리된 ctDNA가 양쪽 눈에서 추출될 수 있기 때문에 눈에 특이적이지 않습니다. 이로 인해 특히 RB 사례의 40%를 차지하는 양측 환자에서 혈액 기반 플랫폼의 임상적 유용성이 제한되는 반면, AH는 특정 눈에 남아 SNV 및 SCNA 수치 모두에서 안간 이질성을 입증할 수 있습니다53.
RB 분야에 대한 AH 액체 생검의 중요성은 매우 중요합니다. AH 액체 생검은 적극적으로 치료를 받고 있는 눈의 종양 내 역학을 더 잘 이해할 수 있는 기회를 제공할 뿐만 아니라 환자 치료를 개선할 수 있는 잠재력도 있습니다. 이전 연구를 바탕으로 MYCN 증폭 또는 중앙값 대비 ≥1.5의 진폭으로 염색체 6p 증가의 존재를 기반으로 하는 분자 서명을 확립했으며, 이는 적출을 필요로 하는 치료 실패 가능성을 16.5배 증가시킬 수 있는 예후입니다 18,20,21. 진단 시점에 이러한 지식을 가지고 있다면, 임상의들은 적절한 치료 옵션과 현재 치료법으로 안구를 구할 가능성에 대해 가족들에게 더 잘 상담할 수 있을 것입니다. RB에 대한 AH 액체 생검 플랫폼의 임상적 타당성은 확립되었지만, 17,18,19,20,21,22,23, 현재는 연구용으로만 승인되었습니다. RB에 대한 직접적인 환자 치료를 돕기 위해 AH 액체 생검을 임상적으로 시행하기 전에 더 큰 규모의 전향적 다기관 연구가 필요합니다. 그럼에도 불구하고, AH 액체 생검은 미래에 RB뿐만 아니라 다른 안구 종양에 대해서도 정밀 종양학을 가능하게 할 수 있는 잠재력을 가지고 있습니다.
공개
제시 베리(Jesse Berry), 리야 수(Liya Xu), 제임스 힉스(James Hicks)는 안과 질환의 진단 및 예후 평가를 위한 수액 무세포 DNA라는 제목의 특허 출원을 제출했습니다. 그렇지 않다면, 저자는 잠재적인 이해 상충을 보고하지 않는다.
감사의 말
이 연구는 다음과 같은 출처의 지원을 받았습니다 : NIH Award K08CA232344의 NCI (J. L. Berry에게); Hyundai Hope on Wheels RGA012351 (J. L. Berry에게); 소아 안구암 신탁(J.L. Berry에게); 미국 암 학회 IRG-16-181-57 (J. L. Berry에게); 라이트 재단 (J.L. Berry 및 M.E. Kim에게); Knights Templar Eye Foundation (J.L. Berry에게); Larry and Celia Moh 재단 (J. L. Berry에게); The Institute for Families, Inc., 로스 앤젤레스 아동 병원 (JL Berry); 실명 방지를 위한 연구(Research to Prevent Blindness)의 무제한 부서 보조금(모두); NCI P30CA014089(모두); Vicky Joseph Research Fund (P. Kuhn에게); Carol Vassiliadis 연구 기금 (P. Kuhn에게); USC Dornsife College of Letters, Arts and Sciences (P. Kuhn에게).
자료
| Name | Company | Catalog Number | Comments |
| 1 cc syringe | surgical grade, whatever available in hospital | ||
| 32 G needle | surgical grade, whatever available in hospital | ||
| Aligner | Authors use Bowtie2 (http://bowtie-bio.sourceforge.net/bowtie2/index.shtml) but other aligners such as BWA or GRCh38 will also work | ||
| Atropos | generic term: adapter remover. https://atropos.readthedocs.io/en/latest/index.html# | ||
| Bioanalyzer High Sensitivity DNA Kit | Agilent | 5067-4626 | generic term: DNA fragment size profiling assay |
| BWA-MEM | generic term: long sequence aligner. http://bio-bwa.sourceforge.net/bwa.shtml | ||
| DNAcopy | Bioconductor | generic term: DNA copy number data analysis. https://bioconductor.org/packages/release/bioc/html/DNAcopy.html | |
| dsDNA High Sensitivity Assay | Qubit | Q32851 | generic term: DNA quantification assay |
| FreeBayes | generic term: sequence variant determiner. https://github.com/freebayes/freebayes | ||
| ichorCNA software | generic term: CNA-based TFx estimation. https://github.com/broadinstitute/ichorCNA | ||
| Illumina platform | Illumina | generic term: NGS platform; please note that other NGS platforms will work in principle, but have not been trialed by these authors | |
| NovoAlign (v3) | Novocraft | generic term: mapping of short reads onto reference genome. http://www.novocraft.com/products/novoalign/ | |
| QIAamp Circulating Nucleic Acid Kit | Qiagen | 55114 | generic term: cfDNA isolation and purification kit |
| QIAseq Ultralow Input Library Kit | Qiagen | 180492 | generic term: DNA library sequencing kit |
| Samtools rmdup | generic term: tool to remove duplicate reads. http://www.htslib.org/doc/samtools-rmdup.html | ||
| VarDict | generic term: variant caller. https://github.com/AstraZeneca-NGS/VarDict | ||
| Variant Effect Predictor | Ensembl | generic term: variant effect determinator. https://uswest.ensembl.org/info/docs/tools/vep/index.html |
참고문헌
- Fernandes, A. G., Pollock, B. D., Rabito, F. A. Retinoblastoma in the United States: A 40-year incidence and survival analysis. Journal of Pediatric Ophthalmology & Strabismus. 55 (3), 182-188 (2018).
- Linn, A. M. Intraocular retinoblastoma: the case for a new group classification. Ophthalmology Clinics of North America. 18 (1), 41-53 (2005).
- Shields, J. A., Shields, C. L., Ehya, H., Eagle, R. C., De Potter, P. Fine-needle aspiration biopsy of suspected intraocular tumors. The 1992 Urwick lecture. Ophthalmology. 100 (11), 1677-1684 (1993).
- Karcioglu, Z. A., Gordon, R. A., Karcioglu, G. L. Tumor seeding in ocular fine needle aspiration biopsy. Ophthalmology. 92 (12), 1763-1767 (1985).
- Karcioglu, Z. A. Fine needle aspiration biopsy (FNAB) for retinoblastoma. Retina. 22 (6), 707-710 (2002).
- Eide, N., Syrdalen, P., Walaas, L., Hagmar, B. Fine needle aspiration biopsy in selecting treatment for inconclusive intraocular disease. Acta Ophthalmologica Scandinavica. 77 (4), 448-452 (1999).
- Eide, N., Walaas, L. Fine-needle aspiration biopsy and other biopsies in suspected intraocular malignant disease: a review. Acta Ophthalmologica. 87 (6), 588-601 (2009).
- Eriksson, O., Hagmar, B., Ryd, W. Effects of fine-needle aspiration and other biopsy procedures on tumor dissemination in mice. Cancer. 54 (1), 73-78 (1984).
- Ali, M. J., Honavar, S. G., Vemuganti, G. K., Singh, A. D. Fine needle aspiration biopsy of retinal tumors. Monographs in Clinical Cytology. 21, 72-81 (2012).
- Di Nicolantonio, F. a. l., et al. The chemosensitivity profile of retinoblastoma. Recent Results in Cancer Research. 161, 73-80 (2003).
- Francis, J. H., et al. Efficacy and toxicity of second-course ophthalmic artery chemosurgery for retinoblastoma. Ophthalmology. 122 (5), 1016-1022 (2015).
- Francis, J. H. Efficacy and toxicity of intravitreous chemotherapy for retinoblastoma: Four-year experience. Ophthalmology. 124 (4), 488-495 (2017).
- Francis, J. H., et al. Current treatment of bilateral retinoblastoma: The impact of intraarterial and intravitreous chemotherapy. Neoplasia. 20 (8), 757-763 (2018).
- Rushlow, D. E., et al. Characterisation of retinoblastomas without RB1 mutations: genomic, gene expression, and clinical studies. The Lancet Oncology. 14 (4), 327-334 (2013).
- Afshar, A. R., et al. Next-generation sequencing of retinoblastoma identifies pathogenic alterations beyond RB1 inactivation that correlate with aggressive histopathologic features. Ophthalmology. 127 (6), 804-813 (2020).
- Kothari, P. Cell-free DNA profiling in retinoblastoma patients with advanced intraocular disease: An MSKCC experience. Cancer Medicine. 9 (17), 6093-6101 (2020).
- Berry, J. L., et al. Potential of aqueous humor as a surrogate tumor biopsy for retinoblastoma. JAMA Ophthalmology. 135 (11), 1221-1230 (2017).
- Berry, J. L., et al. Genomic cfDNA analysis of aqueous humor in retinoblastoma predicts eye salvage: the surrogate tumor biopsy for retinoblastoma. Molecular Cancer Research. 16 (11), 1701-1712 (2018).
- Berry, J. L., et al. Aqueous humor is superior to blood as a liquid biopsy for retinoblastoma. Ophthalmology. 127 (4), 552-554 (2020).
- Xu, L., et al. Chromosome 6p amplification in aqueous humor cell-free DNA Is a prognostic biomarker for retinoblastoma ocular survival. Molecular Cancer Research. 18 (8), 1166-1175 (2020).
- Xu, L., et al. Establishing the clinical utility of ctDNA analysis for diagnosis, prognosis, and treatment monitoring of retinoblastoma: The aqueous humor liquid biopsy. Cancers. 13 (6), (2021).
- Xu, L. Simultaneous identification of clinically relevant RB1 mutations and copy number alterations in aqueous humor of retinoblastoma eyes. Ophthalmic Genetics. 41 (6), 526-532 (2020).
- Polski, A., et al. Longitudinal aqueous humor sampling reflects treatment response in retinoblastoma patients. Investigative Ophthalmology & Visual Science. 61 (7), 1394 (2020).
- . Genome Browser Available from: https://genome.ucsc.edu/cgi-bin/hgTracks?db=hg19&lastVirtModeType=default&lastVirtModeExtraState=&virtModeType=default&3A15578261%_2D15621068&hgsid=1135765621_qIuA0U (2021)
- Navarro Gonzalez, J., et al. The UCSC genome browser database: 2021 update. Nucleic Acids Research. 49 (1), 1046-1057 (2021).
- Li, H. A statistical framework for SNP calling, mutation discovery, association mapping and population genetical parameter estimation from sequencing data. Bioinformatics. 27 (21), 2987-2993 (2011).
- Baslan, T., et al. Genome-wide copy number analysis of single cells. Nature Protocols. 7 (6), 1024-1041 (2012).
- Huber, W., et al. Orchestrating high-throughput genomic analysis with Bioconductor. Nature Methods. 12 (2), 115-121 (2015).
- Baslan, T., et al. Erratum: Genome-wide copy number analysis of single cells. Nature Protocols. 11 (3), 1024-1041 (2016).
- Didion, J. P., Martin, M., Collins, F. S. Atropos: specific, sensitive, and speedy trimming of sequencing reads. PeerJ. 5, 3720 (2017).
- Li, H. Aligning sequence reads, clone sequences and assembly contigs with BWA-MEM. arXiv. , (2013).
- Garrison, E., Marth, G. Haplotype-based variant detection from short-read sequencing. arXiv. , (2012).
- McLaren, W., et al. The ensembl variant effect predictor. Genome Biology. 17 (1), 122 (2016).
- Lai, Z., et al. VarDict: a novel and versatile variant caller for next-generation sequencing in cancer research. Nucleic Acids Research. 44 (11), 108 (2016).
- . GitHub Available from: https://github.com/broadinstitute/ichorCNA (2017)
- Adalsteinsson, V. A., et al. Scalable whole-exome sequencing of cell-free DNA reveals high concordance with metastatic tumors. Nature Communications. 8 (1), 1-13 (2017).
- . GitHub Available from: https://github.com/broadinstitute/ichorCNA/wiki/Interpreting-ichorCNA-results (2017)
- Berry, J. L., et al. Long-term outcomes of Group D retinoblastoma eyes during the intravitreal melphalan era. Pediatric Blood & Cancer. 64 (12), (2017).
- Berry, J. L., et al. Long-term outcomes of Group D eyes in bilateral retinoblastoma patients treated with chemoreduction and low-dose IMRT salvage. Pediatric Blood & Cancer. 60 (4), 688-693 (2013).
- Francis, J. H., et al. Molecular changes in retinoblastoma beyond RB1: Findings from next-generation sequencing. Cancers (Basel). 13 (1), (2021).
- Schwermer, M., et al. Comprehensive characterization of RB1 mutant and MYCN amplified retinoblastoma cell lines. Experimental Cell Research. 375 (2), 92-99 (2019).
- Davies, H. R. Whole-genome sequencing of retinoblastoma reveals the diversity of rearrangements disrupting RB1 and uncovers a treatment-related mutational signature. Cancers. 13 (4), 754 (2021).
- Lillington, D. M., et al. Comparative genomic hybridization of 49 primary retinoblastoma tumors identifies chromosomal regions associated with histopathology, progression, and patient outcome. Genes, Chromosomes and Cancer. 36 (2), 121-128 (2003).
- Greger, V., et al. Frequency and parental origin of hypermethylated RB1 alleles in retinoblastoma. Human Genetics. 94 (5), 491-496 (1994).
- Raizis, A. M., et al. DNA hypermethylation/boundary control loss identified in retinoblastomas associated with genetic and epigenetic inactivation of the RB1 gene promoter. Epigenetics. 1-15, (2020).
- Munier, F. L., et al. Profiling safety of intravitreal injections for retinoblastoma using an anti-reflux procedure and sterilisation of the needle track. British Journal of Ophthalmology. 96 (8), 1084-1087 (2012).
- Chévez-Barrios, P., et al. Response of retinoblastoma with vitreous tumor seeding to adenovirus-mediated delivery of thymidine kinase followed by ganciclovir. Journal of Clinical Oncology. 23 (31), 7927-7935 (2005).
- Shao, W., Khin, S., Kopp, W. C. Characterization of effect of repeated freeze and thaw cycles on stability of genomic DNA using pulsed field gel electrophoresis. Biopreservation and Biobanking. 10 (1), 4-11 (2012).
- Choudhury, A. D., et al. Tumor fraction in cell-free DNA as a biomarker in prostate cancer. JCI Insight. 3 (21), (2018).
- Gerrish, A., et al. Non-invasive diagnosis of retinoblastoma using cell-free DNA from aqueous humour. British Journal of Ophthalmology. 103 (5), 721-724 (2019).
- Wong, E. Y., Xu, L., Shen, L. Inter-eye genomic heterogeneity in bilateral retinoblastoma via aqueous humor liquid biopsy. NPJ Precis Oncol. 5 (1), 73 (2021).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유