このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
せん断力によるアンサンブル力分光法
要約
アンサンブル力分光法(EFS)は、生物物理学およびバイオセンシング分野における生体分子構造のアンサンブルセットの機械的アンフォールディングおよびリアルタイムセンシングのための堅牢な技術です。
要約
蛍光とメカノケミカルの原理に基づく単一分子技術は、生物学的センシングにおいて優れた感度を提供します。しかしながら、高スループット能力の欠如のために、これらの技術の適用は生物物理学において制限されている。アンサンブルフォース分光法(EFS)は、個々の分子のメカノケミカル研究を分子アンサンブルの研究に変換することにより、大量の分子構造の調査において高いスループットを実証しています。このプロトコルでは、DNA二次構造(i-motif)をホモジナイザーチップのローターとステーターの間のせん断流で最大77796/sのせん断速度で展開しました。i-motifが受けるせん断力に対する流量と分子サイズの影響が実証されました。EFS法はまた、DNA iモチーフとリガンドの間の結合親和性を明らかにした。さらに、せん断力によって作動できるクリック化学反応(メカノクリック化学)を実証しました。これらの結果は、せん断力を利用して分子構造の立体構造を制御することの有効性を確立しています。
概要
単一分子力分光法1(SMFS)では、原子間力顕微鏡、光ピンセット、磁気ピンセット2,3,4などの高度な機器によって、個々の分子構造の機械的特性が研究されてきました。力の生成/検出セットアップにおける分子の同じ指向性要件、または磁気ピンセットと小型遠心力顕微鏡(MCF)5、6、7、8の小さな視野によって制限されるため、SMFSを使用して同時に調査できる分子の数は限られています。SMFSのスループットが低いため、多数の分子の関与を必要とする分子認識分野での幅広い用途が妨げられています。
せん断流は、分子の大規模なセットに力を加えるための潜在的な解決策を提供します9。流路内部の液体流では、流路表面に近いほど、流量10は遅くなる。このような流速勾配は、境界面に平行なせん断応力を引き起こします。分子がこのせん断流に配置されると、せん断力が長軸11に加えられるため、分子はその長軸が流れ方向と整列するようにそれ自体を再配向する。この再配向の結果として、同じタイプ(ハンドルのサイズと長さ)のすべての分子は、同じせん断力を受けながら同じ方向に整列することが期待されます。
この研究では、このようなせん断流を使用して、DNA i-motifに例示されるように、大量の分子構造にせん断力を加えるプロトコルについて説明しています。このプロトコルでは、ホモジナイザーチップのローターとステーターの間にせん断流が発生します。本研究では、折り畳まれたDNA i-motif構造が9724-97245 s-1のせん断速度で展開できることを見出した。また、L2H2-4OTD配位子とiモチーフの間には36μMの解離定数が見られた。この値は、ゲルシフトアッセイ12によって測定された31μMの値と一致する。さらに、現在の技術を使用してiモチーフを展開し、キレート銅(I)を露出させてクリック反応を触媒することができます。したがって、このプロトコルにより、低コストの機器を使用して大量のi-motif構造を妥当な時間(30分未満)で展開することができます。せん断力法は力分光法のスループットを大幅に向上させることを考えると、この技術をアンサンブル力分光法(EFS)と呼びます。このプロトコルは、このせん断力ベースのEFSの適用を容易にするための実験ガイドラインを提供することを目的としています。
プロトコル
注:このプロトコルで使用されるすべてのバッファーと化学試薬は、 表の材料に記載されています。
1. せん断力顕微鏡の作製
注:せん断力顕微鏡には、反応ユニット(ホモジナイザー)と検出ユニット(蛍光顕微鏡)の2つの部分があります。接眼レンズの倍率は10倍、対物レンズ(空気)の倍率は4倍です。
- ホモジナイザーと顕微鏡を取り付けテーブルに組み立てます。ゴーグルを着用して蛍光顕微鏡の電源を入れ、ホモジナイザーを調整して、適切な波長(ここでは488nmを使用)の励起光ビームがホモジナイザーの分散先端の中心を通過することを確認します。
- 高さ5 cm、断面1.5 cm 2 x 1.5 cm2の平底反応チャンバーを準備します。バックグラウンドを減らすには、選択したチャンバー材料(ガラスなど)が蛍光を発しないことを確認してください(一部のプラスチックには蛍光があります)。
- 所望のせん断力を提供できる適切なホモジナイザー分散チップを選択してください。
注意: せん断速度は、次の式に従って、固定回転速度11 での回転子と固定子の間の距離に依存します。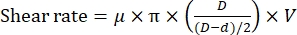
ここで 、μ は20°Cでの水の動的粘度です。 D は固定子の内径です。 d はローターの外径、 V はせん断速度(rpm / s)です。 - 反応チャンバーを蛍光顕微鏡の試料台にクランプし、ホモジナイザーの分散先端を保持するようにチャンバーを調整します(図1)。分散チップが反応チャンバーの底面より少し上(~1 mm)にあることを確認してください。
- ホモジナイザーとチャンバーの垂直位置を一緒に調整して、顕微鏡の焦点が分散チップの表面にあることを確認します。次に、分散チップの水平位置を調整して、ローターとステーターの間に検出領域(視野)が設定されていることを確認します(図1)。
- 実験に使用した蛍光色素に応じて蛍光チャンネルをオンにします。
- 高速せん断実験の前に、脱イオン(DI)水を使用して、低いせん断速度(2,000 rpmなど)でせん断をテストし、分散チップが反応チャンバーに触れることなく適切に機能することを確認します。
2. 配位子の有無にかかわらずiモチーフの展開
- Huら11に記載されているように、DI水中でそれぞれ色素とクエンチャーで標識されたヒトテロメアi-motif DNA(材料表)を準備します。
注:配列を含むI-モチーフ:5'-TAA CCC TAA CCC TAA CCC TAA CCC TAA CCC TAA. - pH 5.5またはpH 7.4の30 mM MESバッファーでDNAを5 μMに希釈します。DNA溶液に、Abraham Punnooseら13に従って合成したリガンドL2H2-4OTDを0-60 μMの濃度範囲で加えます。 溶液を10分間穏やかに混合して、光なしでi-モチーフ構造を折り畳みます。
- せん断せずに蛍光顕微鏡を使用して、脱イオンDI水で満たされた反応チャンバーのバックグラウンド蛍光強度を確認し、最小化します。バックグラウンド蛍光を最小限に抑える簡単な方法は、反応チャンバーをDI水で洗浄することです。バックグラウンド蛍光値は、後でデータ解析で差し引く必要があります。
注意: このステップ以降は迷光を避ける必要があります。 - ソフトウェアを使用してCCDカメラのパラメータを設定します。推奨パラメータは、露光時間=0.5秒、CCD感度=1600、記録時間=20分です。
- 長いピペットを使用して、DNA溶液を空の清潔な反応チャンバーに加えます。反応室をブラックボックスで覆います。次に、ホモジナイザーを起動して、CCDカメラの電源を入れた状態で、9,724 s−1から97,245 s−1(ホモジナイザーに関連付けられたソフトウェアを使用して選択)の範囲の選択されたせん断速度で20分間せん断を実行しますデータを記録します。
- 実験後、チャンバーを取り外し、DI水で洗浄します。
3. せん断力作動クリック反作用
- DI水中でi-モチーフDNAを調製します。150 μM CuClと300 μMアスコルビン酸を添加した300 μLの30 mM Trisバッファー(pH 7.4)中で10 μM i-モチーフDNAを10分間インキュベートし、i-モチーフ構造をフォールディングします(すべての濃度は溶液中の最終濃度です)。
注:CuClはクリック反応の触媒です。アスコルビン酸は銅(I)の酸化を防ぎます。 - 限外ろ過装置で14,300 x gの遠心力で溶液を限外ろ過します。各ろ過後に、300 μMアスコルビン酸を添加した30 mMトリス緩衝液(pH 7.4)を~500 μLに補充します。
- ろ過を3回繰り返します。
- 残留溶液を回収し、300 μMアスコルビン酸を添加した30 mMトリス(pH 7.4)と20 μMカルフルオール488アジ化物、20 μM HPG、および10 μM TBTAを加えて、最終容量300 μLにします。試薬を追加したら、溶液を暗室に移動します。
注意: この手順の後は、光を避けてください。 - せん断実験の前に、顕微鏡を使用してDI水で満たされた反応チャンバーのバックグラウンド蛍光強度を確認し、最小限に抑えます。バックグラウンド蛍光を最小限に抑える簡単な方法は、反応チャンバーをDI水で洗浄することです。
- DNA溶液を長いピペットで空の反応チャンバーに加え、CCDカメラの電源を入れた状態で、せん断速度63,209 s-1 で20分間ホモジナイザーせん断を開始します。
- 実験後、チャンバーを取り外し、DI水で洗浄します。
結果
図1は、EFSにおけるアンサンブル分子の機械的アンフォールディングとリアルタイムセンシングの概要を示しています。図1Bでは、i-motif DNAの蛍光強度は、pH 5.5 MESバッファー中で9,724 s−1から97,245 s−1の範囲のせん断速度とともに増加することが観察された。対照として、同じi-motif DNAをpH 7.4 MES緩衝液中で63,209 s−1
ディスカッション
この原稿に記載されているプロトコルは、せん断力による生体分子構造のアンサンブルセットの展開のリアルタイム調査を可能にします。ここで提示された結果は、DNA i-モチーフ構造がせん断力によって展開できることを強調しています。リガンド結合iモチーフの展開とせん断力作動クリック反応は、このアンサンブル力分光法の概念実証アプリケーションでした。
開示事項
著者には利益相反はありません。
謝辞
この研究成果は、米国国立科学財団 [CBET-1904921] および国立衛生研究所 [NIH R01CA236350] の支援を受けました。
資料
| Name | Company | Catalog Number | Comments |
| 3K MWCO Amicon | Millipore Sigma | ufc900324 | |
| Ascorbic acid | VWR | VWRC0143-100G | |
| Calfluor 488 azide | Click Chemistry Tools | 1369-1 | |
| CuCl | Thermo | ACRO270525000 | |
| Dispersion tip | Switzerland | PT-DA07/2EC-B101 | |
| DNA oligos | IDT | ||
| Dye | IDT | /5Cy5/ | |
| Fluorescence microscope | Janpan | Nikon TE2000-U | |
| Homogenizer | Switzerland | PT 3100D | |
| HPG | Santa Cruz Biotechnology | cs-295271 | |
| KCl | VWR | VWRC26760.295 | |
| MES | VWR | VWRCE169-500G | |
| Quencher | IDT | /3IAbRQSp/ | |
| TBTA | Tokyo Chemical Industry | T2993 | |
| Tris | VWR | VWRCE133-100G |
参考文献
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: Optical tweezers, magnetic tweezers and atomic force microscopy. Nature Methods. 5 (6), 491-505 (2008).
- Woodside, M. T., et al. Nanomechanical measurements of the sequence-dependent folding landscapes of single nucleic acid hairpins. Proceedings of the National Academy of Sciences of the United States of America. 103 (16), 6190-6195 (2006).
- Grandbois, M., Beyer, M., Rief, M., Clausen-Schaumann, H., Gaub, H. E. How strong is a covalent bond. Science. 283 (5408), 1727-1730 (1999).
- Strick, T. R., Allemand, J. F., Bensimon, D., Croquette, V. Behavior of supercoiled DNA. Biophysical Journal. 74 (4), 2016-2028 (1998).
- Yang, D., Ward, A., Halvorsen, K., Wong, W. P. Multiplexed single-molecule force spectroscopy using a centrifuge. Nature Communications. 7, 11026 (2016).
- Su, H., et al. Light-responsive polymer particles as force clamps for the mechanical unfolding of target molecules. Nano Letters. 18 (4), 2630-2636 (2018).
- Kirkness, M. W. H., Forde, N. R. Single-molecule assay for proteolytic susceptibility: Force-induced collagen destabilization. Biophysical Journal. 114 (3), 570-576 (2018).
- Astumian, R. D. Thermodynamics and kinetics of molecular motors. Biophysical Journal. 98 (11), 2401-2409 (2010).
- Bekard, I. B., Asimakis, P., Bertolini, J., Dunstan, D. E. The effects of shear flow on protein structure and function. Biopolymers. 95 (11), 733-745 (2011).
- Chistiakov, D. A., Orekhov, A. N., Bobryshev, Y. V. Effects of shear stress on endothelial cells: go with the flow. Acta Physiologica. 219 (2), 382-408 (2017).
- Hu, C., Jonchhe, S., Pokhrel, P., Karna, D., Mao, H. Mechanical unfolding of ensemble biomolecular structures by shear force. Chemical Science. 12 (30), 10159-10164 (2021).
- Sedghi Masoud, S., et al. Analysis of interactions between telomeric i-motif DNA and a cyclic tetraoxazole compound. ChemBioChem. 19 (21), 2268-2272 (2018).
- Abraham Punnoose, J., et al. Adaptive and specific recognition of telomeric G-quadruplexes via polyvalency induced unstacking of binding units. Journal of the American Chemical Society. 139 (22), 7476-7484 (2017).
- Dhakal, S., et al. Coexistence of an ILPR i-motif and a partially folded structure with comparable mechanical stability revealed at the single-molecule level. Journal of the American Chemical Society. 132 (26), 8991-8997 (2010).
- Hu, C., Tahir, R., Mao, H. Single-molecule mechanochemical sensing. Accounts of Chemical Research. 55 (9), 1214-1225 (2022).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved