Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Ансамблевая силовая спектроскопия по силам сдвига
В этой статье
Резюме
Ансамблевая силовая спектроскопия (EFS) является надежным методом механического разворачивания и зондирования в реальном времени ансамблевого набора биомолекулярных структур в биофизических и биосенсорных полях.
Аннотация
Одномолекулярные методы, основанные на флуоресцентных и механохимических принципах, обеспечивают превосходную чувствительность в биологическом зондировании. Однако из-за отсутствия высоких пропускных возможностей применение этих методов ограничено в биофизике. Ансамблевая силовая спектроскопия (EFS) продемонстрировала высокую пропускную способность при исследовании массивного набора молекулярных структур путем преобразования механохимических исследований отдельных молекул в исследования молекулярных ансамблей. В этом протоколе вторичные структуры ДНК (i-мотивы) разворачивались в сдвиговом потоке между ротором и статором наконечника гомогенизатора со скоростью сдвига до 77796/с. Было продемонстрировано влияние скорости потока и размеров молекул на силы сдвига, испытываемые i-мотивом. Методика EFS также выявила сродство связывания между i-мотивами ДНК и лигандами. Кроме того, мы продемонстрировали химическую реакцию щелчка, которая может быть приведена в действие силой сдвига (т. Е. Химия механо-щелчка). Эти результаты устанавливают эффективность использования силы сдвига для контроля конформации молекулярных структур.
Введение
В одномолекулярной силовой спектроскопии1 (SMFS) механические свойства отдельных молекулярных структур были изучены сложными инструментами, такими как атомно-силовой микроскоп, оптический пинцет и магнитный пинцет 2,3,4. Ограниченные одним и тем же требованием направленности молекул в силообразующих/детектирующих установках или малым полем зрения в магнитных пинцетах и миниатюрном центрифужном силовом микроскопе (MCF)5,6,7,8, только ограниченное количество молекул может быть исследовано одновременно с использованием SMFS. Низкая пропускная способность SMFS препятствует ее широкому применению в области молекулярного распознавания, что требует привлечения большого набора молекул.
Сдвиговой поток обеспечивает потенциальное решение для приложения сил к массивному набору молекул9. В потоке жидкости внутри канала, чем ближе к поверхности канала, тем медленнее скорость потока10. Такой градиент скорости потока вызывает напряжение сдвига, параллельное граничной поверхности. Когда молекула помещается в этот поток сдвига, молекула переориентируется так, что ее длинная ось выравнивается с направлением потока, поскольку сила сдвига прикладывается к длинной оси11. В результате этой переориентации ожидается, что все молекулы одного типа (размер и длина ручек) выровняются в одном направлении, испытывая при этом одинаковую силу сдвига.
Эта работа описывает протокол использования такого сдвигового потока для оказания силы сдвига на массивный набор молекулярных структур, примером чего является i-мотив ДНК. В этом протоколе между ротором и статором в наконечнике гомогенизатора генерируется сдвиговой поток. Настоящее исследование показало, что сложенная структура i-мотива ДНК может быть развернута со скоростью сдвига 9724-97245 s−1. Кроме того, между лигандом L2H2-4OTD и i-мотивом была обнаружена константа диссоциации 36 мкМ. Это значение согласуется с значением 31 мкМ, измеренным анализом сдвигагеля 12. Кроме того, современная техника используется для развертывания i-мотива, который может обнажить хелатную медь (I) для катализации реакции щелчка. Таким образом, этот протокол позволяет развернуть большой набор структур i-motif с недорогими инструментами за разумное время (менее 30 минут). Учитывая, что метод силы сдвига резко увеличивает пропускную способность силовой спектроскопии, мы называем эту технику ансамблевой силовой спектроскопией (EFS). Этот протокол направлен на предоставление экспериментальных руководящих принципов для облегчения применения этой EFS на основе сдвиговой силы.
протокол
ПРИМЕЧАНИЕ: Все буферы и химические реагенты, используемые в этом протоколе, перечислены в таблице материалов.
1. Подготовка силового микроскопа сдвига
ПРИМЕЧАНИЕ: Микроскоп с силой сдвига состоит из двух частей: реакционного блока (гомогенизатора) и блока детектирования (флуоресцентного микроскопа). Увеличение окуляра составляет 10x, а увеличение объектива (воздуха) - 4x.
- Соберите гомогенизатор и микроскоп на монтажном столе. Наденьте очки и включите флуоресцентный микроскоп, а затем отрегулируйте гомогенизатор, чтобы убедиться, что луч света возбуждения соответствующей длины волны (здесь использовался 488 нм) проходит через центр диспергирующего наконечника гомогенизатора.
- Подготовьте реакционную камеру с плоским дном высотой 5 см и поперечным сечением 1,5 см2 х 1,5см2. Чтобы уменьшить фон, убедитесь, что выбранный материал камеры не флуоресцирует, например, стекла (некоторые пластмассы имеют флуоресценцию).
- Выберите подходящий диспергирующий наконечник гомогенизатора, который может обеспечить требуемую силу сдвига.
ПРИМЕЧАНИЕ: Скорость сдвига зависит от расстояния между ротором и статором при фиксированной скорости вращения11 в соответствии со следующим уравнением: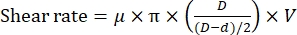
где μ - динамическая вязкость воды при 20°С; D - внутренний диаметр статора; d - наружный диаметр ротора, а V - скорость сдвига (об/мин/с). - Зажмите реакционную камеру на стадии образца флуоресцентного микроскопа, а затем отрегулируйте камеру, чтобы удерживать диспергирующий наконечник гомогенизатора (рисунок 1). Убедитесь, что диспергирующий наконечник находится немного выше (~1 мм) нижней поверхности реакционной камеры.
- Отрегулируйте вертикальное положение гомогенизатора и камеры вместе, чтобы фокус микроскопа находился на поверхности диспергирующего наконечника. Затем отрегулируйте горизонтальное положение диспергирующего наконечника, чтобы убедиться, что область обнаружения (поле зрения) установлена между ротором и статором (рисунок 1).
- Включите флуоресцентные каналы в соответствии с флуоресцентным красителем, используемым в эксперименте.
- Перед экспериментом по высокоскоростному сдвигу используйте деионизированную (DI) воду для проверки сдвига с низкой скоростью сдвига (например, 2000 оборотов в минуту), чтобы убедиться, что диспергирующий наконечник может работать надлежащим образом, не касаясь реакционной камеры.
2. Разворачивание i-мотивов с лигандами и без них
- Получают человеческую теломерную i-мотивную ДНК (Таблицу материалов), помеченную красителем и гасителем на двух концах, соответственно, в воде DI, как описано в Hu et al.11.
ПРИМЕЧАНИЕ: i-мотив, содержащий последовательность: 5'-TAA CCC TAA CCC TAA CCC TAA CCC CCC TAA. - Разбавьте ДНК до 5 мкМ в буфере MES 30 мМ при рН 5,5 или рН 7,4. К раствору ДНК добавляют лиганд L2H2-4OTD, который был синтезирован по данным Abraham Punnoose et al.13, в диапазоне концентраций 0-60 мкМ. Смешивайте раствор осторожно в течение 10 мин, чтобы сложить структуры i-мотива без света.
- Проверьте и минимизируйте интенсивность фоновой флуоресценции реакционной камеры, заполненной деионизированной водой DI, с помощью флуоресцентного микроскопа без сдвига. Простым способом минимизации фоновой флуоресценции является промывка реакционной камеры водой DI. Значение фоновой флуоресценции должно быть вычтено в анализе данных позже.
ПРИМЕЧАНИЕ: С этого шага следует избегать рассеянного света. - Установите параметры ПЗС-камеры с помощью программного обеспечения. Рекомендуемые параметры следующие: время экспозиции = 0,5 с, чувствительность ПЗС = 1600 и время записи = 20 мин.
- Используя длинную пипетку, добавьте раствор ДНК в пустую и чистую реакционную камеру. Накройте реакционную камеру черным ящиком. Затем запустите гомогенизатор для выполнения сдвига с выбранной скоростью сдвига в диапазоне от 9 724 с−1 до 97 245 с−1 (выбранного с помощью программного обеспечения, связанного с гомогенизатором) в течение 20 минут с включенной ПЗС-камерой для записи данных.
- После эксперимента снимите камеру и промыть ее водой DI.
3. Реакция щелчка с силой сдвига
- Подготовьте ДНК i-мотива в воде DI. Инкубировать 10 мкМ i-мотивной ДНК в 300 мкл 30 мМ буфера Tris (рН 7,4), дополненного 150 мкМ CuCl и 300 мкМ аскорбиновой кислоты в течение 10 мин для сворачивания структур i-мотива (все концентрации являются конечными концентрациями в растворе).
ПРИМЕЧАНИЕ: CuCl является катализатором реакции щелчка. Аскорбиновая кислота предотвратит окисление меди (I). - Ультрафильтрация раствора с помощью ультрафильтрационного устройства при центробежной силе 14 300 х г. Пополняйте раствор до ~500 мкл с буфером Tris 30 мМ (рН 7,4), дополненным 300 мкМ аскорбиновой кислотой после каждой фильтрации.
- Повторите фильтрацию 3x.
- Соберите остаточный раствор и сделайте конечный объем 300 мкл, добавив 30 мМ Tris (pH 7,4), дополненный 300 мкМ аскорбиновой кислотой вместе с 20 мкМ Calfluor 488 азида, 20 мкМ HPG и 10 мкМ TBTA. После того, как реагенты добавлены, переместите раствор в темную комнату.
ПРИМЕЧАНИЕ: После этого шага следует избегать освещения. - Проверьте и минимизируйте интенсивность фоновой флуоресценции реакционной камеры, заполненной водой DI, с помощью микроскопа перед экспериментами по сдвигу. Простым способом минимизации фоновой флуоресценции является промывка реакционной камеры водой DI.
- Добавьте раствор ДНК в пустую реакционную камеру с длинной пипеткой, а затем начните сдвиг гомогенизатора со скоростью сдвига 63 209 с−1 в течение 20 мин с включенной ПЗС-камерой.
- После эксперимента снимите камеру и промыть ее водой DI.
Результаты
На рисунке 1 показано механическое развертывание и зондирование в реальном времени ансамблевых молекул в EFS. На рисунке 1B интенсивность флуоресценции ДНК i-мотива увеличивалась со скоростью сдвига в диапазоне от 9 724 с−1 до 97 245 с−1 в б...
Обсуждение
Протокол, описанный в этой рукописи, позволяет в режиме реального времени исследовать разворачивание ансамблевого набора биомолекулярных структур силой сдвига. Представленные здесь результаты подчеркивают, что структуры ДНК i-motif могут быть развернуты силой сдвига. Развертывание лиг...
Раскрытие информации
У авторов нет конфликта интересов.
Благодарности
Эта исследовательская работа была поддержана Национальным научным фондом [CBET-1904921] и Национальными институтами здравоохранения [NIH R01CA236350] для H. M.
Материалы
| Name | Company | Catalog Number | Comments |
| 3K MWCO Amicon | Millipore Sigma | ufc900324 | |
| Ascorbic acid | VWR | VWRC0143-100G | |
| Calfluor 488 azide | Click Chemistry Tools | 1369-1 | |
| CuCl | Thermo | ACRO270525000 | |
| Dispersion tip | Switzerland | PT-DA07/2EC-B101 | |
| DNA oligos | IDT | ||
| Dye | IDT | /5Cy5/ | |
| Fluorescence microscope | Janpan | Nikon TE2000-U | |
| Homogenizer | Switzerland | PT 3100D | |
| HPG | Santa Cruz Biotechnology | cs-295271 | |
| KCl | VWR | VWRC26760.295 | |
| MES | VWR | VWRCE169-500G | |
| Quencher | IDT | /3IAbRQSp/ | |
| TBTA | Tokyo Chemical Industry | T2993 | |
| Tris | VWR | VWRCE133-100G |
Ссылки
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: Optical tweezers, magnetic tweezers and atomic force microscopy. Nature Methods. 5 (6), 491-505 (2008).
- Woodside, M. T., et al. Nanomechanical measurements of the sequence-dependent folding landscapes of single nucleic acid hairpins. Proceedings of the National Academy of Sciences of the United States of America. 103 (16), 6190-6195 (2006).
- Grandbois, M., Beyer, M., Rief, M., Clausen-Schaumann, H., Gaub, H. E. How strong is a covalent bond. Science. 283 (5408), 1727-1730 (1999).
- Strick, T. R., Allemand, J. F., Bensimon, D., Croquette, V. Behavior of supercoiled DNA. Biophysical Journal. 74 (4), 2016-2028 (1998).
- Yang, D., Ward, A., Halvorsen, K., Wong, W. P. Multiplexed single-molecule force spectroscopy using a centrifuge. Nature Communications. 7, 11026 (2016).
- Su, H., et al. Light-responsive polymer particles as force clamps for the mechanical unfolding of target molecules. Nano Letters. 18 (4), 2630-2636 (2018).
- Kirkness, M. W. H., Forde, N. R. Single-molecule assay for proteolytic susceptibility: Force-induced collagen destabilization. Biophysical Journal. 114 (3), 570-576 (2018).
- Astumian, R. D. Thermodynamics and kinetics of molecular motors. Biophysical Journal. 98 (11), 2401-2409 (2010).
- Bekard, I. B., Asimakis, P., Bertolini, J., Dunstan, D. E. The effects of shear flow on protein structure and function. Biopolymers. 95 (11), 733-745 (2011).
- Chistiakov, D. A., Orekhov, A. N., Bobryshev, Y. V. Effects of shear stress on endothelial cells: go with the flow. Acta Physiologica. 219 (2), 382-408 (2017).
- Hu, C., Jonchhe, S., Pokhrel, P., Karna, D., Mao, H. Mechanical unfolding of ensemble biomolecular structures by shear force. Chemical Science. 12 (30), 10159-10164 (2021).
- Sedghi Masoud, S., et al. Analysis of interactions between telomeric i-motif DNA and a cyclic tetraoxazole compound. ChemBioChem. 19 (21), 2268-2272 (2018).
- Abraham Punnoose, J., et al. Adaptive and specific recognition of telomeric G-quadruplexes via polyvalency induced unstacking of binding units. Journal of the American Chemical Society. 139 (22), 7476-7484 (2017).
- Dhakal, S., et al. Coexistence of an ILPR i-motif and a partially folded structure with comparable mechanical stability revealed at the single-molecule level. Journal of the American Chemical Society. 132 (26), 8991-8997 (2010).
- Hu, C., Tahir, R., Mao, H. Single-molecule mechanochemical sensing. Accounts of Chemical Research. 55 (9), 1214-1225 (2022).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены