Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Espectroscopía de fuerza de conjunto por fuerzas de cizallamiento
En este artículo
Resumen
La espectroscopia de fuerza de conjunto (EFS) es una técnica robusta para el despliegue mecánico y la detección en tiempo real de un conjunto de estructuras biomoleculares en campos biofísicos y biosensibles.
Resumen
Las técnicas de molécula única basadas en principios de fluorescencia y mecanoquímicos proporcionan una sensibilidad superior en la detección biológica. Sin embargo, debido a la falta de capacidades de alto rendimiento, la aplicación de estas técnicas es limitada en biofísica. La espectroscopia de fuerza de conjunto (EFS) ha demostrado un alto rendimiento en la investigación de un conjunto masivo de estructuras moleculares mediante la conversión de estudios mecanoquímicos de moléculas individuales en los de conjuntos moleculares. En este protocolo, las estructuras secundarias de ADN (i-motivos) se desplegaron en el flujo de corte entre el rotor y el estator de una punta homogeneizadora a velocidades de cizallamiento de hasta 77796 / s. Se demostraron los efectos de las tasas de flujo y los tamaños moleculares sobre las fuerzas de cizallamiento experimentadas por el i-motivo. La técnica EFS también reveló la afinidad de unión entre los i-motivos de ADN y los ligandos. Además, hemos demostrado una reacción química de clic que puede ser accionada por fuerza de cizallamiento (es decir, química de clic mecánico). Estos resultados establecen la efectividad del uso de la fuerza de cizallamiento para controlar la conformación de estructuras moleculares.
Introducción
En la espectroscopia de fuerza de molécula única1 (SMFS), las propiedades mecánicas de estructuras moleculares individuales han sido estudiadas por instrumentos sofisticados como el microscopio de fuerza atómica, pinzas ópticas y pinzas magnéticas 2,3,4. Restringido por el mismo requisito de direccionalidad de las moléculas en las configuraciones generadoras / de detección de fuerza o el pequeño campo de visión en pinzas magnéticas y el microscopio de fuerza centrífuga en miniatura (MCF)5,6,7,8, solo se puede investigar simultáneamente un número limitado de moléculas utilizando SMFS. El bajo rendimiento de SMFS impide su amplia aplicación en el campo del reconocimiento molecular, que requiere la participación de un gran conjunto de moléculas.
El flujo de cizallamiento proporciona una solución potencial para aplicar fuerzas a un conjunto masivo de moléculas9. En un flujo de líquido dentro de un canal, cuanto más cerca de la superficie del canal, más lento será el caudal10. Tal gradiente de velocidad de flujo causa un esfuerzo cortante que es paralelo a la superficie límite. Cuando una molécula se coloca en este flujo de corte, la molécula se reorienta para que su eje largo se alinee con la dirección del flujo, ya que la fuerza de cizallamiento se aplica al eje largo11. Como resultado de esta reorientación, se espera que todas las moléculas del mismo tipo (tamaño y longitud de los mangos) se alineen en la misma dirección mientras experimentan la misma fuerza de corte.
Este trabajo describe un protocolo para usar dicho flujo de cizallamiento para ejercer fuerza de cizallamiento sobre un conjunto masivo de estructuras moleculares, como lo ejemplifica el motivo i del ADN. En este protocolo, se genera un flujo de corte entre el rotor y el estator en una punta homogeneizadora. El presente estudio encontró que la estructura plegada del i-motivo del ADN podría desplegarse mediante velocidades de cizallamiento de 9724-97245 s−1. Además, se encontró una constante de disociación de 36 μM entre el ligando L2H2-4OTD y el motivo i. Este valor es consistente con el de 31 μM medido por el ensayo de desplazamiento de gel12. Además, la técnica actual se utiliza para desplegar el i-motivo, que puede exponer el cobre quelado (I) para catalizar una reacción de clic. Por lo tanto, este protocolo permite desplegar un gran conjunto de estructuras i-motif con instrumentos de bajo costo en un tiempo razonable (menos de 30 min). Dado que la técnica de fuerza de cizallamiento aumenta drásticamente el rendimiento de la espectroscopia de fuerza, llamamos a esta técnica espectroscopía de fuerza de conjunto (EFS). Este protocolo tiene como objetivo proporcionar directrices experimentales para facilitar la aplicación de este EFS basado en la fuerza de corte.
Protocolo
NOTA: Todos los tampones y los reactivos químicos utilizados en este protocolo se enumeran en la tabla Materiales.
1. Preparación del microscopio de fuerza de cizallamiento
NOTA: El microscopio de fuerza de cizallamiento contiene dos partes, una unidad de reacción (homogeneizador) y una unidad de detección (microscopio de fluorescencia). El aumento del ocular es 10x, y el aumento de la lente del objetivo (aire) es 4x.
- Ensamble el homogeneizador y el microscopio en una mesa de montaje. Use gafas y encienda el microscopio de fluorescencia, y luego ajuste el homogeneizador para asegurarse de que el haz de luz de excitación de una longitud de onda apropiada (aquí, se usó 488 nm) pase a través del centro de la punta dispersa del homogeneizador.
- Prepare una cámara de reacción de fondo plano de 5 cm de altura y 1,5 cm 2 x 1,5 cm2 de sección transversal. Para reducir el fondo, asegúrese de que el material de la cámara seleccionado no emita fluorescencia, como los vidrios (algunos plásticos tienen fluorescencia).
- Elija una punta de dispersión homogeneizadora adecuada que pueda proporcionar la fuerza de cizallamiento deseada.
NOTA: La velocidad de cizallamiento depende de la distancia entre el rotor y el estator a una velocidad de rotación fija11 de acuerdo con la siguiente ecuación: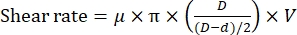
donde μ es la viscosidad dinámica del agua a 20 °C; D es el diámetro interior del estator; d es el diámetro exterior del rotor, y V es la velocidad de cizallamiento (rpm/s). - Sujete la cámara de reacción en la etapa de muestra del microscopio de fluorescencia y luego ajuste la cámara para sostener la punta dispersante del homogeneizador (Figura 1). Asegúrese de que la punta dispersante esté ligeramente por encima (~1 mm) de la superficie inferior de la cámara de reacción.
- Ajuste la posición vertical del homogeneizador y la cámara juntos para asegurarse de que el foco del microscopio esté en la superficie de la punta de dispersión. Luego, ajuste la posición horizontal de la punta de dispersión para asegurarse de que el área de detección (campo de visión) esté establecida entre el rotor y el estator (Figura 1).
- Encienda los canales de fluorescencia de acuerdo con el tinte fluorescente utilizado en el experimento.
- Antes del experimento de cizallamiento de alta velocidad, use agua desionizada (DI) para probar el cizallamiento con una velocidad de cizallamiento baja (por ejemplo, 2,000 rpm) para garantizar que la punta dispersante pueda funcionar adecuadamente sin tocar la cámara de reacción.
2. Despliegue de i-motivos con y sin ligandos
- Preparar un ADN telomérico humano i-motif (Tabla de Materiales) marcado con un tinte y un quencher en sus dos extremos, respectivamente, en agua DI, como se describe en Hu et al.11.
NOTA: i-motivo que contiene la secuencia: 5'-TAA CCC TAA CCC TAA CCC TAA CCC TAA. - Diluir el ADN a 5 μM en el tampón MES de 30 mM a pH 5.5 o pH 7.4. A la solución de ADN, añadir el ligando L2H2-4OTD, que fue sintetizado según Abraham Punnoose et al.13, en un rango de concentración de 0-60 μM. Mezclar la solución suavemente durante 10 min para doblar las estructuras i-motif sin luz.
- Verifique y minimice la intensidad de fluorescencia de fondo de la cámara de reacción que se llena con agua DI desionizada utilizando el microscopio fluorescente sin cizallamiento. Una manera fácil de minimizar la fluorescencia de fondo es lavar la cámara de reacción con agua DI. El valor de fluorescencia de fondo debe restarse en los análisis de datos más adelante.
NOTA: La luz parásita debe evitarse a partir de este paso. - Establezca los parámetros de la cámara CCD utilizando el software. Los parámetros recomendados son los siguientes: tiempo de exposición = 0,5 s, sensibilidad CCD = 1600 y tiempo de grabación = 20 min.
- Usando una pipeta larga, agregue la solución de ADN en la cámara de reacción vacía y limpia. Cubra la cámara de reacción con una caja negra. Luego, inicie el homogeneizador para realizar el cizallamiento a una velocidad de cizallamiento seleccionada que oscile entre 9,724 s−1 y 97,245 s−1 (seleccionada usando el software asociado con el homogeneizador) durante 20 minutos con la cámara CCD encendida para registrar los datos.
- Después del experimento, retire la cámara y lávela con agua DI.
3. Reacción de clic accionada por fuerza de cizallamiento
- Prepare el ADN i-motif en agua DI. Incubar 10 μM de ADN i-motivo en 300 μL de tampón Tris de 30 mM (pH 7.4) suplementado con 150 μM CuCl y 300 μM de ácido ascórbico durante 10 min para plegar las estructuras del i-motivo (todas las concentraciones son concentraciones finales en la solución).
NOTA: CuCl es el catalizador de la reacción de clic. El ácido ascórbico evitará la oxidación del cobre (I). - Ultrafiltre la solución con un dispositivo de ultrafiltración a una fuerza centrífuga de 14.300 x g. Reponer la solución a ~500 μL con tampón Tris de 30 mM (pH 7.4) suplementado con ácido ascórbico de 300 μM después de cada filtración.
- Repita la filtración 3x.
- Recoger la solución residual y hacer un volumen final de 300 μL añadiendo 30 mM de Tris (pH 7,4) suplementado con 300 μM de ácido ascórbico junto con 20 μM de Calfluor 488 azida, 20 μM de HPG y 10 μM de TBTA. Una vez que se agreguen los reactivos, mueva la solución al cuarto oscuro.
NOTA: La luz debe evitarse después de este paso. - Verifique y minimice la intensidad de fluorescencia de fondo de la cámara de reacción llena con agua DI usando el microscopio antes de los experimentos de cizallamiento. Una manera fácil de minimizar la fluorescencia de fondo es lavar la cámara de reacción con agua DI.
- Agregue la solución de ADN en la cámara de reacción vacía con una pipeta larga y luego inicie el cizallamiento del homogeneizador a una velocidad de cizallamiento de 63,209 s−1 durante 20 minutos con la cámara CCD encendida.
- Después del experimento, retire la cámara y lávela con agua DI.
Resultados
La Figura 1 describe el despliegue mecánico y la detección en tiempo real de moléculas de conjunto en EFS. En la Figura 1B, se observó que la intensidad de fluorescencia del ADN i-motif aumentaba con una velocidad de cizallamiento que oscilaba entre 9.724 s−1 y 97.245 s−1 en un tampón MES de pH 5,5. Como control, la intensidad de fluorescencia no aumentó cuando el mismo ADN i-motif se cortó a una velocidad de 63...
Discusión
El protocolo descrito en este manuscrito permite la investigación en tiempo real del despliegue de un conjunto de estructuras biomoleculares por fuerza de corte. Los resultados presentados aquí subrayan que las estructuras del motivo i del ADN pueden desplegarse mediante fuerza de corte. El despliegue del i-motivo ligado al ligando y las reacciones de clic accionadas por fuerza de cizallamiento fueron aplicaciones de prueba de concepto para este método de espectroscopia de fuerza de conjunto.
Divulgaciones
Los autores no tienen conflictos de intereses.
Agradecimientos
Este trabajo de investigación fue apoyado por la National Science Foundation [CBET-1904921] y los Institutos Nacionales de Salud [NIH R01CA236350] a H. M.
Materiales
| Name | Company | Catalog Number | Comments |
| 3K MWCO Amicon | Millipore Sigma | ufc900324 | |
| Ascorbic acid | VWR | VWRC0143-100G | |
| Calfluor 488 azide | Click Chemistry Tools | 1369-1 | |
| CuCl | Thermo | ACRO270525000 | |
| Dispersion tip | Switzerland | PT-DA07/2EC-B101 | |
| DNA oligos | IDT | ||
| Dye | IDT | /5Cy5/ | |
| Fluorescence microscope | Janpan | Nikon TE2000-U | |
| Homogenizer | Switzerland | PT 3100D | |
| HPG | Santa Cruz Biotechnology | cs-295271 | |
| KCl | VWR | VWRC26760.295 | |
| MES | VWR | VWRCE169-500G | |
| Quencher | IDT | /3IAbRQSp/ | |
| TBTA | Tokyo Chemical Industry | T2993 | |
| Tris | VWR | VWRCE133-100G |
Referencias
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: Optical tweezers, magnetic tweezers and atomic force microscopy. Nature Methods. 5 (6), 491-505 (2008).
- Woodside, M. T., et al. Nanomechanical measurements of the sequence-dependent folding landscapes of single nucleic acid hairpins. Proceedings of the National Academy of Sciences of the United States of America. 103 (16), 6190-6195 (2006).
- Grandbois, M., Beyer, M., Rief, M., Clausen-Schaumann, H., Gaub, H. E. How strong is a covalent bond. Science. 283 (5408), 1727-1730 (1999).
- Strick, T. R., Allemand, J. F., Bensimon, D., Croquette, V. Behavior of supercoiled DNA. Biophysical Journal. 74 (4), 2016-2028 (1998).
- Yang, D., Ward, A., Halvorsen, K., Wong, W. P. Multiplexed single-molecule force spectroscopy using a centrifuge. Nature Communications. 7, 11026 (2016).
- Su, H., et al. Light-responsive polymer particles as force clamps for the mechanical unfolding of target molecules. Nano Letters. 18 (4), 2630-2636 (2018).
- Kirkness, M. W. H., Forde, N. R. Single-molecule assay for proteolytic susceptibility: Force-induced collagen destabilization. Biophysical Journal. 114 (3), 570-576 (2018).
- Astumian, R. D. Thermodynamics and kinetics of molecular motors. Biophysical Journal. 98 (11), 2401-2409 (2010).
- Bekard, I. B., Asimakis, P., Bertolini, J., Dunstan, D. E. The effects of shear flow on protein structure and function. Biopolymers. 95 (11), 733-745 (2011).
- Chistiakov, D. A., Orekhov, A. N., Bobryshev, Y. V. Effects of shear stress on endothelial cells: go with the flow. Acta Physiologica. 219 (2), 382-408 (2017).
- Hu, C., Jonchhe, S., Pokhrel, P., Karna, D., Mao, H. Mechanical unfolding of ensemble biomolecular structures by shear force. Chemical Science. 12 (30), 10159-10164 (2021).
- Sedghi Masoud, S., et al. Analysis of interactions between telomeric i-motif DNA and a cyclic tetraoxazole compound. ChemBioChem. 19 (21), 2268-2272 (2018).
- Abraham Punnoose, J., et al. Adaptive and specific recognition of telomeric G-quadruplexes via polyvalency induced unstacking of binding units. Journal of the American Chemical Society. 139 (22), 7476-7484 (2017).
- Dhakal, S., et al. Coexistence of an ILPR i-motif and a partially folded structure with comparable mechanical stability revealed at the single-molecule level. Journal of the American Chemical Society. 132 (26), 8991-8997 (2010).
- Hu, C., Tahir, R., Mao, H. Single-molecule mechanochemical sensing. Accounts of Chemical Research. 55 (9), 1214-1225 (2022).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados