È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Spettroscopia Ensemble Force mediante Shear Forces
In questo articolo
Riepilogo
La spettroscopia di forza d'insieme (EFS) è una tecnica robusta per lo sviluppo meccanico e il rilevamento in tempo reale di un insieme di strutture biomolecolari in campi biofisici e biosensibili.
Abstract
Le tecniche a singola molecola basate sulla fluorescenza e sui principi meccanochimici forniscono una sensibilità superiore nel rilevamento biologico. Tuttavia, a causa della mancanza di elevate capacità di produttività, l'applicazione di queste tecniche è limitata in biofisica. La spettroscopia di forza d'insieme (EFS) ha dimostrato un elevato rendimento nello studio di un massiccio insieme di strutture molecolari convertendo gli studi meccanochimici delle singole molecole in quelli degli insiemi molecolari. In questo protocollo, le strutture secondarie del DNA (i-motivi) sono state spiegate nel flusso di taglio tra il rotore e lo statore di una punta omogeneizzatore a velocità di taglio fino a 77796/s. Sono stati dimostrati gli effetti delle portate e delle dimensioni molecolari sulle forze di taglio sperimentate dall'i-motif. La tecnica EFS ha anche rivelato l'affinità di legame tra i motivi del DNA e i ligandi. Inoltre, abbiamo dimostrato una reazione chimica del clic che può essere azionata dalla forza di taglio (cioè la chimica meccano-clic). Questi risultati stabiliscono l'efficacia dell'uso della forza di taglio per controllare la conformazione delle strutture molecolari.
Introduzione
Nella spettroscopia di forza a singola molecola1 (SMFS), le proprietà meccaniche delle singole strutture molecolari sono state studiate da strumenti sofisticati come il microscopio a forza atomica, le pinzette ottiche e le pinzette magnetiche 2,3,4. Limitato dallo stesso requisito di direzionalità delle molecole nelle configurazioni di generazione/rilevamento della forza o dal piccolo campo visivo nelle pinzette magnetiche e nel microscopio a forza centrifuga miniaturizzato (MCF)5,6,7,8, solo un numero limitato di molecole può essere studiato simultaneamente utilizzando SMFS. Il basso rendimento di SMFS impedisce la sua ampia applicazione nel campo del riconoscimento molecolare, che richiede il coinvolgimento di un ampio insieme di molecole.
Il flusso di taglio fornisce una potenziale soluzione per applicare forze a un massiccio insieme di molecole9. In un flusso di liquido all'interno di un canale, più vicino alla superficie del canale, più lenta è la portata10. Tale gradiente di velocità del flusso causa sollecitazioni di taglio parallele alla superficie limite. Quando una molecola viene posizionata in questo flusso di taglio, la molecola si riorienta in modo che il suo asse lungo si allinei con la direzione del flusso, poiché la forza di taglio viene applicata all'asse lungo11. Come risultato di questo riorientamento, ci si aspetta che tutte le molecole dello stesso tipo (dimensioni e lunghezza delle maniglie) si allineino nella stessa direzione mentre sperimentano la stessa forza di taglio.
Questo lavoro descrive un protocollo per utilizzare un tale flusso di taglio per esercitare una forza di taglio su un massiccio insieme di strutture molecolari, come esemplificato dal motivo del DNA. In questo protocollo, viene generato un flusso di taglio tra il rotore e lo statore in una punta omogeneizzatore. Il presente studio ha scoperto che la struttura ripiegata del DNA i-motif potrebbe essere spiegata da velocità di taglio di 9724-97245 s-1. Inoltre, è stata trovata una costante di dissociazione di 36 μM tra il ligando L2H2-4OTD e il motivo i. Questo valore è coerente con quello di 31 μM misurato dal saggio di spostamento del gel12. Inoltre, la tecnica corrente viene utilizzata per dispiegare il motivo i, che può esporre il rame chelato (I) per catalizzare una reazione di clic. Questo protocollo consente quindi di dispiegare un ampio set di strutture i-motif con strumenti a basso costo in un tempo ragionevole (inferiore a 30 min). Dato che la tecnica della forza di taglio aumenta drasticamente il throughput della spettroscopia di forza, chiamiamo questa tecnica spettroscopia di forza d'insieme (EFS). Questo protocollo mira a fornire linee guida sperimentali per facilitare l'applicazione di questo EFS basato sulla forza di taglio.
Protocollo
NOTA: Tutti i tamponi e i reagenti chimici utilizzati in questo protocollo sono elencati nella tabella Materiali.
1. Preparazione del microscopio a forza di taglio
NOTA: Il microscopio a forza di taglio contiene due parti, un'unità di reazione (omogeneizzatore) e un'unità di rilevamento (microscopio a fluorescenza). L'ingrandimento dell'oculare è 10x e l'ingrandimento della lente dell'obiettivo (aria) è 4x.
- Assemblare l'omogeneizzatore e il microscopio su un tavolo di montaggio. Indossare occhiali e accendere il microscopio a fluorescenza, quindi regolare l'omogeneizzatore per assicurarsi che il fascio di luce di eccitazione di una lunghezza d'onda appropriata (qui, è stato utilizzato 488 nm) passi attraverso il centro della punta di dispersione dell'omogeneizzatore.
- Preparare una camera di reazione a fondo piatto di 5 cm di altezza e 1,5 cm 2 x 1,5 cm2 di sezione trasversale. Per ridurre lo sfondo, assicurarsi che il materiale della camera selezionato non fluoresce, come gli occhiali (alcune materie plastiche hanno fluorescenza).
- Scegliere una punta di dispersione omogeneizzatore appropriata in grado di fornire la forza di taglio desiderata.
NOTA: la velocità di taglio dipende dalla distanza tra il rotore e lo statore a una velocità di rotazione fissa11 secondo la seguente equazione: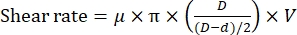
dove μ è la viscosità dinamica dell'acqua a 20 °C; D è il diametro interno dello statore; d è il diametro esterno del rotore e V è la velocità di taglio (rpm/s). - Bloccare la camera di reazione sullo stadio del campione del microscopio a fluorescenza, quindi regolare la camera per contenere la punta disperdente dell'omogeneizzatore (Figura 1). Assicurarsi che la punta di dispersione sia leggermente al di sopra (~1 mm) della superficie inferiore della camera di reazione.
- Regolare la posizione verticale dell'omogeneizzatore e della camera insieme per assicurarsi che la messa a fuoco del microscopio sia sulla superficie della punta disperdente. Quindi, regolare la posizione orizzontale della punta di dispersione per assicurarsi che l'area di rilevamento (campo visivo) sia impostata tra il rotore e lo statore (Figura 1).
- Accendere i canali di fluorescenza in base al colorante fluorescente utilizzato nell'esperimento.
- Prima dell'esperimento di taglio ad alta velocità, utilizzare acqua deionizzata (DI) per testare la cesoiatura con una bassa velocità di taglio (ad esempio, 2.000 giri / min) per garantire che la punta di dispersione possa funzionare in modo appropriato senza toccare la camera di reazione.
2. Spiegamento di i-motivi con e senza ligandi
- Preparare un DNA i-motif telomerico umano (Table of Materials) etichettato con un colorante e un quencher alle sue due estremità, rispettivamente, in acqua DI, come descritto in Hu et al.11.
NOTA: i-motivo contenente sequenza: 5'-TAA CCC TAA CCC TAA CCC TAA CCC TAA. - Diluire il DNA a 5 μM nel tampone MES da 30 mM a pH 5,5 o pH 7,4. Alla soluzione di DNA, aggiungere il ligando L2H2-4OTD, che è stato sintetizzato secondo Abraham Punnoose et al.13, in un intervallo di concentrazione di 0-60 μM. Mescolare delicatamente la soluzione per 10 minuti per piegare le strutture i-motif senza luce.
- Controllare e ridurre al minimo l'intensità della fluorescenza di fondo della camera di reazione riempita con acqua DI deionizzata utilizzando il microscopio fluorescente senza cesoiatura. Un modo semplice per ridurre al minimo la fluorescenza di fondo è lavare la camera di reazione con acqua DI. Il valore di fluorescenza di fondo deve essere sottratto nelle analisi dei dati in un secondo momento.
NOTA: la luce diffusa dovrebbe essere evitata da questo passaggio in poi. - Impostare i parametri della telecamera CCD utilizzando il software. I parametri consigliati sono i seguenti: tempo di esposizione = 0,5 s, sensibilità CCD = 1600 e tempo di registrazione = 20 min.
- Utilizzando un pipet lungo, aggiungere la soluzione di DNA nella camera di reazione vuota e pulita. Coprire la camera di reazione con una scatola nera. Quindi, avviare l'omogeneizzatore per eseguire la cesoiatura a una velocità di taglio selezionata compresa tra 9.724 s-1 e 97.245 s-1 (selezionata utilizzando il software associato all'omogeneizzatore) per 20 minuti con la telecamera CCD accesa per registrare i dati.
- Dopo l'esperimento, rimuovere la camera e lavarla con acqua DI.
3. Reazione del clic azionata dalla forza di taglio
- Preparare il DNA i-motif in acqua DI. Incubare 10 μM di DNA i-motif in 300 μL di tampone Tris da 30 mM (pH 7,4) integrato con 150 μM di CuCl e 300 μM di acido ascorbico per 10 minuti per piegare le strutture i-motif (tutte le concentrazioni sono concentrazioni finali nella soluzione).
NOTA: CuCl è il catalizzatore della reazione del clic. L'acido ascorbico impedirà l'ossidazione del rame (I). - Ultrafiltrare la soluzione con un dispositivo di ultrafiltrazione con una forza centrifuga di 14.300 x g. Reintegrare la soluzione a ~500 μL con 30 mM di tampone Tris (pH 7,4) integrato con acido ascorbico 300 μM dopo ogni filtrazione.
- Ripetere la filtrazione 3x.
- Raccogliere la soluzione residua e ottenere un volume finale di 300 μL aggiungendo 30 mM Tris (pH 7,4) integrati con 300 μM di acido ascorbico insieme a 20 μM di Calfluor 488 azide, 20 μM HPG e 10 μM TBTA. Una volta aggiunti i reagenti, spostare la soluzione nella camera oscura.
NOTA: la luce deve essere evitata dopo questo passaggio. - Controllare e ridurre al minimo l'intensità della fluorescenza di fondo della camera di reazione riempita con acqua DI usando il microscopio prima degli esperimenti di taglio. Un modo semplice per ridurre al minimo la fluorescenza di fondo è lavare la camera di reazione con acqua DI.
- Aggiungere la soluzione di DNA nella camera di reazione vuota con un pipet lungo, quindi avviare la cesoiatura dell'omogeneizzatore a una velocità di taglio di 63.209 s−1 per 20 minuti con la telecamera CCD accesa.
- Dopo l'esperimento, rimuovere la camera e lavarla con acqua DI.
Risultati
La Figura 1 illustra lo sviluppo meccanico e il rilevamento in tempo reale delle molecole dell'insieme in EFS. Nella Figura 1B, è stato osservato che l'intensità di fluorescenza del DNA i-motif aumenta con la velocità di taglio che varia da 9.724 s-1 a 97.245 s-1 in un tampone MES a pH 5,5. Come controllo, l'intensità della fluorescenza non è aumentata quando lo stesso DNA i-motif è stato tagliato ad una velocità di ...
Discussione
Il protocollo descritto in questo manoscritto consente di studiare in tempo reale lo sviluppo di un insieme di strutture biomolecolari mediante forza di taglio. I risultati qui presentati sottolineano che le strutture del DNA i-motif possono essere spiegate dalla forza di taglio. Lo sviluppo del motivo i-legato al ligando e le reazioni a scatto azionate dalla forza di taglio sono state applicazioni proof-of-concept per questo metodo di spettroscopia a forza d'insieme.
La F...
Divulgazioni
Gli autori non hanno conflitti di interesse.
Riconoscimenti
Questo lavoro di ricerca è stato sostenuto dalla National Science Foundation [CBET-1904921] e dal National Institutes of Health [NIH R01CA236350] a H. M.
Materiali
| Name | Company | Catalog Number | Comments |
| 3K MWCO Amicon | Millipore Sigma | ufc900324 | |
| Ascorbic acid | VWR | VWRC0143-100G | |
| Calfluor 488 azide | Click Chemistry Tools | 1369-1 | |
| CuCl | Thermo | ACRO270525000 | |
| Dispersion tip | Switzerland | PT-DA07/2EC-B101 | |
| DNA oligos | IDT | ||
| Dye | IDT | /5Cy5/ | |
| Fluorescence microscope | Janpan | Nikon TE2000-U | |
| Homogenizer | Switzerland | PT 3100D | |
| HPG | Santa Cruz Biotechnology | cs-295271 | |
| KCl | VWR | VWRC26760.295 | |
| MES | VWR | VWRCE169-500G | |
| Quencher | IDT | /3IAbRQSp/ | |
| TBTA | Tokyo Chemical Industry | T2993 | |
| Tris | VWR | VWRCE133-100G |
Riferimenti
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: Optical tweezers, magnetic tweezers and atomic force microscopy. Nature Methods. 5 (6), 491-505 (2008).
- Woodside, M. T., et al. Nanomechanical measurements of the sequence-dependent folding landscapes of single nucleic acid hairpins. Proceedings of the National Academy of Sciences of the United States of America. 103 (16), 6190-6195 (2006).
- Grandbois, M., Beyer, M., Rief, M., Clausen-Schaumann, H., Gaub, H. E. How strong is a covalent bond. Science. 283 (5408), 1727-1730 (1999).
- Strick, T. R., Allemand, J. F., Bensimon, D., Croquette, V. Behavior of supercoiled DNA. Biophysical Journal. 74 (4), 2016-2028 (1998).
- Yang, D., Ward, A., Halvorsen, K., Wong, W. P. Multiplexed single-molecule force spectroscopy using a centrifuge. Nature Communications. 7, 11026 (2016).
- Su, H., et al. Light-responsive polymer particles as force clamps for the mechanical unfolding of target molecules. Nano Letters. 18 (4), 2630-2636 (2018).
- Kirkness, M. W. H., Forde, N. R. Single-molecule assay for proteolytic susceptibility: Force-induced collagen destabilization. Biophysical Journal. 114 (3), 570-576 (2018).
- Astumian, R. D. Thermodynamics and kinetics of molecular motors. Biophysical Journal. 98 (11), 2401-2409 (2010).
- Bekard, I. B., Asimakis, P., Bertolini, J., Dunstan, D. E. The effects of shear flow on protein structure and function. Biopolymers. 95 (11), 733-745 (2011).
- Chistiakov, D. A., Orekhov, A. N., Bobryshev, Y. V. Effects of shear stress on endothelial cells: go with the flow. Acta Physiologica. 219 (2), 382-408 (2017).
- Hu, C., Jonchhe, S., Pokhrel, P., Karna, D., Mao, H. Mechanical unfolding of ensemble biomolecular structures by shear force. Chemical Science. 12 (30), 10159-10164 (2021).
- Sedghi Masoud, S., et al. Analysis of interactions between telomeric i-motif DNA and a cyclic tetraoxazole compound. ChemBioChem. 19 (21), 2268-2272 (2018).
- Abraham Punnoose, J., et al. Adaptive and specific recognition of telomeric G-quadruplexes via polyvalency induced unstacking of binding units. Journal of the American Chemical Society. 139 (22), 7476-7484 (2017).
- Dhakal, S., et al. Coexistence of an ILPR i-motif and a partially folded structure with comparable mechanical stability revealed at the single-molecule level. Journal of the American Chemical Society. 132 (26), 8991-8997 (2010).
- Hu, C., Tahir, R., Mao, H. Single-molecule mechanochemical sensing. Accounts of Chemical Research. 55 (9), 1214-1225 (2022).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon