É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Espectroscopia de Força de Ensemble por Forças de Cisalhamento
Neste Artigo
Resumo
A espectroscopia de força de conjunto (EFS) é uma técnica robusta para desdobramento mecânico e detecção em tempo real de um conjunto de estruturas biomoleculares em campos biofísicos e de biossensoriamento.
Resumo
Técnicas de molécula única baseadas em princípios de fluorescência e mecanoquímicos fornecem sensibilidade superior em sensoriamento biológico. No entanto, devido à falta de recursos de alto rendimento, a aplicação dessas técnicas é limitada em biofísica. A espectroscopia de força de conjunto (EFS) demonstrou alto rendimento na investigação de um conjunto maciço de estruturas moleculares, convertendo estudos mecanoquímicos de moléculas individuais em conjuntos moleculares. Neste protocolo, as estruturas secundárias do DNA (i-motivos) foram desdobradas no fluxo de cisalhamento entre o rotor e o estator de uma ponta do homogeneizador a taxas de cisalhamento de até 77796/s. Os efeitos das taxas de fluxo e tamanhos moleculares sobre as forças de cisalhamento experimentadas pelo motivo i foram demonstrados. A técnica EFS também revelou a afinidade de ligação entre i-motivos de DNA e ligantes. Além disso, demonstramos uma reação química de clique que pode ser acionada por força de cisalhamento (ou seja, química de clique mecano). Esses resultados estabelecem a eficácia do uso da força de cisalhamento para controlar a conformação de estruturas moleculares.
Introdução
Na espectroscopia de força de molécula única1 (SMFS), as propriedades mecânicas de estruturas moleculares individuais têm sido estudadas por instrumentos sofisticados como o microscópio de força atômica, pinças ópticas e pinças magnéticas 2,3,4. Restrito pelo mesmo requisito de direcionalidade das moléculas nas configurações de geração/detecção de força ou pelo pequeno campo de visão em pinças magnéticas e no microscópio de força de centrífuga em miniatura (MCF)5,6,7,8, apenas um número limitado de moléculas pode ser investigado simultaneamente usando SMFS. O baixo rendimento do SMFS impede sua ampla aplicação no campo do reconhecimento molecular, o que requer o envolvimento de um grande conjunto de moléculas.
O fluxo de cisalhamento fornece uma solução potencial para aplicar forças a um conjunto maciço de moléculas9. Em um fluxo de líquido dentro de um canal, quanto mais próximo da superfície do canal, mais lenta a taxa de fluxo10. Tal gradiente de velocidade de fluxo causa tensão de cisalhamento paralela à superfície limite. Quando uma molécula é colocada nesse fluxo de cisalhamento, a molécula se reorienta para que seu eixo longo se alinhe com a direção do fluxo, à medida que a força de cisalhamento é aplicada ao eixo longo11. Como resultado dessa reorientação, espera-se que todas as moléculas do mesmo tipo (tamanho e comprimento das alças) se alinhem na mesma direção enquanto experimentam a mesma força de cisalhamento.
Este trabalho descreve um protocolo para usar tal fluxo de cisalhamento para exercer força de cisalhamento em um conjunto maciço de estruturas moleculares, como exemplificado pelo i-motivo do DNA. Neste protocolo, um fluxo de cisalhamento é gerado entre o rotor e o estator em uma ponta do homogeneizador. O presente estudo descobriu que a estrutura dobrada do i-motivo do DNA poderia ser desdobrada por taxas de cisalhamento de 9724-97245 s−1. Além disso, uma constante de dissociação de 36 μM foi encontrada entre o ligante L2H2-4OTD e o motivo i. Este valor é consistente com o de 31 μM medido pelo ensaio de deslocamento de gel12. Além disso, a técnica atual é usada para desdobrar o i-motivo, que pode expor o cobre quelado (I) para catalisar uma reação de clique. Este protocolo permite, assim, desdobrar um grande conjunto de estruturas i-motif com instrumentos de baixo custo em um tempo razoável (menor que 30 min). Dado que a técnica de força de cisalhamento aumenta drasticamente o rendimento da espectroscopia de força, chamamos essa técnica de espectroscopia de força de conjunto (EFS). Este protocolo tem como objetivo fornecer diretrizes experimentais para facilitar a aplicação deste EFS baseado em força de cisalhamento.
Protocolo
NOTA: Todos os tampões e os reagentes químicos utilizados neste protocolo estão listados na Tabela de Materiais.
1. Preparação do microscópio de força de cisalhamento
NOTA: O microscópio de força de cisalhamento contém duas partes, uma unidade de reação (homogeneizador) e uma unidade de detecção (microscópio de fluorescência). A ampliação da ocular é de 10x, e a ampliação da lente objetiva (ar) é de 4x.
- Monte o homogeneizador e o microscópio em uma mesa de montagem. Use óculos e ligue o microscópio de fluorescência e, em seguida, ajuste o homogeneizador para garantir que o feixe de luz de excitação de um comprimento de onda apropriado (aqui, 488 nm foi usado) passe pelo centro da ponta de dispersão do homogeneizador.
- Prepare uma câmara de reação de fundo plano com 5 cm de altura e 1,5 cm 2 x 1,5 cm2 de seção transversal. Para reduzir o fundo, certifique-se de que o material da câmara selecionado não fluoresce, como vidros (alguns plásticos têm fluorescência).
- Escolha uma ponta de dispersão do homogeneizador apropriada que possa fornecer a força de cisalhamento desejada.
NOTA: A taxa de cisalhamento depende da distância entre o rotor e o estator a uma velocidade de rotação fixa11 de acordo com a seguinte equação: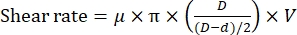
em que μ é a viscosidade dinâmica da água a 20 °C; D é o diâmetro interno do estator; d é o diâmetro externo do rotor e V é a velocidade de cisalhamento (rpm/s). - Aperte a câmara de reação no estágio de amostra do microscópio de fluorescência e, em seguida, ajuste a câmara para segurar a ponta dispersora do homogeneizador (Figura 1). Certifique-se de que a ponta de dispersão esteja ligeiramente acima (~1 mm) da superfície inferior da câmara de reação.
- Ajuste a posição vertical do homogeneizador e da câmara em conjunto para garantir que o foco do microscópio esteja na superfície da ponta de dispersão. Em seguida, ajuste a posição horizontal da ponta de dispersão para garantir que a área de detecção (campo de visão) esteja ajustada entre o rotor e o estator (Figura 1).
- Ligue os canais de fluorescência de acordo com o corante fluorescente usado no experimento.
- Antes do experimento de cisalhamento de alta velocidade, use água deionizada (DI) para testar o cisalhamento com uma baixa velocidade de cisalhamento (por exemplo, 2.000 rpm) para garantir que a ponta de dispersão possa funcionar adequadamente sem tocar na câmara de reação.
2. Desdobramento de i-motivos com e sem ligantes
- Preparar um DNA telomérico humano i-motif (Table of Materials) marcado com um corante e um quencher em suas duas extremidades, respectivamente, em água DI, conforme descrito em Hu et al.11.
NOTA: i-motivo contendo sequência: 5'-TAA CCC TAA CCC TAA CCC TAA CCC CCC TAA. - Diluir o ADN a 5 μM no tampão MES de 30 mM a pH 5,5 ou pH 7,4. À solução de DNA, adicionar o ligante L2H2-4OTD, que foi sintetizado de acordo com Abraham Punnoose et al.13, em uma faixa de concentração de 0-60 μM. Misture a solução suavemente por 10 min para dobrar as estruturas do motivo i sem luz.
- Verifique e minimize a intensidade de fluorescência de fundo da câmara de reação que é preenchida com água DI deionizada usando o microscópio fluorescente sem cisalhamento. Uma maneira fácil de minimizar a fluorescência de fundo é lavar a câmara de reação com água DI. O valor de fluorescência de fundo precisa ser subtraído nas análises de dados posteriormente.
NOTA: A luz dispersa deve ser evitada a partir deste passo. - Defina os parâmetros da câmera CCD usando o software. Os parâmetros recomendados são os seguintes: tempo de exposição = 0,5 s, sensibilidade ao CCD = 1600 e tempo de registro = 20 min.
- Usando uma pipeta longa, adicione a solução de DNA na câmara de reação vazia e limpa. Cubra a câmara de reação com uma caixa preta. Em seguida, inicie o homogeneizador para realizar o cisalhamento a uma taxa de cisalhamento selecionada variando de 9.724 s−1 a 97.245 s−1 (selecionada usando o software associado ao homogeneizador) por 20 minutos com a câmera CCD ligada para registrar os dados.
- Após o experimento, remova a câmara e lave-a com água DI.
3. Reação de clique acionada por força de cisalhamento
- Prepare o DNA i-motif em água DI. Incubar 10 μM de ADN i-motif em 300 μL de tampão Tris de 30 mM (pH 7,4) suplementado com 150 μM de CuCl e 300 μM de ácido ascórbico durante 10 minutos para dobrar as estruturas i-motif (todas as concentrações são concentrações finais na solução).
Observação : CuCl é o catalisador da reação de clique. O ácido ascórbico impedirá a oxidação do cobre (I). - Ultrafiltre a solução com um dispositivo de ultrafiltração a uma força centrífuga de 14.300 x g. Repor a solução até ~500 μL com tampão Tris de 30 mM (pH 7,4) suplementado com ácido ascórbico de 300 μM após cada filtração.
- Repita a filtração 3x.
- Recolher a solução residual e fazer um volume final de 300 μL adicionando 30 mM Tris (pH 7,4) suplementado com ácido ascórbico 300 μM juntamente com 20 μM Calfluor 488 azida, 20 μM HPG e 10 μM TBTA. Uma vez que os reagentes são adicionados, mova a solução para a câmara escura.
NOTA: A luz deve ser evitada após esta etapa. - Verifique e minimize a intensidade de fluorescência de fundo da câmara de reação preenchida com água DI usando o microscópio antes dos experimentos de cisalhamento. Uma maneira fácil de minimizar a fluorescência de fundo é lavar a câmara de reação com água DI.
- Adicione a solução de DNA na câmara de reação vazia com um pipeto longo e, em seguida, inicie o cisalhamento do homogeneizador a uma taxa de cisalhamento de 63.209 s−1 por 20 minutos com a câmera CCD ligada.
- Após o experimento, remova a câmara e lave-a com água DI.
Resultados
A Figura 1 descreve o desdobramento mecânico e a detecção em tempo real das moléculas do conjunto no EFS. Na Figura 1B, observou-se que a intensidade de fluorescência do DNA i-motif aumentou com a taxa de cisalhamento variando de 9.724 s−1 a 97.245 s−1 em um tampão MES de pH 5,5. Como controle, a intensidade da fluorescência não foi aumentada quando o mesmo DNA i-motif foi cortado a uma taxa de 63.209 s−1...
Discussão
O protocolo descrito neste manuscrito permite a investigação em tempo real do desdobramento de um conjunto de estruturas biomoleculares por força de cisalhamento. Os resultados aqui apresentados ressaltam que as estruturas do i-motivo do DNA podem ser desdobradas pela força de cisalhamento. O desdobramento do i-motivo ligado ao ligante e as reações de clique acionadas por força de cisalhamento foram aplicações de prova de conceito para este método de espectroscopia de força de conjunto.
Divulgações
Os autores não têm conflitos de interesse.
Agradecimentos
Este trabalho de pesquisa foi apoiado pela National Science Foundation [CBET-1904921] e pelos Institutos Nacionais de Saúde [NIH R01CA236350] para H. M.
Materiais
| Name | Company | Catalog Number | Comments |
| 3K MWCO Amicon | Millipore Sigma | ufc900324 | |
| Ascorbic acid | VWR | VWRC0143-100G | |
| Calfluor 488 azide | Click Chemistry Tools | 1369-1 | |
| CuCl | Thermo | ACRO270525000 | |
| Dispersion tip | Switzerland | PT-DA07/2EC-B101 | |
| DNA oligos | IDT | ||
| Dye | IDT | /5Cy5/ | |
| Fluorescence microscope | Janpan | Nikon TE2000-U | |
| Homogenizer | Switzerland | PT 3100D | |
| HPG | Santa Cruz Biotechnology | cs-295271 | |
| KCl | VWR | VWRC26760.295 | |
| MES | VWR | VWRCE169-500G | |
| Quencher | IDT | /3IAbRQSp/ | |
| TBTA | Tokyo Chemical Industry | T2993 | |
| Tris | VWR | VWRCE133-100G |
Referências
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: Optical tweezers, magnetic tweezers and atomic force microscopy. Nature Methods. 5 (6), 491-505 (2008).
- Woodside, M. T., et al. Nanomechanical measurements of the sequence-dependent folding landscapes of single nucleic acid hairpins. Proceedings of the National Academy of Sciences of the United States of America. 103 (16), 6190-6195 (2006).
- Grandbois, M., Beyer, M., Rief, M., Clausen-Schaumann, H., Gaub, H. E. How strong is a covalent bond. Science. 283 (5408), 1727-1730 (1999).
- Strick, T. R., Allemand, J. F., Bensimon, D., Croquette, V. Behavior of supercoiled DNA. Biophysical Journal. 74 (4), 2016-2028 (1998).
- Yang, D., Ward, A., Halvorsen, K., Wong, W. P. Multiplexed single-molecule force spectroscopy using a centrifuge. Nature Communications. 7, 11026 (2016).
- Su, H., et al. Light-responsive polymer particles as force clamps for the mechanical unfolding of target molecules. Nano Letters. 18 (4), 2630-2636 (2018).
- Kirkness, M. W. H., Forde, N. R. Single-molecule assay for proteolytic susceptibility: Force-induced collagen destabilization. Biophysical Journal. 114 (3), 570-576 (2018).
- Astumian, R. D. Thermodynamics and kinetics of molecular motors. Biophysical Journal. 98 (11), 2401-2409 (2010).
- Bekard, I. B., Asimakis, P., Bertolini, J., Dunstan, D. E. The effects of shear flow on protein structure and function. Biopolymers. 95 (11), 733-745 (2011).
- Chistiakov, D. A., Orekhov, A. N., Bobryshev, Y. V. Effects of shear stress on endothelial cells: go with the flow. Acta Physiologica. 219 (2), 382-408 (2017).
- Hu, C., Jonchhe, S., Pokhrel, P., Karna, D., Mao, H. Mechanical unfolding of ensemble biomolecular structures by shear force. Chemical Science. 12 (30), 10159-10164 (2021).
- Sedghi Masoud, S., et al. Analysis of interactions between telomeric i-motif DNA and a cyclic tetraoxazole compound. ChemBioChem. 19 (21), 2268-2272 (2018).
- Abraham Punnoose, J., et al. Adaptive and specific recognition of telomeric G-quadruplexes via polyvalency induced unstacking of binding units. Journal of the American Chemical Society. 139 (22), 7476-7484 (2017).
- Dhakal, S., et al. Coexistence of an ILPR i-motif and a partially folded structure with comparable mechanical stability revealed at the single-molecule level. Journal of the American Chemical Society. 132 (26), 8991-8997 (2010).
- Hu, C., Tahir, R., Mao, H. Single-molecule mechanochemical sensing. Accounts of Chemical Research. 55 (9), 1214-1225 (2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados