Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Ensemble Force Spektroskopie durch Scherkräfte
In diesem Artikel
Zusammenfassung
Die Ensemblekraftspektroskopie (EFS) ist eine robuste Technik zur mechanischen Entfaltung und Echtzeiterfassung eines Ensemblesatzes biomolekularer Strukturen in biophysikalischen und biosensorischen Bereichen.
Zusammenfassung
Einzelmolekültechniken, die auf Fluoreszenz und mechanochemischen Prinzipien basieren, bieten eine überlegene Empfindlichkeit in der biologischen Sensorik. Aufgrund des Mangels an Hochdurchsatzkapazitäten ist die Anwendung dieser Techniken in der Biophysik jedoch begrenzt. Die Ensemble-Kraft-Spektroskopie (EFS) hat einen hohen Durchsatz bei der Untersuchung eines massiven Satzes molekularer Strukturen gezeigt, indem mechanochemische Untersuchungen einzelner Moleküle in solche von molekularen Ensembles umgewandelt wurden. In diesem Protokoll wurden die DNA-Sekundärstrukturen (i-Motive) im Scherstrom zwischen Rotor und Stator einer Homogenisatorspitze mit Scherraten bis zu 77796/s entfaltet. Die Auswirkungen von Durchflussraten und Molekülgrößen auf die Scherkräfte des i-Motivs wurden nachgewiesen. Die EFS-Technik zeigte auch die Bindungsaffinität zwischen DNA-i-Motiven und Liganden. Darüber hinaus haben wir eine Klickchemiereaktion demonstriert, die durch Scherkraft (d.h. Mechano-Klick-Chemie) ausgelöst werden kann. Diese Ergebnisse belegen die Wirksamkeit der Verwendung von Scherkraft zur Kontrolle der Konformation molekularer Strukturen.
Einleitung
In der Einzelmolekül-Kraftspektroskopie1 (SMFS) wurden die mechanischen Eigenschaften einzelner Molekülstrukturen mit hochentwickelten Instrumenten wie dem Rasterkraftmikroskop, einer optischen Pinzette und einer Magnetpinzette 2,3,4 untersucht. Eingeschränkt durch die gleiche Richtungsanforderung der Moleküle in den krafterzeugenden/detektierenden Aufbauten oder das kleine Sichtfeld in Magnetpinzetten und dem Miniatur-Zentrifugenkraftmikroskop (MCF)5,6,7,8, kann nur eine begrenzte Anzahl von Molekülen gleichzeitig mit SMFS untersucht werden. Der geringe Durchsatz von SMFS verhindert seine breite Anwendung im Bereich der molekularen Erkennung, was die Beteiligung einer großen Anzahl von Molekülen erfordert.
Die Scherströmung bietet eine mögliche Lösung, um Kräfte auf eine massive Gruppe von Molekülen auszuüben9. In einer Flüssigkeitsströmung innerhalb eines Kanals gilt: Je näher an der Kanaloberfläche, desto langsamer ist die Durchflussrate10. Ein solcher Strömungsgeschwindigkeitsgradient verursacht eine Schubspannung, die parallel zur Grenzfläche verläuft. Wenn ein Molekül in diese Scherströmung gebracht wird, orientiert sich das Molekül so, dass seine Längsachse mit der Strömungsrichtung übereinstimmt, da die Querkraft auf die lange Achse11 aufgebracht wird. Als Ergebnis dieser Neuorientierung wird erwartet, dass sich alle Moleküle des gleichen Typs (Größe und Länge der Griffe) in die gleiche Richtung ausrichten, während sie die gleiche Scherkraft erfahren.
Diese Arbeit beschreibt ein Protokoll, um eine solche Scherströmung zu verwenden, um Scherkraft auf einen massiven Satz molekularer Strukturen auszuüben, wie das DNA-i-Motiv veranschaulicht. Bei diesem Protokoll wird eine Scherströmung zwischen Rotor und Stator in einer Homogenisatorspitze erzeugt. Die vorliegende Studie ergab, dass die gefaltete DNA-i-Motivstruktur durch Scherraten von 9724-97245 s−1 entfaltet werden konnte. Außerdem wurde eine Dissoziationskonstante von 36 μM zwischen dem L2H2-4OTD-Liganden und dem i-Motiv gefunden. Dieser Wert stimmt mit dem Wert von 31 μM überein, gemessen mit dem Gelverschiebungsassay12. Weiterhin wird die aktuelle Technik verwendet, um das i-Motiv zu entfalten, das das chelatierte Kupfer (I) freilegen kann, um eine Klickreaktion zu katalysieren. Dieses Protokoll ermöglicht es somit, einen großen Satz von i-Motiv-Strukturen mit kostengünstigen Instrumenten in angemessener Zeit (kürzer als 30 min) zu entfalten. Da die Querkrafttechnik den Durchsatz der Kraftspektroskopie drastisch erhöht, nennen wir diese Technik Ensemblekraftspektroskopie (EFS). Dieses Protokoll zielt darauf ab, experimentelle Richtlinien bereitzustellen, um die Anwendung dieses auf Scherkraft basierenden EFS zu erleichtern.
Protokoll
HINWEIS: Alle in diesem Protokoll verwendeten Puffer und chemischen Reagenzien sind in der Tabelle Materialien aufgeführt.
1. Vorbereitung des Scherkraftmikroskops
HINWEIS: Das Scherkraftmikroskop besteht aus zwei Teilen, einer Reaktionseinheit (Homogenisator) und einer Detektionseinheit (Fluoreszenzmikroskop). Die Vergrößerung des Okulars beträgt 10x und die Vergrößerung der Objektivlinse (Luft) ist 4x.
- Montieren Sie den Homogenisator und das Mikroskop auf einem Montagetisch. Tragen Sie eine Schutzbrille und schalten Sie das Fluoreszenzmikroskop ein und stellen Sie dann den Homogenisator ein, um sicherzustellen, dass der Anregungslichtstrahl einer geeigneten Wellenlänge (hier wurden 488 nm verwendet) durch die Mitte der Dispergierspitze des Homogenisators geht.
- Bereiten Sie eine Reaktionskammer mit flachem Boden vor, die 5 cm hoch und 1,5 cm 2 x 1,5 cm2 im Querschnitt ist. Um den Hintergrund zu reduzieren, stellen Sie sicher, dass das ausgewählte Kammermaterial nicht fluoresziert, wie z. B. Gläser (einige Kunststoffe haben Fluoreszenz).
- Wählen Sie eine geeignete Homogenisator-Dispergierspitze, die die gewünschte Scherkraft bereitstellen kann.
HINWEIS: Die Scherrate ist abhängig vom Abstand zwischen Rotor und Stator bei einer festen Drehzahl11 gemäß der folgenden Gleichung: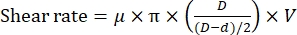
wobei μ die dynamische Viskosität von Wasser bei 20 °C ist; D ist der Innendurchmesser des Stators; d ist der Außendurchmesser des Rotors und V die Schergeschwindigkeit (U/min). - Klemmen Sie die Reaktionskammer auf den Probentisch des Fluoreszenzmikroskops, und stellen Sie die Kammer dann so ein, dass sie die Dispergierspitze des Homogenisators hält (Abbildung 1). Stellen Sie sicher, dass sich die Dispergierspitze etwas über (~ 1 mm) der unteren Oberfläche der Reaktionskammer befindet.
- Stellen Sie die vertikale Position des Homogenisators und der Kammer zusammen ein, um sicherzustellen, dass der Fokus des Mikroskops auf der Oberfläche der Dispergierspitze liegt. Stellen Sie dann die horizontale Position der Dispergierspitze ein, um sicherzustellen, dass der Erfassungsbereich (Sichtfeld) zwischen Rotor und Stator eingestellt ist (Abbildung 1).
- Schalten Sie die Fluoreszenzkanäle entsprechend dem im Experiment verwendeten Fluoreszenzfarbstoff ein.
- Verwenden Sie vor dem Hochgeschwindigkeitsscherexperiment entionisiertes (DI) Wasser, um die Scherung mit einer niedrigen Schergeschwindigkeit (z. B. 2.000 U/min) zu testen, um sicherzustellen, dass die Dispergierspitze ordnungsgemäß arbeiten kann, ohne die Reaktionskammer zu berühren.
2. Entfaltung von i-Motiven mit und ohne Liganden
- Bereiten Sie eine menschliche Telomer-i-Motiv-DNA (Table of Materials) vor, die an ihren beiden Enden mit einem Farbstoff bzw. einem Quencher markiert ist, in DI-Wasser, wie in Hu et al.11 beschrieben.
HINWEIS: i-Motiv mit Sequenz: 5'-TAA CCC TAA CCC TAA CCC TAA CCC TAA. - Die DNA wird im 30 mM MES-Puffer bei pH 5,5 oder pH 7,4 auf 5 μM verdünnt. Der DNA-Lösung wird der Ligand L2H2-4OTD, der nach Abraham Punnoose et al.13 synthetisiert wurde, in einem Konzentrationsbereich von 0-60 μM hinzugefügt. Mischen Sie die Lösung vorsichtig für 10 min, um die i-Motivstrukturen ohne Licht zu falten.
- Überprüfen und minimieren Sie die Hintergrundfluoreszenzintensität der Reaktionskammer, die mit deionisiertem DI-Wasser gefüllt ist, mit dem Fluoreszenzmikroskop ohne Scherung. Eine einfache Möglichkeit, die Hintergrundfluoreszenz zu minimieren, besteht darin, die Reaktionskammer mit DI-Wasser zu waschen. Der Hintergrundfluoreszenzwert muss später in den Datenanalysen subtrahiert werden.
HINWEIS: Streulicht sollte ab diesem Schritt vermieden werden. - Stellen Sie die Parameter der CCD-Kamera über die Software ein. Die empfohlenen Parameter lauten wie folgt: Belichtungszeit = 0,5 s, CCD-Empfindlichkeit = 1600 und Aufnahmezeit = 20 min.
- Geben Sie die DNA-Lösung mit einer langen Pipette in die leere und saubere Reaktionskammer. Decken Sie die Reaktionskammer mit einer schwarzen Box ab. Starten Sie dann den Homogenisator, um die Scherung mit einer ausgewählten Scherrate von 9.724 s− 1 bis 97.245 s−1 (ausgewählt mit der mit dem Homogenisator verbundenen Software) für 20 Minuten durchzuführen, wobei die CCD-Kamera eingeschaltet ist, um die Daten aufzuzeichnen.
- Entfernen Sie nach dem Experiment die Kammer und waschen Sie sie mit DI-Wasser.
3. Scherkraftbetätigte Klickreaktion
- Bereiten Sie i-Motiv-DNA in DI-Wasser vor. Inkubation von 10 μM i-Motiv-DNA in 300 μL 30 mM Tris-Puffer (pH 7,4), ergänzt mit 150 μM CuCl und 300 μM Ascorbinsäure für 10 min, um die i-Motivstrukturen zu falten (alle Konzentrationen sind Endkonzentrationen in der Lösung).
HINWEIS: CuCl ist der Katalysator der Klickreaktion. Ascorbinsäure verhindert die Oxidation von Kupfer (I). - Ultrafiltrieren Sie die Lösung mit einer Ultrafiltrationsvorrichtung bei einer Zentrifugalkraft von 14.300 x g. Die Lösung wird nach jeder Filtration mit 30 mM Tris-Puffer (pH 7,4), ergänzt mit 300 μM Ascorbinsäure, auf ~500 μL aufgefüllt.
- Wiederholen Sie die Filtration 3x.
- Die Restlösung wird gesammelt und ergibt ein Endvolumen von 300 μL durch Zugabe von 30 mM Tris (pH 7,4), ergänzt mit 300 μM Ascorbinsäure, zusammen mit 20 μM Calfluor 488 Azid, 20 μM HPG und 10 μM TBTA. Sobald die Reagenzien hinzugefügt wurden, bringen Sie die Lösung in die Dunkelkammer.
HINWEIS: Licht sollte nach diesem Schritt vermieden werden. - Überprüfen und minimieren Sie die Hintergrundfluoreszenzintensität der mit DI-Wasser gefüllten Reaktionskammer vor den Scherexperimenten mit dem Mikroskop. Eine einfache Möglichkeit, die Hintergrundfluoreszenz zu minimieren, besteht darin, die Reaktionskammer mit DI-Wasser zu waschen.
- Geben Sie die DNA-Lösung mit einer langen Pipette in die leere Reaktionskammer und starten Sie dann die Homogenisatorscherung mit einer Scherrate von 63.209 s−1 für 20 Minuten bei eingeschalteter CCD-Kamera .
- Entfernen Sie nach dem Experiment die Kammer und waschen Sie sie mit DI-Wasser.
Ergebnisse
Abbildung 1 zeigt die mechanische Entfaltung und Echtzeiterfassung von Ensemblemolekülen in EFS. In Abbildung 1B wurde beobachtet, dass die Fluoreszenzintensität der i-Motiv-DNA mit einer Scherrate von 9.724 s−1 bis 97.245 s−1 in einem pH-Puffer von 5,5 MES zunahm. Als Kontrolle wurde die Fluoreszenzintensität nicht erhöht, wenn die gleiche i-Motiv-DNA mit einer Rate von 63.209 s−1 in einem pH-Puffer vo...
Diskussion
Das in diesem Manuskript beschriebene Protokoll ermöglicht die Echtzeituntersuchung der Entfaltung eines Ensemblesatzes biomolekularer Strukturen durch Scherkraft. Die hier vorgestellten Ergebnisse unterstreichen, dass DNA-i-Motivstrukturen durch Scherkraft entfaltet werden können. Die Entfaltung des ligandengebundenen i-Motivs und die Scherkraft-betätigten Klickreaktionen waren Proof-of-Concept-Anwendungen für diese Ensemble-Kraftspektroskopie-Methode.
Abbildung 1
Offenlegungen
Die Autoren haben keine Interessenkonflikte.
Danksagungen
Diese Forschungsarbeiten wurden von der National Science Foundation [CBET-1904921] und den National Institutes of Health [NIH R01CA236350] bis H. M.
Materialien
| Name | Company | Catalog Number | Comments |
| 3K MWCO Amicon | Millipore Sigma | ufc900324 | |
| Ascorbic acid | VWR | VWRC0143-100G | |
| Calfluor 488 azide | Click Chemistry Tools | 1369-1 | |
| CuCl | Thermo | ACRO270525000 | |
| Dispersion tip | Switzerland | PT-DA07/2EC-B101 | |
| DNA oligos | IDT | ||
| Dye | IDT | /5Cy5/ | |
| Fluorescence microscope | Janpan | Nikon TE2000-U | |
| Homogenizer | Switzerland | PT 3100D | |
| HPG | Santa Cruz Biotechnology | cs-295271 | |
| KCl | VWR | VWRC26760.295 | |
| MES | VWR | VWRCE169-500G | |
| Quencher | IDT | /3IAbRQSp/ | |
| TBTA | Tokyo Chemical Industry | T2993 | |
| Tris | VWR | VWRCE133-100G |
Referenzen
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: Optical tweezers, magnetic tweezers and atomic force microscopy. Nature Methods. 5 (6), 491-505 (2008).
- Woodside, M. T., et al. Nanomechanical measurements of the sequence-dependent folding landscapes of single nucleic acid hairpins. Proceedings of the National Academy of Sciences of the United States of America. 103 (16), 6190-6195 (2006).
- Grandbois, M., Beyer, M., Rief, M., Clausen-Schaumann, H., Gaub, H. E. How strong is a covalent bond. Science. 283 (5408), 1727-1730 (1999).
- Strick, T. R., Allemand, J. F., Bensimon, D., Croquette, V. Behavior of supercoiled DNA. Biophysical Journal. 74 (4), 2016-2028 (1998).
- Yang, D., Ward, A., Halvorsen, K., Wong, W. P. Multiplexed single-molecule force spectroscopy using a centrifuge. Nature Communications. 7, 11026 (2016).
- Su, H., et al. Light-responsive polymer particles as force clamps for the mechanical unfolding of target molecules. Nano Letters. 18 (4), 2630-2636 (2018).
- Kirkness, M. W. H., Forde, N. R. Single-molecule assay for proteolytic susceptibility: Force-induced collagen destabilization. Biophysical Journal. 114 (3), 570-576 (2018).
- Astumian, R. D. Thermodynamics and kinetics of molecular motors. Biophysical Journal. 98 (11), 2401-2409 (2010).
- Bekard, I. B., Asimakis, P., Bertolini, J., Dunstan, D. E. The effects of shear flow on protein structure and function. Biopolymers. 95 (11), 733-745 (2011).
- Chistiakov, D. A., Orekhov, A. N., Bobryshev, Y. V. Effects of shear stress on endothelial cells: go with the flow. Acta Physiologica. 219 (2), 382-408 (2017).
- Hu, C., Jonchhe, S., Pokhrel, P., Karna, D., Mao, H. Mechanical unfolding of ensemble biomolecular structures by shear force. Chemical Science. 12 (30), 10159-10164 (2021).
- Sedghi Masoud, S., et al. Analysis of interactions between telomeric i-motif DNA and a cyclic tetraoxazole compound. ChemBioChem. 19 (21), 2268-2272 (2018).
- Abraham Punnoose, J., et al. Adaptive and specific recognition of telomeric G-quadruplexes via polyvalency induced unstacking of binding units. Journal of the American Chemical Society. 139 (22), 7476-7484 (2017).
- Dhakal, S., et al. Coexistence of an ILPR i-motif and a partially folded structure with comparable mechanical stability revealed at the single-molecule level. Journal of the American Chemical Society. 132 (26), 8991-8997 (2010).
- Hu, C., Tahir, R., Mao, H. Single-molecule mechanochemical sensing. Accounts of Chemical Research. 55 (9), 1214-1225 (2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten