Method Article
三次元細胞培養基材のUVフォトパターニングによるマルチキュー細胞微小環境の生成
要約
伝統的に、細胞培養は、 インビボで細胞の自然環境をほとんど模倣しない平面基質上で行われる。ここでは、生理学的に関連する湾曲した形状およびマイクロパターン化された細胞外タンパク質を有する細胞培養基質を作製し、これらの細胞外手がかりの細胞センシングを系統的に調査する方法について説明する。
要約
細胞外マトリックスは、細胞機能の重要な調節因子である。リガンド分布や組織形状などの細胞微小環境に存在する環境手がかりは、細胞表現型および挙動を支配する上で重要な役割を果たすことがますます示されている。しかし、これらの環境手がかりと細胞に対するそれらの影響は、多くの場合、個々の手がかりを分離するin vitro プラットフォームを使用して別々に研究され、複数の手がかりの複雑な in vivo 状況を大幅に単純化する戦略です。エンジニアリングアプローチは、 in vivo 微小環境の複雑さを捉えながら、in vitro システムの精度と操作可能性の程度を保持する実験セットアップを開発することによって、このギャップを埋めるために特に有用であり得る。
この研究は、紫外線(UV)ベースのタンパク質パターニングとリソグラフィーベースの基板微細加工を組み合わせたアプローチを強調しており、マルチキュー環境における細胞挙動のハイスループット調査を可能にします。マスクレスUVフォトパターニングにより、さまざまな明確に定義された幾何学的手がかりを含むチップ上の3次元(3D)細胞培養基板上に複雑で接着性タンパク質分布を作成することが可能です。提案された技術は、異なるポリマー材料から作られ、広範囲のタンパク質の接着パターン化された領域と組み合わせた培養基材に採用することができる。このアプローチにより、単一細胞は、単層と同様に、パターン化された基板によって提示される幾何学的手がかりおよび接触誘導手がかりの組み合わせを受けることができる。したがって、チップ材料、タンパク質パターン、および細胞型の組み合わせを用いた体系的な研究は、マルチキュー環境に対する細胞応答に関する基本的な洞察を提供することができる。
概要
インビボでは、細胞は、細胞外マトリックス(ECM)に由来する機械的、物理的、および生化学的性質のものであり得る多種多様な環境手がかりを受ける。増殖、分化、遊走などの細胞行動の調節に重要な役割を果たしている多数の環境手がかりが同定されている1,2,3,4,5。最も広く調査された現象の1つは接触ガイダンスであり、細胞外基板6、7、8、9、10、11上に存在する異方性生化学的または地形学的パターンに沿った接着媒介性細胞アライメントを記述する。細胞のアライメントを指示するだけでなく、接触ガイダンスの手がかりは、細胞遊走、細胞内タンパク質の組織化、細胞形状、および細胞運命などの他の細胞特性にも影響を与えることが示されている12、13、14、15。さらに、3Dセルラー環境の幾何学的アーキテクチャは、セル挙動に対するその調節的影響についても認められている16,17。人体では、細胞はマイクロスケールのコラーゲン線維、毛細血管、糸球体からメソスケールの肺胞や動脈まで、さまざまな湾曲した形状にさらされています18,19。興味深いことに、最近のインビトロ研究は、細胞がナノスケールからメソスケールまで、そのような物理的手がかりを感知し、応答することができることを示している20、21、22、23。
今日まで、環境手がかりに対する細胞応答を調査するほとんどの研究は、単一の手がかりを分離する実験セットアップを使用して主に行われてきた。このアプローチは、環境手がかりの細胞センシングの背後にある基本的なメカニズムの理解において驚異的な進歩を可能にしましたが、複数の手がかりを同時に提示するin vivo環境をほとんど再現していません。このギャップを埋めるには、複数の環境手がかりを独立して同時に制御できる文化プラットフォームを開発することが有用です。この概念は、マトリックス剛性と配位子密度26,27,28,29、基板剛性と気孔率30、基板剛性と3Dマイクロニッチ体積31、表面地形と接触誘導手がかり32,33,34を組み合わせた研究により、最近24,25の牽引力を高めています。、およびメソスケール曲率ガイダンスキュー23を有するナノスケール接触ガイダンスキュー。しかし、接触誘導の手がかりをさまざまな3D形状と制御された高スループットの方法で組み合わせることは依然として困難です。
この研究プロトコルは、この課題に対処し、ECMタンパク質のパターン化された接着領域(接触誘導手がかり)と基質曲率(幾何学的手がかり)の制御された組み合わせを有する細胞培養基質を作成する方法を導入する。このアプローチは、生物模倣マルチキュー環境における細胞応答の解剖を、体系的かつハイスループットな方法で可能にする。得られた知識は、複雑な環境における細胞挙動のさらなる理解に役立ち、細胞応答を所望の結果に導く特性を有する有益な材料を設計するために使用することができる。
3Dタンパク質フォトパターニング
細胞培養材料上のECMタンパク質の接着領域(接触誘導手がかり)の作成は、例えば、深紫外(deep-UV)パターニングまたはマイクロコンタクト印刷によって、様々な技術を用いて達成することができる35、36。ディープUVパターニングは、ポリマー材料上のマスクを介して投影されるUV光を利用して、細胞培養基板上の特定の位置で不動態化ポリマーを分解する。次いで、パターン化された基板を、目的のリガンドと共にインキュベートし、予め定義された位置12、37、38上で細胞付着および培養を支持する接着領域をもたらす。タンパク質パターンを導入する別の方法は、マイクロコンタクト印刷によるものであり、所望の形状を含むエラストマースタンプを任意のタンパク質でコーティングし、細胞培養基材上にプレスし、それによって細胞が接着できるタンパク質コーティングを転写する35,37,39,40.残念ながら、どちらの手法もマスク調製法とソフトリソグラフィー法に依存しているため、実験には時間と労力がかかり、パターンの柔軟性の点では限られています。さらに、深紫外パターニングとマイクロコンタクト印刷はどちらも平面材料に最も適しており、3D環境での配位子のパターニングには不可能ではないにしても技術的に困難です。
これらの従来の方法を改善するために、Waterkotteらは、マスクレスリソグラフィー、化学気相成長、および熱成形を組み合わせて、マイクロパターン化された3Dポリマー基板41を生成する。しかし、この技術は熱成形可能なポリマーフィルムの使用に依存しており、低いタンパク質パターン分解能(7.5μm)を提供し、細胞は0.1μm2,42という小さな幾何学的タンパク質パターンに応答することが報告されている。Sevcikらは、ナノおよびマイクロメートルの地形図を含む基板上のECMリガンドをナノパターン化する別の有望な方法を記載した43。マイクロコンタクト印刷を用いて、ECMタンパク質をポリジメチルシロキサン(PDMS)スタンプから温度応答性ポリ(N-イソプロピルアクリルアミド)(pNIPAM)基板に転写した。その後、pNIPAMネットワークの温度応答性により、2次元(2D)タンパク質パターンを地形PDMS基板(10-100μmの深溝)に転写することができ、それによって地形学的特徴上の接着部位の局在を制御することができた。しかし、濡れ性の低下の問題により、より深い地形基板をパターン化することがより困難になるため、すべての可能なマイクロトポグラフィーをパターン化できるわけではありません。深さ対幅アスペクト比が2.4のトレンチは、地形基板43にパターンを首尾よく転写するための究極の限界であると報告されている。さらに、さまざまなパターンの柔軟性と生成されたパターンの解像度は、マイクロコンタクト印刷の要件のために貧弱です。
本稿では、上記のボトルネックを克服し、細胞培養に使用できるマルチキュー基質を作成するための柔軟でハイスループットな方法を提示する方法について説明します( 図1参照)。曲率が ĸ = 1/2500 ~ ĸ = 1/125 μm-1 の範囲の生理学的に関連する形状(シリンダー、ドーム、楕円、およびサドル表面)は、PDMSチップで事前に設計および微細加工されています。その後、1.5μm44という小さな解像度のフォトパターニング技術を採用することにより、さまざまなデジタルパターン設計を使用して、3Dジオメトリの上に接触誘導キューが作成されます。この目的のために、PDMSチップは、細胞およびタンパク質が接着するのを防ぐために最初に不動態化される。次いで、このパッシベーション層は、光開始剤4−ベンゾイルベンジルトリメチルアンモニウムクロリド(PLPP)およびUV光露光45の組み合わせによって除去することができる。デジタルマスクは、UV露光の位置、つまりパッシベーション層が除去される領域を指定するように設計されています。タンパク質はその後、これらの領域に接着することができ、細胞付着を可能にする。パターニングは(物理的なマスクではなく)デジタルマスクを使用して実行されるため、追加のフォトマスクの設計と製造に関連する手間とコストをかけずに、さまざまなパターンをすばやく作成できます。さらに、多様な範囲のECMタンパク質(例えば、コラーゲンタイプI、ゼラチン、およびフィブロネクチン)を基板上にパターニングすることができる。このプロトコルはPDMSで作製された細胞培養チップを用いて行われるが、この原理は、関心のある任意の他の材料に適用することができる46。
プロトコル
このプロトコールに記載された研究では、初代ヒト角化細胞が使用された。この研究は、ヘルシンキ宣言の教義に従って行われました。初代角化細胞は、死亡したすべてのドナーの近親者からの同意を得た後、ETB-BISLIFEマルチティッシュセンター(オランダ、Beverwijk)の角膜部門から得られたデスメット膜内皮角層形成術から、残りのヒト死体角膜強膜組織から単離された。
メモ: このプロトコルで使用されるすべての材料、試薬、機器、およびソフトウェアの詳細については、 材料表 を参照してください。
1. 3D細胞培養基材の作製
- 目的のすべてのフィーチャを含む負のガラス型 を作成します (この場合は、フェムト秒レーザー直接書き込み技術を使用してガラスから作成します ( 図 2 参照)。
- マイナスのガラス型をペトリ皿の底に慎重に置きます。
- 空の50mL円錐形チューブをスケール上に置き、スケールを風袋に入れ、所望の量のシリコーンエラストマーベースをチューブに注ぐことによってPDMSプレポリマーを調製する。
- エラストマーベースと硬化剤の最終比が10:1(w / w)になるようにパスツールピペットを使用して硬化剤を追加します。
- ヘラを使用して円錐管内の成分を徹底的に混合する。
- 2,000 × g で 70 秒間遠心分離して、すべての気泡を除去します。
- PDMSプレポリマーをペトリ皿のネガティブガラス型の上に注ぎ、完全に覆います。
- マイナスガラスモールドとPDMSプレポリマーの入ったシャーレを真空デシケータに入れ、真空ポンプを起動します。真空に達したら、5分間待って、金型表面とPDMSプレポリマーとの界面に存在するすべての気泡を除去します。
- 真空を取り除き、ペトリ皿をデシケーターから取り出します。
- PDMSプレポリマーを65°Cのオーブン中で一晩硬化させる。
- ヘラを使用してPDMSの端を持ち上げて、新しく硬化したポジティブPDMSチップをネガティブガラスモールドから慎重に取り外します。ポジティブPDMSチップ()がネガティブガラスモールドにくっつきやすい場合は、持ち上げながらインプリントの端にエタノールまたは水を加えます。
注:流体は2つの層の間を流れ、負のガラスモールドと正のPDMSチップの分離を容易にします。 - ブレードを使用して、長方形のチップが残るように、正のPDMSチップ()の側面を切り取ります。
- 陽性PDMSチップを、シラン化剤であるトリデカフルオロ(1,1,2,2-テトラヒドロオクチル)トリクロロシランの液滴を入れた小さなバイアルの隣のデシケーターに入れ、一晩真空放置する。
メモ: シラン化により、インプリントがプロトコルの後半で他の PDMS レイヤーにバインドされなくなります。他のシラン化剤および/または方法も、PDMSチップの表面が他のPDMS層に固執しないようにするために働き得る。
警告: トリデカフルオロ(1,1,2,2-テトラヒドロオクチル)トリクロロシランは可燃性(H226)で、重度の皮膚火傷や眼の損傷(H314)を引き起こします。個人用保護具を着用し、ヒュームフードで作業し、取り扱い後はよく手を洗ってください。シラン化剤は、熱、高温表面、火花、裸火、その他の発火源から遠ざけてください。15~30°C(P280、P210、P240、P403、P235、P310)の間で保管してください。 - 真空を取り外し、プラスのPDMSチップ()をペトリ皿の底に置きます。PDMSプレポリマー(10:1)を上に注ぎ、複数の負のPDMSモールド(7)を製造します。
- ペトリ皿をデシケータに15分間真空下に置き、すべての気泡を除去します。
- PDMSプレポリマーを65°Cで一晩硬化させ、その後、ヘラを用いて負型PDMSモールドを陽性PDMSチップから剥離することができる。
- 真空デシケーター中のシラン化剤を用いて最終的な負型PDMSモールドを一晩シラン化する。
- マイナスPDMS型にPDMSプレポリマーを流し込み、デシケーターを用いて気泡を除去し、65°Cで3時間硬化させることにより、厚さ約5mmの細胞培養チップ(#4、 図2参照)を複数作製する。 カミソリの刃で、 図2に視覚化されているように、チップを最終的なサイズに切断します。チップを室温で保管してください。
メモ:表面粗さは細胞応答に影響を与える可能性があります。必要に応じて、PDMSの追加の薄層を正のPDMSチップ上のコーティングとして使用して、表面を平滑化することができる(1)。これを行うには、PDMSプレポリマーの小さな液滴をチップ上に注ぎ、加圧空気を使用してチップ全体に広げます。コーティングされたチップを65°Cで3時間硬化させ、ステップ1.12に進みます。
2. フラットPDMSサンプル(対照サンプル)の作製
- PDMSプレポリマー(10:1)をステップ1.3〜1.6に従って調製する。
- スピンコーターの中央にある丸みを帯びた真空ポストにガラスカバースリップを置きます。
- 真空をオンにしてガラスカバースリップを機械に取り付け、パスツールピペットを使用してカバースリップの中央にPDMSの液滴をピペッティングします。
- 以下のプロトコルを用いてPDMSプレポリマーをガラス基板上に分配し、厚さ約10μmの層を得た。
- スピンコート 0.45 × g、加速度: 0.2 × g/sで 10 秒。
- スピンコート 44.8 × g、加速度: 0.54 × g/sで 50 秒。
- 真空を止め、ピンセットでスピンコーターからカバースリップをはがし、シャーレに入れます。PDMSを65°Cのオーブンで一晩硬化させ、その後室温で保存する。
3. 3D細胞培養基板の基質パッシベーション
- PDMSチップの表面の水酸基をO2プラズマを用いて活性化する。ピンセットを使用して、チップをプラズマアッシャーのバスケットに入れます。
- 20 W の電力を 30 秒間使用してアッシング サイクルを実行します。N2を用いて灰化室を通気する。
- チップをバスケットから取り出し、小さなPDMSコンテナに入れます( 図1参照)。
- パスツールピペットを使用して、チップの上部に500μLのポリ-L-リジン(PLL、0.01%)を加え、表面全体がPLL溶液に浸されるようにします。室温で30分間インキュベートする。
- ピペットで細胞培養チップから 450 μL の PLL を取り出し、500 μL の 0.1 M HEPES バッファー (8 < pH < 8.5) でチップ表面を 3 回リンスします。PDMSチップ上に少量の液体を常に残して、サンプルから乾燥し、最終的なパターン品質が低下するのを防ぎます。
- 500 μLの50 mg/mLメトキシポリエチレングリコール-スクシンイミジル吉草酸塩(mPEG-SVA;MW 5,000 Da)溶液を細胞培養チップあたり0.1 M HEPES緩衝液(8注: mPEG-SVA は、溶液が完全に透明になると溶解します。
- マイクロピペットを用いてmPEG-SVA溶液450μLを除去し、チップ表面をリン酸緩衝生理食塩水(PBS)で5回洗浄した。洗浄ごとに複数回ピペットを上下させて、バインドされていないmPEG-SVAがすべて取り除かれるようにしてください。サンプルの乾燥を防ぐため、洗浄工程の間隔を最小限に抑え、洗浄には過剰(500 μL以上)のPBSを使用してください。
- PBSに浸してサンプルを保存するか、プロトコルのパターニングステップに進みます。
メモ:必要に応じて、培養キャビネットで作業し、滅菌溶液および機器で作業するときに、滅菌条件下でパッシベーションを実行できます。
4. パターン化細胞培養基材の保存
注: 3D 細胞培養基材は、プロセスのさまざまなステップで保存できます。
- 硬化PDMS細胞培養チップを室温の乾燥条件下で保存する。
- 不動態化細胞培養チップを次の2つの方法のいずれかで保存します。
- 4°CのPBSに最大7日間保存してください。
- 乾燥した状態で数ヶ月間保管してください。乾燥サンプルを得るには、PBSを除去し、二重蒸留水(ddH2O)を使用して複数回すすぎます。窒素ガンまたはエアガンを使用してブロードライします。
5. フォトパターニング用デジタルマスクの設計
メモ:3D基板のパターニングは、単一または複数の焦点面を使用して実行できます( 図3参照)。単一焦点面は、1つのデジタルミラーデバイス(DMD、約300 μm x 500 μm)よりも大きくなく、高さが高すぎない(50-100 μm)機能に使用できます。その場合は、TIFFモードを使用してデジタルパターンを設計します。1 つの DMD の寸法を超え、比較的高いフィーチャの場合は、基板のパターニングを複数のステップに分割します。この場合、複数のパターンは、すべて単一焦点面に個別に焦点を合わせるPDFモードを使用して設計されています。
- 設計ソフトウェアツールを使用してデジタルマスクを設計します。
- TIFF モードでのパターニング (ピクセル): 1,140 x 1,824 ピクセル (1 DMD の正確なサイズ、約 300 μm x 500 μm) の完全に黒いアートボードを作成し、目的の形状でアートボードを塗りつぶします。アートボードを 8 ビットの TIFF ファイルとしてエクスポートします。
メモ: パターンデザインのグレーレベルが異なると、その場所で実行される露出量が決まります。 - PDF モードでのパターニング(メートル単位):目的のサイズの黒いアートボードを mm 単位で作成し、目的の任意の形状でアートボードを塗りつぶします。3D基板を複数のフォーカルプレーンを使用してパターン化する必要がある場合は、パターンを複数のファイルに分割します。アートボードをPDFファイルとして保存します。
メモ: パターンデザインのグレーレベルが異なると、その場所で実行される露出量が決まります。
- TIFF モードでのパターニング (ピクセル): 1,140 x 1,824 ピクセル (1 DMD の正確なサイズ、約 300 μm x 500 μm) の完全に黒いアートボードを作成し、目的の形状でアートボードを塗りつぶします。アートボードを 8 ビットの TIFF ファイルとしてエクスポートします。
6. 3D細胞培養基板のUVフォトパターニング
- キャリブレーション
メモ:レーザーのキャリブレーションは、目的の材料に適切な焦点を合わせるために行われます。PDMS細胞培養基板は厚すぎてパターンスルーできないため、チップを逆さまに置いたスライドガラスを使用してください。レーザーはスライドガラスに最初に遭遇するので、ガラスを使用してレーザーを校正します。- 蛍光蛍光ハイライターをガラスカバースリップに塗布し、ガラスカバースリップを蛍光顕微鏡のステージに置きます。ハイライト表示されたサーフェスが上を向いていることを確認します。
- 顕微鏡とPRIMO機器の電源を入れ、マイクロマネージャーを開いて「プラグイン」の下でレオナルドソフトウェアにアクセスします。
- 初期メニューで キャリブレーション を選択し、顕微鏡とソフトウェアの両方で20倍対物レンズを選択します。[ 次へ]をクリックします。
- 蛍光蛍光ペン付きのスライドガラスを顕微鏡の光路に置きます。 蛍光 モードに切り替え、表示されるPRIMO画像に慎重に焦点を合わせ、ロゴとテキストの両方に焦点が合っていることを確認します。[ 次へ]をクリックしてキャリブレーション手順を完了します。
- キャリブレーション時にステージのZ位置を書き留め、この位置をプロトコルの後半で参照として使用します。
メモ: キャリブレーション材料は、プロトコルの後半でパターニングに使用される材料と一致する必要があります。上記のステップは、細胞培養基質に必要な較正を記載する。フラットPDMSコントロールサンプルのキャリブレーションには、追加のフラットPDMSサンプルの上に蛍光蛍光ペンを塗布し、ステップ6.1.2-6.1.5を使用してキャリブレーションします。
- 単一焦点面を使用した 3D フィーチャのパターニング
メモ: 単一焦点面を使用したパターニングは、1 つの DMD の寸法(約 300 μm x 500 μm)を超えない 3D フィーチャに対して行われます。フォーカルプレーンのセットアップの概略図を 図3に示します。- キャリブレーションスライドをステージから取り出し、光開始剤(PLPP)の液滴(〜50μL)を含むスライドガラスをステージ内に置きます。
警告: PLPP は目、呼吸器系、および皮膚を刺激します(R36-38)。個人用保護具を着用し、爆発物から遠ざけ、本製品に水を加えないこと、静電気放電に対する予防措置を講じること、衝撃や摩擦を避けること(S26-36)。 - PDMS細胞培養基板を光開始剤の液滴中に逆さまに置く。細胞培養基材の表面上の 3D フィーチャがスライドガラスに面し、適切なパターニングを確実にするために PLPP に完全に浸漬されていることを確認します。
- ソフトウェアで 「パターン 」を選択します。
- 顕微鏡の 明視野 モードに切り替え、ステージを目的のフィーチャに移動します。
- 凸状構造と凹状構造のそれぞれ上部または下部に焦点を合わせます(図3)。
- PRIMO を選択して、選択したパターンを挿入します。ライブ明視野画像の上にオレンジ色のパターンプレビューを確認します。
- 特徴(位置、角度、繰り返し)に応じてパターニング設定を調整し、線量1,000mJ/mm2を選択します。
- [ロック]をクリックし、顕微鏡を蛍光モードに切り替えます。
- 画面の右下にある [再生 ]ボタンをクリックして、パターン化を開始します。終了したら、緑色で表示されているパターンを観察します。
- 細胞培養チップ上のすべての特徴が完成したら、チップをパターニングスライドから取り出し、4°CのPBSに保存する。
- キャリブレーションスライドをステージから取り出し、光開始剤(PLPP)の液滴(〜50μL)を含むスライドガラスをステージ内に置きます。
- 複数の焦点面を使用した 3D フィーチャのパターニング
メモ: 複数のフォーカルプレーンを使用したパターニングは、1 つの DMD より大きい (約 300 μm x 500 μm) または比較的背の高い 3D フィーチャに対して行われます。この場合、3D フィーチャを複数のステップでパターン化する必要があります。つまり、 図 3 の例に従ってパターン デザインを適合させる必要があります。- キャリブレーションスライドをステージから取り出し、光開始剤(PLPP)の液滴(〜50μL)を含むスライドガラスをステージ内に置きます。
- ピンセットを用いてPDMS細胞培養チップを光開始剤の液滴中に逆さまに置いた。
- ソフトウェアで 「パターン 」を選択します。顕微鏡の 明視野 モードに切り替え、ステージを目的のフィーチャに移動します。
- チップの正しい位置の領域に焦点を合わせます。パターニングは単一の焦点面で実行され、フィーチャは単一のDMDよりも大きいため、複数のフォーカルプレーンを使用し、3D基板ごとに複数のラウンドのパターニングを使用して、フィーチャの完全な高さと幅に沿って十分なパターン解像度を確保します( 図3参照)。たとえば、高さが 150 μm のフィーチャの場合、フィーチャを 3 ラウンド (Z トラベル 50 μm あたり 1 つの焦点面を±) にパターン化し、フィーチャの下部から約 25 μm、75 μm、および 125 μm に焦点を合わせます。フィーチャーの下部が、ステップ 6.1.5 で書き留めた値の周りにあることを確認してください。
- PRIMO を選択して、選択したパターンを挿入します。ライブ明視野画像の上にオレンジ色で示されているパターンプレビューを観察します。
- 特徴(位置、角度)に応じてパターニング設定を調整し、線量1,000mJ/mm2を選択します。
- [ロック]をクリックし、顕微鏡を蛍光モードに切り替えます。
- 画面の右下にある [再生 ]ボタンをクリックして、パターン化を開始します。終了したら、緑色で表示されているパターンを観察します。
- 複数のフォーカルプレーンが使用されている場合は、目的のフィーチャーについてステップ 6.3.4-6.3.8 を繰り返します。
- 細胞培養チップ上のすべての機能が完成したら、チップをパターニングスライドから取り出し、4°CのPBSに保存します。
注:必要に応じて、ガラス底のペトリ皿を使用して滅菌条件下でUVフォトパターニングを適用し、滅菌培養キャビネットにすべての基板を準備します。パターン化されたサンプルをPBSに最大数週間保存します。ddH2Oで洗浄し、加圧空気を用いて乾燥させると、試料は4°Cで数ヶ月まで保存することができる。
7. タンパク質インキュベーション
注:細胞培養には、新しくタンパク質インキュベートされた基質を使用することをお勧めします。プロトコルのこの部分に進むのは、セルのシード処理(ステップ8)がその後に直接行われる場合のみです。
- パターン化された細胞培養チップを培養キャビネット内の滅菌PDMS容器に移す。
- パターニングが滅菌条件下で行われなかった場合は、パターニングされた細胞培養チップ3xを過剰の滅菌PBSで洗浄する。
- PBS中の新鮮なタンパク質溶液を調製する。
- フィブロネクチン:ローダミン標識フィブロネクチン20μgのバイアルにマイクロピペットを用いてPBS2mLを添加し、10μg/mLの濃度を得た。タンパク質凝集塊の形成を避け、光から保護するために優しくピペット。
- ゼラチン:溶解したゼラチン - フルオレセインの200 μLアリコートを解凍する。光から守る。
- マイクロピペットを用いて200〜500μLのタンパク質溶液を細胞培養チップに加える。選択したタンパク質に応じてインキュベーション時間と温度を調整します:フィブロネクチン:室温で5分、ゼラチン:37°Cで15分。 サンプルを必ず覆ってください(例えば、アルミ箔で)。
- タンパク質溶液を除去し、500μLの滅菌PBSで5倍を洗浄する。PBSを細胞培養チップのすべての関連する機能の上に複数回上下にピペッティングして、結合していないタンパク質を除去してください。
注:洗浄ステップ中は、サンプルが決して乾燥しないことが重要です。洗浄中にタンパク質溶液またはPBSを除去する場合は、タンパク質凝集塊が形成されないように、直ちに新しいPBSを追加します( 図4参照)。凸状の特徴は、基材表面より上に上昇しているため、乾燥に特に敏感です。 - オプション:蛍光顕微鏡でタンパク質パターンを確認します。サンプルを滅菌し、PBSに浸漬します。
8. 細胞播種
注:このプロトコルは、ヒト初代角化細胞およびヒト皮膚線維芽細胞を使用する。この角化細胞は、材料の二次使用に関するオランダのガイドラインに従って、患者からのヒト角膜組織から採取され、以前は角化細胞として特徴付けられていた47。これらの細胞は、5%ウシ胎児血清(FBS)、1%ペニシリン/ストレプトマイシン(P/S)、および1mM L-アスコルビン酸2リン酸セスキマグネシウム塩水和物(ビタミンC)を添加したDMEM中で、37°Cで最大4継代培養されます。ヒト皮膚線維芽細胞を購入し、10%FBSおよび1%P/Sを添加したDMEM中で、37°Cで最大15継代培養した。光パターン化された細胞培養チップ上の角化細胞および皮膚線維芽細胞の両方の播種のために、チップあたり20,000個の細胞が使用された。
- 目的の細胞を剥離し(例えば、トリプシンを使用して)、チップあたり培養培地中に10,000〜50,000細胞/mL±の細胞懸濁液1mLを調製する。基板ごとに追加されるセルの正確な数は、セルサイズと目的の読み出しに応じて調整します。
- PBSを細胞培養チップから取り出し、1mLの細胞懸濁液を加える。
- 細胞とともに細胞培養チップをインキュベーターに穏やかに輸送し、37°Cで60分間インキュベートする。
- パターン化された細胞培養チップ上の細胞の接着を明視野顕微鏡で確認します。ラインパターンの場合の細長い細胞形態を探します( 図5参照)。細胞がパターン化された領域の外側にも接着し始めた場合は、基板上の細胞の真上に培地を上下にピペッティングしてこれらを除去します。
メモ: パターン化された領域に取り付けられているセルは付着したままになりますが、パターン化された領域の外側のセルは剥離します。 - PDMS容器から細胞培養チップを取り出し、滅菌ピンセットを使用して、約5mLの培養培地で満たされた6ウェルプレートに入れます。
- 細胞を所望の時間培養する。細胞培養培地を2〜3日ごとに交換する。細胞培養後のタンパク質パターンの可視化を確実にするために、培養中のサンプルの光への曝露量を減らす。
9. 染色、画像取得、解析
- 固定と染色
- 所望の培養長の後、ほとんどすべての培地を除去し、過剰なPBSで3回洗浄する。次に、3.7%ホルマリンと共に室温で15分間インキュベートし、続いて室温で洗浄あたり5分間PBSで3回の洗浄ステップを行う。サンプルを乾燥させないでください。
注意:ホルマリンは、飲み込んだり吸入したりすると有害であり(H302、H332)、アレルギー性皮膚反応を引き起こす可能性があり(H317)、遺伝的欠陥を引き起こす疑いがある(H341)、癌を引き起こす可能性がある(H350)。個人用保護具を着用し、ヒュームフードで作業してください。 - 細胞培養基材を所望の染色剤または抗体で染色する。染色剤の量を減らすには、スライドガラス上にパイプでつながれた染色液の液滴に細胞培養チップを逆さまに置きます。
- サンプルをPBS(短期)に保管するか、4°Cでマウント媒体(長期)を使用してカバースリップに取り付けます。
- 所望の培養長の後、ほとんどすべての培地を除去し、過剰なPBSで3回洗浄する。次に、3.7%ホルマリンと共に室温で15分間インキュベートし、続いて室温で洗浄あたり5分間PBSで3回の洗浄ステップを行う。サンプルを乾燥させないでください。
- 画像取得
- 染色したサンプルをスライドガラス上のPBSの液滴に逆さまに置きます。試料を共焦点顕微鏡の段階に入れる。
- 必要な詳細レベルに応じて、適切な目的(10倍、20倍、または40倍)とZ間隔でZスタックを作成し、適切な画像取得を確保します。
- 画像解析・可視化
- 画像解析ソフトウェアで生の画像ファイルを開き、画像のプロパティ(寸法、解像度など)が正しいことを確認します。
- 必要に応じて、チャンネルごとに明るさとコントラストを調整します。
- パターンとセルを含む関心領域をトリミングします。
- オプション: 必要に応じて、デコンボリューション・ステップを実行します。
- 3D レンダリングソフトウェアを使用して、Z スタックの 3D レンダリングを作成します。
- 個々のチャンネルのコントラストとゲインの設定を最適化します。
- 3D レンダリングのイメージを作成するには、スナップショットを作成し、 としてエクスポートします。TIFF ファイル。
- 3Dレンダリングのムービーを作成するには、ムービーを登録する前に、開始フレームと最終フレーム、および時間枠を設定します。.aviとしてエクスポートします。
結果
記載されたプロトコルにより、3D PDMS細胞培養基材をUVフォトパターン化して、細胞接着に適した正確で高スループットの接着領域を作り出すことができます。このようにして、細胞は関連する基質形状と接着リガンドパターンの両方に同時にさらされる。配向、細胞面積、焦点接着の数などの細胞特性を簡単に監視し、複雑な 生体内のような環境での細胞挙動をよりよく理解するために使用できます。
3D PDMS基板上のパターニング事象を検証するために、プロトコルのさまざまな段階における材料の原子表面組成が、原子X線光電子分光法(XPS)48を使用して測定されています。要約すると、XPS測定は、パッシベーションされたサンプル上に増加した炭素シグナルを有するPEG鎖の存在を示し、これは光パターニング後に減少した。フィブロネクチンとのインキュベーションは炭素シグナルの増加をもたらし、細胞培養チップの表面上でのタンパク質接着の成功を再び示した。次に、さまざまな円パターンの凹型ピット(ĸ = 1/250 μm-1、ĸ = 1/1,000 μm-1、ĸ = 1/3,750 μm-1、図6参照)で3Dフィーチャのパターン解像度とアライメントを特徴付けました。最大強度投影から、タンパク質パターンは3つの3D特徴すべてで正常にパターン化されたと結論付けることができます。図6Aの強度プロファイルは、パターン化された領域とパターン化されていない領域との間の急激な遷移を伴う高いパターン解像度を示しています。さらに、ピット内の完全なパターン全体にわたって一貫したタンパク質強度が得られた。
ĸ=1/250μm-1の凹状のピットは単焦点面法(1パターン)を用いてパターニングし、ĸ=1/1,000μm-1及びĸ=1/3,750μm-1のピットはそれぞれ2面と3枚の焦点面(パターン)を用いてパターニングした。図 6 の最大強度投影でわかるように、どちらの方法でも、フィーチャの上にパターンが完全に配置されます。2つの異なる焦点面およびパターン間のずれた遷移は観察できない。
記載されたプロトコルを使用すると、広範囲のタンパク質パターンデザインをさまざまな形状に適用することができます( 図7 および ビデオ1を参照)。この方法の汎用性を説明するために、半円柱(凸面および凹面)、サドル面、およびピットを、様々な幅の線および円を用いてパターン化した。フォトパターン化された材料は、その後、細胞培養に使用することができる( 図7、 図8、ビデオ 2、ビデオ3、および ビデオ4を参照)。パターン化された凹状半円筒(フィブロネクチン線、赤色、幅5μm、および5μmギャップ)の凹状半円筒上で培養された真皮線維芽細胞の一例を、 図8、 図9、および ビデオ4に示す。実験中、細胞はマルチキュー細胞培養基材を感知して接着し、時間の経過とともに生存可能なままである。 図8の免疫蛍光染色から分かるように、細胞は主にフィブロネクチン株上に焦点接着(ビンクリンクラスター)を形成する。
これらの細胞培養材料を利用した別の例の研究が、最近、我々のグループ48によって発表された。この研究では、ヒト筋線維芽細胞および内皮細胞を、接触誘導手がかりおよび幾何学的地形図の組み合わせに供した。 インビボでは、両方のタイプの細胞が、ヒト血管系などの天然組織において湾曲および接触誘導の手がかりを経験する。インビ トロで 細胞を両方の環境手がかりを組み合わせた環境にさらすことによって、 インビボ の状況を反復することができ、細胞挙動に対する微小環境の役割をより深く理解することができる。ヒト筋線維芽細胞は、凹状の円筒形基板48上の接触誘導手がかり(平行なフィブロネクチン線)と位置合わせすることが示された。しかし、曲率が増加する凸構造では、幾何学的手がかりが生化学的手がかりを覆し、筋線維芽細胞が曲率の程度と徴候の両方を感知できることが示唆された。興味深いことに、内皮細胞は凹状のマルチキュー基質にのみ接着でき、凸状のPDMS基質には接着できなかった。凹状のタンパク質パターン化された基質上では、内皮細胞は接触誘導合図の方向に配向している。この基本的な in vitro 知識は、血管組織工学の分野において生理学的関連性を有し、最終的にはスマート組織工学構築物の設計を助けることができる。
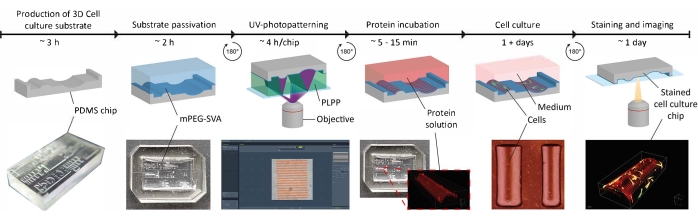
図1:3D細胞培養基材に接触ガイダンスの手がかりを適用する実験タイムライン。 まず、ポジティブな細胞培養チップは、さまざまな形状を含むネガティブPDMSモールドから製造される。未硬化のPDMSを金型に流し込み、65°Cで3時間硬化させた。 続いて、PDMSをO2プラズマで処理し、PLLおよびmPEG−SVA(青色、標識付き)と共にインキュベートして、細胞培養基材の表面を不動態化する。洗浄後、基板を光開始剤(PLPP、緑色、ラベル付き)の液滴中で逆さまに反転させ、LIMAPアプローチを使用してUVフォトパターン化する。ここでは、ユーザ定義のパターンを有するデジタルマスクを使用して、定義された位置でパッシベーション層を切断する。次に、タンパク質溶液(赤色、標識)をインキュベートすることができ、パッシベーション層が除去される位置にのみ付着する。基板上に播種された細胞は、幾何学的形状とタンパク質パターンの両方にさらされ、複雑な in vivo模倣環境での細胞挙動の研究を可能にします。略語:PDMS=ポリジメチルシロキサン;mPEG-SVA=メトキシポリエチレングリコール-スクシンイミジル吉草酸;PLPP=4-ベンゾイルベンジルトリメチルアンモニウムクロリド;LIMAP = タンパク質の光誘導分子吸着。 この図の拡大版を表示するには、ここをクリックしてください。

図2:3D細胞培養基材の製造および不動態化の間の異なる段階。 負のガラスモールドは、コンピュータ支援設計ソフトウェアで設計され、フェムト秒レーザー直接書き込み技術を使用して製造されています。このモールドは、中間の陽性PDMSチップおよび陰性PDMSモールドを製造するために利用され、その後、最終的な細胞培養チップを製造するために使用される。略語:PDMS=ポリジメチルシロキサン。 この図の拡大版を表示するには、ここをクリックしてください。

図3:2つのパターニング方法の概略図。左:UVフォトパターニングは、単一の焦点面とパターンを使用して、より小さなフィーチャ(約1つのDMD)に対して実行されます。その結果、完全な機能が一度にパターン化されます。右:より大きなフィーチャ(1つのDMDより大きい)を使用する場合、パターニングは複数の焦点面とパターンに分割されます。略語:DMD =デジタルミラーデバイス。この図の拡大版を表示するには、ここをクリックしてください。
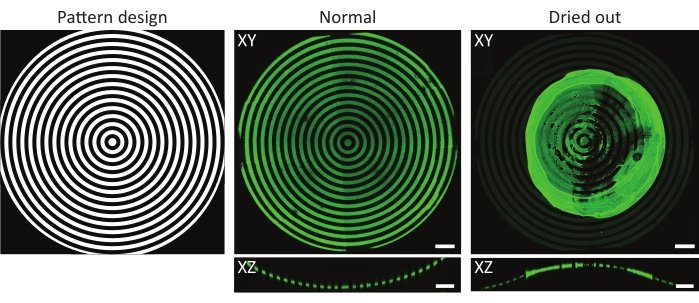
図4:通常および乾燥させたタンパク質インキュベート基質の典型的な例。 正常およびドライアウトされたタンパク質インキュベート基質の最大強度投影(XY)および直交図(XZ)。タンパク質溶液でインキュベーションした後にパターン化された細胞培養基材を洗浄する場合、サンプルを常に濡らしておくことが重要です。パターンは特徴(ĸ = 1/1,000 μm-1)上のすべての画像で同じですが、ゼラチン - フルオレセイン(緑色)は、サンプルを数秒間乾燥させたときに凝集して大きな塊を形成しました。サンプルが常に濡れたままであれば、正しいタンパク質パターンを観察することができます。スケール バー = 100 μm 。この図の拡大版を表示するには、ここをクリックしてください。

図5:シード後の明視野画像初代角化細胞(左)および皮膚線維芽細胞(右)を3D幾何学的特徴(ĸ=1/1,000μm-1の凹状ピットおよびĸ=1/500、1/375、1/250、1/175、および1/125μm-1の半円柱)上に播種してから4時間後。左上の挿入は、ジオメトリのパターン化に使用されるラインパターンを表します。白い矢印は、既に配置が示されているセルが広がっていることを示します。スケール バー = 250 μm。この図の拡大版を表示するには、ここをクリックしてください。
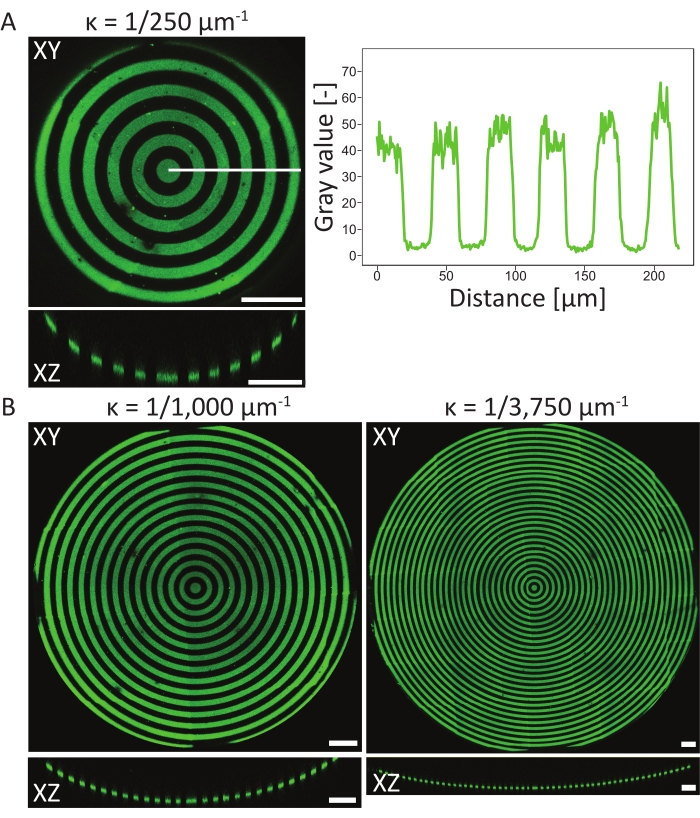
図6:凹状ピット上の円形パターンの特性評価(A)LIMAP(線幅:20μm、ギャップ幅:20μm)を用いてパターン化し、ゼラチン-フルオレセイン(緑色)と共にインキュベートした凹状ピット(ĸ=1/250μm-1)の最大強度投影(XY)および直交図(XZ)。白い線に沿った強度プロファイルが距離に対してプロットされ、一貫したパターン品質と解像度を示します。(B) ĸ = 1/1,000 μm-1 および ĸ = 1/3750 μm-1 の凹状ピットに対して追加のパターニングが行われ、パターニングに使用できる幾何学的特徴の点で柔軟性が示されます。ここでも、最大強度投影(XY)と直交ビュー(XZ)の両方が視覚化されます。スケール バー = 100 μm。この図の拡大版を表示するには、ここをクリックしてください。
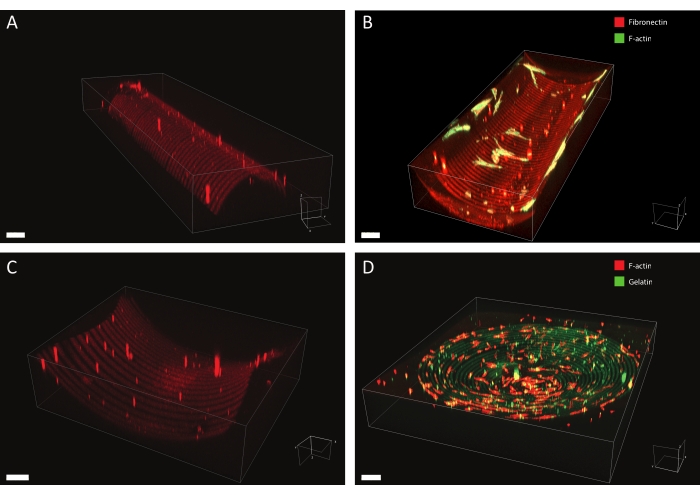
図7:パターン化された構造の3D顕微鏡データ。 光パターニングおよび細胞培養後の3Dパターン化された細胞培養材料の典型的な例を、3Dレンダリングソフトウェアを用いて可視化する。(A)幅10μmの線(ローダミン - フィブロネクチン、赤色)と10μm幅の隙間でパターン化された凸状の半円筒。(B)F-アクチン(緑色)用に染色した皮膚線維芽細胞を、幅20μmの線(ローダミン-フィブロネクチン、赤色)および20μm幅の隙間でパターニングされた凹状の半円筒上で培養した。スケールバー = 5 μm. (C) 幅 20 μm の線 (ローダミン-フィブロネクチン、赤) と幅 20 μm のギャップでパターニングされたサドル表面。スケールバー = 5 μm. (D) 幅 20 μm の線 (ゼラチン - フルオレセイン、緑色) の同心円と幅 20 μm のギャップでパターン化された凹状のピット。ヒト角化細胞のF-アクチン細胞骨格をファロイジンを用いて染色し、赤色で可視化する。スケール バー = 200 μm 。この図の拡大版を表示するには、ここをクリックしてください。

図8:光パターン化された凹状の半円筒上のヒト皮膚線維芽細胞の免疫蛍光染色(A)パターン化された凹状半円筒(フィブロネクチン線、赤色、幅5μm、およびギャップ5μm)上で24時間培養したヒト皮膚線維芽細胞の最大強度投影(XY)および直交切片(XZおよびYZ)。細胞は、F-アクチン(マゼンタ)、ビンクリン(緑色)、および核(青色)について染色される。スケール バー = 100 μm。(B) マルチキュー環境に接着しているセルのズームイン。スケール バー = 50 μm。この図の拡大版を表示するには、ここをクリックしてください。

図9:パターン状の凹状の円柱上のヒト皮膚線維芽細胞の明視野タイムラプス画像。 凹状の半円筒(ĸ = 1/250 μm-1)を平行線(幅5 μm、隙間5 μm)でパターニングし、細胞播種前にローダミン - フィブロネクチンと共にインキュベートした。タイムラプスイメージングは、最初の細胞播種から1時間後(左、0分)に開始され、細胞はまだ丸みを帯びていて接着していない(矢印)。約24時間後(中間、1,420分)、細胞はマルチキュー基板に接着し、接触ガイダンスパターンに従って位置合わせ応答を示す。アライメント応答および細胞生存率の両方が、培養期間全体(右、3,180分)を通して維持される。スケール バー = 200 μm 。この図の拡大版を表示するには、ここをクリックしてください。
ビデオ 1: 3D 円筒形基板上のパターン例。 ローダミン - フィブロネクチン(赤)でパターン化された凸状の円柱の3D表現。 このビデオをダウンロードするには、ここをクリックしてください。
ビデオ2:パターン化された3D円筒形基材(ĸ = 1/500 μm-1)上で培養した皮膚線維芽細胞の3D表現。 皮膚線維芽細胞を、パターン化された(フィブロネクチン線、赤色、幅10μm、および10μmの隙間)凸半円筒上で24時間培養した。細胞は、F-アクチン(マゼンタ)、ビンクリン(緑色)、および核(青色)について染色される。 このビデオをダウンロードするには、ここをクリックしてください。
ビデオ3:パターン化された3Dピット(ĸ = 1/3,750 μm-1)上で培養されたヒト角化細胞の3D表現。 凹状のパターン状のピット(ゼラチン円、緑色、幅20 μm、およびギャップ20 μm)で24時間培養したヒト角化細胞の3D表現。細胞はF-アクチン(赤色)について染色される。 このビデオをダウンロードするには、ここをクリックしてください。
ビデオ4:パターン状の凹状の円柱上のヒト皮膚線維芽細胞の明視野タイムラプスイメージング。 凹状の半円筒(ĸ = 1/250 μm-1)を平行線(幅5 μm、隙間5 μm)でパターニングし、細胞播種前にローダミン - フィブロネクチンと共にインキュベートした。タイムラプスイメージングは、細胞がマルチキュー環境への初期接着を示す最初の細胞播種後1時間で開始されます。完全なタイムラプスの間、細胞は主に接触誘導手がかりに沿って配向し、細胞の生存率は維持される。 このビデオをダウンロードするには、ここをクリックしてください。
ディスカッション
今日、細胞挙動は、天然の細胞微小環境の複雑さを欠いている平坦な培養基質上でしばしば研究される。足場やヒドロゲルなどの3D環境が代替として使用されます。これらの細胞培養環境はin vivoでの関連性を向上させるが、細胞挙動の体系的な研究と読み出し方法の実現可能性の両方は依然として課題である。代表的な培養基質上の細胞挙動を体系的に調査するには、顕微鏡的な読み出しを可能にする一貫したマルチキュー基質が必要です。したがって、このプロトコルでは、生理学的に関連する形状およびパターン化されたECMタンパク質を有するマルチキュー細胞培養基質を作成する方法を記載する。in vitroプラットフォーム上で組織形状や接触誘導キューなどの環境キューを組み合わせる際の主な課題は、主に技術的な性質のものです。接触誘導手がかり(例えば、ソフトリソグラフィー、深紫外パターニング、およびマイクロコンタクトプリンティング35,36)を細胞培養材料に適用する従来の方法は、平面基板用に最適化されてきた。接触ガイダンスの手がかりと組み合わせた3D細胞培養材料の必要性は、パターンアライメントの貧弱さ、解像度、柔軟性など、いくつかの技術的課題を強調しました。これらの課題を克服するために、高スループット、マスクレス、光ベースのパターニング方法を使用することができる45,49。ここで、光学顕微鏡は、正確なパターンアライメントとマイクロメートルオーダーの分解能を可能にします(図6参照)。さらに、デジタルマスクを使用することで、研究者は労働集約的な物理マスクを製造することなく、幅広いパターンで細胞の挙動を研究することができます。
UVフォトパターニングアプローチは、様々な材料48から生成される様々な3D形状(例えば、シリンダー、サドル、ドーム、ピット)と組み合わせて使用することができる。この研究で使用した3D細胞培養基質はPDMSから作られています。ただし、他の材料も使用できます。これは、目的の特徴を含む最終的な細胞培養基材を製造するために異なるステップを必要とするかもしれない。細胞は細胞培養材料の表面粗さに敏感であることが示されているので、観察された細胞の応答が3D幾何学的および接触誘導手がかりに完全に起因するように、滑らかな表面を有する細胞培養チップを作成することが重要です50,51。表面粗さの測定には、光学プロフィロメトリー、走査型電子顕微鏡、原子間力顕微鏡などの測定方法を使用できます。細胞培養材料の作製後、関心のある特徴の特定の寸法に依存する1つまたは複数の焦点面に基づくパターニング方法を選択することができる(図3参照)。通常、単一焦点面を使用して、約50μmのZ範囲内の領域をパターン化します。この経験則を使用して、パターン解決に一貫性があることが示されました(図6参照)。しかし、この方法の欠点は、複数の焦点面とパターンの導入によりパターニング時間が長くなることです。私たちの手では、複数の焦点面を使用して、最大16 mm x 16 mm x 0.17 mm(X x Y x Z)の3D幾何学的特徴が、高いパターン品質で正常にパターン化されています。
さらに、このプロトコルと組み合わせて使用できる幾何学的特徴の高さ(Z軸)には限界があることに言及することが重要です。UVフォトパターニングと多くのセルラー読み出しの両方が顕微鏡セットアップに依存しているため、対物レンズの作動距離によってフィーチャの最大高さが決まります。私たちの手の中では、高さ300μmを超える形状は依然としてUVフォトパターン化することができ、40倍の対物レンズを備えた共焦点顕微鏡を使用して読み出しが行われています。したがって、細胞内から細胞および組織スケールに至るまでのメカノバイオロジカル研究は、記載されたプロトコルを使用して可能である。
考慮する必要がある別の要因は、UV光パターニング49の間または後にサンプルが乾燥するリスクである。これは、凸部が細胞培養材料の外部に露出することが多いため、3D ジオメトリを使用する場合に特に重要です。 図 4 に示すように、これにより、目的のフィーチャの上にタンパク質凝集体が形成される不均一なパターンが生じる可能性があります。タンパク質インキュベーション後および細胞培養中の細胞培養チップの洗浄は、3D形状の適切なコーティングにとって重要です。したがって、細胞培養チップの上には、常に少量の作業溶液(PBS、PLPP、タンパク質溶液、細胞培養培地)を残すことをお勧めします。
これまでのところ、いくつかのタンパク質コーティング(フィブロネクチン、コラーゲンタイプIおよびIV、ゼラチン、FNC)および細胞型(ヒト骨髄間質細胞、ヒト筋線維芽細胞、ヒト内皮細胞、ヒト角化細胞、および皮膚線維芽細胞)が、構造化細胞培養材料に関する記載されたフォトパターニングアプローチと組み合わせて使用されてきた。以前の研究48で示されているように、タンパク質インキュベーションパラメータの最適化は、新しい細胞型における体系的調査のための鍵である。したがって、新しいタンパク質または細胞で新しい実験を行う前に、さまざまなタンパク質濃度、インキュベーション温度、およびインキュベーション時間を試験することをお勧めします。均質でパターン化された平坦な領域での初期接着後の細胞形態を、「正常な」細胞培養条件下での細胞形態と比較することにより、最適化された実験パラメータのセットを得ることができる。さらに、各細胞型は、特定のマルチキュー環境で認識可能な接着形態を示すために、播種後に異なる時間を必要とする場合があります( 図5参照)。この目的のために、ステップ8.4の洗浄中にパターン化された領域に接触事象を提示するために細胞タイプごとに必要な時間を最適化することが極めて重要である。例えば、ラインパターン上では、ヒト角化細胞は播種後最初の30分以内に細長い形態を示すが、内皮細胞および皮膚線維芽細胞は接着形態の変化を示すまでに数時間かかることが観察された。したがって、タンパク質インキュベーション(ステップ7)および細胞播種(ステップ8)に必要な実験パラメータは、選択するタンパク質および細胞タイプに依存する可能性がある。
3Dジオメトリに接触ガイダンスの手がかりを適用するための提示されたアプローチは、複雑なマルチキュー環境での細胞の動作をより深く理解するのに役立ちます。これには、焦点接着や核などの細胞内成分の調査が含まれ、提案された方法を使用して、より大きな、細胞、または組織スケールで実施される実験も含まれ得る。最終的に、得られた知識は、複雑な細胞環境が所望の結果に向かって細胞挙動を導くように設計されている組織工学アプリケーションの設計に使用することができることが期待される。
開示事項
著者らは、開示すべき利益相反はありません。
謝辞
我々は、ヒト初代角化細胞を提供してくれたネロ・フォルミサーノ博士(MERLN技術に触発された再生医療研究所)に感謝する。この研究は、Chemelot InSciTe(プロジェクトBM3.02)によってサポートされています。欧州研究評議会(助成金851960);重力プログラムのための文部科学省024.003.013「材料駆動再生」。著者は、Alvéoleの対応、ヘルプ、トラブルシューティングに感謝します。
資料
| Name | Company | Catalog Number | Comments |
| Anti-vinculin antibody, mouse monoclonal IgG1 | Sigma | V9131 | Dilution: 1/600 |
| Bovine Serum albumin, Fraction V | Roche | 10735086001 | |
| DMEM, high glucose, pyruvate | Gibco | 41966029 | |
| DMEM/F-12 + GlutaMAX (1x) | Gibco | 10565018 | |
| DMi8 epifluorescent microscope | Leica Microsystems | ||
| Ethanol | Biosolve | 0005250210BS | |
| Fetal Bovine Serum | Serana | 758093 | |
| Fiji/ImageJ, version v1.53k | www.imageJ.nih.gov | ||
| Fluorescent highlighter | Stabilo | 4006381333627 | |
| Fluorescin-labeled gelatin | Invitrogen | G13187 | Concentration: 0.01% |
| Formaldehyde solution | Merck | F8775 | |
| Glass coverslips 24 x 60 mm, #1 | VWR | 631-1575 | |
| Glass coverslips, ø = 32 mm, #1 | Menzel-Gläser | ||
| HCX PL fluotar L 20X/0.40na microscope objective | Leica | 11506242 | |
| HEPES | Gibco | 15630080 | |
| Human dermal fibroblasts | Lonza | CC-2511 | |
| Human primary keratocytes | MERLN Institute for Technology-Inspired Regenerative Medicine | ||
| Illustrator, Version 26.0.1 | Adobe | ||
| Laboratory oven | Carbolite | ||
| L-Ascorbic acid 2-phosphate sesquimagnesium salt hydrate | Sigma-Aldrich | A8960 | |
| Leica Application Suite X software, version 3.5.7.23225 | Leica Microsystems | ||
| Leonardo software, version 4.16 | Alvéole | ||
| Micro-manager, version 1.4.23 | Open imaging | ||
| Mowiol 4-88 | Sigma-Aldrich | 81381 | mounting medium |
| mPEG-succinimidyl valerate MW 5,000 Da | Laysan Bio | MPEG-SVA-5000 | Concentration: 50 mg/mL |
| Negative glass mold | FEMTOprint | ||
| NucBlue Live Readyprobes Reagent (Hoechst 33342) | Invitrogen | R37605 | 2 drops/mL |
| Penicillin-Streptomycin (10,000 U/mL) | Gibco | 15140163 | |
| Petri dish (ø=100 mm) | Greiner Bio-one | 664160 | |
| Phalloidin Atto 647N | Sigma | 65906 | Dilution: 1/250 |
| Phosphate Buffered Saline | Sigma | P4417 | |
| Plasma asher | Emitech | K1050X | |
| PLPP (photoinitiator) | Alvéole | ||
| Poly-L-lysine, sterile-filtered | Sigma-Aldrich | P4707 | Concentration: 0.01% |
| PRIMO | Alvéole | ||
| Rhodamine-labeled fibronectin | Cytoskeletn, Inc. | FNR01 | Concentration: 10 µg/mL |
| Secondary antibody with Alexa 488, Goat anti-mouse IgG1 (H) | Molecular Probes | A21121 | Dilution: 1/300 |
| Secondary antibody with Alexa 555, Goat anti-mouse IgG1 (H) | Molecular Probes | A21127 | Dilution: 1/300 |
| Spin coater | Leurell Technologies Corporation | model WS-650MZ-23NPPB | |
| SYLGARD 184 Silicone Elastomer Kit | DOW | 1673921 | |
| TCS SP8X confocal microscope | Leica Microsystems | ||
| tridecafluoro(1,1,2,2-tetrahydrooctyl)trichlorosilane | ABCR | AB111444 | |
| TrypLE Express Enzyme (1x), no phenol red | Gibco | 12604013 | |
| Trypsin-EDTA (0.05%), phenol red | Gibco | 25300054 |
参考文献
- Wang, Y., Wang, G., Luo, X., Qiu, J., Tang, C. Substrate stiffness regulates the proliferation, migration, and differentiation of epidermal cells. Burns. 38, 414-420 (2012).
- Viswanathan, P., et al. 3D surface topology guides stem cell adhesion and differentiation. Biomaterials. 52, 140-147 (2015).
- Peyton, S. R., et al. Marrow-derived stem cell motility in 3D synthetic scaffold is governed by geometry along with adhesivity and stiffness. Biotechnology and Bioengineering. 108 (5), 1181-1193 (2011).
- Vining, K. H., Mooney, D. J. Mechanical forces direct stem cell behaviour in development and regeneration. Nature Reviews Molecular Cell Biology. 18 (12), 728-742 (2017).
- Chaudhuri, O., Cooper-White, J., Janmey, P. A., Mooney, D. J., Shenoy, V. B. Effects of extracellular matrix viscoelasticity on cellular behaviour. Nature. 584, 535-546 (2020).
- Guido, S., Tranquillo, R. T. A methodology for the systematic and quantitative study of cell contact guidance in oriented collagen gels. Correlation of fibroblast orientation and gel birefringence. Journal of Cell Science. 105 (2), 317-331 (1993).
- Teixeira, A. I., Abrams, G. A., Bertics, P. J., Murphy, C. J., Nealey, P. F. Epithelial contact guidance on well-defined micro- and nanostructured substrates. Journal of Cell Science. 116, 1881-1892 (2003).
- Driscoll, M. K., Sun, X., Guven, C., Fourkas, J. T., Losert, W. Cellular contact guidance through dynamic sensing of nanotopography. ACS Nano. 8 (4), 3546-3555 (2014).
- Buskermolen, A. B. C., et al. Cellular contact guidance emerges from gap avoidance. Cell Reports Physical Science. 1 (5), 100055 (2020).
- Thrivikraman, G., et al. Cell contact guidance via sensing anisotropy of network mechanical resistance. Proceedings of the National Academy of Sciences of the United States of America. 118 (29), 1-11 (2021).
- Buskermolen, A. B. C., et al. Entropic forces drive cellular contact guidance. Biophysical Journal. 116 (10), 1994-2008 (2019).
- Vignaud, T., et al. Reprogramming cell shape with laser nano-patterning. Journal of Cell Science. 125 (9), 2134-2140 (2012).
- Pouthas, F., et al. In migrating cells, the Golgi complex and the position of the centrosome depend on geometrical constraints of the substratum. Journal of Cell Science. 121 (14), 2406-2414 (2008).
- Chen, C. S., Mrksich, M., Huang, S., Whitesides, G. M., Ingber, D. E. Geometric control of cell life and death. Science. 276 (5317), 1425-1428 (1997).
- Théry, M., et al. Anisotropy of cell adhesive microenvironment governs cell internal organization and orientation of polarity. Proceedings of the National Academy of Sciences of the United States of America. 103 (52), 19771-19776 (2006).
- Huang, G., et al. Functional and biomimetic materials for engineering of the three-dimensional cell microenvironment. Chemical Reviews. 117 (20), 12764-12850 (2017).
- Callens, S. J. P., Uyttendaele, R. J. C., Fratila-Apachitei, L. E., Zadpoor, A. A. Substrate curvature as a cue to guide spatiotemporal cell and tissue organization. Biomaterials. 232, 119739 (2020).
- Yilmaz, C. O., Xu, Z. S., Gracias, D. H. Curved and Folded Micropatterns in 3D Cell Culture and Tissue Engineering. Methods in Cell Biology. 121, (2014).
- Silver, F. H., Freeman, J. W., Seehra, G. P. Collagen self-assembly and the development of tendon mechanical properties. Journal of Biomechanics. 36 (10), 1529-1553 (2003).
- Werner, M., Kurniawan, N. A., Bouten, C. V. C. Cellular geometry sensing at different length scales and its implications for scaffold design. Materials. 13 (4), 963 (2020).
- Di Cio, S., Bøggild, T. M. L., Connelly, J., Sutherland, D. S., Gautrot, J. E. Differential integrin expression regulates cell sensing of the matrix nanoscale geometry. Acta Biomaterialia. 50, 280-292 (2017).
- Fioretta, E. S., Simonet, M., Smits, A. I. P. M., Baaijens, F. P. T., Bouten, C. V. C. Differential response of endothelial and endothelial colony forming cells on electrospun scaffolds with distinct microfiber diameters. Biomacromolecules. 15 (3), 821-829 (2014).
- Werner, M., Kurniawan, N. A., Korus, G., Bouten, C. V. C., Petersen, A. Mesoscale substrate curvature overrules nanoscale contact guidance to direct bone marrow stromal cell migration. Journal of The Royal Society Interface. 15 (145), 20180162 (2018).
- Ruprecht, V., et al. How cells respond to environmental cues - insights from bio-functionalized substrates. Journal of Cell Science. 130 (1), 51-61 (2017).
- Bao, M., Xie, J., Huck, W. T. S. Recent advances in engineering the stem cell microniche in 3D. Advanced Science. 5 (1800448), 1-16 (2018).
- Zhan, X. Effect of matrix stiffness and adhesion ligand density on chondrogenic differentiation of mesenchymal stem cells. Journal of Biomedical Materials Research - Part A. 108 (3), 675-683 (2020).
- Jiang, T., et al. Untangling the response of bone tumor cells and bone forming cells to matrix stiffness and adhesion ligand density by means of hydrogels. Biomaterials. 188, 130-143 (2019).
- Choi, J. S., Harley, B. A. C. The combined influence of substrate elasticity and ligand density on the viability and biophysical properties of hematopoietic stem and progenitor cells. Biomaterials. 33, 4460-4468 (2012).
- Rape, A. D., Zibinsky, M., Murthy, N., Kumar, S. A synthetic hydrogel for the high-throughput study of cell-ECM interactions. Nature Communications. 6 (8129), 1-9 (2015).
- Camarero-Espinosa, S., et al. 3D printed dual-porosity scaffolds: the combined effect of stiffness and porosity in the modulation of macrophage polarization. Advanced Healthcare Materials. 11 (2101415), 1-16 (2022).
- Bao, M., et al. Cellular volume and matrix stiffness direct stem cell behavior in a 3D microniche. ACS Applied Materials and Interfaces. 11 (2), 1754-1759 (2019).
- Charest, J. L., Eliason, M. T., García, A. J., King, W. P. Combined microscale mechanical topography and chemical patterns on polymer cell culture substrates. Biomaterials. 27, 2487-2494 (2006).
- Bilem, I., et al. Interplay of Geometric Cues and RGD/BMP-2 crosstalk in directing stem cell fate. ACS Biomaterials Science and Engineering. 3 (10), 2514-2523 (2017).
- Nam, K. -. H., et al. Multiscale cues drive collective cell migration. Scientific Reports. 6, 29749 (2016).
- Alom Ruiz, S., Chen, C. S. Microcontact printing: A tool to pattern. Soft Matter. 3 (2), 168-177 (2007).
- Azioune, A., Carpi, N., Tseng, Q., Théry, M., Piel, M. Protein Micropatterns. A Direct Printing Protocol Using Deep UVs. Methods in Cell Biology. 97, (2010).
- Kane, R. S., Takayama, S., Ostuni, E., Ingber, D. E., Whitesides, G. M. Patterning proteins and cells using soft lithography. Biomaterials. 20 (23-24), 2363-2376 (1999).
- Azioune, A., Storch, M., Bornens, M., Théry, M., Piel, M. Simple and rapid process for single cell micro-patterning. Lab on a Chip. 9 (11), 1640 (2009).
- Offenhäusser, A., et al. Microcontact printing of proteins for neuronal cell guidance. Soft Matter. 3 (3), 290-298 (2007).
- Ricoult, S. G., Sanati Nezhad, A., Knapp-Mohammady, M., Kennedy, T. E., Juncker, D. Humidified microcontact printing of proteins: universal patterning of proteins on both low and high energy surfaces. Langmuir. 30 (40), 12002-12010 (2014).
- Waterkotte, B., et al. Biofunctional Micropatterning of Thermoformed 3D Substrates. Advanced Functional Materials. 24 (4), 442-450 (2014).
- Lehnert, D., et al. Cell behaviour on micropatterned substrata: Limits of extracellular matrix geometry for spreading and adhesion. Journal of Cell Science. 117 (1), 41-52 (2004).
- Sevcik, E. N., Szymanski, J. M., Jallerat, Q., Feinberg, A. W. Patterning on topography for generation of cell culture substrates with independent nanoscale control of chemical and topographical extracellular matrix cues. Current Protocols in Cell Biology. 75, 1-25 (2017).
- Micropatterning: Surface functionalization. Alvéole Available from: https://www.alveolelab.com/technology/micropatterning-surface-functionalization/ (2022)
- Strale, P. O., et al. Multiprotein printing by light-induced molecular adsorption. Advanced Materials. 28 (10), 2024-2029 (2016).
- van Gaal, R. C., Miltenburg, R. P. R. S., Kurniawan, N. A., Bouten, C. V. C., Dankers, P. Y. W. Renal epithelial cell responses to supramolecular thermoplastic elastomeric concave and convex structures. Advanced Materials Interfaces. 8 (1), 2001490 (2021).
- Foster, J. W., Gouveia, R. M., Connon, C. J. Low-glucose enhances keratocyte-characteristic phenotype from corneal stromal cells in serum-free conditions. Scientific Reports. 5, 1-16 (2015).
- Van Der Putten, C., et al. Protein micropatterning in 2.5D: an approach to investigate cellular responses in multi-cue environments. ACS Applied Materials and Interfaces. 13 (22), 25589-25598 (2021).
- Melero, C., et al. Light-induced molecular adsorption of proteins using the primo system for micro-patterning to study cell responses to extracellular matrix proteins. Journal of Visualized Experiments. (152), e60092 (2019).
- Hou, Y., et al. Surface roughness gradients reveal topography-specific mechanosensitive responses in human mesenchymal stem cells. Small. 16 (10), 1905422 (2020).
- Bourkoula, A., et al. Roughness threshold for cell attachment and proliferation on plasma micro-nanotextured polymeric surfaces: The case of primary human skin fibroblasts and mouse immortalized 3T3 fibroblasts. Journal of Physics D: Applied Physics. 49 (30), 304002 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved