Method Article
腫瘍微小環境の空間免疫細胞ランドスケープのマルチプレックス免疫組織化学的解析
* これらの著者は同等に貢献しました
要約
このプロトコルは、マルチプレックス免疫組織化学を用いた腫瘍微小環境の免疫細胞特性評価がどのように行われるかを詳細に説明しています。
要約
腫瘍微小環境の免疫細胞ランドスケープには、予後および予測バイオマーカーの発見のための情報が含まれている可能性があります。マルチプレックス免疫組織化学は、空間情報を保持しながら、腫瘍組織内のさまざまな種類の免疫細胞を視覚化および同定するための貴重なツールです。ここでは、組織切片のリンパ球、骨髄、および樹状細胞集団を分析するための詳細なプロトコルを提供します。ホルマリン固定パラフィン包埋切片の切断から始まり、自動プラットフォームでの自動マルチプレックス染色手順、マルチスペクトルイメージング顕微鏡でのスライドのスキャン、自社開発の機械学習アルゴリズムImmuNetを使用した画像分析まで。これらのプロトコルは、腫瘍マーカーを切り替えるだけで、サンプルの異なるコンパートメント(腫瘍と浸潤性マージン)の免疫細胞を分析し、最近傍分析を適用するだけで、さまざまな腫瘍標本に適用できます。この分析は腫瘍サンプルに限定されず、他の(非)病原性組織にも適用できます。過去数年間の機器とワークフローの改善により、スループット時間が大幅に短縮され、診断現場でのこの手順の将来の適用が容易になります。
概要
免疫細胞は、ウイルスや細菌などの病原体に対する防御だけでなく、がん細胞に対する防御にも重要な役割を果たします1。したがって、腫瘍微小環境(TME)内の免疫系は、予後および予測バイオマーカーの発見に多くの期待を寄せています2。免疫細胞の浸潤は、さまざまな種類のがんの予後と相関していますが、これはまだ臨床ケアで実施されていません3,4。ほとんどの腫瘍タイプでは、細胞傷害性T細胞およびヘルパーT細胞の数が多い、および/または制御性T細胞の数が少ないことが良好な予後に関連しています。結腸直腸癌のTNM病期分類にいわゆる「免疫スコア」を組み込み、TNM-I病期分類に変える努力が進行中です5,6。イムノスコアは、腫瘍のコアと腫瘍の浸潤性断端(IM)の2つの異なる腫瘍領域におけるT細胞(CD3で検出)と細胞傷害性T細胞(CD8で検出)の総数から導き出されます。イムノスコアは、黒色腫、肺がん、乳がんなど、他の種類のがんでも予後的価値があることが提案されています6,7,8,9。さらに、免疫細胞浸潤は、チェックポイント遮断免疫療法10に対する応答にも相関し得る。ただし、これらの予測バイオマーカーは、臨床診療で日常的に実施する前に、前向き研究で検証する必要があります。さらに、単一のバイオマーカーでは意味のある予測には不十分であることも提案されています11。したがって、異なるバイオマーカーを組み合わせて患者サンプルの完全なマップを作成することは、いわゆる「がん免疫図」12のより包括的な予測バイオマーカーとして提案されています。
TME内の免疫細胞を研究する方法の中で、最も古く、最もよく知られている技術は免疫組織化学(IHC)であり、いくつかの疾患、特に癌13の診断検査に日常的に使用されています。この技術は、1つまたは少数のマーカー14 を長期間使用することに限られていたため、研究環境では、フローサイトメトリーや遺伝子発現プロファイリング(GEP)などの他の技術に負けていた。しかし、日常的な診断や研究で一般的に使用されるホルマリン固定およびパラフィン包埋(FFPE)腫瘍組織は、フローサイトメトリーやGEPには(最適に)適していません。さらに、GEPとフローサイトメトリーは、細胞の表現型と機能について多くの洞察を提供しますが、空間情報の欠如は大きな欠点です。したがって、腫瘍の免疫細胞浸潤領域と免疫細胞排除領域の違いなど、サンプル内の不均一性が検出されない可能性がある15。マルチプレックスIHC、イメージングマスサイトメトリー、CO-Detection by indEXing(CODEX)など、FFPE組織のマルチプレックス解析のための新しいプラットフォームが開発されており、組織切片16内で複数のマーカーを同時に検出するために使用できます。TMEの免疫細胞は、免疫療法に最適なバイオマーカーを見つけるために広く研究されています。しかし、マルチプレックス技術や自動画像解析には、それぞれにハードルがあります。
当研究室では、オパール/チラミドシグナル増幅法(TSA)法を用いたマルチプレックスIHC染色に豊富な経験を有しており、これをIHCプラットホーム上で自動化しています(材料表参照)17,18,19,20,21,22,23,24,25,26,27,28,29、30、31。私たちは、リンパ球、骨髄細胞、樹状細胞(DC)のさまざまなサブセットを検出するために、免疫細胞パネルを最適化しました。リンパ球や複雑な細胞形態(骨髄細胞やDC)など、免疫細胞の領域が密集している組織は、特に解析が難しく、存在する免疫細胞の数を過大評価または過小評価するリスクがあります。この問題を解決するために、私たちのグループ32によってImmuNet分析ソフトウェアが開発され、この機械学習パイプラインにより、これらのさまざまな種類の免疫細胞の検出品質が大幅に向上しました。ここでは、FFPE材料の取得から、さまざまな組織コンパートメントの免疫細胞密度および免疫細胞タイプ間の距離の分析までの詳細なプロトコルについて説明します。
このプロトコルは、2022年にデジタルパソロジーイメージャーが導入されて以来、ラドバウド大学医療センターでマルチプレックスIHCパネルがどのように行われているかを概説しています。記載されたマルチプレックスIHCパネルは、腫瘍マーカーとして汎サイトケラチン抗体を使用することにより、または腫瘍マーカーとしてメラノサイト関連抗体を使用することにより、異なる癌腫(例えば、肺、前立腺、結腸直腸、膀胱、乳房)に使用することができる。これらのマルチプレックスIHCプロトコールは、一次抗体の濃度、蛍光色素の組み合わせ、および染色手順の順序に関して慎重に最適化されています。私たちや他の人々は、以前にマルチプレックスIHCパネルの最適化について説明しました17,33,34,35。マルチプレックスIHCパネルは適応させることができますが、説明されている分析パイプラインを評価し、それに応じて調整または再トレーニングする必要があります。記載されている7色のマルチプレックスIHCプロトコルは、オパールの蛍光色素Opal480、Opal520、Opal570、Opal620、Opal690、Opal780、および4',6-ジアミジノ-2-フェニルインドール(DAPI)を使用しているため、「マルチスペクトルワンタッチ免疫蛍光」(MOTiF)により、イメージャーでの簡単なアンミキシングと高速スキャンが可能になります。9色染色とスキャンは、実験セットアップのさらに微調整と、液晶チューナブルフィルターを使用するイメージャーでのスキャンの別のモードが必要になるため、このプロトコルでは説明されていません。
プロトコル
このプロトコルで示されている患者資料は、以前に実施された研究の一部であり、オランダの法律と並行して、地元のRadboudumc医療倫理委員会によって医療倫理承認が免除されると公式に見なされました(ファイル番号2017-3164)30。
1. FFPE材料の収集、ブロックの選定、サンプルの調製
- 治療を行う医師や病理医を通じて、患者ファイルから FFPE ブロック識別子を取得します。倫理的な許可が必要かどうかは、地域の規制に確認してください。
- 地域の病理アーカイブまたは外部の病院からFFPEブロックをリクエストします。
注:特定の研究のために腫瘍材料または生検が取得される可能性もあります。これは、小規模な臨床試験や動物実験に当てはまります。このような場合、組織サンプルの処理は研究者の責任となる場合があります。 - 複数のFFPEブロックが利用可能な場合は、ヘマトキシリンおよびエオシン(HE)染色されたスライドを評価することにより、周囲の間質組織が存在する生存腫瘍組織を含む最も代表的なFFPEブロックを選択します(図1)。
注:この選択については、専門家の意見(病理医など)を得ることをお勧めします。FFPE ブロックの内容の評価に HE を使用できず、選択のために新しい HE を作成する必要がある可能性があります。説明については、セクション 2 に進んでください。 - 厚さ4μmのFFPEリボンをミクロトームにカットします。
注:厚さは1μmから6μmの間で、目立った染色の影響はありません。ただし、4μmが最も標準です。 - 以下のいずれかを使用して、オートステイナーの流路系に適した位置にサンプルをスライドガラスにセットします(図2A-C)。
- 蒸留した40°Cの水の表面に切片を置き、伸ばしてスライドガラスで持ち上げます。
又は
スライドガラスを40°Cの加熱プレートに置き、スライドに取り付ける場所を蒸留水の滴で覆うようにします。このドロップの上に鉗子でセクションを置き、伸ばすのを待ちます。ペーパータオルで蒸留水を吸い取り、スライドを軽くたたいて余分な水分を取り除きます。
注:スライドのラベルに近すぎる位置に組織切片を配置すると、染色が最適ではなくなります(図2D、E)。サンプルごとに6〜10枚のスライドガラスを取り付けて、さまざまなマルチプレックスIHCパネルを実行し、バックアップを取る傾向があります。
- 蒸留した40°Cの水の表面に切片を置き、伸ばしてスライドガラスで持ち上げます。
- 取り付けられたスライドガラスを56°Cで1時間、または37°Cで一晩乾燥させます。
- 実験には取り付けられたスライドガラスを使用するか、4°Cの箱に保管してください。
注:これまでの経験では、これらのマウントされたスライドは、マルチプレックスIHC染色が行われる前に何年も保存することができます。
2. ヘマトキシリンおよびエオシン染色スライドの作製
注:セクション2の次の手順はすべて、ドラフト内で実行する必要があります。
- スライドをキシレンで脱パラフィンします(2 x 5分)。
- エタノールで再水和します(99.6%1 x 5分、95%1 x 5分、70%1 x 2分)。あるいは、スライドを99.6%エタノールに3回浸します。
- スライドを蒸留水で洗います(2分)。
- 核をヘマトキシリンで染色します(10分)。
- スライドを蒸留H2Oで洗浄します(5分)。
- スライドをエオシンで染色します(5分)。
- 99.6%エタノールに3倍浸してスライドを脱水します。
- スライドをキシレンに2回浸します。
- 封入用剤を数滴加え、カバースリップで密封します。
- スライドを固め、すべての化学物質が蒸発したらスライドをヒュームフードから取り出します。
3. オートステイナーでのモノプレックスIHCとマルチプレックスIHCの実行
- 染色するサンプルの数に応じて、必要な試薬の量を計算します。
注:1回のランで、オートステイナーの容量は30枚のスライドで、6つの抗体でマルチプレックスIHCプロトコルを完了するには~18時間かかります。より多くのスライドを染色する必要がある場合は、(作業)週の毎晩、複数のバッチを入れることができます。4泊30枚のスライド=週に120枚のスライド。- 週の初めに必要なすべての試薬を準備します。オートステイナーシステムは、スライドごとに150 μLの試薬を分注します。抗体試薬とオパール試薬には6 mL滴定容器を、ブロッキング試薬と二次抗体-西洋ワサビペルオキシダーゼには30 mL容器を使用します。
注:6mL容器には便利なインサートが付いており、簡単に取り出して必要に応じて交換できます。試薬の計算では、30 mL容器または6 mL滴定容器でそれぞれ1.6 mLまたは300 μLのデッドボリュームを考慮する必要があります。 - すべてのオパール蛍光色素とジゴキシゲニン(DIG)を付属の希釈液で1:100希釈します。Opal780を抗体希釈液で1:25に希釈します。すべての一次抗体を抗体希釈液で希釈し、 Supplemental File 1で指定されている希釈液で希釈します。
- 週の初めに必要なすべての試薬を準備します。オートステイナーシステムは、スライドごとに150 μLの試薬を分注します。抗体試薬とオパール試薬には6 mL滴定容器を、ブロッキング試薬と二次抗体-西洋ワサビペルオキシダーゼには30 mL容器を使用します。
- このプロトコルに従うには、実際のマルチプレックスIHC実験を開始する前に、扁桃腺制御組織と他の(腫瘍)組織タイプの両方を含むスライドでモノプレックスIHC(補足ファイル2)を実行し、すべての試薬が適切に準備されていることを確認してください。
注:モノプレックスIHCは~3.5時間かかり、その日の終わりまでに信号パターンと強度を確認できます。特定のシグナルが弱すぎる場合(図3)、試薬を調整することができます。 - 自家蛍光補正のためには、血液やコラーゲンなどの自己蛍光構造を含む(腫瘍)組織を含むスライドを調製します。このスライドは、Monoplex IHCスライドと同時に調製しますが、ブロッキング試薬を抗体とOpal試薬に置き換えて調製します(補足ファイル3)。
注:原則として、このようなスライドは、自家蛍光補正が最適でなくなるまで、マルチスペクトルイメージングに再利用できます。ただし、脳や肝臓などの自己蛍光性の高い組織では、その組織を自家蛍光補正に使用することをお勧めします。 - マルチプレックスIHCの各ランで、1つのコントロール組織スライドを使用して29個のサンプルをオートステイナーシステムにロードし、各マルチプレックスIHCランの性能を確認します。
- マルチプレックスIHCプロトコルは、オートステイナーのウェブサイトの[ ダウンロード ]タブからダウンロードし、カスタマイズされた各マルチプレックスIHCパネル36に合うように調整する。マルチプレックスIHCについては、プロトコルの 補足ファイル4 を参照し、カスタマイズされたマルチプレックスIHCパネルについては、 補足ファイル1を参照してください。
- 染色プロトコルが完了したら、スライドをオートステイナーから取り出し、洗浄バッファーの入った容器に入れます。
- サンプルがすでに非常に低濃度で染色されているため、オートステイナーシステムがDAPIで汚染されるのを防ぐために、スライドをカバースリップで覆う前に手動でDAPIを適用してください。洗浄バッファー1 mLあたり2滴のDAPIを添加し、暗所で室温で5分間インキュベートします。
注:スペクトルライブラリを構築するには、サンプルにDAPIが染色されていないことが重要です。洗浄バッファー1 mLあたり1滴のDAPIと、RTでの10分間のインキュベーションも可能です。 - スライドをウォッシュバッファーで3回洗浄します。
- スライドをペーパータオルの上に置き、余分なウォッシュバッファーをスライドから軽くたたきます。
- 数滴の封入剤をティッシュにピペットで移します。
- 封入剤の上にガラスカバースリップをそっと置き、気泡を避けるためにスライドを斜めに覆います。
- 鉗子または清潔なピペットチップでガラスカバースリップを優しく押して、余分な封入媒体と気泡を取り除きます。
- 封入剤が固まるまで、スライドを~24時間放置し、顕微鏡スライドボード上で水平に固めるか、顕微鏡に直接ロードしてイメージングします。
- 封入剤が固まった後、またはスライドがイメージングされた後、スライドを4°Cの顕微鏡ボックスに保管します。
4. デジタルパソロジーイメージャを用いたイメージングとスキャンファイルのアノテーション
- マシンの右側にある電源ボタンを押して、イメージャーの電源を入れます。少なくとも 20 秒後、ソフトウェアを起動します。
メモ: ハードウェアが正しく起動するまで 20 秒待ちます。 - スライドを4枚ごとにカセットにセットします。
- オプション: テンプレートをダウンロードできる .csv ファイルにスライドを入力します (補足ファイル 5)。.csvファイルをプログラムにロードするには、C:\ Users \ Public \ Akoya \ VectraPolaris \ Statesに保存します。
注意: 最大20個のカセットまたは80枚のスライドを同時にロードできます。
- オプション: テンプレートをダウンロードできる .csv ファイルにスライドを入力します (補足ファイル 5)。.csvファイルをプログラムにロードするには、C:\ Users \ Public \ Akoya \ VectraPolaris \ Statesに保存します。
- リファレンス設定
- メインメニューから [Check Dashboard ]を開きます。
注意: リファレンススライド付きのカセットはメーカーから提供されており、オプションでスロット20に恒久的に保持できます。 - 製造元の指示に従って、付属のスライドに明視野基準を週に一度設定します(数分かかります)。
- 製造元の指示に従って、付属のスライドに蛍光基準を月に1回設定します(1時間以上かかります)。
- メインメニューから [Check Dashboard ]を開きます。
- プロトコールの作成または調整
- メインメニューに戻り、[ プロトコルの編集 ]をクリックしてプロトコルを作成します。
- [新規...] をクリックし、[イメージングモード] として [蛍光]、[マルチスペクトル スライド スキャン]、および [染色] オプションで [Opal Polaris 5, 6, 7 色] を選択します。
- プロトコル名(Protocol Name)でプロトコルに名前を付け、Available Studies(利用可能なスタディ)からスタディを選択してスタディに保存します。または、Create New Study |調査名。
- [ Create Protocol] を選択して終了します。
- このタイプのスキャンでは、左側のウィンドウの マルチスペクトルスライドスキャン設定のみを使用します。右側の マルチスペクトルフィールド設定のウィンドウは無視してください。
- スライドをさまざまな倍率でスキャンします。このプロトコルに従うには、 ピクセル解像度 を 0.50μm(20倍)のままにして、20倍の倍率でスキャンします。
- 「スキャン露出」を選択して、露出時間を設定します。
- スライドが保持されているカセットをロードするには、[ Load Carrier ]オプションで正しいスロットを選択します。
- スライド内を移動するには、「Take Overview」を選択して、キャリアがロードされた後にスライドを含むキャリアの概要画像を取得します。これを自動的にオンまたはオフにするには、右上の歯車アイコンをクリックし、[設定...]に移動し、[ナビゲーション概要画像]の下のオプションをオンまたはオフにして、[インタラクティブタスクの読み込み時に自動的に画像キャリア]を有効にします。
- 対応するモノプレックスIHC染色スライドのフィルターごとに露光時間を設定するには、[ Set Scan Exposures ]を選択し、正の信号を持つさまざまなスポットを見つけます。手動でピントを合わせるか、 オートフォーカス を使用して、その信号に対応するフィルターに切り替えた後に オートエクスポージャー を選択します。露出オーバーを防ぐために最も低い露出時間を選択し、すべての露出時間を設定した後で参照用に各スライドのスナップショットを撮ります(図3)。
注: このタイプのスキャンでは、[ フィールド露出の設定] オプションは無視してください。 - マルチプレックス染色スライドの露光時間を設定するには、信号が正のいくつかの場所にあるすべてのフィルターを確認します。最低自動露出時間を10%短縮して露出過多を防ぎ、すべての露出時間が設定された後にいくつかのスナップショットを撮ります。
- 自家蛍光補正のために未染色スライドのスナップショットを撮影するには、 Sample AFフィルター を使用してナビゲートします(図3H)。
注:赤血球とコラーゲン構造を持つ場所は興味深いものです。Opal480フィルターの露光時間は、強い自己蛍光領域のために短縮する必要があるかもしれません。Opal480の信号が十分に強い場合でも、独自のサンプルAFフィルターが実装されているため、自己蛍光構造から十分に分離する必要があります(セクション6を参照)。 - ソフトウェアを使用して染色とイメージングの品質を評価します(セクション5および6を参照。 図4、 補足ファイル6:補足図S1、補足図S2)。
- 「保存...」ボタンを選択して、プロトコルとその調整された露出時間がプロトコルに保存されていることを確認します。
注:プロトコルがすでに保存されている場合、これまでのところ、ソフトウェアによって未保存の調整に関する追加の通知は提供されません。
- スライドの自動スキャン
- メインメニューに戻り、[ スライドのスキャン] をクリックしてスライドをスキャンします。
- スライド名/ID と対応するタスクとプロトコルを [Configure Tasks] に手動で入力するか、以前に作成した .csv ファイルから [Load Setup] で自動的に入力します。
- [スキャン] をクリックしてスキャンを開始します。
- スキャン設定を保存するためのウィンドウがポップアップするのを待ちます。「 保存 」をクリックしてデフォルト設定を使用し、スキャンを開始します。
注:この方法を使用したスキャンには、スライドごとに10~20分かかります。スライドの数にもよりますが、スキャンには最大で丸一日かかる場合があります。 - エラーメッセージを探して、すべてのスライドでスライドのスキャンが成功したかどうかを確認します。スキャンが成功したかどうかを確認するには、スキャンされた アコヤ全体のスライドスキャンファイル (.qptiff)とスキャンされた組織全体を保存します。
5. スライドビューアによるデータのアノテーション
- メインメニューに戻り、[ フェノチャートの起動 ]をクリックしてスライドビューアを開きます。
- スキャン ファイルが直接表示されない場合は、まず右上隅の 歯車アイコンをクリックして 場所を指定し、[ ブラウザの場所の変更] に移動して、目的のデータセットの .qptiff ファイルの 1 つをランダムに選択します。
注:データはデフォルトでD:\ Data \ VectraPolarisに保存されます。 - スライドをロードするには、スライドを選択して右上隅の「 ロード 」をクリックするか、スライドをダブルクリックします。
- 右上隅の [ログイン ]ボタンをクリックしてログインします。
注:ユーザー名はイニシャルまたは名前のみにすることができ、誰がどの注釈を付けたかを追跡するために使用されます。 - アンミキシングを実行するには、上部の[ アンミキシング ]ボタンをクリックし、[ オパール+AF ]オプションを選択します。
注:これは、Opal 480チャンネルの近くの自家蛍光シグナルの一部を取り除くのに役立ちますが、すべてではありません。 - データをバッチ処理するためのアルゴリズムを生成するには、inForm Projects 1 x 1画像用(画像サイズ:928 μm x 696 μm)オプションを使用したスタンプを使用して、代表的な画像を選択します。
注:腫瘍、間質、背景、およびさまざまな種類の免疫細胞を含むいくつかの代表的なスタンプがデータセット全体で選択され、最終的に20〜30枚の画像が得られます。 - 組織内で何を分析する必要があるかに応じて、 ROI オプションを使用して関心領域を選択し、 inForm Batchを選択します。腫瘍から離れすぎている画像や背景にある画像など、分析する必要のない画像を手動で削除します。
注:腫瘍全体の周囲にROIを描画し、腫瘍領域から1つの追加の画像を選択して、~0.5mmのIMを分析できるようにする傾向があります。
描画されたROIが比較的小さい場合、ROIは2〜9個のマージされた20x画像で構成されます。これは私たちにとって好ましくないので、これを回避するために、目的の組織(inForm Batch用に選択)を手動でスタンプします。 - 注釈付けが終了したら、注釈を自動的に保存し、次のスライドを読み込みます。
- 注釈付けプロセス中に、スライドが正しくスキャンされているかどうかを確認します。
- .qptiffファイルが見つからない場合、またはスライドが正常にスキャンされない場合は、スライドに組織が存在するかどうかを確認し、70%エタノールでスライドをクリーニングして、再度スキャンします。
- 組織が完全にスキャンされていないため、潜在的に重要な(腫瘍)領域が欠落している場合、または重要な領域のスキャンが焦点が合っていない場合は、スライドを70%エタノールで洗浄し、再度スキャンします。
注意: どちらの場合も、カバーガラスの上部にあるマーカーで組織を囲むと、システムが組織を見つけてスキャンを再試行するのに役立ちます(補足ファイル6:補足図S3)。私たちの手の中では、細い赤いマーカーが太い黒いマーカーよりもうまく機能しました。
- すべてのサンプルのスキャンと注釈付けが完了したら、データを別のコンピューターまたは外部ディスクに保存してバックアップします。
6. スペクトルアンミキシング
- inForm自動画像解析ソフトウェアを開きます。
- 画像をソフトウェアにロードします ファイル |画像を開く; .qptiff ファイルを選択します。ステップ5.6で inForm Projects とマークされたスタンプをプロジェクトにロードします。
- 自家蛍光補正用に画像化された .qptiff ファイルを読み込みます。
- 自家蛍光を補正するには、 画像上の自家蛍光選択 ツールを使用して、赤血球やコラーゲンなどの自家蛍光であるさまざまなタイプの構造を通る未染色のスライドから画像上に線を引きます。
- [ Edit Markers and Colors... ] セクションで、Opal fluorophore に対応するマーカー名を割り当て、色を好みのものに調整します。
- 蛍光色素の混合を解除するには、左下隅にある「 すべて準備 」を選択します。
- 画像を調べて、すべての信号が画像に表示されているかどうか、およびアンミキシングがうまくいったかどうかを確認します。 目玉のアイコン を選択して、すべてのマーカーを1つずつオフにしてからオンにして、品質を確認します。
- オプションで、組織セグメンテーション、細胞セグメンテーション、表現型解析のアルゴリズムを学習させます。
- 「エクスポート」タブに移動し、「エクスポートディレクトリ」の下にある「参照...」ボタンをクリックして、新しい空のエクスポートディレクトリを作成します。
- [エクスポートする画像]で、[合成画像]と[コンポーネント画像(マルチ画像TIFF)]を選択します。
- [ファイル] |保存する |[投影] を使用して、アルゴリズムを特定の場所に保存します。
- スライドのバッチ処理のために、左側の垂直な [バッチ分析 ]タブに移動します。
- [エクスポート オプション] で [アイテムごとに個別のディレクトリを作成する] を選択します。
- 分析用のスライドを追加するには、[スライドの追加] ボタンの下にある .qptiff ファイルを選択し、バッチ分析にロードします。
- 「実行」を選択して、スライドのバッチ処理を開始します。
7. ROIの描画
- セクション 6 のコンポーネント ファイルのみを含むフォルダを作成しますが、階層フォルダ構造はそのままにします (コンポーネント ファイルは sample/slide という名前のフォルダにあります)。
- QuPathホールスライドビューアソフトウェアを開きます。
- 左側の[ プロジェクトの作成 ]をクリックし、適切な名前で新しい空のフォルダーを選択/作成します。
- [自動化] をクリックし、[スクリプト エディターの表示] を選択します。
- 補足ファイル 7 にあるスクリプトをコピーして貼り付けます。34 行目で、すべてのコンポーネント ファイルを含むスライド フォルダー (手順 7.1 で作成したフォルダー) の場所に変更します。
- スライドのバッチステッチが終了したら(翌日以降) 実行して戻る を選択して続行します。
- 生成された.ome.tifファイルをQuPathプロジェクトにドラッグし、プロジェクトとして保存します。
- 新しいウィンドウが自動的にポップアップしたら、[ 画像の種類を設定] |Fluorescence をクリックし 、[Import] をクリックします。
- 左側のメニューで、サンプルのリストを確認します。いずれかをダブルクリックしてサンプルを開きます(図5A)。
- チャンネルの強度を調整して見やすくするには、 コントラストアイコンをクリックします。
- すべてのチャンネルを選択し、「 リセット」をクリックします。
- 自家蛍光をオフに切り替えます。
- 腫瘍のROIの描画を開始するには、 コントラストアイコンをクリックして [ グレースケールを表示]を選択します。腫瘍マーカーチャネルを選択し、強度を調整して最適に見えるようにします(図5B)。
- ブラシツールをクリックして、腫瘍のROIを大まかに描画します。
- ワンドツールを選択しながら、Altキーを押しながらROIの外側をクリックすると、ROIが外側から滑らかになります(図5C)。
- 同じROIで分離された腫瘍片をマージします。
- ROI に適切な名前を付けます (tumor など) 左側のリストで注釈を右クリックします。 [プロパティの設定] を選択し 、名前を入力します。
- IM の ROI を作成するには、次を選択して腫瘍領域から既存の ROI を拡張し ます。注釈... |注釈を展開します。
- 拡張半径のサイズを選択し、[ 内部の削除 ] と [ 親に制約] を選択します (図 5D)。
- コントラストアイコンをクリックし、自家蛍光チャンネルを選択し、強度を調整して最適に見えるようにします。
- ワンドをクリックし、Altキーを押しながらROIを調整して、ROIを外側から滑らかにし、このROIの一部にならない背景を削除します。
- ROI に適切な名前を付けます (侵 襲的マージン や IM など) 。その中には、左側の一覧で注釈を右クリックし、 [プロパティの設定] を選択して名前を入力し、必要に応じて色を緑色に変更します。
- 注釈を保存します: ファイル |オブジェクトのエクスポート |すべてのオブジェクトをエクスポートし、デフォルトの選択で [FeatureCollection としてエクスポート] で [OK] をクリックし、任意の場所に保存します。
8. 免疫細胞の検出
- ImmuNetは、学習と推論の両方にコンポーネントデータ(マルチチャネルTIFFファイル)を使用するため、アノテーションを学習セットと検証セットに分割します。モデルをトレーニングするには、リポジトリの Readme ファイルに記載されている手順に従い、サンプル データセットと注釈を目的のデータに置き換えます。異なる免疫細胞とは別に、関心のある細胞として認識されるべきではない部位(腫瘍細胞、他の細胞、または「細胞なし」(関心のある細胞と混同される可能性のある構造)に 背景 注釈を付けることにより、モデルに否定的な例を提供します。詳細については、ImmuNetの出版物を参照してください32。
- 検証アノテーションを使用して、パフォーマンスが十分であることを確認します。アノテーションタイプごとのエラー率(モデルが検出しなかった検証アノテーションの割合)を見てみて、最もわかりやすい評価指標です。誤検出に関するパフォーマンスを評価するには、完全に注釈が付けられたROIをいくつか作成し、Fスコアを計算します。
- 定量的な評価だけでなく、予測を目視で検証することで、モデルがどのような誤差を生じやすいのかを定性的に把握します(図6、補足ファイル6:補足図S4、補足図S5)。モデルのパフォーマンスが不十分であると判断された場合は、リポジトリに記載されているように一部のタイルの予測を視覚化し、最もエラーが発生しやすいサイトを確認します。そのようなサイトでより多くの注釈を作成し、モデルのトレーニングと評価を再実行します。
- 目標のパフォーマンスが達成されたら、リポジトリの Readme の 「データセット全体の推論 」セクションで説明されているように、データセット全体に対して推論を実行します。モデル予測を含む取得した.csvファイルをデータ分析の入力として使用します(そのためにPythonまたはRスクリプトを記述します)。
9. 予測表現型とデータ分析
注:このセクションでは、ImmuNet(セクション8)で同定された免疫細胞の位置とQuPath(セクション7)で示されたROI(セクション7)を組み合わせたリンパ球パネルで染色した単一のメラノーマサンプルの簡単なデータ解析の例を示します。解析は R 4.1.1 で実行されています (スクリプトは 補足ファイル 8 として提供されています)。このスクリプトには、パッケージ plyr 1.8.8、dplyr 1.0.8、tidyr 1.2.0、sf 1.0-7、ggplot2 3.4.0、RANN 2.6.1、および RColorBrewer 1.1-2 が必要です。これらは install.packages() コマンドでインストールできます。入力として、ImmuNetのサンプル予測を含む.csvファイルと、QuPathからエクスポートされたROIを含むファイルを取ります。手順 9.1 から 9.6 では、指定されたスクリプトで実行される 1 つのサンプルの分析について説明し、セクション 9.7 から 9.9 では、複数のサンプルの分析のオプションについて説明します。
- ImmuNet の予測を R に読み込んだ後、表現型を定義するマーカーを相互にプロットし、母集団を最適に分離するしきい値を選択することにより、予測マーカー発現のしきい値を決定します。
注:特定のサンプルに使用されたゲーティング戦略を 図7Bに示します。骨髄系および樹状細胞パネルのゲーティング戦略は、 補足ファイル6:補足図S6および補足図S7に示されています。 - 閾値を決定したら、それらを使用して、各ImmuNet予測にパネルで定義された表現型を割り当てます。一部の予測では、予測されたマーカーのいずれも閾値を上回っていないか、閾値化後に発現すると考えられるマーカーの組み合わせに一貫性がない可能性があることを観察します(例:リンパ球パネルにおけるCD3+ CD20+ 予測)。ステップ 8.3 で良好なモデル性能が達成された場合、そのような予測の割合は小さくなります。分析の前にそれらを除外します。
- QuPathで描画された腫瘍とその浸潤性マージンの最大100μmのROIを個別に分析するには、対応するGeoJSONファイルをRにロードし、各予測について、予測が当てはまるROIを決定します。
- 健全性チェックのため、および探索的データ分析の一部として、サンプル中に見つかった免疫細胞を、対応するROIとROIの境界で別々に視覚化します(図7A)。
- 次に、ROIごとに異なる免疫細胞の密度を個別に計算します。特定のサンプルで見つかった密度を 表1に示します。
- 複数のサンプルが利用可能な場合は、細胞密度の分布を可視化します。密度値を対数変換して、正規分布の値を実現します。
注:特定の表現型のカウントが0の場合、これらはLog変換できず、欠損値につながります。この問題を解決するために、ラプラシアン平滑化を適用するには、まずすべてのセル数に0.5を加算してから、表面積で除算します。 - 密度値を分析し、選択したソフトウェアを使用してプロットします(図8)。
- セルの位置が保持されているため、空間解析が可能になります。例えば、検出された免疫細胞ごとに最近傍細胞を見つけ、次に表現型ごとに、異なる表現型が最近傍として発生するケースの割合を計算します。
注:このサンプルで見つかったナチュラルキラー(NK)細胞の数は非常に少なかったため、この分析から除外しました。腫瘍とIM ROIについて得られた結果を 図9に示します。
結果
腫瘍組織を含むFFPEブロックは、病理レポートとHE染色スライドに基づいて選択されました。患者から複数の腫瘍病変を切除した場合、および/または腫瘍サンプルが大きい場合、これらは複数のFFPEブロックに分割されます。私たちは、腫瘍コンパートメントと腫瘍の浸潤性断端(IM)として知られているものの両方で免疫細胞を分析することを好みます。IMは、腫瘍に隣接する非癌性間質組織です。したがって、1つの腫瘍サンプルに複数のFFPEブロックがある場合は、両方の組織タイプを含むFFPEブロックが選択されます。HE染色スライドに見られるように、1つのFFPEブロックには腫瘍組織と腫瘍に隣接する間質組織が含まれていました(図1A)。同じ腫瘍の別のFFPEブロックは、周囲の間質組織をはるかに少なく含んでいました(図1B)。ただし、一部の組織サンプルでは、FFPEブロックに選択肢がないか、IMがどのFFPEブロックにも存在しません。これは(針)生検によく当てはまり、データの解釈時に留意する必要があります。
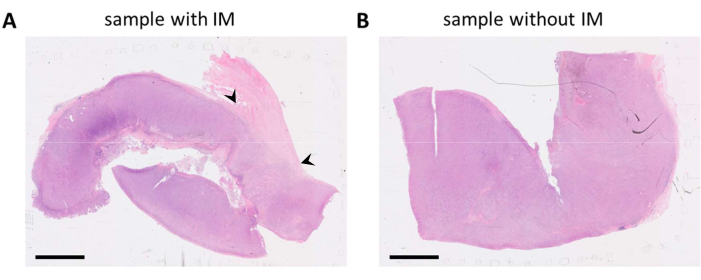
図1:黒色腫腫瘍検体のHE染色スライド(A)サンプルの右上隅に腫瘍(IM)に隣接する間質組織を持つ腫瘍サンプルの例(黒い矢印で示されています)。(B)サンプルに間質組織がほとんどまたはまったく存在しない同じ腫瘍病変からの別のサンプル。スケールバー= 5 mm。略語:HE =ヘマトキシリンとエオシン。IM = 侵襲的マージン。この図の拡大版を表示するには、ここをクリックしてください。
提案された7色パネル(Supplemental File 4)によるマルチプレックスIHC染色は、3日間の染色プロセス(通常の労働時間を考慮した)で手動で行うか、または自動染色器で一晩行うことができる。オートステイナーを使用する場合、システムの最適な流路系を可能にするために、セクションをスライドガラス上の特定の場所に取り付ける必要があります(図2A)。切片がスライドに正しく取り付けられている場合(図2B)、それらは均一に染色されます(図2C)。切片がスライドガラスに最適に取り付けられていない場合(図2D)、オートステイナーの流路系が(完全な)組織に到達しないため、染色パターンが最適でない結果になることがよくあります(図2E)。これは、サンプルが非常に大きい場合や、この問題に気づいていない人によってマウントされたスライドが提供された場合に発生する可能性があります。このような場合は、スライドの染色が良好な部分のみを分析対象として選択する必要があります。これらのタイプのサンプルの別の選択肢は、液体を最適に広げるために手動で染色することです。
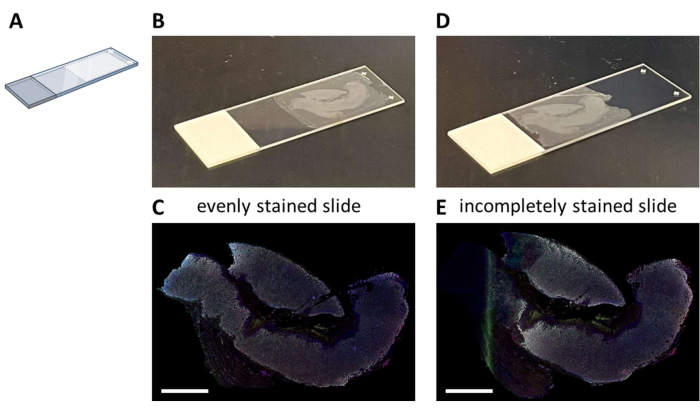
図2:FFPEセクションのスライドガラスへの取り付けと衝撃 (A)オートステイナーでの最適な染色のためにスライドガラスのどこに取り付けるかの概略図。(B)正しく取り付けられたスライドの例。(C)スライドを正しく取り付けると、組織切片が均一に染色されます。(D)最適でない取り付けスライドの例。(E)最適でないマウントされたスライドは、この写真の左側に見られるように、不完全な染色組織切片をもたらす可能性があります。スケールバー= 5 mm。略称:FFPE=ホルマリン固定およびパラフィン包埋。 この図の拡大版を表示するには、ここをクリックしてください。
大規模なマルチプレックスIHC実験を複数回の染色ラウンドで行い、大量の溶液を調製する必要がある場合は、マルチプレックスIHCに進む前に、まずこれらの試薬をモノプレックスIHCランで試験するのが最善です。Monoplex IHCは、デジタルパソロジーイメージャーで予想される染色パターンをチェックし、露光時間はコントロールスライド上の対応するフィルターで設定されます(図3A-H)。扁桃腺組織は、ほとんどの免疫細胞マーカーのポジティブコントロールとして使用されます。扁桃腺コントロール組織におけるDAPI曝露時間は常に他の組織よりも長いため(図3G)、DAPI曝露時間は研究する組織タイプに設定する必要があります。このタイプのスキャンでの通常の露光時間は、蛍光色素分子とフィルターによって異なりますが、1 ms〜30 msです(図3I)。モノプレックスIHCがこれらの数値を超える場合、または染色パターンが思ったほど明確でない場合は、抗体溶液を調整または交換する必要があります。ここに示す例では、FOXP3(図3Cおよび図3I)の濃度を上げて、強度を他のマーカーとより一致させることにしました。自家蛍光は、扁桃腺対照組織よりも他の組織で強くなる場合があります。私たちの設定では、サンプルAFフィルターの露光時間は25ミリ秒から50ミリ秒の間です(図3H、I)。

図3:モノプレックスIHCおよび未染色対照サンプルの露光時間の設定(A)扁桃腺対照組織におけるCD20-オパール480シグナル。(B)扁桃腺制御組織におけるCD3-オパール520シグナル。(C)扁桃腺コントロール組織におけるFOXP3-オパール570シグナル。(D)CD56-扁桃腺コントロール組織におけるオパール620シグナル (E)CD8-扁桃腺コントロール組織におけるオパール690シグナル。(F)腫瘍マーカー - 扁桃腺コントロール組織におけるオパール780シグナル。(G)扁桃腺コントロール組織のDAPIシグナルは、多くの場合、目的の組織タイプよりも弱いです。(H)自家蛍光-腫瘍対照組織におけるサンプルAF信号。(I)10%で調整し、マルチプレックスIHC染色スライドを確認する前の露光時間のスクリーンショット。スケールバー = 100 μm。略語:AF =自家蛍光;IHC = 免疫組織化学;DAPI=4'6-ジアミジノ-2-フェニルリンドール。この図の拡大版を表示するには、ここをクリックしてください。
マルチプレックスIHCを実行した後、モノプレックスIHC設定から、いくつかのマルチプレックスIHCスライドをチェックし、自動露出を選択することにより、露光時間を調整します。このタイプのスキャンでは、飽和保護オプションがないため、露出を高く設定しすぎないようにして、露出過多を避けることが非常に重要です。露出オーバーは、蛍光色素のスペクトルアンミキシングを妨げます。多くの場合、モノプレックスIHCに基づく露光時間を超える露光時間は設定せず、マルチプレックスIHCでより強いマーカーの露光時間のみを減少させます(図3Gおよび図4A)。いくつかのスライドで異なる場所に自動露光することで、いくつかのフィルターの露光時間がまだ長すぎることがわかります。これらは、自動露出設定を使用するときに観察される最小の数に調整し、他の見えない場所での露出過多を防ぐために、値のさらに10%を差し引く必要があります(図4A)。この方法では、特定のフィルターの露光時間をモノプレックスIHCに設定されたものよりも短くすることができます。しかし、マルチプレックスIHC実験が成功すれば、少なくともコントロールスライド上では、すべてのマーカーが観察可能になるはずです(図4B-H、補足ファイル6:補足図S1、および補足図S2)。特定のマーカーがすべてのサンプルに存在するとは限らないことを考慮してください。少なくとも扁桃腺切片を含むコントロールスライドを含めることにより、標準パネルのすべてのマーカーと信号強度の良好な染色を確認できます。
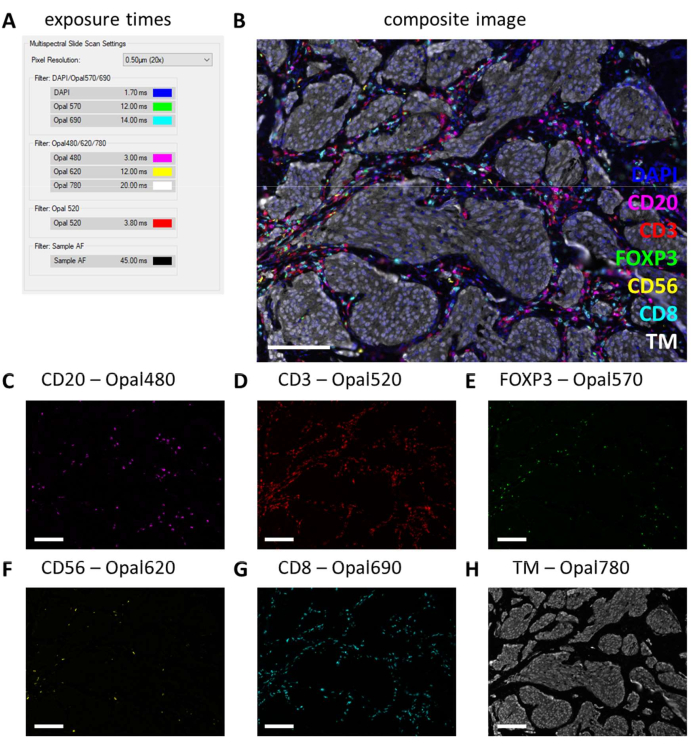
図4:黒色腫腫瘍検体中のリンパ球パネルを用いた切片の染色に成功した場合の例。 (A)このマルチプレックスIHCサンプルの記録に使用した曝露時間。(B)腫瘍組織内のマルチプレックスIHCリンパ球パネルの合成画像。(C)CD20 - マゼンタのオパール480信号。(D)CD3 - 赤のオパール520信号。(E)FOXP3-緑色のオパール570信号。(F)CD56 - 黄色のオパール620信号。(G)CD8 - シアンのオパール690信号。(H)TM-白のオパール780。スケールバー = 100 μm。略語:TM =腫瘍マーカー;IHC = 免疫組織化学。 この図の拡大版を表示するには、ここをクリックしてください。
マルチプレックスIHCスライドは、デジタルイメージャーによって完全にスキャンされます。後続の分析用のタイルは、スライドビューアで選択されます。ただし、腫瘍とIMなど、より具体的な領域を分析する必要がある場合は、QuPathを使用してこれらの関心領域(ROI)を描画できます。スライド ビューアで選択したタイルのバッチ処理が完了すると、コンポーネント ファイルが再びマージされます (図 5A と 補足ファイル 7)。腫瘍マーカーチャネル(図 5B)と QuPath の魔法の杖ツールを使用して、腫瘍の輪郭を追跡して「腫瘍 ROI」(図 5C)を形成できます。次に、腫瘍のROIを一定の距離(この場合は500μm)で拡張して、「侵襲的マージンROI」を作成することができます(図5D)。不要なバックグラウンド(非組織)は、Magic Wandツールを使用して自家蛍光シグナルを調べることにより、このROIから除去します(図5E)。Tumor ROI と IM ROI はどちらも、さらに処理するために GeoJSON ファイルとして保存されます (図 5F)。
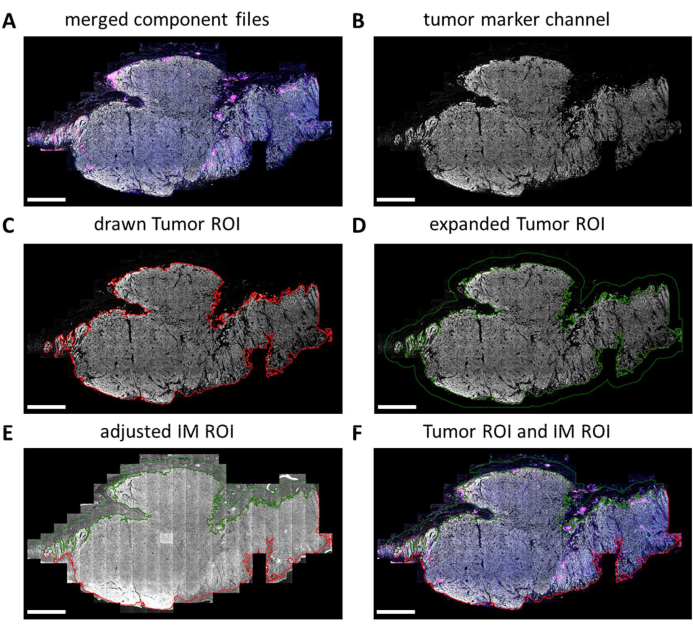
図5:QuPathでの腫瘍ROIおよび浸潤的マージンROI描画プロセス(A)マージされたコンポーネントファイル。(B)腫瘍マーカーチャネルのみを示すグレースケール画像。(C)腫瘍ROIは、腫瘍マーカーシグナルの周囲に描画されます。(D)腫瘍ROIを100〜500μm拡大してIM ROIを形成することにより、新しいROIが作成されます。(E)IM ROIは、バックグラウンド(負のシグナル)や脂肪、血管、毛包などの他の大きな組織構造を除外することにより、間質組織のみを含むように調整されます。(F)結果として得られる腫瘍ROIとIM ROIは保存され、GeoJSONファイルにエクスポートされ、領域をさらに処理します。腫瘍のROIは赤い枠線で表示され、IM ROIは緑の枠線で表示されます。スケールバー= 2 mm。略語: ROI = 関心領域;IM = 侵襲的マージン;GeoJSON = 地理的な JavaScript オブジェクト表記。この図の拡大版を表示するには、ここをクリックしてください。
ImmuNetネットワークは、免疫細胞の検出に使用できます。リンパ球パネルの場合、実験用複合画像(図6A)を、ソフトウェアによって検出された免疫細胞(図6B)と視覚的に比較できます。骨髄パネル(Supplemental File 6:Supplemental Figure S4)と樹状細胞パネル(Supplemental File 6:Supplemental Figure S5)についても、同様の視覚的比較を行うことができます。
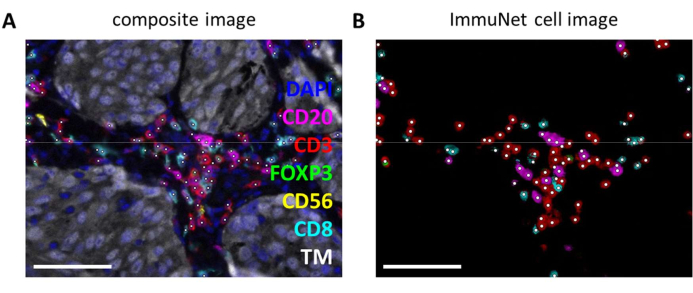
図6:ImmuNetが認識したリンパ球 (A)ImmuNetが認識した細胞を白い点で示した 図4B の合成画像。(B)ImmuNetによって認識され、その後検出されたマーカー発現。スケールバー = 50 μm。略語:TM =腫瘍マーカー。 この図の拡大版を表示するには、ここをクリックしてください。
ImmuNetによって検出され、.csv形式で保存された免疫細胞は、任意のプログラミング言語にインポートしてさらに分析することができます。空間可視化とゲーティングをRで行いました(補足ファイル8〕。その後、検出された細胞を空間的に可視化することができる(図7A、 補足ファイル6:補足図S6 および 補足図S7)。偽マーカー発現のゲーティングは、個々の免疫細胞を表現型決定するために行うことができます(図7B)。
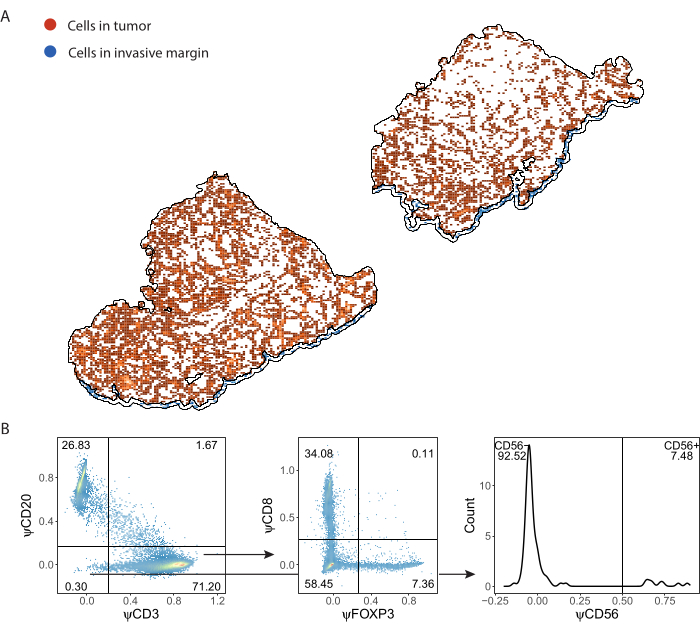
図7:リンパ球パネルのゲーティング戦略。(A)QuPathで描写された腫瘍および浸潤性マージンの関心領域で検出された免疫細胞。(B)パートAからImmuNetによって検出されたすべての細胞のゲーティング。リンパ球は、まずCD20+ B細胞およびCD3+ T細胞にゲートされます。CD3+ T細胞は、CD8およびFOXP3の発現に対してさらにゲーティングされています。CD20-CD3-集団は、CD56+ナチュラルキラー細胞に対してゲーティングされています。この図の拡大版を表示するには、ここをクリックしてください。
予測された細胞の表現型をゲーティングで決定すると、異なる表現型の細胞密度を異なるROI内で計算できます。これは、表現型ごとの細胞の総数をROIの表面積で割ることによって計算されます(表1、図8、および補足ファイル8)。ここで、B細胞はCD3-CD20+、ヘルパーT細胞はCD3+CD20-CD8-FoxP3-、制御性T細胞はCD3+CD20-CD8-FoxP3+、細胞傷害性T細胞はCD3+CD20-CD8+FoxP3-、NK細胞はCD3-CD20-CD56+と定義します。
| 表現型 | 腫瘍の密度(細胞/ mm2) | IM単位の密度(セル/ mm2) |
| Bセル | 185.74 | 145.62 |
| ヘルパーT細胞 | 301.46 | 157.51 |
| 制御性T細胞 | 38.53 | 19.53 |
| 細胞傷害性T細胞 | 185.35 | 83.21 |
| NKセル | 0.18 | 0 |
表1:ROIにおける表現型の密度。 リンパ球パネルで染色した単一の黒色腫サンプルで見つかった異なる表現型の細胞の密度。密度は、腫瘍ROIとIM ROIで別々に計算されます。略語:IM =侵襲的マージン;ROI = 関心領域。
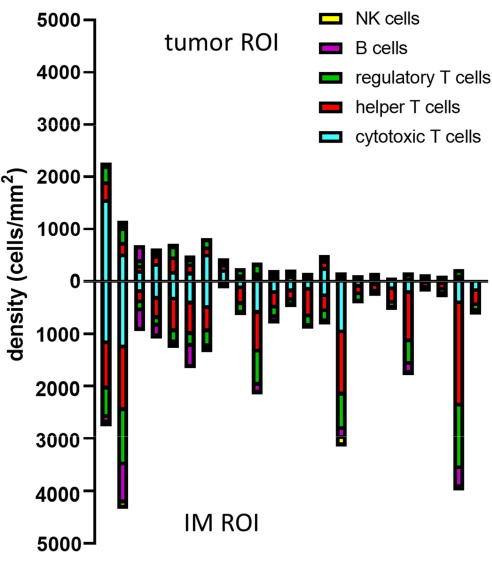
図8:複数のサンプルのデータ分析の例。 腫瘍におけるさまざまなリンパ球表現型の密度分析と、23の原発性黒色腫腫瘍のIM。略語:IM =侵襲的マージン;ROI = 関心領域。 この図の拡大版を表示するには、ここをクリックしてください。
これらの免疫細胞の空間情報をさらに詳しく調べるために、同定された表現型間の距離や、サンプル内の最近傍細胞の表現型の割合を決定することも可能です(図9)。
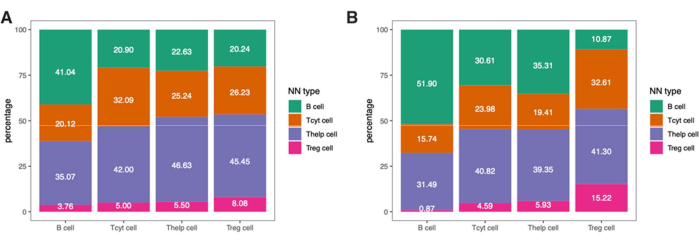
図9:単一サンプルの最近傍分析の例。 リンパ球パネルで染色された単一の黒色腫サンプルで見つかった (A) 腫瘍および (B) IM ROI のさまざまな細胞タイプの最近傍表現型の割合。略語:IM =侵襲的マージン;ROI = 関心領域。 この図の拡大版を表示するには、ここをクリックしてください。
補足ファイル1:プロトコル仕様をまとめたマルチプレックスIHC。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル2:モノプレックスのAutostainerプロトコル。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル3:自家蛍光補償のためのAutostainerプロトコル。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル4:マルチプレックス免疫組織化学のためのAutostainerプロトコル。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル5:テンプレート.csvファイル。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル6:黒色腫組織サンプル中の骨髄系および樹状細胞パネル、スキャンに失敗した場合のスライドのマーキング、ImmuNetによって認識された骨髄系および樹状細胞、骨髄系および樹状細胞パネルのゲーティング戦略。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル7:QuPathステッチスクリプト。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル8:データ分析スクリプト。このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
TMEの空間解析は、免疫細胞コンパートメントについてさらに学び、特に免疫腫瘍学の分野で新しい予後および予測バイオマーカーを発見するための求められている手法です16。この目的のために、タンパク質、mRNA転写産物、またはこれら2つの組み合わせの検出を含む多くの異なる技術が開発されており、最大100〜1,000のターゲットを推定します。しかし、マルチプレックス化が進むと、ハイスループットな実験、実験コスト、技術的な課題が減り、多くの場合、TMEのごく一部しか解析できません。ここで説明するTSAベースの方法を使用したマルチプレックスIHCは、6つの異なるマーカー+ DAPIを同時に検出し、比較的安価に実行でき、組織切片全体を20分以内にイメージングし、完全に分析する準備ができています。この技術は、染色手順の自動化により、複雑さが軽減されました。2つの追加フィルターの追加を含むマルチスペクトル顕微鏡の改良により、スペクトルのアンミキシングとスキャン時間が大幅に改善されました。最大8つの異なるマーカー+DAPIを同時に検出することが可能です。しかし、マルチプレックスをより多くのマーカーで拡張すると、スペクトルのアンミキシングがより困難になり、スライド全体のスキャン時間が大幅に増加するため、前述の利点はなくなります。診断現場での実施を容易にするために、異なる施設間でマルチプレックスIHCを標準化する取り組みが行われています。このマルチプレックスIHCの標準化のために、6つの異なるマーカー+ DAPIを使用して、よりアクセスしやすいプロトコルに従うことをお勧めします。それにもかかわらず、まだかなりの技術的なノウハウが必要であり、ダウンストリーム分析は困難な場合があり、そのためにこのプロトコルで説明されている方法論を開発しました。
標準化は、マルチプレックスIHCパネルの開発から始まります。特定のタンパク質標的を検出する一次抗体を選択することの重要性は、17以前から強調されてきました。当社のマルチプレックスIHCパネルは、主に一次抗体クローンを使用して開発されており、診断部門でもIHCに使用され、検証されています。しかし、樹状細胞マルチプレックスIHCパネルの場合、ほとんどの抗体は診断現場で使用されていませんでした(van der Hoorn et al.、提出原稿)。特異性を確保し、バッチの違いを最小限に抑えるために、ポリクローナル抗体よりもモノクローナル抗体を使用することを選択し、トランスフェクションした細胞株と初代細胞を使用してほとんどの抗体も検証しています。長年にわたり、Vectra 3システム18,21,23,24,25,26,27,28,29,30,31,32を使用した多数の研究で、さまざまなバージョンのマルチプレックスIHCパネルが使用されてきました.これらのマルチプレックスIHCパネルをPhenoImager HTシステムに最適に実装するためには、一次抗体と蛍光色素の組み合わせにいくつかの調整を行う必要がありました。全組織切片のより良いスペクトルアンミキシングとスキャン時間の短縮の恩恵を受けるためには、最新のOpal480およびOpal780蛍光色素を実装し、7色のマルチプレックスIHCパネルでOpal540およびOpal650蛍光色素の使用を避ける必要があります。スキャン時間は、組織切片のサイズにもよりますが、3~10倍速くなります。マルチプレックスIHCパネルの調整は非常に簡単に実現できましたが、いくつかの考慮事項を念頭に置く必要があります。Opal480の蛍光スペクトルは、自家蛍光スペクトルと多く重複するため、赤血球や他の自己蛍光構造のスペクトルアンミキシングを妨げます。一次抗体の濃度を上げてOpal480と組み合わせることで、ほとんどの場合、この問題が解決しました。PhenoImager HTに独自のサンプルAFフィルターを実装すると、Opal480と自家蛍光のアンミキシングが容易になります。ただし、Opal480と併用すると明確なシグナルが得られる一次抗体を使用するのが最善であり、そのシグナルは自家蛍光よりも高くなります。
これらのマルチプレックスIHCパネルは確立されていますが、バッチ間のバリエーションを考慮する必要があります。フルマルチプレックスIHC実験を開始する前にモノプレックスIHCコントロールを行うことで、実験ごとに一次抗体の性能が強いか弱いかが観察されることがありました。この原因としては、ピペッティングエラー、試薬の保管条件が最適でない、保存期間などが考えられます。私たちは、経験に基づいて一次抗体溶液を調整することでこれを解決しました。前述の調整を行う必要がなかった場合でも、マルチプレックスIHCバッチ実験のたびに、モノプレックスIHC染色コントロールスライドに基づいて露光時間を設定することが重要です。
私たちの研究は当初、さまざまな種類のがんと黒色腫に焦点を当てていたため、マルチプレックスIHCパネルは、最小限の調整で腫瘍タイプ間で交換可能である必要がありました。したがって、最適化プロセスには常に複数の(腫瘍)組織タイプを含め、免疫細胞マーカーの一次抗体の希釈は、異なる腫瘍タイプ間で同等に保つことができることを観察しました。ただし、がんと黒色腫の間の腫瘍組織検出には、異なる腫瘍マーカーが必要です。したがって、腫瘍マーカーは常に各マルチプレックスIHCパネルの最後に機能するように最適化されており、現在は常にOpal780と組み合わせて使用されています。Opal780は、偶然にもマルチプレックスIHC染色手順の最後の蛍光色素分子になければなりません。したがって、マルチプレックスIHCの最後に腫瘍マーカーを使用することにより、これらのマルチプレックスIHCパネルは、神経膠芽腫(すなわち、GFAP)およびホジキンリンパ腫(すなわち、CD30)などの他の腫瘍タイプと容易に交換することができる。血管肉腫に対しては、赤芽球形質転換特異的遺伝子(ERG)を用いたこのリンパ球マルチプレックスIHCパネルを腫瘍マーカーとして用い、最適化実験は25回のみであった。最適化には、ERG一次抗体の滴定と、最後にERGを使用したマルチプレックスIHCパネルの試験が含まれていました。
これらのマルチプレックスIHCパネルに対する他の調整は、特定の免疫細胞マーカーを別の免疫マーカーまたは機能マーカーと交換することによっても行うことができます。すべての変更には最適化が必要です。最適化のためのプロトコルは、前に説明した17に従うことができる。提案されたマルチプレックスIHCパネルへの特定の変更は、私たちが作成したImmuNetアルゴリズムに干渉します。十分なデータを生成する必要があり、これらの変更をアルゴリズムに実装するために時間を費やす必要があります(新しいマーカーや細胞表現型ごとに少なくとも750のアノテーション、および以前にトレーニングされたマーカーの検証に150のアノテーション)。本パネルには機能マーカーは含まれていませんが、PD-1やPD-L1などの免疫チェックポイントマーカーのマルチプレックスIHCパネルへの実装は当研究室で行っています。しかし、負のシグナルとポジティブのシグナルでバイナリーが少ないマーカーの解析はより困難であることが証明されており、私たちのグループでは活発な研究が行われています。
マルチプレックスIHCで同時に評価できるマーカーの数は、他の新しい技術と比較して限られています。これは、FFPE ブロックの連続するスライス上の異なるパネルを分析することで回避できますが、これらのスライスを空間的に比較することは困難です。スライドの準備後、向きと折りたたまれたアーティファクトは同じではない可能性があります。それにもかかわらず、マルチプレックスIHCは非常にアクセスしやすいため、より多くの機関や研究者にとって魅力的なツールとなり、したがって、診断環境での将来の実装により適しています。複数の腫瘍タイプと下流の分析パイプラインに対するマルチプレックスIHC免疫細胞パネルの標準化により、患者と腫瘍タイプ間のTMEの違いについてより多くの知識を得ることができます。これにより、例えば、特定の治療に対する抗腫瘍反応におけるTMEの役割について、より深い洞察を得ることができます。これにより、治療への反応や予想生存率などの要因を予測するための新しいバイオマーカーが生まれる可能性さえあります。全体として、これにより、マルチプレックスIHCは、個別化医療アプローチにおいて、臨床上の意思決定を支援する臨床ツールになることができます。確かに、分析手順のより多くのステップは、日常の診断設定で使用できるようにするために自動化および標準化されるべきであり、現時点では、それは主に未来的な視点です。
1つのサンプルスライド上の複数のマーカーの分析は、技術的な課題にもかかわらず、非常に強力なツールになる可能性があります。標準化された実験プロトコルと堅牢な分析方法により、ここでImmuNetを使用して説明したように、複数のマーカーの定量化により、従来のIHCよりも情報量が多くなり、マルチプレックスIHCは新しい高プレックス実験法と比較して比較的ハイスループットのままです。
開示事項
著者には、開示すべき利益相反はありません。
謝辞
PhenoImager HTは、ラドバウド大学医療センターとラドバウド顕微鏡技術センターの資金提供により購入されました。CFは、Dutch Cancer Societyの助成金(10673)とERC Advの助成金ARTimmune(834618)によって財政的にサポートされています。JTは、NWO Vidi助成金(VI.Vidi.192.084)によって財政的にサポートされています。著者らは、マルチプレックスIHCデータを保存するワークフローの作成に協力してくれたEric van Dinther氏とAnkur Ankan氏に感謝し、ROI描画のためにQuPathにマルチプレックスIHCデータを実装する方法についての指示を受けたBengt Pung氏に感謝します。
資料
| Name | Company | Catalog Number | Comments |
| anti-CD14 | Cell Marque | 114R-16 | section 3, clone EPR3653 |
| anti-CD163 | Cell Marque | 163M-15 | section 3, clone MRQ-26 |
| anti-CD19 | Abcam | ab134114 | section 3, clone EPR5906 |
| anti-CD1c (BDCA1) | Thermo Scientific | TA505411 | section 3, clone OTI2F4 |
| anti-CD20 | Thermo Scientific | MS-340-S | section 3, clone L26 |
| anti-CD3 | Thermo Scientific | RM-9107 | section 3, clone sp7 |
| anti-CD303/BDCA2 | Dendritics via Enzo Lifesciences/Axxora | DDX0043 | section 3, clone 124B3.13 |
| anti-CD56 | Cell Marque | 156R-94 | section 3, clone MRQ-42 |
| anti-CD66b | BD Biosciences | 555723 | section 3, clone G10F5 |
| anti-CD68 | Dako Agilent | M087601 | section 3, clone PG-M1 |
| anti-CD8 | Dako Agilent | M7103 | section 3, clone C8/144B |
| anti-Foxp3 | Thermo Scientific | 14-4777 | section 3, clone 236A/E7 |
| anti-Gp100 | Dako Agilent | M063401 | section 3, clone HMB45 |
| anti-HLA-DR, DP, DQ | Santa Cruz | sc-53302 | section 3, clone CR3/43 |
| anti-MART-1 | Thermo Scientific | MS-799 | section 3, clone A103 |
| anti-pan cytokeratin | Abcam | ab86734 | section 3, clone AE1/AE3 + 5D3 |
| anti-SOX10 | Sigma Aldrich | 383R | section 3, clone EP268 |
| anti-Tyrosinase | Sanbio | MONX10591 | section 3, clone T311 |
| anti-XCR1 | Cell Signaling Technologies via Bioké | 44665S | section 3, clone D2F8T |
| antibody diluent | Akoya BioSciences | SKU ARD1001EA | section 3, from Opal 7-Color Automation IHC Kit 50 slide (can optionally also be replaced by TBST with 10% BSA) |
| Bond Aspirating Probe | Leica Biosciences | S21.0605 | section 3 |
| Bond Aspirating Probe Cleaning | Leica Biosciences | CS9100 | section 3 |
| Bond Dewax Solution | Leica Biosciences | AR9222 | section 3 |
| Bond Objectglas label + print lint | Leica Biosciences | S21.4564.A | section 3 |
| Bond Research Detection System 2 | Leica Biosciences | DS9777 | section 3 |
| Bond RX autostainer | Leica Biosciences | - | section 3, automated platform |
| Bond TM Epitope Retrieval 1 - 1 L | Leica Biosciences | AR9961 | section 3 |
| Bond TM Epitope Retrieval 2 - 1 L | Leica Biosciences | AR9640 | section 3 |
| Bond TM Wash Solution 10x - 1 L | Leica Biosciences | AR9590 | section 3 |
| BOND Universal Covertile | Leica Biosciences | S21.4611 | section 3 |
| Bond(TM) Titration Kit | Leica Biosciences | OPT9049 | section 3 |
| Coverslip 24 x 32 mm #1 (0.13-0.16 mm) | Fisher Scientific | 15717592 | section 2 |
| coverslip 24 x 50 mm | VWR | 631-0146 | section 2 |
| DAPI Fluoromount-G | VWR | 0100-20 | section 3, whenever monoplex slides need to be quickly checked, not for official analysis, then DAPI is stained seperately for better results |
| Eosine | section 2, home made | ||
| Ethanol 99.5% | VWR | 4099.9005 | section 2 |
| Fluoromount-G | VWR | 0100-01 | section 3 |
| haematoxyline | - | section 2, home made | |
| ImmuNet | - | immune cell detection and phenotyping pipeline | |
| inForm software 2.4.10 | Akoya BioSciences | - | section 4 & 6 |
| OPAL 480 reagent pack | Akoya BioSciences | FP1500001KT | section 3 |
| OPAL 520 reagent pack | Akoya BioSciences | FP1487001KT | section 3 |
| OPAL 570 reagent pack | Akoya BioSciences | FP1488001KT | section 3 |
| OPAL 620 reagent pack | Akoya BioSciences | FP1495001KT | section 3 |
| OPAL 690 reagent pack | Akoya BioSciences | FP1497001KT | section 3 |
| OPAL 780 reagent pack | Akoya BioSciences | FP1501001KT | section 3 |
| Opal 7-Color Automation IHC Kit 50 slide | Akoya BioSciences | NEL821001KT | section 3 |
| PhenoChart 1.1.0 | Akoya BioSciences | - | section 5 |
| PhenoImagerHT | Akoya BioSciences | CLS143455 | section 4, digital pathology imager with slide viewer and imaging software (formerly known as Vectra Polaris) |
| Quick-D mounting medium | Klinipath | 7280 | section 2 |
| QuPath 0.3.2 | whole slide image analysis software platform | ||
| R 4.1.1 | |||
| Slide boxes | VWR | 631-0737 | section 1 |
| SuperFrost Plus | Thermo Scientific through VWR | 631-9483 | section 1 |
| Vectra Polaris software 1.0.13 | Akoya BioSciences | - | section 4 |
| Xylene | VWR | 4055-9005 | section 2 |
参考文献
- Dunn, G. P., Bruce, A. T., Ikeda, H., Old, L. J., Schreiber, R. D. Cancer immunoediting: from immunosurveillance to tumor escape. Nature Immunology. 3 (11), 991-998 (2002).
- van der Woude, L. L., Gorris, M. A. J., Halilovic, A., Figdor, C. G., de Vries, I. J. M. Migrating into the tumor: a roadmap for T cells. Trends in Cancer. 3 (11), 797-808 (2017).
- Fridman, W. H., Pages, F., Sautes-Fridman, C., Galon, J. The immune contexture in human tumours: impact on clinical outcome. Nature Reviews. Cancer. 12 (4), 298-306 (2012).
- Fridman, W. H., et al. The immune microenvironment of human tumors: general significance and clinical impact. Cancer Microenvironment. 6 (2), 117-122 (2013).
- Pages, F., et al. International validation of the consensus Immunoscore for the classification of colon cancer: a prognostic and accuracy study. Lancet. 391 (10135), 2128-2139 (2018).
- Angell, H. K., Bruni, D., Barrett, J. C., Herbst, R., Galon, J. The Immunoscore: colon cancer and beyond. Clinical Cancer Research. 26 (2), 332-339 (2020).
- Angell, H., Galon, J. From the immune contexture to the Immunoscore: the role of prognostic and predictive immune markers in cancer. Current Opinion in Immunology. 25 (2), 261-267 (2013).
- Galon, J., et al. Cancer classification using the Immunoscore: a worldwide task force. Journal of Translational Medicine. 10, 205 (2012).
- Galon, J., et al. World-wide Immunoscore Task Force: meeting report from the 34;Melanoma Bridge", Napoli, November 30th-December 3rd, 2016. Journal of Translational Medicine. 15 (1), 212 (2017).
- Tumeh, P. C., et al. PD-1 blockade induces responses by inhibiting adaptive immune resistance. Nature. 515 (7528), 568-571 (2014).
- Creemers, J. H. A., et al. A tipping point in cancer-immune dynamics leads to divergent immunotherapy responses and hampers biomarker discovery. Journal for Immunotherapy of Cancer. 9 (5), e002032 (2021).
- Blank, C. U., Haanen, J. B., Ribas, A., Schumacher, T. N. CANCER IMMUNOLOGY. The "cancer immunogram". Science. 352 (6286), 658-660 (2016).
- Teruya-Feldstein, J. The immunohistochemistry laboratory looking at molecules and preparing for tomorrow. Archives of Pathology & Laboratory Medicine. 134 (11), 1659-1665 (2010).
- Ramos-Vara, J. A., Miller, M. A., et al. When tissue antigens and antibodies get along: revisiting the technical aspects of immunohistochemistry--the red, brown, and blue technique. Veterinary Pathology. 51 (1), 42-87 (2014).
- Hegde, P. S., Karanikas, V., Evers, S. The where, the when, and the how of immune monitoring for cancer immunotherapies in the era of checkpoint inhibition. Clinical Cancer Research. 22 (8), 1865-1874 (2016).
- Parra, E. Novel platforms of multiplexed immunofluorescence for study of paraffin tumor tissues. Journal of Cancer Treatment and Diagnosis. 2 (1), 43-53 (2018).
- Gorris, M. A. J., et al. Eight-color multiplex immunohistochemistry for simultaneous detection of multiple immune checkpoint molecules within the tumor microenvironment. Journal of Immunology. 200 (1), 347-354 (2018).
- Roelofsen, T., et al. Spontaneous regression of ovarian carcinoma after septic peritonitis; a unique case report. Frontiers in Oncology. 8, 562 (2018).
- van den Brand, D., et al. Peptide-mediated delivery of therapeutic mRNA in ovarian cancer. European Journal of Pharmaceutics and Biopharmaceutics. 141, 180-190 (2019).
- van den Brand, D., et al. EpCAM-binding DARPins for targeted photodynamic therapy of ovarian cancer. Cancers. 12 (7), 1762 (2020).
- Di Blasio, S., et al. The tumour microenvironment shapes dendritic cell plasticity in a human organotypic melanoma culture. Nature Communications. 11 (1), 2749 (2020).
- van Beek, J. J. P., et al. Human pDCs are superior to cDC2s in attracting cytolytic lymphocytes in melanoma patients receiving DC vaccination. Cell Reports. 30 (4), 1027-1038 (2020).
- Rodriguez-Rosales, Y. A., et al. Immunomodulatory aged neutrophils are augmented in blood and skin of psoriasis patients. Journal of Allergy and Clinical Immunology. 148 (4), 1030-1040 (2021).
- Hoeijmakers, Y. M., et al. Immune cell composition in the endometrium of patients with a complete molar pregnancy: Effects on outcome. Gynecologic Oncology. 160 (2), 450-456 (2021).
- van Ravensteijn, S. G., et al. Immunological and genomic analysis reveals clinically relevant distinctions between angiosarcoma subgroups. Cancers. 14 (23), 5938 (2022).
- van der Woude, L. L., et al. Tumor microenvironment shows an immunological abscopal effect in patients with NSCLC treated with pembrolizumab-radiotherapy combination. Journal for Immunotherapy of Cancer. 10 (10), e005248 (2022).
- Graham Martinez, C., et al. The immune microenvironment landscape shows treatment-specific differences in rectal cancer patients. Frontiers in Immunology. 13, 1011498 (2022).
- Cortenbach, K. R. G., et al. Topography of immune cell infiltration in different stages of coronary atherosclerosis revealed by multiplex immunohistochemistry. International Journal of Cardiology. Heart & Vasculature. 44, 101111 (2023).
- van Wilpe, S., et al. Homologous recombination repair deficient prostate cancer represents an immunologically distinct subtype. Oncoimmunology. 11 (1), 2094133 (2022).
- Gorris, M. A. J., et al. Paired primary and metastatic lesions of patients with ipilimumab-treated melanoma: high variation in lymphocyte infiltration and HLA-ABC expression whereas tumor mutational load is similar and correlates with clinical outcome. Journal for Immunotherapy of Cancer. 10 (5), e004329 (2022).
- van Wilpe, S., et al. Intratumoral T cell depletion following neoadjuvant chemotherapy in patients with muscle-invasive bladder cancer is associated with poor clinical outcome. Cancer Immunology, Immunotherapy. 72 (1), 137-149 (2023).
- Sultan, S., Gorris, M. A. J., Buytenhuijs, F., Lvan de Woude, L. A Segmentation-free machine learning architecture for immune land-scape phenotyping in solid tumors by multichannel imaging. bioRxiv. , (2021).
- Parra, E. R., et al. Immuno-profiling and cellular spatial analysis using five immune oncology multiplex immunofluorescence panels for paraffin tumor tissue. Scientific Reports. 11 (1), 8511 (2021).
- Parra, E. R., et al. Validation of multiplex immunofluorescence panels using multispectral microscopy for immune-profiling of formalin-fixed and paraffin-embedded human tumor tissues. Scientific Reports. 7 (1), 13380 (2017).
- Sun, Z., Nyberg, R., Wu, Y., Bernard, B., Redmond, W. L. Developing an enhanced 7-color multiplex IHC protocol to dissect immune infiltration in human cancers. PLoS One. 16 (2), e0247238 (2021).
- LeicaBiosystems. BOND RX Fully Automated Research Stainer Protocols. , (2022).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved