Method Article
Analisi immunoistochimica multiplex del panorama spaziale delle cellule immunitarie del microambiente tumorale
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo protocollo descrive in dettaglio come viene effettuata la caratterizzazione delle cellule immunitarie del microambiente tumorale utilizzando l'immunoistochimica multiplex.
Abstract
Il panorama delle cellule immunitarie del microambiente tumorale contiene potenzialmente informazioni per la scoperta di biomarcatori prognostici e predittivi. L'immunoistochimica multiplex è uno strumento prezioso per visualizzare e identificare diversi tipi di cellule immunitarie nei tessuti tumorali, conservando le informazioni spaziali. Qui forniamo protocolli dettagliati per analizzare le popolazioni di cellule linfocitarie, mieloidi e dendritiche in sezioni di tessuto. A partire dal taglio di sezioni fissate in formalina e incluse in paraffina, alle procedure automatiche di colorazione multiplex su una piattaforma automatizzata, alla scansione dei vetrini su un microscopio per immagini multispettrale, fino all'analisi delle immagini utilizzando un algoritmo di apprendimento automatico sviluppato internamente ImmuNet. Questi protocolli possono essere applicati a una varietà di campioni tumorali semplicemente cambiando i marcatori tumorali per analizzare le cellule immunitarie in diversi compartimenti del campione (tumore contro margine invasivo) e applicare l'analisi del vicino più vicino. Questa analisi non si limita ai campioni tumorali, ma può essere applicata anche ad altri tessuti (non) patogeni. I miglioramenti apportati alle apparecchiature e al flusso di lavoro negli ultimi anni hanno ridotto significativamente i tempi di produzione, facilitando l'applicazione futura di questa procedura in ambito diagnostico.
Introduzione
Le cellule immunitarie svolgono un ruolo cruciale nella protezione contro agenti patogeni come virus e batteri, ma anche contro le cellule cancerose1. Pertanto, il sistema immunitario all'interno del microambiente tumorale (TME) è molto promettente per la scoperta di biomarcatori prognostici e predittivi2. Gli infiltrati di cellule immunitarie sono stati correlati alla prognosi in vari tipi di cancro, sebbene ciò non sia stato ancora implementato nell'assistenza clinica 3,4. Nella maggior parte dei tipi di tumore, un numero elevato di cellule T citotossiche e di cellule T helper 1 e/o un basso numero di cellule T regolatorie sono legati a buone prognosi. Sono in corso sforzi per incorporare un cosiddetto "Immunoscore" nella stadiazione TNM del cancro del colon-retto, trasformandolo in stadiazione TNM-I 5,6. L'Immunoscore è derivato dal numero totale di cellule T (rilevate con CD3) e cellule T citotossiche (rilevate con CD8) in due diverse regioni tumorali: il nucleo tumorale rispetto al margine invasivo (IM) dei tumori. È stato anche proposto che l'Immunoscore abbia un valore prognostico in altri tipi di cancro, come il melanoma, il cancro ai polmoni e il cancro al seno 6,7,8,9. Inoltre, gli infiltrati di cellule immunitarie possono anche essere correlati alla risposta all'immunoterapia con blocco del checkpoint10. Tuttavia, questi biomarcatori predittivi devono essere convalidati in studi prospettici prima di poter essere implementati di routine nella pratica clinica. Inoltre, è stato anche proposto che un singolo biomarcatore sarà insufficiente per una previsione significativa11. Pertanto, la creazione di una mappa completa di un campione di paziente combinando diversi biomarcatori è stata proposta come biomarcatore predittivo più completo in un cosiddetto "immunogramma del cancro"12.
Tra i metodi per lo studio delle cellule immunitarie nell'ambito della TME, la tecnica più antica e conosciuta è l'immunoistochimica (IHC), utilizzata di routine per i test diagnostici in diverse malattie, in particolare il cancro13. Questa tecnica è stata limitata all'uso di uno o solo pochi marcatori14 per lungo tempo e quindi è stata superata in contesti di ricerca da altre tecniche come la citometria a flusso e il profilo di espressione genica (GEP). Tuttavia, i tessuti tumorali fissati in formalina e inclusi in paraffina (FFPE) tipicamente utilizzati nella diagnostica e nella ricerca di routine non sono (in modo ottimale) adatti per la citometria a flusso e la GEP. Inoltre, sebbene la GEP e la citometria a flusso forniscano molte informazioni sul fenotipo e sulla funzione cellulare, la mancanza di informazioni spaziali è un grave svantaggio. Pertanto, l'eterogeneità all'interno di un campione, come le differenze nelle aree di un tumore infiltrate da cellule immunitarie rispetto a quelle escluse dalle cellule immunitarie, potrebbe non essere rilevata15. Sono state sviluppate nuove piattaforme per l'analisi multiplex dei tessuti FFPE, come l'IHC multiplex, la citometria di massa per imaging e il rilevamento di CO mediante indEXing (CODEX) che possono essere utilizzate per rilevare più marcatori contemporaneamente all'interno di una sezione tissutale16. Le cellule immunitarie contenute nel TME sono oggetto di studi approfonditi per trovare i migliori biomarcatori per l'immunoterapia. Tuttavia, le tecniche multiplex e l'analisi automatizzata delle immagini pongono dei propri ostacoli.
Il nostro laboratorio ha una vasta esperienza nella colorazione IHC multiplex utilizzando il metodo di amplificazione del segnale opalino/tiramide (TSA) e lo ha automatizzato su una piattaforma IHC (vedi la tabella dei materiali)17,18,19,20,21,22,23,24,25,26,27,28, 29,30,31. Abbiamo ottimizzato i pannelli di cellule immunitarie per il rilevamento di diversi sottogruppi di linfociti, cellule mieloidi e cellule dendritiche (DC). I tessuti che contengono aree di cellule immunitarie dense - per i linfociti o le morfologie cellulari complesse (ad esempio, cellule mieloidi e DC) - sono particolarmente difficili da analizzare, con il rischio di sovrastimare o sottostimare il numero di cellule immunitarie presenti. Per superare questo problema, il nostro gruppo32 ha sviluppato il software di analisi ImmuNet e questa pipeline di apprendimento automatico ha migliorato enormemente la qualità del rilevamento di questi diversi tipi di cellule immunitarie. Qui viene descritto un protocollo dettagliato dall'ottenimento del materiale FFPE all'analisi della densità delle cellule immunitarie in diversi compartimenti tissutali e delle distanze tra i tipi di cellule immunitarie.
Questo protocollo delinea le modalità di esecuzione dei pannelli IHC multiplex presso il Radboud University Medical Center dall'implementazione dell'imager di patologia digitale nel 2022. I pannelli IHC multiplex descritti possono essere utilizzati per diversi carcinomi (ad es. polmone, prostata, colon-retto, vescica, mammella) con l'uso di un anticorpo pan-citocheratina come marcatore tumorale o per il melanoma con l'uso di anticorpi associati ai melanociti come marcatori tumorali. Questi protocolli IHC multiplex sono stati accuratamente ottimizzati in termini di concentrazione di anticorpi primari, combinazioni di fluorofori e sequenza della procedura di colorazione. Noi e altri abbiamo descritto l'ottimizzazione del pannello IHC multiplex in precedenza 17,33,34,35. I pannelli IHC multiplex possono essere adattati, ma le pipeline di analisi descritte devono essere valutate ed eventualmente regolate o riaddestrate di conseguenza. I protocolli IHC multiplex a sette colori descritti utilizzano i fluorofori opalini Opal480, Opal520, Opal570, Opal620, Opal690, Opal780 e 4',6-diamidino-2-fenilindolo (DAPI), in modo che la scansione facile e veloce sull'imager sia abilitata con "Multispectral One Touch ImmunoFluorescence" (MOTiF). La colorazione e la scansione a nove colori non sono descritte in questo protocollo, in quanto ciò richiede una messa a punto ancora maggiore della configurazione sperimentale e un'altra modalità di scansione sull'imager che utilizza il filtro sintonizzabile a cristalli liquidi.
Protocollo
Il materiale dei pazienti mostrato per questo protocollo faceva parte di uno studio condotto in precedenza ed è stato ufficialmente ritenuto esente dall'approvazione etica medica dal Comitato etico medico locale di Radboudumc in concomitanza con la legislazione olandese (numero di file 2017-3164)30.
1. Raccolta del materiale FFPE, selezione dei blocchi e preparazione dei campioni
- Recupera gli identificatori di blocco FFPE dalle cartelle cliniche dei pazienti tramite medici curanti o patologi. Verificare con le normative locali se è necessaria un'autorizzazione etica.
- Richiedi i blocchi FFPE all'archivio di patologia locale o all'ospedale esterno.
NOTA: È anche possibile che venga acquisito materiale tumorale o una biopsia per un particolare studio. Questo può essere il caso di piccoli studi clinici o studi sugli animali. In questi casi, l'elaborazione del campione di tessuto può essere responsabilità del ricercatore. - Quando sono disponibili più blocchi FFPE, selezionare il blocco FFPE più rappresentativo contenente tessuto tumorale vitale, preferenzialmente con il tessuto stromale circostante presente valutando i vetrini colorati con ematossilina ed eosina (HE) (Figura 1).
NOTA: Si consiglia di ottenere il parere di un esperto per questa selezione (ad esempio, un patologo). È possibile che gli HE non siano disponibili per la valutazione del contenuto di un blocco FFPE e che ne siano necessari di nuovi per la selezione. Vai alla sezione 2 per una descrizione. - Tagliare nastri FFPE di 4 μm di spessore su un microtomo.
NOTA: Lo spessore può essere compreso tra 1 μm e 6 μm senza un notevole impatto di macchie; Tuttavia, 4 μm è il più standard. - Montare i campioni su vetrini in una posizione favorevole per la fluidica dell'autocoloratore (Figura 2A-C) utilizzando uno dei metodi descritti di seguito:
- Porre le sezioni sulla superficie di acqua distillata a 40 °C a bagnomaria per distenderle e raccoglierle con un vetrino.
O
Posizionare i vetrini su una piastra riscaldante a 40 °C, avendo cura di coprire il punto in cui la sezione deve essere montata sul vetrino con una goccia di acqua distillata. Posiziona la sezione sopra questa goccia con una pinza e lasciala allungare. Assorbire l'acqua distillata utilizzando un tovagliolo di carta e rimuovere l'acqua in eccesso picchiettando il vetrino.
NOTA: Posizionare sezioni di tessuto troppo vicino all'etichetta del vetrino comporterà una colorazione non ottimale (Figura 2D, E). Tendiamo a montare 6-10 vetrini per campione per eseguire i diversi pannelli IHC multiplex e per avere un backup.
- Porre le sezioni sulla superficie di acqua distillata a 40 °C a bagnomaria per distenderle e raccoglierle con un vetrino.
- Lasciare asciugare i vetrini montati a 56 °C per 1 ora o durante la notte a 37 °C.
- Utilizzare i vetrini montati per l'esperimento o conservarli in scatole a 4 °C.
NOTA: In base alla nostra esperienza finora, questi vetrini montati possono essere conservati per anni prima che venga eseguita la colorazione IHC multiplex.
2. Generazione di vetrini colorati con ematossilina ed eosina
NOTA: Tutti i passaggi successivi della sezione 2 devono essere eseguiti in una cappa aspirante.
- Deparaffinare i vetrini in xilene (2 x 5 min).
- Reidratare in etanolo (99,6% 1 x 5 min; 95% 1 x 5 min; 70% 1 x 2 min). In alternativa, immergere i vetrini 3 volte in etanolo al 99,6%.
- Lavare i vetrini in acqua distillata (2 min).
- Colorare i nuclei con ematossilina (10 min).
- Lavare i vetrini con H2O distillato (5 min).
- Colorare i vetrini con eosina (5 min).
- Disidratare i vetrini immergendoli 3 volte in etanolo al 99,6%.
- Immergere i vetrini 2 volte nello xilene.
- Aggiungere alcune gocce di mezzo di montaggio e sigillare con un vetrino coprioggetti.
- Lasciare che i vetrini si induriscano e toglierli dalla cappa aspirante quando tutte le sostanze chimiche sono evaporate.
3. Esecuzione di IHC monoplex e multiplex nell'autostainer
- Calcolare la quantità di reagente necessaria in base al numero di campioni da colorare.
NOTA: Per ogni corsa, l'autostainer ha una capacità di 30 vetrini e impiega ~18 ore per completare il protocollo IHC multiplex con sei anticorpi. Quando è necessario colorare più vetrini, è possibile inserire più lotti ogni sera della settimana (lavorativa); 4 notti di 30 diapositive = 120 diapositive a settimana.- Preparare tutti i reagenti necessari all'inizio della settimana. Il sistema autostainer eroga 150 μL di reagente per vetrino. Utilizzare i contenitori di titolazione da 6 mL per gli anticorpi e i reagenti Opal e i contenitori da 30 mL per il reagente bloccante e l'anticorpo secondario-perossidasi di rafano.
NOTA: I contenitori da 6 ml sono dotati di comodi inserti che possono essere facilmente estratti e sostituiti quando necessario. Con i calcoli dei reagenti, si deve considerare il volume morto di 1,6 mL o 300 μL per il contenitore da 30 mL o il contenitore di titolazione da 6 mL, rispettivamente. - Diluire tutti i fluorofori opalini e la digossigenina (DIG) 1:100 nel diluente fornito; diluire Opal780 1:25 nel diluente anticorpale. Diluire tutti gli anticorpi primari in diluente anticorpale, con le diluizioni specificate nel file supplementare 1.
- Preparare tutti i reagenti necessari all'inizio della settimana. Il sistema autostainer eroga 150 μL di reagente per vetrino. Utilizzare i contenitori di titolazione da 6 mL per gli anticorpi e i reagenti Opal e i contenitori da 30 mL per il reagente bloccante e l'anticorpo secondario-perossidasi di rafano.
- Per seguire questo protocollo, eseguire IHC monoplex (Supplemental File 2) su vetrini contenenti sia il tessuto di controllo delle tonsille che altri tipi di tessuto (tumorale) di interesse prima di iniziare con l'esperimento IHC multiplex vero e proprio per assicurarsi che tutti i reagenti siano preparati bene.
NOTA: L'IHC monoplex impiega ~3,5 ore e può essere controllato prima della fine di quella giornata per i modelli e l'intensità del segnale. Se alcuni segnali sono troppo deboli (Figura 3), è possibile apportare modifiche ai reagenti. - Per la correzione dell'autofluorescenza, preparare un vetrino con tessuto (tumorale) contenente strutture autofluorescenti, come sangue e collagene. Preparare questo vetrino contemporaneamente ai vetrini IHC monoplex, ma con il reagente bloccante che sostituisce l'anticorpo e i reagenti Opal (File supplementare 3).
NOTA: In linea di principio, tale vetrino può essere riutilizzato per l'imaging multispettrale fino a quando la correzione dell'autofluorescenza non è più ottimale. Tuttavia, con tessuti altamente autofluorescenti, come il cervello e il fegato, è consigliabile utilizzare quel tessuto per la correzione dell'autofluorescenza. - Ad ogni ciclo IHC multiplex, caricare 29 campioni nel sistema autostainer con un vetrino di tessuto di controllo per verificare le prestazioni di ogni ciclo IHC multiplex.
- Scaricate i protocolli IHC multiplex dal sito web dell'autostainer nella scheda Download e regolateli per adattarli a ciascun pannello IHC36 multiplex personalizzato. Per il multiplex IHC, vedere il file supplementare 4 per il protocollo e per i pannelli IHC multiplex personalizzati, vedere il file supplementare 1.
- Dopo aver completato il protocollo di colorazione, estrarre i vetrini dall'autocoloratore e metterli in un contenitore con tampone di lavaggio.
- Per evitare la contaminazione del sistema autostainer con DAPI, poiché i campioni sono già colorati a concentrazioni molto basse, applicare DAPI manualmente prima di coprire i vetrini con vetrini. Aggiungere due gocce di DAPI per mL di tampone di lavaggio e incubare per 5 minuti a temperatura ambiente al buio.
NOTA: Per la creazione di librerie spettrali, è importante non avere alcun DAPI colorato nei campioni. È inoltre possibile una goccia di DAPI per mL di tampone di lavaggio e 10 minuti di incubazione a RT. - Lavare i vetrini 3 volte con un tampone di lavaggio.
- Posizionare i vetrini su carta assorbente e picchiettare il tampone di lavaggio in eccesso dai vetrini.
- Pipett alcune gocce di terreno di montaggio sul tessuto.
- Posizionare delicatamente un vetrino coprioggetti di vetro sopra il supporto di montaggio per coprire il vetrino ad angolo per evitare bolle d'aria.
- Rimuovere il mezzo di montaggio in eccesso e le bolle d'aria spingendo delicatamente il vetrino coprioggetti in vetro con una pinza o una punta di pipetta pulita.
- Lasciare i vetrini indisturbati per ~24 ore prima che il mezzo di montaggio si solidifichi, orizzontalmente su una scheda di vetrini per microscopia o caricarli direttamente nel microscopio per l'imaging.
- Dopo che il mezzo di montaggio si è solidificato o dopo che i vetrini sono stati riprodotti, conservare i vetrini in scatole per microscopia a 4 °C.
4. Imaging utilizzando l'imager patologico digitale e annotazione dei file di scansione
- Accendere la termocamera premendo il pulsante di alimentazione a destra della macchina. Dopo almeno 20 s, avviare il software.
NOTA: Attendere 20 s per consentire il corretto avvio dell'hardware. - Caricare i vetrini nelle cassette per quattro vetrini.
- Facoltativo: inserire le diapositive in un file .csv per il quale è possibile scaricare un modello (File supplementare 5). Per caricare il file .csv nel programma, salvarlo in C:\Users\Public\Akoya\VectraPolaris\States.
NOTA: È possibile caricare contemporaneamente un massimo di 20 cassette o 80 vetrini.
- Facoltativo: inserire le diapositive in un file .csv per il quale è possibile scaricare un modello (File supplementare 5). Per caricare il file .csv nel programma, salvarlo in C:\Users\Public\Akoya\VectraPolaris\States.
- Impostazioni di riferimento
- Apri Check Dashboard dal menu principale.
NOTA: Una cassetta con slitte di riferimento è fornita dal produttore e può essere conservata in modo permanente nella fessura 20. - Impostare i riferimenti in campo chiaro sul vetrino fornito una volta alla settimana secondo le istruzioni del produttore (richiede alcuni minuti).
- Impostare i riferimenti di fluorescenza sul vetrino fornito una volta al mese secondo le istruzioni del produttore (richiede più di 1 ora).
- Apri Check Dashboard dal menu principale.
- Creazione o modifica del protocollo
- Torna al menu principale e fai clic su Modifica protocollo per creare un protocollo.
- Fare clic su Nuovo... e selezionare Fluorescenza come Modalità di imaging, Scansione di vetrini multispettrali e Colore Opal Polaris 5, 6 e 7 sotto l'opzione Colorazione .
- Assegna un nome al protocollo in Nome protocollo e salvalo in uno studio selezionando uno studio da Studi disponibili o crea uno studio in Crea nuovo studio | Nome dello studio.
- Termina selezionando Crea protocollo.
- Per questo tipo di scansione, utilizzare solo la finestra di sinistra Impostazioni scansione vetrini multispettrali; ignorare la finestra a destra Impostazioni campo multispettrale.
- Scansiona i vetrini con diversi ingrandimenti. Per seguire questo protocollo, eseguire la scansione con un ingrandimento di 20x lasciando la risoluzione dei pixel a 0,50 μm (20x).
- Impostare i tempi di esposizione selezionando Scansiona esposizioni.
- Caricare la cassetta in cui sono conservate le guide selezionando l'apposita fessura sotto l'opzione Portapacchi .
- Per facilitare la navigazione tra i vetrini, selezionare Acquisisci panoramica per acquisire un'immagine panoramica del supporto contenente i vetrini dopo il caricamento del supporto. Per attivare o disattivare automaticamente questa opzione, fai clic sull'icona a forma di ingranaggio in alto a destra, vai su Preferenze... e seleziona l'opzione attiva o disattivata in Immagine panoramica di navigazione per abilitare Vettore immagine automaticamente durante il caricamento per le attività interattive.
- Impostare i tempi di esposizione per filtro sui vetrini colorati IHC monoplex corrispondenti selezionando Imposta esposizioni di scansione e trovando punti diversi con un segnale positivo. Mettere a fuoco manualmente o utilizzare la messa a fuoco automatica e selezionare Esposizione automatica dopo essere passati al filtro compatibile per quel segnale. Selezionare il tempo di esposizione più basso per evitare la sovraesposizione e scattare istantanee di ciascun vetrino come riferimento dopo aver impostato tutti i tempi di esposizione (Figura 3).
NOTA: Ignorare l'opzione Imposta esposizioni campo per questo tipo di scansione. - Impostare i tempi di esposizione su un vetrino colorato multiplex controllando tutti i filtri in alcune posizioni con segnale positivo. Riduci del 10% il tempo di esposizione automatica più basso per evitare la sovraesposizione e scatta alcune istantanee dopo aver impostato tutti i tempi di esposizione.
- Scattare istantanee del vetrino non colorato per la compensazione dell'autofluorescenza utilizzando il filtro AF campione per navigare (Figura 3H).
NOTA: Le sedi con eritrociti e strutture di collagene sono interessanti. Potrebbe essere necessario ridurre il tempo di esposizione del filtro Opal480 per le regioni con forte autofluorescenza. Se il segnale Opal480 è abbastanza forte, dovrebbe comunque essere ben separato (vedi sezione 6) dalle strutture autofluorescenti a causa dell'implementazione del filtro AF campione proprietario. - Valutare la qualità della colorazione e dell'imaging utilizzando il software (vedere le sezioni 5 e 6; Figura 4, File supplementare 6: Figura supplementare S1 e Figura supplementare S2).
- Selezionare il pulsante Salva... per assicurarsi che il protocollo e i relativi tempi di esposizione regolati vengano salvati nel protocollo.
NOTA: Quando il protocollo è già stato salvato, il software non ha ancora ricevuto alcuna notifica aggiuntiva per le regolazioni non salvate.
- Scansione automatica dei vetrini
- Torna al menu principale e fai clic su Scansiona diapositive per scansionare le diapositive.
- Inserire manualmente i nomi/ID delle diapositive e le attività e il protocollo corrispondenti in Configura attività o automaticamente dal file .csv precedentemente creato con Carica configurazione.
- Fare clic su Scansione per avviare la scansione.
- Attendi che venga visualizzata una finestra per salvare la configurazione della scansione. Fare clic su Salva per utilizzare le impostazioni predefinite e avviare la scansione.
NOTA: La scansione con questo metodo richiede ~10-20 minuti per diapositiva. A seconda del numero di diapositive, la scansione può richiedere fino a un'intera giornata. - Verificare se la scansione delle diapositive è stata eseguita correttamente per tutte le diapositive cercando eventuali messaggi di errore. Per sapere se la scansione è andata a buon fine, cerca un file di scansione dell'intero vetrino Akoya salvato (.qptiff) della scansione e il tessuto completo nella scansione.
5. Annotazione dei dati utilizzando il visualizzatore di diapositive
- Torna al menu principale e fai clic su Avvia Phenochart per aprire il visualizzatore di diapositive.
- Se i file di scansione non sono direttamente visibili, indica la loro posizione facendo prima clic sull'icona a forma di ingranaggio nell'angolo in alto a destra, vai su Cambia posizione browser... e seleziona casualmente uno dei file .qptiff del set di dati di interesse.
NOTA: Per impostazione predefinita, i dati vengono memorizzati in D:\Data\VectraPolaris. - Carica una diapositiva selezionandola e facendo clic su Carica nell'angolo in alto a destra o facendo doppio clic su di essa.
- Accedi facendo clic sul pulsante Accedi nell'angolo in alto a destra.
NOTA: Il nome utente può essere solo le iniziali o il nome e viene utilizzato per tenere traccia di chi ha fatto quali annotazioni. - Per eseguire l'unmixing, fare clic sul pulsante Unmixing in alto e selezionare l'opzione Opal + AF .
NOTA: Questo è utile per eliminare parte del segnale autofluorescente vicino al canale Opal 480, ma non tutto. - Per generare un algoritmo per l'elaborazione batch dei dati, selezionare le immagini rappresentative utilizzando l'opzione Timbro utilizzando l'opzione per le immagini 1 x 1 di inForm Projects (dimensione immagine: 928 μm x 696 μm).
NOTA: Alcuni timbri rappresentativi contenenti tumore, stroma, background e diversi tipi di cellule immunitarie sono selezionati in tutto il set di dati per ottenere ~ 20-30 immagini. - A seconda di ciò che deve essere analizzato nel tessuto, selezionare una regione di interesse utilizzando l'opzione ROI e selezionare inForm Batch. Elimina manualmente le immagini che non devono essere analizzate, come le immagini troppo lontane dal tumore o sullo sfondo.
NOTA: Tendiamo a disegnare una ROI attorno all'intero tumore e selezioniamo un'immagine in più lontano dalla regione del tumore per essere in grado di analizzare un IM di ~0,5 mm.
Se il ROI disegnato è relativamente piccolo, il ROI consisterà in 2-9 immagini 20x unite. Poiché questo non è preferito da noi, timbri manualmente il tessuto di interesse (selezionato per inForm Batch) per aggirare questo problema. - Al termine dell'annotazione, lascia che le annotazioni vengano salvate automaticamente e carica la diapositiva successiva.
- Durante il processo di annotazione, verificare che le diapositive siano state scansionate correttamente.
- Se manca un file .qptiff o se un vetrino non è stato scansionato correttamente, verificare se è presente del tessuto sul vetrino, pulire il vetrino con etanolo al 70% ed eseguire nuovamente la scansione.
- Se il tessuto non è completamente scansionato, mancando quindi una regione potenzialmente importante (tumorale), o se la scansione della regione importante era sfocata, pulire il vetrino con etanolo al 70% ed eseguire nuovamente la scansione.
NOTA: In entrambi i casi, può anche essere utile circondare il tessuto con un pennarello sulla parte superiore del vetrino coprioggetti per aiutare il sistema a localizzare il tessuto e riprovare a scansionare (File supplementare 6: Figura supplementare S3). Nelle nostre mani, un pennarello rosso sottile funzionava meglio di un pennarello nero spesso.
- Una volta completate la scansione e l'annotazione di tutti i campioni, è possibile eseguire il backup dei dati archiviandoli su un computer diverso o su un disco esterno.
6. Rimescolamento spettrale
- Aprire il software di analisi automatizzata delle immagini inForm.
- Caricare le immagini nel software tramite File | Apri immagine; Selezionare File .qptiff. Lascia che i timbri, contrassegnati come progetti inForm nel passaggio 5.6, vengano caricati nel progetto.
- Caricare i file .qptiff di cui viene creata l'immagine per la compensazione dell'autofluorescenza.
- Per compensare l'autofluorescenza, utilizzare l'opzione seleziona autofluorescenza sullo strumento immagine per tracciare una linea sull'immagine dal vetrino non colorato attraverso diversi tipi di strutture autofluorescenti, come eritrociti e collagene.
- Nella sezione Modifica Marcatori e Colori... , assegna i nomi dei marcatori che corrispondono al fluoroforo Opale e regola il colore su quello che preferisci.
- Per dismescolare i fluorofori, seleziona Prepara tutto nell'angolo in basso a sinistra.
- Esamina le immagini e controlla se tutti i segnali sono visibili nelle immagini e se il missaggio è andato bene. Seleziona l'icona del bulbo oculare per spegnere e riaccendere tutti i marcatori uno per uno per verificarne la qualità.
- Facoltativamente, addestrare gli algoritmi per la segmentazione dei tessuti, la segmentazione cellulare e la fenotipizzazione.
- Vai alla scheda Esporta e crea una nuova directory di esportazione vuota facendo clic sul pulsante Sfoglia... sotto la directory di esportazione.
- In Immagini da esportare:, selezionare Immagine composita e Immagini componenti (TIFF multi-immagine).
- Seleziona File | Salva | Progetto per salvare l'algoritmo in una determinata posizione.
- Vai alla scheda Analisi batch verticalmente a sinistra per l'elaborazione batch delle diapositive.
- Seleziona Crea directory separate per ogni elemento in Opzioni di esportazione.
- Per aggiungere diapositive per l'analisi, selezionare i file .qptiff sotto il pulsante Aggiungi diapositive... e caricarli nell'analisi batch.
- Selezionare Esegui per avviare l'elaborazione batch delle diapositive.
7. Disegno del ROI
- Creare una cartella con solo i file dei componenti della sezione 6, ma mantenere intatta la struttura gerarchica delle cartelle (i file dei componenti si trovano in cartelle denominate da campione/diapositiva).
- Apri il software di visualizzazione di diapositive intere QuPath.
- Fai clic su Crea progetto a sinistra e seleziona/crea una nuova cartella vuota con un nome adatto.
- Fare clic su Automatizza e selezionare Mostra editor di script.
- Copiare e incollare lo script disponibile nel file supplementare 7. Alla riga 34, modificare la posizione in cui si trovano le cartelle delle diapositive contenenti tutti i file dei componenti (la cartella creata nel passaggio 7.1.
- Seleziona Esegui e ritorna quando l'unione batch delle diapositive è terminata (il giorno successivo o più tardi) per continuare.
- Trascina i file .ome.tif generati nel progetto QuPath e salvali come progetto.
- Quando viene visualizzata automaticamente una nuova finestra, selezionare Imposta tipo di immagine | Fluorescenza e fare clic su Importa.
- Nel menu a sinistra, osservare l'elenco dei campioni; fare doppio clic su uno di essi per aprire l'esempio (Figura 5A).
- Per regolare l'intensità dei canali in modo da renderli più visibili, fare clic sull'icona del contrasto.
- Seleziona tutti i canali e fai clic su Ripristina.
- Disattiva l'autofluorescenza.
- Per iniziare a tracciare una ROI per il tumore, fai clic sull'icona del contrasto e seleziona Mostra scala di grigi. Selezionare il canale del marcatore tumorale e regolare l'intensità per renderlo visibile in modo ottimale (Figura 5B).
- Fare clic sullo strumento pennello per disegnare approssimativamente il ROI di un tumore.
- Mentre si seleziona lo strumento bacchetta, fare clic all'esterno della ROI mentre si tiene premuto il tasto alt per attenuare la ROI dall'esterno (Figura 5C).
- Unisci pezzi di tumore separati con lo stesso ROI.
- Assegna al ROI un nome appropriato, ad esempio tumore , facendo clic con il pulsante destro del mouse sull'annotazione nell'elenco a sinistra; selezionare Imposta proprietà e immettere il nome.
- Per ottenere una ROI per l'IM, espandere la ROI esistente dalla regione del tumore selezionando: Oggetti | Annotazioni... | Espandi le annotazioni.
- Selezionare le dimensioni del raggio di espansione e selezionare Rimuovi interno e Vincola al genitore (Figura 5D).
- Fare clic sull'icona di contrasto, selezionare il canale di autofluorescenza e regolare l'intensità per renderlo visibile in modo ottimale.
- Fai clic sulla bacchetta e regola la ROI mentre premi il tasto alt per attenuare la ROI dall'esterno e rimuovere qualsiasi sfondo che non dovrebbe far parte di questa ROI.
- Assegna al ROI un nome appropriato, ad esempio margine invasivo o IM , facendo clic con il pulsante destro del mouse sull'annotazione nell'elenco a sinistra, seleziona Imposta proprietà, inserisci il nome e, facoltativamente, cambia il suo colore in verde.
- Salvare le annotazioni: File | Esportazione di oggetti | Esporta tutti gli oggetti e fare clic su OK con la selezione di default su Esporta come raccolta feature e salvarla in una posizione preferita.
8. Rilevamento delle cellule immunitarie
- Poiché ImmuNet utilizza i dati dei componenti (file TIFF multicanale) sia per l'addestramento che per l'inferenza, suddividi le annotazioni in set di addestramento e convalida. Per addestrare il modello, seguire i passaggi descritti nel file Leggimi del repository, sostituendo il set di dati di esempio e le annotazioni con i dati desiderati. Oltre alle diverse cellule immunitarie, fornire al modello esempi negativi effettuando annotazioni di fondo in siti che non dovrebbero essere riconosciuti come cellule di interesse: cellule tumorali, altre cellule o "nessuna cellula" (strutture che potrebbero essere confuse con cellule di interesse); si veda la pubblicazione ImmuNet per i dettagli32.
- Utilizzando le annotazioni di convalida, assicurarsi che le prestazioni siano soddisfacenti. Esamina il tasso di errore per tipo di annotazione, ovvero una quota di annotazioni di convalida che il modello non ha rilevato, la metrica di valutazione più semplice. Valuta le prestazioni rispetto ai falsi positivi effettuando alcune ROI completamente annotate e calcolando i punteggi F.
- Oltre alla valutazione quantitativa, ispezionare visivamente la previsione per avere un'idea qualitativa degli errori che il modello tende a commettere (Figura 6, File supplementare 6: Figura supplementare S4 e Figura supplementare S5). Se le prestazioni del modello vengono giudicate insufficienti, visualizzare la previsione per alcuni riquadri come descritto nel repository e verificare quali siti sono i più soggetti a errori. Effettua più annotazioni in tali siti ed esegui nuovamente l'addestramento e la valutazione del modello.
- Una volta raggiunte le prestazioni target, eseguire l'inferenza per l'intero set di dati come descritto nella sezione Inferenza per l'intero set di dati del file Leggimi del repository. Utilizzare i file .csv ottenuti con la previsione del modello come input per l'analisi dei dati (scrivere uno script Python o R per questo).
9. Fenotipizzazione predittiva e analisi dei dati
NOTA: In questa sezione, forniamo un esempio di semplice analisi dei dati per un singolo campione di melanoma colorato con il pannello dei linfociti, che combina le posizioni delle cellule immunitarie identificate da ImmuNet (sezione 8) e le ROI delineate con QuPath (sezione 7). L'analisi è stata eseguita in R 4.1.1 (uno script viene fornito come file supplementare 8). Lo script richiede i pacchetti: plyr 1.8.8, dplyr 1.0.8, tidyr 1.2.0, sf 1.0-7, ggplot2 3.4.0, RANN 2.6.1 e RColorBrewer 1.1-2, che possono essere installati con il comando install.packages(). Come input, prende un file .csv con la previsione di ImmuNet di un campione e un file con i ROI esportati da QuPath. I passaggi 9.1-9.6 descrivono l'analisi di un singolo campione eseguita nello script fornito, mentre le sezioni 9.7-9.9 descrivono le opzioni per l'analisi di più campioni.
- Dopo aver caricato la previsione di ImmuNet in R, determinare le soglie per l'espressione dei marcatori previsti tracciando i marcatori che definiscono i fenotipi l'uno rispetto all'altro e selezionando le soglie che separano meglio le popolazioni.
NOTA: La strategia di gating utilizzata per il campione dato è mostrata nella Figura 7B. Le strategie di gating per i pannelli cellulari mieloidi e dendritici sono mostrate nel File Supplementare 6: Figura Supplementare S6 e Figura Supplementare S7. - Dopo aver determinato le soglie, utilizzarle per assegnare a ogni previsione ImmuNet un fenotipo definito in un pannello. In alcune previsioni, si osservi che nessuno dei marcatori previsti è al di sopra della soglia o che la combinazione di marcatori considerata espressa dopo la soglia può essere incoerente (ad esempio, previsioni CD3+ CD20+ nei pannelli dei linfociti). Se al passaggio 8.3 si ottengono buone prestazioni del modello, la frazione di tali previsioni sarà piccola; Filtrali prima dell'analisi.
- Per analizzare separatamente le ROI per il tumore e il suo margine invasivo fino a 100 μm disegnato in QuPath, caricare i file GeoJSON corrispondenti in R e, per ogni previsione, determinare la ROI in cui rientra la previsione.
- Per un controllo di integrità e come parte dell'analisi esplorativa dei dati, visualizzare le cellule immunitarie presenti in un campione separatamente nelle ROI corrispondenti insieme ai limiti delle ROI (Figura 7A).
- Ora, calcola le densità delle diverse cellule immunitarie separatamente per ogni ROI. Le densità riscontrate nel campione indicato sono illustrate nella Tabella 1.
- Se sono disponibili più campioni, visualizzare la distribuzione delle densità cellulari. Trasforma logaritmicamente i valori di densità per ottenere valori distribuiti normalmente.
NOTA: quando i conteggi di determinati fenotipi sono 0, questi non possono essere trasformati in log, portando a valori mancanti. Per ovviare a questo problema, il livellamento LaPlasiano può essere applicato aggiungendo 0,5 a tutti i conteggi delle celle prima di dividere per l'area superficiale. - Analizza i valori di densità e tracciali utilizzando il software di tua scelta (Figura 8).
- Le posizioni conservate delle celle consentono l'analisi spaziale. Ad esempio, per ogni cellula immunitaria rilevata, trova un vicino più vicino e quindi per ogni fenotipo, calcola la percentuale di casi in cui i diversi fenotipi si verificano come il vicino più prossimo.
NOTA: Poiché il numero di cellule natural killer (NK) trovate in questo campione era molto piccolo, le abbiamo escluse da questa analisi. I risultati ottenuti per i ROI del tumore e dell'IM sono riportati nella Figura 9.
Risultati
I blocchi FFPE contenenti tessuto tumorale sono stati selezionati sulla base di referti patologici e vetrini colorati con HE. Quando più lesioni tumorali vengono resecate dal paziente e/o i campioni tumorali sono grandi, questi vengono divisi su più blocchi FFPE. Preferiamo analizzare le cellule immunitarie sia nel compartimento tumorale che in quello che è noto come margine invasivo (IM) del tumore. L'IM è un tessuto stromale non canceroso adiacente al tumore. Pertanto, quando sono disponibili più blocchi FFPE per un campione tumorale, vengono selezionati i blocchi FFPE che contengono entrambi i tipi di tessuto. Come si vede sui vetrini colorati con HE, un blocco FFPE conteneva tessuto tumorale e tessuto stromale adiacente al tumore (Figura 1A). Un altro blocco FFPE dello stesso tumore conteneva molto meno tessuto stromale circostante (Figura 1B). Tuttavia, per alcuni campioni di tessuto non c'è scelta nei blocchi FFPE o l'IM non è presente in nessuno dei blocchi FFPE. Questo è spesso il caso delle biopsie (con ago), che devono essere tenute a mente durante l'interpretazione dei dati.
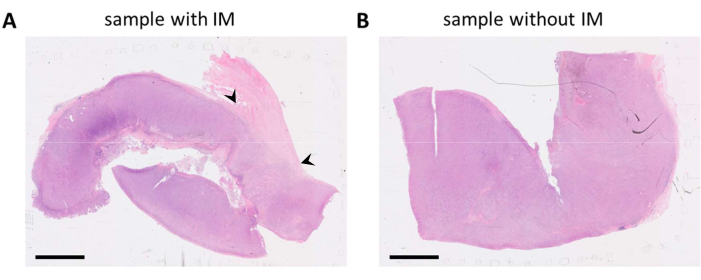
Figura 1: Vetrini colorati con HE di un campione di tumore di melanoma. (A) Un esempio di un campione di tumore con tessuto stromale adiacente al tumore (IM) nell'angolo in alto a destra del campione (indicato con punte di freccia nere). (B) Un altro campione della stessa lesione tumorale con poco o nessun tessuto stromale presente nel campione. Barre della scala = 5 mm. Abbreviazioni: HE = ematossilina ed eosina; IM = margine invasivo. Clicca qui per visualizzare una versione più grande di questa figura.
La colorazione IHC multiplex con un pannello a sette colori proposto (File supplementare 4) può essere eseguita manualmente in un processo di colorazione di 3 giorni (considerando il normale orario di lavoro) o durante la notte in un autostainer. Quando si utilizza l'autostainer, le sezioni devono essere montate in una posizione particolare sul vetrino che consenta una fluidica ottimale del sistema (Figura 2A). Quando le sezioni sono montate correttamente sulle guide (Figura 2B), saranno macchiate uniformemente (Figura 2C). Se le sezioni non sono montate in modo ottimale sul vetrino (Figura 2D), spesso si verifica un modello di colorazione non ottimale (Figura 2E) perché la fluidica dell'autocolorante non raggiunge il tessuto (completo). Questo può accadere quando i campioni sono molto grandi o quando i vetrini montati vengono forniti da qualcuno che non è a conoscenza di questo problema. In questi casi, solo la parte ben colorata del vetrino deve essere selezionata per l'analisi. Un'altra scelta per questi tipi di campioni potrebbe essere quella di colorarli manualmente per distribuire i liquidi in modo ottimale.
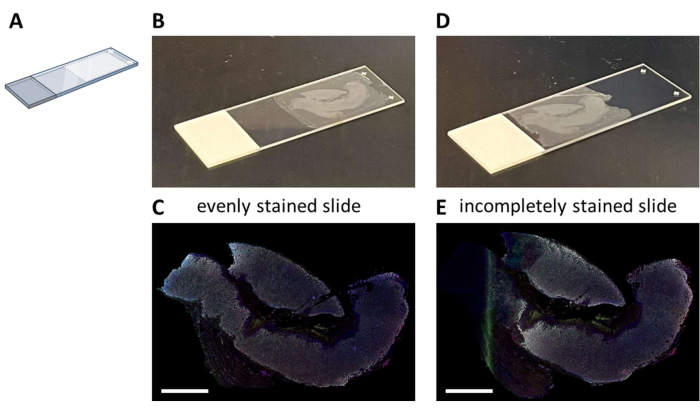
Figura 2: Montaggio della sezione FFPE sul vetrino e impatto. (A) Schema di dove montare sul vetrino per una colorazione ottimale sull'autostainer. (B) Esempio di una guida montata correttamente. (C) I vetrini montati correttamente producono una sezione di tessuto uniformemente colorata. (D) Esempio di una guida montata in modo non ottimale. (E) I vetrini montati in modo non ottimale possono portare a una sezione di tessuto colorata incompleta, come si vede sul lato sinistro di questa immagine. Barre della scala = 5 mm. Abbreviazione: FFPE = fissato in formalina e incluso in paraffina. Clicca qui per visualizzare una versione più grande di questa figura.
Quando si eseguono esperimenti IHC multiplex di grandi dimensioni in più cicli di colorazione ed è necessario preparare grandi quantità di soluzioni, è meglio testare questi reagenti in una corsa IHC monoplex prima di procedere con l'IHC multiplex. L'IHC monoplex viene controllato con l'imager di patologia digitale per verificare la presenza di modelli di colorazione previsti e i tempi di esposizione vengono impostati con i filtri corrispondenti sui vetrini di controllo (Figura 3A-H). Il tessuto tonsillare viene utilizzato come controllo positivo per la maggior parte dei marcatori delle cellule immunitarie. Poiché il tempo di esposizione al DAPI nel tessuto di controllo delle tonsille è sempre più elevato rispetto ad altri tessuti (Figura 3G), il tempo di esposizione al DAPI deve essere impostato sul tipo di tessuto da studiare. I tempi di esposizione regolari con questo tipo di scansione sono compresi tra 1 ms e 30 ms, a seconda del fluoroforo e del filtro (Figura 3I). Quando un IHC monoplex supera questi numeri o il modello di colorazione non è chiaro come previsto, la soluzione anticorpale deve essere aggiustata o sostituita. Nell'esempio qui mostrato, abbiamo deciso di aumentare la concentrazione di FOXP3 (Figura 3C e Figura 3I) per avere l'intensità più in linea con gli altri marcatori. L'autofluorescenza può anche essere più forte in altri tessuti rispetto al tessuto di controllo delle tonsille. Nella nostra impostazione, il tempo di esposizione per il filtro AF campione è compreso tra 25 ms e 50 ms (Figura 3H,I).

Figura 3: Impostazione dei tempi di esposizione su IHC monoplex e campioni di controllo non colorati. (A) Segnale CD20 - Opal 480 nel tessuto di controllo delle tonsille. (B) CD3 - Segnale opale 520 nel tessuto di controllo delle tonsille. (C) FOXP3 - Segnale dell'opale 570 nel tessuto di controllo delle tonsille. (D) CD56 - Segnale dell'opale 620 nel tessuto di controllo delle tonsille (E) CD8 - Segnale dell'opale 690 nel tessuto di controllo delle tonsille. (F) Marcatore tumorale - Segnale opale 780 nel tessuto di controllo delle tonsille. (G) Il segnale DAPI nel tessuto di controllo delle tonsille è spesso più debole del tipo di tessuto di interesse. (H) Autofluorescenza - campione di segnale AF nel tessuto di controllo del tumore. (I) Screenshot dei tempi di esposizione prima di regolarlo con il 10% e controllare su vetrini colorati IHC multiplex. Barre di scala = 100 μm. Abbreviazioni: AF = autofluorescenza; IHC = immunoistochimica; DAPI = 4'6-diamidino-2-fenilidolo. Clicca qui per visualizzare una versione più grande di questa figura.
Dopo aver eseguito l'IHC multiplex, i tempi di esposizione vengono regolati dalle impostazioni IHC monoplex controllando alcuni vetrini IHC multiplex e selezionando l'esposizione automatica. Con questo tipo di scansione, non esiste un'opzione di protezione dalla saturazione e quindi è estremamente importante evitare di impostare un'esposizione troppo alta, evitando così la sovraesposizione. La sovraesposizione ostacola la miscelazione spettrale dei fluorofori. Spesso non impostiamo tempi di esposizione superiori ai tempi di esposizione basati sull'IHC monoplex e diminuiamo i tempi di esposizione solo per i marcatori che sono più forti nell'IHC multiplex (Figura 3G e Figura 4A). Eseguendo l'esposizione automatica in posizioni diverse su alcune diapositive, si può osservare che i tempi di esposizione di alcuni filtri sono ancora troppo elevati. Questi devono essere regolati sul numero più basso che si osserva quando si utilizza l'impostazione di esposizione automatica e sottrarre un altro 10% del valore per evitare la sovraesposizione in altri luoghi invisibili (Figura 4A). Con questo metodo, i tempi di esposizione possono essere inferiori per alcuni filtri rispetto a quelli impostati su IHC monoplex. Tuttavia, con un esperimento IHC multiplex riuscito, tutti i marcatori dovrebbero essere osservabili, almeno sul vetrino di controllo (Figura 4B-H, File supplementare 6: Figura supplementare S1 e Figura supplementare S2). Considera che alcuni marcatori potrebbero non essere presenti in tutti i campioni. Includendo un vetrino di controllo contenente almeno una sezione tonsillare, è possibile verificare la corretta colorazione di tutti i marcatori dei pannelli standard e l'intensità del segnale.
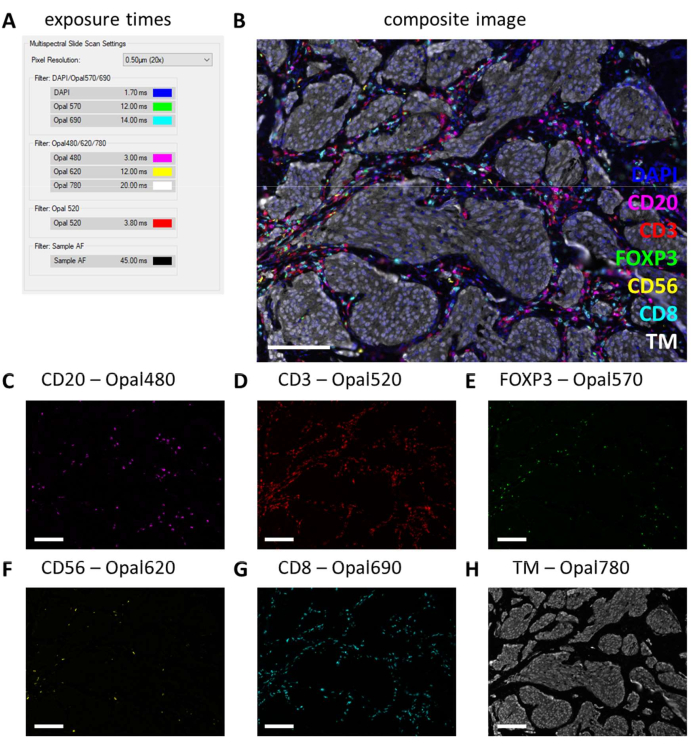
Figura 4: Esempio di una sezione colorata con successo con il pannello dei linfociti in un campione di tumore di melanoma. (A) Tempi di esposizione utilizzati per registrare questo campione IHC multiplex. (B) Immagine composita del pannello linfocitario IHC multiplex all'interno del tessuto tumorale. (C) CD20 - Segnale opale 480 in magenta. (D) CD3 - Segnale opale 520 in rosso. (E) FOXP3 - Segnale opale 570 in verde. (F) CD56 - Segnale opale 620 in giallo. (G) CD8 - Segnale opale 690 in ciano. (H) TM - Opale 780 in bianco. Barre di scala = 100 μm. Abbreviazione: TM = marcatore tumorale; IHC = immunoistochimica. Clicca qui per visualizzare una versione più grande di questa figura.
I vetrini IHC multiplex vengono scansionati completamente dall'imager digitale. I riquadri per l'analisi successiva vengono selezionati nel visualizzatore diapositive. Tuttavia, quando è necessario analizzare regioni più specifiche, come il tumore rispetto all'IM, queste regioni di interesse (ROI) possono essere disegnate utilizzando QuPath. Al termine dell'elaborazione batch delle tessere selezionate nel visualizzatore diapositive, i file dei componenti vengono uniti nuovamente (Figura 5A e File supplementare 7). Utilizzando il canale del marcatore tumorale (Figura 5B) e lo strumento bacchetta magica in QuPath, è possibile tracciare il contorno del tumore per formare il "ROI del tumore" (Figura 5C). Successivamente, la ROI del tumore può essere espansa con una certa distanza, in questo caso 500 μm, per creare una "ROI del margine invasivo" (Figura 5D). Qualsiasi sfondo indesiderato (non tessuto) viene rimosso da questa ROI con lo strumento bacchetta magica osservando il segnale di autofluorescenza (Figura 5E). Sia la ROI del tumore che la ROI IM vengono salvate come file GeoJSON per un'ulteriore elaborazione (Figura 5F).
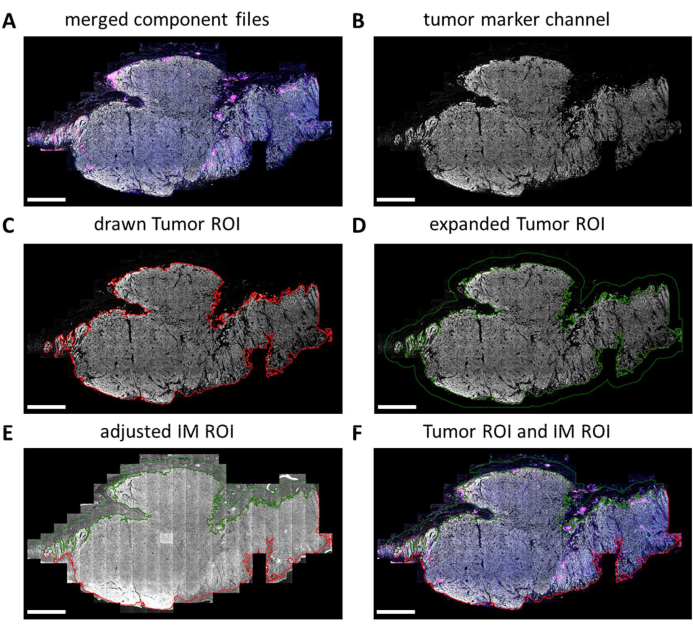
Figura 5: Processo di disegno del ROI del tumore e del margine invasivo in QuPath. (A) File dei componenti uniti. (B) Immagine in scala di grigi che mostra solo il canale del marcatore tumorale. (C) La ROI del tumore viene disegnata attorno al segnale del marcatore tumorale. (D) Un nuovo ROI viene realizzato espandendo il ROI del tumore di 100-500 μm per formare il ROI IM. (E) Il ROI IM viene regolato per includere solo il tessuto stromale escludendo il fondo (segnale negativo) e altre strutture tissutali di grandi dimensioni come grasso, vasi sanguigni e follicoli piliferi. (F) Il ROI del tumore e il ROI IM risultanti vengono salvati ed esportati in file GeoJSON per un'ulteriore elaborazione delle regioni. La ROI del tumore viene visualizzata con un contorno rosso e la ROI IM con un contorno verde. Barre della scala = 2 mm. Abbreviazioni: ROI = regione di interesse; IM = margine invasivo; GeoJSON = Notazione geografica dell'oggetto JavaScript. Clicca qui per visualizzare una versione più grande di questa figura.
Le reti ImmuNet possono essere utilizzate per rilevare le cellule immunitarie. Per il pannello dei linfociti, l'immagine composita sperimentale (Figura 6A) può essere confrontata visivamente con le cellule immunitarie rilevate dal software (Figura 6B). Confronti visivi simili possono essere fatti per il pannello mieloide (File supplementare 6: Figura supplementare S4) e il pannello delle cellule dendritiche (File supplementare 6: Figura supplementare S5).
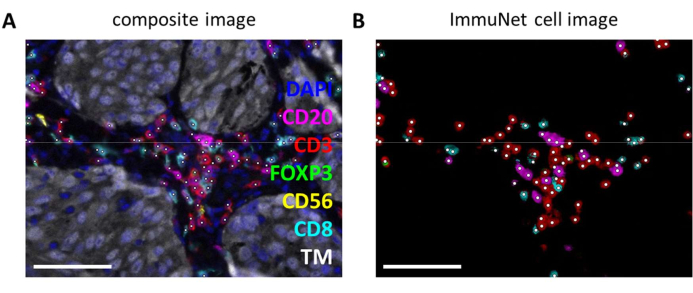
Figura 6: Linfociti riconosciuti da ImmuNet. (A) Immagine composita della Figura 4B che mostra le cellule riconosciute da ImmuNet con punti bianchi. (B) Cellule riconosciute da ImmuNet e successiva espressione di marcatori rilevati. Barre di scala = 50 μm. Abbreviazione: TM = marcatore tumorale. Clicca qui per visualizzare una versione più grande di questa figura.
Le cellule immunitarie rilevate da ImmuNet e salvate in .csv formato possono essere importate in qualsiasi linguaggio di programmazione per ulteriori analisi. Abbiamo eseguito la visualizzazione spaziale e il gating in R (File supplementare 8). Le cellule rilevate possono quindi essere visualizzate spazialmente (Figura 7A, File supplementare 6: Figura supplementare S6 e Figura supplementare S7). Il gating sull'espressione di pseudomarcatori può essere eseguito per fenotipizzare le singole cellule immunitarie (Figura 7B).
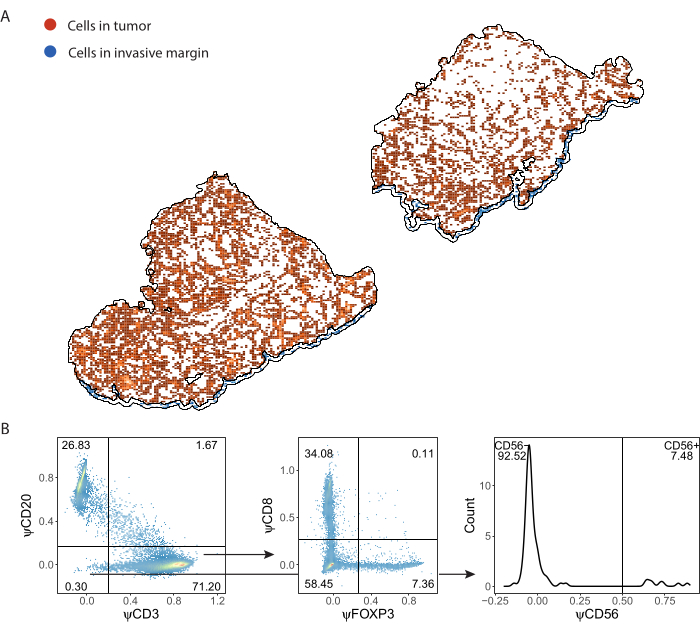
Figura 7: Strategia di gating del pannello linfocitario. (A) Cellule immunitarie rilevate nelle regioni di interesse del tumore e del margine invasivo delineate con QuPath. (B) Gating di tutte le cellule rilevate da ImmuNet dalla parte A. I linfociti vengono prima controllati dalle cellule B CD20+ e dalle cellule T CD3+. Le cellule T CD3+ sono ulteriormente gated per l'espressione di CD8 e FOXP3. La popolazione CD20-CD3- è controllata per le cellule natural killer CD56+. Clicca qui per visualizzare una versione più grande di questa figura.
Quando i fenotipi delle cellule previste vengono determinati con il gating, le densità cellulari di diversi fenotipi possono essere calcolate all'interno di diversi ROI. Questo viene calcolato dividendo il numero totale di cellule per fenotipo per l'area superficiale del ROI (Tabella 1, Figura 8 e File supplementare 8). Qui, le cellule B sono definite come CD3-CD20+, le cellule T helper come CD3+CD20-CD8-FoxP3-, le cellule T regolatorie come CD3+CD20-CD8-FoxP3+, le cellule T citotossiche come CD3+CD20-CD8+FoxP3- e le cellule NK come CD3-CD20-CD56+.
| Fenotipo | Densità nel tumore (cellule/mm2) | Densità in IM (celle/mm2) |
| Cellula B | 185.74 | 145.62 |
| Cellula T helper | 301.46 | 157.51 |
| Cellula T regolatoria | 38.53 | 19.53 |
| Cellula T citotossica | 185.35 | 83.21 |
| Cella NK | 0.18 | 0 |
Tabella 1: Densità dei fenotipi nei ROI. Densità di cellule di diversi fenotipi trovate in un singolo campione di melanoma colorato con il pannello dei linfociti. Le densità sono calcolate separatamente in ROI del tumore e dell'IM. Abbreviazioni: IM = margine invasivo; ROI = regione di interesse.
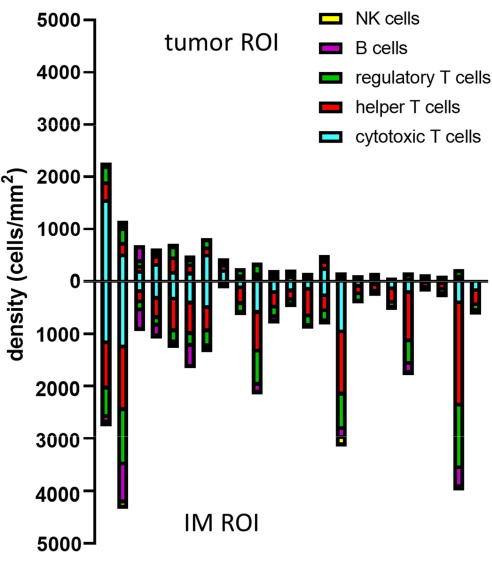
Figura 8: Esempio di analisi dei dati per più campioni. Analisi della densità di diversi fenotipi linfocitari nel tumore e IM di 23 tumori primari del melanoma. Abbreviazioni: IM = margine invasivo; ROI = regione di interesse. Clicca qui per visualizzare una versione più grande di questa figura.
Per approfondire le informazioni spaziali di queste cellule immunitarie, è anche possibile determinare le distanze tra i fenotipi identificati o le percentuali di fenotipi dei vicini più prossimi in un campione (Figura 9).
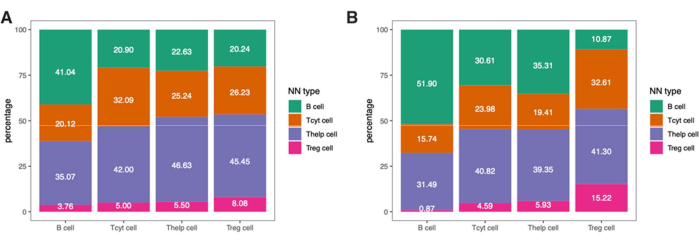
Figura 9: Esempio di analisi del vicino più vicino per un singolo campione. Percentuale di fenotipi più vicini per diversi tipi di cellule in (A) ROI tumorali e (B) IM riscontrati in un singolo campione di melanoma colorato con il pannello linfocitario. Abbreviazioni: IM = margine invasivo; ROI = regione di interesse. Clicca qui per visualizzare una versione più grande di questa figura.
File supplementare 1: Multiplex IHC che riassume le specifiche del protocollo. Clicca qui per scaricare questo file.
File supplementare 2: Protocollo Autostainer per monoplex. Clicca qui per scaricare questo file.
File supplementare 3: Protocollo autostainer per la compensazione dell'autofluorescenza. Clicca qui per scaricare questo file.
File supplementare 4: Protocollo autostainer per immunoistochimica multiplex. Clicca qui per scaricare questo file.
File supplementare 5: Template .csv file. Clicca qui per scaricare questo file.
File 6 supplementare: Pannelli di cellule mieloidi e dendritiche in un campione di tessuto di melanoma; vetrini di marcatura in caso di fallimento della scansione; cellule mieloidi e dendritiche riconosciute da ImmuNet; strategie di gating di pannelli di cellule mieloidi e dendritiche. Clicca qui per scaricare questo file.
File supplementare 7: Script di punto QuPath. Clicca qui per scaricare questo file.
File supplementare 8: script di analisi dei dati. Clicca qui per scaricare questo file.
Discussione
L'analisi spaziale del TME è una tecnica ricercata per saperne di più sul compartimento delle cellule immunitarie e scoprire nuovi biomarcatori prognostici e predittivi, in particolare nel campo dell'immuno-oncologia16. A questo scopo sono in fase di sviluppo molte tecniche diverse, che prevedono la rilevazione di proteine, trascritti di mRNA o una combinazione dei due, con stime fino a 100-1.000 bersagli. Tuttavia, un multiplexing più elevato comporta un minor numero di esperimenti ad alto rendimento, costi sperimentali più elevati e sfide tecniche e, spesso, solo una piccola parte del TME può essere analizzata. L'IHC multiplex che utilizza il metodo basato su TSA che descriviamo qui, rileva sei diversi marcatori + DAPI contemporaneamente, è relativamente meno costoso da eseguire e intere sezioni di tessuto vengono visualizzate in meno di 20 minuti, pronte per essere analizzate completamente. Questa tecnica è diventata meno complessa con l'automazione della procedura di colorazione. I miglioramenti apportati al microscopio multispettrale, che includono l'aggiunta di due filtri aggiuntivi, hanno migliorato enormemente i tempi di demiscelazione e scansione spettrale. È possibile rilevare contemporaneamente fino a otto diversi marcatori + DAPI. Tuttavia, espandendo il multiplexing con più marcatori, i vantaggi di cui sopra scompaiono poiché la miscelazione spettrale diventa più difficile e i tempi di scansione per interi vetrini aumentano notevolmente. Sono in corso sforzi per standardizzare l'IHC multiplex tra diverse istituzioni per facilitare più facilmente l'implementazione in ambito diagnostico. Per questa standardizzazione dell'IHC multiplex, consigliamo agli utenti di aderire al protocollo più accessibile con sei diversi marcatori + DAPI. Tuttavia, è necessario ancora un po' di know-how tecnico e l'analisi a valle può essere impegnativa, per la quale abbiamo sviluppato metodologie descritte in questo protocollo.
La standardizzazione inizia con lo sviluppo di pannelli IHC multiplex. L'importanza della scelta di anticorpi primari che rilevano particolari bersagli proteici è stata sottolineata prima del17. I nostri pannelli IHC multiplex sono per lo più sviluppati con cloni di anticorpi primari che vengono utilizzati e convalidati per IHC presso il nostro reparto diagnostico. Tuttavia, nel caso del pannello IHC multiplex a cellule dendritiche, la maggior parte degli anticorpi non è stata utilizzata in ambito diagnostico (van der Hoorn et al., manoscritto in presentazione). Per garantire la specificità e ridurre al minimo le differenze tra i lotti, abbiamo scelto di utilizzare anticorpi monoclonali rispetto agli anticorpi policlonali e abbiamo anche convalidato la maggior parte degli anticorpi utilizzando linee cellulari trasfettate e cellule primarie. Nel corso degli anni, diverse versioni di pannelli IHC multiplex sono state utilizzate in numerosi studi utilizzando il sistema Vectra 3 18,21,23,24,25,26,27,28,29,30,31,32 . Per implementare in modo ottimale questi pannelli IHC multiplex sul sistema PhenoImager HT, è stato necessario apportare alcune modifiche alle combinazioni di anticorpi primari e fluorofori. Per beneficiare di una migliore miscelazione spettrale e di tempi di scansione più rapidi di intere sezioni di tessuto, è necessaria l'implementazione dei più recenti fluorofori Opal480 e Opal780 ed evitare l'uso di fluorofori Opal540 e Opal650 nei pannelli IHC multiplex a sette colori. I tempi di scansione sono ~3-10 volte più veloci a seconda delle dimensioni della sezione di tessuto. Le regolazioni del pannello IHC multiplex sono state abbastanza facili da ottenere, ma è necessario tenere a mente alcune considerazioni. Lo spettro fluorescente di Opal480 si sovrappone molto allo spettro di autofluorescenza e quindi interferisce con la miscelazione spettrale degli eritrociti e di altre strutture autofluorescenti. L'utilizzo di una maggiore concentrazione dell'anticorpo primario abbinato a Opal480 ha risolto questo problema nella maggior parte dei casi. L'implementazione del filtro AF proprietario Sample AF sul PhenoImager HT facilita la dismiscelazione di Opal480 e l'autofluorescenza. Tuttavia, è meglio utilizzare un anticorpo primario che produca un segnale chiaro quando viene utilizzato con Opal480 in modo che il suo segnale sia superiore all'autofluorescenza.
Anche se questi pannelli IHC multiplex sono stati stabiliti, la variazione da lotto a lotto è qualcosa che deve essere considerato. Eseguendo controlli IHC monoplex prima di iniziare l'esperimento IHC multiplex completo, a volte abbiamo osservato che gli anticorpi primari si comportano più o meno forti da un esperimento all'altro. Le ragioni di ciò potrebbero essere errori di pipettaggio, condizioni di conservazione dei reagenti non ottimali e durata di conservazione. Abbiamo risolto questo problema regolando la soluzione di anticorpi primari in base alla nostra esperienza. Anche quando non è stato necessario effettuare alcuna delle regolazioni di cui sopra, con ogni esperimento batch IHC multiplex, è importante impostare i tempi di esposizione in base ai vetrini di controllo colorati con IHC monoplex.
Poiché la nostra ricerca si è inizialmente concentrata su diversi tipi di carcinomi e melanoma, i pannelli IHC multiplex dovevano essere intercambiabili tra i tipi di tumore con aggiustamenti minimi. Pertanto, abbiamo sempre incluso più tipi di tessuto (tumorale) nel processo di ottimizzazione e abbiamo osservato che le diluizioni per gli anticorpi primari per i marcatori delle cellule immunitarie possono essere mantenute simili tra i diversi tipi di tumore. Tuttavia, il rilevamento del tessuto tumorale tra carcinomi e melanoma necessita di marcatori tumorali diversi. Di conseguenza, il marcatore tumorale è sempre stato ottimizzato per funzionare all'estremità di ogni pannello IHC multiplex e attualmente viene sempre utilizzato in combinazione con Opal780, che per coincidenza deve essere anche all'ultimo fluoroforo in una procedura di colorazione IHC multiplex. Utilizzando il marcatore tumorale di conseguenza alla fine dell'IHC multiplex, questi pannelli IHC multiplex possono essere facilmente scambiati con altri tipi di tumore, come il glioblastoma (cioè GFAP) e il linfoma di Hodgkin (cioè CD30). Per l'angiosarcoma, abbiamo utilizzato questo pannello IHC multiplex linfocitario con gene correlato alla trasformazione specifica dell'eritroblasto (ERG) come marcatore tumorale con solo due esperimenti di ottimizzazione25. L'ottimizzazione ha incluso la titolazione dell'anticorpo primario ERG e il test del pannello IHC multiplex con ERG alla fine.
Altre regolazioni di questi pannelli IHC multiplex possono essere effettuate anche sostituendo un determinato marcatore di cellule immunitarie con un altro marcatore immunitario o funzionale. Ogni modifica richiede ottimizzazione. Il protocollo per l'ottimizzazione potrebbe essere seguito come descritto in precedenza17. Alcune modifiche ai pannelli IHC multiplex proposti interferiranno con gli algoritmi ImmuNet che abbiamo creato. È necessario generare dati sufficienti e dedicare tempo all'implementazione di queste modifiche nell'algoritmo (almeno 750 annotazioni per ogni nuovo marcatore e/o fenotipo cellulare e 150 annotazioni per la convalida di marcatori precedentemente addestrati). I pannelli qui presentati non contengono marcatori funzionali, sebbene l'implementazione di marcatori di checkpoint immunitari come PD-1 e PD-L1 in pannelli IHC multiplex venga eseguita nel nostro laboratorio. Tuttavia, l'analisi dei marcatori che sono meno binari nei segnali negativi e positivi si è dimostrata più difficile ed è un'area di ricerca attiva nel nostro gruppo.
Il numero di marcatori che possono essere valutati contemporaneamente con l'IHC multiplex è limitato rispetto ad altre nuove tecniche. Sebbene ciò possa essere aggirato analizzando diversi pannelli su sezioni consecutive di un blocco FFPE, sarà difficile confrontare queste sezioni spazialmente. L'orientamento e gli artefatti piegati probabilmente non sono gli stessi dopo la preparazione del vetrino. Tuttavia, l'IHC multiplex è abbastanza accessibile, il che lo rende uno strumento attraente per più istituzioni e ricercatori e, quindi, più adatto per l'implementazione futura in un contesto diagnostico. Con la standardizzazione dei pannelli di cellule immunitarie IHC multiplex per diversi tipi di tumore e le pipeline di analisi a valle, è stato possibile acquisire maggiori conoscenze sulle differenze nel TME tra pazienti e tipi di tumore. Ciò può, ad esempio, portare a maggiori informazioni sul ruolo del TME nella risposta antitumorale a trattamenti specifici. Ciò potrebbe anche dare origine a nuovi biomarcatori per prevedere fattori come la risposta al trattamento e la sopravvivenza attesa. Nel complesso, ciò può consentire all'IHC multiplex di diventare uno strumento clinico per aiutare nel processo decisionale clinico, in un approccio di medicina personalizzata. Certo, più fasi della procedura di analisi dovrebbero probabilmente essere automatizzate e standardizzate per essere fattibili per l'uso in un contesto diagnostico quotidiano, quindi per ora è per lo più una prospettiva futuristica.
L'analisi di più marcatori su un singolo vetrino campione può essere uno strumento molto potente nonostante le sue sfide tecniche. Con protocolli sperimentali standardizzati e un metodo di analisi robusto, come abbiamo descritto qui utilizzando ImmuNet, la quantificazione di più marcatori lo rende più informativo rispetto all'IHC classico, mentre l'IHC multiplex rimane relativamente ad alto rendimento rispetto ai nuovi metodi sperimentali a plesso superiore.
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Il PhenoImager HT è stato acquistato grazie ai finanziamenti forniti dal Radboud University Medical Center e dal Radboud Technology Center for Microscopy. La fibrosi cistica è sostenuta finanziariamente da una sovvenzione della Dutch Cancer Society (10673) e da una sovvenzione ERC Adv ARTimmune (834618). JT è sostenuto finanziariamente da una sovvenzione NWO Vidi (VI.Vidi.192.084). Gli autori desiderano ringraziare Eric van Dinther e Ankur Ankan per la loro assistenza nella creazione di flussi di lavoro per archiviare i dati IHC multiplex e Bengt Phung è ringraziato per le istruzioni su come implementare i dati IHC multiplex in QuPath per il disegno ROI.
Materiali
| Name | Company | Catalog Number | Comments |
| anti-CD14 | Cell Marque | 114R-16 | section 3, clone EPR3653 |
| anti-CD163 | Cell Marque | 163M-15 | section 3, clone MRQ-26 |
| anti-CD19 | Abcam | ab134114 | section 3, clone EPR5906 |
| anti-CD1c (BDCA1) | Thermo Scientific | TA505411 | section 3, clone OTI2F4 |
| anti-CD20 | Thermo Scientific | MS-340-S | section 3, clone L26 |
| anti-CD3 | Thermo Scientific | RM-9107 | section 3, clone sp7 |
| anti-CD303/BDCA2 | Dendritics via Enzo Lifesciences/Axxora | DDX0043 | section 3, clone 124B3.13 |
| anti-CD56 | Cell Marque | 156R-94 | section 3, clone MRQ-42 |
| anti-CD66b | BD Biosciences | 555723 | section 3, clone G10F5 |
| anti-CD68 | Dako Agilent | M087601 | section 3, clone PG-M1 |
| anti-CD8 | Dako Agilent | M7103 | section 3, clone C8/144B |
| anti-Foxp3 | Thermo Scientific | 14-4777 | section 3, clone 236A/E7 |
| anti-Gp100 | Dako Agilent | M063401 | section 3, clone HMB45 |
| anti-HLA-DR, DP, DQ | Santa Cruz | sc-53302 | section 3, clone CR3/43 |
| anti-MART-1 | Thermo Scientific | MS-799 | section 3, clone A103 |
| anti-pan cytokeratin | Abcam | ab86734 | section 3, clone AE1/AE3 + 5D3 |
| anti-SOX10 | Sigma Aldrich | 383R | section 3, clone EP268 |
| anti-Tyrosinase | Sanbio | MONX10591 | section 3, clone T311 |
| anti-XCR1 | Cell Signaling Technologies via Bioké | 44665S | section 3, clone D2F8T |
| antibody diluent | Akoya BioSciences | SKU ARD1001EA | section 3, from Opal 7-Color Automation IHC Kit 50 slide (can optionally also be replaced by TBST with 10% BSA) |
| Bond Aspirating Probe | Leica Biosciences | S21.0605 | section 3 |
| Bond Aspirating Probe Cleaning | Leica Biosciences | CS9100 | section 3 |
| Bond Dewax Solution | Leica Biosciences | AR9222 | section 3 |
| Bond Objectglas label + print lint | Leica Biosciences | S21.4564.A | section 3 |
| Bond Research Detection System 2 | Leica Biosciences | DS9777 | section 3 |
| Bond RX autostainer | Leica Biosciences | - | section 3, automated platform |
| Bond TM Epitope Retrieval 1 - 1 L | Leica Biosciences | AR9961 | section 3 |
| Bond TM Epitope Retrieval 2 - 1 L | Leica Biosciences | AR9640 | section 3 |
| Bond TM Wash Solution 10x - 1 L | Leica Biosciences | AR9590 | section 3 |
| BOND Universal Covertile | Leica Biosciences | S21.4611 | section 3 |
| Bond(TM) Titration Kit | Leica Biosciences | OPT9049 | section 3 |
| Coverslip 24 x 32 mm #1 (0.13-0.16 mm) | Fisher Scientific | 15717592 | section 2 |
| coverslip 24 x 50 mm | VWR | 631-0146 | section 2 |
| DAPI Fluoromount-G | VWR | 0100-20 | section 3, whenever monoplex slides need to be quickly checked, not for official analysis, then DAPI is stained seperately for better results |
| Eosine | section 2, home made | ||
| Ethanol 99.5% | VWR | 4099.9005 | section 2 |
| Fluoromount-G | VWR | 0100-01 | section 3 |
| haematoxyline | - | section 2, home made | |
| ImmuNet | - | immune cell detection and phenotyping pipeline | |
| inForm software 2.4.10 | Akoya BioSciences | - | section 4 & 6 |
| OPAL 480 reagent pack | Akoya BioSciences | FP1500001KT | section 3 |
| OPAL 520 reagent pack | Akoya BioSciences | FP1487001KT | section 3 |
| OPAL 570 reagent pack | Akoya BioSciences | FP1488001KT | section 3 |
| OPAL 620 reagent pack | Akoya BioSciences | FP1495001KT | section 3 |
| OPAL 690 reagent pack | Akoya BioSciences | FP1497001KT | section 3 |
| OPAL 780 reagent pack | Akoya BioSciences | FP1501001KT | section 3 |
| Opal 7-Color Automation IHC Kit 50 slide | Akoya BioSciences | NEL821001KT | section 3 |
| PhenoChart 1.1.0 | Akoya BioSciences | - | section 5 |
| PhenoImagerHT | Akoya BioSciences | CLS143455 | section 4, digital pathology imager with slide viewer and imaging software (formerly known as Vectra Polaris) |
| Quick-D mounting medium | Klinipath | 7280 | section 2 |
| QuPath 0.3.2 | whole slide image analysis software platform | ||
| R 4.1.1 | |||
| Slide boxes | VWR | 631-0737 | section 1 |
| SuperFrost Plus | Thermo Scientific through VWR | 631-9483 | section 1 |
| Vectra Polaris software 1.0.13 | Akoya BioSciences | - | section 4 |
| Xylene | VWR | 4055-9005 | section 2 |
Riferimenti
- Dunn, G. P., Bruce, A. T., Ikeda, H., Old, L. J., Schreiber, R. D. Cancer immunoediting: from immunosurveillance to tumor escape. Nature Immunology. 3 (11), 991-998 (2002).
- van der Woude, L. L., Gorris, M. A. J., Halilovic, A., Figdor, C. G., de Vries, I. J. M. Migrating into the tumor: a roadmap for T cells. Trends in Cancer. 3 (11), 797-808 (2017).
- Fridman, W. H., Pages, F., Sautes-Fridman, C., Galon, J. The immune contexture in human tumours: impact on clinical outcome. Nature Reviews. Cancer. 12 (4), 298-306 (2012).
- Fridman, W. H., et al. The immune microenvironment of human tumors: general significance and clinical impact. Cancer Microenvironment. 6 (2), 117-122 (2013).
- Pages, F., et al. International validation of the consensus Immunoscore for the classification of colon cancer: a prognostic and accuracy study. Lancet. 391 (10135), 2128-2139 (2018).
- Angell, H. K., Bruni, D., Barrett, J. C., Herbst, R., Galon, J. The Immunoscore: colon cancer and beyond. Clinical Cancer Research. 26 (2), 332-339 (2020).
- Angell, H., Galon, J. From the immune contexture to the Immunoscore: the role of prognostic and predictive immune markers in cancer. Current Opinion in Immunology. 25 (2), 261-267 (2013).
- Galon, J., et al. Cancer classification using the Immunoscore: a worldwide task force. Journal of Translational Medicine. 10, 205(2012).
- Galon, J., et al. World-wide Immunoscore Task Force: meeting report from the 34;Melanoma Bridge", Napoli, November 30th-December 3rd, 2016. Journal of Translational Medicine. 15 (1), 212(2017).
- Tumeh, P. C., et al. PD-1 blockade induces responses by inhibiting adaptive immune resistance. Nature. 515 (7528), 568-571 (2014).
- Creemers, J. H. A., et al. A tipping point in cancer-immune dynamics leads to divergent immunotherapy responses and hampers biomarker discovery. Journal for Immunotherapy of Cancer. 9 (5), e002032(2021).
- Blank, C. U., Haanen, J. B., Ribas, A., Schumacher, T. N. CANCER IMMUNOLOGY. The "cancer immunogram". Science. 352 (6286), 658-660 (2016).
- Teruya-Feldstein, J. The immunohistochemistry laboratory looking at molecules and preparing for tomorrow. Archives of Pathology & Laboratory Medicine. 134 (11), 1659-1665 (2010).
- Ramos-Vara, J. A., Miller, M. A., et al. When tissue antigens and antibodies get along: revisiting the technical aspects of immunohistochemistry--the red, brown, and blue technique. Veterinary Pathology. 51 (1), 42-87 (2014).
- Hegde, P. S., Karanikas, V., Evers, S. The where, the when, and the how of immune monitoring for cancer immunotherapies in the era of checkpoint inhibition. Clinical Cancer Research. 22 (8), 1865-1874 (2016).
- Parra, E. Novel platforms of multiplexed immunofluorescence for study of paraffin tumor tissues. Journal of Cancer Treatment and Diagnosis. 2 (1), 43-53 (2018).
- Gorris, M. A. J., et al. Eight-color multiplex immunohistochemistry for simultaneous detection of multiple immune checkpoint molecules within the tumor microenvironment. Journal of Immunology. 200 (1), 347-354 (2018).
- Roelofsen, T., et al. Spontaneous regression of ovarian carcinoma after septic peritonitis; a unique case report. Frontiers in Oncology. 8, 562(2018).
- van den Brand, D., et al. Peptide-mediated delivery of therapeutic mRNA in ovarian cancer. European Journal of Pharmaceutics and Biopharmaceutics. 141, 180-190 (2019).
- van den Brand, D., et al. EpCAM-binding DARPins for targeted photodynamic therapy of ovarian cancer. Cancers. 12 (7), 1762(2020).
- Di Blasio, S., et al. The tumour microenvironment shapes dendritic cell plasticity in a human organotypic melanoma culture. Nature Communications. 11 (1), 2749(2020).
- van Beek, J. J. P., et al. Human pDCs are superior to cDC2s in attracting cytolytic lymphocytes in melanoma patients receiving DC vaccination. Cell Reports. 30 (4), 1027-1038 (2020).
- Rodriguez-Rosales, Y. A., et al. Immunomodulatory aged neutrophils are augmented in blood and skin of psoriasis patients. Journal of Allergy and Clinical Immunology. 148 (4), 1030-1040 (2021).
- Hoeijmakers, Y. M., et al. Immune cell composition in the endometrium of patients with a complete molar pregnancy: Effects on outcome. Gynecologic Oncology. 160 (2), 450-456 (2021).
- van Ravensteijn, S. G., et al. Immunological and genomic analysis reveals clinically relevant distinctions between angiosarcoma subgroups. Cancers. 14 (23), 5938(2022).
- van der Woude, L. L., et al. Tumor microenvironment shows an immunological abscopal effect in patients with NSCLC treated with pembrolizumab-radiotherapy combination. Journal for Immunotherapy of Cancer. 10 (10), e005248(2022).
- Graham Martinez, C., et al. The immune microenvironment landscape shows treatment-specific differences in rectal cancer patients. Frontiers in Immunology. 13, 1011498(2022).
- Cortenbach, K. R. G., et al. Topography of immune cell infiltration in different stages of coronary atherosclerosis revealed by multiplex immunohistochemistry. International Journal of Cardiology. Heart & Vasculature. 44, 101111(2023).
- van Wilpe, S., et al. Homologous recombination repair deficient prostate cancer represents an immunologically distinct subtype. Oncoimmunology. 11 (1), 2094133(2022).
- Gorris, M. A. J., et al. Paired primary and metastatic lesions of patients with ipilimumab-treated melanoma: high variation in lymphocyte infiltration and HLA-ABC expression whereas tumor mutational load is similar and correlates with clinical outcome. Journal for Immunotherapy of Cancer. 10 (5), e004329(2022).
- van Wilpe, S., et al. Intratumoral T cell depletion following neoadjuvant chemotherapy in patients with muscle-invasive bladder cancer is associated with poor clinical outcome. Cancer Immunology, Immunotherapy. 72 (1), 137-149 (2023).
- Sultan, S., Gorris, M. A. J., Buytenhuijs, F., Lvan de Woude, L. A Segmentation-free machine learning architecture for immune land-scape phenotyping in solid tumors by multichannel imaging. bioRxiv. , (2021).
- Parra, E. R., et al. Immuno-profiling and cellular spatial analysis using five immune oncology multiplex immunofluorescence panels for paraffin tumor tissue. Scientific Reports. 11 (1), 8511(2021).
- Parra, E. R., et al. Validation of multiplex immunofluorescence panels using multispectral microscopy for immune-profiling of formalin-fixed and paraffin-embedded human tumor tissues. Scientific Reports. 7 (1), 13380(2017).
- Sun, Z., Nyberg, R., Wu, Y., Bernard, B., Redmond, W. L. Developing an enhanced 7-color multiplex IHC protocol to dissect immune infiltration in human cancers. PLoS One. 16 (2), e0247238(2021).
- LeicaBiosystems. BOND RX Fully Automated Research Stainer Protocols. , https://www.leicabiosystems.com/en-nl/ihc-ish/ihc-ish-instruments/bond-rx/ (2022).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon