Method Article
肿瘤微环境空间免疫细胞景观的多重免疫组织化学分析
* 这些作者具有相同的贡献
摘要
该方案详细描述了如何使用多重免疫组织化学对肿瘤微环境进行免疫细胞表征。
摘要
肿瘤微环境的免疫细胞景观可能包含用于发现预后和预测生物标志物的信息。多重免疫组织化学是一种有价值的工具,可以可视化和识别肿瘤组织中不同类型的免疫细胞,同时保留其空间信息。在这里,我们提供了详细的方案来分析组织切片中的淋巴细胞、骨髓和树突状细胞群。从切割福尔马林固定石蜡包埋切片开始,在自动化平台上进行自动多重染色程序,在多光谱成像显微镜上扫描载玻片,再到使用内部开发的机器学习算法 ImmuNet 进行图像分析。这些方案可以应用于各种肿瘤标本,只需切换肿瘤标志物即可分析样本不同区室(肿瘤与侵袭性切缘)中的免疫细胞,并应用最近邻分析。这种分析不仅限于肿瘤样本,还可以应用于其他(非)致病性组织。过去几年对设备和工作流程的改进显著缩短了吞吐时间,这有利于将来在诊断环境中应用该程序。
引言
免疫细胞在抵御病毒和细菌等病原体方面起着至关重要的作用,而且在抵御癌细胞方面也起着至关重要的作用1。因此,肿瘤微环境 (TME) 内的免疫系统在发现预后和预测生物标志物方面有很大的希望2。免疫细胞浸润与各种类型癌症的预后相关,尽管这尚未在临床护理中实施 3,4。在大多数肿瘤类型中,细胞毒性 T 细胞和辅助性 T 细胞数量多和/或调节性 T 细胞数量少与预后良好有关。正在努力将所谓的“免疫评分”纳入结直肠癌的 TNM 分期,将其转变为 TNM-I 分期 5,6。免疫评分来自两个不同肿瘤区域的 T 细胞(用 CD3 检测)和细胞毒性 T 细胞(用 CD8 检测)的总数:肿瘤核心与肿瘤的浸润边缘 (IM)。免疫评分也被认为对其他癌症类型具有预后价值,例如黑色素瘤、肺癌和乳腺癌 6,7,8,9。此外,免疫细胞浸润也可能与对检查点阻断免疫疗法的反应相关10。然而,这些预测性生物标志物必须在前瞻性研究中得到验证,然后才能在临床实践中常规实施。此外,还有人提出,单一生物标志物不足以进行有意义的预测11。因此,通过组合不同的生物标志物来创建患者样本的完整图谱已被提议作为所谓的“癌症免疫图”12 中更全面的预测生物标志物。
在研究 TME 中免疫细胞的方法中,最古老和最著名的技术是免疫组织化学 (IHC),通常用于多种疾病的诊断测试,尤其是癌症13。该技术在很长一段时间内仅限于使用一个或几个标记物14 ,因此在研究环境中被流式细胞术和基因表达谱 (GEP) 等其他技术所取代。然而,通常用于常规诊断和研究的福尔马林固定和石蜡包埋 (FFPE) 肿瘤组织并不(最佳地)适用于流式细胞术和 GEP。此外,尽管 GEP 和流式细胞术提供了对细胞表型和功能的大量见解,但缺乏空间信息是一个主要缺点。因此,样本内的异质性,例如肿瘤免疫细胞浸润区域与免疫细胞排除区域的差异,可能无法被检测到15。已经开发了用于 FFPE 组织多重分析的新型平台,例如多重 IHC、成像质谱流式细胞术和 indEXing 共产阶级检测 (CODEX),可用于同时检测组织切片内的多个标志物16。TME 中的免疫细胞正在被广泛研究,以寻找免疫疗法的最佳生物标志物。然而,多重技术和自动图像分析本身也存在障碍。
我们的实验室在使用蛋白石/酪胺信号放大 (TSA) 方法进行多重 IHC 染色方面拥有丰富的经验,并已在 IHC 平台上实现了自动化(参见材料表)17,18,19,20,21,22,23,24,25,26,27,28, 29,30,31.我们优化了免疫细胞检测组合,用于检测淋巴细胞、骨髓细胞和树突状细胞 (DC) 的不同亚群。包含致密免疫细胞区域的组织 - 对于淋巴细胞或复杂细胞形态 (即骨髓细胞和 DC) - 特别难以分析,存在高估或低估存在的免疫细胞数量的风险。为了克服这个问题,我们第32 小组开发了 ImmuNet 分析软件,这种机器学习管道极大地提高了这些不同类型免疫细胞的检测质量。这里描述了从获得 FFPE 材料到分析不同组织隔室中的免疫细胞密度和免疫细胞类型之间距离的详细方案。
该协议概述了自 2022 年实施数字病理成像仪以来,拉德堡德大学医学中心如何进行多重 IHC 面板。所描述的多重 IHC 面板可用于使用泛细胞角蛋白抗体作为肿瘤标志物的不同癌症(例如,肺癌、前列腺癌、结直肠癌、膀胱癌、乳腺癌)或使用黑色素细胞相关抗体作为肿瘤标志物的黑色素瘤。这些多重 IHC 方案在一抗浓度、荧光团组合和染色程序的顺序方面进行了仔细优化。我们和其他人之前已经描述了多重 IHC panel 优化 17,33,34,35。可以调整多重 IHC 检测组合,但需要对所描述的分析管道进行评估,并可能相应地进行调整或重新训练。所描述的七色多重 IHC 方案利用了蛋白荧光团 Opal480、Opal520、Opal570、Opal620、Opal690、Opal780 和 4',6-二脒基-2-苯基吲哚 (DAPI),因此可以通过“多光谱一键式免疫荧光”(MOTiF) 在成像仪上实现轻松解混和快速扫描。该协议中没有描述九色染色和扫描,因为这需要对实验设置进行更多微调,并在使用液晶可调谐滤光片的成像仪上进行另一种扫描模式。
研究方案
该方案显示的患者材料是先前进行的研究的一部分,并被当地 Radboudumc 医学伦理委员会与荷兰立法同时(文件编号 2017-3164)30 正式视为免于医学伦理批准。
1. FFPE 材料的收集、块的选择和样品的制备
- 通过治疗医生或病理学家从患者档案中检索 FFPE 块标识符。请查阅当地法规是否需要道德许可。
- 从当地病理档案馆或外部医院请求 FFPE 块。
注意:也有可能为特定研究获取肿瘤材料或活检。小型临床试验或动物研究可能就是这种情况。在这些情况下,组织样本的处理可能是研究人员的责任。 - 当有多个 FFPE 块可用时,通过评估苏木精和伊红 (HE) 染色的载玻片,选择包含活肿瘤组织的最具代表性的 FFPE 块,优先存在周围的基质组织(图 1)。
注意:建议获得此选择的专家意见(例如,病理学家)。HE 可能无法用于评估 FFPE 块的内容,需要创建新的 HE 进行选择。请转到第 2 部分以获取说明。 - 在切片机上切割 4 μm 厚的 FFPE 带。
注:厚度可在 1 μm 至 6 μm 之间,无明显的染色影响;但是,4 μm 是最标准的。 - 使用下述方法之一,将样品安装在载玻片上有利于自动染色机液流的位置(图 2A-C):
- 将切片放在水浴中 40 °C 蒸馏水的表面,以伸展并用载玻片将其拾取。
或
将载玻片放在 40 °C 加热板上,确保用一滴蒸馏水覆盖要安装在载玻片上的部分的位置。用镊子将切片放在此液滴的顶部,并使其伸展。用纸巾吸收蒸馏水,然后轻敲载玻片去除多余的水。
注意:将组织切片放置得太靠近载玻片的标签将导致次优染色(图 2D,E)。我们倾向于每个样品安装 6-10 个载玻片,以执行不同的多重 IHC 检测组合并进行备份。
- 将切片放在水浴中 40 °C 蒸馏水的表面,以伸展并用载玻片将其拾取。
- 让安装的载玻片在 56 °C 下干燥 1 小时或在 37 °C 下过夜。
- 使用安装的载玻片进行实验或将它们储存在 4 °C 的盒子中。
注意:根据我们迄今为止的经验,这些封固的载玻片可以在进行多重 IHC 染色之前保存数年。
2. 生成苏木精和伊红染色玻片
注意: 第 2 节的所有后续步骤均应在通风橱中进行。
- 在二甲苯中脱蜡载玻片(2 x 5 分钟)。
- 在乙醇中再水化(99.6% 1 x 5 分钟;95% 1 x 5 分钟;70% 1 x 2 分钟)。或者,将玻片浸入 99.6% 乙醇中 3 次。
- 用蒸馏水清洗载玻片(2 分钟)。
- 用苏木精对细胞核染色(10 分钟)。
- 用蒸馏的 H2O 洗涤载玻片(5 分钟)。
- 用伊红对载玻片染色(5 分钟)。
- 通过在 99.6% 乙醇中浸入 3 次来脱水载玻片。
- 将载玻片浸入二甲苯中 2 次。
- 加入几滴封固剂并用盖玻片密封。
- 让载玻片变硬,并在所有化学物质蒸发后将载玻片从通风橱中取出。
3. 在自动染色仪中进行单重和多重 IHC
- 根据要染色的样品数量计算需要多少试剂。
注:每次运行,自动染色仪可容纳 30 张载玻片,需要 ~18 小时才能完成 6 种抗体的多重 IHC 方案。当需要染色的载玻片较多时,可以在(工作)周的每个晚上放入多个批次;4 晚 30 张幻灯片 = 每周 120 张幻灯片。- 在本周开始时准备所有必要的试剂。Autostainer 系统每张玻片可分配 150 μL 试剂。使用 6 mL 滴定容器放置抗体和 Opal 试剂,使用 30 mL 容器放置封闭试剂和二抗辣根过氧化物酶。
注:6 mL 容器具有方便的插件,必要时可以轻松取出和更换。在进行试剂计算时,必须考虑 30 mL 容器或 6 mL 滴定容器的死体积分别为 1.6 mL 或 300 μL。 - 在提供的稀释剂中以 1:100 稀释所有 Opal 荧光团和地高辛 (DIG);在抗体稀释剂中以 1:25 稀释 Opal780。用抗体稀释剂稀释所有一抗,稀释度在 补充文件 1 中指定。
- 在本周开始时准备所有必要的试剂。Autostainer 系统每张玻片可分配 150 μL 试剂。使用 6 mL 滴定容器放置抗体和 Opal 试剂,使用 30 mL 容器放置封闭试剂和二抗辣根过氧化物酶。
- 为了遵循此方案,在开始实际的多重 IHC 实验之前,在包含扁桃体对照组织和其他(肿瘤)感兴趣组织类型的载玻片上运行单重 IHC(补充文件 2),以确保所有试剂都准备好了。
注意:单链 IHC 需要 ~3.5 小时,可以在当天结束前检查信号模式和强度。如果某些信号太弱(图 3),则可以调整试剂。 - 对于自发荧光校正,准备一张载玻片,其中包含包含自发荧光结构(例如血液和胶原蛋白)的(肿瘤)组织。与单重 IHC 玻片同时制备此玻片,但用封闭试剂代替抗体和 Opal 试剂(补充文件 3)。
注意:原则上,这样的载玻片可以重复用于多光谱成像,直到自发荧光校正不再最佳。然而,对于高度自发荧光的组织,例如大脑和肝脏,建议使用该组织进行自发荧光校正。 - 每次多重 IHC 运行时,使用一张对照组织玻片将 29 份样品加载到自动染色系统中,以检查每次多重 IHC 运行的性能。
- 从自动染色机网站的 “下载 ”选项卡下下载多重 IHC 方案,并调整它们以适应每个定制的多重 IHC panel36。对于多重 IHC,请参阅 补充文件 4 了解方案,对于定制的多重 IHC 面板,请参阅 补充文件 1。
- 完成染色方案后,将载玻片从自动染色仪中取出,放入装有洗涤缓冲液的容器中。
- 为防止 DAPI 污染自动染色系统,因为样品已经以非常低的浓度染色,请在用盖玻片覆盖载玻片之前手动应用 DAPI。每 mL 洗涤缓冲液中加入 2 滴 DAPI,并在室温下避光孵育 5 分钟。
注意:为了构建谱库,重要的是样品中没有任何 DAPI 染色。也可以每 mL 洗涤缓冲液滴 1 滴 DAPI,并在 RT 下孵育 10 分钟。 - 用洗涤缓冲液洗涤载玻片 3 次。
- 将玻片放在纸巾上,然后轻拍玻片上多余的洗涤缓冲液。
- 在组织上滴几滴封固剂。
- 将玻璃盖玻片轻轻地放在封固剂的顶部,以一定角度盖住载玻片,以避免气泡。
- 用镊子或干净的移液管尖端轻轻推动玻璃盖玻片,去除多余的封固剂和气泡。
- 在封固剂凝固之前,将载玻片保持原样 ~24 小时,水平放在显微镜载玻片上或将它们直接加载到显微镜中进行成像。
- 封固剂固化后或对载玻片成像后,将载玻片存放在 4 °C 的显微镜盒中。
4. 使用数字病理成像仪成像和扫描文件注释
- 按下设备右侧的电源按钮打开成像仪。至少 20 秒后,启动软件。
注意: 等待 20 秒,让硬件正确启动。 - 将载玻片每 4 张载玻片装入载玻片盒中。
- 可选:将幻灯片输入到可下载模板的 .csv 文件中(补充文件 5)。要将 .csv 文件加载到程序中,请将其保存在 C:\Users\Public\Akoya\VectraPolaris\States 中。
注意:最多可以同时加载 20 个包埋盒或 80 张玻片。
- 可选:将幻灯片输入到可下载模板的 .csv 文件中(补充文件 5)。要将 .csv 文件加载到程序中,请将其保存在 C:\Users\Public\Akoya\VectraPolaris\States 中。
- 引用设置
- 从主菜单打开 Check Dashboard 。
注意:带有参考玻片的包埋盒由制造商提供,可以选择永久保存在插槽 20 中。 - 根据制造商的说明,每周一次在提供的载玻片上设置明场参考(需要几分钟)。
- 根据制造商的说明,每月一次在提供的载玻片上设置荧光参考(需要 1 小时以上)。
- 从主菜单打开 Check Dashboard 。
- 制定或调整协议
- 返回主菜单并单击 Edit Protocol 创建协议。
- 单击新建...,然后在染色选项下选择荧光作为成像模式、多光谱玻片扫描和 Opal Polaris 5、6 和 7 颜色。
- 在 Protocol Name 下为方案命名,并通过从 Available Studies 中选择研究将其保存在研究下,或在 Create New Study |研究名称。
- 选择 Create Protocol (创建协议) 完成。
- 对于这种类型的扫描,请仅使用左侧窗口 Multispectral Slide Scan Settings;忽略右侧 Multispectral Field Settings 上的窗口。
- 以不同的放大倍数扫描载玻片。要遵循此协议,请将 像素分辨率 保持在 0.50 μm (20x) 以 20 倍放大倍率进行扫描。
- 通过选择 Scan Exposures (扫描曝光) 来设置曝光时间。
- 通过在 Load Carrier 选项下选择正确的插槽来加载保存载玻片的盒。
- 为了帮助浏览幻灯片,请选择 Take Overview(拍摄概述),以在加载载玻片后获取包含载玻片的载具的概览图像。要自动打开或关闭此功能,请单击右上角的齿轮图标,转到首选项...,然后在导航概览图像下勾选选项以打开或关闭,以启用加载交互式任务时自动图像载体。
- 通过选择 Set Scan Exposures 并查找具有阳性信号的不同点,在相应的单体 IHC 染色玻片上设置每个滤光片的曝光时间。手动对焦或使用 Auto Focus 并在切换到该信号的兼容滤波器后选择 Autoexposed 。选择最低曝光时间以防止过度曝光,并在设置所有曝光时间后拍摄每张幻灯片的快照以供参考(图 3)。
注意:忽略此类型扫描的 Set Field Exposures 选项。 - 通过检查几个信号为阳性的位置的所有滤光片,设置多重染色玻片上的曝光时间。将最低自动曝光时间减少 10% 以防止过度曝光,并在设置所有曝光时间后拍摄一些快照。
- 通过使用 Sample AF 过滤器 导航,拍摄未染色载玻片的快照以进行自发荧光补偿(图 3H)。
注意:具有红细胞和胶原结构的位置值得关注。对于强自发荧光区域,可能需要减少 Opal480 滤光片的曝光时间。如果 Opal480 信号足够强,由于实施了专有的 Sample AF 滤波器,它仍应与自发荧光结构很好地分离(参见第 6 节)。 - 使用软件评估染色和成像的质量(参见第 5 节和第 6 节; 图 4, 补充文件 6:补充图 S1 和补充图 S2)。
- 选择 Save...(保存... )按钮,确保将协议及其调整后的曝光时间保存到协议中。
注意:当协议已保存时,到目前为止,软件不会提供未保存调整的额外通知。
- 自动扫描载玻片
- 返回主菜单并单击 扫描幻灯片 扫描幻灯片。
- 在 “配置任务” 下手动输入幻灯片名称/ID 以及相应的任务和协议,或使用 “加载设置”从之前创建的.csv文件中自动输入。
- 单击 Scan 扫描 开始扫描。
- 等待弹出窗口以保存扫描设置。单击 Save 以使用默认设置并开始扫描。
注意:使用此方法扫描每张玻片需要 ~10-20 分钟。根据载玻片的数量,扫描可能需要一整天的时间。 - 通过查找任何错误消息,检查是否成功扫描了所有幻灯片的幻灯片。要了解扫描是否成功,请查找扫描的已保存的 Akoya 完整玻片扫描文件 (.qptiff) 和扫描中的完整组织。
5. 使用幻灯片查看器对数据进行注释
- 返回主菜单并单击 Launch Phenochart 打开幻灯片查看器。
- 如果扫描文件无法直接看到,请首先单击右上角的 齿轮图标 来指定其位置,转到 更改浏览器位置... ,然后随机选择感兴趣数据集的 .qptiff 文件之一。
注意:默认情况下,数据存储在 D:\Data\VectraPolaris 中。 - 通过选择幻灯片并单击右上角的 Load 或双击幻灯片来加载幻灯片。
- 通过单击右上角的 Login 按钮登录。
注意:用户名可以是首字母缩写或姓名,用于跟踪谁进行了哪些注释。 - 要执行解混,请单击 解混 顶部的按钮,然后选择 蛋白石 + AF 选项。
注意:这对于消除 Opal 480 通道附近的一些自发荧光信号很有用,但不是全部。 - 要生成用于批处理数据的算法,请使用 for inForm Projects 1 x 1 图像(图像大小:928 μm x 696 μm)选项的 Stamp 选择代表性图像。
注意:在整个数据集中选择了一些包含肿瘤、基质、背景和不同类型免疫细胞的代表性邮票,最终得到 ~20-30 张图像。 - 根据组织中需要分析的内容,使用 ROI 选项选择感兴趣的区域,然后选择 inForm Batch。手动删除不需要分析的图像,例如离肿瘤太远或位于背景中的图像。
注意:我们倾向于在整个肿瘤周围绘制 ROI,并选择远离肿瘤区域的一张额外图像,以便能够分析 ~0.5 mm 的 IM。
如果绘制的 ROI 相对较小,则 ROI 将由 2-9 张合并的 20 倍图像组成。由于我们不喜欢这样做,因此请手动标记感兴趣的组织(为 inForm Batch 选择)以规避这种情况。 - 完成批注后,让批注自动保存并加载下一张幻灯片。
- 在注释过程中,请检查幻灯片是否被正确扫描。
- 如果 .qptiff 文件丢失或未成功扫描玻片,请检查玻片上是否存在任何组织,用 70% 乙醇清洁玻片,然后再次扫描。
- 如果组织未完全扫描,从而错过可能重要的(肿瘤)区域,或者重要区域的扫描失焦,请用 70% 乙醇清洁载玻片并再次扫描。
注意:在这两种情况下,在盖玻片顶部用标记物圈住组织也有助于系统定位组织并再次尝试扫描(补充文件 6:补充图 S3)。在我们手中,细的红色记号笔比粗的黑色记号笔效果更好。
- 完成所有样品的扫描和注释后,通过将数据存储在不同的计算机或外部磁盘上来备份数据。
6. 频谱解混
- 打开 inForm 自动图像分析软件。
- 通过 File |打开图片;选择 .qptiff 文件。将步骤 5.6 中标记为 inForm Projects 的图章加载到项目中。
- 加载为自发荧光补偿成像的 .qptiff 文件。
- 为了补偿自发荧光,请使用 选择图像上的自发荧光 工具,在图像上从未染色的玻片上画一条线,穿过不同类型的自发荧光结构,例如红细胞和胶原蛋白。
- 在 Edit Markers and Colors... 部分中,分配与 Opal 荧光团相对应的标记名称,并将颜色调整为首选颜色。
- 要解混荧光基团,请选择左下角的 Prepare All 。
- 浏览图像并检查图像中是否可以看到所有信号,以及解混是否顺利。选择 眼球图标 以关闭和逐个打开所有标记以检查质量。
- (可选)训练组织分割、细胞分割和表型分析的算法。
- 转到 Export 选项卡,然后单击 Export Directory 下的 Browse... 按钮创建一个新的空导出目录。
- 在 Images to export:,选择 Composite Image 和 Component Images (multi-image TIFF)。
- 选择 “文件”|”收藏 |Project 将算法保存在某个位置。
- 转到左侧垂直的 Batch Analysis 选项卡,对幻灯片进行批处理。
- 选择 Create separate directories for each item 下 Export Options.
- 要添加幻灯片进行分析,请在 Add Slides... 按钮下选择 .qptiff 文件,然后将其加载到批量分析中。
- 选择 Run 以开始幻灯片的批处理。
7. ROI 图纸
- 创建一个仅包含第 6 节中的组件文件的文件夹,但保持分层文件夹结构不变(组件文件位于由 sample/slide 命名的文件夹中)。
- 打开 QuPath 整个幻灯片查看器软件。
- 单击左侧的 Create project 并选择/创建一个具有合适名称的新空文件夹。
- 单击 Automate 并选择 Show script editor。
- 复制粘贴 Supplemental File 7 中提供的脚本。在第 34 行,将位置更改为包含所有组件文件的幻灯片文件夹所在的位置(在步骤 7.1 中创建的文件夹。
- 选择 Run and return (在幻灯片的批量拼接完成时(第二天或更晚)以继续。
- 将生成的 .ome.tif 文件拖到 QuPath 项目中,并将其保存为项目。
- 当自动弹出新窗口时,选择 设置图像类型 |Fluorescence 并单击 Import。
- 在左侧的菜单中,观察样本列表;双击一个打开样品(图 5A)。
- 要调整通道的强度以使其更明显,请单击 对比度图标。
- 选择所有通道,然后单击 重置。
- 关闭自发荧光。
- 要开始绘制肿瘤的 ROI,请单击 对比度图标 并选择 Show grayscale (显示灰度)。选择肿瘤标志物通道并调整强度以使其最佳可见(图 5B)。
- 单击 画笔工具 以粗略绘制肿瘤 ROI。
- 在选择 魔杖工具时,在按 alt 键的同时单击 ROI 外部以从外部平滑 ROI(图 5C)。
- 合并具有相同 ROI 的分离肿瘤片段。
- 通过右键单击左侧列表中的注释,为 ROI 指定一个合适的名称,例如 tumor ; 选择 Set properties 并输入名称。
- 要为 IM 制作 ROI,请通过选择从肿瘤区域扩展现有 ROI: 对象 |注释... |展开注释。
- 选择扩展半径的大小,然后选择 Remove internal 和 Constrain to parent (图 5D)。
- 单击 对比度图标,选择自发荧光通道,并调整强度以使其最佳可见。
- 单击 魔杖 并调整 ROI,同时按下 alt 键从外部平滑 ROI 并删除任何不应属于此 ROI 的背景。
- 右键单击左侧列表中的注释,选择 Set properties,输入名称,并选择性地将其颜色更改为绿色,为 ROI 指定合适的名称,例如侵入式边距或 IM。
- 保存注释: File |导出对象 |导出所有对象 ,然后单击 确定 , 并在 导出为 FeatureCollection 上默认选择,然后将其保存在首选位置。
8. 免疫细胞检测
- 由于 ImmuNet 使用组件数据(多通道 TIFF 文件)进行训练和推理,因此将注释拆分为训练集和验证集。要训练模型,请按照存储库的自述文件中描述的步骤操作,将示例数据集和注释替换为所需的数据。除了不同的免疫细胞外,通过在不应被识别为感兴趣细胞的位点进行 背景 注释来为模型提供负面示例:肿瘤细胞、其他细胞或“无细胞”(可能与感兴趣细胞混淆的结构);有关详细信息,请参见 ImmuNet 出版物32.
- 使用验证注释,确保性能令人满意。查看每种注释类型的错误率 - 模型尚未检测到的验证注释的份额 - 最直接的评估指标。通过制作一些完全注释的 ROI 并计算 F 分数来评估误报方面的性能。
- 除了定量评估之外,还可以目视检查预测,以获得模型容易犯的误差的定性感觉(图 6、 补充文件 6:补充图 S4 和 补充图 S5)。如果判断模型性能不足,请按照存储库中的说明可视化某些切片的预测,并检查哪些站点最容易出错。在此类站点进行更多注释,并重新运行模型训练和评估。
- 达到目标性能后,按照存储库自述文件的 Inference for the entire dataset 部分中所述,为整个数据集运行推理。使用获取的 .csv 文件和模型预测作为数据分析的输入(为此编写 Python 或 R 脚本)。
9. 预测表型和数据分析
注意:在本节中,我们给出了一个用淋巴细胞面板染色的单个黑色素瘤样本的简单数据分析示例,该示例结合了 ImmuNet 识别的免疫细胞的位置(第 8 节)和用 QuPath 描绘的 ROI(第 7 节)。分析已在 R 4.1.1 中执行(脚本作为 补充文件 8 提供)。该脚本需要以下软件包:plyr 1.8.8、dplyr 1.0.8、tidyr 1.2.0、sf 1.0-7、ggplot2 3.4.0、RANN 2.6.1 和 RColorBrewer 1.1-2,可以使用 install.packages() 命令安装。作为输入,它需要一个 .csv 文件,其中包含 ImmuNet 对样本的预测,以及一个包含从 QuPath 导出的 ROI 的文件。步骤 9.1-9.6 描述了在提供的脚本中对单个样品进行的分析,第 9.7-9.9 节描述了分析多个样品的选项。
- 将 ImmuNet 的预测加载到 R 中后,通过绘制定义表型的标记物并确定预测标记表达的阈值,并选择最好区分种群的阈值来确定预测标记物表达的阈值。
注意:用于给定样品的门控策略如图 7B 所示。髓系和树突状细胞面板的门控策略显示在 补充文件 6:补充图 S6 和补充图 S7 中。 - 确定阈值后,使用它们为每个 ImmuNet 预测分配一个在面板中定义的表型。在某些预测中,观察到预测的标志物均未高于阈值,或者阈值后被认为表达的标志物组合可能不一致(例如,淋巴细胞面板中的 CD3 + CD20 + 预测)。如果在步骤 8.3 中实现了良好的模型性能,则此类预测的比例将很小;在分析之前过滤掉它们。
- 要单独分析肿瘤的 ROI 及其在 QuPath 中绘制的高达 100 μm 的侵袭边缘,请在 R 中加载相应的 GeoJSON 文件,并对于每个预测,确定预测落入的 ROI。
- 为了进行健全性检查并作为探索性数据分析的一部分,在相应的 ROI 中分别可视化样品中发现的免疫细胞以及 ROI 的边界(图 7A)。
- 现在,分别计算每个 ROI 的不同免疫细胞的密度。在给定样品中发现的密度如 表 1 所示。
- 如果有多个样品可用,则可视化细胞密度的分布。对密度值进行对数变换以获得正态分布的值。
注意:当某些表型的计数为 0 时,这些表型无法进行对数转换,从而导致值缺失。为了克服这个问题,可以通过先将所有细胞计数增加 0.5 来应用 LaPlacian 平滑,然后再除以表面积。 - 分析密度值并使用所选软件绘制它们(图 8)。
- 保留的像元位置可用于空间分析。例如,对于每个检测到的免疫细胞,找到一个最近的邻居,然后对于每个表型,计算不同表型作为最近邻居出现的情况百分比。
注:由于该样品中发现的自然杀伤 (NK) 细胞数量非常少,因此我们将其排除在本次分析之外。 图 9 给出了 Tumor 和 IM ROIs 的获得结果。
结果
根据病理报告和 HE 染色玻片选择含有肿瘤组织的 FFPE 块。当从患者身上切除多个肿瘤病灶和/或肿瘤样本很大时,这些病灶被分为多个 FFPE 块。我们更喜欢分析肿瘤隔室和肿瘤侵袭性边缘 (IM) 中的免疫细胞。IM 是与肿瘤相邻的非癌性基质组织。因此,当一个肿瘤样本有多个 FFPE 块可用时,将选择包含两种组织类型的 FFPE 块。从 HE 染色载玻片上看到的,一个 FFPE 块包含肿瘤组织和与肿瘤相邻的基质组织(图 1A)。来自同一肿瘤的另一个 FFPE 块包含的周围基质组织要少得多(图 1B)。然而,对于某些组织样品,FFPE 块中没有选择,或者任何 FFPE 块中都不存在 IM。(穿刺)活检通常就是这种情况,在数据解释过程中必须牢记这一点。
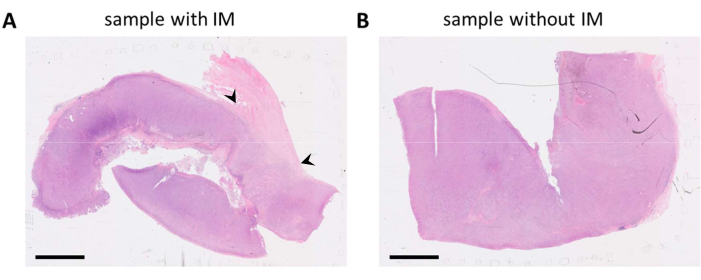
图 1:黑色素瘤肿瘤标本的 HE 染色载玻片。 (A) 肿瘤样本的示例,样本右上角的肿瘤 (IM) 附近有基质组织(用黑色箭头表示)。(B) 来自同一肿瘤病灶的另一个样本,样本中几乎没有基质组织。比例尺 = 5 毫米。缩写:HE = 苏木精和伊红;IM = 侵入性切缘。 请单击此处查看此图的较大版本。
使用建议的七色组合(补充文件 4)进行多重 IHC 染色可以在 3 天的染色过程中(考虑正常工作时间)手动进行,也可以在自动染色仪中过夜进行。使用自动染色仪时,必须将切片安装在载玻片上的特定位置,以实现系统的最佳流路(图 2A)。当切片正确安装在载玻片上(图 2B)时,它们将被均匀染色(图 2C)。如果切片没有最佳地安装在载玻片上(图 2D),则通常会导致次优染色模式(图 2E),因为自动染色仪的液流无法到达(完整)组织。当样品非常大时,或者当安装的玻片由不了解此问题的人提供时,可能会发生这种情况。在这些情况下,应仅选择玻片染色良好的部分进行分析。这些类型样品的另一种选择是手动染色,以最佳方式铺展液体。
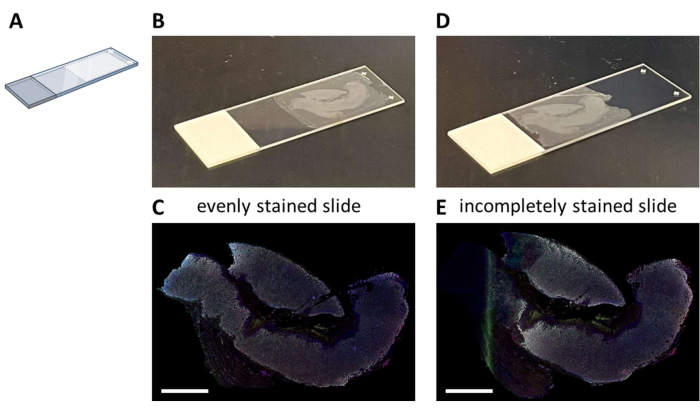
图 2:FFPE 切片安装到载玻片上和撞击。 (A) 在载玻片上安装位置以在自动染色仪上实现最佳染色的示意图。(B) 正确安装的滑轨示例。(C) 正确安装的载玻片可产生均匀染色的组织切片。(D) 次优安装的载玻片示例。(E) 次优的镶嵌玻片会导致染色组织切片不完整,如本图左侧所示。比例尺 = 5 毫米。缩写:FFPE = 福尔马林固定和石蜡包埋。 请单击此处查看此图的较大版本。
当在多个染色轮次中进行大型多重 IHC 实验并且需要制备大量溶液时,最好先在单重 IHC 运行中测试这些试剂,然后再进行多重 IHC。使用数字病理成像仪检查单重 IHC 以获得预期的染色模式,并使用对照载玻片上的相应过滤器设置曝光时间(图 3A-H)。扁桃体组织用作大多数免疫细胞标志物的阳性对照。由于扁桃体对照组织中的 DAPI 暴露时间总是高于其他组织(图 3G),因此必须在要研究的组织类型上设置 DAPI 暴露时间。这种扫描的常规曝光时间在 1 毫秒到 30 毫秒之间,具体取决于荧光团和滤光片(图 3I)。当单链 IHC 超过这些数字或染色模式不如预期清晰时,应调整或更换抗体溶液。在此处显示的示例中,我们决定增加 FOXP3 的浓度(图 3C 和图 3I),以使强度与其他标记物更一致。其他组织的自发荧光也可能比扁桃体对照组织更强。在我们的设置中,样本 AF 滤镜的曝光时间在 25 毫秒到 50 毫秒之间(图 3H,I)。

图 3:单重 IHC 和未染色对照样品的曝光时间设置。 (A) 扁桃体对照组织中的 CD20 - Opal 480 信号。(B) 扁桃体对照组织中的 CD3 - 蛋白石 520 信号。(C) FOXP3 - 扁桃体对照组织中的蛋白 570 信号。(D) 扁桃体对照组织中的 CD56 - 蛋白 620 信号 (E) 扁桃体对照组织中的 CD8 - 蛋白 690 信号。(F) 肿瘤标志物 - 扁桃体对照组织中的 Opal 780 信号。(G) 扁桃体对照组织中的 DAPI 信号通常比感兴趣的组织类型弱。(H) 自发荧光 - 肿瘤对照组织中的样本 AF 信号。(I) 用 10% 调整并检查多重 IHC 染色载玻片之前的曝光时间屏幕截图。比例尺 = 100 μm。缩写:AF = 自发荧光;IHC = 免疫组化;DAPI = 4'6-二脒基-2-苯基吲哚。 请单击此处查看此图的较大版本。
执行多重 IHC 后,通过检查一些多重 IHC 载玻片并选择自动曝光,从单重 IHC 设置调整曝光时间。对于这种类型的扫描,没有饱和保护选项,因此,避免将曝光设置得太高,从而避免过度曝光非常重要。过度曝光会阻碍荧光团的光谱解混。我们通常不会设置超过基于单重 IHC 的曝光时间的曝光时间,我们只减少多重 IHC 中更强的标记物的曝光时间(图 3G 和图 4A)。通过在几张幻灯片的不同位置自动曝光,可以观察到一些滤镜的曝光时间仍然过高。这些必须调整为使用自动曝光设置时观察到的最低数字,并再减去该值的 10% 以防止在其他看不见的位置过度曝光(图 4A)。使用这种方法,某些过滤器的曝光时间可以比在单重 IHC 上设置的曝光时间短。然而,通过成功的多重 IHC 实验,所有标记物都应该是可观察的,至少在对照载玻片上是这样(图 4B-H,补充文件 6:补充图 S1 和补充图 S2)。考虑到某些标志物可能并非每个样本中都存在。通过包括包含至少一个扁桃体切片的对照玻片,可以验证标准面板的所有标记物和信号强度的成功染色。
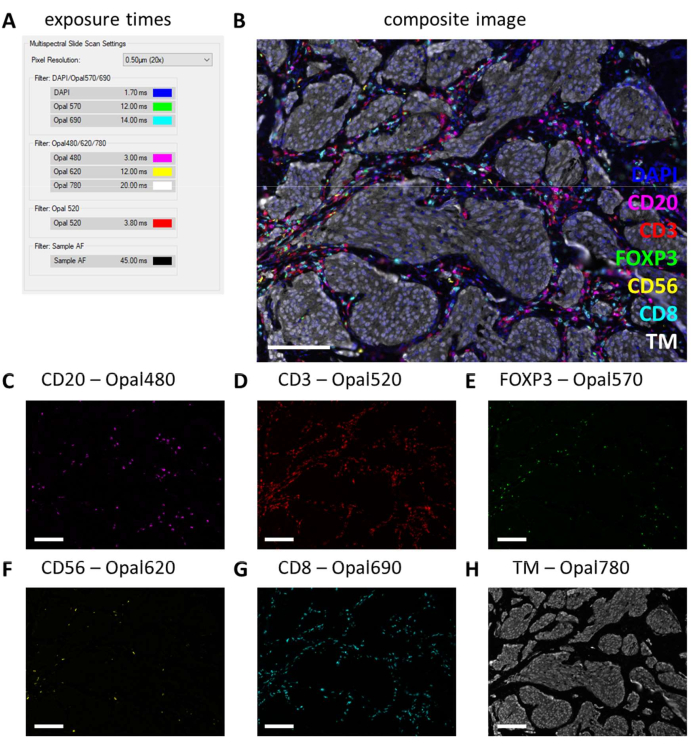
图 4:黑色素瘤肿瘤标本中淋巴细胞面板成功染色切片的示例。 (A) 用于记录此多重 IHC 样品的曝光时间。(B) 肿瘤组织内多重 IHC 淋巴细胞面板的合成图像。(C) CD20 - 洋红色的 Opal 480 信号。(D) CD3 - 红色的 Opal 520 信号。(E) FOXP3 - Opal 570 信号为绿色。(F) CD56 - 黄色的 Opal 620 信号。(G) CD8 - 青色的 Opal 690 信号。(H) TM - 白色蛋白石 780。比例尺 = 100 μm。缩写:TM = 肿瘤标志物;IHC = 免疫组织化学。 请单击此处查看此图的较大版本。
多重 IHC 玻片由数字成像仪完全扫描。在幻灯片查看器中选择用于后续分析的拼贴。然而,当需要分析更具体的区域时,例如肿瘤与 IM,可以使用 QuPath 绘制这些感兴趣区域 (ROI)。在幻灯片查看器中选择的图块的批处理完成后,组件文件将重新合并在一起(图 5A 和 补充文件 7)。使用 QuPath 中的肿瘤标志物通道(图 5B)和魔术棒工具,可以追踪肿瘤轮廓以形成“肿瘤 ROI”(图 5C)。接下来,肿瘤 ROI 可以扩大一定距离,在本例中为 500 μm,以创建“侵入性边缘 ROI”(图 5D)。通过观察自发荧光信号,使用魔术棒工具从该 ROI 中去除任何不需要的背景(非组织)(图 5E)。Tumor ROI 和 IM ROI 都保存为 GeoJSON 文件以供进一步处理(图 5F)。
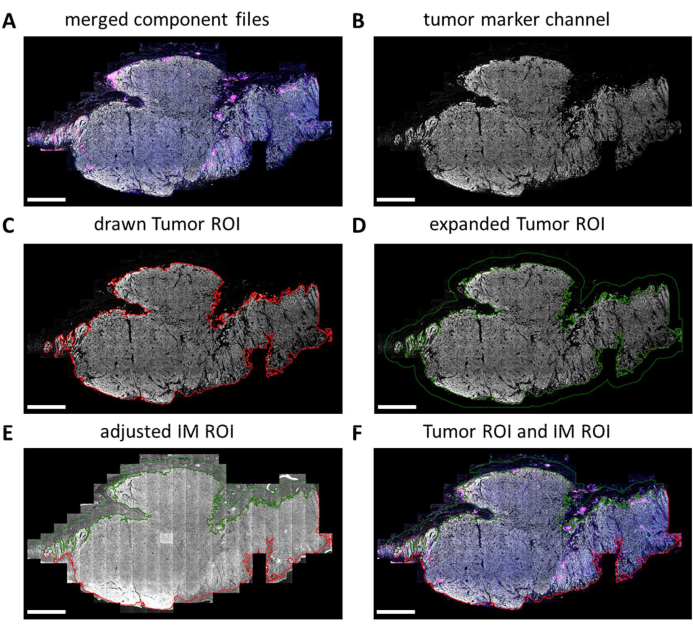
图 5:QuPath 中的肿瘤 ROI 和侵入性边缘 ROI 绘制过程。 (A) 合并的组件文件。(B) 仅显示肿瘤标志物通道的灰度图像。(C) 肿瘤 ROI 围绕肿瘤标志物信号绘制。(D) 通过将肿瘤 ROI 扩大 100-500 μm 以形成 IM ROI 来获得新的 ROI。(E) 通过排除背景(负信号)和其他大组织结构(如脂肪、血管和毛囊),将 IM ROI 调整为仅包括基质组织。(F) 生成的肿瘤 ROI 和 IM ROI 被保存并导出到 GeoJSON 文件中,以便进一步处理区域。肿瘤 ROI 以红色轮廓显示,IM ROI 以绿色轮廓显示。比例尺 = 2 毫米。缩写: ROI = 感兴趣区域;IM = 浸润性切缘;GeoJSON = 地理 JavaScript 对象表示法。 请单击此处查看此图的较大版本。
ImmuNet 网络可用于检测免疫细胞。对于淋巴细胞面板,可以将实验合成图像(图 6A)与软件检测到的免疫细胞(图 6B)进行视觉比较。对于髓样面板(补充文件 6:补充图 S4)和树突状细胞面板(补充文件 6:补充图 S5),可以进行类似的视觉比较。
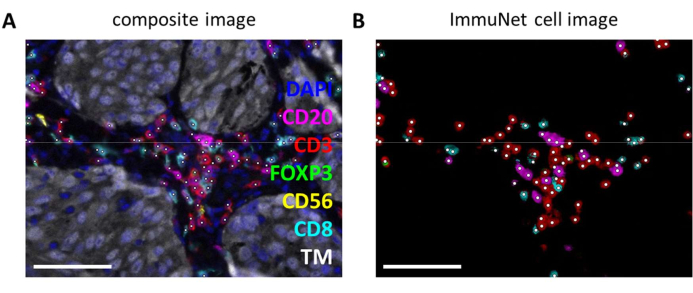
图 6:ImmuNet 识别的淋巴细胞。 (A) 图 4B 的合成图像,显示 ImmuNet 识别的细胞和白点。(B) ImmuNet 识别的细胞和随后检测到的标志物表达。比例尺 = 50 μm。缩写:TM = 肿瘤标志物。 请单击此处查看此图的较大版本。
ImmuNet 检测到并以 .csv 格式保存的免疫细胞可以导入任何编程语言以供进一步分析。我们在 R (补充文件 8) 中执行了空间可视化和门控。然后可以在空间上可视化检测到的细胞(图 7A, 补充文件 6:补充图 S6 和 补充图 S7)。可以对假标志物表达进行门控以对单个免疫细胞进行表型分析(图 7B)。
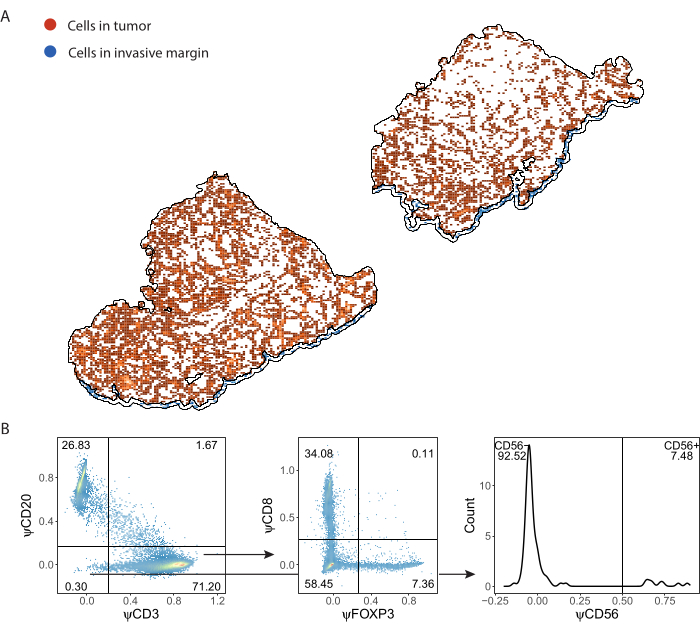
图 7:淋巴细胞面板的门控策略。(A) 使用 QuPath 描绘的感兴趣肿瘤和侵袭边缘区域检测到的免疫细胞。(B) ImmuNet 从 A 部分检测到的所有细胞的门控。淋巴细胞首先在 CD20+ B 细胞和 CD3+ T 细胞上设门。CD3 + T 细胞进一步设门口以获取 CD8 和 FOXP3 表达。CD20-CD3- 群体是 CD56+ 自然杀伤细胞的门控。请单击此处查看此图的较大版本。
当通过门控确定预测细胞的表型时,可以在不同的 ROI 内计算不同表型的细胞密度。这是通过将每个表型的细胞总数除以 ROI 的表面积来计算的(表 1、图 8 和补充文件 8)。在这里,B 细胞定义为 CD3-CD20+,辅助性 T 细胞定义为 CD3+CD20-CD8-FoxP3-,调节性 T 细胞定义为 CD3+CD20-CD8-FoxP3+,细胞毒性 T 细胞定义为 CD3+CD20-CD8+FoxP3-,NK 细胞定义为 CD3-CD20-CD56+。
| 表现型 | 肿瘤密度 (cells/mm2) | IM 密度(单元/mm2) |
| B 细胞 | 185.74 | 145.62 |
| 辅助性 T 细胞 | 301.46 | 157.51 |
| 调节性 T 细胞 | 38.53 | 19.53 |
| 细胞毒性 T 细胞 | 185.35 | 83.21 |
| NK 细胞 | 0.18 | 0 |
表 1:ROI 中表型的密度。 在用淋巴细胞面板染色的单个黑色素瘤样本中发现的不同表型细胞密度。密度在 Tumor 和 IM ROI 中分别计算。缩写: IM = 侵入式切缘;ROI = 感兴趣区域。
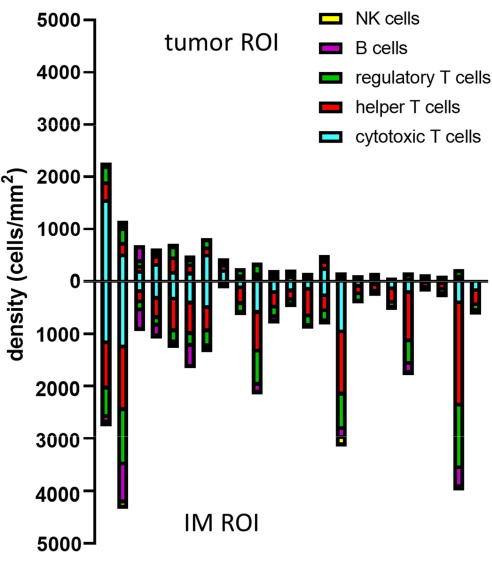
图 8:多个样品的数据分析示例。 23 例原发性黑色素瘤肿瘤和 IM 中不同淋巴细胞表型的密度分析。缩写: IM = 侵入式切缘;ROI = 感兴趣区域。 请单击此处查看此图的较大版本。
为了更深入地了解这些免疫细胞的空间信息,还可以确定样本中已鉴定表型之间的距离或最近邻表型的百分比(图 9)。
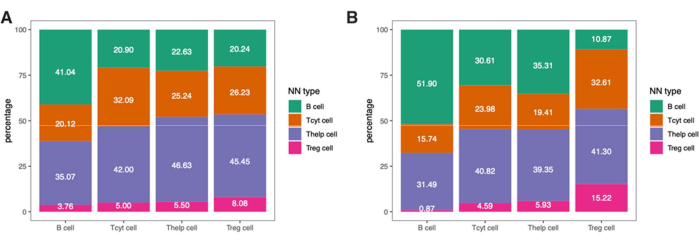
图 9:单个样品的最近邻分析示例。 在用淋巴细胞面板染色的单个黑色素瘤样本中发现的 (A) 肿瘤和 (B) IM ROI 中不同细胞类型的最近邻表型百分比。缩写: IM = 侵入式切缘;ROI = 感兴趣区域。 请单击此处查看此图的较大版本。
补充文件 1:多重 IHC 总结协议规范。请点击此处下载此文件。
补充文件 2:单链体的 Autostainer 协议。请点击此处下载此文件。
补充文件 3:用于自发荧光补偿的 Autostainer 方案。请点击此处下载此文件。
补充文件 4:用于多重免疫组织化学的 Autostainer 方案。请点击此处下载此文件。
补充文件 5:模板 .csv 文件。请点击此处下载此文件。
补充文件 6:黑色素瘤组织样本中的髓系和树突状细胞面板;扫描失败时标记载玻片;ImmuNet 识别的髓系和树突状细胞;髓系和树突状细胞面板的门控策略。请点击此处下载此文件。
补充文件 7:QuPath 拼接脚本。请点击此处下载此文件。
补充文件 8:数据分析脚本。请点击此处下载此文件。
讨论
TME 的空间分析是一种广受欢迎的技术,可以更多地了解免疫细胞区室并发现新的预后和预测生物标志物,尤其是在免疫肿瘤学领域16。为此,人们正在开发许多不同的技术,包括检测蛋白质、mRNA 转录本或两者的组合,估计多达 100-1,000 个靶标。然而,更高的多重检测是以较低的高通量实验、较高的实验成本和技术挑战为代价的,而且通常只能分析一小部分 TME。使用我们在此处描述的基于 TSA 的方法进行多重 IHC,同时检测六种不同的标记物 + DAPI,执行成本相对较低,并且整个组织切片在 20 分钟内成像,准备好进行全面分析。随着染色程序的自动化,这项技术变得不那么复杂。多光谱显微镜的改进,包括增加两个额外的滤光片,极大地改善了光谱分离和扫描时间。可以同时检测多达 8 个不同的标记物 + DAPI。然而,通过使用更多标记物扩展多路复用,随着光谱分离变得更具挑战性并且整个玻片的扫描时间大幅增加,上述好处消失了。正在努力在不同机构之间实现多重 IHC 的标准化,以便于更轻松地在诊断环境中实施。对于多重 IHC 的标准化,我们建议用户遵循使用六种不同标记物 + DAPI 的更易于访问的方案。尽管如此,仍然需要相当多的技术知识,并且下游分析可能具有挑战性,为此我们开发了本协议中描述的方法。
标准化从多重 IHC 检测组合开发开始。选择检测特定蛋白质靶标的一抗的重要性已在17 之前得到强调。我们的多重 IHC 检测组合主要使用一抗克隆开发,这些抗体克隆也在我们的诊断部门用于 IHC 并进行了验证。然而,在树突状细胞多重 IHC panel 的情况下,大多数抗体未用于诊断环境(van der Hoorn 等人,提交的手稿)。为了确保特异性并最大限度地减少批次差异,我们选择使用单克隆抗体而不是多克隆抗体,并且还使用转染细胞系和原代细胞验证了大多数抗体。多年来,使用 Vectra 3 系统的许多研究中使用了不同版本的多重 IHC 检测板18、21、23、24、25、26、27、28、29、30、31、32.为了在 PhenoImager HT 系统上以最佳方式实施这些多重 IHC 检测组合,必须对一抗和荧光团组合进行一些调整。为了获得更好的光谱解混和更快的整个组织切片扫描时间,有必要使用最新的 Opal480 和 Opal780 荧光基团,并避免在七色多重 IHC 检测组合中使用 Opal540 和 Opal650 荧光基团。扫描时间快 ~3-10 倍,具体取决于组织切片的大小。多重 IHC 检测组合调整很容易实现,但需要牢记一些注意事项。Opal480 的荧光光谱与自发荧光光谱重叠很多,因此会干扰红细胞和其他自发荧光结构的光谱解混。在大多数情况下,将更高浓度的一抗与 Opal480 配对可以解决这个问题。在 PhenoImager HT 上实施专有的样品 AF 滤光片有助于 Opal480 和自发荧光的分离。但是,最好使用与 Opal480 一起使用时产生清晰信号的一抗,使其信号高于自发荧光。
即使这些多重 IHC 检测组合已经建立,也需要考虑批次间差异。通过在开始完整的多重 IHC 实验之前进行单重 IHC 对照,我们有时会观察到一抗在不同实验中的表现更强或更弱。造成这种情况的原因可能是移液错误、次优的试剂储存条件和保质期。我们根据我们的经验调整一抗解决方案解决了这个问题。即使不需要进行上述任何调整,对于每个多重 IHC 批量实验,根据单重 IHC 染色对照玻片设置曝光时间也很重要。
因为我们的研究最初集中在不同类型的癌症和黑色素瘤上,所以需要多重 IHC 面板可以在肿瘤类型之间互换,并且调整最小。因此,我们始终在优化过程中包括多种(肿瘤)组织类型,并观察到免疫细胞标志物的一抗稀释度在不同肿瘤类型之间可以保持相似。然而,癌和黑色素瘤之间的肿瘤组织检测需要不同的肿瘤标志物。因此,肿瘤标志物始终经过优化,在每个多重 IHC 面板结束时起作用,并且目前始终与 Opal780 结合使用,巧合的是,Opal780 也必须位于多重 IHC 染色程序的最后一个荧光团。因此,通过在多重 IHC 结束时使用肿瘤标志物,这些多重 IHC 检测组合可以很容易地更换为其他肿瘤类型,例如胶质母细胞瘤(即 GFAP)和霍奇金淋巴瘤(即 CD30)。对于血管肉瘤,我们使用这种淋巴细胞多重 IHC 面板,以成红细胞转化特异性相关基因 (ERG) 作为肿瘤标志物,仅进行了两次优化实验25。优化包括 ERG 一抗的滴定和最后使用 ERG 测试多重 IHC 检测板。
也可以通过将某种免疫细胞标志物交换为另一种免疫或功能标志物来对这些多重 IHC 检测组合进行其他调整。每一次更改都需要优化。可以按照前面17 的描述来遵循优化协议。对提议的多重 IHC panel 的某些更改会干扰我们创建的 ImmuNet 算法。必须生成足够的数据,并且必须花费时间将这些更改实施到算法中(每个新标记和/或细胞表型至少 750 个注释,以及 150 个注释用于验证先前训练的标记)。此处介绍的面板不包含功能标志物,尽管在我们的实验室中将免疫检查点标志物(如 PD-1 和 PD-L1)实施到多重 IHC 面板中。然而,事实证明,分析阴性和阳性信号中二元化较少的标志物更加困难,并且是我们小组积极研究的一个领域。
与其他新技术相比,可以使用多重 IHC 同时评估的标志物数量有限。虽然可以通过分析 FFPE 块的连续切片上的不同面板来规避这种情况,但很难在空间上比较这些切片。载玻片制备后,方向和折叠伪影可能不同。尽管如此,多重 IHC 非常容易获得,这使其成为更多机构和研究人员的有吸引力的工具,因此更适合未来在诊断环境中实施。随着针对多种肿瘤类型的多重 IHC 免疫细胞检测组合和下游分析管道的标准化,可以更多地了解患者和肿瘤类型之间 TME 的差异。例如,这可以导致对 TME 在特定治疗抗肿瘤反应中的作用有更多的了解。这甚至可能产生新的生物标志物来预测对治疗的反应和预期生存期等因素。总体而言,这可以使多重 IHC 成为一种临床工具,以个性化医学方法帮助临床决策。诚然,分析程序的更多步骤可能应该是自动化和标准化的,以便它可以在日常诊断环境中使用,因此到目前为止,它主要是一个未来主义的观点。
尽管存在技术挑战,但在单个样品玻片上分析多个标记物可能是一个非常强大的工具。正如我们在这里使用 ImmuNet 所描述的那样,凭借标准化的实验方案和稳健的分析方法,多个标记物的定量使其比经典 IHC 更具信息量,而与新的更高重实验方法相比,多重 IHC 仍然保持相对较高的通量。
披露声明
作者没有需要披露的利益冲突。
致谢
PhenoImager HT 是通过 Radboud 大学医学中心和 Radboud 显微镜技术中心提供的资金购买的。CF 由荷兰癌症协会赠款 (10673) 和 ERC Adv 赠款 ARTimmune (834618) 提供财政支持。JT 由 NWO Vidi 赠款 (VI.Vidi.192.084) 提供财政支持。作者要感谢 Eric van Dinther 和 Ankur Ankan 在创建工作流程以存储多重 IHC 数据方面提供的帮助,并感谢 Bengt Phung 有关如何在 QuPath 中实施多重 IHC 数据以进行 ROI 绘图的说明。
材料
| Name | Company | Catalog Number | Comments |
| anti-CD14 | Cell Marque | 114R-16 | section 3, clone EPR3653 |
| anti-CD163 | Cell Marque | 163M-15 | section 3, clone MRQ-26 |
| anti-CD19 | Abcam | ab134114 | section 3, clone EPR5906 |
| anti-CD1c (BDCA1) | Thermo Scientific | TA505411 | section 3, clone OTI2F4 |
| anti-CD20 | Thermo Scientific | MS-340-S | section 3, clone L26 |
| anti-CD3 | Thermo Scientific | RM-9107 | section 3, clone sp7 |
| anti-CD303/BDCA2 | Dendritics via Enzo Lifesciences/Axxora | DDX0043 | section 3, clone 124B3.13 |
| anti-CD56 | Cell Marque | 156R-94 | section 3, clone MRQ-42 |
| anti-CD66b | BD Biosciences | 555723 | section 3, clone G10F5 |
| anti-CD68 | Dako Agilent | M087601 | section 3, clone PG-M1 |
| anti-CD8 | Dako Agilent | M7103 | section 3, clone C8/144B |
| anti-Foxp3 | Thermo Scientific | 14-4777 | section 3, clone 236A/E7 |
| anti-Gp100 | Dako Agilent | M063401 | section 3, clone HMB45 |
| anti-HLA-DR, DP, DQ | Santa Cruz | sc-53302 | section 3, clone CR3/43 |
| anti-MART-1 | Thermo Scientific | MS-799 | section 3, clone A103 |
| anti-pan cytokeratin | Abcam | ab86734 | section 3, clone AE1/AE3 + 5D3 |
| anti-SOX10 | Sigma Aldrich | 383R | section 3, clone EP268 |
| anti-Tyrosinase | Sanbio | MONX10591 | section 3, clone T311 |
| anti-XCR1 | Cell Signaling Technologies via Bioké | 44665S | section 3, clone D2F8T |
| antibody diluent | Akoya BioSciences | SKU ARD1001EA | section 3, from Opal 7-Color Automation IHC Kit 50 slide (can optionally also be replaced by TBST with 10% BSA) |
| Bond Aspirating Probe | Leica Biosciences | S21.0605 | section 3 |
| Bond Aspirating Probe Cleaning | Leica Biosciences | CS9100 | section 3 |
| Bond Dewax Solution | Leica Biosciences | AR9222 | section 3 |
| Bond Objectglas label + print lint | Leica Biosciences | S21.4564.A | section 3 |
| Bond Research Detection System 2 | Leica Biosciences | DS9777 | section 3 |
| Bond RX autostainer | Leica Biosciences | - | section 3, automated platform |
| Bond TM Epitope Retrieval 1 - 1 L | Leica Biosciences | AR9961 | section 3 |
| Bond TM Epitope Retrieval 2 - 1 L | Leica Biosciences | AR9640 | section 3 |
| Bond TM Wash Solution 10x - 1 L | Leica Biosciences | AR9590 | section 3 |
| BOND Universal Covertile | Leica Biosciences | S21.4611 | section 3 |
| Bond(TM) Titration Kit | Leica Biosciences | OPT9049 | section 3 |
| Coverslip 24 x 32 mm #1 (0.13-0.16 mm) | Fisher Scientific | 15717592 | section 2 |
| coverslip 24 x 50 mm | VWR | 631-0146 | section 2 |
| DAPI Fluoromount-G | VWR | 0100-20 | section 3, whenever monoplex slides need to be quickly checked, not for official analysis, then DAPI is stained seperately for better results |
| Eosine | section 2, home made | ||
| Ethanol 99.5% | VWR | 4099.9005 | section 2 |
| Fluoromount-G | VWR | 0100-01 | section 3 |
| haematoxyline | - | section 2, home made | |
| ImmuNet | - | immune cell detection and phenotyping pipeline | |
| inForm software 2.4.10 | Akoya BioSciences | - | section 4 & 6 |
| OPAL 480 reagent pack | Akoya BioSciences | FP1500001KT | section 3 |
| OPAL 520 reagent pack | Akoya BioSciences | FP1487001KT | section 3 |
| OPAL 570 reagent pack | Akoya BioSciences | FP1488001KT | section 3 |
| OPAL 620 reagent pack | Akoya BioSciences | FP1495001KT | section 3 |
| OPAL 690 reagent pack | Akoya BioSciences | FP1497001KT | section 3 |
| OPAL 780 reagent pack | Akoya BioSciences | FP1501001KT | section 3 |
| Opal 7-Color Automation IHC Kit 50 slide | Akoya BioSciences | NEL821001KT | section 3 |
| PhenoChart 1.1.0 | Akoya BioSciences | - | section 5 |
| PhenoImagerHT | Akoya BioSciences | CLS143455 | section 4, digital pathology imager with slide viewer and imaging software (formerly known as Vectra Polaris) |
| Quick-D mounting medium | Klinipath | 7280 | section 2 |
| QuPath 0.3.2 | whole slide image analysis software platform | ||
| R 4.1.1 | |||
| Slide boxes | VWR | 631-0737 | section 1 |
| SuperFrost Plus | Thermo Scientific through VWR | 631-9483 | section 1 |
| Vectra Polaris software 1.0.13 | Akoya BioSciences | - | section 4 |
| Xylene | VWR | 4055-9005 | section 2 |
参考文献
- Dunn, G. P., Bruce, A. T., Ikeda, H., Old, L. J., Schreiber, R. D. Cancer immunoediting: from immunosurveillance to tumor escape. Nature Immunology. 3 (11), 991-998 (2002).
- van der Woude, L. L., Gorris, M. A. J., Halilovic, A., Figdor, C. G., de Vries, I. J. M. Migrating into the tumor: a roadmap for T cells. Trends in Cancer. 3 (11), 797-808 (2017).
- Fridman, W. H., Pages, F., Sautes-Fridman, C., Galon, J. The immune contexture in human tumours: impact on clinical outcome. Nature Reviews. Cancer. 12 (4), 298-306 (2012).
- Fridman, W. H., et al. The immune microenvironment of human tumors: general significance and clinical impact. Cancer Microenvironment. 6 (2), 117-122 (2013).
- Pages, F., et al. International validation of the consensus Immunoscore for the classification of colon cancer: a prognostic and accuracy study. Lancet. 391 (10135), 2128-2139 (2018).
- Angell, H. K., Bruni, D., Barrett, J. C., Herbst, R., Galon, J. The Immunoscore: colon cancer and beyond. Clinical Cancer Research. 26 (2), 332-339 (2020).
- Angell, H., Galon, J. From the immune contexture to the Immunoscore: the role of prognostic and predictive immune markers in cancer. Current Opinion in Immunology. 25 (2), 261-267 (2013).
- Galon, J., et al. Cancer classification using the Immunoscore: a worldwide task force. Journal of Translational Medicine. 10, 205(2012).
- Galon, J., et al. World-wide Immunoscore Task Force: meeting report from the 34;Melanoma Bridge", Napoli, November 30th-December 3rd, 2016. Journal of Translational Medicine. 15 (1), 212(2017).
- Tumeh, P. C., et al. PD-1 blockade induces responses by inhibiting adaptive immune resistance. Nature. 515 (7528), 568-571 (2014).
- Creemers, J. H. A., et al. A tipping point in cancer-immune dynamics leads to divergent immunotherapy responses and hampers biomarker discovery. Journal for Immunotherapy of Cancer. 9 (5), e002032(2021).
- Blank, C. U., Haanen, J. B., Ribas, A., Schumacher, T. N. CANCER IMMUNOLOGY. The "cancer immunogram". Science. 352 (6286), 658-660 (2016).
- Teruya-Feldstein, J. The immunohistochemistry laboratory looking at molecules and preparing for tomorrow. Archives of Pathology & Laboratory Medicine. 134 (11), 1659-1665 (2010).
- Ramos-Vara, J. A., Miller, M. A., et al. When tissue antigens and antibodies get along: revisiting the technical aspects of immunohistochemistry--the red, brown, and blue technique. Veterinary Pathology. 51 (1), 42-87 (2014).
- Hegde, P. S., Karanikas, V., Evers, S. The where, the when, and the how of immune monitoring for cancer immunotherapies in the era of checkpoint inhibition. Clinical Cancer Research. 22 (8), 1865-1874 (2016).
- Parra, E. Novel platforms of multiplexed immunofluorescence for study of paraffin tumor tissues. Journal of Cancer Treatment and Diagnosis. 2 (1), 43-53 (2018).
- Gorris, M. A. J., et al. Eight-color multiplex immunohistochemistry for simultaneous detection of multiple immune checkpoint molecules within the tumor microenvironment. Journal of Immunology. 200 (1), 347-354 (2018).
- Roelofsen, T., et al. Spontaneous regression of ovarian carcinoma after septic peritonitis; a unique case report. Frontiers in Oncology. 8, 562(2018).
- van den Brand, D., et al. Peptide-mediated delivery of therapeutic mRNA in ovarian cancer. European Journal of Pharmaceutics and Biopharmaceutics. 141, 180-190 (2019).
- van den Brand, D., et al. EpCAM-binding DARPins for targeted photodynamic therapy of ovarian cancer. Cancers. 12 (7), 1762(2020).
- Di Blasio, S., et al. The tumour microenvironment shapes dendritic cell plasticity in a human organotypic melanoma culture. Nature Communications. 11 (1), 2749(2020).
- van Beek, J. J. P., et al. Human pDCs are superior to cDC2s in attracting cytolytic lymphocytes in melanoma patients receiving DC vaccination. Cell Reports. 30 (4), 1027-1038 (2020).
- Rodriguez-Rosales, Y. A., et al. Immunomodulatory aged neutrophils are augmented in blood and skin of psoriasis patients. Journal of Allergy and Clinical Immunology. 148 (4), 1030-1040 (2021).
- Hoeijmakers, Y. M., et al. Immune cell composition in the endometrium of patients with a complete molar pregnancy: Effects on outcome. Gynecologic Oncology. 160 (2), 450-456 (2021).
- van Ravensteijn, S. G., et al. Immunological and genomic analysis reveals clinically relevant distinctions between angiosarcoma subgroups. Cancers. 14 (23), 5938(2022).
- van der Woude, L. L., et al. Tumor microenvironment shows an immunological abscopal effect in patients with NSCLC treated with pembrolizumab-radiotherapy combination. Journal for Immunotherapy of Cancer. 10 (10), e005248(2022).
- Graham Martinez, C., et al. The immune microenvironment landscape shows treatment-specific differences in rectal cancer patients. Frontiers in Immunology. 13, 1011498(2022).
- Cortenbach, K. R. G., et al. Topography of immune cell infiltration in different stages of coronary atherosclerosis revealed by multiplex immunohistochemistry. International Journal of Cardiology. Heart & Vasculature. 44, 101111(2023).
- van Wilpe, S., et al. Homologous recombination repair deficient prostate cancer represents an immunologically distinct subtype. Oncoimmunology. 11 (1), 2094133(2022).
- Gorris, M. A. J., et al. Paired primary and metastatic lesions of patients with ipilimumab-treated melanoma: high variation in lymphocyte infiltration and HLA-ABC expression whereas tumor mutational load is similar and correlates with clinical outcome. Journal for Immunotherapy of Cancer. 10 (5), e004329(2022).
- van Wilpe, S., et al. Intratumoral T cell depletion following neoadjuvant chemotherapy in patients with muscle-invasive bladder cancer is associated with poor clinical outcome. Cancer Immunology, Immunotherapy. 72 (1), 137-149 (2023).
- Sultan, S., Gorris, M. A. J., Buytenhuijs, F., Lvan de Woude, L. A Segmentation-free machine learning architecture for immune land-scape phenotyping in solid tumors by multichannel imaging. bioRxiv. , (2021).
- Parra, E. R., et al. Immuno-profiling and cellular spatial analysis using five immune oncology multiplex immunofluorescence panels for paraffin tumor tissue. Scientific Reports. 11 (1), 8511(2021).
- Parra, E. R., et al. Validation of multiplex immunofluorescence panels using multispectral microscopy for immune-profiling of formalin-fixed and paraffin-embedded human tumor tissues. Scientific Reports. 7 (1), 13380(2017).
- Sun, Z., Nyberg, R., Wu, Y., Bernard, B., Redmond, W. L. Developing an enhanced 7-color multiplex IHC protocol to dissect immune infiltration in human cancers. PLoS One. 16 (2), e0247238(2021).
- LeicaBiosystems. BOND RX Fully Automated Research Stainer Protocols. , https://www.leicabiosystems.com/en-nl/ihc-ish/ihc-ish-instruments/bond-rx/ (2022).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。