Method Article
Análisis inmunohistoquímico multiplex del paisaje espacial de células inmunitarias del microambiente tumoral
* Estos autores han contribuido por igual
En este artículo
Resumen
Este protocolo describe en detalle cómo se lleva a cabo la caracterización de las células inmunitarias del microambiente tumoral mediante inmunohistoquímica multiplex.
Resumen
El paisaje de células inmunitarias del microambiente tumoral contiene potencialmente información para el descubrimiento de biomarcadores pronósticos y predictivos. La inmunohistoquímica multiplex es una herramienta valiosa para visualizar e identificar diferentes tipos de células inmunitarias en los tejidos tumorales mientras se conserva su información espacial. Aquí proporcionamos protocolos detallados para analizar poblaciones de linfocitos, mieloides y células dendríticas en secciones de tejido. Desde el corte de secciones fijadas en formol e integradas en parafina, los procedimientos automáticos de tinción multiplex en una plataforma automatizada, el escaneo de los portaobjetos en un microscopio de imágenes multiespectral, hasta el análisis de imágenes mediante un algoritmo de aprendizaje automático desarrollado internamente ImmuNet. Estos protocolos se pueden aplicar a una variedad de muestras tumorales simplemente cambiando los marcadores tumorales para analizar las células inmunitarias en diferentes compartimentos de la muestra (tumor frente a margen invasivo) y aplicar el análisis del vecino más cercano. Este análisis no se limita a muestras tumorales, sino que también se puede aplicar a otros tejidos (no) patógenos. Las mejoras en el equipo y el flujo de trabajo en los últimos años han acortado significativamente los tiempos de producción, lo que facilita la aplicación futura de este procedimiento en el entorno de diagnóstico.
Introducción
Las células inmunitarias desempeñan un papel crucial en la protección contra patógenos como virus y bacterias, pero también contra las células cancerosas1. Por lo tanto, el sistema inmunitario dentro del microambiente tumoral (TME) es muy prometedor para descubrir biomarcadores pronósticos y predictivos2. Los infiltrados de células inmunitarias se han correlacionado con el pronóstico en varios tipos de cáncer, aunque esto aún no se ha implementado en la atención clínica 3,4. En la mayoría de los tipos de tumores, un número alto de linfocitos T citotóxicos y linfocitos T auxiliares 1 o un número bajo de linfocitos T reguladores se relacionan con un buen pronóstico. Se están realizando esfuerzos para incorporar el llamado "Immunoscore" en la estadificación TNM del cáncer colorrectal, convirtiéndola en la estadificación TNM-I 5,6. El Immunoscore se deriva del número total de linfocitos T (detectados con CD3) y linfocitos T citotóxicos (detectados con CD8) en dos regiones tumorales diferentes: el núcleo tumoral frente al margen invasivo (MI) de los tumores. También se ha propuesto que el Immunoscore tiene valor pronóstico en otros tipos de cáncer, como el melanoma, el cáncer de pulmón y el cáncer de mama 6,7,8,9. Además, los infiltrados de células inmunitarias también pueden correlacionarse con la respuesta a la inmunoterapia con bloqueo de puntos de control10. Sin embargo, estos biomarcadores predictivos deben ser validados en estudios prospectivos antes de que puedan ser implementados de forma rutinaria en la práctica clínica. Además, también se ha propuesto que un solo biomarcador será insuficiente para una predicción significativa11. Por ello, se ha propuesto la creación de un mapa completo de una muestra de paciente mediante la combinación de diferentes biomarcadores como un biomarcador predictivo más completo en el llamado "inmunograma del cáncer"12.
Entre los métodos para el estudio de las células inmunitarias dentro de la TME, la técnica más antigua y conocida es la inmunohistoquímica (IHQ), utilizada habitualmente para las pruebas diagnósticas de varias enfermedades, especialmente el cáncer13. Esta técnica se limitó al uso de uno o solo unos pocos marcadores14 durante mucho tiempo y, por lo tanto, fue superada en entornos de investigación por otras técnicas como la citometría de flujo y el perfil de expresión génica (GEP). Sin embargo, los tejidos tumorales fijados en formol e incluidos en parafina (FFPE) que se utilizan normalmente en el diagnóstico y la investigación de rutina no son (óptimamente) adecuados para la citometría de flujo y la GEP. Además, aunque la GEP y la citometría de flujo proporcionan mucha información sobre el fenotipo y la función de las células, la falta de información espacial es una gran desventaja. Por lo tanto, la heterogeneidad dentro de una muestra, como las diferencias en las áreas de un tumor infiltradas por células inmunitarias frente a las áreas excluidas por células inmunitarias, podría pasar desapercibida15. Se han desarrollado nuevas plataformas para el análisis multiplex de tejidos FFPE, como la IHQ multiplex, la citometría de masas por imagen y la detección de CO por indEXing (CODEX) que se pueden utilizar para detectar múltiples marcadores simultáneamente dentro de una sección de tejido16. Las células inmunitarias de la EMT se están estudiando ampliamente para encontrar los mejores biomarcadores para la inmunoterapia. Sin embargo, las técnicas multiplex y el análisis automatizado de imágenes plantean sus propios obstáculos.
Nuestro laboratorio tiene una amplia experiencia en la tinción multiplex de IHQ utilizando el método de amplificación de señal de ópalo/tiramida (TSA) y lo ha automatizado en una plataforma de IHQ (ver la Tabla de Materiales)17,18,19,20,21,22,23,24,25,26,27,28, 29,30,31. Hemos optimizado los paneles de células inmunitarias para la detección de diferentes subconjuntos de linfocitos, células mieloides y células dendríticas (CD). Los tejidos que contienen áreas densas de células inmunitarias (linfocitos o morfologías celulares complejas (es decir, células mieloides y CD) son particularmente difíciles de analizar, con el riesgo de sobreestimar o subestimar el número de células inmunitarias presentes. Para superar este problema, nuestro grupo32 desarrolló el software de análisis ImmuNet, y esta línea de aprendizaje automático mejoró enormemente la calidad de la detección de estos diferentes tipos de células inmunitarias. Aquí se describe un protocolo detallado desde la obtención del material FFPE hasta el análisis de las densidades de células inmunitarias en diferentes compartimentos de tejidos y las distancias entre los tipos de células inmunitarias.
Este protocolo describe cómo se realizan los paneles IHC multiplex en el Centro Médico de la Universidad de Radboud desde la implementación del generador de imágenes de patología digital en 2022. Los paneles IHQ multiplex descritos se pueden usar para diferentes carcinomas (por ejemplo, pulmón, próstata, colorrectal, vejiga, mama) con el uso de un anticuerpo pan-citoqueratina como marcador tumoral o para el melanoma con el uso de anticuerpos asociados a melanocitos como marcadores tumorales. Estos protocolos de IHQ multiplex se han optimizado cuidadosamente en términos de concentración de anticuerpos primarios, combinaciones de fluoróforos y la secuencia del procedimiento de tinción. Nosotros y otros hemos descrito anteriormente la optimización del panel IHC multiplex 17,33,34,35. Los paneles IHC multiplex se pueden adaptar, pero las tuberías de análisis descritas deben evaluarse y, potencialmente, ajustarse o reentrenarse en consecuencia. Los protocolos IHC multiplex de siete colores descritos utilizan los fluoróforos Opal Opal480, Opal520, Opal570, Opal620, Opal690, Opal780 y 4',6-diamidino-2-fenilindol (DAPI), de modo que se permite una fácil desmezcla y un escaneo rápido en el generador de imágenes con "Multiespectral One Touch ImmunoFluorescence" (MOTiF). La tinción y el escaneo de nueve colores no se describen en este protocolo, ya que esto requiere un ajuste aún más fino de la configuración experimental y otro modo de escaneo en el generador de imágenes que utiliza el filtro sintonizable de cristal líquido.
Protocolo
El material de pacientes que se muestra para este protocolo fue parte de un estudio realizado previamente y fue considerado oficialmente exento de aprobación ética médica por el Comité de Ética Médica local de Radboudumc concurrente con la legislación holandesa (número de archivo 2017-3164)30.
1. Recolección de material FFPE, selección de bloques y preparación de muestras
- Recupere identificadores de bloques FFPE de los archivos de los pacientes a través de médicos tratantes o patólogos. Consulte con las regulaciones locales si se requiere permiso ético.
- Solicite bloques FFPE del archivo de patología local o de hospitales externos.
NOTA: También es posible que se adquiera material tumoral o una biopsia para un estudio en particular. Este puede ser el caso de pequeños ensayos clínicos o estudios en animales. En estos casos, el procesamiento de la muestra de tejido puede ser responsabilidad del investigador. - Cuando se dispone de varios bloques de FFPE, se debe seleccionar el bloque de FFPE más representativo que contenga tejido tumoral viable, preferiblemente con tejido estromal circundante presente, evaluando los portaobjetos teñidos con hematoxilina y eosina (HE) (Figura 1).
NOTA: Se recomienda obtener la opinión de un experto para esta selección (por ejemplo, un patólogo). Es posible que las ES no estén disponibles para evaluar el contenido de un bloque FFPE y que sea necesario realizar otras nuevas para la selección. Vaya a la sección 2 para obtener una descripción. - Corte cintas FFPE de 4 μm de grosor en un micrótomo.
NOTA: El espesor puede estar entre 1 μm y 6 μm sin que se note el impacto de las manchas; sin embargo, 4 μm es el más estándar. - Monte las muestras en portaobjetos de vidrio en una posición que sea favorable para la fluidez del autotiñido (Figura 2A-C) utilizando uno de los métodos que se describen a continuación:
- Coloque las secciones sobre la superficie de agua destilada a 40 °C en un baño de agua para estirarlas y recójalas con un portaobjetos de vidrio.
O
Coloque los portaobjetos de vidrio en una placa calefactora a 40 °C, asegurándose de cubrir el lugar donde se va a montar la sección en el portaobjetos con una gota de agua destilada. Coloque la sección encima de esta gota con pinzas y deje que se estire. Absorba el agua destilada con una toalla de papel y elimine el exceso de agua golpeando el portaobjetos.
NOTA: Colocar secciones de tejido demasiado cerca de la etiqueta del portaobjetos dará como resultado una tinción subóptima (Figura 2D, E). Tendemos a montar de 6 a 10 portaobjetos de vidrio por muestra para realizar los diferentes paneles IHC multiplex y tener un respaldo.
- Coloque las secciones sobre la superficie de agua destilada a 40 °C en un baño de agua para estirarlas y recójalas con un portaobjetos de vidrio.
- Deje secar los portaobjetos de vidrio montados a 56 °C durante 1 h o toda la noche a 37 °C.
- Utilice los portaobjetos de vidrio montados para el experimento o guárdelos en cajas a 4 °C.
NOTA: En nuestra experiencia hasta ahora, estos portaobjetos montados se pueden almacenar durante años antes de que se lleve a cabo la tinción IHC multiplex.
2. Generación de portaobjetos teñidos con hematoxilina y eosina
NOTA: Todos los pasos siguientes de la sección 2 deben llevarse a cabo en una campana extractora.
- Portaobjetos desparafinados en xileno (2 x 5 min).
- Rehidratar en etanol (99,6% 1 x 5 min; 95% 1 x 5 min; 70% 1 x 2 min). Alternativamente, sumerja los portaobjetos 3 veces en etanol al 99,6%.
- Lave los portaobjetos en agua destilada (2 min).
- Tiñir los núcleos con hematoxilina (10 min).
- Lave los portaobjetos con H2O destilado (5 min).
- Tiñe los portaobjetos con eosina (5 min).
- Deshidrate los portaobjetos sumergiéndolos 3 veces en etanol al 99,6%.
- Sumerge los portaobjetos 2 veces en xileno.
- Agregue unas gotas de medio de montaje y selle con un cubreobjetos.
- Deje que los portaobjetos se endurezcan y sáquelos de la campana extractora cuando todos los productos químicos se hayan evaporado.
3. Realización de IHC monoplex y multiplex en el autostainer
- Calcule la cantidad de reactivo que se necesita en función del número de muestras que se van a teñir.
NOTA: Por ejecución, el autotinificador tiene una capacidad de 30 portaobjetos y tarda ~ 18 h en completar el protocolo IHC multiplex con seis anticuerpos. Cuando es necesario teñir más portaobjetos, se pueden colocar varios lotes cada noche de la semana (laboral); 4 noches de 30 diapositivas = 120 diapositivas por semana.- Prepare todos los reactivos necesarios al comienzo de la semana. El sistema de autotinción dispensa 150 μL de reactivo por portaobjetos. Utilice los recipientes de valoración de 6 ml para los reactivos de anticuerpos y ópalo y los recipientes de 30 ml para el reactivo de bloqueo y el anticuerpo secundario-peroxidasa de rábano picante.
NOTA: Los recipientes de 6 ml tienen insertos convenientes que se pueden quitar y reemplazar fácilmente cuando sea necesario. Con los cálculos de reactivos, se debe considerar el volumen muerto de 1,6 mL o 300 μL para el contenedor de 30 mL o el recipiente de valoración de 6 mL, respectivamente. - Diluya todos los fluoróforos Opal y la digoxigenina (DIG) 1:100 en el diluyente proporcionado; diluya Opal780 1:25 en el diluyente de anticuerpos. Diluya todos los anticuerpos primarios en el diluyente de anticuerpos, con las diluciones especificadas en el Archivo Suplementario 1.
- Prepare todos los reactivos necesarios al comienzo de la semana. El sistema de autotinción dispensa 150 μL de reactivo por portaobjetos. Utilice los recipientes de valoración de 6 ml para los reactivos de anticuerpos y ópalo y los recipientes de 30 ml para el reactivo de bloqueo y el anticuerpo secundario-peroxidasa de rábano picante.
- Para seguir este protocolo, ejecute IHC monoplex (Archivo suplementario 2) en portaobjetos que contengan tejido de control de amígdalas y otros tipos de tejido (tumoral) de interés antes de comenzar con el experimento de IHQ multiplex real para asegurarse de que todos los reactivos estén bien preparados.
NOTA: Monoplex IHC tarda ~ 3.5 h y se puede verificar antes del final de ese día para ver los patrones de señal y la intensidad. Si ciertas señales son demasiado débiles (Figura 3), se pueden realizar ajustes en los reactivos. - Para la corrección de la autofluorescencia, prepare un portaobjetos con tejido (tumoral) que contenga estructuras autofluorescentes, como sangre y colágeno. Prepare este portaobjetos simultáneamente con los portaobjetos monoplex de IHC, pero con un reactivo de bloqueo que sustituya al anticuerpo y a los reactivos Opal (Archivo complementario 3).
NOTA: En principio, un portaobjetos de este tipo se puede reutilizar para obtener imágenes multiespectrales hasta que la corrección de la autofluorescencia ya no sea óptima. Sin embargo, con tejidos altamente autofluorescentes, como el cerebro y el hígado, es aconsejable utilizar ese tejido para la corrección de la autofluorescencia. - Con cada corrida de IHQ múltiplex, cargue 29 muestras en el sistema de tinción automática con un portaobjetos de tejido de control para comprobar el rendimiento de cada corrida de IHC multiplex.
- Descargue los protocolos IHC multiplex desde el sitio web del autostainer en la pestaña Descargas y ajústelos para que se ajusten a cada panel IHC multiplex personalizado36. Para IHC múltiplex, consulte el Archivo Suplementario 4 para el protocolo y para los paneles IHC multiplex personalizados, consulte el Archivo Suplementario 1.
- Una vez finalizado el protocolo de tinción, saque los portaobjetos del autotinción y colóquelos en un recipiente con tampón de lavado.
- Para evitar la contaminación del sistema de autotinción con DAPI, ya que las muestras ya están teñidas a concentraciones muy bajas, aplique DAPI manualmente antes de cubrir los portaobjetos con cubreobjetos. Añadir dos gotas de DAPI por mL de tampón de lavado e incubar durante 5 min a temperatura ambiente en la oscuridad.
NOTA: Para construir bibliotecas espectrales, es importante no tener ningún DAPI manchado en las muestras. También es posible una gota de DAPI por ml de tampón de lavado y 10 min de incubación en RT. - Lave los portaobjetos 3 veces con tampón de lavado.
- Coloque los portaobjetos sobre toallas de papel y retire el exceso de tampón de lavado de los portaobjetos.
- Pipetear unas gotas de medio de montaje sobre el tejido.
- Coloque suavemente un cubreobjetos de vidrio sobre el medio de montaje para cubrir la corredera en ángulo y evitar burbujas de aire.
- Elimine el exceso de medio de montaje y las burbujas de aire empujando suavemente el cubreobjetos de vidrio con pinzas o una punta de pipeta limpia.
- Deje los portaobjetos sin tocar durante ~ 24 h antes de que el medio de montaje se solidifique, ya sea horizontalmente en una placa de portaobjetos de microscopía o cárguelos directamente en el microscopio para obtener imágenes.
- Después de que el medio de montaje se solidifique o después de obtener imágenes de los portaobjetos, almacene los portaobjetos en cajas de microscopía a 4 °C.
4. Obtención de imágenes mediante el generador de imágenes de patología digital y anotación de archivos de escaneo
- Encienda el generador de imágenes presionando el botón de encendido a la derecha de la máquina. Después de al menos 20 segundos, inicie el software.
NOTA: Espere 20 s para permitir que el hardware se inicie correctamente. - Cargue las diapositivas en los casetes por cada cuatro diapositivas.
- Opcional: Introduzca las diapositivas en un archivo .csv para el que se pueda descargar una plantilla (Archivo complementario 5). Para cargar el archivo .csv en el programa, guárdelo en C:\Users\Public\Akoya\VectraPolaris\States.
NOTA: Se puede cargar un máximo de 20 casetes u 80 diapositivas simultáneamente.
- Opcional: Introduzca las diapositivas en un archivo .csv para el que se pueda descargar una plantilla (Archivo complementario 5). Para cargar el archivo .csv en el programa, guárdelo en C:\Users\Public\Akoya\VectraPolaris\States.
- Configuración de referencia
- Abra Check Dashboard desde el menú principal.
NOTA: El fabricante proporciona un casete con correderas de referencia y, opcionalmente, se puede guardar de forma permanente en la ranura 20. - Configure las referencias de campo claro en el portaobjetos provisto una vez por semana de acuerdo con las instrucciones del fabricante (tarda unos minutos).
- Configure las referencias de fluorescencia en el portaobjetos provisto una vez al mes de acuerdo con las instrucciones del fabricante (tarda más de 1 h).
- Abra Check Dashboard desde el menú principal.
- Elaboración o ajuste del protocolo
- Regrese al menú principal y haga clic en Editar protocolo para crear un protocolo.
- Haga clic en Nuevo... y seleccione Fluorescencia como modo de imagen, escaneo de portaobjetos multiespectral y color Opal Polaris 5, 6 y 7 en la opción Tinción .
- Asigne un nombre al protocolo en Nombre del protocolo y guárdelo en un estudio seleccionando un estudio de Estudios disponibles o cree un estudio en Crear nuevo estudio | Nombre del estudio.
- Para finalizar, seleccione Crear protocolo.
- Para este tipo de escaneo, use solo la ventana de la izquierda Configuración de escaneo de diapositivas multiespectrales; ignore la ventana de la derecha Configuración de campo multiespectral.
- Escanee las diapositivas con diferentes aumentos. Para seguir este protocolo, escanee con un aumento de 20x dejando la resolución de píxeles en 0,50 μm (20x).
- Establezca los tiempos de exposición seleccionando Exposiciones de escaneo.
- Cargue el casete en el que se guardan las correderas seleccionando la ranura correcta en la opción Portador de carga .
- Para ayudar a navegar por las diapositivas, seleccione Tomar información general para obtener una imagen general del portador que contiene las diapositivas después de cargar el portador. Para activar o desactivar esta opción automáticamente, haga clic en el icono de engranaje en la parte superior derecha, vaya a Preferencias..., y marque la opción de activar o desactivar en Imagen de descripción general de la navegación para habilitar el portador de imagen automáticamente al cargar para tareas interactivas.
- Establezca los tiempos de exposición por filtro en los portaobjetos monoplex teñidos con IHC correspondientes seleccionando Establecer exposiciones de escaneo y buscando diferentes puntos con una señal positiva. Enfoque manual o use Enfoque automático y seleccione Exposición automática después de cambiar al filtro compatible para esa señal. Seleccione el tiempo de exposición más bajo para evitar la sobreexposición y tome instantáneas de cada diapositiva como referencia después de establecer todos los tiempos de exposición (Figura 3).
NOTA: Ignore la opción Establecer exposiciones de campo para este tipo de escaneo. - Establezca los tiempos de exposición en un portaobjetos teñido multiplex comprobando todos los filtros en algunas ubicaciones con señal positiva. Reduzca el tiempo de exposición automática más bajo en un 10% para evitar la sobreexposición y tome algunas instantáneas después de establecer todos los tiempos de exposición.
- Tome instantáneas del portaobjetos sin teñir para la compensación de autofluorescencia utilizando el filtro AF de muestra para navegar (Figura 3H).
NOTA: Las localizaciones con eritrocitos y estructuras de colágeno son de interés. Es posible que sea necesario reducir el tiempo de exposición del filtro Opal480 para regiones autofluorescentes fuertes. Si la señal Opal480 es lo suficientemente fuerte, aún debe separarse bien (consulte la sección 6) de las estructuras autofluorescentes debido a la implementación del filtro Sample AF patentado. - Evalúe la calidad de la tinción y la obtención de imágenes utilizando el software (ver secciones 5 y 6; Figura 4, Archivo Suplementario 6: Figura Suplementaria S1 y Figura Suplementaria S2).
- Seleccione el botón Guardar... para asegurarse de que el protocolo y sus tiempos de exposición ajustados se guardan en el protocolo.
NOTA: Cuando el protocolo ya está guardado, el software no proporciona ninguna notificación adicional de los ajustes no guardados hasta el momento.
- Escaneo automático de portaobjetos
- Vuelve al menú principal y haz clic en Escanear diapositivas para escanear las diapositivas.
- Introduzca manualmente los nombres/ID de las diapositivas y las tareas y el protocolo correspondientes en Configurar tareas o automáticamente desde el archivo .csv creado anteriormente con Configuración de carga.
- Haga clic en Escanear para iniciar el escaneo.
- Espere a que aparezca una ventana para guardar la configuración del escaneo. Haga clic en Guardar para usar la configuración predeterminada y comenzar a escanear.
NOTA: El escaneo con este método tarda ~10-20 minutos por diapositiva. Dependiendo del número de diapositivas, el escaneo puede tardar hasta un día completo. - Compruebe si el escaneo de las diapositivas se ha realizado correctamente para todas las diapositivas buscando cualquier mensaje de error. Para saber si el escaneo se ha realizado correctamente, busque un archivo de escaneo de portaobjetos completo de Akoya guardado (.qptiff) del escaneo y el tejido completo en el escaneo.
5. Anotación de datos mediante el visor de diapositivas
- Regrese al menú principal y haga clic en Iniciar Phenochart para abrir el visor de diapositivas.
- Si los archivos escaneados no son directamente visibles, designe su ubicación haciendo clic primero en el icono de ajustes en la esquina superior derecha, vaya a Cambiar ubicación del navegador... y seleccione aleatoriamente uno de los archivos .qptiff del conjunto de datos de interés.
NOTA: Los datos se almacenan de forma predeterminada en D:\Data\VectraPolaris. - Cargue una diapositiva seleccionándola y haciendo clic en Cargar en la esquina superior derecha o haciendo doble clic en ella.
- Inicie sesión haciendo clic en el botón Iniciar sesión en la esquina superior derecha.
NOTA: El nombre de usuario puede ser solo las iniciales o el nombre y se usa para realizar un seguimiento de quién hizo qué anotaciones. - Para realizar la desmezcla, haga clic en el botón Desmezclar en la parte superior y seleccione la opción Ópalo + AF .
NOTA: Esto es útil para deshacerse de parte de la señal autofluorescente cerca del canal Opal 480, pero no toda. - Para generar un algoritmo para el procesamiento por lotes de los datos, seleccione imágenes representativas utilizando el sello utilizando la opción para imágenes de proyectos inForm 1 x 1 (tamaño de imagen: 928 μm x 696 μm).
NOTA: Se seleccionan algunos sellos representativos que contienen tumores, estromas, antecedentes y diferentes tipos de células inmunitarias en todo el conjunto de datos para terminar con ~ 20-30 imágenes. - En función de lo que se necesite analizar en el tejido, seleccione una región de interés utilizando la opción ROI y seleccione inForm Batch. Elimine manualmente las imágenes que no necesitan ser analizadas, como las imágenes que están demasiado lejos del tumor o en el fondo.
NOTA: Tendemos a dibujar un ROI alrededor de todo el tumor y seleccionamos una imagen adicional lejos de la región tumoral para poder analizar un IM de ~0,5 mm.
Si el ROI dibujado es relativamente pequeño, el ROI consistirá en 2-9 imágenes 20x combinadas. Como no lo preferimos, selle manualmente el tejido de interés (seleccionado para inForm Batch) para evitar esto. - Cuando termine de anotar, deje que las anotaciones se guarden automáticamente y cargue la siguiente diapositiva.
- Durante el proceso de anotación, compruebe si las diapositivas se han escaneado correctamente.
- Si falta un archivo .qptiff o un portaobjetos no se ha escaneado correctamente, compruebe si hay algún tejido en el portaobjetos, limpie el portaobjetos con etanol al 70% y vuelva a escanear.
- Si el tejido no se escanea completamente, por lo que se pierde una región potencialmente importante (tumor), o si la exploración de la región importante estaba desenfocada, limpie el portaobjetos con etanol al 70% y vuelva a escanear.
NOTA: En ambos casos, también puede ser útil rodear el tejido con un marcador en la parte superior del cubreobjetos para ayudar al sistema a localizar el tejido e intentar escanear nuevamente (Archivo Suplementario 6: Figura Suplementaria S3). En nuestras manos, un rotulador rojo fino funcionaba mejor que un rotulador negro grueso.
- Una vez que se completen el escaneo y la anotación de todas las muestras, haga una copia de seguridad de los datos almacenándolos en una computadora diferente o en un disco externo.
6. Desmezcla espectral
- Abra el software de análisis automatizado de imágenes inForm.
- Cargue las imágenes en el software por Archivo | Imagen abierta; Seleccione Archivos .qptiff. Deje que los sellos, marcados como inForm Projects en el paso 5.6, se carguen en el proyecto.
- Cargue los archivos .qptiff de los que se genera una imagen para la compensación de autofluorescencia.
- Para compensar la autofluorescencia, utilice la herramienta Seleccionar autofluorescencia en la imagen para dibujar una línea en la imagen desde el portaobjetos sin teñir a través de diferentes tipos de estructuras autofluorescentes, como los eritrocitos y el colágeno.
- En la sección Editar marcadores y colores... , asigne nombres de marcadores que correspondan al fluoróforo Opal y ajuste el color al preferido.
- Para desmezclar los fluoróforos, seleccione Preparar todo en la esquina inferior izquierda.
- Revise las imágenes y verifique si todas las señales son visibles en las imágenes y si la desmezcla salió bien. Seleccione el icono del globo ocular para apagar y encender todos los marcadores uno por uno para comprobar la calidad.
- Opcionalmente, entrene los algoritmos para la segmentación de tejidos, la segmentación celular y el fenotipado.
- Vaya a la pestaña Exportar y cree un nuevo directorio de exportación vacío haciendo clic en el botón Examinar... debajo del Directorio de exportación.
- En Imágenes para exportar:, seleccione Imagen compuesta e imágenes de componentes (TIFF de varias imágenes).
- Seleccione Archivo | Guardar | Proyecto para guardar el algoritmo en una ubicación determinada.
- Vaya a la pestaña Análisis de lotes verticalmente a la izquierda para el procesamiento por lotes de portaobjetos.
- Seleccione Crear directorios separados para cada elemento en Opciones de exportación.
- Para agregar diapositivas para el análisis, seleccione archivos .qptiff en el botón Agregar diapositivas... y cárguelos en el análisis por lotes.
- Seleccione Ejecutar para iniciar el procesamiento por lotes de diapositivas.
7. Dibujo del ROI
- Cree una carpeta con solo los archivos de componentes de la sección 6, pero mantenga intacta la estructura jerárquica de carpetas (los archivos de componentes están en carpetas nombradas por muestra/diapositiva).
- Abra el software de visualización de diapositivas completo QuPath.
- Haga clic en Crear proyecto a la izquierda y seleccione/cree una nueva carpeta vacía con un nombre adecuado.
- Haga clic en Automatizar y seleccione Mostrar editor de scripts.
- Copie y pegue el script que está disponible en el archivo complementario 7. En la línea 34, cambie la ubicación a donde están las carpetas de diapositivas que contienen todos los archivos componentes (la carpeta creada en el paso 7.1.
- Seleccione Ejecutar y volver cuando finalice la costura por lotes de diapositivas (al día siguiente o más tarde) para continuar.
- Arrastre los archivos de .ome.tif generados al proyecto QuPath y guárdelos como un proyecto.
- Cuando aparezca automáticamente una nueva ventana, selecciona Establecer tipo de imagen | Fluorescencia y haga clic en Importar.
- En el menú de la izquierda, observe la lista de muestras; haga doble clic en uno para abrir la muestra (Figura 5A).
- Para ajustar la intensidad de los canales y hacerlos más visibles, haga clic en el icono de contraste.
- Seleccione todos los canales y haga clic en Restablecer.
- Desactiva la autofluorescencia.
- Para empezar a dibujar un ROI para el tumor, haga clic en el icono de contraste y seleccione Mostrar escala de grises. Seleccione el canal marcador tumoral y ajuste la intensidad para que sea óptimamente visible (Figura 5B).
- Haga clic en la herramienta de pincel para dibujar un ROI del tumor de forma aproximada.
- Mientras selecciona la herramienta de varita, haga clic fuera del ROI mientras presiona la tecla alt para suavizar el ROI desde el exterior (Figura 5C).
- Fusione piezas tumorales separadas con el mismo retorno de la inversión.
- Asigne al ROI un nombre adecuado, como tumor, haciendo clic con el botón derecho en la anotación de la lista de la izquierda; seleccione Establecer propiedades y escriba el nombre.
- Para realizar un ROI para el IM, expanda el ROI existente de la región tumoral seleccionando: Objetos | Anotaciones... | Expandir anotaciones.
- Seleccione el tamaño que debe tener el radio de expansión y seleccione Eliminar interior y Restringir a padre (Figura 5D).
- Haga clic en el icono de contraste, seleccione el canal de autofluorescencia y ajuste la intensidad para que sea visible de forma óptima.
- Haga clic en la varita y ajuste el ROI mientras presiona la tecla alt para suavizar el ROI desde el exterior y eliminar cualquier fondo que no deba ser parte de este ROI.
- Asigne al ROI un nombre adecuado, como margen invasivo o mensajería instantánea , haciendo clic con el botón derecho en la anotación de la lista de la izquierda, seleccione Establecer propiedades, escriba el nombre y, opcionalmente, cambie su color a verde.
- Guarde las anotaciones: Archivo | Exportar objetos | Exportar todos los objetos y haga clic en Aceptar con la selección predeterminada en Exportar como FeatureCollection y guárdelo en una ubicación preferida.
8. Detección de células inmunitarias
- Dado que ImmuNet utiliza datos de componentes (archivos TIFF multicanal) tanto para el entrenamiento como para la inferencia, divida las anotaciones en conjuntos de entrenamiento y validación. Para entrenar el modelo, siga los pasos descritos en el archivo Léame del repositorio, sustituyendo el conjunto de datos de ejemplo y las anotaciones con los datos deseados. Aparte de las diferentes células inmunitarias, proporcione al modelo ejemplos negativos haciendo anotaciones de fondo en sitios que no deben reconocerse como una célula de interés: células tumorales, otras células o "no células" (estructuras que podrían confundirse con células de interés); véase la publicación de ImmuNet para más detalles32.
- Con las anotaciones de validación, asegúrese de que el rendimiento sea satisfactorio. Observe la tasa de error por tipo de anotación, una parte de las anotaciones de validación que el modelo no ha detectado, la métrica de evaluación más sencilla. Evalúe el rendimiento con respecto a los falsos positivos realizando algunos ROI completamente anotados y calculando las puntuaciones F.
- Además de la evaluación cuantitativa, inspeccione visualmente la predicción para obtener una idea cualitativa de los errores que tiende a cometer el modelo (Figura 6, Archivo complementario 6: Figura complementaria S4 y Figura complementaria S5). Si se considera que el rendimiento del modelo es insuficiente, visualice la predicción de algunos mosaicos como se describe en el repositorio y verifique qué sitios son los más propensos a errores. Realice más anotaciones en dichos sitios y vuelva a ejecutar el entrenamiento y la evaluación del modelo.
- Cuando se logre el rendimiento objetivo, ejecute la inferencia para todo el conjunto de datos como se describe en la sección Inferencia para todo el conjunto de datos del archivo Léame del repositorio. Utilice los archivos de .csv obtenidos con la predicción del modelo como entrada para el análisis de datos (escriba un script de Python o R para eso).
9. Predicción, fenotipado y análisis de datos
NOTA: En esta sección, ofrecemos un ejemplo de análisis de datos simple para una sola muestra de melanoma teñida con el panel de linfocitos, que combina las ubicaciones de las células inmunitarias identificadas por ImmuNet (sección 8) y los ROI delineados con QuPath (sección 7). El análisis se ha realizado en R 4.1.1 (se proporciona un script como Archivo Suplementario 8). El script requiere los paquetes: plyr 1.8.8, dplyr 1.0.8, tidyr 1.2.0, sf 1.0-7, ggplot2 3.4.0, RANN 2.6.1 y RColorBrewer 1.1-2, que se pueden instalar con el comando install.packages(). Como entrada, toma un archivo .csv con la predicción de ImmuNet de una muestra y un archivo con ROI exportados desde QuPath. Los pasos 9.1-9.6 describen el análisis de una sola muestra realizado en el script proporcionado, y las secciones 9.7-9.9 describen las opciones para el análisis de múltiples muestras.
- Después de cargar la predicción de ImmuNet en R, determine los umbrales para la expresión de marcadores predichos trazando los marcadores que definen los fenotipos entre sí y seleccionando los umbrales que separan mejor las poblaciones.
NOTA: La estrategia de compuerta utilizada para la muestra dada se muestra en la Figura 7B. Las estrategias de activación para los paneles de células mieloides y dendríticas se muestran en el Archivo Suplementario 6: Figura Suplementaria S6 y Figura Suplementaria S7. - Después de determinar los umbrales, utilícelos para asignar a cada predicción de ImmuNet un fenotipo definido en un panel. En algunas predicciones, observe que ninguno de los marcadores predichos está por encima del umbral o que la combinación de marcadores considerados expresados después del umbral puede ser inconsistente (por ejemplo, predicciones de CD3+ CD20+ en los paneles de linfocitos). Si se logra un buen rendimiento del modelo en el paso 8.3, la fracción de tales predicciones será pequeña; Filtrarlos antes del análisis.
- Para analizar por separado el ROI del tumor y su margen invasivo de hasta 100 μm dibujado en QuPath, cargue los archivos GeoJSON correspondientes en R y, para cada predicción, determine el ROI en el que se encuentra la predicción.
- Para una comprobación de la cordura y como parte del análisis exploratorio de datos, visualice las células inmunitarias que se encuentran en una muestra por separado en los ROI correspondientes junto con los límites de los ROI (Figura 7A).
- Ahora, calcule las densidades de diferentes células inmunitarias por separado para cada ROI. Las densidades encontradas en la muestra dada se muestran en la Tabla 1.
- Si hay varias muestras disponibles, visualice la distribución de las densidades de celdas. Transforme logarítmicamente los valores de densidad para obtener valores distribuidos normalmente.
NOTA: Cuando los recuentos de ciertos fenotipos son 0, estos no se pueden transformar en Log, lo que lleva a que falten valores. Para superar este problema, el suavizado de LaPlacian se puede aplicar añadiendo primero 0,5 a todos los recuentos de células antes de dividirlo por el área de la superficie. - Analice los valores de densidad y represúrelos utilizando el software de su elección (Figura 8).
- Las ubicaciones conservadas de las celdas permiten el análisis espacial. Por ejemplo, para cada célula inmunitaria detectada, encuentre un vecino más cercano y, a continuación, para cada fenotipo, calcule el porcentaje de casos en los que los diferentes fenotipos se presentan como el vecino más cercano.
NOTA: Dado que el número de células asesinas naturales (NK) encontradas en esta muestra era muy pequeño, las excluimos de este análisis. Los resultados obtenidos para el ROIs tumoral e IM se muestran en la Figura 9.
Resultados
Los bloques de FFPE que contenían tejido tumoral se seleccionaron sobre la base de informes de patología y portaobjetos teñidos con HE. Cuando se resecan múltiples lesiones tumorales del paciente y/o las muestras tumorales son grandes, estas se dividen en múltiples bloques FFPE. Preferimos analizar las células inmunitarias tanto en el compartimento tumoral como en lo que se conoce como margen invasivo (MI) del tumor. El MI es tejido estromal no canceroso adyacente al tumor. Por lo tanto, cuando hay varios bloques FFPE disponibles para una muestra tumoral, se seleccionan los bloques FFPE que contienen ambos tipos de tejido. Como se observa en los portaobjetos teñidos con HE, un bloque de FFPE contenía tejido tumoral y tejido estromal adyacente al tumor (Figura 1A). Otro bloque de FFPE del mismo tumor contenía mucho menos tejido estromal circundante (Figura 1B). Sin embargo, para algunas muestras de tejido no hay opción en los bloques FFPE o el IM no está presente en ninguno de los bloques FFPE. Este suele ser el caso de las biopsias (con aguja), que deben tenerse en cuenta durante la interpretación de los datos.
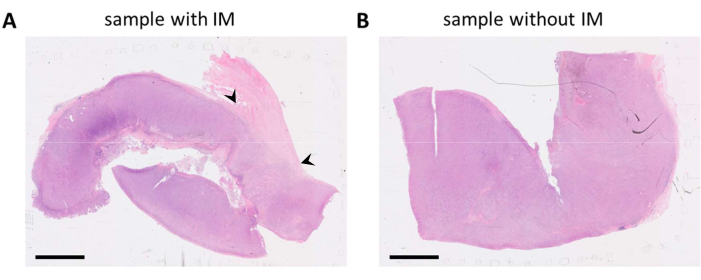
Figura 1: Portaobjetos teñidos con HE de un espécimen de tumor de melanoma. (A) Un ejemplo de una muestra de tumor con tejido estromal adyacente al tumor (como primario) en la esquina superior derecha de la muestra (indicada con puntas de flecha negras). (B) Otra muestra de la misma lesión tumoral con poco o ningún tejido estromal presente en la muestra. Barras de escala = 5 mm. Abreviaturas: HE = hematoxilina y eosina; IM = margen invasivo. Haga clic aquí para ver una versión más grande de esta figura.
La tinción IHQ multiplex con un panel propuesto de siete colores (Archivo Suplementario 4) se puede realizar manualmente en un proceso de tinción de 3 días (considerando las horas normales de trabajo) o durante la noche en un autotintor. Al utilizar el autostainer, las secciones deben montarse en una ubicación particular del portaobjetos de vidrio que permita una fluidicidad óptima del sistema (Figura 2A). Cuando las secciones están correctamente montadas en portaobjetos (Figura 2B), se teñirán uniformemente (Figura 2C). Si las secciones no están montadas de manera óptima en el portaobjetos de vidrio (Figura 2D), a menudo se produce un patrón de tinción subóptimo (Figura 2E) porque los fluidos del autotinción no alcanzan el tejido (completo). Esto puede suceder cuando las muestras son muy grandes o cuando los portaobjetos montados son proporcionados por alguien que no está al tanto de este problema. En estos casos, solo se debe seleccionar la parte bien manchada del portaobjetos para el análisis. Otra opción para este tipo de muestras podría ser teñirlas manualmente para esparcir los líquidos de manera óptima.
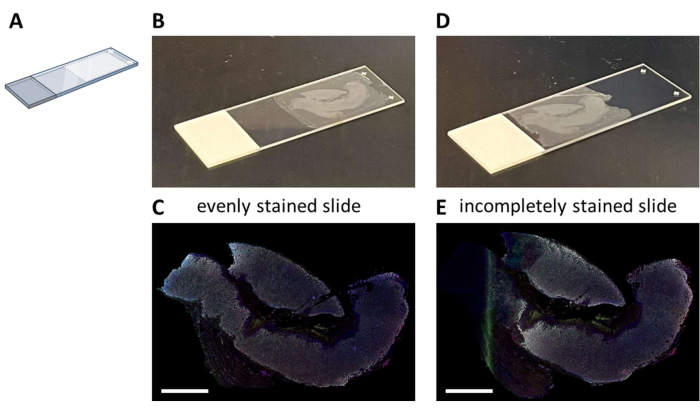
Figura 2: Montaje de la sección FFPE en el portaobjetos de vidrio e impacto. (A) Esquema de dónde montar en el portaobjetos de vidrio para una tinción óptima en el autostainer. (B) Ejemplo de una corredera montada correctamente. (C) Los portaobjetos montados correctamente dan como resultado una sección de tejido teñida uniformemente. (D) Ejemplo de una corredera montada subóptima. (E) Los portaobjetos montados subóptimos pueden dar como resultado una sección de tejido teñida incompleta, como se ve en el lado izquierdo de esta imagen. Barras de escala = 5 mm. Abreviatura: FFPE = fijada en formol e integrada en parafina. Haga clic aquí para ver una versión más grande de esta figura.
Cuando se realizan grandes experimentos de IHQ multiplex en múltiples rondas de tinción y es necesario preparar grandes cantidades de soluciones, es mejor probar primero estos reactivos en una serie de IHQ monoplex antes de proceder a la IHQ multiplex. La IHQ monoplex se verifica con el generador de imágenes de patología digital para determinar los patrones de tinción esperados y los tiempos de exposición se establecen con los filtros correspondientes en portaobjetos de control (Figura 3A-H). El tejido de las amígdalas se utiliza como control positivo para la mayoría de los marcadores de células inmunitarias. Dado que el tiempo de exposición a DAPI en el tejido control de las amígdalas es siempre mayor que en otros tejidos (Figura 3G), el tiempo de exposición a DAPI debe ajustarse al tipo de tejido que se va a estudiar. Los tiempos de exposición regulares con este tipo de escaneo están entre 1 ms y 30 ms, dependiendo del fluoróforo y el filtro (Figura 3I). Cuando una IHQ monoplex supera estos números o el patrón de tinción no es tan claro como se esperaba, se debe ajustar o reemplazar la solución de anticuerpos. En el ejemplo que se muestra aquí, decidimos aumentar la concentración de FOXP3 (Figura 3C y Figura 3I) para tener la intensidad más en el rango de los otros marcadores. La autofluorescencia también puede ser más fuerte en otros tejidos que en el tejido de control de las amígdalas. En nuestra configuración, el tiempo de exposición para el filtro AF de muestra está entre 25 ms y 50 ms (Figura 3H,I).

Figura 3: Ajuste de los tiempos de exposición en muestras IHQ monoplex y muestras de control sin teñir. (A) CD20 - Señal de Opal 480 en el tejido de control de las amígdalas. (B) CD3 - Señal de ópalo 520 en el tejido de control de las amígdalas. (C) FOXP3 - Señal de Opal 570 en el tejido de control de las amígdalas. (D) CD56 - Señal de Opal 620 en el tejido de control de las amígdalas (E) CD8 - Señal de Opal 690 en el tejido de control de las amígdalas. (F) Marcador tumoral: señal de Opal 780 en el tejido de control de las amígdalas. (G) La señal DAPI en el tejido de control de las amígdalas suele ser más débil que el tipo de tejido de interés. (H) Autofluorescencia: muestra la señal de AF en el tejido de control tumoral. (I) Captura de pantalla de los tiempos de exposición antes de ajustarlo al 10% y comprobar en portaobjetos teñidos con IHC múltiplex. Barras de escala = 100 μm. Abreviaturas: AF = autofluorescencia; IHQ = inmunohistoquímica; DAPI = 4'6-diamidino-2-fenilindol. Haga clic aquí para ver una versión más grande de esta figura.
Después de realizar la IHC multiplexada, los tiempos de exposición se ajustan desde la configuración de IHC monoplex comprobando algunas diapositivas IHC multiplex y seleccionando la exposición automática. Con este tipo de escaneo, no hay opción de protección contra la saturación y, por lo tanto, es extremadamente importante evitar establecer la exposición demasiado alta, evitando así la sobreexposición. La sobreexposición dificulta la desmezcla espectral de los fluoróforos. A menudo no establecemos tiempos de exposición que excedan los tiempos de exposición que se basaron en la IHC monoplex y solo disminuimos los tiempos de exposición para los marcadores que son más fuertes en la IHC múltiplex (Figura 3G y Figura 4A). Al autoexponer en diferentes ubicaciones de algunas diapositivas, se puede observar que los tiempos de exposición de algunos filtros siguen siendo demasiado altos. Estos deben ajustarse al número más bajo que se observa cuando se usa la configuración de exposición automática y restar otro 10% del valor para evitar la sobreexposición en otras ubicaciones invisibles (Figura 4A). Con este método, los tiempos de exposición pueden ser más bajos para ciertos filtros que los que se establecieron en el IHC monoplex. Sin embargo, con un experimento de IHQ múltiplex exitoso, todos los marcadores deben ser observables, al menos en el portaobjetos de control (Figura 4B-H, Archivo Suplementario 6: Figura Suplementaria S1 y Figura Suplementaria S2). Tenga en cuenta que ciertos marcadores pueden no estar presentes en todas las muestras. Al incluir un portaobjetos de control que contiene al menos una sección de amígdalas, se puede verificar el éxito de la tinción de todos los marcadores de los paneles estándar y la intensidad de la señal.
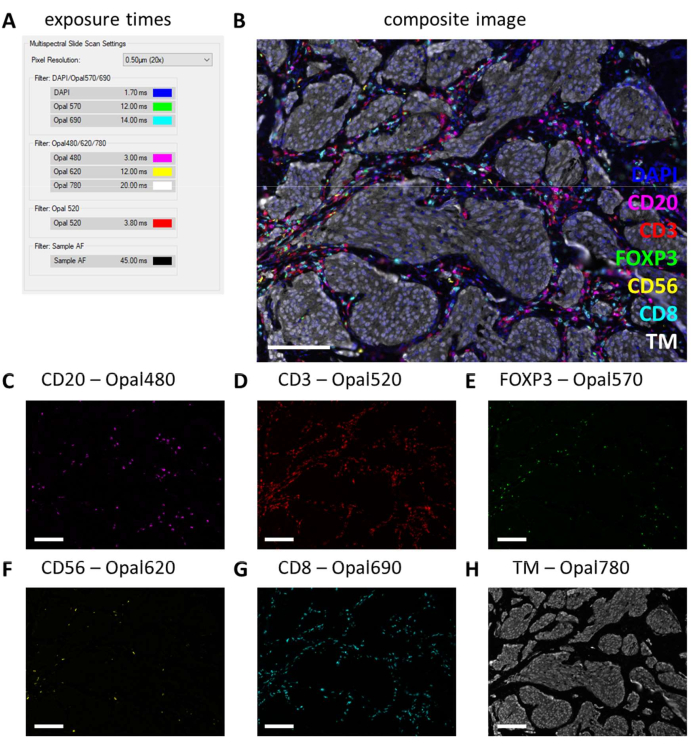
Figura 4: Ejemplo de una sección teñida con éxito con el panel de linfocitos en un espécimen de tumor de melanoma. (A) Tiempos de exposición utilizados para registrar esta muestra múltiple de IHQ. (B) Imagen compuesta del panel de linfocitos IHQ multiplex dentro del tejido tumoral. (C) CD20 - Señal Opal 480 en magenta. (D) CD3 - Señal Opal 520 en rojo. (E) FOXP3 - Señal Opal 570 en verde. (F) CD56 - Señal Opal 620 en amarillo. (G) CD8 - Señal Opal 690 en cian. (H) TM - Opal 780 en blanco. Barras de escala = 100 μm. Abreviatura: TM = marcador tumoral; IHQ = inmunohistoquímica. Haga clic aquí para ver una versión más grande de esta figura.
Los portaobjetos IHC multiplex son escaneados completamente por el generador de imágenes digitales. Los mosaicos para el análisis posterior se seleccionan en el visor de diapositivas. Sin embargo, cuando es necesario analizar regiones más específicas, como el tumor frente a la IM, estas regiones de interés (ROI) se pueden dibujar mediante QuPath. Una vez completado el procesamiento por lotes de los mosaicos seleccionados en el visor de diapositivas, los archivos de componentes se vuelven a combinar (Figura 5A y Archivo complementario 7). Utilizando el canal marcador tumoral (Figura 5B) y la herramienta de varita mágica en QuPath, se puede trazar el contorno del tumor para formar el "ROI del tumor" (Figura 5C). A continuación, el ROI del tumor se puede ampliar con una cierta distancia, en este caso, 500 μm, para crear un "ROI de margen invasivo" (Figura 5D). Cualquier fondo no deseado (no tejido) se elimina de este ROI con la herramienta de varita mágica observando la señal de autofluorescencia (Figura 5E). Tanto el ROI del tumor como el ROI de IM se guardan como un archivo GeoJSON para su posterior procesamiento (Figura 5F).
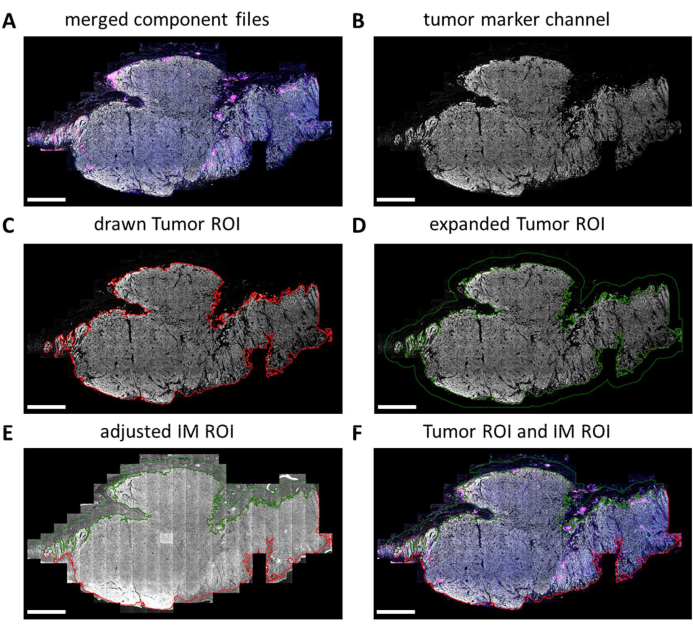
Figura 5: Proceso de dibujo del ROI del tumor y del ROI del margen invasivo en QuPath. (A) Archivos de componentes combinados. (B) Imagen en escala de grises que muestra solo el canal marcador tumoral. (C) El ROI del tumor se dibuja alrededor de la señal del marcador tumoral. (D) Se obtiene un nuevo ROI expandiendo el ROI del tumor en 100-500 μm para formar el ROI IM. (E) El ROI de IM se ajusta para incluir solo el tejido estromal al excluir el fondo (señal negativa) y otras estructuras de tejido grandes como la grasa, los vasos sanguíneos y los folículos pilosos. (F) El ROI del tumor y el ROI de IM resultantes se guardan y exportan a archivos GeoJSON para su posterior procesamiento de las regiones. El ROI del tumor se muestra con un contorno rojo y el ROI de IM con un contorno verde. Barras de escala = 2 mm. Abreviaturas: ROI = región de interés; IM = margen invasivo; GeoJSON = Notación geográfica de objetos JavaScript. Haga clic aquí para ver una versión más grande de esta figura.
Las redes ImmuNet se pueden utilizar para detectar células inmunitarias. Para el panel de linfocitos, la imagen compuesta experimental (Figura 6A) se puede comparar visualmente con las células inmunitarias detectadas por el software (Figura 6B). Se pueden hacer comparaciones visuales similares para el panel mieloide (Archivo suplementario 6: Figura suplementaria S4) y el panel de células dendríticas (Archivo suplementario 6: Figura suplementaria S5).
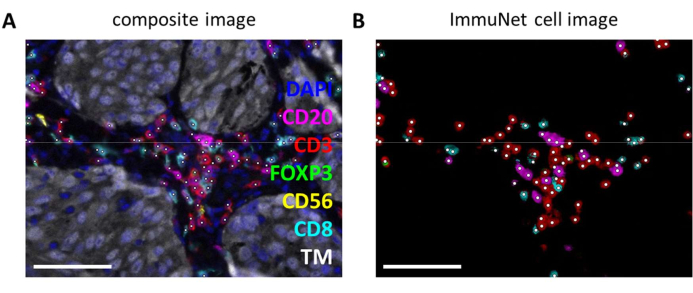
Figura 6: Linfocitos reconocidos por ImmuNet. (A) Imagen compuesta de la Figura 4B que muestra células reconocidas por ImmuNet con puntos blancos. (B) Células reconocidas por ImmuNet y posterior expresión de marcadores detectados. Barras de escala = 50 μm. Abreviatura: TM = marcador tumoral. Haga clic aquí para ver una versión más grande de esta figura.
Las células inmunitarias detectadas por ImmuNet y guardadas en formato .csv pueden importarse a cualquier lenguaje de programación para su posterior análisis. Se realizó la visualización espacial y el compuerta en R (Archivo Suplementario 8). A continuación, las células detectadas se pueden visualizar espacialmente (Figura 7A, Archivo complementario 6: Figura complementaria S6 y Figura complementaria S7). La activación de la expresión de pseudomarcadores se puede realizar para fenotipar las células inmunitarias individuales (Figura 7B).
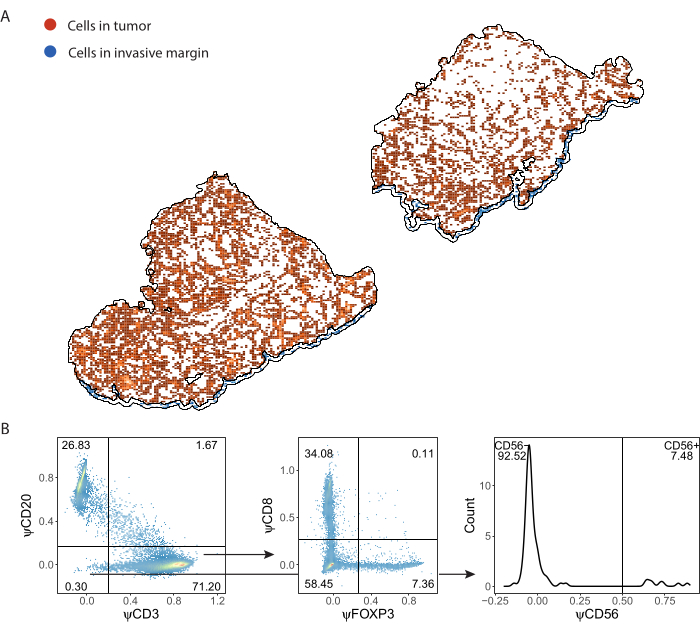
Figura 7: Estrategia de activación del panel de linfocitos. (A) Células inmunitarias detectadas en regiones de interés tumorales y márgenes invasivos delineadas con QuPath. (B) Activación de todas las células detectadas por ImmuNet de la parte A. Los linfocitos se activan primero en los linfocitos B CD20+ y en los linfocitos T CD3+. Los linfocitos T CD3+ están activados para la expresión de CD8 y FOXP3. La población CD20-CD3- está controlada para las células asesinas naturales CD56+. Haga clic aquí para ver una versión más grande de esta figura.
Cuando los fenotipos de las células predichas se determinan con gating, las densidades celulares de diferentes fenotipos se pueden calcular dentro de diferentes ROI. Esto se calcula dividiendo el número total de células por fenotipo por el área de superficie del ROI (Tabla 1, Figura 8 y Archivo Suplementario 8). Aquí, los linfocitos B se definen como CD3-CD20+, los linfocitos T auxiliares como CD3+CD20-CD8-FoxP3-, los linfocitos T reguladores como CD3+CD20-CD8-FoxP3+, los linfocitos T citotóxicos como CD3+CD20-CD8+FoxP3-y los linfocitos NK como CD3-CD20-CD56+.
| Fenotipo | Densidad en el tumor (células/mm2) | Densidad en IM (células/mm2) |
| Célula B | 185.74 | 145.62 |
| Célula T auxiliar | 301.46 | 157.51 |
| Célula T reguladora | 38.53 | 19.53 |
| Células T citotóxicas | 185.35 | 83.21 |
| Célula NK | 0.18 | 0 |
Tabla 1: Densidades de fenotipos en ROIs. Densidades de células de diferentes fenotipos encontradas en una sola muestra de melanoma teñida con el panel de linfocitos. Las densidades se calculan por separado en las ROI tumorales y de IM. Abreviaturas: IM = margen invasivo; ROI = región de interés.
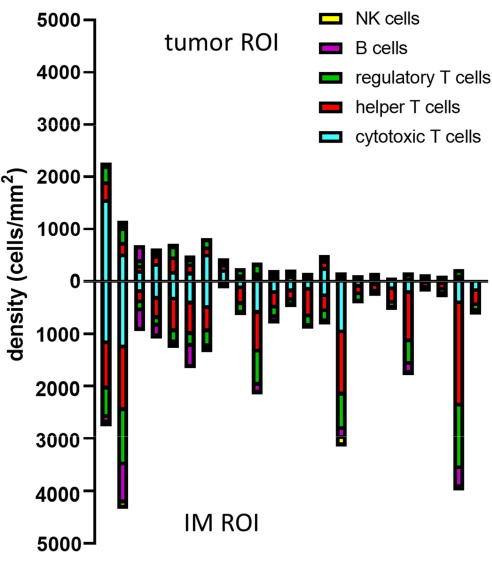
Figura 8: Ejemplo de análisis de datos para varias muestras. Análisis de densidad de diferentes fenotipos de linfocitos en tumores e IM de 23 tumores primarios de melanoma. Abreviaturas: IM = margen invasivo; ROI = región de interés. Haga clic aquí para ver una versión más grande de esta figura.
Para profundizar en la información espacial de estas células inmunitarias, también es posible determinar distancias entre fenotipos identificados o porcentajes de fenotipos de los vecinos más cercanos en una muestra (Figura 9).
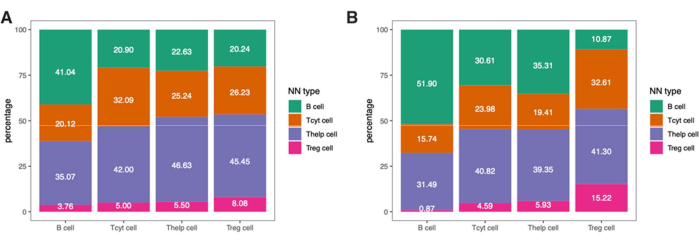
Figura 9: Ejemplo de análisis del vecino más cercano para una sola muestra. Porcentaje de fenotipos del vecino más cercano para diferentes tipos de células en (A) ROI tumoral y (B) IM encontrados en una sola muestra de melanoma teñida con el panel de linfocitos. Abreviaturas: IM = margen invasivo; ROI = región de interés. Haga clic aquí para ver una versión más grande de esta figura.
Archivo complementario 1: Resumen de las especificaciones del protocolo IHC múltiplex. Haga clic aquí para descargar este archivo.
Archivo complementario 2: Protocolo de autotinción para monoplex. Haga clic aquí para descargar este archivo.
Archivo complementario 3: Protocolo de autotinción para la compensación de autofluorescencia. Haga clic aquí para descargar este archivo.
Archivo complementario 4: Protocolo de autotinción para inmunohistoquímica multiplex. Haga clic aquí para descargar este archivo.
Archivo complementario 5: Plantilla .csv archivo. Haga clic aquí para descargar este archivo.
Archivo suplementario 6: Paneles de células mieloides y dendríticas en una muestra de tejido de melanoma; marcaje de portaobjetos en caso de fracaso de la exploración; células mieloides y dendríticas reconocidas por ImmuNet; estrategias de activación de paneles de células mieloides y dendríticas. Haga clic aquí para descargar este archivo.
Archivo complementario 7: Script de puntada de QuPath. Haga clic aquí para descargar este archivo.
Archivo complementario 8: Guión de análisis de datos. Haga clic aquí para descargar este archivo.
Discusión
El análisis espacial de la EMT es una técnica muy solicitada para conocer mejor el compartimento de las células inmunitarias y descubrir nuevos biomarcadores pronósticos y predictivos, especialmente en el campo de la inmunooncología16. Se están desarrollando muchas técnicas diferentes para este propósito, que involucran la detección de proteínas, transcripciones de ARNm o una combinación de ambas, con estimaciones de hasta 100-1.000 objetivos. Sin embargo, una mayor multiplexación se produce a costa de experimentos de menor rendimiento, mayores costos experimentales y desafíos técnicos, y a menudo, solo se puede analizar una pequeña parte del TME. La IHQ multiplex utilizando el método basado en TSA que describimos aquí, detecta seis marcadores diferentes + DAPI simultáneamente, es relativamente menos costoso de realizar y las secciones completas de tejido se obtienen en menos de 20 minutos, listas para ser analizadas por completo. Esta técnica se ha vuelto menos compleja con la automatización del procedimiento de tinción. Las mejoras en el microscopio multiespectral, que incluyen la adición de dos filtros adicionales, han mejorado enormemente los tiempos de desmezcla y escaneo espectral. Es posible detectar hasta ocho marcadores diferentes + DAPI simultáneamente. Sin embargo, al expandir la multiplexación con más marcadores, los beneficios antes mencionados desaparecen a medida que la desmezcla espectral se vuelve más desafiante y los tiempos de escaneo de diapositivas completas aumentan sustancialmente. Se están realizando esfuerzos para normalizar la IHC múltiplex entre diferentes instituciones a fin de facilitar su aplicación en el entorno de diagnóstico. Para esta estandarización de la IHC multiplex, aconsejamos a los usuarios que se adhieran al protocolo más accesible con seis marcadores diferentes + DAPI. Sin embargo, todavía se necesitan bastantes conocimientos técnicos y el análisis posterior puede ser un desafío, para lo cual hemos desarrollado metodologías que se describen en este protocolo.
La estandarización comienza con el desarrollo de paneles IHC multiplex. La importancia de la elección de los anticuerpos primarios que detectan determinadas proteínas diana se ha destacado anteriormente17. Nuestros paneles multiplex de IHQ se desarrollan principalmente con clones de anticuerpos primarios que también se utilizan y validan para IHQ en nuestro departamento de diagnóstico. Sin embargo, en el caso del panel de IHQ múltiplex de células dendríticas, la mayoría de los anticuerpos no se utilizaron en el entorno diagnóstico (van der Hoorn et al., manuscrito en presentación). Para garantizar la especificidad y minimizar las diferencias entre lotes, elegimos usar anticuerpos monoclonales en lugar de anticuerpos policlonales y también hemos validado la mayoría de los anticuerpos utilizando líneas celulares transfectadas y células primarias. A lo largo de los años, se han utilizado diferentes versiones de paneles IHC multiplex en numerosos estudios utilizando el sistema Vectra 3 18,21,23,24,25,26,27,28,29,30,31,32 . Para implementar estos paneles IHC multiplex de manera óptima en el sistema PhenoImager HT, se tuvieron que realizar algunos ajustes en las combinaciones de anticuerpos primarios y fluoróforos. Para beneficiarse de una mejor desmezcla espectral y tiempos de escaneo más rápidos de secciones de tejido completo, es necesaria la implementación de los últimos fluoróforos Opal480 y Opal780 y evitar el uso de fluoróforos Opal540 y Opal650 en paneles IHC multiplex de siete colores. Los tiempos de escaneo son ~ 3-10 veces más rápidos dependiendo del tamaño de la sección de tejido. Los ajustes del panel IHC múltiplex fueron bastante fáciles de lograr, pero se deben tener en cuenta algunas consideraciones. El espectro fluorescente de Opal480 se superpone mucho con el espectro de autofluorescencia y, por lo tanto, interfiere con la desmezcla espectral de eritrocitos y otras estructuras autofluorescentes. El uso de una mayor concentración del anticuerpo primario junto con Opal480 resolvió este problema en la mayoría de los casos. La implementación del filtro Sample AF patentado en el PhenoImager HT facilita la desmezcla de Opal480 y la autofluorescencia. Sin embargo, es mejor usar un anticuerpo primario que produzca una señal clara cuando se usa con Opal480 para que su señal sea más alta que la autofluorescencia.
A pesar de que estos paneles IHC multiplex están establecidos, la variación de lote a lote es algo que debe tenerse en cuenta. Al realizar controles de IHQ monoplex antes de comenzar el experimento de IHQ multiplex completo, a veces observamos que los anticuerpos primarios se comportan más fuertes o más débiles de un experimento a otro. Las razones de esto podrían ser errores de pipeteo, condiciones de almacenamiento de reactivos subóptimas y vida útil. Resolvimos esto ajustando la solución de anticuerpos primarios en función de nuestra experiencia. Incluso cuando no se tuvo que realizar ninguno de los ajustes antes mencionados, con cada experimento de lote de IHC múltiple, es importante establecer los tiempos de exposición en función de los portaobjetos de control teñidos con IHC monoplex.
Debido a que nuestra investigación se centró inicialmente en diferentes tipos de carcinomas y melanoma, se requirió que los paneles IHQ multiplex fueran intercambiables entre los tipos de tumores con ajustes mínimos. Por lo tanto, siempre incluimos múltiples tipos de tejido (tumoral) en el proceso de optimización y observamos que las diluciones de los anticuerpos primarios para los marcadores de células inmunitarias pueden mantenerse similares entre los diferentes tipos de tumores. Sin embargo, la detección de tejido tumoral entre carcinomas y melanoma necesita diferentes marcadores tumorales. En consecuencia, el marcador tumoral siempre se optimizó para funcionar al final de cada panel de IHQ multiplex y actualmente siempre se usa junto con Opal780, que casualmente también tiene que estar en el último fluoróforo en un procedimiento de tinción de IHQ multiplex. Al utilizar el marcador tumoral al final de la IHQ multiplex, estos paneles de IHQ multiplex se pueden intercambiar fácilmente por otros tipos de tumores, como el glioblastoma (es decir, GFAP) y el linfoma de Hodgkin (es decir, CD30). Para el angiosarcoma, utilizamos este panel IHQ multiplex de linfocitos con gen relacionado con la transformación específica del eritroblasto (ERG) como marcador tumoral con solo dos experimentos de optimización25. La optimización incluyó la titulación del anticuerpo primario ERG y la prueba del panel IHQ multiplex con ERG al final.
Otros ajustes a estos paneles multiplex de IHQ también se pueden realizar mediante el intercambio de un determinado marcador de células inmunitarias por otro marcador inmunitario o funcional. Cada cambio requiere optimización. Se podría seguir el protocolo de optimización descrito anteriormente17. Ciertos cambios en los paneles IHC multiplex propuestos interferirán con los algoritmos de ImmuNet que hemos creado. Se deben generar suficientes datos y se debe dedicar tiempo a implementar estos cambios en el algoritmo (al menos 750 anotaciones para cada nuevo marcador y/o fenotipos de células, y 150 anotaciones para la validación de marcadores previamente entrenados). Los paneles aquí presentados no contienen marcadores funcionales, aunque la implementación de marcadores de puntos de control inmune como PD-1 y PD-L1 en paneles IHQ multiplex se realiza en nuestro laboratorio. Sin embargo, el análisis de marcadores que son menos binarios en señales negativas y positivas ha demostrado ser más difícil y es un área de investigación activa en nuestro grupo.
El número de marcadores que se pueden evaluar simultáneamente con IHQ multiplex es limitado en comparación con otras técnicas novedosas. Si bien esto se puede eludir analizando diferentes paneles en porciones consecutivas de un bloque FFPE, será difícil comparar estas rebanadas espacialmente. Es probable que la orientación y los artefactos plegados no sean los mismos después de la preparación de la diapositiva. Sin embargo, la IHC múltiple es bastante accesible, lo que la convierte en una herramienta atractiva para más instituciones e investigadores y, por lo tanto, más adecuada para su futura implementación en un entorno de diagnóstico. Con la estandarización de los paneles multiplexados de células inmunitarias IHQ para múltiples tipos de tumores y los canales de análisis posteriores, se podría obtener más conocimiento sobre las diferencias en la EMT entre los pacientes y los tipos de tumores. Esto puede, por ejemplo, conducir a más conocimientos sobre el papel de la TME en la respuesta antitumoral a tratamientos específicos. Esto puede incluso dar lugar a nuevos biomarcadores para predecir factores como la respuesta al tratamiento y la supervivencia esperada. En general, esto puede permitir que la IHQ multiplex se convierta en una herramienta clínica para ayudar en la toma de decisiones clínicas, en un enfoque de medicina personalizada. Es cierto que probablemente se deberían automatizar y estandarizar más pasos del procedimiento de análisis para que sea factible su uso en un entorno de diagnóstico diario, por lo que, hasta ahora, es principalmente una perspectiva futurista.
El análisis de múltiples marcadores en un solo portaobjetos de muestra puede ser una herramienta muy poderosa a pesar de sus desafíos técnicos. Con protocolos experimentales estandarizados y un método de análisis robusto, como describimos aquí utilizando ImmuNet, la cuantificación de múltiples marcadores la hace más informativa que la IHQ clásica, mientras que la IHC múltiple sigue teniendo un rendimiento relativamente alto en comparación con los nuevos métodos experimentales de plex superior.
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
El PhenoImager HT fue adquirido a través de fondos proporcionados por el Centro Médico de la Universidad de Radboud y el Centro de Tecnología de Microscopía de Radboud. La FQ cuenta con el apoyo financiero de una subvención de la Sociedad Holandesa contra el Cáncer (10673) y una subvención ARTimmune (834618) del Consejo Europeo de Defensa. JT cuenta con el apoyo financiero de una subvención de NMO VIDI (VI.Vidi.192.084). Los autores desean agradecer a Eric van Dinther y Ankur Ankan por su ayuda en la creación de flujos de trabajo para almacenar datos IHC multiplex y se agradece a Bengt Phung por las instrucciones sobre cómo implementar datos IHC multiplex en QuPath para el dibujo de ROI.
Materiales
| Name | Company | Catalog Number | Comments |
| anti-CD14 | Cell Marque | 114R-16 | section 3, clone EPR3653 |
| anti-CD163 | Cell Marque | 163M-15 | section 3, clone MRQ-26 |
| anti-CD19 | Abcam | ab134114 | section 3, clone EPR5906 |
| anti-CD1c (BDCA1) | Thermo Scientific | TA505411 | section 3, clone OTI2F4 |
| anti-CD20 | Thermo Scientific | MS-340-S | section 3, clone L26 |
| anti-CD3 | Thermo Scientific | RM-9107 | section 3, clone sp7 |
| anti-CD303/BDCA2 | Dendritics via Enzo Lifesciences/Axxora | DDX0043 | section 3, clone 124B3.13 |
| anti-CD56 | Cell Marque | 156R-94 | section 3, clone MRQ-42 |
| anti-CD66b | BD Biosciences | 555723 | section 3, clone G10F5 |
| anti-CD68 | Dako Agilent | M087601 | section 3, clone PG-M1 |
| anti-CD8 | Dako Agilent | M7103 | section 3, clone C8/144B |
| anti-Foxp3 | Thermo Scientific | 14-4777 | section 3, clone 236A/E7 |
| anti-Gp100 | Dako Agilent | M063401 | section 3, clone HMB45 |
| anti-HLA-DR, DP, DQ | Santa Cruz | sc-53302 | section 3, clone CR3/43 |
| anti-MART-1 | Thermo Scientific | MS-799 | section 3, clone A103 |
| anti-pan cytokeratin | Abcam | ab86734 | section 3, clone AE1/AE3 + 5D3 |
| anti-SOX10 | Sigma Aldrich | 383R | section 3, clone EP268 |
| anti-Tyrosinase | Sanbio | MONX10591 | section 3, clone T311 |
| anti-XCR1 | Cell Signaling Technologies via Bioké | 44665S | section 3, clone D2F8T |
| antibody diluent | Akoya BioSciences | SKU ARD1001EA | section 3, from Opal 7-Color Automation IHC Kit 50 slide (can optionally also be replaced by TBST with 10% BSA) |
| Bond Aspirating Probe | Leica Biosciences | S21.0605 | section 3 |
| Bond Aspirating Probe Cleaning | Leica Biosciences | CS9100 | section 3 |
| Bond Dewax Solution | Leica Biosciences | AR9222 | section 3 |
| Bond Objectglas label + print lint | Leica Biosciences | S21.4564.A | section 3 |
| Bond Research Detection System 2 | Leica Biosciences | DS9777 | section 3 |
| Bond RX autostainer | Leica Biosciences | - | section 3, automated platform |
| Bond TM Epitope Retrieval 1 - 1 L | Leica Biosciences | AR9961 | section 3 |
| Bond TM Epitope Retrieval 2 - 1 L | Leica Biosciences | AR9640 | section 3 |
| Bond TM Wash Solution 10x - 1 L | Leica Biosciences | AR9590 | section 3 |
| BOND Universal Covertile | Leica Biosciences | S21.4611 | section 3 |
| Bond(TM) Titration Kit | Leica Biosciences | OPT9049 | section 3 |
| Coverslip 24 x 32 mm #1 (0.13-0.16 mm) | Fisher Scientific | 15717592 | section 2 |
| coverslip 24 x 50 mm | VWR | 631-0146 | section 2 |
| DAPI Fluoromount-G | VWR | 0100-20 | section 3, whenever monoplex slides need to be quickly checked, not for official analysis, then DAPI is stained seperately for better results |
| Eosine | section 2, home made | ||
| Ethanol 99.5% | VWR | 4099.9005 | section 2 |
| Fluoromount-G | VWR | 0100-01 | section 3 |
| haematoxyline | - | section 2, home made | |
| ImmuNet | - | immune cell detection and phenotyping pipeline | |
| inForm software 2.4.10 | Akoya BioSciences | - | section 4 & 6 |
| OPAL 480 reagent pack | Akoya BioSciences | FP1500001KT | section 3 |
| OPAL 520 reagent pack | Akoya BioSciences | FP1487001KT | section 3 |
| OPAL 570 reagent pack | Akoya BioSciences | FP1488001KT | section 3 |
| OPAL 620 reagent pack | Akoya BioSciences | FP1495001KT | section 3 |
| OPAL 690 reagent pack | Akoya BioSciences | FP1497001KT | section 3 |
| OPAL 780 reagent pack | Akoya BioSciences | FP1501001KT | section 3 |
| Opal 7-Color Automation IHC Kit 50 slide | Akoya BioSciences | NEL821001KT | section 3 |
| PhenoChart 1.1.0 | Akoya BioSciences | - | section 5 |
| PhenoImagerHT | Akoya BioSciences | CLS143455 | section 4, digital pathology imager with slide viewer and imaging software (formerly known as Vectra Polaris) |
| Quick-D mounting medium | Klinipath | 7280 | section 2 |
| QuPath 0.3.2 | whole slide image analysis software platform | ||
| R 4.1.1 | |||
| Slide boxes | VWR | 631-0737 | section 1 |
| SuperFrost Plus | Thermo Scientific through VWR | 631-9483 | section 1 |
| Vectra Polaris software 1.0.13 | Akoya BioSciences | - | section 4 |
| Xylene | VWR | 4055-9005 | section 2 |
Referencias
- Dunn, G. P., Bruce, A. T., Ikeda, H., Old, L. J., Schreiber, R. D. Cancer immunoediting: from immunosurveillance to tumor escape. Nature Immunology. 3 (11), 991-998 (2002).
- van der Woude, L. L., Gorris, M. A. J., Halilovic, A., Figdor, C. G., de Vries, I. J. M. Migrating into the tumor: a roadmap for T cells. Trends in Cancer. 3 (11), 797-808 (2017).
- Fridman, W. H., Pages, F., Sautes-Fridman, C., Galon, J. The immune contexture in human tumours: impact on clinical outcome. Nature Reviews. Cancer. 12 (4), 298-306 (2012).
- Fridman, W. H., et al. The immune microenvironment of human tumors: general significance and clinical impact. Cancer Microenvironment. 6 (2), 117-122 (2013).
- Pages, F., et al. International validation of the consensus Immunoscore for the classification of colon cancer: a prognostic and accuracy study. Lancet. 391 (10135), 2128-2139 (2018).
- Angell, H. K., Bruni, D., Barrett, J. C., Herbst, R., Galon, J. The Immunoscore: colon cancer and beyond. Clinical Cancer Research. 26 (2), 332-339 (2020).
- Angell, H., Galon, J. From the immune contexture to the Immunoscore: the role of prognostic and predictive immune markers in cancer. Current Opinion in Immunology. 25 (2), 261-267 (2013).
- Galon, J., et al. Cancer classification using the Immunoscore: a worldwide task force. Journal of Translational Medicine. 10, 205 (2012).
- Galon, J., et al. World-wide Immunoscore Task Force: meeting report from the 34;Melanoma Bridge", Napoli, November 30th-December 3rd, 2016. Journal of Translational Medicine. 15 (1), 212 (2017).
- Tumeh, P. C., et al. PD-1 blockade induces responses by inhibiting adaptive immune resistance. Nature. 515 (7528), 568-571 (2014).
- Creemers, J. H. A., et al. A tipping point in cancer-immune dynamics leads to divergent immunotherapy responses and hampers biomarker discovery. Journal for Immunotherapy of Cancer. 9 (5), e002032 (2021).
- Blank, C. U., Haanen, J. B., Ribas, A., Schumacher, T. N. CANCER IMMUNOLOGY. The "cancer immunogram". Science. 352 (6286), 658-660 (2016).
- Teruya-Feldstein, J. The immunohistochemistry laboratory looking at molecules and preparing for tomorrow. Archives of Pathology & Laboratory Medicine. 134 (11), 1659-1665 (2010).
- Ramos-Vara, J. A., Miller, M. A., et al. When tissue antigens and antibodies get along: revisiting the technical aspects of immunohistochemistry--the red, brown, and blue technique. Veterinary Pathology. 51 (1), 42-87 (2014).
- Hegde, P. S., Karanikas, V., Evers, S. The where, the when, and the how of immune monitoring for cancer immunotherapies in the era of checkpoint inhibition. Clinical Cancer Research. 22 (8), 1865-1874 (2016).
- Parra, E. Novel platforms of multiplexed immunofluorescence for study of paraffin tumor tissues. Journal of Cancer Treatment and Diagnosis. 2 (1), 43-53 (2018).
- Gorris, M. A. J., et al. Eight-color multiplex immunohistochemistry for simultaneous detection of multiple immune checkpoint molecules within the tumor microenvironment. Journal of Immunology. 200 (1), 347-354 (2018).
- Roelofsen, T., et al. Spontaneous regression of ovarian carcinoma after septic peritonitis; a unique case report. Frontiers in Oncology. 8, 562 (2018).
- van den Brand, D., et al. Peptide-mediated delivery of therapeutic mRNA in ovarian cancer. European Journal of Pharmaceutics and Biopharmaceutics. 141, 180-190 (2019).
- van den Brand, D., et al. EpCAM-binding DARPins for targeted photodynamic therapy of ovarian cancer. Cancers. 12 (7), 1762 (2020).
- Di Blasio, S., et al. The tumour microenvironment shapes dendritic cell plasticity in a human organotypic melanoma culture. Nature Communications. 11 (1), 2749 (2020).
- van Beek, J. J. P., et al. Human pDCs are superior to cDC2s in attracting cytolytic lymphocytes in melanoma patients receiving DC vaccination. Cell Reports. 30 (4), 1027-1038 (2020).
- Rodriguez-Rosales, Y. A., et al. Immunomodulatory aged neutrophils are augmented in blood and skin of psoriasis patients. Journal of Allergy and Clinical Immunology. 148 (4), 1030-1040 (2021).
- Hoeijmakers, Y. M., et al. Immune cell composition in the endometrium of patients with a complete molar pregnancy: Effects on outcome. Gynecologic Oncology. 160 (2), 450-456 (2021).
- van Ravensteijn, S. G., et al. Immunological and genomic analysis reveals clinically relevant distinctions between angiosarcoma subgroups. Cancers. 14 (23), 5938 (2022).
- van der Woude, L. L., et al. Tumor microenvironment shows an immunological abscopal effect in patients with NSCLC treated with pembrolizumab-radiotherapy combination. Journal for Immunotherapy of Cancer. 10 (10), e005248 (2022).
- Graham Martinez, C., et al. The immune microenvironment landscape shows treatment-specific differences in rectal cancer patients. Frontiers in Immunology. 13, 1011498 (2022).
- Cortenbach, K. R. G., et al. Topography of immune cell infiltration in different stages of coronary atherosclerosis revealed by multiplex immunohistochemistry. International Journal of Cardiology. Heart & Vasculature. 44, 101111 (2023).
- van Wilpe, S., et al. Homologous recombination repair deficient prostate cancer represents an immunologically distinct subtype. Oncoimmunology. 11 (1), 2094133 (2022).
- Gorris, M. A. J., et al. Paired primary and metastatic lesions of patients with ipilimumab-treated melanoma: high variation in lymphocyte infiltration and HLA-ABC expression whereas tumor mutational load is similar and correlates with clinical outcome. Journal for Immunotherapy of Cancer. 10 (5), e004329 (2022).
- van Wilpe, S., et al. Intratumoral T cell depletion following neoadjuvant chemotherapy in patients with muscle-invasive bladder cancer is associated with poor clinical outcome. Cancer Immunology, Immunotherapy. 72 (1), 137-149 (2023).
- Sultan, S., Gorris, M. A. J., Buytenhuijs, F., Lvan de Woude, L. A Segmentation-free machine learning architecture for immune land-scape phenotyping in solid tumors by multichannel imaging. bioRxiv. , (2021).
- Parra, E. R., et al. Immuno-profiling and cellular spatial analysis using five immune oncology multiplex immunofluorescence panels for paraffin tumor tissue. Scientific Reports. 11 (1), 8511 (2021).
- Parra, E. R., et al. Validation of multiplex immunofluorescence panels using multispectral microscopy for immune-profiling of formalin-fixed and paraffin-embedded human tumor tissues. Scientific Reports. 7 (1), 13380 (2017).
- Sun, Z., Nyberg, R., Wu, Y., Bernard, B., Redmond, W. L. Developing an enhanced 7-color multiplex IHC protocol to dissect immune infiltration in human cancers. PLoS One. 16 (2), e0247238 (2021).
- LeicaBiosystems. BOND RX Fully Automated Research Stainer Protocols. , (2022).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados